Method Article
Purification d’affinité d’une protéine marquée 6X-His à l’aide d’un système de chromatographie liquide à protéines rapides
Dans cet article
Résumé
Cet article fournit une procédure pour la purification d’affinité d’une protéine recombinante humaine, l’endonucléase de lambeau 1 (FEN1), qui a été marquée avec un marqueur 6X-histidine. Le protocole implique l’utilisation de deux colonnes d’ions métalliques immobilisées distinctes pour la purification de la protéine marquée.
Résumé
La caractérisation fonctionnelle des protéines nécessite qu’elles soient exprimées et purifiées en quantités substantielles avec une grande pureté pour effectuer des analyses biochimiques. Le système de chromatographie liquide rapide des protéines (FPLC) permet la séparation à haute résolution de mélanges de protéines complexes. En ajustant divers paramètres dans FPLC, tels que la sélection de la matrice de purification appropriée, la régulation de la température de l’échantillon de protéine et la gestion du débit de l’échantillon sur la matrice et du taux d’élution, il est possible d’assurer la stabilité et la fonctionnalité de la protéine. Dans ce protocole, nous démontrerons la polyvalence du système FPLC pour purifier la protéine FEN1 (6X-His-tagged flap endonucléase 1), produite dans des cultures bactériennes. Pour améliorer l’efficacité de la purification des protéines, nous nous concentrerons sur de multiples considérations, notamment l’emballage et la préparation appropriés de la colonne, l’injection d’échantillons à l’aide d’une boucle d’échantillon, le débit d’application de l’échantillon dans la colonne et les paramètres d’élution de l’échantillon. Enfin, le chromatogramme sera analysé afin d’identifier les fractions contenant des rendements élevés de protéines et les considérations pour un stockage approprié à long terme des protéines recombinantes. L’optimisation des méthodes de purification des protéines est cruciale pour améliorer la précision et la fiabilité de l’analyse des protéines.
Introduction
De nombreuses stratégies sont disponibles pour comprendre la biologie cellulaire. Une approche implique une stratégie descendante, dans laquelle des mutations génétiques sont introduites dans un gène, suivies de l’évaluation des changements phénotypiques qui en résultent dans un organisme modèle. À l’inverse, une approche réductionniste implique l’élucidation initiale des mécanismes moléculaires et des fonctions enzymatiques d’une protéine particulière, accompagnée de la caractérisation de ses interactions avec d’autres composants cellulaires. Par la suite, l’impact de cette protéine sur une voie biologique est évalué. Bien que chaque approche de recherche possède ses avantages et ses limites inhérents, la compréhension complète d’une voie biologique nécessite des investigations interdisciplinaires.
L’ADN étant le modèle génétique de la vie, la compréhension des mécanismes de duplication de l’ADN et de maintenance du génome est un domaine d’intérêt actif depuis plus de sept décennies. Des études dans le domaine de la réplication de l’ADN ont fourni de nombreuses données concernant les structures et les fonctions individuelles de nombreuses protéines de réplication. Ces recherches, qui englobent les aspects mécanistes et les dosages d’activité biochimique, ont été rendues possibles grâce à la purification de ces protéines, ce qui a permis leur examen méticuleux dans un environnement in vitro . Par conséquent, la purification des protéines apparaît comme une technique indispensable et omniprésente dans la majorité des efforts de recherche visant à démêler les connaissances mécanistes de la réplication de l’ADN.
Cet article présente une méthodologie permettant d’isoler une protéine de réplication de l’ADN marquée avec de la 6X-histidine, qui a été surexprimée dans les cellules bactériennes. La protéine d’intérêt est l’endonucléase de lambeau humain 1 (FEN1), une nucléase spécifique à la structure qui joue un rôle central dans la réplication des brins retardés et qui est également un participant essentiel dans les voies de réparation de l’ADN comme la réparation par excision de base (BER)1,2,3. La fonction principale de FEN1 est de cliver à la base d’une structure de lambeau déplacée de 5', un intermédiaire qui se produit lors de la réplication de l’ADN ou BER. Initialement, des études biochimiques évaluant l’activité enzymatique de FEN1 ont suggéré un mécanisme de « suivi », dans lequel la nucléase reconnaîtrait l’extrémité libre 5'-phosphate d’une structure de lambeau, puis suivrait le lambeau le long de sa base avant de le fendre4. Des recherches ultérieures ont révélé que FEN1 fonctionne via un mécanisme de « filetage », dans lequel il se lie d’abord à la base du rabat, puis enfile l’extrémité libre de 5' à travers son site actif avant le clivage (Figure 1)5. La capacité de surexprimer et d’isoler FEN1 recombinant a facilité ces percées, permettant aux chercheurs de l’utiliser dans des études biochimiques et structurales.
La chromatographie d’affinité est une méthode de séparation couramment utilisée pour purifier l’ADN. Cette technique utilise l’affinité de liaison réversible des protéines cibles envers des ligands immobilisés sur une résine pour piéger spécifiquement la protéine d’intérêt. L’une des bio-affinités les plus largement utilisées est l’interaction robuste entre l’acide aminé, l’histidine et les ions métalliques tels que le nickel et le cobalt et, par conséquent, peut être capturée sur une résine chargée de Ni2+ ou de Co2+.
La séquence d’ADN codant pour une chaîne de 6 à 9 résidus d’histidine (His) est fréquemment incorporée dans la construction plasmidique qui code pour la protéine d’intérêt (soit à l’extrémité N-terminale, soit à l’extrémité C), en marquant la protéine avec une étiquette 6X-His-Tag ou une étiquette poly-His. La protéine marquée par His peut ensuite être facilement purifiée par chromatographie d’affinité métallique immobilisée (IMAC), un sous-type de chromatographie d’affinité où les ions métalliques sur la résine capturent des protéines avec un marqueur d’affinité, qui peuvent ensuite être éluées à l’aide d’agents d’élution appropriés. Les ions de métaux de transition tels que le Ni2+ et le Co2+ peuvent être immobilisés sur des matrices d’agarose ou de gel de silice dérivées des groupes6 de la N,N,N'-tris-(carboxyméthyl)-éthylènediamine ou de l’acide nitrilotriacétique (NTA).
Les ligands métalliques sont connus pour être robustes contre la dégradation par des facteurs physiques, chimiques et biologiques et présentent cet avantage sur les autres types de ligands 6,7. De plus, le His-tag est un marqueur relativement petit et n’a pas d’impact significatif sur la structure ou la fonction des protéines7. Cependant, dans un système d’expression bactérien, de nombreuses protéines exprimées chromosomiquement ont une affinité pour les ions métalliques et peuvent co-se purifier avec la protéine cible. Le nickel et le cobalt sont les ions métalliques typiques utilisés dans les matrices IMAC. La résine Ni-NTA et la résine à base de cobalt TALON sont couramment utilisées pour la purification des protéines marquées His.
Ni-NTA contre TALON
Les ions métalliques respectifs du Ni-NTA et du TALON sont immobilisés sur la résine par des ligands NTA. On pense que le Ni-NTA a une capacité de liaison plus élevée, se liant jusqu’à 100 mg/mL de protéines. Cela peut entraîner un rendement en protéines plus élevé, avec la mise en garde que les protéines contaminantes peuvent être co-purifiées. En revanche, la résine a une spécificité de liaison plus élevée envers les protéines marquées par His et peut être capable de produire des fractions de plus grande pureté. Dans cette étude, nous visons à comparer l’efficacité de purification des deux résines à l’aide du système automatisé de chromatographie liquide à protéines rapides référencé, le système NGC (voir le tableau des matériaux).
Tampons et compatibilité
Des tampons sont nécessaires lors de la purification des protéines pour la lyse cellulaire, la préparation des échantillons, l’équilibrage de la résine et l’élution de la protéine capturée de la résine. Les tampons Tris, MOPS et HEPES jusqu’à une concentration de 100 mM sont les tampons compatibles connus pour la résine Ni-NTA. Les tampons comprennent souvent des agents réducteurs pour empêcher l’oxydation des protéines et l’agrégation des protéines. Cependant, au-delà d’une limite seuil, les agents réducteurs pourraient débarrasser la résine des ions métalliques. La concentration recommandée d’agents réducteurs tels que le bêta-mercaptoéthanol (BME) ou le dithiothréitol (DTT) est inférieure à 1 mM pour les résines à base de nickel et de cobalt mentionnées ci-dessus.
Les tampons pour la purification de hFEN1 sont des tampons Tris contenant du NaCl, du BME, du fluorure de phénylméthylsulfonyle (PMSF), de l’EDTA et du glycérol. Le NaCl maintient la protéine sous forme soluble et perturbe les interactions moléculaires telles que la liaison à l’ADN. Le BME réduit les protéines oxydées et empêche ainsi l’agrégation des protéines. PMSF) est un inhibiteur de la protéase qui empêche la dégradation de la protéine cible par la protéase. L’EDTA élimine les cations divalents de l’échantillon, empêchant leur accès aux nucléases et aux protéases. Le glycérol améliore la stabilité de la protéine sous forme aqueuse. De plus, le tampon de lyse contient des comprimés complets d’inhibiteur de protéase pour assurer une protection maximale de la protéine cible contre la dégradation des protéases pendant la lyse cellulaire. Les tampons d’équilibrage et d’élution contiennent de l’imidazole, le tampon d’élution contenant des quantités plus élevées pour que l’imidazole déplace la protéine liée de la résine pendant l’élution.
Système de chromatographie de nouvelle génération (NGC)
Ce système automatisé de chromatographie à moyenne pression conçu pour la chromatographie liquide protéique à débit rapide (FPLC) utilise deux pompes pour pomper simultanément deux tampons différents et est capable d’injecter une large gamme de volumes d’échantillons allant de 250 μL à 100 ml. La boucle d’échantillonnage (appelée Dynaloop dans ce système), permet d’injecter des volumes d’échantillons plus importants. Le système peut être utilisé à l’aide du logiciel Chromlab, ce qui facilite la création de méthodes personnalisées, la manipulation des cycles de purification et l’analyse des pics UV et des fractions de protéines.
Protocole
1. Préparation de l’échantillon
- Pour purifier FEN1 recombinant, exprimez la construction (pET-FCH-FEN1) dans les cellules BL21(DE3) comme décrit précédemment par Ononye et al.8.
- Inocuculer le milieu LB (tableau 1) avec une culture de nuit à 1 % et faire croître les cellules à 37 °C jusqu’à ce que la DO atteigne 0,6.
- Induire avec 0,4 M d’isopropyl-bêta-D-thiogalactoside (IPTG) et faire pousser la culture pendant 3 h supplémentaires.
- Récoltez les cellules en centrifugeant la culture à 5 000 × g pendant 15 min à 4 °C. Conservez les granulés dans un congélateur à -80°C.
- Le jour de la purification, retirer la pastille cellulaire du congélateur à -80 °C, la laisser décongeler sur de la glace et remettre la pastille en suspension dans 100 mL de tampon de lyse (tableau 1).
REMARQUE : Le lysat cellulaire doit toujours être placé sur de la glace pendant la manipulation pour éviter la protéolyse. Une fois décongelé, l’ensemble du processus de purification doit être terminé le même jour. Le lysat cellulaire ne doit pas être conservé au réfrigérateur pendant la nuit.
- Transférez la suspension dans un bécher de 250 mL et sonicate sur de la glace avec 30 s ON et 30 s OFF à 35 % d’amplitude pendant 15 min.
REMARQUE : Consultez les paramètres du fabricant pour identifier l’amplitude. - Transférez l’échantillon soniqué dans un tube à centrifuger et faites-le tourner à 27 000 × g pendant 30 min à 4 °C pour obtenir un lysat clair. Placez le lysat cellulaire sur de la glace dans le réfrigérateur à 4 °C jusqu’à l’injection de l’échantillon.
2. Préparation des colonnes d’affinité
- Montez les colonnes (une pour la résine Ni-NTA et une pour la résine à base de cobalt) sur un support de serrage de sorte que les colonnes soient maintenues aussi droites que possible, en évitant toute inclinaison des colonnes. Assurez-vous que le bas des colonnes est bouché à l’aide d’un bouchon et ajoutez 1 à 2 ml de tampon de départ au bas de chaque colonne pour éviter la formation de bulles d’air pendant l’emballage des colonnes.
- Secouez doucement les flacons de résine jusqu’à ce que la résine soit complètement remise en suspension. Ouvrez le haut des colonnes et versez 10 ml de chaque boue de manière continue pour éviter l’inclusion de bulles d’air. Versez la résine contre une spatule métallique ou une tige de verre maintenue au bord de la colonne pour éviter les bulles d’air dans la colonne.
3. Garnissage de colonne à l’aide d’un adaptateur de débit
- Débranchez le bouchon de la colonne et connectez le bas à une extrémité du tube. Connectez l’autre extrémité du tube au port UV du système FPLC.
- Assurez-vous que le boîtier du support de lit a été inséré dans le corps de l’adaptateur de débit et faites glisser le corps de l’adaptateur de débit sur le dessus de la colonne (Figure 2A). Avec le loquet à came en position 1, abaissez l’adaptateur de débit en prenant soin de ne pas l’insérer dans la mémoire tampon (Figure 2B). Mettez le loquet de la came en position 2 (Figure 2B). Connectez le tube au système et démarrez le débit du tampon de démarrage pendant 1 à 2 minutes pour débarrasser le tube de l’air et éviter les bulles d’air pendant l’emballage.
- Remettez le loquet en position 1 et abaissez l’adaptateur de débit dans le tampon d’un seul mouvement fluide, en évitant l’inclusion de bulles d’air, puis remettez le loquet en position 2. Cela permet à une partie de la mémoire tampon d’entrer dans l’adaptateur de flux et de supprimer toutes les bulles restantes.
- Mettez le loquet en position 3 et serrez la base de came et la bague de verrouillage (Figure 2B).
- Démarrer le flux de tampon à 10 mL/min pour tasser la colonne. Continuez cette étape pendant 3 minutes ou jusqu’à ce que la résine soit bien tassée.
4. Amorçage de la boucle d’échantillonnage du système FPLC
- Connectez le tube du haut de la boucle d’échantillonnage à la boucle E du système FPLC (Figure 3).
- Connectez le tube du bas de la boucle d’échantillonnage à Waste 2 sur le système FPLC.
- Insérez une extrémité du tube dans l’orifice de la colonne du système FPLC et placez l’autre extrémité dans la bouteille de déchets.
- Insérez le tube de la pompe A dans de l’eau double distillée filtrée (ddH2O).
- Double-cliquez sur les paramètres de la pompe à l’aide du logiciel (Figure 4A, étiqueté 2) et sélectionnez la boucle d’injection de la pompe du système (Figure 4C).
- Cliquez sur les paramètres de débit (Figure 4A, étiqueté 1) et réglez le débit à 10 mL/min. Entrez 0 % dans la zone %B (Figure 4B).
- Démarrez l’écoulement pour rincer la boucle d’échantillonnage avec de l’eau, ce qui poussera le joint coulissant vers le bas.
- Une fois que le joint coulissant atteint le bas de la boucle d’échantillonnage, arrêtez l’écoulement. Basculez la pompe A de ddH2O au tampon de démarrage. Connectez le tube du haut de la boucle d’échantillonnage au déchet 2 et celui du bas à la boucle E. Laissez le tube de l’orifice de la colonne dans le déchet.
- Démarrer le débit à 10 mL/min. Une fois que le joint coulissant poussé vers le haut par le tampon de démarrage entrant atteint le haut de la boucle d’échantillonnage, arrêtez l’écoulement. La boucle d’échantillonnage est maintenant prête pour l’injection d’échantillon.
5. Injection d’échantillon
- Double-cliquez sur les paramètres de la pompe et sélectionnez Boucle de chargement manuelle (Figure 4C).
- Dévissez le tube de la boucle E et placez-le dans le conteneur à déchets.
- Connectez le tube de l’orifice de la colonne au tube situé sur le dessus de l’adaptateur de débit.
- Prélever l’échantillon à l’aide d’une seringue de 30 ml ; Ensuite, fixez la seringue à l’adaptateur d’orifice d’injection.
- Vissez la seringue dans l’orifice d’injection et injectez l’échantillon en vous assurant qu’il est injecté de manière visible dans la boucle d’échantillon. Stocker 100 μL de l’échantillon ( lysat d’entrée d’étiquette) dans un tube de microcentrifugation à -20 °C pour l’analyse SDS-PAGE
REMARQUE : Selon le volume total de l’échantillon, 2-3 injections peuvent être nécessaires. - Bouchez l’orifice d’injection avec le capuchon de colonne.
- Reconnectez le tube du bas de la boucle d’échantillonnage dans le port Loop E.
- Placez le tube de la pompe B dans le tampon d’élution.
6. Création et analyse de la méthode à l’aide du logiciel lié au système FPLC
- Accédez à l’onglet Accueil et sélectionnez Nouvelle méthode.
- Sélectionnez l’onglet Paramètres de méthode dans la colonne de gauche, accédez au menu déroulant Type de colonne , puis sélectionnez Personnalisé (Figure 5).
- Entrez le volume de la colonne (5 ml).
- Entrez le débit requis pour le cycle (1 mL/min).
- Accédez à Sélection d’unité et sélectionnez CV (volumes de colonnes) comme unité de base de méthode.
- Assurez-vous que l’entrée A est définie sur la mémoire tampon A et que l’entrée B est définie sur la mémoire tampon B (paramètre par défaut).
- Sélectionnez l’onglet Plan de la méthode dans le menu de gauche. Ajoutez les étapes de la purification dans l’ordre suivant : équilibre, application de l’échantillon, lavage de la colonne et élution.
- Sous Equilibration, réglez le volume d’équilibrage sur 10 CV. Assurez-vous que le paramètre de mémoire tampon est maintenu à 0 % B.
- Sous l’onglet Exemple d’application , définissez le volume d’échantillon en observant la boucle d’échantillonnage. La position du joint coulissant indique le volume de l’échantillon.
- Sous le lavage en colonne, réglez le volume de lavage sur 5 CV. Désactiver la collecte de fractions pour cette étape ; Recueillir le lavage entier dans un bécher pendant la course, plutôt que de le recueillir sous forme de fractions.
- Pour l’élution, réglez le volume de gradient sur 14 CV en commençant par 0 % de tampon B à 100 % de tampon B.
- Enregistrez la méthode et lancez l’exécution.
- Assurez-vous que le collecteur de fractions commence à la position de la première fraction.
- Entrez le nom de l’exécution et cliquez sur Démarrer.
- Pendant la phase d’équilibrage, surveillez la courbe UV en temps réel. Assurez-vous que toutes les connexions sont sécurisées et qu’il n’y a pas de fuite ; Confirmez un circuit complet en recherchant le tampon qui s’écoule dans le conteneur à déchets. Si la courbe UV ne s’est pas stabilisée pendant les dernières minutes d’équilibrage, prolongez l’étape d’équilibrage jusqu’à ce que la stabilisation soit obtenue (Figure 6) .
- Au moment de l’application de l’échantillon, placez les tubes dans le conteneur à déchets par un bécher propre. Au cours de cette étape, récupérez les protéines non liées qui s’écoulent des tubes de déchets sous forme de flux (étiquetez comme FT).
- Recherchez une forte augmentation de la courbe UV qui atteint probablement la lecture maximale possible de 3 000 maU (Figure 6).
REMARQUE : Une courbe typique se stabilise à 3 000 maU pendant la majeure partie de l’étape d’application de l’échantillon. - Pendant le lavage de la colonne, alors que les protéines résiduelles non liées sont lavées et que seules les protéines liées restent sur la résine, recherchez une chute brutale de la courbe UV (Figure 6). Si la courbe UV n’a pas baissé à la fin de l’étape de lavage, prolongez l’étape en cliquant sur le bouton de maintien jusqu’à ce que l’UV ait suffisamment baissé. Au début de cette étape, transférez les tubes de déchets du bécher FT vers un autre bécher propre étiqueté Wash.
- Lorsque l’étape d’élution commence, assurez-vous que le collecteur de fractions se met en position et commencez à collecter les fractions. Visualisez l’élution de la protéine en recherchant des pics dans la courbe UV (Figure 6).
- Une fois l’exécution terminée, accédez à l’onglet Accueil et sélectionnez Ouvrir l’exécution. Ouvrez le cycle pour afficher le chromatogramme et sélectionnez l’intégration des pics dans le menu supérieur pour marquer les pics obtenus pendant le cycle.
- Sélectionnez l’onglet Fractions pour visualiser les fractions contenant la protéine éluée.
7. Analyse de la page FDS
- Mélanger 15 μL de chaque fraction contenant des protéines avec 5 μL de tampon de chargement d’échantillon SDS (tableau 1). Chauffer les échantillons à 95 °C pendant 5 min.
- Chargez les échantillons dans des gels prémoulés et faites-les fonctionner à 160 V pendant 45 minutes avec un marqueur protéique précoloré.
- Teindre les gels dans la solution de coloration bleu brillant Coomassie pendant 30 min (tableau 1).
- Lavez le gel dans ddH2O (répétez 3x).
- Décolorer le gel dans la solution de décoloration pendant 1 h (tableau 1).
- Observer les bandes protéiques ; détecter FEN1 à la marque de 42 kDa.
- Stocker les fractions contenant du FEN1 et concentrer la piscine à l’aide d’un filtre centrifuge de 10 kDa. Aliquotes et conservez-les au congélateur à -80° °C.
Résultats
Les lysats cellulaires BL21 (DE3) exprimant hFEN1 ont été passés à travers des résines Ni-NTA et TALON équilibrées. La résine Ni-NTA est chargée en ions Ni2+ et a une capacité de liaison élevée. Les résultats montrent que la résine Ni-NTA produit une quantité plus élevée de FEN1 par rapport à la résine TALON (Figure 7). La résine Ni-NTA est également connue pour se lier de manière non spécifique à d’autres protéines exprimées chromosomiquement. Le lysat cellulaire a traversé la résine à base de cobalt, a été purifié avec une grande pureté mais un rendement inférieur, par rapport au Ni-NTA. Comme beaucoup d’autres protéines de liaison à l’ADN, FEN1 a ensuite été passée à travers une résine d’exclusion stérique pour éliminer les impuretés. hFEN1 peut être identifié à la marque de 43 kDa.
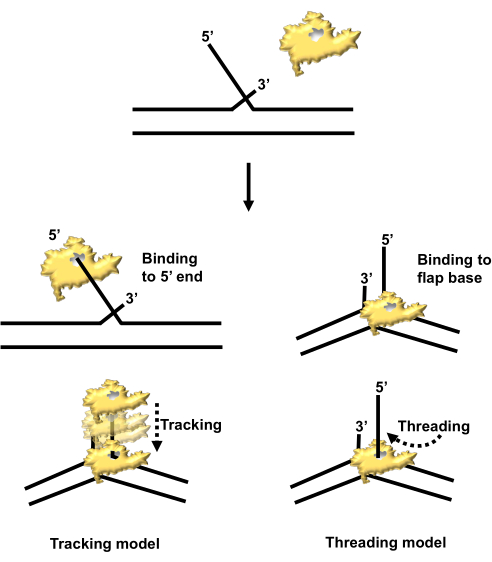
Figure 1 : Mécanisme de clivage du rabat par hFEN1 : suivi et filetage. On pensait initialement que FEN1 (représenté en jaune) se fixait à l’extrémité 5 pi de la structure du volet et la traversait jusqu’à la base du volet (à gauche). Des études ultérieures ont révélé que FEN1 se lie d’abord à la base du rabat et enfile le rabat de 5 pi à travers son site actif (à droite). Abréviation : hFEN1 = lambeau humain endonucléase 1. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Adaptateur de débit. (A) Composants de l’adaptateur de débit utilisés pour le processus d’emballage et de purification des colonnes ; (B) les trois positions du loquet à came utilisé pour serrer/desserrer le joint torique et fixer l’adaptateur en place. L’image agrandie montre l’état du joint torique (desserré/serré) aux positions 1, 2 et 3 de haut en bas. Les images ont été prises avec la permission de 9. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Module d’injection du système de chromatographie liquide protéique rapide. Les différents orifices du module d’injection relient le système de chromatographie liquide rapide en protéines à la colonne, à la boucle d’échantillonnage et aux déchets. L’image a été prise avec la permission de 10. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Contrôle du système. (A) Onglet de contrôle du système dans lequel les paramètres de débit peuvent être manipulés et le chemin d’écoulement peut être vu ; (B) fenêtre de réglage du débit ; (C) fenêtre de paramètres de la pompe. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure 5 : fenêtre des paramètres de la méthode. Une fenêtre permettant d’entrer des détails de purification tels que le type de colonne, le volume de colonne, l’unité de base de la méthode et le débit pour le cycle. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 6 : Schéma de la courbe UV pendant la FPLC : La courbe UV monte et descend en fonction de la phase de purification. Au début de l’équilibre, la courbe UV peut être déformée en raison de la présence de bulles d’air ou de contaminants dans le tube. Au fur et à mesure que l’équilibre se poursuit, la courbe devrait se stabiliser sous la forme d’une ligne plate ; Lors de l’injection de l’échantillon, à mesure que les protéines se détachent de la résine et s’écoulent, la courbe UV augmente jusqu’à ce qu’un seuil maximal soit atteint. L’étape de lavage, qui élimine les protéines résiduelles non liées, montre une diminution progressive des mesures UV. Enfin, pendant la phase d’élution, lorsque la protéine cible est éluée, un pic est observé dans la courbe UV. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 7 : Image gel de la FDS PAGE. Voie 1-Marqueur de poids moléculaire ; Voie 2-1% Entrée Ni-NTA (lysat cellulaire) ; Voie 3-1 % d’écoulement de la résine Ni-NTA ; Lane 4-hFEN1 purifié à partir de résine Ni-NTA ; Voie 5-1% d’entrée (lysat cellulaire) ; Voie 6-1% d’écoulement de la résine à base de cobalt ; Lane 7-hFEN1 purifié à partir de résine à base de cobalt ; la flèche noire représente FEN1 à 42,5 kDa. Abréviations : SDS-PAGE = dodécylsulfate de sodium ; MW = poids moléculaire ; hFEN1 = lambeau humain endonucléase 1 ; NTA = acide nitrilotriacétique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Tampons et solutions | Composants |
| Tampon de lyse | 50 mM de Tris-HCl [pH 8,0] |
| 500 mM de NaCl | |
| 1 mM de bêta-mercaptoéthanol (BME) | |
| 1 mM de fluorure de phényl-méthylsulfonyle (PMSF) | |
| 1 mM d’EDTA | |
| 1 comprimé d’inhibiteur complet de la protéase | |
| 10 mM d’imidazole | |
| 10% de glycérol | |
| Tampon d’équilibration/lavage | 50 mM de Tris-HCl [pH 8,0] |
| 500 mM de NaCl | |
| 1 mM d’EMB | |
| 1 mM de PMSF | |
| 10 mM d’imidazole | |
| 10% de glycérol | |
| Tampon d’élution | 50 mM de Tris-HCl [pH 8,0] |
| 500 mM de NaCl | |
| 1 mM d’EMB | |
| 1 mM de PMSF | |
| 1 M d’imidazole | |
| 10% de glycérol | |
| Tampon de chargement d’échantillon SDS | 900 μL 4x tampon d’échantillon Laemmli |
| 100 μL BME | |
| Coomassie solution de coloration bleu brillant | 50% ddH2O |
| 40% Méthanol | |
| 10% d’acide acétique | |
| 0.1% Coomassie bleu brillant R-250 | |
| Solution de décoloration | 50% ddH2O |
| 40% Méthanol | |
| 10% d’acide acétique |
Tableau 1 : Composition du tampon.
Discussion
La chromatographie d’affinité est une technique largement utilisée pour purifier les protéines de liaison à l’ADN. La chromatographie d’affinité métallique immobilisée (IMAC) est un type spécifique de chromatographie d’affinité qui utilise des ions métalliques pour capturer les résidus d’histidine d’une séquence peptidique. C’est pourquoi le « 6X-His tag » ou « poly-His tag » est attaché au N-terminal ou au C-terminal des protéines à purifier. Le nickel et le cobalt sont les ions métalliques les plus couramment utilisés et varient dans leur compatibilité avec des réactifs tels que le BME et le DTT normalement utilisés dans la purification. Bien que des purifications aient été effectuées à des concentrations de 1 mM de BME ou de DTT, l’utilisation de ces réactifs peut encore être peu fiable. Une incompatibilité peut être observée lors de l’équilibre. Si le tampon qui traverse la résine apparaît de couleur brune, cela indique l’oxydation des ions métalliques et peut enlever les ions métalliques de la résine. Cela pourrait réduire considérablement la capacité de liaison de la résine.
Pour garantir une pureté et un rendement élevés de protéines, les interactions d’affinité entre les ions métalliques et le His-tag doivent être privilégiées par rapport à d’autres interactions possibles basées sur la charge. Cela garantira que toute la protéine est éluée en même temps au lieu d’éluer sur les différentes phases de l’élution. Pour y parvenir, la protéine doit porter une charge très faible lors de la purification. Si les valeurs de pH des tampons sont plus proches du point isoélectrique de la protéine cible, la charge de la protéine peut être maintenue à un faible niveau. Comme le point isoélectrique de FEN1 est d’environ 8,8, nos tampons sont maintenus à un pH de 8.
On pense que la résine Ni-NTA, chargée d’ions Ni, a une plus grande capacité de liaison aux protéines et devrait produire une plus grande quantité de protéines par rapport à la résine à base de cobalt TALON. Cela signifie également qu’il y a un plus grand potentiel de liaison de protéines non spécifiques et donc une pureté plus faible. La capacité de liaison plus faible de TALON est complétée par une liaison spécifique plus élevée et un rendement en protéines de haute pureté. Ceci est cohérent avec nos résultats montrant un rendement plus élevé de protéines purifiées à partir de Ni-NTA par rapport au TALON.
Indépendamment de la résine, la pureté et le rendement sont considérablement affectés par l’emballage de la colonne. Un garnissage efficace peut être obtenu à l’aide de l’adaptateur de débit connecté au système de chromatographie automatisé NGC9. À des débits suffisamment élevés, l’adaptateur de débit peut efficacement tasser la résine et empêcher l’accumulation de tampon dans la colonne. L’adaptateur de débit associé au NGC permet une comparaison précise des deux résines grâce à la régulation du débit, de la pression et du gradient d’élution, ainsi qu’à la collecte efficace des fractions protéiques.
Grâce au logiciel Chromlab, les conditions de fonctionnement peuvent être surveillées en temps réel10. Le débit peut être modifié à tout moment tout au long du parcours. Lors de l’application de l’échantillon, la pression du système peut dépasser le seuil (600 psi). Dans ce cas, la réduction du débit pendant l’étape d’application de l’échantillon peut réduire la pression. Le logiciel dispose également d’une fonction Hold step grâce à laquelle n’importe quelle étape peut être étendue selon les besoins. Cette fonction est particulièrement utile lors des étapes de lavage et d’élution de la colonne. Pendant le lavage de la colonne, le graphique UV devrait descendre et atteindre presque 0, ce qui signifie que la plupart des protéines non liées ont été lavées de la résine. Si la ligne UV continue d’augmenter ou ne parvient pas à diminuer, l’étape de lavage peut être prolongée jusqu’à ce que le graphique attendu soit visible. De même, pendant l’élution, l’élution de la protéine cible peut être visualisée à travers des pics dans le graphique UV. Si, de manière inattendue, aucun pic n’est visible, l’étape d’élution peut être prolongée jusqu’à ce que des pics anticipés soient observés.
Trucs et astuces
Au cours de l’étape d’équilibrage, le tampon doit être injecté dans la colonne et le tampon de déchets doit être libéré en continu dans la bouteille à déchets ; Cela permet de s’assurer que toutes les connexions sont étanches et étanches. En cas de fuite de n’importe quel site de la NGC, tous les ports entourant la zone doivent être vérifiés, car l’origine de la fuite peut être différente de celle où la fuite est remarquée. Si l’écoulement a été démarré mais qu’aucun tampon n’entre dans la colonne, il est probable que de l’air soit emprisonné à l’intérieur du tube. Purgez le système avant de continuer.
Lors de l’injection de l’échantillon, si la contre-pression du système résiste à l’injection, le tube doit être dévissé de la boucle E et placé dans la bouteille de déchets pendant toute la durée de l’injection. Il est important de le revisser dans la boucle E avant de commencer le programme. Si le tampon d’équilibrage ou l’échantillon est accumulé dans la colonne pendant l’exécution, l’exécution doit être interrompue et l’adaptateur de débit retiré. Le tube doit être dévissé du bas de la colonne et le tampon ou l’échantillon doit pouvoir s’écouler à travers. Si l’échantillon s’écoule, assurez-vous de le collecter en tant que flux.
Si les fractions analysées sur le gel SDS PAGE présentent des niveaux élevés de protéines contaminantes, les fractions pourraient être analysées de chaque côté des pics observés dans le chromatogramme. Souvent, pour les protéines à faible expression, les fractions pures ne montrent pas de pics significatifs dans les UV.
Comprendre les différences entre les propriétés de la résine à base de nickel et de la résine à base de cobalt permet de sélectionner la résine la mieux adaptée aux applications futures de la protéine purifiée. Par exemple, les protéines utilisées dans les essais biochimiques in vitro doivent être de la plus grande pureté possible, une situation dans laquelle la résine à base de cobalt convient. Cependant, si la protéine est utilisée pour la production d’anticorps, un rendement significatif de protéines purifiées est nécessaire pour le processus. La résine Ni-NTA pourrait constituer une meilleure option dans ce cas. Cet article avait pour but de mettre en évidence les différences entre ces résines grâce à la purification de FEN1.
Déclarations de divulgation
Les auteurs n’ont pas d’intérêts financiers concurrents ou d’autres conflits d’intérêts.
Remerciements
Ce travail a été financé par des subventions de la National Science Foundation (1929346) et de l’American Cancer Society (RSG-21-028-01). Nous tenons également à remercier les membres du laboratoire Balakrishnan pour les discussions utiles.
matériels
| Name | Company | Catalog Number | Comments |
| 4x Laemmli sample buffer | BioRad | 1610747 | |
| Acetic acid | Merck | UN2789 | |
| Beta-mercaptoethanol (BME) | Sigma | M-6250 | |
| Chromlab software version 6.1.27.0 | BioRad | operates the NGC system | |
| Complete MINI protease inhibitor tablet | Roche | 11836153001 | |
| Coomassie Brilliant Blue R | Sigma | B0149 | |
| Dithiothreitol (DTT) | Dot Scientific | DSD11000 | |
| Econo-Column glass | BioRad | 7371512 | |
| Ethylene diamine tetraacetic acid (EDTA) | Dot Scientific | DSE57020 | |
| Flow adaptor | BioRad | 7380014 | |
| Glycerol | Dot Scientific | DSG22020 | |
| Imidazole | Dot Scientific | DSI52000 | |
| Methanol | Fisher Scientific | A412 | |
| Mini PROTEAN TGX gels | BioRad | 4561084 | |
| NGC Chromatography System | BioRad | automated liquid chromatography system | |
| Ni-NTA Agarose | Qiagen | 1018244 | |
| Phenyl-methyl-sulfonyl fluoride | Dot Scientific | DSP20270 | |
| PreScission Plus Protein Dual Color Standards | BioRad | 1610374 | |
| Sodium chloride | Dot Scientific | DSS23020 | |
| TALON metal affinity resin | Takara | 635502 | |
| Tris Base | DST60040 |
Références
- Balakrishnan, L., Bambara, R. A. Flap endonuclease 1. Annu Rev Biochem. 82, 119-138 (2013).
- Asagoshi, K. FEN1 functions in long patch base excision repair under conditions of oxidative stress in vertebrate cells. Mol Cancer Res. 8 (2), 204-215 (2010).
- Lyamichev, V., Brow, M. A., Dahlberg, J. E. Structure-specific endonucleolytic cleavage of nucleic acids by eubacterial DNA polymerases. Science. 260 (5109), 778-783 (1993).
- Bornarth, C. J., Ranalli, T. A., Henricksen, L. A., Wahl, A. F., Bambara, R. A. Effect of flap modifications on human FEN1 cleavage. Biochemistry. 38 (40), 13347-13354 (1999).
- Xu, Y., Potapova, O., Leschziner, A. E., Grindley, N. D., Joyce, C. M. Contacts between the 5' nuclease of DNA polymerase I and its DNA substrate. J Biol Chem. 276 (32), 30167-30177 (2001).
- Wen-Hui, K., Kuo, K., Chase, H. A. Exploiting the interactions between poly-histidine fusion tagsand immobilized metal ions. Biotechnol Lett. 33 (6), 1075-1084 (2011).
- Charlton, A., Zachariou, M. Immobilized metal ion affinity chromatography of native proteins. Methods Mol Biol. 421, 25-35 (2008).
- Ononye, O. E., Njeri, C. W., Balakrishnan, L. Analysis of DNA processing enzyme FEN1 and its regulation by protein lysine acetylation. Methods Mol Biol. 1983, 207-224 (2019).
- Econo-column flow adaptor instruction manual. Bio-Rad Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/M7380014.pdf (2015)
- NGC Chromatography systems and ChromLab software instrument guide version 3.3. Bio-Rad Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/10026252.pdf (2015)
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon