Method Article
Fast Protein Liquid Chromatographyシステムを用いた6X-Hisタグ付きタンパク質のアフィニティー精製(英語)
要約
この記事では、6X-ヒスチジンタグで標識されたヒト組換えタンパク質であるフラップエンドヌクレアーゼ1(FEN1)のアフィニティー精製の手順について説明します。このプロトコルでは、タグ付きタンパク質の精製に2つの異なる固定化金属イオンカラムを使用します。
要約
タンパク質の機能特性評価には、生化学的アッセイを行うために、タンパク質を高純度で大量に発現および精製する必要があります。高速タンパク質液体クロマトグラフィー(FPLC)システムにより、複雑なタンパク質混合物を高分解能で分離できます。FPLCでは、適切な精製マトリックスの選定、タンパク質サンプルの温度制御、マトリックスへのサンプルの流量や溶出速度の管理など、さまざまなパラメータを調整することで、タンパク質の安定性と機能性を確保することができます。このプロトコールでは、細菌培養で産生された6X-Hisタグ付きフラップエンドヌクレアーゼ1(FEN1)タンパク質を精製するためのFPLCシステムの汎用性を実証します。タンパク質精製効率を向上させるために、適切なカラムの充填と調製、サンプルループを使用したサンプル注入、カラムへのサンプル適用の流速、サンプル溶出パラメータなど、複数の考慮事項に焦点を当てます。最後に、クロマトグラムを分析して、高収率のタンパク質を含む画分を特定し、適切な組換えタンパク質の長期保存に関する考慮事項を特定します。タンパク質精製法の最適化は、タンパク質分析の精度と信頼性を向上させるために重要です。
概要
細胞生物学を理解するためには、数多くの戦略が考えられます。1つのアプローチには、遺伝子変異を遺伝子に導入し、その後、モデル生物に結果として生じる表現型の変化を評価するトップダウン戦略が含まれます。逆に、還元主義的アプローチでは、特定のタンパク質の分子メカニズムと酵素機能の最初の解明を伴い、他の細胞成分との相互作用の特性評価を伴います。その後、このタンパク質が生物学的経路に与える影響が評価されます。それぞれの研究アプローチには固有の利点と限界がありますが、生物学的経路を包括的に理解するには、学際的な研究が必要です。
DNAは生命の遺伝的設計図であり、DNA複製とゲノム維持のメカニズムを理解することは、70年以上にわたって活発な関心が寄せられてきた分野です。DNA複製の分野における研究は、多数の複製タンパク質の個々の構造と機能に関する豊富なデータを生み出しています。これらの調査は、機構的側面と生化学的活性アッセイを包含しており、これらのタンパク質の精製を通じて実現可能になり、 in vitro 環境での綿密な検査が可能になりました。その結果、タンパク質の精製は、DNA複製のメカニズムの洞察を解明することを目的とした研究の大部分において、不可欠でユビキタスな技術として浮上しています。
この記事では、細菌細胞で過剰発現している6X-ヒスチジンでタグ付けされたDNA複製タンパク質を単離する方法を紹介します。関心のあるタンパク質は、ヒト皮弁エンドヌクレアーゼ1(FEN1)であり、これは構造特異的なヌクレアーゼであり、遅延する鎖複製において極めて重要な役割を果たし、塩基切除修復(BER)1,2,3のようなDNA修復経路にも重要な関与しています。FEN1の主な機能は、DNA複製またはBER中に生じる中間体である5'変位フラップ構造の基部で切断することです。当初、FEN1の酵素活性を評価する生化学的研究は、ヌクレアーゼがフラップ構造の遊離5'-リン酸末端を認識し、フラップに沿ってその基部まで追跡してから切断する「トラッキング」メカニズムを示唆した4。その後の研究により、FEN1は、最初にフラップの基部に結合し、次に切断前にその活性部位に遊離5'末端を通す「スレッディング」メカニズムを介して動作することが明らかになりました(図1)5。組換えFEN1を過剰発現して単離する能力は、これらのブレークスルーを促進し、研究者が生化学的および構造的研究にFEN1を使用することを可能にしました。
アフィニティークロマトグラフィーは、DNAを精製するために一般的に使用される分離法です。この技術は、標的タンパク質が樹脂に固定化されたリガンドに対して可逆的に結合する親和性を利用して、目的のタンパク質を特異的にトラップします。最も広く使用されているバイオアフィニティーの1つは、アミノ酸、ヒスチジン、およびニッケルやコバルトなどの金属イオンとの間の強固な相互作用であり、したがって、Ni2+ またはCo2+を充填した樹脂に捕捉できます。
6-9ヒスチジン残基(His)のストリングをコードするDNA配列は、目的タンパク質(N末端またはC末端)をコードするプラスミドコンストラクトに頻繁に組み込まれ、タンパク質に6X-Hisタグまたはポリ-Hisタグをタグ付けします。その後、Hisタグ付きタンパク質は、アフィニティークロマトグラフィーのサブタイプである固定化金属アフィニティークロマトグラフィー(IMAC)によって容易に精製でき、これにより、樹脂上の金属イオンがアフィニティータグを持つタンパク質を捕捉し、後で適切な溶出剤を使用して溶出することができます。Ni2+ やCo2+ などの遷移金属イオンは 、N,N,N'-トリス-(カルボキシメチル)-エチレンジアミンまたはニトリロトリアセチン(NTA)基6に由来するアガロースまたはシリカゲルマトリックスに固定化できます。
金属配位子は、物理的、化学的、および生物学的要因による分解に対して堅牢であることが知られており、この利点を他のタイプの配位子6,7よりも保持しています。さらに、Hisタグは比較的小さなタグであり、タンパク質の構造や機能に大きな影響を与えません7。しかし、細菌発現系では、染色体で発現した多くのタンパク質が金属イオンに対して親和性を持ち、標的タンパク質と共精製することがあります。ニッケルとコバルトは、IMACマトリックスで使用される典型的な金属イオンです。Ni-NTA樹脂とTALONコバルトベース樹脂は、Hisタグ付きタンパク質の精製に一般的に使用されます。
Ni-NTAとTALONの比較
Ni-NTAとTALONのそれぞれの金属イオンは、NTA配位子を介して樹脂に固定化されます。Ni-NTAは、最大100 mg / mLのタンパク質に結合する、より高い結合能を持つと考えられています。これにより、タンパク質の収量が増加する可能性がありますが、汚染物質のタンパク質が共精製される可能性があるという注意点があります。対照的に、この樹脂はHisタグ付きタンパク質に対してより高い結合特異性を有し、より高純度の画分を産生することができる可能性がある。この研究では、参照されている自動高速タンパク質液体クロマトグラフィーシステムであるNGCシステムを使用して、両方の樹脂の精製効率を比較することを目指しています( 材料表を参照)。
バッファと互換性
バッファーは、細胞溶解、サンプル調製、樹脂平衡化、および樹脂から捕捉されたタンパク質の溶出のために、タンパク質精製中に必要です。100 mMまでのTris、MOPS、およびHEPESバッファーは、Ni-NTA樹脂に適合することが知られています。バッファーには、タンパク質の酸化やタンパク質凝集を防ぐための還元剤が含まれていることがよくあります。ただし、しきい値を超えると、還元剤は樹脂から金属イオンを剥がす可能性があります。β-メルカプトエタノール(BME)やジチオスレイトール(DTT)などの還元剤の推奨濃度は、上記のニッケルおよびコバルトベースの樹脂で1 mM未満です。
hFEN1の精製用バッファーは、NaCl、BME、フェニルメチルスルホニルフルオリド(PMSF)、EDTA、およびグリセロールを含むTrisバッファーです。NaClはタンパク質を可溶性の形で保持し、DNA結合などの分子相互作用を破壊します。BMEは酸化タンパク質を減少させ、それによってタンパク質の凝集を防ぎます。PMSF)は、プロテアーゼを介した標的タンパク質の分解を防ぐプロテアーゼ阻害剤です。EDTAは、サンプルから二価カチオンを排除し、ヌクレアーゼやプロテアーゼへのアクセスを防ぎます。グリセロールは、水性タンパク質の安定性を高めます。さらに、溶解バッファーには完全なプロテアーゼ阻害剤錠剤が含まれており、細胞溶解中の分解プロテアーゼから標的タンパク質を最大限に保護します。平衡化バッファーと溶出バッファーにはイミダゾールが含まれており、溶出バッファーには、溶出中に結合タンパク質を樹脂から置換するためのイミダゾールが大量に含まれています。
次世代クロマトグラフィー(NGC)システム
この自動化された中圧クロマトグラフィーシステムは、高速フロータンパク質液体クロマトグラフィー(FPLC)用に設計されたもので、2つのポンプを使用して2つの異なるバッファーを同時にポンピングし、250 μLから100 mLまでの幅広いサンプル量を注入できます。サンプルループ(このシステムではDynaloopと呼ばれます)により、より多くのサンプルを注入することができます。このシステムは、カスタマイズされたメソッドの作成、精製ランの操作、UVピークとタンパク質画分の分析を容易にするChromlabソフトウェアを使用して操作できます。
プロトコル
1. サンプル調製
- 組換えFEN1を精製するには、Ononyeらによって以前に報告されたように、BL21(DE3)細胞でコンストラクト(pET-FCH-FEN1)を発現します.8。
- LB培地(表1)に1%一晩培養液を接種し、ODが0.6に達するまで細胞を37°Cで増殖させます。
- 0.4 Mイソプロピル-β-D-チオガラクトシド(IPTG)で誘導し、さらに3時間培養します。
- 培養物を5,000 × g で4°Cで15分間遠心分離することにより、細胞を回収します。 ペレットを-80°Cの冷凍庫に保管します。
- 精製当日に、細胞ペレットを-80°Cの冷凍庫から取り出し、氷上で解凍した後、ペレットを100 mLの溶解緩衝液に再懸濁します(表1)。
注:細胞溶解物は、タンパク質分解を防ぐために、取り扱い中は常に氷の上に置く必要があります。解凍後、すべての精製プロセスを同じ日に完了する必要があります。細胞溶解物は冷蔵庫に一晩保存しないでください。
- 懸濁液を250mLビーカーに移し、30秒オンおよび30秒オフで35%の振幅で15分間氷上で超音波処理します。
注意: 振幅を特定するには、製造元の設定を参照してください。 - 超音波処理したサンプルを遠心チューブに移し、27,000 × g で4°Cで30分間遠心して、透明なライセートを得ます。細胞ライセートを氷上、4°Cの冷蔵庫に置き、サンプル注入まで待ちます。
2. アフィニティーカラムの準備
- カラム(Ni-NTA樹脂用とコバルト系樹脂用)をクランプスタンドに取り付け、カラムができるだけまっすぐに保持されるようにし、カラムが斜めにならないようにします。カラムの底部がストッパーで差し込まれていることを確認し、各カラムの底部に1〜2 mLの開始バッファーを追加して、カラム充満中の気泡を防ぎます。
- レジンが完全に再懸濁されるまで、レジンボトルを静かに振ってください。カラムの上部を開き、気泡の混入を避けるために、各スラリーを10 mL連続的に注ぎます。カラムの縁に固定された金属スパチュラまたはガラス棒に樹脂を注ぎ、カラム内の気泡を防ぎます。
3. フローアダプターによるカラムパッキング
- ストッパーをカラムから抜き、底部をチューブの一方の端に接続します。チューブのもう一方の端をFPLCシステムのUVポートに接続します。
- ベッドサポートハウジングがフローアダプター本体に引き込まれていることを確認し、フローアダプター本体をカラムの上部にスライドさせます(図2A)。カムラッチを1の位置に置いた状態で、フローアダプターをバッファーに挿入しないように注意しながら下げます(図2B)。カムラッチを2の位置に切り替えます(図2B)。チューブをシステムに接続し、開始バッファーの流れを1〜2分間開始して、チューブの空気を取り除き、パッキング中の気泡を防ぎます。
- ラッチを位置 1 に戻し、気泡が含まれないように 1 回のスムーズな動きでフロー アダプターをバッファーに下げ、ラッチを位置 2 に戻します。これにより、一部のバッファーがフローアダプターに入り、残っている気泡を取り除くことができます。
- ラッチを位置3に切り替え、カムベースとロックリングを締めます(図2B)。
- バッファーの流液を10 mL/分で開始し、カラムを充填します。この手順を3分間、または樹脂が十分に梱包されるまで続けます。
4. FPLCシステムのサンプルループのプライミング
- サンプルループの上部からチューブをFPLCシステムのループEに接続します(図3)。
- サンプルループの底部からチューブをFPLCシステムの廃棄物2に接続します。
- チューブの一方の端をFPLCシステムのカラムポートに挿入し、もう一方の端を廃液ボトルに入れます。
- ポンプAのチューブをろ過した二重蒸留水(ddH2O)に挿入します。
- ソフトウェアを使用して ポンプ設定を ダブルクリックし(図4A、ラベル2)、 システムポンプ注入ループ (図4C)を選択します。
- 流量設定(図4A、1とラベル付け)をクリックし、流量を10 mL/minに変更します。[%B] ボックスに 0% と入力します (図 4B)。
- 流れを開始してサンプルループを水で洗い流し、スライドシールを下に押し下げます。
- スライドシールがサンプルループの底に到達したら、流れを停止します。ポンプAをddH2Oから開始バッファに切り替えます。サンプルループの上部から廃棄物2にチューブを接続し、下部からループEにチューブを接続します。
- 10 mL/minでフローを開始します。入力開始バッファによって上向きに押し上げられたスライディングシールがサンプルループの上部に到達したら、流れを停止します。これで、サンプルループをサンプル注入する準備が整いました。
5.サンプル注入
- ポンプの設定をダブルクリックし、[手動負荷ループ]を選択します(図4C)。
- ループEからチューブを緩め、廃棄物容器に入れます。
- カラムポートからのチューブをフローアダプター上部のチューブに接続します。
- 30 mLシリンジを使用してサンプルを引き上げます。次に、シリンジを注入ポートアダプターに取り付けます。
- シリンジを注入ポートにねじ込み、サンプルを注入して、サンプルループに目に見える形で注入されていることを確認します。100 μL のサンプル (標識 インプットライセート) を -20 °C の微量遠心チューブに保管し、SDS-PAGE 分析を行います
注:総サンプル量によっては、2〜3回の注入が必要になる場合があります。 - 注入ポートをカラムキャップで塞ぎます。
- サンプルループの底部からチューブをループEポートに戻します。
- ポンプBのチューブを溶出バッファーに入れます。
6. FPLCシステム連携ソフトウェアによるメソッド作成・解析
- [ホーム] タブに移動し、[ 新しい方法] を選択します。
- 左側の列にある [ メソッド設定 ] タブを選択し、[ 列の種類 ] ドロップダウン メニューに移動して、[ カスタム ] を選択します (図 5) 。
- カラム容量(5 mL)を入力します。
- 分析に必要な 流量 (1 mL/分)を入力します。
- ユニット選択に移動し、メソッドの基本ユニットとしてCV(カラムボリューム)を選択します。
- インレット A がバッファ A に設定され、インレット B がバッファ B (デフォルト設定) に設定されていることを確認します。
- 左側のメニューの「 メソッドアウトライン 」タブを選択します。精製のステップを、平衡化、サンプル適用、カラム洗浄、溶出の順序で追加します。
- 平衡化の下で、平衡化ボリュームを10CVに設定します。バッファ設定が0%Bに保たれていることを確認してください。
- [sample application] タブで、サンプル ループを観察してサンプル量を設定します。スライドシールの位置は、サンプル量を示します。
- 「カラム洗浄」で、洗浄量を 5 CV に設定します。この手順では、分数収集を無効にします。フラクションとして収集するのではなく、実行中に全体として洗浄をビーカーに収集します。
- 溶出するには、グラジエントボリュームを 0% バッファー B から 100% バッファー B まで 14 CV に設定します。
- メソッドを保存し、実行を開始します。
- フラクションコレクターが最初のフラクションの位置から始まることを確認します。
- 実行名を入力し、[開始] をクリックします。
- 平衡化フェーズでは、リアルタイムのUVカーブを監視します。すべての接続が漏れなく安全であることを確認してください。廃棄物容器に流れ込むバッファーを探して、回路全体を確認します。平衡化の最後の数分間でUV曲線が安定していない場合は、安定化が達成されるまで平衡化ステップを延長します(図6)。
- サンプル塗布の際は、廃液容器に入るチューブを清潔なビーカーに交換してください。このステップでは、廃液チューブから流出する未結合のタンパク質をフロースルーとして収集します( FTとラベル付けします)。
- UVカーブの急激な増加が、最大読み取り値の3,000 maUに達する可能性が高いことを確認します(図6)。
注:典型的な曲線は、サンプルアプリケーションステップのほとんどを通じて3,000 maUで横ばいになります。 - カラム洗浄中、レジン上に結合したタンパク質のみが残った状態で残留した未結合タンパク質が洗い流されるため、UV曲線が急激に低下するのを探します(図6)。ウォッシュステップの終了までにUVカーブがドロップしていない場合は、UVが十分にドロップするまで ステップを押し続ける ことでステップを延長します。この手順の開始時に、廃棄物チューブを FTビーカー から Washというラベルの付いた別のきれいなビーカーに移します。
- 溶出ステップが開始されたら、フラクションコレクターが所定の位置に移動し、フラクションの収集を開始することを確認します。UV曲線のピークを探すことにより、タンパク質の溶出を視覚化します(図6)。
- 実行が完了したら、[ ホーム ] タブに移動し、[ 実行を開く] を選択します。ランを開いてクロマトグラムを表示し、トップメニューで [ピークインテグレーション ]を選択して、ラン中に得られたピークをマークします。
- 「フラクション」タブを選択して、溶出タンパク質を含むフラクションを視覚化します。
7. SDS PAGE分析
- タンパク質を含む各画分15 μLを5 μLのSDSサンプルローディングバッファーと混合します(表1)。サンプルを95°Cで5分間加熱します。
- サンプルをプレキャストゲルにロードし、染色済みタンパク質マーカーとともに160 Vで45分間分析します。
- ゲルをCoomassieブリリアントブルー染色溶液で30分間染色します(表1)。
- ゲルをddH2Oで洗浄します(3回繰り返します)。
- ゲルを脱染溶液中で1時間脱染します(表1)。
- タンパク質バンドを観察します。42kDaのマークでFEN1を検出します。
- FEN1を含むプール画分をプールし、10 kDaの遠心フィルターを使用してプールを濃縮します。分注して-80°Cの冷凍庫に保管します。
結果
hFEN1を発現するBL21(DE3)細胞ライセートを平衡化したNi-NTAおよびTALON樹脂に通しました。Ni-NTA樹脂はNi2+ イオンを帯電させ、高い結合能を有しています。その結果、Ni-NTA樹脂はTALON樹脂に比べてFEN1の量が多いことがわかりました(図7)。Ni-NTA樹脂は、他の染色体発現タンパク質に非特異的に結合することも知られています。細胞溶解物をコバルトベースの樹脂に通し、Ni-NTAと比較して高純度で精製されましたが、収率は低くなりました。他の多くのDNA結合タンパク質と同様に、FEN1はさらにサイズ排除樹脂を通過して不純物を除去しました。hFEN1は43 kDaのマークで識別できます。
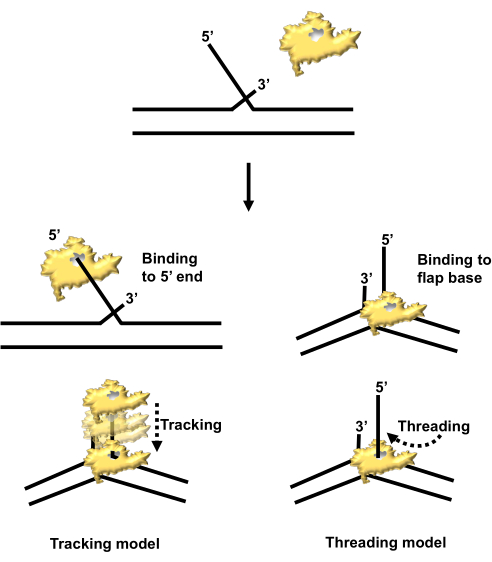
図1:hFEN1によるフラップ切断メカニズム:トラッキングとスレッディング。 FEN1(黄色で描かれている)は、当初、フラップ構造の5'端に結合し、フラップベース(左)まで追跡すると考えられていました。その後の研究で、FEN1はまずフラップ基部に結合し、5フィートフラップをその活性部位に通すことが明らかになりました(右)。略称:hFEN1 = ヒト皮弁エンドヌクレアーゼ1。 この図の拡大版を表示するには、ここをクリックしてください。

図2:フローアダプター (A)カラムの充塡および精製プロセスに使用されるフローアダプターのコンポーネント。(B)Oリングを締めたり緩めたり、アダプターを所定の位置に固定したりするために使用されるカムラッチの3つの位置。拡大した画像は、上から下に位置 1、2、3 の O リングの状態 (緩い/締まった) を示しています。画像は 9の許可を得て撮影しました。 この図の拡大版を表示するには、ここをクリックしてください。

図3:Fastタンパク質液体クロマトグラフィーシステムの注入モジュール。 注入モジュールのさまざまなポートは、高速タンパク質液体クロマトグラフィーシステムをカラム、サンプルループ、および廃棄物に接続します。画像は 10号から許可を得て撮影しました。 この図の拡大版を表示するには、ここをクリックしてください。

図4:システム制御 (A)フロー設定を操作し、流路を確認できるシステム制御タブ。(B)流量設定ウィンドウ。(C)ポンプ設定ウィンドウ。 この図の拡大版を表示するには、ここをクリックしてください。
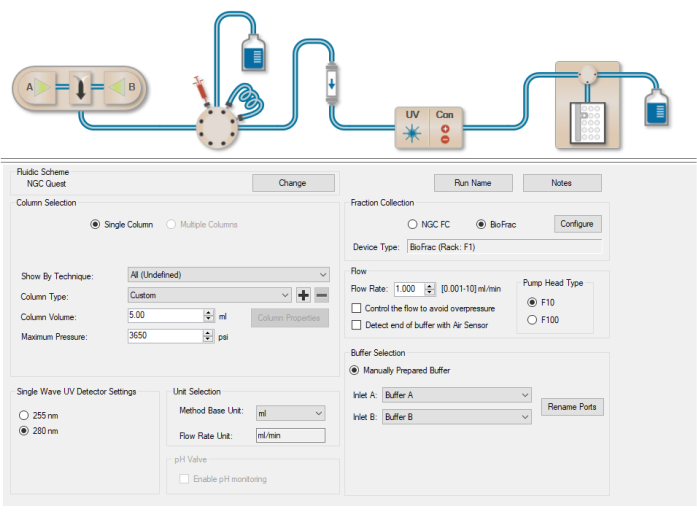
図5:メソッド設定ウィンドウ。 カラムタイプ、カラム容量、メソッドベースユニット、分析法の流量などの精製の詳細を入力するウィンドウ。 この図の拡大版を表示するには、ここをクリックしてください。

図6:FPLC中のUVカーブの概略図: UVカーブは精製フェーズに応じて上昇および下降します。平衡化の開始時には、チューブ内の気泡や汚染物質の存在により、UV曲線が歪むことがあります。平衡化が続くと、曲線は平らな線として安定するはずです。サンプル注入中、樹脂に結合していないタンパク質が流出すると、UV曲線は最大閾値に達するまで上昇します。残留した未結合タンパク質を洗い流す洗浄ステップでは、UV測定値が徐々に減少します。最後に、溶出段階では、標的タンパク質が溶出されると、UVカーブにピークが見られます。 この図の拡大版を表示するには、ここをクリックしてください。

図7:SDS PAGEゲル画像。 レーン1-分子量マーカー;レーン 2-1% Ni-NTA インプット (細胞ライセート);Ni-NTA樹脂からのレーン3-1%のフロースルー。Ni-NTA樹脂から精製されたレーン4-hFEN1。レーン5-1%インプット(細胞溶解物);コバルトベースの樹脂からのレーン6-1%のフロースルー。コバルトベースの樹脂から精製されたレーン7-hFEN1。黒矢印は42.5kDaのFEN1を表しています。略語:SDS-PAGE =ドデシル硫酸ナトリウム;MW =分子量;hFEN1 = ヒト皮弁エンドヌクレアーゼ 1;NTA = ニトリロ三酢酸。 この図の拡大版を表示するには、ここをクリックしてください。
| バッファーと溶液 | コンポーネント |
| 溶解バッファー | 50 mM トリス-HCl [pH 8.0] |
| 500 mM NaCl | |
| 1 mMベータメルカプトエタノール(BME) | |
| 1 mM フェニル-メチルスルホニルフッ化物 (PMSF) | |
| 1 mM EDTAの | |
| 1錠完全プロテアーゼ阻害剤 | |
| 10 mM イミダゾール | |
| 10%グリセロール | |
| 平衡化/洗浄バッファー | 50 mM トリス-HCl [pH 8.0] |
| 500 mM NaCl | |
| 1 mM BME | |
| 1 mM PMSF | |
| 10 mM イミダゾール | |
| 10%グリセロール | |
| 溶出バッファー | 50 mM トリス-HCl [pH 8.0] |
| 500 mM NaCl | |
| 1 mM BME | |
| 1 mM PMSF | |
| 1 M イミダゾール | |
| 10%グリセロール | |
| SDS サンプルローディングバッファ | 900 μL 4x Laemmli サンプルバッファー |
| 100 μL BME | |
| クマシーブリリアントブルー染色液 | 50% ddH2O |
| 40%メタノール | |
| 10%酢酸 | |
| 0.1% クーマシーブリリアントブルー R-250 | |
| 脱染ソリューション | 50% ddH2O |
| 40%メタノール | |
| 10%酢酸 |
表 1: バッファーの構成。
ディスカッション
アフィニティークロマトグラフィーは、DNA結合タンパク質を精製するために広く使用されている手法です。固定化金属アフィニティークロマトグラフィー(IMAC)は、金属イオンを使用してペプチド配列のヒスチジン残基を捕捉する特定のタイプのアフィニティークロマトグラフィーです。そのため、精製するタンパク質のN末端やC末端に「6X-Hisタグ」や「poly-Hisタグ」がつけられています。ニッケルとコバルトは最も一般的に使用される金属イオンであり、精製に通常使用されるBMEやDTTなどの試薬との適合性が異なります。精製は1 mM BMEまたはDTTの濃度で行われていますが、これらの試薬の使用はまだ信頼性が低い場合があります。平衡化中に非互換性が観察される可能性があります。レジンを流れるバッファーの色が茶色に見える場合は、金属イオンの酸化を示しており、レジンから金属イオンが剥がれる可能性があります。これにより、樹脂の結合能力が大幅に低下する可能性があります。
タンパク質の高純度と高収率を確保するためには、金属イオンとHisタグとの間の親和性相互作用を、他の電荷ベースの相互作用よりも優先する必要があります。これにより、すべてのタンパク質が溶出の異なる段階で溶出するのではなく、一度に溶出するようになります。これを達成するために、タンパク質は精製中に非常に低い電荷を持つ必要があります。緩衝液のpH値が標的タンパク質の等電点に近い場合、タンパク質の電荷を低く抑えることができます。FEN1の等電点は約8.8であるため、当社の緩衝液はpH 8に維持されています。
Niイオンを充填したNi-NTA樹脂は、コバルト系樹脂TALONに比べてタンパク質結合能が高く、タンパク質量が多いとされています。これは、非特異的タンパク質が結合する可能性が高くなり、したがって純度が低下することも意味します。TALONの結合能が低いことは、より高い特異的結合と高純度タンパク質の収量によって補完されます。これは、NI-NTAから精製されたタンパク質の収量がTALONと比較して高いことを示す結果と一致しています。
樹脂に関係なく、純度と歩留まりはカラム充塡剤によって大きく影響を受けます。効率的な充填は、NGC自動クロマトグラフィーシステム9に接続されたフローアダプターを使用して達成できます。十分に高い流量では、フローアダプターは樹脂を効果的に充填し、カラム内のバッファーの蓄積を防ぐことができます。フローアダプターとNGCの組み合わせにより、流量、圧力、溶出勾配の調節、およびタンパク質画分の効率的な収集により、両方の樹脂を正確に比較することができます。
Chromlabソフトウェアを通じて、実行条件をリアルタイムで監視できます10。流量は、運転中いつでも変更できます。サンプルの適用中に、システム圧力がしきい値(600 psi)を超える場合があります。この場合、サンプル塗布ステップ中に流量を下げると、圧力を下げることができます。このソフトウェアは、必要に応じて任意のステップを拡張できる ホールドステップ 機能も備えています。この機能は、カラム洗浄および溶出ステップで特に役立ちます。カラム洗浄中、UVグラフは下降し、ほぼ0に達すると予想されますが、これは未結合のタンパク質のほとんどが樹脂から洗い流されたことを意味します。UVラインが上昇し続けるか、ランプダウンに失敗した場合は、予想されるグラフが表示されるまで洗浄ステップを延長できます。同様に、溶出中、標的タンパク質の溶出はUVグラフのピークを通じて視覚化できます。予期せぬことにピークが見えない場合は、予想されるピークが観察されるまで溶出ステップを延長できます。
ヒントとテクニック
平衡化ステップでは、バッファーをカラムに注入し、廃液バッファーを廃液ボトルに連続的に放出する必要があります。これにより、すべての接続がしっかりと漏れないようにします。NGCの任意のサイトから漏れが発生した場合は、漏れの原因が漏れに気付いた場所とは異なる可能性があるため、そのエリアを囲むすべてのポートを確認する必要があります。フローが開始されているにもかかわらず、バッファーがカラムに流入していない場合は、チューブ内に空気が閉じ込められている可能性があります。先に進む前に、システムをパージしてください。
サンプル注入中に、システムからの背圧が注入に抵抗している場合は、チューブをループEから緩め、注入中は廃液ボトルに入れる必要があります。プログラムを開始する前に、ループEにねじ込むことが重要です。分析中に平衡化バッファーまたはサンプルがカラムに蓄積された場合は、分析を一時停止し、フローアダプターを取り外す必要があります。チューブをカラムの底から緩め、バッファーまたはサンプルを流す必要があります。サンプルが流れる場合は、必ずフロースルーとして収集してください。
SDS PAGE ゲルで分析したフラクションが高レベルの汚染物質タンパク質を示した場合、クロマトグラムで確認されたピークの両側でフラクションを分析できます。多くの場合、低発現タンパク質の場合、純粋な画分はUVで有意なピークを示さない。
ニッケルベース樹脂とコバルトベース樹脂の特性の違いを理解することで、精製タンパク質の将来の用途に最適な樹脂を選択することができます。例えば、 in vitro 生化学的アッセイで使用されるタンパク質は、可能な限り高い純度であることが期待され、コバルトベースの樹脂が適している状況です。しかし、タンパク質を抗体の産生に使用する場合、そのプロセスにはかなりの量の精製タンパク質が必要です。この場合、Ni-NTA樹脂がより良いオプションとして機能する可能性があります。本稿では、FEN1の精製を通じて、これらの樹脂の違いを引き出すことを目指しました。
開示事項
著者には、競合する金銭的利益やその他の利益相反はありません。
謝辞
この研究は、全米科学財団(1929346)と米国がん協会(RSG-21-028-01)からの助成金によって資金提供されました。また、バラクリシュナン研究室の皆さんにも、有益な議論をいただき、ありがとうございました。
資料
| Name | Company | Catalog Number | Comments |
| 4x Laemmli sample buffer | BioRad | 1610747 | |
| Acetic acid | Merck | UN2789 | |
| Beta-mercaptoethanol (BME) | Sigma | M-6250 | |
| Chromlab software version 6.1.27.0 | BioRad | operates the NGC system | |
| Complete MINI protease inhibitor tablet | Roche | 11836153001 | |
| Coomassie Brilliant Blue R | Sigma | B0149 | |
| Dithiothreitol (DTT) | Dot Scientific | DSD11000 | |
| Econo-Column glass | BioRad | 7371512 | |
| Ethylene diamine tetraacetic acid (EDTA) | Dot Scientific | DSE57020 | |
| Flow adaptor | BioRad | 7380014 | |
| Glycerol | Dot Scientific | DSG22020 | |
| Imidazole | Dot Scientific | DSI52000 | |
| Methanol | Fisher Scientific | A412 | |
| Mini PROTEAN TGX gels | BioRad | 4561084 | |
| NGC Chromatography System | BioRad | automated liquid chromatography system | |
| Ni-NTA Agarose | Qiagen | 1018244 | |
| Phenyl-methyl-sulfonyl fluoride | Dot Scientific | DSP20270 | |
| PreScission Plus Protein Dual Color Standards | BioRad | 1610374 | |
| Sodium chloride | Dot Scientific | DSS23020 | |
| TALON metal affinity resin | Takara | 635502 | |
| Tris Base | DST60040 |
参考文献
- Balakrishnan, L., Bambara, R. A. Flap endonuclease 1. Annu Rev Biochem. 82, 119-138 (2013).
- Asagoshi, K. FEN1 functions in long patch base excision repair under conditions of oxidative stress in vertebrate cells. Mol Cancer Res. 8 (2), 204-215 (2010).
- Lyamichev, V., Brow, M. A., Dahlberg, J. E. Structure-specific endonucleolytic cleavage of nucleic acids by eubacterial DNA polymerases. Science. 260 (5109), 778-783 (1993).
- Bornarth, C. J., Ranalli, T. A., Henricksen, L. A., Wahl, A. F., Bambara, R. A. Effect of flap modifications on human FEN1 cleavage. Biochemistry. 38 (40), 13347-13354 (1999).
- Xu, Y., Potapova, O., Leschziner, A. E., Grindley, N. D., Joyce, C. M. Contacts between the 5' nuclease of DNA polymerase I and its DNA substrate. J Biol Chem. 276 (32), 30167-30177 (2001).
- Wen-Hui, K., Kuo, K., Chase, H. A. Exploiting the interactions between poly-histidine fusion tagsand immobilized metal ions. Biotechnol Lett. 33 (6), 1075-1084 (2011).
- Charlton, A., Zachariou, M. Immobilized metal ion affinity chromatography of native proteins. Methods Mol Biol. 421, 25-35 (2008).
- Ononye, O. E., Njeri, C. W., Balakrishnan, L. Analysis of DNA processing enzyme FEN1 and its regulation by protein lysine acetylation. Methods Mol Biol. 1983, 207-224 (2019).
- Econo-column flow adaptor instruction manual. Bio-Rad Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/M7380014.pdf (2015)
- NGC Chromatography systems and ChromLab software instrument guide version 3.3. Bio-Rad Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/10026252.pdf (2015)
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved