Method Article
Um ensaio de recuo de molécula única otimizado para quantificação da fosforilação de proteínas
Neste Artigo
Resumo
O presente protocolo descreve a preparação da amostra e a análise de dados para quantificar a fosforilação proteica usando um ensaio de recuo de molécula única (SiMPull).
Resumo
A fosforilação é uma modificação pós-transinal necessária que regula a função proteica e direciona os resultados da sinalização celular. Os métodos atuais para medir a fosforilação proteica não podem preservar a heterogeneidade na fosforilação entre proteínas individuais. O ensaio de tração única de moléculas (SiMPull) foi desenvolvido para investigar a composição de complexos macromoleculares através da imunoprecipitação de proteínas em um deslizamento de vidro seguido de imagem de molécula única. A técnica atual é uma adaptação do SiMPull que fornece quantificação robusta do estado de fosforilação de receptores de membrana de comprimento total no nível de molécula única. A imagem de milhares de receptores individuais dessa forma permite quantificar padrões de fosforilação de proteínas. O presente protocolo detalha o procedimento SiMPull otimizado, desde a preparação da amostra até a imagem. A otimização dos protocolos de preparação de vidro e fixação de anticorpos melhora ainda mais a qualidade dos dados. O protocolo atual fornece código para a análise de dados de molécula única que calcula a fração de receptores fosforilados dentro de uma amostra. Enquanto este trabalho se concentra na fosforilação do receptor do fator de crescimento epidérmico (EGFR), o protocolo pode ser generalizado para outros receptores de membrana e moléculas de sinalização citostónica.
Introdução
A sinalização associada à membrana é ajustada por uma combinação de ativação do receptor de membrana induzida por ligante e recrutamento de proteínas acessórias a jusante que propagam o sinal. A fosforilação das tyrosinas-chave nas caudas citoplasmáticas receptoras é fundamental para iniciar a formação de complexos de sinalização, ou signalossomos 1,2. Portanto, uma questão importante na biologia é como os padrões de fosforilação são criados e mantidos para recrutar parceiros de sinalização e ditar resultados celulares. Isso inclui compreender a heterogeneidade da fosforilação receptora, tanto em abundância quanto nos padrões específicos de fosfotimosina que podem fornecer um meio de manipular saídas de sinalização ditando a composição do sinalosome 3,4,5,6,7. No entanto, existem limitações nos métodos atuais para interrogar a fosforilação proteica. A análise de manchas ocidentais é excelente para descrever tendências de fosforilação proteica, mas é semi-quantitativa8 e não fornece informações sobre a heterogeneidade do sistema porque milhares a milhões de receptores são mediados juntos. Embora as manchas ocidentais permitam sondar uma amostra usando anticorpos específicos do fosfo para tirasinas específicas, elas não podem fornecer informações sobre padrões de fosforilação multisite dentro da mesma proteína. A fosfoproteômica quantitativa relata a abundância de fosfotyrosina, mas há limitações para detectar fosforilação multisite, pois os resíduos de interesse precisam estar localizados dentro do mesmo peptídeo (tipicamente 7-35 aminoácidos) que é gerado pela digestão enzimática 9,10,11.
Para superar as limitações mencionadas acima, o ensaio de uma única molécula pull-down (SiMPull) foi adaptado para quantificar os estados de fosforilação de receptores intactos no nível de molécula única. O SiMPull foi demonstrado pela primeira vez como uma poderosa ferramenta para interrogar complexos macromoleculares por Jain et al.12,13. No SiMPull, os complexos macromoleculares foram imunoprecipitados (IP) em tampas de vidro funcionalizadas por anticorpos e, em seguida, analisados através de microscopia de molécula única para número de subunidade proteica e co-IP com componentes complexos12. Uma modificação de Kim et al.14, denominada SiMBlot, foi a primeira a usar uma variação de SiMPull para analisar a fosforilação de proteínas desnaturadas. O protocolo SiMBlot baseia-se na captura de proteínas de superfície celular biotinilada usando tampas revestidas de NeutrAvidin, que são então sondadas para fosforilação com rotulagem de anticorpos específicos do fosfo14. Apesar desses avanços, foram necessárias melhorias para tornar a quantificação da modificação pós-transinal mais robusta e aplicável a uma gama mais ampla de proteínas.
O presente protocolo descreve uma abordagem simpull otimizada que foi usada para quantificar padrões de fosforilação do receptor de fator de crescimento epidérmico intacto (EGFR) em resposta a uma série de condições de ligantes e mutações oncogênicas15. Embora este trabalho se concentre no EGFR, essa abordagem pode ser aplicada a qualquer receptor de membrana e proteínas citosóicas de interesse (POI), para as quais anticorpos de qualidade estão disponíveis. O protocolo inclui etapas para reduzir a autofluorescência da amostra, um projeto de matriz de amostra que requer volume mínimo de amostra com preparação simultânea de até 20 amostras e otimização das condições de rotulagem e fixação de anticorpos. Algoritmos de análise de dados foram desenvolvidos para detecção e quantificação de proteínas fosfoiladas.
Protocolo
1. Preparação do deslizamento de cobertura
NOTA: Para esta etapa, é preciso usar equipamentos de proteção individual (EPI), que inclui uma camada dupla de luvas de nitrito, óculos de segurança ou escudo facial, e um jaleco.
- Realize a gravura de piranha para remover detritos orgânicos do vidro.
ATENÇÃO: A solução piranha é um agente oxidante forte que é corrosivo e altamente reativo quando em contato com materiais orgânicos. A reação com detritos orgânicos é exotérmica e potencialmente explosiva. Assim, o procedimento deve ser realizado em um capô de fumaça química com a faixa abaixada. O vidro Pyrex é necessário para lidar com a solução piranha.- Prepare o espaço de trabalho dentro de um capô de fumaça química. Disponha tampas sem sobreposição no fundo de um copo de vidro 4 L, o béquer "reação" e coloque o béquer de reação em uma placa quente com calor suave. Deixe os vidros aquecerem por 10 minutos. Coloque um béquer de vidro "desperdício" 1 L com 500 mL de ddH2O proximal ao béquer de reação.
- Adicione 49 mL de ácido sulfúrico de 12 N (H2SO4) lentamente ao béquer de reação com uma pipeta sorológica de vidro. Enxágüe a pipeta no béquer antes do descarte.
- Adicione 21 mL de peróxido de hidrogênio de 30% (H2O2) dropwise com uma pipeta sorológica de vidro ao béquer de reação. Distribua lentamente as gotículas H2O2 uniformemente através da parte inferior do frasco de reação para evitar a sacieda localizada da reação de gravação de piranha. Enxágüe a pipeta no béquer antes do descarte.
ATENÇÃO: Adicione sempre H2O2 no H2SO4, e nunca vice-versa. - Piranha grava as tampas por 30 min. Agitar suavemente o conteúdo do béquer de reação a cada 5 minutos.
- Sacie a solução de piranha despejando o conteúdo do béquer de resíduos no béquer de reação. Transfira o líquido lentamente pela parede do béquer de reação para minimizar o espirro. Remova o béquer de reação da placa de aquecimento.
- Quando a reação for saciada e resfriada, despeje a solução de piranha de volta no béquer para neutralização sem remover as tampas gravadas do béquer de reação.
- Neutralizar a solução de piranha com a adição gradual de uma base fraca. Por exemplo, use uma massa excessiva de 20 g de bicarbonato de sódio (NaHCO3)/100 mL solução de piranha.
ATENÇÃO: Não armazene soluções de piranha em recipientes de resíduos lacrados. A solução deve ser sempre neutralizada antes do descarte. A reação de neutralização produz bolhas vigorosas e pode ser explosiva se não controlada pela adição gradual da base fraca. - Mexa a solução neutralizada com uma haste de vidro e deixe-a reagir por 2h. Levante o pH para >4 e descarte a solução.
- Entre as adições de base fraca à solução piranha, transfira os clipes gravados do béquer de reação para um funil Buchner com uma haste de vidro e enxágue por 5 minutos na execução ddH2O.
NOTA: Prossiga imediatamente para o próximo passo ou armazene as tampas gravadas por piranha por até 2 semanas em ddH2O em um frasco de vidro selado ou placa de Petri (enrole com filme de vedação, veja Tabela de Materiais).
- Sonicar as tampas em solventes orgânicos seguindo os passos abaixo.
- Coloque as tampas em um frasco de vidro Coplin (ver Tabela de Materiais) e cubra com metanol (CH3OH). Sele a tampa do frasco com filme de vedação e sonicato de banho por 10 minutos. Despeje cuidadosamente o metanol do frasco coplin em uma garrafa de armazenamento de vidro.
- Encha o frasco de Coplin com acetona (C3H6O), sele a tampa e sônica de banho por 10 minutos. Despeje cuidadosamente a acetona do frasco Coplin em uma garrafa de armazenamento de vidro.
ATENÇÃO: O metanol é inflamável e extremamente tóxico. Use em um capuz de fumaça química. Acetona é inflamável e irritante. Portanto, manuseie com e guarde em vidro, e use-o em um capô de fumaça química. Descarte como resíduos perigosos de acordo com as normas e diretrizes locais.
NOTA: Metanol e acetona podem ser reutilizados até cinco vezes cada.
- Ative a superfície de deslizamento de tampa para a funcionalização do silano.
- Sonicato de banho com hidróxido de potássio de 1 M (KOH) por 20 min. Despeje cuidadosamente o KOH do Frasco de Coplin em um tubo cônico de 50 mL para reutilização.
ATENÇÃO: KOH é corrosivo e irritante. Use em um capô de fumaça química e não guarde em vidro. Guarde-o em tubos de polipropileno. Descarte como resíduos perigosos de acordo com as normas e diretrizes locais.
NOTA: O KOH pode ser reutilizado em até cinco vezes. - Enxágüe duas vezes com ddH2O. Drene ddH2O das tampas e, em seguida, aqueça cada mancha de cobertura acenando através da chama de um queimador de Bunsen para afastar toda a umidade da superfície. Coloque as tampas em um frasco de Coplin seco.
- Sonicato de banho com hidróxido de potássio de 1 M (KOH) por 20 min. Despeje cuidadosamente o KOH do Frasco de Coplin em um tubo cônico de 50 mL para reutilização.
- Executar coberturalip aminosilanização.
- Prepare a solução de aminosilano misturando 69,4 mL de metanol com 3,6 mL de ácido acético (CH3COOH) em um frasco cônico. Adicione 720 μL de N-(2-aminoetil)-3-aminopropyltrimethoxysilane (aminosilano) e misture bem (ver Tabela de Materiais).
ATENÇÃO: O ácido acético é inflamável e corrosivo. Manuseie com pipetas de vidro e armazene em vidro. Trabalhe com ácido acético em um capô de fumaça química. Aminosilano é um risco de inalação agudamente tóxico, um sensibilizador e um irritante. É prejudicial à vida aquática. Use em um capuz de fumaça química. Descarte os produtos químicos como resíduos perigosos de acordo com as normas e diretrizes locais.
NOTA: Aminosilano é fotossensíveis e hidrólises rapidamente na água. Todas as etapas com este reagente precisam ser executadas sob condições mínimas de luz para reter a atividade. Limpe a garrafa com gás nitrogênio e aplique filme de vedação antes de armazenar em um desiccator escuro. Substitua a cada 6-9 meses. - Adicione imediatamente a solução de aminosilano ao frasco de Coplin. Cubra e aplique o filme de vedação, continuando a proteger da luz.
- Incubar as tampas na solução de aminosilano por 10 minutos no escuro à temperatura ambiente (RT). Sonicato de banho por 1 min e depois incubar por mais 10 minutos. Despeje cuidadosamente a solução de aminosilano em um recipiente de resíduos designado para CH3OH com aminosilano de traço e CH3COOH.
- Enxágüe as tampas com metanol e despeje a solução em um recipiente de resíduos designado para metanol.
- Enxágüe as tampas três vezes por 2 min cada com ddH2O. Escorra as tampas, destrua o excesso de umidade e o ar seque completamente por 10 minutos.
- Prepare a solução de aminosilano misturando 69,4 mL de metanol com 3,6 mL de ácido acético (CH3COOH) em um frasco cônico. Adicione 720 μL de N-(2-aminoetil)-3-aminopropyltrimethoxysilane (aminosilano) e misture bem (ver Tabela de Materiais).
- Realize a preparação do array/a funcionalidade biotin-PEG das tampas.
- Prepare 1 M NaHCO3 (pH 8.5) de estoque de trabalho dissolvendo 84,5 mg de NaHCO3 em 1 mL de ddH2O. Para concentração final de 10 mM NaHCO3, diluir 1 M NaHCO3 em ddH2O (1:100).
- Desenhe uma matriz de grade nas tampas aminosilanizadas secas com uma caneta de barreira hidrofóbica (ver Tabela de Materiais) e aguarde que a tinta seque. Escreva um identificador no deslizamento de tampas para marcar a orientação adequada. Coloque as tampas em uma câmara umidificada.
NOTA: A matriz deve ser composta por 16-20 quadrados, aproximadamente 4 mm x 4 mm de tamanho. - Para fazer a solução de valerate de succinimidyl biotina-PEG (biotin-PEG)/mPEG succinimidyl valerate (mPEG), primeiro, remova mPEG e biotin-PEG (ver Tabela de Materiais) do congelador e equilibre-se à RT. Adicione 153 mg de mPEG e 3,9 mg de biotin-PEG (~1:39 biotin-PEG:mPEG) a um tubo de microcentrifuuge de 1,5 mL e resuspenque em 609 μL de 10 mM NaHCO3 por tubulação suave. Centrifugar a 10.000 x g por 1 min no RT para remover bolhas.
NOTA: A meia-vida hidrolise de valerate de succinimidyl em pH 8,5 tampão é ~30 min. Depois de adicionar o buffer ao mPEG, proceda com as seguintes etapas o mais rápido possível. Este passo é crítico. - Aplique a solução biotin-PEG/mPEG para cobrir completamente cada quadrado nas matrizes de deslizamento de cobertura, tipicamente de 10-15 μL por quadrado. Não permita que o líquido transborde o espaço definido. Armazene as tampas em uma câmara de umidade no escuro por 3-4 h no RT.
- Lave as tampas com quantidades abundantes de água mergulhando-as sequencialmente em 3x 250 mL de bicos de vidro preenchidos com ddH2O por 10 s cada.
- Retire toda a umidade das tampas com gás nitrogênio. Armazene as tampas de costas para trás em um tubo cônico de 50 mL cheio de nitrogênio embrulhado com filme de vedação a -20 °C.
NOTA: Proceda imediatamente para a etapa 2 ou as tampas da loja deslizes a -20 °C por até 1 semana antes do uso.
2. Preparação de SiMPull lysate
ATENÇÃO: Os EPI necessários para as etapas restantes do protocolo são luvas de nitrito, óculos de segurança e jalecos.
NOTA: Os lises foram preparados a partir das células CHO aderentes expressando EGFR-GFP. As células foram banhadas em um prato de cultura tecidual de 60 mm (TC60) durante a noitede 12,13. As células CHO foram cultivadas em DMEM suplementadas com 10% de soro bovino fetal, 1% L-glutamina, 1% penicilina-estreptomicina e 500 ng/mL de geneticina (ver Tabela de Materiais). Outras linhas de células aderentes ou células de suspensão também podem ser usadas.
- Emplaque as células seguindo os passos abaixo.
- Lave o prato de cultura (contendo as células) com 1 mL de 1x PBS. Adicione 1 mL de trippsina 1x e incubar por 5 min a 37 °C para desprender as células. Usando uma pipeta, transfira as células separadas do prato para um tubo centrífuga de 1,5 mL.
- Pegue 10 μL de azul trypan e misture com 10 μL de suspensão celular em um tubo de centrífuga separado. Conte as células usando 10 μL da mistura celular em um contador automático de células, de acordo com as instruções do fabricante (ver Tabela de Materiais).
- Placa 8 x 105 células durante a noite em uma placa de petri TC60. Prato um prato por condição.
NOTA: Para o presente estudo, as células não foram tratadas ou tratadas com Pervanadate e EGF, conforme descrito na etapa 2.4.1.
- Prepare as seguintes soluções para a preparação do lise celular.
- Prepare o 1x PBS gelado (pH 7.4).
- Prepare o tampão de lise, uma solução de 1% detergente não iônico e não degenerador (ver Tabela de Materiais) em 50 mM Tris-HCl (pH 7.2) e 150 mM NaCl complementado com Inibidor de Protease/Fosfattase (PPI) (1:100 de estoque). Coloque o tubo em um nutador e deixe o tampão misturar por 15 minutos. Mantenha a solução preparada no gelo.
- Prepare o tampão do Tyrode para uma concentração final de 135 mM NaCl, 10 mM KCl, 0,4 mM MgCl2, 1 mM CaCl2, 10 mM HEPES (pH 7.2), 20 mM de glicose e 0,1% Bovine Serum Albumin (BSA) (ver Tabela de Materiais). Aqueça a solução a 37 °C.
- Prepare o controle positivo para fosforilação - 1 mM de tratamento pervanadate.
NOTA: Esta etapa é opcional. Pervandate é a forma peroxidizada de vanadate-an inibidor de fosfates de proteína tyrosina16. Prevenir a desfosforilação proteica inibindo a atividade fosfatase resulta em uma amostra altamente fosfoilada.- Prepare um estoque de 200 mM de ortovanadate de sódio ativado (Na3VO4).
- Para preparar a solução de 100 mL, adicione 3,89 g de Na3VO4 (ver Tabela de Materiais) a 90 mL de ddH2O e dissolva enquanto mexe. Ajuste o pH para 10 adicionando HCl ou NaOH dropwise. Adicionar HCl vai tornar a solução amarela.
- Leve o volume para 100 mL com ddH2O. Ferva a solução aquecendo-a no micro-ondas. Depois de ferver, a solução será incolor.
- Esfrie a solução para RT e reajuste o pH para 10. Repita o ajuste de ebulição, resfriamento e pH de duas a quatro vezes, até que o pH se estabilize em 10. Aliquot e armazenar a -20 °C.
- Prepare um estoque de 30 mM pervanadate (PV) misturando 20,4 μL de 3% H2O2 com 100 μL de 200 mM Na3VO4 e 546,8 μL de ddH2O (concentrações equimolar de H2O2 e ativada naVO 34). Incubar no escuro em RT por 15 minutos.
- Prepare 1 mM PV no buffer de Tyrode. Para a solução de 10 mL, adicione 0,33 mL de estoque PV de 30 mM a 9,67 mL de 37 °C Tyrode tampão. Trate as células imediatamente.
- Lave células uma vez com 3 mL de tampão de Tyrode. Adicione 3 mL de 1 mM PV no tampão do Tyrode às células e incubar por 15 min a 37 °C.
- Prepare um estoque de 200 mM de ortovanadate de sódio ativado (Na3VO4).
- Realize a estimulação de ligas.
- Estimule as células com o ligante de interesse usando concentração, tempo e temperatura adequadas. Para estimulação máxima de EGFR, incubar com 1 mL de 50 nM Epidérmico Growth Factor (EGF, ver Tabela de Materiais) + 1 mM de PV no buffer de Tyrode por 5 min a 37 °C.
- Faça lise celular.
- Após o tratamento celular desejado, coloque o prato no gelo e lave com 1x PBS gelado. Remova completamente o volume total de PBS usando uma pipeta.
- Adicione 180 μL de tampão de lise (passo 2.2.2) à placa. Use um raspador de célula para puxar o tampão ao redor da placa para cobrir as células completamente. Aplique pressão firme e consistente com o raspador de células em toda a superfície cultivada para lise totalmente as células.
NOTA: O volume de tampão de lise precisa ser mantido no mínimo para garantir uma alta concentração proteica. - Pipeta as células lísedas e transferi-las para um tubo de 1,5 mL. Mantenha o tubo no gelo por 30 minutos. Vórtice os lises a cada 5 minutos.
NOTA: Se o POI for composto por múltiplas subunidades ou for sensível à dissociação, não faça o vórtice dos lises. - Centrifugar as células líssedas a 16.000 x g por 20 min a 4 °C. Transfira o supernatante para um novo tubo de 1,5 mL usando uma pipeta. Este contém o total de proteínas lysate.
- Reserve 10 μL do liseto e dilua-o em 90 μL do tampão de lise para análise de ensaio colorimétrico bicinchonínico (BCA)17. Armazene o restante da proteína total a -80 °C.
- Determine a concentração total de proteínas por meio da análise de BCA (ver Tabela de Materiais).
NOTA: Os lisatos totais de proteína podem ser preparados no dia do experimento e usados frescos ou armazenados como alíquotas de uso único a -80 °C por até 12 semanas. Não congele/descongele.
3. Funcionalização da matriz com o anticorpo biotinilado
- Prepare as seguintes soluções.
- T50 Buffer, uma solução de 10 mM Tris-HCl (pH 8.0) e 50 mM NaCl. A solução é estável por 1 mês na RT.
- T50-BSA suplementando tampão T50 com 0,1 mg/mL de BSA. Mantenha a solução preparada no gelo.
- 10 mg/mL de boroidido de sódio (NaBH4) em 1x PBS. Prepare isso imediatamente antes de usar.
- 0,2 mg/mL de NeutrAvidin (ver Tabela de Materiais) no buffer T50.
ATENÇÃO: O NaBH4 é um agente redutor e é inflamável. Limpe sempre o recipiente com gás nitrogênio após o uso e armazene-o em um desiccador.
- Funcionalize a matriz com o anticorpo biotinilado.
- Remova as matrizes funcionalizadas PEG-biotin do congelador e equilibre o tubo cônico para RT antes de abrir. Coloque o deslizamento com a matriz orientada para um filme de vedação forrado de 100 mm de cultura tecidual (TC100).
NOTA: Minimizar a iluminação aérea. Todas as soluções devem "subir" nos quadrados definidos pela matriz hidrofóbica. Adicione um volume apropriado de solução para cobrir completamente cada quadrado (tipicamente de 10-15 μL) e não permita que o líquido transborde o espaço definido. Para remover rapidamente os líquidos, use uma linha de vácuo interna presa a um frasco de vácuo para capturar resíduos. Deixe naBH4 para degas por 1 h antes de descarte, deixando o tubo aberto no capô da fumaça química. O tratamento naBH4 é necessário para reduzir a autofluorescência de fundo, reduzindo assim detecções de moléculas simples falsas e positivas. - Trate cada quadrado da matriz com 10 mg/mL de NaBH4 em 1x PBS por 4 min na RT. Lave três vezes com PBS.
- Incubar cada quadrado por 5 min com 0,2 mg/mL de NeutrAvidin em T50. Lave três vezes com T50-BSA.
NOTA: NeutrAvidin se liga ao PEG-biotina e fornece um local de ligação para anticorpos biotinylated 12,13,15. - Incubar cada quadrado por 10 min com 2 μg/mL de anticorpo específico poi biotinilado em T50-BSA; lavar três vezes com T50-BSA.
NOTA: O presente protocolo usa o IgG anti-EGFR biotinilado (ver Tabela de Materiais) para capturar EGFR-GFP.
- Remova as matrizes funcionalizadas PEG-biotin do congelador e equilibre o tubo cônico para RT antes de abrir. Coloque o deslizamento com a matriz orientada para um filme de vedação forrado de 100 mm de cultura tecidual (TC100).
4. SiMPull de POI de lises de células inteiras
NOTA: Coloque o prato TC100 de matrizes simpull funcionalizadas no gelo para o restante da preparação do SiMPull. Este passo é a retirada de um POI do total de proteínas lysate. O lise não deve ser reutilizado após o descongelamento.
- Prepare as seguintes soluções.
- Prepare 4% de paraformaldeído (PFA)/0,1% glutaraldeído (GA) em 1x PBS.
ATENÇÃO: PFA e GA são fixadores químicos tóxicos e potenciais cancerígenos. Use EPI. Descarte os produtos químicos como resíduos perigosos de acordo com as normas e diretrizes locais. - Prepare 10 mM Tris-HCl, pH 7.4.
- Prepare 4% de paraformaldeído (PFA)/0,1% glutaraldeído (GA) em 1x PBS.
- Descongele e misture o lise por tubos suavemente para cima e para baixo. Mantenha-se no gelo.
- Diluir 1 μL do lysate em 100 μL de gelo-frio T50-BSA/PPI.
NOTA: Se necessário, determine o fator de diluição adequado do liseto proteico total, aplicando uma série de diluições à matriz. A densidade ideal dos receptores SiMPull por área de matriz é de 0,04-0,08/μm2. As diluições de lise podem ser avaliadas na etapa 6 (análise de dados). - Incubar o lysate na matriz por 10 min; em seguida, lave quatro vezes com T50-BSA/PPI gelado.
- Diluir o anticorpo antifosfotyrosina conjugado AF647 (ver Tabela de Materiais) no frio do gelo T50-BSA/PPI e incubar na matriz por 1h.
NOTA: No presente protocolo, um Pan anti-pTyr (PY99)-AF647 IgG é usado para identificar a população fosfoilada de EGFR-GFP. O uso de anticorpos diretamente rotulados elimina a necessidade de anticorpos secundários, aumentando as opções de rotulagem e melhorando a consistência dos resultados. Anticorpos fluorescentes podem ser obtidos de fontes comerciais. Se não estiverem disponíveis comercialmente, os anticorpos podem ser rotulados sob medida usando técnicas padrão de bioconjugação e kits de bioconjugação comercial. Cada lote de anticorpos fluorescentes rotulados precisa ser testado para condições ideais de rotulagem, realizando SiMPull para medir uma curva de dose e encontrar o ponto de saturação. - Lave seis vezes com T50-BSA gelada para um total de 6-8 min.
- Lave duas vezes com 1x PBS gelado.
- Incubar a matriz com solução PFA/0,1% GA de 4% por 10 minutos para evitar a dissociação de anticorpos.
- Lave duas vezes por 5 min cada com 10 mM Tris-HCl, pH 7.4/PBS para inativar os fixativos.
NOTA: Para experimentos que utilizam mais de um anticorpo ( por exemplo, detectando vários sítios de fosforosclerose), repita as etapas 4.5-4.9. Consulte o passo 6.2.9 para obter informações sobre como determinar o obstáculo estérico entre dois anticorpos.
5. Aquisição de imagens
NOTA: A aquisição de imagem de molécula única é realizada usando um objetivo TIRF de 150x e um divisor de imagens que captura cada canal espectral em um quadrante específico da câmera emCCD (ver Tabela de Materiais). As imagens de calibração são adquiridas pela primeira vez para permitir o registro do canal e a calibração do ganho da câmera com uma grade de alinhamento de canal nanopatada (nanogrid) que contém 20 x 20 matrizes de 200 ± buracos de 50 nm a uma distância intrahole de 3 ± 1 μm (tamanho total ~60 μm × 60 μm).
- Realize o registro do canal seguindo as etapas abaixo.
NOTA: É necessário um registro preciso do canal para calcular adequadamente a colocalização dos emissores. Este passo é crítico.- Limpe o objetivo do óleo e deposite uma gota de óleo no objetivo. Coloque a nanogrid no palco para imagens. Usando luz branca transmitida, concentre-se no padrão da grade.
NOTA: As imagens com a nanogrid são adquiridas usando luz transmitida, que passa pela nanogrid e é detectada em todos os canais espectrais. Alternativamente, pode-se usar contas multifluorescentes que emitem fluorescência detectada em cada canal. A aquisição de imagens precisará ser otimizada de acordo com cada configuração de microscópio. - Adquira uma série de 20 imagens da grade. Certifique-se de que os pixels não estão saturados. Salve a série de imagens como "Fiducial".
- Desfoque a nanogrid para criar um padrãoarejado 18. Adquira uma série de 20 imagens para obter calibrações. Salve a imagem como "Ganho".
- Adquira uma série de 20 imagens para a câmera compensada bloqueando toda a luz de ir para a câmera. Salve a imagem como "Fundo".
- Limpe o objetivo do óleo e deposite uma gota de óleo no objetivo. Coloque a nanogrid no palco para imagens. Usando luz branca transmitida, concentre-se no padrão da grade.
- Adquira imagens SiMPull.
NOTA: Antes de imaginar o array de deslizamento de cobertura, troque a solução Tris por T50-BSA e equilibre a matriz para RT.- Limpe o objetivo do óleo e deposite óleo adicional no objetivo. Fixar a matriz de deslizamento de tampas no estágio do microscópio.
- Otimize o poder de excitação de cada fluoróforo, o ângulo TIRF e o tempo de integração da câmera. O objetivo é alcançar o maior sinal-ruído, minimizando o fotobleaching da amostra. Regisso laser para consistência em medições futuras.
NOTA: O presente estudo utilizou 300 ms tempo de exposição para o canal vermelho distante e 1 s para o canal verde. O laser de 642 nm foi usado a aproximadamente 500 μW de potência laser, enquanto o laser de 488 nm foi usado a 860 μW, medido antes da lente do tubo. - Adquira imagens para cada amostra. Imagem o canal vermelho primeiro, seguido por cada fluorophore de comprimento de onda inferior para reduzir o fotobleaching. Devido ao baixo volume utilizado para cada amostra, verifique o nível do buffer a cada 30-45 min e reponha conforme necessário.
6. Análise de dados
- Baixe os códigos de demonstração.
NOTA: Os conjuntos de dados de demo e exemplo fornecidos demonstram o fluxo de trabalho completo de análise de dados (Arquivos de Codificação Suplementar 1-4). Os requisitos do sistema listados no SiMPullMain.m são encontrados no Arquivo de Codificação Suplementar 1.-
Descompactar e salvar em um diretório de Documentos/MATLAB pessoais (MacOS/Linux) ou Documentos\MATLAB (Windows).
NOTA: Isso gera quatro novas pastas: SiMPull_class, smite, Dados de Amostra, Saídas de Análise de Amostras. - Abra o arquivo "ReadMe_Setup.txt" encontrado na pasta SiMPull_class.
- Instale caixas de ferramentas MATLAB e MATLAB: Caixa de ferramentas de ajuste de curvas, caixa de ferramentas de computação paralela e caixa de ferramentas estatísticas e machine learning.
- Instale o DipImage19 de acordo com as instruções de download.
- Instale o pacote de análise de molécula única "smite", conforme descrito em ReadMe_setup.txt.
NOTA: "smite" está disponível no repositório do GitHub (ver Tabela de Materiais). - Abra o SiMPullMain.m, encontrado em SiMPull_class pasta, arrastando o arquivo para a janela MATLAB.
- Alterar diretório para ...\MATLAB\Sample Data\ clicando no ícone Procurar pasta e selecionando a pasta.
-
Descompactar e salvar em um diretório de Documentos/MATLAB pessoais (MacOS/Linux) ou Documentos\MATLAB (Windows).
- Visão geral das etapas de processamento de dados
- Execute SiMPullMain.m - seguindo instruções para cada seção. Execute cada seção individualmente colocando o cursor nessa seção e clicando no ícone Executar seção.
NOTA: As etapas gerais para análise de dados estão descritas nesta seção. Instruções detalhadas são encontradas no código SiMPullMain.m que acompanha. - Execute a seção "Inicialização" para definir o caminho para definir canais espectrais e tamanho da imagem.
- Execute a seção "Encontre ganho de câmera e deslocamento" para converter ganho de câmera em fótons usando os conjuntos de dados Gain and Background.
- Execute a seção "Registro de Canal" para calcular a transformação média ponderada local usada para registro de imagem.
- Formate e faça a curadoria dos dados. Execute a seção "Junte canais sequenciais em uma imagem quádrupla". Execute "Remova quadros ruins".
- Execute a seção "Ajuste moléculas simples e encontre moléculas sobrepostas".
NOTA: Esta seção executa múltiplas funções para localização de moléculas únicas em cada canal e determina eventos de colocalização entre canais espectrais. - Para determinar a contagem mínima de fótons por ajuste GFP verdadeiro, execute o "Optimize Minimum Photon Threshold". Este é um processo iterativo.
- Primeiro, coloque a SMF. Thresholding_MinPhotons = [0, 0, 0] e executar a seção. Selecione arquivos "Dados em branco" quando solicitado. Repita com os arquivos "CHO-EGFR-GFP".
- Selecione um valor mínimo de limite apropriado comparando os dois histogramas. Definir smf. Thresholding_MinPhotons = [475, 0, 0] e executar a seção novamente.
- Execute a seção "Porcentagem de cálculo do GFP se encaixa positivo para sinal FR" para corrigir as localizações de fundo e calcular os valores finais.
- Execute a Opção 1 (etapa 6.2.10) ou a Opção 2 (etapa 6.2.11) conforme necessidade experimental.
- Opção 1: Corrija o número de receptores disponíveis na membrana plasmática para ligação de ligas, conforme descrito na Referência15.
- Primeiro, rotular receptores de superfície com níveis saturados de corante NHS Ester fluorescente (NHS-AF647, ver Tabela de Materiais). Em seguida, realize um experimento de SiMPull para determinar a porcentagem de localização do GFP que se colocalize com AF647.
NOTA: Isso fornece uma estimativa da fração de receptores disponíveis para rotulagem do NHS e a razão de receptores na superfície (RS). - Aplique a correção de RS no cálculo final: NGFP = (NLOC - NBG)*SR, onde o NGFP é o número corrigido de localizações de GFP, NBG é o número de localização em segundo plano, e NLOC é localizações totais.
NOTA: No presente exemplo, essa correção não é aplicada porque Pervanadate é permeável à membrana16 e, portanto, a ação da inibição da fosfatase não se limita aos receptores superficiais.
- Primeiro, rotular receptores de superfície com níveis saturados de corante NHS Ester fluorescente (NHS-AF647, ver Tabela de Materiais). Em seguida, realize um experimento de SiMPull para determinar a porcentagem de localização do GFP que se colocalize com AF647.
- Opção 2: Para medições de fosforilação multisite, considere o potencial de obstáculo estérico quando dois anticorpos são usados.
NOTA: O empecilho estérico pode causar uma redução no percentual observado de receptores fosforilalatados para anticorpo 1 quando na presença de Anticorpo 2 (P12), em comparação com o anticorpo 1 sozinho (P1).- Use o SiMPull para determinar P1 e P12 e calcular o fator de correção para impedimento estérico, seguindo a referência15 publicada anteriormente.
- Execute SiMPullMain.m - seguindo instruções para cada seção. Execute cada seção individualmente colocando o cursor nessa seção e clicando no ícone Executar seção.
Resultados
Um desenho animado retratando o processo SiMPull é mostrado na Figura 1A. As manchas são funcionalizadas usando NeutrAvidin como uma âncora para anticorpos anti-EGFR biotinilados para capturar EGFR-GFP de lisatos totais de proteínas. Após a lavagem da proteína não ligada, os receptores fosforilalatados são rotulados com um anticorpo anti-fosfotyrosina (anti-PY)15. A Figura 1B mostra uma imagem da matriz hidrofóbica, onde várias amostras podem ser preparadas e imagens na mesma mancha. Uma vantagem deste suporte de amostra é que são necessários volumes mínimos de amostra de ~10 μL. O deslizamento de tampa pode ser imageado colocando-o diretamente no estágio do microscópio. No entanto, é útil estabilizar o deslizamento de tampa usando um suporte de deslizamento de tampa. O suporte de deslizamento de tampas mostrado na Figura 1B foi criado usando uma impressora 3D, e o projeto é fornecido no Arquivo de Codificação Suplementar 5. A autofluorescência da tinta hidrofóbica é um guia útil para encontrar o plano focal da amostra (Figura 1C). Um exemplo de uma imagem bruta multicanal é mostrado na Figura 1D. Uma sobreposição dos canais verde e vermelho cru é mostrada na Figura 1E.
A Figura 2 descreve o fluxo de trabalho da análise e fornece dados representativos. A aquisição de dados começa primeiro com a aquisição de fiduciários para registro de canais, que é usado para sobrepor os dados dos canais espectrais individuais (Figura 2A). Imagens de campo brilhante são tiradas usando um padrão nanogrid que passa a luz branca e é detectado em cada canal espectral do divisor de imagens (não mostrado). O canal verde atua como canal de referência, e o canal vermelho é o canal deslocado. A transformação média ponderada local é calculada usando a função fitgeotrans20 no MATLAB e é usada para deslocar coordenadas muito vermelhas para o quadro de coordenadas do canal verde. Esta transformação usa um modelo polinomial de segunda ordem em cada ponto de controle. Os dados multicanais da matriz SiMPull são então adquiridos. Este fluxo de trabalho consistia em uma aquisição semi-automatizada, onde um ROI inicial foi selecionado para o quadrado de amostra específico, e três regiões ao redor desta área foram imagens, de tal forma que cada conjunto de dados contém a imagem de exibição quádrupla completa de três ROIs independentes (Figura 1D). Em cada canal espectral, os locais dos candidatos emitter são encontrados aplicando uma diferença do filtro gaussiano às imagens e identificando máximas locais. Sub-regiões (caixas, Figura 2B) são desenhadas em torno de maxima local, e as contagens de fótons emissores são estimadas assumindo que cada sub-região contém apenas um emissor. Sub-regiões que mantêm candidatos emissores com contagem de fótons acima de um valor mínimo são mantidas para a montagem. Uma função de spread de ponto gaussiano (PSF) se encaixa em cada candidato emissor dentro de pequenas sub-regiões aproximadamente centradas em torno de cada emissor. As localizações resultantes são limiares com base em sua contagem de fótons, fundo, Cramér-Rao inferior do limite das coordenadas de ajuste, variância PSF (ou seja, largura PSF) e um valor p descrevendo a bondade do ajuste do modelo PSF. Uma imagem gaussiana é criada para cada canal espectral, com bolhas gaussianas de intensidade uniforme colocadas nas coordenadas para cada bom ajuste (Figura 2C). A colocalização é visualizada sobrepondo as imagens gaussianas de cada canal espectral utilizando a transformação calculada a partir da amostra fiduciária (Figura 2D). É importante rotular fluorescentemente o receptor para identificação, uma vez que ainda há ligação não específica dos anticorpos anti-fosfotyrosina à superfície quando o linsato celular está presente. O EGFR-GFP (canal verde) é usado para gerar uma máscara dos locais do receptor, e apenas o sinal AF647-anti-PY (canal vermelho distante) dentro dessa máscara é contado (Figura 2D). Pares dentro de 1 pixel (tamanho de pixel de 106,7 nm) são considerados colocalizados e salvos em uma lista contendo as coordenadas do canal de referência. A porcentagem de AF647 colocalized com GFP é calculada para determinar a fração de receptores fosforilalatados (Figura 2E).
Existem várias etapas críticas para garantir uma boa qualidade de dados. Um desses esforços é incubar a matriz de deslizamentos com o NaBH4 , conforme descrito no protocolo para saciar a autofluorescência no canal verde. Esta autofluorescência refere-se ao sinal não específico devido a possíveis impurezas no vidro, contendo ligações π únicas ou conjugadas21. Tais impurezas são potencialmente dos reagentes de aminoselano e PEG usados no processo de funcionalização ou poeira do ar, e tendem a fluorescer no canal espectral verde. Apesar dos esforços para manter o vidro armazenado sob nitrogênio, essas moléculas também podem ser geradas através da oxidação que ocorre no armazenamento. O NaBH4 também tem sido usado para reduzir a fluorescência de impurezas em slides e microarrays, incluindo aqueles com revestimento de silano16. A Figura 3A mostra a redução no número de detecções de fundo que ocorrem quando o vidro gravado de piranha é tratado com NaBH4. Enquanto o NaBH4 reduz drasticamente a fluorescência de fundo, alguns emissores ainda são detectados no canal verde. Pode-se corrigir isso adquirindo imagens de fundo de amostras sem sabor (Figura 3D) e subtraindo o número médio de localizações de fundo das amostras contendo GFP. A fluorescência das impurezas não foi detectada no canal vermelho distante. Se a densidade do receptor for muito alta, vários emissores de GFP podem ser encontrados dentro de um único ponto limitado por difração (dados não mostrados). Usando o fotobleaching passo para identificar o número de GFPs por ponto, descobrimos que uma densidade receptora entre 0,04-0,08 proteínas/μm2 forneceu espaçamento suficiente entre emissores únicos para remover o potencial de encontrar vários emissores por ponto12. A densidade do receptor pode ser otimizada variando a quantidade de anticorpo IP ligado à superfície do vidro ou a quantidade de lysate adicionada. É fundamental garantir que o anticorpo direcionado ao POI seja usado em níveis saturados. Recomenda-se adquirir uma curva de concentração de anticorpos em amostras fosfolalatadas para determinar as condições de rotulagem adequadas (Figura 3B). Além disso, a especificidade fosfo de um anticorpo precisa ser validada com amostras de repouso e/ou tratamento com inibidores de quinase específicos para proteínas (Figura 3B). Os anticorpos se dissociarão do receptor durante a janela de tempo de imagem. O tratamento da amostra com uma combinação de PFA e GA impediu a perda de sinal (Figura 3C).
Por fim, é importante otimizar os parâmetros de montagem de molécula única. A primeira etapa de "encontrar caixa" que identifica potenciais candidatos emissores (Figura 2B) precisa ser generosa para permitir que muitos candidatos se submetam ao Ajuste Gaussiano. Assim, o limiar mínimo de fótons para encontrar caixa pode ser relativamente baixo para capturar todos os emissores reais e alguns pontos de fundo. Também é importante não definir o tamanho da caixa e sobrepor a mesada muito pequena. Manter o tamanho da caixa dentro de 5-7 pixels e permitir a sobreposição de dois pixels é ideal para emissores na densidade recomendada. Após a descoberta da caixa, o limiar mínimo de fótons na etapa de encaixe precisa ser otimizado. O parâmetro mínimo de fótons contribui para determinar quais emissores gaussianos passam como um verdadeiro ajuste. Para determinar o limite mínimo adequado de fótons para os verdadeiros ajustes de GFP, o código inclui uma função de plotagem de histograma para examinar os fótons/localização em ambos os fundos (sem lise celular) e amostras contendo GFP (mais células lysate) (Figura 3D). Este passo é importante porque, embora o NaBH4 reduza a quantidade de fluorescência das impurezas, ela não remove todas as localizações de fundo. A Figura 3D demonstra a necessidade de definir um limiar mínimo de fótons para reduzir o número de detecções de impurezas. Para determinar esse limiar, um histograma de intensidades de emissor de fundo é calculado a partir de uma imagem de uma amostra que não está exposta ao liseto celular (Figura 3D, superior esquerda). A maioria dos emissores de fundo foi encontrada com valores inferiores a 475 fótons. Em comparação, a amostra contendo verdadeiros emissores de GFP mostrou uma fração significativa da distribuição acima de 475 (Figura 3D, superior direita). O limiar é escolhido por inspeção para remover o maior número possível de contagens de fundo, minimizando a quantidade de perda de sinal da amostra de lise (Figura 3D, linha inferior). A densidade restante da contagem de antecedentes neste limiar é contabilizada na análise quantitativa.
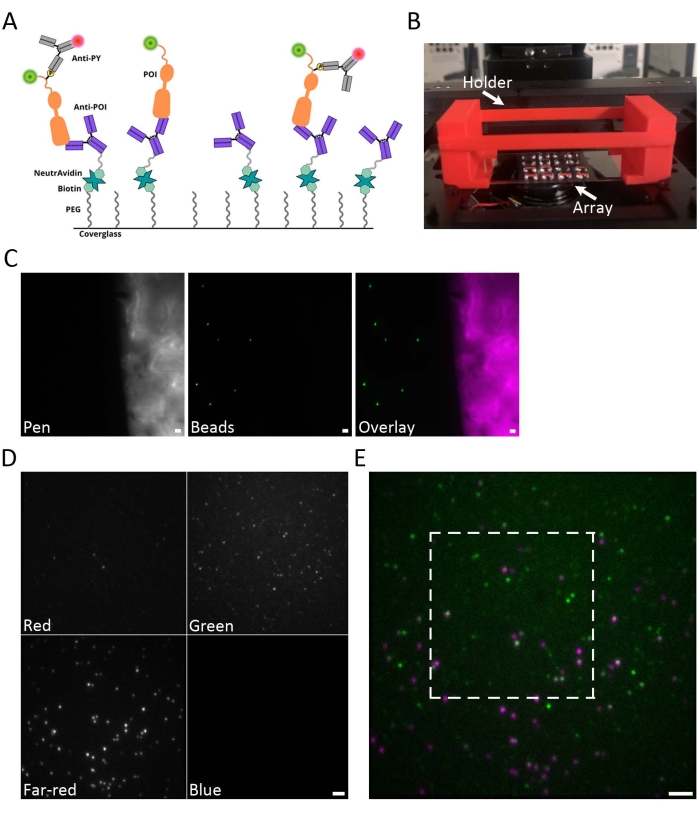
Figura 1: Visão geral da preparação da amostra. (A) Desenho animado representando a abordagem SiMPull. As manchas são funcionais com um anticorpo que reconhece o POI para capturar esse POI de lisatos celulares inteiros. O vidro é revestido pela primeira vez com PEG e biotin-PEG. NeutrAvidin é então vinculado ao biotina-PEG e age como uma âncora para o anticorpo anti-POI biotinylated. Proteínas fosforiladas são então detectadas com um anticorpo anti-PY rotulado fluorescentemente. (B) Fotografia do suporte de deslizamento de tampa (vermelho) com matriz de deslizamento de tampa no lugar e montado no estágio do microscópio. As matrizes multiamostra são geradas usando tinta hidrofóbica para criar até 20 quadrados de amostra individuais em uma única mancha de vidro. O deslizamento de tampas é de 60 mm x 24 mm. (C) Exemplos de imagens da autofluorescência de tinta hidrofóbica (magenta) e contas fluorescentes (verde). A autofluorescência da tinta hidrofóbica é um guia útil para encontrar o plano focal na superfície da fenda. (D) Exemplo de uma imagem de dados brutos com canais espectrais separados no chip da câmera pelo divisor de imagens quádruplo. O conjunto de filtros quádruplo inclui os seguintes filtros de emissão: azul (445/45 nm), verde (525/45 nm), vermelho (600/37 nm), vermelho distante (685/40 nm). (E) Sobreposição bruta de canais verdes e vermelhos. A caixa branca indica a região ainda mais examinada na Figura 2B-D. Barra de escala (C-E) = 2 μm. Clique aqui para ver uma versão maior desta figura.
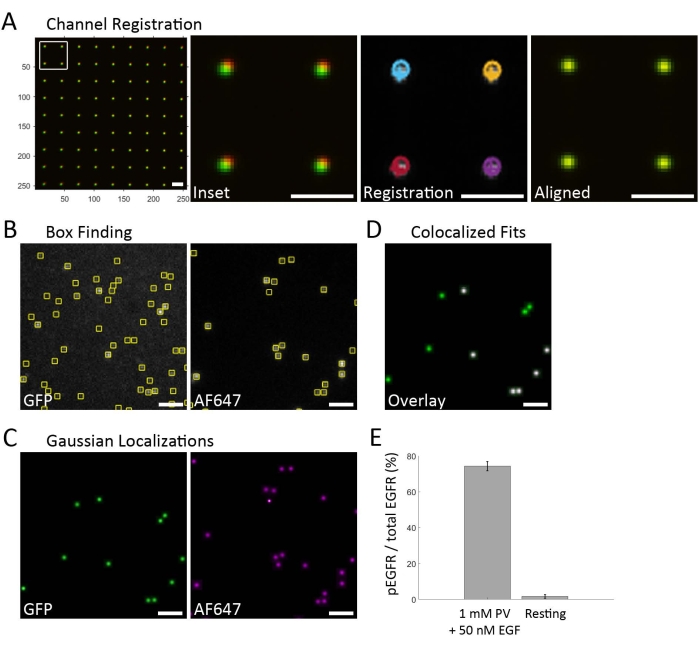
Figura 2: Fluxo de trabalho de análise de dados. (A) O registro do canal é realizado pela primeira vez em imagens adquiridas do nanogrid. Depois de cortar os dois canais espectrais de interesse (aqui, verde e vermelho), as imagens fiduciárias para cada canal são sobrepostas (esquerda). O alargamento da caixa na imagem esquerda (Inset) mostra que as imagens ainda não estão realmente registradas. Os emissores de cada canal se encaixam em um modelo gaussiano e são localizados (Registro). A localização dos emissores é mostrada como círculos para o canal vermelho e cruzes para o canal verde. O passo final é aplicar uma transformação média ponderada local para deslocar as coordenadas de localização do canal vermelho para o quadro de referência do canal verde (Alinhado). A transformação média ponderada local calculada é então usada para registrar os dados subsequentes do SiMPull. (B) Imagens representativas do canal verde/EGFR-GFP e do canal anti-PY, muito vermelho/AF647. Os emissores únicos acima da contagem de fótons de fundo são identificados e marcados com caixas. (C) O perfil de emissão dentro de cada caixa selecionada é adequado a um modelo gaussiano, e os emissores que se encaixam no modelo de um único PSF fluorophore são mantidos. (D) Uma máscara é criada a partir dos emissores GFP para identificar a localização do EGFR-GFP (verde). A colocalização do EGFR-GFP e do AF647-anti-PY identifica receptores fosforilalatados (brancos). (E) A fração de receptores fosfoilados é calculada a partir dos ajustes EGFR-GFP e AF647-anti-PY. O gráfico da barra compara o tratamento PV + EGF com as células de repouso, em média para múltiplas medidas. As barras de erro representam erro padrão calculado assumindo uma distribuição binomial. Barra de escala = 2 μm. Clique aqui para ver uma versão maior desta figura.

Figura 3: Passos críticos para garantir a qualidade dos dados. (A) Da esquerda para a direita, os três primeiros painéis são imagens representativas da autofluorescência no vidro nas respectivas condições: após a gravura de piranha, com PEG, e peg mais tratamento NaBH4 (indicado com +). Além disso, a funcionalidade superficial é mantida após o tratamento naBH4 , como demonstrado pela ligação mínima não específica PY99-AF647, mantendo a vinculação robusta do EGFR-GFP do lysate. (B) Uma curva de saturação deve ser adquirida para cada lote de anticorpos utilizados para garantir a rotulagem ideal de anticorpos. Esta figura mostra a curva de concentração para rotular EGFR com o anticorpo fosfotyrosina específico do local, anti-EGFR-pY1173. A fosforilação mínima é detectada em células não tratadas (Descansando, diamante cinza). Como controle para ligação não específica, as células também foram tratadas com o inibidor de quinase EGFR, Lapatinib, antes de adicionar 100 nM de EGF (triângulo magenta), o que mostra a prevenção esperada da fosforilação EGFR. As barras de erro representam erro padrão assumindo uma distribuição binomial. (C) Fixação da amostra com uma combinação de PFA e GA impede a dissociação de anticorpos ao longo do tempo. As barras de erro representam erro padrão assumindo uma distribuição binomial. (D) Falsos positivos são excluídos selecionando o limiar apropriado para a montagem gaussiana. Comparar o histograma de intensidades de ajuste em um limiar baixo (Limiar = 0; superior) entre fundo (sem lise) e dados reais (mais lise celular) permite a seleção de valor apropriado (Limiar = 475; inferior) para remover os ajustes de pontos autofluorescentes no canal verde. A linha magenta vertical indica um limiar de 475 fótons. Os histogramas são calculados a partir do mesmo número de ROIs para cada tipo de amostra (n = 3). Barra de escala = 2 μm. Clique aqui para ver uma versão maior desta figura.
Arquivo de codificação suplementar 1: arquivo zip contendo scripts e utilitários para executar a análise do SiMPull. Clique aqui para baixar este Arquivo.
Arquivo de codificação suplementar 2: arquivo zip contendo o pacote de análise de molécula única smite . Clique aqui para baixar este Arquivo.
Arquivo de codificação suplementar 3: arquivo zip contendo os dados da amostra. Clique aqui para baixar este Arquivo.
Arquivo de codificação suplementar 4: arquivo zip contendo saídas representativas de análise de dados de amostra. Clique aqui para baixar este Arquivo.
Arquivo de codificação suplementar 5: Projeto do suporte de deslizamento de cobertura para impressão 3D. Clique aqui para baixar este Arquivo.
Discussão
O protocolo descrito aqui foi otimizado para permitir medições quantitativas de fosforilação receptora no nível único da proteína. Várias modificações simples, mas importantes no protocolo SiMPull foram desenvolvidas que melhoraram a confiabilidade da medição para detecção de fosfo-tyrosina, incluindo redução da autofluorescência com tratamento NaBH4 e pós-fixação da amostra para evitar a dissociação de anticorpos. O uso da máscara de canal verde para identificar os locais do receptor para o cálculo da colocalização com o anticorpo anti-PY também melhora a precisão da medição, removendo artefatos potenciais da ligação não específica do anticorpo ao lysato celular. A imagem de duas cores foi utilizada para detectar a fração de receptores fosfoilados. Nesse cenário, o receptor foi geneticamente marcado com GFP, e o anticorpo foi diretamente rotulado com um corante vermelho distante. A abordagem SiMPull se aplica a outros alvos proteicos para os quais anticorpos específicos estão disponíveis, incluindo proteínas intracelulares. Além disso, como as condições de desnaturação não são necessárias, receptores/complexos multisubunitas também podem ser capturados. No entanto, a desnaturação pode ser incorporada se os PTMs de interesse estiverem localizados em regiões estruturadas da proteína14. Em última análise, o SiMPull pode ser facilmente expandido para incluir rotulagem simultânea de fosfo-tirasinas distintas em receptores individuais para quantificar padrões de fosforilação multisite15. O interrogatório de receptores intactos de comprimento completo de tal forma não pode ser alcançado por outros métodos padrão, incluindo mancha ocidental e espectrometria de massa fosfo.
Junto com as vantagens do SiMPull, algumas limitações precisam ser consideradas. Como em qualquer técnica baseada em anticorpos, a afinidade e especificidade dos anticorpos utilizados são fundamentais para o sucesso da medição. Por isso, é importante otimizar as condições de rotulagem de anticorpos e, idealmente, evitar anticorpos secundários usando anticorpos primários diretamente rotulados. Além disso, os anticorpos ligados à superfície precipitarão proteínas localizadas na membrana plasmática e dentro de compartimentos citosóicos. Isso pode subestimar a fosforilação, uma vez que as proteínas localizadas com citosol não são acessíveis ao ligante exógeno adicionado. Medidas extras devem ser tomadas para corrigir os níveis de superfície do receptor (passo 6.2.10). Os anticorpos antifosfotyrosina exibiram alguma ligação não específica uma vez que o lysato estava presente. Para evitar este artefato, o EGFR foi geneticamente marcado com GFP para identificar a localização dos receptores, o que nos permitiu excluir o sinal anti-PY do receptor. Se as proteínas endógenas forem interrogadas, então a contra-mancha com um anticorpo proteico total pode fornecer a imagem da máscara, com correção adequada para qualquer ligação não específica. Finalmente, enquanto o SiMPull fornece informações sobre heterogeneidade no nível da proteína, o lysate gerado neste protocolo é de milhares de células, e a variabilidade célula-celular é perdida. No entanto, os avanços em direção ao SiMPull unicelular foram feitos usando uma câmara de fluxo consistindo em um deslizamento de fenda e um lado do microscópio com uma lacuna de 10 μm; as bactérias foram pouco banhadas no deslizamento enquanto o slide foi funcionalizado com anticorpos para capturar as proteínas desejadas. Após a lise das bactérias, as proteínas de cada célula foram capturadas em uma área confinada no slide revestido deanticorpos 22. Análises semelhantes de SiMPull de células mamíferas e fosforilação proteica podem ser possíveis no futuro.
O protocolo SiMPull contém várias etapas críticas necessárias para garantir dados de alta qualidade. Por exemplo, o protocolo inclui uma elaboração elaborada do vidro de deslizamento de tampas. As tampas de gravação de piranha limpam completamente o vidro e aumentam grupos de hidroxílico e hidrofilialidade, que são necessárias para otimizar a superfície do deslizamento de cobertura. Após várias lavagens com solventes orgânicos, o tratamento KOH fornece grupos hidroxil adicionais para aminosilanização13,23, que reveste o vidro com grupos de amina para ligação PEG e biotin-PEG. A limpeza ou a funcionalidade inadequadas em qualquer uma dessas etapas interferirão com a retirada de proteínas. O controle da razão molar de PEG:biotin-PEG, juntamente com a concentração de liseto, são fatores-chave na obtenção da densidade de IP proteica adequada no substrato SiMPull. Como em qualquer ensaio biológico, há variabilidade entre as preparações de liseto celular, e pequenas diferenças entre os percentuais de fosforilação podem ser vistas entre as réplicas da amostra. Por isso, é importante medir os níveis de fosforilação de diferentes sítios de tirasina dentro da mesma amostra. A câmara de amostra descrita neste protocolo fornece um sistema para coletar muitos pontos de dados em uma sessão de imagem e permite uma média sobre vários experimentos simpull.
No lado da aquisição de imagens, é importante obter a amostra fiduciária para garantir uma sobreposição precisa do canal; caso contrário, a colocalização não será precisa. Também é importante otimizar a potência do laser e as configurações da câmera para maximizar o sinal-ruído e, ao mesmo tempo, minimizar o fotobleaching. Por último, enquanto o conjunto amostral requer uma pequena quantidade de amostra e reagentes, os baixos volumes são suscetíveis à evaporação durante a sessão de imagem. É importante verificar periodicamente o conjunto amostral (~30-45 min) e adicionar tampão conforme necessário para evitar que as amostras sequem.
O presente protocolo demonstrou o uso do SiMPull para quantificar os estados de fosforilação do receptor de membrana. Embora focada no EGFR, a abordagem pode ser aplicada a outros receptores de superfície celular e proteínas intracelulares e complexos proteicos, desde que os anticorpos apropriados estejam disponíveis. Outro uso potencial para o SiMPull é interrogar o conteúdo e o status de fosforilação de condensados separados de fases. Além disso, o SiMPull pode ser usado para medir outros PTMs, como a ubiquização. Portanto, o SiMPull fornece uma ferramenta única para biólogos celulares interrogarem PTMs em proteínas intactas e correlacionar padrões de PTM com desfecho celular.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado pelos Institutos Nacionais de Saúde R35GM126934, R01AI153617 e R01CA248166 para DSL. A EMB foi apoiada através do programa ASERT-IRACDA (NIH K12GM088021) e jar pelo programa UNM MARC (NIH 2T34GM008751-20). Agradecemos o uso do recurso compartilhado de microscopia de fluorescência do Centro de Câncer Integral da Universidade do Novo México, apoiado pelo NIH P30CA118100. Queremos reconhecer os Drs. Ankur Jain e Taekijip Ha, cujo desenvolvimento original do SiMPull inspirou este trabalho.
ES-C apresentar endereço: Grupo de Imunodinâmica, Laboratório de Imunologia Integrativa do Câncer, Centro de Pesquisa do Câncer, Instituto Nacional de Câncer, Bethesda
Materiais
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes | MTC Bio | C2000 | |
| 10 mM Tris-HCl pH 7.4 | |||
| 10 mM Tris-HCl pH 8.0/ 50 mM NaCl | T50 Buffer | ||
| 100 mm Tissue Culture dish | CELLTREAT | 229620 | Storage of piranha etched glass/arrays |
| 15 mL conical tube | |||
| 16% Paraformaldehyde Aqueous Solution | Electron Microscopy Sciences | 15710 | Hazardous |
| 50 mL conical tube | Functionalized Glass storage/ KOH reuse | ||
| 50 mM Tris-HCl pH 7.2/150 mM NaCl | Lysis Buffer Component | ||
| 60 mm Tissue Culture dish | Corning | 430166 | |
| 8% Glutaraldehyde Aqueous Solution | Electron Microscopy Sciences | 16020 | Hazardous |
| Acetone (C3H6O) | Millipore Sigma | 270725 | Hazardous |
| Alexa Fluor 647 NHS Ester | Thermo Fisher Scientific | A-20006 | |
| Animal-Free Recombinant Human EGF | Peprotech | AF-100-15 | |
| Anti-Human EGFR (External Domain) – Biotin | Leinco Technologies, Inc | E101 | |
| Anti-p-Tyr Antibody (PY99) Alexa Fluor 647 | Santa Cruz Biotechnology | sc-7020 AF647 | |
| Bath-sonicator | Branson | 1200 | |
| BCA Protein Assay Kit | Pierce | 23227 | |
| Biotin-PEG | Laysan Bio | Biotin-PEG-SVA, MW 5,000 | |
| Bovine serum albumin | Gold Biotechnology | A-420-1 | Tyrode's Buffer Component |
| Buchner funnel | |||
| Bunsen burner | |||
| Calcium Chloride (CaCl2) | Millipore Sigma | C4901 | Tyrode's Buffer Component |
| Cell Scraper | Bioworld | 30900017-1 | |
| Conical Filtering Flask | Fisher Scientific | S15464 | |
| Coplin Jar | WHEATON | 900470 | |
| Countess II Automated Cell Counter | Thermo Fisher Scientific | AMQAX1000 | |
| Coverslips 24 x 60 #1.5 | Electron Microscopy Sciences | 63793 | |
| DipImage | https://diplib.org/ | ||
| DMEM | Caisson Labs | DML19-500 | |
| emCCD camera | Andor iXon | ||
| Fetal Bovine Serum, Optima | Bio-Techne | S12450H | Heat Inactivated |
| Fusion 360 software | Autodesk | ||
| Geneticin G418 Disulfate | Caisson Labs | G030-5GM | |
| Glacial Acetic Acid (CH3COOH) | JT Baker | JTB-9526-01 | Hazardous |
| Glass serological pipettes | |||
| Glass Stir Rod | |||
| Glucose (D-(+)-Glucose) | Millipore Sigma | D9434 | Tyrode's Buffer Component |
| Halt Phosphotase and Protease Inhibitor Cocktail (100X) | Thermo Fisher Scientific | 78446 | Lysis Buffer Component |
| HEPES | Millipore Sigma | H3375 | Tyrode's Buffer Component |
| Hydrochloric Acid (HCl) | VWR | BDH7204-1 | Hazardous |
| Hydrogen Peroxide (H2O2) (3%) | HX0645 | ||
| Hydrogen Peroxide (H2O2) (30%) | EMD Millipore | HX0635-2 | |
| Ice | |||
| IGEPAL CA-630 (NP-40) | Sigma Aldrich | I8896 | Lysis Buffer Component |
| ImmEdge Hydrophobic Barrier Pen | Vector Laboratories | H-4000 | |
| Immersol 518F immersion oil | Zeiss | 444960-0000-000 | |
| in-house vacuum line | |||
| L-glutamine | Thermo Fisher Scientific | 25030-164 | |
| Magnessium Chloride Hexahydrate (MgCl2-6H2O) | MPBio | 2191421 | Tyrode's Buffer Component |
| Matlab | Mathworks | Curve Fitting Toolbox, Parallel Computing Toolbox, and Statistics and Machine Learning toolbox | |
| Methanol (CH3OH) | IBIS Scientific | MX0486-1 | Hazardous |
| Milli-Q water | |||
| Mix-n-Stain CF Dye Antibody Labeling Kits | Biotium | 92245 | Suggested conjugation kit |
| mPEG | Laysan Bio | mPEG-succinimidyl valerate, MW 5,000 | |
| N-(2-aminoethyl)-3-aminopropyltrimethoxysilane | UCT United Chemical | A0700 | Hazardous |
| Nanogrid | Miraloma Tech | ||
| NeutrAvidin Biotin Binding Protein | Thermo Fisher Scientific | 31000 | |
| Nitrogen (compressed gas) | |||
| NVIDIA GPU with CUDA | Look for compatibility at https://www.mathworks.com/help/parallel-computing/gpu-support-by-release.html | ||
| Olympus iX71 Microscope | Olympus | ||
| Parafilm M Sealing Film | The Lab Depot | HS234526C | |
| PBS pH 7.4 | Caisson Labs | PBL06 | |
| PC-200 Analog Hot Plate | Corning | 6795-200 | |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher Scientific | 15140-163 | |
| Phospho-EGF Receptor (Tyr1068) (1H12) Mouse mAb | Cell Signaling Technology | 2236BF | |
| Potassium Chloride (KCl) | Millipore Sigma | 529552 | Tyrode's Buffer Component |
| Potassium Hydroxide (KOH) | Millipore Sigma | 1050330500 | Hazardous |
| Premium PLA Filament, 1.75 mm diameter | Raise 3D | PMS:2035U/RAL:3028 | Printing temperature range: 205-235 °C |
| Pro2 3D printer | Raise 3D | ||
| Pyrex 1 L beaker | |||
| PYREX 100 mL storage bottles | Corning | 1395-100 | CH3OH/C3H6O reuse |
| Pyrex 250 mL beakers | |||
| Pyrex 4 L beaker | |||
| Quad-view Image Splitter | Photometrics | Model QV2 | |
| Refrigerated centrifuge | Eppendorf | EP-5415R | |
| RevCount Cell Counters, EVE Cell Counting Slides | VWR | 10027-446 | |
| Semrock emission filters: blue (445/45 nm), green (525/45 nm), red (600/37 nm), far-red (685/40 nm) | Semrock | LF405/488/561/635-4X4M-B-000 | |
| Serological pipette controller | |||
| Serological Pipettes | |||
| smite single molecule analysis package | https://github.com/LidkeLab/smite.git | ||
| Sodium Bicarbonate (NaHCO3) | Sigma Aldrich | S6014 | Hazardous |
| Sodium Borohydride (NaBH4) | Millipore Sigma | 452874 | Tyrode's Buffer Component |
| Sodium Chloride (NaCl) | Millipore Sigma | S9625 | Activate by successive heat and pH cycling |
| Sodium Hydroxide | VWR | BDH3247-1 | |
| Sodium Orthovanadate (Na3VO4) | Millipore Sigma | S6508 | Hazardous |
| Sulfuric Acid (H2SO4) | Millipore Sigma | 258105 | Hazardous |
| TetraSpeck Microspheres | Thermo Fisher Scientific | T7279 | multi-fluorescent beads |
| Tris (Trizma) base | Millipore Sigma | T1503 | |
| Trypan blue stain, 0.4% | Thermo Fisher Scientific | 15250061 | |
| Trypsin-EDTA 0.05% | Thermo Fisher Scientific | 25300120 |
Referências
- Lemmon, M. A., Schlessinger, J. Cell Signaling by Receptor Tyrosine Kinases. Cell. 141 (7), 1117-1134 (2010).
- Seet, B. T., Dikic, I., Zhou, M. M., Pawson, T. Reading protein modifications with interaction domains. Nature Reviews Molecular Cell Biology. 7 (7), 473-483 (2006).
- Coba, M. P., et al. Neurotransmitters drive combinatorial multistate postsynaptic density networks. Science Signaling. 2 (68), (2009).
- Gibson, S. K., Parkes, J. H., Liebman, P. A. Phosphorylation modulates the affinity of light-activated rhodopsin for g protein and arrestin. Biochemistry. 39 (19), 5738-5749 (2000).
- Stites, E. C., et al. Use of mechanistic models to integrate and analyze multiple proteomic datasets. Biophysical Journal. 108 (7), 1819-1829 (2015).
- Hause, R. J., et al. Comprehensive binary interaction mapping of SH2 domains via fluorescence polarization reveals novel functional diversification of ErbB receptors. PLOS ONE. 7 (9), 44471(2012).
- Lau, E. K., et al. Quantitative encoding of the effect of a partial agonist on individual opioid receptors by multisite phosphorylation and threshold detection. Science Signaling. 4 (185), (2011).
- Mishra, M., Tiwari, S., Gomes, A. V. Protein purification and analysis: next generation Western blotting techniques. Expert Review of Proteomics. 14 (11), 1037-1053 (2017).
- Brunner, A. M., et al. Benchmarking multiple fragmentation methods on an orbitrap fusion for top-down phospho-proteoform characterization. Analytical Chemistry. 87 (8), 4152-4158 (2015).
- Swaney, D. L., Wenger, C. D., Coon, J. J. Value of using multiple proteases for large-scale mass spectrometry-based proteomics. Journal of Proteome Research. 9 (3), 1323-1329 (2010).
- Curran, T. G., Zhang, Y., Ma, D. J., Sarkaria, J. N., White, F. M. MARQUIS: A multiplex method for absolute quantification of peptides and posttranslational modifications. Nature Communications. 6 (1), 1-11 (2015).
- Jain, A., et al. Probing cellular protein complexes using single-molecule pull-down. Nature. 473 (7348), 484-488 (2011).
- Jain, A., Liu, R., Xiang, Y. K., Ha, T. Single-molecule pull-down for studying protein interactions. Nature Protocols. 7 (3), 445-452 (2012).
- Kim, K. L., et al. Pairwise detection of site-specific receptor phosphorylations using single-molecule blotting. Nature Communications. 7 (1), 1-10 (2016).
- Salazar-Cavazos, E., et al. Multisite EGFR phosphorylation is regulated by adaptor protein abundances and dimer lifetimes. Molecular Biology of the Cell. 31 (7), 695(2020).
- Huyer, G., et al. Mechanism of inhibition of protein-tyrosine phosphatases by vanadate and pervanadate. Journal of Biological Chemistry. 272 (2), 843-851 (1997).
- Smith, P. K., et al. Measurement of protein using bicinchoninic acid. Analytical Biochemistry. 150 (1), 76-85 (1985).
- Keller, H. E. Objective lenses for confocal microscopy. Handbook of Biological Confocal Microscopy. , 145-161 (2006).
- Hendriks, C. L. L., van Vliet, L. J., Rieger, B., van Kempen, G. M. P., van Ginkel, M. Dipimage: a scientific image processing toolbox for MATLAB. , (1999).
- fitgeotrans: Fit geometric transformation to control point pairs. The MathWorks Inc. , Available from: https://www.mathworks.com/images/ref/fitgeotrans.html (2013).
- Raghavachari, N., Bao, Y. P., Li, G., Xie, X., Müller, U. R. Reduction of autofluorescence on DNA microarrays and slide surfaces by treatment with sodium borohydride. Analytical Biochemistry. 312 (2), 101-105 (2003).
- Wang, X., Park, S., Zeng, L., Jain, A., Ha, T. Toward single-cell single-molecule pull-down. Biophysical Journal. 115 (2), 283-288 (2018).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. Journal of Visualized Experiments. (86), e50549(2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados