Method Article
Un test de traction optimisé à molécule unique pour la quantification de la phosphorylation des protéines
Dans cet article
Résumé
Le présent protocole décrit la préparation des échantillons et l’analyse des données pour quantifier la phosphorylation des protéines à l’aide d’un test amélioré d’extraction à molécule unique (SiMPull).
Résumé
La phosphorylation est une modification post-traductionnelle nécessaire qui régule la fonction des protéines et dirige les résultats de la signalisation cellulaire. Les méthodes actuelles de mesure de la phosphorylation des protéines ne peuvent pas préserver l’hétérogénéité de la phosphorylation entre les protéines individuelles. Le test SiMPull (Single-Molecule Pull-Down) a été développé pour étudier la composition de complexes macromoléculaires par immunoprécipitation de protéines sur un couvercle en verre suivie d’une imagerie à molécule unique. La technique actuelle est une adaptation de SiMPull qui fournit une quantification robuste de l’état de phosphorylation des récepteurs membranaires pleine longueur au niveau de la molécule unique. L’imagerie de milliers de récepteurs individuels de cette manière permet de quantifier les modèles de phosphorylation des protéines. Le présent protocole détaille la procédure SiMPull optimisée, de la préparation de l’échantillon à l’imagerie. L’optimisation des protocoles de préparation du verre et de fixation des anticorps améliore encore la qualité des données. Le protocole actuel fournit un code pour l’analyse des données à molécule unique qui calcule la fraction des récepteurs phosphorylés dans un échantillon. Bien que ces travaux se concentrent sur la phosphorylation du récepteur du facteur de croissance épidermique (EGFR), le protocole peut être généralisé à d’autres récepteurs membranaires et molécules de signalisation cytosoliques.
Introduction
La signalisation associée à la membrane est réglée par une combinaison d’activation des récepteurs membranaires induits par les ligands et de recrutement de protéines accessoires en aval qui propagent le signal. La phosphorylation des tyrosines clés dans les queues cytoplasmiques des récepteurs est essentielle pour initier la formation de complexes de signalisation, ou signalosomes 1,2. Par conséquent, une question importante en biologie est de savoir comment les modèles de phosphorylation sont créés et maintenus pour recruter des partenaires de signalisation et dicter les résultats cellulaires. Cela comprend la compréhension de l’hétérogénéité de la phosphorylation des récepteurs, à la fois en abondance et dans les modèles spécifiques de phosphotyrosine qui peuvent fournir un moyen de manipuler les sorties de signalisation en dictant la composition du signalosome 3,4,5,6,7. Cependant, il existe des limites dans les méthodes actuelles pour interroger la phosphorylation des protéines. L’analyse par transfert de Western est excellente pour décrire les tendances de la phosphorylation des protéines, mais elle est semi-quantitative8 et ne fournit pas d’informations sur l’hétérogénéité du système, car des milliers à des millions de récepteurs sont moyennés ensemble. Bien que les transferts de Western permettent de sonder un échantillon à l’aide d’anticorps spécifiques aux phosphos contre des tyrosines spécifiques, ils ne peuvent pas fournir d’informations sur les modèles de phosphorylation multisite au sein d’une même protéine. La phosphoprotéomique quantitative rend compte de l’abondance de la phosphotyrosine, mais il existe des limites à la détection de la phosphorylation multisite, car les résidus d’intérêt doivent être situés dans le même peptide (généralement 7-35 acides aminés) qui est généré par la digestion enzymatique 9,10,11.
Pour surmonter les limites mentionnées ci-dessus, le test SiMPull (Single-Molecule Pull-Down) a été adapté pour quantifier les états de phosphorylation des récepteurs intacts au niveau de la molécule unique. SiMPull a été démontré pour la première fois comme un outil puissant pour interroger les complexes macromoléculaires par Jain et al.12,13. Dans SiMPull, les complexes macromoléculaires ont été immunoprécipités (IP) sur des couvercles de verre fonctionnalisés par anticorps, puis analysés par microscopie à molécule unique pour le nombre de sous-unités protéiques et la co-IP avec des composants complexes12. Une modification de Kim et al.14, appelée SiMBlot, a été la première à utiliser une variante de SiMPull pour analyser la phosphorylation des protéines dénaturées. Le protocole SiMBlot repose sur la capture de protéines de surface cellulaire biotinylées à l’aide de couvercles recouverts de NeutrAvidin, qui sont ensuite sondés pour la phosphorylation avec un marquage d’anticorpsspécifiques au phospho 14. Malgré ces progrès, des améliorations étaient nécessaires pour rendre la quantification de la modification post-traductionnelle plus robuste et applicable à un plus large éventail de protéines.
Le présent protocole décrit une approche SiMPull optimisée qui a été utilisée pour quantifier les modèles de phosphorylation du récepteur du facteur de croissance épidermique intact (EGFR) en réponse à une gamme de conditions de ligand et de mutations oncogènes15. Bien que ces travaux se concentrent sur l’EGFR, cette approche peut être appliquée à n’importe quel récepteur membranaire et protéine cytosolique d’intérêt (POI), pour lesquels des anticorps de qualité sont disponibles. Le protocole comprend des étapes pour réduire l’autofluorescence des échantillons, une conception de réseau d’échantillons qui nécessite un volume d’échantillon minimal avec la préparation simultanée de jusqu’à 20 échantillons, et l’optimisation des conditions de marquage et de fixation des anticorps. Des algorithmes d’analyse de données ont été développés pour la détection et la quantification de protéines phosphorylées à molécule unique.
Protocole
1. Préparation du couvercle
REMARQUE: Pour cette étape, il faut porter un équipement de protection individuelle (EPI), qui comprend une double couche de gants en nitrile, des lunettes de sécurité ou un écran facial, et une blouse de laboratoire.
- Effectuez une gravure au piranha pour éliminer les débris organiques du verre.
ATTENTION: La solution de piranha est un agent oxydant puissant qui est corrosif et très réactif au contact de matières organiques. La réaction avec les débris organiques est exothermique et potentiellement explosive. Ainsi, la procédure doit être effectuée dans une hotte chimique avec le châssis abaissé. La verrerie Pyrex est nécessaire pour manipuler la solution de piranha.- Préparez l’espace de travail à l’intérieur d’une hotte chimique. Disposer les couvercles sans chevauchement dans le fond d’un bécher en verre de 4 L, le bécher « réactionnel », et placer le bécher réactionnel sur une plaque chauffante à feu doux. Laissez la verrerie chauffer pendant 10 min. Placer un bécher en verre « déchets » de 1 L avec 500 mL de ddH2O proximal au bécher réactionnel.
- Ajouter lentement 49 mL d’acide sulfurique 12 N (H2SO4) au bécher réactionnel à l’aide d’une pipette sérologique en verre. Rincez la pipette dans le bécher avant de l’éliminer.
- Ajouter 21 mL de peroxyde d’hydrogène à 30 % (H2 O2) goutte à goutte avec une pipette sérologique en verre au bécher réactionnel. Répartir lentement les gouttelettes H2O2 uniformément sur le fond de la fiole réactionnelle pour éviter la trempe localisée de la réaction de gravure du piranha. Rincez la pipette dans le bécher avant de l’éliminer.
ATTENTION : Ajoutez toujours H2O2 dans le H2SO4, et jamais l’inverse. - Piranha graver les couvercles pendant 30 min. Agiter doucement le contenu du bécher réactionnel toutes les 5 minutes.
- Trempez la solution de piranha en versant le contenu du bécher à déchets dans le bécher réactionnel. Transférez le liquide lentement le long de la paroi du bécher de réaction pour minimiser les éclaboussures. Retirez le bécher réactionnel de la plaque chauffante.
- Lorsque la réaction est trempée et refroidie, versez la solution de piranha dans le bécher à déchets pour la neutralisation sans retirer les couvercles gravés du bécher réactionnel.
- Neutraliser la solution de piranha avec l’ajout progressif d’une base faible. Par exemple, utilisez une masse excessive de 20 g de bicarbonate de sodium (NaHCO3)/100 mL de solution de piranha.
ATTENTION : Ne conservez pas les solutions de piranha dans des conteneurs à déchets scellés. La solution doit toujours être neutralisée avant d’être éliminée. La réaction de neutralisation produit des bulles vigoureuses et peut être explosive si elle n’est pas contrôlée par l’ajout progressif de la base faible. - Remuer la solution neutralisée avec une tige d’agitation en verre et laisser réagir pendant 2 h. Augmentez le pH à >4 et éliminez la solution.
- Entre les ajouts de base faible à la solution de piranha, transférer les couvercles gravés du bécher réactionnel dans un entonnoir Buchner avec une tige d’agitation en verre et rincer pendant 5 min en ddH2O.
REMARQUE: Passez immédiatement à l’étape suivante ou conservez les couvercles gravés au piranha jusqu’à 2 semaines dans ddH2O dans un bocal en verre scellé ou une boîte de Petri (envelopper avec un film d’étanchéité, voir Tableau des matériaux).
- Bain-soniquer les couvercles dans des solvants organiques en suivant les étapes ci-dessous.
- Placez les couvercles dans un bocal Coplin en verre (voir Tableau des matériaux) et recouvrez de méthanol (CH3OH). Scellez le couvercle au pot avec un film d’étanchéité et soniquez le bain pendant 10 min. Versez soigneusement le méthanol du pot Coplin dans une bouteille de stockage en verre.
- Remplissez le pot Coplin avec de l’acétone (C3H6O), scellez le couvercle et faites du bain sonique pendant 10 min. Versez soigneusement l’acétone du pot Coplin dans une bouteille de stockage en verre.
ATTENTION : Le méthanol est inflammable et extrêmement toxique. Utilisation dans une hotte chimique. L’acétone est inflammable et irritante. Par conséquent, manipulez-le et stockez-le dans du verre et utilisez-le dans une hotte à fumée chimique. Éliminer les déchets dangereux conformément aux réglementations et directives locales.
REMARQUE: Le méthanol et l’acétone peuvent être réutilisés jusqu’à cinq fois chacun.
- Activez la surface du couvercle pour la fonctionnalisation du silane.
- Bain-sonicate avec 1 M d’hydroxyde de potassium (KOH) pendant 20 min. Versez soigneusement le KOH hors du pot Coplin dans un tube conique de 50 mL pour le réutiliser.
ATTENTION : Le KOH est corrosif et irritant. Utiliser dans une hotte à fumée chimique et ne pas stocker dans du verre. Conservez-le dans des tubes en polypropylène. Éliminer les déchets dangereux conformément aux réglementations et directives locales.
REMARQUE: KOH peut être réutilisé jusqu’à cinq fois. - Rincez deux fois avec ddH2O. Égouttez ddH2O des couvercles, puis chauffez chaque couvercle en agitant à travers la flamme d’un brûleur Bunsen pour éliminer toute humidité de surface. Placez les couvercles dans un pot Coplin sec.
- Bain-sonicate avec 1 M d’hydroxyde de potassium (KOH) pendant 20 min. Versez soigneusement le KOH hors du pot Coplin dans un tube conique de 50 mL pour le réutiliser.
- Effectuer l’aminosilanisation coverslip.
- Préparer la solution d’aminosilane en mélangeant 69,4 mL de méthanol avec 3,6 mL d’acide acétique (CH3COOH) dans une fiole conique. Ajouter 720 μL de N-(2-aminoéthyl)-3-aminopropyltriméthoxysilane (aminosilane) et bien mélanger (voir tableau des matériaux).
ATTENTION : L’acide acétique est inflammable et corrosif. Manipuler avec des pipettes en verre et stocker dans du verre. Travailler avec de l’acide acétique dans une hotte chimique. L’aminosilane est un danger d’inhalation extrêmement toxique, un sensibilisant et un irritant. Il est nocif pour la vie aquatique. Utilisation dans une hotte chimique. Éliminer les produits chimiques en tant que déchets dangereux conformément aux réglementations et directives locales.
REMARQUE: Aminosilane est photosensible et s’hydrolyse rapidement dans l’eau. Toutes les étapes avec ce réactif doivent être effectuées dans des conditions d’éclairage minimales pour conserver l’activité. Purgez la bouteille avec de l’azote gazeux et appliquez un film d’étanchéité avant de le stocker dans un dessiccateur sombre. Remplacer tous les 6-9 mois. - Ajouter immédiatement la solution d’aminosilane dans le pot Coplin. Couvrir et appliquer le film d’étanchéité en continuant à protéger de la lumière.
- Incuber les couvercles dans la solution d’aminosilane pendant 10 min dans l’obscurité à température ambiante (RT). Bain-soniquer pendant 1 min, puis incuber pendant encore 10 min. Versez soigneusement la solution d’aminosilane dans un récipient à déchets désigné pour CH3OH avec trace aminosilane et CH3COOH.
- Rincez les couvercles avec du méthanol et versez la solution dans un récipient à déchets destiné au méthanol.
- Rincez les couvercles trois fois pendant 2 minutes chacun avec ddH2O. Égouttez les couvercles, tamponnez l’excès d’humidité et séchez complètement à l’air libre pendant 10 minutes.
- Préparer la solution d’aminosilane en mélangeant 69,4 mL de méthanol avec 3,6 mL d’acide acétique (CH3COOH) dans une fiole conique. Ajouter 720 μL de N-(2-aminoéthyl)-3-aminopropyltriméthoxysilane (aminosilane) et bien mélanger (voir tableau des matériaux).
- Effectuer la préparation de la matrice/ fonctionnalisation biotine-PEG des couvercles.
- Préparer 1 M de NaHCO3 (pH 8,5) sur un bouillon de travail en dissolvant 84,5 mg de NaHCO3 en 1 mL de ddH2O. Pour une concentration finale de 10 mM de NaHCO3, diluer 1 M de NaHCO3 en ddH2O (1:100).
- Dessinez un réseau de grilles sur les couvercles aminosilanisés secs avec un stylo barrière hydrophobe (voir Tableau des matériaux) et attendez que l’encre sèche. Écrivez un identificateur sur le couvercle pour marquer l’orientation appropriée. Placez les couvercles dans une chambre humidifiée.
REMARQUE: Le réseau doit être composé de 16 à 20 carrés, d’une taille d’environ 4 mm x 4 mm. - Pour fabriquer la solution de valérate de succinimidyle biotine-PEG (biotine-PEG)/mPEG valérate de succinimidyle (mPEG), il faut d’abord retirer le mPEG et le biotin-PEG (voir Tableau des matériaux) du congélateur et équilibrer à RT. Ajouter 153 mg de mPEG et 3,9 mg de biotine-PEG (~1:39 biotine-PEG:mPEG) dans un tube de microcentrifugation de 1,5 mL, et remettre en suspension dans 609 μL de 10 mM NaHCO3 par pipetage doux. Centrifuger à 10 000 x g pendant 1 min à RT pour éliminer les bulles.
REMARQUE: La demi-vie d’hydrolyse du valérate de succinimidyle dans un tampon de pH 8,5 est d’environ 30 min. Après avoir ajouté la mémoire tampon à mPEG, procédez comme suit le plus rapidement possible. Cette étape est essentielle. - Appliquez la solution de biotine-PEG/mPEG pour couvrir complètement chaque carré dans les réseaux de couvercles, généralement 10-15 μL par carré. Ne laissez pas le liquide déborder de l’espace défini. Conservez les couvercles dans une chambre d’humidité dans l’obscurité pendant 3-4 h à RT.
- Lavez les couvercles avec de grandes quantités d’eau en les trempant séquentiellement dans 3 béchers en verre de 250 mL remplis de ddH2O pendant 10 s chacun.
- Éliminez toute l’humidité des couvercles avec de l’azote gazeux. Conservez les couvercles dos à dos dans un tube conique rempli d’azote de 50 mL enveloppé d’un film d’étanchéité à -20 °C.
REMARQUE: Passez immédiatement à l’étape 2 ou conservez les couvercles à -20 ° C pendant 1 semaine avant utilisation.
2. Préparation du lysat de SiMPull
ATTENTION : Les EPI requis pour les autres étapes du protocole sont des gants en nitrile, des lunettes de sécurité et des blouses de laboratoire.
REMARQUE: Les lysats ont été préparés à partir des cellules CHO adhérentes exprimant l’EGFR-GFP. Les cellules ont été plaquées dans une boîte de culture tissulaire de 60 mm (TC60) pendant la nuit12,13. Les cellules CHO ont été cultivées dans du DMEM complété par 10 % de sérum bovin fœtal, 1 % de L-glutamine, 1 % de pénicilline-streptomycine et 500 ng/mL de généticine (voir tableau des matériaux). D’autres lignées cellulaires adhérentes ou cellules de suspension peuvent également être utilisées.
- Plaquez les cellules en suivant les étapes ci-dessous.
- Laver la boîte de culture (contenant les cellules) avec 1 mL de 1x PBS. Ajouter 1 mL de 1x trypsine et incuber pendant 5 min à 37 °C pour détacher les cellules. À l’aide d’une pipette, transférer les cellules détachées de la boîte dans un tube centrifuge de 1,5 mL.
- Prendre 10 μL de bleu de trypan et mélanger avec 10 μL de suspension cellulaire dans un tube de centrifugeuse séparé. Compter les cellules en utilisant 10 μL du mélange cellulaire dans un compteur de cellules automatique, conformément aux instructions du fabricant (voir tableau des matériaux).
- Plaque 8 x 105 cellules pendant la nuit dans une boîte de Pétri TC60. Assiette un plat par condition.
NOTE: Pour la présente étude, les cellules n’ont pas été traitées ou traitées avec du pervanadate et de l’EGF comme décrit à l’étape 2.4.1.
- Préparez les solutions suivantes pour la préparation du lysat cellulaire.
- Préparez 1x PBS glacé (pH 7,4).
- Préparer un tampon de lyse, une solution de détergent non ionique et non dégénérant à 1 % (voir tableau des matériaux) dans 50 mM de Tris-HCl (pH 7,2) et 150 mM de NaCl complété par un inhibiteur de la protéase/phosphatase (IPP) (1:100 à partir du stock). Placer le tube sur un nutateur et laisser le tampon mélanger pendant 15 min. Gardez la solution préparée sur la glace.
- Préparer le tampon du Tyrode pour une concentration finale de 135 mM de NaCl, 10 mM de KCl, 0,4 mM deMgCl2, 1 mM de CaCl2, 10 mM de HEPES (pH 7,2), 20 mM de glucose et 0,1 % d’albumine sérique bovine (BSA) (voir tableau des matériaux). Réchauffer la solution à 37 °C.
- Préparer le témoin positif pour le traitement par phosphorylation - 1 mM de pervanadate.
REMARQUE : cette étape est facultative. Le pervandate est la forme peroxydée du vanadate, un inhibiteur de la protéine tyrosine phosphatases16. La prévention de la déphosphorylation des protéines en inhibant l’activité de la phosphatase donne un échantillon hautement phosphorylé.- Préparer un stock de 200 mM d’orthovanadate de sodium activé (Na3VO4).
- Pour préparer une solution de 100 mL, ajouter 3,89 g de Na3VO4 (voir tableau des matériaux) à 90 mL de ddH2O et dissoudre en remuant. Ajustez le pH à 10 en ajoutant HCl ou NaOH goutte à goutte. L’ajout de HCl rendra la solution jaune.
- Porter le volume à 100 mL avec ddH2O. Faire bouillir la solution en la chauffant au micro-ondes. Après ébullition, la solution sera incolore.
- Refroidir la solution à RT et réajuster le pH à 10. Répétez l’ébullition, le refroidissement et l’ajustement du pH deux à quatre fois de plus, jusqu’à ce que le pH se stabilise à 10. Aliquote et conserver à -20 °C.
- Préparer un stock de 30 mM de pervanadate (PV) en mélangeant 20,4 μL de 3 %H2O2 avec 100 μL de 200 mM Na3VO4 et 546,8 μL de ddH2O (concentrations équimolaires deH2O2 et de Na3VO4 activé). Incuber dans le noir à RT pendant 15 min.
- Préparez 1 mM de PV dans le tampon de Tyrode. Pour une solution de 10 mL, ajouter 0,33 mL de stock PV de 30 mM à 9,67 mL de tampon de Tyrode à 37 °C. Traiter les cellules immédiatement.
- Lavez les cellules une fois avec 3 mL de tampon de Tyrode. Ajouter 3 mL de PV de 1 mM dans le tampon du Tyrode aux cellules et incuber pendant 15 min à 37 °C.
- Préparer un stock de 200 mM d’orthovanadate de sodium activé (Na3VO4).
- Effectuez la stimulation du ligand.
- Stimuler les cellules avec le ligand d’intérêt en utilisant la concentration, le temps et la température appropriés. Pour une stimulation maximale de l’EGFR, incuber avec 1 mL de facteur de croissance épidermique (EGF, voir tableau des matériaux) + 1 mM de PV dans le tampon de Tyrode pendant 5 min à 37 °C.
- Effectuer une lyse cellulaire.
- Après le traitement cellulaire souhaité, placez le plat sur de la glace et lavez-le avec du PBS glacé 1x. Retirez complètement le volume total de PBS à l’aide d’une pipette.
- Ajouter 180 μL de tampon de lyse (étape 2.2.2) à la plaque. Utilisez un grattoir à cellules pour tirer le tampon autour de la plaque afin de couvrir complètement les cellules. Appliquez une pression ferme et constante avec le grattoir à cellules sur toute la surface cultivée pour lyser complètement les cellules.
REMARQUE: Le volume du tampon de lyse doit être maintenu au minimum pour assurer une concentration élevée en protéines. - Pipeter les cellules lysées et les transférer dans un tube de 1,5 mL. Gardez le tube sur la glace pendant 30 min. Vortex les lysats toutes les 5 min.
REMARQUE: Si le POI se compose de plusieurs sous-unités ou est sensible à la dissociation, ne pas vortexer les lysats. - Centrifuger les cellules lysées à 16 000 x g pendant 20 min à 4 °C. Transférer le surnageant dans un nouveau tube de 1,5 mL à l’aide d’une pipette. Celui-ci contient le lysat protéique total.
- Réservez 10 μL du lysat et diluez-le dans 90 μL du tampon de lyse pour l’analyse colorimétrique bicinchoninique (BCA)17. Conserver le lysat protéique total restant à -80 °C.
- Déterminer la concentration de protéines totales de lysat à l’aide de l’analyse BCA (voir tableau des matériaux).
REMARQUE: Les lysats protéiques totaux peuvent être préparés le jour de l’expérience et utilisés frais ou stockés comme aliquotes à usage unique à -80 ° C pendant 12 semaines. Ne pas congeler/décongeler.
3. Fonctionnalisation du réseau avec l’anticorps biotinylé
- Préparez les solutions suivantes.
- T50 Buffer, une solution de 10 mM de Tris-HCl (pH 8,0) et de 50 mM de NaCl. La solution est stable pendant 1 mois à RT.
- T50-BSA en complétant le tampon T50 avec 0,1 mg/mL de BSA. Gardez la solution préparée sur la glace.
- 10 mg/mL de borohydrure de sodium (NaBH4) dans 1x PBS. Préparez-le immédiatement avant utilisation.
- 0,2 mg/mL de NeutrAvidin (voir tableau des matériaux) dans le tampon T50.
ATTENTION : NaBH4 est un agent réducteur et est inflammable. Purgez toujours le récipient avec de l’azote gazeux après utilisation et stockez-le dans un dessiccateur.
- Fonctionnaliser le réseau avec l’anticorps biotinylé.
- Retirez les matrices fonctionnalisées PEG-biotine du congélateur et équilibrez le tube conique en RT avant de l’ouvrir. Placez le couvercle avec le réseau orienté vers le haut sur un film d’étanchéité doublé de 100 mm de boîte de culture tissulaire (TC100).
REMARQUE: Minimisez l’éclairage aérien. Toutes les solutions doivent « perler » sur les carrés définis par le réseau hydrophobe. Ajouter un volume approprié de solution pour couvrir complètement chaque carré (généralement 10-15 μL) et ne pas laisser le liquide déborder de l’espace défini. Pour éliminer rapidement les liquides, utilisez une conduite de vide interne fixée à une fiole à vide pour capturer les déchets. Laisser le NaBH4 dégazer pendant 1 h avant l’élimination en laissant le tube ouvert dans la hotte de fumée chimique. Le traitement NaBH4 est nécessaire pour réduire l’autofluorescence de fond, réduisant ainsi les détections de molécules uniques faussement positives. - Traiter chaque carré de la matrice avec 10 mg/mL de NaBH4 dans 1x PBS pendant 4 min à TA. Laver trois fois avec PBS.
- Incuber chaque carré pendant 5 min avec 0,2 mg/mL de NeutrAvidin dans la T50. Laver trois fois avec T50-BSA.
REMARQUE: NeutrAvidin se lie à la BIOTINE PEG et fournit un site de liaison pour les anticorps biotinylés 12,13,15. - Incuber chaque carré pendant 10 min avec 2 μg/mL d’anticorps biotinylés spécifiques au POI dans le T50-BSA; laver trois fois avec du T50-BSA.
REMARQUE : Le présent protocole utilise des IgG anti-EGFR biotinylées (voir tableau des matériaux) pour capturer l’EGFR-GFP.
- Retirez les matrices fonctionnalisées PEG-biotine du congélateur et équilibrez le tube conique en RT avant de l’ouvrir. Placez le couvercle avec le réseau orienté vers le haut sur un film d’étanchéité doublé de 100 mm de boîte de culture tissulaire (TC100).
4. SiMPull de POI à partir de lysats de cellules entières
REMARQUE: Placez la parabole TC100 des matrices SiMPull fonctionnalisées sur de la glace pour le reste de la préparation SiMPull. Cette étape est l’extraction d’un POI à partir du lysat protéique total. Le lysat ne doit pas être réutilisé après décongélation.
- Préparez les solutions suivantes.
- Préparer 4 % de paraformaldéhyde (PFA)/0,1 % de glutaraldéhyde (GA) dans 1x PBS.
ATTENTION : Les PFA et les AG sont des fixateurs chimiques toxiques et des cancérogènes potentiels. Portez de l’EPI. Éliminer les produits chimiques en tant que déchets dangereux conformément aux réglementations et directives locales. - Préparer 10 mM de Tris-HCl, pH 7,4.
- Préparer 4 % de paraformaldéhyde (PFA)/0,1 % de glutaraldéhyde (GA) dans 1x PBS.
- Décongeler et mélanger le lysat en pipetant doucement de haut en bas. Restez sur la glace.
- Diluer 1 μL du lysat dans 100 μL de T50-BSA/IPP glacé.
REMARQUE: Si nécessaire, déterminez le facteur de dilution approprié du lysat protéique total en appliquant une gamme de dilutions à la matrice. La densité optimale des récepteurs SiMPull par zone de réseau est de 0,04-0,08/μm2. Les dilutions de lysat peuvent être évaluées à l’étape 6 (Analyse des données). - Incuber le lysat sur le tableau pendant 10 min; puis laver quatre fois avec du T50-BSA/IPP glacé.
- Diluer l’anticorps anti-phosphotyrosine conjugué AF647 (voir tableau des matériaux) dans du T50-BSA/IPP glacé et incuber sur la matrice pendant 1 h.
NOTE: Dans le présent protocole, un pan anti-pTyr (PY99)-AF647 IgG est utilisé pour identifier la population phosphorylée d’EGFR-GFP. L’utilisation d’anticorps directement marqués élimine le besoin d’anticorps secondaires, augmentant les options de marquage et améliorant la cohérence des résultats. Les anticorps marqués par fluorescence peuvent être obtenus à partir de sources commerciales. S’ils ne sont pas disponibles dans le commerce, les anticorps peuvent être étiquetés sur mesure à l’aide de techniques de bioconjugation standard, et des kits de bioconjugation commerciaux sont disponibles. Chaque lot d’anticorps marqués par fluorescence doit être testé pour des conditions de marquage optimales en effectuant SiMPull pour mesurer une courbe de dose et trouver le point de saturation. - Laver six fois avec du T50-BSA glacé pendant un total de 6 à 8 min.
- Laver deux fois avec du PBS glacé 1x.
- Incuber le réseau avec une solution à 4 % de PFA/0,1 % d’AG pendant 10 min pour éviter la dissociation des anticorps.
- Laver deux fois pendant 5 min chacun avec 10 mM de Tris-HCl, pH 7,4/PBS pour inactiver les fixateurs.
REMARQUE : Pour les expériences utilisant plus d’un anticorps (p. ex., détection de plusieurs sites de phosphotyrosine), répétez les étapes 4.5 à 4.9. Voir l’étape 6.2.9 pour obtenir des renseignements sur la détermination de l’obstacle stérique entre deux anticorps.
5. Acquisition d’images
REMARQUE: L’acquisition d’images à molécule unique est effectuée à l’aide d’un objectif TIRF 150x et d’un séparateur d’images qui capture chaque canal spectral dans un quadrant spécifique de la caméra EMCCD (voir Tableau des matériaux). Les images d’étalonnage sont d’abord acquises pour permettre l’enregistrement des canaux et l’étalonnage du gain de la caméra avec une grille d’alignement des canaux nanomotifs (nanogrilles) qui contient 20 x 20 réseaux de trous de 200 ± 50 nm à une distance intra-trou de 3 ± 1 μm (taille totale ~ 60 μm × 60 μm).
- Effectuez l’enregistrement du canal en suivant les étapes ci-dessous.
REMARQUE: Un enregistrement précis des canaux est nécessaire pour calculer correctement la colocalisation des émetteurs. Cette étape est essentielle.- Nettoyez l’objectif d’huile et déposez une goutte d’huile sur l’objectif. Placez le nanogrille sur la scène pour l’imagerie. À l’aide de la lumière blanche transmise, concentrez-vous sur le motif de la grille.
REMARQUE: Les images avec le nanoréseau sont acquises à l’aide de la lumière transmise, qui traverse le nanoréseau et est détectée dans tous les canaux spectraux. Alternativement, on peut utiliser des perles multifluorescentes qui émettent de la fluorescence détectée dans chaque canal. L’acquisition d’images devra être optimisée en fonction de chaque configuration de microscope. - Acquérir une série de 20 images de la grille. Assurez-vous que les pixels ne sont pas saturés. Enregistrez la série d’images sous le nom « Fiducial ».
- Décentrez le nanogrille pour créer un motif Airy18. Acquérez une série de 20 images pour des étalonnages de gain. Enregistrez l’image sous le nom « Gain ».
- Acquérir une série de 20 images pour le décalage de la caméra en empêchant toute la lumière d’aller à la caméra. Enregistrez l’image en tant que « Arrière-plan ».
- Nettoyez l’objectif d’huile et déposez une goutte d’huile sur l’objectif. Placez le nanogrille sur la scène pour l’imagerie. À l’aide de la lumière blanche transmise, concentrez-vous sur le motif de la grille.
- Acquérir des images SiMPull.
REMARQUE : Avant d’imager la matrice de couvercles, échangez la solution Tris contre T50-BSA et équilibrez la matrice sur RT.- Nettoyez l’objectif d’huile et déposez de l’huile supplémentaire sur l’objectif. Fixez le réseau de couvercles sur l’étage du microscope.
- Optimisez la puissance d’excitation de chaque fluorophore, l’angle TIRF et le temps d’intégration de la caméra. L’objectif est d’obtenir le rapport signal-bruit le plus élevé tout en minimisant le photoblanchiment de l’échantillon. Enregistrez la puissance du laser pour plus de cohérence dans les mesures futures.
REMARQUE: La présente étude a utilisé un temps d’exposition de 300 ms pour le canal rouge lointain et de 1 s pour le canal vert. Le laser de 642 nm a été utilisé à une puissance laser d’environ 500 μW, tandis que le laser de 488 nm a été utilisé à 860 μW, mesuré avant la lentille du tube. - Acquérir des images pour chaque échantillon. Imagez d’abord le canal rouge lointain, suivi de chaque fluorophore de longueur d’onde inférieure pour réduire le photoblanchiment. En raison du faible volume utilisé pour chaque échantillon, vérifiez le niveau de tampon toutes les 30 à 45 minutes et réapprovisionnez-le au besoin.
6. Analyse des données
- Téléchargez les codes de démonstration.
REMARQUE : Le code de démonstration fourni et les exemples d’ensembles de données illustrent le flux de travail complet de l’analyse des données (fichiers de codage supplémentaires 1 à 4). La configuration système requise répertoriée dans SiMPullMain.m se trouve dans le fichier de codage supplémentaire 1.-
Décompressez et enregistrez dans un répertoire personnel Documents/MATLAB (MacOS/Linux) ou Documents\MATLAB (Windows).
REMARQUE: Cela génère quatre nouveaux dossiers: SiMPull_class, smite, Sample Data, Sample Analysis Outputs. - Ouvrez le fichier « ReadMe_Setup.txt » qui se trouve dans le dossier SiMPull_class.
- Installez MATLAB et MATLAB Toolboxes : Curve Fitting Toolbox, Parallel Computing Toolbox et Statistics and Machine Learning toolbox.
- Installez DipImage19 conformément aux instructions de téléchargement.
- Installez le package d’analyse monomoléculaire « smite » comme décrit dans ReadMe_setup.txt.
REMARQUE: « smite » est disponible sur le référentiel GitHub (voir Table des matériaux). - Ouvrez SiMPullMain.m, qui se trouve dans SiMPull_class dossier, en faisant glisser le fichier dans la fenêtre MATLAB.
- Remplacez le répertoire par ...\MATLAB\Sample Data\ en cliquant sur l’icône Rechercher un dossier et en sélectionnant le dossier.
-
Décompressez et enregistrez dans un répertoire personnel Documents/MATLAB (MacOS/Linux) ou Documents\MATLAB (Windows).
- Vue d’ensemble des étapes de traitement des données
- Exécutez SiMPullMain.m - suivez les instructions pour chaque section. Exécutez chaque section individuellement en plaçant le curseur dans cette section et en cliquant sur l’icône Exécuter la section.
REMARQUE : Les étapes générales de l’analyse des données sont décrites dans cette section. Vous trouverez des instructions détaillées dans le code SiMPullMain.m qui l’accompagne. - Exécutez la section « Initialisation » pour définir le chemin à suivre pour définir les canaux spectraux et la taille de l’image.
- Exécutez la section « Rechercher le gain et le décalage de la caméra » pour convertir la gain de la caméra en photons à l’aide des jeux de données Gain et Arrière-plan.
- Exécutez la section « Enregistrement du canal » pour calculer la transformation moyenne pondérée locale utilisée pour l’enregistrement de l’image.
- Formatez et organisez les données. Exécutez la section « Joindre des canaux séquentiels dans une image quadruple ». Exécutez « Supprimer les images incorrectes ».
- Exécutez la section « Ajuster les molécules uniques et trouver les molécules qui se chevauchent ».
REMARQUE: Cette section exécute plusieurs fonctions pour localiser des molécules uniques dans chaque canal et déterminer les événements de colocalisation entre les canaux spectraux. - Pour déterminer le nombre minimal de photons par ajustement GFP réel, exécutez le « Optimiser le seuil minimal de photons ». Il s’agit d’un processus itératif.
- Tout d’abord, définissez smf. Thresholding_MinPhotons = [0, 0, 0] et exécutez la section. Sélectionnez les fichiers « Données vides » lorsque vous y êtes invité. Répétez l’opération avec les fichiers « CHO-EGFR-GFP ».
- Sélectionnez une valeur seuil minimale appropriée en comparant les deux histogrammes. Définissez smf. Thresholding_MinPhotons = [475, 0, 0] et exécutez à nouveau la section.
- Exécutez la section « Calculer le pourcentage de GFP correspond positivement au signal FR » pour corriger les localisations en arrière-plan et calculer les valeurs finales.
- Exécutez l’option 1 (étape 6.2.10) ou l’option 2 (étape 6.2.11) selon les besoins expérimentaux.
- Option 1 : Corriger le nombre de récepteurs disponibles au niveau de la membrane plasmique pour la liaison aux ligands, comme décrit dans la référence15.
- Tout d’abord, étiqueter les récepteurs de surface avec des niveaux saturants de colorant fluorescent NHS Ester (NHS-AF647, voir Table des matériaux). Effectuez ensuite une expérience SiMPull pour déterminer le pourcentage de localisation GFP qui colocalise avec AF647.
REMARQUE: Cela fournit une estimation de la fraction de récepteurs disponibles pour le marquage NHS et du rapport des récepteurs à la surface (SR). - Appliquez la correction SR dans le calcul final : NGFP = (NLOC - NBG)*SR, où NGFP est le nombre corrigé de localisations GFP, NBG est le numéro de localisation d’arrière-plan et NLOC est le nombre total de localisations.
NOTE: Dans le présent exemple, cette correction n’est pas appliquée car le pervanadate est perméable à la membrane16 et, par conséquent, l’action de l’inhibition de la phosphatase ne se limite pas aux récepteurs de surface.
- Tout d’abord, étiqueter les récepteurs de surface avec des niveaux saturants de colorant fluorescent NHS Ester (NHS-AF647, voir Table des matériaux). Effectuez ensuite une expérience SiMPull pour déterminer le pourcentage de localisation GFP qui colocalise avec AF647.
- Option 2 : Pour les mesures de phosphorylation multisite, tenez compte du potentiel d’entrave stérique lorsque deux anticorps sont utilisés.
REMARQUE: L’obstacle stérique peut entraîner une réduction du pourcentage observé de récepteurs phosphorylés pour l’anticorps 1 en présence de l’anticorps 2 (P12), par rapport à l’anticorps 1 seul (P1).- Utilisez SiMPull pour déterminer P1 et P12 et calculer le facteur de correction de l’obstacle stérique, conformément à la référence15 publiée précédemment.
- Exécutez SiMPullMain.m - suivez les instructions pour chaque section. Exécutez chaque section individuellement en plaçant le curseur dans cette section et en cliquant sur l’icône Exécuter la section.
Résultats
Un dessin animé illustrant le processus SiMPull est illustré à la figure 1A. Les couvercles sont fonctionnalisés à l’aide de NeutrAvidin comme point d’ancrage pour les anticorps anti-EGFR biotinylés afin de capturer l’EGFR-GFP à partir de lysats de protéines totales. Après avoir éliminé les protéines non liées, les récepteurs phosphorylés sont marqués avec un anticorps anti-phosphotyrosine (anti-PY)15. La figure 1B montre une image du réseau hydrophobe, où plusieurs échantillons peuvent être préparés et imagés sur la même couverture. L’un des avantages de ce porte-échantillon est que des volumes d’échantillon minimaux d’environ 10 μL sont nécessaires. Le couvercle peut être imagé en le plaçant directement sur la scène du microscope. Cependant, il est utile de stabiliser le couvercle en utilisant un porte-couvercle. Le porte-couvercle illustré à la figure 1B a été créé à l’aide d’une imprimante 3D et le plan est fourni dans le fichier de codage supplémentaire 5. L’autofluorescence de l’encre hydrophobe est un guide utile pour trouver le plan focal de l’échantillon (Figure 1C). Un exemple d’image brute multicanal est illustré à la figure 1D. Une superposition des canaux verts bruts et rouge lointain est illustrée à la figure 1E.
La figure 2 illustre le flux de travail d’analyse et fournit des données représentatives. L’acquisition de données commence d’abord par l’acquisition de fiducials pour l’enregistrement des canaux, qui sont utilisés pour superposer les données des canaux spectraux individuels (Figure 2A). Les images en champ clair sont prises à l’aide d’un motif de nanogrille qui transmet la lumière blanche et est détecté dans chaque canal spectral du séparateur d’image (non montré). Le canal vert sert de canal de référence et le canal rouge lointain est le canal décalé. La transformation moyenne pondérée locale est calculée à l’aide de la fonction fitgeotrans20 dans MATLAB et est utilisée pour déplacer les coordonnées rouge lointain dans le cadre de coordonnées du canal vert. Cette transformation utilise un modèle polynomial de second ordre à chaque point de contrôle. Les données multicanaux de la baie SiMPull sont ensuite acquises. Ce flux de travail consistait en une acquisition semi-automatisée, où un retour sur investissement de départ était sélectionné pour le carré d’échantillon spécifique et trois régions autour de cette zone étaient imagées, de sorte que chaque jeu de données contienne l’image complète à quatre vues de trois ROI indépendants (Figure 1D). Dans chaque canal spectral, les emplacements candidats émetteurs sont trouvés en appliquant une différence de filtre gaussien aux images et en identifiant les maxima locaux. Les sous-régions (boîtes, figure 2B) sont dessinées autour de maxima locaux, et le nombre de photons émetteurs est estimé en supposant que chaque sous-région ne contient qu’un seul émetteur. Les sous-régions contenant des émetteurs candidats dont le nombre de photons est supérieur à une valeur minimale sont conservées pour l’ajustement. Une fonction d’étalement de points gaussien (PSF) s’adapte à chaque émetteur candidat dans de petites sous-régions à peu près centrées autour de chaque émetteur. Les localisations résultantes sont seuillées en fonction de leur nombre de photons, de leur arrière-plan, de la limite inférieure de Cramér-Rao des coordonnées d’ajustement, de la variance PSF (c’est-à-dire la largeur PSF) et d’une valeur p décrivant la qualité de l’ajustement du modèle PSF. Une image gaussienne est créée pour chaque canal spectral, avec des taches gaussiennes d’intensité uniforme placées aux coordonnées de chaque bon ajustement (Figure 2C). La colocalisation est visualisée en superposant les images gaussiennes de chaque canal spectral à l’aide de la transformation calculée à partir de l’échantillon fiduciaire (Figure 2D). Il est important de marquer par fluorescence le récepteur pour l’identification car il y a encore une liaison non spécifique des anticorps anti-phosphotyrosine à la surface lorsque le lysat cellulaire est présent. L’EGFR-GFP (canal vert) est utilisé pour générer un masque des emplacements des récepteurs, et seul le signal AF647-anti-PY (canal rouge lointain) à l’intérieur de ce masque est compté (Figure 2D). Les paires d’un pixel inférieur à 1 (taille de pixel de 106,7 nm) sont considérées comme colocalisées et enregistrées dans une liste contenant les coordonnées du canal de référence. Le pourcentage d’AF647 colocalisé avec la GFP est calculé pour déterminer la fraction des récepteurs phosphorylés (Figure 2E).
Il existe plusieurs étapes critiques pour assurer une bonne qualité des données. L’un de ces efforts consiste à incuber le réseau de couvercles avec Du NaBH4 comme décrit dans le protocole pour éteindre l’autofluorescence dans le canal vert. Cette autofluorescence fait référence au signal non spécifique dû à d’éventuelles impuretés sur le verre, contenant des liaisons π simples ou conjuguées21. Ces impuretés proviennent potentiellement des réactifs aminosilane et PEG utilisés dans le processus de fonctionnalisation ou de la poussière de l’air, et ont tendance à fluorescence dans le canal spectral vert. Malgré les efforts déployés pour maintenir le verre stocké sous azote, ces molécules peuvent également être générées par oxydation qui se produit pendant le stockage. NaBH4 a également été utilisé pour réduire la fluorescence des impuretés sur les lames et les microréseaux, y compris ceux avec revêtement de silane16. La figure 3A montre la réduction du nombre de détections de fond qui se produisent lorsque le verre gravé de piranha est traité avec NaBH4. Alors que NaBH4 réduit considérablement la fluorescence de fond, certains émetteurs sont encore détectés dans le canal vert. On peut corriger cela en acquérant des images d’arrière-plan à partir d’échantillons sans lysat (Figure 3D) et en soustrayant le nombre moyen de localisations d’arrière-plan des échantillons contenant des GFP. La fluorescence des impuretés n’a pas été détectée dans le canal rouge lointain. Si la densité du récepteur est trop élevée, plusieurs émetteurs GFP peuvent être trouvés dans un seul endroit limité par diffraction (données non montrées). En utilisant le photoblanchiment par étapes pour identifier le nombre de GFPs par spot, nous avons constaté qu’une densité de récepteur comprise entre 0,04 et 0,08 protéines / μm2 fournissait un espacement suffisant entre les émetteurs uniques pour éliminer le potentiel de trouver plusieurs émetteurs par point12. La densité du récepteur peut être optimisée en faisant varier la quantité d’anticorps IP liés à la surface du verre ou la quantité de lysat ajoutée. Il est essentiel de s’assurer que l’anticorps ciblant le POI est utilisé à des niveaux saturés. Il est recommandé d’acquérir une courbe de concentration d’anticorps sur des échantillons phosphorylés afin de déterminer les conditions de marquage appropriées (figure 3B). De plus, la spécificité phospho d’un anticorps doit être validée avec des échantillons au repos et/ou un traitement avec des inhibiteurs de kinases spécifiques à la protéine (figure 3B). Les anticorps se dissocieront du récepteur pendant la fenêtre de temps d’imagerie. Le traitement de l’échantillon avec une combinaison de PFA et d’AG a empêché la perte de signal (Figure 3C).
Enfin, il est important d’optimiser les paramètres d’ajustement d’une seule molécule. La première étape de « recherche de boîte » qui identifie les candidats émetteurs potentiels (Figure 2B) doit être généreuse pour permettre à de nombreux candidats de subir le raccord gaussien. Ainsi, le seuil minimal de photons pour la recherche de boîtes peut être relativement bas pour capturer tous les émetteurs réels et certains points d’arrière-plan. Il est également important de ne pas définir la taille de la boîte et l’allocation de chevauchement trop petites. Garder la taille de la boîte dans les 5-7 pixels et permettre le chevauchement de deux pixels est idéal pour les émetteurs à la densité recommandée. Après la recherche de la boîte, le seuil minimal de photons dans l’étape d’ajustement doit être optimisé. Le paramètre des photons minimaux contribue à déterminer quels émetteurs ajustés gaussiens passent pour un véritable ajustement. Pour déterminer le seuil minimal de photons approprié pour les véritables ajustements GFP, le code comprend une fonction de traçage d’histogramme pour examiner les photons / localisation dans les échantillons de fond (pas de lysat cellulaire) et contenant du GFP (plus le lysat cellulaire) (Figure 3D). Cette étape est importante car, bien que NaBH4 réduise la quantité de fluorescence des impuretés, elle ne supprime pas toutes les localisations de fond. La figure 3D montre la nécessité de définir un seuil minimal de photons pour réduire le nombre de détections d’impuretés. Pour déterminer ce seuil, un histogramme des intensités des émetteurs de fond est calculé à partir de l’imagerie d’un échantillon qui n’est pas exposé au lysat cellulaire (Figure 3D, en haut à gauche). La majorité des émetteurs de fond ont des valeurs inférieures à 475 photons. En comparaison, l’échantillon contenant de véritables émetteurs de GFP a montré une fraction significative de la distribution au-dessus de 475 (Figure 3D, en haut à droite). Le seuil est choisi par inspection pour supprimer autant de comptes d’arrière-plan que possible tout en minimisant la quantité de perte de signal de l’échantillon de lysat (Figure 3D, ligne du bas). La densité de comptage de fond restante à ce seuil est prise en compte dans l’analyse quantitative.
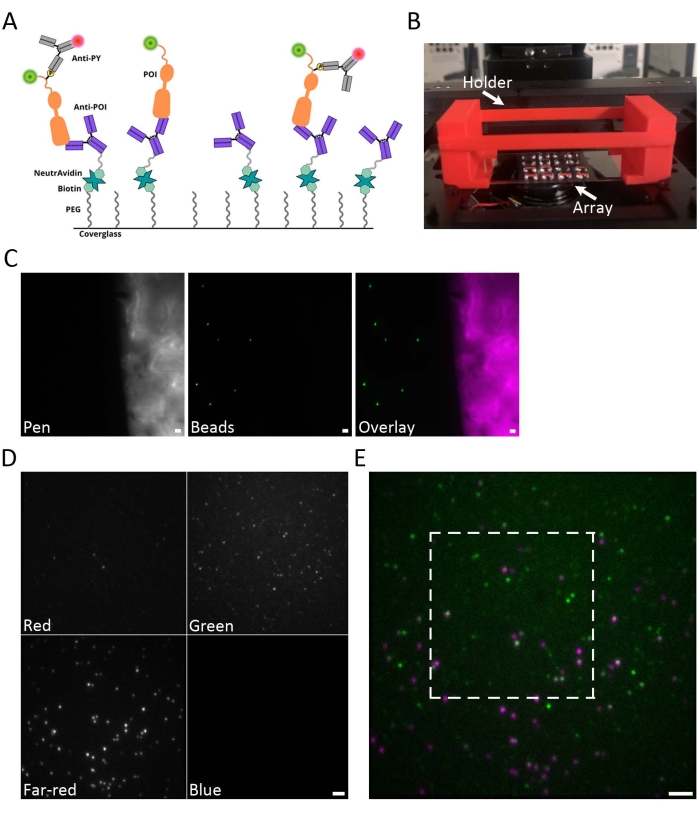
Figure 1 : Vue d’ensemble de la préparation de l’échantillon. (A) Dessin animé illustrant l’approche SiMPull. Les coverslips sont fonctionnalisés avec un anticorps qui reconnaît le POI pour capturer ce POI des lysats de cellules entières. Le verre est d’abord recouvert de PEG et de biotine-PEG. NeutrAvidin est ensuite lié à la biotine-PEG et agit comme un point d’ancrage pour l’anticorps anti-POI biotinylé. Les protéines phosphorylées sont ensuite détectées avec un anticorps anti-PY marqué par fluorescence. (B) Photographie du porte-couvercle (rouge) avec le réseau de couvercles en place et monté sur la scène du microscope. Les réseaux multiéchantillons sont générés à l’aide d’encre hydrophobe pour créer jusqu’à 20 carrés d’échantillon individuels sur un seul couvercle en verre. La glissière de couverture est de 60 mm x 24 mm. (C) Exemples d’images de l’autofluorescence d’encre hydrophobe (magenta) et des perles fluorescentes (vert). L’autofluorescence de l’encre hydrophobe est un guide utile pour trouver le plan focal à la surface du couvercle. (D) Exemple d’une image de données brutes avec des canaux spectraux séparés sur la puce de la caméra par le séparateur d’images Quad-view. L’ensemble de filtres Quad-view comprend les filtres d’émission suivants : bleu (445/45 nm), vert (525/45 nm), rouge (600/37 nm), rouge lointain (685/40 nm). (E) Superposition brute de canaux verts et rouges lointains. La boîte blanche indique la région examinée plus en détail à la figure 2B-D. Barre d’échelle (C-E) = 2 μm. Veuillez cliquer ici pour afficher une version agrandie de cette figure.
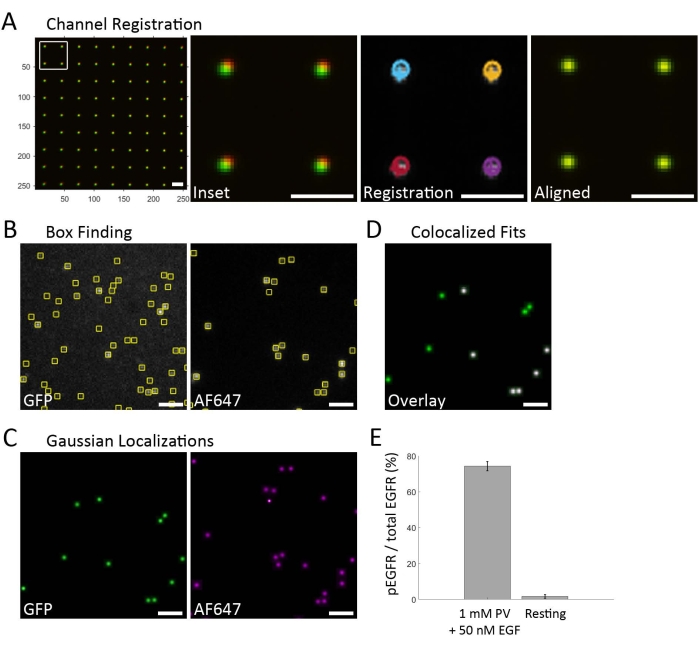
Figure 2 : Flux de travail d’analyse des données. (A) L’enregistrement du canal est d’abord effectué sur les images acquises à partir du nanogrille. Après avoir recadré les deux canaux spectraux d’intérêt (ici, vert et rouge lointain), les images fiduciales pour chaque canal sont superposées (à gauche). L’agrandissement de la boîte dans l’image de gauche (encadré) montre que les images ne sont pas encore vraiment enregistrées. Les émetteurs de chaque canal correspondent à un modèle gaussien et sont localisés (Enregistrement). La localisation des émetteurs est représentée par des cercles pour le canal rouge lointain et des croix pour le canal vert. La dernière étape consiste à appliquer une transformation moyenne pondérée locale pour déplacer les coordonnées de localisation du canal rouge lointain dans le cadre de référence du canal vert (aligné). La transformation moyenne pondérée locale calculée est ensuite utilisée pour enregistrer les données SiMPull suivantes. (B) Images représentatives du canal vert/EGFR-GFP et du canal rouge lointain/AF647-anti-PY. Les émetteurs uniques au-dessus du nombre de photons de fond sont identifiés et marqués avec des boîtes. (C) Le profil d’émission à l’intérieur de chaque boîte sélectionnée est adapté à un modèle gaussien, et les émetteurs qui correspondent au modèle d’un seul fluorophore PSF sont conservés. (D) Un masque est créé à partir des émetteurs de GFP pour identifier l’emplacement de l’EGFR-GFP (vert). La colocalisation de l’EGFR-GFP et de l’AF647-anti-PY identifie les récepteurs phosphorylés (blancs). (E) La fraction des récepteurs phosphorylés est calculée à partir des coupes COLOCALISÉES EGFR-GFP et AF647-anti-PY. Le graphique à barres compare le traitement PV + EGF aux cellules au repos, moyennées pour plusieurs mesures. Les barres d’erreur représentent l’erreur type calculée en supposant une distribution binomiale. Barre d’échelle = 2 μm. Veuillez cliquer ici pour afficher une version agrandie de cette figure.

Figure 3 : Étapes critiques pour assurer la qualité des données. (A) De gauche à droite, les trois premiers panneaux sont des images représentatives de l’autofluorescence sur verre dans les conditions respectives : après gravure au piranha, avec PEG, et traitement PEG plus NaBH4 (indiqué par +). De plus, la fonctionnalisation de surface est conservée après le traitement NaBH4, comme le démontre la liaison PY99-AF647 non spécifique minimale tout en conservant une liaison robuste de l’EGFR-GFP du lysat. (B) Une courbe de saturation doit être acquise pour chaque lot d’anticorps utilisé afin d’assurer un marquage optimal des anticorps. Cette figure montre la courbe de concentration pour le marquage de l’EGFR avec l’anticorps phosphotyrosine spécifique au site, anti-EGFR-pY1173. Une phosphorylation minimale est détectée dans les cellules non traitées (au repos, diamant gris). Comme contrôle de la liaison non spécifique, les cellules ont également été traitées avec l’inhibiteur de la kinase EGFR, le lapatinib, avant d’ajouter 100 nM d’EGF (triangle magenta), ce qui montre la prévention attendue de la phosphorylation de l’EGFR. Les barres d’erreur représentent une erreur type supposant une distribution binomiale. (C) La fixation de l’échantillon avec une combinaison de PFA et d’AG empêche la dissociation des anticorps au fil du temps. Les barres d’erreur représentent une erreur type supposant une distribution binomiale. (D) Les faux positifs sont exclus en sélectionnant le seuil approprié pour l’ajustement gaussien. La comparaison de l’histogramme des intensités d’ajustement à un seuil bas (Seuil = 0; haut) entre l’arrière-plan (pas de lysat) et les données réelles (plus le lysat de cellule) permet de sélectionner la valeur appropriée (Seuil = 475; en bas) pour éliminer les ajustements des taches autofluorescentes dans le canal vert. La ligne magenta verticale indique un seuil de 475 photons. Les histogrammes sont calculés à partir du même nombre de retours sur investissement pour chaque type d’échantillon (n = 3). Barre d’échelle = 2 μm. Veuillez cliquer ici pour afficher une version agrandie de cette figure.
Fichier de codage supplémentaire 1: fichier zip contenant des scripts et des utilitaires pour exécuter l’analyse SiMPull. Veuillez cliquer ici pour télécharger ce fichier.
Fichier de codage supplémentaire 2: fichier zip contenant le package d’analyse de molécule unique smite . Veuillez cliquer ici pour télécharger ce fichier.
Fichier de codage supplémentaire 3 : fichier zip contenant les exemples de données. Veuillez cliquer ici pour télécharger ce fichier.
Fichier de codage supplémentaire 4 : fichier zip contenant des résultats représentatifs d’analyse d’échantillons de données. Veuillez cliquer ici pour télécharger ce fichier.
Fichier de codage supplémentaire 5 : Plan de support de couvercle pour l’impression 3D. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Le protocole décrit ici a été optimisé pour permettre des mesures quantitatives de la phosphorylation des récepteurs au niveau d’une seule protéine. Plusieurs modifications simples mais importantes du protocole SiMPull ont été développées pour améliorer la fiabilité de la mesure pour la détection de la phospho-tyrosine, y compris la réduction de l’autofluorescence avec le traitement NaBH4 et la postfixation de l’échantillon pour prévenir la dissociation des anticorps. L’utilisation du masque de canal vert pour identifier les emplacements des récepteurs pour le calcul de la colocalisation avec l’anticorps anti-PY améliore également la précision de la mesure en éliminant les artefacts potentiels de la liaison non spécifique de l’anticorps au lysat cellulaire. L’imagerie bicolore a été utilisée pour détecter la fraction des récepteurs phosphorylés. Dans ce scénario, le récepteur a été génétiquement marqué avec GFP, et l’anticorps a été directement marqué avec un colorant rouge lointain. L’approche SiMPull s’applique à d’autres cibles protéiques pour lesquelles des anticorps spécifiques sont disponibles, y compris les protéines intracellulaires. En outre, comme les conditions de dénaturation ne sont pas nécessaires, des récepteurs / complexes multisopuces peuvent également être capturés. Cependant, la dénaturation peut être incorporée si les PTM d’intérêt sont situés dans des régions structurées de la protéine14. En fin de compte, SiMPull peut être facilement étendu pour inclure le marquage simultané de phospho-tyrosines distinctes sur des récepteurs individuels afin de quantifier les modèles de phosphorylation multisite15. L’interrogation de récepteurs intacts et de pleine longueur de cette manière ne peut pas être réalisée par d’autres méthodes standard, y compris le transfert western et la spectrométrie de masse phospho.
Outre les avantages de SiMPull, certaines limitations doivent être prises en compte. Comme pour toute technique à base d’anticorps, l’affinité et la spécificité des anticorps utilisés sont essentielles au succès de la mesure. Par conséquent, il est important d’optimiser les conditions de marquage des anticorps et, idéalement, d’éviter les anticorps secondaires en utilisant des anticorps primaires directement marqués. De plus, les anticorps liés à la surface précipiteront les protéines localisées dans la membrane plasmique et dans les compartiments cytosoliques. Cela peut sous-estimer la phosphorylation puisque les protéines localisées dans le cytosol ne sont pas accessibles au ligand ajouté de manière exogène. Des mesures supplémentaires doivent être prises pour corriger les niveaux de surface du récepteur (étape 6.2.10). Les anticorps anti-phosphotyrosine ont présenté une certaine liaison non spécifique une fois que le lysat était présent. Pour éviter cet artefact, l’EGFR a été génétiquement marqué avec GFP pour identifier l’emplacement des récepteurs, ce qui nous a permis d’exclure le signal anti-PY du récepteur. Si des protéines endogènes doivent être interrogées, alors le contre-maintien avec un anticorps protéique total peut fournir l’image du masque, avec une correction appropriée pour toute liaison non spécifique. Enfin, alors que SiMPull fournit des informations sur l’hétérogénéité au niveau des protéines, le lysat généré dans ce protocole provient de milliers de cellules et la variabilité de cellule à cellule est perdue. Cependant, des progrès vers le SiMPull à cellule unique ont été réalisés à l’aide d’une chambre d’écoulement composée d’un couvercle et d’un côté microscope avec un espace de 10 μm; les bactéries étaient peu plaquées sur le couvercle tandis que la lame était fonctionnalisée avec des anticorps pour capturer les protéines souhaitées. Lors de la lyse des bactéries, les protéines de chaque cellule ont été capturées dans une zone confinée sur la lame22 recouverte d’anticorps. Une analyse SiMPull unicellulaire similaire des cellules de mammifères et de la phosphorylation des protéines pourrait être possible à l’avenir.
Le protocole SiMPull contient plusieurs étapes critiques requises pour garantir des données de haute qualité. Par exemple, le protocole comprend une préparation élaborée du verre à glissement de couverture. Les couvercles de gravure Piranha nettoient en profondeur le verre et augmentent les groupes hydroxyles et l’hydrophilie, qui sont nécessaires pour optimiser la surface du couvercle. Après plusieurs lavages avec des solvants organiques, le traitement KOH fournit des groupes hydroxyles supplémentaires pour l’aminosilanisation13,23, qui recouvre le verre de groupes amines pour la liaison PEG et biotine-PEG. Un nettoyage ou une fonctionnalisation inadéquats à l’une de ces étapes interférera avec l’extraction des protéines. Le contrôle du rapport molaire PEG:biotine-PEG, ainsi que la concentration de lysat, sont des facteurs clés pour obtenir une densité IP protéique appropriée sur le substrat SiMPull. Comme pour tout essai biologique, il existe une variabilité entre les préparations de lysat cellulaire, et de petites différences entre les pourcentages de phosphorylation peuvent être observées entre les répliques de l’échantillon. Par conséquent, il est important de mesurer les niveaux de phosphorylation de différents sites de tyrosine dans le même échantillon. La chambre d’échantillonnage décrite dans ce protocole fournit un système permettant de collecter de nombreux points de données en une seule séance d’imagerie et permet de faire la moyenne sur plusieurs expériences SiMPull.
Du côté de l’acquisition d’images, il est important d’obtenir l’échantillon fiducial pour assurer une superposition de canal précise; sinon, la colocalisation ne sera pas exacte. Il est également important d’optimiser la puissance laser et les paramètres de la caméra pour maximiser le rapport signal-bruit tout en minimisant le photoblanchiment. Enfin, alors que le réseau d’échantillons nécessite une petite quantité d’échantillon et de réactifs, les faibles volumes sont sensibles à l’évaporation pendant la séance d’imagerie. Il est important de vérifier périodiquement le réseau d’échantillons (~30-45 min) et d’ajouter un tampon au besoin pour empêcher les échantillons de sécher.
Le présent protocole a démontré l’utilisation de SiMPull pour quantifier les états de phosphorylation des récepteurs membranaires. Bien que axée sur l’EGFR, l’approche peut être appliquée à d’autres récepteurs de surface cellulaire et à des protéines intracellulaires et complexes protéiques, à condition que des anticorps appropriés soient disponibles. Une autre utilisation potentielle de SiMPull est d’interroger le contenu et l’état de phosphorylation des condensats séparés par phase. En outre, SiMPull peut être utilisé pour mesurer d’autres PTM, tels que l’ubiquitination. Par conséquent, SiMPull fournit un outil unique aux biologistes cellulaires pour interroger les PTM sur des protéines intactes et corréler les modèles de PTM avec les résultats cellulaires.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par les National Institutes of Health R35GM126934, R01AI153617 et R01CA248166 à DSL. EMB a été soutenu par le programme ASERT-IRACDA (NIH K12GM088021) et JAR par le programme UNM MARC (NIH 2T34GM008751-20). Nous remercions l’utilisation de la ressource partagée de microscopie à fluorescence du Comprehensive Cancer Center de l’Université du Nouveau-Mexique, soutenue par NIH P30CA118100. Nous tenons à remercier les Drs Ankur Jain et Taekijip Ha, dont le développement original de SiMPull a inspiré ce travail.
ES-C adresse actuelle: Groupe immunodynamique, Laboratoire d’immunologie intégrative du cancer, Centre de recherche sur le cancer, Institut national du cancer, Bethesda
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes | MTC Bio | C2000 | |
| 10 mM Tris-HCl pH 7.4 | |||
| 10 mM Tris-HCl pH 8.0/ 50 mM NaCl | T50 Buffer | ||
| 100 mm Tissue Culture dish | CELLTREAT | 229620 | Storage of piranha etched glass/arrays |
| 15 mL conical tube | |||
| 16% Paraformaldehyde Aqueous Solution | Electron Microscopy Sciences | 15710 | Hazardous |
| 50 mL conical tube | Functionalized Glass storage/ KOH reuse | ||
| 50 mM Tris-HCl pH 7.2/150 mM NaCl | Lysis Buffer Component | ||
| 60 mm Tissue Culture dish | Corning | 430166 | |
| 8% Glutaraldehyde Aqueous Solution | Electron Microscopy Sciences | 16020 | Hazardous |
| Acetone (C3H6O) | Millipore Sigma | 270725 | Hazardous |
| Alexa Fluor 647 NHS Ester | Thermo Fisher Scientific | A-20006 | |
| Animal-Free Recombinant Human EGF | Peprotech | AF-100-15 | |
| Anti-Human EGFR (External Domain) – Biotin | Leinco Technologies, Inc | E101 | |
| Anti-p-Tyr Antibody (PY99) Alexa Fluor 647 | Santa Cruz Biotechnology | sc-7020 AF647 | |
| Bath-sonicator | Branson | 1200 | |
| BCA Protein Assay Kit | Pierce | 23227 | |
| Biotin-PEG | Laysan Bio | Biotin-PEG-SVA, MW 5,000 | |
| Bovine serum albumin | Gold Biotechnology | A-420-1 | Tyrode's Buffer Component |
| Buchner funnel | |||
| Bunsen burner | |||
| Calcium Chloride (CaCl2) | Millipore Sigma | C4901 | Tyrode's Buffer Component |
| Cell Scraper | Bioworld | 30900017-1 | |
| Conical Filtering Flask | Fisher Scientific | S15464 | |
| Coplin Jar | WHEATON | 900470 | |
| Countess II Automated Cell Counter | Thermo Fisher Scientific | AMQAX1000 | |
| Coverslips 24 x 60 #1.5 | Electron Microscopy Sciences | 63793 | |
| DipImage | https://diplib.org/ | ||
| DMEM | Caisson Labs | DML19-500 | |
| emCCD camera | Andor iXon | ||
| Fetal Bovine Serum, Optima | Bio-Techne | S12450H | Heat Inactivated |
| Fusion 360 software | Autodesk | ||
| Geneticin G418 Disulfate | Caisson Labs | G030-5GM | |
| Glacial Acetic Acid (CH3COOH) | JT Baker | JTB-9526-01 | Hazardous |
| Glass serological pipettes | |||
| Glass Stir Rod | |||
| Glucose (D-(+)-Glucose) | Millipore Sigma | D9434 | Tyrode's Buffer Component |
| Halt Phosphotase and Protease Inhibitor Cocktail (100X) | Thermo Fisher Scientific | 78446 | Lysis Buffer Component |
| HEPES | Millipore Sigma | H3375 | Tyrode's Buffer Component |
| Hydrochloric Acid (HCl) | VWR | BDH7204-1 | Hazardous |
| Hydrogen Peroxide (H2O2) (3%) | HX0645 | ||
| Hydrogen Peroxide (H2O2) (30%) | EMD Millipore | HX0635-2 | |
| Ice | |||
| IGEPAL CA-630 (NP-40) | Sigma Aldrich | I8896 | Lysis Buffer Component |
| ImmEdge Hydrophobic Barrier Pen | Vector Laboratories | H-4000 | |
| Immersol 518F immersion oil | Zeiss | 444960-0000-000 | |
| in-house vacuum line | |||
| L-glutamine | Thermo Fisher Scientific | 25030-164 | |
| Magnessium Chloride Hexahydrate (MgCl2-6H2O) | MPBio | 2191421 | Tyrode's Buffer Component |
| Matlab | Mathworks | Curve Fitting Toolbox, Parallel Computing Toolbox, and Statistics and Machine Learning toolbox | |
| Methanol (CH3OH) | IBIS Scientific | MX0486-1 | Hazardous |
| Milli-Q water | |||
| Mix-n-Stain CF Dye Antibody Labeling Kits | Biotium | 92245 | Suggested conjugation kit |
| mPEG | Laysan Bio | mPEG-succinimidyl valerate, MW 5,000 | |
| N-(2-aminoethyl)-3-aminopropyltrimethoxysilane | UCT United Chemical | A0700 | Hazardous |
| Nanogrid | Miraloma Tech | ||
| NeutrAvidin Biotin Binding Protein | Thermo Fisher Scientific | 31000 | |
| Nitrogen (compressed gas) | |||
| NVIDIA GPU with CUDA | Look for compatibility at https://www.mathworks.com/help/parallel-computing/gpu-support-by-release.html | ||
| Olympus iX71 Microscope | Olympus | ||
| Parafilm M Sealing Film | The Lab Depot | HS234526C | |
| PBS pH 7.4 | Caisson Labs | PBL06 | |
| PC-200 Analog Hot Plate | Corning | 6795-200 | |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher Scientific | 15140-163 | |
| Phospho-EGF Receptor (Tyr1068) (1H12) Mouse mAb | Cell Signaling Technology | 2236BF | |
| Potassium Chloride (KCl) | Millipore Sigma | 529552 | Tyrode's Buffer Component |
| Potassium Hydroxide (KOH) | Millipore Sigma | 1050330500 | Hazardous |
| Premium PLA Filament, 1.75 mm diameter | Raise 3D | PMS:2035U/RAL:3028 | Printing temperature range: 205-235 °C |
| Pro2 3D printer | Raise 3D | ||
| Pyrex 1 L beaker | |||
| PYREX 100 mL storage bottles | Corning | 1395-100 | CH3OH/C3H6O reuse |
| Pyrex 250 mL beakers | |||
| Pyrex 4 L beaker | |||
| Quad-view Image Splitter | Photometrics | Model QV2 | |
| Refrigerated centrifuge | Eppendorf | EP-5415R | |
| RevCount Cell Counters, EVE Cell Counting Slides | VWR | 10027-446 | |
| Semrock emission filters: blue (445/45 nm), green (525/45 nm), red (600/37 nm), far-red (685/40 nm) | Semrock | LF405/488/561/635-4X4M-B-000 | |
| Serological pipette controller | |||
| Serological Pipettes | |||
| smite single molecule analysis package | https://github.com/LidkeLab/smite.git | ||
| Sodium Bicarbonate (NaHCO3) | Sigma Aldrich | S6014 | Hazardous |
| Sodium Borohydride (NaBH4) | Millipore Sigma | 452874 | Tyrode's Buffer Component |
| Sodium Chloride (NaCl) | Millipore Sigma | S9625 | Activate by successive heat and pH cycling |
| Sodium Hydroxide | VWR | BDH3247-1 | |
| Sodium Orthovanadate (Na3VO4) | Millipore Sigma | S6508 | Hazardous |
| Sulfuric Acid (H2SO4) | Millipore Sigma | 258105 | Hazardous |
| TetraSpeck Microspheres | Thermo Fisher Scientific | T7279 | multi-fluorescent beads |
| Tris (Trizma) base | Millipore Sigma | T1503 | |
| Trypan blue stain, 0.4% | Thermo Fisher Scientific | 15250061 | |
| Trypsin-EDTA 0.05% | Thermo Fisher Scientific | 25300120 |
Références
- Lemmon, M. A., Schlessinger, J. Cell Signaling by Receptor Tyrosine Kinases. Cell. 141 (7), 1117-1134 (2010).
- Seet, B. T., Dikic, I., Zhou, M. M., Pawson, T. Reading protein modifications with interaction domains. Nature Reviews Molecular Cell Biology. 7 (7), 473-483 (2006).
- Coba, M. P., et al. Neurotransmitters drive combinatorial multistate postsynaptic density networks. Science Signaling. 2 (68), (2009).
- Gibson, S. K., Parkes, J. H., Liebman, P. A. Phosphorylation modulates the affinity of light-activated rhodopsin for g protein and arrestin. Biochemistry. 39 (19), 5738-5749 (2000).
- Stites, E. C., et al. Use of mechanistic models to integrate and analyze multiple proteomic datasets. Biophysical Journal. 108 (7), 1819-1829 (2015).
- Hause, R. J., et al. Comprehensive binary interaction mapping of SH2 domains via fluorescence polarization reveals novel functional diversification of ErbB receptors. PLOS ONE. 7 (9), 44471(2012).
- Lau, E. K., et al. Quantitative encoding of the effect of a partial agonist on individual opioid receptors by multisite phosphorylation and threshold detection. Science Signaling. 4 (185), (2011).
- Mishra, M., Tiwari, S., Gomes, A. V. Protein purification and analysis: next generation Western blotting techniques. Expert Review of Proteomics. 14 (11), 1037-1053 (2017).
- Brunner, A. M., et al. Benchmarking multiple fragmentation methods on an orbitrap fusion for top-down phospho-proteoform characterization. Analytical Chemistry. 87 (8), 4152-4158 (2015).
- Swaney, D. L., Wenger, C. D., Coon, J. J. Value of using multiple proteases for large-scale mass spectrometry-based proteomics. Journal of Proteome Research. 9 (3), 1323-1329 (2010).
- Curran, T. G., Zhang, Y., Ma, D. J., Sarkaria, J. N., White, F. M. MARQUIS: A multiplex method for absolute quantification of peptides and posttranslational modifications. Nature Communications. 6 (1), 1-11 (2015).
- Jain, A., et al. Probing cellular protein complexes using single-molecule pull-down. Nature. 473 (7348), 484-488 (2011).
- Jain, A., Liu, R., Xiang, Y. K., Ha, T. Single-molecule pull-down for studying protein interactions. Nature Protocols. 7 (3), 445-452 (2012).
- Kim, K. L., et al. Pairwise detection of site-specific receptor phosphorylations using single-molecule blotting. Nature Communications. 7 (1), 1-10 (2016).
- Salazar-Cavazos, E., et al. Multisite EGFR phosphorylation is regulated by adaptor protein abundances and dimer lifetimes. Molecular Biology of the Cell. 31 (7), 695(2020).
- Huyer, G., et al. Mechanism of inhibition of protein-tyrosine phosphatases by vanadate and pervanadate. Journal of Biological Chemistry. 272 (2), 843-851 (1997).
- Smith, P. K., et al. Measurement of protein using bicinchoninic acid. Analytical Biochemistry. 150 (1), 76-85 (1985).
- Keller, H. E. Objective lenses for confocal microscopy. Handbook of Biological Confocal Microscopy. , 145-161 (2006).
- Hendriks, C. L. L., van Vliet, L. J., Rieger, B., van Kempen, G. M. P., van Ginkel, M. Dipimage: a scientific image processing toolbox for MATLAB. , (1999).
- fitgeotrans: Fit geometric transformation to control point pairs. The MathWorks Inc. , Available from: https://www.mathworks.com/images/ref/fitgeotrans.html (2013).
- Raghavachari, N., Bao, Y. P., Li, G., Xie, X., Müller, U. R. Reduction of autofluorescence on DNA microarrays and slide surfaces by treatment with sodium borohydride. Analytical Biochemistry. 312 (2), 101-105 (2003).
- Wang, X., Park, S., Zeng, L., Jain, A., Ha, T. Toward single-cell single-molecule pull-down. Biophysical Journal. 115 (2), 283-288 (2018).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. Journal of Visualized Experiments. (86), e50549(2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon