Method Article
用于蛋白质磷酸化定量的优化单分子下拉测定
摘要
本方案描述了样品制备和数据分析,以使用改进的单分子下拉(SiMPull)测定来量化蛋白质磷酸化。
摘要
磷酸化是一种必要的翻译后修饰,可调节蛋白质功能并指导细胞信号传导结果。目前测量蛋白质磷酸化的方法不能保持单个蛋白质磷酸化的异质性。单分子下拉(SiMPull)测定是通过在玻璃盖玻片 上免疫沉淀 蛋白质然后进行单分子成像来研究大分子复合物的组成。目前的技术是对SiMPull的改编,可在单分子水平上对全长膜受体的磷酸化状态进行强有力的定量。以这种方式对数千个个体受体进行成像可以量化蛋白质磷酸化模式。本实验方案详细介绍了优化的SiMPull程序,从样品制备到成像。玻璃制备和抗体固定方案的优化进一步提高了数据质量。目前的方案为单分子数据分析提供了代码,该数据分析计算样品中磷酸化的受体的比例。虽然这项工作的重点是表皮生长因子受体(EGFR)的磷酸化,但该方案可以推广到其他膜受体和胞质信号分子。
引言
膜相关信号传导通过配体诱导的膜受体激活和募集传播信号的下游辅助蛋白的组合进行调整。受体细胞质尾部中关键酪氨酸的磷酸化对于启动信号复合物或信号体1,2的形成至关重要。因此,生物学中的一个重要问题是如何创建和维护磷酸化模式以招募信号伙伴并决定细胞结果。这包括了解受体磷酸化的异质性,无论是在丰度上还是在特定的磷酸酪氨酸模式中,都可以通过决定信号体3,4,5,6,7的组成来提供操纵信号输出的方法。然而,目前询问蛋白质磷酸化的方法存在局限性。蛋白质印迹分析非常适合描述蛋白质磷酸化的趋势,但半定量8,并且不能提供有关系统异质性的信息,因为数千到数百万个受体被平均在一起。虽然蛋白质印迹允许使用磷酸特异性抗体对特定酪氨酸进行探查样品,但它们不能提供有关同一蛋白质内多位磷酸化模式的信息。定量磷酸化蛋白质组学报告了磷酸酪氨酸丰度,但检测多位磷酸化存在局限性,因为感兴趣的残基需要位于酶消化产生的相同肽(通常为7-35个氨基酸)内9,10,11。
为了克服上述限制,单分子下拉(SiMPull)测定已被调整为在单分子水平上量化完整受体的磷酸化状态。SiMPull首先被耆那教等人证明是询问大分子复合物的强大工具。12,13。在SiMPull中,在抗体功能化玻璃盖玻片上对大分子复合物进行免疫沉淀(IP),然后通过单分子显微镜分析蛋白质亚基数和与复合组分12的共IP。Kim等人14的一项修改,称为SiMBlot,是第一个使用SiMPull的变体来分析变性蛋白质的磷酸化。SiMBlot方案依赖于使用NeutrAvidin包被的盖玻片捕获生物素化的细胞表面蛋白,然后用磷酸特异性抗体标记14探测磷酸化。尽管取得了这些进展,但仍需要改进,以使翻译后修饰的定量更加稳健,并适用于更广泛的蛋白质。
本方案描述了一种优化的SiMPull方法,该方法用于量化完整表皮生长因子受体(EGFR)的磷酸化模式,以响应一系列配体条件和致癌突变15。虽然这项工作侧重于EGFR,但这种方法可以应用于任何膜受体和感兴趣的胞质蛋白(POI),其具有高质量的抗体。该协议包括减少样品自发荧光的步骤,需要最小样品体积的样品阵列设计,同时制备多达20个样品,以及优化抗体标记和固定条件。已经开发了用于磷酸化蛋白的单分子检测和定量的数据分析算法。
研究方案
1. 盖玻片准备
注意:对于此步骤,需要穿戴个人防护装备(PPE),其中包括双层丁腈手套,安全眼镜或面罩以及实验室外套。
- 进行食人鱼蚀刻以清除玻璃上的有机碎屑。
注意:食人鱼溶液是一种强氧化剂,与有机材料接触时具有腐蚀性和高度反应性。与有机碎片的反应是放热的,具有潜在的爆炸性。因此,该过程必须在降低窗扇的化学通风橱中进行。需要Pyrex玻璃器皿来处理食人鱼溶液。- 在化学通风橱内准备工作空间。将不重叠的盖玻片放在4 L玻璃烧杯("反应"烧杯)的底部,并将反应烧杯放在加热的温板上。让玻璃器皿加热10分钟。将一个"废物"1 L玻璃烧杯与500 mL ddH2O放在反应烧杯的近端。
- 用玻璃血清移液管将49mL12N硫酸(H 2 SO4)缓慢加入反应烧杯中。在处理之前,在废烧杯中冲洗移液器。
- 用玻璃血清移液管滴加21mL 30%过氧化氢(H2 O 2)到反应烧杯中。将H2O2液滴均匀地分布在反应瓶的底部,以防止食人鱼蚀刻反应的局部淬火。在处理之前,在废烧杯中冲洗移液器。
注意:始终将 H2O2 添加到 H2SO4 中,反之亦然。 - 食人鱼蚀刻盖玻片30分钟。每5分钟轻轻搅拌反应烧杯的内容物。
- 通过将废烧杯的内容物倒入反应烧杯中来淬灭食人鱼溶液。将液体缓慢地沿反应烧杯壁向下转移,以尽量减少飞溅。从电炉中取出反应烧杯。
- 当反应淬灭并冷却时,将食人鱼溶液倒回废烧杯中进行中和,而不从反应烧杯中取出蚀刻的盖玻片。
- 通过逐渐添加弱碱来中和食人鱼溶液。例如,使用过量的20克碳酸氢钠(NaHCO3)/ 100毫升食人鱼溶液。
注意:不要将食人鱼溶液储存在密封的废物容器中。在处置之前,溶液必须始终中和。中和反应产生剧烈的气泡,如果不通过逐渐加入弱碱来控制,可能会爆炸。 - 用玻璃搅拌棒搅拌中和的溶液,让它反应2小时。将pH值提高到>4并处理溶液。
- 在向食人鱼溶液中加入弱碱之间,将蚀刻的盖玻片从反应烧杯转移到带有玻璃搅拌棒的Buchner漏斗中,并在运行ddH2O中冲洗5分钟。
注意:立即进入下一步,或将食人鱼蚀刻盖玻片在ddH 2 O中密封的玻璃罐或培养皿中存放长达2周(用密封膜包裹,参见 材料表)。
- 按照以下步骤在有机溶剂中对盖玻片进行浴超声处理。
- 将盖玻片放入玻璃Coplin罐中(见 材料表),并用甲醇(CH3OH)覆盖。用密封膜将盖子密封到罐子上,并浸泡超声处理10分钟。小心地将甲醇从Coplin罐中倒入玻璃储存瓶中。
- 用丙酮(C3H6O)填充Coplin罐,密封盖子,然后浴超声处理10分钟。小心地将丙酮从科普林罐中倒入玻璃储存瓶中。
注意:甲醇易燃且剧毒。在化学通风橱中使用。丙酮易燃且有刺激性。因此,请将其处理并存放在玻璃中,并在化学通风橱中使用它。根据当地法规和准则作为危险废物处置。
注意:甲醇和丙酮可重复使用多达五次。
- 激活盖玻片表面以进行硅烷功能化。
- 用1M氢氧化钾(KOH)沐浴超声处理20分钟。小心地将KOH从科普林罐中倒入50毫升锥形管中以重复使用。
注意:KOH具有腐蚀性和刺激性。在化学通风橱中使用,不要存放在玻璃中。将其存放在聚丙烯管中。根据当地法规和准则作为危险废物处置。
注意:KOH最多可以重复使用五次。 - 用ddH2O冲洗两次,从盖玻片上沥干ddH2O,然后通过本生燃烧器的火焰挥舞来加热每个盖玻片,以驱走所有表面水分。将盖玻片放入干燥的科普林罐中。
- 用1M氢氧化钾(KOH)沐浴超声处理20分钟。小心地将KOH从科普林罐中倒入50毫升锥形管中以重复使用。
- 进行盖玻片氨基硅烷化。
- 通过在锥形烧瓶中将69.4mL甲醇与3.6mL乙酸(CH3COOH)混合来制备氨基硅烷溶液。加入720μLN-(2-氨基乙基)-3-氨基丙基三甲氧基硅烷(氨基硅烷)并充分混合(见 材料表)。
注意:醋酸易燃且具有腐蚀性。用玻璃移液器处理并储存在玻璃中。在化学通风橱中使用乙酸。氨基硅烷是一种急性毒性吸入危险剂、致敏剂和刺激物。它对水生生物有害。在化学通风橱中使用。根据当地法规和准则将化学品作为危险废物处置。
注意:氨基硅烷具有光敏性,在水中迅速水解。使用该试剂的所有步骤都需要在最小的光照条件下进行,以保持活性。用氮气吹扫瓶子,并在储存在黑暗干燥器中之前涂上密封膜。每 6-9 个月更换一次。 - 立即将氨基硅烷溶液加入椰干林罐中。盖上并涂上密封膜,继续避光。
- 将盖玻片在氨基硅烷溶液中在室温(RT)的黑暗中孵育10分钟。浴超声处理1分钟,然后再孵育10分钟。小心地将氨基硅烷溶液倒入指定用于CH3OH的废物容器中,其中含有痕量氨基硅烷和CH3COOH。
- 用甲醇冲洗盖玻片,并将溶液倒入指定用于甲醇的废物容器中。
- 用ddH 2 O冲洗盖玻片三次,每次2分钟,沥干盖玻片,轻拍掉多余的水分,完全风干10分钟。
- 通过在锥形烧瓶中将69.4mL甲醇与3.6mL乙酸(CH3COOH)混合来制备氨基硅烷溶液。加入720μLN-(2-氨基乙基)-3-氨基丙基三甲氧基硅烷(氨基硅烷)并充分混合(见 材料表)。
- 执行盖玻片的阵列制备/生物素-PEG功能化。
- 通过将84.5毫克钠HCO3溶解到1毫升ddH2O中来制备1M NaHCO3(pH 8.5)工作储备。对于10mM NaHCO3的最终浓度,将1M NaHCO3稀释到ddH 2O(1:100)中。
- 用疏水性阻隔笔在干燥的氨基硅化盖玻片上绘制网格阵列(参见 材料表),并等待墨水干燥。在盖玻片上写一个标识符以标记正确的方向。将盖玻片放在加湿的腔室中。
注:阵列应由 16-20 个正方形组成,大小约为 4 mm x 4 mm。 - 要使生物素- PEG琥珀酰戊酸酯(生物素-PEG)/ mPEG琥珀酰戊酸酯(mPEG)溶液,首先,从冰箱中取出mPEG和生物素- PEG(参见 材料表)并平衡到室温。 在室温下以10,000× g 离心1分钟以除去气泡。
注意:戊酸琥珀酰亚胺在pH 8.5缓冲液中的水解半衰期为〜30分钟。将缓冲液添加到 mPEG 后,请尽快继续执行以下步骤。此步骤至关重要。 - 应用生物素-PEG / mPEG溶液完全覆盖盖玻片阵列中的每个正方形,通常每平方10-15μL。不要让液体溢出定义的空间。将盖玻片在黑暗中的湿度室中在室温下放置3-4小时。
- 将盖玻片依次浸入装满ddH 2O 的 3x 250 mL 玻璃烧杯中,每次浸泡 10 秒,用大量水清洗盖玻片。
- 用氮气排出盖玻片上的所有水分。将盖玻片背靠背存放在氮气填充的50 mL锥形管中,该管在-20°C下用密封膜包裹。
注意:立即继续执行步骤 2 或在使用前将盖玻片储存在 -20 °C 下长达 1 周。
2. 裂解物的制备
注意:实验方案其余步骤所需的个人防护用品是丁腈手套、安全眼镜和实验室外套。
注意:裂解物是从表达EGFR-GFP的贴壁CHO细胞制备的。将细胞接种在60mm组织培养物(TC60)培养皿中过夜12,13。在DMEM中培养CHO细胞,并补充10%胎牛血清,1%L-谷氨酰胺,1%青霉素 - 链霉素和500ng / mL遗传素(见 材料表)。也可以使用其他贴壁细胞系或悬浮细胞。
- 按照以下步骤对细胞进行平板。
- 用1毫升1x PBS洗涤培养皿(含有细胞)。加入1mL 1x胰蛋白酶,并在37°C下孵育5分钟以分离细胞。使用移液管,将分离的细胞从培养皿中转移到1.5 mL离心管中。
- 取10μL台盼蓝,在单独的离心管中与10μL细胞悬浮液混合。根据制造商的说明,在自动细胞计数器中使用10μL细胞混合物对细胞进行计数(参见 材料表)。
- 将8 x 105 个细胞在TC60培养皿中过夜。每个条件盘一道菜。
注意:对于本研究,细胞未经处理或用高钒酸盐和EGF处理,如步骤2.4.1所述。
- 为细胞裂解物制备准备以下溶液。
- 准备冰冷的1倍PBS(pH值为7.4)。
- 制备裂解缓冲液,1%非离子,非变性洗涤剂(见 材料表)的溶液,在50mM Tris-HCl(pH 7.2)和150mM NaCl中补充蛋白酶/磷酸酶抑制剂(PPI)(从库存中1:100)。将试管放在螺母上,让缓冲液混合15分钟。将准备好的溶液保存在冰上。
- 准备Tyrode的缓冲液,最终浓度为135mM氯化钠,10mM KCl,0.4mMMgCl2,1mM钙氯化物2,10mMHEPES(pH 7.2),20mM葡萄糖和0.1%牛血清白蛋白(BSA)(参见 材料表)。将溶液加热至37°C。
- 准备磷酸化阳性对照 - 1 mM钒酸盐处理。
注意:此步骤是可选的。钒酸盐是钒酸盐的过氧化形式 - 蛋白质酪氨酸磷酸酶的抑制剂16。通过抑制磷酸酶活性来防止蛋白质去磷酸化,导致高度磷酸化的样品。- 准备200 mM的活化原钒酸钠储备(Na3VO4)。
- 要制备100 mL溶液,将3.89g Na3VO4 (参见 材料表)加入90 mL ddH2O中,并在搅拌时溶解。通过滴加盐酸或钠氧混合将pH调节至10。添加HCl会使溶液变黄。
- 用ddH2O将体积降至100 mL,通过在微波炉中加热溶液将其煮沸。煮沸后,溶液将是无色的。
- 将溶液冷却至室温,并将pH值重新调整至10。重复煮沸,冷却和pH调节两到四次,直到pH稳定在10。等分试样并储存在-20°C。
- 通过将20.4μL 3%H 2O 2与100μL200mM Na3VO4和546.8μLdH 2O(等摩尔浓度为H 2O 2和活化的Na3VO4)混合,制备30mM高钒酸盐(PV)的储备。在室温下在黑暗中孵育15分钟。
- 在泰罗德的缓冲液中准备1 mM PV。对于 10 mL 溶液,将 0.33 mL 30 mM PV 储备液加入 9.67 mL 37 °C Tyrode 缓冲液中。立即处理细胞。
- 用3 mL泰罗德缓冲液洗涤细胞一次。在Tyrode的缓冲液中向细胞中加入3mL的1mM PV,并在37°C下孵育15分钟。
- 准备200 mM的活化原钒酸钠储备(Na3VO4)。
- 进行配体刺激。
- 使用适当的浓度、时间和温度刺激感兴趣的配体细胞。为了获得最大的EGFR刺激,在37°C下与1mL的50nM表皮生长因子(EGF,参见 材料表)+ 1mM PV一起在Tyrode的缓冲液中孵育5分钟。
- 进行细胞裂解。
- 进行所需的细胞处理后,将培养皿放在冰上,用冰冷的1x PBS洗涤。使用移液器完全去除整个体积的PBS。
- 向板中加入180μL裂解缓冲液(步骤2.2.2)。使用细胞刮刀在板周围拉动缓冲液以完全覆盖细胞。用细胞刮刀在整个培养表面上施加牢固,一致的压力,以完全裂解细胞。
注意:裂解缓冲液的体积需要保持在最小值,以确保高蛋白质浓度。 - 移取裂解的细胞并将其转移到1.5 mL管中。将管子放在冰上30分钟。每5分钟涡旋裂解物。
注意:如果POI由多个亚基组成或对解离敏感,请不要涡旋裂解物。 - 在4°C下以16,000× g 离心裂解的细胞20分钟。 使用移液管将上清液转移到新的1.5 mL管中。这含有总蛋白裂解物。
- 保留10μL裂解物并将其稀释到90μL裂解缓冲液中,用于双新春花比色测定(BCA)分析17。将剩余的总蛋白质裂解物储存在-80°C。
- 使用BCA分析测定裂解物总蛋白浓度(参见 材料表)。
注意:总蛋白质裂解物可以在实验日制备,并新鲜使用或在-80°C下作为一次性等分试样储存长达12周。不要冷冻/解冻。
3. 用生物素化抗体使阵列功能化
- 准备以下解决方案。
- T50缓冲液,10 mM 三氯化碳 (pH 8.0) 和 50 mM 氯化钠的溶液。该溶液在室温下稳定1个月。
- T50-BSA通过补充0.1毫克/毫升的BSA来补充T50缓冲液。将准备好的溶液保存在冰上。
- 10 毫克/毫升硼氢化钠 (NaBH4) 在 1x PBS 中。使用前请立即准备。
- 0.2 mg / mL中性萘丁(见 材料表)在T50缓冲液中。
注意:NaBH4 是一种还原剂,易燃。使用后始终用氮气吹扫容器,并将其储存在干燥器中。
- 用生物素化抗体使阵列功能化。
- 从冰箱中取出PEG-生物素功能化阵列,并在打开前将锥形管平衡至RT。将盖玻片与阵列朝上放在衬有100 mm组织培养(TC100)的密封膜上。
注:尽量减少头顶照明。所有溶液都应在疏水阵列定义的方块上"串珠"。加入适当体积的溶液以完全覆盖每个正方形(通常为10-15μL),并且不允许液体溢出定义的空间。要快速去除液体,请使用连接到真空瓶的内部真空管线来捕获废物。让NaBH4 脱气1小时,然后在化学通风橱中打开管子,然后处理。NaBH4 处理对于减少背景自发荧光是必要的,从而减少假阳性单分子检测。 - 用10mg / mL NaBH4 在1x PBS中处理阵列的每个方块,在室温下洗涤4分钟。
- 将每个正方形与0.2mg / mL的纽特罗维丁在T50中孵育5分钟。用 T50-BSA 洗涤三次。
注意:NeutrAvidin与PEG生物素结合,并为生物素化抗体12,13,15提供结合位点。 - 在T50-BSA中用2μg/ mL生物素化POI特异性抗体孵育每个正方形10分钟;用 T50-BSA 洗涤三次。
注:本方案使用生物素化抗EGFR IgG(见 材料表)来捕获EGFR-GFP。
- 从冰箱中取出PEG-生物素功能化阵列,并在打开前将锥形管平衡至RT。将盖玻片与阵列朝上放在衬有100 mm组织培养(TC100)的密封膜上。
4. 来自全细胞裂解物的POI的抽吸
注意:将功能化SiMUll阵列的TC100培养皿放在冰上,用于剩余的SiMPull制备。此步骤是从总蛋白裂解物中拉出POI。裂解物在解冻后不得重复使用。
- 准备以下解决方案。
- 在1x PBS中制备4%多聚甲醛(PFA)/ 0.1%戊二醛(GA)。
注意:PFA和GA是有毒的化学固定剂和潜在的致癌物质。穿戴个人防护用品。根据当地法规和准则将化学品作为危险废物处置。 - 制备10 mM三盐酸,pH 7.4。
- 在1x PBS中制备4%多聚甲醛(PFA)/ 0.1%戊二醛(GA)。
- 解冻并通过轻轻地上下移液来混合裂解物。保持在冰上。
- 将1μL裂解物稀释成100μL冰冷的T50-BSA / PPI。
注意:如果需要,通过对阵列施加一系列稀释来确定总蛋白裂解物的适当稀释因子。每个阵列面积SiMPull受体的最佳密度为0.04-0.08 / μm2。裂解液稀释度可以在步骤6(数据分析)中评估。 - 将裂解物在阵列上孵育10分钟;然后用冰冷的 T50-牛血清白蛋白/PPI 洗涤四次。
- 在冰冷的T50-BSA / PPI中稀释AF647偶联的抗磷酸酪氨酸抗体(见 材料表),并在阵列上孵育1小时。
注意:在本实验方案中,泛抗pTyr (PY99)-AF647 IgG用于鉴定EGFR-GFP的磷酸化群体。直接标记抗体的使用消除了对二抗的需求,增加了标记选项并提高了结果的一致性。荧光标记的抗体可以从商业来源获得。如果没有市售,可以使用标准生物偶联技术定制标记抗体,并且可以使用商业生物偶联试剂盒。每批荧光标记的抗体都需要通过执行SiMPull来测量剂量曲线并找到饱和点,以达到最佳的标记条件。 - 用冰冷的T50-BSA洗涤六次,共6-8分钟。
- 用冰冷的 1x PBS 清洗两次。
- 将阵列与4%PFA / 0.1%GA溶液孵育10分钟以防止抗体解离。
- 用10mM Tris-HCl,pH 7.4 / PBS洗涤两次5分钟,使固定剂失活。
注意:对于使用一种以上抗体的实验(例如,检测多个磷酸酪氨酸位点),重复步骤4.5-4.9。有关确定两种抗体之间的空间位阻的信息,请参见步骤6.2.9。
5. 图像采集
注意:单分子图像采集是使用150倍TIRF物镜和图像分离器进行的,该图像分离器可捕获eMCD相机特定象限中的每个光谱通道(参见 材料表)。首先获取校准图像,以便使用纳米图案通道对准网格(nanogrid)进行通道配准和相机增益校准,该网格包含20 x 20个200±50 nm孔的阵列,孔内距离为3± 1 μm(总尺寸〜60 μm×60 μm)。
- 按照以下步骤执行频道注册。
注意:需要精确的通道配准才能正确计算发射器的共定域。此步骤至关重要。- 清洁油目标,并在物镜上沉积一滴油。将纳米网格放在舞台上进行成像。使用透射的白光,聚焦在网格图案上。
注意:使用透射光获取具有纳米网格的图像,透射光通过纳米网格并在所有光谱通道中检测到。或者,可以使用多荧光珠,在每个通道中发射检测到的荧光。图像采集需要根据每个显微镜设置进行优化。 - 获取网格的一系列20张图像。确保像素未饱和。将图像系列另存为"基准"。
- 散焦纳米网格以创建通风图案18.采集一系列20张图像用于增益校准。将图像另存为"增益"。
- 获取一系列20张图像,通过阻挡所有光线进入相机来偏移相机。将图像另存为"背景"。
- 清洁油目标,并在物镜上沉积一滴油。将纳米网格放在舞台上进行成像。使用透射的白光,聚焦在网格图案上。
- 获取超脉冲图像。
注:在对盖玻片阵列进行成像之前,请将Tris溶液换成T50-BSA,并将阵列平衡至RT。- 清洁油物镜并在物镜上沉积额外的油。将盖玻片阵列固定在显微镜载物台上。
- 优化每个荧光团的激发功率、TIRF 角度和相机积分时间。目标是实现最高的信噪比,同时最大限度地减少样品的光漂白。记录激光功率,以确保将来测量的一致性。
注:本研究对远红色通道使用300 ms曝光时间,对绿色通道使用1 s。642 nm激光器以大约500 μW的激光功率使用,而488 nm激光器在860 μW的激光器下使用,在管透镜之前测量。 - 采集每个样品的图像。首先对远红色通道进行成像,然后对每个较低波长的荧光团进行成像,以减少光漂白。由于每个样品使用的体积较小,因此每30-45分钟检查一次缓冲液水平,并根据需要补充。
6. 数据分析
- 下载演示代码。
注意:提供的演示代码和示例数据集演示了完整的数据分析工作流程(补充编码文件 1-4)。SiM普尔主要.m中列出的系统要求可在 补充编码文件1中找到。-
解压缩 并 保存到 个人文档/材料实验室 (MacOS/Linux) 或文档\材料实验室(视窗)目录中。
注意:这将生成四个新文件夹:SiMPull_class、smite、样本数据、样本分析输出。 - 打开在SiMPull_class文件夹中找到的"ReadMe_Setup.txt"文件。
- 安装 MATLAB 和 MATLAB 工具箱:曲线拟合工具箱、并行计算工具箱以及统计和机器学习工具箱。
- 根据下载说明安装双图像19 。
- 安装"smite"单分子分析包,如ReadMe_setup.txt中所述。
注意:"smite"在 GitHub 存储库上可用(请参阅 材料表)。 - 通过将文件拖动到 MATLAB 窗口中,打开位于SiMPull_class文件夹中的 SiM普尔主屏幕。
- 将目录更改为 ...\MATLAB\示例数据\,方法是单击" 浏览文件夹 "图标并选择文件夹。
-
解压缩 并 保存到 个人文档/材料实验室 (MacOS/Linux) 或文档\材料实验室(视窗)目录中。
- 数据处理步骤概述
- 运行 SiM普尔主要.m - 按照每个部分的说明进行操作。通过将光标放在该部分中并单击"运行部分"图标来单独执行每个部分。
注意:本节介绍了数据分析的一般步骤。详细说明可在随附的 SiM 普尔主干代码中找到。 - 运行"初始化"部分以设置定义光谱通道和图像大小的路径。
- 运行"查找相机增益和偏移"部分,使用"增益"和"背景"数据集将相机增益转换为光子。
- 运行"通道配准"部分,计算用于图像配准的局部加权均值变换。
- 格式化和整理数据。运行"将顺序通道加入四映像"部分。运行"删除坏帧"。
- 运行"拟合单个分子并找到重叠分子"部分。
注意:本节执行多个函数来定位每个通道中的单个分子,并确定光谱通道之间的共定位事件。 - 要确定每个真实GFP拟合的最小光子计数,请运行"优化最小光子阈值"。这是一个迭代过程。
- 首先,设置Thresholding_MinPhotons = [0, 0, 0] 并运行 该部分。出现提示时选择"空白数据"文件。对"乔·埃格弗雷·格普法尔"文件重复上述步骤。
- 通过比较两个直方图来选择适当的最小阈值。设置Thresholding_MinPhotons = [475, 0, 0],然后再次运行该部分。
- 运行"计算 GFP 拟合对 FR 信号的正值百分比"部分以更正背景定位并计算最终值。
- 根据实验需要执行选项1(步骤6.2.10)或选项2(步骤6.2.11)。
- 选项1:校正质膜上可用于配体结合的受体数量,如参考文献15所述。
- 首先,用饱和水平的荧光NHS酯染料标记表面受体(NHS-AF647,参见 材料表)。然后执行SiMPull实验,以确定与AF647共定位的GFP定位的百分比。
注意:这提供了可用于NHS标记的受体分数和表面受体比例(SR)的估计值。 - 在最终计算中应用 SR 校正:NGFP = (NLOC - NBG)*SR,其中 NGFP 是校正后的 GFP 本地化数,NBG 是后台本地化编号,NLOC 是总本地化数。
注:在本例中,不应用此校正,因为钒酸盐是膜可渗透的16 ,因此,磷酸酶抑制的作用不限于表面受体。
- 首先,用饱和水平的荧光NHS酯染料标记表面受体(NHS-AF647,参见 材料表)。然后执行SiMPull实验,以确定与AF647共定位的GFP定位的百分比。
- 选项2:对于多位点磷酸化测量,考虑使用两种抗体时空间位阻的可能性。
注意:与单独的抗体 1 (P1) 相比,空间位阻可能导致在抗体 2 (P12) 存在的情况下观察到的抗体 1 磷酸化受体百分比降低。- 使用SiMPull确定P1和P12,并计算空间位阻的校正因子,遵循先前发布的参考文献15。
- 运行 SiM普尔主要.m - 按照每个部分的说明进行操作。通过将光标放在该部分中并单击"运行部分"图标来单独执行每个部分。
结果
图 1A显示了一幅描绘SiMPull过程的卡通。使用新链阿维丁作为生物素化抗EGFR抗体的锚点,从总蛋白裂解物中捕获EGFR-GFP,盖玻片被功能化。洗去未结合的蛋白质后,磷酸化的受体用抗磷酸酪氨酸(anti-PY)抗体15标记。 图1B 显示了疏水阵列的图像,其中可以制备多个样品并在同一盖玻片上成像。该样品架的一个优点是需要~10 μL的最小样品体积。盖玻片可以通过将其直接放在显微镜载物台上来成像。但是,使用盖玻片支架稳定盖玻片是有帮助的。 图1B 所示的盖玻片支架是使用3D打印机创建的,蓝图在 补充编码文件5中提供。疏水性油墨的自发荧光是找到样品焦平面的有用指南(图1C)。多通道原始图像的示例如图 1D 所示。原始绿色和远红色通道的叠加 如图1E所示。
图 2 概述了分析工作流程,并提供了具有代表性的数据。数据采集首先从采集信道配准基准开始,用于叠加各个频谱信道数据(图2A)。使用纳米网格图案拍摄明场图像,该图案通过白光并在图像分配器的每个光谱通道中检测到(未显示)。绿色通道充当参考通道,远红色通道是移位通道。局部加权均值变换是使用 MATLAB 中的 拟合矩阵20 函数计算的,用于将远红色坐标移动到绿色通道的坐标系中。此变换在每个控制点使用二阶多项式模型。然后采集SiMPull阵列的多通道数据。该工作流程包括半自动采集,其中为特定样本方块选择起始ROI,并对该区域周围的三个区域进行成像,使得每个数据集都包含来自三个独立ROI的完整四视图图像(图1D)。在每个光谱通道中,通过将高斯滤波器的差异应用于图像并识别局部最大值来找到发射器候选位置。子区域(框, 图2B)围绕局部最大值绘制,发射器光子计数通过假设每个子区域仅包含一个发射器来估计。保留光子计数高于最小值的发射极候选子区域的子区域以进行拟合。高斯点扩散函数 (PSF) 将每个候选发射极拟合到大致以每个发射器为中心的小子区域中。生成的定位基于其光子计数、背景、拟合坐标的克拉梅尔-拉奥下限、PSF 方差(即 PSF 宽度)和描述 PSF 模型拟合优度的 p 值进行阈值。为每个光谱通道创建高斯图像,在每个良好拟合的坐标处放置均匀强度的高斯斑点(图2C)。通过使用从基准样本计算的变换叠加来自每个光谱通道的高斯图像来可视化共定位(图2D)。荧光标记受体以进行鉴定很重要,因为当细胞裂解物存在时,抗磷酸酪氨酸抗体仍与表面存在非特异性结合。EGFR-GFP(绿色通道)用于生成受体位置的掩模,并且仅计算该掩模内的AF647-抗PY信号(远红色通道)(图2D)。1 像素(106.7 nm 像素大小)内的对被视为共定位并保存到包含参考通道坐标的列表中。计算与GFP共定位的AF647的百分比以确定磷酸化受体的比例(图2E)。
有几个关键步骤可以确保良好的数据质量。其中一项努力是将盖玻片阵列与NaBH4 孵育,如实验方案中所述,以淬灭绿色通道中的自发荧光。这种自发荧光是指由于玻璃上可能存在的杂质而产生的非特异性信号,含有单一或共轭π键21。这些杂质可能来自功能化过程中使用的氨基硅烷和PEG试剂或空气中的灰尘,并且倾向于在绿色光谱通道中发出荧光。尽管努力将玻璃储存在氮气下,但这些分子也可能通过在储存过程中发生的氧化产生。NaBH4 还用于减少载玻片和微阵列上杂质的荧光,包括带有硅烷涂层16的载玻片和微阵列。 图3A 显示了用NaBH4处理食人鱼蚀刻玻璃时发生的背景检测次数的减少。虽然NaBH4 显着降低了背景荧光,但在绿色通道中仍然检测到一些发射器。可以通过从无裂解物的样品中获取背景图像(图3D)并从含有GFP的样品中减去平均背景定位次数来纠正这一点。在远红色通道中未检测到来自杂质的荧光。如果受体密度太高,可以在单个衍射极限点内找到多个GFP发射器(数据未显示)。使用步进光漂白来识别每个点的GFP数量,我们发现0.04-0.08 proteins / μm2 之间的受体密度在单个发射器之间提供了足够的间距,以消除每个点12找到多个发射器的可能性。受体密度可以通过改变与玻璃表面结合的IP抗体的量或加入裂解物的量来优化。确保靶向POI的抗体在饱和水平下使用至关重要。建议在磷酸化样品上获取抗体浓度曲线,以确定适当的标记条件(图3B)。此外,抗体的磷酸特异性需要通过静息样品和/或用蛋白质特异性激酶抑制剂处理来验证(图3B)。抗体将在成像时间窗口内从受体中解离。用PFA和GA的组合处理样品可防止信号丢失(图3C)。
最后,优化单分子拟合参数非常重要。识别潜在候选发射极的第一个"方框查找"步骤(图2B)需要慷慨大方,以允许许多候选者进行高斯拟合。因此,用于发现盒子的最小光子阈值可能相对较低,以捕获所有真实发射器和一些背景点。同样重要的是,不要将箱子大小和重叠余量设置得太小。将框大小保持在 5-7 像素以内并允许两个像素重叠,是推荐密度的发射器的理想选择。找到盒子后,需要优化拟合步骤中的最小光子阈值。最小光子参数有助于确定哪些高斯拟合射极作为真拟合通过。为了确定真GFP拟合的适当最小光子阈值,该代码包括一个直方图绘图函数,用于检查背景(无细胞裂解物)和含GFP(加细胞裂解物)样品中的光子/定位(图3D)。此步骤很重要,因为虽然NaBH4 减少了杂质的荧光量,但它不会去除所有背景定位。 图3D 演示了需要设置最小光子阈值以减少杂质检测的数量。为了确定该阈值,通过对未暴露于细胞裂解物的样品进行成像来计算背景发射器强度的直方图(图3D,左上角)。大多数背景发射器的值小于475光子。相比之下,包含真GFP发射器的样品显示出475以上分布的很大一部分(图3D,右上角)。通过检查选择阈值以消除尽可能多的背景计数,同时最大限度地减少裂解物样品的信号损失量(图3D,底行)。此阈值处的剩余背景计数密度在定量分析中被考虑在内。
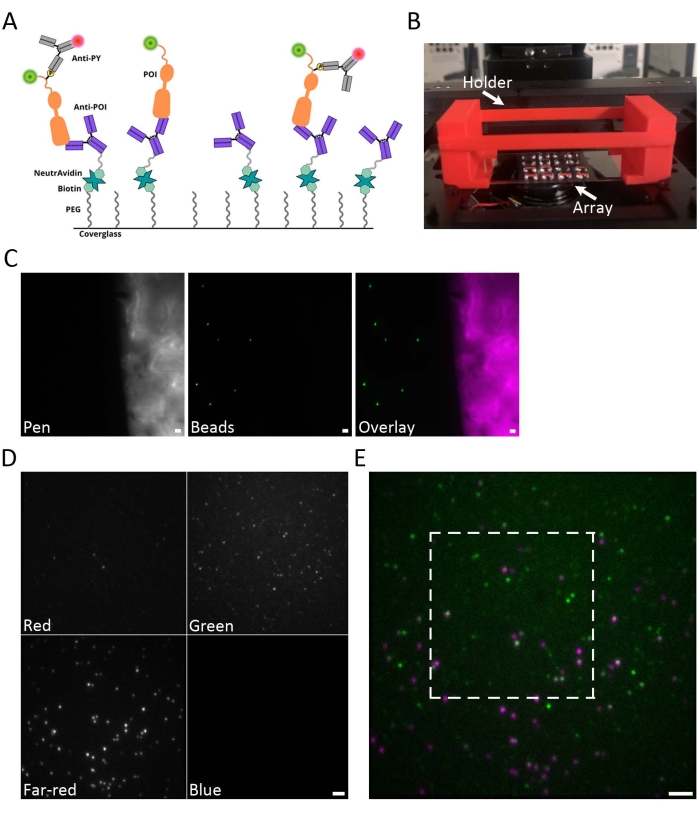
图 1:样品制备概述 (A) 描绘 SiMPull 方法的卡通。盖玻片用一种抗体功能化,该抗体识别POI以从整个细胞裂解物中捕获该POI。玻璃首先涂有聚乙二醇和生物素聚乙二醇。然后,NeutrAvidin与生物素-PEG结合,并充当生物素化抗POI抗体的锚点。然后用荧光标记的抗PY抗体检测磷酸化蛋白质。(B)盖玻片支架(红色)的照片,盖玻片阵列已到位并安装在显微镜载物台上。多样本阵列使用疏水性油墨生成,在单个玻璃盖玻片上创建多达20个单独的样品方块。盖玻片为60 mm x 24 mm.(C)疏水性油墨自发荧光(洋红色)和荧光珠(绿色)的示例图像。疏水性油墨的自发荧光是找到盖玻片表面焦平面的有用指南。(D) 原始数据图像示例,其光谱通道在相机芯片上由四视图图像分离器分隔。四视图滤光片集包括以下发射滤光片:蓝色 (445/45 nm)、绿色 (525/45 nm)、红色 (600/37 nm)、远红 (685/40 nm)。(E) 绿色和远红色通道的原始叠加。白色框表示图2B-D中进一步检查的区域。比例尺 (C-E) = 2 μm.请单击此处查看此图的大图。
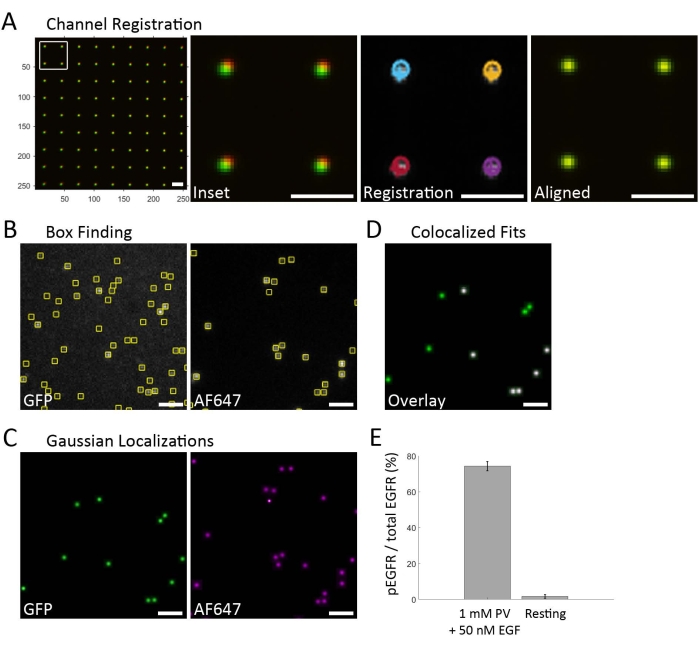
(A)首先对从纳米网格获取的图像进行通道配准。裁剪感兴趣的两个光谱通道(此处为绿色和远红色)后,将覆盖每个通道的基准图像(左图)。左图(插图)中框的放大显示图像尚未真正注册。每个通道中的发射器都符合高斯模型并进行局部化(配准)。发射器的定位显示为远红色通道的圆圈和绿色通道的十字。最后一步是应用局部加权均值变换,将远红色通道定位坐标移动到绿色通道参考系(对齐)中。然后,计算出的局部加权均值变换用于注册后续的SiMPull数据。(B) 绿色/EGFR-GFP 通道和远红色/AF647 反 PY 通道的代表性图像。背景光子计数以上的单个发射器被识别并用框标记。(C) 每个选定框中的发射曲线适合高斯模型,并保留适合单个荧光团PSF模型的发射器。(D) 从 GFP 发射器创建掩模以识别 EGFR-GFP 的位置(绿色)。EGFR-GFP 和 AF647-抗 PY 的共定位可识别磷酸化受体(白色)。(E)磷酸化受体的比例是根据共定位的EGFR-GFP和AF647-抗PY拟合计算的。条形图将PV + EGF处理与静息细胞进行比较,并进行了多次测量的平均值。误差线表示假设二项式分布计算的标准误差。比例尺 = 2 μm.请点击此处查看此图的大图。

(A)从左到右,前三个面板是在各自条件下玻璃上自发荧光的代表性图像:食人鱼蚀刻后,用PEG和PEG加NaBH4处理(用+表示)。此外,在NaBH4处理后保留了表面官能化,如最小的非特异性PY99-AF647结合所示,同时保留了裂解物中EGFR-GFP的稳健结合。(B)必须为每批抗体获取饱和度曲线,以确保最佳的抗体标记。该图显示了用位点特异性磷酸酪氨酸抗体抗EGFR-pY1173标记EGFR的浓度曲线。在未经处理的细胞(静止,灰色钻石)中检测到最小的磷酸化。作为非特异性结合的对照,在加入100 nM的EGF(品红色三角形)之前,还用EGFR激酶抑制剂拉帕替尼处理细胞,这表明EGFR磷酸化的预期预防。误差线表示假设二项式分布的标准误差。(C)用PFA和GA的组合固定样品可防止抗体随时间推移解离。误差线表示假设二项式分布的标准误差。(D) 通过为高斯拟合选择适当的阈值来排除误报。比较背景(无裂解物)和真实数据(加上细胞裂解物)之间的低阈值(阈值 = 0; top)下拟合强度的直方图允许选择适当的值(阈值 = 475; 底部)以从绿色通道中的自发荧光点移除拟合。垂直洋红色线表示475光子阈值。直方图是根据每种样本类型的相同 ROI 数计算得出的 (n = 3)。比例尺 = 2 μm.请点击此处查看此图的大图。
补充编码文件 1:包含用于运行 SiMPull 分析的脚本和实用程序的 zip 文件。 请按此下载此档案。
补充编码文件2: 包含 smite 单分子分析包的zip文件。 请按此下载此档案。
补充编码文件 3: 包含示例数据的 zip 文件。 请按此下载此档案。
补充编码文件4: 包含代表性样本数据分析输出的zip文件。 请按此下载此档案。
补充编码文件 5: 用于3D打印的盖玻片支架蓝图。 请按此下载此档案。
讨论
这里描述的方案经过优化,可以在单一蛋白质水平上定量测量受体磷酸化。对SiMPull方案进行了一些简单但重要的修改,提高了磷酸酪氨酸检测测量的可靠性,包括通过NaBH4 处理减少自发荧光和后固定样品以防止抗体解离。使用绿色通道掩模识别受体位置以计算与抗PY抗体的共定位,还可以通过消除抗体与细胞裂解物的非特异性结合中的潜在伪影来提高测量精度。利用双色成像来检测磷酸化受体的比例。在这种情况下,受体用GFP进行遗传标记,抗体直接用远红色染料标记。SiMPull方法适用于具有特定抗体的其他蛋白质靶标,包括细胞内蛋白质。此外,由于不需要变性条件,因此也可以捕获多苏布尼受体/复合物。然而,如果感兴趣的PTM位于蛋白质14的结构区域中,则可以掺入变性。最终,SiMPull可以很容易地扩展为包括同时标记单个受体上不同的磷酸酪氨酸,以量化多位磷酸化模式15。以这种方式询问全长,完整的受体不能通过其他标准方法实现,包括蛋白质印迹和磷酸化质谱。
除了SiMPull的优势外,还需要考虑一些限制。与任何基于抗体的技术一样,所用抗体的亲和力和特异性对于测量的成功至关重要。因此,重要的是优化抗体标记条件,并通过使用直接标记的一抗来理想地避免二抗。此外,表面结合的抗体将沉淀定位于质膜和胞质区室内的蛋白质。这可能会低估磷酸化,因为外源性添加的配体无法进入细胞质基质定位的蛋白质。必须采取额外的步骤来纠正受体表面水平(步骤6.2.10)。抗磷酸酪氨酸抗体一旦存在裂解物就表现出一些非特异性结合。为了避免这种伪影,EGFR被用GFP进行基因标记以识别受体的位置,这使我们能够从受体上排除抗PY信号。如果要询问内源性蛋白质,则用总蛋白抗体进行复染可以提供掩模图像,并对任何非特异性结合进行适当的校正。最后,虽然SiMPull提供了有关蛋白质水平异质性的信息,但该方案中产生的裂解物来自数千个细胞,并且细胞间变异性丢失。然而,单细胞SiMPull的发展已经使用由盖玻片和具有10μm间隙的显微镜侧组成的流动室;将细菌稀疏地接种在盖玻片上,同时用抗体对载玻片进行功能化以捕获所需的蛋白质。在细菌裂解后,来自每个细胞的蛋白质在抗体包被的载玻片22上的密闭区域捕获。类似的单细胞SiMPull哺乳动物细胞和蛋白质磷酸化分析可能在未来成为可能。
SiMPull协议包含确保高质量数据所需的几个关键步骤。例如,该协议包括盖玻片玻璃的精心制备。食人鱼蚀刻盖玻片彻底清洁玻璃并增加羟基和亲水性,这是优化盖玻片表面所必需的。在用有机溶剂洗涤几次之后,KOH处理为氨基硅烷化13,23提供额外的羟基,其用胺基团涂覆玻璃,用于PEG和生物素- PEG结合。任何这些步骤的不当清洁或功能化都会干扰蛋白质的下拉。控制PEG:生物素-PEG的摩尔比以及裂解物浓度是在SiMPull底物上获得适当蛋白质IP密度的关键因素。与任何生物测定一样,细胞裂解物制剂之间存在可变性,并且在样品重复之间可以看到磷酸化百分比之间的微小差异。因此,测量同一样品中不同酪氨酸位点的磷酸化水平非常重要。该协议中描述的样品室提供了一个系统,用于在一个成像会话中收集许多数据点,并允许对多个SiMPull实验进行平均。
在图像采集方面,重要的是获得基准样本,以确保准确的通道叠加;否则,共定位将不准确。优化激光功率和相机设置以最大化信噪比,同时最大限度地减少光漂白也很重要。最后,虽然样品阵列需要少量的样品和试剂,但在成像过程中,低体积容易蒸发。重要的是定期检查样品阵列(约30-45分钟)并根据需要加入缓冲液以防止样品干燥。
本方案证明了使用SiMPull来量化膜受体磷酸化状态。虽然专注于EGFR,但只要有适当的抗体可用,该方法可以应用于其他细胞表面受体和细胞内蛋白质和蛋白质复合物。SiMPull的另一个潜在用途是询问相分离冷凝物的内容物和磷酸化状态。此外,SiMPull可用于测量其他PTM,例如泛素化。因此,SiMPull为细胞生物学家提供了一种独特的工具,可以询问完整蛋白质上的PTM,并将PTM模式与细胞结果相关联。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了美国国立卫生研究院R35GM126934,R01AI153617和R01CA248166的支持。EMB通过阿塞特-伊拉克开发区计划(NIH K12GM088021)和UNM MARC计划(NIH 2T34GM008751-20)提供支持。我们非常感谢使用新墨西哥大学综合癌症中心荧光显微镜共享资源,由NIH P30CA118100支持。我们要感谢安库尔·贾恩博士和哈太基吉普博士,他们最初对SiMPull的开发启发了这项工作。
ES-C现在地址:免疫动力学组,综合癌症免疫学实验室,癌症研究中心,国家癌症研究所,贝塞斯达
材料
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes | MTC Bio | C2000 | |
| 10 mM Tris-HCl pH 7.4 | |||
| 10 mM Tris-HCl pH 8.0/ 50 mM NaCl | T50 Buffer | ||
| 100 mm Tissue Culture dish | CELLTREAT | 229620 | Storage of piranha etched glass/arrays |
| 15 mL conical tube | |||
| 16% Paraformaldehyde Aqueous Solution | Electron Microscopy Sciences | 15710 | Hazardous |
| 50 mL conical tube | Functionalized Glass storage/ KOH reuse | ||
| 50 mM Tris-HCl pH 7.2/150 mM NaCl | Lysis Buffer Component | ||
| 60 mm Tissue Culture dish | Corning | 430166 | |
| 8% Glutaraldehyde Aqueous Solution | Electron Microscopy Sciences | 16020 | Hazardous |
| Acetone (C3H6O) | Millipore Sigma | 270725 | Hazardous |
| Alexa Fluor 647 NHS Ester | Thermo Fisher Scientific | A-20006 | |
| Animal-Free Recombinant Human EGF | Peprotech | AF-100-15 | |
| Anti-Human EGFR (External Domain) – Biotin | Leinco Technologies, Inc | E101 | |
| Anti-p-Tyr Antibody (PY99) Alexa Fluor 647 | Santa Cruz Biotechnology | sc-7020 AF647 | |
| Bath-sonicator | Branson | 1200 | |
| BCA Protein Assay Kit | Pierce | 23227 | |
| Biotin-PEG | Laysan Bio | Biotin-PEG-SVA, MW 5,000 | |
| Bovine serum albumin | Gold Biotechnology | A-420-1 | Tyrode's Buffer Component |
| Buchner funnel | |||
| Bunsen burner | |||
| Calcium Chloride (CaCl2) | Millipore Sigma | C4901 | Tyrode's Buffer Component |
| Cell Scraper | Bioworld | 30900017-1 | |
| Conical Filtering Flask | Fisher Scientific | S15464 | |
| Coplin Jar | WHEATON | 900470 | |
| Countess II Automated Cell Counter | Thermo Fisher Scientific | AMQAX1000 | |
| Coverslips 24 x 60 #1.5 | Electron Microscopy Sciences | 63793 | |
| DipImage | https://diplib.org/ | ||
| DMEM | Caisson Labs | DML19-500 | |
| emCCD camera | Andor iXon | ||
| Fetal Bovine Serum, Optima | Bio-Techne | S12450H | Heat Inactivated |
| Fusion 360 software | Autodesk | ||
| Geneticin G418 Disulfate | Caisson Labs | G030-5GM | |
| Glacial Acetic Acid (CH3COOH) | JT Baker | JTB-9526-01 | Hazardous |
| Glass serological pipettes | |||
| Glass Stir Rod | |||
| Glucose (D-(+)-Glucose) | Millipore Sigma | D9434 | Tyrode's Buffer Component |
| Halt Phosphotase and Protease Inhibitor Cocktail (100X) | Thermo Fisher Scientific | 78446 | Lysis Buffer Component |
| HEPES | Millipore Sigma | H3375 | Tyrode's Buffer Component |
| Hydrochloric Acid (HCl) | VWR | BDH7204-1 | Hazardous |
| Hydrogen Peroxide (H2O2) (3%) | HX0645 | ||
| Hydrogen Peroxide (H2O2) (30%) | EMD Millipore | HX0635-2 | |
| Ice | |||
| IGEPAL CA-630 (NP-40) | Sigma Aldrich | I8896 | Lysis Buffer Component |
| ImmEdge Hydrophobic Barrier Pen | Vector Laboratories | H-4000 | |
| Immersol 518F immersion oil | Zeiss | 444960-0000-000 | |
| in-house vacuum line | |||
| L-glutamine | Thermo Fisher Scientific | 25030-164 | |
| Magnessium Chloride Hexahydrate (MgCl2-6H2O) | MPBio | 2191421 | Tyrode's Buffer Component |
| Matlab | Mathworks | Curve Fitting Toolbox, Parallel Computing Toolbox, and Statistics and Machine Learning toolbox | |
| Methanol (CH3OH) | IBIS Scientific | MX0486-1 | Hazardous |
| Milli-Q water | |||
| Mix-n-Stain CF Dye Antibody Labeling Kits | Biotium | 92245 | Suggested conjugation kit |
| mPEG | Laysan Bio | mPEG-succinimidyl valerate, MW 5,000 | |
| N-(2-aminoethyl)-3-aminopropyltrimethoxysilane | UCT United Chemical | A0700 | Hazardous |
| Nanogrid | Miraloma Tech | ||
| NeutrAvidin Biotin Binding Protein | Thermo Fisher Scientific | 31000 | |
| Nitrogen (compressed gas) | |||
| NVIDIA GPU with CUDA | Look for compatibility at https://www.mathworks.com/help/parallel-computing/gpu-support-by-release.html | ||
| Olympus iX71 Microscope | Olympus | ||
| Parafilm M Sealing Film | The Lab Depot | HS234526C | |
| PBS pH 7.4 | Caisson Labs | PBL06 | |
| PC-200 Analog Hot Plate | Corning | 6795-200 | |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher Scientific | 15140-163 | |
| Phospho-EGF Receptor (Tyr1068) (1H12) Mouse mAb | Cell Signaling Technology | 2236BF | |
| Potassium Chloride (KCl) | Millipore Sigma | 529552 | Tyrode's Buffer Component |
| Potassium Hydroxide (KOH) | Millipore Sigma | 1050330500 | Hazardous |
| Premium PLA Filament, 1.75 mm diameter | Raise 3D | PMS:2035U/RAL:3028 | Printing temperature range: 205-235 °C |
| Pro2 3D printer | Raise 3D | ||
| Pyrex 1 L beaker | |||
| PYREX 100 mL storage bottles | Corning | 1395-100 | CH3OH/C3H6O reuse |
| Pyrex 250 mL beakers | |||
| Pyrex 4 L beaker | |||
| Quad-view Image Splitter | Photometrics | Model QV2 | |
| Refrigerated centrifuge | Eppendorf | EP-5415R | |
| RevCount Cell Counters, EVE Cell Counting Slides | VWR | 10027-446 | |
| Semrock emission filters: blue (445/45 nm), green (525/45 nm), red (600/37 nm), far-red (685/40 nm) | Semrock | LF405/488/561/635-4X4M-B-000 | |
| Serological pipette controller | |||
| Serological Pipettes | |||
| smite single molecule analysis package | https://github.com/LidkeLab/smite.git | ||
| Sodium Bicarbonate (NaHCO3) | Sigma Aldrich | S6014 | Hazardous |
| Sodium Borohydride (NaBH4) | Millipore Sigma | 452874 | Tyrode's Buffer Component |
| Sodium Chloride (NaCl) | Millipore Sigma | S9625 | Activate by successive heat and pH cycling |
| Sodium Hydroxide | VWR | BDH3247-1 | |
| Sodium Orthovanadate (Na3VO4) | Millipore Sigma | S6508 | Hazardous |
| Sulfuric Acid (H2SO4) | Millipore Sigma | 258105 | Hazardous |
| TetraSpeck Microspheres | Thermo Fisher Scientific | T7279 | multi-fluorescent beads |
| Tris (Trizma) base | Millipore Sigma | T1503 | |
| Trypan blue stain, 0.4% | Thermo Fisher Scientific | 15250061 | |
| Trypsin-EDTA 0.05% | Thermo Fisher Scientific | 25300120 |
参考文献
- Lemmon, M. A., Schlessinger, J. Cell Signaling by Receptor Tyrosine Kinases. Cell. 141 (7), 1117-1134 (2010).
- Seet, B. T., Dikic, I., Zhou, M. M., Pawson, T. Reading protein modifications with interaction domains. Nature Reviews Molecular Cell Biology. 7 (7), 473-483 (2006).
- Coba, M. P., et al. Neurotransmitters drive combinatorial multistate postsynaptic density networks. Science Signaling. 2 (68), (2009).
- Gibson, S. K., Parkes, J. H., Liebman, P. A. Phosphorylation modulates the affinity of light-activated rhodopsin for g protein and arrestin. Biochemistry. 39 (19), 5738-5749 (2000).
- Stites, E. C., et al. Use of mechanistic models to integrate and analyze multiple proteomic datasets. Biophysical Journal. 108 (7), 1819-1829 (2015).
- Hause, R. J., et al. Comprehensive binary interaction mapping of SH2 domains via fluorescence polarization reveals novel functional diversification of ErbB receptors. PLOS ONE. 7 (9), 44471(2012).
- Lau, E. K., et al. Quantitative encoding of the effect of a partial agonist on individual opioid receptors by multisite phosphorylation and threshold detection. Science Signaling. 4 (185), (2011).
- Mishra, M., Tiwari, S., Gomes, A. V. Protein purification and analysis: next generation Western blotting techniques. Expert Review of Proteomics. 14 (11), 1037-1053 (2017).
- Brunner, A. M., et al. Benchmarking multiple fragmentation methods on an orbitrap fusion for top-down phospho-proteoform characterization. Analytical Chemistry. 87 (8), 4152-4158 (2015).
- Swaney, D. L., Wenger, C. D., Coon, J. J. Value of using multiple proteases for large-scale mass spectrometry-based proteomics. Journal of Proteome Research. 9 (3), 1323-1329 (2010).
- Curran, T. G., Zhang, Y., Ma, D. J., Sarkaria, J. N., White, F. M. MARQUIS: A multiplex method for absolute quantification of peptides and posttranslational modifications. Nature Communications. 6 (1), 1-11 (2015).
- Jain, A., et al. Probing cellular protein complexes using single-molecule pull-down. Nature. 473 (7348), 484-488 (2011).
- Jain, A., Liu, R., Xiang, Y. K., Ha, T. Single-molecule pull-down for studying protein interactions. Nature Protocols. 7 (3), 445-452 (2012).
- Kim, K. L., et al. Pairwise detection of site-specific receptor phosphorylations using single-molecule blotting. Nature Communications. 7 (1), 1-10 (2016).
- Salazar-Cavazos, E., et al. Multisite EGFR phosphorylation is regulated by adaptor protein abundances and dimer lifetimes. Molecular Biology of the Cell. 31 (7), 695(2020).
- Huyer, G., et al. Mechanism of inhibition of protein-tyrosine phosphatases by vanadate and pervanadate. Journal of Biological Chemistry. 272 (2), 843-851 (1997).
- Smith, P. K., et al. Measurement of protein using bicinchoninic acid. Analytical Biochemistry. 150 (1), 76-85 (1985).
- Keller, H. E. Objective lenses for confocal microscopy. Handbook of Biological Confocal Microscopy. , 145-161 (2006).
- Hendriks, C. L. L., van Vliet, L. J., Rieger, B., van Kempen, G. M. P., van Ginkel, M. Dipimage: a scientific image processing toolbox for MATLAB. , (1999).
- fitgeotrans: Fit geometric transformation to control point pairs. The MathWorks Inc. , Available from: https://www.mathworks.com/images/ref/fitgeotrans.html (2013).
- Raghavachari, N., Bao, Y. P., Li, G., Xie, X., Müller, U. R. Reduction of autofluorescence on DNA microarrays and slide surfaces by treatment with sodium borohydride. Analytical Biochemistry. 312 (2), 101-105 (2003).
- Wang, X., Park, S., Zeng, L., Jain, A., Ha, T. Toward single-cell single-molecule pull-down. Biophysical Journal. 115 (2), 283-288 (2018).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. Journal of Visualized Experiments. (86), e50549(2014).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。