Method Article
Subcultura e Criopreservação de Organoides Adenocarcinoma Esofágico: Prós e Contras para Digestão Celular Única
Neste Artigo
Resumo
Este protocolo descreve os métodos de subcultura e criopreservação de organoides adenocarcinoma esofágico com e sem digestão celular única para permitir que os pesquisadores escolham estratégias apropriadas com base em seu desenho experimental.
Resumo
A falta de modelos de pesquisa translacional adequados que refam doenças primárias para explorar a tumorigênese e estratégias terapêuticas é um grande obstáculo no adenocarcinoma esofágico (EAC). Organoides derivados do paciente (PDOs) surgiram recentemente como um modelo pré-clínico notável em uma variedade de cânceres. No entanto, ainda há protocolos limitados disponíveis para o desenvolvimento de PDOs EAC. Uma vez estabelecidos os PDOs, a propagação e a criopreservação são essenciais para novas análises a jusante. Aqui, dois métodos diferentes foram padronizados para subcultura e criopreservação de PDOs EAC, ou seja, com e sem digestão celular única. Ambos os métodos podem obter de forma confiável a viabilidade celular apropriada e são aplicáveis para uma configuração experimental diversificada. O presente estudo demonstrou que subculturar PDOs EAC com digestão celular única é adequado para a maioria dos experimentos que requerem controle de número de células, densidade uniforme e uma estrutura oca que facilita o rastreamento de tamanho. No entanto, o método baseado em células únicas mostra um crescimento mais lento na cultura, bem como após o reap cultivo de estoques congelados. Além disso, subcultura com digestão celular única é caracterizada pela formação de estruturas ocas com um núcleo oco. Em contrapartida, processar PDOs EAC sem digestão celular única é favorável para criopreservação, expansão e caracterização histológica. Neste protocolo, as vantagens e desvantagens da subcultura e criopreservação de PDOs EAC com e sem digestão celular única são descritas para permitir que os pesquisadores escolham um método adequado para processar e investigar seus organoides.
Introdução
O câncer de esôfago (CE) é a décima causa mais comum e a sexta principal causa de morte por câncer em todo o mundo1. O adenocarcinoma esofágico (EAC) é um dos principais subtipos histológicos da CE e ocorre principalmente nos países ocidentais2. Na última década, a incidência do EAC aumentou significativamente em muitos países desenvolvidos, incluindo a Alemanha3. Devido à agressividade do câncer e à falta de sintomas durante o estágio inicial do desenvolvimento do tumor, o prognóstico geral em pacientes com EAC é ruim, mostrando uma taxa de sobrevivência de 5 anos de cerca de 20%2,4,5.
Desde o final do século XX, vários modelos foram estabelecidos para a pesquisa biomédica da EAC. As clássicas linhas celulares humanas que foram estabelecidas na década de 19906, ampliam nosso conhecimento sobre biologia tumoral EAC, genética tumoral, bem como estratégias anti-tumor, e são comumente utilizadas em pesquisas da EAC. Além disso, alguns grupos de pesquisa desenvolveram com sucesso modelos animais do EAC ou do esôfago de Barrett, expondo os animais a fatores de risco conhecidos, como o refluxo gastroesofágico através de abordagens cirúrgicas ou inflamatórias 7,8,9. Além disso, modelos de xenoenxerto derivados do paciente (PDX) que engrafam tecidos primários de câncer EAC subcutâneamente ou ortotopicamente em camundongos imunodeficientes, foram desenvolvidos para simular o comportamento biológico do tumor EAC humano e o ambiente tumoral 10,11,12. No entanto, apesar desses modelos melhorarem as aplicações clínicas e da nossa compreensão dos mecanismos moleculares por trás da tumorigênese e da progressão do EAC, ainda há um grande desafio para extrapolar os resultados desses modelos de pesquisa para os seres humanos.
Os organoides tumorais derivados do paciente (PDOs) são cultivados em um sistema de cultura 3D que imita o desenvolvimento humano e a regeneração de órgãos in vitro. Gerados a partir do tecido primário dos pacientes, os PDOs recapitulam as características moleculares e fenotípicas do tumor humano e têm mostrado aplicações promissoras no desenvolvimento de medicamentos e tratamento personalizado do câncer13,14. Comparando dez casos de PDOs EAC com seu tecido tumoral emparelhado, os PDOs do EAC relatam compartilhar características histopatológicas semelhantes e paisagem genômica com o tumor primário, reter heterogeneidade intra-tumor e facilitar a triagem eficiente de medicamentos in vitro15. Os PDOs do EAC também foram utilizados no estudo da interação das células tumorais do EAC com os fibroblastos associados ao câncer (CAFs) derivados do paciente, indicando uma aplicação poderosa no campo da pesquisa de microambiente tumoral16. Infelizmente, existem protocolos limitados disponíveis para o desenvolvimento e propagação de PDOs EAC. Aqui, dois métodos diferentes são descritos para subculturar e preservar os PDOs EAC em detalhes: com e sem digestão celular única. Os métodos padronizados para manutenção de PDOs EAC e suas aplicações podem apoiar os pesquisadores a escolher métodos apropriados para diferentes propósitos em suas pesquisas de PDO EAC.
Protocolo
Uma cultura PDO estabelecida e bem crescente representa a base para uma subcultura e criopreservação bem sucedida descritas neste protocolo. Aqui, os PDOs do EAC foram gerados a partir do tecido tumoral primário dos pacientes da EAC usando o protocolo descrito por Karakasheva T. A. et al17. Os tecidos EAC foram coletados do biobanco sob a aprovação do BioMaSOTA (aprovado pelo Comitê de Ética da Universidade de Colônia, ID: 13-091).
NOTA: Os PDOs do EAC foram cultivados em uma incubadora umidificada a 37 °C e 5% de CO2 utilizando um meio de cultura PDO (Tabela 1). Nas etapas seguintes, dois métodos da subcultura são descritos em detalhes. Uma placa de 12 poços é recomendada para subculturar os PDOs com uma densidade de semeadura de três cúpulas de gel de matriz extracelular (ECM) por bem, pois permite o uso flexível de cada bem e quantidade apropriada de PDOs para diferentes propósitos. Uma técnica asséptica é obrigatória durante o manuseio dos PDOs.
1. Preparativos com antecedência
- Pré-aqueça uma placa de 12 poços colocando-a em uma incubadora de CO2 de 37 °C durante a noite antes da subcultura para garantir o aquecimento completo da placa. Se disponível, use poços vazios de um prato com a cultura PDO atual.
NOTA: Recomenda-se o armazenamento contínuo de 1-2 placas frescas a 37 °C para o planejamento flexível da subcultura. - Pré-cool 1.000 μL e 200 μL dicas com um orifício largo a -20 °C (armazenamento contínuo recomendado). Centrífuga pré-fria a 4 °C.
- Configure a temperatura da incubadora rotativa até 37 °C (se for realizada uma digestão celular única).
- Incubar um volume apropriado de gel ECM por 1 h no gelo para liquefazer. Coloque a solução de recuperação celular no gelo.
2. Colhendo organoides
- Remova a placa com PDOs em crescimento da incubadora de CO2.
- Aspirar o meio velho usando uma bomba de vácuo.
NOTA: Evite tocar nas cúpulas. - Adicione um volume apropriado de solução de recuperação de células geladas (500 μL/cúpula) no poço.
- Desintegrem o gel ECM ao escoar várias vezes para fragmentar cúpulas de gel ECM em pequenos pedaços usando pontas de 1.000 μL com um orifício largo.
- Misture a mistura de PDO, gel ECM e solução de recuperação celular a partir de um máximo de dois poços (seis cúpulas) e transfira-o para um tubo de ligação baixa de 5 mL (use um segundo tubo no caso de mais poços serem usados para subcultura).
NOTA: Opcionalmente, se o gel ECM não foi completamente dissolvido, adicione uma solução adicional de 1,5 mL de recuperação celular à mistura de PDO, gel ECM e solução de recuperação celular. - Incubar o tubo contendo a mistura na etapa 2.5 no gelo por 20 minutos, misture a cada 5 minutos invertendo o tubo cinco vezes para garantir a liquefação do gel ECM.
- Centrifugar a 500 x g por 4 min a 4° C.
- Se houver uma pelota visível e estável após a centrifugação, proceda com a etapa 2.10. Caso contrário, continue com o passo 2.9.
- Se não houver pelota visível e os PDOs ainda parecerem estar presos em uma fase de gel, remova cuidadosamente o supernasce com uma bomba de vácuo até que a fase contendo eCM gel-PDO-Solution seja atingida e adicione 3 mL de solução de recuperação de células geladas.
- Inverta o tubo algumas vezes e incubar no gelo por mais 10 minutos. Misture invertendo o tubo de vez em quando.
- Centrifugar a 500 x g para 4 min a 4 °C e continuar com a etapa 2.10.
- Descarte o supernatante cuidadosamente usando uma bomba de vácuo ou uma pipeta de 1.000 μL. Tente remover o supernatante o máximo possível.
NOTA: Devido à superfície de baixa ligação do tubo, a pelota não será tão estável como de costume. - Armazene a pelota PDO no gelo e proceda com o passo 3 (sem digestão) ou o passo 4 (com digestão celular única) dependendo dos diferentes propósitos.
3. Subcultura sem digestão
NOTA: Este método visa aumentar o tamanho e a densidade dos PDOs. O tamanho maior e a densidade maior facilitam o processo de incorporação, caracterização histológica e expansão do PDO. Dependendo das razões de divisão do PDO (com base na densidade dos PDOs, recomenda-se uma razão entre 1:3 e 1:6), resuspenque a pelota da etapa 2.8 em um volume apropriado de gel ECM líquido.
- Remova as pontas pré-resfriadas de 200 μL e 1.000 μL com um orifício largo do congelador -20 °C e coloque-as em um banco limpo.
- Resuspenda a pelota da etapa 2.11 em gel ECM usando pontas pré-resfriadas de 1.000 μL. Misture por pipetar para cima e para baixo cerca de 10 vezes para garantir que os PDOs não estejam desajeitados e sejam distribuídos uniformemente no gel ECM.
NOTA: Use gel/cúpula de 50 μL ECM. Calcule sempre para uma cúpula mais do que o necessário (por exemplo, para nove cúpulas (ou seja, três poços), resuspense a pelota em 500 μL de gel ECM líquido (450 + 50 μL extra). Tente evitar produzir bolhas durante a ressuspensão! - Remova a placa pré-aquecida de 12 poços da incubadora antes de semear as cúpulas.
- Cúpulas de sementes contendo gel ECM de 50 μL na placa quente (três cúpulas/bem). Evite bolhas de tubulação nas cúpulas de gel ECM.
- Coloque a placa de volta na incubadora de 37 °C e 5% de CO2e incubar por 20-30 min para solidificar o gel ECM.
- Adicione cuidadosamente o meio PDO pré-aquecido (Tabela 1) sem perturbar as cúpulas.
- Cultura os PDOs por 7-14 dias até que a densidade necessária e a morfologia ocorram.
4. Subcultura com digestão celular única
NOTA: As seguintes etapas visam aumentar o número de PDOs por cúpula. A digestão celular única facilita o controle de números de células e a expansão do PDO.
- Prepare o meio de digestão misturando 2 mL de 0,25% Trypsin-EDTA e 20 μL DNase I (para digestão de três cúpulas).
- Resuspenque a pelota a partir do passo 2.11 com um volume apropriado de unha pré-aquecida de 0,25% Trypsin-EDTA + DNase I e misture-a cerca de 10 vezes por pipetar para cima e para baixo usando uma pipeta de 1.000 μL (use dicas normais de 1.000 μL).
- Incubar por 10 min a 37 °C em uma incubadora rotativa com uma velocidade de rotação de no mínimo 28 rpm.
- Prepare um tubo de 15 mL contendo 6 mL de solução inibidora de trippsina de soja (IST, Tabela 2) (por 2 mL de 0,25% Trypsin-EDTA).
- Após a digestão, misture bem os PDOs digeridos algumas vezes com uma pipeta de 1.000 μL para interromper os PDOs.
- Transfira os PDOs digeridos para o tubo de 15 mL contendo solução IST para interromper o processo de digestão.
- Centrifugar a 500 x g para 4 min a 4 °C. Descarte o supernatante cuidadosamente usando uma bomba de vácuo ou uma pipeta de 1.000 μL. Resuspense a pelota em 1 mL de meio basal (Tabela 3).
- Determine a concentração e a viabilidade celular usando um contador celular automatizado ou um Hemocitómetro.
- Seed digeriu PDO em uma placa de 12 poços com 2 x 104 células por cúpula.
- Calcule o número do celular de acordo com as cúpulas planejadas para semeadura e transfira-os para um novo tubo de ligação baixa de 1,5 mL.
NOTA: Calcule mais uma cúpula (+ 2 x 104 células extras). Por exemplo, para semear três cúpulas em um poço, pegue células 8 x 104 (2 x 104* 3 + 2 x 104 extra). - Centrifugar a 500 x g para 4 min a 4 °C.
- Caso não haja pelota visível, lembre-se da orientação do tubo dentro da centrífuga para saber onde a pelota está localizada.
- Descarte cuidadosamente o supernatante usando uma pipeta de 1.000 μL. Remova o supernatante o máximo possível sem perturbar a pelota.
- Adicione o volume apropriado de gel ECM à pelota usando uma pipeta de 1.000 μL com ponta de orifício de 1.000 μL (50 μL/dome + 50 μL extra).
- Siga os passos 3.3-3.7.
- Calcule o número do celular de acordo com as cúpulas planejadas para semeadura e transfira-os para um novo tubo de ligação baixa de 1,5 mL.
5. Criopreservação dos PDOs digeridos e não digeridos
NOTA: PDS digeridos e não digeridos de célula única são adequados para a preparação de estoques de backup congelados. Observe que os PDOs ressumados dos estoques congelados de célula única requerem um tempo maior para se recuperar e atingir um certo tamanho.
- Criopreservação dos PDOs não digeridos.
- Inicie o processo de criopreservação com a pelota a partir da etapa 2.8. Use 500 μL de meio de congelamento frio para resuspensar a pelota e transferi-la para um frasco criogênico.
NOTA: Armazene duas cúpulas por frasco. - Congele os PDOs durante a noite em um congelador de -80 °C usando um recipiente de congelamento de células apropriado.
- Inicie o processo de criopreservação com a pelota a partir da etapa 2.8. Use 500 μL de meio de congelamento frio para resuspensar a pelota e transferi-la para um frasco criogênico.
- Criopreservação dos PDOs digeridos por célula única
- Após a colheita e digestão dos PDOs, comece a criopreservação a partir do passo 4.8.
- Para armazenar um frasco criogênico, transfira 4-5 x 105 células para um tubo fresco de ligação baixa de 1,5 mL.
NOTA: Armazene três cúpulas/frascos. - Centrifugar a 500 x g para 4 min a 4 °C. Descarte o supernatante cuidadosamente usando uma pipeta de 1.000 μL. Remova o supernatante o máximo possível sem perturbar a pelota.
- Resuspenda a pelota em um volume apropriado de meio de congelamento (500 μL/frasco) e transfira-a para um frasco criogênico.
- Congele os PDOs durante a noite em um congelador de -80 °C usando um recipiente de congelamento celular apropriado e transfira-os para um congelador de -150 °C ou nitrogênio líquido para armazenamento a longo prazo.
Resultados
Este protocolo apresenta os procedimentos que incluem subcultura e criopreservação de PDOs EAC com e sem digestão celular única.
A Figura 1 mostra imagens representativas de contraste de fase das duas estratégias diferentes de subcultura. Os PDOs do EAC atingiram densidade adequada para subcultura (Figura 1, à esquerda). Subcultura sem digestão celular única leva menos tempo para atingir densidade comparável e leva principalmente a estruturas compactas (Figura 1, linha superior). Em contraste, os PDOs digeridos por célula única mostram estruturas ocas com um núcleo oco (Figura 1, linha inferior). A Figura 2 mostra a coloração hematoxilina-eosina (H&E) e a coloração imunohistoquímica (IHC) de PDOs EAC embutidos em parafina com estruturas compactas e ocas. A pan-citokeratina (Pan-CK) permite a identificação de células tumorais epiteliais18. A citokeratina 7 (CK7) destaca as células tumorais diferenciadasglandulares 19. A estrutura compacta (linha superior) existe predominantemente na cultura não digerida, enquanto a estrutura oca (linha inferior) é dominante na cultura que sofreu digestão celular única.
A Figura 3 mostra a coloração da imunofluorescência (IHC) de tecido EAC emparelhado e PDOs com estrutura compacta e estrutura oca. O Ki67 destaca as populações celulares com maior proliferação celular20. Os Ki67 (vermelho) e Pan-CK (verde) foram distribuídos da mesma forma entre tecido primário EAC, estrutura compacta EAC PDO e estrutura oca EAC PDO. A Figura 4 mostra as características morfológicas dos PDOs EAC no primeiro dia de recuperação do estoque congelado com criopreservação baseada em célula única (esquerda) e criopreservação baseada em PDO não digerida (à direita).
A Figura 5 resume um fluxograma do processo de subcultura de PDOs EAC com e sem digestão celular única. Resumidamente, um PDO EAC em crescimento está pronto para ser aprovado. Os PDOs do EAC foram colhidos e pelotas. Para digestão de células únicas, os PDOs foram digeridos enzimáticamente por 5-10 minutos para obter células únicas, que provavelmente crescerão em estruturas ocas que facilitam experimentos que requerem controle de número de células, densidade uniforme e rastreamento de tamanho. Para subcultura não digerida, os PDOs foram divididos para ganhar mais espaço de crescimento sem perturbar enzimaticamente, que provavelmente crescerão em estruturas compactas que facilitam análises histológicas, expansão rápida e recuperação mais rápida da criopreservação.
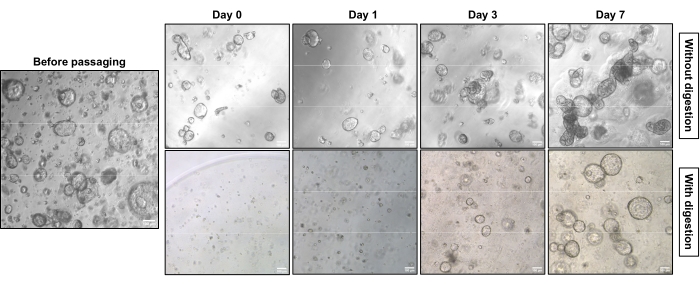
Figura 1: Características morfológicas da subcultura PDOs EAC com e sem digestão celular única sob um microscópio de contraste de fase. Os PDOs EAC crescem a uma certa densidade antes da subcultura (esquerda). Ao subculturar PDOs EAC sem digestão celular única, os PDOs gradualmente crescem de estruturas ocas para estruturas compactas (direita, linha superior), enquanto os PDOs cultivados a partir de células únicas mostram estruturas predominantemente ocas (direita, linha inferior). As fotos foram tiradas com microscópio de luz invertida usando um objetivo de 5x. Barra de escala: 100 μm. Clique aqui para ver uma versão maior desta figura.
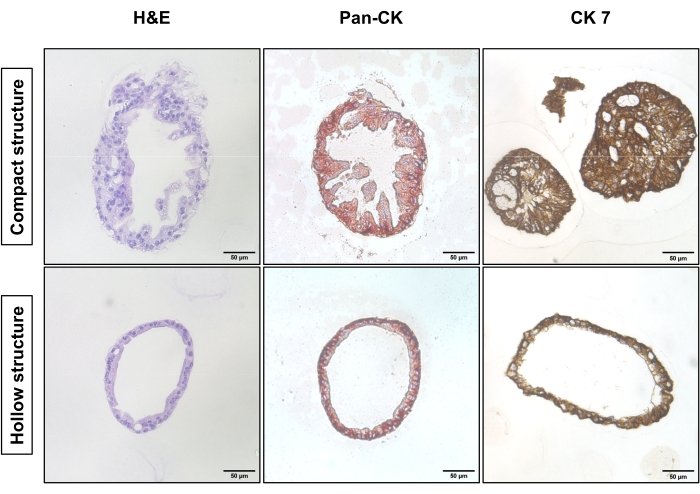
Figura 2: Características histológicas da estrutura compacta e estrutura oca dos PDOs do EAC. A coloração de H&E (esquerda), a mancha Pan-CK (meio) e a coloração CK7 (direita) da estrutura compacta (linha superior) e estrutura oca (linha inferior). As fotos foram tiradas com microscópio de luz invertida usando um objetivo de 20x. Barra de escala: 50 μm. Clique aqui para ver uma versão maior desta figura.

Figura 3: Mancha de imunohistoquímica de tecido EAC emparelhado e PDOs. A coloração de imunofluorescência (IF) de tecido EAC emparelhado (linha superior), estrutura compacta (linha média) e estrutura oca (linha inferior) com Pan-CK (verde), Ki67 (vermelho) e DAPI (azul). As fotos foram tiradas com microscópio de fluorescência automatizada invertido usando um objetivo de 20x. Barra de escala: 50 μm. Clique aqui para ver uma versão maior desta figura.

Figura 4: Características morfológicas dos PDOs EAC no primeiro dia de recuperação do estoque congelado. Imagens de contraste de fase de recultivação a partir de criopreservação baseada em célula única (esquerda) e criopreservação baseada em PDO não digerida (direita) no primeiro dia de recuperação. As fotos foram tiradas com microscópio de luz invertida usando um objetivo de 5x. Barra de escala: 100 μm. Clique aqui para ver uma versão maior desta figura.

Figura 5: O fluxograma do processo de subcultura de PDOs EAC com e sem digestão celular única. Por favor, clique aqui para ver uma versão maior desta figura.
| Estoque | Concentração Final | 50 mL | |||
| Meio basal (ver Tabela 3) | 24 mL | ||||
| Meio condicionado WNT-3A | 12 mL | ||||
| Meio condicionado R-Spondin1 das células Cultrex R-Spondin | 12 mL | ||||
| N-2 | 100x | 1x | 500 μL | ||
| B-27 | 50x | 1x | 1 mL | ||
| N-Acetilcisteína | 0,5 M | 1 mM | 100 μL | ||
| CHIR-99021 | 5 mM | 0,5 μM | 5 μL | ||
| Fator de crescimento epidérmico humano recombinante (EGF) | 100 μg/mL | 250 ng/mL | 125 μL | ||
| A83-01 | 25 mM | 0,5 μM | 1 μL | ||
| SB202190 | 10 mM | 1 μM | 5 μL | ||
| Gastrin | 100 μM | 0,1 μM | 50 μL | ||
| Nicotinamida | 1 M | 20 μM | 1 mL | ||
| Gentamicina | 50 mg/mL | 10 μM | 5 μL | ||
| Penicilina/Estreptomicina | 100x | 1x | 500 μL | ||
| Anfotericina B | 250 μg/mL | 0.60% | 300 μL | ||
| Adicione recentemente em bem: | |||||
| Cabeça | 100 μg/mL | 50 μL | |||
| Y-27632 | 10,5 mM | 50 μL | |||
| Adicione ao estabelecer novos PDOs do tecido primário ou se recuperar de estoques congelados | |||||
| FGF-10a | 100 μg/mL | 100 ng/mL | 50 μL | ||
Tabela 1: Preparação do meio de cultura EAC PDO.
| Inibidor de trippsina de soja (IST) | 12,5 mgs |
| Ajuste para 50 mL com DPBS | |
| Filtrar através de 0,2 μm filtro estéril |
Tabela 2: Preparação da solução inibidora de trippsina de soja (IST).
| Reagente | Volume | Concentração final |
| DMEM avançado/F-12 | 48,2 mL | |
| HEPES (1 M) | 500 μL | 10 mM |
| L-Glutamina (100X) | 500 μL | 1X |
| Penicilina-Estreptomicina (100X) | 500 μL | 1X |
| Anfotericina B | 300 μL | 0.60% |
| Gentamicina (50 mg/mL) | 5 μL | 5 μg/mL |
Tabela 3: Preparação do meio basal.
| Digestão celular única | |
| Profissionais | Contras |
| Controle de números de células | Frágil durante a incorporação |
| Verificação de viabilidade | Tempo mais longo necessário entre passagens |
| Aplicável para, por exemplo, Triagem de Drogas, Citometria de Fluxo | Tempo de recuperação mais longo dos estoques congelados |
| Sem digestão celular única | |
| Profissionais | Contras |
| A morfologia é benéfica para análises histológicas | Expansão de PDOs em tamanho mais do que em número |
| Maior estabilidade no processo de incorporação | Não é aplicável para análises onde a suspensão de célula única é obrigatória |
| Recuperação rápida de estoques congelados | Falta de controle de número de celular e rastreamento de tamanho |
Tabela 4: Prós e contras para subculturar PDOs EAC com e sem digestão celular única.
Discussão
Neste protocolo, são descritos dois métodos diferentes de subcultura e criopreservação de PDOs EAC, ou seja, com e sem digestão celular única. Vários estudos recomendaram a passagem de PDOs EAC com digestãocelular única 15,17, o que é benéfico para a maioria dos experimentos que requerem controle de número de células, densidade uniforme e uma estrutura oca que facilita o rastreamento de tamanho. No entanto, o método baseado em célula única é caracterizado por um crescimento mais lento após a recultivação de estoques congelados e morfologia menos compacta durante o período de cultura. A experiência indica 2-3 semanas para que a recultura baseada em célula única atinja a densidade aplicável para o processo de subcultura. Em contraste, os PDOs EAC congelados sem digestão celular única podem atingir o mesmo tamanho em um período mais curto (cerca de 1 semana) após a recultivação. Uma das razões pode ser o estresse extra da digestão da trippsina por um tempo relativamente longo (10 min). Portanto, recomenda-se preservar PDOs EAC não digdiges em uma proporção de 1:1.5 (congelando duas cúpulas de PDOs EAC não digdigesos e semeando de volta em três cúpulas para a recultivação). Além disso, o uso de PDOs EAC não digeridos é recomendado para rápida expansão e caracterização histológica por IHC ou re coloração IF devido à estrutura compacta. Os prós e contras dos dois métodos de subcultura são resumidos na Tabela 4.
Várias etapas críticas requerem atenção neste protocolo. Em primeiro lugar, as placas para a cultura PDO precisam ser pré-aquecidas durante a noite em uma incubadora de 37 °C para garantir o processo de solidificação de cúpulas de gel ECM recém-semeadas. Recomenda-se o uso de uma placa quente para manter a placa a 37 °C enquanto lida com a duração prolongada da semeadura. Em segundo lugar, são necessários tubos de baixa ligação durante o processo de subcultura para evitar perdas significativas de PDO. Para evitar a perda de gel ECM, as pontas com uma abertura de furo largo podem ser pré-resfriadas no congelador de -20 °C antes de usar. Aqui, a ampla abertura das pontas evita o dano das estruturas de PDO durante a etapa de colheita. Em seguida, recomenda-se incubar PDOs por 20 minutos no gelo antes da primeira etapa de centrifugação, para garantir a liquefação completa do gel ECM. Observe que a centrífuga precisa ser definida a 4 °C durante as etapas de centrifugação para manter o gel ECM residual no estado líquido. Além disso, para o método unicelular, recomenda-se misturar completamente os PDOs após a incubação de trypsin usando uma ponta normal de 1.000 μL para quebrar aglomerados celulares antes de adicionar a IST, em vez de filtrar diretamente a suspensão celular com coador de células, para evitar a perda celular.
Algumas modificações podem ser feitas neste protocolo. A solução de recuperação celular pode ser substituída por DPBS gelado para dissolver o gel ECM na etapa de colheita. No entanto, as experiências mostraram uma melhor capacidade de dissolver o gel ECM usando a solução de recuperação celular. Portanto, o DPBS gelado é bastante recomendado apenas como um método alternativo de backup. Se o laboratório não estiver equipado com uma incubadora rotativa, os PDOs EAC podem ser incubados com trippsina em um banho de água de 37 °C, juntamente com a mistura, invertendo o tubo a cada 2-3 min. 10% DMSO com soro bovino fetal (FBS) pode ser usado como alternativa para congelar meio para preparar estoques de PDO congelados. No entanto, é preferível um meio de congelamento comercial com soro inferior ou não devido a uma melhor recuperação do PDO.
Algumas limitações precisam ser abordadas neste protocolo. Uma vez que esses métodos foram testados apenas em PDOs EAC, a aplicação deste protocolo a outros tipos de PDOs não é clara. Embora os procedimentos para a passagem de PDOs com e sem digestão celular único sejam padronizados para a maioria dos tipos organoides21,22, ainda há a necessidade de tentar protocolos atuais em outros tipos de câncer para garantir a reprodutibilidade. Além disso, uma incubação de trippsina de 10 min 0,25% pode estressar as células durante a digestão; portanto, o tempo de incubação pode variar de acordo com a condição de PDO pré-subcultura e a diversidade de PDO individual. Durante as tentativas iniciais, sugere-se definir diferentes tempos de incubação de trippsina para cada PDO EAC.
Em conclusão, este é o primeiro protocolo que descreve e discute subcultura e criopreservação de PDOs EAC com e sem digestão celular única. Subculturar PDOs EAC com digestão celular única é aplicável para experimentos de comparação entre grupos, enquanto PDOs EAC não digeridos são benéficos para caracterização histológica, criopreservação e expansão rápida. Aqui, a manutenção rotineira dos PDOs EAC é padronizada, fornecendo um guia para que os pesquisadores escolham métodos adequados para a geração organoide do EAC.
Divulgações
Os autores não declaram conflitos de interesse neste trabalho.
Agradecimentos
Este trabalho foi apoiado pelo Köln Fortune Program/Faculty of Medicine da Universidade de Colônia. Agradecemos a assistência técnica de Susanne Neiss, Michaela Heitmann e Anke Wienand-Dorweiler. Ningbo Fan foi apoiado financeiramente pelo Guangzhou Elite Scholarship Council (GESC). Os autores agradecem ao Dr. Joshua D'Rozario por sua ajuda na edição linguística.
Materiais
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| -20°C Freezer | Bosch | Economic | |
| -80°C Freezer | Panasonic | MDF DU500VH-PE | |
| Automated Cell counter | Thermo Fisher | AMQAX1000 | Countess II |
| Biological Safety Cabinet Class II | Thermo Scientific | 51022482 | Herasafe KS12 |
| Centrifuge | Heraeus | 75003060 | Megafuge 1.0R |
| CO2 Incubator | Thermo Scientific | 50116048 | Heracell 150i |
| Inverted automated fluorescence microscope | Olympus | IX83 | |
| Inverted light microscope | Leica | DMIL LED Fluo | |
| Pipette 1000 µL | Eppendorf | 3123000063 | Research Plus |
| Pipette 200 µL | Eppendorf | 3123000039 | Research Plus |
| Rotating Incubator | Scientific Industries, sc. | SI-1200 | Enviro-genie |
| Shaker | Eppendorf | 5355 000.011 | Thermomixer Comfort |
| Vacuum pump | Vacuubrand | 20727200 | BVC control |
| Waterbath | Medingen | p2725 | W22 |
| Material | |||
| 15 mL tube | Sarstedt | 62.554.502 | Inc Screw cap tube PP 15 mL |
| Cryo vial 2 mL | Sarstedt | 72.379 | CryoPure 2.0 mL tube |
| Low bind tube 1.5 mL | Sarstedt | 72.706.600 | Micro tube 1.5 mL protein LB |
| Low bind tube 5 mL | Eppendorf | 0030 108.302 | Protein LoBind Tube 5.0 mL |
| Pipette tip 200 µL | Starlab | E1011-8000 | 200 µL Graduated tip, wide orifice |
| Pipette tip 1000 µL | Starlab | E1011-9000 | 1000 µL Graduated tip, wide orifice |
| Pipette tip 1000 µL | Sarstedt | 70.3050 | Pipette tip 1000 µL |
| Sterile filter 0.2 µm | Sarstedt | 83.1826.001 | Filtropur 0.2 µm sterile filter |
| Tissue culture plate | Sarstedt | 83.3921 | 12 well-plate |
| Reagent/Chemical | |||
| A83-01 | Tocris | 2939 | |
| Advanced DMEM/F-12 | Thermo Fisher Scientific | 12634010 | |
| Amphotericin B | Thermo Fisher Scientific | 15290026 | |
| B-27 | Thermo Fisher Scientific | 17504001 | |
| Cell Recovery Solution | Corning | 354253 | |
| CHIR-99021 | MedChemExpress | HY-10182/CS-0181 | |
| DNase I grade II, from bovine pancreas | Sigma-Aldrich | 10104159001 | |
| Dulbecco's phosphate-buffered saline (DPBS) | Thermo Fisher Scientific | 14190094 | |
| Extracellular matrix (ECM) gel: Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix | Corning | 356231 | |
| FGF-10a | Peprotech | 100-26-100 | |
| Freezing medium: Recovery Cell Freezing Medium | Thermo Fisher Scientific | 12648010 | |
| Gastrin | Sigma | G9020 | |
| Gentamicin-25 (25 mg/ 500 µL) | PromoCell | C-36030 | |
| HEPES (1 M) | Thermo Fisher Scientific | 15630080 | |
| L-Glutamine 200 mM (100X) | Thermo Fisher Scientific | 25030024 | |
| N-2 | Thermo Fisher Scientific | 17502-048 | |
| N-Acetylcysteine | Sigma | A9165 | |
| Nicotinamide | Sigma | N0636-100 | |
| Noggin | Peprotech | 120-10C-50 | |
| Penicillin-Streptomycin 10,000 U/ mL (100X) | Thermo Fisher Scientific | 15140122 | |
| Recombinant human epidermal growth factor (EGF) | Peprotech | AF-100-15 | |
| R-Spondin1 conditioned medium from Cultrex R-Spondin Cells | Biotechne | 3710-001-01 | |
| SB202190 | MedChemExpress | 152121-30-7 | |
| Trypsin inhibitor from Glycine max (soybean) | Sigma-Aldrich | 93620-1G | |
| Trypsin-EDTA (0.25 %), phenol red | Thermo Fisher Scientific | 25200056 | |
| Wnt-3A conditioned medium | Wnt-3A expressing cell line was kindly provided by Prof. Hans Clevers' group | ||
| Y-27632 | Sigma | Y0503 |
Referências
- Sung, H., et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: A Cancer Journal for Clinicians. 71 (3), 209-249 (2021).
- Coleman, H. G., Xie, S. -. H., Lagergren, J. The epidemiology of esophageal adenocarcinoma. Gastroenterology. 154 (2), 390-405 (2018).
- Rumgay, H., et al. International trends in esophageal squamous cell carcinoma and adenocarcinoma incidence. The American Journal of Gastroenterology. 116 (5), 1072-1076 (2021).
- Qian, H., et al. Clinical characteristics, prognosis, and nomogram for esophageal cancer based on adenosquamous carcinoma: a seer database analysis. Frontiers in Oncology. 11, 603349 (2021).
- Lagergren, J., Smyth, E., Cunningham, D., Lagergren, P. Oesophageal cancer. Lancet. 390 (10110), 2383-2396 (2017).
- Rockett, J. C., Larkin, K., Darnton, S. J., Morris, A. G., Matthews, H. R. Five newly established oesophageal carcinoma cell lines: phenotypic and immunological characterization. British Journal of Cancer. 75 (2), 258-263 (1997).
- Hashimoto, N. Expression of COX2 and p53 in rat esophageal cancer induced by reflux of duodenal contents. ISRN Gastroenterology. 2012, 1-5 (2012).
- Quante, M., et al. Bile acid and inflammation activate gastric cardia stem cells in a mouse model of barrett-like metaplasia. Cancer Cell. 21 (1), 36-51 (2012).
- Kapoor, H., Lohani, K. R., Lee, T. H., Agrawal, D. K., Mittal, S. K. Animal models of Barrett's esophagus and esophageal adenocarcinoma-past, present, and future. Clinical and Translational Science. 8 (6), 841-847 (2015).
- Lan, T., Xue, X., Dunmall, L. C., Miao, J., Wang, Y. Patient-derived xenograft: a developing tool for screening biomarkers and potential therapeutic targets for human esophageal cancers. Aging. 13 (8), 12273-12293 (2021).
- Liu, D. S. H., et al. APR-246 potently inhibits tumour growth and overcomes chemoresistance in preclinical models of oesophageal adenocarcinoma. Gut. 64 (10), 1506-1516 (2015).
- Ebbing, E. A., et al. Esophageal adenocarcinoma cells and xenograft tumors exposed to Erb-b2 receptor tyrosine kinase 2 and 3 inhibitors activate transforming growth factor beta signaling, which induces epithelial to mesenchymal transition. Gastroenterology. 153 (1), 63-76 (2017).
- Simian, M., Bissell, M. J. Organoids: A historical perspective of thinking in three dimensions. The Journal of Cell Biology. 216 (1), 31-40 (2017).
- Drost, J., Clevers, H. Organoids in cancer research. Nature Reviews Cancer. 18 (7), 407-418 (2018).
- Li, X., et al. Organoid cultures recapitulate esophageal adenocarcinoma heterogeneity providing a model for clonality studies and precision therapeutics. Nature Communications. 9, 2983 (2018).
- Ebbing, E. A., et al. Stromal-derived interleukin 6 drives epithelial-to-mesenchymal transition and therapy resistance in esophageal adenocarcinoma. Proceedings of the National Academy of Sciences of the United States of America. 116 (6), 2237-2242 (2019).
- Karakasheva, T. A., et al. Generation and characterization of patient-derived head and neck, oral, and esophageal cancer organoids. Current Protocols in Stem Cell Biology. 53 (1), 109 (2020).
- Ordóñez, N. G. Broad-spectrum immunohistochemical epithelial markers: a review. Human Pathology. 44 (7), 1195-1215 (2013).
- Maniar, K. P., Umpires, B. Cytokeratin 7 (CK7, K7). Pathology Outlines.com website. , (2021).
- Sun, X., Kaufman, P. D. Ki-67: more than a proliferation marker. Chromosoma. 127 (2), 175-186 (2018).
- Driehuis, E., Kretzschmar, K., Clevers, H. Establishment of patient-derived cancer organoids for drug-screening applications. Nature Protocols. 15 (10), 3380-3409 (2020).
- Sachs, N., et al. Long-term expanding human airway organoids for disease modeling. The EMBO Journal. 38 (4), 100300 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados