Method Article
Imagem confocal de alta taxa de imagem confocal de sars-cov-2 trimers de pico conjugados de alta throughput para rastrear a vinculação e a endocitose em células HEK293T
Neste Artigo
Resumo
Neste protocolo, os pontos quânticos conjugados ao pico SARS-CoV-2 recombinante permitem que ensaios baseados em células monitorem a ligação de espigão para hACE2 na membrana plasmática e a endocitose subsequente das proteínas ligadas ao citoplasma.
Resumo
O desenvolvimento de novas tecnologias para microscopia de fluorescência celular facilitou métodos de triagem de alto rendimento para a descoberta de drogas. Pontos quânticos são nanopartículas fluorescentes com excelentes propriedades fotofísicas imbuídas de fotoluminescência brilhante e estável, bem como faixas de emissão estreitas. Os pontos quânticos são esféricos em forma, e com a modificação adequada da química da superfície, podem ser usados para conjugar biomoléculas para aplicações celulares. Essas propriedades ópticas, combinadas com a capacidade de funcionalizá-las com biomoléculas, as tornam uma excelente ferramenta para investigar interações receptor-ligantes e tráfico celular. Aqui, apresentamos um método que usa pontos quânticos para rastrear a ligação e a endocitose da proteína de pico SARS-CoV-2. Este protocolo pode ser usado como um guia para experimentalistas que procuram utilizar pontos quânticos para estudar interações proteína-proteína e tráfico no contexto da fisiologia celular.
Introdução
A microscopia de fluorescência permite que os pesquisadores espiem o funcionamento interno da célula usando corantes especializados1, proteínas fluorescentes geneticamente codificadas2 e nanopartículas fluorescentes na forma de pontos quânticos (QDs)3. Para a síndrome respiratória aguda grave coronavírus de 2019 (SARS-CoV-2) pandemia global, pesquisadores têm usado microscopia de fluorescência para entender como o vírus interage com a célula tanto na membrana plasmática quanto no citoplasma. Por exemplo, os pesquisadores têm sido capazes de obter insights sobre a ligação da proteína SARS-CoV-2 Spike na superfície do virion à enzima conversor de angiotensina humana 2 (hACE2) na superfície das células humanas, internalização subsequente via fusão na membrana plasmática e endocitose do complexo de proteína Spike:hACE24,5. Grandes insights também foram obtidos na saída do SARS-CoV-2 das células através do lisosome usando imagens de fluorescência celular, uma característica única de coronavírus previamente pensados para ocorrer através da vesícula tradicional brotando do Golgi, como é com muitos outros vírus6. Um dos pilares de quase todos os aspectos da pesquisa biológica, a técnica de microscopia de fluorescência celular tem necessariamente avançado em sua amplitude e escopo de aplicações desde imagens de super-resolução de animais inteiros até imagens multi-paramétricas automatizadas de alto conteúdo para triagem de medicamentos. Aqui, a microscopia confocal automatizada de alto conteúdo é aplicada ao estudo da entrada de células SARS-CoV-2 usando QDs fluorescentes conjugados à proteína do pico viral.
A análise de alto teor de imagens geradas por plataformas de imagem biológica permite maior extração de insights biológicos valiosos do que parâmetros únicos, como a intensidade de todo-poço, que se obteria usando um leitor de placas multimodal7. Ao separar os objetos em um campo de visão usando algoritmos automatizados de segmentação, cada objeto ou uma população de objetos pode ser analisado para parâmetros como intensidade, área e textura em cada canal de fluorescência disponível8. Combinar muitas medidas em conjuntos de dados multivariados é uma abordagem útil para perfis fenotípicos. Quando o fenótipo desejado é conhecido, como a internalização de QD na forma de puncta, pode-se usar as medidas relacionadas a puncta, como tamanho, número e intensidade para avaliar a eficácia de um tratamento.
O software de análise de imagens de alto conteúdo baseado em nuvem pode acomodar uma grande variedade de saídas de dados de instrumentos, incluindo a plataforma de imagem de alto conteúdo. Usando um servidor baseado em nuvem para armazenamento de imagens e análise on-line, o usuário é capaz de carregar seus dados do instrumento de imagem ou da unidade de rede onde os dados são armazenados. A parte de análise do protocolo é conduzida dentro do ambiente de software em nuvem, e os dados podem ser exportados em uma variedade de formatos de arquivo para visualização de dados a jusante.
O vírus SARS-CoV-2 é composto por proteínas não estruturais e estruturais que auxiliam em sua montagem e replicação. O pico SARS-CoV-2 possui dois domínios chamados S1 e S2, com S1 contendo o domínio de ligação receptor responsável pelas interações hACE2 na membrana plasmática9. O pico também foi encontrado para interagir com outras moléculas na membrana plasmática que podem atuar como co-receptores, além de hACE210,11. Ao longo da sequência de proteína de pico e particularmente na interface S1/S2, existem locais de decote protease que permitem fusão na membrana após a protease de serina transmembrana 2 (TMPRSS2)12. Várias proteínas sars-cov-2 spike recombinantes foram produzidas a partir de domínios individuais de ligação de receptores, para S1, S2, S1 com S2, e aparadores de pico inteiros de vários fornecedores comerciais para uso em atividades de pesquisa13.
Neste trabalho, a superfície dos QDs foi funcionalizada com aparadores de espigão recombinantes que contêm uma tag histidina (QD-Spike). Os QDs produzidos pela Seção de Nanomateriais Ópticos do Laboratório de Pesquisa Naval contêm um núcleo de selenida de cádmio e um shell de sulfeto de zinco14,15. O zinco na superfície QD coordena os resíduos de histidina dentro da proteína recombinante para formar um QD funcionalizado que se assemelha a uma partícula viral SARS-CoV-2 em forma e função. A geração das nanopartículas e conjugação de proteínas foi descrita anteriormente usando o domínio de ligação do receptor conjugado QD15. Este método descreve as preparações de cultura celular, tratamento de QD, aquisição de imagens e protocolo de análise de dados que podem orientar um pesquisador no estudo da atividade sars-cov-2 no contexto fisiológico de uma célula humana.
Protocolo
A linha celular HEK293T usada neste estudo é uma linha celular imortalizada. Não foram utilizados indivíduos humanos ou animais neste estudo.
1. Cultivo de células e semeadura
- Dentro de um armário de biossegurança estéril, usando equipamentos de proteção individual (incluindo luvas de laboratório, jaleco e óculos de segurança), prepare o meio de cultura celular suplementando o Meio Águia Modificada (DMEM) de Dulbecco com 10% de soro bovino fetal (FBS), penicilina/estreptomicina (P/S) e 250 μg/mL G418.
- Para 500 mL de mídia, adicione 443,75 mL de DMEM, 50 mL de FBS, 5 mL de P/S e 1,25 mL de G418.
- Filtre através de um frasco de filtro de 0,2 μm e mantenha a esterilidade para evitar contaminação bacteriana.
- Dentro de um armário de biossegurança estéril, semente os 60 poços interiores de uma placa preta, de fundo claro, poli-D-lysina revestida de 96 poços com 20.000 células em 100 μL por poço de cultura celular média. Encha os 36 poços externos com 100 μL por poço de soro fisiológico tamponado de fosfato (PBS).
- Para contar as células, adicione 2 μL de laranja acridina e mancha de iodeto de propídio a 18 μL de suspensão celular.
- Carregue 10 μL desta solução em um lado de um slide de contagem de células.
- Repita as etapas 1.2.1. e 1.2.2. para carregar ambos os lados do slide.
- Coloque o slide em um contador automático de células.
- Selecione o protocolo de contagem de fluorescência e pressione Count. Conte ambos os lados do slide e calcule a média das duas densidades de células vivas.
- Para calcular o volume de suspensão celular necessário, divida o número total de células necessárias pela densidade celular média.
- Inspecione os poços após a semeadura sob um microscópio leve para garantir que a densidade e distribuição adequadas tenham sido alcançadas.
- Incubar a placa durante a noite em uma incubadora umidificada a 37 °C com 5% de CO2.
2. Tratamento de células com QD-Spike
- Prepare 0,1% de albumina de soro bovino (BSA) diluindo em mídia de imagem.
- Para 10 mL de mídia de imagem, adicione 130 μL de 7,5% de BSA e misture.
- Em um reservatório de 12 poços ou placa de ensaio, dilui o estoque de 440 nM QD-Spike (SARS-CoV-2, Isolate USA-WA1/2020) para 20 nM usando 0,1% BSA em mídia de imagem.
- Para 1 poço de QD-Spike, adicione 2,27 μL de 440 nM QD-Spike a 47,73 μL de 0,1% de mídia de imagem BSA para uma concentração final de 20 nM.
- Para gerar uma diluição seriada de seis pontos 1:3 (em triplicado), adicione 14,26 μL de QD-Spike a 285,74 μL de 0,1% de mídia de imagem BSA para fazer a maior concentração de 20 nM. Adicione 100 μL de 20 nM QD-Spike a 200 μL de mídia BSA de 0,1% para fazer a segunda diluição de 6,67 nM QD-Spike. Repita quatro vezes para gerar os seis pontos de concentração.
- Usando um aspirador multicanal, remova todas as mídias gastas de cada poço. Usando uma pipeta multicanal, lave uma vez com mídia de imagem (100 μL/well).
- Aspire 100 μL de mídia de imagem e adicione de volta 50 μL/bem da solução QD-Spike.
- Incubar a placa por 3h em uma incubadora umidificada a 37 °C com 5% de CO2. Continue a passo 4.
3. Fixação e coloração de núcleos
- Dentro de um armário de biossegurança estéril, usando equipamentos de proteção individual (incluindo luvas de laboratório, jaleco e óculos de segurança), prepare 4% de paraformaldeído (PFA) em 0,1% de mídia de imagem BSA.
- Aspire 50 μL de QD-Spike de cada poço e adicione 100 μL/well de 4% PFA.
- Não deixe os poços secarem. Use uma pipeta multicanal automatizada para evitar a secagem dos poços (recomendado).
- Incubar por 15 minutos em temperatura ambiente.
- Lave três vezes com soro fisiológico tamponado de fosfato 1x (PBS).
- Prepare o corante nuclear vermelho profundo diluindo a solução de estoque de 5 mM para diluição de 1:1000 em PBS.
- Aspire o PBS e adicione de volta 50 μL/poço de corante nuclear diluído.
- Incubar por 30 minutos à temperatura ambiente.
- Lave três vezes com PBS.
- Imagem da placa ou vedação usando um selador de placa para imagem posteriormente. Armazene a placa a 4 °C. A fluorescência deve ser estável por várias semanas ou mais.
4. Configuração de aquisição e imagem
- Inicie o software para a plataforma de imagem. Ligue a plataforma de imagem de alto conteúdo e a luz na barra de status deve estar acesa. Se a máquina não estiver conectada, o software entrará no modo de análise offline.
- Faça login na plataforma de imagem.
- Crie um novo protocolo de aquisição.
- Selecione o tipo de placa como placa de imagem inferior 96 bem clara.
NOTA: A seleção da placa no instrumento pode variar e pode ser personalizada carregando as definições corretas da placa que incluem dimensões da placa, como altura, largura e outras distâncias para definir bem o espaçamento e os pontos focais. - Selecione o Modo Óptico como confocal e ampliação como imersão de água de 40x.
- Selecione Binning como 1.
- Escolha os canais e os comprimentos de onda de excitação/emissão fluorescentes, conforme descrito nas etapas 4.3.5-4.3.8. Tire um instantâneo quando as configurações forem selecionadas para visualizar a imagem e garantir que as configurações sejam apropriadas.
NOTA: O DPC (Digital Phase Contrast) permitirá a visualização de células vivas sem uma mancha de célula ou corante fluorescente. Não é recomendado para células fixas. - Selecione o modo para DPC, como Alto Contraste, para produzir corpos celulares bem definidos. Tire uma foto para confirmar que essa configuração é apropriada, como visto na janela da imagem.
- Selecione o Canal FITC para uso com a linha celular ACE2-GFP que permite a visualização do tráfico DE ACE2 dentro da célula.
- Para fazer um canal personalizado para o canal QD, selecione o menu suspenso do triângulo para o canal e escolha a excitação na faixa de 405 nm e emissão na faixa de 608 nm.
- Se as células foram fixadas e um corante nuclear vermelho profundo foi usado, selecione o canal vermelho distante com emissão superior a 630 nm para delinear ainda mais o corpo celular e agir como uma máscara celular.
- Selecione um poço com um forte sinal QD citoplasmado para definir o tempo de exposição, a potência do laser e a posição de altura Z. Siga os passos de 4.3.10-4.3.13.
- Verifique se a altura padrão produz uma imagem onde as células estão no plano focal desejado. Se a puncta endócitosa for a organela alvo, a posição Z será menor do que se a membrana plasmática for a organela alvo.
- Escolha um tempo de exposição que produza uma imagem brilhante com níveis cinzentos pelo menos três vezes maior que o sinal de fundo. Isso é tipicamente entre 100 e 300 ms. A 20 nM, os níveis cinzentos devem ser de aproximadamente 6000 a.u. usando um tempo de exposição de 200 ms e potência laser de 80%.
- Verifique os níveis cinzentos clicando com o botão direito do mouse na imagem produzida com o recurso Snapshot em cada canal e selecionando Mostrar intensidade. Em seguida, clique à esquerda no objeto de interesse ou fundo para ver o nível cinza para esse pixel.
- Ajuste o poder do laser para ajustar a intensidade dos objetos de interesse.
- Selecione o tipo de placa como placa de imagem inferior 96 bem clara.
- Guarde o protocolo de aquisição.
- Mude para Executar experimento e digite o Nome da placa.
- Executar o experimento.
5. Análise de dados
- Importar dados para o software de imagem.
- Após a aquisição de todas as imagens, exporte medições para o software de imagem. Isso requer que um servidor seja estabelecido e uma conta seja criada. Crie uma tela para que os dados sejam exportados para o software de imagem.
- No software de imagem, selecione Análise de Imagem para começar a construir o protocolo de análise.
- Carregue as medidas do experimento de imagem clicando com o botão direito do mouse no arquivo na lista de telas e clicando em Selecionar. O mapa da placa dos poços de imagem e seus campos associados devem ser visíveis no canto inferior esquerdo da janela.
- Escolha um bem com pontos de QD citoplasmica brilhantes para iniciar a segmentação de imagens.
- No bloco de construção de imagem de entrada, selecione Correção de campo plano.
- Adicione o próximo bloco de construção ao protocolo selecionando "Encontrar núcleos".
- Aqui, use o DPC ou o canal vermelho distante como marcador de Núcleos.
- Selecione o método que segmenta os objetos com precisão primeiro e, em seguida, ajuste a segmentação usando o menu suspenso e ajuste os controles deslizantes.
NOTA: Uma boa segmentação define com precisão a região de interesse (ROI) para o núcleo, celular e manchas. Apenas os pixels positivos para o marcador devem ser capturados dentro de um ROI individual. O sinal de fundo deve ser excluído. Se o fundo for capturado no ROI, a sensibilidade do método pode ser diminuída, ou o limiar de intensidade pode ser aumentado para aumentar a stringency do algoritmo de segmentação. Isso é aplicável a todos os blocos de construção Find.
- Em seguida, adicione o Bloco de Construção De Citoplasma para identificar o citoplasma.
- Neste caso, use o canal ACE2-GFP para segmentação citoplasmática.
- Selecione o método que melhor identifica o citoplasma de cada célula.
- Em seguida, adicione o Find Spots Building Block para identificar o puncta QD-Spike.
- Selecione os Núcleos como a população do ROI.
- Selecione a célula como a região do ROI. Isso captura a puncta dentro de toda a célula.
- Selecione o método que melhor identifica o puncta QD em cada célula.
- Uma vez que todos os objetos da imagem tenham sido segmentados e identificados com precisão neste poço, escolha outros poços para garantir que os blocos e configurações de construção possam ser geralmente aplicados aos outros poços e outras condições.
- Adicione as Propriedades de Intensidade de Cálculo para todos os objetos segmentados (núcleos, citoplasma/células e manchas).
- Adicione as Propriedades de Morfologia de Cálculo para todos os objetos segmentados (núcleos, citoplasma/células e manchas).
- Adicione as Propriedades de Textura de Cálculo para todos os objetos segmentados (núcleos, citoplasma/células e manchas).
- Defina os resultados selecionando os parâmetros para cada população, incluindo núcleos e manchas. O número de objetos pode ser usado como uma medida indireta da viabilidade celular.
6. Dados de exportação
- Clique na análise batch. Aguarde que a análise termine antes de prosseguir para a próxima etapa (etapa 6.2).
NOTA: A análise em lote permite que o servidor de software de análise de imagens carregue o protocolo de análise usando as medidas selecionadas para analisar os dados remotamente. - Exporte o conjunto de dados para um computador local ou unidade de rede.
- Conecte o software de análise de imagens a um aplicativo de ajuda no computador baixando e abrindo um arquivo de conexão.
- Selecione o tipo de arquivo de resultados (.txt, .csv, .html ou XML nativo).
- Escolha as configurações da pasta de arquivos com o menu suspenso.
7. Analisar os dados em uma planilha
- Abra o arquivo exportado. Se for um arquivo .csv, salve-o como um formato de arquivo .xls para permitir o uso de tabelas dinâmicas. Se o caminho do arquivo for muito longo, salve o arquivo .xls mais acima na hierarquia da pasta para evitar erros na salvação.
- Adicione colunas à planilha que designam as condições para cada poço. Por exemplo, o tipo de célula, variantes QD-Spike, concentrações de QD-Spike, tempo de incubação, etc.
- Escolha a função Tabela Dinâmica e construa a tabela usando as condições adicionadas como Linhas e os parâmetros medidos como Colunas. Selecione o cálculo para cada parâmetro, ou seja, Média, Desvio Padrão, Mediana, Min, Max ou Contagem.
- Normalize os dados para controlar amostras, incluindo QDs não julgados como 0% e a maior concentração de QD-Spike em 100%.
NOTA: Se avaliar a eficácia de inibidores como anticorpos neutralizantes, normalize os dados para células tratadas somente com mídia (100% de eficácia) e QD-Spike sem inibidor (0% de eficácia). - Plote os dados no software de grafação.
Resultados
Após o tratamento, os QDs serão internalizados, pois a nanopartícula se ligará ao ACE2 na membrana plasmática e induzirá a endocitose. Usando uma linha celular de expressão ACE2-GFP, a translocação de QDs e ACE2 pode ser visualizada usando microscopia de fluorescência. Uma vez internalizados, os dois sinais QD e ACE2 mostram forte colocalização. A partir dessas imagens, a segmentação de imagens e a análise subsequente podem ser realizadas para extrair parâmetros relevantes, como contagem de pontos (Figura 1, Figura 2B). Figura 1A é uma montagem de células tratadas com diferentes concentrações de QD-Spike ou mídia apenas como um controle. Foram utilizados três canais, incluindo contraste de fase digital, FITC e o canal QD personalizado com excitação a 405 nm e emissão a 608 nm. O DPC fornece uma imagem da forma geral da célula. A imagem DPC fornece uma indicação aproximada de toda a morfologia celular. A área de interesse se sobrepõe ao sinal DPC e é suficiente para detectar QDs internalizados. O canal FITC mostra o ACE2-GFP mudando a localização e acumulando-se em regiões que se colocam com QD-Spike. À medida que a concentração de QD-Spike diminui, os QDs não são mais visíveis e o sinal ACE2-GFP é semelhante ao controle. O canal de fusão demonstra a colocalização do ACE2-GFP com QD-Spike. Esses objetos são uma mistura de manchas e acumulações maiores que podem ser segmentadas com o software de análise. O ajuste fino do método de análise de software utilizado para segmentação de imagens pode ser usado para segmentar os objetos de interesse.
Aqui foi realizado um experimento de concentração-resposta com seis concentrações diferentes de QD-Spike, a partir de 20 nM, para determinar as concentrações ideais de QD (Figura 2A). QDs podem ser usados a partir de 2,22 nM e ainda mostram vinculação e internalização. No entanto, recomenda-se usar concentrações de 20 nM ou superior para garantir uma resposta robusta.
Durante a conjugação ao QD, pode ocorrer agregação de proteínas. A principal questão com os agregados é que eles se acumularão em cima das células e causarão artefatos na etapa de segmentação de imagens. Os agregados não serão capazes de entrar nas células, e as nanopartículas como um todo não se assemelharão mais a uma partícula viral (Figura 3). Agregados podem ser vistos na solução QD como precipitados brilhantes e desajeitados.
Este ensaio também pode ser usado para avaliar os biológicos, como neutralizar anticorpos que bloqueiam a entrada viral. Os QDs foram incubados com anticorpos neutralizantes (Figura 4A), a partir de 30 μg/mL, por 30 minutos à temperatura ambiente antes da adição às células. Anticorpos levantados contra a cepa de referência de Washington, SARS-CoV-2 RBD, foram usados. Bloquearam a vinculação, a internalização e causaram uma redução na contagem de manchas medidas em comparação com as células tratadas apenas com QD (Figura 4B).
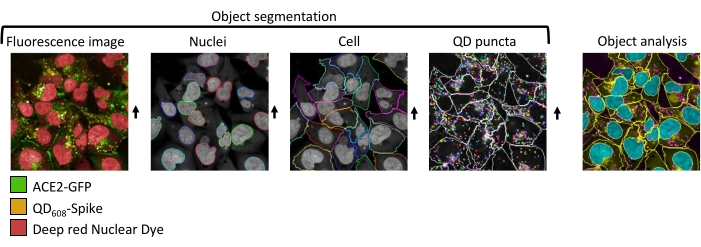
Figura 1: Análise de imagem de alto conteúdo e segmentação de células ACE2-GFP tratadas com QD608-Spike. Imagens representativas de segmentação precisa. Primeiro, os núcleos são segmentados a partir do canal contendo o marcador nuclear para criar uma população de ROI. Da população do ROI, uma região do ROI delineando a célula é segmentada usando o canal ACE2-GFP. Por fim, os pontos QD608-Spike são segmentados a partir da região do ROI celular. Clique aqui para ver uma versão maior desta figura.

Figura 2: QD608-Spike liga hACE2 e é endocittosed em células hACE2-GFP HEK293T. (A) montagem de imagem de células hACE2-GFP (amarela) HEK293T tratadas com várias concentrações de QD608-Spike (magenta) ou somente mídia. O contraste de fase digital (ciano) foi usado para visualizar o corpo celular. Barra de escala: 20 μm. (B) Análise de alto teor de QD608-Spike Spot Counts normalizada para 20 nM QD608-Spike (100%) e o controle (0%). N = poços triplicados. As barras de erro indicam desvio padrão (S.D.). Clique aqui para ver uma versão maior desta figura.
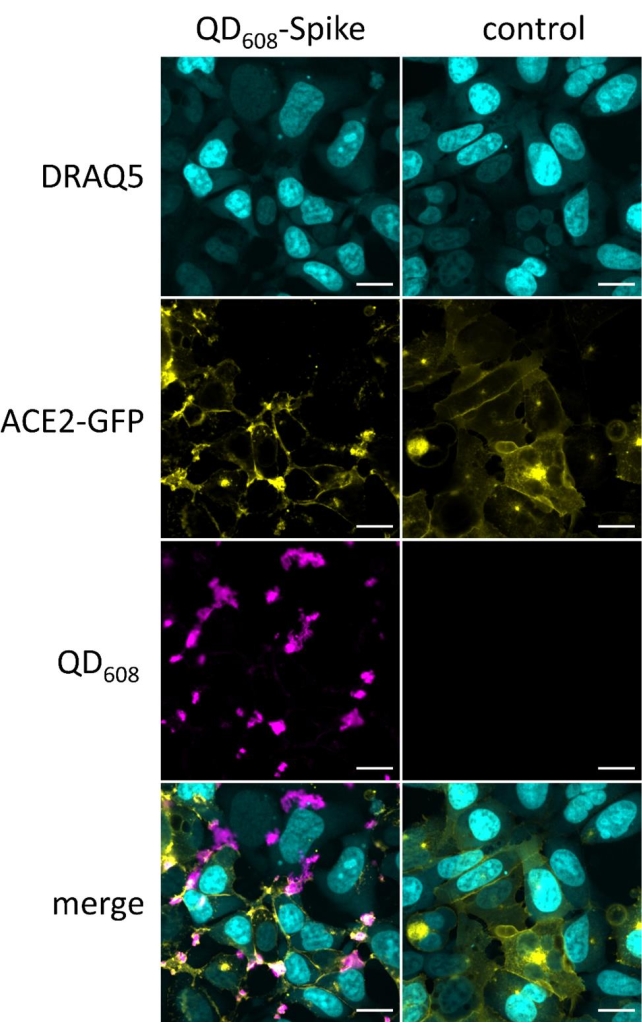
Figura 3: O QD608-Spike agregado não é internalizado nas células hACE2-GFP HEK293T. Montagem de imagem de células hACE2-GFP HEK293T tratadas com 10 nM QD608-Spike ou somente mídia. Barra de escala: 20 μm. Clique aqui para ver uma versão maior desta figura.

Figura 4: Neutralizar blocos de anticorpos endocitose de QD608-Spike em células hACE2-GFP HEK293T. (A) Montagem de imagem de células hACE2-GFP (amarela) HEK293T tratadas com QD608-Spike (magenta) pré-insinuladas com concentrações de neutralização de anticorpos, usando contraste de fase digital (ciano) para identificar corpos celulares. Barra de escala: 20 μm. (B) Análise de alto teor de QD608-Spike Spot Counts normalizada para controle somente de mídia (100%) e 10 nM QD608-Spike apenas (0%). N = poços triplicados. Clique aqui para ver uma versão maior desta figura.
Discussão
O método descrito neste artigo fornece as etapas necessárias para a imagem de QDs funcionalizados em células humanas usando microscopia confocal de alta produtividade. Este método é mais adequado para células onde a endocitose é a principal rota de entrada viral em vez da atividade de TMPRSS2 e fusão de membrana, pois permite o estudo do SarS-CoV-2 Spike e hACE2 endocitose. Devido à natureza do modelo QD e da tag C-terminal No aparador spike comercialmente disponível, qualquer decote TMPRSS2 dos domínios Spike S1 e S2 deixaria o QD ligado apenas ao domínio S212. Isso pode evitar a internalização, uma vez que o RBD é encontrado em S1. Portanto, se a sequência de eventos na superfície celular fosse precisa, onde hACE2 está vinculado e, em seguida, TMPRSS2 cleaves Spike, um sinal negativo sem internalização é esperado.
Como este protocolo trata de processos celulares de imagem, as células cultivadas devem ser permissivas à infecção viral com a expressão de hACE2. hACE2 pode ser transitoriamente transfectado em células, ou uma linha celular estável expressando hACE2 pode ser gerada17. Recomenda-se verificar a expressão e localização hACE2 usando imunofluorescência com anticorpos contra hACE2, a menos que o hACE2 tenha uma etiqueta de proteína fluorescente como o GFP que pode ser observada usando microscopia. O hACE2 marcado pela GFP confere o benefício adicional de visualizar o tráfico de hACE2 após a endocitose QD-Spike. Algumas linhas celulares com expressão endógena hACE2 podem ser usadas, mas isso deve ser confirmado usando imunocytoquímica. Em alguns casos, a expressão endógena não fornece parceiros de ligação hACE2 suficientes para QD-Spike e pode resultar em um sinal que é mal detectado por microscopia confocícal de alto throughput.
Um passo crítico no protocolo inclui a aquisição de QDs de alta qualidade conjugados ao pico SARS-CoV-2 recombinante. O pré-requisito para isso é uma proteína devidamente purificada que pode ser conjugada aos QDs sem causar agregação. QDs agregados terão vários problemas que impedem um experimento bem sucedido. Portanto, o teste de QD-Spike usando ferramentas de análise (por exemplo, espectroscopia visível uv, microscopia eletrônica de transmissão ou dispersão dinâmica de luz) antes da experimentação completa é altamente recomendado para preservar recursos valiosos se testar reagentes preciosos16. Durante a rotulagem de QDs com pico, concentrações específicas de QD e Spike são misturadas para alcançar uma proporção específica de moléculas. Apenas QD e Spike são misturados, e ambas as soluções são altamente purificadas. Para avaliar a rotulagem de QDs, um gel de acrilamida pode ser lançado e carregado apenas com QD, bem como QD conjugado para Spike, o que resulta em faixas de peso molecular mais pesadas.
Os QDs utilizados neste protocolo têm um espectro de excitação/emissão de fluorescência sintonizado com emissão de 608 nm após excitação UV (≤405 nm) já que a maioria dos QDs têm alta absorção perto da faixa UV produzindo alta fotoluminescência18. Esta é uma combinação não convencional de excitação/emissão que requer um microscópio para ter canais personalizáveis. Muitos microscópios confocal tradicionais podem ser configurados com os filtros certos e linhas laser para alcançar essa excitação/emissão. Alternativamente, a excitação do QD no primeiro máximo de absorção, aproximadamente 10 a 20 nm de distância do pico de emissão (por exemplo, 592 nm para QD608 usado aqui), também será capaz de produzir fotoluminescência suficiente.
O software baseado em nuvem usado neste protocolo de análise de alto conteúdo usa um esquema de nomeação que se baseia em etapas anteriores. Por exemplo, os primeiros objetos segmentados são núcleos, que criam uma população chamada Núcleos. Depois disso, a célula ou citoplasma pode ser identificada como uma região de ROI dentro dos Núcleos populacionais. A terminologia utilizada no software de análise de imagens define o nome da população de objetos segmentados utilizando o bloco de construção do núcleo como Núcleos. No entanto, eles não necessariamente têm que ser núcleos e podem ser corpos celulares se não houver corante nuclear disponível. Este esquema de nomeação também pode ser alterado e personalizado dentro do nome de saída de cada bloco de construção.
Nosso protocolo não contava com interações de membrana, mas isso poderia ser feito adicionando uma mancha de membrana adicional que é independente do tráfico ACE2-GFP. Para bloquear a vinculação não específica, 0,1% BSA está incluído na mídia de ensaio (DMEM + 0,1% BSA), e as células são cultivadas em 10% FBS também. Proteínas mutantes de pico poderiam ser usadas, mas estávamos limitados a proteínas de pico disponíveis comercialmente. Neste caso, não estava disponível uma proteína spike mutante não-ACE2 mutante SARS-CoV-2.
O método de nanopartículas QD apresenta uma tecnologia poderosa para estudar a ligação e internalização de vírus que dependem da entrada mediada por Spike. Este ensaio pode ser usado para triagem de alto rendimento de forma análoga, como mostrado na Figura 4, para reutilizar e identificar antivirais potentes que bloqueiam a entrada celular. Embora este protocolo só tenha demonstrado o uso de QDs conjugados à linha de referência Washington WA-1 de SARS-CoV-2, este ensaio pode ser facilmente adaptado para estudar as propriedades vinculantes de variantes recém-emergentes como Alpha, Beta, Gamma e Delta através do uso de suas respectivas proteínas de pico conjugadas a QDs.
Divulgações
Os autores não têm conflitos de interesse para divulgar.
Agradecimentos
Esta pesquisa foi apoiada em parte pelo Programa de Pesquisa Intramural do Centro Nacional de Promoção de Ciências Translacionais, NIH. O Laboratório de Pesquisa Naval forneceu financiamento através de seu Instituto interno de Nanociências. A preparação do reagente foi apoiada através do fundo base NRL COVID-19.
Materiais
| Name | Company | Catalog Number | Comments |
| 32% Paraformaldehyde | Electron Microscopy Sciences | 15714 | Used for fixing cells after quantum dot treatment, final concentration 3.2% Used for stabilizing QDs in Optimem I and preventing non-specific interactions, final concentration 0.1% |
| 7.5% Bovine Serum Albumin | Gibco | 15260-037 | Used as a cell viability dye for fluorescence cell counting |
| Acridine Orange / Propidium Iodide Stain | Logos Biosystems | F23001 | Microwell plates used for seeding cells and assaying QD-Spike |
| Black clear bottom 96 well coated plate coated with poly-D-lysine | Greiner | 655946 | Used to support cell culture, DMEM supplement |
| Characterized Fetal Bovine Serum | Cytiva/HyClone | SH30071.03 | Cloud-based high-content image analysis software; V2.9.1 |
| Columbus Analyzer | Perkin Elmer | NA | Used for labeling cell nuclei and cell bodies after fixation, deep red nuclear dye |
| DRAQ5 (5 mM) | ThermoFisher Scientific | 62252 | Basal media for HEK293T cell culture |
| Dulbecco's Minimal Essential Media, D-glucose (4.5g/L), L-glutamine, sodium pyruvate (110 mg/L), phenol red | Gibco | 11995-065 | Used for arranging data after export from Columbus; V2110 Microsoft 365 |
| Excel | Microsoft | NA | Used to continue selection of hACE2-GFP positive cells, DMEM supplement |
| G418 | InvivoGen | ant-gn-5 | Human embryonic kidney cell line stably expression human angiotensin converting enzyme 2 tagged with GFP |
| HEK293T hACE2-GFP | Codex Biosolutions | CB-97100-203 | Automated cell counter |
| Luna Automated Cell Counter | Logos Biosystems | NA | Used for fluorescence cell counting |
| Luna Cell Counting Slides | Logos Biosystems | L12001 | High-content imaging platform |
| Opera Phenix | Perkin Elmer | NA | Imaging media, used for incubating cells with quantum dots |
| Opti-MEM I Reduced Serum Medium | Gibco | 11058-021 | Phosphate-buffered saline without calcium or magnesium used for washing cells during passaging and assaying |
| PBS -/- | Gibco | 10010-023 | Used to prevent bacterial contamination of cell culture, DMEM supplement |
| Penicillin Streptomycin | Gibco | 15140-122 | Used for graphing, data visualization, and statistical analysis;V9.1.0 |
| Prism | GraphPad | NA | Used for assaying SARS-Cov-2 Spike binding to hACE2 and monitoring Spike endocytosis |
| Quantum Dot 608 nm-Spike (QD608-Spike) | custom made by Naval Research Laboratory | Used for inhibition of SARS-Cov-2 Spike binding to hACE2 | |
| SARS-CoV-2 (2019-nCoV) Spike Neutralizing Antibody, Mouse Mab | Sino Biological | 40592-MM57 | Used to dissociate cells from flask during passaging |
| TrypLE Express | Gibco | 12605-010 |
Referências
- Chazotte, B. Labeling Lysosomes in Live Cells with LysoTracker. Cold Spring Harbor Protocols. 2011 (2), 5571(2011).
- Mehta, S., Zhang, J. Biochemical activity architectures visualized-using genetically encoded fluorescent biosensors to map the spatial boundaries of signaling compartments. Accounts of Chemical Research. 54 (10), 2409-2420 (2021).
- Barroso, M. M. Quantum dots in cell biology. The Journal of Histochemistry and Cytochemistry: Official Journal of the Histochemistry Society. 59 (3), 237-251 (2011).
- Cuervo, N. Z., Grandvaux, N. ACE2: Evidence of role as entry receptor for SARS-CoV-2 and implications in comorbidities. eLife. 9, 61390(2020).
- Shang, J., et al. Cell entry mechanisms of SARS-CoV-2. Proceedings of the National Academy of Sciences of the United States of America. 117 (21), 11727-11734 (2020).
- Ghosh, S., et al. β-Coronaviruses use lysosomes for egress instead of the biosynthetic secretory pathway. Cell. 183 (6), 1520-1535 (2020).
- Buchser, W., et al. Assay development guidelines for image-based high content screening, high content analysis and high content imaging. Assay Guidance Manual. Markossian, S., et al. , Eli Lilly & Company and the National Center for Advancing Translational Sciences. (2012).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews. Drug Discovery. 20 (2), 145-159 (2021).
- Huang, Y., Yang, C., Xu, X. F., Xu, W., Liu, S. W. Structural and functional properties of SARS-CoV-2 spike protein: potential antivirus drug development for COVID-19. Acta Pharmacologica Sinica. 41 (9), 1141-1149 (2020).
- Gao, C., et al. SARS-CoV-2 spike protein interacts with multiple innate immune receptors. bioRxiv: the preprint server for biology. , 227462(2020).
- Zhang, Q., et al. Heparan sulfate assists SARS-CoV-2 in cell entry and can be targeted by approved drugs in vitro. Cell Discovery. 6 (1), 80(2020).
- Hoffmann, M., et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and Is blocked by a clinically proven protease inhibitor. Cell. 181 (2), 271-280 (2020).
- Cai, Y., et al. Distinct conformational states of SARS-CoV-2 spike protein. Science. 369 (6511), New York, N.Y. 1586-1592 (2020).
- Oh, E., et al. Meta-analysis of cellular toxicity for cadmium-containing quantum dots. Nature Nanotechnology. 11 (5), 479-486 (2016).
- Gorshkov, K., et al. Quantum dot-conjugated SARS-CoV-2 spike pseudo-virions enable tracking of angiotensin Converting enzyme 2 binding and endocytosis. ACS Nano. 14 (9), 12234-12247 (2020).
- Narayanan, S. S., Pal, S. K. Aggregated CdS quantum dots: Host of biomolecular ligands. The Journal of Physical Chemistry B. 110 (48), 24403-24409 (2006).
- Wang, S., et al. Endocytosis of the receptor-binding domain of SARS-CoV spike protein together with virus receptor ACE2. Virus Research. 136 (1), 8-15 (2008).
- Hildebrandt, N., et al. Energy transfer with semiconductor quantum dot bioconjugates: A versatile platform for biosensing, energy harvesting, and other developing applications. Chemical Reviews. 117 (2), 536(2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados