Method Article
HEK293T 세포에서 결합 및 엔도사이토시스를 추적하기 위한 양자점 접합 SARS-CoV-2 스파이크 삼량체의 고처리량 공초점 이미징
요약
이 프로토콜에서, 재조합 SARS-CoV-2 스파이크에 접합된 양자점은 세포-기반 분석을 가능하게 하여 원형질막에서 hACE2에 대한 스파이크 결합 및 세포질 내로의 결합된 단백질의 후속 엔도사이토시스를 모니터링한다.
초록
세포 형광 현미경 검사를위한 새로운 기술의 개발은 약물 발견을위한 고처리량 스크리닝 방법을 촉진했습니다. 양자점은 밝고 안정적인 광발광뿐만 아니라 좁은 발광 대역으로 스며든 우수한 광물리학적 특성을 가진 형광 나노입자입니다. 양자점은 구형이며, 표면 화학의 적절한 변형과 함께, 세포 응용을 위해 생체분자를 접합하는데 사용될 수 있다. 이러한 광학 특성은 생체 분자로 기능화 할 수있는 능력과 결합하여 수용체 - 리간드 상호 작용 및 세포 밀매를 조사하기위한 훌륭한 도구입니다. 여기에서는 양자점을 사용하여 SARS-CoV-2 스파이크 단백질의 결합 및 엔도사이토시스를 추적하는 방법을 제시합니다. 이 프로토콜은 양자점을 활용하여 세포 생리학의 맥락에서 단백질-단백질 상호작용 및 인신매매를 연구하고자 하는 실험가들을 위한 가이드로 사용될 수 있다.
서문
형광 현미경을 통해 연구원은 특수 염료1, 유전적으로 인코딩된 형광 단백질2 및 양자점(QD) 형태의 형광 나노입자를 사용하여 세포의 내부 작용을 조사할 수 있습니다3. 2019 년 심각한 급성 호흡기 증후군 코로나 바이러스 (SARS-CoV-2) 세계적인 유행병의 경우, 연구자들은 바이러스가 원형질막과 세포질에서 세포와 어떻게 상호 작용하는지 이해하기 위해 형광 현미경을 사용했습니다. 예를 들어, 연구자들은 비리온 표면의 SARS-CoV-2 스파이크 단백질과 인간 세포 표면의 인간 안지오텐신 전환 효소 2 (hACE2)의 결합, 원형질막에서의 융합을 통한 후속 내재화 및 스파이크 : hACE2 단백질 복합체의 내시세포증4,5에 대한 통찰력을 얻을 수있었습니다. 또한 세포 형광 이미징을 사용하여 리소좀을 통해 세포로부터 SARS-CoV-2 유출 에 대한 훌륭한 통찰력을 얻었는데, 이는 이전에 골지에서 싹트는 전통적인 소포를 통해 발생하는 것으로 생각되었던 코로나 바이러스의 독특한 특징입니다6 다른 많은 바이러스와 마찬가지로. 생물학적 연구의 거의 모든 측면의 주류 인 세포 형광 현미경 검사 기술은 전체 동물의 초해상도 이미징에서 약물 스크리닝을위한 자동화 된 고함량 다중 파라 메트릭 이미징에 이르기까지 광범위한 응용 분야에서 필연적으로 발전해 왔습니다. 여기서, 자동화된 고함량 공초점 현미경은 바이러스 스파이크 단백질에 접합된 형광 QDs를 사용하는 SARS-CoV-2 세포 진입의 연구에 적용된다.
생물학적 이미징 플랫폼에 의해 생성된 이미지의 고함량 분석은 다중 모드 플레이트 리더를 사용하여 얻을 수 있는 전체 웰 강도와 같은 단일 파라미터보다 귀중한 생물학적 통찰력을 더 크게 추출할 수 있게 해줍니다7. 자동화된 세분화 알고리즘을 사용하여 시야에서 객체를 분리함으로써, 각각의 오브젝트 또는 오브젝트 집단은 각각의 이용가능한 형광 채널8에서 강도, 면적 및 텍스처와 같은 파라미터에 대해 분석될 수 있다. 많은 측정값을 다변량 데이터 세트에 결합하는 것은 표현형 프로파일링에 유용한 접근 방식입니다. 원하는 표현형, 예를 들어 누점 형태의 QD 내재화가 알려져 있는 경우, 치료의 효능을 평가하기 위해 크기, 수 및 강도와 같은 누자와 관련된 측정을 사용할 수 있다.
클라우드 기반 고콘텐츠 이미징 분석 소프트웨어는 고콘텐츠 이미징 플랫폼을 포함한 다양한 계측기 데이터 출력을 수용할 수 있습니다. 이미지 저장 및 온라인 분석을 위해 클라우드 기반 서버를 사용함으로써 사용자는 이미징 장비 또는 데이터가 저장된 네트워크 드라이브에서 데이터를 업로드 할 수 있습니다. 프로토콜의 분석 부분은 클라우드 소프트웨어 환경 내에서 수행되며 다운스트림 데이터 시각화를 위해 다양한 파일 형식으로 데이터를 내보낼 수 있습니다.
SARS-CoV-2 바이러스는 조립 및 복제를 돕는 비구조 및 구조 단백질로 구성됩니다. SARS-CoV-2 스파이크는 S1과 S2라는 두 개의 도메인을 가지고 있으며, S1은 원형질막에서 hACE2 상호 작용을 담당하는 수용체 결합 도메인을 포함합니다9. 스파이크는 또한 hACE210,11 이외에 공동-수용체로서 작용할 수 있는 원형질막에서 다른 분자들과 상호작용하는 것으로 밝혀졌다. 스파이크 단백질 서열 전체에서, 특히 S1/S2 계면에서, 막횡단 세린 프로테아제 2 (TMPRSS2)12 이후 막에서 융합을 가능하게 하는 프로테아제 절단 부위가 존재한다. 다양한 재조합 SARS-CoV-2 스파이크 단백질은 개별 수용체 결합 도메인으로부터 S1, S2, S1 및 S2를 갖는 S2 및 전체 스파이크 트리머로부터 연구 활동에 사용하기 위해 여러 상업 벤더로부터 생산되었다13.
이 작업에서, QDs의 표면은 히스티딘 태그 (QD-Spike)를 포함하는 재조합 스파이크 트리머로 기능화되었다. 해군 연구소 광학 나노 물질 섹션에서 생산 한 QD에는 카드뮴 셀레나이드 코어와 황화아연 쉘14,15가 포함되어 있습니다. QD 표면 상의 아연은 재조합 단백질 내의 히스티딘 잔기를 좌표화하여 형태와 기능면에서 SARS-CoV-2 바이러스 입자와 유사한 기능화된 QD를 형성한다. 나노입자의 생성과 컨쥬게이션된 단백질은 QD 컨쥬게이션된 수용체 결합 도메인15를 사용하여 이전에 기술되었다. 이 방법은 인간 세포의 생리적 맥락에서 SARS-CoV-2 스파이크 활성을 연구하는 연구자를 안내 할 수있는 세포 배양 준비, QD 치료, 이미지 수집 및 데이터 분석 프로토콜을 설명합니다.
프로토콜
본 연구에 사용된 HEK293T 세포주는 불멸화 세포주이다. 이 연구에서는 인간 또는 동물 피험자가 사용되지 않았다.
1. 세포 배양 및 파종
- 멸균 생물 안전 캐비닛 내부에는 개인 보호 장비 (실험실 장갑, 실험실 코트 및 안전 안경 포함)를 착용하고 Dulbecco의 변형 이글 배지 (DMEM)에 10 % 태아 소 혈청 (FBS), 1 % 페니실린 / 스트렙토 마이신 (P / S) 및 250 μg / mL G418을 보충하여 세포 배양 배지를 준비하십시오.
- 500mL의 배지에 DMEM 443.75mL, FBS 50mL, P/S 5mL, G418 1.25mL를 첨가합니다.
- 0.2 μm 필터 플라스크를 통해 여과하고 박테리아 오염을 피하기 위해 멸균성을 유지한다.
- 멸균된 생물안전 캐비닛 내부에, 세포 배양 배지의 웰 당 100 μL에 20,000개의 세포가 있는 검은색, 투명한 바닥, 폴리-D-라이신이 코팅된 96-웰 플레이트의 내부 60웰을 시드한다. 외부 36 웰을 웰 당 100 μL의 포스페이트 완충 식염수 (PBS)로 채운다.
- 세포를 계수하려면 2μL의 아크리딘 오렌지 및 프로피듐 요오드화 염색을 18μL의 세포 현탁액에 첨가한다.
- 이 용액 10 μL를 세포 카운팅 슬라이드의 한쪽 면에 로딩한다.
- 1.2.1단계를 반복합니다. 및 1.2.2. 을 눌러 슬라이드의 양쪽을 로드합니다.
- 슬라이드를 자동화된 셀 카운터에 놓습니다.
- 형광 계수 프로토콜을 선택하고 Count를 누릅니다. 슬라이드의 양쪽을 계산하고 두 라이브 셀 밀도의 평균을 계산합니다.
- 필요한 세포 현탁액의 부피를 계산하려면 평균 세포 밀도로 필요한 총 세포 수를 나눕니다.
- 적절한 밀도와 분포가 달성되었는지 확인하기 위해 광 현미경으로 시딩 한 후 우물을 검사하십시오.
- 플레이트를 5% CO2와 함께 37°C의 가습된 인큐베이터에서 하룻밤 동안 인큐베이션한다.
2. QD-스파이크로 세포의 처리
- 이미징 배지에서 희석하여 0.1% 소 혈청 알부민(BSA)을 제조하였다.
- 10mL의 이미징 배지에 130μL의 7.5% BSA를 넣고 혼합합니다.
- 12-웰 저장소 또는 분석 플레이트에서, 이미징 매질에서 0.1% BSA를 사용하여 440 nM QD-스파이크 (SARS-CoV-2, Isolate USA-WA1/2020) 스톡을 20 nM으로 희석한다.
- QD-스파이크의 1 웰에 대해, 2.27 μL의 440 nM QD-스파이크를 20 nM의 최종 농도를 위해 0.1% BSA 이미징 매질의 47.73 μL에 첨가한다.
- 6-포인트 1:3 연속 희석액(3배)을 생성하려면, 14.26 μL의 QD-스파이크를 0.1% BSA 이미징 매질의 285.74 μL에 첨가하여 20 nM의 최고 농도를 만든다. 100 μL의 20 nM QD-스파이크를 0.1% BSA 매질의 200 μL에 첨가하여 6.67 nM QD-스파이크의 두 번째 희석을 만든다. 네 번 반복하여 여섯 개의 농도 점을 생성합니다.
- 다중 채널 흡입기를 사용하여 각 웰에서 사용된 모든 미디어를 제거합니다. 다중 채널 피펫을 사용하여 이미징 매체(100μL/웰)로 한 번 세척합니다.
- 100μL의 이미징 미디어를 흡인하고 QD-스파이크 용액 50μL/웰을 다시 추가합니다.
- 플레이트를 5% CO2와 함께 37°C의 가습된 인큐베이터에서 3시간 동안 인큐베이션한다. 4단계를 계속합니다.
3. 고정 및 핵 염색
- 멸균 된 생물 안전 캐비닛 내부에서 개인 보호 장비 (실험실 장갑, 실험실 코트 및 안전 안경 포함)를 착용하고 0.1 % BSA 이미징 매체에 4 % 파라 포름 알데히드 (PFA)를 준비하십시오.
- 각 웰에서 50μL의 QD-스파이크를 흡인하고 4% PFA의 100μL/웰을 첨가합니다.
- 우물이 마르지 않도록하십시오. 자동화된 다중 채널 피펫을 사용하여 웰이 건조되지 않도록 합니다(권장).
- 실온에서 15분 동안 인큐베이션한다.
- 1x 인산완충식염수(PBS)로 세 번 세척한다.
- 5 mM 원액을 PBS로 1:1000 희석하여 딥 레드 핵 염료를 제조하였다.
- PBS를 흡인하고 희석된 핵 염료 50 μL/웰을 다시 첨가한다.
- 실온에서 30분 동안 인큐베이션한다.
- PBS로 세 번 씻으십시오.
- 나중에 이미징을 위해 플레이트 실러를 사용하여 플레이트 또는 씰을 이미지화하십시오. 플레이트를 4°C에서 보관한다. 형광은 몇 주 이상 안정해야 한다.
4. 획득 설정 및 이미징
- 이미징 플랫폼용 소프트웨어를 시작합니다. 고콘텐츠 이미징 플랫폼을 켜면 상태 표시줄의 표시등이 켜져 있어야 합니다. 기기가 연결되어 있지 않으면 소프트웨어가 오프라인 분석 모드로 전환됩니다.
- 이미징 플랫폼에 로그인합니다.
- 새 획득 프로토콜을 만듭니다.
- 플레이트 유형을 96웰 클리어 바닥 이미징 플레이트로 선택합니다.
참고: 계측기의 플레이트 선택은 다양할 수 있으며 높이, 너비 및 기타 거리와 같은 플레이트 치수를 포함하는 올바른 플레이트 정의를 로드하여 간격과 초점이 잘 정해져 있는 것을 사용자 지정할 수 있습니다. - 광학 모드를 공초점으로 선택하고 배율을 40x 수침으로 선택합니다.
- 비닝을 1로 선택합니다.
- 4.3.5-4.3.8 단계에 설명된 대로 채널 및 형광 여기/방출 파장을 선택합니다. 이미지를 보고 설정이 적절한지 확인하기 위해 설정을 선택할 때 스냅숏을 만듭니다.
참고: DPC(Digital Phase Contrast)를 사용하면 세포 얼룩이나 형광 염료 없이 살아있는 세포를 시각화할 수 있습니다. 고정 셀에는 권장하지 않습니다. - DPC에 대한 모드 (예: 고대비)를 선택하여 잘 정의된 세포체를 생성합니다. 스냅숏을 만들어 이미지 창에 표시된 대로 이 설정이 적절한지 확인합니다.
- 세포 내에서 ACE2 트래피킹의 시각화를 허용하는 ACE2-GFP 세포주와 함께 사용할 FITC 채널을 선택하십시오.
- QD 채널에 대한 사용자 지정 채널을 만들려면 채널의 삼각형 드롭다운 메뉴를 선택하고 405nm 범위에서 여기와 608nm 범위의 방출을 선택합니다.
- 세포가 고정되고 깊은 적색 핵 염료가 사용 된 경우, 630 nm 이상의 방출을 가진 원간 적색 채널을 선택하여 세포 몸체를 더 묘사하고 세포 마스크로 작용하십시오.
- 강한 세포질 QD 신호가 있는 웰을 선택하여 노출 시간, 레이저 파워 및 Z 높이 위치를 설정합니다. 4.3.10-4.3.13의 단계를 따릅니다.
- 기본 높이가 셀이 원하는 초점면에 있는 이미지를 생성하는지 확인합니다. 엔도사이토시드된 누자가 표적 소기관인 경우, Z-위치는 원형질막이 표적 소기관인 경우보다 낮을 것이다.
- 배경 신호보다 적어도 세 배 더 큰 회색 레벨의 밝은 이미지를 생성하는 노출 시간을 선택합니다. 일반적으로 100ms에서 300ms 사이입니다. 20nM에서 회색 레벨은 200ms의 노출 시간과 80%의 레이저 파워를 사용하여 약 6000a.u.여야 합니다.
- 각 채널의 스냅샷 기능으로 생성된 이미지를 마우스 오른쪽 단추로 클릭하고 강도 표시를 선택하여 회색 수준을 확인합니다. 그런 다음 관심 있는 개체 또는 배경을 마우스 왼쪽 단추로 클릭하여 해당 픽셀의 회색 수준을 봅니다.
- 레이저 파워를 조정하여 관심 물체의 강도를 미세 조정하십시오.
- 플레이트 유형을 96웰 클리어 바닥 이미징 플레이트로 선택합니다.
- 획득 프로토콜을 저장합니다.
- 실험 실행으로 전환하고 플레이트 이름을 입력합니다.
- 실험을 실행합니다.
5. 데이터 분석
- 이미징 소프트웨어로 데이터를 가져옵니다.
- 모든 이미지를 획득한 후 측정값을 이미징 소프트웨어로 내보냅니다. 이렇게 하려면 서버를 설정하고 계정을 만들어야 합니다. 이미징 소프트웨어로 내보낼 데이터의 화면을 만듭니다.
- 이미징 소프트웨어에서 이미지 분석을 선택하여 분석 프로토콜 구축을 시작합니다.
- 화면 목록에서 파일을 마우스 오른쪽 단추로 클릭하고 선택을 클릭하여 이미징 실험에서 측정값을 로드 합니다. 이미지화된 우물과 관련 필드의 플레이트 맵은 창의 왼쪽 하단에 표시되어야 합니다.
- 밝은 세포질 QD 반점이있는 우물을 선택하여 이미지 세분화를 시작하십시오.
- 입력 이미지 빌딩 블록에서 플랫필드 보정을 선택합니다.
- 핵 찾기를 선택하여 프로토콜에 다음 빌딩 블록을 추가합니다.
- 여기서, 핵 마커로서 DPC 또는 원적색 채널을 사용한다.
- 먼저 객체를 정확하게 분할하는 방법을 선택한 다음 드롭다운 메뉴를 사용하여 세그멘테이션을 미세 조정하고 슬라이더를 조정합니다.
참고: 양호한 세분화는 핵, 세포 및 반점에 대한 관심 영역(ROI)을 정확하게 정의합니다. 마커에 대해 양수인 픽셀만 개별 ROI 내에서 캡처해야 합니다. 배경 신호는 제외해야 합니다. 배경이 ROI에서 캡처되는 경우, 방법의 감도가 감소될 수 있거나, 강도의 임계값이 증가하여 세그멘테이션 알고리즘의 엄격성이 증가될 수 있다. 이는 모든 Find 빌딩 블록에 적용할 수 있습니다.
- 다음으로, 세포질 빌딩 블록 찾기 를 추가하여 세포질을 확인합니다.
- 이 경우, 세포질 세분화를 위해 ACE2-GFP 채널을 사용한다.
- 각 세포의 세포질을 가장 잘 식별하는 방법을 선택하십시오.
- 그런 다음 찾기 스팟 빌딩 블록을 추가하여 QD-스파이크 누점을 식별합니다.
- ROI 모집단으로 핵을 선택합니다.
- 셀을 ROI 영역으로 선택합니다. 이것은 전체 세포 내의 누점을 캡처합니다.
- 각 셀에서 QD 누자를 가장 잘 식별하는 방법을 선택합니다.
- 이미지의 모든 객체가 분할되고 이 웰에서 정확하게 식별되면 다른 웰을 선택하여 빌딩 블록과 설정이 일반적으로 다른 웰 및 기타 조건에 적용될 수 있도록 합니다.
- 분할된 모든 개체(핵, 세포질/세포 및 반점)에 대한 강도 계산 특성을 추가합니다.
- 분할된 모든 개체(핵, 세포질/세포 및 반점)에 대한 형태학 특성 계산 을 추가합니다.
- 분할된 모든 개체(핵, 세포질/세포 및 반점)에 대한 텍스처 특성 계산 을 추가합니다.
- 핵과 반점을 포함한 각 집단에 대한 매개 변수를 선택하여 결과를 정의합니다. 개체의 수는 세포 생존력의 간접적 척도로서 사용될 수 있다.
6. 데이터 내보내기6.
- 배치 분석을 클릭하십시오. 다음 단계로 진행하기 전에 분석이 완료될 때까지 기다립니다(단계 6.2).
참고: 배치 분석을 통해 이미지 분석 소프트웨어 서버는 선택한 측정값을 사용하여 분석 프로토콜을 로드하여 데이터를 원격으로 분석할 수 있습니다. - 데이터 세트를 로컬 컴퓨터 또는 네트워크 드라이브로 내보냅니다.
- 연결 파일을 다운로드하여 열어 이미지 분석 소프트웨어를 컴퓨터의 도우미 응용 프로그램에 연결합니다.
- 결과 파일 형식(.txt, .csv, .html 또는 기본 XML)을 선택합니다.
- 드롭다운 메뉴를 사용하여 파일 폴더 설정을 선택합니다.
7. 스프레드시트의 데이터 분석
- 내보낸 파일을 엽니다. .csv 파일인 경우 피벗 테이블을 사용할 수 있도록 .xls 파일 형식으로 저장합니다. 파일 경로가 너무 긴 경우 저장 오류를 방지하기 위해 .xls 파일을 폴더 계층 구조에서 위쪽으로 저장하십시오.
- 스프레드시트에 각 웰의 조건을 지정하는 열을 추가합니다. 예를 들어, 세포 유형, QD-스파이크 변이체, QD-스파이크 농도, 배양 시간 등이 있다.
- 피벗 테이블 함수를 선택하고 추가된 조건을 행으로, 측정된 매개 변수를 열로 사용하여 테이블을 빌드합니다. 각 매개변수에 대한 계산(예: 평균, 표준 편차, 중앙값, 최소값, 최대 또는 개수)을 선택합니다.
- 데이터를 정규화하여 비접합 QD를 0%로, 최고 농도의 QD-Spike를 100%로 포함하여 샘플을 제어합니다.
참고: 중화 항체와 같은 억제제의 효능을 평가하는 경우, 데이터를 배지 전용 처리된 세포(100% 효능) 및 억제제가 없는 QD-Spike(0% 효능)로 정규화합니다. - 그래프 소프트웨어에 데이터를 플로팅합니다.
결과
처리시, QDs는 나노입자가 원형질막 상의 ACE2에 결합하고 엔도사이토시스를 유도할 것이기 때문에 내재화될 것이다. ACE2-GFP 발현 세포주를 사용하여, QD 및 ACE2 둘 다의 전좌는 형광 현미경을 사용하여 시각화될 수 있다. 일단 내부화되면, 두 개의 QD 및 ACE2 신호는 강력한 공동 로컬라이제이션을 보여준다. 이러한 이미지로부터 이미지 세분화 및 후속 분석을 수행하여 스폿 카운트와 같은 관련 파라미터를 추출할 수 있습니다(그림 1, 그림 2B). 도 1A는 대조군으로서 상이한 농도의 QD-Spike 또는 배지만으로 처리된 세포의 몽타주이다. 디지털 위상 콘트라스트, FITC 및 405nm에서 여기, 608nm에서 방출되는 맞춤형 QD 채널을 포함하여 세 가지 채널이 사용되었습니다. DPC는 일반적인 세포 형상의 이미지를 제공한다. DPC 이미지는 전체 세포 형태학의 대략적인 표시를 제공한다. 관심 영역은 DPC 신호와 겹치며 내부화된 QD를 감지하기에 충분합니다. FITC 채널은 ACE2-GFP가 QD-스파이크와 함께 현지화되고 지역화되는 지역에서 축적되는 것을 보여줍니다. QD-스파이크의 농도가 감소함에 따라 QD는 더 이상 보이지 않으며 ACE2-GFP 신호는 제어와 유사합니다. 병합 채널은 ACE2-GFP와 QD-Spike의 공동 현지화를 보여줍니다. 이러한 물체는 분석 소프트웨어로 세분화 할 수있는 반점과 더 큰 축적의 혼합물입니다. 이미지 세분화에 사용되는 소프트웨어 분석 방법의 미세 조정은 관심 대상을 세그먼트화하는 데 사용될 수 있습니다.
여기에서 QD의 최적 농도를 결정하기 위해 20 nM에서 시작하여 여섯 가지 농도의 QD-Spike를 사용한 농도-반응 실험을 수행하였다(도 2A). QD는 2.22nM만큼 낮게 사용할 수 있으며 여전히 결합 및 내재화를 보여줍니다. 그러나 강력한 반응을 보장하기 위해 20 nM 이상의 농도를 사용하는 것이 좋습니다.
QD에 컨쥬게이션하는 동안, 단백질 응집이 발생할 수 있다. 집계의 주요 문제점은 셀 위에 축적되어 이미지 세분화 단계에서 아티팩트를 발생시킨다는 것입니다. 응집체는 세포에 들어갈 수 없으며 나노 입자 전체는 더 이상 바이러스 입자와 닮지 않습니다 (그림 3). 응집체는 QD 용액에서 밝고 뭉친 침전물로서 발견될 수 있다.
이 분석은 또한 바이러스 유입을 차단하는 중화 항체와 같은 생물학적 제제를 평가하는데 사용될 수 있다. QDs를 중화 항체와 함께 인큐베이션하였고 (도 4A), 30 μg/mL에서 시작하여, 세포에 첨가하기 전에 실온에서 30분 동안 인큐베이션하였다. 기준 워싱턴 균주인 SARS-CoV-2 RBD에 대해 제기된 항체가 사용되었다. 이들은 결합, 내재화를 차단하고, QD 전용으로 처리된 세포에 비해 측정된 스팟 카운트의 감소를 야기하였다(도 4B).
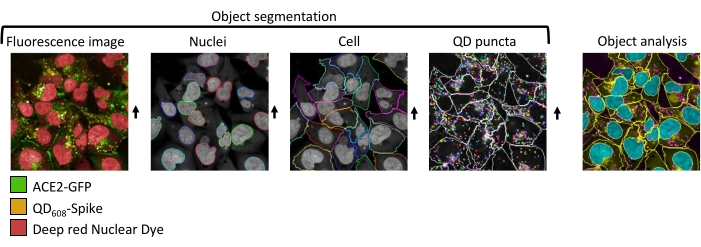
그림 1: QD608-스파이크로 처리된 ACE2-GFP 세포의 고함량 이미지 분석 및 세분화. 정확한 세분화의 대표 이미지. 첫째, 핵은 핵 마커를 함유하는 채널로부터 분할되어 ROI 집단을 생성한다. ROI 집단으로부터, 세포를 윤곽을 그리는 ROI 영역은 ACE2-GFP 채널을 사용하여 분절된다. 마지막으로, QD608-스파이크 스팟은 셀 ROI 영역에서 분할됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

도 2: QD608-스파이크는 hACE2에 결합하고 hACE2-GFP HEK293T 세포에서 엔도사이토시스화된다. (A) QD608-스파이크(마젠타) 또는 배지만의 여러 농도로 처리된 hACE2-GFP(황색) HEK293T 세포의 몽타주 이미지. 디지털 위상 콘트라스트(cyan)를 사용하여 세포체를 시각화하였다. 스케일 바: 20 μm. (B) 20 nM QD608-스파이크 (100%) 및 대조군 (0%)으로 정규화된 QD608-스파이크 스팟 카운트의 고함량 분석. N = 웰을 삼중으로 늘립니다. 오류 막대는 표준 편차(S.D.)를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
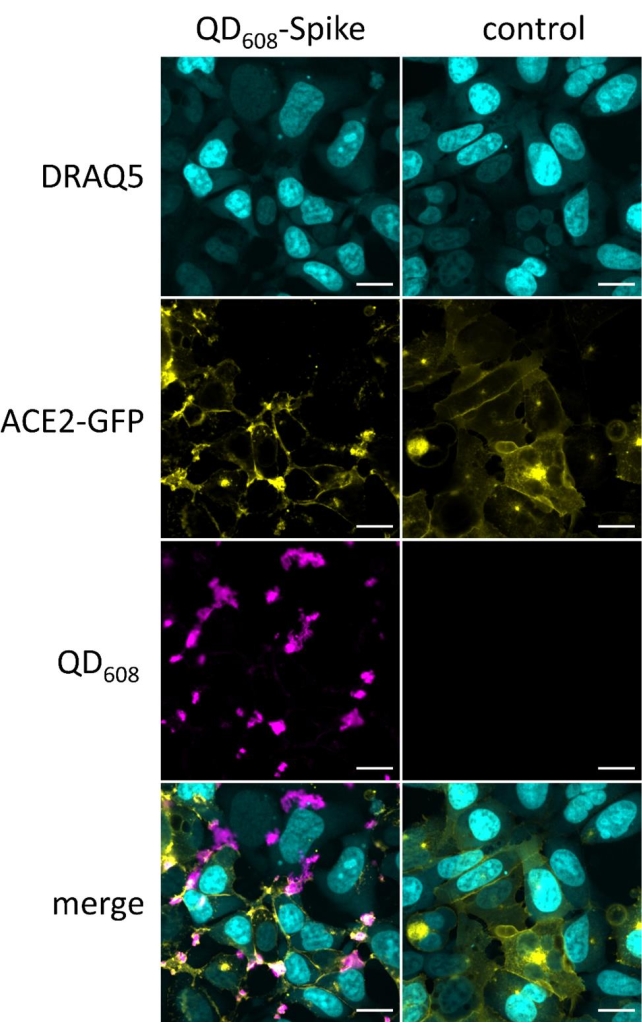
그림 3: 응집된 QD608-스파이크는 hACE2-GFP HEK293T 세포 내로 내재화되지 않는다. 10 nM QD608-스파이크 또는 배지만으로 처리된 hACE2-GFP HEK293T 세포의 이미지 몽타주. 스케일 바: 20 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

도 4: 중화 항체는 hACE2-GFP HEK293T 세포에서 QD608-스파이크의 엔도사이토시스를 차단한다. (A) QD608-스파이크(자홍색)로 처리된 hACE2-GFP(노란색) HEK293T 세포의 이미지 몽타주를 디지털 위상 조영제(cyan)를 사용하여 세포체를 확인하기 위해 디지털 위상 조영제(cyan)를 사용하여 중화 항체의 농도를 감소시키면서 예비배양하였다. 스케일 바: 20 μm. (B) 미디어 전용 제어(100%) 및 10 nM QD608-스파이크만(0%)으로 정규화된 QD608-스파이크 스팟 카운트의 고함량 분석. N = 웰을 삼중으로 늘립니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
이 기사에 설명 된 방법은 고처리량 공초점 현미경을 사용하여 인간 세포에서 기능화 된 QD를 이미징하는 데 필요한 단계를 제공합니다. 이 방법은 SARS-CoV-2 스파이크 및 hACE2 엔도사이토시스에 대한 연구를 가능하게 하기 때문에 TMPRSS2 및 막 융합의 활성보다는 엔도사이토시스가 바이러스 진입의 주요 경로인 세포에 가장 적합합니다. QD 모델 및 상업적으로 입수가능한 스파이크 삼량체 상의 C-말단 His-태그의 특성 때문에, 스파이크 S1 및 S2 도메인의 임의의 TMPRSS2 절단은 S2 도메인에만 부착된 QD를 남겨둘 것이다12. 이는 RBD가 S1에서 발견된다는 점을 감안할 때 내재화를 방지할 수 있다. 따라서 hACE2가 결합되고 TMPRSS2가 스파이크를 절단하는 셀 표면에서의 이벤트 시퀀스가 정확하다면 내재화가없는 음의 신호가 예상됩니다.
이 프로토콜은 이미징 세포 과정을 다루기 때문에 배양 된 세포는 hACE2의 발현으로 인한 바이러스 감염에 허용되어야합니다. hACE2는 세포 내로 일시적으로 형질감염될 수 있거나, 또는 hACE2를 발현하는 안정한 세포주가 생성될 수 있다17. hACE2에 현미경을 사용하여 관찰 할 수있는 GFP와 같은 형광 단백질 태그가 없으면 hACE2에 대한 항체가있는 면역 형광을 사용하여 hACE2 발현 및 국소화를 확인하는 것이 좋습니다. GFP 태그 hACE2는 QD-스파이크 엔도사이토시스 이후 hACE2 밀매를 시각화하는 추가적인 이점을 제공합니다. 내인성 hACE2 발현을 갖는 일부 세포주가 사용될 수 있지만, 이것은 면역세포화학을 사용하여 확인되어야 한다. 일부 경우에, 내인성 발현은 QD-스파이크에 대해 충분한 hACE2 결합 파트너를 제공하지 않으며, 고처리량 공초점 현미경에 의해 저조하게 검출되는 신호를 초래할 수 있다.
프로토콜의 한 가지 중요한 단계는 재조합 His-태그가 달린 SARS-CoV-2 스파이크에 컨쥬게이션된 고품질 QD의 조달을 포함한다. 이를 위한 전제 조건은 응집을 일으키지 않고 QD에 접합될 수 있는 적절하게 정제된 단백질이다. 집계된 QD에는 성공적인 실험을 방해하는 몇 가지 문제가 있습니다. 따라서 전체 실험 전에 분석 도구(예: UV 가시 분광법, 투과 전자 현미경 또는 동적 광산란)를 사용하여 QD-스파이크를 테스트하는 것은 귀중한 시약을 테스트하는 경우 귀중한 자원을 보존하는 것이 좋습니다16. 스파이크가있는 QD의 라벨링 중에 QD와 스파이크의 특정 농도가 혼합되어 분자의 특정 비율을 달성합니다. QD와 스파이크만 혼합되며 두 솔루션 모두 고도로 정제됩니다. QD의 라벨링을 평가하기 위해, 아크릴아미드 겔은 QD 단독으로뿐만 아니라 QD로 캐스팅 및 로딩 될 수 있으며 QD는 스파이크에 접합되어 분자량 밴드가 더 무거워집니다.
이 프로토콜에 사용되는 QD는 대부분의 QD가 높은 광발광을 생성하는 UV 범위 근처에서 높은 흡수율을 갖기 때문에 UV 여기 (≤405nm) 후 608nm 방출로 조정 된 형광 여기 / 방출 스펙트럼을 가지고 있습니다18. 이것은 현미경이 사용자 정의 가능한 채널을 가져야하는 비 전통적인 여기 / 방출 조합입니다. 많은 전통적인 공초점 현미경은 이러한 여기 / 방출을 달성하기 위해 올바른 필터와 레이저 라인으로 설정할 수 있습니다. 대안적으로, 방출 피크로부터 대략 10 내지 20 nm 떨어진 제1 흡수 최대치(예를 들어, 여기서 사용된 QD608 의 경우 592 nm)에서의 QD의 여기는 또한 충분한 광발광을 생성할 수 있을 것이다.
이 고콘텐츠 분석 프로토콜에 사용되는 클라우드 기반 소프트웨어는 이전 단계를 기반으로 하는 명명 체계를 사용합니다. 예를 들어, 분할되는 첫 번째 객체는 Nuclei이며, Nuclei라는 모집단을 만듭니다. 이어서, 세포 또는 세포질은 집단 핵 내의 ROI 영역으로서 확인될 수 있다. 이미지 분석 소프트웨어에 사용되는 용어는 핵 빌딩 블록을 사용하여 분할된 개체 집단의 이름을 Nuclei로 설정합니다. 그러나, 그들은 반드시 핵일 필요는 없으며 핵 염료가 없는 경우 세포체가 될 수 있다. 이 명명 체계는 각 빌딩 블록 출력 이름 내에서 변경하고 사용자 정의 할 수도 있습니다.
우리의 프로토콜은 막 상호 작용을 설명하지 않았지만, ACE2-GFP 트래피킹과 독립적 인 추가 멤브레인 얼룩을 추가하여 수행 할 수 있습니다. 비특이적 결합을 차단하기 위해, 0.1% BSA가 분석 배지 (DMEM + 0.1% BSA)에 포함되고, 세포는 또한 10% FBS에서 성장한다. 돌연변이 스파이크 단백질을 사용할 수 있었지만, 상업적으로 이용가능한 스파이크 단백질로 제한하였다. 이 경우, SARS-CoV-2 돌연변이체 비-ACE2 결합 스파이크 단백질은 이용가능하지 않았다.
QD 나노입자 방법은 스파이크 매개 진입에 의존하는 바이러스의 결합 및 내재화를 연구하는 강력한 기술을 제시합니다. 이 분석은 도 4에 도시된 바와 같이, 유사한 방식으로 고처리량 스크리닝에 사용될 수 있으며, 세포 진입을 차단하는 강력한 항바이러스제를 용도 변경하고 동정할 수 있다. 이 프로토콜은 SARS-CoV-2의 워싱턴 WA-1 기준 균주에 접합된 QD의 사용을 입증했을 뿐이지만, 이 분석은 QD에 접합된 각각의 스파이크 단백질의 사용을 통해 알파, 베타, 감마 및 델타와 같은 새로 출현하는 변이체의 결합 특성을 연구하기 위해 쉽게 적응할 수 있다.
공개
저자는 공개 할 이해 상충이 없습니다.
감사의 말
이 연구는 NIH 국립 번역 과학 발전 센터의 교내 연구 프로그램에 의해 부분적으로 지원되었습니다. 해군 연구소는 내부 나노 과학 연구소를 통해 자금을 제공했습니다. 시약 준비는 NRL COVID-19 기본 기금을 통해 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 32% Paraformaldehyde | Electron Microscopy Sciences | 15714 | Used for fixing cells after quantum dot treatment, final concentration 3.2% Used for stabilizing QDs in Optimem I and preventing non-specific interactions, final concentration 0.1% |
| 7.5% Bovine Serum Albumin | Gibco | 15260-037 | Used as a cell viability dye for fluorescence cell counting |
| Acridine Orange / Propidium Iodide Stain | Logos Biosystems | F23001 | Microwell plates used for seeding cells and assaying QD-Spike |
| Black clear bottom 96 well coated plate coated with poly-D-lysine | Greiner | 655946 | Used to support cell culture, DMEM supplement |
| Characterized Fetal Bovine Serum | Cytiva/HyClone | SH30071.03 | Cloud-based high-content image analysis software; V2.9.1 |
| Columbus Analyzer | Perkin Elmer | NA | Used for labeling cell nuclei and cell bodies after fixation, deep red nuclear dye |
| DRAQ5 (5 mM) | ThermoFisher Scientific | 62252 | Basal media for HEK293T cell culture |
| Dulbecco's Minimal Essential Media, D-glucose (4.5g/L), L-glutamine, sodium pyruvate (110 mg/L), phenol red | Gibco | 11995-065 | Used for arranging data after export from Columbus; V2110 Microsoft 365 |
| Excel | Microsoft | NA | Used to continue selection of hACE2-GFP positive cells, DMEM supplement |
| G418 | InvivoGen | ant-gn-5 | Human embryonic kidney cell line stably expression human angiotensin converting enzyme 2 tagged with GFP |
| HEK293T hACE2-GFP | Codex Biosolutions | CB-97100-203 | Automated cell counter |
| Luna Automated Cell Counter | Logos Biosystems | NA | Used for fluorescence cell counting |
| Luna Cell Counting Slides | Logos Biosystems | L12001 | High-content imaging platform |
| Opera Phenix | Perkin Elmer | NA | Imaging media, used for incubating cells with quantum dots |
| Opti-MEM I Reduced Serum Medium | Gibco | 11058-021 | Phosphate-buffered saline without calcium or magnesium used for washing cells during passaging and assaying |
| PBS -/- | Gibco | 10010-023 | Used to prevent bacterial contamination of cell culture, DMEM supplement |
| Penicillin Streptomycin | Gibco | 15140-122 | Used for graphing, data visualization, and statistical analysis;V9.1.0 |
| Prism | GraphPad | NA | Used for assaying SARS-Cov-2 Spike binding to hACE2 and monitoring Spike endocytosis |
| Quantum Dot 608 nm-Spike (QD608-Spike) | custom made by Naval Research Laboratory | Used for inhibition of SARS-Cov-2 Spike binding to hACE2 | |
| SARS-CoV-2 (2019-nCoV) Spike Neutralizing Antibody, Mouse Mab | Sino Biological | 40592-MM57 | Used to dissociate cells from flask during passaging |
| TrypLE Express | Gibco | 12605-010 |
참고문헌
- Chazotte, B. Labeling Lysosomes in Live Cells with LysoTracker. Cold Spring Harbor Protocols. 2011 (2), 5571(2011).
- Mehta, S., Zhang, J. Biochemical activity architectures visualized-using genetically encoded fluorescent biosensors to map the spatial boundaries of signaling compartments. Accounts of Chemical Research. 54 (10), 2409-2420 (2021).
- Barroso, M. M. Quantum dots in cell biology. The Journal of Histochemistry and Cytochemistry: Official Journal of the Histochemistry Society. 59 (3), 237-251 (2011).
- Cuervo, N. Z., Grandvaux, N. ACE2: Evidence of role as entry receptor for SARS-CoV-2 and implications in comorbidities. eLife. 9, 61390(2020).
- Shang, J., et al. Cell entry mechanisms of SARS-CoV-2. Proceedings of the National Academy of Sciences of the United States of America. 117 (21), 11727-11734 (2020).
- Ghosh, S., et al. β-Coronaviruses use lysosomes for egress instead of the biosynthetic secretory pathway. Cell. 183 (6), 1520-1535 (2020).
- Buchser, W., et al. Assay development guidelines for image-based high content screening, high content analysis and high content imaging. Assay Guidance Manual. Markossian, S., et al. , Eli Lilly & Company and the National Center for Advancing Translational Sciences. (2012).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews. Drug Discovery. 20 (2), 145-159 (2021).
- Huang, Y., Yang, C., Xu, X. F., Xu, W., Liu, S. W. Structural and functional properties of SARS-CoV-2 spike protein: potential antivirus drug development for COVID-19. Acta Pharmacologica Sinica. 41 (9), 1141-1149 (2020).
- Gao, C., et al. SARS-CoV-2 spike protein interacts with multiple innate immune receptors. bioRxiv: the preprint server for biology. , 227462(2020).
- Zhang, Q., et al. Heparan sulfate assists SARS-CoV-2 in cell entry and can be targeted by approved drugs in vitro. Cell Discovery. 6 (1), 80(2020).
- Hoffmann, M., et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and Is blocked by a clinically proven protease inhibitor. Cell. 181 (2), 271-280 (2020).
- Cai, Y., et al. Distinct conformational states of SARS-CoV-2 spike protein. Science. 369 (6511), New York, N.Y. 1586-1592 (2020).
- Oh, E., et al. Meta-analysis of cellular toxicity for cadmium-containing quantum dots. Nature Nanotechnology. 11 (5), 479-486 (2016).
- Gorshkov, K., et al. Quantum dot-conjugated SARS-CoV-2 spike pseudo-virions enable tracking of angiotensin Converting enzyme 2 binding and endocytosis. ACS Nano. 14 (9), 12234-12247 (2020).
- Narayanan, S. S., Pal, S. K. Aggregated CdS quantum dots: Host of biomolecular ligands. The Journal of Physical Chemistry B. 110 (48), 24403-24409 (2006).
- Wang, S., et al. Endocytosis of the receptor-binding domain of SARS-CoV spike protein together with virus receptor ACE2. Virus Research. 136 (1), 8-15 (2008).
- Hildebrandt, N., et al. Energy transfer with semiconductor quantum dot bioconjugates: A versatile platform for biosensing, energy harvesting, and other developing applications. Chemical Reviews. 117 (2), 536(2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유