Method Article
HEK293T細胞における結合およびエンドサイトーシスを追跡するための量子ドット共役SARS-CoV-2スパイク三量体のハイスループット共焦点イメージング
要約
このプロトコルでは、組換えSARS-CoV-2スパイクにコンジュゲートした量子ドットは、細胞ベースのアッセイを可能にし、原形質膜でのhACE2へのスパイク結合およびその後の細胞質への結合タンパク質のエンドサイトーシスをモニターする。
要約
細胞蛍光顕微鏡法の新技術の開発により、創薬のためのハイスループットスクリーニング方法が容易になりました。量子ドットは、明るく安定したフォトルミネッセンスと狭い発光帯を吹き込んだ優れた光物性を有する蛍光ナノ粒子である。量子ドットは球状の形状であり、表面化学を適切に修正して、細胞用途のために生体分子を共役するために使用することができる。これらの光学特性は、生体分子でそれらを機能化する能力と相まって、受容体 - リガンド相互作用および細胞輸送を調査するための優れたツールとなる。ここでは、量子ドットを使用してSARS-CoV-2スパイクタンパク質の結合およびエンドサイトーシスを追跡する方法を提示する。このプロトコルは、細胞生理学の文脈におけるタンパク質間相互作用およびトラッキングを研究するために量子ドットを利用しようとしている実験家のためのガイドとして使用することができる。
概要
蛍光顕微鏡により、研究者は特殊な色素1、遺伝的にコードされた蛍光タンパク質2、および量子ドット(QD)3の形の蛍光ナノ粒子を使用して、細胞の内部の仕組みを覗くことができます。2019年の重症急性呼吸器症候群コロナウイルス(SARS-CoV-2)の世界的なパンデミックのために、研究者は蛍光顕微鏡法を使用して、ウイルスが原形質膜と細胞質の両方で細胞とどのように相互作用するかを理解しました。例えば、研究者らは、ビリオン表面のSARS-CoV-2スパイクタンパク質とヒト細胞表面のヒトアンジオテンシン変換酵素2(hACE2)との結合、その後の原形質膜での融合によるインターナリゼーション、およびスパイク:hACE2タンパク質複合体のエンドサイトーシスに関する洞察を得ることができました4,5。また、他の多くのウイルスと同様に、ゴルジ体から出芽する伝統的な小胞を介して発生すると考えられていたコロナウイルスのユニークな特徴である細胞蛍光イメージングを使用して、リソソームを介して細胞からSARS-CoV-2が出ることについても大きな洞察が得られました6。生物学的研究のほぼすべての側面の柱である細胞蛍光顕微鏡技術は、動物全体の超解像イメージングから薬物スクリーニングのための自動高含有量マルチパラメトリックイメージングまで、その幅広さと応用範囲において必然的に進歩してきました。ここでは、自動高含量共焦点顕微鏡法が、ウイルススパイクタンパク質にコンジュゲートした蛍光QDを用いたSARS-CoV-2細胞侵入の研究に適用される。
生物学的イメージングプラットフォームによって生成された画像のハイコンテンツ分析は、マルチモーダルプレートリーダーを使用して取得するウェル全体の強度などの単一のパラメータよりも、貴重な生物学的洞察のより大きな抽出を可能にします7。自動セグメンテーションアルゴリズムを使用して視野内のオブジェクトを分離することにより、各オブジェクトまたはオブジェクトの集団を、利用可能な各蛍光チャネルの強度、面積、テクスチャなどのパラメータについて分析できます8。多くの測定値を多変量データセットに結合することは、表現型プロファイリングに便利なアプローチです。プンクタの形態のQDインターナリゼーションなどの所望の表現型が知られている場合、サイズ、数、強度などのプンクタに関連する測定値を使用して、治療の有効性を評価することができる。
クラウドベースのハイコンテンツイメージング解析ソフトウェアは、ハイコンテンツイメージングプラットフォームを含む多種多様な機器データ出力に対応できます。画像ストレージとオンライン分析にクラウドベースのサーバーを使用することで、ユーザーはイメージング機器またはデータが保存されているネットワークドライブからデータをアップロードできます。プロトコルの分析部分はクラウドソフトウェア環境内で行われ、ダウンストリームのデータ視覚化のためにさまざまなファイル形式でデータをエクスポートできます。
SARS-CoV-2ウイルスは、その集合および複製を助ける非構造的および構造的タンパク質からなる。SARS-CoV-2スパイクはS1とS2と呼ばれる2つのドメインを有し、S1は原形質膜におけるhACE2相互作用に関与する受容体結合ドメインを含む9。スパイクはまた、hACE210,11に加えて共受容体として作用し得る原形質膜において他の分子と相互作用することが見出されている。スパイクタンパク質配列全体、特にS1/S2界面には、膜貫通後の膜における融合を可能にするプロテアーゼ切断部位がセリンプロテアーゼ2(TMPRSS2)12に存在する。個々の受容体結合ドメインから、S1、S2、S1、およびスパイク三量体全体を研究活動に使用するために、複数の商業ベンダーからS1、S2、およびスパイク三量体全体に対して、さまざまな組換えSARS-CoV-2スパイクタンパク質が産生されています13。
この研究において、QDsの表面を、ヒスチジンタグを含む組換えスパイク三量体(QD-Spike)で官能化した。海軍研究所光学ナノ材料部門によって製造されたQDは、セレン化カドミウムコアと硫化亜鉛シェル14,15を含む。QD表面上の亜鉛は、組換えタンパク質内のヒスチジン残基を調整して、SARS-CoV-2ウイルス粒子に形態および機能的に類似した機能化されたQDを形成する。ナノ粒子の生成およびタンパク質コンジュゲーションは、QD結合型受容体結合ドメインを用いて先に記載した15。この方法は、ヒト細胞の生理学的文脈におけるSARS-CoV-2スパイク活性を研究する際に研究者を導くことができる細胞培養調製物、QD処理、画像取得、およびデータ解析プロトコルを記述する。
プロトコル
本研究で用いたHEK293T細胞株は、不死化細胞株である。ヒトまたは動物の被験者は、この研究において使用されなかった。
1. 細胞の培養と播種
- 滅菌バイオセーフティキャビネット内で、個人用保護具(ラボグローブ、ラボコート、安全メガネを含む)を着用し、ダルベッコの改変イーグル培地(DMEM)に10%ウシ胎児血清(FBS)、1%ペニシリン/ストレプトマイシン(P/S)、および250μg/mL G418を補充して細胞培養培地を準備します。
- 500 mL の培地に、443.75 mL の DMEM、50 mL の FBS、5 mL の P/S、および 1.25 mL の G418 を加えます。
- 0.2μmのフィルターフラスコを通してろ過し、細菌汚染を避けるために無菌性を維持してください。
- 滅菌バイオセーフティキャビネットの内部に、黒色の透明底のポリD-リジンコーティングされた96ウェルプレートの内部60ウェルに、細胞培養培地1ウェルあたり100μLで20,000個の細胞を播種する。外側の36ウェルを1ウェルあたり100 μLのリン酸緩衝生理食塩水(PBS)で満たします。
- 細胞を計数するために、2 μLのアクリジンオレンジおよびヨウ化プロピジウム染色剤を18 μLの細胞懸濁液に加える。
- この溶液10 μLを細胞計数スライドの片側にロードします。
- 手順 1.2.1 を繰り返します。および 1.2.2.をクリックしてスライドの両側を読み込みます。
- スライドを自動セルカウンターに置きます。
- 蛍光カウントプロトコルを選択し、カウントを押し ます。スライドの両側を数え、2 つの生細胞密度の平均を計算します。
- 必要な細胞懸濁液の体積を計算するには、必要な細胞の総数を平均細胞密度で除算する。
- 播種後のウェルを光学顕微鏡で検査し、適切な密度と分布が達成されていることを確認します。
- プレートを5%CO2で37°Cの加湿インキュベーターで一晩インキュベートする。
2. QDスパイクによる細胞の処理
- 1%ウシ血清アルブミン(BSA)をイメージング培地で希釈して調製する。
- 10 mL のイメージング培地に、130 μL の 7.5% BSA を加えて混合します。
- 12 ウェルリザーバーまたはアッセイプレートで、イメージング培地中の 0.1% BSA を使用して、440 nM QD-スパイク (SARS-CoV-2, Isolate USA-WA1/2020) ストックを 20 nM に希釈します。
- 1ウェルのQD-スパイクについて、2.27 μL の 440 nM QD-スパイクを 47.73 μL の 0.1% BSA イメージング培地に添加し、最終濃度を 20 nM にします。
- 6 点 1:3 の連続希釈 (3 連) を生成するには、14.26 μL の QD-Spike を 285.74 μL の 0.1% BSA イメージング培地に追加して、最高濃度の 20 nM にします。200 μL の 0.1% BSA 培地に 100 μL の 20 nM QD-スパイクを加え、6.67 nM QD-スパイクの第 2 希釈を行います。4 回繰り返して、6 つの濃度ポイントを生成します。
- マルチチャンネルアスピレーターを使用して、使用済みのメディアをすべて各ウェルから取り出します。マルチチャンネルピペットを使用して、イメージングメディア(100 μL/ウェル)で1回洗浄します。
- 100 μL のイメージング培地を吸引し、50 μL/ウェルの QD-スパイク溶液を再び添加します。
- プレートを5%CO2で37°Cの加湿インキュベーターで3時間インキュベートする。ステップ 4 に進みます。
3. 固定と核染色
- 滅菌バイオセーフティキャビネット内で、個人用保護具(ラボグローブ、ラボコート、安全メガネを含む)を着用し、0.1%BSAイメージングメディアに4%パラホルムアルデヒド(PFA)を準備します。
- 各ウェルから50 μLのQD-スパイクを吸引し、100 μL/ウェルの4% PFAを加える。
- 井戸を乾かさないでください。ウェルの乾燥を避けるために、自動マルチチャンネルピペットを使用してください(推奨)。
- 室温で15分間インキュベートする。
- 1xリン酸緩衝生理食塩水(PBS)で3回洗浄する。
- 5mM原液をPBSで1:1000希釈に希釈して深赤色核色素を調製した。
- PBSを吸引し、希釈した核色素を50 μL/ウェル戻します。
- 室温で30分間インキュベートする。
- PBSで3回洗ってください。
- 後日撮像するためのプレートシーラーを用いてプレートまたはシールを撮像する。プレートを4°Cで保管する。 蛍光は数週間以上安定している必要があります。
4. 集録のセットアップとイメージング
- イメージングプラットフォーム用のソフトウェアを起動します。ハイコンテンツイメージングプラットフォームをオンにすると、ステータスバーのライトが点灯します。マシンが接続されていない場合、ソフトウェアはオフライン分析モードに入ります。
- イメージングプラットフォームにログインします。
- 新しい集録プロトコルを作成します。
- 96 ウェルクリアボトムイメージングプレートとして プレートタイプ を選択します。
メモ:機器のプレート選択はさまざまであり、高さ、幅、その他の距離などのプレート寸法を含む正しいプレート定義をロードして、ウェル間隔と焦点を定義することによってカスタマイズできます。 - 共焦点として 光学モード を選択し、倍率を40倍の水浸漬として選択します。
- [ビン分割] を 1 として選択します。
- ステップ4.3.5-4.3.8で説明されているようにチャンネルと蛍光励起/発光波長を選択します。設定が選択されたときにスナップショットを撮って画像を表示し、設定が適切であることを確認します。
注:デジタル位相コントラスト(DPC)により、細胞染色や蛍光色素を使用せずに生細胞を視覚化できます。固定セルにはお勧めできません。 - 「ハイコントラスト」などの DPC の モード を選択して、明確に定義された細胞体を生成します。スナップショットを撮って、イメージウィンドウに表示されるように、この設定が適切であることを確認します。
- 細胞内のACE2トラフィッキングの可視化を可能にするACE2-GFP細胞株で使用する FITCチャネル を選択します。
- QDチャンネルのカスタムチャンネルを作成するには、チャンネルの三角形のドロップダウンメニューを選択し、405nm範囲の励起と608nm範囲の発光を選択します。
- 細胞を固定し、深赤色の核色素を使用した場合は、630nmを超える発光を有する遠赤色チャネルを選択して、細胞体をさらに描写し、細胞マスクとして作用させる。
- 細胞質QD信号が強いウェルを選択して、露光時間、レーザーパワー、Z高さ位置を設定します。4.3.10 から 4.3.13 までの手順に従います。
- デフォルトの高さで、セルが目的の焦点面にある画像が生成されるかどうかを確認します。エンドサイトーシス穿刺が標的細胞小器官である場合、Z位は原形質膜が標的細胞小器官である場合よりも低くなる。
- 背景信号の少なくとも 3 倍のグレーレベルを持つ明るい画像を生成する露光時間を選択します。これは通常、100 ~ 300 ミリ秒です。20 nM では、グレー レベルは 200 ミリ秒の露光時間と 80 ミリ秒のレーザー出力を使用して約 6000 a.u. になります。
- 各チャンネルのスナップショット機能で生成された画像を右クリックし、[ 強度を表示]を選択してグレーレベルを確認します。次に、目的のオブジェクトまたは背景を左クリックして、そのピクセルのグレーレベルを表示します。
- レーザー出力を調整して、目的のオブジェクトの強度を微調整します。
- 96 ウェルクリアボトムイメージングプレートとして プレートタイプ を選択します。
- 集録プロトコルを保存します。
- [ 実験の実行 ] に切り替えて、[ プレート名] を入力します。
- 実験を実行します。
5. データ解析
- イメージングソフトウェアにデータをインポートします。
- すべての画像を取得したら、測定値をイメージングソフトウェアにエクスポートします。これには、サーバーを確立し、アカウントを作成する必要があります。イメージングソフトウェアにエクスポートするデータの画面を作成します。
- イメージング ソフトウェアで、[ 画像解析 ] を選択して解析プロトコルの構築を開始します。
- 画像化実験の測定値を読み込むには、画面リストでファイルを右クリックし、[ 選択]をクリックします。画像化された井戸とそれに関連するフィールドのプレートマップは、ウィンドウの左下隅に表示されます。
- 明るい細胞質QDスポットのあるウェルを選択して、画像セグメンテーションを開始します。
- 入力イメージの構成要素で、「フラットフィールド補正」を選択します。
- 次の構成要素をプロトコルに追加するには、[ Nuclei の検索] を選択します。
- ここでは、DPCまたは遠赤色チャネルをNucleiマーカーとして使用します。
- 最初にオブジェクトを正確にセグメント化する方法を選択し、ドロップダウンメニューを使用してスライダを調整してセグメンテーションを微調整します。
注: 適切なセグメンテーションは、核、細胞、およびスポットの関心領域 (ROI) を正確に定義します。マーカーに対して正のピクセルのみが、個々のROI内でキャプチャする必要があります。バックグラウンド信号は除外する必要があります。バックグラウンドがROIに取り込まれる場合、本方法の感度を低下させることができ、または強度の閾値を増加させて、セグメンテーションアルゴリズムのストリンジェンシーを増加させることができる。これは、すべての検索ビルディングブロックに適用されます。
- 次に、[ 細胞質の検索] ビルディング ブロック を追加して、細胞質を識別します。
- この場合、細胞質セグメンテーションにACE2-GFPチャネルを使用する。
- 各細胞の細胞質を最もよく識別する方法を選択します。
- 次に、[ スポットの検索] ビルディング ブロック を追加して、QD スパイク プンクタを識別します。
- ROI 母集団として [核 ] を選択します。
- ROI 領域としてセルを選択します。これは、細胞全体のプンクタをキャプチャします。
- 各セルの QD 穿刺を最もよく識別する方法を選択します。
- 画像内のすべてのオブジェクトがセグメント化され、このウェル内で正確に識別されたら、他のウェルを選択して、ビルディングブロックと設定を他のウェルやその他の条件に一般的に適用できるようにします。
- セグメント化されたすべてのオブジェクト(核、細胞質/細胞、およびスポット)の 強度の計算プロパティ を追加します。
- セグメント化されたすべてのオブジェクト(核、細胞質/細胞、およびスポット)の 形態計算プロパティ を追加します。
- セグメント化されたすべてのオブジェクト(核、細胞質/細胞、スポット)の テクスチャプロパティの計算 を追加します。
- 核や斑点を含む各母集団のパラメータを選択して、結果を定義します。オブジェクトの数は、細胞生存率の間接的な尺度として使用できます。
6. データのエクスポート
- バッチ分析をクリックします。分析が完了するのを待ってから、次のステップに進みます (ステップ 6.2)。
メモ: バッチ分析により、画像解析ソフトウェアサーバーは、選択した測定値を使用して解析プロトコルをロードし、データをリモートで分析できます。 - データセットをローカル コンピューターまたはネットワーク ドライブにエクスポートします。
- 接続ファイルをダウンロードして開いて、画像解析ソフトウェアをコンピュータ上のヘルパーアプリケーションに接続します。
- 結果ファイルの種類 (.txt、.csv、.html、またはネイティブ XML) を選択します。
- ドロップダウンメニューで ファイルフォルダ 設定を選択します。
7. スプレッドシート内のデータを分析する
- エクスポートしたファイルを開きます。.csvファイルの場合は、.xlsファイル形式で保存して、ピボットテーブルを使用できるようにします。ファイル パスが長すぎる場合は、保存時のエラーを回避するために、フォルダー階層の上位にある.xls ファイルを保存します。
- 各ウェルの条件を指定する列をスプレッドシートに追加します。例えば、細胞型としては、QD−スパイク変異体、QD−スパイク濃度、インキュベーション時間等が挙げられる。
- ピボットテーブル関数を選択し、追加された条件を行として、測定パラメータを列として使用してテーブルを作成します。各パラメータの計算(平均、標準偏差、中央値、最小値、最大値、またはカウント)を選択します。
- 非共役QDを0%、最高濃度のQD-スパイクを含む対照サンプルにデータを正規化します。
注:中和抗体などの阻害剤の有効性を評価する場合は、データを培地のみの処理細胞(100%有効性)および阻害剤なしのQD-スパイク(0%有効性)に正規化します。 - グラフ作成ソフトウェアでデータをプロットします。
結果
治療すると、ナノ粒子が原形質膜上のACE2に結合し、エンドサイトーシスを誘導するため、QDは内在化される。ACE2-GFP発現細胞株を用いて、QDおよびACE2の両方の転座を蛍光顕微鏡を用いて可視化することができる。一旦内部化されると、2つのQDおよびACE2シグナルは強い共局在化を示す。これらの画像から、画像セグメンテーションおよびその後の分析を実行して、スポットカウントなどの関連パラメータを抽出することができる(図1、 図2B)。 図1A は、対照として異なる濃度のQD−スパイクまたは培地のみで処理した細胞のモンタージュである。デジタル位相コントラスト、FITCおよび405nmでの励起と608nmでの発光を備えたカスタムQDチャネルを含む3つのチャネルが使用された。DPC は、一般的なセル形状のイメージを提供します。DPC画像は、細胞形態全体のおおよその指標を提供する。関心領域はDPC信号と重なり合い、内部化されたQDを検出するのに十分です。FITCチャネルは、ACE2-GFPが局在化を変化させ、QD-スパイクと共局在化する領域で蓄積することを示している。QDスパイクの濃度が低下すると、QDはもはや見えなくなり、ACE2-GFP信号は制御に似ています。マージチャネルは、ACE2-GFPとQD-スパイクの共局在化を示しています。これらのオブジェクトは、解析ソフトウェアでセグメント化できるスポットと大きな蓄積が混在しています。画像セグメンテーションに使用されるソフトウェア解析方法の微調整を使用して、目的のオブジェクトをセグメント化できます。
ここでは、QDの最適濃度を決定するために、20nMから開始して、6つの異なる濃度のQD-スパイクを用いた濃度応答実験が行われた(図2A)。QDは2.22nMという低さで使用でき、それでもバインディングとインターナリゼーションを示します。ただし、堅牢な応答を確保するために、20nM以上の濃度を使用することをお勧めします。
QDへのコンジュゲーション中に、タンパク質凝集が起こり得る。集計の主な問題は、それらがセルの上に蓄積し、画像セグメンテーションステップでアーチファクトを引き起こすことです。凝集体は細胞に入ることができず、ナノ粒子全体はもはやウイルス粒子に似ていません(図3)。凝集体は、QD溶液中に明るく凝集した沈殿物としてスポットすることができる。
このアッセイは、ウイルスの侵入をブロックする中和抗体などの生物製剤を評価するためにも使用できます。QDを中和抗体(図4A)と共に、30μg/mLから始めて室温で30分間インキュベートしてから細胞に添加した。参照ワシントン株、SARS−CoV−2 RBDに対して産生された抗体が使用された。それらは、結合を遮断し、インターナリゼーションし、QDのみで処理した細胞と比較して測定スポット数の減少を引き起こした(図4B)。
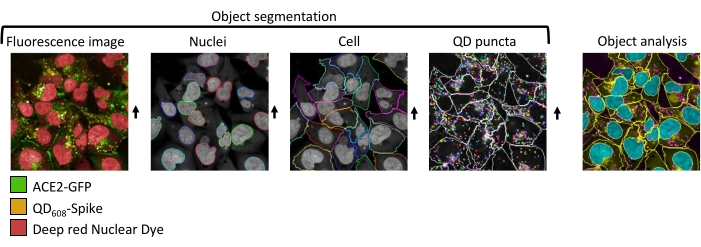
図1:QD608-スパイクで処理したACE2-GFP細胞のハイコンテント画像解析とセグメンテーション。 正確なセグメンテーションの代表的な画像。まず、核マーカーを含むチャネルから核をセグメント化して、ROI集団を作成します。ROI集団から、細胞を輪郭を描くROI領域を、ACE2−GFPチャネルを用いてセグメント化する。最後に、QD608スパイクスポットは、セルROI領域からセグメント化される。 この図の拡大版を表示するには、ここをクリックしてください。

図2:QD608-スパイクはhACE2と結合し、hACE2-GFP HEK293T細胞においてエンドサイトーシスされる。 (A)hACE2-GFP(黄色)HEK293T細胞の画像モンタージュを数濃度のQD608-スパイク(マゼンタ)または培地のみで処理した。デジタル位相差(シアン)を用いて、細胞体を可視化した。スケールバー:20μm. (B)QD608-スパイクスポットカウントの高含有量分析は、20nM QD608-スパイク(100%)および対照(0%)に正規化されました。N = 3 連の井戸。エラーバーは標準偏差(S.D.)を示す。 この図の拡大版を表示するには、ここをクリックしてください。
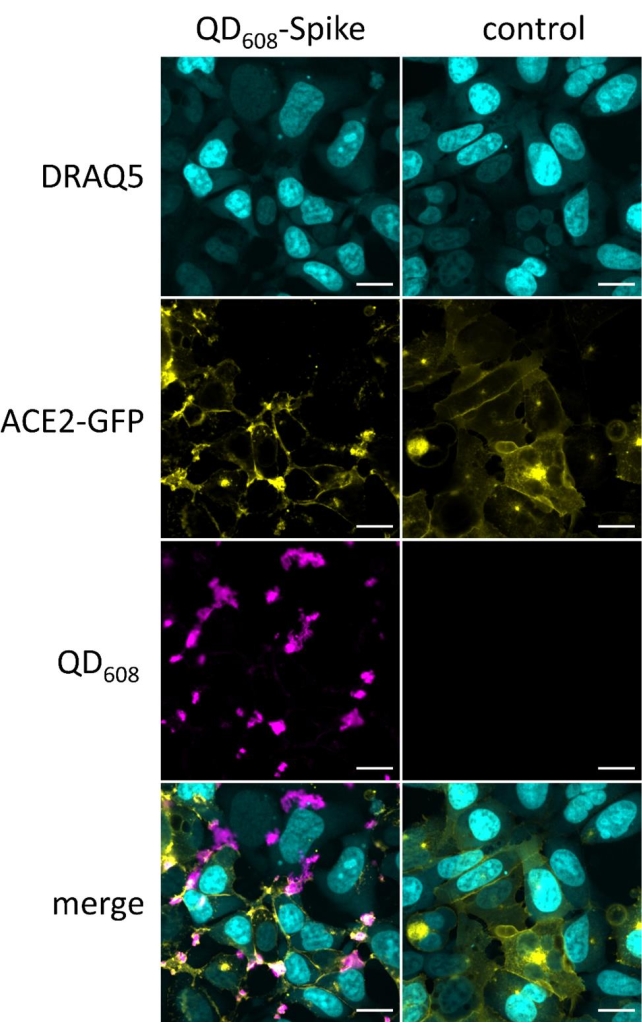
図3:凝集したQD608-スパイクは、hACE2-GFP HEK293T細胞にインターナライズされていない。 10nM QD608-スパイクまたは培地のみで処理したhACE2-GFP HEK293T細胞の画像モンタージュ。スケール バー: 20 μm。 この図の拡大版を表示するには、ここをクリックしてください。

図4:中和抗体は、hACE2-GFP HEK293T細胞におけるQD608-スパイクのエンドサイトーシスを遮断する。 (A)hACE2-GFP(黄色)の画像モンタージュQD608-スパイク(マゼンタ)で処理したHEK293T細胞を中和抗体の濃度を低下させるとともにプレインキュベートし、デジタル位相差(シアン)を使用して細胞体を同定した。スケールバー:20 μm. (B) QD608-スパイクスポットカウントの高含有量分析をメディアのみのコントロール(100%)と10 nM QD608-スパイクのみ(0%)に正規化。N = 3 連の井戸。この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
この記事で説明する方法は、ハイスループット共焦点顕微鏡を使用してヒト細胞中の機能化QDをイメージングするために必要な手順を提供します。この方法は、SARS-CoV-2スパイクおよびhACE2エンドサイトーシスの研究を可能にするため、TMPRSS2の活性および膜融合ではなく、エンドサイトーシスがウイルス侵入の主な経路である細胞に最も適している。QDモデルの性質と市販のスパイク三量体上のC末端Hisタグのため、スパイクS1ドメインとS2ドメインのTMPRSS2切断は、QDをS2ドメインに結合したままにするだけです12。これは、RBD が S1 で見つかった場合、内部化を防ぐことができます。したがって、細胞表面での一連の事象が正確であった場合、hACE2が結合し、TMPRSS2がスパイクを切断する場合、インターナリゼーションのない負のシグナルが予想される。
このプロトコルは細胞プロセスのイメージングを扱うので、培養細胞はhACE2の発現を伴うウイルス感染に対して許容性でなければならない。hACE2は、細胞に一過性にトランスフェクトされ得るか、またはhACE2を発現する安定な細胞株が生成され得る17。hACE2に顕微鏡で観察できるGFPなどの蛍光タンパク質タグがない限り、hACE2に対する抗体を用いて免疫蛍光を用いてhACE2の発現および局在を検証することが推奨される。GFPタグ付きhACE2は、QDスパイクエンドサイトーシス後のhACE2トラフィッキングを視覚化するという追加の利点を与える。内因性hACE2発現を有するいくつかの細胞株が使用され得るが、これは免疫細胞化学を用いて確認されるべきである。場合によっては、内因性発現は、QD−スパイクのための十分なhACE2結合パートナーを提供せず、ハイスループット共焦点顕微鏡によって不十分に検出されるシグナルをもたらし得る。
プロトコルの重要なステップの1つには、組換えHisタグ付きSARS-CoV-2スパイクにコンジュゲートされた高品質のQDの調達が含まれます。このための前提条件は、凝集を引き起こすことなくQDにコンジュゲートすることができる適切に精製されたタンパク質である。集約されたQDには、実験の成功を妨げるいくつかの問題があります。したがって、完全な実験の前に分析ツール(例えば、紫外可視分光法、透過型電子顕微鏡、または動的光散乱)を使用してQD-スパイクをテストすることは、貴重な試薬をテストする場合に貴重な資源を保存するために強く推奨されます16。スパイクによるQDの標識の間、特定の濃度のQDおよびスパイクが混合され、分子の特定の比率を達成する。QDとスパイクのみが混合され、両方の溶液が高度に精製される。QDの標識を評価するために、アクリルアミドゲルをキャストしてQD単独で装填し、スパイクにQDコンジュゲートさせることができ、その結果、分子量バンドが重くなります。
このプロトコルで使用されるQDは、ほとんどのQDがUV範囲付近に高い吸収を持ち、高いフォトルミネッセンスを生成するため、UV励起(≤405nm)に続いて608nmの発光に調整された蛍光励起/発光スペクトルを有する18。これは型破りな励起/発光の組み合わせであり、顕微鏡にカスタマイズ可能なチャンネルが必要です。多くの従来の共焦点顕微鏡は、この励起/放出を達成するために、適切なフィルターとレーザーラインでセットアップすることができます。あるいは、第1の吸収極大におけるQDの励起は、発光ピークから約10〜20nm離れた場所(例えば、ここで使用されるQD608 については592nm)でも、十分なフォトルミネッセンスを生成することができるであろう。
この高コンテンツ分析プロトコルで使用されるクラウドベースのソフトウェアは、前の手順に基づいて構築された命名スキームを使用します。たとえば、セグメント化された最初のオブジェクトは nuclei で、Nuclei という母集団を作成します。これに続いて、細胞または細胞質は、集団核内のROI領域として同定され得る。画像解析ソフトウェアで使用される用語では、核ビルディング ブロックを使用してセグメント化されたオブジェクトの母集団の名前を Nuclei として設定します。しかし、それらは必ずしも核である必要はなく、核色素が利用可能でなければ細胞体であり得る。この命名スキームは、各ビルディングブロック出力名内で変更およびカスタマイズすることもできます。
我々のプロトコルは膜相互作用を考慮していなかったが、これはACE2-GFPトラフィッキングとは無関係の追加の膜染色剤を追加することによって行うことができる。非特異的結合をブロックするために、0.1%BSAをアッセイ培地(DMEM+0.1%BSA)に含ませ、細胞を同様に10%FBS中で増殖させる。変異型スパイクタンパク質も使用可能であったが、市販のスパイクタンパク質に限られていた。この場合、SARS-CoV-2変異体である非ACE2結合スパイクタンパク質は利用できなかった。
QDナノ粒子法は、スパイク媒介性侵入に依存するウイルスの結合とインターナリゼーションを研究するための強力な技術を提示する。このアッセイは、 図4に示すように、同様の方法でハイスループットスクリーニングに使用して、細胞侵入をブロックする強力な抗ウイルス薬を再利用および同定することができます。このプロトコルは、SARS-CoV-2のワシントンWA-1参照株にコンジュゲートしたQDの使用を実証したにすぎないが、このアッセイは、QDにコンジュゲートしたそれぞれのスパイクタンパク質の使用を通じて、アルファ、ベータ、ガンマ、デルタなどの新たに出現した変異体の結合特性を研究するために容易に適合させることができる。
開示事項
著者らは、開示する利益相反はありません。
謝辞
この研究は、NIHの国立トランスレーショナルサイエンス推進センターの学内研究プログラムによって部分的に支援されました。海軍研究所は、内部のナノサイエンス研究所 を通じて 資金を提供しました。試薬調製は、NRL COVID-19基本基金 を通じて 支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 32% Paraformaldehyde | Electron Microscopy Sciences | 15714 | Used for fixing cells after quantum dot treatment, final concentration 3.2% Used for stabilizing QDs in Optimem I and preventing non-specific interactions, final concentration 0.1% |
| 7.5% Bovine Serum Albumin | Gibco | 15260-037 | Used as a cell viability dye for fluorescence cell counting |
| Acridine Orange / Propidium Iodide Stain | Logos Biosystems | F23001 | Microwell plates used for seeding cells and assaying QD-Spike |
| Black clear bottom 96 well coated plate coated with poly-D-lysine | Greiner | 655946 | Used to support cell culture, DMEM supplement |
| Characterized Fetal Bovine Serum | Cytiva/HyClone | SH30071.03 | Cloud-based high-content image analysis software; V2.9.1 |
| Columbus Analyzer | Perkin Elmer | NA | Used for labeling cell nuclei and cell bodies after fixation, deep red nuclear dye |
| DRAQ5 (5 mM) | ThermoFisher Scientific | 62252 | Basal media for HEK293T cell culture |
| Dulbecco's Minimal Essential Media, D-glucose (4.5g/L), L-glutamine, sodium pyruvate (110 mg/L), phenol red | Gibco | 11995-065 | Used for arranging data after export from Columbus; V2110 Microsoft 365 |
| Excel | Microsoft | NA | Used to continue selection of hACE2-GFP positive cells, DMEM supplement |
| G418 | InvivoGen | ant-gn-5 | Human embryonic kidney cell line stably expression human angiotensin converting enzyme 2 tagged with GFP |
| HEK293T hACE2-GFP | Codex Biosolutions | CB-97100-203 | Automated cell counter |
| Luna Automated Cell Counter | Logos Biosystems | NA | Used for fluorescence cell counting |
| Luna Cell Counting Slides | Logos Biosystems | L12001 | High-content imaging platform |
| Opera Phenix | Perkin Elmer | NA | Imaging media, used for incubating cells with quantum dots |
| Opti-MEM I Reduced Serum Medium | Gibco | 11058-021 | Phosphate-buffered saline without calcium or magnesium used for washing cells during passaging and assaying |
| PBS -/- | Gibco | 10010-023 | Used to prevent bacterial contamination of cell culture, DMEM supplement |
| Penicillin Streptomycin | Gibco | 15140-122 | Used for graphing, data visualization, and statistical analysis;V9.1.0 |
| Prism | GraphPad | NA | Used for assaying SARS-Cov-2 Spike binding to hACE2 and monitoring Spike endocytosis |
| Quantum Dot 608 nm-Spike (QD608-Spike) | custom made by Naval Research Laboratory | Used for inhibition of SARS-Cov-2 Spike binding to hACE2 | |
| SARS-CoV-2 (2019-nCoV) Spike Neutralizing Antibody, Mouse Mab | Sino Biological | 40592-MM57 | Used to dissociate cells from flask during passaging |
| TrypLE Express | Gibco | 12605-010 |
参考文献
- Chazotte, B. Labeling Lysosomes in Live Cells with LysoTracker. Cold Spring Harbor Protocols. 2011 (2), 5571(2011).
- Mehta, S., Zhang, J. Biochemical activity architectures visualized-using genetically encoded fluorescent biosensors to map the spatial boundaries of signaling compartments. Accounts of Chemical Research. 54 (10), 2409-2420 (2021).
- Barroso, M. M. Quantum dots in cell biology. The Journal of Histochemistry and Cytochemistry: Official Journal of the Histochemistry Society. 59 (3), 237-251 (2011).
- Cuervo, N. Z., Grandvaux, N. ACE2: Evidence of role as entry receptor for SARS-CoV-2 and implications in comorbidities. eLife. 9, 61390(2020).
- Shang, J., et al. Cell entry mechanisms of SARS-CoV-2. Proceedings of the National Academy of Sciences of the United States of America. 117 (21), 11727-11734 (2020).
- Ghosh, S., et al. β-Coronaviruses use lysosomes for egress instead of the biosynthetic secretory pathway. Cell. 183 (6), 1520-1535 (2020).
- Buchser, W., et al. Assay development guidelines for image-based high content screening, high content analysis and high content imaging. Assay Guidance Manual. Markossian, S., et al. , Eli Lilly & Company and the National Center for Advancing Translational Sciences. (2012).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews. Drug Discovery. 20 (2), 145-159 (2021).
- Huang, Y., Yang, C., Xu, X. F., Xu, W., Liu, S. W. Structural and functional properties of SARS-CoV-2 spike protein: potential antivirus drug development for COVID-19. Acta Pharmacologica Sinica. 41 (9), 1141-1149 (2020).
- Gao, C., et al. SARS-CoV-2 spike protein interacts with multiple innate immune receptors. bioRxiv: the preprint server for biology. , 227462(2020).
- Zhang, Q., et al. Heparan sulfate assists SARS-CoV-2 in cell entry and can be targeted by approved drugs in vitro. Cell Discovery. 6 (1), 80(2020).
- Hoffmann, M., et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and Is blocked by a clinically proven protease inhibitor. Cell. 181 (2), 271-280 (2020).
- Cai, Y., et al. Distinct conformational states of SARS-CoV-2 spike protein. Science. 369 (6511), New York, N.Y. 1586-1592 (2020).
- Oh, E., et al. Meta-analysis of cellular toxicity for cadmium-containing quantum dots. Nature Nanotechnology. 11 (5), 479-486 (2016).
- Gorshkov, K., et al. Quantum dot-conjugated SARS-CoV-2 spike pseudo-virions enable tracking of angiotensin Converting enzyme 2 binding and endocytosis. ACS Nano. 14 (9), 12234-12247 (2020).
- Narayanan, S. S., Pal, S. K. Aggregated CdS quantum dots: Host of biomolecular ligands. The Journal of Physical Chemistry B. 110 (48), 24403-24409 (2006).
- Wang, S., et al. Endocytosis of the receptor-binding domain of SARS-CoV spike protein together with virus receptor ACE2. Virus Research. 136 (1), 8-15 (2008).
- Hildebrandt, N., et al. Energy transfer with semiconductor quantum dot bioconjugates: A versatile platform for biosensing, energy harvesting, and other developing applications. Chemical Reviews. 117 (2), 536(2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved