Method Article
Imágenes confocales de alto rendimiento de trímeros de púas de SARS-CoV-2 conjugados con puntos cuánticos para rastrear la unión y la endocitosis en células HEK293T
En este artículo
Resumen
En este protocolo, los puntos cuánticos conjugados con el pico recombinante del SARS-CoV-2 permiten que los ensayos basados en células monitoreen la unión del pico a hACE2 en la membrana plasmática y la posterior endocitosis de las proteínas unidas al citoplasma.
Resumen
El desarrollo de nuevas tecnologías para la microscopía de fluorescencia celular ha facilitado métodos de detección de alto rendimiento para el descubrimiento de fármacos. Los puntos cuánticos son nanopartículas fluorescentes con excelentes propiedades fotofísicas imbuidas de fotoluminiscencia brillante y estable, así como de bandas de emisión estrechas. Los puntos cuánticos son de forma esférica, y con la modificación adecuada de la química de la superficie, se pueden utilizar para conjugar biomoléculas para aplicaciones celulares. Estas propiedades ópticas, combinadas con la capacidad de funcionalizarlas con biomoléculas, las convierten en una excelente herramienta para investigar las interacciones receptor-ligando y el tráfico celular. Aquí, presentamos un método que utiliza puntos cuánticos para rastrear la unión y endocitosis de la proteína espiga del SARS-CoV-2. Este protocolo se puede utilizar como una guía para los experimentalistas que buscan utilizar puntos cuánticos para estudiar las interacciones proteína-proteína y el tráfico en el contexto de la fisiología celular.
Introducción
La microscopía de fluorescencia permite a los investigadores observar el funcionamiento interno de la célula utilizando colorantes especializados1, proteínas fluorescentes codificadas genéticamente2 y nanopartículas fluorescentes en forma de puntos cuánticos (QD)3. Para la pandemia mundial del coronavirus del síndrome respiratorio agudo severo de 2019 (SARS-CoV-2), los investigadores han empleado la microscopía de fluorescencia para comprender cómo el virus interactúa con la célula tanto en la membrana plasmática como en el citoplasma. Por ejemplo, los investigadores han podido obtener información sobre la unión de la proteína Espiga del SARS-CoV-2 en la superficie del virión a la enzima convertidora de angiotensina humana 2 (hACE2) en la superficie de las células humanas, la internalización posterior a través de la fusión en la membrana plasmática y la endocitosis del complejo proteico Spike:hACE24,5. También se han obtenido grandes conocimientos sobre la salida del SARS-CoV-2 de las células a través del lisosoma utilizando imágenes de fluorescencia celular, una característica única de los coronavirus que anteriormente se pensaba que ocurrían a través de la vesícula tradicional que brota del Golgi, como ocurre con muchos otros virus6. Un pilar de casi todos los aspectos de la investigación biológica, la técnica de microscopía de fluorescencia celular ha avanzado necesariamente en su amplitud y alcance de aplicaciones, desde imágenes de superresolución de animales enteros hasta imágenes multiparamétricas automatizadas de alto contenido para la detección de drogas. Aquí, la microscopía confocal automatizada de alto contenido se aplica al estudio de la entrada de células del SARS-CoV-2 utilizando QD fluorescentes conjugados con la proteína espiga viral.
El análisis de alto contenido de las imágenes generadas por las plataformas de imágenes biológicas permite una mayor extracción de valiosos conocimientos biológicos que los parámetros individuales, como la intensidad de todo el pozo, que se obtendría utilizando un lector de placas multimodal7. Al separar los objetos en un campo de visión utilizando algoritmos de segmentación automatizados, cada objeto o una población de objetos se puede analizar en busca de parámetros como la intensidad, el área y la textura en cada canal de fluorescencia disponible8. La combinación de muchas mediciones en conjuntos de datos multivariados es un enfoque útil para la elaboración de perfiles fenotípicos. Cuando se conoce el fenotipo deseado, como la internalización de QD en forma de puntcta, se pueden usar las mediciones relacionadas con puncta como el tamaño, el número y la intensidad para evaluar la eficacia de un tratamiento.
El software de análisis de imágenes de alto contenido basado en la nube puede acomodar una gran variedad de salidas de datos de instrumentos, incluida la plataforma de imágenes de alto contenido. Mediante el uso de un servidor basado en la nube para el almacenamiento de imágenes y el análisis en línea, el usuario puede cargar sus datos desde el instrumento de imágenes o desde la unidad de red donde se almacenan los datos. La parte de análisis del protocolo se lleva a cabo dentro del entorno de software en la nube, y los datos se pueden exportar en una variedad de formatos de archivo para la visualización de datos posteriores.
El virus SARS-CoV-2 está compuesto por proteínas no estructurales y no estructurales que ayudan en su ensamblaje y replicación. El pico del SARS-CoV-2 tiene dos dominios llamados S1 y S2, con S1 que contiene el dominio de unión al receptor responsable de las interacciones hACE2 en la membrana plasmática9. También se ha encontrado que Spike interactúa con otras moléculas en la membrana plasmática que pueden actuar como co-receptores además de hACE210,11. A lo largo de la secuencia de proteínas espiga y particularmente en la interfaz S1/S2, hay sitios de escisión de la proteasa que permiten la fusión en la membrana después de la serina proteasa transmembrana 2 (TMPRSS2)12. Se han producido varias proteínas recombinantes de pico de SARS-CoV-2 a partir de dominios de unión a receptores individuales, a S1, S2, S1 con S2 y trímeros de pico enteros de múltiples proveedores comerciales para su uso en actividades de investigación13.
En este trabajo, la superficie de los QD se funcionalizó con trímeros de pico recombinantes que contienen una etiqueta de histidina (QD-Spike). Los QD producidos por la Sección de Nanomateriales Ópticos del Laboratorio de Investigación Naval contienen un núcleo de seleniuro de cadmio y una carcasa de sulfuro de zinc14,15. El zinc en la superficie QD coordina los residuos de histidina dentro de la proteína recombinante para formar un QD funcionalizado que se asemeja a una partícula viral del SARS-CoV-2 en forma y función. La generación de las nanopartículas y la conjugación de proteínas se describió previamente utilizando el dominio de unión al receptor conjugado QD15. Este método describe las preparaciones de cultivo celular, el tratamiento QD, la adquisición de imágenes y el protocolo de análisis de datos que pueden guiar a un investigador en el estudio de la actividad del pico del SARS-CoV-2 en el contexto fisiológico de una célula humana.
Protocolo
La línea celular HEK293T utilizada en este estudio es una línea celular inmortalizada. No se utilizaron sujetos humanos o animales en este estudio.
1. Cultivo y siembra celular
- Dentro de un gabinete de bioseguridad estéril, usando equipo de protección personal (incluidos guantes de laboratorio, bata de laboratorio y gafas de seguridad), prepare el medio de cultivo celular complementando el Medio Águila Modificada (DMEM) de Dulbecco con suero bovino fetal (FBS) al 10%, penicilina / estreptomicina (P / S) y 250 μg / ml G418.
- Para 500 mL de medios, agregue 443.75 mL de DMEM, 50 mL de FBS, 5 mL de P/S y 1.25 mL de G418.
- Filtre a través de un matraz de filtro de 0,2 μm y mantenga la esterilidad para evitar la contaminación bacteriana.
- Dentro de un gabinete de bioseguridad estéril, sembra el interior 60 pocillos de una placa de 96 pocillos negra, de fondo transparente, recubierta de poli-D-lisina con 20,000 células en 100 μL por pozo de medio de cultivo celular. Llene los 36 pocillos externos con 100 μL por pozo de solución salina tamponada con fosfato (PBS).
- Para contar las células, agregue 2 μL de naranja acridina y tinción de yoduro de propidio a 18 μL de suspensión celular.
- Cargue 10 μL de esta solución en un lado de una diapositiva de conteo de celdas.
- Repita los pasos 1.2.1. y 1.2.2. para cargar ambos lados de la diapositiva.
- Coloque la diapositiva en un contador de celdas automatizado.
- Seleccione el protocolo de conteo de fluorescencia y presione Count. Cuente ambos lados de la diapositiva y calcule el promedio de las dos densidades de células vivas.
- Para calcular el volumen de suspensión celular requerido, divida el número total de células necesarias por la densidad celular promedio.
- Inspeccione los pozos después de la siembra bajo un microscopio de luz para asegurarse de que se haya logrado la densidad y distribución adecuadas.
- Incubar la placa durante la noche en una incubadora humidificada a 37 °C con un 5% de CO2.
2. Tratamiento de células con QD-Spike
- Preparar albúmina sérica bovina (BSA) al 0,1% diluyéndola en medios de imagen.
- Para 10 ml de medios de imagen, agregue 130 μL de BSA al 7,5% y mezcle.
- En un reservorio o placa de ensayo de 12 pocillos, diluya el stock de 440 nM QD-Spike (SARS-CoV-2, Isolate USA-WA1/2020) a 20 nM utilizando BSA al 0,1% en medios de imagen.
- Para 1 pocillo de QD-Spike, agregue 2.27 μL de 440 nM QD-Spike a 47.73 μL de medios de imagen BSA al 0.1% para una concentración final de 20 nM.
- Para generar una dilución seriada de seis puntos 1:3 (por triplicado), agregue 14.26 μL de QD-Spike a 285.74 μL de medios de imagen BSA al 0.1% para hacer la concentración más alta de 20 nM. Agregue 100 μL de 20 nM QD-Spike a 200 μL de medios BSA al 0.1% para hacer la segunda dilución de 6.67 nM QD-Spike. Repita cuatro veces para generar los seis puntos de concentración.
- Usando un aspirador multicanal, retire todos los medios gastados de cada pozo. Con una pipeta multicanal, lavar una vez con soportes de imagen (100 μL/pocillo).
- Aspire 100 μL de medios de imagen y vuelva a agregar 50 μL/pocillo de solución QD-Spike.
- Incubar la placa durante 3 h en una incubadora humidificada a 37 °C con un 5% de CO2. Continúe con el paso 4.
3. Fijación y tinción de núcleos
- Dentro de un gabinete de bioseguridad estéril, usando equipo de protección personal (incluidos guantes de laboratorio, bata de laboratorio y gafas de seguridad), prepare paraformaldehído al 4% (PFA) en medios de imágenes BSA al 0.1%.
- Aspire 50 μL de QD-Spike de cada pozo y agregue 100 μL/pocillo de PFA al 4%.
- No deje que los pozos se sequen. Utilice una pipeta multicanal automatizada para evitar el secado de los pozos (recomendado).
- Incubar durante 15 min a temperatura ambiente.
- Lavar tres veces con 1 solución salina tamponada con fosfato (PBS).
- Prepare el colorante nuclear de color rojo intenso diluyendo la solución madre de 5 mM a una dilución de 1:1000 en PBS.
- Aspire el PBS y vuelva a agregar 50 μL/pocillo de colorante nuclear diluido.
- Incubar durante 30 min a temperatura ambiente.
- Lavar tres veces con PBS.
- Tome una imagen de la placa o el sello con un sellador de placas para obtener imágenes en una fecha posterior. Guarde la placa a 4 °C. La fluorescencia debe ser estable durante varias semanas o más.
4. Configuración de adquisición e imágenes
- Inicie el software para la plataforma de imágenes. Encienda la plataforma de imágenes de alto contenido y la luz de la barra de estado debería estar encendida. Si la máquina no está conectada, el software entrará en modo de análisis sin conexión.
- Inicie sesión en la plataforma de imágenes.
- Cree un nuevo protocolo de adquisición.
- Seleccione Tipo de placa como placa de imagen inferior transparente de 96 pocillos.
NOTA: La selección de la placa en el instrumento puede variar y se puede personalizar cargando las definiciones correctas de la placa que incluyen las dimensiones de la placa, como la altura, el ancho y otras distancias para definir el espaciado del pozo y los puntos focales. - Seleccione Modo óptico como confocal y aumento como inmersión en agua 40x.
- Seleccione Binning como 1.
- Elija los canales y las longitudes de onda fluorescentes de excitación/emisión como se describe en los pasos 4.3.5-4.3.8. Tome una instantánea cuando se seleccione la configuración para ver la imagen y asegúrese de que la configuración sea adecuada.
NOTA: El contraste de fase digital (DPC) permitirá la visualización de células vivas sin una mancha celular o tinte fluorescente. No se recomienda para células fijas. - Seleccione el modo para DPC, como Alto contraste, para producir cuerpos celulares bien definidos. Tome una instantánea para confirmar que esta configuración es adecuada, como se ve en la ventana de la imagen.
- Seleccione el canal FITC para su uso con la línea celular ACE2-GFP que permite la visualización del tráfico ACE2 dentro de la célula.
- Para crear un canal personalizado para el canal QD, seleccione el menú desplegable triangular para el canal y elija la excitación en el rango de 405 nm y la emisión en el rango de 608 nm.
- Si se fijaron las células y se utilizó un tinte nuclear de color rojo intenso, seleccione el canal rojo lejano con una emisión superior a 630 nm para delinear aún más el cuerpo celular y actuar como una máscara celular.
- Seleccione un pozo con una señal QD citoplasmática fuerte para establecer el tiempo de exposición, la potencia del láser y la posición de altura Z. Siga los pasos de 4.3.10-4.3.13.
- Compruebe si la altura predeterminada produce una imagen en la que las celdas se encuentran en el plano focal deseado. Si los puntos endocitosados son el orgánulo objetivo, la posición Z será menor que si la membrana plasmática es el orgánulo objetivo.
- Elija un tiempo de exposición que produzca una imagen brillante con niveles de gris al menos tres veces mayores que la señal de fondo. Esto es típicamente entre 100 y 300 ms. A 20 nM, los niveles de gris deben ser de aproximadamente 6000 u.a.u. utilizando un tiempo de exposición de 200 ms y una potencia láser del 80%.
- Compruebe los niveles de gris haciendo clic con el botón derecho en la imagen producida con la función Instantánea en cada canal y seleccionando Mostrar intensidad. Luego, haga clic izquierdo en el objeto de interés o fondo para ver el nivel de gris de ese píxel.
- Ajuste la potencia del láser para ajustar la intensidad de los objetos de interés.
- Seleccione Tipo de placa como placa de imagen inferior transparente de 96 pocillos.
- Guarde el protocolo de adquisición.
- Cambie a Ejecutar experimento e introduzca el nombre de la placa.
- Ejecute el experimento.
5. Análisis de datos
- Importe datos en el software de imágenes.
- Después de adquirir todas las imágenes, exporte las mediciones al software de imágenes. Esto requiere que se establezca un servidor y se cree una cuenta. Cree una pantalla para que los datos se exporten al software de imágenes.
- En el software de imágenes, seleccione Análisis de imágenes para comenzar a crear el protocolo de análisis.
- Cargue las mediciones del experimento de imágenes haciendo clic con el botón derecho en el archivo en la lista de pantallas y haciendo clic en Seleccionar. El mapa de placas de los pozos fotografiados y sus campos asociados deben ser visibles en la esquina inferior izquierda de la ventana.
- Elija un pozo con puntos QD citoplasmáticos brillantes para comenzar la segmentación de la imagen.
- En el bloque de creación de imagen de entrada, seleccione Corrección de campo plano.
- Agregue el siguiente bloque de creación al protocolo seleccionando Buscar núcleos.
- Aquí, use el DPC o el canal rojo lejano como el marcador Nuclei.
- Seleccione primero el método que segmenta con precisión los objetos y, a continuación, ajuste la segmentación mediante el menú desplegable y ajustando los controles deslizantes.
NOTA: Una buena segmentación define con precisión la región de interés (ROI) para el núcleo, la célula y las manchas. Solo los píxeles que son positivos para el marcador deben capturarse dentro de un ROI individual. Debe excluirse la señal de fondo. Si el fondo se captura en el ROI, se puede disminuir la sensibilidad del método o aumentar el umbral de intensidad para aumentar la rigurosidad del algoritmo de segmentación. Esto es aplicable a todos los bloques de creación de Buscar.
- A continuación, agregue el bloque de construcción Find Cytoplasm para identificar el citoplasma.
- En este caso, utilice el canal ACE2-GFP para la segmentación citoplasmática.
- Seleccione el método que mejor identifique el citoplasma de cada célula.
- A continuación, agregue el bloque de construcción Buscar puntos para identificar el punto QD-Spike.
- Seleccione los núcleos como la población de ROI.
- Seleccione la celda como región de ROI. Esto captura la puncta dentro de toda la célula.
- Seleccione el método que mejor identifique la puntuación QD en cada celda.
- Una vez que todos los objetos de la imagen se hayan segmentado e identificado con precisión en este pozo, elija otros pozos para garantizar que los bloques de construcción y la configuración se puedan aplicar generalmente a los otros pozos y otras condiciones.
- Agregue las propiedades de calcular intensidad para todos los objetos segmentados (núcleos, citoplasma/células y manchas).
- Agregue las propiedades de cálculo de morfología para todos los objetos segmentados (núcleos, citoplasma/células y manchas).
- Agregue la opción Calcular propiedades de textura para todos los objetos segmentados (núcleos, citoplasma/células y manchas).
- Defina los resultados seleccionando los parámetros para cada población, incluidos los núcleos y las manchas. El número de objetos se puede utilizar como una medida indirecta de la viabilidad celular.
6. Exportar datos
- Haga clic en Análisis por lotes. Espere a que finalice el análisis antes de continuar con el siguiente paso (paso 6.2).
NOTA: El análisis por lotes permite que el servidor de software de análisis de imágenes cargue el protocolo de análisis utilizando las mediciones seleccionadas para analizar los datos de forma remota. - Exporte el conjunto de datos a un equipo local o unidad de red.
- Conecte el software de análisis de imágenes a una aplicación auxiliar en el equipo descargando y abriendo un archivo de conexión.
- Seleccione el tipo de archivo de resultados (.txt, .csv, .html o XML nativo).
- Elija la configuración de la carpeta de archivos con el menú desplegable.
7. Analiza los datos en una hoja de cálculo
- Abra el archivo exportado. Si se trata de un archivo .csv, guárdelo como un formato de archivo .xls para habilitar el uso de tablas dinámicas. Si la ruta del archivo es demasiado larga, guarde el archivo .xls más arriba en la jerarquía de carpetas para evitar errores en el guardado.
- Agregue columnas a la hoja de cálculo que designen las condiciones para cada pozo. Por ejemplo, el tipo de célula, las variantes de QD-Spike, las concentraciones de QD-Spike, el tiempo de incubación, etc.
- Elija la función Tabla dinámica y cree la tabla utilizando las condiciones agregadas como filas y los parámetros medidos como columnas. Seleccione el cálculo para cada parámetro, es decir, Promedio, Desviación estándar, Mediana, Mín, Máx. o Recuento.
- Normalizar los datos para controlar las muestras, incluidos los QD no conjugados como 0% y la mayor concentración de QD-Spike como 100%.
NOTA: Si evalúa la eficacia de inhibidores como los anticuerpos neutralizantes, normalice los datos a células tratadas solo con medios (100% de eficacia) y QD-Spike sin inhibidor (0% de eficacia). - Trazar los datos en un software de gráficos.
Resultados
Tras el tratamiento, los QD se internalizarán ya que la nanopartícula se unirá a ACE2 en la membrana plasmática e inducirá endocitosis. Utilizando una línea celular que expresa ACE2-GFP, la translocación de QDs y ACE2 se puede visualizar utilizando microscopía de fluorescencia. Una vez internalizadas, las dos señales QD y ACE2 muestran una fuerte colocalización. A partir de estas imágenes, se puede realizar la segmentación de imágenes y el análisis posterior para extraer parámetros relevantes como el recuento de puntos (Figura 1, Figura 2B). La Figura 1A es un montaje de células tratadas con diferentes concentraciones de QD-Spike o medios solo como control. Se utilizaron tres canales, incluido el contraste de fase digital, FITC y el canal QD personalizado con excitación a 405 nm y emisión a 608 nm. DPC proporciona una imagen de la forma general de la celda. La imagen DPC proporciona una indicación aproximada de toda la morfología celular. El área de interés se superpone con la señal DPC y es suficiente para detectar QD internalizados. El canal FITC muestra la localización cambiante de ACE2-GFP y acumulándose en regiones que se colocalizan con QD-Spike. A medida que disminuye la concentración de QD-Spike, los QD ya no son visibles y la señal ACE2-GFP es similar al control. El canal de fusión demuestra la colocalización de ACE2-GFP con QD-Spike. Estos objetos son una mezcla de manchas y acumulaciones más grandes que se pueden segmentar con el software de análisis. El ajuste fino del método de análisis de software utilizado para la segmentación de imágenes se puede utilizar para segmentar los objetos de interés.
Aquí se realizó un experimento de concentración-respuesta con seis concentraciones diferentes de QD-Spike, a partir de 20 nM, para determinar las concentraciones óptimas de QD (Figura 2A). Los QD se pueden usar tan bajo como 2.22 nM y aún muestran enlace e internalización. Sin embargo, se recomienda utilizar concentraciones de 20 nM o más para garantizar una respuesta robusta.
Durante la conjugación con el QD, puede ocurrir la agregación de proteínas. El principal problema con los agregados es que se acumularán en la parte superior de las celdas y causarán artefactos en el paso de segmentación de la imagen. Los agregados no podrán entrar en las células, y las nanopartículas en su conjunto ya no se parecerán a una partícula viral (Figura 3). Los agregados se pueden detectar en la solución QD como precipitados brillantes y agrupados.
Este ensayo también se puede utilizar para evaluar productos biológicos, como anticuerpos neutralizantes que bloquean la entrada viral. Los QD se incubaron con anticuerpos neutralizantes (Figura 4A), a partir de 30 μg/ml, durante 30 min a temperatura ambiente antes de la adición a las células. Se utilizaron anticuerpos levantados contra la cepa de referencia de Washington, SARS-CoV-2 RBD. Bloquearon la unión, la internalización y causaron una reducción en el recuento de puntos medido en comparación con las células tratadas con QD solo (Figura 4B).
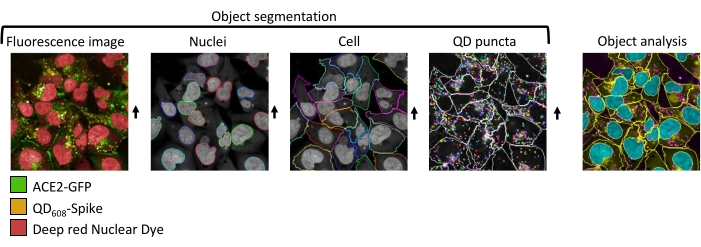
Figura 1: Análisis de imágenes de alto contenido y segmentación de células ACE2-GFP tratadas con QD608-Spike. Imágenes representativas de segmentación precisa. Primero, los núcleos se segmentan desde el canal que contiene el marcador nuclear para crear una población ROI. A partir de la población de ROI, una región de ROI que describe la célula se segmenta utilizando el canal ACE2-GFP. Por último, los puntos QD608-Spike están segmentados desde la región de ROI celular. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: QD608-Spike se une a hACE2 y se endocitosiza en células hACE2-GFP HEK293T. (A) Montaje de imágenes de células hACE2-GFP (amarillas) HEK293T tratadas con varias concentraciones de QD608-Spike (magenta) o solo medios. Se utilizó contraste de fase digital (cian) para visualizar el cuerpo celular. Barra de escala: 20 μm. (B) Análisis de alto contenido de QD608-Spike Spot Counts normalizado a 20 nM QD608-Spike (100%) y el control (0%). N = pozos triplicados. Las barras de error indican desviación estándar (S.D.). Haga clic aquí para ver una versión más grande de esta figura.
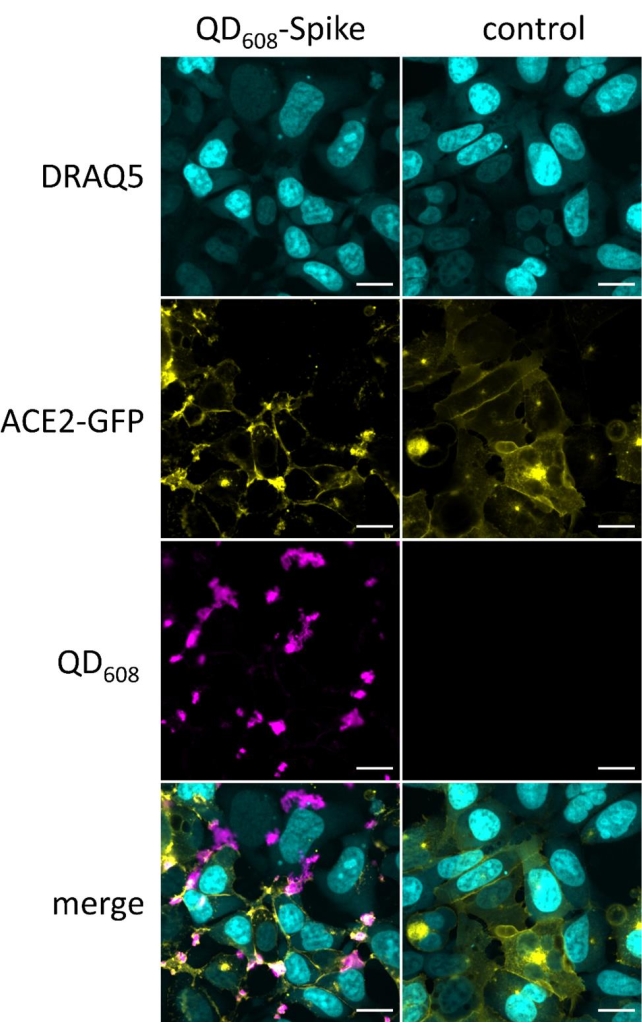
Figura 3: QD608-Spike agregado no se internaliza en células hACE2-GFP HEK293T. Montaje de imágenes de células hACE2-GFP HEK293T tratadas con 10 nM QD608-Spike o solo medios. Barra de escala: 20 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Anticuerpo neutralizante bloquea la endocitosis de QD608-Spike en células hACE2-GFP HEK293T. (A) Montaje de imagen de células hACE2-GFP (amarillas) HEK293T tratadas con QD608-Spike (magenta) preincutadas con concentraciones decrecientes de anticuerpos neutralizantes, utilizando contraste de fase digital (cian) para identificar cuerpos celulares. Barra de escala: 20 μm. (B) Análisis de alto contenido de QD608-Spike Spot Counts normalizado a control solo de medios (100%) y 10 nM QD608-Spike solamente (0%). N = pozos triplicados. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El método descrito en este artículo proporciona los pasos necesarios para obtener imágenes de QD funcionalizados en células humanas mediante microscopía confocal de alto rendimiento. Este método es más adecuado para las células donde la endocitosis es la principal vía de entrada viral en lugar de la actividad de TMPRSS2 y la fusión de membrana, ya que permite el estudio de la endocitosis del pico del SARS-CoV-2 y la endocitosis hACE2. Debido a la naturaleza del modelo QD y la etiqueta C-terminal His-tag en el trímero Spike disponible comercialmente, cualquier escisión TMPRSS2 de los dominios Spike S1 y S2 dejaría el QD conectado al dominio S2 solamente12. Esto puede evitar la internalización, dado que el RBD se encuentra en S1. Por lo tanto, si la secuencia de eventos en la superficie celular fuera precisa, donde hACE2 se une y luego TMPRSS2 escinde Spike, se espera una señal negativa sin internalización.
Como este protocolo se ocupa de los procesos celulares de imagen, las células cultivadas deben ser permisivas a la infección viral con la expresión de hACE2. hACE2 puede ser transfectado transitoriamente en células, o puede generarse una línea celular estable que exprese hACE217. Se recomienda verificar la expresión y localización de hACE2 mediante inmunofluorescencia con anticuerpos contra hACE2 a menos que el hACE2 tenga una etiqueta de proteína fluorescente como GFP que se pueda observar mediante microscopía. HACE2 marcado con GFP confiere el beneficio adicional de visualizar el tráfico de hACE2 después de la endocitosis QD-Spike. Se pueden utilizar algunas líneas celulares con expresión endógena de hACE2, pero esto debe confirmarse mediante inmunocitoquímica. En algunos casos, la expresión endógena no proporciona suficientes socios de unión a hACE2 para QD-Spike y puede dar lugar a una señal que se detecta mal mediante microscopía confocal de alto rendimiento.
Un paso crítico en el protocolo incluye la adquisición de QD de alta calidad conjugados con el pico recombinante de SARS-CoV-2 etiquetado con His. El requisito previo para esto es una proteína debidamente purificada que pueda conjugarse con los QD sin causar agregación. Los QD agregados tendrán varios problemas que impiden un experimento exitoso. Por lo tanto, se recomienda encarecidamente realizar pruebas de QD-Spike utilizando herramientas de análisis (por ejemplo, espectroscopía UV-visible, microscopía electrónica de transmisión o dispersión dinámica de luz) antes de la experimentación completa para preservar recursos valiosos si se prueban reactivos preciosos16. Durante el etiquetado de QDs con spike, las concentraciones específicas de QD y Spike se mezclan para lograr una proporción específica de moléculas. Solo QD y Spike se mezclan, y ambas soluciones están altamente purificadas. Para evaluar el etiquetado de los QD, se puede fundir un gel de acrilamida y cargarlo con QD solo, así como conyugal QD a Spike, lo que resulta en bandas de peso molecular más pesadas.
Los QD utilizados en este protocolo tienen un espectro de excitación/emisión de fluorescencia sintonizado con la emisión de 608 nm después de la excitación UV (≤405 nm) ya que la mayoría de los QD tienen una alta absorción cerca del rango UV produciendo una alta fotoluminiscencia18. Esta es una combinación de excitación / emisión no convencional que requiere que un microscopio tenga canales personalizables. Muchos microscopios confocales tradicionales se pueden configurar con los filtros y líneas láser adecuados para lograr esta excitación / emisión. Alternativamente, la excitación del QD en el primer máximo de absorción, aproximadamente a 10 a 20 nm del pico de emisión (por ejemplo, 592 nm para QD608 utilizado aquí), también podrá producir suficiente fotoluminiscencia.
El software basado en la nube utilizado en este protocolo de análisis de alto contenido utiliza un esquema de nomenclatura que se basa en pasos anteriores. Por ejemplo, los primeros objetos que se segmentan son núcleos, que crean una población llamada Núcleos. Después de esto, la célula o citoplasma se puede identificar como una región ROI dentro de los núcleos de población. La terminología utilizada en el software de análisis de imágenes establece el nombre de la población de objetos segmentados utilizando el bloque de construcción del núcleo como Núcleos. Sin embargo, no necesariamente tienen que ser núcleos y pueden ser cuerpos celulares si no hay un tinte nuclear disponible. Este esquema de nomenclatura también se puede cambiar y personalizar dentro de cada nombre de salida de bloque de creación.
Nuestro protocolo no tuvo en cuenta las interacciones de membrana, pero esto podría hacerse agregando una tinción de membrana adicional que sea independiente del tráfico ace2-GFP. Para bloquear la unión inespecífica, se incluye un 0,1% de BSA en los medios de ensayo (DMEM + 0,1% de BSA), y las células también se cultivan en un 10% de FBS. Se podían usar proteínas de espiga mutantes, pero estábamos limitados a las proteínas de espiga disponibles comercialmente. En este caso, no se disponía de una proteína Spike mutante no fijadora al SARS-CoV-2.
El método de nanopartículas QD presenta una poderosa tecnología para estudiar la unión e internalización de virus que dependen de la entrada mediada por Spike. Este ensayo se puede utilizar para la detección de alto rendimiento de manera análoga, como se muestra en la Figura 4, para reutilizar e identificar antivirales potentes que bloquean la entrada celular. Si bien este protocolo solo demostró el uso de QD conjugados con la cepa de referencia Washington WA-1 de SARS-CoV-2, este ensayo se puede adaptar fácilmente para estudiar las propiedades de unión de nuevas variantes emergentes como Alpha, Beta, Gamma y Delta mediante el uso de sus respectivas proteínas espiga conjugadas con QD.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Esta investigación fue apoyada en parte por el Programa de Investigación Intramuros del Centro Nacional para el Avance de las Ciencias Traslacionales, NIH. El Laboratorio de Investigación Naval proporcionó fondos a través de su Instituto de Nanociencia interno. La preparación de reactivos se apoyó a través del fondo base NRL COVID-19.
Materiales
| Name | Company | Catalog Number | Comments |
| 32% Paraformaldehyde | Electron Microscopy Sciences | 15714 | Used for fixing cells after quantum dot treatment, final concentration 3.2% Used for stabilizing QDs in Optimem I and preventing non-specific interactions, final concentration 0.1% |
| 7.5% Bovine Serum Albumin | Gibco | 15260-037 | Used as a cell viability dye for fluorescence cell counting |
| Acridine Orange / Propidium Iodide Stain | Logos Biosystems | F23001 | Microwell plates used for seeding cells and assaying QD-Spike |
| Black clear bottom 96 well coated plate coated with poly-D-lysine | Greiner | 655946 | Used to support cell culture, DMEM supplement |
| Characterized Fetal Bovine Serum | Cytiva/HyClone | SH30071.03 | Cloud-based high-content image analysis software; V2.9.1 |
| Columbus Analyzer | Perkin Elmer | NA | Used for labeling cell nuclei and cell bodies after fixation, deep red nuclear dye |
| DRAQ5 (5 mM) | ThermoFisher Scientific | 62252 | Basal media for HEK293T cell culture |
| Dulbecco's Minimal Essential Media, D-glucose (4.5g/L), L-glutamine, sodium pyruvate (110 mg/L), phenol red | Gibco | 11995-065 | Used for arranging data after export from Columbus; V2110 Microsoft 365 |
| Excel | Microsoft | NA | Used to continue selection of hACE2-GFP positive cells, DMEM supplement |
| G418 | InvivoGen | ant-gn-5 | Human embryonic kidney cell line stably expression human angiotensin converting enzyme 2 tagged with GFP |
| HEK293T hACE2-GFP | Codex Biosolutions | CB-97100-203 | Automated cell counter |
| Luna Automated Cell Counter | Logos Biosystems | NA | Used for fluorescence cell counting |
| Luna Cell Counting Slides | Logos Biosystems | L12001 | High-content imaging platform |
| Opera Phenix | Perkin Elmer | NA | Imaging media, used for incubating cells with quantum dots |
| Opti-MEM I Reduced Serum Medium | Gibco | 11058-021 | Phosphate-buffered saline without calcium or magnesium used for washing cells during passaging and assaying |
| PBS -/- | Gibco | 10010-023 | Used to prevent bacterial contamination of cell culture, DMEM supplement |
| Penicillin Streptomycin | Gibco | 15140-122 | Used for graphing, data visualization, and statistical analysis;V9.1.0 |
| Prism | GraphPad | NA | Used for assaying SARS-Cov-2 Spike binding to hACE2 and monitoring Spike endocytosis |
| Quantum Dot 608 nm-Spike (QD608-Spike) | custom made by Naval Research Laboratory | Used for inhibition of SARS-Cov-2 Spike binding to hACE2 | |
| SARS-CoV-2 (2019-nCoV) Spike Neutralizing Antibody, Mouse Mab | Sino Biological | 40592-MM57 | Used to dissociate cells from flask during passaging |
| TrypLE Express | Gibco | 12605-010 |
Referencias
- Chazotte, B. Labeling Lysosomes in Live Cells with LysoTracker. Cold Spring Harbor Protocols. 2011 (2), 5571(2011).
- Mehta, S., Zhang, J. Biochemical activity architectures visualized-using genetically encoded fluorescent biosensors to map the spatial boundaries of signaling compartments. Accounts of Chemical Research. 54 (10), 2409-2420 (2021).
- Barroso, M. M. Quantum dots in cell biology. The Journal of Histochemistry and Cytochemistry: Official Journal of the Histochemistry Society. 59 (3), 237-251 (2011).
- Cuervo, N. Z., Grandvaux, N. ACE2: Evidence of role as entry receptor for SARS-CoV-2 and implications in comorbidities. eLife. 9, 61390(2020).
- Shang, J., et al. Cell entry mechanisms of SARS-CoV-2. Proceedings of the National Academy of Sciences of the United States of America. 117 (21), 11727-11734 (2020).
- Ghosh, S., et al. β-Coronaviruses use lysosomes for egress instead of the biosynthetic secretory pathway. Cell. 183 (6), 1520-1535 (2020).
- Buchser, W., et al. Assay development guidelines for image-based high content screening, high content analysis and high content imaging. Assay Guidance Manual. Markossian, S., et al. , Eli Lilly & Company and the National Center for Advancing Translational Sciences. (2012).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews. Drug Discovery. 20 (2), 145-159 (2021).
- Huang, Y., Yang, C., Xu, X. F., Xu, W., Liu, S. W. Structural and functional properties of SARS-CoV-2 spike protein: potential antivirus drug development for COVID-19. Acta Pharmacologica Sinica. 41 (9), 1141-1149 (2020).
- Gao, C., et al. SARS-CoV-2 spike protein interacts with multiple innate immune receptors. bioRxiv: the preprint server for biology. , 227462(2020).
- Zhang, Q., et al. Heparan sulfate assists SARS-CoV-2 in cell entry and can be targeted by approved drugs in vitro. Cell Discovery. 6 (1), 80(2020).
- Hoffmann, M., et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and Is blocked by a clinically proven protease inhibitor. Cell. 181 (2), 271-280 (2020).
- Cai, Y., et al. Distinct conformational states of SARS-CoV-2 spike protein. Science. 369 (6511), New York, N.Y. 1586-1592 (2020).
- Oh, E., et al. Meta-analysis of cellular toxicity for cadmium-containing quantum dots. Nature Nanotechnology. 11 (5), 479-486 (2016).
- Gorshkov, K., et al. Quantum dot-conjugated SARS-CoV-2 spike pseudo-virions enable tracking of angiotensin Converting enzyme 2 binding and endocytosis. ACS Nano. 14 (9), 12234-12247 (2020).
- Narayanan, S. S., Pal, S. K. Aggregated CdS quantum dots: Host of biomolecular ligands. The Journal of Physical Chemistry B. 110 (48), 24403-24409 (2006).
- Wang, S., et al. Endocytosis of the receptor-binding domain of SARS-CoV spike protein together with virus receptor ACE2. Virus Research. 136 (1), 8-15 (2008).
- Hildebrandt, N., et al. Energy transfer with semiconductor quantum dot bioconjugates: A versatile platform for biosensing, energy harvesting, and other developing applications. Chemical Reviews. 117 (2), 536(2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados