Method Article
Fazendo medições de FRET de molécula única precisas e precisas usando o smfBox de código aberto
Neste Artigo
Resumo
Este artigo fornece instruções passo a passo para fazer medições de FRET precisas totalmente corrigidas em biomoléculas individuais, difundindo livremente biomoléculas usando o smfBox de código aberto e barato, desde ligar, através do alinhamento e foco, até a coleta e análise de dados.
Resumo
O smfBox é um instrumento de código aberto e econômico recentemente desenvolvido para transferência de energia de ressonância förster de molécula única (smFRET), que torna as medições em biomoléculas de difusão livremente mais acessíveis. Essa visão geral inclui um protocolo passo-a-passo para o uso deste instrumento para fazer medições de eficiências precisas de FRET em amostras de DNA duplex, incluindo detalhes da preparação e alinhamento da amostra, configuração e alinhamento de instrumentos, aquisição de dados e rotinas completas de análise. A abordagem apresentada, que inclui como determinar todos os fatores de correção necessários para medições precisas de distância derivadas do FRET, baseia-se em um grande conjunto de trabalhos colaborativos recentes em toda a Comunidade FRET, que visa estabelecer protocolos padrão e abordagens de análise. Este protocolo, que é facilmente adaptável a uma gama de sistemas biomoleculares, aumenta os esforços crescentes na democratização do SMFRET para a comunidade científica mais ampla.
Introdução
A transferência de energia de ressonância Förster de uma molécula única (smFRET) é uma técnica que mede a eficiência do FRET entre dois corantes - um doador e um aceitador -ao nível de moléculas individuais. FRET é um processo fotofísico decorrente do espectro de energia sobreposto de dois corantes: o doador está animado com a luz de um comprimento de onda específico e transfere energia sem radiação para o aceitor, resultando em emissão do aceitador. A eficiência dessa transferência é inversamente proporcional à sexta potência da distância entre os dois corantes, de modo que a eficiência de transferência varia de acordo com a distância1. Assim, essa eficiência de FRET pode ser usada para determinar informações espaciais sobre a molécula(s)2 à qual os corantes estão ligados, dentro de uma faixa de 3-10 nm. Essa escala, e o fato de que as mudanças na eficiência do FRET são sensíveis aos movimentos moleculares de Angstrom3, torna a técnica adequada para investigar informações estruturais sobre biomoléculas - como ácidos nucleicos e proteínas - sem as complicações do conjunto em média4,5,6. Embora mudanças na eficiência relativa do FRET possam ser usadas para monitorar interações biomoleculares e dinâmicas conformacionais, lançando luz sobre os principais processos celulares, como proteína (un)dobrável, transcrição e replicação e reparo de DNA, eficiências absolutas de FRET têm sido usadas para determinar distâncias precisas para determinação da estrutura biomolecular7,8,9,10,11 , superando a necessidade de cristalização ou congelamento, como é necessário para alguns outros métodos estruturais4,12.
os experimentos smFRET geralmente tomam duas formas, microscopia de fluorescência de reflexão interna confocal ou total (TIRF). Entre ambas as abordagens, a dinâmica molecular das biomoléculas pode ser tipicamente investigada em escalas de tempo do pico- a milissegundo (moléculas confocal, difusas livremente) até milissegundos a horas (TIRF, moléculas imobilizadas da superfície). Isso se deve às diferentes configurações envolvidas em cada técnica. Na microscopia TIRF, as moléculas são imobilizadas na superfície de um slide e excitadas por uma onda evanescente (Figura 1A). Aqui, no entanto, o foco é na microscopia confocal, pois este é o formato do smfBox. Na microscopia confocal, as moléculas não são imobilizadas e, em vez disso, livremente difusas através do movimento browniano através do volume confocal (~1 fL), formado pelo foco de um raio laser através de uma lente de abertura numérica alta em um ponto em alguma profundidade designada dentro da solução (Figura 1B). A emissão resultante é focada de volta através da mesma abertura e filtrada através de um espelho dicroico (Figura 1C para esquema completo). Em seguida, é focado através de um orifício para remover qualquer luz fora de foco e em um fotodiodo de avalanche (APD). Quando o APD detecta um fóton, ele produz um pulso TTL, o tempo do qual pode ser registrado com resolução de até picosegundo. O tempo de observação dessas moléculas de difusão livre nas proximidades do volume confocal é comumente dentro da ordem dos milissegundos.
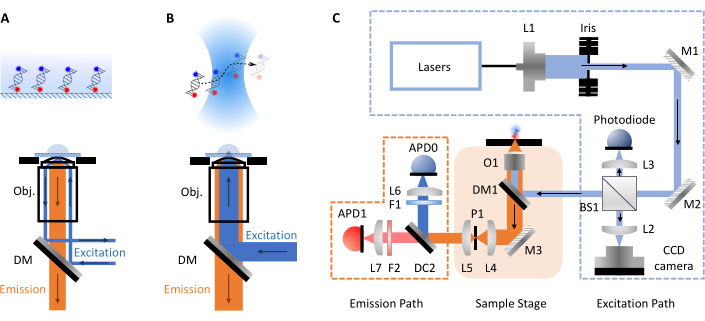
Figura 1: Esquemas que mostram princípios da microscopia e da configuração smfBox. (A) Princípio de Microscopia de Reflexão Interna Total (TIRF): a luz de excitação é direcionada para a borda do objetivo (Obj.) e sofre total reflexão interna na interface de tampão de tampa gerando um campo de evanescência exponencialmente decadente para excitar moléculas conectadas à superfície. (B) Microscopia Confocal: Moléculas de difusão livre são animadas por um ponto quase difração limitado focado na amostra. (C) A configuração smfBox utilizada neste protocolo, mostrando todos os componentes-chave: fotodiodos de avalanche (APD), divisor de feixe (BS), espelhosdicróicos (DM), filtros (F), espelhos (M), objetivo (O) e pinhole (P). Clique aqui para ver uma versão maior desta figura.
Mais recentemente, as técnicas smFRET incorporaram duas excitações de cores, onde lasers que correspondem ao doador e comprimentos de onda de excitação aceitadores são alternados5. Isso pode ser feito de uma das duas maneiras, a primeira modulando lasers de ondas contínuas na escala de tempo KHz, que é conhecida como excitação a laser alternada (ALEX)13,14. O segundo método intercala pulsos rápidos na escala de tempo MHz; este é nanossegundo-ALEX15 ou excitação intercalada pulsada (PIE)16. Em todas essas abordagens, a informação do laser aceitador leva ao cálculo da chamada estequiometria, que pode discriminar entre moléculas com baixa eficiência de FRET e aquelas que não possuem um aceitador (seja através de rotulagem incompleta ou fotobleaching). O uso do PIE/ns-ALEX também dá acesso a vidas fluorescentes no nível de molécula única, e as anisotropias podem ser medidas quando associadas à ótica polarizadora. Esta combinação de medidas é conhecida como detecção de fluorescência multiparmétrica (MFD)9.
Apesar das muitas vantagens da SMFRET, ela não é amplamente utilizada fora de laboratórios especializados devido aos altos custos de instrumentos comerciais e à falta de alternativas simples e auto-construídas. Uma tendência crescente para o desenvolvimento de microscopia de origem aberta de baixo custo está ocorrendo e outras plataformas surgiram recentemente, incluindo Plânctonscope17, Microscópio OpenFlexure18, Flexiscope19, miCube20, liteTIRF21 e Squid22. Aqui, o estudo descreve o protocolo para o uso do smfBox, uma configuração confocal recentemente desenvolvida e econômica capaz de medir a eficiência do FRET entre dois corantes em moléculas únicas de difusão livre. Instruções detalhadas de construção e todos os softwares operacionais necessários estão disponíveis gratuitamente em: https://craggslab.github.io/smfBox/ 23. O arranjo óptico do smfBox é montado a partir de componentes prontamente disponíveis comprados de fabricantes acessíveis e amplamente acessíveis, enquanto o corpo do microscópio (responsável pela maior parte da despesa em uma configuração confocal padrão) foi substituído por uma caixa de alumínio anodizado sob medida (permitindo que as medidas sejam feitas sob condições de luz ambiente). Esta caixa abriga componentes ópticos chave, incluindo odicróico de excitação, objetivo e pinalho, e um intertravamento de laser mecânico, permitindo sua operação segura como um produto laser classe I (ver Figura 1C para um esquema completo). O smfBox usa ALEX para validar a estequiometria de corante e determinar fatores precisos de correção de FRET. É operado usando software de código aberto personalizado (smOTTER), que controla todos os aspectos da aquisição de dados e produz os dados no formato fóton-HDF5 de código aberto24, compatível com muitas ferramentas de análise de terceiros. O smfBox e os protocolos de aquisição e análise de dados foram recentemente testados contra >20 outros instrumentos (tanto confocal quanto TIRF) em um estudo cego multi-laboratório25. As eficiências da FRET obtidas estavam em excelente concordância com todos os outros instrumentos, apesar do smfBox custar apenas uma fração do preço das configurações disponíveis comercialmente.
Aqui, um protocolo passo-a-passo é delineado para a aquisição e análise de eficiências irissivas e absolutas precisas sobre a difusão livre de duplexes de DNA usando o smfBox, desde o desligamento, passando pelo alinhamento e foco, até a coleta e análise de dados. As amostras utilizadas aqui são três DNAs duplex (exibindo eficiências de alto, médio e baixo FRET, ver Tabela 1) que foram avaliadas no estudo cego mundial25; no entanto, o método é adaptável a muitos sistemas moleculares, incluindo proteínas e outros ácidos nucleicos. A esperança é que um protocolo tão detalhado, juntamente com as instruções de construção já existentes para o smfBox23, ajude a tornar essa técnica poderosa ainda mais acessível a uma ampla gama de laboratórios.
Protocolo
1. Componentes de alimentação
- Potência nos seis plugues (sem ordem específica): Piezoconcept (z-focus), APD0, APD1, laser verde, laser vermelho e fotodetetor.
2. Configuração de software 1-Experimental
- Lance o centro de controle a laser.
- Certifique-se de onda contínua - O modo CW-ACC (Alternating Constant Current, corrente constante alternada) está selecionado.
- Ligue para os lasers.
- Verifique/defina os poderes de laser.
NOTA: A potência do laser precisará ser ajustada conforme medido pouco antes dodicróico de excitação, pois os filtros ND e divisores de feixe no caminho de excitação reduzirão a potência do laser do número dado no painel de controle a laser. Os números aqui dados são de potência nodicróico de excitação, mas a potência do controle de laser que corresponde a isso precisará ser trabalhada.- Laser verde de 220 μW (515 nm, 40 mW)
- Μ70 W para laser vermelho (638 nm, 10 mW)
- software de aquisição smOTTER
- Conecte os lasers, detectores, z-stage e câmera. Configure-os corretamente (pode variar dependendo das configurações da configuração específica do cartão NI).
3. Alinhamento do caminho de emissão (não rotineiramente necessário)
- Configuração do alinhamento
- Pipeta 10 μL de corante Cy3B gratuito (~100 nM) no microscópio e foco conforme descrito nas etapas 4.3-4.5 abaixo.
NOTA: Outro corante doador com algum vazamento no canal de aceitação também funcionaria. - Abra a guia Alinhamento no smOTTER, baixe a potência do laser e altere a escala do eixo y até que a leitura seja vista nos detectores.
NOTA: O objetivo aqui é aumentar o sinal, de modo que a escala pode precisar ser alterada novamente após o sinal aumentar. - Desaparafusar os quatro parafusos na frente do smfBox e remover o painel frontal.
- Pipeta 10 μL de corante Cy3B gratuito (~100 nM) no microscópio e foco conforme descrito nas etapas 4.3-4.5 abaixo.
- Alinhamento pinhole
- Gire o botão x no posicionador do orifício enquanto observa o sinal na guia Alinhamento, tentando aumentar o sinal em verde e vermelho.
- Agora gire o botão y para alinhar o orifício na outra direção.
- Volte para o botão x para verificar se há mais aumento no sinal.
- Alinhamento da lente pinhole
- Gire a lente x botão em uma direção, isso diminuirá o sinal.
- Gire o botão pinhole x na mesma direção para aumentar o sinal novamente.
- Se o novo sinal máximo for mais alto do que antes, continue a mover iterativamente tanto o orifício quanto a lente nessa direção. Se for menor do que antes, se mova iterativamente na direção oposta.
- Repita acima para y.
- Alinhamento da lente APD
- Começando com o APD verde, mova o botão x até que o sinal verde esteja no máximo.
- Repita para o botão y.
- Volte para o botão x, mova-se para frente e para trás para encontrar os pontos limiares onde o sinal começa a cair, e deixe-o em uma posição no meio do caminho entre esses dois pontos.
- Repita para o botão x.
- Repita os passos acima para a lente APD vermelha, observando o sinal vermelho.
- Coloque o painel frontal de volta no smfBox e substitua os parafusos.
NOTA: Realize o mensal de alinhamento acima e, como medida de precaução, se suspeita-se que o microscópio tenha desenvolvido uma falha de alinhamento entre as medições.
4. Aquisição de dados de medição
- Para a primeira amostra, limpe o estágio com tecido de limpeza de lentes embebido em metanol; limpar suavemente através do objetivo de uma extremidade para a outra.
- Aplicar 3-4 gotas de óleo de imersão para microscopia no centro do objetivo; repor conforme necessário entre as amostras.
- Preparação da amostra
- Pipeta 10 μL de amostra (primeiro uso de água ultrapura tipo I) no centro de um copo de cobertura limpa.
- Coloque cuidadosamente o vidro de cobertura sobre a lente objetiva baixando-o em um ângulo para o óleo para evitar a captura de bolhas de ar entre o vidro de cobertura e o objetivo.
NOTA: Se necessário, deslize o vidro para empurrar as bolhas de ar no óleo para longe do ponto focal.
- Para realizar experimentos de excitação a laser alternada (ALEX) configuram o laser no painel de ciclos de serviço a laser da seguinte forma.
- Doador (laser verde) 0 off, 45 on, 55 off
- Acceptor (laser vermelho) 50 off, 45 on, 5 off
- Período ALEX (μs): 100
- Clique na guia foco Z na fita; no painel de aquisição, troque os lasers para Live e inicie a câmera; ajustar a exposição para que um ponto brilhante apareça centralmente cercado por preto.
NOTA: A câmera captura a luz refletida pela interface de vidro/água entre o deslizamento de tampas e a amostra e o círculo visível de luz está em seu menor diâmetro quando o ponto focal está nesta interface. Portanto, é necessário começar abaixo desta interface, aumentar a altura z para a interface e, em seguida, um pouco mais para se concentrar acima do deslizamento de tampas e na amostra que está sendo medida.- A partir de uma posição Z baixa, aumente a altura até que o ponto brilhante atinja seu tamanho mínimo, em seguida, eleve a altura mais em até 20 μm para focar o laser acima do óleo e cobrir o vidro e na amostra.
- Pare a câmera uma vez em foco.
- Para confirmar que a configuração foi realizada adequadamente, monitore o traço da água ultrauso tipo I para não ver sinais de fluorescência; confirmação repetindo a pureza para os buffers em que as amostras são preparadas.
- Remova cuidadosamente o vidro de cobertura com pinças de borracha para evitar o objetivo prejudicial e, em seguida, coloque a amostra de interesse preparada acima sobre o objetivo. Reponha o óleo de imersão conforme necessário para manter uma área uniforme de contato entre o vidro de cobertura e o objetivo (atenção especial dada à área de contato da amostra no vidro de cobertura e área focal do objetivo)
- Bingo de concentração
- Para garantir a obtenção de dados de molécula única, as amostras precisam estar em uma concentração na qual 1-5 rajadas por segundo são observadas no painel de rastreamento vivo do smOTTER (isso minimiza a chance de mais de uma molécula estar presente no volume de detecção de uma só vez). Faça as medidas quando a concentração apropriada tiver sido determinada.
- Para evitar a evaporação durante longos experimentos, prepare uma câmara de amostra hermética. Pressione os isoladores de silicone (orifício de 8-9 mm de diâmetro) para baixo no centro de um vidro de cobertura (colorido com uma ponta de pipeta). Em seguida, cuidadosamente pipeta uma amostra para o centro, evitando qualquer contato com o silicone. Em seguida, coloque um segundo copo de cobertura em cima e pressione para formar um selo.
- Verifique o histograma de stoquiometria ao vivo vs ES para ver se a amostra está se comportando como esperado, ou seja, a eficiência esperada do FRET e a estequiometria razoável (~0,5).
- Quando a amostra estiver pronta, insira os detalhes do experimento no painel de configurações de salvamento. Escolha um diretório e nome de arquivo apropriados clicando no ícone de elipse (arquivos salvos no formato hdf5/h5) no painel de configurações de salvamento.
- Insira informações para nome da amostra, detalhes da amostra, rótulos de doadores e aceitadores, comprimentos de onda de buffer, doadores e aceitadores, comprimentos de onda de detecção e poder laser e afiliação de usuários e usuários.
- Retorne à guia de rastreamento ao vivo na fita e, no painel de aquisição, digite o comprimento do experimento (em minutos), selecione um intervalo de salvamento apropriado para mitigar a perda de dados potencial em caso de erro. Selecione Salvar poderes laser, se necessário.
- Pressione Comece a pegar os dados.
NOTA: Para permitir a determinação precisa do FRET, pelo menos duas amostras precisarão ser medidas contendo o mesmo doador e corantes aceitadores, mas com eficiências fret suficientemente diferentes.
5. Análise/software 2
- Inicie o notebook Jupyter com o pacote PYTHON FRETBursts (instruções de configuração podem ser encontradas aqui: https://craggslab.github.io/smfBox/anasoftware.html)
- Se a correção para a eficiência absoluta do FRET não for necessária (ou seja, apenas alterações relativas no FRET são suficientes para a medição) inicie a análise DO caderno DE PRODUTOS 1.4 Uncorrected.ipynb. Use isso para exportar números ou dados como arquivos csv para análise suplementar ou plotagem em outros softwares.
NOTA: Cada caderno contém instruções detalhadas para orientar o usuário através dos procedimentos de análise. Para uma discussão mais detalhada dos procedimentos de análise, incluindo algoritmos de pesquisa de explosão, correção de fundo e todos os parâmetros de correção, veja23,25. - Se for necessária a correção para a eficiência absoluta do FRET, comece pelo cálculo dos parâmetros crosstalk alfa (vazamento de emissão de doadores no canal aceitor) e delta (proporção de excitação direta do aceitador sob excitação de doadores).
- Primeiro inicie o notebook Jupyter Correction Factor Finder Alpha-Delta.ipynb e determine os parâmetros alfa e delta.
NOTA: Se estes são inconsistentes entre as amostras, o microscópio pode ter desenvolvido um problema de alinhamento entre as medidas ou o espectro dos corantes difere entre as amostras. - Se os parâmetros alfa e delta forem consistentes, inicie o fator de correção do notebook Jupyter Gamma-Beta.ipynb para determinar os parâmetros gama e beta (que explicam as diferenças na eficiência de excitação e detecção entre os corantes).
NOTA: Se o enredo gama e beta não se encaixar bem, o microscópio pode ter desenvolvido um problema de alinhamento entre as medições, ou os rendimentos quânticos, ou os coeficientes de extinção dos corantes diferem entre as amostras. - Com os quatro parâmetros de crosstalk determinados, esses fatores podem ser usados no caderno FRET Analysis 1.4 Corrected.ipynb Jupyter para determinar a eficiência absoluta do FRET.
- Primeiro inicie o notebook Jupyter Correction Factor Finder Alpha-Delta.ipynb e determine os parâmetros alfa e delta.
6. Solução de problemas
- Se todos os sinais forem baixos ou as contagens por explosão forem inferiores ao esperado (isto é dependente de corante-mas para ATTO-550 e ATTO-647N no smfBox os valores típicos estão entre 50 e 100 contagens por ms durante uma explosão de uma única molécula), realinhar o smfBox.
- Ponte entre populações duplamente e singly rotuladas no histograma ES.
NOTA: Isso pode ser causado por trabalhar em uma concentração muito alta (remediar isso diluindo a amostra), ou por fotobleaching, que pode ser causado pelo uso muito alto de uma potência laser (remediar isso reduzindo a potência do laser). - Se a taxa de estouro cair ao longo do experimento (moléculas fluorescentes rotuladas provavelmente estão aderindo ao deslizamento de tampa de vidro), use uma concentração aumentada de BSA para corrigir.
- Se a taxa de estouro aumentar ao longo do experimento (a amostra provavelmente está evaporando), prepare uma câmara de amostra apertada a ar como descrito acima.
- Se o sinal falhar para zero durante o alinhamento (o software provavelmente está sendo sobrecarregado pelo sinal dos detectores), baixe a potência do laser ou use uma amostra de alinhamento mais diluída.
- Se o foco Z mostrar anéis concêntricos (pode acontecer se várias manchas de cobertura tiverem sido colocadas no objetivo), verifique se há várias falhas no objetivo.
- Se a amostra contiver rajadas longas e brilhantes de estequiometrias intermediárias (causadas por moléculas agregadas), use detergentes ou modifique protocolos de purificação de amostras.
NOTA: Obter um tampão limpo pode ser um problema, pois alguns componentes tampão muitas vezes contêm quantidades muito pequenas de contaminantes moderadamente fluorescentes que são suficientes para apresentar como rajadas de cor única no traço de tempo. Se há muito disso no buffer, então ele pode coincidir com rajadas de amostra e alterar a eficiência do FRET ou a estequiometria sendo medida. A BSA, em particular, pode muitas vezes ser problemática a esse respeito, por isso é útil expor uma solução BSA de estoque a uma fonte de luz forte para fotobleach os contaminantes.
Resultados
O protocolo requer avaliação crítica das condições experimentais durante a configuração (ver protocolo passo 4.8). Os primeiros resultados adquiridos que determinam o sucesso ou o fracasso do experimento são alcançados nesta fase. Um resultado positivo seria ter entre cinco e uma rajadas por segundo (ver Figura 2B,C). Um resultado negativo seria ter muitas (Figura 2A) ou poucas rajadas (Figura 2D) dentro desse período de tempo. Continua sendo possível nesta fase corrigir esses erros: uma amostra com uma concentração muito alta precisa simplesmente ser diluída; se a concentração for muito baixa, no entanto, uma nova amostra pode precisar ser preparada (o determinante é se é possível nesta baixa concentração coletar dados em um período de tempo razoável).

Figura 2: Capturas de tela de rastreamento ao vivo durante a configuração experimental mostrando diferentes concentrações de amostras duplamente rotuladas de DNA duplex. (A) muito alto, (B) limite superior aceitável, (C) concentração de alvo, (D) muito baixa. As contagens de fótons (1 ms bins) são mostradas nos três canais de detecção; emissão de doadores após excitação de doadores (DD), emissão de aceitação após excitação de doadores (DA) e emissão aceitadora após excitação aceitadora (AA). Clique aqui para ver uma versão maior desta figura.
Um sistema estático de espécie única normalmente exigiria 30 a 60 minutos de medição para obter as explosões necessárias de ~1.000 necessárias para uma análise robusta de dados. O tempo e o número de rajadas necessárias aumentarão com várias espécies ou sistemas dinâmicos. Após a coleta e análise de dados utilizando os números do protocolo são exportados dos cadernos Jupyter. O enredo de alternância (Figura 3A) deve coincidir com o período ALEX da configuração experimental. O traço de tempo (Figura 3B) é usado para avaliar qualitativamente que a concentração amostral é razoável. O gráfico de fundo (Figura 3C) mostra a distribuição de períodos de atraso intertona com um ajuste linear para os tempos mais longos para estimar a taxa de fundo26. O traço de fundo (Figura 3D) pode identificar se houve alterações na amostra durante a duração do experimento; principalmente isso seria devido à evaporação durante tempos de aquisição mais longos. Histogramas ES são gerados para todos os fótons (Figura 3E) e espécies duplamente rotuladas (Figura 3F). Finalmente, um histograma 1D E (Figura 3G) é gerado com encaixe gaussiano dos dados de explosão.

Figura 3: Saída de exemplo de dados analisados gerados pelos Cadernos Jupyter. (A) Gráfico de alternação, (B) Traço de tempo, (C) Determinação de fundo, (D) Taxas de fundo, (E) Todos os histogramas ES de fótons, (F) Dual channel ES histograma e (G) 1D E histograma. Clique aqui para ver uma versão maior desta figura.
| Nome | Seqüenciar |
| 1a | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC AAC AAC AAA CTC ACA AAC AGA CC - 5' | |
| 1b | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC AAC AAC AAA CTC ACA AAC AGA CC - 5' | |
| 1c | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC AAC AAC AAA CTC ACA AAC AGA CC - 5' |
Tabela 1: sequências de DNA usadas no protocolo. Os nucleotídeos são destacados em azul e vermelho representando resíduos de timina modificada C2 rotulados com Atto-550 e Atto-647N, respectivamente.
Fator de correção Localizador Alpha-Delta: Clique aqui para baixar este Arquivo.
Fator de correção Localizador Gamma-Beta: Clique aqui para baixar este Arquivo.
Análise FRET 1.4 Corrigido: Clique aqui para baixar este Arquivo.
Análise FRET 1.4 Não corrigida: Clique aqui para baixar este Arquivo.
Discussão
Os passos mais críticos do protocolo são o alinhamento do microscópio e o ajuste da concentração amostral à diluição correta. Se o alinhamento estiver desligado, então pode haver sinal insuficiente para identificar rajadas e traçar histogramas, e se ocorrer desalinhamento entre as amostras, a correção precisa do FRET pode falhar devido a alterações na eficiência de vazamento e detecção/excitação. O uso de uma concentração apropriada também é importante, uma concentração muito alta dará rajadas coincidentes, contendo múltiplas moléculas com eficiências de FRET potencialmente diferentes ou rotulagem de estequiometrias. Uma concentração muito baixa dará poucas rajadas para análise robusta de dados.
O protocolo descrito aqui é para medir distâncias em espécies estáticas de fret único. Se a amostra tiver mais de um pico no histograma de eficiência FRET, ou os picos parecerem largos (o que pode acontecer com espécies dinâmicas), então mais rajadas podem ser necessárias para encaixar histogramas no mesmo grau de precisão. Para dois picos bem separados, aproximadamente o dobro de dados será necessário, mas se as populações se sobreporem ligeiramente, então ainda mais dados são necessários.
Se as duas populações se interconvertem na escala de tempo do experimento, a dinâmica e cinética do sistema podem ser potencialmente determinadas. Testes como BVA27 e 2CDE28 podem confirmar que as rajadas intermediárias são de natureza dinâmica, enquanto análises incluindo dPDA29,30 ou H2MM31 podem determinar as taxas de interconversão. Os notebooks Jupyter para BVA e 2CDE estão disponíveis no site da FRETBursts26, e o software PAM32, baseado em MATLab, pode executar análises BVA, 2CDE e PDA.
O FRET de molécula única confocal pode facilmente observar estados muito mais curtos (~1 ms) do que o TIRF; no entanto, os curtos tempos de observação, limitados pela difusão, não dão história molecular, e por isso não podem determinar tempos de moradia mais longos, ou redes de transição complexas da maneira que experimentos imobilizados pela superfície podem.
Como o protocolo mede a difusão livre de moléculas em uma concentração muito baixa, ele funciona melhor ao medir distâncias intramoleculares na mesma molécula. Distâncias intermoleculares entre moléculas transitoriamente ligadas podem ser medidas desde que o Kd das duas moléculas seja baixo o suficiente para que o complexo exista em uma quantidade significativa na baixa concentração de trabalho exigida pelo experimento (~100 pM). Se o Kd for muito maior do que isso, então apenas moléculas isoladas rotuladas serão vistas. Esse problema pode ser superado usando microfluidos para misturar os dois componentes rotulados em alta concentração e, em seguida, diluindo e fluindo rapidamente sobre o objetivo antes do complexo dissociar33,34.
Medir a eficiência do FRET no nível de molécula única tem uma vantagem significativa sobre as técnicas de conjunto, pois informa sobre subpopulações heterogêneas, que em um experimento de conjunto seria mediada. Além disso, fret de molécula única com ALEX dá acesso a eficiências precisas de FRET, que podem ser convertidas em distâncias precisas. Isso permite determinar informações estruturais mais detalhadas em vez de simplesmente sondar mudanças relativas de distância. O smfBox carrega todos esses benefícios e capacidades, mas pode ser construído com um orçamento muito menor do que microscópios comercialmente disponíveis comparáveis capazes de confocal smFRET23.
O smfBox representa uma barreira muito menor para a entrada de técnicas de smFRET, permitindo aos pesquisadores medir mudanças conformais e distâncias precisas dentro e entre proteínas e ácidos nucleicos7,8,9,10,11,35.
Divulgações
Os autores não declaram interesses concorrentes.
Agradecimentos
Os autores reconhecem com gratidão as seguintes fontes de financiamento: BBSRC (BB/T008032/1); EPSRC (Studentship to B.A.) e MRC (Studentship to A. R.-T.).
Materiais
| Name | Company | Catalog Number | Comments |
| Amino modified oligonucleotide | Eurogentec | N/A | May be ordered from various suppliers or synthesised; amino modification enables labeling with NHS-ester modified dyes |
| Avalanche photodiode (APD) | Excelitas | SPCM-AQRH-14 | Two APDs are required for the smfBox setup |
| Bovine Serum Albumin (BSA) | Merck | A2153 | System dependant; imaging buffer component (0.1 mg/mL in buffer) |
| Compact Laser Combiner | OMICRON | LightHUB-2 | 515 nm (80 mW) and 638 nm (100 mW) lasers |
| Coverglass | VWR | 630-2742 | Thickness: 0.17 ± 0.01 mm, LxW: 22x22 mm |
| Cy3B | Cytiva | PA63101 | 1 mg, PA63100 (5 mg), PA96106 (25 mg) |
| FRETBursts Python Package | N/A | N/A | Open-source python package for burst analysis of freely-diffusing single-molecule FRET data: https://fretbursts.readthedocs.io |
| Imaging Buffer | N/A | System dependant; 5 mM NaCl, 20 mM MgCl2, 5 mM Tris pH 7.5 and 0.1 mg/mL BSA | |
| Immersion Oil | Olympus | IMMOIL-F30CC | |
| Jupyter notebooks | Project Jupyter | N/A | Open-source web application to create and share documents that contain live code, equations, visualizations and text; data analysis notebooks for smfBox can be found in the SI |
| Lens Tissue | ThorLabs | MC-5 | MC-50E is same item in bulk |
| Magnesium Chloride | Merck | M2670 | System dependant; imaging buffer component (20 mM in buffer) |
| MilliQ/Ultrapure water | N/A | ||
| Nanopoistioner | Piezoconcept | FOC300 | Nanopositioner for accurate positioning of microscope objective |
| NHS-ester modified ATTO-550 | ATTO-TEC | AD 550-31 | 1 mg, AD 550-35 (5 mg) |
| NHS-ester modified ATTO-647N | ATTO-TEC | AD 647N-31 | 1 mg, AD 647N-35 (5 mg) |
| Objective lens | Olympus | N1480700 | Olympus objective series from orignal smfBox discontinued; replaced by N5702300 |
| OMICRON Control Center (OCC)- laser control center | OMICRON | N/A | v3.5.34 - OMICRON laser driver software |
| Press-To-Seal silicone isolator | Grace Bio-Labs | 664201 | 8-9 mm Diameter x 1.7 mm Depth |
| smOTTER | N/A | N/A | Open-source acquisition software for the Craggs Lab smfBox: https://github.com/craggslab/smOTTER |
| Sodium Chloride | Merck | S7653 | System dependant; imaging buffer component (5 mM in buffer) |
| Tris base | Merck | 93362 | System dependant; imaging buffer component (5 mM, pH 7.5 in buffer) |
| Type I ultrapure water | Merck | ZIQ7000T0 | Milli-Q® IQ 7000 Ultrapure Water System |
Referências
- Forster, T. Intermolecular energy migration and fluorescence. Annals of Physics. 437 (1-2), 55-75 (1948).
- Stryer, L., Haugland, R. P. Energy transfer: a spectroscopic ruler. Proceedings of the National Academy of Sciences. 58 (2), 719-726 (1967).
- Hohlbein, J., et al. Conformational landscapes of DNA polymerase I and mutator derivatives establish fidelity checkpoints for nucleotide insertion. Nature Communications. 4 (1), 2131 (2013).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), (2018).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chemical Society Reviews. 43 (4), 1156-1171 (2014).
- Lerner, E., et al. The FRET-based structural dynamics challenge -- community contributions to consistent and open science practices. arXiv. , (2020).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nature Methods. 14 (2), 174-180 (2017).
- Craggs, T. D., et al. Substrate conformational dynamics facilitate structure-specific recognition of gapped DNA by DNA polymerase. Nucleic Acids Research. 47 (20), 10788-10800 (2019).
- Tsytlonok, M., et al. Dynamic anticipation by Cdk2/Cyclin A-bound p27 mediates signal integration in cell cycle regulation. Nature Communications. 10 (1), 1676 (2019).
- Nagy, J., et al. Complete architecture of the archaeal RNA polymerase open complex from single-molecule FRET and NPS. Nature Communications. 6 (1), 6161 (2015).
- LeBlanc, S. J., et al. Coordinated protein and DNA conformational changes govern mismatch repair initiation by MutS. Nucleic Acids Research. 46 (20), 10782-10795 (2018).
- Segal, M., et al. High-throughput smFRET analysis of freely diffusing nucleic acid molecules and associated proteins. Methods. 169, 21-45 (2019).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: Analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences. 101 (24), 8936-8941 (2004).
- Kapanidis, A. N., et al. Alternating-laser excitation of single molecules. Accounts of Chemical Research. 38 (7), 523-533 (2005).
- Müller, B. K., Zaychikov, E., Brauchle, C., Lamb, D. C. Pulsed interleaved excitation. Biophysical Journal. 89 (5), 3508-3522 (2005).
- Laurence, T. A., Kong, X., Jager, M., Weiss, S. Probing structural heterogeneities and fluctuations of nucleic acids and denatured proteins. Proceedings of the National Academy of Sciences of the United States of America. 102 (48), 17348-17353 (2005).
- Pollina, T., et al. PlanktonScope: Affordable modular imaging platform for citizen oceanography. bioRxiv. , 056978 (2020).
- Collins, J. T., et al. Robotic microscopy for everyone: the OpenFlexure microscope. Biomedical Optics Express. 11 (5), 2447-2460 (2020).
- Courtney, A., Alvey, L. M., Merces, G. O. T., Burke, N., Pickering, M. The Flexiscope: a low cost, flexible, convertible and modular microscope with automated scanning and micromanipulation. Royal Society Open Science. 7 (3), 191949 (2020).
- Martens, K. J. A., et al. Visualisation of dCas9 target search in vivo using an open-microscopy framework. Nature Communications. 10 (1), 3552 (2019).
- Auer, A., et al. Nanometer-scale multiplexed super-resolution imaging with an economic 3D-DNA-PAINT microscope. ChemPhysChem. 19 (22), 3024-3034 (2018).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , 424613 (2020).
- Ambrose, B., et al. The smfBox is an open-source platform for single-molecule FRET. Nature Communications. 11 (1), 5641 (2020).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An open file format for timestamp-based single-molecule fluorescence experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-a multi-laboratory benchmark study. Nature Methods. 15 (9), 669-676 (2018).
- Ingargiola, A., Lerner, E., Chung, S., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing single-molecule FRET. PLOS One. 11 (8), 0160716 (2016).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule FRET experiments with burst variance analysis. Biophysical Journal. 100 (6), 1568-1577 (2011).
- Tomov, T. E., et al. Disentangling subpopulations in single-molecule FRET and ALEX experiments with photon distribution analysis. Biophysical Journal. 102 (5), 1163-1173 (2012).
- Santoso, Y., Torella, J. P., Kapanidis, A. N. Characterizing single-molecule FRET dynamics with probability distribution analysis. ChemPhysChem. 11 (10), 2209-2219 (2010).
- Kalinin, S., Valeri, A., Antonik, M., Felekyan, S., Seidel, C. A. M. Detection of structural dynamics by FRET: A photon distribution and fluorescence lifetime analysis of systems with multiple states. The Journal of Physical Chemistry B. 114 (23), 7983-7995 (2010).
- Pirchi, M., et al. Photon-by-photon hidden Markov model analysis for microsecond single-molecule FRET kinetics. The Journal of Physical Chemistry B. 120 (51), 13065-13075 (2016).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. PAM: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophysical Journal. 114 (7), 1518-1528 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry B. 122 (49), 11554-11560 (2018).
- Bennet, I. A., et al. Regional conformational flexibility couples substrate specificity and scissile phosphate diester selectivity in human flap endonuclease 1. Nucleic Acids Research. 46 (11), 5618-5633 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados