Method Article
Präzise und genaue Einzelmolekül-FRET-Messungen mit der Open-Source-smfBox
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieser Artikel enthält Schritt-für-Schritt-Anleitungen für die Durchführung vollständig korrigierter genauer FRET-Messungen an einzelnen, frei streuenden Biomolekülen mit der quelloffenen, kostengünstigen smfBox, vom Einschalten über die Ausrichtung und Fokussierung bis hin zur Datenerfassung und -analyse.
Zusammenfassung
Die smfBox ist ein kürzlich entwickeltes kostengünstiges Open-Source-Instrument für den Einzelmolekül-Förster-Resonanz-Energietransfer (smFRET), das Messungen an frei streuenden Biomolekülen zugänglicher macht. Diese Übersicht enthält ein Schritt-für-Schritt-Protokoll für die Verwendung dieses Instruments zur Messung präziser FRET-Wirkungsgrade in Duplex-DNA-Proben, einschließlich Details zur Probenvorbereitung, geräteeinrichtung und -ausrichtung, Datenerfassung und vollständigen Analyseroutinen. Der vorgestellte Ansatz, der die Bestimmung aller Korrekturfaktoren beinhaltet, die für genaue FRET-abgeleitete Entfernungsmessungen erforderlich sind, baut auf einer vielzahl von jüngsten kollaborativen Arbeiten in der gesamten FRET-Community auf, die darauf abzielen, Standardprotokolle und Analyseansätze zu etablieren. Dieses Protokoll, das leicht an eine Reihe von biomolekularen Systemen angepasst werden kann, trägt zu den wachsenden Bemühungen bei, smFRET für die breitere wissenschaftliche Gemeinschaft zu demokratisieren.
Einleitung
Der Einzelmolekül-Förster-Resonanz-Energietransfer (smFRET) ist eine Technik, die die FRET-Effizienz zwischen zwei Farbstoffen - einem Donor und einem Akzeptor - auf der Ebene einzelner Moleküle misst. FRET ist ein photophysikalischer Prozess, der aus den überlappenden Energiespektren zweier Farbstoffe entsteht: Der Donor wird durch Licht einer bestimmten Wellenlänge angeregt und überträgt Energie nicht strahlend an den Akzeptor, was zu einer Emission vom Akzeptor führt. Die Effizienz dieser Übertragung ist umgekehrt proportional zur sechsten Potenz des Abstands zwischen den beiden Farbstoffen, so dass die Übertragungseffizienz mit der Entfernung1 variiert. Somit kann diese FRET-Effizienz verwendet werden, um räumliche Informationen über das(e)Molekül(e)2, an das/die Farbstoffe gebunden sind, in einem Bereich von 3-10 nm zu bestimmen. Diese Skala und die Tatsache, dass Änderungen der FRET-Effizienz empfindlich auf molekulare Bewegungen von Angstrom reagieren3, macht die Technik gut geeignet, um strukturelle Informationen über Biomoleküle - wie Nukleinsäuren und Proteine - ohne die Komplikationen der Ensemble-Mittelung zu untersuchen4,5,6. Während Änderungen der relativen FRET-Effizienz verwendet werden können, um biomolekulare Interaktionen und Konformationsdynamik zu überwachen und Wichtige zelluläre Prozesse wie Protein(un)faltung, Transkription und DNA-Replikation und -Reparatur zu beleuchten, wurden absolute FRET-Effizienzen verwendet, um genaue Abstände für die Biomolekularstrukturbestimmung zu bestimmen7,8,9,10,11 , wobei die Notwendigkeit der Kristallisation oder des Einfrierens überwunden wird, wie es für einige andere Strukturverfahren erforderlich ist4,12.
smFRET-Experimente nehmen am häufigsten zwei Formen an, konfokale oder totale interne Reflexionsfluoreszenzmikroskopie (TIRF). Zwischen beiden Ansätzen kann die molekulare Dynamik von Biomolekülen typischerweise auf Zeitskalen von Pico- bis Millisekunde (konfokale, frei diffundierende Moleküle) bis zu Millisekunden bis Stunden (TIRF, surface immobilized molecules) untersucht werden. Dies liegt an den unterschiedlichen Setups, die an jeder Technik beteiligt sind. In der TIRF-Mikroskopie werden Moleküle auf der Oberfläche eines Objektträgers immobilisiert und durch eine evaneszente Welle angeregt (Abbildung 1A). Hier liegt der Fokus jedoch auf der konfokalen Mikroskopie, da dies das Format der smfBox ist. In der konfokalen Mikroskopie werden Moleküle nicht immobilisiert und diffundieren stattdessen frei über brownsche Bewegung durch das konfokale Volumen (~ 1 fL), das durch Fokussieren eines Laserstrahls durch eine Linse mit hoher numerischer Apertur auf einen Punkt in einer bestimmten Tiefe innerhalb der Lösung entsteht (Abbildung 1B). Die resultierende Emission wird durch die gleiche Öffnung zurückfokussiert und durch einen dichroitischen Spiegel gefiltert (Abbildung 1C für den vollständigen Schaltplan). Es wird dann durch ein Loch fokussiert, um unscharfes Licht zu entfernen und auf eine Lawinenphotodiode (APD) zu fokussieren. Wenn der APD ein Photon detektiert, gibt er einen TTL-Impuls aus, dessen Timing mit einer Auflösung von bis zu Pikosekunden aufgezeichnet werden kann. Die Beobachtungszeit dieser frei diffundierenden Moleküle in der Nähe des konfokalen Volumens liegt üblicherweise in der Größenordnung von Millisekunden.
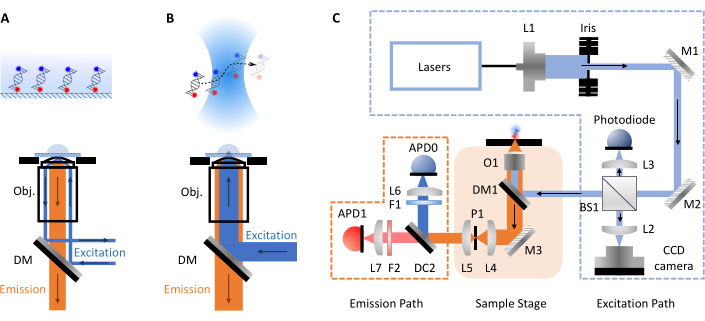
Abbildung 1: Schemata, die die Prinzipien der Mikroskopie und des smfBox-Aufbaus zeigen. (A) Total Internal Reflection Fluorescence (TIRF) Mikroskopisches Prinzip: Anregungslicht wird in den Rand des Objektivs (Obj.) geleitet und durchläuft eine totale interne Reflexion an der Deckglas-Puffer-Grenzfläche, wodurch ein exponentiell zerfallendes Evaneszenzfeld erzeugt wird, um an der Oberfläche befestigte Moleküle anzuregen. (B) Konfokale Mikroskopie: Frei diffundierende Moleküle werden durch einen nahezu beugungsbegrenzten Punkt angeregt, der in die Probe fokussiert ist. (C) Das in diesem Protokoll verwendete smfBox-Setup, das alle Schlüsselkomponenten anzeigt: Lawinenphotodioden (APD), Strahlteiler (BS), dichroitische Spiegel (DM), Filter (F), Spiegel (M), Objektiv (O) und Lochblende (P). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
In jüngerer Zeit enthielten smFRET-Techniken zwei Farbanregungen, bei denen Laser, die den Wellenlängen der Donor- und Akzeptoranregung entsprechen, abgewechselt werden5. Dies kann auf zwei Arten geschehen, die erste durch Modulation von Dauerstrichlasern auf der KHz-Zeitskala, die als alternierende Laseranregung (ALEX)13,14 bekannt ist. Die zweite Methode verschachtelt schnelle Impulse auf der MHz-Zeitskala; dies ist Nanosekunden-ALEX15 oder gepulste interleaved Excitation (PIE)16. Bei all diesen Ansätzen führen Informationen aus dem Akzeptorlaser zur Berechnung der sogenannten Stöchiometrie, die zwischen Molekülen mit einem niedrigen FRET-Wirkungsgrad und solchen ohne Akzeptor unterscheiden kann (entweder durch unvollständige Markierung oder Photobleichen). Die Verwendung von PIE/ns-ALEX ermöglicht zusätzlich den Zugang zu Fluoreszenzlebensdauern auf Einzelmolekülebene, und Anisotropien können gemessen werden, wenn sie mit polarisierenden Optiken gekoppelt sind. Diese Kombination von Messungen wird als Multiparameter-Fluoreszenzdetektion (MFD)9 bezeichnet.
Trotz der vielen Vorteile von smFRET ist es außerhalb von Speziallaboren aufgrund der hohen Kosten für kommerzielle Instrumente und des Mangels an einfachen, selbst gebauten Alternativen nicht weit verbreitet. Ein wachsender Trend zur Entwicklung einer kostengünstigen Open-Source-Mikroskopie findet statt und in letzter Zeit sind andere Plattformen entstanden, darunter Planktonscope17, OpenFlexure Microscope18, Flexiscope19, miCube20, liteTIRF21 und Squid22. Hierin beschreibt die Studie das Protokoll für die Verwendung der smfBox, einem kürzlich entwickelten kostengünstigen konfokalen Aufbau, der in der Lage ist, die FRET-Effizienz zwischen zwei Farbstoffen auf frei diffundierenden Einzelmolekülen zu messen. Detaillierte Bauanleitungen und die notwendige Betriebssoftware sind frei erhältlich unter: https://craggslab.github.io/smfBox/ 23. Die optische Anordnung der smfBox besteht aus leicht verfügbaren Komponenten, die von erschwinglichen und allgemein zugänglichen Herstellern gekauft wurden, während der Mikroskopkörper (der für den Großteil der Kosten in einem konfokalen Standardaufbau verantwortlich ist) durch eine kundenspezifische lichtdichte eloxierte Aluminiumbox ersetzt wurde (so dass Messungen unter Umgebungslichtbedingungen durchgeführt werden können). Diese Box enthält wichtige optische Komponenten, einschließlich der Anregung dichroitisch, des Objektivs und des Lochs sowie eine mechanische Laserverriegelung, die einen sicheren Betrieb als Laserprodukt der Klasse I ermöglicht (siehe Abbildung 1C für einen vollständigen Schaltplan). Die smfBox verwendet ALEX, um die Farbstoffstöchiometrie zu validieren und genaue FRET-Korrekturfaktoren zu bestimmen. Es wird mit einer eigens geschriebenen Open-Source-Software (smOTTER) betrieben, die alle Aspekte der Datenerfassung steuert und die Daten im Open-Source-Photon-HDF5-Format24 ausgibt, das mit vielen Analysetools von Drittanbietern kompatibel ist. Die smfBox und die Erfassungs- und Datenanalyseprotokolle wurden kürzlich in einer Multi-Lab-Blindstudie gegen >20 andere Instrumente (sowohl konfokal als auch TIRF) getestet25. Die erzielten FRET-Wirkungsgrade stimmten hervorragend mit allen anderen Instrumenten überein, obwohl die smfBox nur einen Bruchteil des Preises handelsüblicher Setups kostete.
Hier wird ein Schritt-für-Schritt-Protokoll zur Erfassung und Analyse genauer, absoluter FRET-Wirkungsgrade bei frei diffundierenden DNA-Duplexen mit der smfBox beschrieben, vom Einschalten über die Ausrichtung und Fokussierung bis hin zur Datenerfassung und -analyse. Bei den hier verwendeten Proben handelt es sich um drei Duplex-DNAs (mit hohen, mittleren und niedrigen FRET-Wirkungsgraden, siehe Tabelle 1), die in der weltweiten Blindenstudie25 bewertet wurden; Die Methode ist jedoch an viele molekulare Systeme anpassbar, einschließlich Proteine und andere Nukleinsäuren. Die Hoffnung ist, dass ein so detailliertes Protokoll zusammen mit den bereits vorhandenen Bauanleitungen für die smfBox23 dazu beitragen wird, diese leistungsstarke Technik für eine Vielzahl von Laboren noch zugänglicher zu machen.
Protokoll
1. Einschaltkomponenten
- Schalten Sie die sechs Stecker ein (keine bestimmte Reihenfolge): Piezoconcept (z-Fokus), APD0, APD1, grüner Laser, roter Laser und Photodetektor.
2. Software 1-Versuchsaufbau
- Starten Sie das Laser-Kontrollzentrum.
- Stellen Sie sicher , dass der Modus Dauerwelle - Konstanter Wechselstrom (CW-ACC) ausgewählt ist.
- Schalten Sie beide Laser ein.
- Überprüfen/einstellen Sie die Laserleistungen.
HINWEIS: Die Laserleistung muss so eingestellt werden, wie sie kurz vor der Anregungsdiroitis gemessen wurde, da ND-Filter und Strahlteiler im Anregungspfad die Laserleistung von der auf dem Laserbedienfeld angegebenen Zahl reduzieren. Die hier angegebenen Zahlen sind die Leistung bei der Anregung dichroitisch, aber die Leistung auf der Lasersteuerung, die dem entspricht, muss herausgearbeitet werden.- 220 μW grüner Laser (515 nm, 40 mW)
- μ70 W für roter Laser (638 nm, 10 mW)
- smOTTER Akquisitionssoftware
- Schließen Sie die Laser, Detektoren, die Z-Stufe und die Kamera an. Konfigurieren Sie sie korrekt (kann je nach Den Einstellungen des jeweiligen NI-Karten-Setups variieren).
3. Ausrichtung des Emissionspfads (nicht routinemäßig erforderlich)
- Einrichten der Ausrichtung
- 10 μL des freien Cy3B-Farbstoffs (~100 nM) werden auf das Mikroskop pipettiert und wie in den Schritten 4.3-4.5 beschrieben fokussiert.
HINWEIS: Ein weiterer Spenderfarbstoff mit etwas Leckage in den Akzeptorkanal würde ebenfalls funktionieren. - Öffnen Sie die Registerkarte Ausrichtung in smOTTER, verringern Sie die Laserleistung und ändern Sie die y-Achsenskala, bis die Anzeige von den Detektoren sichtbar ist.
HINWEIS: Das Ziel hier ist es, das Signal zu erhöhen, so dass die Skala möglicherweise erneut geändert werden muss, nachdem das Signal erhöht wurde. - Schrauben Sie die vier Schrauben an der Vorderseite der smfBox ab und entfernen Sie die Frontplatte.
- 10 μL des freien Cy3B-Farbstoffs (~100 nM) werden auf das Mikroskop pipettiert und wie in den Schritten 4.3-4.5 beschrieben fokussiert.
- Pinhole-Ausrichtung
- Drehen Sie den x-Knopf am Lochlochpositionierer, während Sie das Signal auf der Registerkarte Ausrichtung beobachten und versuchen, das Signal in Grün und Rot zu erhöhen.
- Drehen Sie nun den Y-Knopf, um das Loch in die andere Richtung auszurichten.
- Kehren Sie zum x-Knopf zurück, um zu überprüfen, ob das Signal weiter zunimmt.
- Pinhole-Objektiv-Ausrichtung
- Drehen Sie den Objektiv x Knopf in eine Richtung, dies verringert das Signal.
- Drehen Sie den Pinhole x-Knopf in die gleiche Richtung, um das Signal wieder zu erhöhen.
- Wenn das neue max-Signal höher als zuvor ist, bewegen Sie sowohl das Loch als auch die Linse iterativ in diese Richtung. Wenn es niedriger als zuvor ist, bewegen Sie sich iterativ in die entgegengesetzte Richtung.
- Wiederholen Sie den obigen Vorgang für y.
- Ausrichtung des APD-Objektivs
- Beginnen Sie mit dem grünen APD, bewegen Sie den x-Knopf, bis das grüne Signal maximal ist.
- Wiederholen Sie den Vorgang für den Y-Knopf.
- Kehren Sie zum x-Knopf zurück, bewegen Sie sich hin und her, um die Schwellenwertpunkte zu finden, an denen das Signal zu fallen beginnt, und lassen Sie es an einer Position auf halbem Weg zwischen diesen beiden Punkten.
- Wiederholen Sie den Vorgang für den x-Knopf.
- Wiederholen Sie die obigen Schritte für das rote APD-Objektiv und beobachten Sie das rote Signal.
- Setzen Sie die Frontplatte wieder auf die smfBox und setzen Sie die Schrauben wieder ein.
HINWEIS: Führen Sie die obige Ausrichtung monatlich und als Vorsichtsmaßnahme durch, wenn der Verdacht besteht, dass das Mikroskop zwischen den Messungen einen Ausrichtungsfehler entwickelt hat.
4. Messdatenerfassung
- Für die erste Probe reinigen Sie die Stufe mit Linsenreinigungsgewebe, das in Methanol getränkt ist; wischen Sie vorsichtig über das Objektiv von einem Ende zum anderen.
- Tragen Sie 3-4 Tropfen Tauchöl für die Mikroskopie auf die Mitte des Objektivs auf; nach Bedarf zwischen den Proben auffüllen.
- Probenvorbereitung
- 10 μL Probe (erstmalige Verwendung von Reinstwasser Typ I) auf die Mitte eines sauberen Deckglases pipettieren.
- Legen Sie das Deckglas vorsichtig auf die Objektivlinse und senken Sie es in einem Winkel zum Öl ab, um luftblasen zwischen dem Deckglas und dem Objektiv einzufangen.
HINWEIS: Schieben Sie bei Bedarf das Glas ab, um Luftblasen im Öl vom Brennpunkt weg zu drücken.
- Um ALEX-Experimente (Alternating Laser Excitation) durchzuführen, konfigurieren Sie den Laser im Laser-Tastverhältnis wie folgt.
- Spender (grüner Laser) 0 aus, 45 ein, 55 aus
- Akzeptor (roter Laser) 50 aus, 45 an, 5 aus
- ALEX-Periode (μs): 100
- Klicken Sie im Menüband auf die Registerkarte Z-Fokus . Schalten Sie im Erfassungsbereich die Laser auf Live um und starten Sie die Kamera. Passen Sie die Belichtung so an, dass ein heller Fleck zentral von Schwarz umgeben erscheint.
HINWEIS: Die Kamera erfasst Licht, das von der Glas/Wasser-Grenzfläche zwischen dem Deckglas und der Probe reflektiert wird, und der sichtbare Lichtkreis hat seinen kleinsten Durchmesser, wenn sich der Brennpunkt an dieser Schnittstelle befindet. Es ist daher notwendig, unterhalb dieser Schnittstelle zu beginnen, die z-Höhe zur Schnittstelle zu erhöhen und dann etwas weiter über dem Deckglas und in der zu messenden Probe zu fokussieren.- Ausgehend von einer niedrigen Z-Position erhöhen Sie die Höhe, bis der helle Punkt seine minimale Größe erreicht, und erhöhen Sie dann die Höhe um bis zu 20 μm, um den Laser über dem Öl- und Deckglas und in der Probe zu fokussieren.
- Stoppen Sie die Kamera, sobald sie scharf ist.
- Um zu bestätigen, dass der Aufbau ordnungsgemäß durchgeführt wurde, überwachen Sie die Spur von Reinstwasser Typ I, um keine Fluoreszenzsignale zu sehen. Wiederholung der Reinheitsbestätigung für die Puffer, in denen Proben hergestellt werden.
- Entfernen Sie das Deckglas vorsichtig mit einer Pinzette mit Gummiende, um eine Beschädigung des Objektivs zu vermeiden, und legen Sie dann die wie oben vorbereitete Probe von Interesse auf das Objektiv. Füllen Sie das Tauchöl nach Bedarf auf, um einen gleichmäßigen Kontaktbereich zwischen Deckglas und Objektiv aufrechtzuerhalten (besonderes Augenmerk auf die Kontaktfläche der Probe auf dem Deckglas und den Fokusbereich des Objektivs)
- Konzentration Bingo
- Um sicherzustellen, dass Einzelmoleküldaten gewonnen werden, müssen die Proben in einer Konzentration sein, bei der 1-5 Ausbrüche pro Sekunde im Live-Trace-Panel von smOTTER beobachtet werden (dies minimiert die Wahrscheinlichkeit, dass mehr als ein Molekül gleichzeitig im Nachweisvolumen vorhanden ist). Führen Sie die Messungen durch, wenn die entsprechende Konzentration bestimmt wurde.
- Um die Verdunstung während langer Experimente zu verhindern, bereiten Sie eine luftdichte Probenkammer vor. Drücken Sie die Silikonisolatoren (Loch mit 8-9 mm Durchmesser) auf die Mitte eines Deckglases (eingefärbt mit einer Pipettenspitze). Dann pipettieren Sie eine Probe vorsichtig in die Mitte und vermeiden Sie jeglichen Kontakt mit dem Silikon. Legen Sie dann ein zweites Deckglas darauf und drücken Sie, um eine Versiegelung zu bilden.
- Überprüfen Sie das Live-Stöchiometrie- und FRET-Effizienz-Histogramm (ES), um festzustellen, ob sich die Probe wie erwartet verhält, d. H. Erwartete FRET-Effizienz und angemessene Stöchiometrie (~ 0,5).
- Wenn das Beispiel fertig ist, geben Sie die Details für den Versuch in das Bedienfeld "Einstellungen speichern" ein. Wählen Sie ein geeignetes Verzeichnis und einen Dateinamen aus, indem Sie in den Speichereinstellungen auf das Auslassungssymbol (Dateien im hdf5/h5-Format) klicken.
- Geben Sie Informationen für Probenname, Probendetails, Spender- und Akzeptoretiketten, Puffer-, Donor- und Akzeptoranregungswellenlängen, Detektionswellenlängen und Laserleistung sowie Benutzer- und Benutzerzugehörigkeit ein.
- Kehren Sie zur Registerkarte Live-Ablaufverfolgung im Menüband zurück, und geben Sie im Erfassungsbereich die Testlänge (in Minuten) ein geeignetes Speicherintervall aus, um potenzielle Datenverluste im Fehlerfall zu minimieren. Wählen Sie bei Bedarf Laserstärken speichern aus.
- Drücken Sie Start , um die Daten zu übernehmen.
HINWEIS: Um eine genaue FRET-Bestimmung zu ermöglichen, müssen mindestens zwei Proben gemessen werden, die die gleichen Donor- und Akzeptorfarbstoffe enthalten, jedoch mit ausreichend unterschiedlichen FRET-Wirkungsgraden.
5. Analyse/Software 2
- Starten Sie das Jupyter-Notebook mit dem FRETBursts-Python-Paket (Setup-Anweisungen finden Sie hier: https://craggslab.github.io/smfBox/anasoftware.html)
- Wenn eine Korrektur für absolute FRET-Wirkungsgrade nicht erforderlich ist (d.h. nur relative Änderungen des FRET für die Messung ausreichen), starten Sie Jupyter notebook FRET Analysis 1.4 Uncorrected.ipynb. Verwenden Sie diese Option, um Zahlen oder Daten als CSV-Dateien zur weiteren Analyse oder Darstellung in anderer Software zu exportieren.
HINWEIS: Jedes Notizbuch enthält detaillierte Anweisungen, um den Benutzer durch die Analyseverfahren zu führen. Eine ausführlichere Erörterung der Analyseverfahren, einschließlich Burst-Suchalgorithmen, Hintergrundkorrektur und aller Korrekturparameter, finden Sie unter 23,25. - Wenn eine Korrektur für absolute FRET-Wirkungsgrade erforderlich ist, beginnen Sie mit der Berechnung der Übersprechparameter alpha (Leckage der Donoremission in den Akzeptorkanal) und delta (Anteil der direkten Anregung des Akzeptors unter Donoranregung).
- Starten Sie zunächst jupyter notebook Correction Factor Finder Alpha-Delta.ipynb und bestimmen Sie die Alpha- und Delta-Parameter.
HINWEIS: Wenn diese zwischen den Proben inkonsistent sind, kann das Mikroskop ein Ausrichtungsproblem zwischen den Messungen entwickelt haben oder die Spektren der Farbstoffe unterscheiden sich zwischen den Proben. - Wenn die Alpha- und Delta-Parameter konsistent sind, starten Sie jupyter notebook Correction Factor Finder Gamma-Beta.ipynb , um die Gamma- und Beta-Parameter zu bestimmen (die Unterschiede in der Anregung und Detektionseffizienz zwischen den Farbstoffen berücksichtigen).
HINWEIS: Wenn das Gamma- und Beta-Diagramm nicht gut zusammenpasst, hat das Mikroskop möglicherweise ein Ausrichtungsproblem zwischen den Messungen entwickelt, oder die Quantenausbeuten oder die Extinktionskoeffizienten der Farbstoffe unterscheiden sich zwischen den Proben. - Wenn die vier Übersprechparameter bestimmt sind, können diese Faktoren im FRET Analysis 1.4 Corrected.ipynb Jupyter Notebook verwendet werden, um absolute FRET-Wirkungsgrade zu bestimmen.
- Starten Sie zunächst jupyter notebook Correction Factor Finder Alpha-Delta.ipynb und bestimmen Sie die Alpha- und Delta-Parameter.
6. Fehlerbehebung
- Wenn alle Signale niedrig sind oder die Anzahl pro Burst niedriger ist als erwartet (dies ist farbstoffabhängig - aber für ATTO-550 und ATTO-647N auf der smfBox liegen die typischen Werte zwischen 50 und 100 Zählungen pro ms während eines Einzelmolekül-Bursts), richten Sie die smfBox neu aus.
- Brücke zwischen doppelt und einzeln gekennzeichneten Populationen auf dem ES-Histogramm.
HINWEIS: Dies kann entweder durch Arbeiten mit einer zu hohen Konzentration (Abhilfe durch Verdünnen der Probe) oder durch Photobleichen verursacht werden, das durch die Verwendung einer zu hohen Laserleistung verursacht werden kann (Beheben Sie dies durch Verringerung der Laserleistung). - Wenn die Berstrate während des gesamten Experiments sinkt (fluoreszierend markierte Moleküle haften wahrscheinlich am Glasdeckglas), verwenden Sie eine erhöhte Konzentration von BSA, um zu korrigieren.
- Wenn die Berstrate während des Experiments zunimmt (die Probe verdampft wahrscheinlich), bereiten Sie eine luftdichte Probenkammer wie oben beschrieben vor.
- Wenn das Signal während der Ausrichtung auf Null abstürzt (die Software wird wahrscheinlich vom Signal der Detektoren überwältigt), verringern Sie die Laserleistung oder verwenden Sie eine verdünntere Ausrichtungsprobe.
- Wenn der Z-Fokus konzentrische Ringe zeigt (kann passieren, wenn mehrere Deckgläser auf dem Objektiv platziert wurden), suchen Sie nach mehreren Deckgläsern auf dem Objektiv.
- Wenn die Probe helle, lange Ausbrüche von intermediären Stöchiometrien (verursacht durch aggregierte Moleküle) enthält, verwenden Sie Detergenzien oder ändern Sie die Probenreinigungsprotokolle.
HINWEIS: Einen sauberen Puffer zu erhalten, kann ein Problem sein, da einige Pufferkomponenten oft sehr kleine Mengen mäßig fluoreszierender Verunreinigungen enthalten, die ausreichen, um als einfarbige Bursts auf der Zeitspur zu präsentieren. Wenn sich zu viel davon im Puffer befindet, kann dies mit Probenausbrüchen zusammenfallen und die gemessene FRET-Effizienz oder Stöchiometrie ändern. Insbesondere BSA kann in dieser Hinsicht oft problematisch sein, daher ist es hilfreich, eine Standard-BSA-Lösung einer starken Lichtquelle auszusetzen, um die Verunreinigungen zu photobleichen.
Ergebnisse
Das Protokoll erfordert eine kritische Bewertung der experimentellen Bedingungen während des Aufbaus (siehe Protokollschritt 4.8). Die ersten gewonnenen Ergebnisse, die über Erfolg oder Misserfolg des Experiments entscheiden, werden in diesem Stadium erreicht. Ein positives Ergebnis wäre, zwischen fünf und einem Burst pro Sekunde zu haben (siehe Abbildung 2B,C). Ein negatives Ergebnis wäre zu viele (Abbildung 2A) oder zu wenige Bursts (Abbildung 2D) innerhalb dieses Zeitrahmens. In diesem Stadium ist es noch möglich, diese Fehler zu beheben: Eine Probe mit einer zu hohen Konzentration muss einfach verdünnt werden; Wenn die Konzentration jedoch zu niedrig ist, muss möglicherweise eine neue Probe vorbereitet werden (entscheidend ist, ob es bei dieser niedrigen Konzentration weiterhin möglich ist, Daten in einem angemessenen Zeitrahmen zu sammeln).

Abbildung 2: Screenshots von Live-Spuren während des Versuchsaufbaus, die unterschiedliche Konzentrationen von doppelt markierten Duplex-DNA-Proben zeigen. (A) zu hoch, (B) obere akzeptable Grenze, (C) Zielkonzentration, (D) zu niedrig. Photonenzahlen (1 ms Bins) werden in den drei Detektionskanälen angezeigt; Donoremission nach Donoranregung (DD), Akzeptoremission nach Donoranregung (DA) und Akzeptoremission nach Akzeptoranregung (AA). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ein statisches Einzelspezies-System würde typischerweise 30 bis 60 Minuten Messung benötigen, um die notwendigen ~ 1.000 Bursts zu erhalten, die für eine robuste Datenanalyse erforderlich sind. Die Dauer und Anzahl der erforderlichen Bursts nimmt mit mehreren Spezies oder dynamischen Systemen zu. Anschließend werden Datenerhebungen und -analysen anhand des Protokolls Zahlen aus den Jupyter Notebooks exportiert. Das Wechseldiagramm (Abbildung 3A) sollte mit der ALEX-Periode des Versuchsaufbaus übereinstimmen. Die Zeitspur (Abbildung 3B) wird verwendet, um qualitativ zu beurteilen, ob die Probenkonzentration angemessen ist. Das Hintergrunddiagramm (Abbildung 3C) zeigt die Verteilung der Interphotonen-Verzögerungsperioden mit einer linearen Anpassung an die längeren Zeiten, um die Hintergrundrate zu schätzen26. Die Hintergrundspur (Abbildung 3D) kann identifizieren, ob es während der Dauer des Experiments Veränderungen in der Probe gab; dies wäre in erster Linie auf die Verdunstung bei längeren Erfassungszeiten zurückzuführen. ES-Histogramme werden für alle Photonen (Abbildung 3E) und doppelt markierten Spezies (Abbildung 3F) erzeugt. Schließlich wird ein 1D-E-Histogramm (Abbildung 3G) mit Gauß-Anpassung der Burst-Daten generiert.

Abbildung 3: Beispiel für die Ausgabe analysierter Daten, die von den Jupyter Notebooks generiert wurden. (A) Alternationsdiagramm, (B) Zeitablaufverfolgung, (C) Hintergrundbestimmung, (D) Hintergrundraten, (E) All photon ES-Histogramm, (F) Zweikanal-ES-Histogramm und (G) 1D E-Histogramm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Name | Reihenfolge |
| 1a | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAA CTC ACA AAC AGA CC - 5' | |
| 1b | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAA CTC ACA AAC AGA CC - 5' | |
| 1c | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAA CTC ACA AAC AGA CC - 5' |
Tabelle 1: DNA-Sequenzen, die im Protokoll verwendet werden. Nukleotide sind blau und rot hervorgehoben, was C2-aminomodifizierte Thyminreste darstellt, die mit Atto-550 bzw. Atto-647N markiert sind.
Korrekturfaktor-Finder Alpha-Delta: Bitte klicken Sie hier, um diese Datei herunterzuladen.
Korrekturfaktor-Finder Gamma-Beta: Bitte klicken Sie hier, um diese Datei herunterzuladen.
FRET Analysis 1.4 Korrigiert: Bitte klicken Sie hier, um diese Datei herunterzuladen.
FRET Analysis 1.4 Unkorrigiert: Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die kritischsten Schritte im Protokoll sind die Ausrichtung des Mikroskops und die Anpassung der Probenkonzentration an die richtige Verdünnung. Wenn die Ausrichtung deaktiviert ist, liegt möglicherweise kein ausreichendes Signal vor, um Bursts zu identifizieren und Histogramme darzustellen, und wenn eine Fehlausrichtung zwischen den Proben auftritt, kann die genaue FRET-Korrektur aufgrund von Änderungen der Leckage- und Erkennungs- / Anregungseffizienz fehlschlagen. Die Verwendung einer angemessenen Konzentration ist ebenfalls wichtig, eine zu hohe Konzentration führt zu zufälligen Bursts, die mehrere Moleküle mit potenziell unterschiedlichen FRET-Wirkungsgraden oder Markierungsstöchiometrien enthalten. Eine zu niedrige Konzentration führt zu wenig Ausbrüchen für eine robuste Datenanalyse.
Das hier beschriebene Protokoll dient zur Messung von Abständen in statischen Einzel-FRET-Spezies. Wenn die Probe mehr als einen Peak im FRET-Effizienzhistogramm aufweist oder Peaks breit erscheinen (was bei dynamischen Spezies vorkommen kann), sind möglicherweise mehr Bursts erforderlich, um Histogramme mit der gleichen Präzision anzupassen. Für zwei gut getrennte Peaks werden dann etwa doppelt so viele Daten benötigt, aber wenn sich die Populationen leicht überlappen, werden noch mehr Daten benötigt.
Wenn sich die beiden Populationen auf der Zeitskala des Experiments interkonvertieren, kann die Dynamik und Kinetik des Systems möglicherweise bestimmt werden. Tests wie BVA27 und 2CDE28 können bestätigen, dass die Zwischenausbrüche dynamischer Natur sind, während Analysen mit dPDA29,30 oder H2MM31 die Interkonversionsraten bestimmen können. Jupyter Notebooks für BVA und 2CDE sind auf der FRETBursts26-Website verfügbar, und die MATLab-basierte Software PAM32 kann BVA-, 2CDE- und PDA-Analysen ausführen.
Konfokales Einzelmolekül-FRET kann leicht Zustände beobachten, die viel kurzlebiger sind (~ 1 ms) als TIRF; Die kurzen Beobachtungszeiten, die durch die Diffusion begrenzt sind, geben jedoch keine molekulare Geschichte und können daher keine längeren Verweilzeiten oder komplexe Übergangsnetzwerke so bestimmen, wie es oberflächenimmobilisierte Experimente können.
Da das Protokoll frei diffundierende Moleküle in einer sehr geringen Konzentration misst, funktioniert es am besten, wenn intramolekulare Abstände auf demselben Molekül gemessen werden. Intermolekulare Abstände zwischen vorübergehend gebundenen Molekülen können gemessen werden, vorausgesetzt, dass die Kd der beiden Moleküle niedrig genug ist, dass der Komplex bei der für das Experiment erforderlichen niedrigen Arbeitskonzentration (~ 100 pM) in einer signifikanten Menge vorhanden ist. Wenn die Kd viel höher ist, werden nur einzeln markierte Moleküle gesehen. Dieses Problem kann überwunden werden, indem die Mikrofluidik verwendet wird, um die beiden markierten Komponenten in einer hohen Konzentration miteinander zu mischen und dann schnell zu verdünnen und über das Objektiv zu fließen, bevor der Komplex dissoziiert33,34.
Die Messung der FRET-Effizienz auf Einzelmolekülebene hat einen signifikanten Vorteil gegenüber Ensemble-Techniken, da sie auf heterogene Subpopulationen informiert, die in einem Ensemble-Experiment gemittelt würden. Darüber hinaus bietet Einzelmolekül-FRET mit ALEX Zugang zu genauen FRET-Wirkungsgraden, die in genaue Entfernungen umgewandelt werden können. Dies ermöglicht die Bestimmung detaillierterer Strukturinformationen, anstatt nur relative Entfernungsänderungen zu untersuchen. Die smfBox bietet all diese Vorteile und Fähigkeiten, kann aber mit einem viel geringeren Budget konstruiert werden als vergleichbare kommerziell erhältliche Mikroskope, die konfokales smFRET23 verarbeiten können.
Die smfBox stellt eine viel niedrigere Eintrittsbarriere für smFRET-Techniken dar, die es den Forschern ermöglicht, Konformationsänderungen und genaue Abstände innerhalb und zwischen Proteinen und Nukleinsäuren zu messen7,8,9,10,11,35.
Offenlegungen
Die Autoren erklären keine gegensätzlichen Interessen.
Danksagungen
Die Autoren danken den folgenden Finanzierungsquellen: BBSRC (BB/T008032/1); EPSRC (Studentship to B.A.) und MRC (Studentship to A. R.-T.).
Materialien
| Name | Company | Catalog Number | Comments |
| Amino modified oligonucleotide | Eurogentec | N/A | May be ordered from various suppliers or synthesised; amino modification enables labeling with NHS-ester modified dyes |
| Avalanche photodiode (APD) | Excelitas | SPCM-AQRH-14 | Two APDs are required for the smfBox setup |
| Bovine Serum Albumin (BSA) | Merck | A2153 | System dependant; imaging buffer component (0.1 mg/mL in buffer) |
| Compact Laser Combiner | OMICRON | LightHUB-2 | 515 nm (80 mW) and 638 nm (100 mW) lasers |
| Coverglass | VWR | 630-2742 | Thickness: 0.17 ± 0.01 mm, LxW: 22x22 mm |
| Cy3B | Cytiva | PA63101 | 1 mg, PA63100 (5 mg), PA96106 (25 mg) |
| FRETBursts Python Package | N/A | N/A | Open-source python package for burst analysis of freely-diffusing single-molecule FRET data: https://fretbursts.readthedocs.io |
| Imaging Buffer | N/A | System dependant; 5 mM NaCl, 20 mM MgCl2, 5 mM Tris pH 7.5 and 0.1 mg/mL BSA | |
| Immersion Oil | Olympus | IMMOIL-F30CC | |
| Jupyter notebooks | Project Jupyter | N/A | Open-source web application to create and share documents that contain live code, equations, visualizations and text; data analysis notebooks for smfBox can be found in the SI |
| Lens Tissue | ThorLabs | MC-5 | MC-50E is same item in bulk |
| Magnesium Chloride | Merck | M2670 | System dependant; imaging buffer component (20 mM in buffer) |
| MilliQ/Ultrapure water | N/A | ||
| Nanopoistioner | Piezoconcept | FOC300 | Nanopositioner for accurate positioning of microscope objective |
| NHS-ester modified ATTO-550 | ATTO-TEC | AD 550-31 | 1 mg, AD 550-35 (5 mg) |
| NHS-ester modified ATTO-647N | ATTO-TEC | AD 647N-31 | 1 mg, AD 647N-35 (5 mg) |
| Objective lens | Olympus | N1480700 | Olympus objective series from orignal smfBox discontinued; replaced by N5702300 |
| OMICRON Control Center (OCC)- laser control center | OMICRON | N/A | v3.5.34 - OMICRON laser driver software |
| Press-To-Seal silicone isolator | Grace Bio-Labs | 664201 | 8-9 mm Diameter x 1.7 mm Depth |
| smOTTER | N/A | N/A | Open-source acquisition software for the Craggs Lab smfBox: https://github.com/craggslab/smOTTER |
| Sodium Chloride | Merck | S7653 | System dependant; imaging buffer component (5 mM in buffer) |
| Tris base | Merck | 93362 | System dependant; imaging buffer component (5 mM, pH 7.5 in buffer) |
| Type I ultrapure water | Merck | ZIQ7000T0 | Milli-Q® IQ 7000 Ultrapure Water System |
Referenzen
- Forster, T. Intermolecular energy migration and fluorescence. Annals of Physics. 437 (1-2), 55-75 (1948).
- Stryer, L., Haugland, R. P. Energy transfer: a spectroscopic ruler. Proceedings of the National Academy of Sciences. 58 (2), 719-726 (1967).
- Hohlbein, J., et al. Conformational landscapes of DNA polymerase I and mutator derivatives establish fidelity checkpoints for nucleotide insertion. Nature Communications. 4 (1), 2131 (2013).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), (2018).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chemical Society Reviews. 43 (4), 1156-1171 (2014).
- Lerner, E., et al. The FRET-based structural dynamics challenge -- community contributions to consistent and open science practices. arXiv. , (2020).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nature Methods. 14 (2), 174-180 (2017).
- Craggs, T. D., et al. Substrate conformational dynamics facilitate structure-specific recognition of gapped DNA by DNA polymerase. Nucleic Acids Research. 47 (20), 10788-10800 (2019).
- Tsytlonok, M., et al. Dynamic anticipation by Cdk2/Cyclin A-bound p27 mediates signal integration in cell cycle regulation. Nature Communications. 10 (1), 1676 (2019).
- Nagy, J., et al. Complete architecture of the archaeal RNA polymerase open complex from single-molecule FRET and NPS. Nature Communications. 6 (1), 6161 (2015).
- LeBlanc, S. J., et al. Coordinated protein and DNA conformational changes govern mismatch repair initiation by MutS. Nucleic Acids Research. 46 (20), 10782-10795 (2018).
- Segal, M., et al. High-throughput smFRET analysis of freely diffusing nucleic acid molecules and associated proteins. Methods. 169, 21-45 (2019).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: Analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences. 101 (24), 8936-8941 (2004).
- Kapanidis, A. N., et al. Alternating-laser excitation of single molecules. Accounts of Chemical Research. 38 (7), 523-533 (2005).
- Müller, B. K., Zaychikov, E., Brauchle, C., Lamb, D. C. Pulsed interleaved excitation. Biophysical Journal. 89 (5), 3508-3522 (2005).
- Laurence, T. A., Kong, X., Jager, M., Weiss, S. Probing structural heterogeneities and fluctuations of nucleic acids and denatured proteins. Proceedings of the National Academy of Sciences of the United States of America. 102 (48), 17348-17353 (2005).
- Pollina, T., et al. PlanktonScope: Affordable modular imaging platform for citizen oceanography. bioRxiv. , 056978 (2020).
- Collins, J. T., et al. Robotic microscopy for everyone: the OpenFlexure microscope. Biomedical Optics Express. 11 (5), 2447-2460 (2020).
- Courtney, A., Alvey, L. M., Merces, G. O. T., Burke, N., Pickering, M. The Flexiscope: a low cost, flexible, convertible and modular microscope with automated scanning and micromanipulation. Royal Society Open Science. 7 (3), 191949 (2020).
- Martens, K. J. A., et al. Visualisation of dCas9 target search in vivo using an open-microscopy framework. Nature Communications. 10 (1), 3552 (2019).
- Auer, A., et al. Nanometer-scale multiplexed super-resolution imaging with an economic 3D-DNA-PAINT microscope. ChemPhysChem. 19 (22), 3024-3034 (2018).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , 424613 (2020).
- Ambrose, B., et al. The smfBox is an open-source platform for single-molecule FRET. Nature Communications. 11 (1), 5641 (2020).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An open file format for timestamp-based single-molecule fluorescence experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-a multi-laboratory benchmark study. Nature Methods. 15 (9), 669-676 (2018).
- Ingargiola, A., Lerner, E., Chung, S., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing single-molecule FRET. PLOS One. 11 (8), 0160716 (2016).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule FRET experiments with burst variance analysis. Biophysical Journal. 100 (6), 1568-1577 (2011).
- Tomov, T. E., et al. Disentangling subpopulations in single-molecule FRET and ALEX experiments with photon distribution analysis. Biophysical Journal. 102 (5), 1163-1173 (2012).
- Santoso, Y., Torella, J. P., Kapanidis, A. N. Characterizing single-molecule FRET dynamics with probability distribution analysis. ChemPhysChem. 11 (10), 2209-2219 (2010).
- Kalinin, S., Valeri, A., Antonik, M., Felekyan, S., Seidel, C. A. M. Detection of structural dynamics by FRET: A photon distribution and fluorescence lifetime analysis of systems with multiple states. The Journal of Physical Chemistry B. 114 (23), 7983-7995 (2010).
- Pirchi, M., et al. Photon-by-photon hidden Markov model analysis for microsecond single-molecule FRET kinetics. The Journal of Physical Chemistry B. 120 (51), 13065-13075 (2016).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. PAM: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophysical Journal. 114 (7), 1518-1528 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry B. 122 (49), 11554-11560 (2018).
- Bennet, I. A., et al. Regional conformational flexibility couples substrate specificity and scissile phosphate diester selectivity in human flap endonuclease 1. Nucleic Acids Research. 46 (11), 5618-5633 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten