Method Article
Effettuare misurazioni FRET a singola molecola precise e accurate utilizzando l'smfBox open source
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo articolo fornisce istruzioni dettagliate per effettuare misurazioni FRET accurate e completamente corrette su singole biomolecole a diffusione libera utilizzando l'smfBox open source ed economico, dall'accensione, attraverso l'allineamento e la messa a fuoco, alla raccolta e all'analisi dei dati.
Abstract
SmfBox è uno strumento open source economico di recente sviluppo per il trasferimento di energia di risonanza Förster a singola molecola (smFRET), che rende più accessibili le misurazioni su biomolecole a diffusione libera. Questa panoramica include un protocollo passo-passo per l'utilizzo di questo strumento per effettuare misurazioni di precise efficienze FRET in campioni di DNA duplex, inclusi i dettagli della preparazione del campione, la configurazione e l'allineamento dello strumento, l'acquisizione dei dati e le routine di analisi complete. L'approccio presentato, che include come determinare tutti i fattori di correzione necessari per accurate misurazioni della distanza derivate da FRET, si basa su un ampio corpus di recenti lavori collaborativi in tutta la comunità FRET, che mira a stabilire protocolli standard e approcci di analisi. Questo protocollo, che è facilmente adattabile a una serie di sistemi biomolecolari, si aggiunge ai crescenti sforzi di democratizzazione di smFRET per la più ampia comunità scientifica.
Introduzione
Il trasferimento di energia di risonanza Förster a singola molecola (smFRET) è una tecnica che misura l'efficienza FRET tra due coloranti - un donatore e un accettore - a livello di singole molecole. FRET è un processo fotofisico derivante dalla sovrapposizione di spettri di energia di due coloranti: il donatore è eccitato dalla luce di una specifica lunghezza d'onda e trasferisce energia in modo non radiativo all'accettore, con conseguente emissione dall'accettore. L'efficienza di questo trasferimento è inversamente proporzionale alla sesta potenza della distanza tra i due coloranti, quindi l'efficienza di trasferimento varia con la distanza1. Pertanto, questa efficienza FRET può essere utilizzata per determinare le informazioni spaziali sulle molecole 2 a cui sono attaccati i coloranti, entro un intervallo di 3-10 nm. Questa scala, e il fatto che i cambiamenti nell'efficienza di FRET sono sensibili ai movimenti molecolari di Angstrom3, rende la tecnica adatta a studiare le informazioni strutturali sulle biomolecole - come gli acidi nucleici e le proteine - senza le complicazioni della media dell'insieme4,5,6. Mentre i cambiamenti nelle efficienze FRET relative possono essere utilizzati per monitorare le interazioni biomolecolari e le dinamiche conformazionali, facendo luce su processi cellulari chiave come il (s)ripiegamento delle proteine, la trascrizione e la replicazione e riparazione del DNA, le efficienze fret assolute sono state utilizzate per determinare distanze precise per la determinazione della struttura biomolecolare7,8,9,10,11 , superando la necessità di cristallizzazione o congelamento come richiesto per alcuni altri metodi strutturali4,12.
Gli esperimenti smFRET assumono più comunemente due forme, microscopia a fluorescenza a riflessione interna totale (TIRF) o confocale o a riflessione interna totale. Tra i due approcci la dinamica molecolare delle biomolecole può essere tipicamente studiata su scale temporali da pico- a millisecondi (molecole confocali, liberamente diffuse) fino a millisecondi a ore (TIRF, molecole immobilizzate di superficie). Ciò è dovuto alle diverse configurazioni coinvolte in ciascuna tecnica. Nella microscopia TIRF, le molecole sono immobilizzate sulla superficie di un vetrino ed eccitate da un'onda evanescente (Figura 1A). Qui, tuttavia, l'attenzione si concentra sulla microscopia confocale in quanto questo è il formato di smfBox. Nella microscopia confocale, le molecole non sono immobilizzate e invece si diffondono liberamente attraverso il movimento browniano attraverso il volume confocale (~ 1 fL), formato focalizzando un raggio laser attraverso una lente ad alta apertura numerica in un punto a una profondità designata all'interno della soluzione (Figura 1B). L'emissione risultante viene focalizzata indietro attraverso la stessa apertura e filtrata attraverso uno specchio dicroico (Figura 1C per schema completo). Viene quindi focalizzato attraverso un foro stenopeico per rimuovere qualsiasi luce sfocata e su un fotodiodo a valanga (APD). Quando l'APD rileva un fotone, emette un impulso TTL, la cui temporizzazione può essere registrata con una risoluzione fino a picosecondi. Il tempo di osservazione di queste molecole liberamente diffuse nelle vicinanze del volume confocale è comunemente nell'ordine dei millisecondi.
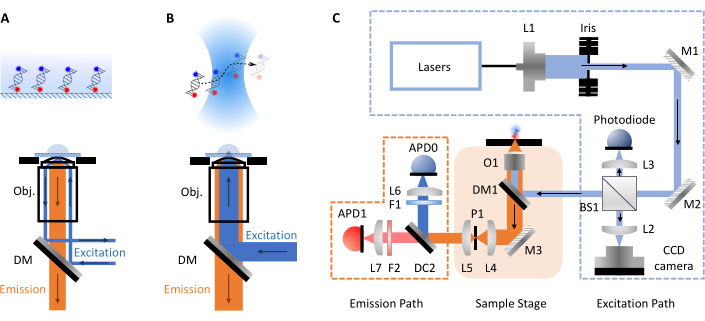
Figura 1: Schemi che mostrano i principi della microscopia e la configurazione smfBox. (A) Principio della microscopia a riflessione interna totale (TIRF): la luce di eccitazione è diretta verso il bordo dell'obiettivo (Obj.) e subisce una riflessione interna totale all'interfaccia coverslip-buffer generando un campo di evanescenza in decadimento esponenziale per eccitare le molecole attaccate alla superficie. (B) Microscopia confocale: le molecole che si diffondono liberamente sono eccitate da un punto vicino alla diffrazione focalizzato nel campione. (C) La configurazione smfBox utilizzata in questo protocollo, che mostra tutti i componenti chiave: fotodiodi a valanga (APD), beam splitter (BS), specchi dicroici (DM), filtri (F), specchi (M), obiettivo (O) e foro stenopeico (P). Fare clic qui per visualizzare una versione più grande di questa figura.
Più recentemente, le tecniche smFRET hanno incorporato due eccitazioni di colore, in cui si alternano laser che corrispondono alle lunghezze d'onda di eccitazione del donatore e dell'accettore5. Questo può essere fatto in uno dei due modi, il primo modulando i laser a onda continua sulla scala temporale KHz, che è nota come eccitazione laser alternata (ALEX)13,14. Il secondo metodo interlascia impulsi veloci sulla scala temporale MHz; questo è nanosecondo-ALEX15 o eccitazione interlacciata pulsata (PIE)16. In tutti questi approcci, le informazioni provenienti dal laser accettore portano al calcolo della cosiddetta stechiometria, che può discriminare tra molecole con una bassa efficienza FRET e quelle prive di un accettore (sia attraverso l'etichettatura incompleta o il fotosbiancamento). L'utilizzo di PIE/ns-ALEX dà inoltre accesso a durate fluorescenti a livello di singola molecola e le anisotropie possono essere misurate se accoppiate con ottiche polarizzanti. Questa combinazione di misurazioni è nota come rilevamento multiparametrico della fluorescenza (MFD)9.
Nonostante i numerosi vantaggi di smFRET, non è ampiamente utilizzato al di fuori dei laboratori specializzati a causa degli alti costi degli strumenti commerciali e della mancanza di alternative semplici e autocostruite. Una crescente tendenza verso lo sviluppo di microscopia opensource a basso costo è in corso e recentemente sono emerse altre piattaforme, tra cui Planktonscope17, OpenFlexure Microscope18, Flexiscope19, miCube20, liteTIRF21 e Squid22. Qui lo studio descrive il protocollo per l'utilizzo di smfBox, un set-up confocale economico di recente sviluppo in grado di misurare l'efficienza FRET tra due coloranti su singole molecole che si diffondono liberamente. Istruzioni dettagliate per la compilazione e tutto il software operativo necessario sono disponibili gratuitamente all'indirizzo: https://craggslab.github.io/smfBox/ 23. La disposizione ottica dell'smfBox è assemblata da componenti prontamente disponibili acquistati da produttori convenienti e ampiamente accessibili, mentre il corpo del microscopio (responsabile della maggior parte della spesa in un set-up confocale standard) è stato sostituito da una scatola personalizzata in alluminio anodizzato a tenuta di luce leggera (che consente di effettuare misurazioni in condizioni di luce ambientale). Questa scatola ospita componenti ottici chiave, tra cui l'eccitazione dicroica, l'obiettivo e il foro stenopeico, e un interblocco laser meccanico, che ne consente il funzionamento sicuro come prodotto laser di Classe I (vedere la Figura 1C per uno schema completo). SmfBox utilizza ALEX per convalidare la stechiometria del colorante e per determinare accurati fattori di correzione FRET. Viene gestito utilizzando un software open source scritto su misura (smOTTER), che controlla tutti gli aspetti dell'acquisizione dei dati e restituisce i dati nel formato open source fotone-HDF524, compatibile con molti strumenti di analisi di terze parti. L'smfBox e i protocolli di acquisizione e analisi dei dati sono stati recentemente testati contro >20 altri strumenti (sia confocali che TIRF) in uno studio multi-lab in cieco25. Le efficienze FRET ottenute erano in ottimo accordo con tutti gli altri strumenti, nonostante l'smfBox costasse solo una frazione del prezzo delle configurazioni disponibili in commercio.
Qui, viene delineato un protocollo passo-passo per acquisire e analizzare efficienze FRET accurate e assolute su duplex di DNA a diffusione libera utilizzando smfBox, dall'accensione, attraverso l'allineamento e la messa a fuoco, alla raccolta e all'analisi dei dati. I campioni utilizzati qui sono tre DNA duplex (che presentano efficienze FRET alte, medie e basse, vedi Tabella 1) che sono stati valutati nello studio mondiale sui ciechi25; tuttavia, il metodo è adattabile a molti sistemi molecolari, comprese le proteine e altri acidi nucleici. La speranza è che un protocollo così dettagliato, insieme alle istruzioni di costruzione già esistenti per smfBox23, contribuirà a rendere questa potente tecnica ancora più accessibile a una vasta gamma di laboratori.
Protocollo
1. Componenti di accensione
- Accensione delle sei spine (nessun ordine particolare): Piezoconcept (z-focus), APD0, APD1, laser verde, laser rosso e fotorivelatore.
2. Software 1-Configurazione sperimentale
- Avviare il centro di controllo laser.
- Assicurarsi che la modalità Onda continua - Corrente costante alternata (CW-ACC) sia selezionata.
- Accendi entrambi i laser.
- Controllare/impostare le potenze del laser.
NOTA: la potenza del laser dovrà essere regolata come misurato appena prima dell'eccitazione dicroica, poiché i filtri ND e gli splitter del fascio nel percorso di eccitazione ridurranno la potenza del laser dal numero indicato sul pannello di controllo laser. I numeri qui indicati sono potenza all'eccitazione dicroica, ma la potenza sul controllo laser che corrisponde a questo dovrà essere elaborata.- Laser verde 220 μW (515 nm, 40 mW)
- μ70 W per laser rosso (638 nm, 10 mW)
- Software di acquisizione smOTTER
- Collega i laser, i rilevatori, lo z-stage e la fotocamera. Configurali correttamente (può variare a seconda delle impostazioni di una particolare configurazione della scheda NI).
3. Allineamento del percorso di emissione (non richiesto di routine)
- Impostazione dell'allineamento
- Pipettare 10 μL di colorante Cy3B libero (~100 nM) sul microscopio e messa a fuoco come descritto nei passaggi 4.3-4.5 di seguito.
NOTA: anche un altro colorante donatore con qualche perdita nel canale accettore funzionerebbe. - Aprire la scheda Allineamento in smOTTER, abbassare la potenza del laser e modificare la scala dell'asse y fino a quando non viene visualizzata la lettura dai rilevatori.
NOTA: l'obiettivo qui è aumentare il segnale, quindi potrebbe essere necessario cambiare nuovamente la scala dopo l'aumento del segnale. - Svitare le quattro viti nella parte anteriore dell'smfBox e rimuovere il pannello frontale.
- Pipettare 10 μL di colorante Cy3B libero (~100 nM) sul microscopio e messa a fuoco come descritto nei passaggi 4.3-4.5 di seguito.
- Allineamento foro stenopeico
- Ruotare la manopola x sul posizionatore stenopeico mentre si guarda il segnale nella scheda Allineamento, cercando di aumentare il segnale in verde e rosso.
- Ora ruota la manopola y per allineare il foro stenopeico nella direzione opposta.
- Tornare alla manopola x per verificare eventuali ulteriori aumenti di segnale.
- Allineamento foro stenopeico-lente
- Ruota la manopola dell'obiettivo x in una direzione, questo diminuirà il segnale.
- Ruotare la manopola stenopeica x nella stessa direzione per aumentare nuovamente il segnale.
- Se il nuovo segnale massimo è superiore a prima, continua a spostare iterativamente sia il foro stenopeico che l'obiettivo in quella direzione. Se è inferiore a prima, muoviti iterativamente nella direzione opposta.
- Ripeti sopra per y.
- Allineamento lenti APD
- A partire dall'APD verde, spostare la manopola x fino a quando il segnale verde non è al massimo.
- Ripetere l'operazione per la manopola y.
- Tornate alla manopola x, spostatevi avanti e indietro per trovare i punti di soglia in cui il segnale inizia a cadere e lasciatelo in una posizione a metà strada tra questi due punti.
- Ripetere l'operazione per la manopola x.
- Ripeti i passaggi precedenti per l'obiettivo APD rosso, osservando il segnale rosso.
- Riposizionare il pannello frontale sull'smfBox e sostituire le viti.
NOTA: Eseguire il mese di allineamento di cui sopra e come misura precauzionale se si sospetta che il microscopio abbia sviluppato un errore di allineamento tra le misurazioni.
4. Acquisizione dati di misura
- Per il primo campione, pulire il palco con tessuto per la pulizia delle lenti imbevuto di metanolo; pulire delicatamente l'obiettivo da un'estremità all'altra.
- Applicare 3-4 gocce di olio ad immersione per microscopia sul centro dell'obiettivo; ricostituire secondo necessità tra i campioni.
- Preparazione del campione
- Pipettare 10 μL di campione (primo utilizzo di acqua ultrapura di tipo I) al centro di un vetro di copertura pulito.
- Posizionare con attenzione il vetro di copertura sulla lente dell'obiettivo abbassandolo ad angolo rispetto all'olio per evitare di intrappolare bolle d'aria tra il vetro di copertura e l'obiettivo.
NOTA: Se necessario, far scorrere il vetro di copertura per spingere le bolle d'aria nell'olio lontano dal punto focale.
- Per eseguire esperimenti di eccitazione laser alternato (ALEX), configurare il laser nel pannello dei cicli di lavoro laser come segue.
- Donatore (laser verde) 0 off, 45 on, 55 off
- Accettore (laser rosso) 50 off, 45 on, 5 off
- Periodo ALEX (μs): 100
- Fare clic sulla scheda Z di messa a fuoco nella barra multifunzione; nel pannello di acquisizione, passare i laser a Live e avviare la fotocamera; regolare l'esposizione in modo che un punto luminoso appaia circondato centralmente dal nero.
NOTA: la fotocamera cattura la luce riflessa dall'interfaccia vetro/acqua tra il coperchio e il campione e il cerchio visibile di luce è al suo diametro più piccolo quando il punto focale si trova su questa interfaccia. È quindi necessario iniziare al di sotto di questa interfaccia, aumentare l'altezza z dell'interfaccia e quindi concentrarsi leggermente oltre sopra il coverslip e nel campione da misurare.- Partendo da una posizione Z bassa, aumentare l'altezza fino a quando il punto luminoso raggiunge la sua dimensione minima, quindi aumentare ulteriormente l'altezza fino a 20 μm per focalizzare il laser sopra l'olio e coprire il vetro e nel campione.
- Arrestare la fotocamera una volta a fuoco.
- Per confermare che la configurazione è stata eseguita in modo appropriato, monitorare la traccia di acqua ultrapura di tipo I per non vedere segnali di fluorescenza; ripetere la conferma di purezza per i tamponi in cui vengono preparati i campioni.
- Rimuovere con attenzione il vetro di copertura con pinzette di gomma per evitare di danneggiare l'obiettivo, quindi posizionare il campione di interesse preparato come sopra sull'obiettivo. Ricostituire l'olio di immersione se necessario per mantenere un'area uniforme di contatto tra il vetro di copertura e l'obiettivo (particolare attenzione all'area di contatto del campione sul vetro di copertura e all'area focale dell'obiettivo)
- Bingo di concentrazione
- Per garantire che si ottengano dati su una singola molecola, i campioni devono essere a una concentrazione alla quale si osservano 1-5 esplosioni al secondo nel pannello di tracce vive di smOTTER (questo riduce al minimo la possibilità che più di una molecola sia presente nel volume di rilevamento contemporaneamente). Effettuare le misurazioni quando è stata determinata la concentrazione appropriata.
- Per evitare l'evaporazione durante lunghi esperimenti, preparare una camera di campionamento ermetica. Premere gli isolatori in silicone (foro diametro 8-9 mm) verso il basso sul centro di un vetro di copertura (colorato con una punta della pipetta). Quindi, pipettare con cura un campione al centro, evitando qualsiasi contatto con il silicone. Quindi, posizionare un secondo vetro di copertura sulla parte superiore e premere per formare un sigillo.
- Controllare l'istogramma di stechiometria dal vivo rispetto all'efficienza FRET (ES) per vedere se il campione si sta comportando come previsto, cioè l'efficienza FRET prevista e la stechiometria ragionevole (~ 0,5).
- Quando l'esempio è pronto, immettete i dettagli dell'esperimento nel pannello delle impostazioni di salvataggio. Scegliere una directory e un nome file appropriati facendo clic sull'icona con i puntini di sospensione (file salvati in formato hdf5/h5) nel pannello delle impostazioni di salvataggio.
- Immettere le informazioni per il nome del campione, i dettagli del campione, le etichette del donatore e dell'accettore, le lunghezze d'onda di eccitazione del buffer, del donatore e dell'accettore, le lunghezze d'onda di rilevamento e la potenza del laser e l'affiliazione dell'utente e dell'utente.
- Tornate alla scheda Traccia in tempo reale nella barra multifunzione e nel pannello di acquisizione immettete la lunghezza dell'esperimento (in minuti), selezionate un intervallo di salvataggio appropriato per mitigare la potenziale perdita di dati in caso di errore. Selezionare Salva potenze laser, se necessario.
- Premere Start per acquisire i dati.
NOTA: per consentire una determinazione accurata del FRET, sarà necessario misurare almeno due campioni contenenti gli stessi coloranti donatore e accettore, ma con efficienze FRET sufficientemente diverse.
5. Analisi/software 2
- Avvia jupyter notebook con il pacchetto python FRETBursts (le istruzioni di configurazione sono disponibili qui: https://craggslab.github.io/smfBox/anasoftware.html)
- Se non è necessaria la correzione per le efficienze FRET assolute (ad esempio, solo le variazioni relative in FRET sono sufficienti per la misurazione) avviare Jupyter notebook FRET Analysis 1.4 Uncorrected.ipynb. Usalo per esportare figure o dati come file csv per ulteriori analisi o grafici in altri software.
NOTA: Ogni notebook contiene istruzioni dettagliate per guidare l'utente attraverso le procedure di analisi. Per una discussione più dettagliata delle procedure di analisi, inclusi gli algoritmi di ricerca burst, la correzione in background e tutti i parametri di correzione vedere23,25. - Se è necessaria una correzione per le efficienze FRET assolute, iniziare calcolando i parametri di diafonia alfa (perdita di emissione del donatore nel canale accettore) e delta (proporzione di eccitazione diretta dell'accettore sotto eccitazione del donatore).
- Per prima cosa avviare Jupyter notebook Correction Factor Finder Alpha-Delta.ipynb e determinare i parametri alfa e delta.
NOTA: Se questi sono incoerenti tra i campioni, il microscopio potrebbe aver sviluppato un problema di allineamento tra le misurazioni o gli spettri dei coloranti differiscono tra i campioni. - Se i parametri alfa e delta sono coerenti, avviare Jupyter notebook Correction Factor Finder Gamma-Beta.ipynb per determinare i parametri gamma e beta (che tengono conto delle differenze nell'eccitazione e nell'efficienza di rilevamento tra i coloranti).
NOTA: Se il grafico gamma e beta non si adatta bene, il microscopio potrebbe aver sviluppato un problema di allineamento tra le misurazioni, o le rese quantistiche, o i coefficienti di estinzione dei coloranti differiscono tra i campioni. - Con i quattro parametri di diafonia determinati, questi fattori possono essere utilizzati nel notebook FRET Analysis 1.4 Corrected.ipynb Jupyter per determinare le efficienze FRET assolute.
- Per prima cosa avviare Jupyter notebook Correction Factor Finder Alpha-Delta.ipynb e determinare i parametri alfa e delta.
6. Risoluzione dei problemi
- Se tutti i segnali sono bassi o i conteggi per burst sono inferiori al previsto (questo dipende dal colorante, ma per ATTO-550 e ATTO-647N su smfBox i valori tipici sono compresi tra 50 e 100 conteggi per ms durante un burst a singola molecola), riallineare l'smfBox.
- Ponte tra popolazioni doppiamente e singolarmente etichettate sull'istogramma ES.
NOTA: Questo può essere causato sia lavorando a una concentrazione troppo alta (rimediare a questo diluendo il campione), sia dalla fotosbiancamento, che può essere causato dall'utilizzo di una potenza laser troppo elevata (rimediare riducendo la potenza del laser). - Se la velocità di scoppio diminuisce durante l'esperimento (è probabile che le molecole marcate fluorescenti aderiscano al coperchio di vetro), utilizzare una maggiore concentrazione di BSA per rettificare.
- Se la velocità di scoppio aumenta durante l'esperimento (il campione è probabile che evapori), preparare una camera di campionamento ermetica come descritto sopra.
- Se il segnale si blocca a zero durante l'allineamento (il software è probabilmente sopraffatto dal segnale dei rilevatori), abbassare la potenza del laser o utilizzare un campione di allineamento più diluito.
- Se Z-focus mostra anelli concentrici (può accadere se sono stati posizionati più coverslip sull'obiettivo), verificare la presenza di più coverslip sull'obiettivo.
- Se il campione contiene raffiche luminose e lunghe di stechiometrie intermedie (causate da molecole aggregate), utilizzare detergenti o modificare i protocolli di purificazione del campione.
NOTA: ottenere un buffer pulito può essere un problema, poiché alcuni componenti del buffer contengono spesso quantità molto piccole di contaminanti moderatamente fluorescenti che sono sufficienti per presentarsi come esplosioni di colore singolo sulla traccia temporale. Se c'è troppo di questo nel buffer, allora può coincidere con esplosioni di campioni e modificare l'efficienza FRET o la stechiometria misurata. BSA in particolare può spesso essere problematico in questo senso, quindi è utile esporre una soluzione BSA stock a una forte fonte di luce per fotosbiancare i contaminanti.
Risultati
Il protocollo richiede una valutazione critica delle condizioni sperimentali durante l'installazione (vedere il passaggio 4.8 del protocollo). I primi risultati acquisiti che determinano il successo o il fallimento dell'esperimento sono raggiunti in questa fase. Un risultato positivo sarebbe quello di avere tra cinque e una raffica al secondo (vedi Figura 2B,C). Un risultato negativo sarebbe avere troppe (Figura 2A) o troppo poche esplosioni (Figura 2D) in quel lasso di tempo. Rimane possibile in questa fase correggere questi errori: un campione con una concentrazione troppo elevata deve semplicemente essere diluito; se la concentrazione è troppo bassa, tuttavia, potrebbe essere necessario preparare un nuovo campione (il determinante è se rimane possibile a questa bassa concentrazione raccogliere dati in un lasso di tempo ragionevole).

Figura 2: Schermate da tracce dal vivo durante la configurazione sperimentale che mostrano diverse concentrazioni di campioni di DNA duplex doppiamente etichettati. (A) troppo alto, (B) limite superiore accettabile, (C) concentrazione target, (D) troppo basso. I conteggi dei fotoni (contenitori da 1 ms) sono mostrati nei tre canali di rilevamento; emissione del donatore dopo l'eccitazione del donatore (DD), emissione dell'accettore dopo l'eccitazione del donatore (DA) ed emissione dell'accettore dopo l'eccitazione dell'accettore (AA). Fare clic qui per visualizzare una versione più grande di questa figura.
Un sistema statico a singola specie richiederebbe in genere da 30 a 60 minuti di misurazione per ottenere i ~ 1.000 burst necessari per una solida analisi dei dati. Il periodo di tempo e il numero di raffiche richieste aumenteranno con più specie o sistemi dinamici. Dopo la raccolta e l'analisi dei dati utilizzando il protocollo, le figure vengono esportate dai notebook Jupyter. Il grafico di alternanza (Figura 3A) deve corrispondere al periodo ALEX della configurazione sperimentale. La traccia temporale (Figura 3B) viene utilizzata per valutare qualitativamente che la concentrazione del campione è ragionevole. Il grafico di sfondo (Figura 3C) mostra la distribuzione dei periodi di ritardo interfotoni con un adattamento lineare ai tempi più lunghi per stimare il tasso di sfondo26. La traccia di sfondo (Figura 3D) può identificare se ci sono stati cambiamenti nel campione per tutta la durata dell'esperimento; principalmente ciò sarebbe dovuto all'evaporazione durante tempi di acquisizione più lunghi. Gli istogrammi ES sono generati per tutti i fotoni (Figura 3E) e le specie doppiamente etichettate (Figura 3F). Infine, viene generato un istogramma 1D E (Figura 3G) con adattamento gaussiano dei dati di burst.

Figura 3: Esempio di output di dati analizzati generati dai Jupyter Notebooks. (A) Grafico di alternanza, (B) Traccia temporale, (C) Determinazione dello sfondo, (D) Tassi di sfondo, (E) Istogramma ES di tutti i fotoni, (F) Istogramma ES a doppio canale e (G) Istogramma 1D E. Fare clic qui per visualizzare una versione più grande di questa figura.
| Nome | Sequenza |
| 1a | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAA CTC ACA AAC AGA CC - 5' | |
| 1b | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAA CTC ACA AAC AGA CC - 5' | |
| 1c | 5'- GAG CTG AAA GTG TCG AGT TTG TTT GAG TGT TTG TCT GG - 3' |
| 3'- CTC GAC TTT CAC AGC TCA AAC AAA CTC ACA AAC AGA CC - 5' |
Tabella 1: Sequenze di DNA utilizzate nel protocollo. I nucleotidi sono evidenziati in blu e rosso che rappresentano i residui di timina modificata con aminoAcidi C2 etichettati rispettivamente con Atto-550 e Atto-647N.
Correction Factor Finder Alpha-Delta: Fare clic qui per scaricare questo file.
Correction Factor Finder Gamma-Beta: Fare clic qui per scaricare questo file.
ANALISI FRET 1.4 Corretta: Fare clic qui per scaricare questo file.
ANALISI FRET 1.4 Non corretta: Fare clic qui per scaricare questo file.
Discussione
I passaggi più critici del protocollo sono l'allineamento del microscopio e la regolazione della concentrazione del campione alla corretta diluizione. Se l'allineamento è disattivato, potrebbe esserci un segnale insufficiente per identificare le esplosioni e tracciare gli istogrammi, e se si verifica un disallineamento tra i campioni, la correzione FRET accurata potrebbe fallire a causa di cambiamenti nelle perdite e nelle efficienze di rilevamento / eccitazione. Anche l'uso di una concentrazione appropriata è importante, una concentrazione troppo alta darà esplosioni coincidenti, contenenti più molecole con efficienze FRET potenzialmente diverse o che etichettano stechiometrie. Una concentrazione troppo bassa darà troppo poche esplosioni per una solida analisi dei dati.
Il protocollo qui descritto è per misurare le distanze in singole specie fret statiche. Se il campione ha più di un picco nell'istogramma di efficienza FRET, o i picchi appaiono ampi (cosa che può accadere con le specie dinamiche), potrebbero essere necessari più burst per adattare gli istogrammi allo stesso grado di precisione. Per due picchi ben separati saranno necessari circa il doppio dei dati, ma se le popolazioni si sovrappongono leggermente, sono necessari ancora più dati.
Se le due popolazioni si interconvertono sulla scala temporale dell'esperimento, la dinamica e la cinetica del sistema possono potenzialmente essere determinate. Test come BVA27 e 2CDE28 possono confermare che i burst intermedi sono di natura dinamica, mentre le analisi tra cui dPDA29,30 o H2MM31 possono determinare i tassi di interconversione. I notebook Jupyter per BVA e 2CDE sono disponibili sul sito Web FRETBursts26 e il software basato su MATLab PAM32 può eseguire analisi BVA, 2CDE e PDA.
La singola molecola confocale FRET può facilmente osservare stati molto più brevi (~1 ms) rispetto al TIRF; tuttavia, i brevi tempi di osservazione, limitati dalla diffusione, non forniscono alcuna storia molecolare, e quindi non possono determinare tempi di permanenza più lunghi, o reti di transizione complesse nel modo in cui possono fare gli esperimenti immobilizzati in superficie.
Poiché il protocollo misura molecole liberamente diffuse a una concentrazione molto bassa, funziona meglio quando si misurano le distanze intramolecolari sulla stessa molecola. Le distanze intermolecolari tra molecole legate transitoriamente possono essere misurate a condizione che il Kd delle due molecole sia abbastanza basso da far sì che il complesso esista in quantità significativa alla bassa concentrazione di lavoro richiesta dall'esperimento (~100 pM). Se il Kd è molto più alto di questo, allora si vedranno solo molecole marcate singolarmente. Questo problema può essere superato utilizzando la microfluidica per mescolare i due componenti etichettati insieme ad alta concentrazione e quindi diluire rapidamente e fluire sull'obiettivo prima che il complesso si dissocia33,34.
Misurare l'efficienza FRET a livello di singola molecola ha un vantaggio significativo rispetto alle tecniche di ensemble, in quanto informa su sottopopolazioni eterogenee, che in un esperimento di ensemble sarebbero mediate. Inoltre, fret a singola molecola con ALEX dà accesso a efficienze FRET accurate, che possono essere convertite in distanze accurate. Ciò consente di determinare informazioni strutturali più dettagliate piuttosto che semplicemente sondare i cambiamenti relativi alla distanza. L'smfBox offre tutti questi vantaggi e capacità, ma può essere costruito con un budget molto inferiore rispetto a microscopi comparabili disponibili in commercio in grado di smFRET23 confocale.
L'smfBox rappresenta una barriera all'ingresso molto più bassa per le tecniche smFRET, consentendo ai ricercatori di misurare i cambiamenti conformazionali e le distanze accurate all'interno e tra le proteine e gli acidi nucleici7,8,9,10,11,35.
Divulgazioni
Gli autori non dichiarano interessi concorrenti.
Riconoscimenti
Gli autori riconoscono con gratitudine le seguenti fonti di finanziamento: BBSRC (BB/T008032/1); EPSRC (Studentship to B.A.) e MRC (Studentship to A. R.-T.).
Materiali
| Name | Company | Catalog Number | Comments |
| Amino modified oligonucleotide | Eurogentec | N/A | May be ordered from various suppliers or synthesised; amino modification enables labeling with NHS-ester modified dyes |
| Avalanche photodiode (APD) | Excelitas | SPCM-AQRH-14 | Two APDs are required for the smfBox setup |
| Bovine Serum Albumin (BSA) | Merck | A2153 | System dependant; imaging buffer component (0.1 mg/mL in buffer) |
| Compact Laser Combiner | OMICRON | LightHUB-2 | 515 nm (80 mW) and 638 nm (100 mW) lasers |
| Coverglass | VWR | 630-2742 | Thickness: 0.17 ± 0.01 mm, LxW: 22x22 mm |
| Cy3B | Cytiva | PA63101 | 1 mg, PA63100 (5 mg), PA96106 (25 mg) |
| FRETBursts Python Package | N/A | N/A | Open-source python package for burst analysis of freely-diffusing single-molecule FRET data: https://fretbursts.readthedocs.io |
| Imaging Buffer | N/A | System dependant; 5 mM NaCl, 20 mM MgCl2, 5 mM Tris pH 7.5 and 0.1 mg/mL BSA | |
| Immersion Oil | Olympus | IMMOIL-F30CC | |
| Jupyter notebooks | Project Jupyter | N/A | Open-source web application to create and share documents that contain live code, equations, visualizations and text; data analysis notebooks for smfBox can be found in the SI |
| Lens Tissue | ThorLabs | MC-5 | MC-50E is same item in bulk |
| Magnesium Chloride | Merck | M2670 | System dependant; imaging buffer component (20 mM in buffer) |
| MilliQ/Ultrapure water | N/A | ||
| Nanopoistioner | Piezoconcept | FOC300 | Nanopositioner for accurate positioning of microscope objective |
| NHS-ester modified ATTO-550 | ATTO-TEC | AD 550-31 | 1 mg, AD 550-35 (5 mg) |
| NHS-ester modified ATTO-647N | ATTO-TEC | AD 647N-31 | 1 mg, AD 647N-35 (5 mg) |
| Objective lens | Olympus | N1480700 | Olympus objective series from orignal smfBox discontinued; replaced by N5702300 |
| OMICRON Control Center (OCC)- laser control center | OMICRON | N/A | v3.5.34 - OMICRON laser driver software |
| Press-To-Seal silicone isolator | Grace Bio-Labs | 664201 | 8-9 mm Diameter x 1.7 mm Depth |
| smOTTER | N/A | N/A | Open-source acquisition software for the Craggs Lab smfBox: https://github.com/craggslab/smOTTER |
| Sodium Chloride | Merck | S7653 | System dependant; imaging buffer component (5 mM in buffer) |
| Tris base | Merck | 93362 | System dependant; imaging buffer component (5 mM, pH 7.5 in buffer) |
| Type I ultrapure water | Merck | ZIQ7000T0 | Milli-Q® IQ 7000 Ultrapure Water System |
Riferimenti
- Forster, T. Intermolecular energy migration and fluorescence. Annals of Physics. 437 (1-2), 55-75 (1948).
- Stryer, L., Haugland, R. P. Energy transfer: a spectroscopic ruler. Proceedings of the National Academy of Sciences. 58 (2), 719-726 (1967).
- Hohlbein, J., et al. Conformational landscapes of DNA polymerase I and mutator derivatives establish fidelity checkpoints for nucleotide insertion. Nature Communications. 4 (1), 2131 (2013).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), (2018).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chemical Society Reviews. 43 (4), 1156-1171 (2014).
- Lerner, E., et al. The FRET-based structural dynamics challenge -- community contributions to consistent and open science practices. arXiv. , (2020).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nature Methods. 14 (2), 174-180 (2017).
- Craggs, T. D., et al. Substrate conformational dynamics facilitate structure-specific recognition of gapped DNA by DNA polymerase. Nucleic Acids Research. 47 (20), 10788-10800 (2019).
- Tsytlonok, M., et al. Dynamic anticipation by Cdk2/Cyclin A-bound p27 mediates signal integration in cell cycle regulation. Nature Communications. 10 (1), 1676 (2019).
- Nagy, J., et al. Complete architecture of the archaeal RNA polymerase open complex from single-molecule FRET and NPS. Nature Communications. 6 (1), 6161 (2015).
- LeBlanc, S. J., et al. Coordinated protein and DNA conformational changes govern mismatch repair initiation by MutS. Nucleic Acids Research. 46 (20), 10782-10795 (2018).
- Segal, M., et al. High-throughput smFRET analysis of freely diffusing nucleic acid molecules and associated proteins. Methods. 169, 21-45 (2019).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: Analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences. 101 (24), 8936-8941 (2004).
- Kapanidis, A. N., et al. Alternating-laser excitation of single molecules. Accounts of Chemical Research. 38 (7), 523-533 (2005).
- Müller, B. K., Zaychikov, E., Brauchle, C., Lamb, D. C. Pulsed interleaved excitation. Biophysical Journal. 89 (5), 3508-3522 (2005).
- Laurence, T. A., Kong, X., Jager, M., Weiss, S. Probing structural heterogeneities and fluctuations of nucleic acids and denatured proteins. Proceedings of the National Academy of Sciences of the United States of America. 102 (48), 17348-17353 (2005).
- Pollina, T., et al. PlanktonScope: Affordable modular imaging platform for citizen oceanography. bioRxiv. , 056978 (2020).
- Collins, J. T., et al. Robotic microscopy for everyone: the OpenFlexure microscope. Biomedical Optics Express. 11 (5), 2447-2460 (2020).
- Courtney, A., Alvey, L. M., Merces, G. O. T., Burke, N., Pickering, M. The Flexiscope: a low cost, flexible, convertible and modular microscope with automated scanning and micromanipulation. Royal Society Open Science. 7 (3), 191949 (2020).
- Martens, K. J. A., et al. Visualisation of dCas9 target search in vivo using an open-microscopy framework. Nature Communications. 10 (1), 3552 (2019).
- Auer, A., et al. Nanometer-scale multiplexed super-resolution imaging with an economic 3D-DNA-PAINT microscope. ChemPhysChem. 19 (22), 3024-3034 (2018).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , 424613 (2020).
- Ambrose, B., et al. The smfBox is an open-source platform for single-molecule FRET. Nature Communications. 11 (1), 5641 (2020).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An open file format for timestamp-based single-molecule fluorescence experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-a multi-laboratory benchmark study. Nature Methods. 15 (9), 669-676 (2018).
- Ingargiola, A., Lerner, E., Chung, S., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing single-molecule FRET. PLOS One. 11 (8), 0160716 (2016).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule FRET experiments with burst variance analysis. Biophysical Journal. 100 (6), 1568-1577 (2011).
- Tomov, T. E., et al. Disentangling subpopulations in single-molecule FRET and ALEX experiments with photon distribution analysis. Biophysical Journal. 102 (5), 1163-1173 (2012).
- Santoso, Y., Torella, J. P., Kapanidis, A. N. Characterizing single-molecule FRET dynamics with probability distribution analysis. ChemPhysChem. 11 (10), 2209-2219 (2010).
- Kalinin, S., Valeri, A., Antonik, M., Felekyan, S., Seidel, C. A. M. Detection of structural dynamics by FRET: A photon distribution and fluorescence lifetime analysis of systems with multiple states. The Journal of Physical Chemistry B. 114 (23), 7983-7995 (2010).
- Pirchi, M., et al. Photon-by-photon hidden Markov model analysis for microsecond single-molecule FRET kinetics. The Journal of Physical Chemistry B. 120 (51), 13065-13075 (2016).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. PAM: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophysical Journal. 114 (7), 1518-1528 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry B. 122 (49), 11554-11560 (2018).
- Bennet, I. A., et al. Regional conformational flexibility couples substrate specificity and scissile phosphate diester selectivity in human flap endonuclease 1. Nucleic Acids Research. 46 (11), 5618-5633 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon