Method Article
Quantificando a organização do colágeno fibrilar com ferramentas baseadas em curvas
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para usar uma ferramenta de software MATLAB baseada em curvas baseada em curvas, de código aberto para quantificar a organização de colágeno fibrilar na matriz extracelular de tecidos normais e doentes. Esta ferramenta pode ser aplicada a imagens com fibras de colágeno ou outros tipos de estruturas semelhantes a linhas.
Resumo
As colagens fibrilares são componentes proeminentes da matriz extracelular (ECM), e suas alterações na topologia têm se mostrado associadas à progressão de uma ampla gama de doenças, incluindo câncer de mama, ovário, rim e pâncreas. As ferramentas de software de quantificação de fibras disponíveis livremente são focadas principalmente no cálculo do alinhamento ou orientação da fibra, e estão sujeitas a limitações como a exigência de etapas manuais, imprecisão na detecção da borda de fibra em fundo barulhento ou falta de caracterização de recursos localizados. A ferramenta de quantitação de fibra de colágeno descrita neste protocolo é caracterizada pelo uso de uma representação de imagem multiescala ideal habilitada pela curvelet transform (CT). Esta abordagem algorítmica permite a remoção do ruído de imagens de colágeno fibrilar e o aprimoramento das bordas de fibra para fornecer informações de localização e orientação diretamente de uma fibra, em vez de usar as informações indiretas em termos de pixels ou janelas obtidas de outras ferramentas. Esta estrutura baseada em CT contém dois pacotes separados, mas vinculados, chamados "CT-FIRE" e "CurveAlign" que podem quantificar a organização de fibras em uma base global, região de interesse (ROI) ou fibra individual. Esta estrutura de quantificação foi desenvolvida há mais de dez anos e agora evoluiu para uma plataforma abrangente e orientada pelo usuário de quantificação de colágeno. Usando esta plataforma, pode-se medir até cerca de trinta recursos de fibra, incluindo propriedades individuais de fibra, como comprimento, ângulo, largura e retidão, bem como medidas em massa, como densidade e alinhamento. Além disso, o usuário pode medir o ângulo de fibra em relação a limites segmentados manualmente ou automaticamente. Esta plataforma também fornece vários módulos adicionais, incluindo os para análise de ROI, criação automática de limites e pós-processamento. O uso desta plataforma não requer experiência prévia de programação ou processamento de imagens, e pode lidar com grandes conjuntos de dados, incluindo centenas ou milhares de imagens, permitindo quantificação eficiente da organização de fibras de colágeno para aplicações biológicas ou biomédicas.
Introdução
As collagens fibrilares são componentes ECM proeminentes e estruturais. Suas mudanças de organização afetam a função tecidual e provavelmente estão associadas à progressão de muitas doenças que vão desde a osteogênese imperfeita1,disfunção cardíaca2, e cicatrização de feridas3 a diferentes tipos de câncer, incluindo mama4,5,6,ovário7,8,rim9e cânceres pancreáticos10. Muitas modalidades de imagem estabelecidas podem ser usadas para visualizar colágeno fibrilar, como microscopia de segunda geração harmônica11,manchas ou corantes em conjunto com microscopia de campo brilhante ou fluorescência ou microscopia de luz polarizada12,microscopia de polarização baseada em cristal líquido (LC-PolScope)13, e microscopia eletrônica14. À medida que a importância da organização do colágeno fibrilar tornou-se mais clara, e o uso desses métodos aumentou, a necessidade de melhores abordagens de análise de fibra de colágeno também cresceu.
Tem havido muitos esforços para desenvolver métodos computacionais para medição automatizada do colágeno fibrilar. As ferramentas de software disponíveis gratuitamente são focadas principalmente no cálculo do alinhamento ou orientação da fibra, adotando o primeiro tensor derivativo ou estrutura para pixels15,16ou análise de espectro baseado em transformação fourier para telhas de imagem17. Todas essas ferramentas estão sujeitas a limitações como a exigência de etapas manuais, imprecisão na detecção da borda de fibra em fundo barulhento ou falta de caracterização de características localizadas.
Em comparação com outras ferramentas de software livre de código aberto disponíveis livremente, os métodos descritos neste protocolo usam CT — um método ideal, multiescala e direcional de representação de imagens — para remover o ruído de imagens de colágeno fibrilar e melhorar ou rastrear bordas de fibra. Informações sobre localização e orientação podem ser fornecidas diretamente de uma fibra, em vez de usar as informações indiretas em termos de pixels ou janelas para inferir as métricas da organização da fibra. Esta estrutura baseada em CT18,19,20,21 pode quantificar a organização de fibras em uma base global, ROI ou fibra, principalmente através de dois pacotes separados, mas vinculados, chamados "CT-FIRE"18,21 e "CurveAlign"19,21. No que diz respeito à implementação do software, no CT-FIRE, coeficientes ct em múltiplas escalas podem ser usados para reconstruir uma imagem que melhore as bordas e reduza o ruído. Em seguida, um algoritmo individual de extração de fibras é aplicado à imagem reconstruída pela TC para rastrear fibras para encontrar seus pontos centrais representativos, estendendo os ramos de fibra dos pontos centrais e ligando galhos de fibra para formar uma rede de fibras. Em CurveAlign, coeficientes de TC em uma escala especificada pelo usuário podem ser usados para rastrear a orientação local da fibra, onde a orientação e os locais das curvas são extraídos e agrupados para estimar a orientação da fibra nos locais correspondentes. Essa estrutura de quantificação resultante foi desenvolvida há mais de dez anos e evoluiu muito em muitos aspectos, como funcionalidade, interface do usuário e modularidade. Por exemplo, esta ferramenta pode visualizar a orientação local de fibras e permite que o usuário meça até trinta recursos de fibra, incluindo propriedades individuais de fibra, como comprimento, ângulo, largura e retidão, bem como medidas a granel, como densidade e alinhamento. Além disso, o usuário pode medir o ângulo de fibra em relação a limites segmentados manualmente ou automaticamente, o que, por exemplo, desempenha um papel importante no desenvolvimento de biomarcadores baseados em imagem no câncer de mama22 e estudos de câncer de pâncreas10. Esta plataforma fornece vários módulos de recursos, incluindo os para análise de ROI, criação automática de limites e pós-processamento. O módulo ROI pode ser usado para anotar diferentes formas de ROI e realizar análises correspondentes de ROI. Como exemplo de aplicação, o módulo de criação de limites automáticos pode ser usado para registrar imagens de campo brilhante de hematoxilina e eosina (H&E) com imagens de segunda geração harmônica (SHG) e gerar a máscara de imagem dos limites do tumor a partir das imagens de H&E registradas. O módulo pós-processamento pode ajudar a facilitar o processamento e integração de arquivos de dados de saída a partir de imagens individuais para possíveis análises estatísticas.
Esta plataforma de quantificação não requer experiência prévia de programação ou processamento de imagens e pode lidar com grandes conjuntos de dados, incluindo centenas ou milhares de imagens, permitindo quantificação eficiente da organização de colágeno para aplicações biológicas ou biomédicas. Tem sido amplamente utilizado em diferentes campos de pesquisa por muitos pesquisadores em todo o mundo, incluindo nós mesmos. Existem quatro publicações principais no CT-FIRE e CurveAlign18,19,20,21, das quais as três primeiras foram citadas 272 vezes (a partir de 2020-05-04 de acordo com o Google Scholar). Uma revisão das publicações que citavam esta plataforma (CT-FIRE ou CurveAlign) indica que há aproximadamente 110 artigos de revistas que a utilizaram diretamente para sua análise, nas quais aproximadamente 35 publicações colaboravam com nosso grupo, e as outras (~ 75) foram escritas por outros grupos. Por exemplo, esta plataforma foi utilizada para os seguintes estudos: câncer de mama22,23,24, câncer de pâncreas10,25, câncer de rim9,26, cicatrização de feridas3,27,28,29,30, câncer de ovário8,31,7, ligamento uterosacral32, hipofosfatomico dentin33, carcinoma basal celular34, sarcoma hipoxic35, tecido cartilagem36, disfunção cardíaca37, neurônios38, glioblastoma39, contrações linfáticas40,cacffolds fibrosas41,câncer gástrico42, microtúbulo43, e fibrose bexiga44. A Figura 1 demonstra a aplicação de imagem de câncer do CurveAlign para encontrar as assinaturas de colágeno associadas ao tumor do câncer de mama19 a partir da imagem shg. A Figura 2 descreve um fluxo de trabalho esquemático típico desta plataforma. Embora essas ferramentas tenham sido revisadas tecnicamente18,19,21, e um protocolo regular20 para análise de alinhamento com CurveAlign também está disponível, um protocolo visual que demonstra que todas as características essenciais poderiam ser úteis. Um protocolo visualizado, como aqui apresentado, facilitará o processo de aprendizagem do uso desta plataforma, bem como abordará de forma mais eficiente preocupações e perguntas que os usuários possam ter.
Protocolo
NOTA: Este protocolo descreve o uso de CT-FIRE e CurveAlign para quantificação de colágeno. Essas duas ferramentas têm objetivos complementares, mas diferentes, principais e estão ligadas em conjunto até certo ponto. O CT-FIRE pode ser lançado a partir da interface CurveAlign para realizar a maioria das operações, exceto para análise avançada de pós-processamento e ROI. Para uma operação completa de CT-FIRE, deve ser lançado separadamente.
1. Coleta de imagens e exigência de imagem
NOTA: A ferramenta pode processar qualquer arquivo de imagem com estruturas semelhantes a linhas legíveis pelo MATLAB, independentemente da modalidade de imagem usada para recolhê-lo.
- Use escala de cinza de 8 bits como o tipo de imagem, pois os parâmetros de execução padrão são baseados neste formato.
NOTA: A imagem SHG é um método de imagem de colágeno fibrilar livre de rótulos e de alta resolução. Imagens shg de um estudo de câncer de mama19 serão usadas aqui com o propósito de demonstração.
2. Instalação de software e exigência do sistema
NOTA: Ambas as versões autônomas e de código-fonte estão disponíveis gratuitamente. A versão de código-fonte requer uma instalação COMPLETA DO MATLAB, incluindo caixas de ferramentas de processamento de sinal, processamento de imagem, análise de estatísticas e computação paralela. Para executar a versão de código-fonte, todas as pastas necessárias, incluindo algumas das fontes de terceiros, devem ser adicionadas ao caminho MATLAB. O uso do aplicativo autônomo (APP) é recomendado para a maioria dos usuários, o que requer a instalação de um MCR (Matlab Compiler Runtime, compilador de MATLAB) livremente disponível da versão especificada. O procedimento de instalação e lançamento do APP está descrito abaixo.
- Baixe os pacotes de APP CT-FIRE versão 3.0 (CTF3.0) e CurveAlign Versão 5.0 (CA5.0) de https://eliceirilab.org/software/ctfire/ e https://eliceirilab.org/software/curvealign/, respectivamente.
NOTA: Cada pacote inclui as imagens autônomas de APP, manual e teste. - Siga os requisitos detalhados e instruções de instalação dos sites acima para instalar o MATLAB MCR 2018b.
- Inicie o APP.
- Para um sistema com Windows de 64 bits, clique duas vezes no ícone APP para lançá-lo.
- Para um sistema Mac, siga as seguintes etapas para lançá-lo: Clique com o botão direito do mouse no APP (ctrl-click) | Mostrar conteúdo do pacote | Conteúdo | | MacOS applauncher (clique com o botão direito do mouse e escolha aberto).
NOTA: Outros detalhes podem ser vistos nos sites de software listados em 2.1.
3. Extração individual de fibra com CT-FIRE
NOTA: CT-FIRE usa CT para denoizar a imagem, melhorar as bordas de fibra e, em seguida, usa um algoritmo de extração de fibras para rastrear fibras individuais. Comprimento, ângulo, largura e retidão são calculados para fibras individuais.
- CT-FIRE em imagem única ou múltiplas imagens
- Inicie o APP conforme descrito em 2.3.
- Clique no botão Arquivo Aberto(s) na interface principal do usuário gráfico (GUI, Figura 3A)e selecione uma ou mais pilhas de imagens/ou imagens na janela prompt. Use a técnica apropriada para o sistema operacional para selecionar várias imagens na caixa de diálogo (por exemplo, no Windows, segure CTRL enquanto seleciona vários arquivos).
NOTA: Se dois ou mais arquivos de imagem forem selecionados, todas as imagens devem usar os mesmos parâmetros de execução para a análise. Certifique-se de que todas as imagens sejam adquiridas sob as mesmas condições ou similares. - Selecione opções de computação paralela verificando a caixa de seleção Paralela no canto superior direito para análise de múltiplas imagens.
- Para a pilha de imagens, mova o controle deslizante de fatias sob a caixa de lista de arquivos para selecionar a fatia a ser analisada.
- Defina propriedades em execução. Use parâmetros padrão para uma análise inicial de algumas imagens. Se estiver usando parâmetros padrão, pule para a etapa 3.1.6. Para definir diferentes parâmetros, clique no botão Atualizar no painel Parâmetros. Siga o manual para ajustar corretamente os parâmetros.
NOTA: Os parâmetros mais ajustados incluem o limiar de fundo(thresh_im2)e o raio de busca de nucleação(s_xlinkbox). Se o nível de ruído de fundo for alto, defina thresh_im2 para um valor maior; s_xlinkbox está associada ao raio médio das fibras, estabelecendo um valor menor para detectar fibras finas. - Clique no botão Executar.
NOTA: As informações de progresso serão exibidas tanto na janela de informações quanto na janela de comando. Após a conclusão da análise, a tabela de saída será exibida(Figura 3B). - Clique em qualquer item na tabela de saída para ver o histograma de medidas de fibra(Figura 3C e Figura 3F) da imagem, incluindo comprimento, largura, ângulo e retidão.
NOTA: As imagens de fibra com fibras sobrepostas na imagem original também serão exibidas(Figura 3E). - Verifique a subpasta chamada ctFIREout sob a pasta de imagem para obter os arquivos de saída, incluindo o arquivo ".tiff" de imagem sobreposta, o arquivo ".csv" e o arquivo ".mat".
- Análise da região de interesse do CT-FIRE (ROI)
- Anotação do ROI usando o ROI Manager
- Clique no botão Arquivo Aberto(s) na GUI principal (Figura 3A) para carregar uma ou mais imagens.
- Selecione a imagem a ser anotada na lista de arquivos.
- Selecione o GERENCIADOR de ROI no menu suspenso do painel Opções de ROI.
- Clique no botão RUN para iniciar o módulo ROI Manager (Figura 3A).
- No gui gerenciador do ROI(Figura 4A),clique no menu suspenso abaixo Do Menu do ROI(d)para desenhar os ROIs, um a um.
NOTA: A forma do ROI pode ser retângulo, à mão livre, elipse, polígono ou retângulo especificado. Siga as instruções na tela para desenhar, salvar e deixar a anotação do ROI. - Depois de selecionar o método para desenhar o ROI, arraste o retângulo amarelo que aparece na imagem original para a posição desejada e clique no botão Save ROI(s) ou pressione a tecla s para adicionar este ROI à lista do ROI. Este ROI será automaticamente nomeado.
- Desenhe um novo ROI arrastando o ROI anterior para uma nova posição e salve-o como mencionado em 3.2.1.6, ou repita os passos 3.2.1.5-3.2.1.6 para desenhar um novo ROI.
- Pressione x ou selecione Novo ROI? no menu suspenso de forma de ROI para sair da anotação do ROI.
- Verifique as caixas de seleção Mostrar tudo e rótulos para mostrar todos os ROIs definidos na lista e seus nomes na imagem original.
- Selecione o ROI na lista do ROI para realizar operações básicas de ROI, incluindo ROI de renome, Delete ROI, Save ROI Text, Load ROI from Text, Save ROI Mask, Load ROI from Maske Combine ROIs.
- Verifique o arquivo de saída do gerenciador de ROI salvo como arquivo ".mat" em uma subpasta chamada ROI_management sob a pasta de imagem original.
- Para anotar outra imagem na lista de arquivos abertos, repita as etapas 3.2.1.2-3.2.1.11.
- Após a anotação, feche a GUI do gerenciador de ROI e reinicie a gui principal clicando no botão Redefinir na GUI principal.
- Análise de ROI para uma única imagem no ROI Manager
- Se a análise CT-FIRE de imagem completa for conduzida e os resultados forem salvos no diretório padrão, clique em um ou mais ROIs na lista de ROI e clique no botão ctFIRE ROI Analyzer para iniciar o módulo de análise Pós-ROI.
NOTA: Os resultados serão salvos automaticamente em uma subpasta localizada em \\[pasta de imagem]\ CTF_ROI\Individual\ROI_post_analysis\. - Na janela pop-up, clique no botão Verificar fibras para exibir fibras dentro dos ROIs selecionados (Figura 4B).
- Clique em Plot Statistics para exibir histogramas de cada ROI (Figura 4C). As figuras de saída correspondentes serão exibidas.
- Se a análise CT-FIRE da imagem completa não tiver sido realizada, clique em um ou mais ROIs na lista do ROI e clique no botão Aplicar ctFIRE no ROI para aplicar diretamente a análise CT-FIRE aos ROIs selecionados.
- Siga as instruções na janela de solicitação para executar a análise.
NOTA: Os parâmetros para execução do CT-FIRE são passados pela GUI principal, e o usuário pode atualizar os parâmetros de execução conforme descrito na etapa 3.1.5, conforme necessário. Após a conclusão da análise, as estatísticas sumárias das medidas de fibra serão exibidas na tabela de saída. Os resultados serão salvos automaticamente em uma subpasta localizada em \\[pasta de imagem]\ CTF_ROI\Individual\ROI_analysis\.
- Se a análise CT-FIRE de imagem completa for conduzida e os resultados forem salvos no diretório padrão, clique em um ou mais ROIs na lista de ROI e clique no botão ctFIRE ROI Analyzer para iniciar o módulo de análise Pós-ROI.
- Análise de ROI para múltiplas imagens usando o ROI Analyzer
- Siga os passos em 3.2.1 para anotar ROIs para que as imagens sejam analisadas.
- Abra uma ou mais imagens clicando no botão Arquivo Aberto(s).
- Para executar a análise pós-ROI quando os resultados completos de análise de imagem estiverem disponíveis, clique no menu suspenso no painel Opções de execução e selecione a opção analisador pós-ROI ctf.
- Clique no botão EXECUTAR para executar a análise do ROI para todas as imagens carregadas.
- Verifique as informações de progresso exibidas na janela da mensagem na parte inferior da GUI e na janela de comando.
- Após a conclusão da análise, verifique as estatísticas de resumo de cada ROI exibido em uma tabela de saída.
NOTA: Os arquivos de saída detalhados são salvos automaticamente em uma subpasta localizada em \\ [pasta de imagem]\CTF_ROI\Batch\ROI_post_analysis\. - Para fazer uma análise direta quando os resultados completos da análise de imagem não estiverem disponíveis, siga as etapas 3.2.3.1-3.2.3.6, exceto que na etapa 3.2.3.3, selecione a opção CTF ROI analisarr; na etapa 3.2.3.4, antes de clicar no botão RUN, atualize os parâmetros de execução conforme descrito na etapa 3.1.5. Depois de clicar no botão RUN, em uma janela de diálogo imediata, escolha entre ROI Retangular e máscara ROI de qualquer forma.
NOTA: Se todos os ROIs anotados forem retangulares, o usuário poderá escolher o "ROI Retangular". Na etapa 3.2.3.6, os resultados da análise do ROI são salvos em uma subpasta localizada em \\[pasta de imagem]\CTF_ROI\Batch\ROI_analysis\.
- Anotação do ROI usando o ROI Manager
- Pós-processamento com CT-FIRE
NOTA: Após a análise regular do CT-FIRE descrita em 3.1, o usuário pode realizar mais pós-processamento. Sem executar a extração de fibras demorada novamente, o pós-processamento regular, descrito em 3.3.1, pode atualizar algumas propriedades básicas da figura de saída, enquanto o pós-processamento avançado descrito em 3.3.2 pode visualizar fibras individuais e suas propriedades, realizar limiar complexo entre todas as quatro propriedades de fibras, gerar estatísticas sumárias das fibras selecionadas e visualizar as fibras selecionadas usando um mapa de cores personalizado.- Pós-processamento regular com CT-FIRE
- Inicie o aplicativo CT-FIRE ou clique no botão Redefinir após outras operações para inicializar o GUI principal CT-FIRE (Figura 3A).
- Verifique a caixa de seleção .mat na parte superior da GUI principal.
- Clique no botão Arquivo Aberto para selecionar o arquivo CT-FIRE output .mat na subpaste ctFIREout.
NOTA: Se vários arquivos forem selecionados, a caixa de seleção Batch será verificada automaticamente. O nome do arquivo das imagens correspondentes será exibido na lista de caixas. - Atualize as opções no painel Controle de Figura de Saída.
- Mantenha as opções padrão nas Opções de Saída,o que garantirá que todos os arquivos de saída serão atualizados de acordo com o novo conjunto de parâmetros definidos em 3.3.1.4.
- Clique no botão Pós-processamento. Verifique as informações de progresso na janela de mensagens na parte inferior da GUI principal, bem como na janela de comando.
- Após a conclusão da análise, clique em qualquer item da tabela de saída para ver o histograma das medidas de fibra da imagem, incluindo comprimento, largura, ângulo e retidão.
NOTA: Novos arquivos de saída substituirão os antigos na subpastos ctFIREout.
- Pós-processamento avançado de CT-FIRE
- Inicie o aplicativo CT-FIRE ou clique no botão Redefinir após outras operações para inicializar o GUI principal CT-FIRE (Figura 3A).
- Verifique a caixa de seleção OUT.adv na parte superior da GUI principal (Figura 3A).
- Clique no botão Pós-processamento para iniciar o avançado GUI pós-processamento chamado " Módulo deAnálise" (Figura 5A).
- Clique no botão Selecionar Arquivo para selecionar uma imagem.
- Clique no botão Visualise Fibers para inserir o número de fibra com base nas etiquetas na figura de guia Origina-fibras.
NOTA: As medidas das fibras selecionadas serão exibidas em uma tabela de saída (Figura 5B), e as fibras correspondentes serão sobrepostas na imagem original mostrada na figura da guia denominada Fibras Medidas (Figura 5C). - Clique no botão Confirmar/Atualizar para passar para a operação de limiar.
- Verifique a caixa de limiar para habilitar as configurações de limiar.
- Escolha uma das quatro opções de limiar no menu suspenso.
- Digite os limiares desejados no painel Limiares para uma ou mais propriedades de fibra.
- Clique no botão Limiar Agora para aplicar as condições de limiar acima.
- Verifique a figura pronta com seu nome terminando com visualização de métricas para ver as fibras selecionadas sobrepostas nas imagens originais com os mapas de cores personalizados, como mostrado na Figura 5E.
- Repetir passos 3.3.2.9-3.3.2.11 para definir os limites desejáveis.
- Clique no botão Salvar fibras para salvar as informações de fibra selecionadas.
NOTA: As fibras selecionadas correspondentes serão exibidas na figura da guia nomeada Após o Limiar. - Clique no botão Gerar estatísticas e clique no botão OK na janela pop-up para gerar estatísticas de resumo.
NOTA: Uma tabela de saída(Figura 5D) mostrará o valor médio das fibras selecionadas. Outras estatísticas das fibras selecionadas serão salvas em um arquivo excel cujo local é exibido na janela de status na parte inferior desta GUI. - Para incluir as informações de fibra individuais selecionadas no arquivo de saída, verifique a folha Gerar para caixa de dados bruta antes de clicar no botão OK.
- Para combinar resultados de várias imagens, na etapa 3.3.2.4, verifique a caixa de modo de lote ou modo de pilha e selecione as várias imagens ou pilhas a serem analisadas; pular as etapas 3.3.2.5-3.3.2.6. Nas etapas 3.3.2.8-3.3.2.9, defina condições de limiar, mas como os botões limiar agora e Save Fibers estão desativados, pule as etapas 3.3.2.10-3.3.2.13; e, por último, siga as instruções da etapa 3.3.2.14 para gerar estatísticas sumárias e propriedades individuais de fibras das fibras selecionadas.
- Pós-processamento regular com CT-FIRE
4. Análise de fibras com CurveAlign
NOTA: O CurveAlign foi inicialmente desenvolvido para medir automaticamente ângulos de fibras em relação aos limites definidos pelo usuário. A versão atual do CurveAlign pode ser usada para avaliação em massa de recursos baseados em densidade e alinhamento, além da medição relativa do ângulo, carregando as informações individuais de fibra extraídas pelo CT-FIRE ou usando diretamente a orientação local das curvas. O CurveAlign calcula até trinta características relacionadas a características globais ou locais, incluindo principalmente densidade e alinhamento, bem como propriedades individuais de fibra quando o CT-FIRE é adotado como o método de rastreamento de fibras.
- Análise de fibras com curvelets
- Inicie o APP conforme descrito em 2.3.
- Clique no botão Redefinir para redefinir o APP ao seu status inicial se outras operações tiverem sido realizadas.
- Na gui principal (Figura 6A),verifique a opção método de análise de fibra para certificar-se de que a CT está selecionada (opção padrão).
NOTA: Neste modo, a TC é realizada na imagem, e a orientação de cada curvelet representa a direção de uma fibra no local correspondente. - Clique no menu suspenso do método Limite e selecione o modo de processamento de limite das seguintes opções de menu suspenso: Sem limite, limite CSV e limite TIFF.
NOTA: Se não for necessário um limite, pule esta etapa. Consulte 4.3 para saber como calcular ângulos de fibra em relação a um limite. - Clique no botão Obter imagem(s) na GUI principal(Figura 6A)e, em seguida, selecione uma ou mais pilhas de imagens/ou imagens na janela prompt. Use a técnica apropriada para o seu sistema operacional para selecionar várias imagens na caixa de diálogo (por exemplo, no Windows, segure CTRL enquanto seleciona vários arquivos).
NOTA: Se dois ou mais arquivos de imagem forem selecionados, todas as imagens devem usar os mesmos parâmetros de execução para análise. Certifique-se de que todas as imagens sejam adquiridas sob as mesmas condições ou similares. - Para a pilha de imagens, mova o controle deslizante de fatias sob a caixa de lista de arquivos para selecionar a fatia a ser analisada.
- Digite a Fração de coefs para manter. Este valor é a fração dos maiores coeficientes de TC que serão utilizados na análise de fibras.
NOTA: Se a imagem tiver uma grande variação na intensidade ou contraste da fibra, anote regiões de interesse com até mesmo contraste para a análise da fibra, pois este modo só detecta as fibras mais brilhantes de uma imagem. Além disso, quanto maior for o tamanho da imagem, defina um valor menor para esta fração. - Mantenha todos os parâmetros nas Opções de Saída e outros na opção Avançado como padrão; os arquivos de saída podem ser necessários em outras operações futuras.
- Clique no botão Executar na parte inferior da GUI principal (Figura 6A).
NOTA: As informações de progresso serão exibidas em uma janela de mensagem destacada na cor verde na parte inferior. Após o processo ser feito, algumas estatísticas de resumo de cada imagem serão exibidas na tabela de saída (Figura 6B), e todos os arquivos de saída serão salvos automaticamente em uma subpasa chamada CA_Out no diretório da imagem original.s. - Clique em qualquer item na tabela de saída (Figura 6B) para ver o histograma (Figura 6E) ou o gráfico da bússola(Figura 6F) dos ângulos de fibra.
NOTA: A imagem de sobreposição(Figura 6C) e o mapa de calor(Figura 6D) de alinhamento ou ângulo também serão exibidos. - Clique no botão Redefinir para executar outras operações ou feche a GUI principal para sair do APP.
- Análise individual de fibras com CT-FIRE
NOTA: O procedimento é o mesmo descrito na seção 4.1 exceto que na etapa 4.1.3, selecione o modo de análise de fibras relacionadas ao CT-FIREe pule a etapa 4.1.7, pois não é aplicável e é desativado no modo CT-FIRE. Especificamente, na etapa 4.1.3, selecione um entre os seguintes três métodos de análise de fibras individuais baseados em CT-FIRE:- Selecione As fibras CT-FIRE para usar o ponto central de fibra e o ângulo de fibra para representar a fibra.
NOTA: Esta opção não considera as mudanças na orientação da fibra ao longo do comprimento da fibra. - Selecione os pontos finais CT-FIRE para usar os dois pontos finais de uma fibra e ângulo de fibra correspondente para representar a fibra.
NOTA: Em comparação com 4.2.1, esta opção usa duas posições para representar uma fibra em vez de uma (ponto central da fibra). - Selecione segmentos CT-FIRE para usar segmentos de uma fibra para representar a fibra.
NOTA: Cada segmento tem um comprimento igual (definido para 5 pixels por padrão no CT-FIRE) bem como sua orientação e localização, o que reflete a mudança de orientação ao longo de toda a extensão da fibra. Essa opção seria a mais demorada, mas seria a melhor opção entre os três métodos de análise de fibras baseados em CT-FIRE para acompanhar mudanças na orientação local de uma fibra curvilínea.
- Selecione As fibras CT-FIRE para usar o ponto central de fibra e o ângulo de fibra para representar a fibra.
- Análise de alinhamento relativo com limite
NOTA: Em comparação com a análise regular sem condições de limite descritas nas seções 4.2 e 4.3, a análise de alinhamento relativo com condições de limite precisa do seguinte:- Na etapa 4.1.3, selecione a condição de limite de tiff.
NOTA: O usuário precisará de um arquivo de limite correspondente para cada imagem ou cada pilha. Siga as instruções na tela para anotar manualmente um arquivo de limite CSV (formato de valores separados por círias baseado, coordenadas x-y) ou um arquivo de limite Tiff. Os arquivos de limite criados no CurveAlign serão salvos automaticamente de acordo com o diretório de arquivos e convenções de nomeação de arquivos descritos no manual. Se um par de imagens de campo brilhante e SHG forem fornecidas, use o módulo de criação de limite automático descrito na seção 4.4 para gerar o arquivo de limite. - No painel Parâmetros Primários, insira a distância do limite mais próximo para avaliar apenas as fibras dentro dessa distância.
- No painel Opções de saída, verifique a caixa de associação de limite Bdry Assoc para visualizar o ponto no limite que está associado a uma fibra, segmento de fibra ou curvelet.
- Na etapa 4.1.3, selecione a condição de limite de tiff.
- Criação automática de limites
- Inicie o APP conforme descrito em 2.3.
- Clique no botão Redefinir para redefinir o APP ao seu status inicial se outras operações já tiverem sido realizadas.
- Clique no botão BD Creation para iniciar o módulo de criação de limites automáticos.
- Siga as instruções/pistas na tela para criar arquivo de limite para uma ou mais imagens com base em um par de imagens de campo brilhante e SHG de H&E.
- Feche a janela do módulo ou clique no botão Redefinir na GUI principal (Figura 6A)para sair deste módulo.
- Região de análise de juros da CurveAlign
- Anotação do ROI usando o ROI Manager
- Clique no botão Obter imagem(s) na GUI principal (Figura 6A) para carregar uma ou mais imagens.
- Selecione a imagem a ser anotada na lista de arquivos.
- Clique no ROI Manager para lançar o módulo ROI Manager ( Figura7A).
- Siga os passos 3.2.1.5-3.2.1.13 na seção 3.2.1.
- Análise de ROI para uma única imagem no ROI Manager
- Se a análise do CurveAlign de imagem completa tiver sido conduzida e os resultados forem salvos no diretório padrão, clique em um ou mais ROIs na lista de ROI e clique no botão ANALISADOr de ROI do CA para executar a análise pós-ROI.
NOTA: Após a conclusão da análise, as estatísticas sumárias serão exibidas em uma tabela de saída (Figura 7C) bem como uma figura histograma(Figura 7D) mostrando a distribuição do ângulo. - Clique em qualquer item da tabela de saída para visualizar as fibras em um determinado ROI (Figura 7B)bem como o histograma dos ângulos de fibra.
- Verifique os arquivos de saída salvos em uma subpasta localizada em \\[pasta de imagem]\ CA_ROI\Individual\ROI_post_analysis\.
- Se a análise de CA de imagem completa não tiver sido conduzida, clique em um ou mais ROIs na lista de ROI e clique no botão Aplicar CA no ROI para aplicar diretamente a análise de CA nos ROIs selecionados. Siga as instruções na janela de solicitação para executar a análise.
NOTA: Os parâmetros para execução da análise de CA são passados pela gui principal; atualizar os parâmetros de execução descritos na etapa 4.1.7 conforme necessário. Após a conclusão da análise, os resultados das estatísticas sumárias das medidas de fibra serão exibidos na tabela de saída. Os resultados serão salvos automaticamente em uma subpasta localizada em \\[pasta de imagem]\ CA_ROI\Individual\ROI_analysis\.
- Se a análise do CurveAlign de imagem completa tiver sido conduzida e os resultados forem salvos no diretório padrão, clique em um ou mais ROIs na lista de ROI e clique no botão ANALISADOr de ROI do CA para executar a análise pós-ROI.
- Análise de ROI para múltiplas imagens usando o ROI Analyzer
- Siga os passos em 4.5.1 para anotar ROIs para que as imagens sejam analisadas.
- Abra uma ou mais imagens clicando no botão Get Image(s).
- Para executar a análise pós-ROI quando os resultados completos de análise de imagem estiverem disponíveis, clique no botão análise do ROI e selecione a opção ROI pós-processamento.
- Verifique as informações de progresso exibidas na janela da mensagem na parte inferior da GUI e na janela de comando.
- Após a conclusão da análise, verifique as estatísticas de resumo de cada ROI exibido em uma tabela de saída.
NOTA: Os arquivos de saída detalhados são salvos automaticamente em uma subpasta localizada em \\ [pasta de imagem]\CA_ROI\Batch\ROI_post_analysis\. - Para executar uma análise direta com o modo CT quando os resultados completos da análise de imagem não estiverem disponíveis, siga as etapas 4.5.3.1-4.5.3.5, exceto para as seguintes alterações: modifique a etapa 4.5.3.3 selecionando a opção CA no ROI retangular cortado ou CA na máscara com ROI de qualquer forma. Se todos os ROIs anotados forem de forma retangular, escolha a opção ROI Retangular. Após a etapa 4.5.3.2, atualize os parâmetros em execução, conforme descrito em 4.1.7.
NOTA: Os resultados da análise do ROI serão salvos em uma subpasta localizada em \\ [pasta de imagem]\CA_ROI\Batch\ROI_analysis\.
- Anotação do ROI usando o ROI Manager
- Pós-processamento do CurveAlign
- Inicie o APP conforme descrito na seção 2.3.
- Clique no botão Redefinir para redefinir o APP ao seu status inicial se outras operações já tiverem sido realizadas.
- Clique no botão Pós-Processamento para iniciar o módulo pós-processamento.
- Siga as instruções/pistas na tela para combinar os recursos de saída ou valores de diferentes imagens.
- Feche a janela do módulo ou clique no botão Redefinir na GUI principal (Figura 6A)para sair deste módulo.
5. Tempo estimado de execução
- Aguarde o tempo estimado de funcionamento para processar uma imagem com tamanho de 1024 pixels x 1024 pixels com densidade de fibra moderada. O tempo computacional real geralmente depende de vários fatores, incluindo o tamanho do arquivo, o modo de análise, os recursos a serem implantados, o tipo de CPU (Central Processing Unit, unidade de processamento central) e a quantidade de memória de acesso aleatório disponível (ou RAM). A extração individual de fibras CT-FIRE leva alguns minutos. O modo CT CurveAlign sem limite leva alguns segundos. O modo de fibra ct-fire de ajuste de curvas ou extremidades de fibra sem limite leva dezenas de segundos. O modo de fibra CT-FIRE curveAlign sem limite leva centenas de segundos. Análise CurveAlign com limite leva dezenas de segundos a vários minutos, dependendo da complexidade dos limites.
Resultados
Esses métodos têm sido aplicados com sucesso em inúmeros estudos. Algumas aplicações típicas incluem: 1) Conklin et al.22 usaram CurveAlign para calcular assinaturas de colágeno associadas ao tumor, e descobriram que as fibras de colágeno estavam mais frequentemente alinhadas perpendicularmente ao perímetro do ducto em lesões de carcinoma ductal in situ (DCIS); 2) Drifka et al.10 utilizaram o modo CT-FIRE em CurveAlign para quantificar o alinhamento de colágeno estromamal para adenocarcinoma ductal pancreático e tecidos de pancreatite normal/crônica, e descobriram que houve um alinhamento aumentado nos tecidos cancerígenos em comparação com o dos tecidos normais/crônicos; 3) Alkmin et al.7 usaram o CurveAlign para quantificar a distribuição angular das fibras F-actin e o alinhamento geral do colágeno a partir das imagens shg do colágeno estromagênico ovariano, e mostraram que a morfologia matricial desempenha um papel importante na motilidade celular e alinhamento f-actin; 4) LeBert et al.3 aplicaram CT-FIRE nas imagens shg de um modelo de reparação de feridas de zebrafish e encontraram um aumento na espessura das fibras de colágeno após ferimentos agudos; 5) Devine et al.45 utilizaram o modo CT-FIRE em CurveAlign para imagens shg de colágeno de dobra vocal de diferentes modelos animais para medir propriedades individuais de fibra e alinhamento geral, e mostraram que o colágeno porcino e canino da dobra vocal tinha maior alinhamento e retidão inferiormente; 6) Keikhosravi et al.13 usaram o CurveAlign para quantificar o alinhamento do colágeno em amostras histopatologias imagens com LC-PolScope, e mostraram que LC-PolScope e SHG são comparáveis em termos de alinhamento e medição de orientação para alguns tipos de tecido.

Figura 1: Usando o CurveAlign para encontrar assinaturas de colágeno associadas ao tumor a partir de imagens de SHG de uma microarray de tecido de câncer de mama humano (TMA). (A) Imagem de sobreposição de um núcleo TMA com imagem SHG (amarela) sobreposta na imagem de campo brilhante correspondente de H&E. (B) A região de interesse de (A). (C) A imagem de campo brilhante de (B). (D) A imagem shg de (B). (F) A máscara associada à imagem de campo brilhante(C). (E) A imagem de sobreposição da saída CurveAlign mostrando os limites do tumor (amarelo) de (F), locais de fibra representativo e orientação (linhas verdes); as linhas azuis são usadas para associar fibras com seus limites mais próximos. As setas verdes em (B) e (E) mostram as fibras paralelas ao limite do tumor, enquanto as setas vermelhas lá mostram as fibras perpendiculares ao limite. A barra de escala em (A) equivale a 200 μm. As imagens em(B)–(F) são exibidas na mesma escala, e a barra de escala representativa em(D)é igual a 50 μm. Abreviaturas: SHG = segunda geração harmônica; H&E = hematoxilina e eosina. Clique aqui para ver uma versão maior desta figura.

Figura 2: Fluxo de trabalho esquemático de quantificação de uma imagem de colágeno fibrilar. (A) Imagem SHG a ser analisada por CT-FIRE e/ou CurveAlign. (B) Sobreposição de saída de imagem por CT-FIRE. (C) O limite da máscara de (A) é uma entrada curvealign opcional. (D) Sobreposição de saída de imagem por CurveAlign. As linhas coloridas em (B) representam as fibras extraídas. Em (D), as linhas verdes indicam os locais e orientações das fibras que estão fora dos limites (linhas amarelas) e estão dentro da distância especificada de seus limites mais próximos, as linhas vermelhas são as de outras fibras, e as linhas azuis são usadas para associar fibras com seus limites mais próximos. As imagens em (A)–(D) são exibidas na mesma escala, e a barra de escala representativa em (A) é igual a 200 μm. Por favor clique aqui para ver uma versão maior desta figura.

Figura 3: Análise regular ct-fire. (A) Gui principal. (B) Tabela de saída mostrando as estatísticas de resumo. (C) e (F) mostram os histogramas de ângulo e largura, respectivamente. (E) Imagem de saída mostrando as fibras extraídas (linhas de cor) sobrepostas na imagem SHG original(D). Abreviaturas: GUI = CT = curva transformada; interface gráfica do usuário; SHG = segunda geração harmônica. Clique aqui para ver uma versão maior desta figura.
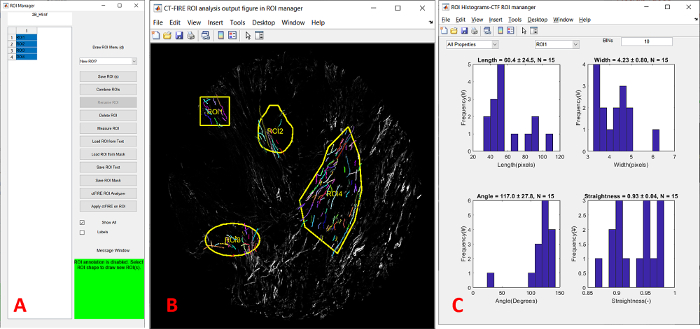
Figura 4: Módulo de gerenciamento do ROI CT-FIRE. (A) Módulo GUI. (B) ROI pós-análise de quatro ROIs com formas diferentes mostrando as fibras dentro de cada ROI. (C) Histogramas de ROI de diferentes propriedades de fibras. Abreviaturas: CT = curvelet transform; GUI = interface gráfica do usuário; ROI = região de interesse. Clique aqui para ver uma versão maior desta figura.

Figura 5: Módulo avançado de pós-processamento CT-FIRE. (A) Módulo GUI. (B) Medições de três fibras selecionadas. (C) Visualização das três fibras selecionadas em (B). (D) Estatísticas sumárias após a aplicação de um limite de comprimento (>60 pixels). (E) Visualização das fibras selecionadas em (D) com barra de cor baseada em comprimento. Abreviaturas: CT = curvelet transform; GUI = interface gráfica do usuário. Clique aqui para ver uma versão maior desta figura.

Figura 6: Análise regular do CurveAlign. (A) Gui principal. (B) Tabela de saída mostrando as estatísticas de resumo. (C) Imagem de saída mostrando os locais e orientação de fibras representativas (linhas verdes) e limites (linhas amarelas) sobrepostos na imagem original do SHG, as linhas azuis são usadas para associar fibras com seus limites mais próximos, linhas vermelhas mostram os locais e orientações das fibras dentro de um limite ou fora do limite (>usuário especificado distância, por exemplo, 250 pixels aqui). (D) Mapa de calor dos ângulos: vermelho (> 60 graus), amarelo (45-60] graus, verde (10, 45] graus. (E)–(F ) mostrar a distribuição de ângulo usando histograma e trama de bússola, respectivamente. Abreviaturas: GUI = interface gráfica do usuário; SHG = segunda geração harmônica. Clique aqui para ver uma versão maior desta figura.
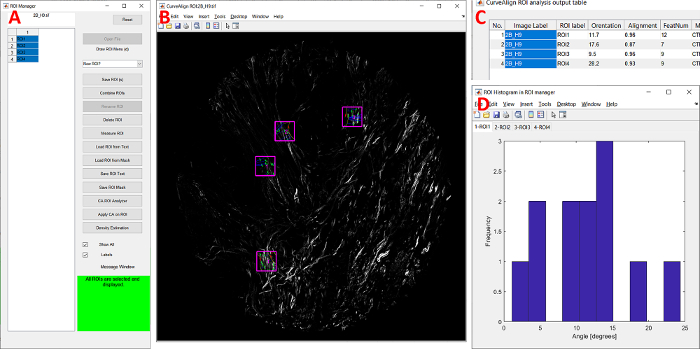
Figura 7: Módulo de gerenciamento do ROI CurveAlign. (A) O módulo GUI. (B) Quatro ROIs retangulares anotados sobrepostas na imagem original. (C) TABELA de saída pós-análise do ROI. (D) Ângulo histograma de cada ROI. Abreviaturas: ROI = região de interesse; GUI = interface gráfica do usuário. Clique aqui para ver uma versão maior desta figura.
Discussão
Este protocolo descreve o uso de CT-FIRE e CurveAlign para quantificação de colágeno fibrilar e pode ser aplicado a qualquer imagem com fibras de colágeno ou outras estruturas alongadas semelhantes a linhas ou fibras adequadas para análise por CT-FIRE ou CurveAlign. Por exemplo, elastina ou fibras elásticas podem ser processadas de forma semelhante nesta plataforma. Testamos ambas as ferramentas em fibras sintéticas geradas computacionalmente21. Dependendo do aplicativo, os usuários devem escolher o modo de análise mais adequado para seus dados. O modo de análise de fibras ct pode usar diretamente as curvas na TC para representar a localização e orientação da fibra, e é sensível às mudanças na estrutura de fibra local. O modo CT pode ser usado para localizar fibras e sua orientação em condições complexas, por exemplo, onde o nível de ruído é alto, a fibra é curvilínea, ou a variação na espessura da fibra é alta. No entanto, como o modo CT só capta as partes mais brilhantes de uma imagem, ele perderia algumas fibras com menor intensidade quando há uma grande variação na intensidade da imagem.
Além disso, o modo CT não fornece informações sobre fibras individuais. Em contraste com o modo CT, o modo CT-FIRE calcula propriedades individuais de fibras e pode analisar todas as fibras cuja intensidade está acima de um limiar especificado. Os desafios associados ao modo CT-FIRE incluem: 1) a precisão de uma extração de fibras intactas pode ser reduzida ou comprometida quando houver grande variação na intensidade ao longo de uma fibra ou na espessura da fibra através de uma imagem; e 2) a análise padrão atual é computacionalmente exigente como mencionado no protocolo. Mais detalhes sobre as vantagens e limitações desses métodos podem ser vistos em nossas publicações anteriores20,21.
No que diz respeito à precisão do rastreamento de fibras, o usuário pode contar principalmente com a inspeção visual para verificar a imagem sobreposta onde as fibras extraídas ou orientações representativas são sobrepostas na imagem original. Além disso, para CT-FIRE, o usuário pode usar o módulo avançado de pós-processamento para identificar as propriedades das fibras individuais selecionadas e compará-las com medições usando outras ferramentas de análise de imagem, como o Fiji46. Para o CurveAlign, o usuário pode comparar os resultados de orientação ou alinhamento com os calculados por outras ferramentas como OrientationJ16 e CytoSpectre17.
Entre os recursos disponíveis para saída pela plataforma, os recursos relacionados ao alinhamento são mais usados e são os mais confiáveis e robustos. Para usar recursos individuais de fibra, o usuário precisa confirmar a extração de recursos representativos de fibra. Note-se que uma fibra intacta pode ser dividida em vários segmentos mais curtos em algumas circunstâncias, que o usuário deve levar em consideração ao selecionar o modo de análise de fibras ou realizar uma análise estatística mais aprofundada. Mesmo quando o comprimento da fibra não pode ser usado diretamente como uma propriedade comparável, a orientação ou largura dos segmentos de fibra ponderados contra seus comprimentos ainda pode indicar informações úteis. No que diz respeito à imagem SHG, a abertura numérica (NA) da lente objetiva pode afetar significativamente a detecção da largura e comprimento de uma fibra, mas tem menos impacto nas medidas de orientação e alinhamento. Em nossa experiência em imagem SHG, geralmente precisamos usar lentes objetivas com ampliação de 40x ou superior com na ≥ 1.0 para obter uma medida robusta de espessura de fibra.
"Alinhamento" pode ser interpretado de três formas diferentes: 1) alinhamento com relação à direção horizontal positiva denominada "ângulo", variando de 0 a 180 graus, onde ângulos próximos a 0 têm orientação semelhante a ângulos próximos a 180 graus; 2) alinhamento em relação a um limite chamado "ângulo relativo", variando de 0 a 90 graus, com 0 graus indicando uma fibra paralela ao limite e 90 indicando uma fibra perpendicular; e 3) alinhamento das fibras em relação umas às outras denominadas "coeficiente de alinhamento", variando de 0 a 1, com 1 indicando fibras perfeitamente alinhadas e valores menores representando fibras mais distribuídas aleatoriamente.
Além das características de fibra calculadas nesta plataforma, algumas métricas baseadas na análise da textura47,48,49 também foram propostas para quantificar padrões de ECM. Esses recursos relacionados à textura podem fornecer um descritor alternativo ou adicional do ECM em algumas aplicações. Os desafios para o desenvolvimento desse tipo de métrica residem na interoperação da possível relevância biológica, caracterização localizada e na precisão do rastreamento de fibras individuais.
Para otimizar os parâmetros de execução e realizar a solução de problemas, o usuário pode consultar o manual, publicações relevantes20,21, bem como as barras laterais faq nas páginas do GitHub Wiki do repositório curvelets: https://github.com/uw-loci/curvelets/wiki. Para alguns botões, uma dica de função pode aparecer para orientar o usuário para a operação atual ou próxima quando o usuário move o ícone do mouse acima de um botão. Siga as informações na gui ou janela de comando para realizar a solução de problemas.
Para processar um grande conjunto de dados, o usuário é encorajado a usar opções de computação paralela, que permitem que a ferramenta processe várias imagens simultaneamente. Uma opção é usar vários núcleos de CPU se disponível no computador que está sendo utilizado. Alternativamente, uma versão sem cabeça de ambos os APPs é fornecida e foi compilada com sucesso no nó de compilação através do servidor mantido no50 Center for High Throughput Computing (CHTC) baseado em CONDOR na Universidade de Wisconsin-Madison. O fluxo de trabalho CHTC para quantificação de fibras em larga escala foi desenvolvido, testado e usado com sucesso em conjuntos de imagens reais que consistem em milhares de imagens. O usuário poderia adaptar as funções MATLAB sem cabeça tanto do CT-FIRE quanto do CurveAlign para executar quantificação em outros sistemas de computação em nuvem, incluindo serviços comerciais, como os oferecidos pela Amazon, Google e Microsoft.
As direções de desenvolvimento contínuas e futuras incluem: 1) incorporação de rede neural de aprendizagem profunda para extrair ou gerar imagens de fibra de colágeno sintético de alta qualidade e melhorar a robustez e precisão do algoritmo de rastreamento de fibras; 2) integração de todos os módulos em uma plataforma abrangente, otimizando o código e a documentação seguindo as melhores práticas de engenharia de software; 3) implantação de todos os recursos principais em uma plataforma de computação em nuvem; 4) aprimoramento do fluxo de trabalho da análise de fibras utilizando o serviço CHTC; e 5) melhoria da funcionalidade do gerador de fibra sintética.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos a muitos colaboradores e usuários ao CT-FIRE e CurveAlign ao longo dos anos, incluindo dr. Rob Nowak, Dr. Carolyn Pehlke, Dr. Jeremy Bredfeldt, Guneet Mehta, Andrew Leicht, Dr. Adib Keikhosravi, Dr. Matt Conklin, Dr. Jayne Squirrell, Dr. Paolo Provenzano, Dr. Brenda Ogle, Dr. Patricia Keely, Dr. Joseph Szulczewski, Dr. Suzanne Ponik e contribuições técnicas adicionais de Swati Anand e Curtis Rueden. Este trabalho foi apoiado por financiamento da Semiconductor Research Corporation, Morgridge Institute for Research, e nih grants R01CA199996, R01CA181885 e U54CA210190 para K.W.E.
Materiais
| Name | Company | Catalog Number | Comments |
| CT-FIRE | Univerity of Wisconsin-Madison | N/A | open source software available from https://eliceirilab.org/software/ctfire/ |
| CurveAlign | University of Wisconsin-Madison | N/A | open source software available from https://eliceirilab.org/software/curvealign/ |
Referências
- Nadiarnykh, O., et al. Second harmonic generation imaging microscopy studies of osteogenesis imperfecta. Journal of Biomedical Optics. 12 (5), 051805(2007).
- Kouris, N. A., et al. A nondenatured, noncrosslinked collagen matrix to deliver stem cells to the heart. Regenerative Medicine. 6 (5), 569-582 (2011).
- LeBert, D. C., et al. Matrix metalloproteinase 9 modulates collagen matrices and wound repair. Development. 142 (12), 2136-2146 (2015).
- Provenzano, P. P., et al. Collagen reorganization at the tumor-stromal interface facilitates local invasion. BMC Medicine. 4 (1), 38(2006).
- Provenzano, P. P., et al. Collagen density promotes mammary tumor initiation and progression. BMC Medicine. 6 (1), 1(2008).
- Conklin, M. W., et al. Aligned collagen is a prognostic signature for survival in human breast carcinoma. The American Journal of Pathology. 178 (3), 1221-1232 (2011).
- Alkmin, S., et al. Migration dynamics of ovarian epithelial cells on micro-fabricated image-based models of normal and malignant stroma. Acta Biomaterialia. 100, 92-104 (2019).
- Campbell, K. R., Campagnola, P. J. Assessing local stromal alterations in human ovarian cancer subtypes via second harmonic generation microscopy and analysis. Journal of Biomedical Optics. 22 (11), 116008(2017).
- Best, S. L., et al. Collagen organization of renal cell carcinoma differs between low and high grade tumors. BMC Cancer. 19 (1), 490(2019).
- Drifka, C. R., et al. Periductal stromal collagen topology of pancreatic ductal adenocarcinoma differs from that of normal and chronic pancreatitis. Modern Pathology. 28 (11), 1470-1480 (2015).
- Campagnola, P. J., et al. Three-dimensional high-resolution second-harmonic generation imaging of endogenous structural proteins in biological tissues. Biophysical Journal. 82 (1), 493-508 (2002).
- Arun Gopinathan, P., et al. Study of collagen birefringence in different grades of oral squamous cell carcinoma using picrosirius red and polarized light microscopy. Scientifica. 2015, 1-7 (2015).
- Keikhosravi, A., et al. Quantification of collagen organization in histopathology samples using liquid crystal based polarization microscopy. Biomedical Optics Express. 8 (9), 4243-4256 (2017).
- Quan, B. D., Sone, E. D. Cryo-TEM analysis of collagen fibrillar structure. Methods in Enzymology. 532, 189-205 (2013).
- Boudaoud, A., et al. FibrilTool, an ImageJ plug-in to quantify fibrillar structures in raw microscopy images. Nature Protocols. 9 (2), 457-463 (2014).
- Rezakhaniha, R., et al. Experimental investigation of collagen waviness and orientation in the arterial adventitia using confocal laser scanning microscopy. Biomechanics and Modeling in Mechanobiology. 11 (3-4), 461-473 (2012).
- Kartasalo, K., et al. CytoSpectre: a tool for spectral analysis of oriented structures on cellular and subcellular levels. BMC Bioinformatics. 16 (1), 1(2015).
- Bredfeldt, J. S., et al. Computational segmentation of collagen fibers from second-harmonic generation images of breast cancer. Journal of Biomedical Optics. 19 (1), 016007(2014).
- Bredfeldt, J. S., et al. Automated quantification of aligned collagen for human breast carcinoma prognosis. Journal of Pathology Informatics. 5 (1), 28(2014).
- Liu, Y., Keikhosravi, A., Mehta, G. S., Drifka, C. R., Eliceiri, K. W. Methods for quantifying fibrillar collagen alignment. Methods in Molecular Biology. 1627, 429-451 (2017).
- Liu, Y., et al. Fibrillar collagen quantification with curvelet transform based computational methods. Frontiers in Bioengineering and Biotechnology. 8, 198(2020).
- Conklin, M. W., et al. Collagen alignment as a predictor of recurrence after ductal carcinoma in situ. Cancer Epidemiology and Prevention Biomarkers. 27 (2), 138-145 (2018).
- Jallow, F., et al. Dynamic interactions between the extracellular matrix and estrogen activity in progression of ER+ breast cancer. Oncogene. 38 (43), 6913-6925 (2019).
- Smirnova, T., et al. Serpin E2 promotes breast cancer metastasis by remodeling the tumor matrix and polarizing tumor associated macrophages. Oncotarget. 7 (50), 82289(2016).
- Fanous, M., Keikhosravi, A., Kajdacsy-Balla, A., Eliceiri, K. W., Popescu, G. Quantitative phase imaging of stromal prognostic markers in pancreatic ductal adenocarcinoma. Biomedical Optics Express. 11 (3), 1354-1364 (2020).
- Jiménez-Torres, J. A., Virumbrales-Muñoz, M., Sung, K. E., Lee, M. H., Abel, E. J., Beebe, D. J. Patient-specific organotypic blood vessels as an in vitro model for anti-angiogenic drug response testing in renal cell carcinoma. EBioMedicine. 42, 408-419 (2019).
- Govindaraju, P., Todd, L., Shetye, S., Monslow, J., Puré, E. CD44-dependent inflammation, fibrogenesis, and collagenolysis regulates extracellular matrix remodeling and tensile strength during cutaneous wound healing. Matrix Biology. 75, 314-330 (2019).
- Henn, D., et al. Cryopreserved human skin allografts promote angiogenesis and dermal regeneration in a murine model. International Wound Journal. 17 (4), 925-936 (2020).
- Rico-Jimenez, J., et al. Non-invasive monitoring of pharmacodynamics during the skin wound healing process using multimodal optical microscopy. BMJ Open Diabetes Research and Care. 8 (1), 000974(2020).
- Israel, J. S., et al. Quantification of collagen organization after nerve repair. Plastic and Reconstructive Surgery Global Open. 5 (12), (2017).
- Rentchler, E. C., Gant, K. L., Drapkin, R., Patankar, M., Campagnola, P. J. Imaging collagen alterations in STICs and high grade ovarian cancers in the fallopian tubes by second harmonic generation microscopy. Cancers. 11 (11), 1805(2019).
- Hu, C., et al. Imaging collagen properties in the uterosacral ligaments of women with pelvic organ prolapse using spatial light interference microscopy (SLIM). Frontiers in Physics. 7, 72(2019).
- Guirado, E., et al. Disrupted protein expression and altered proteolytic events in hypophosphatemic dentin can be rescued by dentin matrix protein 1. Frontiers in Physiology. 11, 82(2020).
- Kiss, N., et al. Quantitative analysis on ex vivo nonlinear microscopy images of basal cell carcinoma samples in comparison to healthy skin. Pathology & Oncology Research. 25 (3), 1015-1021 (2019).
- Lewis, D. M., et al. Collagen fiber architecture regulates hypoxic sarcoma cell migration. ACS Biomaterials Science & Engineering. 4 (2), 400-409 (2018).
- Moura, C. C., Bourdakos, K. N., Tare, R. S., Oreffo, R. O., Mahajan, S. Live-imaging of Bioengineered Cartilage Tissue using Multimodal Non-linear Molecular Imaging. Scientific Reports. 9 (1), 1-9 (2019).
- Murtada, S. I., et al. Paradoxical aortic stiffening and subsequent cardiac dysfunction in Hutchinson-Gilford progeria syndrome. Journal of The Royal Society Interface. 17 (166), 0066(2020).
- Nichol, R. H., Catlett, T. S., Onesto, M. M., Hollender, D., Gómez, T. M. Environmental elasticity regulates cell-type specific RHOA signaling and neuritogenesis of human neurons. Stem Cell Reports. 13 (6), 1006-1021 (2019).
- Pointer, K. B., et al. Association of collagen architecture with glioblastoma patient survival. Journal of Neurosurgery. 126 (6), 1812-1821 (2016).
- Razavi, M. S., Leonard-Duke, J., Hardie, B., Dixon, J. B., Gleason, R. L. Axial stretch regulates rat tail collecting lymphatic vessel contractions. Scientific Reports. 10 (1), 1-11 (2020).
- Xue, Y., et al. Valve leaflet-inspired elastomeric scaffolds with tunable and anisotropic mechanical properties. Polymers for Advanced Technologies. 31 (1), 94-106 (2020).
- Zhou, Z. H., et al. Reorganized collagen in the tumor microenvironment of gastric cancer and its association with prognosis. Journal of Cancer. 8 (8), 1466(2017).
- Zinn, A., et al. The small GTPase RhoG regulates microtubule-mediated focal adhesion disassembly. Scientific Reports. 9 (1), 1-15 (2019).
- Zwaans, B. M., et al. Radiation cystitis modeling: A comparative study of bladder fibrosis radio-sensitivity in C57BL/6, C3H, and BALB/c mice. Physiological Reports. 8 (4), (2020).
- Devine, E. E., Liu, Y., Keikhosravi, A., Eliceiri, K. W., Jiang, J. J. Quantitative second harmonic generation imaging of leporine, canine, and porcine vocal fold collagen. The Laryngoscope. 129 (11), 2549-2556 (2019).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Zeitoune, A. A., et al. Epithelial ovarian cancer diagnosis of second-harmonic generation images: A semiautomatic collagen fibers quantification protocol. Cancer Informatics. 16, (2017).
- Wershof, E., et al. Matrix feedback enables diverse higher-order patterning of the extracellular matrix. PLoS Computational Biology. 15 (10), 1007251(2019).
- Mostaço-Guidolin, L. B., et al. Fractal dimension and directional analysis of elastic and collagen fiber arrangement in unsectioned arterial tissues affected by atherosclerosis and aging. Journal of Applied Physiology. 126 (3), 638-646 (2019).
- Thain, D., Tannenbaum, T., Livny, M. Distributed computing in practice: the Condor experience. Concurrency and Computation: Practice and Experience. 17 (2-4), 323-356 (2005).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados