Method Article
Cuantificación de la organización del colágeno fibrilador con herramientas basadas en curvelet transform
En este artículo
Resumen
Aquí, presentamos un protocolo para utilizar una herramienta de software MATLAB de código abierto basada en transformaciones curvas para cuantificar la organización del colágeno fibrilador en la matriz extracelular de los tejidos normales y enfermos. Esta herramienta se puede aplicar a imágenes con fibras de colágeno u otros tipos de estructuras similares a líneas.
Resumen
Los colágenos fibriladores son componentes prominentes de la matriz extracelular (ECM), y se ha demostrado que sus cambios en la topología se asocian con la progresión de una amplia gama de enfermedades, incluyendo cánceres de mama, ovario, riñón y páncreas. Las herramientas de software de cuantificación de fibra disponibles libremente se centran principalmente en el cálculo de la alineación u orientación de la fibra, y están sujetas a limitaciones tales como el requisito de pasos manuales, la imprecisión en la detección del borde de fibra en un fondo ruidoso, o la falta de caracterización de características localizadas. La herramienta de cuantificación de fibra de colágeno descrita en este protocolo se caracteriza por utilizar una representación óptima de imagen multiescala habilitada por transformación curva (TC). Este enfoque algorítmico permite la eliminación del ruido de las imágenes de colágeno fibrilador y la mejora de los bordes de fibra para proporcionar información de ubicación y orientación directamente desde una fibra, en lugar de utilizar la información indirecta en cuanto a píxeles o en la ventana obtenida de otras herramientas. Este marco basado en TC contiene dos paquetes separados, pero vinculados, denominados "CT-FIRE" y "CurveAlign" que pueden cuantificar la organización de fibra a nivel global, región de interés (ROI) o fibra individual. Este marco de cuantificación se ha desarrollado durante más de diez años y ahora se ha convertido en una plataforma integral de cuantificación de colágeno impulsada por el usuario. Con esta plataforma, se pueden medir hasta una treintena de características de fibra, incluyendo propiedades de fibra individuales como longitud, ángulo, anchura y recudimiento, así como mediciones a granel como densidad y alineación. Además, el usuario puede medir el ángulo de fibra en relación con los límites segmentados manual o automáticamente. Esta plataforma también proporciona varios módulos adicionales, incluidos los para el análisis de ROI, la creación automática de límites y el postprocesamiento. El uso de esta plataforma no requiere experiencia previa en programación o procesamiento de imágenes, y puede manejar grandes conjuntos de datos, incluyendo cientos o miles de imágenes, lo que permite una cuantificación eficiente de la organización de fibra de colágeno para aplicaciones biológicas o biomédicas.
Introducción
Los colágenos fibriladores son componentes estructurales prominentes del ECM. Su organización cambia la función del tejido de impacto y probablemente se asocian con la progresión de muchas enfermedades que van desde osteogénesis imperfecta1,disfunción cardíaca2,y cicatrización de heridas3 a diferentes tipos de cáncer incluyendo mama4,5,6,ovario7,8,riñón9,y cánceres de páncreas10. Muchas modalidades de imagen establecidas se pueden utilizar para visualizar colágeno fibrilador como microscopía de segunda generación armónica11,manchas o tintes junto con microscopía de campo brillante o fluorescencia o microscopía de luz polarizada12,microscopía de polarización basada en cristal líquido (LC-PolScope)13y microscopía electrónica14. A medida que la importancia de la organización del colágeno fibrilador se ha vuelto más clara, y el uso de estos métodos ha aumentado, la necesidad de mejorar los enfoques de análisis de fibra de colágeno también ha crecido.
Ha habido muchos esfuerzos para desarrollar métodos computacionales para la medición automatizada del colágeno fibrilador. Las herramientas de software de libre disposición se centran principalmente en el cálculo de la alineación u orientación de la fibra mediante la adopción de primer tensor derivado o estructura para píxeles15,16o fourier transform-based spectrum analysis for image tiles17. Todas estas herramientas están sujetas a limitaciones como el requisito de pasos manuales, la imprecisión en la detección del borde de fibra en un fondo ruidoso o la falta de caracterización de características localizadas.
En comparación con otras herramientas de software libre de código abierto disponibles libremente, los métodos descritos en este protocolo utilizan TC ,un método óptimo, multiescala y de representación de imágenes direccionales, para eliminar el ruido de las imágenes de colágeno fibrilador y mejorar o rastrear los bordes de fibra. La información sobre la ubicación y la orientación se puede proporcionar directamente desde una fibra en lugar de utilizar la información indirecta en píxeles o en función de las ventanas para inferir las métricas de la organización de fibra. Este marco basado en TC18,19,20,21 puede cuantificar la organización de la fibra a nivel global, ROI o fibra, principalmente a través de dos paquetes separados, pero vinculados, llamados "CT-FIRE"18,21 y "CurveAlign"19,21. En lo que respecta a la implementación del software, en CT-FIRE, los coeficientes de TC en múltiples escalas se pueden utilizar para reconstruir una imagen que mejora los bordes y reduce el ruido. A continuación, se aplica un algoritmo de extracción de fibra individual a la imagen reconstruida por TC para realizar un seguimiento de las fibras para encontrar sus puntos centrales representativos, extender las ramas de fibra desde los puntos centrales y vincular las ramas de fibra para formar una red de fibra. En CurveAlign, los coeficientes ct en una escala especificada por el usuario se pueden utilizar para realizar un seguimiento de la orientación de la fibra local, donde se extraen y agrupan la orientación y las ubicaciones de las curvas para estimar la orientación de la fibra en las ubicaciones correspondientes. Este marco de cuantificación resultante se ha desarrollado durante más de diez años y ha evolucionado mucho en muchos aspectos como la funcionalidad, la interfaz de usuario y la modularidad. Por ejemplo, esta herramienta puede visualizar la orientación local de la fibra y permite al usuario medir hasta treinta entidades de fibra, incluidas propiedades de fibra individuales como longitud, ángulo, anchura y recudez, así como mediciones a granel como densidad y alineación. Además, el usuario puede medir el ángulo de fibra en relación con los límites segmentados manual o automáticamente, lo que, por ejemplo, desempeña un papel importante en el desarrollo de biomarcador basado en imágenes en el cáncer de mama22 y estudios de cáncer de páncreas10. Esta plataforma proporciona varios módulos de características, incluidos los de análisis de ROI, creación automática de límites y postprocesamiento. El módulo roi se puede utilizar para anotar diferentes formas de ROI y realizar el análisis de ROI correspondiente. Como ejemplo de aplicación, el módulo de creación automática de límites se puede utilizar para registrar imágenes de campo brillante de hematoxilina y eosina (H&E) con imágenes de segunda generación armónica (SHG) y generar la máscara de imagen de los límites tumorales a partir de las imágenes H&E registradas. El módulo de postprocesamiento puede ayudar a facilitar el procesamiento y la integración de archivos de datos de salida a partir de imágenes individuales para un posible análisis estadístico.
Esta plataforma de cuantificación no requiere experiencia previa en programación o procesamiento de imágenes y puede manejar grandes conjuntos de datos, incluidas cientos o miles de imágenes, lo que permite una cuantificación eficiente de la organización del colágeno para aplicaciones biológicas o biomédicas. Ha sido ampliamente utilizado en diferentes campos de investigación por muchos investigadores de todo el mundo, incluyendo nosotros mismos. Hay cuatro publicaciones principales en CT-FIRE y CurveAlign18,19,20,21, de las cuales las tres primeras han sido citadas 272 veces (a partir de 2020-05-04 según Google Scholar). Una revisión de las publicaciones que citaron esta plataforma (CT-FIRE o CurveAlign) indica que hay aproximadamente 110 artículos de revistas que la utilizaron directamente para su análisis, en los que aproximadamente 35 publicaciones fueron en colaboración con nuestro grupo, y las otras (~ 75) fueron escritas por otros grupos. Por ejemplo, esta plataforma se utilizó para los siguientes estudios: cáncer de mama22,23,24,cáncer de páncreas10,25,cáncer de riñón9,26,cicatrización de heridas3,27,28,29,30,cáncer de ovario8,31,7,ligamento uterosacral32,antítesis hipofosfatémicotin33,carcinoma basocelular34,sarcoma hipoxico35,tejido cartílago36,disfunción cardíaca37,neuronas38,glioblastoma39,contracciones linfáticas40,cacffolds fibrosos41,cáncer gástrico42,microtúbulo43y fibrosis vesical44. Figura 1 demuestra la aplicación de imágenes de cáncer de CurveAlign para encontrar las firmas de colágeno asociadas al tumor de cáncer de mama19 de la imagen SHG. En la figura 2 se describe un flujo de trabajo esquemático típico de esta plataforma. Aunque estas herramientas se han revisado técnicamente18,19,21y también está disponible un protocolo regular20 para el análisis de alineación con CurveAlign, también está disponible un protocolo visual que demuestra que todas las características esenciales podrían ser útiles. Un protocolo visualizado, tal como se presenta aquí, facilitará el proceso de aprendizaje del uso de esta plataforma, así como abordar de manera más eficiente las preocupaciones y preguntas que los usuarios podrían tener.
Protocolo
NOTA: Este protocolo describe el uso de CT-FIRE y CurveAlign para la cuantificación de colágeno. Estas dos herramientas tienen objetivos complementarios, pero diferentes, principales y están vinculadas en cierta medida. CT-FIRE se puede lanzar desde la interfaz CurveAlign para llevar a cabo la mayoría de las operaciones, excepto para el postprocesamiento avanzado y el análisis de ROI. Para un funcionamiento completo de CT-FIRE, debe iniciarse por separado.
1. Colección de imágenes y requisito de imagen
NOTA: La herramienta puede procesar cualquier archivo de imagen con estructuras similares a líneas legibles por MATLAB independientemente de la modalidad de imagen utilizada para recopilarlo.
- Utilice la escala de grises de 8 bits como tipo de imagen, ya que los parámetros de ejecución predeterminados se basan en este formato.
NOTA: Shg imaging es un método de imágenes de colágeno fibrilador de alta resolución y sin etiquetas ampliamente utilizado. Las imágenes shg de un estudio de cáncer de mama19 se utilizarán aquí con el propósito de demostrar.
2. Instalación de software y requisito del sistema
NOTA: Las versiones independientes y de código fuente están disponibles libremente. La versión del código fuente requiere una instalación completa de MATLAB que incluya cajas de herramientas de procesamiento de señales, procesamiento de imágenes, análisis de estadísticas e informática paralela. Para ejecutar la versión de código fuente, todas las carpetas necesarias, incluidas algunas de los orígenes de terceros, deben agregarse a la ruta de acceso de MATLAB. Se recomienda el uso de la aplicación independiente (APP) para la mayoría de los usuarios, lo que requiere la instalación de un tiempo de ejecución del compilador (MCR) de MATLAB disponible libremente de la versión especificada. El procedimiento de instalación e inicio de la APLICACIÓN se describe a continuación.
- Descargue los paquetes de aplicaciones CT-FIRE versión 3.0 (CTF3.0) y CurveAlign versión 5.0 (CA5.0) de https://eliceirilab.org/software/ctfire/ y https://eliceirilab.org/software/curvealign/, respectivamente.
NOTA: Cada paquete incluye las imágenes independientes de la APLICACIÓN, manuales y de prueba. - Siga los requisitos detallados y las instrucciones de instalación de los sitios web anteriores para instalar MATLAB MCR 2018b.
- Inicie la APLICACIÓN.
- Para un sistema de Windows de 64 bits, haga doble clic en el icono de la APLICACIÓN para iniciarlo.
- Para un sistema Mac, siga los pasos siguientes para iniciarlo: Haga clic con el botón derecho en la aplicación (pulse ctrl) | Mostrar contenido del paquete | Contenido | MacOS | applauncher (haga clic con el botón derecho y elija abrir).
NOTA: Otros detalles se pueden ver en los sitios web de software enumerados en 2.1.
3. Extracción individual de fibra con CT-FIRE
NOTA: CT-FIRE utiliza TC para denoizar la imagen, mejorar los bordes de fibra y luego utiliza un algoritmo de extracción de fibra para rastrear fibras individuales. La longitud, el ángulo, la anchura y la recudedad se calculan para las fibras individuales.
- CT-FIRE en una sola imagen o múltiples imágenes
- Inicie la APLICACIÓN como se describe en 2.3.
- Haga clic en el botón Abrir archivo (s) en la interfaz gráfica de usuario principal (GUI, Figura 3A)y, a continuación, seleccione una o más imágenes/o pilas de imágenes en la ventana del prompt. Utilice la técnica adecuada para que el sistema operativo seleccione varias imágenes en el cuadro de diálogo (por ejemplo, en Windows, mantenga presionada la tecla CTRL mientras selecciona varios archivos).
NOTA: Si se seleccionan dos o más archivos de imagen, todas las imágenes deben utilizar los mismos parámetros en ejecución para el análisis. Asegúrese de que todas las imágenes se adquieren en las mismas condiciones o condiciones similares. - Seleccione las opciones informáticas paralelas marcando la casilla Paralelo en la esquina superior derecha para realizar varios análisis de imágenes.
- Para la pila de imágenes, mueva el control deslizante de sector debajo del cuadro de lista de archivos para seleccionar el sector que se va a analizar.
- Establezca las propiedades en ejecución. Utilice parámetros predeterminados para un análisis inicial de algunas imágenes. Si utiliza parámetros predeterminados, vaya al paso 3.1.6. Para establecer diferentes parámetros, haga clic en el botón Actualizar del panel Parámetros. Siga el manual para ajustar correctamente los parámetros.
NOTA: Los parámetros ajustados con mayor frecuencia incluyen el umbral de fondo (thresh_im2) y el radio de búsqueda de nucleación (s_xlinkbox). Si el nivel de ruido de fondo es alto, establezca thresh_im2 en un valor mayor; s_xlinkbox se asocia con el radio promedio de las fibras, establecer un valor más pequeño para detectar fibras delgadas. - Haga clic en el botón Ejecutar.
NOTA: La información de progreso se mostrará tanto en la ventana de información como en la ventana de comandos. Una vez completado el análisis, se mostrará la tabla de salida (Figura 3B). - Haga clic en cualquier elemento de la tabla de salida para ver el histograma de medidas de fibra (Figura 3C y Figura 3F) de la imagen incluyendo longitud, anchura, ángulo y recado.
NOTA: También se mostrarán las imágenes de fibra con fibras superpuestas en la imagen original(Figura 3E). - Compruebe la subcarpeta denominada ctFIREout en la carpeta de imágenes para ver los archivos de salida, incluido el archivo de imagen superpuesta ".tiff", el archivo ".csv" y el archivo ".mat".
- Análisis de la región de interés (ROI) de CT-FIRE
- Anotación de ROI con ROI Manager
- Haga clic en el botón Abrir archivo (s) de la GUI principal (Figura 3A) para cargar una o más imágenes.
- Seleccione la imagen que desea anotar en la lista de archivos.
- Seleccione roi manager en el menú desplegable del panel Opciones de ROI.
- Haga clic en el botón RUN para iniciar el módulo ROI Manager (Figura 3A).
- En la GUI del administrador de ROI (Figura 4A), haga clic en el menú desplegable debajo de Dibujar menú(d) ROI (d)para dibujar los ROIs, uno por uno.
NOTA: La forma del ROI puede ser rectángulo, mano a mano alzada, elipse, polígono o rectángulo especificado. Siga las instrucciones que aparecen en pantalla para dibujar, guardar y salir de la anotación de ROI. - Después de seleccionar el método para dibujar el ROI, arrastre el rectángulo amarillo que aparece en la imagen original a la posición deseada y, a continuación, haga clic en el botón Guardar ROI o pulse la tecla s para agregar este ROI a la lista de ROI. Este ROI se nombrará automáticamente.
- Dibuje un nuevo ROI arrastrando el ROI anterior a una nueva posición y guárdelo como se mencionó en 3.2.1.6, o repita los pasos 3.2.1.5–3.2.1.6 para dibujar un nuevo ROI.
- Pulse x o seleccione Nuevo ROI? en el menú desplegable Forma de ROI para salir de la anotación de ROI.
- Marque las casillas de verificación Mostrar todo y etiquetas para mostrar todas las ROIs definidas en la lista y sus nombres en la imagen original.
- Seleccione el ROI en la lista de ROI para realizar operaciones básicas de ROI, como Cambiar nombre de ROI, Eliminar ROI, Guardar texto de ROI, Cargar ROI desde texto,Guardar máscara de ROI, Cargar ROI desde Máscaray Combinar ROIs.
- Compruebe el archivo de salida del archivo ROI manager guardado como ".mat" en una subcarpeta denominada ROI_management en la carpeta de imagen original.
- Para anotar otra imagen en la lista de archivos abiertos, repita los pasos 3.2.1.2–3.2.1.11.
- Después de que se haga la anotación, cierre la GUI del administrador del ROI, y reajuste la GUI principal haciendo clic en el botón Reset en el GUI principal.
- Análisis de ROI para una sola imagen en ROI Manager
- Si se realiza un análisis CT-FIRE de imagen completa y los resultados se guardan en el directorio predeterminado, haga clic en uno o varios ROIs de la lista de ROI y, a continuación, haga clic en el botón ctFIRE ROI Analyzer para iniciar el módulo de análisis post-ROI.
NOTA: Los resultados se guardarán automáticamente en una subcarpeta ubicada en \\[carpeta de imagen]\ CTF_ROI\Individual\ROI_post_analysis\. - En la ventana emergente, haga clic en el botón Comprobar fibras para mostrar fibras dentro de los ROIs seleccionados (Figura 4B).
- Haga clic en Estadísticas de trazado para mostrar histogramas de cada ROI ( Figura4C). Se mostrarán las cifras de salida correspondientes.
- Si no se ha realizado el análisis CT-FIRE de imagen completa, haga clic en uno o varios ROIs en la lista de ROI y haga clic en el botón Aplicar ctFIRE on ROI para aplicar directamente el análisis CT-FIRE a los ROIs seleccionados.
- Siga las instrucciones de la ventana de solicitud para ejecutar el análisis.
NOTA: Los parámetros para ejecutar el CT-FIRE se pasan a través de la GUI principal, y el usuario puede actualizar los parámetros en ejecución como se describe en el paso 3.1.5 según sea necesario. Una vez completado el análisis, las estadísticas de resumen de las medidas de fibra se mostrarán en la tabla de salida. Los resultados se guardarán automáticamente en una subcarpeta ubicada en \\[carpeta de imagen]\ CTF_ROI\Individual\ROI_analysis\.
- Si se realiza un análisis CT-FIRE de imagen completa y los resultados se guardan en el directorio predeterminado, haga clic en uno o varios ROIs de la lista de ROI y, a continuación, haga clic en el botón ctFIRE ROI Analyzer para iniciar el módulo de análisis post-ROI.
- Análisis de ROI para múltiples imágenes utilizando ROI Analyzer
- Siga los pasos descritos en 3.2.1 para anotar los ROIs para analizar las imágenes.
- Abra una o más imágenes haciendo clic en el botón Abrir archivo(s).
- Para ejecutar el análisis posterior al ROI cuando los resultados completos del análisis de imágenes estén disponibles, haga clic en el menú desplegable del panel Ejecutar opciones y seleccione la opción Analizador post-ROI CTF.
- Haga clic en el botón EJECUTAR para ejecutar el análisis de ROI de todas las imágenes cargadas.
- Marque la información de progreso mostrada en la ventana del mensaje en la parte inferior de la GUI y en la ventana del comando.
- Una vez completado el análisis, compruebe las estadísticas de resumen de cada ROI que se muestra en una tabla de salida.
NOTA: Los archivos de salida detallados se guardan automáticamente en una subcarpeta ubicada en \\ [carpeta de imagen]\CTF_ROI\Batch\ROI_post_analysis\. - Para ejecutar un análisis directo cuando los resultados completos del análisis de imágenes no están disponibles, siga los pasos 3.2.3.1–3.2.3.6, excepto que en el paso 3.2.3.3, seleccione la opción CTF ROI analyzer; en el paso 3.2.3.4, antes de hacer clic en el botón EJECUTAR, actualice los parámetros en ejecución como se describe en el paso 3.1.5. Después de hacer clic en el botón EJECUTAR, en una ventana de diálogo de solicitud, elija entre Roi rectangular y máscara de ROI de cualquier forma.
NOTA: Si todos los ROIs anotados son rectangulares, el usuario puede elegir el "ROI rectangular". En el paso 3.2.3.6, los resultados del análisis de ROI se guardan en una subcarpeta ubicada en \\[carpeta de imágenes]\CTF_ROI\Batch\ROI_analysis\.
- Anotación de ROI con ROI Manager
- Postprocesamiento con CT-FIRE
NOTA: Después del análisis ct-fire regular descrito en 3.1, el usuario puede realizar más postprocesamiento. Sin volver a ejecutar la extracción de fibra que consume mucho tiempo, el postprocesamiento regular, descrito en 3.3.1, puede actualizar algunas propiedades básicas de la figura de salida, mientras que el postprocesamiento avanzado descrito en 3.3.2 puede visualizar fibras individuales y sus propiedades, realizar umbrales complejos entre las cuatro propiedades de fibra, generar estadísticas de resumen de las fibras seleccionadas y visualizar las fibras seleccionadas utilizando un mapa de color personalizado.- Postprocesamiento regular con CT-FIRE
- Inicie la aplicación CT-FIRE, o haga clic en el botón Restablecer después de otras operaciones para inicializar la GUI principal ct-FIRE (Figura 3A).
- Marque la casilla de verificación .mat en la parte superior de la GUI principal.
- Haga clic en el botón Abrir archivo(s) para seleccionar el archivo .mat de salida CT-FIRE en la subcarpeta ctFIREout.
NOTA: Si se seleccionan varios archivos, la casilla de verificación Lote se comprobará automáticamente. El nombre de archivo de las imágenes correspondientes se mostrará en la lista de cuadros. - Actualice las opciones en el panel Control de figura de salida.
- Mantenga las opciones predeterminadas en Opciones de salida,que se asegurarán de que todos los archivos de salida se actualizarán de acuerdo con el nuevo conjunto de parámetros establecido en 3.3.1.4.
- Haga clic en el botón Postprocesamiento. Marque la información de progreso en la ventana del mensaje en la parte inferior de la GUI principal así como en la ventana del comando.
- Una vez completado el análisis, haga clic en cualquier elemento de la tabla de salida para ver el histograma de las medidas de fibra de la imagen, incluida la longitud, anchura, ángulo y recura.
NOTA: Los nuevos archivos de salida sobrescribirán los antiguos en la subcarpeta ctFIREout.
- Postprocesamiento avanzado de CT-FIRE
- Inicie la aplicación CT-FIRE, o haga clic en el botón Restablecer después de otras operaciones para inicializar la GUI principal ct-FIRE (Figura 3A).
- Marque la casilla de verificación OUT.adv en la parte superior de la GUI principal (Figura 3A).
- Haga clic en el botón Postprocesamiento para iniciar la GUI avanzada posterior al procesamiento denominada "Módulo de análisis" ( Figura5A).
- Haga clic en el botón Seleccionar archivo para seleccionar una imagen.
- Haga clic en el botón Visualizar fibras para introducir el número de fibra en función de las etiquetas de la figura de ficha Fibras originales.
NOTA: Las mediciones de las fibras seleccionadas se mostrarán en una tabla de salida(Figura 5B),y las fibras correspondientes se superpondrán en la imagen original que se muestra en la figura de tabulación denominada Fibras medidas (Figura 5C). - Haga clic en el botón Confirmar/Actualizar para pasar a la operación de umbral.
- Marque el cuadro de umbral para habilitar la configuración del umbral.
- Elija una de las cuatro opciones de umbral en el menú desplegable.
- Introduzca los umbrales deseados en el panel Umbrales para obtener una o más propiedades de fibra.
- Haga clic en el botón Umbral ahora para aplicar las condiciones de umbral anteriores.
- Compruebe la figura de solicitud con su nombre terminando con la visualización de métricas para ver las fibras seleccionadas superpuestas en las imágenes originales con los mapas de color personalizados como se muestra en la Figura 5E.
- Repita los pasos 3.3.2.9–3.3.2.11 para establecer los umbrales deseables.
- Haga clic en el botón Guardar fibras para guardar la información de fibra seleccionada.
NOTA: Las fibras seleccionadas correspondientes se mostrarán en la figura de tabulación denominada After-Thresholding. - Haga clic en el botón Generar estadísticas y, a continuación, haga clic en el botón Aceptar de la ventana emergente para generar estadísticas de resumen.
NOTA: Una tabla de salida (Figura 5D) mostrará el valor medio de las fibras seleccionadas. Otras estadísticas de las fibras seleccionadas se guardarán en un archivo de Excel cuya ubicación se muestra en la ventana de estado en la parte inferior de esta GUI. - Para incluir la información de fibra individual seleccionada en el archivo de salida, active el cuadro Generar hoja para datos sin procesar antes de hacer clic en el botón Aceptar.
- Para combinar los resultados de varias imágenes, en el paso 3.3.2.4, marque el cuadro Modo por lotes o Modo pila y seleccione las múltiples imágenes o pilas que se analizarán; omita los pasos 3.3.2.5–3.3.2.6. En los pasos 3.3.2.8–3.3.2.9, establezca las condiciones de umbral, pero como los botones umbral ahora y Guardar fibras están desactivados, omita los pasos 3.3.2.10–3.3.2.13; y por último, siga las instrucciones del paso 3.3.2.14 para generar estadísticas de resumen y propiedades de fibra individuales de las fibras seleccionadas.
- Postprocesamiento regular con CT-FIRE
4. Análisis de fibra con CurveAlign
NOTA: CurveAlign fue desarrollado inicialmente para medir automáticamente los ángulos de las fibras con respecto a los límites definidos por el usuario. La versión actual de CurveAlign se puede utilizar para la evaluación a granel de entidades basadas en densidad y alineación, además de la medición relativa del ángulo mediante la carga de la información de fibra individual extraída por CT-FIRE o directamente utilizando la orientación local de las curvas. CurveAlign calcula hasta treinta características relacionadas con características globales o locales, principalmente incluyendo densidad y alineación, así como propiedades de fibra individuales cuando CT-FIRE se adopta como el método de seguimiento de fibra.
- Análisis de fibra con curvas
- Inicie la APLICACIÓN como se describe en 2.3.
- Haga clic en el botón Restablecer para restablecer la APLICACIÓN a su estado inicial si se han llevado a cabo otras operaciones.
- En la GUI principal (Figura 6A), marque la opción del método de análisis de fibra para asegurarse de que la TC está seleccionada (opción predeterminada).
NOTA: En este modo, la TC se realiza en la imagen y la orientación de cada curva representa la dirección de una fibra en la ubicación correspondiente. - Haga clic en el menú desplegable Método de límite y seleccione el modo de procesamiento de límites en las siguientes opciones de menú desplegable: Sin límite, Límite CSV y Límite TIFF.
NOTA: Si no se necesita ningún límite, omita este paso. Refiera a 4.3 para cómo calcular los ángulos de fibra con respecto a un límite. - Haga clic en el botón Obtener imágenes en la GUI principal (Figura 6A) y, a continuación, seleccione una o más imágenes/o pilas de imágenes en la ventana del prompt. Utilice la técnica adecuada para que el sistema operativo seleccione varias imágenes en el cuadro de diálogo (por ejemplo, en Windows, mantenga presionada la tecla CTRL mientras selecciona varios archivos).
NOTA: Si se seleccionan dos o más archivos de imagen, todas las imágenes deben utilizar los mismos parámetros en ejecución para el análisis. Asegúrese de que todas las imágenes se adquieren en las mismas condiciones o condiciones similares. - Para la pila de imágenes, mueva el control deslizante de sector debajo del cuadro de lista de archivos para seleccionar el sector que se va a analizar.
- Ingrese la fracción de coefs para mantener. Este valor es la fracción de los coeficientes más grandes de TC que se utilizarán en el análisis de fibra.
NOTA: Si la imagen tiene una gran variación en la intensidad o contraste de la fibra, anota regiones de interés con incluso contraste para el análisis de fibra, ya que este modo solo detecta las fibras más brillantes de una imagen. Además, cuanto mayor sea el tamaño de la imagen, establezca un valor menor para esta fracción. - Mantenga todos los parámetros en las Opciones de salida y otros en la opción Avanzadas como valor predeterminado; los archivos de salida pueden ser necesarios en otras operaciones futuras.
- Haga clic en el botón Ejecutar en la parte inferior de la GUI principal (Figura 6A).
NOTA: La información de progreso se mostrará en una ventana de mensaje resaltada en color verde en la parte inferior. Una vez realizado el proceso, se mostrarán algunas estadísticas de resumen para cada imagen en la tabla de salida(figura 6B),y todos los archivos de salida se guardarán automáticamente en una subcarpeta denominada CA_Out en el directorio de las imágenes originales. - Haga clic en cualquier elemento de la tabla de salida (Figura 6B) para ver el histograma (Figura 6E) o gráfica de brújula (Figura 6F) de ángulos de fibra.
NOTA: También se mostrará la imagen superpuesta(Figura 6C)y el mapa de calor (Figura 6D) de alineación o ángulo. - Haga clic en el botón Restablecer para ejecutar otras operaciones, o cierre la GUI principal para salir de la APLICACIÓN.
- Análisis individual de fibra con CT-FIRE
NOTA: El procedimiento es el mismo que el descrito en la sección 4.1 excepto que en el paso 4.1.3, seleccione el modo de análisis de fibra relacionado con CT-FIREy omita el paso 4.1.7, ya que no es aplicable y está deshabilitado en el modo CT-FIRE. En concreto, en el paso 4.1.3, seleccione uno de los tres métodos de análisis de fibra individuales basados en CT-FIRE siguientes:- Seleccione Fibras CT-FIRE para utilizar el punto central de fibra y el ángulo de fibra para representar la fibra.
NOTA: Esta opción no tiene en cuenta los cambios en la orientación de la fibra a lo largo de la longitud de la fibra. - Seleccione Puntos finales CT-FIRE para utilizar los dos puntos finales de un ángulo de fibra y fibra correspondiente para representar la fibra.
NOTA: En comparación con 4.2.1, esta opción utiliza dos posiciones para representar una fibra en lugar de una (punto central de la fibra). - Seleccione Segmentos CT-FIRE para utilizar segmentos de una fibra para representar la fibra.
NOTA: Cada segmento tiene una longitud igual (establecida en 5 píxeles de forma predeterminada en CT-FIRE), así como su orientación y ubicación, lo que refleja el cambio de orientación a lo largo de toda la longitud de la fibra. Esta opción sería la más lenta, pero sería la mejor opción entre los tres métodos de análisis de fibra basados en CT-FIRE para realizar un seguimiento de los cambios en la orientación local de una fibra curva.
- Seleccione Fibras CT-FIRE para utilizar el punto central de fibra y el ángulo de fibra para representar la fibra.
- Análisis de alineación relativa con límite
NOTA: En comparación con el análisis regular sin condiciones límite descritas en las secciones 4.2 y 4.3, el análisis relativo de alineación con condiciones límite necesita lo siguiente:- En el paso 4.1.3, seleccione la condición de contorno tiff.
NOTA: El usuario necesitará un archivo de límite correspondiente para cada imagen o cada pila. Siga las instrucciones que aparecen en pantalla para anotar manualmente un archivo de límite CSV (formato de valores separados por comas, coordenadas x-y) o un archivo de límite Tiff. Los archivos de límite creados en CurveAlign se guardarán automáticamente según el directorio de archivos y las convenciones de nomenclatura de archivos descritas en el manual. Si se proporcionan un par de imágenes de campo brillante e SHG de H&E, utilice el módulo de creación automática de límites descrito en la sección 4.4 para generar el archivo de límite. - En el panel Parámetros primarios, introduzca la distancia desde el límite más cercano para evaluar únicamente las fibras dentro de este rango de distancia.
- En el panel Opciones de salida, active la casilla de asociación de límites Bdry Assoc para visualizar el punto del límite asociado a una fibra, un segmento de fibra o una curva.
- En el paso 4.1.3, seleccione la condición de contorno tiff.
- Creación automática de límites
- Inicie la APLICACIÓN como se describe en 2.3.
- Haga clic en el botón Restablecer para restablecer la APLICACIÓN a su estado inicial si ya se han llevado a cabo otras operaciones.
- Haga clic en el botón Creación de BD para iniciar el módulo de creación automática de límites.
- Siga las instrucciones/pistas que aparecen en pantalla para crear un archivo de contorno para una o más imágenes basadas en un par de imágenes de campo brillante e SHG de H&E.
- Cierre la ventana del módulo, o haga clic en el botón reset en el GUI principal (figura 6A)para salir de este módulo.
- CurveAlign región de análisis de intereses
- Anotación de ROI con ROI Manager
- Haga clic en el botón Obtener imágenes en el GUI principal (Figura 6A) para cargar una o más imágenes.
- Seleccione la imagen que desea anotar en la lista de archivos.
- Haga clic en el Administrador de ROI para iniciar el módulo ROI Manager ( Figura7A).
- Siga los pasos 3.2.1.5–3.2.1.13 de la sección 3.2.1.
- Análisis de ROI para una sola imagen en ROI Manager
- Si se ha realizado el análisis CurveAlign de imagen completa y los resultados se guardan en el directorio predeterminado, haga clic en uno o varios ROIs de la lista roi y, a continuación, haga clic en el botón Analizador de ROI de CA para ejecutar el análisis posterior al ROI.
NOTA: Una vez completado el análisis, se mostrarán estadísticas de resumen en una tabla de salida (Figura 7C) así como una figura de histograma (Figura 7D) que muestra la distribución del ángulo. - Haga clic en cualquier elemento de la tabla de salida para visualizar las fibras en un ROI determinado(Figura 7B),así como en el histograma de los ángulos de fibra.
- Compruebe los archivos de salida guardados en una subcarpeta ubicada en \\[carpeta de imagen]\ CA_ROI\Individual\ROI_post_analysis\.
- Si no se ha realizado el análisis de CA de imagen completa, haga clic en uno o varios ROIs de la lista ROI y haga clic en el botón Aplicar CA en ROI para aplicar directamente el análisis de CA a los ROIs seleccionados. Siga las instrucciones de la ventana de solicitud para ejecutar el análisis.
NOTA: Los parámetros para ejecutar el análisis de CA se pasan a través de la GUI principal; actualizar los parámetros en ejecución descritos en el paso 4.1.7 según sea necesario. Una vez completado el análisis, los resultados de las estadísticas de resumen de las medidas de fibra se mostrarán en la tabla de salida. Los resultados se guardarán automáticamente en una subcarpeta ubicada en \\[carpeta de imágenes]\ CA_ROI\Individual\ROI_analysis\.
- Si se ha realizado el análisis CurveAlign de imagen completa y los resultados se guardan en el directorio predeterminado, haga clic en uno o varios ROIs de la lista roi y, a continuación, haga clic en el botón Analizador de ROI de CA para ejecutar el análisis posterior al ROI.
- Análisis de ROI para múltiples imágenes utilizando ROI Analyzer
- Siga los pasos descritos en 4.5.1 para anotar los ROIs para analizar las imágenes.
- Abra una o más imágenes haciendo clic en el botón Obtener imágenes.
- Para ejecutar el análisis posterior al ROI cuando estén disponibles los resultados completos del análisis de imágenes, haga clic en el botón Análisis de ROI y seleccione la opción Posterior al procesamiento del ROI.
- Marque la información de progreso mostrada en la ventana del mensaje en la parte inferior de la GUI y en la ventana del comando.
- Una vez completado el análisis, compruebe las estadísticas de resumen de cada ROI que se muestra en una tabla de salida.
NOTA: Los archivos de salida detallados se guardan automáticamente en una subcarpeta ubicada en \\ [carpeta de imagen]\CA_ROI\Batch\ROI_post_analysis\. - Para ejecutar un análisis directo con modo CT cuando los resultados completos del análisis de imagen no están disponibles, siga los pasos 4.5.3.1–4.5.3.5, excepto los siguientes cambios: modifique el paso 4.5.3.3 seleccionando la opción CA en el ROI rectangular recortado o CA en máscara con ROI de cualquier forma. Si todos los ROIs anotados son de forma rectangular, elija la opción ROI rectangular. Después del paso 4.5.3.2, actualice los parámetros en ejecución, como se describe en 4.1.7.
NOTA: Los resultados del análisis de ROI se guardarán en una subcarpeta ubicada en \\ [carpeta de imagen]\CA_ROI\Batch\ROI_analysis\.
- Anotación de ROI con ROI Manager
- Postprocesamiento de CurveAlign
- Inicie la APLICACIÓN como se describe en la sección 2.3.
- Haga clic en el botón Restablecer para restablecer la APLICACIÓN a su estado inicial si ya se han realizado otras operaciones.
- Haga clic en el botón Postprocesamiento para iniciar el módulo posterior al procesamiento.
- Siga las instrucciones/pistas que aparecen en pantalla para combinar las funciones o valores de salida de diferentes imágenes.
- Cierre la ventana del módulo, o haga clic en el botón reset en el GUI principal (figura 6A)para salir de este módulo.
5. Tiempo de ejecución estimado
- Espere el tiempo de ejecución estimado para procesar una imagen con un tamaño de 1024 píxeles x 1024 píxeles con densidad de fibra moderada. El tiempo computacional real generalmente depende de varios factores, incluyendo el tamaño del archivo, el modo de análisis, las características que se implementarán, el tipo de unidad de procesamiento central (CPU) y la cantidad de memoria de acceso aleatorio (o RAM) disponible. La extracción individual de fibra CT-FIRE tarda un par de minutos. CurveAlign ct-mode sin límite toma unos segundos. CurveAlign CT-FIRE fibra o fibra termina el modo sin límite toma decenas de segundos. El modo de fibra CurveAlign CT-FIRE sin límite tarda cientos de segundos. El análisis CurveAlign con contorno tarda de decenas de segundos a varios minutos, dependiendo de la complejidad de los límites.
Resultados
Estos métodos se han aplicado con éxito en numerosos estudios. Algunas aplicaciones típicas incluyen: 1) Conklin et al.22 utilizó CurveAlign para calcular las firmas de colágeno asociadas al tumor, y encontró que las fibras de colágeno se alineaban con mayor frecuencia perpendicularmente al perímetro del conducto en lesiones de carcinoma ductal in situ (DCIS); 2) Drifka et al.10 utilizaron el modo CT-FIRE en CurveAlign para cuantificar la alineación estromal de colágeno para el adenocarcinoma ductal pancreático y los tejidos de pancreatitis normal/crónica, y encontraron que había una mayor alineación en los tejidos cancerosos en comparación con la de los tejidos normales/crónicos; 3) Alkmin et al.7 utilizaron CurveAlign para cuantificar la distribución angular de las fibras de F-actin y la alineación general de colágeno de las imágenes SHG de colágeno estromal ovárico, y mostraron que la morfología matricial juega un papel importante en la conducción de la motilidad celular y la alineación de F-actin; 4) LeBert et al.3 aplicaron CT-FIRE a las imágenes SHG de un modelo de reparación de heridas de pez cebra y encontraron un aumento en el grosor de las fibras de colágeno después de heridas agudas; 5) Devine et al.45 utilizó el modo CT-FIRE en CurveAlign para imágenes SHG de colágeno de pliegue vocal de diferentes modelos animales para medir las propiedades individuales de la fibra y la alineación general, y mostró que el colágeno de pliegue vocal porcino y canino tenía una mayor alineación y reclino inferior; 6) Keikhosravi et al.13 utilizaron CurveAlign para cuantificar la alineación de colágeno en muestras de histopatología imágenes con LC-PolScope, y mostraron que LC-PolScope y SHG son comparables en términos de alineación y medición de orientación para algunos tipos de tejido.

Figura 1: Uso de CurveAlign para encontrar firmas de colágeno asociadas al tumor a partir de imágenes SHG de un microarray de tejido de cáncer de mama humano (TMA). (A) Imagen superpuesta de un núcleo TMA con imagen SHG (amarillo) superpuesta en la imagen de campo brillante H&E correspondiente. (B) La región de interés de (A). (C) La imagen de campo brillante de (B). (D) La imagen SHG de (B). (F) La máscara asociada a la imagen de campo brillante (C). (E) La imagen de superposición de salida CurveAlign que muestra los límites del tumor (amarillo) de (F), ubicaciones representativas de fibra y orientación (líneas verdes); las líneas azules se utilizan para asociar fibras con sus límites más cercanos. Las flechas verdes en (B) y (E) muestran las fibras paralelas al límite del tumor, mientras que las flechas rojas allí muestran las fibras perpendiculares al límite. La barra de escala en (A) es igual a 200 μm. Las imágenes en (B)–(F) se muestran en la misma escala, y la barra de escala representativa en (D) es igual a 50 μm. Abreviaturas: SHG = segunda generación armónica; H&E = hematoxilina y eosina. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Flujo de trabajo esquemático de cuantificación de una imagen de colágeno fibrilador. (A) Imagen SHG que se analizará mediante CT-FIRE y/o CurveAlign. (B) Superposición de salida de imagen por CT-FIRE. (C) Límite de máscara de (A) es una entrada CurveAlign opcional. (D) Superposición de salida de imagen por CurveAlign. Las líneas de color en (B) representan las fibras extraídas. En (D), las líneas verdes indican las ubicaciones y orientaciones de las fibras que están fuera de los límites (líneas amarillas) y están dentro de la distancia especificada de sus límites más cercanos, las líneas rojas son las de otras fibras, y las líneas azules se utilizan para asociar fibras con sus límites más cercanos. Las imágenes en (A)–(D) se muestran en la misma escala, y la barra de escala representativa en (A) es igual a 200 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Análisis regular de CT-FIRE. (A) GUI principal. (B) Tabla de salida que muestra las estadísticas de resumen. (C) y (F) muestran los histogramas de ángulo y anchura, respectivamente. (E) Imagen de salida que muestra las fibras extraídas (líneas de color) superpuestas en la imagen SHG original (D). Abreviaturas: GUI = CT = transformación de curvas; interfaz gráfica de usuario; SHG = segunda generación armónica. Haga clic aquí para ver una versión más grande de esta figura.
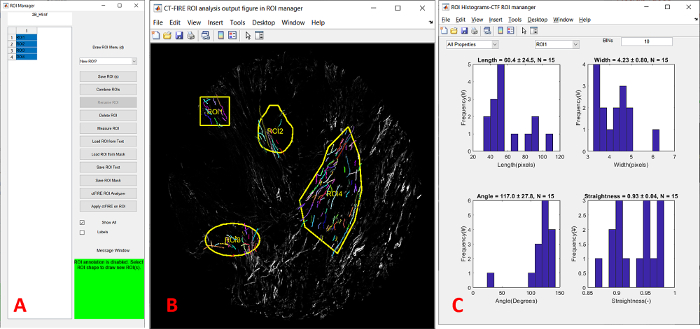
Figura 4: Módulo de gestión de ROI CT-FIRE. (A) Módulo GUI. (B) ROI post-análisis de cuatro ROIs con diferentes formas que muestran las fibras dentro de cada ROI. (C) histogramas roi de diferentes propiedades de fibra. Abreviaturas: CT = transformación de curvas; GUI = interfaz gráfica de usuario; ROI = región de interés. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Módulo avanzado de postprocesamiento CT-FIRE. (A) Módulo GUI. (B) Mediciones de tres fibras seleccionadas. (C) Visualización de las tres fibras seleccionadas en (B). (D) Estadísticas de resumen después de aplicar un umbral de longitud (>60 píxeles). (E) Visualización de las fibras seleccionadas en (D) con barra de color basada en longitud. Abreviaturas: CT = transformación de curvas; GUI = interfaz gráfica de usuario. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Análisis regular curvealign. (A) GUI principal. (B) Tabla de salida que muestra las estadísticas de resumen. (C) Imagen de salida que muestra las ubicaciones y la orientación de las fibras representativas (líneas verdes) y los límites (líneas amarillas) superpuestas en la imagen SHG original, las líneas azules se utilizan para asociar fibras con sus límites más cercanos, las líneas rojas muestran las ubicaciones y orientaciones de las fibras dentro de un límite o fuera lejos del límite (>distancia especificada por el usuario, por ejemplo, 250 píxeles aquí). (D) Mapa de calor de los ángulos: rojo (> 60 grados), amarillo (45-60] grados, verde (10, 45] grados. (E)–(F) mostrar la distribución del ángulo utilizando histograma y gráfica de brújula, respectivamente. Abreviaturas: GUI = interfaz gráfica de usuario; SHG = segunda generación armónica. Haga clic aquí para ver una versión más grande de esta figura.
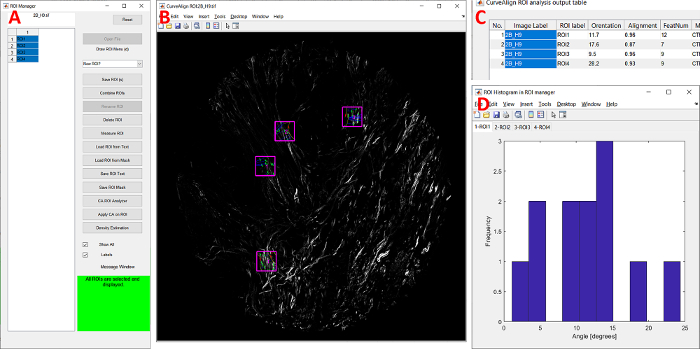
Figura 7: Módulo de gestión curvealign ROI. (A) El módulo GUI. (B) Cuatro ROIs rectangulares anotados superpuestos en la imagen original. (C) Tabla de salida posterior al análisis del ROI. (D) Histograma angular de cada ROI. Abreviaturas: ROI = región de interés; GUI = interfaz gráfica de usuario. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Este protocolo describe el uso de CT-FIRE y CurveAlign para la cuantificación de colágeno fibrilador y se puede aplicar a cualquier imagen con fibras de colágeno u otras estructuras alargadas similares a líneas o similares a fibras adecuadas para su análisis por CT-FIRE o CurveAlign. Por ejemplo, las fibras elastinas o elásticas podrían procesarse de manera similar en esta plataforma. Hemos probado ambas herramientas en fibras sintéticas generadas computacionalmente21. Dependiendo de la aplicación, los usuarios deben elegir el modo de análisis que sea más adecuado para sus datos. El modo de análisis de fibra CT puede utilizar directamente curvas en TC para representar la ubicación y orientación de la fibra, y es sensible a los cambios en la estructura de fibra local. El modo TC se puede utilizar para localizar fibras y su orientación en condiciones complejas, por ejemplo, donde el nivel de ruido es alto, la fibra es curva o la variación en el espesor de la fibra es alta. Sin embargo, como el modo TC sólo recoge las partes más brillantes de una imagen, se perdería algunas fibras con menor intensidad cuando hay una gran variación en la intensidad de la imagen.
Además, el modo TC no proporciona información sobre fibras individuales. A diferencia del modo CT, el modo CT-FIRE calcula las propiedades individuales de la fibra y puede analizar todas las fibras cuya intensidad está por encima de un umbral especificado. Los desafíos asociados con el modo CT-FIRE incluyen: 1) la precisión de una extracción de fibra intacta puede reducirse o verse comprometida cuando hay una gran variación en la intensidad a lo largo de una fibra o el grosor de la fibra a través de una imagen; y 2) el análisis estándar actual es computacionalmente exigente como se menciona en el protocolo. Más detalles sobre las ventajas y limitaciones de estos métodos se pueden ver en nuestras publicaciones anteriores20,21.
En lo que respecta a la precisión del seguimiento de la fibra, el usuario puede basarse principalmente en la inspección visual para comprobar la imagen superpuesta donde las fibras extraídas u orientaciones representativas están superpuestas en la imagen original. Además, para CT-FIRE, el usuario puede utilizar el módulo avanzado posterior al procesamiento para identificar las propiedades de determinadas fibras individuales y compararlas con mediciones utilizando otras herramientas de análisis de imágenes como Fiji46. En CurveAlign, el usuario puede comparar los resultados de orientación o alineación con los calculados por otras herramientas como OrientationJ16 y CytoSpectre17.
Entre las características disponibles para la salida por la plataforma, las características relacionadas con la alineación se utilizan con mayor frecuencia y son las más fiables y robustas. Para utilizar características de fibra individuales, el usuario debe confirmar la extracción de características representativas de fibra. Cabe destacar que una fibra intacta puede dividirse en varios segmentos más cortos en algunas circunstancias, que el usuario debe tener en cuenta al seleccionar el modo de análisis de fibra o realizar más análisis estadísticos. Incluso cuando la longitud de la fibra no se puede utilizar directamente como una propiedad comparable, la orientación o anchura de los segmentos de fibra ponderados contra sus longitudes todavía puede indicar información útil. En lo que respecta a las imágenes SHG, la apertura numérica (NA) de la lente objetiva puede afectar significativamente a la detección de la anchura y longitud de una fibra, pero tiene menos impacto en las mediciones de orientación y alineación. En nuestra experiencia en imágenes SHG, normalmente necesitamos usar lente objetiva con ampliación de 40x o superior con NA ≥ 1.0 para lograr una medición robusta del espesor de la fibra.
"Alineación" se puede interpretar de tres maneras diferentes: 1) alineación con respecto a la dirección horizontal positiva denominada "ángulo", que va de 0 a 180 grados, donde los ángulos cercanos a 0 tienen una orientación similar a los ángulos cercanos a 180 grados; 2) alineación con respecto a un límite denominado "ángulo relativo", que oscila entre 0 y 90 grados, con 0 grados que indican una fibra paralela al límite y 90 que indica una fibra perpendicular; y 3) alineación de fibras con respecto entre sí denominada "coeficiente de alineación", que van de 0 a 1, con 1 que indica fibras perfectamente alineadas y valores más pequeños que representan fibras distribuidas más aleatoriamente.
Además de las características de fibra calculadas en esta plataforma, también se propusieron algunas métricas basadas en el análisis de texturas47,48,49 para cuantificar los patrones ecm. Esas entidades relacionadas con la textura pueden proporcionar un descriptor alternativo o adicional del ECM en algunas aplicaciones. Los desafíos para el desarrollo de este tipo de métricas radican en la interoperación de la posible relevancia biológica, la caracterización localizada y la precisión del rastreo de fibras individuales.
Para optimizar los parámetros en ejecución y realizar la solución de problemas, el usuario puede consultar las publicaciones manuales y relevantes20,21, así como las barras laterales de preguntas frecuentes en las páginas wiki de GitHub del repositorio curvelets: https://github.com/uw-loci/curvelets/wiki. Para algunos botones, puede aparecer una sugerencia de función para guiar al usuario para la operación actual o siguiente cuando el usuario mueve el icono del ratón por encima de un botón. Siga la información en el GUI o la ventana de comandos para realizar el Troubleshooting.
Para procesar un conjunto de datos grande, se recomienda al usuario utilizar opciones informáticas paralelas, que permiten a la herramienta procesar varias imágenes simultáneamente. Una opción es el uso de varios núcleos de CPU si está disponible en el equipo que se está utilizando. Alternativamente, se proporciona una versión sin cabeza de ambas APPs y se ha compilado correctamente en el nodo de compilación a través del servidor que se mantiene en el Centro50 de Computación de Alto Rendimiento (CHTC) basado en CONDOR en la Universidad de Wisconsin-Madison. El flujo de trabajo chtc para cuantificación de fibra a gran escala se ha desarrollado, probado y utilizado con éxito en conjuntos de imágenes reales que constan de miles de imágenes. El usuario podría adaptar las funciones matlab sin cabeza de CT-FIRE y CurveAlign para ejecutar la cuantificación en otros sistemas de computación en la nube, incluidos los servicios comerciales como los ofrecidos por Amazon, Google y Microsoft.
Las instrucciones de desarrollo en curso y futuras incluyen: 1) incorporación de la red neuronal de aprendizaje profundo para extraer o generar imágenes de fibra de colágeno sintético de alta calidad y mejorar la robustez y precisión del algoritmo de trazado de fibra; 2) integración de todos los módulos en una plataforma completa mientras optimiza el código y la documentación siguiendo las mejores prácticas de ingeniería de software; 3) implementación de todas las características principales en una plataforma de computación en la nube; 4) mejora del flujo de trabajo de análisis de fibra utilizando el servicio CHTC; y 5) mejora de la funcionalidad del generador de fibra sintética.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a muchos contribuyentes y usuarios a CT-FIRE y CurveAlign a lo largo de los años, incluyendo el Dr. Rob Nowak, la Dra. Carolyn Pehlke, el Dr. Jeremy Bredfeldt, Guneet Mehta, Andrew Leicht, el Dr. Adib Keikhosravi, el Dr. Matt Conklin, el Dr. Jayne Squirrell, el Dr. Paolo Provenzano, la Dra. Brenda Ogle, la Dra. Patricia Keely, el Dr. Joseph Szulczewski, la Dra. Suzanne Ponik y contribuciones técnicas adicionales de Swati Anand y Curtis Rueden. Este trabajo fue apoyado por fondos de Semiconductor Research Corporation, Morgridge Institute for Research y NIH grants R01CA199996, R01CA181385 y U54CA210190 a K.W.E.
Materiales
| Name | Company | Catalog Number | Comments |
| CT-FIRE | Univerity of Wisconsin-Madison | N/A | open source software available from https://eliceirilab.org/software/ctfire/ |
| CurveAlign | University of Wisconsin-Madison | N/A | open source software available from https://eliceirilab.org/software/curvealign/ |
Referencias
- Nadiarnykh, O., et al. Second harmonic generation imaging microscopy studies of osteogenesis imperfecta. Journal of Biomedical Optics. 12 (5), 051805(2007).
- Kouris, N. A., et al. A nondenatured, noncrosslinked collagen matrix to deliver stem cells to the heart. Regenerative Medicine. 6 (5), 569-582 (2011).
- LeBert, D. C., et al. Matrix metalloproteinase 9 modulates collagen matrices and wound repair. Development. 142 (12), 2136-2146 (2015).
- Provenzano, P. P., et al. Collagen reorganization at the tumor-stromal interface facilitates local invasion. BMC Medicine. 4 (1), 38(2006).
- Provenzano, P. P., et al. Collagen density promotes mammary tumor initiation and progression. BMC Medicine. 6 (1), 1(2008).
- Conklin, M. W., et al. Aligned collagen is a prognostic signature for survival in human breast carcinoma. The American Journal of Pathology. 178 (3), 1221-1232 (2011).
- Alkmin, S., et al. Migration dynamics of ovarian epithelial cells on micro-fabricated image-based models of normal and malignant stroma. Acta Biomaterialia. 100, 92-104 (2019).
- Campbell, K. R., Campagnola, P. J. Assessing local stromal alterations in human ovarian cancer subtypes via second harmonic generation microscopy and analysis. Journal of Biomedical Optics. 22 (11), 116008(2017).
- Best, S. L., et al. Collagen organization of renal cell carcinoma differs between low and high grade tumors. BMC Cancer. 19 (1), 490(2019).
- Drifka, C. R., et al. Periductal stromal collagen topology of pancreatic ductal adenocarcinoma differs from that of normal and chronic pancreatitis. Modern Pathology. 28 (11), 1470-1480 (2015).
- Campagnola, P. J., et al. Three-dimensional high-resolution second-harmonic generation imaging of endogenous structural proteins in biological tissues. Biophysical Journal. 82 (1), 493-508 (2002).
- Arun Gopinathan, P., et al. Study of collagen birefringence in different grades of oral squamous cell carcinoma using picrosirius red and polarized light microscopy. Scientifica. 2015, 1-7 (2015).
- Keikhosravi, A., et al. Quantification of collagen organization in histopathology samples using liquid crystal based polarization microscopy. Biomedical Optics Express. 8 (9), 4243-4256 (2017).
- Quan, B. D., Sone, E. D. Cryo-TEM analysis of collagen fibrillar structure. Methods in Enzymology. 532, 189-205 (2013).
- Boudaoud, A., et al. FibrilTool, an ImageJ plug-in to quantify fibrillar structures in raw microscopy images. Nature Protocols. 9 (2), 457-463 (2014).
- Rezakhaniha, R., et al. Experimental investigation of collagen waviness and orientation in the arterial adventitia using confocal laser scanning microscopy. Biomechanics and Modeling in Mechanobiology. 11 (3-4), 461-473 (2012).
- Kartasalo, K., et al. CytoSpectre: a tool for spectral analysis of oriented structures on cellular and subcellular levels. BMC Bioinformatics. 16 (1), 1(2015).
- Bredfeldt, J. S., et al. Computational segmentation of collagen fibers from second-harmonic generation images of breast cancer. Journal of Biomedical Optics. 19 (1), 016007(2014).
- Bredfeldt, J. S., et al. Automated quantification of aligned collagen for human breast carcinoma prognosis. Journal of Pathology Informatics. 5 (1), 28(2014).
- Liu, Y., Keikhosravi, A., Mehta, G. S., Drifka, C. R., Eliceiri, K. W. Methods for quantifying fibrillar collagen alignment. Methods in Molecular Biology. 1627, 429-451 (2017).
- Liu, Y., et al. Fibrillar collagen quantification with curvelet transform based computational methods. Frontiers in Bioengineering and Biotechnology. 8, 198(2020).
- Conklin, M. W., et al. Collagen alignment as a predictor of recurrence after ductal carcinoma in situ. Cancer Epidemiology and Prevention Biomarkers. 27 (2), 138-145 (2018).
- Jallow, F., et al. Dynamic interactions between the extracellular matrix and estrogen activity in progression of ER+ breast cancer. Oncogene. 38 (43), 6913-6925 (2019).
- Smirnova, T., et al. Serpin E2 promotes breast cancer metastasis by remodeling the tumor matrix and polarizing tumor associated macrophages. Oncotarget. 7 (50), 82289(2016).
- Fanous, M., Keikhosravi, A., Kajdacsy-Balla, A., Eliceiri, K. W., Popescu, G. Quantitative phase imaging of stromal prognostic markers in pancreatic ductal adenocarcinoma. Biomedical Optics Express. 11 (3), 1354-1364 (2020).
- Jiménez-Torres, J. A., Virumbrales-Muñoz, M., Sung, K. E., Lee, M. H., Abel, E. J., Beebe, D. J. Patient-specific organotypic blood vessels as an in vitro model for anti-angiogenic drug response testing in renal cell carcinoma. EBioMedicine. 42, 408-419 (2019).
- Govindaraju, P., Todd, L., Shetye, S., Monslow, J., Puré, E. CD44-dependent inflammation, fibrogenesis, and collagenolysis regulates extracellular matrix remodeling and tensile strength during cutaneous wound healing. Matrix Biology. 75, 314-330 (2019).
- Henn, D., et al. Cryopreserved human skin allografts promote angiogenesis and dermal regeneration in a murine model. International Wound Journal. 17 (4), 925-936 (2020).
- Rico-Jimenez, J., et al. Non-invasive monitoring of pharmacodynamics during the skin wound healing process using multimodal optical microscopy. BMJ Open Diabetes Research and Care. 8 (1), 000974(2020).
- Israel, J. S., et al. Quantification of collagen organization after nerve repair. Plastic and Reconstructive Surgery Global Open. 5 (12), (2017).
- Rentchler, E. C., Gant, K. L., Drapkin, R., Patankar, M., Campagnola, P. J. Imaging collagen alterations in STICs and high grade ovarian cancers in the fallopian tubes by second harmonic generation microscopy. Cancers. 11 (11), 1805(2019).
- Hu, C., et al. Imaging collagen properties in the uterosacral ligaments of women with pelvic organ prolapse using spatial light interference microscopy (SLIM). Frontiers in Physics. 7, 72(2019).
- Guirado, E., et al. Disrupted protein expression and altered proteolytic events in hypophosphatemic dentin can be rescued by dentin matrix protein 1. Frontiers in Physiology. 11, 82(2020).
- Kiss, N., et al. Quantitative analysis on ex vivo nonlinear microscopy images of basal cell carcinoma samples in comparison to healthy skin. Pathology & Oncology Research. 25 (3), 1015-1021 (2019).
- Lewis, D. M., et al. Collagen fiber architecture regulates hypoxic sarcoma cell migration. ACS Biomaterials Science & Engineering. 4 (2), 400-409 (2018).
- Moura, C. C., Bourdakos, K. N., Tare, R. S., Oreffo, R. O., Mahajan, S. Live-imaging of Bioengineered Cartilage Tissue using Multimodal Non-linear Molecular Imaging. Scientific Reports. 9 (1), 1-9 (2019).
- Murtada, S. I., et al. Paradoxical aortic stiffening and subsequent cardiac dysfunction in Hutchinson-Gilford progeria syndrome. Journal of The Royal Society Interface. 17 (166), 0066(2020).
- Nichol, R. H., Catlett, T. S., Onesto, M. M., Hollender, D., Gómez, T. M. Environmental elasticity regulates cell-type specific RHOA signaling and neuritogenesis of human neurons. Stem Cell Reports. 13 (6), 1006-1021 (2019).
- Pointer, K. B., et al. Association of collagen architecture with glioblastoma patient survival. Journal of Neurosurgery. 126 (6), 1812-1821 (2016).
- Razavi, M. S., Leonard-Duke, J., Hardie, B., Dixon, J. B., Gleason, R. L. Axial stretch regulates rat tail collecting lymphatic vessel contractions. Scientific Reports. 10 (1), 1-11 (2020).
- Xue, Y., et al. Valve leaflet-inspired elastomeric scaffolds with tunable and anisotropic mechanical properties. Polymers for Advanced Technologies. 31 (1), 94-106 (2020).
- Zhou, Z. H., et al. Reorganized collagen in the tumor microenvironment of gastric cancer and its association with prognosis. Journal of Cancer. 8 (8), 1466(2017).
- Zinn, A., et al. The small GTPase RhoG regulates microtubule-mediated focal adhesion disassembly. Scientific Reports. 9 (1), 1-15 (2019).
- Zwaans, B. M., et al. Radiation cystitis modeling: A comparative study of bladder fibrosis radio-sensitivity in C57BL/6, C3H, and BALB/c mice. Physiological Reports. 8 (4), (2020).
- Devine, E. E., Liu, Y., Keikhosravi, A., Eliceiri, K. W., Jiang, J. J. Quantitative second harmonic generation imaging of leporine, canine, and porcine vocal fold collagen. The Laryngoscope. 129 (11), 2549-2556 (2019).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Zeitoune, A. A., et al. Epithelial ovarian cancer diagnosis of second-harmonic generation images: A semiautomatic collagen fibers quantification protocol. Cancer Informatics. 16, (2017).
- Wershof, E., et al. Matrix feedback enables diverse higher-order patterning of the extracellular matrix. PLoS Computational Biology. 15 (10), 1007251(2019).
- Mostaço-Guidolin, L. B., et al. Fractal dimension and directional analysis of elastic and collagen fiber arrangement in unsectioned arterial tissues affected by atherosclerosis and aging. Journal of Applied Physiology. 126 (3), 638-646 (2019).
- Thain, D., Tannenbaum, T., Livny, M. Distributed computing in practice: the Condor experience. Concurrency and Computation: Practice and Experience. 17 (2-4), 323-356 (2005).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados