Method Article
Quantification de l’organisation du collagène fibrillar avec des outils curvelet à base de transformation
Dans cet article
Résumé
Ici, nous présentons un protocole pour utiliser un outil logiciel MATLAB à base de transformation de courbes et open-source pour quantifier l’organisation du collagène fibrillar dans la matrice extracellulaire des tissus normaux et maladie. Cet outil peut être appliqué sur des images avec des fibres de collagène ou d’autres types de structures en ligne.
Résumé
Les collagènes fibrillaires sont des composants proéminents de matrice extracellulaire (ECM), et leurs changements de topologie se sont montrés associés à la progression d’un large éventail de maladies comprenant des cancers du sein, de l’ovaire, du rein, et du pancréas. Les outils logiciels de quantification des fibres librement disponibles sont principalement axés sur le calcul de l’alignement ou de l’orientation des fibres, et ils sont soumis à des limitations telles que l’exigence d’étapes manuelles, l’inexactitude dans la détection du bord de la fibre en arrière-plan bruyant, ou le manque de caractérisation localisée des fonctionnalités. L’outil de quantitation de fibre de collagène décrit dans ce protocole est caractérisé par l’utilisation d’une représentation optimale d’image multiscale permise par la transformation de courbe (CT). Cette approche algorithmique permet d’enlever le bruit des images de collagène fibrillaire et l’amélioration des bords de fibre pour fournir des informations d’emplacement et d’orientation directement à partir d’une fibre, plutôt que d’utiliser les informations indirectes pixel-sage ou fenêtre-sage obtenues à partir d’autres outils. Ce cadre basé sur le CT contient deux paquets distincts, mais liés, nommés « CT-FIRE » et « CurveAlign » qui peuvent quantifier l’organisation de la fibre sur une base globale, région d’intérêt (ROI), ou de fibre individuelle. Ce cadre de quantification a été développé depuis plus de dix ans et a maintenant évolué en une plate-forme complète et axée sur l’utilisateur quantification du collagène. En utilisant cette plate-forme, on peut mesurer jusqu’à une trentaine de caractéristiques de fibre, y compris les propriétés individuelles de fibre telles que la longueur, l’angle, la largeur et la droitité, ainsi que des mesures en vrac telles que la densité et l’alignement. En outre, l’utilisateur peut mesurer l’angle de fibre par rapport aux limites segmentées manuellement ou automatiquement. Cette plate-forme fournit également plusieurs modules supplémentaires, y compris ceux pour l’analyse du retour sur investissement, la création automatique des limites et le post-traitement. L’utilisation de cette plate-forme ne nécessite pas d’expérience préalable de la programmation ou du traitement d’image, et elle peut gérer de grands ensembles de données, y compris des centaines ou des milliers d’images, permettant une quantification efficace de l’organisation des fibres de collagène pour les applications biologiques ou biomédicales.
Introduction
Les collagènes fibrillaires sont des composants ECM structurels proéminents. Leur organisation modifie la fonction des tissus d’impact et sont probablement associés à la progression de nombreuses maladies allant de l’ostéogenèse imparfaite1, dysfonctioncardiaque 2, et la cicatrisationdes plaies 3 à différents types decancer,y compris lesein 4,5,6,7 ovarien,8, rein9, et les cancers du pancréas10. De nombreuses modalités d’imagerie établies peuvent être utilisées pour visualiser le collagène fibrillar tel que la microscopie de deuxième génération harmonique11,les taches ou les colorants en conjonction avec la microscopie à champ lumineux ou fluorescence ou la microscopie lumineuse polarisée12,la microscopie polarisation à base de cristaux liquides (LC-PolScope)13, et la microscopie électronique14. Comme l’importance de l’organisation du collagène fibrillaire est devenue plus claire, et l’utilisation de ces méthodes a augmenté, le besoin d’approches améliorées d’analyse des fibres de collagène a également augmenté.
Il y a eu beaucoup d’efforts pour développer des méthodes computationnelles pour la mesure automatisée du collagène fibrillar. Les outils logiciels librement disponibles sont principalement axés sur le calcul de l’alignement ou de l’orientation des fibres en adoptant soit le premier dérivé, soit le tenseur de structure pour les pixels15,16, ou l’analyse du spectre basé sur la transformation fourier pour les tuiles d’image17. Tous ces outils sont soumis à des limitations telles que l’exigence d’étapes manuelles, l’inexactitude dans la détection du bord de la fibre en arrière-plan bruyant, ou le manque de caractérisation localisée des fonctionnalités.
Par rapport à d’autres outils logiciels libres open source librement disponibles, les méthodes décrites dans ce protocole utilisent la Tomodensive — une méthode optimale, multiscale et directionnelle de représentation de l’image — pour éliminer le bruit des images de collagène fibrillaire et améliorer ou suivre les bords des fibres. Les informations sur l’emplacement et l’orientation peuvent être fournies directement à partir d’une fibre plutôt qu’en utilisant les informations indirectes pixel-sage ou fenêtre-sage pour déduire les mesures de l’organisation de fibre. Ce cadre basé sur la CT18,19,20,21 peut quantifier l’organisation de la fibre sur une base globale, roi, ou fibre, principalement via deux ensembles distincts, mais liés, nommés « CT-FIRE »18,21 et « CurveAlign »19,21. En ce qui concerne la mise en œuvre du logiciel, dans CT-FIRE, des coefficients de CT sur plusieurs échelles peuvent être utilisés pour reconstruire une image qui améliore les bords et réduit le bruit. Ensuite, un algorithme individuel d’extraction de fibres est appliqué à l’image reconstruite par CT pour suivre les fibres pour trouver leurs points de centre représentatifs, étendre les branches de fibre des points du centre, et relier les branches de fibre pour former un réseau de fibres. Dans CurveAlign, les coefficients de CT sur une échelle spécifiée par l’utilisateur peuvent être utilisés pour suivre l’orientation des fibres locales, où l’orientation et l’emplacement des courbes sont extraits et regroupés pour estimer l’orientation de la fibre aux emplacements correspondants. Ce cadre de quantification qui en résulte est développé depuis plus de dix ans et a beaucoup évolué dans de nombreux aspects tels que la fonctionnalité, l’interface utilisateur et la modularité. Par exemple, cet outil peut visualiser l’orientation locale de fibre et permet à l’utilisateur de mesurer jusqu’à trente dispositifs de fibre comprenant des propriétés individuelles de fibre telles que la longueur, l’angle, la largeur, et la droitivité, aussi bien que des mesures en vrac telles que la densité et l’alignement. En outre, l’utilisateur peut mesurer l’angle de fibre par rapport aux limites segmentées manuellement ou automatiquement, qui, par exemple, joue un rôle important dans le développement de biomarqueurs basés sur l’image dans le cancer dusein 22 et les études sur le cancer du pancréas10. Cette plate-forme fournit plusieurs modules de fonctionnalités, y compris ceux pour l’analyse du retour sur investissement, la création automatique des limites et le post-traitement. Le module roi peut être utilisé pour annoter différentes formes de retour sur investissement et effectuer une analyse de retour sur investissement correspondante. À titre d’exemple d’application, le module automatique de création de limites peut être utilisé pour enregistrer des images en champ lumineux d’hématoxyline et d’éosine (H&E) avec des images de deuxième génération harmonique (SHG) et générer le masque d’image des limites tumorales à partir des images H&E enregistrées. Le module de post-traitement peut faciliter le traitement et l’intégration des fichiers de données de sortie à partir d’images individuelles pour une analyse statistique possible.
Cette plate-forme de quantification ne nécessite pas d’expérience préalable de la programmation ou du traitement d’images et peut gérer de grands ensembles de données, y compris des centaines ou des milliers d’images, permettant une quantification efficace de l’organisation du collagène pour les applications biologiques ou biomédicales. Il a été largement utilisé dans différents domaines de recherche par de nombreux chercheurs du monde entier, y compris nous-mêmes. Il existe quatre publications principales sur CT-FIRE et CurveAlign18,19,20,21, dont les trois premières ont été citées 272 fois (en 2020-05-04 selon Google Scholar). Un examen des publications qui ont cité cette plate-forme (CT-FIRE ou CurveAlign) indique qu’environ 110 articles de revues l’ont directement utilisé pour leur analyse, dans lesquels environ 35 publications ont été en collaboration avec notre groupe, et les autres (~ 75) ont été écrites par d’autres groupes. Par exemple, cette plate-forme a été utilisée pour les études suivantes: cancer dusein 22,23,24, cancer du pancréas10,25, cancer du rein9,26, cicatrisation desplaies 3,27,28,29,30, cancer del’ovaire 8,31,7, ligament utérinacral32, dentine hypophosphatemique33, carcinome basocellulaire34, sarcome hypoxique35, tissu cartilagineux36, dysfonction cardiaque37, neurones38, glioblastome39, contractions lymphatiques40, cacffolds fibreux41, cancer gastrique42, microtubule43, et fibrose de la vessie44. La figure 1 démontre l’application d’imagerie cancéreuse de CurveAlign pour trouver les signatures de collagène associées à la tumeur du cancer dusein 19 à partir de l’image SHG. La figure 2 décrit un flux de travail schématique typique de cette plate-forme. Bien que ces outils aient été examinéstechniquement 18,19,21, et un protocole régulier20 pour l’analyse d’alignement avec CurveAlign est également disponible, un protocole visuel qui démontre toutes les caractéristiques essentielles pourrait être utile. Un protocole visualisé, tel que présenté ici, facilitera le processus d’apprentissage de l’utilisation de cette plate-forme ainsi que de répondre plus efficacement aux préoccupations et aux questions que les utilisateurs pourraient avoir.
Protocole
REMARQUE : Ce protocole décrit l’utilisation de CT-FIRE et curveAlign pour la quantification du collagène. Ces deux outils ont des objectifs principaux complémentaires, mais différents, et sont liés dans une certaine mesure. CT-FIRE peut être lancé à partir de l’interface CurveAlign pour effectuer la plupart des opérations, à l’exception d’une analyse avancée du post-traitement et du retour sur investissement. Pour une opération complète de CT-FIRE, il doit être lancé séparément.
1. Collection d’images et exigence d’image
REMARQUE : L’outil peut traiter n’importe quel fichier d’image avec des structures en ligne lisibles par MATLAB indépendamment de la modalité d’imagerie utilisée pour le collecter.
- Utilisez l’échelle grise 8 bits comme type d’image car les paramètres de fonctionnement par défaut sont basés sur ce format.
REMARQUE : L’imagerie SHG est une méthode d’imagerie par collagène fibrillaire sans étiquette et haute résolution largement utilisée. Les images SHG d’une étude sur le cancerdu sein 19 seront utilisées ici à des fins de démonstration.
2. Installation logicielle et exigence du système
REMARQUE : Les versions autonomes et source-code sont disponibles gratuitement. La version de code source nécessite une installation MATLAB complète comprenant des boîtes à outils de traitement de signal, de traitement d’image, d’analyse statistique et d’informatique parallèle. Pour exécuter la version code source, tous les dossiers nécessaires, y compris certains provenant de sources tierce, doivent être ajoutés au chemin MATLAB. L’utilisation de l’application autonome (APP) est recommandée pour la plupart des utilisateurs, ce qui nécessite l’installation d’un runtime compilateur MATLAB (MCR) librement disponible de la version spécifiée. La procédure d’installation et de lancement de l’APP est décrite ci-dessous.
- Téléchargez les paquets APP CT-FIRE version 3.0 (CTF3.0) et CurveAlign Version 5.0 (CA5.0) de https://eliceirilab.org/software/ctfire/ et https://eliceirilab.org/software/curvealign/, respectivement.
REMARQUE : Chaque paquet comprend les images app, manuelles et de test autonomes. - Suivez les exigences détaillées et les instructions d’installation des sites Web ci-dessus pour installer MATLAB MCR 2018b.
- Lancez l’APP.
- Pour un système Windows 64 bits, cliquez deux fois sur l’icône APP pour le lancer.
- Pour un système Mac, suivez les étapes suivantes pour le lancer : Cliquez à droite sur l’APP (ctrl-click) | Afficher le contenu du paquet | Contenu | MacOS | applauncher (clic droit et choisir ouvert).
REMARQUE : D’autres détails peuvent être vus dans les sites Web logiciels répertoriés dans 2.1.
3. Extraction individuelle de fibre avec CT-FIRE
REMARQUE : CT-FIRE utilise le CT pour dénoiser l’image, améliorer les bords de fibre, puis utilise un algorithme d’extraction de fibres pour suivre les fibres individuelles. La longueur, l’angle, la largeur et la droitité sont calculés pour les fibres individuelles.
- CT-FIRE sur une seule image ou plusieurs images
- Lancez l’APP tel que décrit dans 2.3.
- Cliquez sur le bouton Open File(s) dans l’interface utilisateur graphique principale (GUI, Figure 3A),puis sélectionnez une ou plusieurs piles d’images/ou d’images à partir de la fenêtre d’invite. Utilisez la technique appropriée pour que le système d’exploitation sélectionne plusieurs images dans le dialogue (par exemple, dans Windows, maintenez CTRL tout en sélectionnant plusieurs fichiers).
REMARQUE : Si deux fichiers d’image ou plus sont sélectionnés, toutes les images doivent utiliser les mêmes paramètres de fonctionnement pour l’analyse. Assurez-vous que toutes les images sont acquises dans les mêmes conditions ou dans des conditions similaires. - Sélectionnez des options informatiques parallèles en vérifiant la case à cocher Parallel dans le coin supérieur droit pour l’analyse de plusieurs images.
- Pour la pile d’images, déplacez le curseur de tranches sous la boîte de liste de fichiers pour sélectionner la tranche à analyser.
- Définissez les propriétés en cours d’exécution. Utilisez des paramètres par défaut pour une analyse initiale de certaines images. Si vous utilisez des paramètres par défaut, passez à l’étape 3.1.6. Pour définir différents paramètres, cliquez sur le bouton Mise à jour dans le panneau Paramètres. Suivez le manuel pour régler correctement les paramètres.
REMARQUE : Les paramètres les plus fréquemment ajustés incluent le seuild’arrière-plan (thresh_im2)et le rayon de recherche de nucléation(s_xlinkbox). Si le niveau de bruit de fond est élevé, réglez thresh_im2 à une plus grande valeur; s_xlinkbox est associé au rayon moyen des fibres, définir une plus petite valeur pour détecter les fibres minces. - Cliquez sur le bouton Exécuter.
REMARQUE : Les informations de progression seront affichées à la fois dans la fenêtre d’information et dans la fenêtre de commande. Une fois l’analyse terminée, la table de sortie sera affichée(figure 3B). - Cliquez sur n’importe quel élément de la table de sortie pour voir l’histogramme des mesures de fibre (figure 3C et figure 3F) de l’image, y compris la longueur, la largeur, l’angle et la droitité.
REMARQUE : Les images en fibre avec des fibres superposées sur l’image d’origine seront également affichées (Figure 3E). - Vérifiez le sous-foldeur nommé ctFIREout sous le dossier d’image pour les fichiers de sortie, y compris le fichier d’image superposé « .tiff », « .csv » fichier, et « .mat » fichier.
- Analyse de la région d’intérêt CT-FIRE (ROI)
- Annotation roi à l’aide de ROI Manager
- Cliquez sur le bouton Open File(s) de l’interface graphique principale(figure 3A)pour charger une ou plusieurs images.
- Sélectionnez l’image à annoter dans la liste des fichiers.
- Sélectionnez le gestionnaire de retour sur investissement dans le menu dropdown du panneau Options roi.
- Cliquez sur le bouton RUN pour lancer le module ROI Manager (Figure 3A).
- Dans l’interface graphique du gestionnaire roi (Figure 4A), cliquez sur le menu dropdown ci-dessous Draw ROI Menu(d) pour dessiner les ROM, un par un.
REMARQUE : La forme roi peut être rectangle, à main levée, ellipse, polygone ou rectangle spécifié. Suivez les instructions à l’écran pour dessiner, enregistrer et arrêter l’annotation du retour sur investissement. - Après avoir sélectionné la méthode pour dessiner le roi, faites glisser le rectangle jaune qui apparaît sur l’image d’origine à la position désirée, puis cliquez sur le bouton Save ROI(s) ou appuyez sur la touche s pour ajouter ce retour sur investissement à la liste roi. Ce retour sur investissement sera automatiquement nommé.
- Tirez un nouveau roi en faisant glisser le roi précédent à une nouvelle position, et enregistrez-le comme mentionné dans 3.2.1.6, ou répétez les étapes 3.2.1.5-3.2.1.6 pour tirer un nouveau roi.
- Appuyez sur x ou sélectionnez Nouveau ROI? dans le menu de dropdown forme ROI pour quitter l’annotation roi.
- Consultez les cases à cocher Afficher tous et les étiquettes pour afficher toutes les IA définies sur la liste et leurs noms sur l’image originale.
- Sélectionnez le retour sur investissement sur la liste roi pour mener des opérations de retour sur investissement de base,y compris Rename ROI , Delete ROI, Save ROI Text, Load ROI from Text, Save ROI Mask, Load ROI from Mask, et Combiner ROIs.
- Vérifiez le fichier de sortie du gestionnaire roi enregistré sous forme de fichier « .mat » dans un sous-foldeur nommé ROI_management le dossier d’image d’origine.
- Pour annoter une autre image dans la liste de fichiers ouverte, répétez les étapes 3.2.1.2-3.2.1.11.
- Une fois l’annotation terminée, fermez l’interface graphique du gestionnaire roi et réinitialisez l’interface graphique principale en cliquant sur le bouton Réinitialisation de l’interface graphique principale.
- Analyse du retour sur investissement pour une seule image dans ROI Manager
- Si l’analyse CT-FIRE à image complète est effectuée et que les résultats sont enregistrés dans l’annuaire par défaut, cliquez sur un ou plusieurs ROM de la liste roi, cliquez sur le bouton analyseur ctFIRE ROI pour lancer le module d’analyse Post-ROI.
REMARQUE : Les résultats seront automatiquement enregistrés dans un sous-foldeur situé à \\[dossier d’image]\ CTF_ROI\Individual\ROI_post_analysis\. - Dans la fenêtre contextée, cliquez sur le bouton Check Fibers pour afficher les fibres dans les IA sélectionnés (Figure 4B).
- Cliquez sur Les statistiques de parcelle pour afficher les histogrammes de chaque retour sur investissement( Figure 4C). Les chiffres de sortie correspondants seront affichés.
- Si l’analyse CT-FIRE complète de l’image n’a pas été effectuée, cliquez sur un ou plusieurs ROM de la liste roi et cliquez sur le bouton Appliquer ctFIRE sur roi pour appliquer directement l’analyse CT-FIRE aux ROM sélectionnés.
- Suivez les instructions dans la fenêtre d’invite pour exécuter l’analyse.
REMARQUE : Les paramètres d’exécution du CT-FIRE sont passés par l’interface graphique principale, et l’utilisateur peut mettre à jour les paramètres d’exécution tels que décrits à l’étape 3.1.5 au besoin. Une fois l’analyse terminée, les statistiques sommaires des mesures de fibre seront affichées dans le tableau de sortie. Les résultats seront automatiquement enregistrés dans un sous-foldeur situé à \\[dossier d’image]\ CTF_ROI\Individual\ROI_analysis\.
- Si l’analyse CT-FIRE à image complète est effectuée et que les résultats sont enregistrés dans l’annuaire par défaut, cliquez sur un ou plusieurs ROM de la liste roi, cliquez sur le bouton analyseur ctFIRE ROI pour lancer le module d’analyse Post-ROI.
- Analyse du retour sur investissement pour plusieurs images à l’aide de ROI Analyzer
- Suivez les étapes en 3.2.1 pour annoter les IA pour que les images soient analysées.
- Ouvrez une ou plusieurs images en cliquant sur le bouton Open File(s).
- Pour exécuter l’analyse de la publication roi lorsque les résultats complets de l’analyse d’image sont disponibles, cliquez sur le menu dropdown dans le panneau Options d’exécuter et sélectionnez l’analyseur post-ROI de la FCE.
- Cliquez sur le bouton RUN pour exécuter l’analyse du retour sur investissement pour toutes les images chargées.
- Vérifiez les informations de progression affichées dans la fenêtre de message en bas de l’interface graphique et dans la fenêtre de commande.
- Une fois l’analyse terminée, consultez les statistiques sommaires pour chaque retour sur investissement affiché dans un tableau de sortie.
REMARQUE : Les fichiers de sortie détaillés sont automatiquement enregistrés dans un sous-foldeur situé à \\ [dossier d’image]\CTF_ROI\Batch\ROI_post_analysis\. - Pour exécuter une analyse directe lorsque les résultats complets de l’analyse d’image ne sont pas disponibles, suivez les étapes 3.2.3.1-3.2.3.6, sauf qu’à l’étape 3.2.3.3, sélectionnez l’option CTF ROI analyserr; à l’étape 3.2.3.4, avant de cliquer sur le bouton RUN, mettez à jour les paramètres de fonctionnement décrits à l’étape 3.1.5. Après avoir cliqué sur le bouton RUN, dans une fenêtre de dialogue rapide, choisissez entre le roi rectangulaire et le masque roi de n’importe quelle forme.
REMARQUE : Si toutes les IA annotées sont rectangulaires, l’utilisateur peut choisir le « retour sur investissement rectangulaire ». Dans l’étape 3.2.3.6, les résultats de l’analyse roi sont enregistrés dans un sous-foldeur situé à \\[dossier d’image]\CTF_ROI\Batch\ROI_analysis\.
- Annotation roi à l’aide de ROI Manager
- Post-traitement avec CT-FIRE
REMARQUE : Après l’analyse CT-FIRE régulière décrite dans 3.1, l’utilisateur peut effectuer d’autres post-traitement. Sans exécuter l’extraction de fibre de temps encore, le post-traitement régulier, décrit dans 3.3.1, peut mettre à jour quelques propriétés de figure de sortie de base, tandis que le post-traitement avancé décrit dans 3.3.2 peut visualiser les fibres individuelles et leurs propriétés, exécuter le seuil complexe parmi les quatre propriétés de fibre, générer des statistiques sommaires des fibres sélectionnées, et visualiser les fibres sélectionnées à l’aide d’une carte de couleur personnalisée.- Post-traitement régulier avec CT-FIRE
- Lancez l’application CT-FIRE ou cliquez sur le bouton Réinitialisation après d’autres opérations pour paraphér l’interface graphique principale CT-FIRE (Figure 3A).
- Vérifiez la case à cocher .mat sur le dessus de l’interface graphique principale.
- Cliquez sur le bouton Open File(s) pour sélectionner le fichier CT-FIRE output .mat dans le sous-document ctFIREout.
REMARQUE : Si plusieurs fichiers sont sélectionnés, la case à cocher batch sera vérifiée automatiquement. Le nom du fichier des images correspondantes sera affiché dans la liste des boîtes. - Mettez à jour les options dans le panneau de contrôle des chiffres de sortie.
- Conservez les options par défaut dans options desortie , qui s’assurera que tous les fichiers de sortie seront mis à jour en fonction du nouvel ensemble de paramètres définis dans 3.3.1.4.
- Cliquez sur le bouton Post-traitement. Vérifiez les informations de progression dans la fenêtre de message au bas de l’interface graphique principale ainsi que dans la fenêtre de commande.
- Une fois l’analyse terminée, cliquez sur n’importe quel élément de la table de sortie pour voir l’histogramme des mesures de fibre de l’image, y compris la longueur, la largeur, l’angle et la droitité.
REMARQUE : Les nouveaux fichiers de sortie écraseront les anciens dans le sous-foldeur ctFIREout.
- Post-traitement avancé de CT-FIRE
- Lancez l’application CT-FIRE ou cliquez sur le bouton Réinitialisation après d’autres opérations pour paraphér l’interface graphique principale CT-FIRE (Figure 3A).
- Consultez la case à cocher OUT.adv en haut de l’interface graphique principale (Figure 3A).
- Cliquez sur le bouton Post-traitement pour lancer l’interface graphique post-traitement avancée nommée «Module d’analyse» (Figure 5A).
- Cliquez sur le bouton Sélectionnez fichier pour sélectionner une image.
- Cliquez sur le bouton Visualise Fibers pour entrer le numéro de fibre en fonction des étiquettes de la figure Tab Original-fibers.
REMARQUE : Les mesures des fibres sélectionnées seront affichées dans une table de sortie (figure 5B), et les fibres correspondantes seront superposées sur l’image originale indiquée dans la figure de l’onglet nommée Fibres mesurées (Figure 5C). - Cliquez sur le bouton Confirmer/Mise à jour pour passer à l’opération de seuil.
- Cochez la case de seuil pour activer les paramètres du seuil.
- Choisissez l’une des quatre options de seuil du menu dropdown.
- Entrez les seuils désirés dans le panneau Seuils pour une ou plusieurs propriétés de fibre.
- Cliquez sur le bouton Threshold Now pour appliquer les conditions de seuil ci-dessus.
- Vérifiez la figure rapide avec son nom se terminant par la visualisation des mesures pour voir les fibres sélectionnées superposées sur les images originales avec les cartes couleur personnalisées comme indiqué dans la figure 5E.
- Répétez les étapes 3.3.2.9-3.3.2.11 pour fixer les seuils souhaitables.
- Cliquez sur le bouton Enregistrer les fibres pour enregistrer les informations fibre sélectionnées.
REMARQUE : Les fibres sélectionnées correspondantes seront affichées dans la figure de l’onglet nommée After-Thresholding. - Cliquez sur le bouton Générer des statistiques, puis cliquez sur le bouton OK dans la fenêtre contexturée pour générer des statistiques sommaires.
REMARQUE : Une table de sortie( Figure 5D) affichera la valeur moyenne des fibres sélectionnées. D’autres statistiques des fibres sélectionnées seront enregistrées dans un fichier Excel dont l’emplacement est affiché dans la fenêtre d’état au bas de cette interface graphique. - Pour inclure les informations de fibre individuelles sélectionnées dans le fichier de sortie, cochez la feuille Générer pour la boîte de données brutes avant de cliquer sur le bouton OK.
- Pour combiner les résultats de plusieurs images, à l’étape 3.3.2.4, cochez la case mode batch ou le mode Stack et sélectionnez les multiples images ou stack(s) à analyser; sauter les étapes 3.3.2.5-3.3.2.6. Dans les étapes 3.3.2.8-3.3.2.9, définissez les conditions de seuil, mais comme le seuil de boutons maintenant et Save Fibers sont désactivés, sautez les étapes 3.3.2.10-3.3.2.13; et enfin, suivez les instructions dans l’étape 3.3.2.14 pour générer des statistiques sommaires et des propriétés individuelles de fibre des fibres sélectionnées.
- Post-traitement régulier avec CT-FIRE
4. Analyse de fibre avec CurveAlign
REMARQUE : CurveAlign a d’abord été développé pour mesurer automatiquement les angles des fibres par rapport aux limites définies par l’utilisateur. La version actuelle de CurveAlign peut être utilisée pour l’évaluation en vrac des caractéristiques basées sur la densité et l’alignement en plus de la mesure de l’angle relatif en chargeant les informations individuelles sur les fibres extraites par CT-FIRE ou en utilisant directement l’orientation locale des courbes. CurveAlign calcule jusqu’à trente fonctionnalités liées à des caractéristiques globales ou locales, y compris principalement la densité et l’alignement ainsi que les propriétés individuelles des fibres lorsque CT-FIRE est adopté comme méthode de suivi des fibres.
- Analyse des fibres avec des courbes
- Lancez l’APP tel que décrit dans 2.3.
- Cliquez sur le bouton Réinitialisation pour réinitialiser l’APP à son état initial si d’autres opérations ont été effectuées.
- Dans l’interface graphique principale (Figure 6A), vérifiez l’option méthode d’analyse des fibres pour vous assurer que CT est sélectionné (option par défaut).
REMARQUE : Dans ce mode, le CT est effectué sur l’image, et l’orientation de chaque courbe représente la direction d’une fibre à l’emplacement correspondant. - Cliquez sur le menu dropdown de la méthode Boundary et sélectionnez le mode de traitement des limites à partir des options de menu de dropdown suivantes : No Boundary, CSV Boundary et TIFF Boundary.
REMARQUE : Si aucune limite n’est nécessaire, sautez cette étape. Reportez-vous à 4,3 pour calculer les angles de fibre par rapport à une limite. - Cliquez sur le bouton Get Image(s) dans l’interface graphique principale(figure 6A),puis sélectionnez une ou plusieurs images/ou piles d’images à partir de la fenêtre d’invite. Utilisez la technique appropriée pour votre système d’exploitation pour sélectionner plusieurs images dans le dialogue (par exemple, dans Windows, maintenez CTRL tout en sélectionnant plusieurs fichiers).
REMARQUE : Si deux fichiers d’image ou plus sont sélectionnés, toutes les images doivent utiliser les mêmes paramètres d’exécution pour l’analyse. Assurez-vous que toutes les images sont acquises dans les mêmes conditions ou dans des conditions similaires. - Pour la pile d’images, déplacez le curseur de tranches sous la boîte de liste de fichiers pour sélectionner la tranche à analyser.
- Entrez la fraction des coefs à garder. Cette valeur est la fraction des plus grands coefficients de CT qui seront utilisés dans l’analyse des fibres.
REMARQUE : Si l’image a une grande variation dans l’intensité ou le contraste de fibre, annotez des régions d’intérêt avec le contraste même pour l’analyse de fibre car ce mode détecte seulement les fibres les plus lumineuses dans une image. En outre, plus la taille de l’image est grande, définir une valeur plus petite pour cette fraction. - Gardez tous les paramètres dans les options de sortie et d’autres dans l’option Avancée comme valeur par défaut; les fichiers de sortie peuvent être nécessaires dans d’autres opérations futures.
- Cliquez sur le bouton Exécuter en bas de l’interface graphique principale (Figure 6A).
REMARQUE : Les informations de progression seront affichées dans une fenêtre de message mise en surbrillance en couleur verte en bas. Une fois le processus terminé, certaines statistiques sommaires pour chaque image seront affichées dans la table de sortie(figure 6B),et tous les fichiers de sortie seront automatiquement enregistrés dans un sous-dossier nommé CA_Out dans l’annuaire de l’image originale. - Cliquez sur n’importe quel élément de la table de sortie (Figure 6B) pour voir l’histogramme (Figure 6E) ou l’intrigue de boussole (Figure 6F) des angles de fibre.
REMARQUE : L’image de superposition (figure 6C) et la carte thermique (figure 6D) de l’alignement ou de l’angle seront également affichées. - Cliquez sur le bouton Réinitialisation pour exécuter d’autres opérations ou fermez l’interface graphique principale pour quitter l’APPLICATION.
- Analyse individuelle des fibres avec CT-FIRE
REMARQUE : La procédure est la même que celle décrite à la section 4.1, sauf qu’à l’étape 4.1.3, sélectionnez le mode d’analyse des fibres liées au CT-FIREet sautez l’étape 4.1.7 car elle n’est pas applicable et est désactivée en mode CT-FIRE. Plus précisément, à l’étape 4.1.3, sélectionnez l’une des trois méthodes d’analyse de fibres individuelles basées sur le CT-FIRE suivantes :- Sélectionnez fibres CT-FIRE pour utiliser le point central de la fibre et l’angle de la fibre pour représenter la fibre.
REMARQUE : Cette option ne tient pas compte des changements dans l’orientation des fibres le long de la longueur de la fibre. - Sélectionnez les critères d’évaluation CT-FIRE pour utiliser les deux points d’évaluation d’une fibre et l’angle de fibre correspondant pour représenter la fibre.
REMARQUE : Par rapport à 4,2,1, cette option utilise deux positions pour représenter une fibre plutôt qu’une (point central de la fibre). - Sélectionnez segments CT-FIRE pour utiliser des segments d’une fibre pour représenter la fibre.
REMARQUE : Chaque segment a une longueur égale (réglée à 5 pixels par défaut dans CT-FIRE) ainsi que son orientation et son emplacement, ce qui reflète le changement d’orientation sur toute la longueur de la fibre. Cette option serait la plus longue, mais serait la meilleure option parmi les trois méthodes d’analyse de fibres à base de CT-FIRE pour suivre les changements dans l’orientation locale d’une fibre sinueuse.
- Sélectionnez fibres CT-FIRE pour utiliser le point central de la fibre et l’angle de la fibre pour représenter la fibre.
- Analyse relative de l’alignement avec la limite
REMARQUE : Par rapport à l’analyse régulière sans conditions limites décrites dans les sections 4.2 et 4.3, l’analyse relative de l’alignement avec les conditions limites doit être la suivante :- À l’étape 4.1.3, sélectionnez l’état limite du tiff.
REMARQUE : L’utilisateur aura besoin d’un fichier de limite correspondant pour chaque image ou chaque pile. Suivez les instructions à l’écran pour annoter manuellement soit un fichier de limite CSV (format de valeurs séparées de virgule, coordonnées x-y) ou un fichier de limite Tiff. Les fichiers limites créés dans CurveAlign seront automatiquement enregistrés en fonction de l’annuaire des fichiers et des conventions de nommage de fichiers décrites dans le manuel. Si une paire d’images H&E bright-field et SHG sont fournies, utilisez le module de création automatique des limites décrit à la section 4.4 pour générer le fichier limite. - Dans le panneau Paramètres primaires, entrez la distance par rapport à la limite la plus proche pour évaluer uniquement les fibres dans cette plage de distance.
- Dans le panneau Options de sortie, cochez la case d’association de limite Bdry Assoc pour visualiser le point sur la limite qui est associé à une fibre, un segment de fibre ou un curvelet.
- À l’étape 4.1.3, sélectionnez l’état limite du tiff.
- Création automatique des limites
- Lancez l’APP tel que décrit dans 2.3.
- Cliquez sur le bouton Réinitialisation pour réinitialiser l’APP à son état initial si d’autres opérations ont déjà été menées.
- Cliquez sur le bouton Création BD pour lancer le module de création de limites automatique.
- Suivez les instructions/indices à l’écran pour créer un fichier limite pour une ou plusieurs images basées sur une paire d’images H&E bright-field et SHG.
- Fermez la fenêtre du module ou cliquez sur le bouton Réinitialisation de l’interface graphique principale (Figure 6A) pour quitter ce module.
- CurveAlign région d’analyse d’intérêt
- Annotation roi à l’aide de ROI Manager
- Cliquez sur le bouton Get Image(s) dans l’interface graphique principale (Figure 6A) pour charger une ou plusieurs images.
- Sélectionnez l’image à annoter dans la liste des fichiers.
- Cliquez sur le gestionnaire roi pour lancer le module ROI Manager (Figure 7A).
- Suivez les étapes 3.2.1.5-3.2.1.13 dans la section 3.2.1.
- Analyse du retour sur investissement pour une seule image dans ROI Manager
- Si l’analyse courbe d’image complète a été effectuée et que les résultats sont enregistrés dans l’annuaire par défaut, cliquez sur une ou plusieurs IA dans la liste roi, puis cliquez sur le bouton Analyseur CA ROI pour exécuter l’analyse post-ROI.
REMARQUE : Une fois l’analyse terminée, des statistiques sommaires seront affichées dans un tableau de sortie (figure 7C) ainsi qu’une figure d’histogramme (figure 7D) montrant la répartition de l’angle. - Cliquez sur n’importe quel élément de la table de sortie pour visualiser les fibres dans un retour sur investissement donné (Figure 7B) ainsi que l’histogramme des angles de fibre.
- Vérifiez les fichiers de sortie enregistrés dans un sous-foldeur situé à \\[dossier d’image]\ CA_ROI\Individual\ROI_post_analysis\.
- Si l’analyse ca pleine image n’a pas été effectuée, cliquez sur un ou plusieurs ROM dans la liste roi, et cliquez sur le bouton Appliquer CA sur roi pour appliquer directement l’analyse CA aux ROM sélectionnés. Suivez les instructions dans la fenêtre d’invite pour exécuter l’analyse.
REMARQUE : Les paramètres d’exécution de l’analyse CA sont passés par l’interface graphique principale; mettre à jour les paramètres de fonctionnement décrits à l’étape 4.1.7 au besoin. Une fois l’analyse terminée, les résultats des statistiques sommaires des mesures de fibre seront affichés dans le tableau de sortie. Les résultats seront automatiquement enregistrés dans un sous-foldeur situé à \\[dossier d’image]\ CA_ROI\Individual\ROI_analysis\.
- Si l’analyse courbe d’image complète a été effectuée et que les résultats sont enregistrés dans l’annuaire par défaut, cliquez sur une ou plusieurs IA dans la liste roi, puis cliquez sur le bouton Analyseur CA ROI pour exécuter l’analyse post-ROI.
- Analyse du retour sur investissement pour plusieurs images à l’aide de ROI Analyzer
- Suivez les étapes en 4.5.1 pour annoter les IA pour que les images soient analysées.
- Ouvrez une ou plusieurs images en cliquant sur le bouton Get Image(s).
- Pour exécuter l’analyse de la publication roi lorsque les résultats complets de l’analyse d’image sont disponibles, cliquez sur le bouton Analyse roi et sélectionnez l’option ROI post-traitement.
- Vérifiez les informations de progression affichées dans la fenêtre de message en bas de l’interface graphique et dans la fenêtre de commande.
- Une fois l’analyse terminée, consultez les statistiques sommaires pour chaque retour sur investissement affiché dans un tableau de sortie.
REMARQUE : Les fichiers de sortie détaillés sont automatiquement enregistrés dans un sous-foldeur situé à \\ [dossier d’image]\CA_ROI\Batch\ROI_post_analysis\. - Pour exécuter une analyse directe avec le mode CT lorsque les résultats complets de l’analyse d’image ne sont pas disponibles, suivez les étapes 4.5.3.1-4.5.3.5, à l’exception des modifications suivantes : modifiez l’étape 4.5.3.3 en sélectionnant l’option CA sur le roi rectangulaire rectangulaire recadré ou CA sur masque avec roi de n’importe quelle forme. Si toutes les IA annotées sont de forme rectangulaire, choisissez l’option roi rectangulaire. Après l’étape 4.5.3.2, mettez à jour les paramètres de fonctionnement, tels que décrits dans 4.1.7.
REMARQUE : Les résultats de l’analyse roi seront enregistrés dans un sous-foldeur situé à \\ [dossier d’image]\CA_ROI\Batch\ROI_analysis\.
- Annotation roi à l’aide de ROI Manager
- Post-traitement de CurveAlign
- Lancez l’APP tel que décrit à la section 2.3.
- Cliquez sur le bouton Réinitialisation pour réinitialiser l’APP à son état initial si d’autres opérations ont déjà été effectuées.
- Cliquez sur le bouton Post-Processing pour lancer le module post-traitement.
- Suivez les instructions/indices à l’écran pour combiner les fonctionnalités de sortie ou les valeurs de différentes images.
- Fermez la fenêtre du module ou cliquez sur le bouton Réinitialisation de l’interface graphique principale (Figure 6A) pour quitter ce module.
5. Durée de fonctionnement estimée
- Attendez le temps de fonctionnement estimé pour le traitement d’une image d’une taille de 1024 pixels x 1024 pixels avec une densité de fibre modérée. Le temps de calcul réel dépend généralement de plusieurs facteurs, y compris la taille du fichier, le mode d’analyse, les fonctionnalités à déployer, le type d’unité centrale de traitement (Processeur) et la quantité de mémoire d’accès aléatoire (ou RAM) disponible. L’extraction individuelle de fibre de CT-FIRE prend quelques minutes. CurveAlign CT-mode sans limite prend quelques secondes. CurveAlign CT-FIRE fibre ou fibre se termine mode sans limite prend des dizaines de secondes. CurveAlign CT-FIRE mode fibre sans limite prend des centaines de secondes. L’analyse CurveAlign avec limite prend des dizaines de secondes à plusieurs minutes, selon la complexité des limites.
Résultats
Ces méthodes ont été appliquées avec succès dans de nombreuses études. Quelques applications typiques incluent : 1) Conklin et autres22 ont employé CurveAlign pour calculer des signatures de collagène tumeur-associées, et ont constaté que les fibres de collagène ont été plus fréquemment alignées perpendiculairement au périmètre de conduit dans les lésions in situ (DCIS) de carcinome ductal ; 2) Drifka et autres10 ont employé le mode CT-FIRE dans CurveAlign pour quantifier l’alignement stromal de collagène pour l’adénocarcinome ductal pancréatique et les tissus normaux/chroniques de pancréatite, et ont constaté qu’il y avait un alignement accru dans des tissus de cancer comparés à cela dans les tissus normaux/chroniques ; 3) Alkmin et autres7 ont employé CurveAlign pour quantifier la distribution angulaire des fibres de F-actin et l’alignement global de collagène des images de SHG du collagène stromal ovarien, et ont prouvé que la morphologie de matrice joue un rôle important dans la motilité de cellules de conduite et l’alignement de F-actin ; 4) LeBert et coll.3 ont appliqué CT-FIRE aux images SHG d’un modèle de réparation de blessure de poisson zèbre et ont trouvé une augmentation de l’épaisseur des fibres de collagène après blessure aiguë ; 5) Devine et coll.45 ont utilisé le mode CT-FIRE dans CurveAlign pour les images SHG de collagène pli vocal de différents modèles animaux pour mesurer les propriétés individuelles de la fibre et l’alignement global, et ont montré que le collagène vocal porcin et canin avait un alignement et une droité inférieurs à l’alignement et à la droitité plus élevés; 6) Keikhosravi et coll.13 ont utilisé CurveAlign pour quantifier l’alignement du collagène dans les échantillons d’histopathologie photographiés avec LC-PolScope, et ont montré que LC-PolScope et SHG sont comparables en termes d’alignement et de mesure d’orientation pour certains types de tissus.

Figure 1 : Utilisation de CurveAlign pour trouver des signatures de collagène associées à la tumeur à partir d’images SHG d’un microrésoré de tissu du cancer du sein humain (TMA). (A) Superposition d’un noyau TMA avec image SHG (jaune) superposée sur l’image de champ lumineux H&E correspondante. (B) La région d’intérêt de (A). (C) L’image de champ lumineux de (B). (D) L’image SHG de (B). (F) Le masque associé à l’image de champ lumineux (C). (E) L’image de superposition de sortie CurveAlign montrant les limites tumorales (jaune) de (F), emplacements de fibres représentatives, et l’orientation (lignes vertes); les lignes bleues sont utilisées pour associer les fibres à leurs limites les plus proches. Les flèches vertes dans (B) et ( E )montrentles fibres parallèles à la limite de la tumeur, tandis que les flèches rouges il montrent les fibres perpendiculaires à la frontière. La barre d’échelledans ( A) équivaut à 200 μm. Les images dans (B)-(F) sont affichées dans la même échelle, et la barre d’échelle représentative dans (D) équivaut à 50 μm. Abréviations : SHG = deuxième génération harmonique ; H&E = hématoxyline et éosine. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 2 : Flux de travail schématique de quantification d’une image fibrillaire de collagène. (A) Image SHG à analyser par CT-FIRE et/ou CurveAlign. (B) Superposition de sortie d’image par CT-FIRE. (C) Masque limite de (A) est une entrée curvealign en option. (D) Sortie d’image de superposition par CurveAlign. Les lignes de couleur dans (B) représentent les fibres extraites. Dans (D), les lignes vertes indiquent les emplacements et les orientations des fibres qui sont en dehors des limites (lignes jaunes) et sont dans la distance spécifiée de leurs limites les plus proches, les lignes rouges sont celles d’autres fibres, et les lignes bleues sont utilisées pour associer les fibres à leurs limites les plus proches. Les images dans (A)-(D) sont affichées dans la même échelle, et la barre d’échelle représentative dans (A) équivaut à 200 μm. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 3 : Analyse régulière de CT-FIRE. (A) Gui principal. (B) Tableau de sortie montrant les statistiques sommaires. (C) et (F) montrent les histogrammes d’angle et de largeur, respectivement. (E) Image de sortie montrant les fibres extraites (lignes de couleur) superposées sur l’image SHG d’origine (D). Abréviations : GUI = CT = transformation courbe; interface utilisateur graphique; SHG = deuxième génération harmonique. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
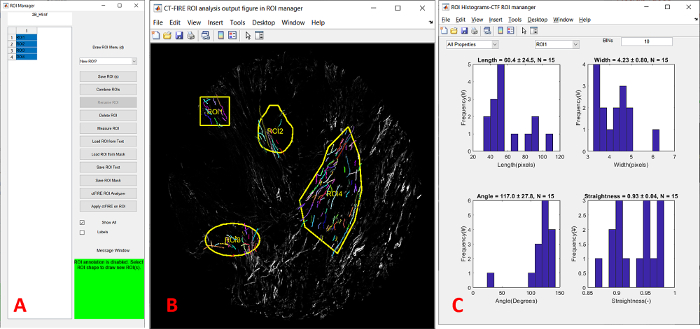
Figure 4 : Module de gestion du roi CT-FIRE. (A) Module GUI. (B) retour sur investissement post-analyse de quatre IA avec des formes différentes montrant les fibres dans chaque roi. (C) histogrammes roi de différentes propriétés de fibres. Abréviations : CT = transformation courbe; GUI = interface utilisateur graphique; ROI = région d’intérêt. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 5 : Module avancé de post-traitement CT-FIRE. (A) Module GUI. (B) Mesures de trois fibres sélectionnées. (C) Visualisation des trois fibres sélectionnées dans (B). (D) Statistiques sommaires après l’application d’un seuil de longueur (>60 pixels). (E) Visualisation des fibres sélectionnées dans ( D )avecbarre de couleur à base de longueur. Abréviations : CT = transformation courbe; GUI = interface utilisateur graphique. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 6 : Analyse régulière curvealign. (A) Gui principal. (B) Tableau de sortie montrant les statistiques sommaires. (C) Image de sortie montrant les emplacements et l’orientation des fibres représentatives (lignes vertes) et des limites (lignes jaunes) superposées sur l’image SHG d’origine, les lignes bleues sont utilisées pour associer les fibres à leurs limites les plus proches, les lignes rouges montrent les emplacements et les orientations des fibres à l’intérieur d’une limite ou à l’extérieur loin de la limite (>distance spécifiée par l’utilisateur, par exemple, 250 pixels ici). (D) Heatmap des angles: rouge (> 60 degrés), jaune (45-60] degrés, vert (10, 45] degrés. (E)-( F )montrent ladistribution d’angle à l’aide de l’histogramme et de l’intrigue de boussole, respectivement. Abréviations: GUI = interface utilisateur graphique; SHG = deuxième génération harmonique. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
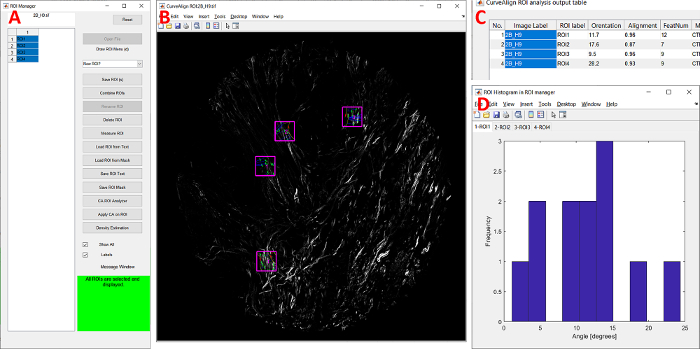
Figure 7 : Module de gestion du roi CurveAlign. (A) Le module GUI. (B) Quatre ROM rectangulaires annotées superposées à l’image d’origine. (C) tableau de sortie post-analyse roi. (D) Histogramme d’angle de chaque retour sur investissement. Abréviations: ROI = région d’intérêt; GUI = interface utilisateur graphique. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Discussion
Ce protocole décrit l’utilisation de CT-FIRE et CurveAlign pour la quantification du collagène fibrillar et peut être appliqué à n’importe quelle image avec des fibres de collagène ou d’autres structures allongées en forme de ligne ou de fibre adaptées à l’analyse par CT-FIRE ou CurveAlign. Par exemple, l’élastine ou les fibres élastiques peuvent être traitées de la même manière sur cette plate-forme. Nous avons testé les deux outils sur des fibres synthétiques générées par calcul21. Selon l’application, les utilisateurs doivent choisir le mode d’analyse le plus approprié pour leurs données. Le mode d’analyse des fibres CT peut utiliser directement des courbes en CT pour représenter l’emplacement et l’orientation des fibres, et il est sensible aux changements dans la structure des fibres locales. Le mode CT peut être utilisé pour localiser les fibres et leur orientation dans des conditions complexes, par exemple, lorsque le niveau de bruit est élevé, la fibre est sinueuse, ou la variation de l’épaisseur de la fibre est élevée. Cependant, comme le mode CT ne capte que les parties les plus brillantes d’une image, il manquerait certaines fibres avec une intensité inférieure lorsqu’il y a une grande variation de l’intensité de l’image.
En outre, le mode CT ne fournit pas d’informations sur les fibres individuelles. Contrairement au mode CT, le mode CT-FIRE calcule les propriétés individuelles des fibres et peut analyser toutes les fibres dont l’intensité est supérieure à un seuil spécifié. Les défis associés au mode CT-FIRE incluent : 1) la précision d’une extraction intacte de fibre peut être réduite ou compromise quand il y a une grande variation dans l’intensité le long d’une fibre ou de l’épaisseur de fibre à travers une image ; et 2) l’analyse standard actuelle est exigeante sur le plan informatique, comme mentionné dans le protocole. Plus de détails sur les avantages et les limites de ces méthodes peuvent être vus dans nos publicationsprécédentes 20,21.
En ce qui concerne la précision du suivi des fibres, l’utilisateur peut principalement compter sur l’inspection visuelle pour vérifier l’image qui se chevauche où les fibres extraites ou les orientations représentatives sont superposées sur l’image originale. En outre, pour CT-FIRE, l’utilisateur peut utiliser le module de post-traitement avancé pour identifier les propriétés de certaines fibres individuelles, et les comparer aux mesures en utilisant d’autres outils d’analyse d’image tels que Fidji46. Pour CurveAlign, l’utilisateur peut comparer les résultats d’orientation ou d’alignement à ceux calculés par d’autres outils tels que OrientationJ16 et CytoSpectre17.
Parmi les fonctionnalités disponibles pour la sortie par la plate-forme, les fonctionnalités liées à l’alignement sont les plus fréquemment utilisées et sont les plus fiables et robustes. Pour utiliser des caractéristiques de fibre individuelles, l’utilisateur doit confirmer l’extraction de caractéristiques de fibre représentatives. Il convient de noter qu’une fibre intacte peut être divisée en plusieurs segments plus courts dans certaines circonstances, que l’utilisateur doit prendre en considération lors de la sélection du mode d’analyse de la fibre ou de la réalisation d’une analyse statistique plus approfondie. Même lorsque la longueur de la fibre ne peut pas être directement utilisée comme propriété comparable, l’orientation ou la largeur des segments de fibres pondérés par rapport à leurs longueurs peuvent encore indiquer des informations utiles. En ce qui concerne l’imagerie SHG, l’ouverture numérique (NA) de la lentille objective peut affecter considérablement la détection de la largeur et de la longueur d’une fibre, mais elle a moins d’impact sur les mesures d’orientation et d’alignement. Dans notre expérience dans l’imagerie SHG, nous avons généralement besoin d’utiliser objectif objectif avec grossissement 40x ou plus avec NA ≥ 1.0 pour atteindre une mesure robuste de l’épaisseur de la fibre.
L'« alignement » peut être interprété de trois façons différentes : 1) l’alignement par rapport à la direction horizontale positive appelée « angle », allant de 0 à 180 degrés, où les angles proches de 0 ont une orientation similaire à des angles proches de 180 degrés; 2) alignement par rapport à une limite appelée « angle relatif », allant de 0 à 90 degrés, avec 0 degrés indiquant une fibre parallèle à la limite et 90 indiquant une fibre perpendiculaire; et 3) l’alignement des fibres les uns par rapport aux autres appelé « coefficient d’alignement », allant de 0 à 1, avec 1 indiquant des fibres parfaitement alignées et des valeurs plus petites représentant des fibres plus distribuées au hasard.
Outre les caractéristiques fibre calculées dans cette plate-forme, certaines mesures basées sur l’analyse de texture47,48,49 ont également été proposées pour quantifier les modèles ECM. Ces fonctionnalités liées à la texture peuvent fournir un descripteur alternatif ou supplémentaire de l’ECM dans certaines applications. Les défis pour le développement de ce type de mesures résident dans l’interopération de la pertinence biologique possible, la caractérisation localisée, et l’exactitude du traçage des fibres individuelles.
Pour optimiser les paramètres d’exécution et effectuer le dépannage, l’utilisateur peut se référer au manuel, aux publicationspertinentes 20,21 ainsi qu’aux barres latérales FAQ sur les pages Wiki GitHub du référentiel curvelets : https://github.com/uw-loci/curvelets/wiki. Pour certains boutons, un indice de fonction peut apparaître pour guider l’utilisateur pour l’opération en cours ou suivante lorsque l’utilisateur déplace l’icône de la souris au-dessus d’un bouton. Suivez les informations sur l’interface graphique ou la fenêtre de commande pour effectuer le dépannage.
Pour traiter un grand ensemble de données, l’utilisateur est encouragé à utiliser des options informatiques parallèles, qui permettent à l’outil de traiter plusieurs images simultanément. Une option est d’utiliser plusieurs cœurs CPU s’il est disponible sur l’ordinateur utilisé. Alternativement, une version sans tête des deux APPs est fournie et a été compilée avec succès dans le nœud de compilation à travers le serveur détenu auCONDOR 50 Center for High Throughput Computing (CHTC) à l’Université du Wisconsin-Madison. Le workflow CHTC pour la quantification des fibres à grande échelle a été développé, testé et utilisé avec succès sur des ensembles d’images réelles composés de milliers d’images. L’utilisateur pourrait adapter les fonctions MATLAB sans tête de CT-FIRE et CurveAlign pour exécuter la quantification sur d’autres systèmes de cloud computing, y compris les services commerciaux tels que ceux offerts par Amazon, Google et Microsoft.
Les orientations de développement en cours et à l’avenir comprennent: 1) l’incorporation d’un réseau neuronal d’apprentissage profond pour extraire ou générer des images synthétiques de haute qualité en fibre de collagène et améliorer la robustesse et la précision de l’algorithme de traçage des fibres; 2) l’intégration de tous les modules dans une plate-forme complète tout en optimisant le code et la documentation en suivant les meilleures pratiques d’ingénierie logicielle; 3) déploiement de toutes les fonctionnalités de base sur une plate-forme de cloud computing; 4) amélioration du flux de travail de l’analyse des fibres à l’aide du service CHTC; et 5) l’amélioration de la fonctionnalité du générateur de fibres synthétiques.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions de nombreux contributeurs et utilisateurs à CT-FIRE et CurveAlign au fil des ans, dont le Dr Rob Nowak, le Dr Carolyn Pehlke, le Dr Jeremy Bredfeldt, Guneet Mehta, Andrew Leicht, le Dr Adib Keikhosravi, le Dr Matt Conklin, le Dr Jayne Squirrell, le Dr Paolo Provenzano, la Dre Brenda Ogle, la Dre Patricia Keely, le Dr Joseph Szulczewski, la Dre Suzanne Ponik et d’autres contributions techniques de Swati Anand et Curtis Rueden. Ces travaux ont été soutenus par le financement de Semiconductor Research Corporation, Morgridge Institute for Research et NIH subventions R01CA199996, R01CA181385 et U54CA210190 à K.W.E.
matériels
| Name | Company | Catalog Number | Comments |
| CT-FIRE | Univerity of Wisconsin-Madison | N/A | open source software available from https://eliceirilab.org/software/ctfire/ |
| CurveAlign | University of Wisconsin-Madison | N/A | open source software available from https://eliceirilab.org/software/curvealign/ |
Références
- Nadiarnykh, O., et al. Second harmonic generation imaging microscopy studies of osteogenesis imperfecta. Journal of Biomedical Optics. 12 (5), 051805(2007).
- Kouris, N. A., et al. A nondenatured, noncrosslinked collagen matrix to deliver stem cells to the heart. Regenerative Medicine. 6 (5), 569-582 (2011).
- LeBert, D. C., et al. Matrix metalloproteinase 9 modulates collagen matrices and wound repair. Development. 142 (12), 2136-2146 (2015).
- Provenzano, P. P., et al. Collagen reorganization at the tumor-stromal interface facilitates local invasion. BMC Medicine. 4 (1), 38(2006).
- Provenzano, P. P., et al. Collagen density promotes mammary tumor initiation and progression. BMC Medicine. 6 (1), 1(2008).
- Conklin, M. W., et al. Aligned collagen is a prognostic signature for survival in human breast carcinoma. The American Journal of Pathology. 178 (3), 1221-1232 (2011).
- Alkmin, S., et al. Migration dynamics of ovarian epithelial cells on micro-fabricated image-based models of normal and malignant stroma. Acta Biomaterialia. 100, 92-104 (2019).
- Campbell, K. R., Campagnola, P. J. Assessing local stromal alterations in human ovarian cancer subtypes via second harmonic generation microscopy and analysis. Journal of Biomedical Optics. 22 (11), 116008(2017).
- Best, S. L., et al. Collagen organization of renal cell carcinoma differs between low and high grade tumors. BMC Cancer. 19 (1), 490(2019).
- Drifka, C. R., et al. Periductal stromal collagen topology of pancreatic ductal adenocarcinoma differs from that of normal and chronic pancreatitis. Modern Pathology. 28 (11), 1470-1480 (2015).
- Campagnola, P. J., et al. Three-dimensional high-resolution second-harmonic generation imaging of endogenous structural proteins in biological tissues. Biophysical Journal. 82 (1), 493-508 (2002).
- Arun Gopinathan, P., et al. Study of collagen birefringence in different grades of oral squamous cell carcinoma using picrosirius red and polarized light microscopy. Scientifica. 2015, 1-7 (2015).
- Keikhosravi, A., et al. Quantification of collagen organization in histopathology samples using liquid crystal based polarization microscopy. Biomedical Optics Express. 8 (9), 4243-4256 (2017).
- Quan, B. D., Sone, E. D. Cryo-TEM analysis of collagen fibrillar structure. Methods in Enzymology. 532, 189-205 (2013).
- Boudaoud, A., et al. FibrilTool, an ImageJ plug-in to quantify fibrillar structures in raw microscopy images. Nature Protocols. 9 (2), 457-463 (2014).
- Rezakhaniha, R., et al. Experimental investigation of collagen waviness and orientation in the arterial adventitia using confocal laser scanning microscopy. Biomechanics and Modeling in Mechanobiology. 11 (3-4), 461-473 (2012).
- Kartasalo, K., et al. CytoSpectre: a tool for spectral analysis of oriented structures on cellular and subcellular levels. BMC Bioinformatics. 16 (1), 1(2015).
- Bredfeldt, J. S., et al. Computational segmentation of collagen fibers from second-harmonic generation images of breast cancer. Journal of Biomedical Optics. 19 (1), 016007(2014).
- Bredfeldt, J. S., et al. Automated quantification of aligned collagen for human breast carcinoma prognosis. Journal of Pathology Informatics. 5 (1), 28(2014).
- Liu, Y., Keikhosravi, A., Mehta, G. S., Drifka, C. R., Eliceiri, K. W. Methods for quantifying fibrillar collagen alignment. Methods in Molecular Biology. 1627, 429-451 (2017).
- Liu, Y., et al. Fibrillar collagen quantification with curvelet transform based computational methods. Frontiers in Bioengineering and Biotechnology. 8, 198(2020).
- Conklin, M. W., et al. Collagen alignment as a predictor of recurrence after ductal carcinoma in situ. Cancer Epidemiology and Prevention Biomarkers. 27 (2), 138-145 (2018).
- Jallow, F., et al. Dynamic interactions between the extracellular matrix and estrogen activity in progression of ER+ breast cancer. Oncogene. 38 (43), 6913-6925 (2019).
- Smirnova, T., et al. Serpin E2 promotes breast cancer metastasis by remodeling the tumor matrix and polarizing tumor associated macrophages. Oncotarget. 7 (50), 82289(2016).
- Fanous, M., Keikhosravi, A., Kajdacsy-Balla, A., Eliceiri, K. W., Popescu, G. Quantitative phase imaging of stromal prognostic markers in pancreatic ductal adenocarcinoma. Biomedical Optics Express. 11 (3), 1354-1364 (2020).
- Jiménez-Torres, J. A., Virumbrales-Muñoz, M., Sung, K. E., Lee, M. H., Abel, E. J., Beebe, D. J. Patient-specific organotypic blood vessels as an in vitro model for anti-angiogenic drug response testing in renal cell carcinoma. EBioMedicine. 42, 408-419 (2019).
- Govindaraju, P., Todd, L., Shetye, S., Monslow, J., Puré, E. CD44-dependent inflammation, fibrogenesis, and collagenolysis regulates extracellular matrix remodeling and tensile strength during cutaneous wound healing. Matrix Biology. 75, 314-330 (2019).
- Henn, D., et al. Cryopreserved human skin allografts promote angiogenesis and dermal regeneration in a murine model. International Wound Journal. 17 (4), 925-936 (2020).
- Rico-Jimenez, J., et al. Non-invasive monitoring of pharmacodynamics during the skin wound healing process using multimodal optical microscopy. BMJ Open Diabetes Research and Care. 8 (1), 000974(2020).
- Israel, J. S., et al. Quantification of collagen organization after nerve repair. Plastic and Reconstructive Surgery Global Open. 5 (12), (2017).
- Rentchler, E. C., Gant, K. L., Drapkin, R., Patankar, M., Campagnola, P. J. Imaging collagen alterations in STICs and high grade ovarian cancers in the fallopian tubes by second harmonic generation microscopy. Cancers. 11 (11), 1805(2019).
- Hu, C., et al. Imaging collagen properties in the uterosacral ligaments of women with pelvic organ prolapse using spatial light interference microscopy (SLIM). Frontiers in Physics. 7, 72(2019).
- Guirado, E., et al. Disrupted protein expression and altered proteolytic events in hypophosphatemic dentin can be rescued by dentin matrix protein 1. Frontiers in Physiology. 11, 82(2020).
- Kiss, N., et al. Quantitative analysis on ex vivo nonlinear microscopy images of basal cell carcinoma samples in comparison to healthy skin. Pathology & Oncology Research. 25 (3), 1015-1021 (2019).
- Lewis, D. M., et al. Collagen fiber architecture regulates hypoxic sarcoma cell migration. ACS Biomaterials Science & Engineering. 4 (2), 400-409 (2018).
- Moura, C. C., Bourdakos, K. N., Tare, R. S., Oreffo, R. O., Mahajan, S. Live-imaging of Bioengineered Cartilage Tissue using Multimodal Non-linear Molecular Imaging. Scientific Reports. 9 (1), 1-9 (2019).
- Murtada, S. I., et al. Paradoxical aortic stiffening and subsequent cardiac dysfunction in Hutchinson-Gilford progeria syndrome. Journal of The Royal Society Interface. 17 (166), 0066(2020).
- Nichol, R. H., Catlett, T. S., Onesto, M. M., Hollender, D., Gómez, T. M. Environmental elasticity regulates cell-type specific RHOA signaling and neuritogenesis of human neurons. Stem Cell Reports. 13 (6), 1006-1021 (2019).
- Pointer, K. B., et al. Association of collagen architecture with glioblastoma patient survival. Journal of Neurosurgery. 126 (6), 1812-1821 (2016).
- Razavi, M. S., Leonard-Duke, J., Hardie, B., Dixon, J. B., Gleason, R. L. Axial stretch regulates rat tail collecting lymphatic vessel contractions. Scientific Reports. 10 (1), 1-11 (2020).
- Xue, Y., et al. Valve leaflet-inspired elastomeric scaffolds with tunable and anisotropic mechanical properties. Polymers for Advanced Technologies. 31 (1), 94-106 (2020).
- Zhou, Z. H., et al. Reorganized collagen in the tumor microenvironment of gastric cancer and its association with prognosis. Journal of Cancer. 8 (8), 1466(2017).
- Zinn, A., et al. The small GTPase RhoG regulates microtubule-mediated focal adhesion disassembly. Scientific Reports. 9 (1), 1-15 (2019).
- Zwaans, B. M., et al. Radiation cystitis modeling: A comparative study of bladder fibrosis radio-sensitivity in C57BL/6, C3H, and BALB/c mice. Physiological Reports. 8 (4), (2020).
- Devine, E. E., Liu, Y., Keikhosravi, A., Eliceiri, K. W., Jiang, J. J. Quantitative second harmonic generation imaging of leporine, canine, and porcine vocal fold collagen. The Laryngoscope. 129 (11), 2549-2556 (2019).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Zeitoune, A. A., et al. Epithelial ovarian cancer diagnosis of second-harmonic generation images: A semiautomatic collagen fibers quantification protocol. Cancer Informatics. 16, (2017).
- Wershof, E., et al. Matrix feedback enables diverse higher-order patterning of the extracellular matrix. PLoS Computational Biology. 15 (10), 1007251(2019).
- Mostaço-Guidolin, L. B., et al. Fractal dimension and directional analysis of elastic and collagen fiber arrangement in unsectioned arterial tissues affected by atherosclerosis and aging. Journal of Applied Physiology. 126 (3), 638-646 (2019).
- Thain, D., Tannenbaum, T., Livny, M. Distributed computing in practice: the Condor experience. Concurrency and Computation: Practice and Experience. 17 (2-4), 323-356 (2005).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon