Method Article
Investigação de dependências genéticas utilizando ensaios de competição baseados em CRISPR-Cas9
Neste Artigo
Resumo
Este manuscrito descreve um método de CRISPR-Cas9-baseado em cluster regularmente intercaladas curta palíndromo repete (CRISPR) para investigação célere e simples do papel dos vários genes candidatos na proliferação de células de leucemia mieloide aguda (LMA) em paralelo. Esta técnica é escalável e pode ser aplicada em outras linhas de células de câncer também.
Resumo
Estudos de perturbação do gene têm sido amplamente utilizados para investigar o papel dos genes individuais na patogênese da LMA. Para alcançar a interrupção completa do gene, muitos desses estudos fizeram uso de modelos de complexo gene nocaute. Enquanto estes estudos com camundongos knockout para oferecer um sistema elegante e já testado para investigar as relações genótipo-para-fenótipo, um método rápido e escalável para avaliar genes candidatos que jogar um papel na proliferação celular de LMA ou sobrevivência em modelos de LMA ajudará a acelerar o interrogatório paralelo de múltiplos genes candidatos. Recentes avanços em tecnologias de edição de genoma melhoraram drasticamente nossa capacidade de executar perturbações genéticas em uma escala sem precedentes. Um tal sistema de edição de genoma é o método CRISPR Cas9-baseada que pode ser usado para fazer alterações rápidas e eficazes no genoma de célula de destino. A facilidade e a escalabilidade do gene-exclusão CRISPR/Cas9-mediada torna uma das técnicas mais atraentes para o interrogatório de um grande número de genes em ensaios fenotípicos. Aqui, apresentamos um ensaio simples usando CRISPR/Cas9 mediada gene-interrupção combinado com alta produtividade fluxo cytometry-concorrência baseada nos ensaios para investigar o papel dos genes que podem desempenhar um papel importante na proliferação ou sobrevivência de humanos e linhas de célula murino AML.
Introdução
Nas últimas décadas tem visto inúmeros esforços de pesquisa focados em identificar a contribuição das principais vias moleculares na patogênese da leucemia mieloide aguda (LMA). Tradicionalmente, gene-interrupção nas células da LMA foi realizado utilizando camundongos knockout condicional ou RNA curto-gancho de cabelo (shRNA). Enquanto ratos do KO oferecem um sistema sofisticado para controle espaço-temporal de gene-exclusão, gerar os ratos nocaute do gene é trabalhoso, demorado e caro. Além disso, gene-nocautes usar estratégias de recombinação não é facilmente escalável; Estas estratégias não se prestam bem para o interrogatório de vários genes em paralelo. Após a descoberta de métodos de interferência do RNA de mRNAs endógenos de knock-down usando o RNA de interferência pequeno (siRNA) ou shRNA, muitos grupos começaram a usar técnicas de interferência do RNA para investigar o papel de genes específicos na LMA. Desde que as células de LMA murino e humanas são notoriamente difíceis de transfect usando métodos tradicionais baseados em lipídios transfeccao, maioria estuda shRNA trabalhador assalariado, lentivirally ou retrovirally-codificado para estudar a função dos genes em células de LMA. A recente descoberta do cluster regularmente intercaladas curtas palíndromos repetições (CRISPR) e as associado nucleases de Cas (CRISPR-Cas9) revolucionou a gene-alvo tecnologias1,2,3. Usando CRISPR-Cas9, genes específicos ou regiões genômicas podem ser excluídas, edição ou marcadas com facilidade e eficiência. CRISPR Cas9-gene-edição baseada está a emergir como o método de escolha para investigar relações genótipo-para-fenótipo em tipos de células diferentes devido a simplicidade, eficácia e ampla aplicabilidade desta técnica. Métodos baseados em CRISPR-Cas9 também estão se tornando o método de escolha na LMA, não só para interrogar os genes individuais, mas também como uma maneira de atingir múltiplos genes na matriz ou em pool genéticos telas destinadas a investigar vários genes em paralelo como potencial AML-dependências4,5,6.
Este manuscrito, descrevemos um ensaio de crescimento competitivo simples para medir o impacto do gene-perturbação no crescimento de células da LMA, com base em estável CRISPR Cas9-mediada por gene-edição seguido por citometria de fluxo elevado-throughput. Este método é simples, eficiente e escalável para médio-taxa de transferência experiências para investigar o papel de vários genes em paralelo nas células da LMA.
Protocolo
1. gerar Clones de linha celular AML com alta expressão de Cas9 estável e ativo
- Produção de lentivirus Cas9
- Dia 0: Placa de 4 x 106 293T células em 10 mL de DMEM com 10% de soro fetal bovino (FBS) e a penicilina e a L-glutamina em um prato de cultura de tecido de 10 cm em um nível 2 de biossegurança (BSL2) certificado capa de cultura de células. Coloque o prato em uma incubadora de 37 ° C.
- Dia 1: As células 293T chapeado devem ser confluente de 70 a 80% no dia 1. Execute o transfeccao usando o protocolo seguinte à tarde.
- Aquecer a transfeccao, meios de cultura e reagente de transfeccao à temperatura ambiente. Descongele todos os plasmideos exigidos para transfeccao.
- Mix 9 µ g de psPAX2, 0,9 µ g de pMD2.G e 9 µ g de pLenti-Cas9 plasmídeos com 500 µ l de meio de transfeccao em um tubo de 5 mL.
- Adicione 1,7 mL de meio de Transfeccao para um 14 mL redonda tubo inferior. Adicione 57 µ l de reagente de transfeccao diretamente no meio de transfeccao dentro do tubo para evitar tocar a parede do tubo.
- Acrescente a mistura inteira do plasmídeo para solução de reagente de transfeccao e misture batendo suavemente o tubo do lado. Incube a mistura à temperatura ambiente por 20 a 30 min. Enquanto isso, mude o meio da placa de 293T semeado.
- Adicione a mistura de transfeccao, gota a gota, a placa e agitar suavemente a placa para o lado para uma mistura eficiente. Coloque a placa em uma incubadora de 37 ° C.
- Dia 2: Aspire o sobrenadante da placa de transfeccao suavemente tal que as células não são perturbadas ou desalojadas da placa. Desprezar o sobrenadante. Adicione 10 mL de meio fresco de DMEM 10% deslizando suavemente para baixo dos lados da placa para evitar desalojar as células.
- Dia 3: Colete o vírus contendo sobrenadante da placa de transfeccao lentamente por desenhá-lo para uma seringa estéril de 10 cm. Depois que o sobrenadante é recolhido para a seringa, coloque um filtro estéril de µM 0.45 a seringa e mantenha a seringa e o filtro sobre um tubo cónico polipropileno de fresco, estéril 15 mL. Mergulhar suavemente a seringa para filtrar o vírus contendo sobrenadante através do filtro de 0,45 µM no tubo polipropileno cônico de 15 mL.
- Loja meio condicionado viral em alíquotas de 2 mL em 2 mL cryovials a-80 ° C.
- Transdução da linha celular de LMA
- Dia -1: Diluir 1 mg/mL caldo de fragmento fibronectina humana recombinante a 10 µ g/mL com solução salina tampão fosfato (PBS). Casaco de uma cultura de não-tecidos tratados 6 placa bem com 2 mL da solução de trabalho de 10 µ g/mL fibronectina humana recombinante fragmento em uma capa de cultura de células BSL2 aprovado. Embrulhe o prato em filme plástico para evitar a perda por evaporação e armazenar à temperatura ambiente durante a noite.
- Dia 0: Descongele o sobrenadante viral contendo Cas9. Aspire a solução do fragmento recombinante humano fibronectina completamente da placa revestida antes spinfection. Adicionar 2 mL do meio condicionado viral com Cas9 à placa e girá-lo em 1.300 x g durante 90 min a 35 ° C. Isso ajuda as partículas virais anexar para o spinfected bem.
- Enquanto isso, contar as células para ser transformados e spin-down 2 x 106 células em um tubo cónico polipropileno de 15 mL. Aspirar o sobrenadante e ressuspender o precipitado em 2 mL de meio de cultura fresco.
- Após spinfection, retire todo o sobrenadante viral a spinfected bem e descarte. Retrovirais partículas do líquido sobrenadante estão ligadas à parte inferior do spinfected bem. Para isso, adicione as 2 x 106 células resuspended em 2 mL do meio da etapa acima.
- Gire a placa novamente a 1.300 x g muito rapidamente (1 a 2 min) suficiente para permitir que as células de assentar no fundo. Coloque a placa de volta para a incubadora e deixar durante a noite para transdução com partículas virais spinfected anexadas ao poço.
- Dia 1: Dependendo da densidade da célula, adicione mais meio às células transduzidas para evitar o crescimento excessivo.
- Dia 2: Desde o plasmídeo pLenti-Cas9 tem um marcador de resistência Blasticidin, adicione Blasticidin em uma dose de 10 µ g/mL para transduzidas e untransduced (controle) MOLM13 ou células de leucemia do MLL-AF9 para a seleção de Cas9 expressando as células.
Nota: Seleção de Blasticidin dos Cas9-transfectadas MOLM13 ou MLL-AF9 células de leucemia é considerada completa quando todas as células de untransduced controle foram eliminadas. Para linhas de célula diferente MOLM13 e MLL-AF9 leucemia, uma curva de resposta de dose deve ser realizada antes desta experiência para que uma dose ideal pode ser empregada. Titulação do sobrenadante viral também pode ser executada no caso de taxas de baixo-transdução.
- Seleção de células expressando alta-Cas9 de clone.
Nota: Notamos que em algumas linhas de célula AML, seleção de clone pode não ser necessária: a maior população de Cas9-Blasticidin selecionado já tem alta eficiência de edição de genoma. Neste caso, é possível pular a etapa 1.3 e passar para a etapa 1.4 para avaliar Cas9 expressão nas células AML granel Cas9-blasticidin pela mancha ocidental. Ainda seria importante avaliar a eficiência de edição de gene naquelas celas de granel Cas9-AML (passo 1.5) antes de prosseguir para os concurso de ensaios. Supomos que a seleção de clones de alta-Cas9 única reduz a variabilidade de edição de genoma, que é especialmente importante quando o teste um número de guia único diferente RNAs (sgRNAs).- Realize uma única célula tipo do Blasticidin-Cas9 selecionado as células de leucemia MOLM13 e MLL-AF9 doravante chamadas MOLM13-Cas9 e células MLL-AF9-Cas9, respectivamente, usando um classificador de FACS contido em uma capa de BSL2 aprovado. Classificação pode ser realizada em 5 – 10 fundo redondo cultura de não-tecidos 96 bem placas tratadas.
- Uma vez única MOLM13-Cas9 e clones de MLL-AF9-Cas9 tem sido escolhidos e nomeado individualmente, expanda clones de 10 – 20 com 10 µ g/mL de Blasticidin em meios de cultura para garantir a manutenção da expressão Cas9.
- Verificar expressão da proteína de Cas9 estável immunoblotting.
- Fazer extratos nucleares de todos os clones selecionado de Blasticidin de leucemia MOLM13 e MLL-AF9 usando o Kit de extração Nuclear conforme o protocolo do fabricante. Verifica a expressão de proteínas Cas9 usando antibandeira M2 anticorpo desde o Cas9 no plasmídeo pLenti-Cas9 está ligado a um epítopo do N-terminal bandeira.
- Carrega 50 µ g de proteínas totais de cada extrato nuclear para 10%, Bis-Tris gel e funcione o gel em 120 V até bandas superiores são bem separadas.
- Bloquear a membrana com solução de leite de 5% em tampão TBST por 1h.
- Incube a membrana com 1:1,000 diluição do antibody de anti-Flag M2 em uma concentração final de 1 µ g/mL a 4 ° C durante a noite.
- No dia seguinte, lave a membrana com buffer TBST e incubar com HRP anti-rato secundário anticorpo conjugado a uma diluição de 1:5,000 (concentração final de 0,16 µ g/mL) à temperatura ambiente durante 1 h.
- Lavar a membrana cuidadosamente com TBST e desenvolver o blot utilizando substrato ECL.
- Escolha 3 – 4 clones com a mais alta expressão da proteína de Cas9 visto pela mancha ocidental para analise posterior funcional (Figura 1).
- Garantindo alta atividade Cas9 em clones selecionados
- Preparar Lentivirus sobrenadante de um vetor de codificação de sgRNA com sgRNAs visando o ser humano ou mouse AAVS1 locus de porto seguro, como descrito no passo 1.1.
- Transduce topo MOLM13 Cas9-expressando ou leucemia MLL-AF9 clones com o sobrenadante Lentivirus de anti-AAVS1 sgRNA usando o spinfection método descrito acima. Selecione com puromicina de 2,5 µ g/mL durante 4 dias.
- Purifica o DNA genômico dos clones de leucemia MOLM13-Cas9 ou MLL-AF9-Cas9 após puromicina juntamente com os respectivos controles de tipo selvagem usando o kit de extração de DNA genômico conforme as instruções do fabricante. Uso 50 ng de DNA como um modelo para realizar uma PCR com o AAVS1_test_primers usando 2 x mestre mistura do Polymerase de Taq e o programa PCR listados na tabela 1.
- Executar o produto do PCR em um gel de agarose 2% e gel-purificar a banda 268 pares de base usando um kit de extração de gel de acordo com o protocolo do fabricante. Sequência de Sanger gel purificada produto do PCR usando o primer AAVS1_target-frag_F.
- Avalie a eficiência de edição pela comparação entre o AAVS1 editada e sua sequências usando ferramentas baseadas na web on-line (Figura 2).
2. clonagem e transdução de sgRNAs nas células da LMA-Cas9
-
Rendimento médio de clonagem de sgRNAs
- Projeto 4 – 6 sgRNAs para o gene de interesse usando um software de design CRISPR baseado na web. Há um número de ferramentas de design CRISPR disponíveis on-line tais como http://crispr.mit.edu/ que geram sequências de sgRNA com base em uma sequência de DNA a entrada.
- Preencha uma informações apropriadas nos campos com asteriscos. Clique no genoma alvo para o projeto de sgRNA. Cole a sequência de destino na caixa sequência e clique no botão Enviar .
- Para clonagem na pKLV2-U6gRNA5 (BbsI) - PGKpuro2ABFP - plasmídeo de expressão sgRNA W, preceder os nucleotídeos "CACC" para o sentido do oligo e "AAAC" para o reverso oligo antisentido complementado antes de encomendar. Os oligos sentido e antiordem sentido pré-misturado em uma placa de 96 poços rotulados Oligo-Mix de uma empresa de síntese do oligonucleotide.
Nota: Temos feito uso da pKLV2-U6gRNA5 (BbsI) - PGKpuro2ABFP - plasmídeo de W, que tem o site de restrição BbsI para sgRNA clonagem. No caso de outro plasmídeo de expressão sgRNA é usado, as saliências usadas para os oligonucleotides sgRNA precisam ser alterado em conformidade. - Pegue uma separado placa de 96 poços de fundo U rotulada Placa de recozimento. Faça uma mistura de mestre de 1 µ l T4 PNK, 1 µ l de 10x buffer de ligadura do ADN T4 e 6 µ l de água por reação de recozimento.
- Adicione 8 µ l do mix mestre para cada poço da placa de recozimento. Adicione 1 µ l de 100 µM cada, sentido e antisentido oligo (ou 2 µ l de oligos mistos de sentido-antisentido) à mistura de mestre. Pipetar 2 – 3 vezes suavemente para misturar bem, girar a placa brevemente para obter a mistura para o fundo dos poços.
- Use o seguinte programa de recozimento em uma máquina PCR: 30 min a 37 ° C, 2 min 30 s a 95 ° C seguido de resfriamento lento a 22 ° C, à taxa de 0,1 ° C/s. Após recozimento, leve um prato de 96 poços fresco rotulado OligoMix diluído. Esta placa, dilua os oligos fosforilada e recozido da reação acima com água (1: 200), usando uma pipeta multicanal.
- Digest 5 µ g de pKLV2-U6gRNA5 (BbsI) - PGKpuro2ABFP - vetor da W, com 1,5 µ l de enzima de restrição BbsI (10.000 unidades/mL), usando um amortecedor apropriado a 37 ° C por 2 h para linearizar-para ligadura mais tarde.
- Pegue um prato bem fresco 96 rotulados Ligadura da placa e adicione 20 ng da BbsI digerido (BbsI) - PGKpuro2ABFP - pKLV2-U6gRNA5 vetor W a um poço por ligadura sgRNA desejado. Para isso, adicione 2 µ l de oligos fosforilados, recozidos da placa OligoMix diluído .
- Adicione 1 µ l de tampão de ligase x T4 10 e 1 µ l da enzima T4 DNA Ligase. Delicadamente, pipetar com uma pipeta multicanal e incubar a mistura de ligadura em temperatura ambiente por 2 h.
Nota: Misturas de ligadura podem ser armazenadas a-20 ° C durante a etapa de transformação mais tarde. - Enquanto isso, o degelo 90 µ l de quimicamente competentes de Escherichia coli células no gelo 10 min antes do final da etapa de ligadura.
- Usando uma pipeta multicanal, fazer 10 alíquotas µ l de células competentes em cada poço da placa 96 bem separados.
- Adicione a mistura de ligadura da etapa 2.1.8 no bem contendo as células competentes, pipeta de cima e para baixo suavemente e incubar durante 10 minutos à temperatura ambiente.
- Pipeta de 5 µ l do mix bactérias-DNA diretamente da reação acima em um 6 bem placa contendo LB-ágar com 100 mg/mL ampicilina. Repita para cada uma das reações de transformação.
- Cada reação de transformação em um separado etiquetado bem de uma placa de 6 da placa. Adicione aproximadamente os grânulos de vidro de 5 a 8 para cada bem e agitar a placa toda 6 8 a 10 vezes em um movimento circular.
- Escolha 1 – 2 colônias única de cada poço com uma ponta de pipeta estéril 20 µ l e raia-lo em um local cuidadosamente rotulado em uma estéril 10cm placa de Petri com ágar LB e 100 mg/mL ampicilina. Depois de estrias em placa de LB, simplesmente Ejete a ponta usada para clone estrias em 3 mL de LB-Ampicillin médio contendo em um mL 14 redondo tubo inferior marcado com o número correspondente de clone bacteriana.
- Enviar a placa bacteriana diretamente para Sanger sequenciando com primer promotor U6 humana para a frente.
- Após a confirmação da sequência de sgRNAs clonado, purifica o DNA do correspondente tubo 14 mL usando um kit de mini-preparação de acordo com as instruções do fabricante.
- Medir a concentração e a qualidade de cada um dos minipreparações com um espectrofotômetro. Normalize a cada mini-preparação a uma concentração de 15 ng / µ l e pipeta em um prato bem 96 rotulados sgRNA placa de Clones.
Nota: Este prato pode ser armazenado a-20 ° C ou usado diretamente para a preparação de vírus.
- Projeto 4 – 6 sgRNAs para o gene de interesse usando um software de design CRISPR baseado na web. Há um número de ferramentas de design CRISPR disponíveis on-line tais como http://crispr.mit.edu/ que geram sequências de sgRNA com base em uma sequência de DNA a entrada.
-
Produção viral de sgRNA construções e transdução
- Para a produção de particulas de Lentivirus de sgRNA no formato de 96 poços, siga o "shRNA/sgRNA/ORF alto Throughput produção Viral (96 bem)" protocolo da plataforma de perturbação genética (web Portal do GPP) do Instituto Broad (URL: https:// Portals.broadinstitute.org/GPP/Public/Resources/Protocols).
- Transferência de 200 µ l do sobrenadante viral para tubos estéreis e congelar a-80 ° C imediatamente.
- Dia -1: Revesti uma parte inferior lisa Non-tecido tratada 96 bem placa de cultura com 100 µ l de fragmento de fibronectina humana recombinante em uma concentração de 10 µ g/mL em uma capa de cultura de células BSL2. Embrulhe o prato com filme plástico para evitar perda de evaporação e deixá-lo no banco durante a noite.
- Dia 0: Remova o fragmento de fibronectina humana recombinante de cada poço. Descongele o sobrenadante viral de cada sgRNA à temperatura ambiente. Adicione 50 µ l do sobrenadante viral de cada tubo a cada revestido bem da placa de transdução. Girar a placa de transdução em 1.300 x g durante 90 minutos a 35 ° C.
- No final do spinfection, conta MOLM13-Cas9 (clone B3) células do frasco de cultura. Use a 10.000 células por poço no volume 100 µ l. Conte as células de todos os poços de transdução, atendendo volume morto.
- Após o spinfection de 90 min, remova o sobrenadante de todos os poços utilizando uma pipeta multicanal ajustada a 50 µ l lentamente pela inclinação da placa. Adicione 100 µ l de cultura de células de MOLM13-Cas9 clone B3 para cada bem lentamente deslizando para baixo da borda.
- Gire a placa a 1.300 x g por 2 min a 35 ° C, para permitir que as células resolver na parte inferior. Transfira a placa para a incubadora de grau de cultura de tecidos de 37 ° C.
- Dia 1: Adicione 100 µ l de meio fresco para cada bem contendo sgRNA transfectadas Cas9-MOLM13 ou células de leucemia Cas9-MLL-AF9 tal que o volume médio final é 200 µ l.
3. ensaio de crescimento competitiva
- Dia 3: 72 h (dia 3) post transdução, verificar a porcentagem de sgRNA contendo células positivas BFP em cada poço por citometria de fluxo (FACS). Use uma tintura de viabilidade celular para marcar e excluir as células mortas da análise.
- Continue a re-chapeamento uma proporção das células em novos poços com meio fresco após cada análise de FACS para evitar o crescimento excessivo durante o ensaio.
-
Repeti a análise de FACS cada 2-3 dias para verificar a proporção relativa de BFP positivo (BFP + ve) células em comparação com as contrapartes BFP negativas (BFP-ve) (Figura 3). Analise a porcentagem de células positivas BFP para cada ponto de tempo (usando o FlowJo ou o software de análise de FACS similar).
- Arraste arquivos Fcs para cada amostra a FlowJo software. Clique duas vezes em qualquer arquivo de uma amostra e plotar um borrão do ponto de dispersão para a frente (FSC) vs lado dispersão (CCD) com FSC na X eixo e SSC no eixo Y. Portão de todas as células.
- Clique duas vezes sobre as células fechadas e parcela-las na mancha de viabilidade (no eixo Y) vs FSC altura (H) ou borrão do ponto de área (A). Portão a viabilidade mancha negativas células (ao vivo).
- Clique duas vezes em células vivas fechadas e plotar BFP (no eixo de Y) vs FSC (A) no eixo X. Portão BFP + ve as células. Aplicam-se todas estas portas para todas as amostras, arrastando a amostra selecionada para Todas as amostras sob a guia do grupo .
- Clique no Editor de tabela para fazer uma tabela de análise das amostras. Arraste o BFP + ve contagem de uma amostra para a tabela e clique no botão Exibir no Editor de tabela para criar um relatório de lote das amostras com uma tabela que exibe a porcentagem de células BFP + ve . Salve a tabela como um xls.
- Calcular o proporcional aumento ou diminuição de células positivas de % BFP dentro da população de células vivas ao longo do tempo e comparar esta relação com a proporção de células positivas BFP no sgRNAs de controle (como luciferase, mexidos ou sgRNA GFP).
- Traça a proporção de células positivas BFP para os sgRNAs diferentes, incluindo os genes de interesse, bem como controles usando dia 3 como linha de base.
Resultados
Em nosso estudo, nós primeiro transfectadas MOLM13 humana AML linhagem celular que leva a translocação de MLL-AF9 com vírus de alta-Título codificação o plasmídeo de Lentivirus Cas9-blasticidin. Em nossas mãos, em massa de células de MOLM13-Cas9 não seleccionadas não exibir alto nível Cas9 expressão pela mancha ocidental e também não executou bem quando analisada por gene eficiente edição-usando o método descrito anteriormente7. Por conseguinte, procedeu-se a estabelecer a única célula clones e selecionar apenas os clones com altos níveis de Cas9 visto pela mancha ocidental (Figura 1). Nós escolhemos 2 clones distintos e eles transfectadas com sgRNAs como alvo um site no locus de porto seguro AAVS1 como descrito anterior7. Usando o DNA genômico extraído a puromicina selecionados MOLM13-Cas9-AAVS1 sgRNA clones, realizamos uma PCR utilizando primers abrangendo o sgRNA de AAVS1 corte local e sanger sequenciado um produto específico do PCR 268 bp de 10 colônias isoladas para cada clone MOLM13. Após o alinhamento com a sequência dos pais, achamos que o clone MOLM13-Cas9 B3 e MLL-AF9-Cas9 clone 8 tinham uma eficiência de 100% (dados não mostrados). Então selecionamos estes clones exibindo alta mediada por Cas9 edição de genoma capacidade para nossos ensaios de competição de sgRNA. Enquanto estes estudos foram sendo realizados, ferramentas baseadas na web para rapidamente testar a eficiência de edição de genoma de Sanger sequências foram desenvolvidas por diferentes grupos como maré8 (https://www.deskgen.com/landing/tide.html) ou gelo (https://ice.synthego.com/#/). Estas ferramentas que seja extremamente fácil e cost-effective para avaliar a eficiência de edição de genoma sem precisar clonar fragmentos individuais e realizar o sequenciamento de vários clones. Portanto, as células em massa Cas9-expressando primeiro devem ser testadas para genoma eficiência usando estes métodos de edição. No caso da eficiência de edição de genoma é alta, então monocelulares clonagem não é necessária. Também, no caso da clonagem de célula única é necessária, como visto em nossos estudos, essas ferramentas de estimativa de frequência mutacional baseados na web oferecem um método muito mais rápido para testar vários clones em paralelo.
Para sgRNA clonagem, fizemos uso da clonagem do plasmídeo pKLV2-U6gRNA5 (BbsI) - PGKpuro2ABFP - o sgRNA W, que abriga uma proteína fluorescente azul (BFP) para acompanhamento de células transfectada de sgRNA e um orientado a RNA polimerase III humano U6 promotor que impulsiona a expressão de o clonado sgRNAs juntamente com o andaime de RNA (tracRNA) de rastreador. Usamos este sistema para clonar 2 sgRNAs DOT1L, uma proteína conhecida por desempenhar um papel importante na regulação da expressão genética epigenética de direcionamento. DOT1L é uma histona methyltransferase que os depósitos de mono, di e tri-metilação no alvo genes9,10. Estudos têm mostrado que DOT1L desempenha um papel importante na LMA, conduzida por oncogenes MLL-fusão. Portanto, DOT1L nocaute usando CRISPR-Cas9 é esperado para liderar de prejudicar significativamente a proliferação de células de leucemia rato MLL-AF9 em consonância com estudos anteriores11,12,13,14. Nós usamos dois sgRNAs visando o AAVS1 locus de porto seguro, que é do intron do gene PPP1R12C. sgRNAs produzir alterações genômicas neste site não são susceptíveis de ter efeitos sobre a capacidade proliferativa de linhas de células de leucemia-MLL. Como um controle positivo, nós clonado sgRNAs como alvo o gene associado a replicação de DNA RPA3. RPA3 é um gene de pan-essenciais que tem demonstrado ser importante para a proliferação de vários AML célula linhas4,15,16 . SgRNAs anti-RPA3, portanto, exibir fortes efeitos antiproliferativa em células da LMA e são normalmente usados como controle positivo. Após a transdução com plasmídeos de sgRNA anti-AAVS1 e anti-RPA3, medimos a proporção relativa de BFP + ve sgRNA transfectadas células em comparação com suas contrapartes BFP-ve cada 2-3 dias em cultura (Figura 3). Com o protocolo descrito acima, a transdução de células MOLM13 ou LMA AF9 MLL resultou em uma eficiência de transdução de 60 – 70% com nossas preparações virais, deixando as células restantes de 30 – 40% untransduced ou BFP-ve em cada poço. Isto permite o estudo de relativo proliferação de células do genoma-edição com o selvagem-tipo pilhas no mesmo bem. O proporcional aumento ou diminuição da percentagem de células sgRNA que nao foi usada como uma medida para refletir o efeito funcional do sgRNA mediada por gene-exclusão nas células MOLM13-Cas9. Se o gene de interesse é importante para a proliferação das células da LMA de teste, então mediada por sgRNA interrupção deste gene levará até a diminuição relativa da BFP sgRNA-expressando + ve células em comparação com as células BFP-ve no decorrer do ensaio, Considerando que sgRNAs como alvo do luciferase, GFP ou genes não essenciais reterá o BFP + ve-ve relação ao longo do tempo.
Usando 2 sgRNAs anti-RPA3 separado, observamos um declínio progressivo e significativo da percentagem de BFP + ve células em comparação com o BFP-ve untransduced homólogos. Em contraste, a porcentagem de sgRNA anti-AAVS1 expressando células BFP manteve-se relativamente constante ao longo do tempo, demonstrando que AAVS1 direcionamento não tem efeito sobre a proliferação de células MOLM13 (figura 4a). Da mesma forma, nas células MLL-AF9-Cas9 rato, nós testamos os efeitos de sgRNAs visando a rodopsina (Rho1), o gene de pigmentação do olho como um controle negativo e Dot1l, um regulador epigenético, conhecido por ser necessária para a proliferação de células de leucemia MLL-AF9 ao longo do tempo . No presente estudo, BFP + ve células de rato MLL-AF9-Cas9 transfectadas com 2 separado anti-DOT1L sgRNAs mostraram uma perda dramática e progressiva ao longo do tempo, em comparação com células de sgRNA não-transfectadas BFP-ve (figura 4b). Em contraste, a relação de sgRNA anti-Rho1 permaneceu relativamente inalterada ao longo do tempo. Estes resultados demonstram a vulnerabilidade do MLL-AF9 expressando células de leucemia do mouse ao esgotamento de Dot1l, confirmando os resultados publicados anteriormente.

Figura 1: borrão ocidental representante resultados apresentando diferentes níveis de Cas9. Clones de única célula da linha MOLM13 AML células transfectadas com CAS9 foram analisados os níveis de expressão de bandeira-Cas9 usando anticorpos anti-Flag. Mostrando a mais alta expressão da Cas9 de clones foram selecionados para estudos adicionais. Células HEK293-T transfected com um Cas9 plasmídeo de expressão foram usados como controle positivo e Cas9 não transfectadas MOLM13 células como controles negativos. Clique aqui para ver uma versão maior desta figura.

Figura 2: análise representativa do genoma de edição para o locus Dot1l avaliado pela análise de ICE. Uma PCR amplicons centrado em torno do local de destino de sgRNA Dot1l foi Sanger sequenciado e analisado usando análise de gelo. Uma comparação de sequência de sgDot1l (linha laranja) a sequência de ADN não específico (linha verde) demonstra o elevado nível de eficiência em células sgDot1l direcionados ao redor do local de destino de sgRNA de edição. Clique aqui para ver uma versão maior desta figura.

Figura 3: fluxo de trabalho esquemático dos métodos propostos. sgRNAs são clonados em vetor de expressão sgRNA co expressar uma proteína fluorescente como BFP. Nas células-alvo são transfectadas em 30 – 60% as taxas de transdução e seguidas por citometria de fluxo a cada 2-3 dias. sgRNAs alvejando genes necessários para a proliferação das células AML mostrará o esgotamento relativo ao longo do tempo como mostrado. Clique aqui para ver uma versão maior desta figura.

Figura 4: representante resultados de ensaios de competição em humanos e células de rato AML. (um) resultados mostram um significativo estatisticamente altamente progressivo declínio na RPA3 transfectadas células de MOLM13-Cas9 clone B3. Em contraste, sgRNAs, direcionando o site AAVS1 não têm efeito. sgRNAs (b) direcionamento Dot1l mostram um declínio notável e progressivo na proliferação do competidor, em contraste com sgRNAs segmentação rodopsina (Rho). p > 0,05. p < 0,05. Barras de erro representam o desvio padrão da média (SD). Clique aqui para ver uma versão maior desta figura.
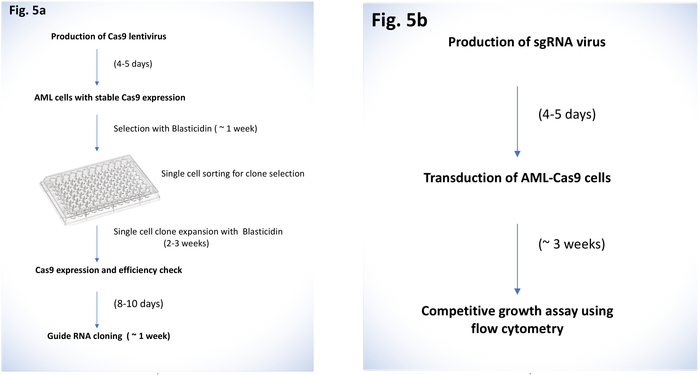
Figura 5: esquema geral do protocolo. (uma) hora para cada etapa na produção de vírus Cas9 e sgRNA clonagem descrito. Design e clonagem de sgRNAs podem ser executadas ao mesmo tempo gerando clones única de linhas de celulares AML com expressao Cas9. (b) protocolo geral para a transdução de linhas de celulares AML com sgRNAs e ensaio de crescimento competitivo usando FACS é mostrado. Clique aqui para ver uma versão maior desta figura.
| Ciclos de | Duração do ciclo | Temperatura |
| 1 | 2 min | 95 ° C |
| 35 | 30 s | 95 ° C |
| 30 s | 55 ° C | |
| 30 s | 72 ° C | |
| 1 | 5 min | 72 ° C |
| Segure | infinita | 4 ° C |
Tabela 1: programa PCR para a amplificação de AAVS1 locus de DNA genômico. Programa PCR utilizado para a amplificação de AAVS1 locus de DNA genômico para teste eficiência de corte de Cas9 em Cas9 expressando clones.
Arquivo complementar: sequências de oligo sgRNA. Clique aqui para baixar este arquivo.
Discussão
Este manuscrito, descrevemos um protocolo detalhado para a realização de um ensaio de crescimento competitivo CRISPR-Cas9-baseado para investigar o papel de genes candidatos em linhas de células de LMA usando citometria de fluxo em células de LMA murino/humano (Figura 5). O objetivo do ensaio é identificar o efeito de supressão do gene na manutenção da proliferação celular AML mais de duas a três semanas em uma escala de rendimento médio. Alguns passos críticos precisam ser seguidos cuidadosamente para facilitar a ampliação do protocolo descrito. Na produção de lentivirus Cas9, é necessário filtrar o meio condicionado de vírus através de um filtro de 0,45 µM para evitar a transição de células 293T ao vírus contendo sobrenadante. Durante spinfection, envolvendo a placa de spinfection com filme plástico ou parafilm ajuda a evitar a contaminação potencial durante a centrifugação. No entanto, o envoltório deve ser removido antes de colocar a placa de spinfected volta na incubadora de cultura de tecidos. É muito importante avaliar clones Cas9-expressando para eficiência de edição. Clones com eficiência de edição de baixo-genoma refletem a atividade inadequada do Cas9 e podem afetar a taxa de sucesso do experimento. Em nossos experimentos, nós primeiro transfectadas clones humanos AML Cas9 com sgRNAs visando o locus de AAVS1 e sequenciados seus DNAs genomic para eficiência de edição. Comparação entre o AAVS1 editada e sua sequências podem ser avaliadas usando ferramentas online baseado na web, tais como a maré8 (https://www.deskgen.com/landing/tide.html) ou gelo (https://ice.synthego.com/#/). Estes programas comparam Sanger vestígios de sequência de PCR potencialmente editado fragmentam a sequência inéditos de sua ou referência e estimam a frequência de alterações. Esta é uma maneira rápida de avaliar alterações mutacional provocadas pela sgRNA teste, qual é a medida da atividade celular Cas9. Alternativamente, clonagem de PCR produto em um plasmídeo de clonagem por PCR como o TOPO sem corte vetor de clonagem pode ser executada, seguido por Sanger sequenciamento das colônias individuais para avaliar a eficiência de edição de genoma pelo alinhamento da sequência de cada clone para o selvagem-tipo. Nós preferimos o método antigo, dado a sua facilidade e custo-eficácia em comparação com o método de clonagem. Normalmente, a descoberta de pares de base alterações tais como mutações pontuais, puntuais, etc., em ou ao redor do local de corte de sgRNA em uma grande maioria de clones sequenciados indicam a presença de um Cas9 altamente ativo. Assim, optamos por leucemia MOLM13 ou MLL-AF9 clones B3 e 8, respectivamente, com a mais alta eficiência de edição para novas experiências.
Nós projetamos o direcionamento de diferentes genes mencionados no protocolo usando http://crispr.mit.edu/, um software baseado em web que fornece uma lista de sgRNAs de sgRNAs. Recomenda-se a pontuação superior sgRNAs selecione na lista a fim de eliminar aqueles com efeitos de fora do alvo previstos. O sgRNAs projetado pode ser clonado usando qualquer um dos protocolos publicados como o site (http://www.addgene.org/67974/). Sentido e antisentido oligos primeiro são fosforilados e recozidos conforme o passo 2.1.5. A primeira etapa a 37 ° C é importante para pressões os oligos com a quinase de T4 e ciclos PCR subsequentes são importantes para recozimento do sentido e oligonucleotídeos antisenso em dsDNA. É importante ter uma proteína fluorescente do vetor de clonagem de sgRNA que é crucial para acompanhamento de células sgRNA transformado mais tarde nas ensaios de competição. sgRNA clonagem é dimensionado com o uso de 96 placa bem em todas as etapas, incluindo o recozimento e ligadura. Para ligadura, limpe o PCR microtubo tiras também podem ser usadas, desde que sejam compatíveis com pipetas multicanais. No caso que é necessária a transformação de um conjunto maior de clones de sgRNA, uma placa de 96 poços, no qual 10 μL de células competentes são pre-aliquotadas em cada poço e congeladas a-80 ° C podem ser usado para agilizar todo o processo. A finalidade do chapeamento as reações de ligadura é escolher colónias individuais, que é difícil de executar no formato de 96 poços. Daí, para transformação bacteriana, há uma ligeira perda de escalabilidade. Ainda, mesmo para o chapeamento de reações de transformação de um prato inteiro de 96 poços, apenas necessita de um total de dezesseis placas 6-boas para o projeto inteiro. Um tem que ter cuidado na próxima etapa de arrancar as colônias as chapas de transformação. Enquanto estrias uma colônia em um ponto rotulado, certifique-se de que o local é claramente isolado os outros pontos. Marcar uma grade de quadrados na parte inferior da caixa de Petri com um marcador permanente escuro ajuda a evitar a contaminação cruzada dos clones bacterianos.
Uma vez que o minipreps de sgRNA construções estão prontos, o vírus pode ser feito no formato de 96 poços. A este nível de rendimento, é impraticável para filtrar 200 µ l de vírus contendo o sobrenadante. Daí, o sobrenadante viral deve ser congelado pelo menos durante a noite para evitar a contaminação cruzada de quaisquer células 293T transitadas no sobrenadante. Também pode ser armazenado em 50 alíquotas µ l em tubo estéril de PCR tiras para evitar congelam descongelar do líquido sobrenadante. Além disso, estes sobrenadantes de vírus condicionado são utilizados para transducing células da LMA. No caso que baixa concentração de transdução viral são observados, como avaliado pela baixa frequência de BFP + ve as células e, em seguida, pode ser importante determinar o título viral e testes transductions em diferente multiplicidade de infecções (MOI) para garantir a 40-60% taxas de transdução. Determinação da concentração viral e MOI cálculo é descrito em detalhe aqui17. No ensaio de competição, conforme descrito na etapa 3 do protocolo, é altamente importante excluir células não viáveis para evitar artefactos espúrios do autofluorescência o BFP à relação de não-BFP na análise. No momento da FACS, antes de analisar amostras de teste, o uso de BFP negativo e controlos positivos BFP é altamente recomendado para definir com precisão as tensões. Em cada ponto de tempo re-chapeamento, o volume das células para ser re-chapeado depende da concentração de células no ponto de tempo determinado. Nós normalmente re-chapeado 20 μL de um total de 200 µ l em cada ponto de tempo em um bem fresco com 180 µ l de meio fresco. Mistura completa das células pipetando delicadamente acima e para baixo antes de re-chapeamento é altamente recomendado. Normalmente, temos observado que o ensaio precisa ser realizado ao longo de 15-20 dias para aviso forte muda no BFP + ve-ve rácios, embora isso varia de gene para gene.
Esta montagem experimental é bem adequada para testar vários genes em paralelo em várias linhas de célula AML para rapidamente identificar a função de genes candidatos na sobrevivência ou proliferação das células da LMA. Uma ressalva do ensaio do competidor proliferação descrito aqui é que não considera potenciais fatores extrínsecos-célula como parácrina sinalização eventos do gene-alvo as células que podem influenciar a população de células não específico dentro do mesmo bem. Mesmo que isso possa ser uma ocorrência rara, este fator deve ter em mente quando conduzindo este ensaio. Embora nós usamos AML como um exemplo para os ensaios de competição baseados em CRISPR-Cas9 descritos neste manuscrito, este método pode ser usado para qualquer linha de células de câncer para identificar o papel dos genes múltiplos em paralelo. Existem várias vantagens de gene-exclusão usando CRISPR-Cas9 sobre gene nocaute usando a interferência do RNA. Em primeiro lugar, em contraste com o shRNAs, que normalmente mostram uma ampla gama de inibição de mRNA alvo, sgRNAs, juntamente com a nuclease Cas9 effectuate nocaute completo de genes-alvo. Isso pode resultar em fenótipos mais dramáticos e consistentes com os métodos baseados em CRISPR-Cas9, mesmo que ele deve ser advertido de que a eficiência de direcionamento de sgRNAs individuais, bem como shRNAs pode variar amplamente.
O ensaio de fluxo cytometry-concorrência baseada oferece diversas vantagens sobre os ensaios de proliferação tradicional em que um número fixo de células é banhado para medir a atividade proliferativa relativa das células de gene-perturbado. Uma vantagem é que a relativa atividade proliferativa das células pode ser facilmente e rapidamente medida por citometria de fluxo por vários dias, ignorando a necessidade de célula contando a cada passo do chapeamento. Isso é útil quando um grande número de candidatos sgRNAs segmentação de vários genes, como contando todos os poços de teste é complicado e pode levar a imprecisões. Ao contrário, ensaios de medição baseado em ATP para crescimento celular, citometria de fluxo pode ser executada em uma amostragem de células vivas, o que torna este método útil para a análise a longo prazo. Isto é especialmente benéfico no estudo de fatores tais como moduladores epigenéticas, que podem mostrar efeitos de ação final sobre a proliferação de células da LMA e podem exigir ensaios a longo prazo. Em segundo lugar, a presença de células transfectadas não dentro do mesmo bem permite uma população celular bem controlados que pode ser usada para definir a linha de base para o ensaio. Em terceiro lugar, quando o set-up em um formato de 96 poços, o ensaio pode ser realizado quase completamente usando pipetas multicanais, substancialmente acelerar todo o processo de clonagem de sgRNAs a preparação do vírus e, eventualmente, fluxo cytometric avaliação de fluorescência.
O método descrito aqui pode ser eficientemente dimensionado para a investigação de até 96 sgRNAs em paralelo em uma tela de matriz. Supondo que 4-6 sgRNAs por gene, este método, portanto, pode ser usado para o interrogatório rápido de pelo menos 16 – 24 genes em paralelo. Em caso de maior número de genes, como um todo caminho molecular que precisa ser interrogado em células da LMA, telas CRISPR-Cas9-baseado em pool será mais útil.
Divulgações
A.J.D é consultor de fármacos A2A (Nova Jersey) e Salgomed Therapeutics (San Diego). Outros autores não têm nenhum conflito para declarar.
Agradecimentos
O plasmídeo pCW-Cas9 foi um presente de Eric Lander & David Sabatini (plasmídeo Addgene # 50661) e o pKLV2-U6gRNA5 (BbsI) - PGKpuro2ABFP - plasmídeo W do laboratório Yusa (plasmídeo Addgene #67974. Gostaríamos de agradecer o núcleo de citometria de fluxo no SBP Medical Discovery Institute ajuda oportuna com análise de fluxo e triagem. Nós gostaríamos de reconhecer o apoio da Fundação Memorial Lady Tata a A.D. Gostaríamos de agradecer também o apoio das seguintes fontes de financiamento: NIH/ICN P30 CA030199 Cancer Center patrocinado Grant, a V-Fundação e centros de câncer NCI de San Diego (C3) #PTC2017to A.J.D.
Materiais
| Name | Company | Catalog Number | Comments |
| FLAG-M2 Antibody | sigma-aldrich | F3165, lot # SLBS3530V | |
| Anti-mouse Antibody | Invitrogen | 31446, lot # TA2514341 | |
| SuperSignal West Femto Maximum Sensitivity Substrate | Thermo Fisher | 34095 | |
| ChemiDoc Imaging System | BIO RAD | 17001401 | |
| Sorvall Legend RT centrifuge | Thermo Scientific | ||
| Blasticidin | Thermo Fisher | R21001 | |
| SYTOX Red | Thermo Fisher | S34859 | |
| Opti-MEM | Thermo Fisher | 31985062 | |
| DMEM | Thermo Fisher | 11965-092 | |
| RPMI | Thermo Fisher | 11875-093 | |
| Penicillin-Streptomycin | Thermo Fisher | 15140122 | |
| L-Glutamine (200 mM) | Thermo Fisher | 25030081 | |
| Fetal Bovine Serum (FBS) | SAFC | 12303C | |
| single gRNA vector | Addgene #67974 | pKLV2-U6gRNA5(BbsI)-PGKpuro2ABFP-W | |
| CelLytic Nuclear extraction kit | sigma-alorich | NXTRACT | |
| XtremeGENE 9 | sigma-alorich | 6365787001 | |
| Retronectin | Takara | T100B | |
| Flow cytometer | BD Biosciences | ||
| T4 PNK | NEBioLabs | M0201S | |
| T4 DNA ligation buffer | NEBioLabs | B0202S | |
| T4 DNA Ligase enzyme | NEBioLabs | M0202S | |
| Ampicillin | Fisher scientific | BP1760-25 | |
| LB agar | Fisher scientific | BP9724-500 | |
| LB Broth | Fisher scientific | BP9731-500 | |
| Qiagen mini-prep kit | Qiagen | 27104 | |
| NanoDrop Spectrophotometer | Thermo Fisher | NanoDrop One | |
| Recombinant Murine IL-3 | Peprotech | 213-13 | |
| Recombinant Murine IL-6 | Peprotech | 216-16 | |
| Recombinant Murine M-CSF | Peprotech | 315-02 | |
| Stable competent cells | NEBiolabs | C3040I | |
| 10 cm Tissue Culture dishes | Fisher Scientific | 353003 | |
| Cell lysis solution | Qiagen | 158906 | |
| Protein precipitation solution | Qiagen | 158910 | |
| DNA hydration solution | Qiagen | 158914 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| BbSI | New England BioLabs | R0539S | |
| APEX 2.0x Taq Red Master Mix Kit | Genessee Scientific | 42-138 | |
| Puromycin | Fisher scientific | BP2956100 | |
| 50 mL polypropylene conical tubes | Fisher scientific | 1495949A | |
| 15 mL polypropylene conical tubes | Fisher scientific | 1495970C |
Referências
- Mali, P., Esvelt, K. M., Church, G. M. Cas9 as a versatile tool for engineering biology. Nature Method. 10 (10), 957-963 (2013).
- Doudna, J. A., Charpentier, E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096(2014).
- Wang, H., La Russa, M., Qi, L. S. CRISPR/Cas9 in Genome Editing and Beyond. Annual Review of Biochemistry. 85, 227-264 (2016).
- Shi, J., et al. Discovery of cancer drug targets by CRISPR-Cas9 screening of protein domains. Nature Biotechnology. 33 (6), 661-667 (2015).
- Kuhn, M. W., et al. Targeting Chromatin Regulators Inhibits Leukemogenic Gene Expression in NPM1 Mutant Leukemia. Cancer Discovery. 6 (10), 1166-1181 (2016).
- Erb, M. A., et al. Transcription control by the ENL YEATS domain in acute leukaemia. Nature. 543 (7644), 270-274 (2017).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Brinkman, E. K., et al. Easy quantification of template-directed CRISPR/Cas9 editing. Nucleic Acids Research. 46 (10), 58(2018).
- Nguyen, A. T., Zhang, Y. The diverse functions of Dot1 and H3K79 methylation. Genes & Development. 25 (13), 1345-1358 (2011).
- Vlaming, H., van Leeuwen, F. The upstreams and downstreams of H3K79 methylation by DOT1L. Chromosoma. 125 (4), 593-605 (2016).
- Bernt, K. M., et al. MLL-rearranged leukemia is dependent on aberrant H3K79 methylation by DOT1L. Cancer Cell. 20 (1), 66-78 (2011).
- Daigle, S. R., et al. Selective killing of mixed lineage leukemia cells by a potent small-molecule DOT1L inhibitor. Cancer Cell. 20 (1), 53-65 (2011).
- Jo, S. Y., Granowicz, E. M., Maillard, I., Thomas, D., Hess, J. L. Requirement for Dot1l in murine postnatal hematopoiesis and leukemogenesis by MLL translocation. Blood. 117 (18), 4759-4768 (2011).
- Nguyen, A. T., Taranova, O., He, J., Zhang, Y. DOT1L, the H3K79 methyltransferase, is required for MLL-AF9-mediated leukemogenesis. Blood. 117 (25), 6912-6922 (2011).
- Zuber, J., et al. Toolkit for evaluating genes required for proliferation and survival using tetracycline-regulated RNAi. Nature Biotechnology. 29 (1), 79-83 (2011).
- Wang, T., et al. Identification and characterization of essential genes in the human genome. Science. 350 (6264), 1096-1101 (2015).
- Li, M. J., Rossi, J. J. Lentivirus transduction of hematopoietic cells. CSH Protocols. 2007, pdb prot4755 (2007).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados