Method Article
Indagine delle dipendenze genetiche utilizzando dosaggi di concorrenza basati su CRISPR-Cas9
In questo articolo
Riepilogo
Questo manoscritto descrive un cluster regolarmente intervallate breve palindromo si ripete (CRISPR) CRISPR Cas9 metodo basato per indagine semplice e rapida del ruolo di geni candidati multipli nella proliferazione delle cellule di leucemia mieloide acuta (LMA) in parallelo. Questa tecnica è scalabile e può essere applicata in altre linee cellulari di cancro pure.
Abstract
Gene perturbazione studi sono stati ampiamente utilizzati per studiare il ruolo dei singoli geni nella patogenesi AML. Per la realizzazione completa del gene, molti di questi studi hanno fatto ricorso a modelli di knockout del gene complesso. Mentre questi studi con topi knockout offrono un sistema elegante e time-tested per indagare le relazioni genotipo-fenotipo, un metodo veloce e scalabile per la valutazione di geni candidati che gioca un ruolo nella proliferazione delle cellule AML o sopravvivenza nei modelli AML aiuterà ad accelerare l'interrogatorio parallela di più geni candidati. I recenti progressi nelle tecnologie di modifica del genoma hanno drammaticamente migliorato la nostra capacità di eseguire perturbazioni genetiche in una scala senza precedenti. Uno di questi sistemi di editing genomico è il metodo di CRISPR-Cas9-based che può essere utilizzato per apportare modifiche rapide ed efficaci nel genoma della cellula bersaglio. La facilità e la scalabilità di omissione del gene per CRISPR/Cas9-mediata rende una delle tecniche più attraente per l'interrogatorio di un gran numero di geni nelle analisi fenotipiche. Qui, presentiamo un'analisi semplice utilizzando rottura CRISPR/Cas9 mediato del gene combinata con saggi di alto-rendimento flusso cytometry-concorrenza basata per indagare il ruolo dei geni che possono svolgere un ruolo importante nella proliferazione o la sopravvivenza dell'essere umano e murine linee cellulari AML.
Introduzione
Degli ultimi decenni hanno visto numerosi sforzi di ricerca focalizzati sull'identificazione il contributo dei principali meccanismi molecolari nella patogenesi della leucemia mieloide acuta (AML). Tradizionalmente, rottura del gene in cellule di AML è stata eseguita utilizzando topi knock-out condizionale o short hairpin RNA (shRNA). Mentre i topi knockout offrono un sofisticato sistema per il controllo spazio-temporale di omissione del gene, generazione di topi knockout del gene è laborioso, lungo e costoso. Inoltre, gene-Ko utilizzando strategie di ricombinazione non è facilmente scalabile; Queste strategie non si prestano bene per l'interrogatorio di parecchi geni in parallelo. Dopo la scoperta di metodi di interferenza del RNA di colpo mRNAs endogeno utilizzando piccolo RNA interferente (siRNA) o shRNA, molti gruppi iniziato utilizzando tecniche di interferenza del RNA per indagare il ruolo dei geni specifici in AML. Poiché le cellule AML sia murine ed umane sono notoriamente difficili a transfect utilizzando metodi di trasfezione basata sui lipidi tradizionali, la maggior parte studia lentivirally autonomo o con codifica retrovirally shRNA per lo studio della funzione del gene in cellule di AML. La recente scoperta di cluster regolarmente interspaziati breve palindromi ripetizioni (CRISPR) e l'associato nucleasi Cas (CRISPR-Cas9) ha rivoluzionato la gene-targeting tecnologie1,2,3. Utilizzando CRISPR-Cas9, specifici geni o regioni genomiche possono essere eliminate, modificate o taggate con efficienza e semplicità. CRISPR Cas9-editing basato su gene ora sta emergendo come il metodo di scelta per indagare le relazioni genotipo-fenotipo in tipi cellulari diversi a causa della semplicità, efficacia e ampia applicabilità di questa tecnica. Metodi basati su CRISPR-Cas9 stanno diventando il metodo di scelta in AML, non solo per interrogare i singoli geni, ma anche come un modo di indirizzare i geni multipli in disposti o pooled schermi genetici finalizzati ad indagare diversi geni in parallelo come potenziale AML-dipendenze4,5,6.
In questo manoscritto, descriviamo un'analisi semplice crescita competitiva per misurare l'impatto della rottura del gene sulla crescita delle cellule di AML, basato sulla stabile CRISPR-Cas9-mediated gene-modifica seguita da citometria a flusso ad alta velocità. Questo metodo è semplice, efficiente e scalabile per esperimenti di media-velocità di trasmissione per investigare il ruolo di parecchi geni in parallelo in cellule AML.
Protocollo
1. generare cloni di linea delle cellule AML con alta espressione di Cas9 stabili e attivi
- Produzione di Cas9 lentivirus
- Giorno 0: Piastra 4 x 106 293T cellule in 10 mL di DMEM con 10% siero bovino fetale (FBS) e penicillina e L-Glutammina in un piatto di coltura del tessuto di 10cm in un livello 2 di biosicurezza (BSL2) certificata cappa di cultura cellulare. Mettere il piatto in un incubatore a 37 ° C.
- Giorno 1: Le cellule 293T placcato dovrebbero essere il 70 – 80% confluenti il giorno 1. Eseguire la transfezione utilizzando il seguente protocollo nel pomeriggio.
- Riscaldare il mezzo di transfezione, terreni di coltura e reagente di transfezione a temperatura ambiente. Scongelare tutti i plasmidi richiesti per la transfezione.
- Mix 9 µ g di psPAX2, 0,9 µ g di pMD2.G e 9 µ g di plasmidi pLenti-Cas9 con 500 µ l di terreno di transfezione in una provetta da 5 mL.
- Aggiungere 1,7 mL di mezzo di transfezione di un 14 mL provetta a fondo sferico. Aggiungere 57 µ l di reagente di transfezione direttamente nel mezzo di transfezione nel tubo per evitare di toccare la parete del tubo.
- Delicatamente aggiungere il mix di intero plasmide alla soluzione di reagente di transfezione e mescolare scuotendo leggermente il tubo sul lato. Incubare la miscela a temperatura ambiente per 20-30 min. Nel frattempo, è possibile modificare il mezzo dalla piastra 293T seminato.
- Aggiungere il mix di transfezione goccia a goccia alla piastra e scuotere delicatamente la piastra lateralmente per la miscelazione efficiente. Posizionare la piastra in un incubatore a 37 ° C.
- Giorno 2: Aspirare il supernatante dalla piastra di transfezione delicatamente tale che le cellule non sono disturbate o sloggiate dalla piastra. Scartare il surnatante. Aggiungere 10 mL di mezzo DMEM 10% fresco facendo scorrere delicatamente verso il basso dai lati della piastra per evitare di staccare le cellule.
- Giorno 3: Raccogliere il virus contenente surnatante dalla piastra di transfezione lentamente disegnandola in una siringa sterile 10cm. Dopo il supernatante viene raccolto nella siringa, collegare un filtro sterile 0,45 µM alla siringa e tenere la siringa e il filtro sopra un tubo conico in polipropilene 15ml fresco, sterile. Immergere delicatamente la siringa per filtrare il virus contenente surnatante attraverso il filtro 0,45 µM nella provetta conica 15 mL in polipropilene.
- Memorizzi il medium condizionato virale nelle aliquote di 2 mL ciascuno in 2 mL cryovials a-80 ° C.
- Trasduzione di linee cellulari AML
- Giorno -1: Diluire 1 mg/mL di brodo di frammento ricombinante umano fibronectina a 10 µ g/mL con soluzione salina sterile tampone fosfato (PBS). Cappotto di una non-tessuto cultura trattati ben 6 piastra con 2 mL di soluzione di lavoro di 10 µ g/mL fibronectina umana ricombinante frammento in una cappa di cultura cellulare BSL2 approvato. Avvolgere la piastra in pellicola trasparente per evitare la perdita per evaporazione e conservare a temperatura ambiente durante la notte.
- Giorno 0: Scongelare il surnatante virale contenente Cas9. Aspirare la soluzione di frammento di fibronectina umana ricombinante completamente dalla piastra rivestita poco prima di spinfection. Aggiungere 2 mL di medium condizionato virale con Cas9 alla piastra e girarla a 1.300 x g per 90 minuti a 35 ° C. Questo aiuta le particelle virali allegare alla spinfected bene.
- Nel frattempo, contare le cellule essere transduced e spin-down 2 x 106 cellule in un tubo conico in polipropilene da 15 mL. Aspirare il supernatante e risospendere il pellet in 2 mL di terreno di coltura fresco.
- Dopo spinfection, rimuovere tutto il surnatante virale dal spinfected bene ed eliminare. Particelle retrovirali dal surnatante sono attaccate alla parte inferiore della spinfected bene. A questo bene, aggiungere le 2 x 106 cellule risospese in 2 mL di terreno dal passaggio precedente.
- Girare la piastra nuovamente a 1.300 x g molto brevemente (1 – 2 min) sufficiente per permettere alle cellule di stabilirsi nella parte inferiore. Posizionare la piastra nuovamente dentro l'incubatrice e lasciare durante la notte per trasduzione con particelle virali spinfected attaccate al pozzo.
- Giorno 1: A seconda della densità delle cellule, aggiungere più terreno alle cellule trasdotte per evitare la crescita eccessiva.
- Giorno 2: Poiché il plasmide pLenti-Cas9 ha un indicatore di resistenza di Dyfed, aggiungere Consciousness ad una dose di 10 µ g/mL per trasdotte e untransduced (controllo) MOLM13 o cellule di leucemia del topo MLL-AF9 per la selezione di Cas9 che esprimono le cellule.
Nota: Dyfed selezione delle cellule di leucemia MOLM13 o MLL-AF9 Cas9 trasdotte è considerato completo quando tutte le celle del controllo untransduced sono state eliminate. Per linee cellulari diversi da leucemia MOLM13 e MLL-AF9, una curva dose-risposta deve essere eseguita prima di questo esperimento che può essere impiegata una dose ottimale. Titolazione del surnatante virale può essere eseguita anche in caso di tariffe basse-trasduzione.
- Clonare la selezione delle cellule che esprimono alta-Cas9.
Nota: Abbiamo notato che in alcune linee cellulari AML, selezione clonale potrebbe non essere necessario: la maggior parte della popolazione Cas9-Consciousness selezionato già ha alta efficienza modifica del genoma. In questo caso, è possibile saltare il passaggio 1.3 e passare al passo 1.4 per valutare l'espressione Cas9 nelle cellule AML massa Cas9-Consciousness mediante western blotting. Sarebbe comunque importante valutare l'efficienza modifica del gene in quelle celle di massa Cas9-AML (passo 1,5) prima di procedere all'analisi della concorrenza. Congetturiamo che la selezione di cloni di singola alta-Cas9 riduce la variabilità di modifica del genoma, che è particolarmente importante quando si verifica un numero di diversa singola guida RNAs (sgRNAs).- Eseguire una singola cellula sorta del Cas9-Consciousness selezionato MOLM13 e MLL-AF9 cellule di leucemia che d'ora in poi chiamate MOLM13-Cas9 e cellule di MLL-AF9-Cas9, rispettivamente, utilizzando un sorter FACS contenuto in un cappuccio BSL2 approvato. L'ordinamento può essere eseguita in 5 – 10 fondo tondo non-tessuto cultura trattata 96 pozzetti.
- Una volta singola MOLM13-Cas9 e cloni di MLL-AF9-Cas9 sono state raccolte e denominato singolarmente, espandere cloni di 10 – 20 10 µ g/ml di consciousness in terreni di coltura per garantire il mantenimento dell'espressione Cas9.
- Espressione di verificare di stabile Cas9 proteina dall'immunoblotting.
- Rendere gli estratti nucleari di tutti i cloni di Dyfed selezionato della leucemia MOLM13 e MLL-AF9 utilizzando nucleare Kit di estrazione secondo il protocollo del produttore. Controllare l'espressione della proteina Cas9 utilizzando Anti-Flag M2 anticorpo dal Cas9 nel plasmide pLenti-Cas9 è collegato ad un epitopo Flag N-terminale.
- Caricare 50 µ g di proteina totale da ogni estratto nucleare sul 10% Bis-Tris del gel ed eseguire il gel a 120 V fino a bande superiore sono ben separati.
- Bloccare la membrana con soluzione al 5% latte nel buffer di TBST per 1 h.
- Incubare la membrana con 1:1,000 diluizione di anticorpo di Anti-Flag M2 ad una concentrazione finale di 1 µ g/mL a 4 ° C durante la notte.
- Giorno successivo, lavare la membrana con buffer di TBST e incubare con l'anticorpo secondario dei HRP coniugato anti-topo ad una diluizione di 1:5,000 (concentrazione finale di 0,16 µ g/mL) a temperatura ambiente per 1 h.
- Lavare la membrana accuratamente con TBST e sviluppare la macchia usando il substrato di ECL.
- Pick 3 – 4 cloni con la più alta espressione della proteina di Cas9 come visto mediante Western blotting per l'ulteriore analisi funzionale (Figura 1).
- Garantendo un'elevata attività Cas9 in cloni selezionati
- Preparare il surnatante lentivirali da un vettore di codifica sgRNA con sgRNAs targeting per l'essere umano o del mouse AAVS1 luogo di approdo sicuro come descritto nel passaggio 1.1.
- Trasducono alto MOLM13 Cas9-esprimendo o MLL-AF9 leucemia cloni con il surnatante di vettori lentivirali sgRNA anti-AAVS1 utilizzando il metodo di spinfection sopra descritto. Selezionare con 2,5 µ g/mL con puromicina per 4 giorni.
- Purificare il DNA genomico dai cloni di leucemia MOLM13-Cas9 o MLL-AF9-Cas9 dopo selezione con puromicina insieme ai rispettivi controlli di selvaggio-tipo utilizzando il kit di estrazione del DNA genomico seguendo le istruzioni del produttore. Uso 50 ng di DNA come un modello per eseguire una PCR con il AAVS1_test_primers utilizzando 2 x mix master di Taq polimerasi e il programma PCR elencati nella tabella 1.
- Eseguire il prodotto PCR su un gel di agarosio al 2% e gel-purificare la band di 268 base pair utilizzando un kit di estrazione del gel secondo il protocollo del produttore. Sequenza di Sanger il gel purificato prodotto di PCR utilizzando il primer AAVS1_target-frag_F.
- Valutare l'efficienza di editing dal confronto tra il AAVS1 modificato e wildtype sequenze utilizzando strumenti web-based online (Figura 2).
2. clonazione e trasduzione di sgRNAs in cellule di AML-Cas9
-
Velocità media effettiva clonazione di sgRNAs
- Progettazione sgRNAs di 4 – 6 per il gene di interesse utilizzando un software di progettazione CRISPR basata sul web. Ci sono una serie di strumenti di progettazione CRISPR disponibili online come http://crispr.mit.edu/ che genera sequenze di sgRNA basato su una sequenza di DNA input.
- Compilare un informazioni appropriate nei campi con gli asterischi. Fare clic sul genoma bersaglio per sgRNA design. Incollare alla sequenza di destinazione nella casella sequenza e fare clic sul pulsante Invia .
- Per la clonazione nella pKLV2-U6gRNA5 (BbsI) - PGKpuro2ABFP - plasmide di espressione sgRNA W, anteporre i nucleotidi "CACC" al senso oligo e "AAAC" per l'inverso oligo antisenso completato prima di ordinare. I oligos ordine premiscelato di senso e antisenso in una piastra a 96 pozzetti etichettati Oligo-Mix da un'azienda di sintesi del oligonucleotide.
Nota: Abbiamo fatto uso del pKLV2-U6gRNA5 (BbsI) - PGKpuro2ABFP - plasmide W, che è il sito di restrizione BbsI per sgRNA clonazione. Nel caso in cui vengono utilizzati altri plasmidi di espressione sgRNA, strapiombi utilizzati per i oligonucleotides sgRNA bisogno di essere cambiato di conseguenza. - Prendere una separato piastra a 96 pozzetti fondo U etichettata Piastra di ricottura. Fare un mix master di 1 µ l T4 PNK, 1 µ l di tampone di legatura del DNA T4 10x e 6 µ l di acqua per reazione di ricottura.
- Aggiungere 8 µ l di master mix in ciascun pozzetto della piastra ricottura. Aggiungere 1 µ l di 100 µM ciascuna, senso e oligo antisenso (o 2 µ l di misto senso-anti-senso oligos) per il mix master. Pipettare 2 – 3 volte delicatamente per amalgamare bene, far girare la piastra brevemente per ottenere le miscele di fondo dei pozzetti.
- Utilizzare il seguente programma di ricottura su una macchina PCR: 30 min a 37 ° C, 2 min 30 s a 95 ° C seguita da un raffreddamento lento a 22 ° C alla velocità di 0,1 ° C/s. Dopo la ricottura, prendere una piastra a 96 pozzetti fresca etichettata OligoMix diluito. In questo piatto, diluire i oligos fosforilati e temprato dalla reazione sopra con acqua (1: 200) usando una pipetta multicanale.
- Digest 5 µ g di pKLV2-U6gRNA5 (BbsI) - PGKpuro2ABFP - vettore W, con 1,5 µ l di enzima di restrizione BbsI (10.000 unità/mL), utilizzando un amplificatore adatto a 37 ° C per 2 h per linearizzare la per legatura più tardi.
- Prendere un piatto ben fresco 96 etichettati Piastra di legatura e aggiungere 20 ng della BbsI digerito pKLV2-U6gRNA5 (BbsI) - PGKpuro2ABFP - vettore di W da un pozzetto per la legatura sgRNA desiderato. Per questo, è necessario aggiungere 2 µ l di oligos fosforilato, temprato dalla piastra OligoMix diluito .
- Aggiungere 1 µ l di tampone di ligasi x T4 10 e 1 µ l di enzima T4 DNA ligasi. Delicatamente la pipetta con una pipetta multicanale e incubare il mix di legatura a temperatura ambiente per 2 h.
Nota: Miscele di legatura sono memorizzabili a-20 ° C per il passaggio di trasformazione più tardi. - Nel frattempo, disgelo 90 µ l di chimicamente competente Escherichia coli cellule su ghiaccio 10 min prima della fine della fase di legatura.
- Usando una pipetta multicanale, fare 10 aliquote µ l delle cellule competenti in ciascun pozzetto di una piastra ben 96 separata.
- Aggiungere la miscela di legatura da passo 2.1.8 nel bene contenente le cellule competenti, pipetta su e giù dolcemente e incubare per 10 minuti a temperatura ambiente.
- Pipettare 5 µ l della miscela di batteri-DNA direttamente dalla reazione di cui sopra in un 6 piastra ben contenente LB-agar con 100 µ g/mL di ampicillina. Ripetere per ciascuna delle reazioni di trasformazione.
- Ogni reazione di trasformazione in un separatamente etichettati bene di una piastra a 6 pozzetti della piastra. Aggiungere circa 5 – 8 perle di vetro a ogni bene e scuotere la piastra intera 6 pozzetti 8 – 10 volte in un movimento circolare.
- Scegli 1 – 2 singole colonie da ogni pozzetto con una pipetta sterile 20 µ l e striscia in un spot accuratamente etichettato su un sterile 10cm capsula di Petri con LB agar e ampicillina 100mg/mL. Dopo striature nella piastra LB, semplicemente espellere la punta utilizzata per clone striature in 3 mL di LB-ampicillina contenente il mezzo in un 14 mL provetta a fondo sferico contrassegnato con il numero corrispondente di clone batterica.
- Inviare la piastra batterica direttamente su Sanger sequenziamento con il primer forward del promotore umano U6.
- Dopo la conferma di sequenza di sgRNAs clonato, purificare il DNA dal tubo 14 mL corrispondenti utilizzando un kit di mini-preparazione secondo le istruzioni del produttore.
- Misurare la concentrazione e la qualità di ogni mini-preps con uno spettrofotometro. Normalizzare ogni mini-prep a una concentrazione di 15 ng / µ l e pipette in una piastra ben 96 etichettati sgRNA piastra di cloni.
Nota: Questo piatto può essere conservato a-20 ° C o utilizzato direttamente per la preparazione di virus.
- Progettazione sgRNAs di 4 – 6 per il gene di interesse utilizzando un software di progettazione CRISPR basata sul web. Ci sono una serie di strumenti di progettazione CRISPR disponibili online come http://crispr.mit.edu/ che genera sequenze di sgRNA basato su una sequenza di DNA input.
-
Produzione virale di sgRNA costrutti e trasduzione
- Per la produzione di particelle lentivirali sgRNA nel formato 96 pozzetti, seguire il "shRNA/sgRNA/ORF High Throughput produzione virale (ben 96)" protocollo dalla genetica piattaforma di perturbazione (GPP web Portal) del Broad Institute (URL: https:// Portals.broadinstitute.org/GPP/public/Resources/Protocols).
- Trasferire 200 µ l del surnatante virale in provette sterili e congelare a-80 ° C immediatamente.
- Giorno -1: Cappotto una piastra ben 96 di coltura trattata fondo piatto Non-tessuto con 100 µ l del frammento ricombinante fibronectina umana ad una concentrazione di 10 µ g/mL in una cappa di cultura cellulare BSL2. Avvolgere la piastra utilizzando pellicola trasparente per evitare la perdita di evaporazione e lasciarlo tutta la notte al banco.
- Giorno 0: Rimuovere il frammento di fibronectina umana ricombinante da ogni pozzetto. Scongelare il surnatante virale di ogni sgRNA a temperatura ambiente. Aggiungere 50 µ l del surnatante virale da ciascuna provetta a ciascuno rivestito bene della piastra di trasduzione. Girare la piastra di trasduzione a 1.300 x g per 90 minuti a 35 ° C.
- Verso la fine del spinfection, contare le cellule MOLM13-Cas9 (clone B3) dal matraccio di cultura. Utilizzare 10.000 cellule per pozzetto in 100 µ l di volume. Contare le celle per tutti i pozzi di trasduzione, tenendo conto di volume morto.
- Dopo il spinfection di 90 min, rimuovere il surnatante da tutti i pozzetti usando una pipetta multicanale regolata a 50 µ l lentamente inclinando la piastra. Aggiungere 100 µ l della coltura delle cellule clone B3 MOLM13-Cas9 per ogni bene lentamente scivolare giù dal cerchio.
- Girare la piastra a 1.300 x g per 2 minuti a 35 ° C e lasciare che le celle di depositarsi sul fondo. Trasferire la piastra per la coltura del tessuto grado Incubatore 37 ° C.
- Giorno 1: Aggiungere 100 µ l di terreno nuovo per ogni sgRNA contenente trasdotte Cas9-MOLM13 o Cas9-MLL-AF9 leucemia cellule tale che il volume medio finale è di 200 µ l.
3. saggio di crescita competitivo
- Giorno 3: 72 h (giorno 3) post di trasduzione, controllare la percentuale di sgRNA contenenti cellule positive di BFP in ciascun pozzetto mediante citometria a flusso (FACS). Utilizzare un colorante di attuabilità delle cellule per contrassegnare ed escludere le cellule morte dall'analisi.
- Continuare a ri-placcatura una proporzione delle cellule in nuovi pozzi con medium fresco dopo ogni analisi FACS per evitare la crescita eccessiva durante il dosaggio.
-
Ripetere l'analisi di FACS ogni 2 – 3 giorni per controllare la proporzione relativa delle cellule positive (BFP + ve) di BFP rispetto alla controparte (BFP-ve) negativa BFP (Figura 3). Analizzare la percentuale di cellule positive di BFP per ciascun punto di tempo (usando il FlowJo o simile software di analisi di FACS).
- Trascinare i file Fcs per ogni campione al software FlowJo. Fare doppio clic su qualsiasi file di un campione e tracciare una macchia del puntino di forward scatter (FSC) vs lato a dispersione (SSC) con FSC su asse X e SSC sull'asse Y. Tutte le celle del cancello.
- Fare doppio clic sulle celle con cancello e tracciarle su macchia di redditività (sull'asse Y) vs FSC altezza (H) o macchia del puntino di zona (A). Cancello l'attuabilità macchia cellule negative (dal vivo).
- Fare doppio clic su cellule vive con cancello e trama BFP (sull'asse Y) vs FSC (A) su asse X. Gate BFP + ve cellule. Applicare tutti questi cancelli a tutti i campioni trascinando il campione selezionato per Tutti i campioni sotto scheda gruppo .
- Fare clic su Editor di tabelle per fare un tavolo di analisi di tutti i campioni. Trascinare conteggio BFP + ve da un campione nella tabella e fare clic sul pulsante di visualizzazione nell' Editor tabella per creare un report di batch di tutti i campioni con una tabella che mostra la percentuale di cellule BFP + ve . Salvare la tabella come un file xls.
- Calcolare il proporzionale aumento o diminuzione cellule positive di BFP % all'interno della popolazione di cellule vive nel tempo, confrontare questo rapporto il rapporto delle cellule positive di BFP nel sgRNAs di controllo (ad esempio luciferasi, uova strapazzate o GFP sgRNA).
- Tracciare il rapporto delle cellule positive di BFP per i diversi sgRNAs tra cui i geni di interesse, nonché i controlli utilizzando il giorno 3 come base di riferimento.
Risultati
Nel nostro studio, abbiamo transduced in primo luogo la linea umana AML cellulare di MOLM13 che porta la traslocazione MLL-AF9 con alto-titolo virus codifica il plasmide lentivirale Cas9-Consciousness. Nelle nostre mani, alla rinfusa indifferenziati MOLM13-Cas9 cellule non venivano visualizzate alto livello Cas9 espressione mediante Western blotting e inoltre non hanno eseguito bene quando analizzati per efficiente gene modifica-utilizzando il metodo descritto in precedenza7. Di conseguenza, abbiamo proceduto a stabilire cloni singola cella e selezionare solo i cloni con alti livelli di Cas9 visto da Western blotting (Figura 1). Abbiamo scelto 2 cloni distinti e li trasdotte con sgRNAs attaccare un sito nel locus di approdo sicuro AAVS1 come descritto precedente7. Usando il DNA genomic estratto dal sgRNA selezionato con puromicina Cas9-MOLM13-AAVS1 cloni, abbiamo effettuato una PCR utilizzando primers che attraversa il sgRNA AAVS1 tagliata sito e sanger sequenziato un prodotto PCR bp 268 specifico da 10 colonie isolate per ogni clone di MOLM13. Dopo l'allineamento con la sequenza dei genitori, abbiamo trovato che il clone MOLM13-Cas9 B3 e MLL-AF9-Cas9 clone 8 aveva un'efficienza del 100% (dati non mostrati). Abbiamo quindi scelto questi cloni visualizzati da elevata capacità di editing genomico Cas9-mediata per nostri saggi di concorrenza sgRNA. Mentre questi studi sono stati condotti, strumenti web-based per verificare rapidamente l'efficienza editing genomico da sequenze di Sanger sono stati sviluppati da diversi gruppi come marea8 (https://www.deskgen.com/landing/tide.html) o ghiaccio (https://ice.synthego.com/#/). Questi strumenti rendono estremamente facile e conveniente per valutare efficienza modifica del genoma senza dover clonare frammenti individuali ed eseguire sequenziamento di più cloni. Pertanto, alla rinfusa che esprimono Cas9 cellule dovrebbero essere prima testate per genoma editing efficiente utilizzando questi metodi. Nel caso in cui l'efficienza di editing genomico è alta, quindi cella singola clonazione non è necessaria. Inoltre, nel caso che la clonazione singola cella è necessaria come visto nei nostri studi, questi strumenti di stima di frequenza mutazionale basata sul web offrono un metodo molto più veloce per testare diversi cloni in parallelo.
Per la clonazione di sgRNA, abbiamo fatto uso di sgRNA clonazione del plasmide pKLV2-U6gRNA5 (BbsI) - PGKpuro2ABFP - W, che ospita una proteina fluorescente blu (BFP) per tenere traccia delle cellule trasformata sgRNA e un U6 promotore umano RNA polimerasi III-driven che spinge l'espressione di il sgRNAs clonato insieme l'impalcatura di RNA (tracRNA) di tracciante. Abbiamo usato questo sistema per clonare 2 sgRNAs DOT1L, una proteina conosciuta per svolgere un ruolo importante nella regolazione epigenetica dell'espressione genica di targeting. DOT1L è un'istone metiltransferasi che deposita mono, di e tri-metilazione a destinazione geni9,10. Studi hanno dimostrato che DOT1L gioca un ruolo importante in AML guidato dagli oncogeni MLL-fusione. Pertanto, DOT1L knockout utilizzando CRISPR-Cas9 dovrebbe portare ad per alterare significativamente la proliferazione delle cellule del mouse MLL-AF9 leucemia in linea con precedenti studi11,12,13,14. Abbiamo usato due sgRNAs il locus di porto sicuro di AAVS1, che è nel introne del gene di PPP1R12C di targeting. sgRNAs producendo alterazioni genomiche in questo sito non sono suscettibili di avere effetti sulla capacità proliferativa delle linee cellulari di MLL-leucemia. Come controllo positivo, abbiamo clonato sgRNAs targeting per il gene di replica-collegato del DNA RPA3. RPA3 è un gene di pan-essenziale che ha dimostrato di essere importanti per la proliferazione di diversi AML cella linee4,15,16 . Anti-RPA3 sgRNAs pertanto visualizzare forti effetti anti-proliferativi in cellule di AML e vengono in genere utilizzati come controllo positivo. Dopo la trasduzione con plasmidi sgRNA anti-AAVS1 e anti-RPA3, abbiamo misurato la proporzione relativa di BFP + ve sgRNA trasdotte cellule rispetto alle loro controparti di BFP-ve ogni 2 – 3 giorni nella cultura (Figura 3). Con il protocollo descritto in precedenza, la trasduzione delle cellule MOLM13 o MLL-AF9 AML è provocato da un'efficienza di trasduzione di 60 – 70% con le nostre preparazioni virale, lasciando le cellule restanti di 30 – 40% untransduced o BFP-ve in ogni pozzetto. In questo modo per lo studio della relativa proliferazione delle cellule di genoma-modificato con selvaggio-tipo celle nello stesso bene. Il proporzionale aumento o diminuzione nella percentuale delle cellule trasformata sgRNA è stato usato come una misura per riflettere l'effetto funzionale di sgRNA mediato omissione del gene nelle cellule MOLM13-Cas9. Se il gene di interesse è importante per la proliferazione delle cellule AML del test, quindi rottura sgRNA-mediata di questo gene porterà alla diminuzione relativa del BFP che esprimono sgRNA + ve cellule rispetto alle cellule del BFP-ve nel corso del test, considerando che sgRNAs targeting luciferasi, GFP o geni non essenziali manterrà il BFP + ve-ve rapporto nel corso del tempo.
Utilizzando 2 separati anti-RPA3 sgRNAs, abbiamo osservato un declino progressivo e significativo della percentuale di BFP + ve cellule rispetto al BFP-ve untransduced controparti. Al contrario, la percentuale di sgRNA anti-AAVS1 che esprimono le cellule BFP rimasta relativamente costante nel tempo, dimostrando che il targeting di AAVS1 non ha alcun effetto sulla proliferazione delle cellule MOLM13 (Figura 4a). Allo stesso modo, nelle cellule del mouse MLL-AF9-Cas9, abbiamo testato gli effetti di sgRNAs rodopsina (Rho1), il gene di pigmentazione dell'occhio come controllo negativo e Dot1l, un regolatore epigenetico, noto per essere necessaria per la proliferazione delle cellule di leucemia di MLL-AF9 nel tempo . In questo studio, BFP + ve mouse MLL-AF9-Cas9 cellule trasformate con 2 separati anti-DOT1L sgRNAs hanno mostrato una perdita drammatica e progressiva nel tempo rispetto alle cellule di BFP-ve sgRNA non trasformata (Figura 4b). Al contrario, il rapporto di anti-Rho1 sgRNA è rimasto relativamente immutato nel tempo. Questi risultati dimostrano la vulnerabilità di MLL-AF9 che esprimono le cellule di leucemia del mouse alla deplezione di Dot1l, confermando i risultati precedentemente pubblicati.

Figura 1: rappresentante Western blot risultati mostrando diversi livelli Cas9. Cloni di singola cellula della linea cellulare MOLM13 AML trasformata con CAS9 sono stati sondati per i livelli di espressione di bandiera-Cas9 usando gli anticorpi Anti-Flag. Cloni mostrando la massima espressione del Cas9 sono stati selezionati per ulteriori studi. HEK293-T cellule trasfettate con un Cas9 plasmide di espressione sono stati utilizzati come controllo positivo e Cas9 non trasdotte MOLM13 cellule come controlli negativi. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: analisi rappresentativa del genoma editing presso il locus Dot1l valutato dall'analisi di ICE. Un amplicone PCR centrato intorno al sito di destinazione sgRNA Dot1l Sanger sequenziato e analizzati usando l'analisi di ghiaccio. Un confronto tra la sequenza di sgDot1l (linea arancione) alla sequenza di DNA non mirati (linea verde) dimostra l'alto livello di efficienza in cellule di sgDot1l mirati attorno al sito di destinazione sgRNA di editing. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: flusso di lavoro schematico dei metodi proposti. sgRNAs vengono clonati nel vettore di espressione sgRNA co-che esprime una proteina fluorescente come BFP. Cellule bersaglio sono transduced al prezzo di 30 – 60% trasduzione e seguite da citometria a flusso ogni 2 – 3 giorni. sgRNAs geni richiesti per la proliferazione delle cellule AML di targeting mostrerà relativo svuotamento nel tempo come mostrato. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: rappresentante deriva da saggi di competizione in umani e cellule di topo AML. (un) risultati mostrano un progressivo statisticamente altamente significativo declino in RPA3 trasdotte cellule clone B3 MOLM13-Cas9. Al contrario, sgRNAs targeting per il sito AAVS1 non hanno effetto. (b) sgRNAs Dot1l di targeting mostrano un notevole e progressivo declino nella proliferazione competitiva, in contrasto con sgRNAs rodopsina (Rho). p > 0.05. p < 0.05. Barre di errore rappresentano la deviazione standard della media (deviazione standard). Clicca qui per visualizzare una versione più grande di questa figura.
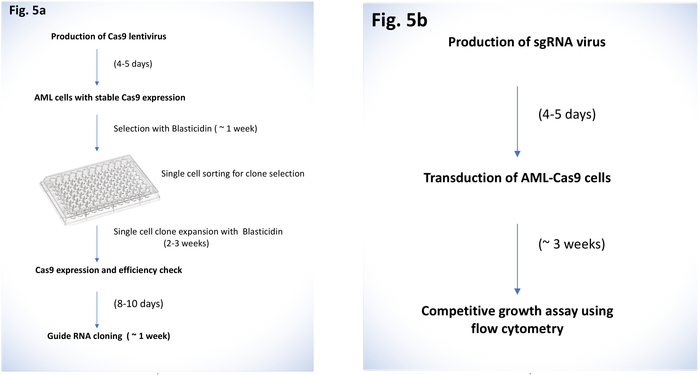
Figura 5: schema generale del protocollo. (una) volta per ogni passo nella produzione di virus Cas9 e sgRNA clonazione descritto. Design e clonazione di sgRNAs può essere eseguite durante la generazione di singoli cloni di linee cellulari AML con espressione Cas9 stabile. il protocollo generale (b) per la trasduzione delle linee cellulari AML con sgRNAs e crescita competitiva analisi usando FACS è mostrato. Clicca qui per visualizzare una versione più grande di questa figura.
| Cicli | Durata del ciclo | Temperatura |
| 1 | 2 min | 95 ° C |
| 35 | 30 s | 95 ° C |
| 30 s | 55 ° C | |
| 30 s | 72 ° C | |
| 1 | 5 min | 72 ° C |
| Tenere premuto | infinito | 4 ° C |
Tabella 1: programma PCR per l'amplificazione locus AAVS1 da DNA Genomic. Programma PCR per l'amplificazione del locus AAVS1 da DNA genomic per test efficienza di taglio di Cas9 in Cas9 esprimendo cloni.
File supplementari: sequenze oligo sgRNA. Per favore clicca qui per scaricare questo file.
Discussione
In questo manoscritto, descriviamo un protocollo dettagliato per lo svolgimento di un'analisi di crescita competitiva basati su CRISPR-Cas9 per studiare il ruolo di geni candidati in linee cellulari AML mediante citometria a flusso in cellule di AML umano/murino (Figura 5). L'obiettivo del test è di identificare l'effetto della delezione del gene sulla manutenzione di proliferazione delle cellule AML oltre due o tre settimane su scala medio-velocità di trasmissione. Alcuni passaggi critici devono essere seguite attentamente per facilitare la scalabilità del protocollo descritto. Nella produzione di Cas9 lentivirus, è necessario filtrare il mezzo virus condizionata attraverso un filtro da 0,45 µM per evitare il riporto delle cellule 293T al virus contenente il surnatante. Durante spinfection, avvolgendo la piastra spinfection con pellicola trasparente o parafilm consente di evitare potenziali contaminazioni durante la centrifugazione. Tuttavia, l'impacco dovrebbe essere rimosso prima di posizionare la piastra di spinfected torna in incubatrice di coltura del tessuto. È molto importante valutare i cloni che esprimono Cas9 per efficienza di editing. Cloni con efficienza di editing di basso-genoma riflettono insufficiente Cas9 attività e possono influenzare il tasso di successo dell'esperimento. Nei nostri esperimenti, abbiamo prima trasdotte cloni umani AML Cas9 con targeting per il locus AAVS1 sgRNAs ed ordinato loro DNAs genomic per editing efficiente. Confronto tra il AAVS1 modificato e wildtype sequenze possono essere valutati utilizzando strumenti basati sul web come marea8 (https://www.deskgen.com/landing/tide.html) o ghiaccio (https://ice.synthego.com/#/). Questi programmi confrontare Sanger sequenza tracce di PCR potenzialmente modificato frammentano alla sequenza wildtype o riferimento inedita e stimano la frequenza delle modifiche. Si tratta di un modo rapido di valutazione mutational cambiamenti apportati dalla sgRNA prova, che è la misura dell'attività cellulare Cas9. In alternativa, la clonazione del PCR prodotto in un plasmide di clonazione di PCR come il TOPO smussato clonazione vettoriale può essere eseguito, seguita da Sanger sequenziamento di diverse colonie di valutare efficienza modifica del genoma di allineamento di sequenza di ogni clone per la selvaggio-tipo. Noi preferiamo il metodo precedente, dato la sua facilità e la convenienza economica rispetto al metodo di clonazione. La scoperta della coppia di base cambia in genere, quali mutazioni puntiformi, indels, ecc., alle, o intorno al sito di taglio sgRNA nella maggior parte dei cloni sequenziati indicano la presenza di un Cas9 altamente attiva. Così, abbiamo scelto di leucemia MOLM13 o MLL-AF9 cloni B3 e 8, rispettivamente, con la più alta efficienza di editing per ulteriori esperimenti.
Abbiamo progettato sgRNAs targeting diversi geni menzionati nel protocollo utilizzando http://crispr.mit.edu/, un software web-based che fornisce un elenco di sgRNAs. Si consiglia di selezionare sgRNAs massimi dall'elenco al fine di eliminare quelli con probabili effetti fuori bersaglio. Il sgRNAs progettato può essere clonata utilizzando i protocolli pubblicati come quello sul sito Web (http://www.addgene.org/67974/). Senso e antisenso oligos sono prima fosforilato e ricotto secondo il punto 2.1.5. Il primo passo a 37 ° C è importante per fosforilante oligos con la chinasi T4 e successivi cicli di PCR sono importanti per la ricottura del senso e oligonucleotidi anti-senso in dsDNA. È importante avere una proteina fluorescente nel vettore di clonazione di sgRNA che è fondamentale per tenere traccia delle cellule trasdotte sgRNA più tardi nelle analisi concorrenza. sgRNA clonazione è scalata con l'uso di ben 96 PORTATARGA presso tutti i passaggi tra cui ricottura e legatura. Per la legatura, pulire microprovetta PCR strisce possono essere utilizzati anche dal momento che sono compatibili con pipette multicanale. In caso che la trasformazione di un insieme più ampio di cloni sgRNA è necessario, una piastra a 96 pozzetti in cui 10 μL delle cellule competenti sono pre-aliquotato in ciascun pozzetto e congelati a-80 ° C può essere utilizzato per accelerare l'intero processo. Lo scopo della placcatura le reazioni di legatura è di scegliere diverse colonie, che è difficile da eseguire nel formato 96 pozzetti. Quindi, per la trasformazione batterica, c'è una leggera perdita in termini di scalabilità. Ancora, anche per la placcatura di reazioni di trasformazione da un'intera piastra a 96 pozzetti, occorrerà solo un totale di sedici piastre da 6 pozzetti per l'intero progetto. Bisogna stare attenti nella fase successiva di picking colonie delle placche in trasformazione. Mentre striarsi una colonia su un posto etichettato, assicurarsi che il posto sia chiaramente isolato da altri punti. Marcatura di una griglia di quadrati sul fondo del piatto Petri con un pennarello scuro aiuta a prevenire la contaminazione incrociata di cloni batterici.
Una volta pronti i minipreps di sgRNA costrutti, il virus può avvenire nel formato 96 pozzetti. A questo livello di velocità effettiva, non è pratico per filtrare 200 µ l di virus contenente il surnatante. Quindi, il surnatante virale deve essere congelato almeno una notte per evitare la contaminazione incrociata da eventuali cellule 293T esportato nel surnatante. Può anche essere conservato in 50 aliquote µ l nella provetta sterile da PCR strisce per evitare congelamento scongelamento del surnatante. Ulteriormente, questi surnatanti di virus condizionata sono utilizzati per trasdurre cellule AML. Nel caso in cui che bassi titoli di trasduzione virale sono osservati, come valutato dalla bassa frequenza di BFP + ve le cellule, quindi può essere importante determinare il titolo virale e trasduzioni test alle diverse molteplicità delle infezioni (MOI) per garantire il 40-60% tariffe di trasduzione. Determinazione del titolo virale e MOI calcolo è descritto in dettaglio qui17. Nell'analisi della concorrenza, come descritto nel passaggio 3 del protocollo, è altamente importante escludere le cellule non vitali per evitare artefatti spurie da auto-fluorescenza mentre si analizza il BFP rapporto non-BFP. Al momento di FACS, prima di analizzare campioni di prova, l'uso di BFP negativo e controlli positivi BFP è altamente consigliato di impostare con precisione le tensioni. Ogni punto di tempo ri-placcatura, il volume delle cellule per essere ri-placcato dipende dalla concentrazione delle cellule presso il punto di tempo determinato. Abbiamo in genere ri-placcato μL 20 su un totale di 200 μL in ogni momento in un pozzo fresco con 180 µ l di terreno nuovo. Si consiglia vivamente di accurata miscelazione delle cellule pipettando delicatamente su e giù prima di ri-placcatura. In genere, abbiamo osservato che il dosaggio deve essere effettuato oltre il 15 – 20 giorni di preavviso forte cambia nel BFP + ve-ve rapporti, anche se questo varia da un gene per gene.
Questo set-up sperimentale si presta bene alla sperimentazione di diversi geni in parallelo in linee cellulari AML multiple per identificare rapidamente il ruolo di geni candidati nella sopravvivenza o la proliferazione delle cellule di AML. Un avvertimento del dosaggio competitivo proliferazione descritto qui è che non considera fattori potenziali cella-estrinseca come paracrine segnalazione eventi dalle cellule gene targeting che possono influenzare la popolazione di cellule non mirati all'interno dello stesso bene. Anche se questo potrebbe essere un avvenimento raro, questo fattore da tener presente quando lo svolgimento di questo test. Anche se abbiamo usato AML come esempio per le analisi di concorrenza basati su CRISPR-Cas9 descritte in questo manoscritto, questo metodo può essere utilizzato per qualsiasi linea cellulare di cancro per identificare il ruolo dei geni multipli in parallelo. Ci sono diversi vantaggi di omissione del gene usando CRISPR-Cas9 sopra gene-atterramento mediante RNA interference. In primo luogo, in contrasto con shRNA, che in genere mostra una vasta gamma di inibizione di mRNA bersaglio, sgRNAs accoppiato con la nucleasi Cas9 effettuare le operazioni di eliminazione completa dei geni bersaglio. Ciò potrebbe comportare fenotipi più drammatici e coerenti con i metodi basati su CRISPR-Cas9, anche se devono essere avvertito che l'efficienza di targeting di sgRNAs individuali così come shRNA può variare ampiamente.
L'analisi di flusso cytometry-concorrenza basata offre parecchi vantaggi sopra analisi di proliferazione tradizionale in cui un numero fisso di cellule è placcato per misurare la relativa attività proliferativa delle cellule gene perturbato. Un vantaggio è che la relativa attività proliferativa delle cellule può essere facilmente e rapidamente misurata da citometria a flusso per diversi giorni, evitando la necessità per cella conteggio ad ogni passo di placcatura. Questo è utile quando si prova un gran numero di candidati sgRNAs parecchi geni, come il conteggio di tutti i pozzetti di targeting è ingombrante e può condurre alle inesattezze. A differenza di, misura basate su ATP dosaggi per la crescita cellulare, citometria a flusso può essere eseguita su un campione di cellule vive, che rende questo metodo utile per analisi a lungo termine. Questo è particolarmente utile nello studio dei fattori come modulatori epigenetici, che possono mostrare effetti fine effetto sulla proliferazione delle cellule AML e possono richiedere saggi a lungo termine. In secondo luogo, la presenza di cellule non trasdotte all'interno dello stesso bene permette per una popolazione di cellule ben controllato che può essere utilizzata per impostare la previsione per il dosaggio. In terzo luogo, quando il set-up in un formato di 96 pozzetti, l'analisi può essere condotta quasi completamente utilizzando pipette multicanale, sostanzialmente accelerare l'intero processo dalla clonazione di sgRNAs per preparazione di virus e, infine, valutazione di flusso-cytometric di fluorescenza.
Il metodo qui descritto può essere efficientemente su vasta scala per l'indagine fino a 96 sgRNAs in parallelo in un schermo disposte. Supponendo che sgRNAs 4 – 6 per gene, questo metodo può essere pertanto utilizzato per l'interrogatorio rapida di almeno 16 – 24 geni in parallelo. In caso di più grande numero di geni, come un intero percorso molecolare che deve essere interrogato in cellule di AML, schermi basati su CRISPR-Cas9 pool sarà più utili.
Divulgazioni
A.J.D è un consulente di A2A pharmaceuticals (New Jersey) e Salgomed Therapeutics (San Diego). Altri autori non hanno nessun conflitto di dichiarare.
Riconoscimenti
Il plasmide pCW-Cas9 era un regalo da Eric Lander & David Sabatini (plasmide Addgene # 50661) e il pKLV2-U6gRNA5 (BbsI) - PGKpuro2ABFP - plasmide W dal laboratorio Yusa (plasmide Addgene #67974. Vorremmo ringraziare il nucleo di citometria a flusso a SBP Medical Discovery Institute per il tempestivo aiuto con l'analisi di flusso e l'ordinamento. Vorremmo riconoscere il sostegno di Lady Tata Memorial Foundation a A.D. Vorremmo anche riconoscere il sostegno delle seguenti fonti di finanziamento: NIH/NCI P30 CA030199 Cancer Center sponsorizzato Grant, la V-Fondazione e i centri di cancro NCI (C3) di San Diego #PTC2017to A.J.D.
Materiali
| Name | Company | Catalog Number | Comments |
| FLAG-M2 Antibody | sigma-aldrich | F3165, lot # SLBS3530V | |
| Anti-mouse Antibody | Invitrogen | 31446, lot # TA2514341 | |
| SuperSignal West Femto Maximum Sensitivity Substrate | Thermo Fisher | 34095 | |
| ChemiDoc Imaging System | BIO RAD | 17001401 | |
| Sorvall Legend RT centrifuge | Thermo Scientific | ||
| Blasticidin | Thermo Fisher | R21001 | |
| SYTOX Red | Thermo Fisher | S34859 | |
| Opti-MEM | Thermo Fisher | 31985062 | |
| DMEM | Thermo Fisher | 11965-092 | |
| RPMI | Thermo Fisher | 11875-093 | |
| Penicillin-Streptomycin | Thermo Fisher | 15140122 | |
| L-Glutamine (200 mM) | Thermo Fisher | 25030081 | |
| Fetal Bovine Serum (FBS) | SAFC | 12303C | |
| single gRNA vector | Addgene #67974 | pKLV2-U6gRNA5(BbsI)-PGKpuro2ABFP-W | |
| CelLytic Nuclear extraction kit | sigma-alorich | NXTRACT | |
| XtremeGENE 9 | sigma-alorich | 6365787001 | |
| Retronectin | Takara | T100B | |
| Flow cytometer | BD Biosciences | ||
| T4 PNK | NEBioLabs | M0201S | |
| T4 DNA ligation buffer | NEBioLabs | B0202S | |
| T4 DNA Ligase enzyme | NEBioLabs | M0202S | |
| Ampicillin | Fisher scientific | BP1760-25 | |
| LB agar | Fisher scientific | BP9724-500 | |
| LB Broth | Fisher scientific | BP9731-500 | |
| Qiagen mini-prep kit | Qiagen | 27104 | |
| NanoDrop Spectrophotometer | Thermo Fisher | NanoDrop One | |
| Recombinant Murine IL-3 | Peprotech | 213-13 | |
| Recombinant Murine IL-6 | Peprotech | 216-16 | |
| Recombinant Murine M-CSF | Peprotech | 315-02 | |
| Stable competent cells | NEBiolabs | C3040I | |
| 10 cm Tissue Culture dishes | Fisher Scientific | 353003 | |
| Cell lysis solution | Qiagen | 158906 | |
| Protein precipitation solution | Qiagen | 158910 | |
| DNA hydration solution | Qiagen | 158914 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| BbSI | New England BioLabs | R0539S | |
| APEX 2.0x Taq Red Master Mix Kit | Genessee Scientific | 42-138 | |
| Puromycin | Fisher scientific | BP2956100 | |
| 50 mL polypropylene conical tubes | Fisher scientific | 1495949A | |
| 15 mL polypropylene conical tubes | Fisher scientific | 1495970C |
Riferimenti
- Mali, P., Esvelt, K. M., Church, G. M. Cas9 as a versatile tool for engineering biology. Nature Method. 10 (10), 957-963 (2013).
- Doudna, J. A., Charpentier, E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096(2014).
- Wang, H., La Russa, M., Qi, L. S. CRISPR/Cas9 in Genome Editing and Beyond. Annual Review of Biochemistry. 85, 227-264 (2016).
- Shi, J., et al. Discovery of cancer drug targets by CRISPR-Cas9 screening of protein domains. Nature Biotechnology. 33 (6), 661-667 (2015).
- Kuhn, M. W., et al. Targeting Chromatin Regulators Inhibits Leukemogenic Gene Expression in NPM1 Mutant Leukemia. Cancer Discovery. 6 (10), 1166-1181 (2016).
- Erb, M. A., et al. Transcription control by the ENL YEATS domain in acute leukaemia. Nature. 543 (7644), 270-274 (2017).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Brinkman, E. K., et al. Easy quantification of template-directed CRISPR/Cas9 editing. Nucleic Acids Research. 46 (10), 58(2018).
- Nguyen, A. T., Zhang, Y. The diverse functions of Dot1 and H3K79 methylation. Genes & Development. 25 (13), 1345-1358 (2011).
- Vlaming, H., van Leeuwen, F. The upstreams and downstreams of H3K79 methylation by DOT1L. Chromosoma. 125 (4), 593-605 (2016).
- Bernt, K. M., et al. MLL-rearranged leukemia is dependent on aberrant H3K79 methylation by DOT1L. Cancer Cell. 20 (1), 66-78 (2011).
- Daigle, S. R., et al. Selective killing of mixed lineage leukemia cells by a potent small-molecule DOT1L inhibitor. Cancer Cell. 20 (1), 53-65 (2011).
- Jo, S. Y., Granowicz, E. M., Maillard, I., Thomas, D., Hess, J. L. Requirement for Dot1l in murine postnatal hematopoiesis and leukemogenesis by MLL translocation. Blood. 117 (18), 4759-4768 (2011).
- Nguyen, A. T., Taranova, O., He, J., Zhang, Y. DOT1L, the H3K79 methyltransferase, is required for MLL-AF9-mediated leukemogenesis. Blood. 117 (25), 6912-6922 (2011).
- Zuber, J., et al. Toolkit for evaluating genes required for proliferation and survival using tetracycline-regulated RNAi. Nature Biotechnology. 29 (1), 79-83 (2011).
- Wang, T., et al. Identification and characterization of essential genes in the human genome. Science. 350 (6264), 1096-1101 (2015).
- Li, M. J., Rossi, J. J. Lentivirus transduction of hematopoietic cells. CSH Protocols. 2007, pdb prot4755 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon