Method Article
Síntese e determinação da estrutura de µ-conotoxina PIIIA isômeros com bissulfeto diferentes conetividades
* Estes autores contribuíram igualmente
Neste Artigo
Erratum Notice
Resumo
Rica em cisteína peptídeos dobram em distintas estruturas tridimensionais dependendo sua conectividade de dissulfeto. Síntese alvo de isómeros de bissulfeto individual é necessária quando oxidação de reserva não implica que a conectividade de bissulfeto desejado. O protocolo lida com a síntese seletiva de peptídeos 3-bissulfeto-ligado e sua análise estrutural usando estudos NMR e MS/MS.
Resumo
Peptídeos com um elevado número de cisteínas geralmente são influenciados em relação a estrutura tridimensional por sua conectividade de bissulfeto. Assim, é altamente importante para evitar a formação de ligação dissulfeto indesejáveis durante a síntese do peptide, porque pode resultar em uma estrutura completamente diferente do peptide e consequentemente alterado Bioatividade. No entanto, a correta formação de múltiplas ligações de bissulfeto em um peptídeo é difícil de obter por meio de métodos Self dobramento padrão tais como protocolos de oxidação do amortecedor convencional, porque vários conetividades dissulfeto podem ser formadas. Este protocolo representa uma estratégia avançada necessária para a síntese de alvo de múltiplas em ponte dissulfeto de peptídeos que não pode ser sintetizado através da oxidação de reserva na alta qualidade e quantidade. O estudo demonstra a aplicação de uma estratégia de grupo protegendo distintas para a síntese de todos os isómeros possíveis do peptide 3-bissulfeto-ligado de µ-conotoxina PIIIA de forma orientada. Os peptídeos são preparados pela síntese de peptídeo de fase sólida baseada em Fmoc usando uma estratégia de grupo protegendo para formação de ligação dissulfeto definidos. Os respectivos pares de cisteínas estão protegidos com trityl (Trt), acetamidomethyl (Acm) e tert-butílico (tBu) protegendo grupos para certificar-se que, durante cada etapa de oxidação somente as necessárias cisteínas são deprotected e ligadas. Além da síntese de alvo, uma combinação de vários métodos analíticos é usada para esclarecer o correto dobramento e geração das estruturas de peptídeo desejado. A comparação dos diferentes isômeros 3-bissulfeto-ligado indica a importância da determinação precisa e conhecimento da conectividade bissulfeto para o cálculo da estrutura tridimensional e a interpretação do biológico atividade dos isômeros do peptide. A caracterização analítica inclui a elucidação de ligação dissulfeto exata através de análise de espectrometria de massa (MS/MS) em tandem, que é realizado com derivados alquilados e parcialmente reduzidos do isômero intacta peptídeo produzido por um protocolo adaptado. Além disso, as estruturas do peptide são determinadas usando 2D da ressonância magnética nuclear (NMR) experiências e os conhecimentos resultantes da análise de MS/MS.
Introdução
O uso de peptídeos bioativos em pesquisa farmacêutica e desenvolvimento é altamente reconhecido, porque eles representam compostos potentes e altamente seletivos para alvos biológicos específicos1. Para sua bioatividade, no entanto, a estrutura tridimensional é de grande importância a fim de realizar a relação estrutura-atividade estudos2,3,de4. Além da sequência de aminoácidos primários que influencia a conformação geral, ligações de bissulfeto significativamente estabilizem a estrutura de peptídeos ricos em cisteína5. Vários peptídeos em ponte dissulfeto incluem conotoxins como µ-PIIIA de Conus purpurascens que contém seis cisteínas na sua sequência. Este conteúdo elevado de cisteína, teoricamente, permite a formação de 15 isómeros de dissulfeto. A conectividade de bissulfeto correta é muito importante para a atividade biológica6,7. No entanto, a pergunta que surge é se há mais de uma conformação Bioativo de peptídeos naturais e se então, que os isómeros possui a maior atividade biológica? No caso de µ-conotoxins, os alvos biológicos são canais de íon sódio voltagem-dependentes, e µ-PIIIA em particular é mais potente para subtipos de nd ndV1.2,V1.4 e atV1.73.
A síntese de peptídeos em ponte dissulfeto pode ser conseguida usando vários métodos. O método mais conveniente para a formação de ligações de bissulfeto dentro de um peptídeo é a chamada abordagem Self dobramento oxidativa. Aqui, o precursor linear do peptide cíclico desejado é sintetizado primeiro usando a síntese do peptide de fase sólida, que é após a clivagem do suporte polimérico submetidos a oxidação em um sistema de buffer. Agentes de redox-ativo como glutationa oxidada e reduzida (GSH/GSSG) são frequentemente adicionados para promover a formação das ligações de bissulfeto. A principal desvantagem do self com suporte buffer de dobramento é que as ligações dissulfureto são formadas não seletivamente de forma gradual. Comparado com o peptídeo nativo, para o qual muitas vezes é descrito isômero bissulfeto específico apenas um, é possível obter inúmeros outros isômeros com esta abordagem8. µ-PIIIA já foi mostrado para resultar em pelo menos três isômeros diferentemente dobrados sobre self dobrar em um estudo anterior3. A separação de tal uma mistura de isômero é um pouco difícil devido a tempos de retenção similares se utilizando de métodos cromatográficos de purificação9. A síntese de alvo de um isómero específico, portanto, é vantajosa. Para especificamente produzir que um isômero com conectividade de bissulfeto definido, uma estratégia especial é necessário que o dissulfeto títulos sucessivamente estão fechadas. Portanto, o precursor linear carregando grupos distintos protegendo os pares individuais cisteína é sintetizado com o apoio do polímero. Após a eliminação, os pares de cisteína são individualmente e sucessivamente deprotected e ligados em uma reação de oxidação para produzir o desejado dissulfeto títulos10,11,12,13, 14 , 15 , 16. após a síntese e a purificação do produto da reação, é necessário para confirmar a identidade e a conectividade de dissulfeto por métodos analíticos apropriados. Numerosos métodos analíticos estão disponíveis para a elucidação da sequência primária de aminoácidos, por exemplo., MS/MS, enquanto a determinação da conectividade bissulfeto continua a ser muito menos investigada. Além da complexidade de tais vários peptídeos dissulfeto-ligado, impurezas relacionados com o produto (ex., de bissulfeto lutando), devido à amostra preparação e check-up podem complicar ainda mais as análises. Neste trabalho, mostramos que o uso de uma combinação de diferentes técnicas analíticas é necessário clarificar inequivocamente a identidade das ligações de bissulfeto nos isómeros µ-PIIIA. Nós temos combinado métodos de cromatografia em fase gasosa com espectrometria de massa e desde que as mesmas amostras para espectroscopia RMN. Em matrix-assisted laser desorption/ionization(MALDI) MS/MS análise, identificamos as ligações de bissulfeto usando redução parcial e iodoacetamide derivatização porque análise top-down, não é possível para este peptídeo. 2D NMR experimentos foram realizados a fim de obter uma estrutura tridimensional de cada isômero. Assim, combinando diferentes métodos analíticos sofisticados, é possível elucidar corretamente a conectividade de bissulfeto e estrutura tridimensional do complexo vários peptídeos dissulfeto-ligado7.
Protocolo
Nota: Todos os aminoácidos utilizados neste documento foram na L-configuração. As abreviaturas de aminoácidos e aminoácidos derivados foram utilizadas de acordo com as recomendações do Comité de nomenclatura de IUB e a Comissão conjunta de IUPAC-IUB na nomenclatura Biochemical.
1. síntese do Peptide solid-Phase (SPPS)
Nota: Efectuar a síntese com um sintetizador de peptídeo de fase sólida. Realize a síntese dos precursores do peptide linear da sequência geral ZRLCCGFOKSCRSRQCKOHRCC-NH2 usando um protocolo padrão para a química de 9-fluorenylmethyloxycarbonyl (Fmoc). Aplicar os seguintes aminoácidos protegidos: Pyr (Boc (terc-butyloxycarbonyl)), Arg (Pbf (2,2,4,6,7-pentamethyldihydrobenzofuran-5-sulfonil)), Asn(Trt), Asp(tBu), Hyp(tBu), Lys(Boc), Ser(tBu), Gln(Trt), Glu(tBu), Trp(Boc), Tyr(tBu), Thr(tBu) e seus (Trt). Protege os pares de cisteína com Trt, Acm ou tBu-grupos de acordo com a conectividade de bissulfeto pretendido.
-
Preparação
- Secar a resina Fmoc Rink-Amida (carregamento: 0,28 mmol/g) usando um Liofilizador durante a noite.
- Digite a sequência de peptídeo desejado (código 1-carta) para o programa do sintetizador. O programa calcula a quantidade necessária para cada reagente individual e indica as quantidades de solventes.
- Pesar os reagentes individuais (aminoácidos, HBTU (2-(1H-benzotriazol-1-yl)-1,1,3,3-tetramethyluronium hexafluorofosfato)) de acordo com o protocolo e dissolvê-los em dimetilformamida (DMF) a uma concentração final de 2,4 M (aminoácidos) e 0,6 M ( HBTU), respectivamente.
- Transferir os reagentes diferentes (aminoácidos, HBTU, N-methylmorpholine (NMM, 50% em DMF), piperidina (20% em DMF), DMF, diclorometano (DCM)) para os vasos correspondentes e colocá-los na venda de apropriado do sintetizador peptídeo de fase sólida.
- Adicione 100 mg de resina seca para as colunas de reação e colocá-los no ramo do sintetizador. Inicie a síntese do peptide de fase sólida.
-
SPPS-protocolo (fornecido pelo sintetizador)
Nota: O protocolo aplica-se a 100 mg de resina (carregamento: 0,28 mmol/g) adicionado à coluna uma reação para 28 escala µmol.- Preparação da resina
- Enxágue a resina com 2500 µ l de DMF, 1400 µ l do DCM e 1400 µ l de DMF.
- Irrigue a resina com ar até o solvente é removido e passe a resina com 2500 µ l de DMF.
- Clivagem do Fmoc protegendo o grupo
- Adicionar 20% piperidina em DMF (1000 µ l) para a resina e espere por 6 min. remover a solução da resina. Repita a etapa 1.2.2.1.
- Lave duas vezes com DMF (1 µ lst 4000, 2 µ lnd1400), irrigue a resina com ar até o solvente é removido e lave duas vezes com 2000 µ l de DMF.
- Reação de acoplamento
- Misture os seguintes reagentes em um frasco separado: HBTU (450 µ l; 0,6 M em DMF; 270 µmol; 9.6 EQ.), NMM (125 µ l; 50% em DMF; µmol 568; 20 EQ.), Fmoc-amino ácido (420 µ l; 2,4 M em DMF; 1.01 mmol; 36 EQ.). Adicione a mistura de resina e esperar por 13 min. remover a solução da resina. Repita a etapa 1.2.3.1.
- Lavar com 3000 µ l de DMF. Lave duas vezes com 1400 µ l de DMF. Lave duas vezes com 2000 µ l de DMF.
- Repita as etapas 1.2.2 e 1.2.3, de acordo com o número de aminoácidos na sequência do peptide.
- Lavagem de clivagem e resina Fmoc final
- Adicionar 20% piperidina em DMF (1000 µ l) para a resina e espere por 6 min. remover a solução da resina. Repita a etapa 1.2.4.1.
- Lave duas vezes com DMF (1 µ lst 4000, 2 µ lnd 1400) e lave a resina com ar. Lave duas vezes com 2000 µ l de DMF, 4 vezes com 1400 µ l do DCM e lave duas vezes com o ar.
- Preparação da resina
-
Check-up
- Lyophilize a resina da reação colunas durante a noite após a conclusão da síntese.
2. o peptídeo clivagem da resina (Figura 1A)
Nota: Durante o processo de clivagem, todas as cadeias laterais de aminoácidos excepto Cys(Acm) e Cys (tBu) vão ser deprotected. O protocolo aplica-se a 100 mg de resina.
- Combinar a resina congelada em um tubo de 12 ml e resfriá-lo para 0 ° C, no gelo.
- Adicione 150 µ l de uma mistura ao tesouro (preparada a partir de fenol 0,75 g, thioanisole de 0,5 mL, Etanoditiol 0,25 mL) e 1 mL de ácido trifluoroacético (TFA, 95% em água (H2O)) no gelo para a resina. Deixe agitando suavemente para 3h em temperatura ambiente.
- Filtrar a mistura através de uma fritas de vidro e recolher o filtrado em tubos individualmente preenchido com éter dietílico gelado (1 mL da mistura de clivagem por 8-10 mL de éter dietílico). O peptídeo precipita-se como um sólido branco.
- Enxágue a torta de filtro com TFA adicional (95% em H2O, aproximadamente 1-3 mL).
- Fechar os tubos contendo o precipitado e centrifugar (3400 x g) as suspensões por 1 min, decantar o sobrenadante e lavar as pelotas com 8-10 mL de gelado de éter dietílico. Repita este passo 3 vezes.
- Deixe as bolinhas de pé sem tampa por 5 min para remover os restos de éter dietílico. Dissolver as pelotas de produto bruto em 1 mL de terc-butanol (80% em H2O). Congela os peptídeos (-80 ° C).
3. purificação do Precursor Linear com semipreparativa alta cromatografia líquida (HPLC)
Nota: Purificar os peptídeos brutos por semipreparativa fase reversa (RP) HPLC equipada com uma coluna C18 (tamanho de partícula 5 µm, 100 Å tamanho do pore, 250 x 32 mm) e um loop de injeção 3,6 mL. Usar um sistema de gradiente de eluição de 0,1% TFA em H2O (eluente A) e 0,1% TFA em acetonitrilo (MeCN) / h2O (9:1, eluente B). Detectar os picos a 220 nm.
- Adicionar aproximadamente 70 mg do peptide bruto a um tubo de 15 mL e dissolver o peptídeo sólido no volume do loop amostra HPLC (EG., 3,6 mL). Usar uma mistura de 0,1% TFA em MeCN/H2O (1:1). Vórtice até completa dissolução e centrífuga (3400 x g) a solução por 1 min.
- Elaborar a amostra (3,6 mL) em uma seringa de 5 mL e injete a amostra sem quaisquer bolhas de ar para o loop de injeção. Injecte no sistema HPLC. Separe a mistura de peptídeo usando um gradiente de 0-50% eluente B mais de 120 min com um caudal de 10 mL/min.
- Recolha as frações em tubos individuais como eles aparecem. Após a execução completa, prepare frações selecionadas para análise HPLC (etapa 6.1-6.2) e espectrometria de massa (MS). Congela as frações e armazená-los a-20 ° C.
- Após análise de MS e HPLC, combinar as frações puras de peptídeo linear e preparar as amostras para a oxidação de primeira.
4. seletiva formação das ligações de bissulfeto
-
1 st oxidação (Figura 1B)
Nota: Durante a clivagem do péptido da resina, são deprotected o Cys(Trt), levando a dois resíduos de CIS desprotegidos que são posteriormente submetidos à oxidação para formar a primeira ligação dissulfeto. O seguinte protocolo aplica-se a 15 mg de peptídeo purificado linear (2864.5 g/mol; 5.2 µmol; 1 EQ.).- Dissolver o peptídeo linear (15 mg) em 105 mL de um isopropanol/H2O-mistura (1:2; pH 8,5 ajustado com hidróxido de sódio (NaOH) 0,05 mM) e deixar a tremer delicadamente no ar sob condições básicas de 12-48 h.
- Monitore a reação de oxidação via HPLC e MS. Confirm a formação da primeira ponte iodoacetamide (IAA) derivatização (etapa 6).
- Congela o peptídeo e usá-lo sem mais purificação para a oxidação de 2nd .
-
2nd oxidação (Figura 1C)
Nota: Durante a oxidação de segunda, a desproteção das cisteínas Acm-protegidos e a formação da segunda ponte são catalisadas por iodo. O protocolo aplica-se a 15 mg de peptídeo após a 1 oxidação dest (2862.5 g/mol; 5.2 µmol; 1 EQ.)- Dissolva o peptídeo (15 mg; concentração final de 0,05 mM) em 105 mL de um isopropanol/H2M O/1 mistura de ácido clorídrico (HCl) (80:12.5:7.5).
- Adicionar 158 µ l de uma solução de iodo 0,1 M em metanol (MeOH) (15,7 µmol; 3 eq.) para a solução. Misture a reação na temperatura de quarto para 3-52 h, i. e., até que a oxidação é concluída.
- Monitore a reação de oxidação via HPLC e MS. Confirm a formação da segunda ligação dissulfeto de derivatização iodoacetamide (IAA) (etapa 6).
- Pare a reação adicionando 79 µ l de uma solução de ácido ascórbico de 1 M em H2O (µmol 78.8; 15 EQ.). Congela a mistura de reação e usar o pó para a oxidação de 3rd .
-
3rd oxidação (Figura 1D)
Nota: A última oxidação leva a desproteção de cisteínas de Bu-protegido a te a formação da terceira ponte dissulfeto. O protocolo aplica-se a 15 mg de peptídeo após a oxidação 2nd (2718.3 g/mol; 5,5 µmol; 1 EQ.).- Dissolva o peptídeo (15 mg, concentração final de 1 mM) em 5,5 mL de TFA. Contém uma mistura de scavenger consistindo de 11,2 mg de diphenylsulfoxide (55 µmol; 10 EQ.), 60,2 µ l de anisol (0,6 mmol; 100 EQ.) e 97,2 µ l de Triclorometilsilanos (0,8 mmol; 150 EQ.). Agite a mistura por 3-5 h à temperatura ambiente.
- Monitore a reação de oxidação via HPLC e MS. Confirm a formação da terceira ligação dissulfeto iodoacetamide (IAA) derivatização (etapa 6).
- Precipitar o peptídeo nos tubos contendo frio éter dietílico (0 ° C, 1 mL da solução de reação por 8-10 mL de éter dietílico).
- Centrifugar as suspensões (3400 x g, 1 min), decante o sobrenadante e lavar as pelotas repetidamente (4 vezes) com 8-10 mL de éter dietílico frio (0 ° C). Seque as pelotas no ar (3 min).
- Dissolver as bolinhas em 1 mL de 80% terc-butanol (em H2O), congela o peptídeo e armazená-lo a-20 ° C.
5. o peptídeo purificação
Nota: Purificar os peptídeos oxidados por semi preparative RP HPLC equipado com uma coluna C18 (tamanho da partícula de 10 µm, 300 Å tamanho do pore, 250 x 22 mm) e um loop de injeção 3,6 mL. Usar um sistema de gradiente de eluição de 0,1% TFA em H2O (eluente A) e 0,1% TFA em MeCN/H2O (9:1, eluente B). Detectar os picos a 220 nm.
- Adicione 15 mg de liofilizado produto bruto da etapa 4.3.5 para um tubo de 15 mL. Dissolver o produto bruto no volume do loop amostra HPLC (EG., 3,6 mL). Usar uma mistura de 0,1% TFA em MeCN/H2O (1:1). Vórtice até completa dissolução e centrífuga (3400 x g) a solução por 1 min.
- Elaborar a 3,6 mL de uma mistura em uma seringa de 5 mL e injete a amostra sem quaisquer bolhas de ar para o loop de injeção. Inicie a injeção no sistema HPLC. Purifica a mistura de peptídeo usando um gradiente de 0-50% eluente B mais de 120 min com um caudal de 10 mL/min.
- Recolha as frações em tubos individuais como eles aparecem. Após a execução completa, preparação das fracções selecionadas para análise de MS e HPLC (etapa 6.1-6.2). Congelar todas as frações e armazená-los a-20 ° C.
- Após a execução completa, preparação das fracções selecionadas para análise de MS e HPLC (etapa 6.1-6.2). Congelar todas as frações e armazená-los a-20 ° C.
6. o peptídeo Analytics
-
HPLC analítico
Nota: Confirmar a pureza de um peptídeo por analítico RP HPLC equipado com uma coluna C18 (tamanho de partícula 5 µm, 300 Å tamanho do pore, 250 x 4,6 mm) e um loop de injeção de 500 µ l. Usar um sistema de gradiente de eluição de 0,1% TFA em H2O (eluente A) e 0,1% TFA em MeCN (eluente B). Detectar os picos a 220 nm.- Transferir uma amostra das frações peptídicas ou controles de reação em uma HPLC frasco e dissolvem-lo em uma mistura de 0,1% TFA em MeCN/H2O (1:1, 300-500 µ l). Coloque o frasco HPLC para o mostruário do RP HPLC analítico.
- Injete 250 µ l de cada amostra. Usar um sistema de gradiente de eluição de 0,1% TFA em H2O (eluente A) e 0,1% TFA em MeCN (eluente B). Purifica o peptídeo usando um gradiente de 10-40% eluente B mais de 30 min a uma taxa de fluxo de 1,0 mL/min.
-
Espectrometria de massa MALDI TOF
Nota: Confirme a identidade de um peptídeo por espectrometria de massa MALDI TOF (tempo de voo) usando o ácido α- cyano-4-hidroxicinâmicos como matriz.- Transferir uma quantidade visível do peptide em um tubo de microcentrifugadora de 1,5 mL e dissolvê-lo em 10 µ l de uma solução ácida 37mm α- cyano-4-hidroxicinâmicos numa mistura de 0,1% TFA em H2O/MeCN (1:1).
- Vórtice a solução por 10 s, aplicar 2 µ l da amostra a um alvo de aço do chão e a amostra secar ao ar livre.
- Use o modo de refletor para as medições e uma calibração de peptídeo padrão para massas molares abaixo 6000 g/mol.
-
Iodoacetamide de acetila
Nota: Como iodoacetamide reage com grupos tiol, iodoacetamide de derivatização dos peptides indica grupos tiol livre. Portanto, a ausência de grupos tiol livre serve como um controle de reação através de MS durante a oxidação dest 1.- Dissolva o peptídeo em tampão de fosfato 10 mM (0,05 mM, 100 µ l; pH 7,8) em um tubo de microcentrifugadora de 1,5 mL. Adicionar 100 µ l de iodoacetamide em tampão de fosfato 10 mM (4 mM) para a solução de peptídeo e agite suavemente a reação para 2 h à temperatura ambiente no escuro. Congela a mistura de reação e armazená-lo a-20 ° C.
- Use uma ponta de pipeta de filtro C18-concentração e condicionar a ponta com 10 µ l de 80% (3 vezes), 50% (3 vezes), 30% (3 vezes) e 0% (3 vezes) MeCN em H2O (+ 0,1% TFA).
- Dissolver a amostra da etapa 6.3.1 em 1 µ l de 0,1% TFA em H2O e adicionar a solução para a ponta da pipeta de filtro. Pipete cuidadosamente acima e para baixo para que o peptídeo vincula o talão. Remova o H2O fora a ponta da pipeta e lave a ponta da pipeta filtro com 10 µ l de 0.1% TFA em H2O.
- Adicionar 2 µ l de 0,1% TFA em H2O/MeCN (1:1) com a outra ponta da pipeta (sem filtro) em cima do grânulo que contém o peptídeo. Aplicar o filtrado para o destino de chão de aço e a amostra secar ao ar livre.
- Aplica 1 µ l de uma solução de ácido α-cyano-4-hidroxicinâmicos 37 milímetros em uma mistura de 0,1% TFA em H2O/MeCN (1:1) para o aço de terreno alvo com a amostra e a amostra secar ao ar livre.
- Use o modo de refletor para as medições e uma calibração de peptídeo padrão para massas molares abaixo 6000 g/mol.
-
Análise de aminoácidos
Nota: Analise a concentração exata do peptide, bem como a composição de aminoácidos do peptide usando um analisador de aminoácidos.- Transferir 100 µ g do peptide puro (2604 g/mol; 0,04 µmol) para um tubo de microcentrifugadora de 1,5 mL e dissolver o pó em 200 µ l de 6 M de HCl.
- Transferir 200 µ l da solução em uma ampola de vidro e enxaguar o tubo de 1,5 mL microcentrifuga duas vezes com 200 µ l de 6 M de HCl. transferir a solução de lavagem para a ampola de vidro também.
- Feche a ampola aquecendo o pescoço da ampola com uma chama do bico de Bunsen. Coloque a ampola em um tubo de vidro. Coloque-o em um bloco de aquecimento para 24 h a 110 ° C para a hidrólise.
- Abra a ampola e transferir a solução para um tubo de microcentrifugadora de 2 mL. Lave a ampola (3 x 200 µ l) e o tampão (3 x 100 µ l) com bidestilada H2O e transferi-lo para o tubo de microcentrifugadora.
- Centrifugue a solução para 6 h a 60 ° C e 210 x g em um concentrador de vácuo rotacional. Dissolver o produto hidrolisado em 192 µ l de tampão de diluição da amostra (200 µM) e transferir a solução para um filtro microcentrífuga.
- Centrifugar a amostra para 1 min a 2300 x g e transferir 100 µ l do filtrado para um tubo de amostra análise de aminoácidos. Colocar o tubo para o analisador de aminoácidos e iniciar a análise. É utilizado um padrão de aminoácidos para calibração.
7. MS/MS análise de conectividade de bissulfeto
-
Parcial de redução e alquilação17
- Dissolver a 600 µ g do peptide puro (2604 g/mol; 0,23 µmol) em 1,2 mL de tampão de citrato 0,05 M (pH 3.0; concentração de peptídeo de 0,14 mM) que contém 7,5 mg de tris(2-carboxyethyl) fosfina (TCEP; 0,02 M e 0,03 mmol).
- Incubar a mistura à temperatura ambiente e leve reação várias amostras de controle (100 µ l) variando de 0 min a 30 min.
- Misturar as amostras em um tubo de microcentrifugadora de 1,5 mL com 300 µ l de tampão de alquilação (0,5 M tris-acetato; pH 8.0; ácido de 2mm ácido etilenodiaminotetracético (EDTA); 1,1 M iodoacetamide) para parar a reação e executar carbamidomethylation dos grupos tiol livre.
- Parar a reacção após 5 min adicionando-se 100 µ l de 10% TFA (em H2O) e armazenar as amostras em gelo seco. Preparar amostras para HPLC, conforme descrito na etapa 6.1.2 e injetar 400 µ l.(Figura 2)
- Usar um sistema de gradiente de eluição de 0,1% TFA em H2O (eluente A) e 0,1% TFA em MeCN (eluente B). Analisar os peptídeos usando a combinação de uma separação de isocrática (10% eluente B por 15 min) e em seguida um gradiente subsequente de 10-35% eluente B mais 25 min a uma taxa de fluxo de 10 mL/min. detectar os picos a 220 nm.
- Recolher as frações em tubos de 1,5 mL microcentrifuga e congela os peptídeos. (Figura 2B)
- Transferi uma pequena quantidade de cada fração para um tubo de microcentrifugadora de 1,5 mL para análise de MS/MS das formas oxidadas (continue no passo 7.1.12).
- Dissolver o restante do peptide (10-100 µ g) em 0,1% TFA em H2O (10-50 µ l) e adicionar um volume adequado de solução TCEP 100 mM (em H2O) para obter uma concentração final de TCEP de 10 mM.
- Incube a reação de 1 h a 37 ° C (Figura 2C). Congela a mistura de reação e armazená-lo a-20 ° C.
- Prepare amostras de MS/MS, conforme descrito no passo 6.3.2-6.3.5.
- Realizar as medições de MS/MS em um espectrômetro de massa MALDI-TOF/TOF. Use a tampa MS/MS (deterioração induzida por laser) para fragmentação do peptide e selecione as massas precursor das espécies de carbamidomethylated 2 - e 4-tempos. Processar e avaliar os dados MALDI para confirmar a conectividade de bissulfeto desejado.
8. NMR experimentos e análise da estrutura
- Dissolver aproximadamente 0.3-2 mg do produto puro peptídeo em 500 µ l de H2o/d2O (90:10) e transferir a mistura para um microtubo de NMR.
- Preparar a amostra para medições em um espectrômetro de NMR
- Disco 2-dimensional [1H,1H] - DQF - COSY (double espectroscopia de correlação quântica filtrada), [1H,1H] - TOCSY (espectroscopia de correlação total), [1H,1H] - NOESY (nuclear Overhauser realce espectroscopia) e [1H,13C]-espectros HSQC (coerência quântica única de heteronuclear) usando água supressão.
- Atribuir as ressonâncias de prótons dos espectros gravados e criar a atribuição de átomo de espectros NOESY. Compare as intensidades no espectro NOESY para definir as restrições de limite superior distância entre átomos diferentes do peptide. Use a intensidade dos prótons germinais para calibração de pico de intensidade.
- Realizar o cálculo da estrutura e requinte com um programa de computador para a visualização molecular e usar as conetividades identificados bissulfeto do passo 7 como restrições adicionais (Figura 3).
- Selecione as estruturas com as mais baixas energias e usá-lo para simulações de dinâmica molecular (MD).
Resultados
15 diferentes isômeros em ponte dissulfeto de PIIIA o µ-conotoxina são sintetizados e caracterizados em detalhes (Figura 1). Ligações dissulfureto são identificadas pela redução parcial e posterior análise de MS/MS (Figura 2). NMR análise dos diferentes isômeros é executada (Figura 3) para revelar as estruturas individuais do peptide. Notavelmente, uma combinação de análise NMR, fragmentação de MS/MS e RP HPLC é necessária para uma identificação inequívoca da conectividade dissulfeto.

Figura 1 : A formação de ligação dissulfeto seletiva de µ-PIIIA nativo através da estratégia de grupo protegendo. (A) a desproteção das cisteínas Trt-protegido durante a clivagem do péptido da resina. (B) a primeira bissulfeto ponte formação de cisteínas as desprotegido. (C) formação de ponte dissulfeto da Acm e desproteção protegido cisteínas. (D) formação de ponte dissulfeto do tBu e desproteção protegido cisteínas. Clique aqui para ver uma versão maior desta figura.
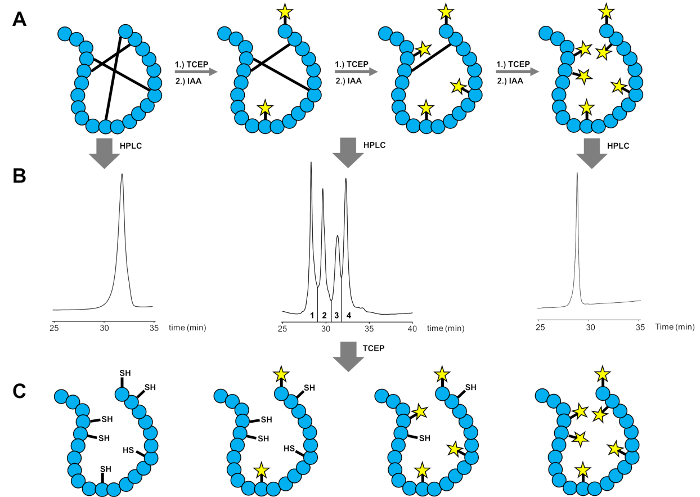
Figura 2 : Redução parcial fluxo de trabalho para a atribuição de ligação dissulfeto por análise de MS/MS. (A) redução parcial e alquilação do peptide. Purificação de HPLC (B) das amostras de controle de reação diferente. (C) redução das amostras purificadas. Parcialmente carbamidomethylated espécies (dois peptídeos no meio) Porto informações sobre a conectividade de dissulfeto que é determinada pela análise de MS/MS. Esta figura foi adaptada com permissão da Heimer, p. et al. Conformacional µ-conotoxina PIIIA isómeros revisitados: o impacto do emparelhamento de cisteína na atribuição de ligação dissulfeto e elucidação da estrutura. Química analítica. 90 (5), 3321-3327 (2018). Direitos autorais (2018) sociedade química americana. Clique aqui para ver uma versão maior desta figura.
Figura 3 : A caminhada sequencial e a estrutura de solução NMR resultante de uma rígida (esquerda) e um isômero flexível (à direita) µ-PIIIa. As 20 estruturas com as energias mais baixas, bem como a conectividade de dissulfeto dos diferentes isômeros são mostradas. A comparação dos valores de raiz quadrada de desvio (RMSD) esclarece que um peptide rígido principalmente conduz a uma melhor estrutura NMR resolvida. Clique aqui para ver uma versão maior desta figura.
Discussão
O método aqui descrito para a síntese de peptídeos ricos em cisteína como µ-PIIIA representa uma possibilidade de produzir seletivamente dissulfeto-ligado de isômeros da mesma sequência de aminoácidos. Portanto, estabeleceu métodos como sólido baseado em Fmoc fase de síntese do peptide18 e uma estratégia de grupo protegendo definida para a formação de regioselective de ligações de bissulfeto foram usados16. A síntese do peptide de fase sólida pode produzir sequências de aminoácidos num suporte de polímero (resina) via síntese automatizado. Estes aminoácidos são protegidos contra reações colaterais indesejáveis durante a montagem da sequência por grupos especiais de proteção em suas cadeias laterais, que são - dependendo do protocolo usado - deprotected após a clivagem do péptido da resina. Essa proteção aplica-se também para as cadeias laterais de cisteína dentro da cadeia peptídica, por exemplo., proteção de trityl. No entanto, protegendo distintos grupos para pares individuais de cisteínas como acetamidomethyl e tert-butílico concomitantemente não são clivados através desta etapa de eliminação. Estes grupos de proteção podem ser clivados fora sob condições que permitam a subsequente oxidação formando dissulfeto dos grupos tiol deprotected. Desta forma, todos os 15 isómeros de bissulfeto de um peptídeo contendo seis resíduos de cisteína podem ser seletivamente formado7,16. O fator limitante, no entanto, é que, com um número crescente de grupos diferentes de proteger ortogonais aumenta o esforço sintético, que tem um alto impacto sobre o rendimento do peptide dissulfeto desejado em ponte. Assim, em casos selecionados e, em especial por menos complexos peptídeos, possuindo apenas uma ou duas ligações de bissulfeto a abordagem Self dobramento oxidativa na verdade pode ser preferencial sobre a estratégia de grupo protegendo.
Neste documento, os grupos do Trt de par uma cisteína são removidos pela ação do TFA após a finalização da montagem do peptide e final Fmoc-desproteção do aminoácido N-terminal. As condições ácidas, no entanto, deixar o Acm e tBu proteger grupos em outros dois pares de resíduos de cisteína intacta. Posteriormente, purificação do peptide linear contendo duas cisteínas desprotegidas é executada usando HPLC preparativa sob condições ligeiramente ácidas para manter os grupos tiol na forma protonada. O protocolo continua com a primeira etapa de oxidação após análise analítico de HPLC e MS verificado síntese bem sucedida e desproteção do Trt com o par de cisteína de interesse. As etapas adicionais de desproteção e oxidação da ligação dissulfeto de segunda e terceira são realizados e confirmados da mesma forma, usando o protocolo apropriado para Acm e tBu, respectivamente. Essas reações de oxidação são, portanto, reações molhado-químicos simples realizadas em solução que não requerem reagentes caros. A desvantagem, no entanto, é que certas reações, i. e., conexões de resíduos de cisteína distintas, não avance suavemente e completamente levando a subprodutos de mexidos conectividade de dissulfeto. Desde que estas não são removidas após a conclusão das reações oxidação individuais, esses subprodutos podem se acumular na mistura de produto bruto. Alguns destes podem possuir propriedades físico-químicas semelhantes como o peptídeo real, por exemplo., eluição de HPLC, que aumenta o esforço para a purificação do peptide dobrada corretamente. Embora a síntese e purificação podem ser difícil, como é o caso, para µ-PIIIA, esse método pode ser utilizado com sucesso, ainda requer boas habilidades de observação e manual. Finalmente, ele precisa ser considerado que cada sequência do peptide é diferente e, portanto, pode ser tratada de forma diferente para ser bem sucedido na geração a conectividade de bissulfeto correto.
Além da síntese desafiador, é essencial verificar se as ligações de bissulfeto produzidas são corretas, i. e., representam a versão pretendida do isômero respectivos dissulfeto. Isto é realizado neste documento, usando uma combinação de análise MALDI MS/MS e elucidação de estrutura NMR. Análise de MS/MS é realizada usando diferentes parcialmente reduzidos e alquilados derivados (carbamidomethylation) do peptídeo totalmente oxidado17. Na tampa de MS/MS MALDI TOF/TOF espectros de que um número par de carbamidomethylated cisteínas é sempre encontraram, i.e., carbamidomethylated 2, 4 ou 6 vezes. Isto pode ser explicado pela redução gradual dos peptides completamente oxidados, desde que em cada ocasião duas funções tiol por ligação dissulfeto são sempre reduzidas (aberto) e em situ alquilados usando iodoacetamide. Este carbamidomethylation de duas cisteínas cada um pode ser detectada no espectro MALDI MS/MS e, por sua vez, refere-se a ligação de dissulfeto respectivos que essas cisteínas estavam envolvidas no peptídeo intacto. Por exemplo, a ocorrência de quatro cisteínas carbamidomethylated em uma sequência com três dissulfeto títulos ajuda a identificar um vínculo específico, ou seja aquele formado entre as dois não-alquilados cisteínas, que não foram abertas durante o tempo de reação a redução parcial suficiente para abrir os outros dois títulos. Assim, pela análise de MALDI MS/MS dos vários derivados de carbamidomethylated 2 - e 4-tempos do isômero de bissulfeto do peptide, as conetividades dissulfeto podem ser completamente elucidado e confirmado.
Outra possibilidade para elucidar o padrão de ligação dissulfeto é a análise de MS/MS de peptídeos digeridos enzimaticamente, onde o peptídeo tem que ser clivada proteoliticamente em aminoácidos distintos para produzir fragmentos menores. Estes fragmentos curtos ainda podem ser ligados através de ligações de bissulfeto, que é a razão que o padrão de dissulfeto pode ser elucidado de MS/MS análise desses fragmentos vinculado. Em caso de µ-PIIIA, no entanto, esta estratégia não pôde ser aplicada porque alguns cisteínas da sequência são diretamente adjacentes uns aos outros e, portanto, a digestão dos peptides não separa as cisteínas uns dos outros. Identificação da ponte dissulfeto específico, portanto, é difícil.
A elucidação da estrutura pela análise de NMR 2D claramente é facilitada no caso de uma conectividade de bissulfeto conhecido, porque esse conhecimento permite a atribuição de efeito de Overhauser nuclear (do NOE) de prótons específicos, isto é., referindo-se ao espacial distância entre dois átomos, determinada a partir as intensidades dos sinais de NOESY (a experiência NMR através do espaço). A análise, no entanto, começa com o pé sequencial19 aplicado para os espectros COSY, TOCSY e NOESY, desta forma, é possível atribuir um sinal específico para o H-átomo correspondente dos aminoácidos (sistema de rotação) na sequência. As restrições de distância acima do experimento NOESY são utilizadas para o cálculo da estrutura do peptídeo7. Os sinais mais identificados, quanto mais precisa a estrutura será. No entanto, a dificuldade desta missão aumenta com o número de aminoácidos do peptide e com a ocorrência de cisteínas adjacentes, à medida que aumenta a probabilidade de que os sinais de vários átomos se sobrepõem e podem já não ser precisamente atribuídos devidos ao fechar proximidade. Além disso, a flexibilidade da cadeia peptídica é decisiva para se sinais no NMR são facilmente ou dificilmente identificáveis. Quanto mais flexível uma região de peptídeo é, as mudanças conformacionais mais estão ocorrendo, permitindo que múltiplos sinais obtidos para um mesmo átomo. Assim, a intensidade diminui com o número de conformações, o que faz com que os sinais de desaparecer no ruído de fundo. Isto significa que a elucidação da estrutura tridimensional através de NMR se torna muito mais fácil se a sequência conformationally é restrito.
Finalmente, este protocolo torna possível gerar vários peptídeos em ponte dissulfeto, monitorando a formação de ligação dissulfeto por análise de MALDI MS/MS e concomitante 2D NMR estrutura elucidação7.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Gostaríamos de agradecer a r. Resemann, F. J. Mayer e D. Suckau da Bruker Daltonics GmbH Bremen; D. Tietze, r. r. Tietze, Schmidts V. e C. Thiele da Universidade de tecnologia de Darmstadt; O. Ohlenschläger o Jena FLI, M. Engeser, da Universidade de Bona; K. Kramer, r. Harzen e H. Nakagami, do Instituto Max Planck para pesquisa de reprodução da planta, Colónia; Susanne Neupert do Instituto de Zoologia, Colónia; e as instalações de espectroscopia de ressonância magnética Biomolecular da Universidade de Frankfurt para técnico suporte, módulos, de formação e acesso aos instrumentos. Apoio financeiro da Universidade de Bonn ao D.I. com gratidão é reconhecido.
Materiais
| Name | Company | Catalog Number | Comments |
| PS PEG2000 Fmoc Rink-amide Resin | Varian, Inc. | PL3867-3764 | AmphiSpheres 40 RAM |
| Pyr(Boc) | Bachem | A-3850 | |
| Arg(Pbf) | Iris Biotech | FSC1010 | |
| Asn(Trt) | Bachem | B-1785 | |
| Asp(tBu) | Iris Biotech | FSP1020 | |
| Hyp(tBu) | Iris Biotech | FAA1627 | |
| Lys(Boc) | Bachem | B-1080 | |
| Ser(tBu) | Iris Biotech | FSC1190 | |
| Gln(Trt) | Iris Biotech | FSC1043 | |
| Glu(tBu) | Iris Biotech | FSP1045 | |
| Trp(Boc) | Iris Biotech | FSC1225 | |
| Tyr(tBu) | Sigma Aldrich | 47623 | |
| Thr(tBu) | Iris Biotech | FSP1210 | |
| His(Trt) | Iris Biotech | FDP1200 | |
| 2-(1H-Benzotriazol-1-yl)-1,1,3,3-tetramethyluroniumhexafluorphosphat | Sigma Aldrich | 8510060 | Flammable |
| DMF | Fisher Scientific | D119 | Flammable, Toxic |
| DCM | Fisher Scientific | D37 | Carcinogenic |
| Piperidine | Alfa Aesar | A12442 | Flammable, Toxic, Corrosive |
| N-Methyl-Morpholin | Sigma Aldrich | 224286 | |
| Cys(Acm) | Iris Biotech | FAA1506 | |
| Cys(Trt) | Bachem | E-2495 | |
| Cys(tBu) | Bachem | B-1220 | |
| trifluoruacetic acid | Sigma Aldrich | 74564 | Toxic, Corrosive |
| phenol | Merck | 1002060 | Toxic |
| thioanisol | Alfa Aesar | A14846 | |
| ethanedithiol | Fluka Analytical | 2390 | |
| diethyl ether | VWR | 100,921 | Flammable |
| tert-butanol | Alfa Aesar | L12338 | Flammable |
| acetonitrile | Fisher Scientific | A998 | Flammable |
| water | Fisher Scientific | W5 | |
| isopropanol | VWR | ACRO42383 | Flammable |
| sodium hydroxide | AppliChem | A6579,1000 | Corrosive |
| iodoacetamide | Sigma Aldrich | I6125 | |
| iodine | Sigma Aldrich | I0385 | |
| Hydrochloric acid | Merck | 110165 | Corrosive |
| ascorbic acid | Sigma Aldrich | A4403 | |
| diphenylsulfoxide | Sigma Aldrich | P35405 | |
| anisol | Sigma Aldrich | 96109 | Flammable |
| trichloromethylsilane | Sigma Aldrich | M85301 | Flammable |
| sample dilution buffer | Laborservice Onken | ||
| sodium dihydrogen phosphate | Sigma Aldrich | 106370 | |
| disodium hydrogen phosphate | Sigma Aldrich | 795410 | |
| (2-carboxyethyl)phosphine hydrochloride | Sigma Aldrich | C4706 | |
| citric acid | Sigma Aldrich | 251275 | |
| sodium citrate dihydrate | Sigma Aldrich | W302600 | |
| tris-acetate | Carl Roth, | 7125 | |
| Ethylenediaminetetraacetic acid | Sigma Aldrich | E26282 | |
| peptide calibration standard II | Bruker Daltonics GmbH | 8222570 | |
| Name of Equipment | Company | ||
| solid-phase peptide synthesizer | Intavis Bioanalytical Instruments AG | EPS 221 | |
| lyophilizer | Martin Christ GmbH | Alpha 1-2 Ldplus | |
| semipreparative HPLC | Jasco | system PV-987 | |
| Eurospher 100 C18 column (RP, 5 µm particle size, 100 Å pore size, 250 x 32 mm) | Knauer | 25QE181E2J | purification of the linear peptide |
| Vydac 218TP1022 column (RP C18, 10 µm particle size, 300 Å pore size, 250 x 22 mm) | Hichrom-VWR | HICH218TP1022 | purification of the oxidized peptide |
| analytical HPLC | Shimadzu | system LC-20AD | |
| Vydac 218TP54 column (C18 RP, 5 µm particle size, 300 Å pore size, 250 x 4.6 mm) | Hichrom-VWR | HICH218TP54 | analytical column |
| ground steel target (MTP 384) | Bruker Daltonics GmbH | NC0910436 | MALDI preparation |
| C18-concentration filter (ZipTip) | Merck KGaA | ZTC18S096 | MALDI preparation |
| MALDI mass spectrometer | Bruker Daltonics GmbH | ultraflex III TOF/TOF | |
| amino acid analyzer | Eppendorf-Biotronik GmbH | LC 3000 system | |
| NMR spectrometer Bruker Avance III | Bruker Daltonics GmbH | Bruker Avance III 600 MHz | |
| computer program for molecular visualising | YASARA Biosciences GmbH | Yasara structures | NMR structure calculation |
| computer program for MALDI data evaluation | Bruker Daltonics GmbH | flexAnalysis, BioTools | MS/MS fragmentation |
| analog vortex mixer | VWR | VM 3000 | |
| Microcentrifuge | Eppendorf | 5410 | |
| Centrifuge | Hettich | EBA 20 | |
| Rotational vacuum concentrator | Christ | 2-18 Cdplus | |
| Analytical Balance | A&D Instruments | GR-202-EC |
Referências
- Fosgerau, K., Hoffmann, T. Peptide therapeutics: Current status and future directions. Drug Discovery Today. 20 (1), 122-128 (2015).
- Gongora-Benítez, M., Tulla-Puche, J., Albericio, F. Multifaceted roles of disulfide bonds. peptides as therapeutics. Chemical Reviews. 114 (2), 901-926 (2014).
- Tietze, A. A., et al. Structurally diverse µ-conotoxin PIIIA isomers block sodium channel NaV1.4. Angewandte Chemie - International Edition. 51 (17), 4058-4061 (2012).
- Carstens, B. B., et al. Structure-Activity Studies of Cysteine-Rich α-Conotoxins that Inhibit High-Voltage-Activated Calcium Channels via GABABReceptor Activation Reveal a Minimal Functional Motif. Angewandte Chemie - International Edition. 55 (15), 4692-4696 (2016).
- Zhang, Y., Schulten, K., Gruebele, M., Bansal, P. S., Wilson, D., Daly, N. L. Disulfide bridges: Bringing together frustrated structure in a bioactive peptide. Biophysical Journal. 110 (8), 1744-1752 (2016).
- Nielsen, K. J., et al. Solution structure of µ-conotoxin PIIIA, a preferential inhibitor of persistent tetrodotoxin-sensitive sodium channels. Journal of Biological Chemistry. 277 (30), 27247-27255 (2002).
- Heimer, P., et al. Conformational µ-Conotoxin PIIIA Isomers Revisited: Impact of Cysteine Pairing on Disulfide-Bond Assignment and Structure Elucidation. Analytical Chemistry. 90 (5), 3321-3327 (2018).
- Chang, J. Y. Diverse pathways of oxidative folding of disulfide proteins: Underlying causes and folding models. Biochemistry. 50 (17), 3414-3431 (2011).
- Böhm, M., et al. Novel insights into structure and function of factor XIIIa-inhibitor tridegin. Journal of Medicinal Chemistry. 57 (24), 10355-10365 (2014).
- Postma, T. M., Albericio, F. Disulfide Formation Strategies in Peptide Synthesis. European Journal of Organic Chemistry. 2014 (17), 3519-3530 (2014).
- Albericio, F., Isidro-llobet, A., Mercedes, A. Amino Acid-Protecting Groups. Chemical Reviews. 109 (6), 2455-2504 (2009).
- Annis, I., Hargittai, B., Barany, G. Disulfide bond formation in peptides. Methods in Enzymology. 289 (1988), 198-221 (1997).
- Kamber, B., et al. The Synthesis of Cystine Peptides by Iodine Oxidation of S-Trityl-cysteine and S-Acetamidomethyl-cysteine Peptides. Helvetica Chimica Acta. 63 (4), 899-915 (1980).
- Bosch, D. E., Zielinski, T., Lowery, R. G., Siderovski, D. P. Evaluating Modulators of Regulator of G-Protein Signaling (RGS) Proteins. Current Protocols in Pharmacology. 56 (2.8), 1-15 (2012).
- Mochizuki, M., Tsuda, S., Tanimura, K., Nishiuchi, Y. Regioselective formation of multiple disulfide bonds with the aid of postsynthetic S-tritylation. Organic Letters. 17 (9), 2202-2205 (2015).
- Peigneur, S., et al. δ-conotoxins synthesized using an acid-cleavable solubility tag approach reveal key structural determinants for NaV subtype selectivity. Journal of Biological Chemistry. 289 (51), 35341-35350 (2014).
- Heimer, P., et al. Application of Room-Temperature Aprotic and Protic Ionic Liquids for Oxidative Folding of Cysteine-Rich Peptides. ChemBioChem. 15 (18), 2754-2765 (2014).
- Kates, S. A., Albericio, F. Solid-Phase Synthesis: A Practical Guide. , Marcel Dekker Inc. New York. (2000).
- Wüthrich, K. NMR studies of structure and function of biological macromolecules (Nobel lecture). Angewandte Chemie - International Edition. 42 (29), 3340-3363 (2003).
Erratum
Formal Correction: Erratum: Synthesis and Structure Determination of µ-Conotoxin PIIIA Isomers with Different Disulfide Connectivities
Posted by JoVE Editors on 11/01/2018. Citeable Link.
An erratum was issued for: Synthesis and Structure Determination of µ-Conotoxin PIIIA Isomers with Different Disulfide Connectivities. The Protocol and Materials sections were updated.
Step 1.1.3 in the Protocol section was updated from:
Weigh the individual reagents (amino acids, HBTU (2-(1H-benzotriazol-1-yl)-1,1,3,3-tetramethyluronium hexafluorophosphate)) according to the protocol and dissolve them in dimethylformamide (DMF) to a final concentration of 2.4 M (amino acids) and 0.6 M (HBTU), respectively.
to:
Weigh the individual reagents (amino acids, HBTU (2-(1H-benzotriazol-1-yl)-1,1,3,3-tetramethyluronium hexafluorophosphate)) according to the protocol and dissolve them in dimethylformamide (DMF) to a final concentration of 0.6 M (amino acids) and 0.6 M (HBTU), respectively.
Step 1.2 in the Protocol section was updated from:
NOTE: The protocol applies to 100 mg of resin (loading: 0.28 mmol/g) added to one reaction column for 28 μmol scale.
to:
NOTE: The standardized protocol usually applies to 100 mg of resin (loading: 0.53 mmol/g) added to one reaction column for 53 μmol scale leading to the following equivalents: 5 eq. HBTU, 10 eq. NMM, 5 eq. amino acid. In case of PIIIA, however, a loading of 0.28 mmol/g (28 µmol scale) is used, which results in the specified higher equivalents.
Step 1.2.3.1 in the Protocol section was updated from:
HBTU (450 µL; 0.6 M in DMF; 270 µmol; 9.6 eq.), NMM (125 µL; 50% in DMF; 568 µmol; 20 eq.), Fmoc-amino acid (420 µL; 2.4 M in DMF; 1.01 mmol; 36 eq.).
to:
HBTU (415 µL; 0.6 M in DMF; 249 µmol; 9 eq.), NMM (112 µL; 50% in DMF; 510 µmol; 18 eq.), Fmoc-amino acid (420 µL; 0.6 M in DMF; 252 µmol; 9 eq.).
The first item in the Materials table was updated from:
| Fmoc Rink amide resin | Novabiochem | 855001 |
to:
| PS PEG2000 Fmoc Rink-amide Resin | Varian, Inc. | PL3867-3764 | AmphiSpheres 40 RAM |
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados
Usamos cookies para melhorar sua experiência em nosso site.
Ao continuar usando nosso site ou clicando em 'continuar', você concorda em aceitar nossos cookies.