Method Article
Synthèse et détermination de Structure des isomères PIIIA µ-conotoxine avec disulfure différentes connectivités
* Ces auteurs ont contribué à parts égales
Dans cet article
Erratum Notice
Résumé
Peptides riches en cystéine incorporer des structures en trois dimensions distinctes selon leur connectivité disulfure. Synthèse ciblée des isomères individuels disulfure est nécessaire lors de l’oxydation de la mémoire tampon ne conduit pas à la connectivité de disulfure désiré. Le protocole porte sur la synthèse sélective des peptides 3-disulfure-collé et leur analyse structurale à l’aide d’études RMN et SM/SM.
Résumé
Peptides avec un nombre élevé de cystéines sont habituellement influencés au sujet de la structure tridimensionnelle par leur connectivité disulfure. Il est donc très important d’éviter la formation de la liaison disulfure indésirables au cours de la synthèse peptidique, car elle peut entraîner dans une structure complètement différente de peptide et par conséquent modifié bioactivité. Toutefois, la formation correcte de multiples liaisons disulfide dans un peptide est difficile à obtenir en utilisant des méthodes pliantes automatique standards tels que les protocoles de tampons classiques d’oxydation, parce que plusieurs des connectivités disulfure peuvent se former. Ce protocole représente une stratégie avancée nécessaire pour la synthèse ciblée de plusieurs peptides avec un pont disulfure qui ne peuvent pas être synthétisés par l’oxydation de la mémoire tampon en qualité et en quantité. L’étude montre l’application d’une stratégie de groupe protecteur distincts pour la synthèse de tous les isomères possibles peptide 3-liaisons disulfure de µ-conotoxine PIIIA de manière ciblée. Les peptides sont préparées par synthèse de peptide de phase solide Fmoc-basé en utilisant une stratégie de groupe protecteur pour la formation de la liaison disulfure définis. Les paires respectives des cystéines sont protégés avec trityl (Trt), acétamidométhyle (Acm) et tert-butyle (t-Bu) protégeant les groupes pour s’assurer qu’au cours de chaque étape de l’oxydation seulement les cystéines requis sont déprotégés et liés. Outre la synthèse ciblée, une combinaison de plusieurs méthodes d’analyse sert à clarifier le pliage correct et la génération des structures peptidiques désirée. La comparaison entre les différents isomères de liaison disulfure-3 indique l’importance de déterminer avec précision et la connaissance de la connectivité de disulfure pour le calcul de la structure tridimensionnelle et interprétation de la Commission activité des isomères peptide. La caractérisation analytique comprend l’élucidation de liaison disulfure exacte via l’analyse de spectrométrie de masse (MS/MS) en tandem qui s’effectue avec des dérivés alkylés et partiellement réduits de l’isomère de peptide intact produites par un protocole adapté. En outre, les structures de peptide sont déterminées à l’aide d’expériences 2D par résonance magnétique nucléaire (RMN) et les connaissances obtenues par l’analyse MS/MS.
Introduction
L’utilisation de peptides bioactifs dans la recherche pharmaceutique et développement est hautement reconnue, parce qu’ils représentent les composés puissants et hautement sélectifs pour des cibles biologiques spécifiques1. Pour leur bioactivité, cependant, la structure tridimensionnelle est d’une grande importance pour réaliser la structure-activité relation études2,3,4. En dehors de la séquence primaire des acides aminés qui influe sur la conformation générale, liaisons disulfide stabilisent sensiblement la structure de peptides riches en cystéine5. Plusieurs peptides avec un pont disulfure incluent conotoxines comme µ-PIIIA de Conus purpurascens qui contient six cystéines dans sa séquence. Cette teneur élevée de cystéine permet théoriquement la formation des 15 disulfure isomères. La connectivité de disulfure correct est très importante pour l’activité biologique6,7. Toutefois, la question qui se pose est si il n’y a plus d’une conformation bioactive peptides naturels et si oui, lequel de ces isomères possède la plus forte activité biologique ? Dans le cas de µ-conotoxines, les cibles biologiques sont des canaux ioniques de sodium voltage-dépendants, et µ-PIIIA en particulier est plus puissant pour les sous-types NaV1.2, NaV1.4 et NaV1.73.
La synthèse des peptides avec un pont disulfure est possible en utilisant différentes méthodes. La méthode la plus pratique pour la formation de ponts disulfures dans un peptide est l’approche dite de pliage automatique oxydatif. Ici, le linéaire précurseur du peptide cyclique désiré est synthétisé tout d’abord à l’aide de synthèse de peptide de phase solide, qui est après que le clivage de l’appui de polymère soumis à l’oxydation dans un système de tampon. Agents actifs en oxydoréduction comme le glutathion réduite et oxydée (GSH/GSSG) sont souvent ajoutés pour promouvoir la formation des liaisons disulfide. Le principal inconvénient du tampon-soutenu pliage automatique, c’est que les ponts disulfures ne forment pas sélectivement de façon progressive. Par rapport au peptide natif, pour lesquels, isomère disulfure spécifiques qu’un seul est souvent décrite, il est possible d’obtenir de nombreux autres isomères avec cette approche8. µ-PIIIA a déjà été montré se traduire par au moins trois isomères différemment pliés à pliage automatique dans une précédente étude3. La séparation d’un tel mélange d’isomère est assez difficile en raison des temps de rétention similaires si l’utilisation de méthodes de purification chromatographique9. La synthèse ciblée d’un isomère est donc avantageuse. Plus précisément les produits qu'un isomère avec connectivité disulfure définie, une stratégie spéciale est requise à laquelle adhère le disulfure sont successivement fermées. Par conséquent, le précurseur linéaire transportant des groupes protecteurs distinctes aux paires individuelles de cystéine est synthétisé sur le soutien de polymère. Après l’élimination, les paires de cystéine sont individuellement et successivement déprotégé et liée à une réaction d’oxydation pour donner le disulfure désiré obligations10,11,12,13, 14 , 15 , 16. après la synthèse et la purification du produit de la réaction, il est nécessaire de confirmer l’identité et la connectivité du disulfure de méthodes analytiques appropriées. Il existe plusieurs méthodes d’analyse pour l’élucidation de la séquence primaire des acides aminés, par exemple., MS/MS, alors que la détermination de la connectivité de disulfure reste encore bien moins étudiée. En dehors de la complexité de ces multiples liaisons disulfure de peptides, impuretés issues du produit (p. ex.., du disulfure de brouillage), en raison de l’échantillon préparation et marche à suivre peuvent compliquer l’analyse. Dans cet article, nous montrons que l’utilisation d’une combinaison de différentes techniques d’analyse est nécessaire de préciser clairement l’identité des liaisons disulfide dans les isomères de µ-PIIIA. Nous avons combiné des méthodes chromatographiques avec la spectrométrie de masse et fourni les mêmes échantillons de spectroscopie RMN. En laser assistée par matrice desorption/ionization(MALDI) analyse MS/MS, nous avons identifié les ponts disulfures en utilisant la réduction partielle et dérivatisation iodoacétamide car analyse descendante n’est pas possible pour ce peptide. Des expériences RMN 2D ont été réalisées afin d’obtenir une structure tridimensionnelle de chaque isomère. Ainsi, en combinant différentes méthodes analytiques sophistiqués, il est possible d’élucider correctement le disulfure connectivité et la structure tridimensionnelle du complexe de plusieurs peptides disulfure-collé7.
Protocole
Remarque : Tous les acides aminés utilisés dans les présentes étaient dans la configuration L. Les abréviations des acides aminés et les acides aminés dérivés ont été utilisées conformément aux recommandations de la Commission de la Nomenclature de IUB et la Commission mixte de l’IUPAC-IUB sur la Nomenclature biochimique.
1. Phase solide de Peptide synthèse (SPPS)
Remarque : Réaliser la synthèse avec un synthétiseur de peptide de phase solide. Effectuer la synthèse des précurseurs peptide linéaire de la séquence générale ZRLCCGFOKSCRSRQCKOHRCC-NH2 en utilisant un protocole standard de chimie 9-fluorenylmethyloxycarbonyl (Fmoc). Appliquer les aminoacides protégées suivantes : Pyr (Boc (tert-butyloxycarbonyl)), Arg (Pbf (2,2,4,6,7-pentamethyldihydrobenzofuran-5-sulfonyl)), Asn(Trt), Asp(tBu), Hyp(tBu), Lys(Boc), Ser(tBu), Gln(Trt), Glu(tBu), Trp(Boc), Tyr(tBu), Thr(tBu) et son (Trt). Protéger les paires de cystéine avec Trt, Acm ou tBu-groupes selon la connectivité disulfure prévue.
-
Préparation
- Sécher la résine Fmoc Rink-amide (chargement : 0.28 mmol/g) à l’aide d’un lyophilisateur du jour au lendemain.
- Entrez la séquence peptidique souhaitée (code 1-lettres) au programme du synthétiseur. Le programme calcule la quantité requise pour chaque réactif individuel et indique les quantités de solvants.
- Peser les réactifs individuels (acides aminés, HBTU (2-(1H-benzotriazol-1-yl)-1,1,3,3-tetramethyluronium hexafluorophosphate)) selon le protocole et les dissoudre dans le diméthylformamide (DMF) à une concentration finale de 2,4 M (acides aminés) et (de 0,6 M HBTU), respectivement.
- Transférer les différents réactifs (acides aminés, HBTU, N-méthylmorpholine (NMM, 50 % dans le DMF), pipéridine (20 % dans le DMF), DMF, dichlorométhane (DCM)) aux navires correspondants et placez-les dans les raquettes appropriées du synthétiseur peptide en phase solide.
- Ajoute les colonnes de la réaction de 100 mg de résine sèche et mettez-les dans la raquette du synthétiseur. Commencer la synthèse de peptide de phase solide.
-
RCR-protocole (fourni par le synthétiseur)
Remarque : Le protocole s’applique à 100 mg de résine (chargement : 0.28 mmol/g) ajouté dans une réaction colonne pour 28 µmol échelle.- Préparation de la résine
- Rincer la résine avec 2500 µL de DMF, 1400 µL de DCM et 1400 µL du DMF.
- Rincer la résine avec de l’air jusqu'à l’élimination du solvant et rincer la résine avec 2500 µL du DMF.
- Clivage de le Fmoc groupe protecteur
- Ajouter 20 % pipéridine dans le DMF (1000 µL) à la résine et attendez de 6 min. enlever la solution de la résine. Répétez l’étape 1.2.2.1.
- Rincer deux fois avec DMF (1 µL dest 4000, 2nd1400 µL), rincez la résine avec de l’air jusqu'à l’élimination du solvant et rincer deux fois avec 2000 µL du DMF.
- Réaction de couplage
- Mélanger les réactifs suivants dans une cuvette séparée : HBTU (450 µL ; 0,6 M dans le DMF ; 270 µmol ; 9,6 EQ.), NMM (125 µL ; 50 % dans le DMF ; 568 µmol ; 20 EQ.), Fmoc-aminoacide (420 µL ; 2,4 M dans le DMF ; 1,01 mmol ; 36 EQ.). Ajouter le mélange à la résine et attendre 13 min. enlever la solution de la résine. Répétez l’étape 1.2.3.1.
- Laver avec 3000 µL du DMF. Laver deux fois avec 1400 µL du DMF. Laver deux fois avec 2000 µL du DMF.
- Répétez les étapes 1.2.2 et 1.2.3 selon le nombre d’acides aminés dans la séquence peptidique.
- Lavage de clivage et de résine Fmoc final
- Ajouter 20 % pipéridine dans le DMF (1000 µL) à la résine et attendez de 6 min. enlever la solution de la résine. Répétez l’étape 1.2.4.1.
- Rincer deux fois avec DMF (1 µL dest 4000, 2nd 1400 µL) et rincer la résine avec de l’air. Rincer deux fois avec 2000 µL du DMF, 4 fois avec 1400 µL de DCM et rincer deux fois avec de l’air.
- Préparation de la résine
-
Marche à suivre
- Lyophiliser la résine de la réaction de colonnes pour la nuit après que la synthèse est terminée.
2. le peptide clivage de la résine (Figure 1A)
Remarque : Au cours de la procédure de clivage, tous les acides aminés des chaînes latérales à l’exception de Cys(Acm) et Cys (t-Bu) vont être déprotégés. Le protocole s’applique à 100 mg de résine.
- Combinez la résine lyophilisée dans un tube de 12 ml et refroidir à 0 ° C sur la glace.
- Ajouter 150 µL d’un mélange de charognard (préparé à partir de phénol 0,75 g, 0,5 mL thioanisole, éthanedithiol de 0,25 mL) et 1 mL d’acide trifluoroacétique (TFA, 95 % dans l’eau (H2O)) sur la glace à la résine. Laissez le secouant doucement pendant 3 h à température ambiante.
- Filtrer le mélange à travers une fritte de verre et recueillir le filtrat dans les tubes remplis individuellement avec éther diethylique glacée (1 mL de mélange de clivage par 8-10 mL d’éther diéthylique). Le peptide précipite sous forme de solide blanc.
- Rincez le gâteau de filtration avec supplémentaire TFA (95 % en H2O, environ 1 à 3 mL).
- Fermer les tubes contenant le précipité et centrifuger (3400 x g) les suspensions pendant 1 min, éliminer le surnageant et laver les pellets avec 8 à 10 mL de diéthyléther glacée. Répétez cette opération 3 fois.
- Laissez les pellets debout sans arrêt pendant 5 min pour éliminer les traces restantes de l’éther diéthylique. Dissoudre les granules de brut produit dans 1 mL de tert-butanol (80 % en H2O). Lyophiliser les peptides (-80 ° C).
3. purification du précurseur linéaire avec la chromatographie liquide semi-préparative haute performance (CLHP)
NOTE : Purifier les peptides bruts en semi-préparative phase inverse (RP) HPLC équipé d’une colonne C18 (taille des particules 5 µm, 100 Å taille des pores, 250 x 32 mm) et une boucle d’injection de 3,6 mL. Utiliser un système de gradient d’élution de 0,1 % TFA dans H2O (éluant A) et 0,1 % TFA dans l’acétonitrile (MeCN) / h2O (9:1, éluant B). Détecter les pics à 220 nm.
- Ajouter environ 70 mg de peptide brut dans un tube de 15 mL et dissoudre le peptide solid dans le volume de la boucle d’échantillonnage HPLC (e.g., 3,6 mL). Utilisez un mélange de 0,1 % TFA dans MeCN/H2O (1:1). Vortex jusqu'à complète dissolution et centrifuger (3400 x g) la solution pendant 1 min.
- Tirer vers le haut de l’échantillon (3,6 mL) dans une seringue de 5 mL et injecter l’échantillon sans bulles d’air dans la boucle de l’injection. Injecter dans le système de CLHP. Séparer le mélange de peptides utilisant un gradient de 0-50 % éluant B pendant 120 min à un débit de 10 mL/min.
- Recueillir les fractions dans des tubes individuels tels qu’ils apparaissent. Une fois la course terminée, préparer les fractions sélectionnées pour la spectrométrie de masse (MS) et analyse par CLHP (étape 6.1 et 6.2). Lyophiliser les fractions et les stocker à-20 ° C.
- Après une analyse MS et HPLC, combiner les fractions pures du peptide linéaire et préparer les échantillons pour l’oxydation en premier.
4. Formation sélective des liaisons disulfide
-
1 st oxydation (Figure 1 B)
Remarque : Durant le clivage du peptide de la résine, les Cys(Trt) sont déprotégé, menant à deux résidus Cys non protégés qui sont ensuite soumis à l’oxydation pour former le premier pont disulfure. Le protocole suivant s’applique à 15 mg de peptide purifié linéaire (2864.5 g/mol ; 5,2 µmol ; 1 éq.).- Dissoudre le peptide linéaire (15 mg) en 105 mL de l’isopropanol/H2O-mélange (1:2 ; pH 8,5 ajusté avec l’hydroxyde de sodium (NaOH), 0,05 mM) et laisser le secouant doucement dans l’air dans des conditions basiques pendant 12 à 48 heures.
- Surveiller la réaction d’oxydation par HPLC et MS. confirmer la formation du premier pont de dérivatisation iodoacétamide (IAA) (étape 6).
- Freeze-Dry le peptide et l’utiliser sans davantage de purification pour l’oxydation du 2nd .
-
2nd oxydation (Figure 1C)
NOTE : Lors de l’oxydation du deuxième, la déprotection des cystéines protégé par Acm et la formation du deuxième pont sont catalysées par l’iode. Le protocole s’applique à 15 mg de peptide après la 1st oxydation (2862.5 g/mol ; 5,2 µmol ; 1 éq.)- Dissoudre le peptide (15 mg, la concentration finale de 0,05 mM) en 105 mL de l’isopropanol/H2M O/1 mélange d’acide chlorhydrique (HCl) (80:12.5:7.5).
- Ajouter 158 µL d’une solution d’iode 0,1 M dans du méthanol (MeOH) (15,7 µmol ; 3 EQ.) à la solution. Remuer la réaction à température ambiante pendant 3-52 h, i.e., jusqu'à ce que l’oxydation est terminée.
- Surveiller la réaction d’oxydation par HPLC et MS. confirmer la formation de la deuxième liaison disulfure de dérivatisation iodoacétamide (IAA) (étape 6).
- Arrêter la réaction en ajoutant 79 µL d’une solution d’acide ascorbique 1 M H2O (78,8 µmol ; 15 EQ.). Freeze-Dry le mélange réactionnel et utiliser la poudre pour l’oxydation de la 3rd .
-
3rd oxydation (Figure 1D)
NOTE : La dernière oxydation conduit à la déprotection des tBu protégé cystéines et à la formation du troisième pont disulfure. Le protocole s’applique à 15 mg de peptide après l’oxydation de 2nd (2718.3 g/mol ; 5,5 µmol ; 1 éq.).- Dissoudre le peptide (15 mg, la concentration finale de 1 mM) à 5,5 mL de TFA. Il contient un mélange de charognard consistant à 11,2 mg de diphénylsulfoxyde (µmol 55 ; 10 EQ.), 60,2 µL d’anisole (0,6 mmol ; 100 EQ) et 97,2 µL de trichloromethylsilane (0,8 mmol ; 150 EQ.). Agiter le mélange pendant 3-5 h à température ambiante.
- Surveiller la réaction d’oxydation par HPLC et MS. confirmer la formation de la troisième liaison disulfure de dérivatisation iodoacétamide (IAA) (étape 6).
- Précipiter le peptide dans les tubes contenant de l’éther diéthylique froid (0 ° C, 1 mL de solution de réaction / 8-10 mL d’éther diéthylique).
- Centrifuger les suspensions (3400 x g, 1 min), éliminer le surnageant et laver les pellets à plusieurs reprises (4 fois) avec 8 à 10 mL d’éther diéthylique froid (0 ° C). Sécher les boulettes sur l’air (3 min).
- Dissoudre les granules dans 1 mL de 80 % de tert-butanol (en H2O), lyophiliser le peptide et conserver à-20 ° C.
5. Purification de peptides
NOTE : Purifier les peptides oxydés en semi-préparative RP HPLC équipé d’une colonne C18 (taille des particules 10 µm, 300 Å taille des pores, 250 x 22 mm) et une boucle d’injection de 3,6 mL. Utiliser un système de gradient d’élution de 0,1 % TFA dans H2O (éluant A) et 0,1 % TFA dans MeCN/H2O (9:1, éluant B). Détecter les pics à 220 nm.
- Ajouter 15 mg du produit brut lyophilisé de l’étape 4.3.5 dans un tube de 15 mL. Dissoudre le brut produit dans le volume de la boucle d’échantillonnage HPLC (e.g., 3,6 mL). Utilisez un mélange de 0,1 % TFA dans MeCN/H2O (1:1). Vortex jusqu'à complète dissolution et centrifuger (3400 x g) la solution pendant 1 min.
- Dresser le mélange de 3,6 mL dans une seringue de 5 mL et injecter l’échantillon sans bulles d’air dans la boucle de l’injection. Commencer l’injection dans le système de CLHP. Purifier le mélange de peptides utilisant un gradient de 0-50 % éluant B pendant 120 min à un débit de 10 mL/min.
- Recueillir les fractions dans des tubes individuels tels qu’ils apparaissent. Une fois la course terminée, préparer les fractions sélectionnées pour analyse MS et HPLC (étape 6.1 et 6.2). Freeze-Dry de toutes les fractions et les stocker à-20 ° C.
- Une fois la course terminée, préparer les fractions sélectionnées pour analyse MS et HPLC (étape 6.1 et 6.2). Freeze-Dry de toutes les fractions et les stocker à-20 ° C.
6. peptide Analytique
-
HPLC analytique
Remarque : Vérifier la pureté d’un peptide par CLHP analytique équipé d’une colonne C18 (taille des particules 5 µm, 300 Å taille des pores, 250 x 4,6 mm) et une boucle d’injection de 500 µL. Utiliser un système de gradient d’élution de 0,1 % TFA dans H2O (éluant A) et 0,1 % TFA dans MeCN (éluant B). Détecter les pics à 220 nm.- Transférer un échantillon des fractions peptidiques ou contrôles de réaction dans une HPLC cuvette et dissolvent dans un mélange de 0,1 % TFA dans MeCN/H2O (1:1, 300-500 µL). Placez le flacon HPLC dans l’échantillonneur automatique de la RP HPLC analytique.
- Injecter 250 µL de chaque échantillon. Utiliser un système de gradient d’élution de 0,1 % TFA dans H2O (éluant A) et 0,1 % TFA dans MeCN (éluant B). Purifier le peptide utilisant un gradient de 10 à 40 % éluant B pendant 30 min à un débit de 1,0 mL/min.
-
Spectrométrie de masse MALDI TOF
NOTE : Confirmer l’identité d’un peptide par spectrométrie de masse MALDI TOF (temps de vol) à l’aide de α- cyano-4-hydroxycinnamique comme matrice.- Transférer un montant visible du peptide dans un tube de microtubes de 1,5 mL et dissoudre dans 10 µL d’une solution acide de 37 mM α- cyano-4-hydroxycinnamique dans un mélange de 0,1 % TFA dans H2O/MeCN (1:1).
- Vortex la solution pendant 10 s, appliquez 2 µL de l’échantillon sur une cible au sol en acier et sécher l’échantillon.
- Utilisez le mode réflecteur pour les mesures et un étalon de peptide pour masses molaires inférieure à 6000 g/mol.
-
Dérivatisation iodoacétamide
NOTE : Comme l’iodoacétamide réagit avec les groupements thiol, iodoacétamide dérivation des peptides indique des groupements thiol libre. Par conséquent, l’absence de groupes thiol libre sert un contrôle de la réaction par l’intermédiaire de MS lors de l’oxydation dest 1.- Dissoudre le peptide dans le tampon de phosphate de 10 mM (pH 7,8, 100 µL ; 0,05 mM) dans un tube de microtubes de 1,5 mL. Ajouter 100 µL de l’iodoacétamide dans un tampon phosphate 10 mM (4 mM) à la solution de peptide et secouez légèrement la réaction pendant 2 h à température ambiante dans l’obscurité. Freeze-Dry le mélange réactionnel et conserver à-20 ° C.
- Utiliser un embout de pipette de filtre C18-concentration et conditionner la pointe 10 µl de 80 % (3 fois), de 50 % (3 fois), de 30 % (3 fois) et de 0 % (3 fois) MeCN en H2O (+ 0,1 % TFA).
- Dissoudre l’échantillon d’étape 6.3.1 dans 1 µL de 0,1 % TFA dans H2O et ajouter la solution à la pointe de pipette de filtre. Pipetter soigneusement de haut en bas afin que le peptide se lie à la perle. Retirez le H2O sur l’embout de la pipette et rincer la pipette de filtre avec 10 µL de 0,1 % TFA dans H2O.
- Ajouter 2 µl de 0,1 % TFA dans H2O/MeCN (1:1) avec un autre embout de la pipette (sans filtre) sur le dessus de la perle qui contient le peptide. Appliquez le filtrat à la cible au sol en acier et sécher l’échantillon.
- Appliquer 1 µL d’une solution d’acide α-cyano-4-hydroxycinnamique 37 mM dans un mélange de 0,1 % TFA dans H2O/MeCN (1:1) à l’acier sol cibler avec l’échantillon et sécher l’échantillon.
- Utilisez le mode réflecteur pour les mesures et un étalon de peptide pour masses molaires inférieure à 6000 g/mol.
-
Analyse des acides aminés
NOTE : Analyser la concentration de peptide exact ainsi que la composition en acides aminés du peptide à l’aide d’un analyseur d’acides aminés.- Transférer 100 µg du peptide pure (2604 g/mol ; 0,04 µmol) à un tube de microtubes de 1,5 mL et dissoudre la poudre dans 200 µL de 6 M HCl.
- Transférer 200 µL de la solution dans une ampoule de verre et rincer le tube de microtubes de 1,5 mL deux fois avec 200 µL de 6 M HCl. transférer la solution de rinçage dans l’ampoule de verre aussi bien.
- Fermer l’ampoule en chauffant le cou de l’ampoule avec une flamme de bec Bunsen. Mettez l’ampoule dans un tube de verre. Placez-le dans un bloc chauffant pendant 24 h à 110 ° C pour l’hydrolyse.
- Ouvrir l’ampoule et transvaser la solution dans un tube de microcentrifuge de 2 mL. Lavez l’ampoule (3 x 200 µL) et le cap (3 x 100 µL) avec bidistillée H2O et transférez-le dans le tube de microcentrifuge.
- Centrifuger la solution pendant 6 h à 60 ° C et 210 x g dans une rotation pompe à vide. Dissoudre le produit hydrolysé en 192 µL du tampon de dilution de l’échantillon (200 µM) et transvaser la solution dans un filtre micro-centrifuge.
- Centrifuger l’échantillon pendant 1 min à 2300 x g et le transfert de 100 µL de filtrat dans un tube à essais analyse des acides aminés. Placer le tube dans l’analyseur d’acides aminés et commencer l’analyse. Une norme de l’acide aminé est utilisée pour l’étalonnage.
7. MS/MS Analysis of Connectivity disulfure
-
Réduction et alkylation partielle17
- Dissoudre 600 µg du peptide pure (2604 g/mol ; 0.23 µmol) à 1,2 mL de tampon citrate de 0,05 M (pH 3,0 ; concentration de peptide de 0,14 mM) contient 7,5 mg de tris(2-carboxyethyl) phosphine (TCEP ; M 0,02 0,03 mmol).
- Incuber le mélange à la température ambiante et prendre plusieurs réaction des échantillons de contrôle (100 µL) allant de 0 min à 30 min.
- Mélanger les échantillons dans un tube de microtubes de 1,5 mL à 300 µL de tampon d’alkylation (tris-acétate de 0,5 M ; pH 8.0 ; l’acide éthylènediaminetétraacétique (EDTA) de 2 mM ; 1,1 M iodoacétamide) pour arrêter la réaction et effectuer les carbamidomethylation des groupes thiol libre.
- Arrêter la réaction après 5 min en ajoutant 100 µL de 10 % TFA (en H2O) et Banque d’échantillons sur la glace sèche. Préparer les échantillons HPLC comme indiqué au point 6.1.2 et injecter 400 µL. (Figure 2A)
- Utiliser un système de gradient d’élution de 0,1 % TFA dans H2O (éluant A) et 0,1 % TFA dans MeCN (éluant B). Analyser les peptides à l’aide de la combinaison d’une séparation isocratique (10 % l’éluant B pendant 15 min) et ensuite un dégradé subséquent de 10 à 35 % éluant B pendant 25 min à un débit de 10 mL/min. détecter les pics à 220 nm.
- Recueillir les fractions en tubes de microcentrifuge de 1,5 mL et lyophiliser les peptides. (Figure 2B)
- Transférer une petite quantité de chaque fraction d’un tube de microtubes de 1,5 mL pour l’analyse MS/MS des formes oxydées (continuez à l’étape 7.1.12).
- Dissoudre le peptide restant (10-100 µg) dans 0,1 % TFA dans H2O (10-50 µL) et ajouter un volume approprié d’un mM 100 PTCE solution (H2O) pour obtenir une concentration finale de PTCE de 10 mM.
- Incubez la réaction pendant 1 h à 37 ° C (Figure 2C). Freeze-Dry le mélange réactionnel et conserver à-20 ° C.
- Préparer les échantillons de MS/MS comme décrit dans l’étape 6.3.2-6.3.5.
- Effectuer les mesures MS/MS sur un spectromètre de masse MALDI-TOF/TOF. Utilisation du couvercle de MS/MS (désintégration induite par laser) pour la fragmentation du peptide et sélectionnez les masses de précurseur des espèces déterminées 2 et 4 fois. Traiter et évaluer les données MALDI pour confirmer la connectivité disulfure désiré.
8. RMN et analyse de la Structure
- Dissoudre environ 0,3-2 mg de produit pur peptide dans 500 µL d’H2o/d2O (90/10) et transvaser le mélange dans un microtube NMR.
- Préparation des échantillons pour des mesures à un spectromètre RMN
- Enregistrement 2D [1H,1H] - DQF - COSY (double spectroscopie de corrélation quantique filtrée), [1H,1H] - TOCSY (spectroscopie corrélation totale), [1H,1H] - NOESY (accentuation des Overhauser nucléaire la spectroscopie) et [1H,13C]-spectres HSQC (hétéronucléaire une cohérence quantique unique) à l’aide de la suppression de l’eau.
- Assigner les résonances des protons des spectres enregistrés et créer l’affectation de l’atome de spectres NOESY. Comparer les intensités dans les spectres NOESY pour définir les contraintes de distance de la limite supérieure entre atomes différents du peptide. Utiliser l’intensité des protons germinales pour l’étalonnage de l’intensité du pic.
- Effectuer le calcul de structure et de raffinement avec un logiciel de visualisation moléculaire et servir les connectivités identifiés disulfure de Step 7 de retenue supplémentaire (Figure 3).
- Sélectionner les structures avec des énergies plus faibles et utilisez-la pour les simulations de dynamique moléculaire (DM).
Résultats
15 différents isomères avec un pont disulfure de la µ-conotoxine PIIIA sont synthétisés et caractérisés en détail (Figure 1). Les liaisons disulfide sont identifiés par une réduction partielle et l’analyse ultérieure de MS/MS (Figure 2). Analyse par RMN des différents isomères est effectuée (Figure 3) pour révéler les structures peptidiques individuels. Notamment, une combinaison de RP HPLC, la fragmentation de la SM/SM et analyse RMN est requise pour une identification sans équivoque de la connectivité de disulfure.

Figure 1 : La formation de la liaison disulfure sélective de µ-PIIIA natif via la stratégie de groupe protecteur. (A) la déprotection des cystéines Trt protégé durant le clivage du peptide de la résine. (B) le disulfure de premier pont de formation des cystéines non protégés. (C) déprotection et formation de ponts disulfure de l’Acm protégé cystéines. (D) la déprotection et formation de ponts disulfure de la tBu protégé cystéines. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Figure 2 : "Workflow" réduction partielle pour l’assignation de liaison disulfure par analyse MS/MS. (A) une réduction partielle et alkylation du peptide. Purification HPLC de (B) des échantillons de contrôle de réaction différents. (C) réduction des échantillons purifiés. Partiellement déterminées (deux peptides au milieu) harbor information sur l’espèce sur la connectivité de disulfure qui est déterminée par l’analyse MS/MS. Ce chiffre a été adapté avec la permission de Heimer, P. et al. µ-Conotoxine conformationnelle PIIIA isomères revisités : Impact de l’appariement de cystéine sur pont disulfure affectation et l’élucidation de la Structure. Chimie analytique. 90 (5), 3321-3327 (2018). Droit d’auteur (2018) American Chemical Society. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
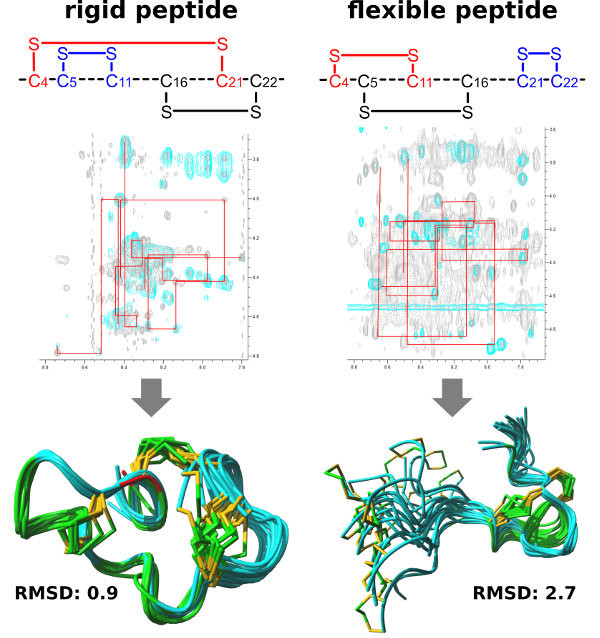
Figure 3 : La marche séquentielle et la structure de solution résultante NMR de rigide (gauche), un isomère de flexible (à droite) µ-PIIIa. Les 20 structures avec les énergies plus faibles ainsi que la connectivité de disulfure des différents isomères sont indiqués. La comparaison des valeurs quadratiques moyennes déviation (RMSD) précise qu’un peptide rigide surtout conduit à une meilleure structure de NMR résolue. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
La méthode décrite ici pour la synthèse des peptides riches en cystéine tels que µ-PIIIA représente une possibilité pour produire sélectivement les isomères de liaison disulfure de la même séquence d’acides aminés. Par conséquent, mis en place des méthodes telles que solide axée sur le Fmoc phase de synthèse de peptide18 et une stratégie de groupe protecteur définie pour la formation régiosélective des ponts disulfures ont utilisé16. La synthèse de peptide de phase solide peut produire des séquences d’acides aminés sur un support de polymère (résine) par l’intermédiaire de synthèse automatisée. Ces acides aminés sont protégés contre les réactions secondaires indésirables lors du montage de la séquence par groupes protecteurs spéciaux à leurs chaînes latérales, qui sont - en fonction du protocole utilisé - déprotégé par clivage du peptide de la résine. Cette protection s’applique également pour les chaînes latérales de cystéine dans la chaîne peptidique, e.g., protection trityle. Cependant, protection distincts groupes pour différentes paires de cystéines tels qu’acétamidométhyle et tert-butyle sont clivés pas en même temps que par le biais de cette étape de l’élimination. Ces groupes protecteurs peuvent être clivés au large dans des conditions qui permettent l’oxydation ultérieure en formant des ponts disulfures des groupes thiol déprotégé. De cette façon, tous les isomères de disulfure 15 d’un peptide contenant six résidus de cystéine peuvent être sélectivement formé7,16. Le facteur limitant, cependant, est qu’avec un nombre croissant de différents groupes protecteurs orthogonaux les augmentations d’effort synthétique, qui a une forte incidence sur le rendement du peptide désiré disulfure ponté. Ainsi, dans certains cas précis et en particulier aux meilleurs prix complexes peptides possédant uniquement un ou deux ponts disulfures l’approche automatique pliant oxydatif en effet peut être préférée au fil de la stratégie de groupe protecteur.
Dans les présentes, les groupes de Trt de paire une cystéine sont éliminés par l’action d’AGT après achèvement de l’Assemblée de peptide et finale Fmoc-déprotection de l’acide aminé N-terminal. Les conditions acides, cependant, laissent l’Acm et tBu groupes protecteurs dans les deux autres paires de résidus cystéine intacte. Par la suite, purification du peptide linéaire contenant deux cystéines non protégés est effectuée par HPLC préparative des conditions légèrement acides à maintenir les groupes thiol dans la forme protonée. Le protocole se poursuit avec la première étape de l’oxydation après que analyse analyse HPLC et MS vérifié la synthèse réussie et la déprotection de la Trt à la paire de cystéine d’intérêt. Les nouvelles mesures de déprotection et oxydation de la liaison disulfure de deuxième et troisième sont effectués et confirmés de la même manière en utilisant le protocole approprié pour Acm et tBu, respectivement. Ces réactions d’oxydation sont donc des réactions chimiques humides simples effectuées dans une solution qui ne nécessitent pas de réactifs coûteux. L’inconvénient, cependant, est que certaines réactions, i.e., connexions distinctes des résidus de cystéine, ne passez pas sans heurts et complètement menant aux sous-produits de brouillés connectivité disulfure. Étant donné que ceux-ci ne sont pas supprimés après les réactions d’oxydation individuels, ces sous-produits peuvent s’accumuler dans le mélange de produit brut. Certaines d'entre elles peuvent posséder des propriétés physico-chimiques similaires comme le peptide réels, par exemple., élution HPLC, ce qui augmente l’effort pour la purification du peptide correctement pliée. Même si la synthèse et la purification peuvent être difficiles, comme c’est le cas pour µ-PIIIA, cette méthode peut être utilisée avec succès, mais exige de bonnes compétences manuel et observation. Enfin, il faut considérer que chaque séquence peptidique est différente et peut donc être traitée différemment pour réussir à générer la connectivité correcte disulfure.
Outre la synthèse difficile, il est essentiel de vérifier si les ponts disulfures produites sont corrects, i.e., représentent la version de l’isomère de disulfure respectifs. Ceci est réalisé dans les présentes en utilisant une combinaison d’analyse MALDI-MS/MS et élucidation de structures NMR. Analyse MS/MS est effectué à l’aide de différents dérivés alkylés et partiellement réduits (carbamidomethylation) du peptide entièrement oxydé17. Dans le couvercle de MS/MS de MALDI TOF/TOF spectres qu'un nombre pair de cystéines déterminées est toujours trouvent, c'est-à-dire, 2, 4 ou 6-fois déterminées. Cela s’explique par la réduction progressive des peptides complètement oxydés, depuis à chaque fois deux fonctions thiols par liaison disulfure sont toujours réduites (ouvert) et in situ alkylés en utilisant iodoacétamide. Cette carbamidomethylation de deux cystéines chacun peut être détectée dans les spectres de MALDI-MS/MS et, à son tour, fait référence à la liaison disulfure respectifs que ces cystéines exerçaient dans le peptide intact. Par exemple, la présence de quatre cystéines déterminées dans une séquence avec le disulfure de trois obligations aide à identifier une liaison spécifique, à savoir celui formé entre les deux cystéines non alkylés, qui n’étaient pas ouvert pendant le temps de réaction de la réduction partielle suffisante pour ouvrir les deux autres liaisons. Ainsi, par l’analyse de MALDI-MS/MS des dérivés divers 2 et 4 fois déterminées de l’isomère de disulfure de peptide, les connectivités disulfure peuvent être complètement élucidée et confirmés.
Une autre possibilité pour élucider le modèle de liaison disulfure est l’analyse MS/MS des peptides enzymatiquement digérées, où le peptide doit être clivage protéolytique clivé à distinctes des acides aminés pour produire de plus petits fragments. Ces courts fragments peuvent toujours être reliés par l’intermédiaire de liaisons disulfide, qui est la raison que le modèle de disulfure peut être élucidé d’analyse MS/MS de ces fragments liés. Dans le cas de µ-PIIIA, cependant, cette stratégie ne pourrait pas être appliquée car certains cystéines de la séquence sont directement adjacents les uns aux autres et donc la digestion des peptides ne sépare pas les cystéines les uns des autres. Identification du pont disulfure spécifiques est donc difficile.
L’élucidation de la structure par des analyses RMN 2D est nettement facilitée dans le cas d’une connectivité de disulfure connus, parce que cette connaissance permet l’attribution de l’effet Overhauser nucléaire (NOE) aux protons spécifiques, c’est à dire., se référant à la spatiale distance entre deux atomes, déterminée à partir de l’intensité des signaux NOESY (expérience de NMR à travers l’espace). L’analyse, cependant, commence par la marche séquentielle19 appliqué aux spectres COSY, TOCSY et NOESY, de cette façon, il est possible d’assigner un signal spécifique à l’atome de H-correspondant des acides aminés (système de spin) dans la séquence. Les contraintes de distance susmentionnés de l’expérience NOESY sont utilisés dans le calcul de la structure du peptide7. Les signaux plus déterminé, plus exacte la structure sera. Toutefois, la difficulté de cette tâche augmente avec le nombre d’acides aminés du peptide et l’occurrence des cystéines adjacentes, comme la probabilité augmente que les signaux de plusieurs atomes se chevauchent et peuvent n’est plus précisément affectés dû fermer proximité. En outre, la flexibilité de la chaîne peptidique est déterminante pour savoir si les signaux de la RMN sont facilement ou difficilement identifiables. Plus souple une région de peptide est, les changements de conformation plus sont produisent, permettant plusieurs signaux à obtenir pour un même atome. Ainsi, l’intensité diminue avec le nombre de conformations, qui provoque des signaux à disparaître en bruit de fond. Cela signifie que l’élucidation de la structure tridimensionnelle par l’intermédiaire de NMR devienne beaucoup plus facile si la séquence est conformationnellement contraints.
Enfin, ce protocole permet de générer plusieurs peptides avec un pont disulfure en contrôlant la formation de la liaison disulfure par analyse de MALDI-MS/MS et concomitante 2D NMR structure elucidation7.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous tenons à remercier A. Resemann, F. J. Mayer et D. de Suckau de Bruker Daltonics GmbH Brême ; D. Tietze, A. A. Tietze, Schmidts V. et C. Thiele de l’Université de technologie de Darmstadt ; O. Ohlenschläger de l’Iéna FLI, M. Engeser de l’Université de Bonn ; K. Kramer, A. Harzen et H. Nakagami, de l’Institut Max Planck pour Plant Breeding Research, Cologne ; Susanne Neupert de l’Institut de zoologie, Cologne ; et les installations biomoléculaire de spectroscopie par résonance magnétique de l’Université de Francfort pour technique prend en charge, modules, de formation et d’accès aux instruments. Soutien financier de l’Université de Bonn à D.I. tient à reconnaître.
matériels
| Name | Company | Catalog Number | Comments |
| PS PEG2000 Fmoc Rink-amide Resin | Varian, Inc. | PL3867-3764 | AmphiSpheres 40 RAM |
| Pyr(Boc) | Bachem | A-3850 | |
| Arg(Pbf) | Iris Biotech | FSC1010 | |
| Asn(Trt) | Bachem | B-1785 | |
| Asp(tBu) | Iris Biotech | FSP1020 | |
| Hyp(tBu) | Iris Biotech | FAA1627 | |
| Lys(Boc) | Bachem | B-1080 | |
| Ser(tBu) | Iris Biotech | FSC1190 | |
| Gln(Trt) | Iris Biotech | FSC1043 | |
| Glu(tBu) | Iris Biotech | FSP1045 | |
| Trp(Boc) | Iris Biotech | FSC1225 | |
| Tyr(tBu) | Sigma Aldrich | 47623 | |
| Thr(tBu) | Iris Biotech | FSP1210 | |
| His(Trt) | Iris Biotech | FDP1200 | |
| 2-(1H-Benzotriazol-1-yl)-1,1,3,3-tetramethyluroniumhexafluorphosphat | Sigma Aldrich | 8510060 | Flammable |
| DMF | Fisher Scientific | D119 | Flammable, Toxic |
| DCM | Fisher Scientific | D37 | Carcinogenic |
| Piperidine | Alfa Aesar | A12442 | Flammable, Toxic, Corrosive |
| N-Methyl-Morpholin | Sigma Aldrich | 224286 | |
| Cys(Acm) | Iris Biotech | FAA1506 | |
| Cys(Trt) | Bachem | E-2495 | |
| Cys(tBu) | Bachem | B-1220 | |
| trifluoruacetic acid | Sigma Aldrich | 74564 | Toxic, Corrosive |
| phenol | Merck | 1002060 | Toxic |
| thioanisol | Alfa Aesar | A14846 | |
| ethanedithiol | Fluka Analytical | 2390 | |
| diethyl ether | VWR | 100,921 | Flammable |
| tert-butanol | Alfa Aesar | L12338 | Flammable |
| acetonitrile | Fisher Scientific | A998 | Flammable |
| water | Fisher Scientific | W5 | |
| isopropanol | VWR | ACRO42383 | Flammable |
| sodium hydroxide | AppliChem | A6579,1000 | Corrosive |
| iodoacetamide | Sigma Aldrich | I6125 | |
| iodine | Sigma Aldrich | I0385 | |
| Hydrochloric acid | Merck | 110165 | Corrosive |
| ascorbic acid | Sigma Aldrich | A4403 | |
| diphenylsulfoxide | Sigma Aldrich | P35405 | |
| anisol | Sigma Aldrich | 96109 | Flammable |
| trichloromethylsilane | Sigma Aldrich | M85301 | Flammable |
| sample dilution buffer | Laborservice Onken | ||
| sodium dihydrogen phosphate | Sigma Aldrich | 106370 | |
| disodium hydrogen phosphate | Sigma Aldrich | 795410 | |
| (2-carboxyethyl)phosphine hydrochloride | Sigma Aldrich | C4706 | |
| citric acid | Sigma Aldrich | 251275 | |
| sodium citrate dihydrate | Sigma Aldrich | W302600 | |
| tris-acetate | Carl Roth, | 7125 | |
| Ethylenediaminetetraacetic acid | Sigma Aldrich | E26282 | |
| peptide calibration standard II | Bruker Daltonics GmbH | 8222570 | |
| Name of Equipment | Company | ||
| solid-phase peptide synthesizer | Intavis Bioanalytical Instruments AG | EPS 221 | |
| lyophilizer | Martin Christ GmbH | Alpha 1-2 Ldplus | |
| semipreparative HPLC | Jasco | system PV-987 | |
| Eurospher 100 C18 column (RP, 5 µm particle size, 100 Å pore size, 250 x 32 mm) | Knauer | 25QE181E2J | purification of the linear peptide |
| Vydac 218TP1022 column (RP C18, 10 µm particle size, 300 Å pore size, 250 x 22 mm) | Hichrom-VWR | HICH218TP1022 | purification of the oxidized peptide |
| analytical HPLC | Shimadzu | system LC-20AD | |
| Vydac 218TP54 column (C18 RP, 5 µm particle size, 300 Å pore size, 250 x 4.6 mm) | Hichrom-VWR | HICH218TP54 | analytical column |
| ground steel target (MTP 384) | Bruker Daltonics GmbH | NC0910436 | MALDI preparation |
| C18-concentration filter (ZipTip) | Merck KGaA | ZTC18S096 | MALDI preparation |
| MALDI mass spectrometer | Bruker Daltonics GmbH | ultraflex III TOF/TOF | |
| amino acid analyzer | Eppendorf-Biotronik GmbH | LC 3000 system | |
| NMR spectrometer Bruker Avance III | Bruker Daltonics GmbH | Bruker Avance III 600 MHz | |
| computer program for molecular visualising | YASARA Biosciences GmbH | Yasara structures | NMR structure calculation |
| computer program for MALDI data evaluation | Bruker Daltonics GmbH | flexAnalysis, BioTools | MS/MS fragmentation |
| analog vortex mixer | VWR | VM 3000 | |
| Microcentrifuge | Eppendorf | 5410 | |
| Centrifuge | Hettich | EBA 20 | |
| Rotational vacuum concentrator | Christ | 2-18 Cdplus | |
| Analytical Balance | A&D Instruments | GR-202-EC |
Références
- Fosgerau, K., Hoffmann, T. Peptide therapeutics: Current status and future directions. Drug Discovery Today. 20 (1), 122-128 (2015).
- Gongora-Benítez, M., Tulla-Puche, J., Albericio, F. Multifaceted roles of disulfide bonds. peptides as therapeutics. Chemical Reviews. 114 (2), 901-926 (2014).
- Tietze, A. A., et al. Structurally diverse µ-conotoxin PIIIA isomers block sodium channel NaV1.4. Angewandte Chemie - International Edition. 51 (17), 4058-4061 (2012).
- Carstens, B. B., et al. Structure-Activity Studies of Cysteine-Rich α-Conotoxins that Inhibit High-Voltage-Activated Calcium Channels via GABABReceptor Activation Reveal a Minimal Functional Motif. Angewandte Chemie - International Edition. 55 (15), 4692-4696 (2016).
- Zhang, Y., Schulten, K., Gruebele, M., Bansal, P. S., Wilson, D., Daly, N. L. Disulfide bridges: Bringing together frustrated structure in a bioactive peptide. Biophysical Journal. 110 (8), 1744-1752 (2016).
- Nielsen, K. J., et al. Solution structure of µ-conotoxin PIIIA, a preferential inhibitor of persistent tetrodotoxin-sensitive sodium channels. Journal of Biological Chemistry. 277 (30), 27247-27255 (2002).
- Heimer, P., et al. Conformational µ-Conotoxin PIIIA Isomers Revisited: Impact of Cysteine Pairing on Disulfide-Bond Assignment and Structure Elucidation. Analytical Chemistry. 90 (5), 3321-3327 (2018).
- Chang, J. Y. Diverse pathways of oxidative folding of disulfide proteins: Underlying causes and folding models. Biochemistry. 50 (17), 3414-3431 (2011).
- Böhm, M., et al. Novel insights into structure and function of factor XIIIa-inhibitor tridegin. Journal of Medicinal Chemistry. 57 (24), 10355-10365 (2014).
- Postma, T. M., Albericio, F. Disulfide Formation Strategies in Peptide Synthesis. European Journal of Organic Chemistry. 2014 (17), 3519-3530 (2014).
- Albericio, F., Isidro-llobet, A., Mercedes, A. Amino Acid-Protecting Groups. Chemical Reviews. 109 (6), 2455-2504 (2009).
- Annis, I., Hargittai, B., Barany, G. Disulfide bond formation in peptides. Methods in Enzymology. 289 (1988), 198-221 (1997).
- Kamber, B., et al. The Synthesis of Cystine Peptides by Iodine Oxidation of S-Trityl-cysteine and S-Acetamidomethyl-cysteine Peptides. Helvetica Chimica Acta. 63 (4), 899-915 (1980).
- Bosch, D. E., Zielinski, T., Lowery, R. G., Siderovski, D. P. Evaluating Modulators of Regulator of G-Protein Signaling (RGS) Proteins. Current Protocols in Pharmacology. 56 (2.8), 1-15 (2012).
- Mochizuki, M., Tsuda, S., Tanimura, K., Nishiuchi, Y. Regioselective formation of multiple disulfide bonds with the aid of postsynthetic S-tritylation. Organic Letters. 17 (9), 2202-2205 (2015).
- Peigneur, S., et al. δ-conotoxins synthesized using an acid-cleavable solubility tag approach reveal key structural determinants for NaV subtype selectivity. Journal of Biological Chemistry. 289 (51), 35341-35350 (2014).
- Heimer, P., et al. Application of Room-Temperature Aprotic and Protic Ionic Liquids for Oxidative Folding of Cysteine-Rich Peptides. ChemBioChem. 15 (18), 2754-2765 (2014).
- Kates, S. A., Albericio, F. Solid-Phase Synthesis: A Practical Guide. , Marcel Dekker Inc. New York. (2000).
- Wüthrich, K. NMR studies of structure and function of biological macromolecules (Nobel lecture). Angewandte Chemie - International Edition. 42 (29), 3340-3363 (2003).
Erratum
Formal Correction: Erratum: Synthesis and Structure Determination of µ-Conotoxin PIIIA Isomers with Different Disulfide Connectivities
Posted by JoVE Editors on 11/01/2018. Citeable Link.
An erratum was issued for: Synthesis and Structure Determination of µ-Conotoxin PIIIA Isomers with Different Disulfide Connectivities. The Protocol and Materials sections were updated.
Step 1.1.3 in the Protocol section was updated from:
Weigh the individual reagents (amino acids, HBTU (2-(1H-benzotriazol-1-yl)-1,1,3,3-tetramethyluronium hexafluorophosphate)) according to the protocol and dissolve them in dimethylformamide (DMF) to a final concentration of 2.4 M (amino acids) and 0.6 M (HBTU), respectively.
to:
Weigh the individual reagents (amino acids, HBTU (2-(1H-benzotriazol-1-yl)-1,1,3,3-tetramethyluronium hexafluorophosphate)) according to the protocol and dissolve them in dimethylformamide (DMF) to a final concentration of 0.6 M (amino acids) and 0.6 M (HBTU), respectively.
Step 1.2 in the Protocol section was updated from:
NOTE: The protocol applies to 100 mg of resin (loading: 0.28 mmol/g) added to one reaction column for 28 μmol scale.
to:
NOTE: The standardized protocol usually applies to 100 mg of resin (loading: 0.53 mmol/g) added to one reaction column for 53 μmol scale leading to the following equivalents: 5 eq. HBTU, 10 eq. NMM, 5 eq. amino acid. In case of PIIIA, however, a loading of 0.28 mmol/g (28 µmol scale) is used, which results in the specified higher equivalents.
Step 1.2.3.1 in the Protocol section was updated from:
HBTU (450 µL; 0.6 M in DMF; 270 µmol; 9.6 eq.), NMM (125 µL; 50% in DMF; 568 µmol; 20 eq.), Fmoc-amino acid (420 µL; 2.4 M in DMF; 1.01 mmol; 36 eq.).
to:
HBTU (415 µL; 0.6 M in DMF; 249 µmol; 9 eq.), NMM (112 µL; 50% in DMF; 510 µmol; 18 eq.), Fmoc-amino acid (420 µL; 0.6 M in DMF; 252 µmol; 9 eq.).
The first item in the Materials table was updated from:
| Fmoc Rink amide resin | Novabiochem | 855001 |
to:
| PS PEG2000 Fmoc Rink-amide Resin | Varian, Inc. | PL3867-3764 | AmphiSpheres 40 RAM |
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon