Method Article
Livre etiqueta de identificação dos subtipos de linfócito utilizando imagens tridimensionais fase quantitativa e aprendizagem de máquina
Neste Artigo
Resumo
Descreveremos um protocolo para a identificação do rótulo-livre dos subtipos de linfócito usando a imagem latente da fase quantitativa e um algoritmo de aprendizado de máquina. Medições de índice de refração 3D tomograms de linfócitos apresentam 3D informações morfológicas e bioquímicas para células individuais, que é, então, analisadas com um algoritmo de aprendizado de máquina para identificação dos tipos de células.
Resumo
Descrevemos aqui um protocolo para a identificação do rótulo-livre dos subtipos de linfócito usando a imagem latente da fase quantitativa e aprendizado de máquina. Identificação dos subtipos de linfócito é importante para o estudo da imunologia, bem como diagnóstico e tratamento de várias doenças. Atualmente, métodos padrão para a classificação dos tipos de linfócitos dependem de rotulagem de proteínas de membrana específicas via reações antígeno-anticorpo. No entanto, essas técnicas de rotulagem carregam os riscos potenciais de alterar funções celulares. O protocolo descrito aqui supera esses desafios, explorando contrastes ópticos intrínsecos, medidos pela imagem latente 3D fase quantitativa e um algoritmo de aprendizado de máquina. Medição de tomograms 3D Índice de refração (RI) de linfócitos fornece informações quantitativas sobre morfologia 3D e fenótipos das células individuais. Os parâmetros biofísicos extraídos o tomograms 3D medido do RI são então analisados quantitativamente com um algoritmo de aprendizagem de máquina, permitindo a identificação de rótulo livre de tipos de linfócitos em um nível de célula única. Medimos o 3D tomograms RI de linfócitos B, T CD4 + e CD8 + T e identificados os tipos de células com mais de 80% exatidão. Este protocolo, descreveremos as etapas detalhadas para isolamento de linfócitos, imagem 3D fase quantitativa e aprendizado de máquina para identificar tipos de linfócitos.
Introdução
Os linfócitos podem ser classificados em vários subtipos, incluindo B, auxiliar T (CD4 +), citotóxicos (CD8 +) T e T reguladora células. Cada tipo de linfócito tem um papel diferente no sistema imune adaptativo; por exemplo, os linfócitos B produzem anticorpos, Considerando que os linfócitos T detectam antígenos específicos, eliminam as células anormais e regulam os linfócitos B. Regulamento e função de linfócitos é firmemente controlado pelo e relacionados a várias doenças, incluindo infecções virais3, cancros1e doenças auto-imunes2. Assim, a identificação dos tipos de linfócitos é importante entender seus papéis fisiopatológicos em tais doenças e para imunoterapia em clínicas.
Atualmente, métodos de classificação de tipos de linfócitos dependem de reações antígeno-anticorpo alvejando proteínas específicas da membrana de superfície ou marcadores de superfície4. Direcionamento de marcadores de superfície é um método preciso e exato para determinar os tipos de linfócitos. No entanto, requer reagentes caros e procedimentos demorados. Além disso, ele carrega os riscos da modificação das estruturas de proteína de membrana e a alteração das funções celulares.
Para superar esses desafios, o protocolo descrito aqui introduz a identificação de rótulo livre de linfócitos tipos usando 3D fase quantitativa de imagem (QPI) e máquina de aprendizagem5. Esse método permite a classificação dos tipos de linfócitos em um nível de célula única com base nas informações morfológicas extraídas de imagem em 3D livre de rótulo de linfócitos individuais. Ao contrário de técnicas de microscopia de fluorescência convencional, QPI utiliza o índice de refração (RI) distribuições (intrínsecas propriedades óticas de células vivas e tecidos) como contraste óptico6,7. Os tomograms RI de linfócitos individuais representam informação fenotípica específica para os subtipos de linfócitos. Neste caso, para utilizar sistemicamente 3D tomograms RI de linfócitos individuais, foi utilizado um algoritmo de aprendizado de máquina supervisionado.
Usando várias técnicas QPI, os 3D tomograms RI de células têm sido ativamente utilizados para o estudo da fisiopatologia da célula porque eles fornecem um rótulo livre, quantitativa de imagem capacidade8,9,10, 11,12,13. Além disso, as distribuições de RI 3D de células individuais podem fornecer informações morfológicas, bioquímicas e biomecânicas sobre células. 3D do RI tomograms tenha sido previamente utilizados nos campos da hematologia14,15,16,17, doenças infecciosas18,19, 20, imunologia21célula biologia22,23, inflamação24, câncer25, neurociência26,27, biologia do desenvolvimento28, toxicologia 29e Microbiologia12,30,31,32.
Embora 3D RI tomograms fornecem informações detalhadas morfológicas e bioquímicas das células, a classificação dos subtipos de linfócito é difícil de alcançar por simplesmente imagem 3D RI tomograms5. Explorarem sistematicamente e quantitativamente a medida tomograms RI 3D para a classificação do tipo de célula, utilizamos um algoritmo de aprendizado de máquina. Recentemente, diversos trabalhos têm sido relatados em qual fase quantitativa, analisaram-se imagens de células com várias máquina aprendendo algoritmos33, incluindo a detecção de microorganismos34, classificação de género bacteriana35 , 36, detecção rápida e livre de rótulo de esporos de antraz37, automatizado de análise de espermatozoides38, análise de células de câncer39,40e detecção de ativação de macrófagos41.
Este protocolo fornece etapas detalhadas para executar livre de etiqueta de identificação de tipos de linfócitos no nível da célula individual usando 3D QPI e aprendizado de máquina. Isto inclui: isolamento 1) linfócitos do sangue de rato, 2) linfócito classificação através de fluxo cytometry, QPI 3) 3D, 4) quantitativos extração do 3D do RI tomograms e aprendizado 5) supervisionado para a identificação de tipos de linfócitos.
Protocolo
Cuidados com animais e procedimentos experimentais foram realizados sob a aprovação dos cuidados institucionais do Animal e uso Comitê de KAIST (KA2010-21, KA2014-01 e KA2015-03). Todas as experiências neste estudo foram realizadas em conformidade com as diretrizes aprovadas.
1. linfócitos isolado do sangue de rato
- Uma vez um rato C57BL/6J é eutanásia por inalação de CO2 , introduza uma agulha 26-G o coração de rato e recolher 0,3 mL de sangue. Diretamente colocar sangue em um tubo com 100 U/mL de solução de heparina diluído com 1 mL de tampão fosfato salino (PBS).
Nota: Como alternativa, os linfócitos do baço podem ser isolados. - Centrifugar o tubo a 400 x g por 5 min a 4 ° C.
- Adicionar 0,5 mL de tampão de lise de amónio-cloreto de potássio ao tubo e suavemente invertê-lo algumas vezes para misturar a solução.
- Incube o tubo em temperatura ambiente (RT) por 5 min.
- Lave as células adicionando 4,5 mL de PBS e centrifugação a 400 x g por 5 min a 4 ° C, duas vezes.
- Remover o sobrenadante e ressuspender as células em 100 µ l de meio RPMI-1640 fresco com 10% de soro fetal bovino (FBS).
- Adicione 0,1 µ g de anticorpos CD16/32 (2.4G2) para o tubo para o bloqueio.
- Mantenha o tubo no gelo.
2. fluxo Cytometry e classificação dos subtipos de linfócito
Nota: Classificação de linfócitos, dependendo do tipo de célula é essencial para o estabelecimento da célula de chão-verdade (ou seja, correto) digite rótulos para treinar e testar um classificador do tipo de célula no aprendizado supervisionado. Citometria de fluxo, um método padrão-ouro, é usada para identificar e separar os linfócitos42.
- Fazer uma mistura de superfície marcador coloração anticorpos em 100 µ l de meio RPMI-1640 fresco [com 10% FBS, 0,1 µ g de CD3e (17A2), CD8a (53-6,7), CD19 (1D 3), CD45R (B220, RA3-6B2) e NK1.1 (PK136)] e 0,25 µ g de anticorpos CD4 (GK1.5) para o destino B, T CD4 + e CD8 + T linfócitos.
- Adicione 100 µ l da mistura de anticorpos para a suspensão de eritrócitos (obtida na etapa 1.8).
- Incube durante 25 min no gelo.
- Lave as células adicionando 5 mL de PBS e centrifugação a 400 x g por 5 min a 4 ° C, duas vezes.
- Ressuspender as células em 5 mL de meio RPMI-1640 fresco com 10% FBS e 2,5 µ g de DAPI (4', 6-diamidino-2-phenylindole).
- Recolha separadamente cada tipo de linfócito com citometria de fluxo, usando os níveis de fluorescência dos marcadores acima descritos. Ao mesmo tempo excluir células mortas usando os sinais DAPI.
Nota: Os protocolos detalhados sobre fluxo baseados em citometria de célula classificação ter sido descrito anteriormente42.
3. 3D fase quantitativa Imaging
- Manter os linfócitos classificados no gelo durante os procedimentos de imagem, que deve ser concluído no prazo de 5h (desde o isolamento do linfócito do mouse) para evitar danos celulares e alterações bioquímicas.
- Selecione um tipo de célula classificada (entre linfócitos B, T CD4 + e CD8 + T) e diluir a amostra (obtida na etapa 2.6) para 180 células / µ l com meio RPMI para condição de imagem ideal (ou seja, uma célula por um único campo de visão).
- Carrega 120 μL da amostra diluída em uma câmara de imagens por injeção lenta. Verific completamente na presença de bolhas na câmara de imagens com a amostra. Se houver bolhas, cuidadosamente removê-los, como eles vão comprometer a qualidade das medições.
- Adquira 3D RI tomograms usando um comercial 3D quantitativo fase o microscópio, ou holotomography e seu software de imagem.
Nota: Informações detalhadas sobre a instalação experimental podem ser encontradas no manuscrito original5.- Coloque uma gota de água destilada em cima da lente da objetiva do microscópio.
- Coloque a câmara de imagem com a amostra na fase de tradução do microscópio e ajustar a sua localização, para que a amostra se alinha com a lente objetiva.
- Ajuste as posições axiais das lentes objetivas e condensador clicando em foco e superfície, respectivamente, na guia "Calibração" da perspectiva do software da imagem latente "Microscópio".
- Alinhe as lentes objetivas e condensador clicando em Modo automático. Como alternativa, use o Modo de digitalização e manualmente ajustar as lentes para que os padrões de iluminação são localizados na região central do campo de visão.
- Voltar ao Modo Normal e ajustar a fase de tradução para localizar uma célula no campo de visão.
- Encontre o plano focal, ajustando a posição axial da lente da objetiva. Foco perfeito faz com que o limite de amostra visualizado na tela quase invisível.
- Ajuste a fase de tradução para encontrar um local sem um celular.
- Clique em calibrar para medir vários hologramas 2D com diferentes ângulos de iluminação.
- Ajuste a fase de tradução para localizar um celular no centro do campo de visão.
- Mover para a guia "Aquisição" e clique em 3D instantâneo para medir os hologramas da célula com os ângulos de iluminação idêntica como feito na etapa 3.4.8.
- Quando os dados adquiridos são apresentados no painel "Gestão de dados", botão direito do mouse sobre os dados adquiridos e clique em processo para reconstruir uma tomografia 3D do RI dos hologramas medido em etapas 3.4.8 e 3.4.10, usando o algoritmo de tomografia computadorizada de difração 9 , 10 implementado no software da imagem latente.
- Repita as etapas 3.4.5-3.4.11 para medir mais de 100 células para garantir poder estatístico para aprendizado de máquina.
- Todas as imagens são processadas através do passo 3.4.11. pode ser visualizado. No painel "Gestão de dados", os dados com o botão direito e clique em abrir para visualizar os dados. Clique em tomografia de RI no painel "Data Manager". Na guia "Preset", clique em carregar e dê um duplo clique em "lymphocyte.xml", que é uma função de transferência predefinidos fornecida pelo software de imagem para visualizar a tomografia de acordo com as distribuições de RI a 3D.
- Repita as etapas de 3,2-3,4 para medir 3D tomograms RI de todos os subtipos de linfócitos.
4. quantitativo morfológicos e bioquímicos extração de RI 3D Tomograms
- Coloque todos os dados tomográficos medidos acima em uma única pasta. Dividi os tipos de células nas subpastas desta pasta. Prepare cada tomografia para ser um arquivo único Mat .
- Abra o arquivo complementar 1 - extração de recurso (escrito por uma software de processamento de imagem).
- Edite a linha 14 para designar a pasta tomografia preparada no passo 4.1.
- Edite a linha 15 para designar uma pasta para salvar os dados extraídos do recurso.
- Opcionalmente, edite a linha 17 para ajustar os parâmetros de limite de RI para extração de características. A opção padrão é 20 limiares de RI de 1.340-1.378, com um incremento de 0,002 conforme descrito anteriormente,5.
- Execute o código. Para cada tomografia no dataset, o código calcula cinco características: superfície de área (SA), o volume celular (CV), esfericidade (SI), densidade de proteína (PD) e massa seca (DM), por limite de RI. Os algoritmos detalhados para extração de características são descritos em outra parte5.
- A fim de monitorar a extração de características, durante a verificação de execução na tela de visualização de segmentação celular baseado em limite de RI.
- Checar os dados extraídos do recurso, como arquivo de Mat por tomografia, salvo na pasta designada na etapa 4.4.
5. supervisão de aprendizagem e identificação
- Aleatoriamente, dividi os dados de recurso obtidos na moda passo 4.8 para treinamento (70%) e teste (30%) com pastas separadas.
- Abrir arquivo complementar 2 - trem (escrito por uma software de processamento de imagem).
- Edite a linha 14 para designar o conjunto de treinamento preparado na etapa 5.1.
- Edite a linha 16 para designar uma pasta para salvar o classificador treinado.
- Edite a linha 17 para definir um nome de arquivo para o classificador.
- Opcionalmente, edite a linha 19 para selecionar os recursos para o treinamento. A opção padrão, conforme especificado anteriormente5, foi usada para obter os resultados representativos abaixo.
- Execute o código. Usando os recursos selecionados do conjunto de treinamento, o código treina um classificador com o k-algoritmo do vizinho mais próximo de (k -NN; k = 4) e em seguida, salva o classificador (como é chamado na etapa 5.5) na pasta designada na etapa 5.4.
- Verifique na tela de visualização do desempenho de classificador e a precisão de validação cruzada.
- Opcionalmente, treine múltiplos classificadores com combinações diferentes de recurso, repetindo as etapas 5,5-5,7. Em seguida, escolha o classificador com a maior precisão na validação cruzada.
- Abra complementar arquivo 3 - teste de (escrito por uma software de processamento de imagem).
- Edite linhas 14-15-designar o classificador treinado para ser testado.
- Edite a linha 17 para designar o conjunto de teste, preparado na etapa 5.1.
- Execute o código. O classificador descrito acima identifica os tipos de células dos linfócitos individuais no conjunto de teste.
- Verifique na tela de visualização do desempenho de identificação e a precisão do teste.
Resultados
Figura 1 mostra o processo esquemático do protocolo inteiro. Usando o procedimento aqui apresentado, isolamos B (n = 149), CD4 + T (n = 95) e CD8 + T (n = 112) linfócitos. Para obter informações de fase e amplitude em diferentes ângulos de iluminação, múltiplos hologramas 2D de cada linfócito foram medidos, alterando o ângulo de iluminação (de-60 ° a 60 °). Normalmente, 50 hologramas podem ser usados para reconstruir uma tomografia 3D do RI, mas o número de hologramas 2D pode ser ajustado Considerando a velocidade e a qualidade de imagem. Informação de amplitude e fase dos hologramas medidos são recuperados usando um algoritmo de recuperação de campo baseado na transformada de Fourier transform43,44. A 3D tomografia RI de cada linfócito foi reconstruída a partir de múltiplos 2D fase e amplitude informações recuperadas em diferentes ângulos de iluminação usando algoritmo de tomografia computadorizada de difração óptica. Detalhes do processo de imagem como método de reconstrução 3D RI tomografia podem ser encontrados em outros lugares21,,45.
Figura 2 A-2_C mostra representativa 3D renderizada RI tomograms de linfócitos B, T CD4 + e CD8 + T alocando os esquemas de cores diferentes de acordo com os valores de RI através do software da imagem latente. Partir dos valores de RI, quantitativos morfológica (SA, CV e SI) e bioquímicos (PD e DM) características foram calculadas (Figura 2A-2_C). Este resultado demonstra claramente que distribuição de RI 3D permite a análise quantitativa de informação morfológica, bem como bioquímica dos linfócitos.
Aprendizado de máquina supervisionado foi explorado para identificar tipos de linfócitos em um nível de célula única. As medida 3D RI tomograms foram aleatoriamente divididos em 70% e 30% de formação (104 b: t: 66 de CD4 + e CD8 + t: 77) e teste (b: 45, t: 29 de CD4 + e CD8 + t: 35) conjuntos de dados, respectivamente. Nós aperfeiçoamos os classificadores para màxima utilizar as impressões digitais de pilha-tipo-específico codificadas no espaço de recurso. A total precisão, sensibilidade (verdadeiro positivo) e especificidade (verdadeiro negativo) foram calculados comparando o classificador-previu resultados e tipos de células do chão-verdade.
Para demonstrar a prova de conceito do protocolo proposto, realizamos o aprendizado de máquina supervisionado em três casos diferentes: classificação binária de linfócitos B (i) e T (ii) dois subtipos de linfócitos T (CD4 + e CD8 +) e (iii) multiclass classificação de todos os tipos de linfócitos.
Figura 3 mostra o desempenho de identificação de classificadores otimizadas para fases de treinamento e teste. A precisão da classificação T e linfócitos B foi 93.15% e 89.81% para a formação e casos de teste, respectivamente. Os linfócitos CD4 + e CD8 + T estatisticamente foram classificados, e a precisão foi 87.41% e 84.38% para a formação e conjuntos de teste, respectivamente. Por último, a precisão do classificador tipo célula multiclass foi 80.65% e 75.93% para as fases de treinamento e teste, respectivamente.

Figura 1 : Diagramas esquemáticos da identificação dos tipos de linfócito explorando rótulo livre 3 D fase quantitativa imaging e aprendizado de máquina. Clique aqui para ver uma versão maior desta figura.
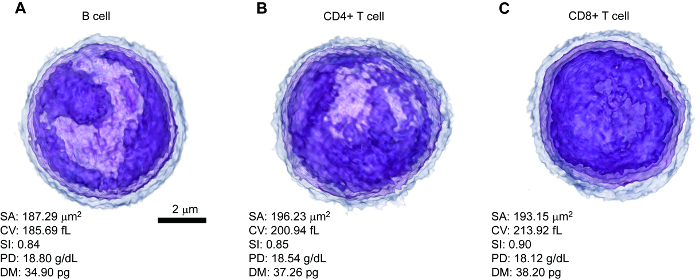
Figura 2 : Representante 3 D processado RI tomograms cada tipo de linfócito de células com características morfológicas e bioquímicas quantitativas. (A) de células B, células b CD4 + T e células (C) CD8 + T. Barra de escala = 2 µm. SA, área de superfície; CV, volume celular; SI, esfericidade; PD, densidade de proteína; DM, massa seca. Esta figura é modificada com permissão5. Clique aqui para ver uma versão maior desta figura.
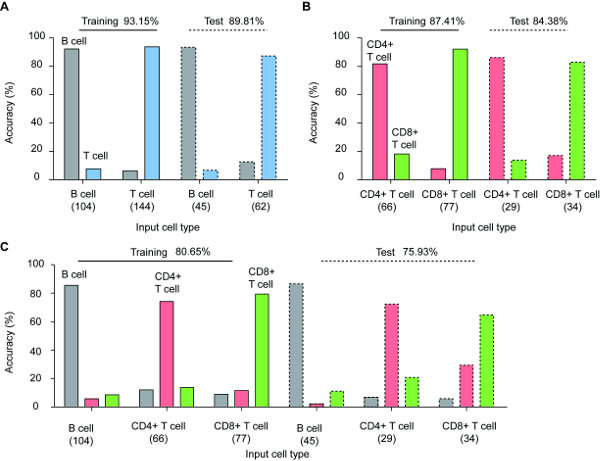
Figura 3 : Identificação do linfócito individual tipos através de aprendizado de máquina supervisionado (A) binária classificação de células B e T, (B) binária classificação de células CD4 + e CD8 + T e (C) multiclass classificação todos os três tipos de células linfócitos; para conjuntos de tanto treinamento e teste. Note a pequena diferença entre a formação e casos de teste, sugerindo boa generalização dos classificadores estabelecidas. Os números abaixo os nomes de cada tipo de célula indicam o número de células utilizadas. Esta figura é modificada com permissão5. Clique aqui para ver uma versão maior desta figura.

Complementares 1 arquivo: apresentam código de extração. Extração de características (SA, CV, SI, PD e DM) depois de segmentação baseado em limite de RI de cada tomografia. Implementado em uma software de processamento de imagem. Clique aqui para baixar este arquivo.

Complementar arquivo 2: código de treinamento. Formação de um k -NN classificador de treinamento baseado nas características selecionadas. Implementado em uma software de processamento de imagem. Clique aqui para baixar este arquivo.

Complementar arquivo 3: testes código. Testando um classificador NN - treinados kpara um novo dataset (i. e., conjunto de teste). Implementado em uma software de processamento de imagem. Clique aqui para baixar este arquivo.
Discussão
Apresentamos um protocolo que permite a identificação de rótulo livre dos tipos de linfócitos, explorando a imagem 3D fase quantitativa e aprendizado de máquina. Passos críticos do presente protocolo são fase quantitativa de imagem e o recurso seleção. Para a ideal de imagem holográfica, a densidade de células deve ser controlada como descrito acima. Estabilidade mecânica das células também é importante para obter uma distribuição de RI 3D precisa porque movimentos celulares flutuantes ou vibracionais perturbará as medições de holograma sobre mudanças de ângulo de iluminação. Nós, portanto esperou vários minutos até que a amostra se tornou estável e estático na câmara de imagem antes de medir os hologramas. Por último, bolhas no interior da câmara de imagem são problemáticas quando medindo hologramas devido a diferenças de RI entre o ar e a amostra; assim, a amostra deve ser cuidadosamente carregada para a câmara de imagem.
Extração de características e seleção ajudam a determinar o desempenho de identificação do classificador. Calculamos 5 quantitativos morfológica (CV, SA, SI) e bioquímico (PD, DM) características da distribuição de RI 3D em 20 diferentes valores de limite do RI; assim, extraímos um total de 100 recursos. Procuramos exaustivamente óptimas combinações característica e classificador, que mostram que a melhor precisão de validação cruzada foi seleccionada. Nós testamos 6 algoritmos de aprendizado de máquina diferente, incluindo k -NN (k = 4 e k = 6), análise linear de discriminação, análise de discriminação quadrática, naïve Bayes e árvore de decisão e encontramos esse k -NN (k = 4 ) mostrou o melhor desempenho de identificação. No entanto, há uma chance de melhorar a precisão de identificação usando outra métodos, incluindo redes neuronais e máquina do vetor da sustentação de aprendizagem de máquina.
Este protocolo medidas intrínsecas propriedades óticas através de imagem 3D fase quantitativa a fim de identificar tipos de linfócitos; assim, não exige um processo de rotulagem baseado em reações antígeno-anticorpo usadas em fluorescência ou magnética do grânulo técnicas baseadas em ordenação de células, que têm riscos de alterar a função celular, modificando as estruturas de proteínas de membrana. Além disso, o presente método mede a distribuição de RI 3D e fornece informações morfológicas e bioquímicas 3D sobre a célula, o que não pode ser obtida por um único tiro holografia método46; Portanto, o desempenho de identificação do protocolo é mais preciso devido a informações de alta dimensão.
Uma limitação menor do presente protocolo é o ajuste manual da fase de amostra e necessário processo de aprendizado de máquina supervisionado de rotulagem. Procuramos um linfócito, ajustando o estágio de translação manual e medidos hologramas, que são as etapas mais demoradas. Esta limitação seria maior empregando um palco motorizado automatizado ou dispositivos de canal microfluidic. Em matéria de aprendizado supervisionado, os tipos de linfócitos conhecidos são obrigados a constituir o classificador ideal; assim, tivemos a primeira isolar e identificar tipos de células de linfócitos com base na classificação técnica baseados no antígeno-anticorpo. Contudo, este protocolo ainda usa o contraste óptico intrínseco de linfócitos e os agentes rotulagem usados para especificar os anticorpos têm efeitos negligenciáveis sobre o sinal medido de RI 3D. Portanto, o classificador estabelecido pode ser utilizado para a identificação de linfócitos em uma forma sem rótulo.
Embora este protocolo utiliza principalmente fenótipos de linfócitos pela medição 3D tomograms RI das células individuais, estes dados RI 3D também podem ser usados em combinação com outras modalidades de endereçamento genótipos ou proteomic informações para a melhor classificação de subtipos. Recentemente, técnicas de microscopia correlativa combinando imagens de fluorescência e QPI têm sido introduzidas47,,48,49. A abordagem apresentada no presente protocolo também pode ser estendida para esses métodos de imagem correlativos.
Livre de etiqueta de identificação de tipos de linfócitos pode ser aplicada para estudar a fisiopatologia ou diagnosticar a doença através da detecção de linfócitos anormais ou rácios entre os tipos de linfócitos. Além disso, este protocolo pode ser aplicado a análise de sangue total através da identificação de várias células, incluindo os glóbulos vermelhos, plaquetas e glóbulos brancos.
Divulgações
Prof Y. Park, Y. Jo, Y. S. Kim e S. Lee tem interesses financeiros em Tomocube, Inc., uma empresa que comercializa quantitativa fase instrumentos de imagem e tomografia computadorizada de difração óptica e é um dos patrocinadores do trabalho.
Agradecimentos
Este trabalho foi apoiado pela KAIST BK21 + programa, Tomocube, Inc. e da National Research Foundation da Coreia (2015R1A3A2066550, 2017M3C1A3013923, 2018K 000396). Y. Jo reconhece apoio do KAIST presidencial Fellowship e bolsa de ciência biomédica Asan Foundation.
Materiais
| Name | Company | Catalog Number | Comments |
| Mouse | Daehan Biolink | C57BL/6J mice | gender and age-matched, 6 – 8 weeks |
| Falcon conical centrifuge tube | ThermoFisher Scientific | 14-959-53A | 15 mL |
| Phosphate-buffered saline | Sigma-Aldrich | 806544-500ML | |
| Ammonium-chloride-potassium lysing buffer | ThermoFisher Scientific | A1049201 | |
| RPMI-1640 medium | Sigma-Aldrich | R8758 | |
| Fetal bovine serum | ThermoFisher Scientific | 10438018 | |
| Antibody | BD Biosciences | 553140 (RRID:AB_394655) | CD16/32 (clone 2.4G2) |
| Antibody | BD Biosciences | 555275 (RRID:AB_395699) | CD3ε (clone 17A2) |
| Antibody | Biolegnd | 100734 (RRID:AB_2075238) | CD8α (clone 53-6.7) |
| Antibody | BD Biosciences | 557655 (RRID:AB_396770) | CD19 (clone 1D3) |
| Antibody | BD Biosciences | 557683 (RRID:AB_396793) | CD45R/B220 (clone RA3-6B2) |
| Antibody | BD Biosciences | 552878 (RRID:AB_394507) | NK1.1 (clone PK136) |
| Antibody | eBioscience | 11-0041-85 (RRID:AB_464893) | CD4 (clone GK1.5) |
| DAPI | Roche | 10236276001 | 4,6-diamidino-2-phenylindole |
| Flow cytometry | BD Biosciences | Aria II or III | |
| Imaging chamber | Tomocube, Inc. | TomoDish | |
| Holotomography | Tomocube, Inc. | HT-1H | |
| Holotomography imaging software | Tomocube, Inc. | TomoStudio | |
| Image professing software | MathWorks | Matlab R2017b |
Referências
- Alizadeh, A. A., et al. Distinct types of diffuse large B-cell lymphoma identified by gene expression profiling. Nature. 403 (6769), 503 (2000).
- Von Boehmer, H., Melchers, F. Checkpoints in lymphocyte development and autoimmune disease. Nature Immunology. 11 (1), 14 (2010).
- Sáez-Cirión, A., et al. HIV controllers exhibit potent CD8 T cell capacity to suppress HIV infection ex vivo and peculiar cytotoxic T lymphocyte activation phenotype. Proceedings of the National Academy of Sciences. 104 (16), 6776-6781 (2007).
- Fischer, K., et al. Isolation and characterization of human antigen-specific TCRαβ+ CD4-CD8-double-negative regulatory T cells. Blood. 105 (7), 2828-2835 (2005).
- Yoon, J., et al. Identification of non-activated lymphocytes using three-dimensional refractive index tomography and machine learning. Scientific Reports. 7 (1), 6654 (2017).
- Popescu, G. . Quantitative phase imaging of cells and tissues. , (2011).
- Lee, K., et al. Quantitative phase imaging techniques for the study of cell pathophysiology: from principles to applications. Sensors. 13 (4), 4170-4191 (2013).
- Kim, D., et al. Refractive index as an intrinsic imaging contrast for 3-D label-free live cell imaging. bioRxiv. , 106328 (2017).
- Kim, K., et al. Optical diffraction tomography techniques for the study of cell pathophysiology. Journal of Biomedical Photonics & Engineering. 2 (2), (2016).
- Wolf, E. Three-dimensional structure determination of semi-transparent objects from holographic data. Optics Communications. 1 (4), 153-156 (1969).
- Kus, A., Dudek, M., Kemper, B., Kujawinska, M., Vollmer, A. Tomographic phase microscopy of living three-dimensional cell cultures. Journal of Biomedical Optics. 19 (4), 046009 (2014).
- Kim, T., et al. White-light diffraction tomography of unlabelled live cells. Nature Photonics. 8 (3), 256 (2014).
- Simon, B., et al. Tomographic diffractive microscopy with isotropic resolution. Optica. 4 (4), 460-463 (2017).
- Kim, Y., et al. Profiling individual human red blood cells using common-path diffraction optical tomography. Scientific Reports. 4, (2014).
- Park, H., et al. Measuring cell surface area and deformability of individual human red blood cells over blood storage using quantitative phase imaging. Scientific Reports. 6, (2016).
- Lee, S., et al. Refractive index tomograms and dynamic membrane fluctuations of red blood cells from patients with diabetes mellitus. Scientific Reports. 7, (2017).
- Merola, F., et al. Tomographic flow cytometry by digital holography. Light-Science & Applications. 6, (2017).
- Park, Y., et al. Refractive index maps and membrane dynamics of human red blood cells parasitized by Plasmodium falciparum. Proceedings of the National Academy of Sciences. 105, 13730-13735 (2008).
- Park, H., et al. Characterizations of individual mouse red blood cells parasitized by Babesia microti using 3-D holographic microscopy. Scientific Reports. 5, 10827 (2015).
- Chandramohanadas, R., et al. Biophysics of malarial parasite exit from infected erythrocytes. Public Library of Science ONE. 6 (6), 20869 (2011).
- Yoon, J., et al. Label-free characterization of white blood cells by measuring 3D refractive index maps. Biomedical Optics Express. 6 (10), 3865-3875 (2015).
- Kim, K., et al. Three-dimensional label-free imaging and quantification of lipid droplets in live hepatocytes. Scientific Reports. 6, 36815 (2016).
- Kim, D., et al. Label-free high-resolution 3-D imaging of gold nanoparticles inside live cells using optical diffraction tomography. Methods. , (2017).
- Lenz, P., et al. Multimodal Quantitative Phase Imaging with Digital Holographic Microscopy Accurately Assesses Intestinal Inflammation and Epithelial Wound Healing. Journal of Visualized Experiments. (115), (2016).
- Huang, J., Guo, P., Moses, M. A. A Time-lapse, Label-free, Quantitative Phase Imaging Study of Dormant and Active Human Cancer Cells. Journal of Visualized Experiments. (132), (2018).
- Yang, S. A., Yoon, J., Kim, K., Park, Y. Measurements of morphological and biochemical alterations in individual neuron cells associated with early neurotoxic effects in Parkinson's disease. Cytometry Part A. 91 (5), 510-518 (2017).
- Cotte, Y., et al. Marker-free phase nanoscopy. Nature Photonics. 7 (2), 113-117 (2013).
- Nguyen, T. H., Kandel, M. E., Rubessa, M., Wheeler, M. B., Popescu, G. Gradient light interference microscopy for 3D imaging of unlabeled specimens. Nature Communications. 8 (1), 210 (2017).
- Kwon, S., et al. Mitochondria-targeting indolizino [3, 2-c] quinolines as novel class of photosensitizers for photodynamic anticancer activity. European Journal of Medicinal Chemistry. 148, 116-127 (2018).
- Bennet, M., Gur, D., Yoon, J., Park, Y., Faivre, D. A Bacteria-Based Remotely Tunable Photonic Device. Advanced Optical Materials. , (2016).
- Kim, T. I., et al. Antibacterial Activities of Graphene Oxide-Molybdenum Disulfide Nanocomposite Films. ACS Applied Materials & Interfaces. 9 (9), 7908-7917 (2017).
- Bedrossian, M., Barr, C., Lindensmith, C. A., Nealson, K., Nadeau, J. L. Quantifying Microorganisms at Low Concentrations Using Digital Holographic Microscopy (DHM). Journal of Visualized Experiments. (129), (2017).
- Jo, Y., et al. Quantitative Phase Imaging and Artificial Intelligence: A Review. arXiv preprint. , (2018).
- Javidi, B., Moon, I., Yeom, S., Carapezza, E. Three-dimensional imaging and recognition of microorganism using single-exposure on-line (SEOL) digital holography. Optics Express. 13 (12), 4492-4506 (2005).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Optics Express. 23 (12), 15792-15805 (2015).
- Jo, Y., et al. Angle-resolved light scattering of individual rod-shaped bacteria based on Fourier transform light scattering. Scientific Reports. 4, 5090 (2014).
- Jo, Y., et al. Holographic deep learning for rapid optical screening of anthrax spores. Science Advances. 3 (8), 1700606 (2017).
- Mirsky, S. K., Barnea, I., Levi, M., Greenspan, H., Shaked, N. T. Automated analysis of individual sperm cells using stain-free interferometric phase microscopy and machine learning. Cytometry Part A. 91 (9), 893-900 (2017).
- Roitshtain, D., et al. Quantitative phase microscopy spatial signatures of cancer cells. Cytometry Part A. 91 (5), 482-493 (2017).
- Lam, V. K., Nguyen, T. C., Chung, B. M., Nehmetallah, G., Raub, C. B. Quantitative assessment of cancer cell morphology and motility using telecentric digital holographic microscopy and machine learning. Cytometry Part A. , (2017).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences. , (2018).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of Specific Cell Population by Fluorescence Activated Cell Sorting (FACS). Journal of Visualized Experiments. (41), e1546 (2010).
- Takeda, M., Ina, H., Kobayashi, S. Fourier-transform method of fringe-pattern analysis for computer-based topography and interferometry. Journal of the Optical Society of America. 72 (1), 156-160 (1982).
- Debnath, S. K., Park, Y. Real-time quantitative phase imaging with a spatial phase-shifting algorithm. Optics Letters. 36 (23), 4677-4679 (2011).
- Kim, K., et al. High-resolution three-dimensional imaging of red blood cells parasitized by Plasmodium falciparum and in situ hemozoin crystals using optical diffraction tomography. Journal of Biomedical Optics. 19 (1), 011005 (2013).
- Vercruysse, D., et al. Three-part differential of unlabeled leukocytes with a compact lens-free imaging flow cytometer. Lab on a Chip. 15 (4), 1123-1132 (2015).
- Kim, K., et al. Correlative three-dimensional fluorescence and refractive index tomography: bridging the gap between molecular specificity and quantitative bioimaging. Biomedical Optics Express. 8 (12), 5688-5697 (2017).
- Shin, S., Kim, D., Kim, K., Park, Y. Super-resolution three-dimensional fluorescence and optical diffraction tomography of live cells using structured illumination generated by a digital micromirror device. arXiv preprint. , (2018).
- Chowdhury, S., Eldridge, W. J., Wax, A., Izatt, J. A. Structured illumination multimodal 3D-resolved quantitative phase and fluorescence sub-diffraction microscopy. Biomedical Optics Express. 8 (5), 2496-2518 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados