Method Article
Máquina de aprendizaje y sin etiqueta de identificación de subtipos de linfocitos usando proyección de imagen tridimensional fase cuantitativa
En este artículo
Resumen
Se describe un protocolo para la identificación de subtipos de linfocitos usando proyección de imagen de fase cuantitativa y un algoritmo de aprendizaje automático libre de etiqueta. Las mediciones de índice de refracción 3D tomogramas de linfocitos presentan 3D información morfológica y bioquímica de células individuales, que luego se analiza con un algoritmo de aprendizaje automático para la identificación de tipos de la célula.
Resumen
Describimos aquí un protocolo para la identificación de subtipos de linfocitos usando proyección de imagen de fase cuantitativa y el aprender de máquina libre de etiqueta. La identificación de subtipos de linfocitos es importante para el estudio de la inmunología, así como diagnóstico y tratamiento de diversas enfermedades. En la actualidad, los métodos estándar para la clasificación de tipos de linfocitos dependen de etiquetado de proteínas de membrana específicas mediante reacciones antígeno-anticuerpo. Sin embargo, estas técnicas de etiquetado llevan los riesgos potenciales de alteración de las funciones celulares. El protocolo descrito aquí supera estos desafíos aprovechando intrínsecos contrastes ópticos mide la proyección de imagen 3D fase cuantitativa y un algoritmo de aprendizaje automático. La medición de los tomogramas 3D índice de refracción (RI) de linfocitos proporciona información cuantitativa sobre morfología 3D y fenotipos de células individuales. Los parámetros biofísicos extraídos de los tomogramas de RI 3D medidos entonces se analizan cuantitativamente con un algoritmo de aprendizaje de máquina, permitiendo libre de etiqueta de identificación de tipos de linfocitos a nivel unicelular. Medimos los tomogramas RI 3D de linfocitos B, T CD4 + y T CD8 + e identifica los tipos de la célula con más del 80% exactitud. En este protocolo, se describen los pasos detallados para aislamiento de linfocitos, la proyección de imagen 3D fase cuantitativa y el aprender de máquina para identificar los tipos de linfocitos.
Introducción
Los linfocitos pueden clasificarse en varios subtipos, incluyendo B, helper (CD4 +) T, T citotóxico (CD8 +) y T reguladora las células. Cada tipo de linfocito tiene un papel diferente en el sistema inmune adaptativo; por ejemplo, los linfocitos B producen anticuerpos, mientras que los linfocitos T detectan antígenos específicos, eliminan las células anormales y regulan los linfocitos B. Regulación y la función del linfocito es firmemente controlado por y relaciona con diversas enfermedades, incluyendo cáncer1, enfermedades autoinmunes2y3de las infecciones virales. Por lo tanto, la identificación de tipos de linfocitos es importante entender su papel fisiopatológico en este tipo de enfermedades y para inmunoterapia en clínicas.
Actualmente, métodos para clasificar los tipos de linfocitos se basan en reacciones antígeno-anticuerpo dirigiéndose a proteínas de membrana específicas de superficie o marcadores de superficie4. Marcadores de superficie de fijación de objetivos son un método preciso y exacto para determinar tipos de linfocitos. Sin embargo, requiere procedimientos desperdiciadores de tiempo y reactivos costosos. Además, conlleva riesgos de la modificación de las estructuras de la proteína de la membrana y la alteración de las funciones celulares.
Para superar estos retos, el protocolo descrito aquí introduce la identificación etiqueta-libre de tipos de linfocitos mediante fase cuantitativa 3D imaging (QPI) y machine learning5. Este método permite la clasificación de los tipos de linfocitos a nivel unicelular, basado en información morfológica extraída de la proyección de imagen 3D sin etiqueta de linfocitos individuales. A diferencia de técnicas de microscopía de fluorescencia convencional, QPI utiliza el índice de refracción (RI) distribuciones (propiedades ópticas intrínsecas de células vivas y tejidos) como contraste óptico6,7. Los tomogramas RI de linfocitos individuales representan fenotípica información específica de los subtipos de linfocitos. En este caso, sistémicamente utilizar 3D RI tomogramas de linfocitos individuales, se utilizó un algoritmo de aprendizaje supervisado de la máquina.
Utilizando diversas técnicas QPI, los tomogramas RI 3D de las células han sido activamente utilizados para el estudio de la fisiopatología celular porque proporcionan una etiqueta libre, cuantitativo de imagen capacidad8,9,10, 11,12,13. También, las distribuciones RI 3D de células individuales pueden proporcionar información morfológica, bioquímica y biomecánica sobre las células. Los tomogramas RI 3D se han utilizado anteriormente en los campos de Hematología14,15,16,17, enfermedades infecciosas18,19, 20, Inmunología21, celular biología22,23, inflamación24, cáncer25, neurociencia26,27,28de la biología del desarrollo, toxicología 29y Microbiología12,30,31,32.
Aunque 3D tomogramas de RI proporcionan toda la información morfológica y bioquímica de las células, la clasificación de subtipos de linfocitos es difícil de lograr por simplemente la proyección de imagen 3D de los tomogramas RI5. Sistemáticamente y cuantitativamente explotar los medida 3D tomogramas de RI para la clasificación del tipo de célula, se utilizó un algoritmo de aprendizaje de máquina. Recientemente, varios trabajos han reportado en que fase cuantitativa se analizaron imágenes de células con varios máquina aprender algoritmos33, incluyendo la detección de microorganismos34, clasificación de Género bacteriano35 , 36, detección rápida y etiqueta-libre de esporas de ántrax37, había automatizado análisis de espermatozoides38, análisis de cáncer las células39,40y la detección de activación de macrófagos41.
Este protocolo proporciona pasos detallados para realizar libre de etiqueta de identificación de tipos de linfocitos a nivel de célula individual utilizando 3D QPI y el aprender de máquina. Esto incluye: aislamiento 1) del linfocito de la sangre de ratón, 2) linfocitos clasificación mediante extracción característica 4) cuantitativos flujo cytometry, QPI 3) 3D, 3D tomogramas de RI y aprendizaje 5) actividades para identificar los tipos de linfocitos.
Protocolo
Cuidado de los animales y los procedimientos experimentales se realizaron bajo la aprobación de la atención institucional del Animal y uso Comité de KAIST (KA2010-21 KA2014-01 y KA2015-03). Todos los experimentos en este estudio se llevaron a cabo conforme a los lineamientos aprobados.
1. aislamiento linfocitos de sangre de ratón
- Una vez un ratón C57BL/6J es eutanasia por inhalación de CO2 , inserte una aguja 26-G en el corazón de ratón y recoger 0,3 mL de sangre. Directamente poner sangre en un tubo con 100 U/mL de solución de heparina diluida con 1 mL de tampón fosfato salino (PBS).
Nota: Como alternativa, los linfocitos del bazo pueden ser aislados. - Centrifugar el tubo a 400 x g durante 5 min a 4 ° C.
- Agregar 0,5 mL de tampón de lisis de amonio cloruro de potasio en el tubo y se invierta suavemente unas pocas veces para mezclar la solución.
- Incubar el tubo a temperatura ambiente (RT) durante 5 minutos.
- Lavar las células agregar 4,5 mL de PBS y centrifugación a 400 x g durante 5 min a 4 ° C, dos veces.
- Quite el sobrenadante y resuspender el precipitado de células en 100 μl de medio fresco RPMI-1640 con 10% suero bovino fetal (FBS).
- Agregar 0.1 μg de anticuerpo CD16/32 (2.4G2) en el tubo de bloqueo.
- Mantenga el tubo en hielo.
2. flujo Cytometry y la clasificación de subtipos de linfocitos
Nota: Clasificación linfocitos dependiendo del tipo de célula es esencial para el establecimiento de la célula de la tierra-verdad (es decir, correcto) tipo etiquetas para entrenar y probar un clasificador de tipo de célula en el aprendizaje supervisado. Citometría de flujo, un método estándar de oro, se utiliza para identificar y separar los linfocitos42.
- Hacer una mezcla de anticuerpos en 100 μl de Medio RPMI-1640 fresco la coloración superficial del marcador [10% FBS, 0,1 μg de CD3e (17A2), CD8a (53-6.7), CD19 (1D 3), CD45R (B220, RA3-6B2) y NK1.1 (PK136)] y 0.25 μg de anticuerpos CD4 (GK1.5) al objetivo B, T CD4 + y T CD8 + linfocitos.
- Añada 100 μl de la mezcla de anticuerpos a la suspensión de células (obtenida en el paso 1.8).
- Incubar durante 25 min sobre hielo.
- Lavar las células añadiendo 5 mL de PBS y centrifugación a 400 x g durante 5 min a 4 ° C, dos veces.
- Resuspender el precipitado de células en 5 mL de medio fresco RPMI-1640 con 10% FBS y 2,5 μg de DAPI (4', 6-diamidino-2-phenylindole).
- Recoger por separado cada tipo de linfocito con citometría de flujo utilizando los niveles de fluorescencia de los marcadores descritos anteriormente. Al mismo tiempo excluir las células muertas usando las señales DAPI.
Nota: Protocolos detallados con respecto a flujo celular basados en citometría de clasificación han sido describen42.
3. proyección de imagen de fase cuantitativa 3D
- Mantener los linfocitos ordenados en el hielo a lo largo de los procedimientos de proyección de imagen, que debe completarse dentro de 5 h (desde aislamiento de linfocitos de ratón) para evitar el daño celular y alteraciones bioquímicas.
- Seleccione un tipo de células clasificadas (entre linfocitos B, T CD4 + y T CD8 +) y diluir la muestra (obtenida en el paso 2.6) a 180 células/μl con Medio RPMI para condición de proyección de imagen óptima (es decir, una célula por campo de visión única).
- Carga 120 μL de la muestra diluida en una cámara de imágenes por inyección lenta. Atentamente para comprobar la presencia de burbujas en la cámara de proyección de imagen con la muestra. Si hay burbujas, retire con cuidado, ya que pondrá en peligro la calidad de las mediciones.
- Adquirir los tomogramas RI 3D utilizando un comercial 3D cuantitativo microscopio de fase, o holotomography y su software de proyección de imagen.
Nota: Toda la información sobre la configuración experimental puede encontrarse en el manuscrito original5.- Coloque una gota de agua destilada encima del objetivo del microscopio.
- Coloque la cámara de proyección de imagen con la muestra en la etapa de la traducción del microscopio y ajustar su ubicación para que la muestra se alinea con el objetivo.
- Ajuste la posición axial de las lentes objetivo y condensador haciendo clic en foco y la superficie, respectivamente, en la pestaña "Calibración" de la perspectiva de "Microscopio" de la proyección de imagen de software.
- Alinear las lentes objetivo y condensador haciendo clic en El modo automático. Como alternativa, utilice el Modo de escaneo y ajustar manualmente los lentes para que los patrones de iluminación se localizan en la región central del campo de visión.
- Volver al Modo Normal y ajustar la etapa de la traducción para localizar un celular en el campo de visión.
- Encontrar el plano focal mediante el ajuste de la posición axial de la lente del objetivo. Perfecto enfoque hace el límite muestra visualizado en la pantalla casi invisible.
- Ajuste de la etapa de la traducción para encontrar un lugar sin un celular.
- Haga clic en calibrar para medir múltiples hologramas 2D con diferentes ángulos de iluminación.
- Ajuste de la etapa de la traducción para localizar un celular en el centro del campo de visión.
- Mover a la pestaña de "Adquisición" y haga clic en instantánea 3D para medir los hologramas de la célula con los ángulos de iluminación idéntica como hecho en el paso 3.4.8.
- Los datos adquiridos se presentaron en el panel "Gestión de datos", haga clic en los datos adquiridos y el proceso para reconstruir un tomograma RI 3D de los hologramas medido en pasos 3.4.8 3.4.10, utilizando el algoritmo de la tomografía de difracción 9 , 10 implementado en el software de imágenes.
- Repita los pasos 3.4.5-3.4.11 para medir más de 100 células para asegurarse de poder estadístico para el aprender de máquina.
- Todas las imágenes que se procesan a través del paso 3.4.11. se pueden visualizar. En el panel "Gestión de datos", haga clic en los datos y haga clic en abrir para visualizar los datos. Haz clic en el tomograma de RI en el panel de "Data Manager". En la ficha "Preset", haga clic en cargar y haga doble clic en "lymphocyte.xml", que es una función de transferencia predefinidos proporcionada por el software de imágenes para visualizar el tomograma según las distribuciones RI 3D.
- Repita los pasos 3.2-3.4 medir tomogramas de RI 3D de todos los subtipos de linfocitos.
4. extracción cuantitativo característica morfológicas y bioquímicas de los tomogramas RI 3D
- Coloque todos los datos tomográficos medidos por encima en una sola carpeta. Dividir los tipos de células en las subcarpetas de esta carpeta. Preparar cada tomograma a ser un archivo solo Mat .
- Abra el archivo complementario 1 - extracción de la característica (escrito para un software de procesamiento de imágenes).
- Editar línea 14 para designar la carpeta tomograma preparada en el paso 4.1.
- Editar la línea 15 para designar una carpeta para guardar los datos extraídos de la característica.
- Opcionalmente, modifique la línea 17 a ajustar los parámetros de umbral de RI para la extracción de la característica. La opción por defecto es 20 umbrales de RI de 1.340-1.378, con incrementos de 0.002 como se describió anteriormente5.
- Ejecutar el código. Para cada tomograma del conjunto de datos, el código calcula cinco características: superficie del área (SA), volumen celular (CV), esfericidad (SI), densidad de proteína (EP) y masa seca (MS), por umbral de RI. Los algoritmos detallados para la extracción de características se describen en otra parte5.
- Con el fin de supervisar la extracción de la característica, en el control de ejecución en la pantalla de visualización de segmentación de células basadas en umbrales de RI.
- Verificación de los datos extraídos de la característica, como archivo .MTL por tomograma, guardado en la carpeta señalada en el paso 4.4.
5. supervisa el aprendizaje y la identificación
- Partida al azar los datos de la función obtenidos en los grupos de paso 4.8 formación (70%) y examen (30%) de carpetas distintas.
- Abrir archivo suplementario 2 - tren (escrito por un software de procesamiento de imágenes).
- Editar línea 14 para designar el conjunto de entrenamiento preparado en el paso 5.1.
- Editar línea 16 para designar una carpeta para guardar el clasificador capacitado.
- Editar línea 17 para establecer un nombre de archivo para el clasificador.
- Opcionalmente, modifique la línea 19 para seleccionar las características para el entrenamiento. La opción predeterminada, como se especifica anteriormente5, se utilizó para obtener los resultados representativos a continuación.
- Ejecutar el código. El código utilizando las funciones seleccionadas del conjunto de entrenamiento, entrena a un clasificador con el k-más cercano algoritmo vecino (k -NN; k = 4) y luego guarda el clasificador (como en el paso 5.5) en la carpeta señalada en el paso 5.4.
- Compruebe en la pantalla visualizar el rendimiento del clasificador y la exactitud de la validación cruzada.
- Opcionalmente, tren de clasificadores múltiples combinaciones de diferentes característica, repitiendo los pasos 5.5-5.7. Continuación, elija el clasificador con la más alta precisión de validación cruzada.
- Abrir archivo complementario 3 - prueba (escrito por un software de procesamiento de imágenes).
- Editar líneas 14-15 para designar el clasificador capacitado para ser probado.
- Editar línea 17 para designar el conjunto de prueba preparado en el paso 5.1.
- Ejecutar el código. El clasificador descrito identifica los tipos celulares de los linfocitos individuales en el equipo de prueba.
- Compruebe en la pantalla visualizar el funcionamiento de la identificación y la exactitud de la prueba.
Resultados
Figura 1 muestra el proceso esquemático de todo protocolo. Utilizando el procedimiento presentado aquí, se aislaron B (n = 149), CD4 + T (n = 95) y T CD8 + (n = 112) los linfocitos. Para obtener información de fase y amplitud en diferentes ángulos de iluminación, se midieron múltiples hologramas 2D de cada linfocito cambiando el ángulo de iluminación (de-60 ° a 60 °). Por lo general, 50 hologramas se pueden utilizar para reconstruir un tomograma RI 3D, pero el número de hologramas 2D puede ajustarse teniendo en cuenta la velocidad de proyección de imagen y calidad. Información de amplitud y fase de los hologramas medidos se recuperan usando un algoritmo de recuperación de campo basado en Fourier transform43,44. El tomograma RI 3D de cada linfocito fue reconstruido a partir de múltiples 2D obtenido fase y amplitud de información en diferentes ángulos de iluminación usando difracción óptica tomografía algoritmo. Detalles del proceso de imagen y método de la reconstrucción 3D de RI tomograma se pueden encontrar en otra parte21,45.
Figura 2 A-2_C muestra representativa 3D renderizada RI tomogramas de linfocitos B, T CD4 + y CD8 + T mediante la asignación de esquemas de color diferentes según los valores de RI mediante el software de imágenes. De los valores de RI, cuantitativos morfológicos (SA, CV y SI) y se calcularon las características bioquímicas (PD y DM) (figura 2A-2_C). Este resultado demuestra claramente que la distribución RI 3D permite análisis cuantitativo de información morfológica y bioquímica de los linfocitos.
Aprendizaje automático supervisado fue explotado para identificar los tipos de linfocitos a nivel unicelular. Los tomogramas de RI 3D medidos fueron aleatoriamente divididos en 70% y 30% de la formación (B: 104, 66 de t de CD4 + y CD8 + T: 77) y prueba (B: 45 T: 29 de CD4 + y CD8 + T: 35) conjuntos de datos, respectivamente. Hemos optimizado los clasificadores para utilizar máximo las huellas específicas de tipo celular codificadas en el espacio de la característica. La exactitud total, sensibilidad (verdaderos positivos) y especificidad (verdaderos negativos) se calcularon comparando el clasificador predice resultados y tipos de la célula de verdad terreno.
Para demostrar la prueba de concepto del protocolo propuesto, realizamos aprendizaje automático supervisado en tres casos diferentes: clasificación binaria de i B y T linfocitos y (ii) dos subtipos de linfocitos T (CD4 + y CD8 +) y (iii) multiclase clasificación de todos los tipos de linfocitos.
Figura 3 muestra el rendimiento de identificación de clasificadores optimizados para las etapas de entrenamiento y prueba. La precisión de la clasificación T y linfocitos B fue 93.15% y 89.81% para la formación y casos de prueba, respectivamente. Los linfocitos CD4 + y T CD8 + fueron clasificados estadísticamente, y la exactitud fue 87.41% y 84.38% para la formación y prueba de sistemas, respectivamente. Por último, la precisión del clasificador de tipo de célula multiclase fue 80.65% y 75.93% para las etapas de entrenamiento y prueba, respectivamente.

Figura 1 : Diagramas esquemáticos de la identificación de etiqueta-libre de tipos de linfocitos explotar 3 D fase cuantitativa la proyección de imagen y el aprender de máquina. Haga clic aquí para ver una versión más grande de esta figura.
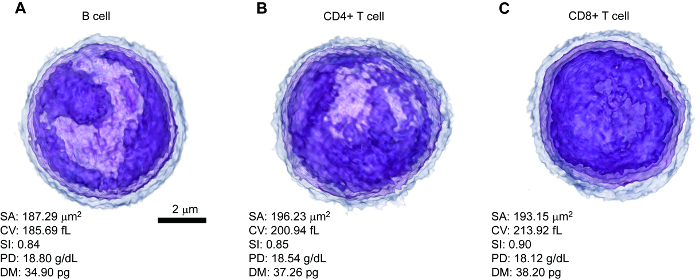
Figura 2 : Representante 3 D prestados los tomogramas RI de cada tipo de célula linfocito con características morfológicas y bioquímicas cuantitativas. (A) la célula de B, célula b CD4 + T y célula de T (C) CD8 +. Barra de escala = 2 μm. SA, área de la superficie; CV, volumen celular; SI, esfericidad; PD, densidad de proteína; DM, masa seca. Esta cifra se modifica con permiso5. Haga clic aquí para ver una versión más grande de esta figura.
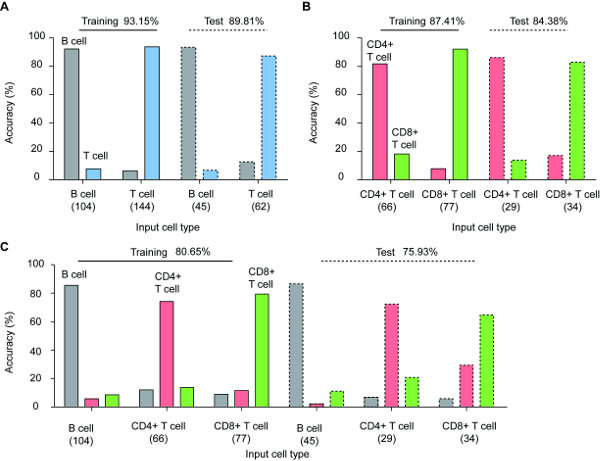
Figura 3 : Tipos de identificación del linfocito individual mediante aprendizaje automático supervisado (A) clasificación binario de B y células T, b binario clasificación de células CD4 + y CD8 + T y (C) multiclase clasificación de los tres tipos de células de linfocitos; para conjuntos de entrenamiento y prueba. Tenga en cuenta la diferencia pequeña entre el entrenamiento y prueba de los casos, sugiriendo buena generalización de los clasificadores establecidos. Los números debajo de los nombres de cada tipo de célula indican el número de células utilizadas. Esta cifra se modifica con permiso5. Haga clic aquí para ver una versión más grande de esta figura.

Archivo complementario 1: cuentan con código de extracción. Extracción de características (SA, CV, SI, PD y DM) después de segmentación basadas en umbrales de RI de cada tomograma. Implementado en un software de procesamiento de imágenes. Haga clic aquí para descargar este archivo.

Archivo complementario 2: código de formación. Un k -NN clasificador entrenamiento basado en las características de la formación. Implementado en un software de procesamiento de imágenes. Haga clic aquí para descargar este archivo.

Archivo complementario 3: código de prueba. Un clasificador de formación k- NN para un nuevo conjunto de datos de prueba (es decir., unidad de prueba). Implementado en un software de procesamiento de imágenes. Haga clic aquí para descargar este archivo.
Discusión
Presentamos un protocolo que permite la identificación de etiqueta-libre de tipos de linfocito, aprovechando la proyección de imagen 3D fase cuantitativa y el aprender de máquina. Pasos críticos de este protocolo son la selección de la proyección de imagen y función fase cuantitativa. Para la proyección de imagen olográfica óptima, la densidad de células debe ser controlada como se describe anteriormente. Estabilidad mecánica de las células es importante para obtener una distribución precisa de RI 3D porque movimientos celulares flotantes o vibracionales disturbará las mediciones de holograma sobre cambios de ángulo de iluminación. Por lo tanto esperamos varios minutos hasta que la muestra se convirtió en estable y estática en la cámara de proyección de imagen antes de medir hologramas. Por último, las burbujas dentro de la cámara de proyección de imagen son problemáticas al medir hologramas debido a las diferencias de RI entre el aire y la muestra; así, la muestra debe colocarse cuidadosamente a la cámara de proyección de imagen.
Selección y extracción de características ayudan a determinar el rendimiento de la identificación del clasificador. Se calcularon 5 cuantitativos morfológicos (CV, SA, SI) y bioquímico (PD, DM) características de la distribución 3D de RI en 20 distintos valores de umbral de RI; así, se obtuvieron un total de 100 características. Buscamos exhaustivamente óptimas combinaciones característica y clasificador, que demuestran que la exactitud mejor validación cruzada fue seleccionada. Probamos 6 algoritmos de aprendizaje de máquina diferentes, incluyendo k -NN (k = 4 y k = 6), análisis de discriminación lineal, análisis de discriminación cuadrática, Naive Bayes y árbol de decisión y encontró que k -NN (k = 4 ) mostró el mejor comportamiento de identificación. Sin embargo, es una oportunidad para mejorar la exactitud de identificación utilizando otros métodos, incluyendo máquinas de vector soporte y redes neuronales de aprendizaje de máquina.
Este protocolo mide propiedades ópticas intrínsecas mediante imágenes 3D fase cuantitativa con el fin de identificar los tipos de linfocitos; por lo tanto, no requiere un proceso de etiquetado basado en reacciones antígeno-anticuerpo utilizadas en fluorescencia o magnéticas grano técnicas basadas en clasificación de células, que tienen riesgos de alterar la función celular mediante la modificación de las estructuras de la proteína de la membrana. Por otra parte, el presente método mide distribución de RI 3D y ofrece 3D información morfológica y bioquímica de la célula, que no puede obtenerse de un solo tiro holografía método46; por lo tanto, el rendimiento de la identificación del protocolo es más preciso debido a la información multidimensional.
Una limitación menor de este protocolo es el ajuste manual de la etapa de muestra y etiquetado proceso de aprendizaje automático supervisado. Buscamos un linfocito mediante el ajuste de la etapa de traslación manual y medidas hologramas, que son los pasos más desperdiciadora de tiempo. Esta limitación se mejorarían mediante el empleo de una etapa motorizada automática o dispositivos de canal microfluídico. En cuanto a aprendizaje supervisado, el tipo de linfocitos conocido se necesitan para establecer el clasificador óptimo; por lo tanto, tuvimos que primero aislar e identificar los tipos de la célula del linfocito basados en la técnica de clasificación basada en el antígeno-anticuerpo. Sin embargo, este protocolo todavía utiliza el contraste óptico intrínseco de los linfocitos, y los agentes Etiquetadoras que se utiliza para especificar los anticuerpos tienen efectos insignificantes en la señal medida de RI 3D. Por lo tanto, el clasificador establecido puede utilizarse para identificar los linfocitos de una manera libre de etiqueta.
Aunque este protocolo utiliza principalmente fenotipos de linfocitos mediante la medición 3D RI tomogramas de células individuales, estos datos RI 3D también pueden utilizarse en combinación con otras modalidades de tratamiento genotipos y proteómicos información para mejor clasificación de subtipos. Recientemente, técnicas de microscopía correlativa combinando imágenes por fluorescencia y QPI han sido introducidas47,48,49. El enfoque presentado en este protocolo también puede extenderse a estos métodos de proyección de imagen correlativos.
Libre de etiqueta de identificación de tipos de linfocitos puede aplicarse para estudiar la fisiopatología o diagnosticar la enfermedad mediante la detección de linfocitos anormales o relaciones entre los tipos de linfocitos. Además, este protocolo se puede aplicar al análisis de sangre entera mediante la identificación de diversas células, incluyendo células de sangre rojas, plaquetas y glóbulos blancos.
Divulgaciones
Prof. Y. Park, Y. Jo, Y. S. Kim y S. Lee tienen intereses financieros en Tomocube, Inc., una empresa que comercializa difracción óptica tomografía y fase cuantitativa instrumentos de la proyección de imagen y es uno de los patrocinadores de la obra.
Agradecimientos
Este trabajo fue apoyado por el KAIST BK21 + programa, Tomocube, Inc. y la nacional investigación Fundación de Corea (2015R1A3A2066550, 2017M3C1A3013923, K 2018 000396). Y. Jo reconoce apoyo de la beca presidencial KAIST y becas de ciencia biomédica de la Fundación de Asan.
Materiales
| Name | Company | Catalog Number | Comments |
| Mouse | Daehan Biolink | C57BL/6J mice | gender and age-matched, 6 – 8 weeks |
| Falcon conical centrifuge tube | ThermoFisher Scientific | 14-959-53A | 15 mL |
| Phosphate-buffered saline | Sigma-Aldrich | 806544-500ML | |
| Ammonium-chloride-potassium lysing buffer | ThermoFisher Scientific | A1049201 | |
| RPMI-1640 medium | Sigma-Aldrich | R8758 | |
| Fetal bovine serum | ThermoFisher Scientific | 10438018 | |
| Antibody | BD Biosciences | 553140 (RRID:AB_394655) | CD16/32 (clone 2.4G2) |
| Antibody | BD Biosciences | 555275 (RRID:AB_395699) | CD3ε (clone 17A2) |
| Antibody | Biolegnd | 100734 (RRID:AB_2075238) | CD8α (clone 53-6.7) |
| Antibody | BD Biosciences | 557655 (RRID:AB_396770) | CD19 (clone 1D3) |
| Antibody | BD Biosciences | 557683 (RRID:AB_396793) | CD45R/B220 (clone RA3-6B2) |
| Antibody | BD Biosciences | 552878 (RRID:AB_394507) | NK1.1 (clone PK136) |
| Antibody | eBioscience | 11-0041-85 (RRID:AB_464893) | CD4 (clone GK1.5) |
| DAPI | Roche | 10236276001 | 4,6-diamidino-2-phenylindole |
| Flow cytometry | BD Biosciences | Aria II or III | |
| Imaging chamber | Tomocube, Inc. | TomoDish | |
| Holotomography | Tomocube, Inc. | HT-1H | |
| Holotomography imaging software | Tomocube, Inc. | TomoStudio | |
| Image professing software | MathWorks | Matlab R2017b |
Referencias
- Alizadeh, A. A., et al. Distinct types of diffuse large B-cell lymphoma identified by gene expression profiling. Nature. 403 (6769), 503 (2000).
- Von Boehmer, H., Melchers, F. Checkpoints in lymphocyte development and autoimmune disease. Nature Immunology. 11 (1), 14 (2010).
- Sáez-Cirión, A., et al. HIV controllers exhibit potent CD8 T cell capacity to suppress HIV infection ex vivo and peculiar cytotoxic T lymphocyte activation phenotype. Proceedings of the National Academy of Sciences. 104 (16), 6776-6781 (2007).
- Fischer, K., et al. Isolation and characterization of human antigen-specific TCRαβ+ CD4-CD8-double-negative regulatory T cells. Blood. 105 (7), 2828-2835 (2005).
- Yoon, J., et al. Identification of non-activated lymphocytes using three-dimensional refractive index tomography and machine learning. Scientific Reports. 7 (1), 6654 (2017).
- Popescu, G. . Quantitative phase imaging of cells and tissues. , (2011).
- Lee, K., et al. Quantitative phase imaging techniques for the study of cell pathophysiology: from principles to applications. Sensors. 13 (4), 4170-4191 (2013).
- Kim, D., et al. Refractive index as an intrinsic imaging contrast for 3-D label-free live cell imaging. bioRxiv. , 106328 (2017).
- Kim, K., et al. Optical diffraction tomography techniques for the study of cell pathophysiology. Journal of Biomedical Photonics & Engineering. 2 (2), (2016).
- Wolf, E. Three-dimensional structure determination of semi-transparent objects from holographic data. Optics Communications. 1 (4), 153-156 (1969).
- Kus, A., Dudek, M., Kemper, B., Kujawinska, M., Vollmer, A. Tomographic phase microscopy of living three-dimensional cell cultures. Journal of Biomedical Optics. 19 (4), 046009 (2014).
- Kim, T., et al. White-light diffraction tomography of unlabelled live cells. Nature Photonics. 8 (3), 256 (2014).
- Simon, B., et al. Tomographic diffractive microscopy with isotropic resolution. Optica. 4 (4), 460-463 (2017).
- Kim, Y., et al. Profiling individual human red blood cells using common-path diffraction optical tomography. Scientific Reports. 4, (2014).
- Park, H., et al. Measuring cell surface area and deformability of individual human red blood cells over blood storage using quantitative phase imaging. Scientific Reports. 6, (2016).
- Lee, S., et al. Refractive index tomograms and dynamic membrane fluctuations of red blood cells from patients with diabetes mellitus. Scientific Reports. 7, (2017).
- Merola, F., et al. Tomographic flow cytometry by digital holography. Light-Science & Applications. 6, (2017).
- Park, Y., et al. Refractive index maps and membrane dynamics of human red blood cells parasitized by Plasmodium falciparum. Proceedings of the National Academy of Sciences. 105, 13730-13735 (2008).
- Park, H., et al. Characterizations of individual mouse red blood cells parasitized by Babesia microti using 3-D holographic microscopy. Scientific Reports. 5, 10827 (2015).
- Chandramohanadas, R., et al. Biophysics of malarial parasite exit from infected erythrocytes. Public Library of Science ONE. 6 (6), 20869 (2011).
- Yoon, J., et al. Label-free characterization of white blood cells by measuring 3D refractive index maps. Biomedical Optics Express. 6 (10), 3865-3875 (2015).
- Kim, K., et al. Three-dimensional label-free imaging and quantification of lipid droplets in live hepatocytes. Scientific Reports. 6, 36815 (2016).
- Kim, D., et al. Label-free high-resolution 3-D imaging of gold nanoparticles inside live cells using optical diffraction tomography. Methods. , (2017).
- Lenz, P., et al. Multimodal Quantitative Phase Imaging with Digital Holographic Microscopy Accurately Assesses Intestinal Inflammation and Epithelial Wound Healing. Journal of Visualized Experiments. (115), (2016).
- Huang, J., Guo, P., Moses, M. A. A Time-lapse, Label-free, Quantitative Phase Imaging Study of Dormant and Active Human Cancer Cells. Journal of Visualized Experiments. (132), (2018).
- Yang, S. A., Yoon, J., Kim, K., Park, Y. Measurements of morphological and biochemical alterations in individual neuron cells associated with early neurotoxic effects in Parkinson's disease. Cytometry Part A. 91 (5), 510-518 (2017).
- Cotte, Y., et al. Marker-free phase nanoscopy. Nature Photonics. 7 (2), 113-117 (2013).
- Nguyen, T. H., Kandel, M. E., Rubessa, M., Wheeler, M. B., Popescu, G. Gradient light interference microscopy for 3D imaging of unlabeled specimens. Nature Communications. 8 (1), 210 (2017).
- Kwon, S., et al. Mitochondria-targeting indolizino [3, 2-c] quinolines as novel class of photosensitizers for photodynamic anticancer activity. European Journal of Medicinal Chemistry. 148, 116-127 (2018).
- Bennet, M., Gur, D., Yoon, J., Park, Y., Faivre, D. A Bacteria-Based Remotely Tunable Photonic Device. Advanced Optical Materials. , (2016).
- Kim, T. I., et al. Antibacterial Activities of Graphene Oxide-Molybdenum Disulfide Nanocomposite Films. ACS Applied Materials & Interfaces. 9 (9), 7908-7917 (2017).
- Bedrossian, M., Barr, C., Lindensmith, C. A., Nealson, K., Nadeau, J. L. Quantifying Microorganisms at Low Concentrations Using Digital Holographic Microscopy (DHM). Journal of Visualized Experiments. (129), (2017).
- Jo, Y., et al. Quantitative Phase Imaging and Artificial Intelligence: A Review. arXiv preprint. , (2018).
- Javidi, B., Moon, I., Yeom, S., Carapezza, E. Three-dimensional imaging and recognition of microorganism using single-exposure on-line (SEOL) digital holography. Optics Express. 13 (12), 4492-4506 (2005).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Optics Express. 23 (12), 15792-15805 (2015).
- Jo, Y., et al. Angle-resolved light scattering of individual rod-shaped bacteria based on Fourier transform light scattering. Scientific Reports. 4, 5090 (2014).
- Jo, Y., et al. Holographic deep learning for rapid optical screening of anthrax spores. Science Advances. 3 (8), 1700606 (2017).
- Mirsky, S. K., Barnea, I., Levi, M., Greenspan, H., Shaked, N. T. Automated analysis of individual sperm cells using stain-free interferometric phase microscopy and machine learning. Cytometry Part A. 91 (9), 893-900 (2017).
- Roitshtain, D., et al. Quantitative phase microscopy spatial signatures of cancer cells. Cytometry Part A. 91 (5), 482-493 (2017).
- Lam, V. K., Nguyen, T. C., Chung, B. M., Nehmetallah, G., Raub, C. B. Quantitative assessment of cancer cell morphology and motility using telecentric digital holographic microscopy and machine learning. Cytometry Part A. , (2017).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences. , (2018).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of Specific Cell Population by Fluorescence Activated Cell Sorting (FACS). Journal of Visualized Experiments. (41), e1546 (2010).
- Takeda, M., Ina, H., Kobayashi, S. Fourier-transform method of fringe-pattern analysis for computer-based topography and interferometry. Journal of the Optical Society of America. 72 (1), 156-160 (1982).
- Debnath, S. K., Park, Y. Real-time quantitative phase imaging with a spatial phase-shifting algorithm. Optics Letters. 36 (23), 4677-4679 (2011).
- Kim, K., et al. High-resolution three-dimensional imaging of red blood cells parasitized by Plasmodium falciparum and in situ hemozoin crystals using optical diffraction tomography. Journal of Biomedical Optics. 19 (1), 011005 (2013).
- Vercruysse, D., et al. Three-part differential of unlabeled leukocytes with a compact lens-free imaging flow cytometer. Lab on a Chip. 15 (4), 1123-1132 (2015).
- Kim, K., et al. Correlative three-dimensional fluorescence and refractive index tomography: bridging the gap between molecular specificity and quantitative bioimaging. Biomedical Optics Express. 8 (12), 5688-5697 (2017).
- Shin, S., Kim, D., Kim, K., Park, Y. Super-resolution three-dimensional fluorescence and optical diffraction tomography of live cells using structured illumination generated by a digital micromirror device. arXiv preprint. , (2018).
- Chowdhury, S., Eldridge, W. J., Wax, A., Izatt, J. A. Structured illumination multimodal 3D-resolved quantitative phase and fluorescence sub-diffraction microscopy. Biomedical Optics Express. 8 (5), 2496-2518 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados