Method Article
Markierungsfreie Identifikation von Lymphozyten-Subtypen mit dreidimensionale Quantitative Phase Bildgebung und maschinelles lernen
In diesem Artikel
Zusammenfassung
Wir beschreiben ein Protokoll für die markierungsfreie Identifizierung von Lymphozyten-Subtypen mit quantitativen Phase Bildgebung und ein maschinelles lernen-Algorithmus. Messungen der 3D Brechungsindex Schichtbilder von Lymphozyten präsentieren 3D morphologische und biochemische Informationen für einzelne Zellen, die dann mit einem Computerlernen Algorithmus zur Identifizierung von Zelltypen analysiert wird.
Zusammenfassung
Wir beschreiben hier ein Protokoll für die markierungsfreie Identifizierung von Lymphozyten-Subtypen mit quantitativen Phase Bildverarbeitung und maschinelles lernen. Identifizierung von Lymphozyten-Subtypen ist wichtig für das Studium der Immunologie sowie Diagnostik und Behandlung von verschiedenen Krankheiten. Standard-Methoden zur Klassifizierung von Lymphozyten Arten setzen derzeit, auf Kennzeichnung bestimmte Membranproteine über Antigen-Antikörper-Reaktionen. Diese Kennzeichnung Techniken tragen jedoch die möglichen Risiken der Zellfunktionen zu verändern. Das hier beschriebene Protokoll überwindet diese Herausforderungen durch die Ausnutzung der intrinsischen optische Kontraste durch 3D quantitativen Phase Bildgebung und eine Maschine Lernalgorithmus gemessen. Messung der 3D Brechungsindex (RI) Schichtbilder von Lymphozyten liefert quantitative Informationen über 3D Morphologie und Phänotypen der einzelnen Zellen. Die biophysikalischen Parameter aus der gemessenen 3D RI Schichtbilder extrahiert werden dann mit einem Algorithmus für maschinelles lernen, die markierungsfreie Identifizierung der Lymphozyten-Typen auf eine einzelne Zelle Ebene ermöglichen quantitativ analysiert. Wir messen die 3D RI Schichtbilder von B, T CD4 + und CD8 + T-Lymphozyten und identifiziert ihre Zelltypen mit über 80 % Genauigkeit. In diesem Protokoll beschreiben wir die einzelnen Schritte zum Lymphozyten isoliert, 3D quantitativen Phase Bildverarbeitung und maschinelles Lernen zur Ermittlung Lymphozyten Typen.
Einleitung
Lymphozyten lassen sich einteilen in verschiedene Subtypen einschließlich B, Helfer (CD4 +) T, zytotoxische (CD8 +) T und regulatorischen T Zellen. Jeder Lymphozyten-Typ hat eine andere Rolle in der adaptiven Immunsystems; zum Beispiel produzieren B-Lymphozyten Antikörper, wobei T-Lymphozyten spezifische Antigene erkennen, Beseitigung von abnormen Zellen und B-Lymphozyten zu regulieren. Lymphozyten Funktion und Regulation ist fest geregelt und im Zusammenhang mit verschiedenen Krankheiten einschließlich Krebs1, Autoimmunerkrankungen2und Virusinfektionen3. So ist die Identifizierung von Lymphozyten Arten wichtig zu verstehen, ihre pathophysiologischen Rollen in solchen Krankheiten und für die Immuntherapie in Kliniken.
Setzen derzeit, Methoden zur Klassifizierung von Lymphozyten Arten auf Antigen-Antikörper-Reaktionen durch gezielte, spezifische Oberfläche Membranproteine oder Oberflächenmarker4. Targeting Oberflächenmarker ist eine exakte und genaue Methode, Lymphozyten Arten zu bestimmen. Es erfordert jedoch teure Reagenzien und zeitaufwändigen Verfahren. Darüber hinaus trägt es Risiken für die Änderung der Membran-Protein-Strukturen und die Veränderung des zellulären Funktionen.
Um diese Herausforderungen zu bewältigen, stellt das hier beschriebene Protokoll die markierungsfreie Identifizierung von Lymphozyten Typen mit 3D quantitativen Phase imaging (QPI) und Machine learning-5. Diese Methode ermöglicht die Klassifizierung der Lymphozyten-Typen auf eine einzellige Ebene basierend auf morphologische Informationen aus markierungsfreie 3D-Bildgebung einzelne Lymphozyten. Im Gegensatz zu herkömmlichen Fluoreszenz-Mikroskopie-Techniken nutzt QPI Brechungsindex (RI) Verteilungen (intrinsische optischen Eigenschaften von lebenden Zellen und Geweben) als optischer Kontrast6,7. Der RI Schichtbilder der einzelnen Lymphozyten darstellen phänotypischen spezifische Informationen für Subtypen von Lymphozyten. In diesem Fall um 3D RI Schichtbilder der einzelnen Lymphozyten systemisch nutzen zu können, wurde ein Algorithmus für betreute maschinelles Lernen genutzt.
Mit verschiedenen QPI-Techniken, die 3D RI Schichtbilder der Zellen aktiv werden seit der Studie der Zelle Pathophysiologie weil sie markierungsfreie, liefern quantitative imaging-Funktion8,9,10, 11,12,13. Darüber hinaus können die 3D RI Verteilungen der einzelnen Zellen morphologische, biochemische und biomechanische über Zellen informieren. 3D RI Schichtbilder wurden früher genutzt, in den Bereichen Hämatologie14,15,16,17, Infektionskrankheiten18,19, 20, Immunologie21, Zelle Biologie22,23, Entzündung24, Krebs25, Neurowissenschaften26,27, Entwicklungsbiologie28, Toxikologie 29und Mikrobiologie12,30,31,32.
Obwohl 3D RI Schichtbilder morphologische und biochemische Zellen ausführlich beschrieben, ist die Klassifizierung der Lymphozyten Subtypen schwer zu erreichen durch einfach bildgebende 3D RI Schichtbilder5. Um systematisch und quantitativ die gemessenen 3D RI Schichtbilder zur Zelle Art Klassifikation auszunutzen, haben wir einen Algorithmus für maschinelles Lernen verwendet. Vor kurzem wurden mehrere Werke beschrieben in welchem quantitativen, die Phase Aufnahmen von Zellen analysiert wurden mit verschiedenen Machine learning Algorithmen33, einschließlich den Nachweis von Mikroorganismen34, Klassifikation der bakteriellen Gattung35 , 36, schnellen und markierungsfreie Nachweis von Anthrax-Sporen-37, automatisierte Analyse von Samenzellen38, Analyse von Krebs Zellen39,40und Erkennung von Makrophagen Aktivierung41.
Dieses Protokoll enthält detaillierte Schritte zum markierungsfreie Identifizierung der Lymphozyten-Typen auf die einzelne Zellenebene mit 3D QPI und maschinelles Lernen führen. Dazu gehören: (1) Lymphozyten isoliert von Maus Blut, 2) Lymphozyten sortieren über Flow Cytometry, 3) 3D QPI 4) quantitative Merkmalsextraktion aus 3D RI Schnittbildern und (5) überwachten Lernen zur Ermittlung Lymphozyten Typen.
Protokoll
Tierbetreuung und experimentelle Verfahren wurden unter Zustimmung des institutionellen Tier Pflege und Nutzung Ausschuss KAIST (KA2010-21, KA2014-01 und KA2015-03) durchgeführt. Alle Versuche in dieser Studie wurden gemäß den genehmigten Richtlinien durchgeführt.
(1) Lymphozyten Isolation aus Maus Blut
- Sobald eine Maus C57BL/6J durch CO2 Einatmen eingeschläfert, 26 G Nadel ins Herz Maus und 0,3 mL Blut zu sammeln. Direkt ins Blut ein Rohr mit 100 U/mL Heparin-Lösung mit 1 mL der Phosphat-gepufferte Kochsalzlösung (PBS) verdünnt.
Hinweis: Alternativ können Lymphozyten aus der Milz isoliert werden. - Zentrifugieren Sie das Rohr auf 400 X g für 5 min bei 4 ° C.
- Das Rohr 0,5 mL Ammonium-Chlorid-Kalium Lysiereinrichtung Puffer hinzu und invertieren Sie es sanft ein paar Mal um die Lösung zu mischen.
- Inkubieren Sie das Rohr bei Raumtemperatur (RT) für 5 min.
- Waschen Sie die Zellen durch Hinzufügen von 4,5 mL PBS und Zentrifugieren bei 400 X g für 5 min bei 4 ° C, zweimal.
- Entfernen Sie den Überstand und Aufschwemmen der Zelle Pellet in 100 µL frisches RPMI-1640 Medium mit 10 % fetalen bovine Serum (FBS).
- Das Rohr für die Sperrung 0,1 µg des Antikörpers CD16/32 (2.4G2) hinzufügen.
- Halten Sie das Rohr auf dem Eis.
(2) flow Cytometry und Sortierung der Lymphozyten-Subtypen
Hinweis: Sortierung Lymphozyten je nach Zelltyp ist wesentlich für den Aufbau der Boden-Wahrheit (d. h. richtige) Zelle geben Sie Beschriftungen zu trainieren und testen Sie eine Zelle Art Klassifizierung im überwachten lernen. Durchflusszytometrie, eine Gold-Standard-Methode wird verwendet, um zu identifizieren und Lymphozyten42zu trennen.
- Machen Sie eine Mischung von Surface Marker Färbung Antikörper in 100 µL frisches RPMI-1640 Medium [mit 10 % FBS, 0,1 µg des CD3e (17A2), CD8a (53-6,7), CD19 (1D 3), CD45R (B220, RA3-6B2) und NK1.1 (PK136)] und 0,25 µg CD4 (GK1.5) Antikörper zu Ziel B, T CD4 + und CD8 + T Lymphozyten.
- Die Zellsuspension (erhältlich im Schritt 1,8) fügen Sie 100 µL der Antikörper-Mischung hinzu.
- Inkubieren Sie für 25 min auf Eis.
- Waschen Sie die Zellen durch Hinzufügen von 5 mL PBS und Zentrifugieren bei 400 X g für 5 min bei 4 ° C, zweimal.
- Aufschwemmen der Zelle Pellet in 5 mL frisches RPMI-1640 Medium mit 10 % FBS und 2,5 µg DAPI (4', 6-Diamidino-2-Phenylindole).
- Sammeln Sie jede Art von Lymphozyten abgesondert mit Durchflusszytometrie mit der Fluoreszenz-Ebenen der oben beschriebenen Marker. Gleichzeitig ausschließen abgestorbene Zellen mit DAPI-Signale.
Hinweis: Ausführliche Protokolle über Flow Cytometry-basierte Zellsortierung gewesen zuvor beschriebenen42.
3. 3D quantitativen Phase Imaging
- Halten Sie die sortierten Lymphozyten auf Eis in den bildgebenden Verfahren, die innerhalb von 5 h (seit Lymphozyten isoliert von der Maus) abgeschlossen sein soll, Zellschäden und biochemischen Veränderungen zu vermeiden.
- Wählen Sie einen sortierte Zelltyp (unter B, T CD4 + und CD8 + T-Lymphozyten) und verdünnen der Probe (in Schritt 2,6), 180 Zellen/µL mit RPMI Medium für optimale Bildgebung Zustand (d. h. eine Zelle pro Feld Einzelansicht).
- Laden Sie 120 μL der verdünnten Probe in einer bildgebenden Kammer durch langsame Injektion. Überprüfen Sie gründlich auf das Vorhandensein von Luftblasen in der bildgebenden Kammer mit der Probe. Treten Bläschen auf, entfernen Sie diese sorgfältig, wie sie die Qualität der Messungen beeinträchtigen werden.
- 3D RI Schichtbilder mit einem kommerziellen 3D quantitativen Phase Mikroskop, oder Holotomography und imaging-Software zu erwerben.
Hinweis: Detaillierte Informationen über den Versuchsaufbau finden Sie in der ursprünglichen Manuskript-5.- Geben Sie einen Tropfen destilliertes Wasser auf das Objektiv des Mikroskops.
- Legen Sie die bildgebende Kammer mit der Probe auf der Bühne der Übersetzung des Mikroskops und einstellen Sie seiner Lage so, dass die Probe mit der Objektivlinse richtet.
- Anpassen der axialen Positionen der Objektive und Kondensator Linsen, indem er Fokus und Oberfläche, bzw. im Register "Kalibrierung" der "Mikroskop" Perspektive der imaging-Software.
- Richten Sie die Ziel und Kondensator-Objektive durch Klicken auf Auto-Modus. Alternativ verwenden Sie den Scan-Modus und passen Sie die Objektive manuell an, damit die Beleuchtung Muster im zentralen Bereich des Sehfeldes lokalisiert sind.
- Zurück in den Normalmodus und passen Sie die Verschiebetisch um eine Zelle in das Blickfeld zu finden.
- Finden Sie die Brennebene durch die axiale Position des Objektivs einstellen. Perfekte Fokussierung macht die Probe-Grenze auf dem Bildschirm visualisiert fast unsichtbar.
- Passen Sie die Übersetzung-Bühne, um einen Standort ohne eine Zelle zu finden.
- Klicken Sie auf kalibrieren um mehrere 2D Hologramme mit unterschiedlicher Beleuchtung Winkel zu messen.
- Passen Sie die Verschiebetisch um eine Zelle in der Mitte des Sehfeldes zu finden.
- Bewegen Sie zur Registerkarte "Erwerb" und klicken Sie auf 3D Snapshot um die Hologramme der Zelle mit der identischen Beleuchtung Winkel zu messen, wie in Schritt 3.4.8 getan.
- Wenn die erfassten Daten im Feld "Data Management" präsentiert werden, mit der rechten Maustaste auf die erfassten Daten und klicken Sie auf Prozess um ein 3D RI-Tomogramm aus der Hologramme, gemessen in den Schritten 3.4.8 und 3.4.10, die Beugung Tomographie Algorithmus zu rekonstruieren 9 , 10 in der imaging-Software implementiert.
- Wiederholen Sie die Schritte 3.4.5-3.4.11 zur Messung von mehr als 100 Zellen um statistische Aussagekraft für maschinelles Lernen zu gewährleisten.
- Alle Bilder, die durch den Schritt 3.4.11 verarbeitet werden. können visualisiert werden. Im Bereich "Data Management" mit der rechten Maustaste der das und klicken Sie auf öffnen, um die Daten zu visualisieren. Bereich "Datenmanager" RI-Tomogramm anklicken. Klicken Sie auf der Registerkarte "Voreinstellungen" auf Laden und doppelklicken Sie auf "lymphocyte.xml", die eine vordefinierte Übertragungsfunktion der imaging Software Tomogramm nach 3D RI Distributionen zu visualisieren ist.
- Wiederholen Sie die Schritte 3.2-3.4, 3D RI Schichtbilder alle Lymphozyten Subtypen zu messen.
4. quantitative morphologische und biochemische Merkmalsextraktion aus 3D RI Schichtbilder
- Legen Sie die tomographischen Daten in einem einzigen Ordner oben gemessen. Die Zelltypen in die Unterordner dieses Ordners aufgeteilt. Bereiten Sie jedes Tomogramm eine einzelne .mat Datei sein.
- Ergänzende Datei 1 - Feature Extraction (geschrieben für ein Bildbearbeitungsprogramm) öffnen.
- Bearbeiten Sie die Zeile 14, vorbereitet im Schritt 4.1 Tomogramm-Ordner zu benennen.
- Bearbeiten Sie Zeile 15, einen Ordner zum Speichern der extrahierten Feature-Daten zu benennen.
- Optional, bearbeiten Sie Zeile 17 RI Schwelle für Merkmalsextraktion Anpassung. Die Standardoption ist 20 RI Schwellenwerte von 1.340-1.378, mit einem Inkrement von 0,002 wie zuvor beschrieben5.
- Führen Sie den Code. Der Code berechnet für jeden Tomogramm im Dataset, fünf Funktionen: Oberflächen-Bereich (SA), zelluläre Volumen (CV), Sphärizität (SI), Protein-Dichte (PD) und Trockenmasse (DM), pro RI Schwelle. Die detaillierte Algorithmen zur Merkmalsextraktion werden an anderer Stelle beschrieben5.
- Um die Merkmalsextraktion zu überwachen, während der Ausführung überprüfen Sie auf dem Bildschirm RI Schwelle-basierte Zelle Segmentierung zu visualisieren.
- Aktivieren Sie den extrahierten Objektdaten als .mat Datei pro Tomogramm, in dem in Schritt 4.4 benannten Ordner gespeichert.
(5) betreutes Lernen und Identifikation
- Nach dem Zufallsprinzip aufgeteilt die Objektdaten in Schritt 4.8 Training (70 %) und Prüfung (30 %)-Sets mit separaten Ordnern erhalten.
- Offenen ergänzende Datei 2 - Zug (geschrieben für ein Bildbearbeitungsprogramm).
- Bearbeiten Sie die Zeile 14, benennen, die die Ausbildung gesetzt, die im Schritt 5.1 vorbereitet.
- Bearbeiten Sie Zeile 16, einen Ordner zum Speichern der ausgebildeten Sichter zu benennen.
- Bearbeiten Sie Zeile 17, einen Dateinamen für die Klassifizierung festlegen.
- Optional, bearbeiten Sie Zeile 19, die Funktionen für die Ausbildung wählen. Die Standardoption wurde wie zuvor angegeben5, verwendet, um die nachstehenden repräsentative Ergebnisse zu erhalten.
- Führen Sie den Code. Mit Hilfe der ausgewählten Funktionen die Ausbildung gesetzt, der Code bildet eine Klassifizierung mit dem k-nächster Nachbar-Algorithmus (k -NN; k = 4) und speichert dann die Klassifizierung (wie in Schritt 5.5 genannt) in den Ordner in Schritt 5.4 bezeichnet.
- Überprüfen Sie auf dem Bildschirm visualisiert die Sichter-Leistung und die Genauigkeit der Kreuzvalidierung.
- Optional, Zug mehrere Klassifikatoren mit verschiedenen Merkmalskombinationen durch Wiederholen der Schritte 5.5-5.7. Wählen Sie dann die Klassifizierung mit der höchsten Genauigkeit der Kreuzvalidierung.
- Öffnen Sie ergänzende Datei 3 - Test (geschrieben für ein Bildbearbeitungsprogramm).
- Bearbeiten Sie die Zeilen 14-15, benennen die trainierten Klassifikator getestet werden.
- Bearbeiten Sie Zeile 17, benennen, die das Test-Set im Schritt 5.1 vorbereitet.
- Führen Sie den Code. Der Sichter, die oben beschriebenen identifiziert die Zelltypen der einzelnen Lymphozyten in den Test.
- Überprüfen Sie auf dem Bildschirm die Identifikation-Leistung und die Prüfgenauigkeit visualisieren.
Ergebnisse
Abbildung 1 zeigt schematisch das gesamte Protokoll. Bei der hier vorgestellten Verfahren wir isolierte B (n = 149), CD4 + T (n = 95), und CD8 + T (n = 112) Lymphozyten. Um Phase und Amplitude Informationen in verschiedenen Winkeln der Beleuchtung zu erhalten, wurden mehrere 2D Hologramme von einzelnen Lymphozyten gemessen durch Veränderung des Winkels der Beleuchtung (von-60 ° bis 60 ° c). In der Regel 50 Hologramme können verwendet werden, um eine 3D RI-Tomogramm zu rekonstruieren, aber die Anzahl der 2D Hologramme kann angepasst werden, unter Berücksichtigung der bildgebenden Geschwindigkeit und Qualität. Amplitude und Phase Informationen von der gemessenen Hologrammen sind mit einem Feld Abruf-Algorithmus basiert auf Fourier Transform43,44abgerufen. Die 3D RI-Tomogramm der einzelnen Lymphozyten wurde aus mehreren abgerufen am Phase und Amplitude 2D-Daten in verschiedenen Winkeln der Beleuchtung mit optischen Beugung Tomographie Algorithmus rekonstruiert. Details der Bildverarbeitung und 3D RI Tomogramm Rekonstruktionsverfahren finden Sie ebenfalls21,45.
Abbildung 2 A-2_C zeigt repräsentative 3D gerenderten RI Schichtbilder von B, T CD4 + und CD8 + T-Lymphozyten durch Zuteilung von verschiedenen Farbschemata nach RI-Werte über die imaging-Software. Von den RI-Werten, quantitative morphologische (SA, Lebenslauf und SI) und biochemische Funktionen (PD und DM) wurden berechnet (Abbildung 2A-2_C). Dieses Ergebnis zeigt deutlich, dass 3D RI Verteilung ermöglicht die Quantitative Analyse von morphologischen sowie biochemische Informationen von Lymphozyten.
Betreute Computerlernen wurde ausgenutzt, um Lymphozyten-Typen auf eine einzelne Zelle Ebene identifizieren. Die gemessenen 3D RI Schichtbilder wurden nach dem Zufallsprinzip in 70 % und 30 % der Ausbildung (B: 104 T: 66 CD4 + und CD8 + t: 77) und Test (B: 45 T: 29 CD4 + und CD8 + T: 35) Datasets, bzw. aufgeteilt. Wir optimieren die Klassifikatoren um die Zelle-Typ-spezifischen Fingerabdrücke in den Merkmalsraum kodiert maximal nutzen zu können. Die Gesamtgenauigkeit, (richtig positiv) Sensitivität und Spezifität, (true negative) wurden durch den Vergleich der Klassifikator vorhergesagt Ergebnisse und Boden-Wahrheit Zelltypen berechnet.
Nachweis der Proof-of-Concept des vorgeschlagenen Protokolls durchgeführt wir betreute maschinelles Lernen auf drei Fälle zu unterscheiden: binäre Klassifikation (i) B- und T-Lymphozyten und T-Lymphozyten-Subtypen (Ii) zwei (CD4 + und CD8 +) und (Iii) Mehrfachklasse Klassifizierung aller Arten von Lymphozyten.
Abbildung 3 zeigt Identifikation Performance optimierte Klassifikatoren für Trainings- und Stufen. Die Genauigkeit der T- und B-Lymphozyten-Klassifikation wurde 93.15 bis 89,81 % für die Ausbildung und die Testfälle, beziehungsweise. Die CD4 + und CD8 + T-Lymphozyten wurden statistisch eingestuft, und die Genauigkeit betrug 87.41 % bzw. 84,38 % für die Ausbildung und Test-Sets. Schließlich betrug die Genauigkeit des Klassifikators multiclass Zelle Typ 80.65 % bzw. 75.93 % für die Ausbildung und Prüfung Stufen.

Abbildung 1 : Schematische Darstellungen der markierungsfreie Identifizierung der Lymphozyten Arten nutzen 3 D quantitative Phase Bildverarbeitung und maschinelles lernen. Bitte klicken Sie hier für eine größere Version dieser Figur.
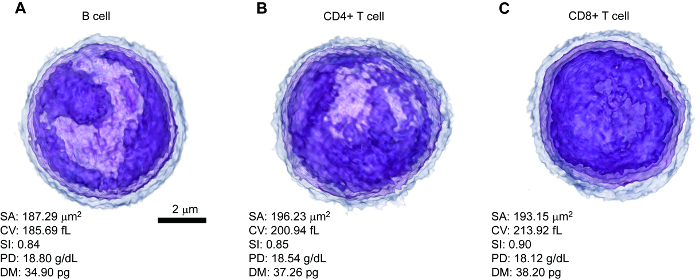
Abbildung 2 : Repräsentative 3 D RI Schichtbilder der jeden Zelltyp Lymphozyten mit quantitativen morphologische und biochemische Eigenschaften gerendert. (A) B-Zell-, (B) CD4 + T-Zellen und (C) CD8 + T-Zellen. Maßstabsleiste = 2 µm. SA, Fläche; CV, zelluläre Volumen; SI, Sphärizität; PD, Protein Dichte; DM, Trockenmasse. Diese Zahl wird mit Erlaubnis5geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
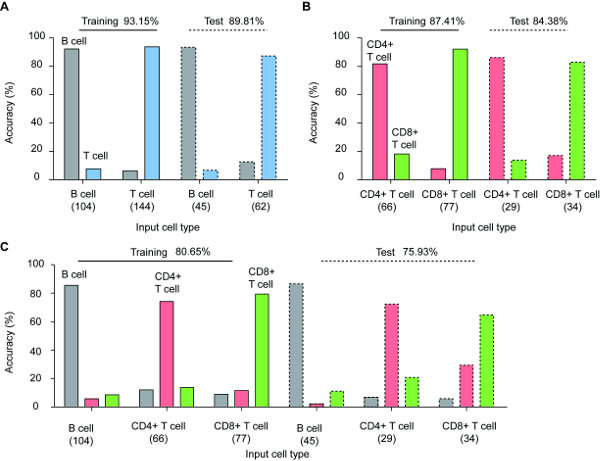
Abbildung 3 : Identifizierung von einzelnen Lymphozyten Typen über betreute maschinelles lernen (A) binäre Klassifikation von B und T-Zellen, (B) binäre Klassifikation der CD4 + und CD8 + T-Zellen und (C) Multiklassenklassifizierung alle drei Lymphozyten Zelltypen; für Training und Test-Sets. Beachten Sie den kleinen Unterschied zwischen Ausbildung und Testfälle, schöne Verallgemeinerung von den etablierten Klassifikatoren vorschlagen. Die Zahlen unter den Namen der einzelnen Zelle geben Sie die Anzahl der verwendeten Zellen. Diese Zahl wird mit Erlaubnis5geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.

Ergänzende Datei 1: Extraktion Code verfügen. Extrahieren von Funktionen (SA, Lebenslauf, SI, PD und DM) nach RI Schwelle-basierte Segmentierung der jedes Tomogramm. Umgesetzt in ein Bildbearbeitungsprogramm. Bitte klicken Sie hier, um diese Datei herunterladen.

Ergänzende Datei 2: Training Code. Training eine k -NN Klassifizierung anhand der ausgewählten Features. Umgesetzt in ein Bildbearbeitungsprogramm. Bitte klicken Sie hier, um diese Datei herunterladen.

Ergänzende Datei 3: Testen von Code. Testen einen ausgebildete k- NN Klassifikator für ein neues Dataset (i.e., Testsatz). Umgesetzt in ein Bildbearbeitungsprogramm. Bitte klicken Sie hier, um diese Datei herunterladen.
Diskussion
Wir präsentieren Ihnen eine Protokoll, die die markierungsfreie Identifizierung von Lymphozyten-Typen, die Nutzung von 3D quantitativen Phase Bildverarbeitung und maschinelles Lernen ermöglicht. Wichtige Schritte dieses Protokolls sind quantitative Phase Bildgebung und Feature-Auswahl. Für die optimale holographische Bildgebung sollte die Dichte der Zellen kontrolliert werden, wie oben beschrieben. Mechanische Stabilität der Zellen ist auch wichtig, eine genaue 3D RI-Verteilung zu erhalten, weil frei verschiebbar oder Schwingungs zelluläre Bewegungen Hologramm Messungen auf Beleuchtung Winkelveränderungen stören werden. Also warteten wir, einige Minuten, bis die Probe stabil und statisch in der bildgebenden Kammer wurde, vor der Messung Hologramme. Zu guter Letzt sind Luftblasen im Inneren der bildgebenden Kammer problematisch, wenn Hologramme auf RI Unterschiede zwischen Luft und der Probe zu messen; die Probe sollte so sorgfältig an die bildgebenden Kammer geladen werden.
Merkmalsextraktion und Auswahl helfen die Identifikation Leistung des Klassifikators zu bestimmen. Wir berechnen 5 quantitative morphologische (CV, SA, SI) und biochemische (PD, DM) Eigenschaften von 3D RI-Distribution bei 20 verschiedenen RI Schwellenwerte; so extrahiert wir insgesamt 100 Funktionen. Wir suchten erschöpfend optimale Funktion und Sichter-Kombinationen, die zeigen, dass die besten Kreuzvalidierung Genauigkeit ausgewählt wurde. Wir haben 6 verschiedene Machine Learning-Algorithmen, einschließlich k -NN getestet (k = 4 und k = 6), lineare Diskriminierung, quadratische Diskriminierung, Naive Bayes, und Entscheidungsstruktur und wir fanden, dass k -NN (k = 4 ) zeigte die beste Leistung der Identifikation. Allerdings gibt es eine Chance zur Verbesserung der Genauigkeit der Identifikation mit anderen Machine learning-Methoden, einschließlich Support Vector Machine und neuronale Netze.
Dieses Protokoll Maßnahmen optischen Eigenschaften über 3D quantitativen Phase imaging um Lymphozyten Arten zu identifizieren; so, es erfordert keine Kennzeichnung beruhend auf Antigen-Antikörper-Reaktionen in Fluoreszenz oder magnetischer Wulst-basierte Zellsortierung Techniken, die Risiken der Veränderung der Zellfunktion durch Ändern von Membran-Protein-Strukturen verwendet. Darüber hinaus ist das vorliegende Verfahren 3D RI Verteilung und 3D morphologische und biochemische informiert über der Zelle, die durch eine Single-Shot-Holographie Methode46erhalten werden kann; Daher ist die Identifizierung Leistung des Protokolls genauer durch hochdimensionale Informationen.
Eine kleine Einschränkung dieses Protokolls ist die manuelle Einstellung der Probe-Bühne und erforderliche Kennzeichnung Prozess für betreute Machine Learning. Wir suchten eine Lymphozyten durch Anpassung der manuellen translationalen Phase und gemessenen Hologramme, die die meiste Zeit Schritte sind. Diese Einschränkung würde durch den Einsatz einer automatisierten motorisierte Phase oder mikrofluidischen Kanal Geräte verbessert werden. Bezug auf überwachten Lernen sind die bekannten Lymphozyten Arten erforderlich, um die optimale Klassifizierung zu etablieren; so, wir mussten erste isolieren und Lymphozyten Zelltypen basiert auf der Antigen-Antikörper-basierte Sortierung Technik zu identifizieren. Dennoch, dieses Protokoll verwendet noch den intrinsischen optischen Kontrast von Lymphozyten, und die Kennzeichnung Agenten verwendet, um Antikörper vernachlässigbare Auswirkungen auf das Messsignal 3D RI. Daher kann der etablierten Sichter zur Identifizierung von Lymphozyten in gewissem Sinne markierungsfreie verwendet werden.
Obwohl dieses Protokoll hauptsächlich Phänotypen von Lymphozyten nutzt durch Messung 3D RI Schichtbilder einzelner Zellen, können diese 3D-Daten RI auch in Kombination mit anderen Modalitäten Genotypen oder Proteomic Informationen für bessere Einstufung der Adressierung verwendet werden Subtypen. Vor kurzem wurden die korrelative Mikroskopiertechniken, die Kombination von Fluoreszenz-Bildgebung und QPI eingeführten47,48,49. Der Ansatz präsentiert in diesem Protokoll kann auch auf diese korrelativen bildgebenden Verfahren ausgedehnt werden.
Markierungsfreie Identifizierung von Lymphozyten Arten kann auf studieren Pathophysiologie oder Diagnose von Krankheiten durch das Erkennen von abnorme Lymphozyten oder Verhältnisse unter den Lymphozyten Typen angewendet werden. Darüber hinaus kann dieses Protokoll auf Vollblut-Analyse angewendet werden, durch die Identifizierung verschiedener Zellen einschließlich der Erythrozyten, Thrombozyten und Leukozyten.
Offenlegungen
Prof. Y. Park, Y.-Jo, Y. S. Kim und S. Lee haben finanzielle Interessen in Tomocube, Inc., ein Unternehmen, das vertreibt optische Beugung Tomographie und quantitativen Phase imaging Instrumente und ist einer der Sponsoren des Werkes.
Danksagungen
Diese Arbeit wurde von der KAIST BK21 + Programm, Tomocube, Inc., und der National Research Foundation of Korea (2015R1A3A2066550, 2017M3C1A3013923, 2018 K 000396) unterstützt. Y. Jo erkennt Unterstützung seitens der KAIST Presidential Fellowship und Asan Stiftung biomedizinische Wissenschaft Stipendium.
Materialien
| Name | Company | Catalog Number | Comments |
| Mouse | Daehan Biolink | C57BL/6J mice | gender and age-matched, 6 – 8 weeks |
| Falcon conical centrifuge tube | ThermoFisher Scientific | 14-959-53A | 15 mL |
| Phosphate-buffered saline | Sigma-Aldrich | 806544-500ML | |
| Ammonium-chloride-potassium lysing buffer | ThermoFisher Scientific | A1049201 | |
| RPMI-1640 medium | Sigma-Aldrich | R8758 | |
| Fetal bovine serum | ThermoFisher Scientific | 10438018 | |
| Antibody | BD Biosciences | 553140 (RRID:AB_394655) | CD16/32 (clone 2.4G2) |
| Antibody | BD Biosciences | 555275 (RRID:AB_395699) | CD3ε (clone 17A2) |
| Antibody | Biolegnd | 100734 (RRID:AB_2075238) | CD8α (clone 53-6.7) |
| Antibody | BD Biosciences | 557655 (RRID:AB_396770) | CD19 (clone 1D3) |
| Antibody | BD Biosciences | 557683 (RRID:AB_396793) | CD45R/B220 (clone RA3-6B2) |
| Antibody | BD Biosciences | 552878 (RRID:AB_394507) | NK1.1 (clone PK136) |
| Antibody | eBioscience | 11-0041-85 (RRID:AB_464893) | CD4 (clone GK1.5) |
| DAPI | Roche | 10236276001 | 4,6-diamidino-2-phenylindole |
| Flow cytometry | BD Biosciences | Aria II or III | |
| Imaging chamber | Tomocube, Inc. | TomoDish | |
| Holotomography | Tomocube, Inc. | HT-1H | |
| Holotomography imaging software | Tomocube, Inc. | TomoStudio | |
| Image professing software | MathWorks | Matlab R2017b |
Referenzen
- Alizadeh, A. A., et al. Distinct types of diffuse large B-cell lymphoma identified by gene expression profiling. Nature. 403 (6769), 503 (2000).
- Von Boehmer, H., Melchers, F. Checkpoints in lymphocyte development and autoimmune disease. Nature Immunology. 11 (1), 14 (2010).
- Sáez-Cirión, A., et al. HIV controllers exhibit potent CD8 T cell capacity to suppress HIV infection ex vivo and peculiar cytotoxic T lymphocyte activation phenotype. Proceedings of the National Academy of Sciences. 104 (16), 6776-6781 (2007).
- Fischer, K., et al. Isolation and characterization of human antigen-specific TCRαβ+ CD4-CD8-double-negative regulatory T cells. Blood. 105 (7), 2828-2835 (2005).
- Yoon, J., et al. Identification of non-activated lymphocytes using three-dimensional refractive index tomography and machine learning. Scientific Reports. 7 (1), 6654 (2017).
- Popescu, G. . Quantitative phase imaging of cells and tissues. , (2011).
- Lee, K., et al. Quantitative phase imaging techniques for the study of cell pathophysiology: from principles to applications. Sensors. 13 (4), 4170-4191 (2013).
- Kim, D., et al. Refractive index as an intrinsic imaging contrast for 3-D label-free live cell imaging. bioRxiv. , 106328 (2017).
- Kim, K., et al. Optical diffraction tomography techniques for the study of cell pathophysiology. Journal of Biomedical Photonics & Engineering. 2 (2), (2016).
- Wolf, E. Three-dimensional structure determination of semi-transparent objects from holographic data. Optics Communications. 1 (4), 153-156 (1969).
- Kus, A., Dudek, M., Kemper, B., Kujawinska, M., Vollmer, A. Tomographic phase microscopy of living three-dimensional cell cultures. Journal of Biomedical Optics. 19 (4), 046009 (2014).
- Kim, T., et al. White-light diffraction tomography of unlabelled live cells. Nature Photonics. 8 (3), 256 (2014).
- Simon, B., et al. Tomographic diffractive microscopy with isotropic resolution. Optica. 4 (4), 460-463 (2017).
- Kim, Y., et al. Profiling individual human red blood cells using common-path diffraction optical tomography. Scientific Reports. 4, (2014).
- Park, H., et al. Measuring cell surface area and deformability of individual human red blood cells over blood storage using quantitative phase imaging. Scientific Reports. 6, (2016).
- Lee, S., et al. Refractive index tomograms and dynamic membrane fluctuations of red blood cells from patients with diabetes mellitus. Scientific Reports. 7, (2017).
- Merola, F., et al. Tomographic flow cytometry by digital holography. Light-Science & Applications. 6, (2017).
- Park, Y., et al. Refractive index maps and membrane dynamics of human red blood cells parasitized by Plasmodium falciparum. Proceedings of the National Academy of Sciences. 105, 13730-13735 (2008).
- Park, H., et al. Characterizations of individual mouse red blood cells parasitized by Babesia microti using 3-D holographic microscopy. Scientific Reports. 5, 10827 (2015).
- Chandramohanadas, R., et al. Biophysics of malarial parasite exit from infected erythrocytes. Public Library of Science ONE. 6 (6), 20869 (2011).
- Yoon, J., et al. Label-free characterization of white blood cells by measuring 3D refractive index maps. Biomedical Optics Express. 6 (10), 3865-3875 (2015).
- Kim, K., et al. Three-dimensional label-free imaging and quantification of lipid droplets in live hepatocytes. Scientific Reports. 6, 36815 (2016).
- Kim, D., et al. Label-free high-resolution 3-D imaging of gold nanoparticles inside live cells using optical diffraction tomography. Methods. , (2017).
- Lenz, P., et al. Multimodal Quantitative Phase Imaging with Digital Holographic Microscopy Accurately Assesses Intestinal Inflammation and Epithelial Wound Healing. Journal of Visualized Experiments. (115), (2016).
- Huang, J., Guo, P., Moses, M. A. A Time-lapse, Label-free, Quantitative Phase Imaging Study of Dormant and Active Human Cancer Cells. Journal of Visualized Experiments. (132), (2018).
- Yang, S. A., Yoon, J., Kim, K., Park, Y. Measurements of morphological and biochemical alterations in individual neuron cells associated with early neurotoxic effects in Parkinson's disease. Cytometry Part A. 91 (5), 510-518 (2017).
- Cotte, Y., et al. Marker-free phase nanoscopy. Nature Photonics. 7 (2), 113-117 (2013).
- Nguyen, T. H., Kandel, M. E., Rubessa, M., Wheeler, M. B., Popescu, G. Gradient light interference microscopy for 3D imaging of unlabeled specimens. Nature Communications. 8 (1), 210 (2017).
- Kwon, S., et al. Mitochondria-targeting indolizino [3, 2-c] quinolines as novel class of photosensitizers for photodynamic anticancer activity. European Journal of Medicinal Chemistry. 148, 116-127 (2018).
- Bennet, M., Gur, D., Yoon, J., Park, Y., Faivre, D. A Bacteria-Based Remotely Tunable Photonic Device. Advanced Optical Materials. , (2016).
- Kim, T. I., et al. Antibacterial Activities of Graphene Oxide-Molybdenum Disulfide Nanocomposite Films. ACS Applied Materials & Interfaces. 9 (9), 7908-7917 (2017).
- Bedrossian, M., Barr, C., Lindensmith, C. A., Nealson, K., Nadeau, J. L. Quantifying Microorganisms at Low Concentrations Using Digital Holographic Microscopy (DHM). Journal of Visualized Experiments. (129), (2017).
- Jo, Y., et al. Quantitative Phase Imaging and Artificial Intelligence: A Review. arXiv preprint. , (2018).
- Javidi, B., Moon, I., Yeom, S., Carapezza, E. Three-dimensional imaging and recognition of microorganism using single-exposure on-line (SEOL) digital holography. Optics Express. 13 (12), 4492-4506 (2005).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Optics Express. 23 (12), 15792-15805 (2015).
- Jo, Y., et al. Angle-resolved light scattering of individual rod-shaped bacteria based on Fourier transform light scattering. Scientific Reports. 4, 5090 (2014).
- Jo, Y., et al. Holographic deep learning for rapid optical screening of anthrax spores. Science Advances. 3 (8), 1700606 (2017).
- Mirsky, S. K., Barnea, I., Levi, M., Greenspan, H., Shaked, N. T. Automated analysis of individual sperm cells using stain-free interferometric phase microscopy and machine learning. Cytometry Part A. 91 (9), 893-900 (2017).
- Roitshtain, D., et al. Quantitative phase microscopy spatial signatures of cancer cells. Cytometry Part A. 91 (5), 482-493 (2017).
- Lam, V. K., Nguyen, T. C., Chung, B. M., Nehmetallah, G., Raub, C. B. Quantitative assessment of cancer cell morphology and motility using telecentric digital holographic microscopy and machine learning. Cytometry Part A. , (2017).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences. , (2018).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of Specific Cell Population by Fluorescence Activated Cell Sorting (FACS). Journal of Visualized Experiments. (41), e1546 (2010).
- Takeda, M., Ina, H., Kobayashi, S. Fourier-transform method of fringe-pattern analysis for computer-based topography and interferometry. Journal of the Optical Society of America. 72 (1), 156-160 (1982).
- Debnath, S. K., Park, Y. Real-time quantitative phase imaging with a spatial phase-shifting algorithm. Optics Letters. 36 (23), 4677-4679 (2011).
- Kim, K., et al. High-resolution three-dimensional imaging of red blood cells parasitized by Plasmodium falciparum and in situ hemozoin crystals using optical diffraction tomography. Journal of Biomedical Optics. 19 (1), 011005 (2013).
- Vercruysse, D., et al. Three-part differential of unlabeled leukocytes with a compact lens-free imaging flow cytometer. Lab on a Chip. 15 (4), 1123-1132 (2015).
- Kim, K., et al. Correlative three-dimensional fluorescence and refractive index tomography: bridging the gap between molecular specificity and quantitative bioimaging. Biomedical Optics Express. 8 (12), 5688-5697 (2017).
- Shin, S., Kim, D., Kim, K., Park, Y. Super-resolution three-dimensional fluorescence and optical diffraction tomography of live cells using structured illumination generated by a digital micromirror device. arXiv preprint. , (2018).
- Chowdhury, S., Eldridge, W. J., Wax, A., Izatt, J. A. Structured illumination multimodal 3D-resolved quantitative phase and fluorescence sub-diffraction microscopy. Biomedical Optics Express. 8 (5), 2496-2518 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten