Method Article
Machine Learning et sans étiquette d’Identification des sous-types de lymphocytes à l’aide de l’imagerie tridimensionnelle Phase Quantitative
Dans cet article
Résumé
Les auteurs décrivent un protocole d’identification des sous-types de lymphocytes en utilisant l’imagerie quantitative phase et un algorithme d’apprentissage machine exempte d’étiquette. Mesures de l’indice de réfraction 3D tomographies des lymphocytes présentent 3D informations morphologiques et biochimiques des cellules individuelles, qui sont ensuite analysées grâce à un algorithme d’apprentissage automatique pour l’identification des types de cellules.
Résumé
Nous décrivons ici un protocole d’identification exempte d’étiquette des sous-types de lymphocytes en utilisant l’imagerie quantitative phase et apprentissage automatique. Identification des sous-types de lymphocytes est importante pour l’étude de l’immunologie ainsi que diagnostic et le traitement de diverses maladies. Actuellement, des méthodes standard pour la classification des types de lymphocytes s’appuient sur l’étiquetage des protéines membranaires spécifiques via des réactions antigène-anticorps. Toutefois, ces techniques d’étiquetage portent les risques potentiels d’altérer les fonctions cellulaires. Le protocole décrit ici permet de surmonter ces défis en exploitant les contrastes optiques intrinsèques, mesurés par imagerie 3D volet quantitatif et un algorithme d’apprentissage automatique. Mesure des tomographies 3D indice de réfraction (RI) des lymphocytes fournit des informations quantitatives sur la morphologie 3D et les phénotypes des cellules individuelles. Les paramètres biophysiques extraites des tomographies de RI 3D mesurées sont ensuite quantitativement analysés avec un algorithme d’apprentissage machine permettant une identification des types de lymphocytes au niveau unicellulaire exempte d’étiquette. Nous mesurons les tomographies RI 3D des lymphocytes B, T CD4 + et CD8 + T et identifié leurs types de cellules avec plus de 80 % exactitude. Dans ce protocole, nous décrivons la procédure détaillée pour l’isolement de lymphocytes, l’imagerie 3D volet quantitatif et apprentissage automatique pour l’identification des types de lymphocytes.
Introduction
Les lymphocytes peuvent être classés en différents sous-types dont B, d’assistance (CD4 +) T cytotoxiques (CD8 +) T et T régulateurs des cellules. Chaque type de lymphocyte a un rôle différent dans le système immunitaire adaptatif ; par exemple, les lymphocytes B produisent des anticorps, alors que les lymphocytes T détecter les antigènes spécifiques, d’éliminer les cellules anormales et réglementent les lymphocytes B. Règlement et fonction lymphocytaire est étroitement contrôlée par et associés à diverses maladies dont les cancers1et maladies auto-immunes2infections virales3. Ainsi, l’identification des types de lymphocytes est importante de comprendre leurs rôles physiopathologiques de ces maladies et d’immunothérapie dans les cliniques.
Actuellement, les méthodes de classification des types de lymphocytes s’appuient sur réactions antigène-anticorps en ciblant les protéines de la membrane de surface spécifique ou de marqueurs de surface4. Ciblage des marqueurs de surface est une méthode précise et exacte pour déterminer les types de lymphocytes. Cependant, elle nécessite des réactifs coûteux et procédures fastidieuses. En outre, il comporte des risques de la modification des structures de protéines membranaires et la modification des fonctions cellulaires.
Pour surmonter ces défis, le protocole décrit ici présente l’identification exempte d’étiquette des types de lymphocytes à l’aide de 3D phase quantitative imaging (QPI) et5en apprentissage automatique. Cette méthode permet la classification des types de lymphocytes au niveau cellule unique basée sur l’information morphologique extraite de l’imagerie 3D exempte d’étiquette de lymphocytes individuels. Contrairement aux techniques de microscopie de fluorescence conventionnels, QPI utilise l’indice de réfraction (RI) des distributions (propriétés optiques intrinsèques des tissus et de cellules vivantes) comme contraste optique6,7. Les tomographies RI des lymphocytes individuels représentent des informations phénotypiques spécifiques aux sous-types des lymphocytes. Dans ce cas, pour pouvoir systématiquement utiliser 3D RI tomographies des lymphocytes individuels, un algorithme d’apprentissage supervisé machine a été utilisé.
En utilisant diverses techniques QPI, les 3D tomographies RI des cellules ont été activement utilisées pour l’étude de la physiopathologie de la cellule parce qu’ils fournissent une étiquette libre, quantitative imaging capacité8,9,10, 11,12,13. Aussi, les distributions de RI 3D de cellules individuelles peuvent fournir des informations morphologiques, biochimiques et biomécaniques sur les cellules. 3D RI tomographies ont été précédemment utilisés dans les domaines de l’hématologie14,15,16,17, maladies infectieuses18,19, 20, immunologie21, cell biology22,23, inflammation24, cancer25, neuroscience26,,27,28de la biologie du développement, toxicologie 29et microbiologie12,30,31,32.
Bien que 3D RI tomographies fournissent des détails morphologiques et biochimiques des cellules, la classification des sous-types de lymphocytes est difficile à réaliser par simplement d’imagerie 3D RI tomographies5. Systématiquement et quantitativement exploiter les tomographies de RI 3D mesurées pour la classification de type de cellule, nous avons utilisé un algorithme d’apprentissage machine. Récemment, plusieurs ouvrages ont été signalés, dans quelle phase quantitative des images de cellules ont été analysées avec divers machine learning algorithmes33, y compris la détection des microorganismes34, classification du genre bactérien35 , 36, détection rapide et sans étiquette de l’anthrax spores37, automatisé d’analyse des spermatozoïdes38, analyse des cellules de cancer39,40et la détection de macrophage activation41.
Ce protocole prévoit la procédure détaillée pour effectuer exempte d’étiquette d’identification des types de lymphocytes au niveau des cellules individuelles à l’aide de 3D QPI et apprentissage automatique. Cela inclut : isolement 1) des lymphocytes de sang de souris, lymphocytes 2) Tri par écoulement cytometry, QPI 3) 3D, 4) quantitative trait extraction de 3D RI tomographies et apprentissage 5) supervisé pour identifier les types de lymphocytes.
Protocole
Soins des animaux et des procédures expérimentales ont été effectuées en vertu de l’approbation de l’animalerie institutionnelle et utilisation Comité du KAIST (KA2010-21, KA2014-01 et KA2015-03). Toutes les expériences dans cette étude ont été effectuées conformément aux principes directeurs adoptés.
1. les lymphocytes isolement du sang de souris
- Une fois une souris C57BL/6J est euthanasiée par inhalation de CO2 , insérer une aiguille 26-G dans le coeur de souris et recueillir 0,3 mL de sang. Directement mettre en sang dans un tube avec 100 U/mL de solution d’héparine dilué avec 1 mL de solution saline tamponnée au phosphate (PBS).
Remarque : Les lymphocytes de la rate peuvent également être isolés. - Centrifuger le tube à 400 x g pendant 5 min à 4 ° C.
- Ajouter 0,5 mL de tampon de lyse d’ammonium-chlorure de potassium dans le tube et il retourner doucement quelques fois pour mélanger la solution.
- Incuber les tubes à température ambiante (RT) pendant 5 min.
- Laver les cellules en ajoutant 4,5 mL de PBS et centrifugation à 400 x g pendant 5 min à 4 ° C, deux fois.
- Retirez le surnageant et Resuspendre le culot dans 100 µL de milieu RPMI 1640 frais avec 10 % sérum fœtal (SVF).
- Ajouter 0,1 µg d’anticorps CD16/32 (2.4G2) dans le tube pour le blocage.
- Maintenir le tube sur la glace.
2. régulateur de cytométrie en flux et tri des sous-types de lymphocytes
NOTE : Tri selon le type de cellules, les lymphocytes est essentiel pour l’établissement de la cellule de réalité-terrain (c'est-à-dire correct) tapez les étiquettes pour former et tester un classifieur de type cellulaire en apprentissage supervisé. Cytométrie en flux, une méthode de l’étalon-or, est utilisé pour identifier et séparer les lymphocytes42.
- Faire un mélange de marqueur de surface anticorps dans 100 µL de milieu RPMI 1640 frais de coloration [10 % FBS, 0,1 µg de CD3e (17A2), CD8a (53-6,7), CD19 (1D 3), CD45R (B220, RA3-6 b 2) et NK1.1 (PK136)] et 0,25 µg d’anticorps anti-CD4 (GK1.5) cible B, lymphocytes T CD4 + et CD8 + T lymphocytes humains.
- Ajouter 100 µL du mélange anticorps à la suspension de cellules (obtenue à l’étape 1.8).
- Incuber pendant 25 minutes sur la glace.
- Laver les cellules en ajoutant 5 mL de PBS et centrifugation à 400 x g pendant 5 min à 4 ° C, deux fois.
- Resuspendre le culot dans 5 mL de milieu RPMI 1640 frais avec 10 % FBS et 2,5 µg de DAPI (4', 6-diamidino-2-phénylindole).
- Collecter séparément chaque type de lymphocyte par cytométrie en flux, en utilisant les niveaux de fluorescence des marqueurs décrites ci-dessus. En même temps exclure les cellules mortes en utilisant les signaux DAPI.
Remarque : Les protocoles détaillés concernant l’écoulement cytometry-base cell tri ont été décrits précédemment42.
3. 3D Phase Quantitative Imaging
- Garder les lymphocytes triés sur la glace tout au long des procédures d’imagerie, qui devrait être terminé dans les 5 h (depuis l’isolement de lymphocytes de la souris) pour éviter des dommages aux cellules et les modifications biochimiques.
- Sélectionnez un type de cellules triées (parmi les lymphocytes B, T CD4 + et CD8 + T) et diluer l’échantillon (obtenu à l’étape 2.6) pour 180 cellules/µL avec milieu RPMI de condition d’imagerie optimale (c.-à-d., une cellule par simple field-of-view).
- Charger les 120 μl de l’échantillon dilué dans une chambre d’imagerie par injection lente. Vérifier soigneusement la présence de bulles dans la chambre d’imagerie avec l’échantillon. S’il y a des bulles, soigneusement supprimer, comme ils vont compromettre la qualité des mesures.
- Acquérir des tomographies de RI 3D à l’aide d’un microscope phase quantitatif 3D commercial, ou holotomography et son logiciel d’imagerie.
NOTE : Des informations détaillées sur l’installation expérimentale se trouvent dans le manuscrit original5.- Déposer une goutte d’eau distillée sur le dessus de la lentille de l’objectif du microscope.
- Placez la chambre d’imagerie avec l’échantillon sur l’étape de traduction du microscope et d’ajuster son emplacement afin d’aligner l’échantillon avec la lentille de l’objectif.
- Ajustez les positions axiales des lentilles objectif et condenseur en cliquant sur mise au point et la Surface, respectivement, sur l’onglet « Calibration » des perspectives « Microscope » du logiciel d’imagerie.
- Aligner les lentilles de l’objectif et condenseur en cliquant sur Mode Auto. Vous pouvez également utiliser le Mode balayage et de régler manuellement les lentilles afin que les modèles d’éclairage sont localisées à la région centrale du champ-de-vue.
- Retournez au Mode Normal et ajuster l’étape de traduction pour localiser une cellule dans le champ de vision.
- Trouver le plan focal en ajustant la position axiale de l’objectif. Mise au point parfaite fait la limite de l’échantillon visualisée à l’écran presque invisible.
- Ajuster l’étape de traduction pour trouver l’emplacement sans une cellule.
- Cliquez sur calibrer pour mesurer plusieurs hologrammes 2D avec des angles d’éclairage variables.
- Ajuster l’étape de traduction pour localiser une cellule au centre du champ-de-vue.
- Accédez à l’onglet « Acquisition » et cliquez sur 3D instantané pour mesurer les hologrammes de la cellule avec les angles d’éclairage identique comme fait à l’étape 3.4.8.
- Lorsque les données acquises sont présentées sur le panneau « Gestion des données », faites un clic droit sur les données acquises et cliquez sur le processus pour reconstruire un tomogramme RI 3D depuis les hologrammes mesurée aux étapes 3.4.8 et 3.4.10, à l’aide de l’algorithme de tomographie par diffraction 9 , 10 mises en œuvre dans le logiciel d’imagerie.
- Répétez les étapes 3.4.5-3.4.11 pour mesurer plus de 100 cellules afin d’assurer une puissance statistique pour l’apprentissage de la machine.
- Toutes les images qui sont traitées par l’étape 3.4.11. peuvent être visualisées. Sur le panneau « Gestion des données », cliquez-droit sur les données et cliquez sur Ouvrir pour visualiser les données. Cliquez sur la tomodensitométrie : RI sur le panneau « Data Manager ». Sous l’onglet « Preset », cliquez sur charger et double-cliquez sur « lymphocyte.xml », qui est une fonction de transfert prédéfinis fournie par le logiciel d’imagerie pour visualiser la tomodensitométrie : selon les distributions RI 3D.
- Répétez les étapes 3.2-3.4 à mesurer 3D tomographies RI de tous les sous-types de lymphocytes.
4. quantitatives caractéristique morphologiques et biochimiques Extraction de tomographies RI 3D
- Placez toutes les données tomographiques mesurées au-dessus dans un seul dossier. Séparer les types de cellules dans les sous-dossiers de ce dossier. Préparer chaque tomogramme à un fichier unique .mat .
- Ouvrir fichier complémentaire 1 - Extraction de caractéristique (écrit pour une logiciel de traitement d’image).
- Modifier la ligne 14 de désigner la tomodensitométrie : dossier préparé à l’étape 4.1.
- Modifier la ligne 15 de désigner un dossier pour enregistrer les données extraites de fonctionnalité.
- Vous pouvez également modifier la ligne 17 pour ajuster les paramètres de seuil RI pour extraction de caractéristiques. L’option par défaut est 20 seuils RI de 1,340-1,378, avec un incrément de 0,002 comme décrit plus haut5.
- Exécuter le code. Pour chaque tomogramme dans le dataset, le code calcule cinq caractéristiques : surface de la zone (SA), volume cellulaire (CV), sphéricité (SI), densité de protéines (pp) et masse sèche (DM), par seuil de RI. Les algorithmes détaillées pour l’extraction de caractéristiques sont décrites ailleurs5.
- Afin de contrôler l’extraction de caractéristique, lors de l’exécution, vérifier sur l’écran de visualisation de segmentation cellulaire basées sur des seuils de RI.
- Vérifier les données extraites, en tant que fichier .mat par tomodensitométrie :, enregistré dans le dossier désigné à l’étape 4.4.
5. sous la supervision d’apprentissage et d’Identification
- Au hasard de fractionner les données de fonctionnalité obtenues dans des ensembles d’étape 4.8 pour examen (30 %) et de formation (70 %) avec dossiers séparés.
- Ouvrir dossier complémentaire 2 - Train (écrit pour une logiciel de traitement d’image).
- Modifiez la ligne 14 pour désigner l’ensemble de la formation établi à l’étape 5.1.
- Modifier la ligne 16 pour désigner un dossier pour enregistrer le classificateur formé.
- Modifier la ligne 17 pour définir un nom de fichier pour le classifieur.
- Vous pouvez également modifier la ligne 19 pour sélectionner les fonctionnalités pour la formation. L’option par défaut, tel que spécifié précédemment5, a été utilisée pour obtenir les résultats représentatifs ci-dessous.
- Exécuter le code. En utilisant les fonctions sélectionnées de l’ensemble de la formation, le code entraîne un classifieur avec le k-plus proches de l’algorithme voisin (k -NN ; k = 4), puis enregistre le classificateur (aussi nommé à l’étape 5.5) dans le dossier désigné à l’étape 5.4.
- Vérifier sur l’écran de visualisation de l’exécution de classificateur et l’exactitude de la validation croisée.
- Éventuellement, former plusieurs classifieurs avec combinaisons de caractéristique différente en répétant les étapes 5,5 à 5,7. Puis choisissez le classifieur avec la plus grande précision de la validation croisée.
- Ouvrir le fichier complémentaire 3 - Test (écrit pour une logiciel de traitement d’image).
- Modifier lignes 14-15 pour désigner le classificateur formé à tester.
- Modifiez la ligne 17 pour désigner l’ensemble d’essai établi à l’étape 5.1.
- Exécuter le code. Le classificateur décrit ci-dessus identifie les types de cellule des lymphocytes dans l’ensemble de tests individuels.
- Vérifier sur l’écran de visualisation de la performance de l’identification et l’exactitude du test.
Résultats
Figure 1 montre le processus schématique de l’ensemble du protocole. À l’aide de la procédure présentée ici, nous avons isolé B (n = 149), lymphocytes T CD4 + (n = 95) et T CD8 + (n = 112) les lymphocytes. Pour obtenir des informations de phase et d’amplitude à différents angles d’éclairage, plusieurs hologrammes 2D de chaque lymphocyte ont été mesurés en changeant l’angle d’éclairage (à partir de-60 ° c à 60 °). En règle générale, 50 hologrammes peuvent servir à reconstruire une tomodensitométrie 3D : les RI, mais le nombre d’hologrammes 2D peut être réglé en tenant compte de la vitesse et la qualité d’imagerie. Informations de phase et d’amplitude des hologrammes mesurées sont récupérées en utilisant un algorithme de récupération de domaine basé sur la transformée de Fourier transform43,44. La tomodensitométrie 3D : les RI de chaque lymphocyte a été reconstruite à partir de multiples 2D phase et amplitude informations extraites à différents angles d’éclairement à l’aide d’algorithme de tomographie optique de diffraction. Détails du processus de l’image et la méthode de reconstruction 3D RI tomogramme peuvent être trouvés ailleurs21,45.
Figure 2 A-2_C montre des tomographies de RI rendu 3D représentant des lymphocytes B, T CD4 + et CD8 + T en attribuant les jeux de couleurs différentes selon les valeurs de RI via le logiciel d’imagerie. Partir des valeurs de RI, quantitatives morphologiques (SA, CV et SI) et on a calculé les caractéristiques biochimiques de (PD et DM) (Figure 2,A-2_C). Ce résultat montre clairement que 3D distribution RI permet une analyse quantitative des informations morphologiques et biochimiques des lymphocytes.
Apprentissage supervisé machine a été exploitée pour identifier les types de lymphocytes au niveau unicellulaire. Les tomographies de RI 3D mesurées étaient aléatoirement divisés en 70 % et 30 % de la formation (B: 104, T: 66 de CD4 + et CD8 + T: 77) et test (B: 45, T: 29 de CD4 + et CD8 + T: 35) datasets, respectivement. Nous avons optimisé les classificateurs pour utiliser au maximum les empreintes digitales de cellule-type spécifique codées dans l’espace de fonction. La précision totale, sensibilité (vrais positifs) et la spécificité (vrai négatif) ont été calculés en comparant les résultats prévus par classifieur et types de cellules de réalité-terrain.
Afin de démontrer la preuve de concept du protocole proposé, nous avons effectué d’apprentissage machine supervisée sur trois cas différents : classification binaire de lymphocytes B (i) et T (ii) deux sous-types de lymphocytes T (CD4 + et CD8 +) et (iii) multiclasse Classification de tous les types de lymphocytes.
Figure 3 affiche les performances d’identification des classificateurs optimisés pour stades de formation et de test. L’exactitude de la classification T et les lymphocytes B était 93.15 et 89,81 % pour la formation et les cas de test, respectivement. Les lymphocytes T CD4 + et CD8 + ont été statistiquement classées et la précision était 87.41 % et 84,38 % pour la formation et les ensembles de test, respectivement. Enfin, la précision du classifieur type cellule multiclass était 80.65 % et 75,93 % pour les stades de la formation et de test, respectivement.

Figure 1 : Des diagrammes schématiques de l’identification exempte d’étiquette de types de lymphocytes exploitant 3 Imagerie quantitative phase D et apprentissage machine. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
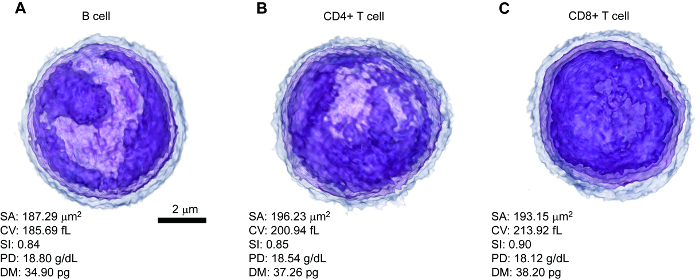
Figure 2 : Représentant 3 D rendu tomographies RI de chaque type de cellules lymphocytes avec des caractéristiques morphologiques et biochimiques quantitatives. (A) les lymphocytes B, des lymphocytes T CD4 (B) et des lymphocytes T CD8 (C) +. Echelle = 2 µm. SA, superficie ; CV, volume cellulaire ; SI, sphéricité ; PD, densité de protéines ; DM, masse sèche. Ce chiffre est modifié avec la permission de5. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
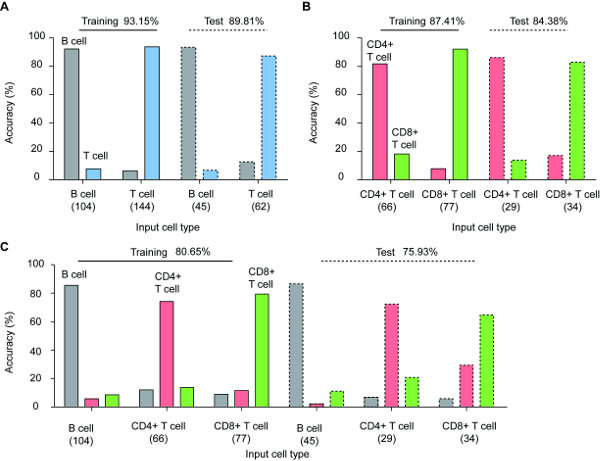
Figure 3 : Identification des lymphocytes différents types par apprentissage supervisé automatique (A) binaire classification des cellules B et T, (B) binaire classification des cellules T CD4 + et CD8 + et c multiclasse classification de tous les trois types de cellules lymphocytes ; pour les jeux de formation et de test. Notez la petite différence entre la formation et les cas de test, suggérant la belle généralisation des classifieurs établies. Les chiffres ci-dessous les noms de chaque type de cellule indiquent le nombre de cellules utilisées. Ce chiffre est modifié avec la permission de5. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Supplémentaire fichier 1 : code d’extraction en vedette. Extraction de caractéristiques (SA, CV, SI, PD et DM) après segmentation de RI basées sur des seuils de chaque tomogramme. Mis en œuvre dans une logiciel de traitement d’image. S’il vous plaît cliquez ici pour télécharger ce fichier.

Complémentaire 2 fichier : code formation. Formation une k -NN classifieur issu des fonctions sélectionnées. Mis en œuvre dans une logiciel de traitement d’image. S’il vous plaît cliquez ici pour télécharger ce fichier.

Supplémentaire 3 fichier : test code. Tester un classifieur NN - formés kpour un nouveau dataset (i.e., l’ensemble de test). Mis en œuvre dans une logiciel de traitement d’image. S’il vous plaît cliquez ici pour télécharger ce fichier.
Discussion
Nous présentons un protocole qui permet l’identification exempte d’étiquette des types de lymphocytes exploitant imagerie 3D volet quantitatif et apprentissage automatique. Les étapes critiques du présent protocole sont volet quantitatif d’imagerie et de la fonctionnalité de sélection. Pour l’imagerie holographique optimale, la densité des cellules doit être contrôlée comme décrit ci-dessus. La stabilité mécanique des cellules est aussi importante d’obtenir une répartition précise des RI 3D parce que des mouvements cellulaires flottantes ou vibrations perturbera mesures hologramme sur les changements d’angle éclairage. Nous avons, par conséquent attendu plusieurs minutes jusqu'à ce que l’échantillon est devenu stable et statique dans la salle d’imagerie avant de mesurer les hologrammes. Enfin, les bulles à l’intérieur de la chambre d’imagerie sont problématiques lorsque vous mesurez des hologrammes en raison des différences de RI entre l’air et de l’échantillon ; ainsi, l’échantillon doit être soigneusement chargé à la chambre d’imagerie.
Extraction de caractéristiques et de sélection à déterminer la performance d’identification du classifieur. Nous avons calculé 5 quantitative de morphologique (SI de CV, SA,) et biochimique (DP, DM) caractéristiques de 3D distribution RI à 20 différentes valeurs de seuil RI ; ainsi, nous avons extrait un total de 100 fonctionnalités. Notre recherche exhaustive des combinaisons caractéristique et classificateur optimales, qui montrent que l’exactitude de la validation croisée meilleur a été sélectionné. Nous avons testé 6 algorithmes d’apprentissage machine différente, y compris k -NN (k = 4 et k = 6), discrimination linéaire analyse, analyse de discrimination quadratique, naïve Bayes et d’arbre de décision et nous a trouvé que k -NN (k = 4 ) a montré les meilleures performances de l’identification. Cependant, il y a une chance d’améliorer l’exactitude d’identification à l’aide d’autre méthodes, y compris la machine de vecteur de soutien et de réseaux de neurones d’apprentissage de la machine.
Ce protocole de mesure des propriétés optiques intrinsèques via 3D volet quantitatif d’imagerie afin d’identifier les types de lymphocytes ; ainsi, il ne nécessite pas un processus d’étiquetage basé sur la réaction antigène-anticorps utilisées en fluorescence ou magnétiques perle techniques basées sur Tri cellulaire, qui ont des risques d’altérer le fonctionnement cellulaire en modifiant les structures des protéines membranaires. En outre, la présente méthode mesure 3D distribution RI et fournit des informations morphologiques et biochimiques 3D sur la cellule, ce qui ne peut être obtenue par une méthode de holographie mono-coup46; par conséquent, la performance d’identification du protocole est plus précise en raison de grande dimension.
Une limitation mineure de ce protocole est le réglage manuel de la scène de l’échantillon et nécessaire processus d’apprentissage supervisé machine d’étiquetage. Nous avons recherché un lymphocyte en ajustant le manuel étape translationnelle et hologrammes mesurées, qui sont les plus longues étapes. Cette limitation s’améliorerait en employant une platine motorisée automatisé ou dispositifs de canaux microfluidiques. Au sujet de l’apprentissage supervisé, les types de lymphocytes connus sont tenus d’établir le classifieur optimal ; ainsi, nous avons dû premier isolat et identifier les types de cellules lymphocytaires, basées sur la technique de tri antigène-anticorps-basée. Néanmoins, ce protocole utilise encore le contraste optique intrinsèque des lymphocytes, et les agents d’étiquetage permet de spécifier les anticorps ont des effets négligeables sur le signal mesuré de RI 3D. Par conséquent, le classifieur établi peut-être servir pour l’identification des lymphocytes dans une manière sans étiquette.
Bien que ce protocole utilise principalement les phénotypes des lymphocytes en mesurant des tomographies RI 3D de cellules individuelles, ces données RI 3D permet également en combinaison avec d’autres modalités traitant des génotypes ou protéomiques informations pour une meilleure classification de sous-types. Récemment, des techniques de microscopie corrélative combinant l’imagerie de fluorescence et QPI ont été introduites47,48,49. L’approche présentée dans le présent protocole peut également être étendue à ces méthodes d’imagerie corrélatives.
Label-free identification des types de lymphocytes peut être appliquée à étudier la physiopathologie ou de diagnostic de la maladie en détectant les lymphocytes anormaux ou les rapports entre les types de lymphocytes. En outre, le présent protocole peut être appliqué à l’analyse de sang en identifiant les diverses cellules dont les globules rouges, des plaquettes et des globules blancs.
Déclarations de divulgation
Prof. Y. Park, Jo Y., Y. S. Kim et S. Lee ont des intérêts financiers dans Tomocube, Inc., une société qui commercialise la tomographie optique de diffraction et volet quantitatif imagérie et est l’un des auteurs de l’ouvrage.
Remerciements
Ce travail a été soutenu par le KAIST BK21 + Program, Tomocube, Inc. et la National Research Foundation of Korea (2015R1A3A2066550, 2017M3C1A3013923, 2018K 000396). Y. Jo reconnaît le soutien du KAIST présidentielle bourse et bourse de sciences biomédicales de la Fondation Asan.
matériels
| Name | Company | Catalog Number | Comments |
| Mouse | Daehan Biolink | C57BL/6J mice | gender and age-matched, 6 – 8 weeks |
| Falcon conical centrifuge tube | ThermoFisher Scientific | 14-959-53A | 15 mL |
| Phosphate-buffered saline | Sigma-Aldrich | 806544-500ML | |
| Ammonium-chloride-potassium lysing buffer | ThermoFisher Scientific | A1049201 | |
| RPMI-1640 medium | Sigma-Aldrich | R8758 | |
| Fetal bovine serum | ThermoFisher Scientific | 10438018 | |
| Antibody | BD Biosciences | 553140 (RRID:AB_394655) | CD16/32 (clone 2.4G2) |
| Antibody | BD Biosciences | 555275 (RRID:AB_395699) | CD3ε (clone 17A2) |
| Antibody | Biolegnd | 100734 (RRID:AB_2075238) | CD8α (clone 53-6.7) |
| Antibody | BD Biosciences | 557655 (RRID:AB_396770) | CD19 (clone 1D3) |
| Antibody | BD Biosciences | 557683 (RRID:AB_396793) | CD45R/B220 (clone RA3-6B2) |
| Antibody | BD Biosciences | 552878 (RRID:AB_394507) | NK1.1 (clone PK136) |
| Antibody | eBioscience | 11-0041-85 (RRID:AB_464893) | CD4 (clone GK1.5) |
| DAPI | Roche | 10236276001 | 4,6-diamidino-2-phenylindole |
| Flow cytometry | BD Biosciences | Aria II or III | |
| Imaging chamber | Tomocube, Inc. | TomoDish | |
| Holotomography | Tomocube, Inc. | HT-1H | |
| Holotomography imaging software | Tomocube, Inc. | TomoStudio | |
| Image professing software | MathWorks | Matlab R2017b |
Références
- Alizadeh, A. A., et al. Distinct types of diffuse large B-cell lymphoma identified by gene expression profiling. Nature. 403 (6769), 503 (2000).
- Von Boehmer, H., Melchers, F. Checkpoints in lymphocyte development and autoimmune disease. Nature Immunology. 11 (1), 14 (2010).
- Sáez-Cirión, A., et al. HIV controllers exhibit potent CD8 T cell capacity to suppress HIV infection ex vivo and peculiar cytotoxic T lymphocyte activation phenotype. Proceedings of the National Academy of Sciences. 104 (16), 6776-6781 (2007).
- Fischer, K., et al. Isolation and characterization of human antigen-specific TCRαβ+ CD4-CD8-double-negative regulatory T cells. Blood. 105 (7), 2828-2835 (2005).
- Yoon, J., et al. Identification of non-activated lymphocytes using three-dimensional refractive index tomography and machine learning. Scientific Reports. 7 (1), 6654 (2017).
- Popescu, G. . Quantitative phase imaging of cells and tissues. , (2011).
- Lee, K., et al. Quantitative phase imaging techniques for the study of cell pathophysiology: from principles to applications. Sensors. 13 (4), 4170-4191 (2013).
- Kim, D., et al. Refractive index as an intrinsic imaging contrast for 3-D label-free live cell imaging. bioRxiv. , 106328 (2017).
- Kim, K., et al. Optical diffraction tomography techniques for the study of cell pathophysiology. Journal of Biomedical Photonics & Engineering. 2 (2), (2016).
- Wolf, E. Three-dimensional structure determination of semi-transparent objects from holographic data. Optics Communications. 1 (4), 153-156 (1969).
- Kus, A., Dudek, M., Kemper, B., Kujawinska, M., Vollmer, A. Tomographic phase microscopy of living three-dimensional cell cultures. Journal of Biomedical Optics. 19 (4), 046009 (2014).
- Kim, T., et al. White-light diffraction tomography of unlabelled live cells. Nature Photonics. 8 (3), 256 (2014).
- Simon, B., et al. Tomographic diffractive microscopy with isotropic resolution. Optica. 4 (4), 460-463 (2017).
- Kim, Y., et al. Profiling individual human red blood cells using common-path diffraction optical tomography. Scientific Reports. 4, (2014).
- Park, H., et al. Measuring cell surface area and deformability of individual human red blood cells over blood storage using quantitative phase imaging. Scientific Reports. 6, (2016).
- Lee, S., et al. Refractive index tomograms and dynamic membrane fluctuations of red blood cells from patients with diabetes mellitus. Scientific Reports. 7, (2017).
- Merola, F., et al. Tomographic flow cytometry by digital holography. Light-Science & Applications. 6, (2017).
- Park, Y., et al. Refractive index maps and membrane dynamics of human red blood cells parasitized by Plasmodium falciparum. Proceedings of the National Academy of Sciences. 105, 13730-13735 (2008).
- Park, H., et al. Characterizations of individual mouse red blood cells parasitized by Babesia microti using 3-D holographic microscopy. Scientific Reports. 5, 10827 (2015).
- Chandramohanadas, R., et al. Biophysics of malarial parasite exit from infected erythrocytes. Public Library of Science ONE. 6 (6), 20869 (2011).
- Yoon, J., et al. Label-free characterization of white blood cells by measuring 3D refractive index maps. Biomedical Optics Express. 6 (10), 3865-3875 (2015).
- Kim, K., et al. Three-dimensional label-free imaging and quantification of lipid droplets in live hepatocytes. Scientific Reports. 6, 36815 (2016).
- Kim, D., et al. Label-free high-resolution 3-D imaging of gold nanoparticles inside live cells using optical diffraction tomography. Methods. , (2017).
- Lenz, P., et al. Multimodal Quantitative Phase Imaging with Digital Holographic Microscopy Accurately Assesses Intestinal Inflammation and Epithelial Wound Healing. Journal of Visualized Experiments. (115), (2016).
- Huang, J., Guo, P., Moses, M. A. A Time-lapse, Label-free, Quantitative Phase Imaging Study of Dormant and Active Human Cancer Cells. Journal of Visualized Experiments. (132), (2018).
- Yang, S. A., Yoon, J., Kim, K., Park, Y. Measurements of morphological and biochemical alterations in individual neuron cells associated with early neurotoxic effects in Parkinson's disease. Cytometry Part A. 91 (5), 510-518 (2017).
- Cotte, Y., et al. Marker-free phase nanoscopy. Nature Photonics. 7 (2), 113-117 (2013).
- Nguyen, T. H., Kandel, M. E., Rubessa, M., Wheeler, M. B., Popescu, G. Gradient light interference microscopy for 3D imaging of unlabeled specimens. Nature Communications. 8 (1), 210 (2017).
- Kwon, S., et al. Mitochondria-targeting indolizino [3, 2-c] quinolines as novel class of photosensitizers for photodynamic anticancer activity. European Journal of Medicinal Chemistry. 148, 116-127 (2018).
- Bennet, M., Gur, D., Yoon, J., Park, Y., Faivre, D. A Bacteria-Based Remotely Tunable Photonic Device. Advanced Optical Materials. , (2016).
- Kim, T. I., et al. Antibacterial Activities of Graphene Oxide-Molybdenum Disulfide Nanocomposite Films. ACS Applied Materials & Interfaces. 9 (9), 7908-7917 (2017).
- Bedrossian, M., Barr, C., Lindensmith, C. A., Nealson, K., Nadeau, J. L. Quantifying Microorganisms at Low Concentrations Using Digital Holographic Microscopy (DHM). Journal of Visualized Experiments. (129), (2017).
- Jo, Y., et al. Quantitative Phase Imaging and Artificial Intelligence: A Review. arXiv preprint. , (2018).
- Javidi, B., Moon, I., Yeom, S., Carapezza, E. Three-dimensional imaging and recognition of microorganism using single-exposure on-line (SEOL) digital holography. Optics Express. 13 (12), 4492-4506 (2005).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Optics Express. 23 (12), 15792-15805 (2015).
- Jo, Y., et al. Angle-resolved light scattering of individual rod-shaped bacteria based on Fourier transform light scattering. Scientific Reports. 4, 5090 (2014).
- Jo, Y., et al. Holographic deep learning for rapid optical screening of anthrax spores. Science Advances. 3 (8), 1700606 (2017).
- Mirsky, S. K., Barnea, I., Levi, M., Greenspan, H., Shaked, N. T. Automated analysis of individual sperm cells using stain-free interferometric phase microscopy and machine learning. Cytometry Part A. 91 (9), 893-900 (2017).
- Roitshtain, D., et al. Quantitative phase microscopy spatial signatures of cancer cells. Cytometry Part A. 91 (5), 482-493 (2017).
- Lam, V. K., Nguyen, T. C., Chung, B. M., Nehmetallah, G., Raub, C. B. Quantitative assessment of cancer cell morphology and motility using telecentric digital holographic microscopy and machine learning. Cytometry Part A. , (2017).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences. , (2018).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of Specific Cell Population by Fluorescence Activated Cell Sorting (FACS). Journal of Visualized Experiments. (41), e1546 (2010).
- Takeda, M., Ina, H., Kobayashi, S. Fourier-transform method of fringe-pattern analysis for computer-based topography and interferometry. Journal of the Optical Society of America. 72 (1), 156-160 (1982).
- Debnath, S. K., Park, Y. Real-time quantitative phase imaging with a spatial phase-shifting algorithm. Optics Letters. 36 (23), 4677-4679 (2011).
- Kim, K., et al. High-resolution three-dimensional imaging of red blood cells parasitized by Plasmodium falciparum and in situ hemozoin crystals using optical diffraction tomography. Journal of Biomedical Optics. 19 (1), 011005 (2013).
- Vercruysse, D., et al. Three-part differential of unlabeled leukocytes with a compact lens-free imaging flow cytometer. Lab on a Chip. 15 (4), 1123-1132 (2015).
- Kim, K., et al. Correlative three-dimensional fluorescence and refractive index tomography: bridging the gap between molecular specificity and quantitative bioimaging. Biomedical Optics Express. 8 (12), 5688-5697 (2017).
- Shin, S., Kim, D., Kim, K., Park, Y. Super-resolution three-dimensional fluorescence and optical diffraction tomography of live cells using structured illumination generated by a digital micromirror device. arXiv preprint. , (2018).
- Chowdhury, S., Eldridge, W. J., Wax, A., Izatt, J. A. Structured illumination multimodal 3D-resolved quantitative phase and fluorescence sub-diffraction microscopy. Biomedical Optics Express. 8 (5), 2496-2518 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon