Method Article
Deteção de colonização Enterohemorrhagic Escherichia Coli murino Host pelo sistema não-invasivo na Vivo bioluminescência
Neste Artigo
Resumo
Um protocolo detalhado de um modelo de mouse para enterohemorrhagic Escherichia coli (EHEC) colonização usando bactérias bioluminescência-etiquetado é apresentado. A detecção destas bactérias bioluminescentes por um não-invasivo na vivo sistema de animais vivos de imagem pode avançar nossa compreensão atual da colonização de EHEC.
Resumo
Enterohemorrhagic Escherichia coli (EHEC) O157: H7, que é um patógeno de origem alimentar que causesdiarrhea, (HS), a colite hemorrágica e Síndrome hemolítico-urêmica (HUS), colonizar para o trato intestinal de seres humanos. Para estudar o mecanismo detalhado da colonização de EHEC in vivo, é essencial ter modelos animais para monitorar e quantificar a colonização de EHEC. Demonstramos aqui um modelo de colonização de rato-EHEC, transformando o plasmídeo expressando bioluminescente de EHEC para monitorar e quantificar a colonização de EHEC em hospedeiros vivos. Animais inoculados com bioluminescência-rotulado EHEC mostram sinais bioluminescentes intensos em camundongos por detecção com um não-invasivo na vivo sistema de imagem. Após 1 a 2 dias pós infecção, sinais bioluminescentes poderiam ainda ser detectados em animais infectados, o que sugere que o EHEC colonizar em hospedeiros pelo menos 2 dias. Também demonstramos que estes EHEC bioluminescentes localizar de intestino de rato, especificamente no ceco e cólon, de imagens ex vivo . Este modelo de colonização de rato-EHEC pode servir como uma ferramenta para fazer avançar o conhecimento atual do mecanismo de colonização EHEC.
Introdução
EHEC O157: H7 é um patógeno que causa diarreia1, HS2, HUS3e insuficiência renal aguda mesmo4 através de água ou alimentos contaminados. EHEC é um enterobacterium patogênico e coloniza para o trato gastrointestinal dos seres humanos1. Quando EHEC primeiro aderir ao epitélio intestinal do hospedeiro, eles injetam os fatores de colonização em células hospedeiras através do sistema de secreção de III do tipo (T3SS) que funciona como uma seringa molecular induzindo um anexar e apagar (A/E) lesão posteriormente para impor adesão (colonização)5. Estes genes envolvidos na formação da lesão de A/E são codificados pelo locus do enterócito (LEE) de apagamento de ilha de patogenicidade5.
Bioluminescência é uma reação química produzindo a luz, na qual luciferase catalisa sua Luciferina de substrato para gerar luz visível6. Este processo enzimático, muitas vezes, requer a presença de oxigênio ou trifosfato de adenosina (ATP)6. Bioluminescência (BLI) de imagem permite que os pesquisadores a visualização e a quantização das interações patógeno-hospedeiro em animais vivos7. BLI pode caracterizar o ciclo de infecção bacteriana em animais vivos, seguindo as bactérias bioluminescentes que migrar para e invadir tecidos diferentes7; Isto revela uma dinâmica progressão da infecção. Além disso, a carga bacteriana nos animais está relacionada com o sinal bioluminescentes8; assim, é um conveniente indicador para estimar as condições patológicas de animais experimentais em uma maneira simples e direta.
O plasmídeo utilizado aqui contidos o operon do luciferase, luxCDABE, que é a partir da bactéria Photorhabdus luminescens que codifica seu próprio substrato de luciferase7,9. Transformando este plasmídeo luciferase-expressando em bactérias, os processos de colonização e infecção podem ser monitorados, observando estas bactérias bioluminescentes em animais vivos. Em geral, BLI e rotulado de bioluminescência bactérias permitem aos investigadores monitorar os números bacterianos e localização, viabilidade bacteriana com antibióticos/terapia tratamento e expressão de gene bacteriano em infecção/colonização6, 7. numerosas bactérias patogênicas têm sido relatadas que expressam o operon luxCDABE para examinar a sua expressão de ciclo e/ou gene infecção na infecção. Estas bactérias, incluindo uropathogenic e. coli10, EHEC8,11,12,13, enteropatogênica Escherichia coli (EPEC)8, Citrobacter rodentium14,15, Salmonella typhimurium16, Listeria monocytogenes17, Yersinia enterocolitica18,19, e Vibrio cholerae20, foram documentados.
Vários modelos experimentais foram desenvolvidos para facilitar o estudo da EHEC colonização in vitro e in vivo21,22,23. No entanto, há uma falta de modelos animais apropriados para estudar o EHEC colonização na vivoe, portanto, uma escassez resultante de detalhes. Para facilitar o estudo da EHEC colonização mecanismo na vivo, que é valioso para construir modelos animais para observar e quantificar a colonização de EHEC em animais vivos em um método não-invasivo.
Este manuscrito descreve um modelo de colonização de rato-EHEC que usa um sistema expressando bioluminescente para monitorar a colonização de EHEC ao longo do tempo em hospedeiros vivos. Ratos intragastrically são inoculados com bioluminescência-rotulado EHEC e o sinal bioluminescente é detectado em ratos com um não-invasivo na vivo de imagem de sistema13. Camundongos infectados com bioluminescência-rotulado EHEC mostrou sinais bioluminescentes significativos em seu intestino, depois de 2 dias pós infecção, que sugeriu que essas bactérias colonizaram no intestino hospedeiro após 2 dias pós infecção. Dados de imagem ex vivo mostram que esta colonização é especificamente no ceco e cólon de ratos. Usando este modelo de rato-EHEC, a colonização de EHEC bioluminescente pode ser detectada no hospedeiro vivo por um na vivo de imagem de sistema, para estudar os mecanismos detalhados da colonização de bactérias entéricas, que pode promover uma compreensão mais em EHEC-induzido por alterações fisiológicas e patológicas.
Protocolo
Cuidado: EHEC O157: H7 é um nível de biossegurança 2 (BSL-2) patógeno de acordo com o Centers for Disease Control and Prevention (CDC) instrução de biossegurança (https://www.cdc.gov/). Portanto, todos os procedimentos experimentais envolvendo EHEC devem ser executados em uma facilidade BSL-2. Use luvas e jalecos de laboratório durante a realização do experimento. Trabalho em uma gabinete de biossegurança certificada (BSC). Desinfete a bancada experimental, antes e após o procedimento experimental com etanol a 70%. Todos os instrumentos ou equipamentos que EHEC contato (ou potencialmente contato) deve ser desinfectado com etanol 70% ou água sanitária. Resíduos contaminados (ou potencialmente contaminados) devem ser selado e esterilizado com cuidado. Use uma máscara, óculos de proteção, luvas duplo ou um macacão, se necessário. Os ratos fêmeas C57BL/6 de 6 semanas de idade foram comprados e mantidos pela Universidade de laboratório Animal centro de Nacional Cheng Kung (NCKU). As experiências com animais foram aprovadas pelo cuidado de Animal institucional e Comitê de uso de NCKU (número de aprovação 104-039).
1. geração de bactéria EHEC Bioluciferase-etiquetado
- Mix 50 ng o Ácido desoxirribonucleico (DNA), 1 μL de cada 10 μm para a frente e reverter as primeiras demão, desoxinucleótidos de 2,5 mM 2 μL (dNTPs), 2 μL de 10x buffer, 0,2 µ l de DNA polimerase e água de bidestilada estéril (ddH2O) para um volume final de 20 μL para amplificar o antigaveta canamicina, de gene nptII 24. O modelo de DNA é de pBSL18024, que é comprado de projeto nacional de BioResource (NBRP). As condições PCR são listadas na tabela 1 e a sequência de cartilha está disponível na tabela 2.
- Ligam os produtos PCR para um vetor de clonagem comercial seguindo o kit protocolo (consulte Tabela de materiais).
- Excisar o fragmento do gene nptII do vetor clonagem por NsiI e SmaI e clonar em pBBR1MCS4, que é digerido pela ScaI e NsiI pAKlux29 -criar o plasmídeo canamicina resistente, pWF27813.
- Excisar o operon luxCDABE , do luciferase expressando de shRNA9, pAKlux2, pela SpeI-HF e ScaI e clone SpeI-HF e SmaI digerido pWF278 para gerar canamicina resistente e luciferase expressando de shRNA, pWF27913.
Nota: Para as extrações do plasmídeo e purificações o fragmento extirpado, use a extração comercial do plasmídeo e o kit de extração do gel, respectivamente. Para a digestão enzimática, misturar 100 ng DNA, 1 μL de buffer x 10, 1 μL de cada enzima de restrição selecionada e ddH estéril2O para um volume total de 10 μL e incubar a 37 ° C para 2,5-3 h. - Transforme o plasmídeo pWF279 em células competentes de Escherichia coli O157: H7 EDL933 por eletroporação com 2.500 V para ms 4.
- Incube as células de bactérias transformadas em 1 mL de meio de Luria-Bertani (LB) a 37 ° C por 1h.
- As bactérias em uma placa de ágar LB suplementado com 50 µ g/mL de canamicina a 37 ° C por 16-18 h da placa.
- Verificar o sinal bioluminescente da placa por um na vivo de imagem máquina no dia seguinte. Escolher uma única colônia da placa e a cultura em 3 mL LB suplementado com 50 canamicina µ g/mL a 37 ° C por 16-18 h.
- Prepare o suporte de ações bacteriana diluindo 100% glicerol em ddH estéril2O tornar a solução de glicerol de 30%.
- Congelar a canamicina resistente bactéria de e. coli O157: H7 EDL933 abrigando o plasmídeo luciferase como um estoque bacteriano em uma tampa de rosca cryovial como uma proporção de 1:1 de solução de glicerol cultura e 30% bactérias a-80 ° C. A concentração final de glicerol é 15%.
2. preparação de bactéria EHEC bioluminescentes para inoculação Oral
Nota: O fluxograma cronograma dos procedimentos experimentais para a preparação de EHEC e gavagem oral rato é apresentado na Figura 1 para auxiliar na preparação experimental.
- Raia e. coli O157: H7 EDL933 as bactérias abrigando plasmídeo luciferase em uma placa de ágar LB com 50 canamicina µ g/mL de estoque-80 ° C. Crescem as bactérias para 16-18 h a 37 ° C.
- Escolha uma única colônia da placa durante a noite e cultura no meio de 3 mL LB com 50 canamicina µ g/mL para 16-18 h em uma incubadora de 37 ° C a 220 rpm.
- Subcultura as bactérias (diluição de 1: 100) em canamicina (50 µ g/mL) contendo caldo LB para 2,5-3 h em uma incubadora de 37 ° C a 200 rpm. (Por exemplo, adicione 2 mL a noite cultivadas bactérias para 200 mL LB suplementado com canamicina (50 µ g/mL)).
- Incubar as bactérias para 2,5-3 h e medir o valor de densidade óptica em 600 nm (OD600) até o valor é entre 0,9 a 1. O número de células bacterianas é em uma densidade de cerca de 108 colônia formando unidades (CFU) / mL.
- Centrifugar as bactérias re-cultivadas em 8.000 x g durante 30 minutos, 4 ° C.
- Desprezar o sobrenadante sem agitar o sedimento de bactérias e lave a pelota com 100 mL 0,9% solução salina normal estéril por agitação suave.
- Repita os passos de 2.5 e 2.6 uma vez.
- Após a lavagem, centrifugar a cultura bacteriana a 8.000 x g por 30 min, 4 ° C e descartar o sobrenadante suavemente.
- Condensado a pelota em 100 vezes com solução salina normal estéril 0,9%. Por exemplo, se são centrifugadas bactérias de 200 mL, adicione salina 2 mL para suspender o sedimento.
- Confirme as bactérias CFU (deve ser aproximadamente 109 UFC/100 μL após a condensação em solução salina normal) com chapeamento concentradas bactérias através de um de 10 vezes de diluição serial25.
3. Mouse gavagem Oral de EHEC
- Trate de camundongos C57BL/6 fêmeas de 6 semanas com água de estreptomicina (5 g/L) por 24 h.
- Após 24h, mude para regular água potável para outro 24h antes de gavage. Após o tratamento com água normal para 24h, os ratos estão prontos para gavagem oral de EHEC.
- Encha a seringa com bactérias EHEC 100 μL, puxando o êmbolo para trás. Não garantir nenhuma bolha de ar na seringa. Remova as bolhas, encaixe a seringa com os dedos.
- Levante o animal delicadamente e coloque-o no topo da gaiola com cuidado.
- Segure o mouse pela cauda cuidadosamente, e o animal vai agarrar o topo da gaiola e tentar te afastar de mim.
- Contenha suavemente o mouse segurando a pele solta do pescoço e costas o animal com o polegar e o indicador para evitar que a cabeça do rato se mova.
- Segure o mouse na posição vertical para inserir a agulha de gavagem e garantir que a cabeça de rato é imobilizado e vertical.
- Insira a agulha de gavagem para a boca do rato após o céu da boca e mover para baixo no esôfago e em direção ao estômago.
- Quando a agulha inserida é metade ou o comprimento de dois terços no mouse, injete 100 bactéria EHEC μL, que contêm aproximadamente 109 células CFU.
4. visualização
- Após a intubação oral, detecta os sinais bioluminescentes em 1 a 2 dias.
- Antes de examinar os sinais bioluminescentes de animais, anestesia, colocando-os em uma câmara de isoflurano em 2,5% 1,5 L/min de oxigênio.
- Espere por 2-5 min até que todos os ratos se tornar inconsciente e parada em movimento. Animais estão prontos para a deteção em vivo de bioluminescência.
- Detectar e imagem bioluminescentes sinais dos animais por um na vivo sistema de imagem. Por favor, veja a seção 5 "Aquisição de dados" para operação de software. Durante o processo de geração de imagens, todos os animais estão sob um fornecimento contínuo de isoflurano em 2,5% 1,5 L/min de oxigênio.
- Clique em arquivo > ao vivo e manualmente o foco do mouse.
- Selecione a intensidade de laser e tempo de exposição.
- Clique em adquirir > capturar.
- Para a imagem latente ex vivo , eutanásia os ratos por deslocamento cervical e retire o intestino inteiro de ratos infectados. Coloque os intestinos em uma placa de Petri-9cm e imagem pelo na vivo sistema de imagem. A configuração é o mesmo que na vivo imagens exceto o campo de visão é definida como A/B. Por favor consulte a seção 5 "Aquisição de dados" para obter detalhes.
Nota: Método por deslocamento cervical: conter os ratos no topo da gaiola agarrando a cauda com uma mão, para que os animais agarrar a gaiola. Coloca uma caneta ou o polegar e o primeiro dedo da outra mão contra o pescoço na base do crânio. Rapidamente empurre para a frente com a mão ou objeto, enquanto a cabeça de restrição e puxar para trás com a mão segurando a cauda.
Olhe com cuidado para confirmar respiratória e sem pulsação.
5. aquisição de dados
Nota: O software utilizado para aquisição de dados é listado na Tabela de materiais.
- Para aquisição de imagem, abra o painel de controle de aquisição do software (Figura 2).
- Selecione "Luminescentes", "fotografia" e "Sobreposição".
- Definir o tempo de exposição, como "Auto". Definir Binning como "Meio".
- Luminescentes e 8 estabelecem ƒ/parar como 1 para fotografia. Ƒ/parar controles a quantidade de luz recebida pelo detector CCD.
- Defina o campo de visão, com base no campo de imagens que são de interesse para adquirir. Opção "D" pode caber cinco ratos de 6 semanas de idade e imagem-los todos em um tempo. "C" pode imagem três ratos de 6 semanas de idade em um campo.
- Uma vez que os ratos/amostras estão prontas para tratamento de imagens, clique em "Adquirir" para aquisição de imagens.
- Abra os dados de imagem que foi adquiridos.
- Abra o painel de paleta de ferramentas (Figura 3).
- Selecione Ferramentas ROI. Recomendamos o círculo (o um mais à esquerda) para variar a área bioluminescente em imagens (Figura 4).
- Clique em "Medida ROIs" (ícone de lápis) para medir a intensidade de superfície bioluminescente (Figura 3). O painel de medições de ROI e os valores de quantificação aparecem (Figura 5).
- Use a medida de configurar no canto esquerdo do painel de medições de ROI para selecionar as valores/informações necessárias (Figura 6), caso contrário clique em "Exportar" para exportar esta tabela de dados e salvar como arquivo. csv (Figura 5).
- Use os valores da coluna "Fluxo Total (p/s)" como a quantificação de intensidade bioluminescentes no arquivo. csv.
Resultados
Nós administramos bioluminescência-rotulado EHEC (~ 109 células bacterianas) para 6 semana de idade camundongos C57BL/6 fêmeas, por gavagem oral. Após a inoculação oral de EHEC aos ratos dentro de 1h, os animais foram examinados pelo sinal bioluminescente, na vivo sistema de imagem como mostrado na Figura 7. Os resultados mostraram um forte sinal bioluminescente em camundongos gavage com bioluminescência-rotulado EHEC. Examinamos os sinais em 2 dias pós infecção. Como mostrado na Figura 8A, os ratos inoculados com bioluminescência-rotulado selvagem-tipo EHEC EDL933 mostrou sinais bioluminescentes intensos, mesmo depois de 2 dias pós infecção, que sugeriu a que EHEC colonizada em hospedeiros por 2 dias. Nós também intragastrically infectados bioluminescência-rotulado EDL933ΔrfaD (ΔrfaD) para ratos (Figura 8A). Este mutante, desertou em lipopolissacarídeo (LPS), tem sido mostrado para reduzir a colonização no acolhimento em nosso estudo anterior. Como mostrado na Figura 8A, não há nenhum sinal bioluminescente detectado em ΔrfaD-ratos infectados, o que sugere que não há nenhum ou menos bactérias células colonizaram nos ratos. Quantificação do sinal fluorescente é mostrada na Figura 8B. Em seguida, determinou-se a localização destas bactérias rotulado de bioluminescência. Os ratos infectados foram sacrificados humanamente e seu intestino todo removido. Os intestinos de ratos 2 dias pós infecção foram posicionados em pratos de Petri de 9 cm e fotografaram ex vivo (Figura 9A). Os tecidos intestinais de bioluminescência-rotulado EDL933 infectado ratos revelaram um aumento significativo em sinais bioluminescentes no ceco e cólon, que sugerem que estes EHEC bioluminescentes colonizado no ceco e cólon de ratos infectados por 2 dias no pelo menos. Em contraste, camundongos infectados com bioluminescência-rotulado ΔrfaD (Figura 9A), revelada diminuíram bioluminescente sinal no tecido intestinal, que é consistente com a imagem na vivo (Figura 8A ). Quantificação do sinal fluorescente é mostrada na Figura 9B.

Figura 1 : Linha do tempo do fluxograma de preparação experimental.
Visão geral do tempo necessário para preparar bioluminescentes bactéria EHEC e pré-tratamento de ratos com estreptomicina. Preparação do (A), EHEC. (B) preparação de ratos. Clique aqui para ver uma versão maior desta figura.
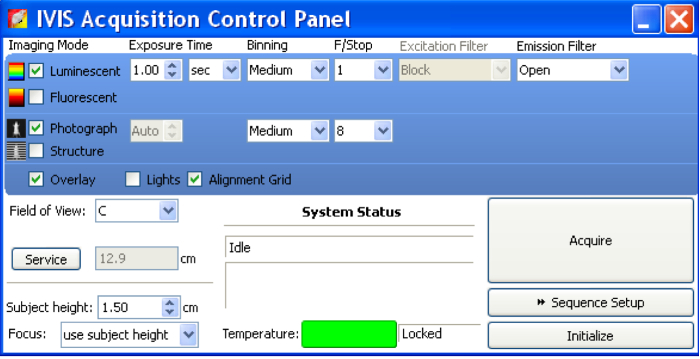
Figura 2 : Na vivo painel de controle de aquisição de sistema de imagem.
Antes de amostras de imagem, abra o painel de controle de aquisição de IVIS. Selecione "Luminescentes", "fotografia" e "Sobreposição". Definir o tempo de exposição, como "Auto". Definir Binning como "Meio". Luminescentes e 8 estabelecem ƒ/parar como 1 para fotografia. Ƒ/parar controles a quantidade de luz recebida pelo detector CCD. Uma vez que as amostras estão prontas para tratamento de imagens, clique em "Adquirir" para adquirir imagens. Clique aqui para ver uma versão maior desta figura.

Figura 3 : Painel paleta de ferramentas.
Após a aquisição de imagem, utilize o painel de paleta de ferramentas para quantificar a intensidade bioluminescente. Abra o painel de paleta de ferramentas e os dados de imagem. Escolha uma das ferramentas de ROI para variar os sinais bioluminescentes em imagens. Clique aqui para ver uma versão maior desta figura.

Figura 4 : Sinal bioluminescente de amostra para quantificação.
Área de sinal bioluminescentes em imagens circundada pelas ferramentas de ROI. Todos os sinais bioluminescentes mostrados aqui estão no círculo vermelho. Clique aqui para ver uma versão maior desta figura.

Figura 5 : Medições de ROI.
Depois circulando sinais bioluminescentes e clicando em "Medida ROIs" no painel paleta de ferramentas, valores são apresentados como mostrado. Os valores da coluna de fluxo Total (p/s) são utilizados para a quantificação de intensidade bioluminescentes. Clique aqui para ver uma versão maior desta figura.

Figura 6 : Adicionar informações de quantificação diferente.
Clicando em configurar medição no canto esquerdo do painel de medições de ROI, você pode selecionar outras valores de quantificação desejado/informações. Clique aqui para ver uma versão maior desta figura.
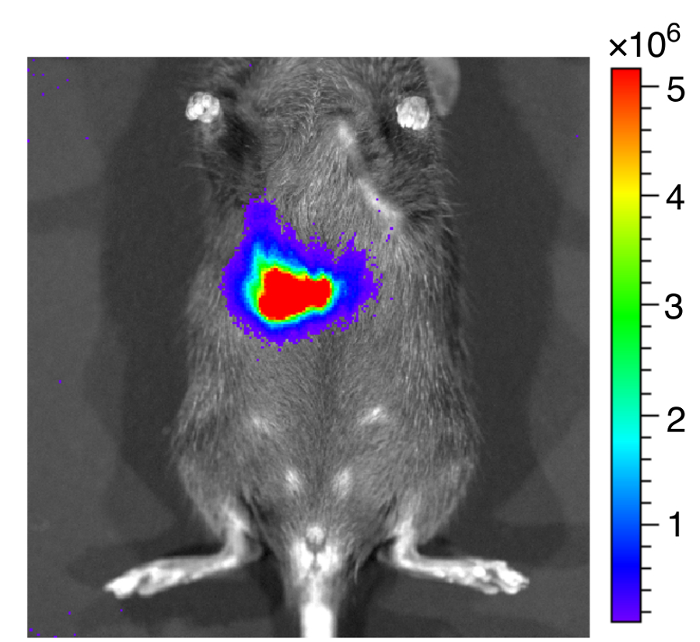
Figura 7 : Imagem representativa dos ratos depois de inoculado com bioluminescente EHEC.
Imagem representativa de camundongos inoculados com EHEC bioluminescente por gavagem oral dentro de 1 h. A escala de cores representa a radiância (p/s/cm2/sr). Clique aqui para ver uma versão maior desta figura.

Figura 8 : Imagens de camundongos inoculados com bioluminescência-rotulado EHEC depois de 2 dias.
(A) representam imagem dos ratos inoculados com bioluminescente selvagem-tipo EHEC EDL933 e EDL933:ΔrfaD por gavagem oral depois de 2 dias pós infecção. (B) a quantificação da intensidade de bioluminescência de camundongos infectados com EHEC. Barras de erro indicam os desvios-padrão. Imagens representativas são mostradas. Todos os experimentos foram conduzidos independentemente três vezes com 2-3 animais cada vez, e as barras de erro indicam os desvios-padrão. P-valores indicam os resultados da análise estatística pelo teste t. A escala de cores representa a radiância (p/s/cm2/sr). Clique aqui para ver uma versão maior desta figura.
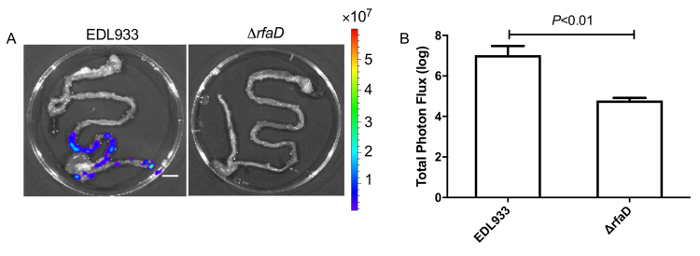
Figura 9 : Imagens dos tecidos intestinais de ratos infectados com bioluminescência-rotulado EHEC.
(A) 2 dias após a inoculação com bioluminescência-rotulado EHEC, os ratos foram sacrificados e todo intestinais tecidos foram removidos e fotografaram ex vivo. Imagens representativas são mostradas. (B) a quantificação da intensidade de bioluminescência dos tecidos intestinais de camundongos infectados com EHEC. Todos os experimentos foram conduzidos independentemente três vezes com 2-3 animais cada vez, e as barras de erro indicam os desvios-padrão. P-valores indicam os resultados da análise estatística pelo teste t. A escala de cores representa a radiância (p/s/cm2/sr). Barra de escala representa 1 cm. clique aqui para ver uma versão maior desta figura.
| Passos | Temperatura | Tempo | Número de ciclos |
| Desnaturação inicial | 95 ° C | 10 min | 1 |
| Desnaturação | 95 ° C | 30 seg | 35 |
| Recozimento | 58,4 ° C | 30 seg | |
| Extensão | 72 ° C | 1,5 min | |
| Extensão final | 72 ° C | 10 min | 1 |
| Segure | 4 ° C | ∞ | 1 |
Tabela 1: Condições de reação em cadeia (PCR) de Polymerase
| Nome de cartilhas | Sequência de |
| nptII F | 5' CCTATGCATAATAATTCCGCTAGCTTCACG3' |
| nptII R | 5' GCTCCACCGATAATATTCCTGAGTCATACT3' |
Tabela 2: sequências de Primer usadas para amplificar nptII
Discussão
Tem sido relatado que EHEC transformada com o plasmídeo luciferase tem sido utilizada para examinar sua localização em hosts ou expressão de gene em vivo8,11,12. O modelo murino demonstrado aqui também tem sido relatado para detectar o sincronismo de EHEC colonizado e localização no anfitrião murino8. Não obstante, nós fornecemos o protocolo de detalhe de como administrar a inoculação de EHEC aos ratos intragastrically e como preparar cuidadosamente as bactérias bioluminescentes para gavagem oral. Nomeadamente, para a gavagem oral de rato de EHEC (etapa 3.7), a posição da cabeça do rato é crítica quando a agulha de gavagem é inserida. Se a posição não é vertical, será difícil passar a agulha, e possivelmente poderia ferir o mouse. Na etapa 3.8, quando a agulha de gavagem é dentro da boca do rato, a língua põe fora da boca ligeiramente. Se a resistência ao passar a agulha de gavagem para o esôfago, parar de se mover a agulha para a frente e retirá-la imediatamente. Altere a posição da agulha, para certificar-se que a agulha está inserindo o esôfago. A agulha pode estar entrando em traqueia quando ocorre resistência, que levaria a injeção de bactérias nos pulmões em vez do estômago.
Aplicação da proteína verde fluorescente (GFP) como um biossensor é comum em experimentos biológicos. No entanto, usando GFP como repórter para observar que a infecção/colonização do patógeno em animais vivos pela imagem latente na vivo não é recomendada, porque a absorvância por hemoglobina, proteínas e água são elevados entre 200-650 nm26, que se sobrepõe com GFP (excitação 480 nm, emissão 510 nm)27. Portanto, usando GFP sinal como um repórter para a imagem latente na vivo pode ser interrompido por hemoglobina, proteínas e água em animais26. Fluorescência infravermelho próximo (NIR) é ideal para na vivo de imagens porque sua janela de absorvância é em torno de 650-900 nm28, que é na região de mais baixo coeficiente de absorção da hemoglobina (< 650 nm) e água (> 900 nm)26 ,28 . Além disso, quando o tecido absorve a luz, há uma chance de induzir autofluorescência. Quando os comprimentos de onda de excitação e emissão variam na janela GFP, induz autofluorescência muito mais do que o NIR29. Uso do NIR pode melhorar o sinal à relação de fundo, comparada com a de GFP, eliminando a autofluorescência fundo29. Bioluminescência não exige a excitação de energia para gerar luz visível. Depende da reação que catalisam Luciferina de substrato por sua luciferase de enzima e gerar luz. Desde que a bioluminescência não requer luz diretamente sobre uma amostra, o sinal de fundo de uma amostra é muito baixo. Portanto, use de bioluminescência como repórter é mais geral e mais fácil para a imagem latente na vivo . Em contraste, a fluorescência requer luz de excitação para induzir a luz de sinal. Quando os tecidos absorvem a luz, há uma chance de que a luz fluorescente será emitida e induzir autofluorescência para que seu sinal-ruído é mais alto comparado de bioluminescência.
Considerando a EHEC é naturalmente menos colonizada em camundongos por infecção oral2,22, um patógeno da mucosa natural de ratinhos, chamado rodentium Citrobacter, tem sido utilizado para estudar o mecanismo de colonização de murino host como um bactéria22,30de aluguel. Ambos de EHEC e rodentium c. colonizam a mucosa intestinal e induzem a formação de lesões A/E no acolhimento de22,30. Eles também contêm a ilha de patogenicidade LEE, que codifica um T3SS e várias proteínas efetoras que induziu A/E lesão22,30. Portanto, o uso de luciferase expressando plasmídeo como um repórter em c. rodentium para detectar a patologia de colonização e estudar o mecanismo de colonização através de um sistema da imagem latente na vivo tem também sido relatado14, 15. no entanto, enquanto a infecção de c. rodentium de ratos é um modelo útil para investigar a função do T3SS e o mecanismo da lesão A/E, rodentium c. não contém Shiga toxina (Stx)30, que é uma posição dominante factor de virulência que causa falência renal em EHEC, particularmente o sorotipo O157: H73. Apesar de uma estirpe de Stx-expressando c. rodentium foi construída recentemente31, que é mais realista a infecção de EHEC, não inclui outros fatores de virulência EHEC potenciais que são cruciais para a colonização e/ou infecção. Além disso, rodentium c. compartilha 67% dos seus genes com EHEC,32, que sugere que o EHEC pode usar uma virulência distinta de c. rodentium durante a colonização e/ou infecção.
A luciferase expressando de shRNA usada aqui, pWF27913, foi modificado de pAKlux29 , cuja espinha dorsal é pBBR1MCS433. Embora pBBR1MCS4 foram testados e replicado em várias bactérias33, é fundamental garantir a origem de replicação (ORI) deste plasmídeo é adequada para o host bacteriano antes de utilizar este sistema baseado em plasmídeo luciferase para o experimento e confirmando que este luciferase expressando de shRNA pode replicar no acolhimento bacteriano. Utilizamos antibiótico stress para manter a estabilidade do plasmídeo nas bactérias. Quando as bactérias entram animais na ausência de antibióticos, o sinal bioluminescente do selvagem-tipo EHEC foi detectado pelo menos 2 dias. No entanto, nós não após infecção por mais de 2 dias, porque já tínhamos visto uma diferença significativa na intensidade luminescente entre EHEC WT e EHEC rfaD (que codifica um gene necessário para EHEC sintetizando LPS intactas) mutante em 2 dias. Para manter o plasmídeo estàvel em bactérias sob a ausência de antibióticos, um plasmídeo pCM1710,34 pode ser usado para essa finalidade. pCM17 codifica um sistema de particionamento de dois-plasmídeo e um mecanismo de matar post-segregational para assegurar a manutenção do plasmídeo em bactérias na ausência de antibióticos10,34. O plasmídeo pCM17 contendo o operon luxCDABE conduzido pelo promotor OmpC pode ser detectado por sinal bioluminescente pelo menos 7 dias8. Um método alternativo para obter uma bactéria contínua expressão bioluminescentes na ausência de antibióticos é para inserir o cromossomo bacteriano35gene luxABCDE . Transposon de Francis et al . usado inserido a gaveta de operon e antibióticos luxABCDE aleatoriamente inserida o cromossomo de Streptococcus pneumoniae para obter a tensão estável de bioluminescência35.
Em nosso estudo anterior13, utilizamos este sistema modelo para examinar a colonização de EHEC em um host e comparar a diferença de capacidade de colonização entre o tipo selvagem EHEC (WT) e mutante13. Quando os ratos foram administrados o mutante de rfaD EHEC bioluminescente, os sinais bioluminescentes diminuídos drasticamente comparado ao que de EHEC WT depois de 2 dias pós infecção. Ele fornece evidência de que este modelo murino pode analisar o efeito da mutação da colonização de EHEC no acolhimento. Além disso, tratamentos terapêuticos para reduzir a colonização de EHEC é uma solução considerada potencial para infecção EHEC desde o uso de antibióticos é contra-indicado5,36. Portanto, é que vale a pena testar se este sistema de modelo pode ser usado para examinar a eficácia da antidrogas colonização / tratamentos contra a colonização de enterobactérias no acolhimento. Acreditamos que usando este modelo de sistema, é possível examinar o calendário e a localização de EHEC não apenas, mas também outros de colonização de enterobactérias em vivo. Usando este modelo animal, o processo de colonização de EHEC em ratos pode ser monitorado e os encargos da colonização no acolhimento podem ser quantificado para determinar a colonização espacial e temporal de EHEC em animais vivos. A visualização e a quantificação da colonização de enterobactérias usando este modelo torna uma ótima ferramenta para investigar e analisar os mecanismos bem de colonização entérobactérienne e assim, compensar a deficiência de pesquisa de colonização e Melhore o conhecimento atual.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Reconhecemos o Chi-Chung Chen do departamento de pesquisa médica, Chi Mei Medical Center (Tainan, Taiwan) pela ajuda no mouse, infecção e o apoio do centro de animais de laboratório da Universidade Nacional de Cheng Kung. Este trabalho é apoiado pelo Ministro da ciência e tecnologia (a maioria) concede (mais 104-2321-B-006-019, 105-2321-B-006-011,-005 and106-2321-B-006) para CC.
Materiais
| Name | Company | Catalog Number | Comments |
| Shaker incubator | YIH DER | LM-570R | bacteria incubation |
| Orbital shaking incubator | FIRSTEK | S300 | bacteria incubation |
| pBSL180 | source of nptII gene | ||
| pAKlux2 | source of luxCDABE operon | ||
| T&A Cloning Kit | Yeastern Biotech | FYC001-20P | use for TA cloning |
| Nsi I | NEB | R0127S | use for plasmid cloning |
| Sca I | NEB | R0122S | use for plasmid cloning |
| Spe I-HF | NEB | R0133S | use for plasmid cloning |
| Sma I | NEB | R0141S | use for plasmid cloning |
| T4 ligase | NEB | M0202S | use for plasmid cloning |
| Ex Taq | TaKaRa | RR001A | use for PCR amplification |
| 10X Ex Taq Buffer | TaKaRa | RR001A | use for PCR amplification |
| dNTP Mixture | TaKaRa | RR001A | use for PCR amplification |
| PCR machine | applied Biosystem | 2720 thermal cycler | for PCR amplification |
| Glycerol | SIGMA | G5516-1L | use for bacteria stocking solution |
| NaCl | Sigma | 31434-5KG-R | chemical for making LB medium, 10 g/L |
| Tryptone | CONDA pronadisa | Cat 1612.00 | chemical for making LB medium, 10 g/L |
| Yeast Extract powder | Affymetrix | 23547-1 KG | chemical for making LB medium, 5 g/L |
| Agar | CONDA pronadisa | Cat 1802.00 | chemical for making LB agar |
| kanamycin | Sigma | K4000-5G | antibiotics, use for seleciton |
| streptomycin | Sigma | S6501-100G | antibiotics, eliminate the microbiota in mice |
| EDL933 competent cell | Homemade | method is on supplemental document | |
| Electroporator | MicroPulser | for electroporation | |
| Electroporation Cuvettes | Gene Pulser/MicroPulser | 1652086 | for electroporation |
| High-speed centrifuge | Beckman Coulter | Avanti, J-26S XP | use for centrifuging bacteria |
| Fixed-Angle Rotor | Beckman Coulter | JA25.5 | use for centrifuging bacteria |
| Fixed-Angle Rotor | Beckman Coulter | JLA10.5 | use for centrifuging bacteria |
| centrifuge bottles | Beckman Coulter | REF357003 | use for centrifuging bacteria |
| centrifuge bottles | Thermo Fisher scientific | 3141-0500 | use for centrifuging bacteria |
| eppendorf biophotometer plus | eppendorf | AG 22331 hamburg | for measuring the OD600 value of bacteria |
| C57BL/6 mice | Laboratory Animal Center of NCKU | ||
| lab coat, gloves | for personnel protection | ||
| isoflurane | Panion & BF Biotech Inc. | G-8669 | for mice anesthesia, pharmaceutical grade |
| 1ml syringe | use for oral gavage of mice | ||
| Reusable 22 G ball-tipped feeding needle | φ0.9 mm X L 50 mm | use for oral gavage of mice | |
| surgical scissors | use for mice experiment | ||
| Xenogen IVIS 200 imaging system | Perkin Elmer | IVIS spectrum | use for bioluminescent image capture |
| Living Image Software | Perkin Elmer | version 4.1 | use for quantifying the image data |
Referências
- Pennington, H. Escherichia coli O157. Lancet. 376 (9750), 1428-1435 (2010).
- Mayer, C. L., Leibowitz, C. S., Kurosawa, S., Stearns-Kurosawa, D. J. Shiga toxins and the pathophysiology of hemolytic uremic syndrome in humans and animals. Toxins (Basel). 4 (11), 1261-1287 (2012).
- Tarr, P. I., Gordon, C. A., Chandler, W. L. Shiga-toxin-producing Escherichia coli and haemolytic uraemic syndrome. Lancet. 365 (9464), 1073-1086 (2005).
- Obrig, T. G. Escherichia coli Shiga Toxin Mechanisms of Action in Renal Disease. Toxins (Basel). 2 (12), 2769-2794 (2010).
- Nguyen, Y., Sperandio, V. Enterohemorrhagic E. coli (EHEC) pathogenesis. Front Cell Infect Microbiol. 2, 90(2012).
- Wiles, S., Robertson, B. D., Frankel, G., Kerton, A. Bioluminescent monitoring of in vivo colonization and clearance dynamics by light-emitting bacteria. Methods Mol Biol. 574, 137-153 (2009).
- Hutchens, M., Luker, G. D. Applications of bioluminescence imaging to the study of infectious diseases. Cell Microbiol. 9 (10), 2315-2322 (2007).
- Rhee, K. J., et al. Determination of spatial and temporal colonization of enteropathogenic E. coli and enterohemorrhagic E. coli in mice using bioluminescent in vivo imaging. Gut Microbes. 2 (1), 34-41 (2011).
- Karsi, A., Lawrence, M. L. Broad host range fluorescence and bioluminescence expression vectors for Gram-negative bacteria. Plasmid. 57 (3), 286-295 (2007).
- Lane, M. C., Alteri, C. J. S., Smith, S. N., Mobley, L. H. Expression of flagella is coincident with uropathogenic Escherichia coli ascension to the upper urinary tract. Proc Natl Acad Sci U S A. 104 (42), 16669-16674 (2007).
- Roxas, J. L., et al. Enterohemorrhagic E. coli alters murine intestinal epithelial tight junction protein expression and barrier function in a Shiga toxin independent manner. Lab Invest. 90 (8), 1152-1168 (2010).
- Siragusa, G. R., Nawotka, K., Spilman, S. D., Contag, P. R., Contag, C. H. Real-Time Monitoring of Escherichia coli O157:H7 Adherence to Beef Carcass Surface Tissues with a Bioluminescent Reporter. , (1999).
- Kuo, C. J., et al. Mutation of the Enterohemorrhagic Escherichia coli Core LPS Biosynthesis Enzyme RfaD Confers Hypersusceptibility to Host Intestinal Innate Immunity In vivo. Front Cell Infect Microbiol. 6, 82(2016).
- Wiles, S., et al. Organ specificity, colonization and clearance dynamics in vivo following oral challenges with the murine pathogen Citrobacter rodentium. Cell Microbiol. 6 (10), 963-972 (2004).
- Wiles, S., Pickard, K. M., Peng, K., MacDonald, T. T., Frankel, G. In vivo bioluminescence imaging of the murine pathogen Citrobacter rodentium. Infect Immun. 74 (9), 5391-5396 (2006).
- Contag, C. H., Contag, P. R., Mullins, J. I., Spillman, S. D., Stevenson, D. K., Benaron, D. A. Photonic detection of bacterial pathogens in living hosts. Mol Microbiol. 18 (4), 593-603 (1995).
- Hardy, J., Francis, K. P., DeBoer, M., Chu, P., Gibbs, K., Contag, C. H. Extracellular replication of Listeria monocytogenes in the murine gall bladder. Science. 303 (5659), 851-853 (2004).
- Kaniga, K., Sory, M. P., Delor, I., Saegerman, C., Limet, J. N., Cornelis, G. R. Monitoring of Yersinia enterocolitica in Murine and Bovine Feces on the Basis of the Chromosomally Integrated luxAB Marker Gene. Appl Environ Microbiol. 58 (3), 1024-1026 (1992).
- Trcek, J., Fuchs, T. M., Trulzsch, K. Analysis of Yersinia enterocolitica invasin expression in vitro and in vivo using a novel luxCDABE reporter system. Microbiology. 156 (Pt 9), 2734-2745 (2010).
- Morin, C. E., Kaper, J. B. Use of stabilized luciferase-expressing plasmids to examine in vivo-induced promoters in the Vibrio cholerae vaccine strain CVD 103-HgR. FEMS Immunol Med Microbiol. 57 (1), 69-79 (2009).
- Law, R. J., Gur-Arie, L., Rosenshine, I., Finlay, B. B. In vitro and in vivo model systems for studying enteropathogenic Escherichia coli infections. Cold Spring Harb Perspect Med. 3 (3), a009977(2013).
- Ritchie, J. M. Animal Models of Enterohemorrhagic Escherichia coli Infection. Microbiol Spectr. 2 (4), EHEC-0022-2013(2014).
- Chou, T. C., et al. Enterohaemorrhagic Escherichia coli O157:H7 Shiga-like toxin 1 is required for full pathogenicity and activation of the p38 mitogen-activated protein kinase pathway in Caenorhabditis elegans. Cell Microbiol. 15 (1), 82-97 (2013).
- Alexeyev, M. F., Shokolenko, I. N. Mini-Tnl 0 transposon derivatives for insertion mutagenesis and gene delivery into the chromosome of Gram-negative bacteria. Gene. 160 (1), 59-62 (1995).
- Wiegand, I., Hilpert, K., Hancock, R. E. Agar and broth dilution methods to determine the minimal inhibitory concentration (MIC) of antimicrobial substances. Nat Protoc. 3 (2), 163-175 (2008).
- Pansare, V., Hejazi, S., Faenza, W., Prud'homme, R. K. Review of Long-Wavelength Optical and NIR Imaging Materials: Contrast Agents, Fluorophores and Multifunctional Nano Carriers. Chem Mater. 24 (5), 812-827 (2012).
- Heim, R., Cubitt, A. B., Tsien, R. Y. Improved green fluorescence. Nature. 373 (6516), 663-664 (1995).
- Weissleder, R. A clearer vision for in vivo imaging. Nat Biotechnol. 19 (4), 316-317 (2001).
- Frangioni, J. In vivo near-infrared fluorescence imaging. Current Opinion in Chemical Biology. 7 (5), 626-634 (2003).
- Collins, J. W., et al. Citrobacter rodentium: infection, inflammation and the microbiota. Nat Rev Microbiol. 12 (9), 612-623 (2014).
- Mallick, E. M., et al. A novel murine infection model for Shiga toxin-producing Escherichia coli. J Clin Invest. 122 (11), 4012-4024 (2012).
- Petty, N. K., et al. The Citrobacter rodentium genome sequence reveals convergent evolution with human pathogenic Escherichia coli. J Bacteriol. 192 (2), 525-538 (2010).
- Kovach, M. E., Elzer, P. H., Hill, D. S., Robertson , G. T., Farris, M. A., Roop, R. M. II, Peterson, K. M. Four new derivatives of the broad-host-range cloning vector pBBR1MCS, carrying different antibiotic-resistance cassettes. Gene. 166 (1), 175-176 (1995).
- Galen, J. E., Nair, J., Wang , J. Y., Wasserman, S. S., Tanner, M. K., Sztein , M. B., Levine, M. M. Optimization of Plasmid Maintenance in the Attenuated Live Vector Vaccine Strain Salmonella typhiCVD 908-htrA. Infect Immun. 67 (12), 6424-6433 (1999).
- Francis, K. P., et al. Visualizing pneumococcal infections in the lungs of live mice using bioluminescent Streptococcus pneumoniae transformed with a novel gram-positive lux transposon. Infect Immun. 69 (5), 3350-3358 (2001).
- Goldwater, P. N., Bettelheim, K. A. Treatment of enterohemorrhagic Escherichia coli (EHEC) infection and hemolytic uremic syndrome (HUS). BMC Med. 10, (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados