Method Article
Análise Quantitativa de Autophagy usando microscopia de fluorescência avançada 3D
Neste Artigo
Resumo
Autophagy é um processo ubíquo que permite que as células para se degradarem e reciclar proteínas e organelas. Aplicamos microscopia de fluorescência para visualizar avançada e quantificar o pequeno, mas, alterações físicas essenciais associados com a indução de autofagia, incluindo a formação e distribuição de autofagossomas e lisossomas, e sua fusão em autolysosomes.
Resumo
O câncer de próstata é a forma principal de tumores malignos entre os homens em os EUA Enquanto a cirurgia acarreta um risco significativo de impotência e incontinência, as abordagens quimioterápicos tradicionais têm falhado. A terapia hormonal é eficaz na fase inicial, mas muitas vezes falha com o eventual desenvolvimento de tumores hormônio-refratário. Nós estávamos interessados no desenvolvimento de terapêuticas dirigidas deficiência metabólica específica de células tumorais. Recentemente, mostrou que as células de tumor da próstata não têm especificamente uma enzima (sintase argininosuccinato ou ASS) envolvida na síntese da arginina, um aminoácido. Esta condição faz com que as células do tumor se tornar dependente de arginina exógena, e eles passam por estresse metabólico quando arginina livre está esgotada pela arginina deiminase (ADI) 1,10. Na verdade, temos demonstrado que as células cancerosas humanas da próstata CWR22 RV1 são efetivamente morto por ADI com apoptose caspase-independente e autopha agressivogy (ou macroautophagy) 1,2,3. A autofagia é um processo evolutivamente conservada que permite que as células de metabolizar proteínas indesejadas pelo colapso lisossômico durante inanição nutricional 4,5. Embora os componentes essenciais desta via são bem caracterizados 6,7,8,9, muitos aspectos do mecanismo molecular não são ainda claras - em particular, o que é o papel de autofagia no-resposta a morte de células de cancro da próstata após o tratamento ADI ? A fim de resolver esta questão, necessário um método experimental para medir o nível ea extensão da resposta autofágica em células - e uma vez que não há marcadores moleculares conhecidos que podem controlar com precisão este processo, optamos por desenvolver uma abordagem baseada em imagens, usando 3D microscopia de fluorescência quantitativa 11,12.
Usando CWR22Rv1 células especificamente marcados com sondas fluorescentes para autophagosomes e lisossomos, mostramos que pilhas de imagens 3D adquirido com ou widefield microscopia desconvolução (e mais tarde, com super-resolução, microscopia de iluminação estruturada) pode capturar claramente as fases iniciais de indução autofagia. Com as aplicações de análise de imagem digitais disponíveis comercialmente, podemos facilmente obter informação estatística sobre o número e autophagosome lisossoma, o tamanho, a distribuição, e do grau de co-localização de qualquer célula de imagens. Esta informação permite-nos acompanhar com precisão o progresso da autofagia em células vivas e permite que nossa investigação contínua sobre o papel da autofagia na quimioterapia do cancro.
Protocolo
1. Parte 1: Cultura de Células e Rotulagem Imuno-fluorescência
- Cresça CWR22 RV1 células tumorais da próstata humana em lamelas de vidro (# 1,5 ou 170 m de espessura), colocadas em placas de 6 poços, com meio RPMI (Mediatech, VA) contendo 10% de soro fetal bovino (FBS) e 1% de penicilina / estreptomicina / glutamina.
- Induzir autofagia amostras seleccionadas por tratamento com arginina deiminase (ADI, 0,3 ug / ml) em solução salina tamponada com fosfato (PBS).
- Fixar as células com paraformaldeído a 4% (PFA, Fisher Scientific, NH), diluídas em PBS, 100 ul por lamela, durante 10 min à TA.
- Lave as células 3x com 1 ml de HBS / BSA.
- Permeabilizar células com 0,05% de saponina (Sigma, MO) em tampão HEPES (HBS) / BSA, com 100 ul por lamela, durante 15 min à TA.
- Antes de imuno-marcação, as amostras do bloco com 2,5% de caseína (Sigma, MO) em HBS / BSA, com 100 ul por lamela, durante 1 hora à temperatura ambiente.
- Incubar as células fixadas com anticorpos primários diluídos em 2,5% de caseína / HBS / BSA, com 100 ul por coverslip, O / N a 4 ° C.
- Lave as células 3x com 1 ml de HBS / BSA.
- Incubar as células fixadas com anticorpos secundários diluídos em 2,5% de caseína / HBS / BSA, com 100 ul por lamela, durante 1 hora à temperatura ambiente.
- Lave as células 3x com 1 ml de HBS / BSA.
- Monte lamelas preparadas em lâminas de vidro para microscópio regulares com ouro SlowFade ou prolongar reagente antifade Gold (Invitrogen, CA), contendo 4 ',6-diamidino-2-phenylindole (DAPI).
- Bordas Seal lamela com unha polonês. Para obter os melhores resultados de imagem, dar tempo (de preferência O / N) para a mídia anti-fade a permear completamente as células.
- Superfície lamela limpo com metanol e / ou clorofórmio.
- Adicionar óleo de imersão (índice de refracção 1,520, quando imagiologia a 37 ° C, ou quando 1,516 imagiologia à TA) à 1,42 NA lente objectiva 60X (Olympus, Japão).
- Posição corrediça na platina do microscópio e estabelecer o foco com as células de interesse.
2. Parte 2: Preparando Células para imagens ao vivo
- Crescer CWR22 RV1 células que expressam a proteína verde fluorescente acoplado Cadeia leve de 3 (LC3-GFP) em meio de cultura RPMI contendo 10% de FBS e 1% de antibióticos de 35 mm revestidas com placas de cultura de vidro com fundo de poli-d-lisina (MatTek, MA). As células devem ser colocadas em placas a uma densidade suficiente para facilitar a proliferação rápida, mas não tanto que as células são cobertas e aglutinado por o tempo de imagiologia.
- Tratar amostras de células seleccionadas com ADI (0,3 ug / ml) em PBS.
- Cerca de 1 hora antes da imagem, diluir 1,5 ul de Lyso Tracker Vermelho DND-99 (Invitrogen, CA), com 20 ml de RPMI. contendo 10% de FBS e 1% de antibióticos. Use soluções contendo ADI para amostras selecionadas. Aquecer todos os suportes a 37 ° C antes da adição à placa de cultura.
- Incubar as células com meio RPMI contendo Lyso Tracker Vermelho DND-99 para 15-45 min a 37 ° C.
- Cerca de 30 minutos antes da imagiologia, ligar o invólucro do ambiente EstaçãoMeteorológica e deixar equilibrar a 37 ° C e 5% de CO 2 (com humidifar IED).
- Lavar as células com PBS e substitua com meio padrão RPMI contendo apenas 10% de FBS e 1% de antibióticos. Adicionar ADI para amostras conforme indicado.
- Pratos de montagem 35 milímetros lamela com fundo de cultura em placa personalizada (Applied Precision, Inc., WA) e posição no palco microscópio. Use óleo de imersão (índice de refração 1,520) no 60X 1,42 NA lente objetiva, e posicionar o prato de cultura montado no palco.
3. Parte 3: Microscopia Deconvolution e Análise
O protocolo nesta seção assume o uso do DeltaVision pessoal DV deconvolution microscópio e associados aplicação suíte Softworx (Applied Precision, Inc., WA).
- Ligue o sistema de microscópio, incluindo o controlador da estação de trabalho e de aquisição de instrumentos. Abra o aplicativo Resolve3D para inicializar o estágio do microscópio e ligue a fonte de luz xenon. Permitir 10 min para a fonte luminosa para aquecer e atingir condições estáveis.
- Adicionegota de óleo de imersão (índice de refração 1,520 para amostras vivas a 37 ° C ou 1,516 para amostras fixas no RT) no 60X 1,42 NA objectiva, e um centro de espécime de slides (tampa de deslizamento para baixo) sobre a lente objetiva.
- Usando qualquer iluminação de campo claro ou externa eo foco grosso botão de ajuste, levante lentamente a objetiva até que o talão de óleo de imersão faz contato com a tampa de vidro invertida. (A partir deste ponto, a camada de células deve entrar em foco em poucas voltas do foco fino botão de ajuste). Ao trabalhar com amostras fixadas sobre as lâminas, é recomendado para evitar a células dentro ou perto de quaisquer áreas com bolhas. Ao visualizar com amostras vivas no prato com fundo de vidro, evitar a movimentação da lente objetiva fora do limite da lamela de vidro.
- Autophagosomes podem ser identificadas por um sinal de eGFP (verde). Os lisossomos são identificados por Lyso Tracker Red (amostra ao vivo) ou imunofluorescência anti-Lamp1 (amostra fixa). Núcleos de células são identificadas por DAPI raining (amostra fixa).
- Selecione o campo de visão desejado. Idealmente, as células devem exibir boa sinal fluorescente em todos os canais apropriados, e suficientemente ligado e espalhar-se sobre a superfície da lamela de modo a que os conteúdos intracelulares é fácil de visualizar. Pequenos ajustes laterais em x, y, e z-focagem podem ser controladas por computador, utilizando a interface Acquire3D. Conjunto para binning 1x1 ou 2x2, dependendo campo de visão desejado (mais células pode ser vista no campo quando binning é definido como 2x2).
- Para uma dada imagem através da barriga de uma célula, defina os parâmetros de exposição (por exemplo,% de transmissão de energia e tempo de exposição) de tal forma que a intensidade máxima de pixel não exceda 3.000 pontos. Geralmente, a% de transmissão deve ser ajustado tão baixo quanto possível, sem aumentar o tempo de exposição correspondente ao longo de 1 seg. Repita esse procedimento para cada cor de fluorescência para ser trabalhada e para cada campo separado de vista para obter as imagens da mais alta qualidade.
- Defina o upper limites e inferiores do Z-pilha, ajustando o foco apenas ligeiramente ao longo do topo e da parte inferior da amostra de células, respectivamente. O número total de imagens de uma dada pilha Z será, assim, determinado por estes limites e por o espaçamento entre as camadas (valor padrão: 0,2 um).
- Para minimizar artefatos de movimento durante a aquisição de imagem, recomendamos definir o modo de aquisição de imagem para "comprimento de onda, então z-stack" para amostras fixas, e "z-stack depois de comprimento de onda" para amostras vivas.
- As imagens de fluorescência são deconvolved usando Softworx (Precision Aplicada, WA) e depois analisados utilizando Volocity (Improvision, agora Perkin Elmer, MA).
4. Parte 4: Super-resolução, Structured-iluminação (OMX) Microscopia
O protocolo nesta seção aplica-se ao uso da OMX Microscópio iluminação estruturada (Applied Precision, WA).
- Ligue a alimentação principal e lasers desejados (410 nm, 488 nm e / ou 532 nm). Aguarde 20 min para os lasers de ser estabilizado termicamente.
- Adicione o óleo de imersão (índice de refração 1,516 para amostras fixas no RT) no 60X 1,42 NA lente objetiva. Se as bolhas são vistos na gota de óleo, limpe o objetivo e adicione o óleo novo.
- Coloque amostra de slides no palco e, se necessário, permitir que 10 min para que as células se contentar em lamela.
- Inicializar o programa de aquisição. Use iluminação de fluorescência para ajustar o foco até um contorno nítido da célula pode ser visto. (Deve ter cuidado em todos os momentos para minimizar a exposição das células a excitação de fluorescência, até a aquisição de imagem real).
- Uma varredura de mosaico em espiral podem ser adquiridos para visualizar áreas maiores da amostra para selecionar células ou regiões de interesse. Recomenda-se usar tempos de exposição de curta duração (1-10 ms) e baixo consumo de energia de excitação (0,1-1% de transmissão) durante a digitalização da amostra ou encontrar alvos.
- Identificar autofagossomas por fluorescência de GFP-LC3. Identificar lisossomos por Alexa Fluor 555 anti-LAMP(Proteína de membrana associada ao lisossoma) e núcleos de células utilizando DAPI (amostras fixas).
- Escolha condições experimentais, incluindo comprimento de onda de excitação, a área de imagem (por exemplo, 512x512), espessura de pilha, tempo de exposição e taxa de transmissão laser. Para imagens de super-resolvida, verifique se a opção de iluminação estruturada está habilitado. Para a microscopia deconvolution na OMX, selecione a opção de iluminação convencional.
- Para obter os melhores resultados de reconstrução, seleccionar as condições experimentais tais que a contagem de intensidade máxima está entre 10.000 e 15.000, durante a aquisição. Se fotodegradação é uma preocupação, a imagem latente numa menor exposição (contagens entre 5.000 e 10.000), podem ser usados. Idealmente, as contagens não deve diminuir mais do que um factor de dois durante toda a aquisição, e diminui em mais de um factor de quatro deve ser evitada. Se necessário, reduzir a espessura da pilha para assegurar uma contagem intensidade sustentada. A câmera satura a 64.000 contagens, então no máximointensidade nunca deve exceder esse valor. Para bons exemplos, encontramos as condições experimentais em 10 ~ 50 ms de tempo de exposição com transmissão do laser de 1%. São preferidos amostras brilhantes e fotoestável que pode ser visualizada, repetidamente. A mancha estável é necessária para time-lapse, imagens de super resolvidos.
- Para reduzir o ruído, a velocidade de leitura de 95MHz (opção de velocidade média) e tempo de aquisição de 2 ms ou mais é recomendado. Aumento dos artefactos são obtidos na fotografada reconstituídas, quando a amostra se move durante a aquisição. Devido a este efeito, as exposições curtas são recomendados para as células não aderentes ou de movimento rápido.
- Multicanal imagiologia pode ser realizado simultaneamente ou sequencialmente. Rendimentos de modo seqüencial menos cross-talk e é, portanto, recomendado.
- Para reduzir a fotodegradação, é geralmente recomendado para a imagem com comprimentos de onda maiores primeiro excitação (532 nm, 488 nm, 410 nm e em seguida). No entanto, os resultados experimentais mostrados na Figura 3, essa ordem era inversad (ou seja, 48 nm, 532 nm e 410 nm), devido à fotoestabilidade desta amostra particular.
- Defina o limite superior / inferior do Z-stack movendo o estágio do microscópio até a parte superior / inferior das células é um pouco fora de foco. A distância desejada entre cada imagem deve ser mantida constante em 0,125 mm para super-resolução de imagem, como o software de reconstrução tomar este valor como padrão.
- Reconstruir pilhas de imagem usando Softworx (Applied Precision, WA) e analisar usando 6.0 Volocity (Perkin Elmer, MA).
Resultados
A sequência da imagem mostrada na Figura 1 mostra as mudanças físicas que ocorrem em CWR22 células durante os primeiros 80 minutos de indução autofagia. Neste e em outros estudos (não mostrado), consistentemente, observada: (1) de deslocamento do núcleo de distância do centro da célula, (2) redução dos pontos de adesão focal, e (3) a translocação geral autofagossomas e lisossomas em direcção ao centro da célula. Além disso, também observamos um pequeno aumento na co-localização (indicado em amarelo) entre autophagosomes (verde) e lisossomos (vermelho), em momentos posteriores.
Como uma demonstração de citometria baseada em imagem, foi utilizado o Volocity digital de aplicação conjunto de imagens (versão 6.0, Improvision / Perkin-Elmer) para identificar, contar e recolher dados estatísticos sobre autophagosomes rotulados e lisossomos nas imagens dos CWR22 células mostradas na Figura 1. Como mostrado nos gráficos da Figura 2, o número eo tamanho dos tele autophagosome (após a sua formação inicial e aparência) que variam com o tempo: após 80 minutos de indução autofagia, houve uma diminuição gradual mas líquido do número de autofagossomas (Figura 2A), com um correspondente aumento do tamanho médio das autofagossomas ( Figura 2B). Além disso, houve um aumento mensurável na co-localização de autofagossomas e lisossomas, com base na análise de correlação de Pearson (Figura 2C). Juntos, esses resultados sugerem que a estimulação da autofagia, numerosos pequenos autophagosomes fundem para formar autophagosomes maiores ao longo do tempo (Figura 2D). Enquanto Figuras 1 e 2 refletiu os resultados de apenas um estudo de imagem única, e análise quantitativa mais rigorosos serão necessários para tirar uma conclusão firme, que ilustram as características e vantagens de microscopia de fluorescência quantitativa. Estamos ativamente usando esta técnica em UOr contínua investigação do mecanismo molecular e processo de autofagia celular.
A Figura 3A mostra uma comparação lado a lado das imagens adquiridas e reconstruídas no modo DV (simulado widefield deconvolution, à esquerda) contra o modo de iluminação estruturada usando o microscópio OMX (direita). Resolução lateral foi melhorada para até 120 nm, o dobro da resolução de microscopia convencional difração limitada 14,15. Co-localização em pequena escala de autophagosomes e lisossomos (Figura 3B, indicado por setas) foram claramente com microscopia de super-resolução, enquanto eles estavam quase evidente com imagens de fluorescência convencional.
Uma das vantagens da utilização de Microscopia Deconvolution é reconstruir todas as imagens em um modelo 3D que vai revelar informação espacial mais precisa entre moléculas diferentes. Filme 1 mostra uma visão geral de um CWR22 Vr1 célula undergoing autofagia no ponto de tempo posterior, em que quantidades significativas de co-localização entre autophagosome lisossoma e começam a ocorrer. A coloração de E-caderina (como indicado na cor branca), revela o contorno da célula alvo. Em Movie 2, o modelo reconstruído em 3D fornece mais detalhes espaciais da fusão entre autophagosomes e lisossomos (como indicado na cor amarela). Podemos ver claramente a interação entre verdes LC3 sinais e sinais Lisosoma vermelhas, ea fusão dos dois sinais para produzir sinais amarelos.

Figura 1. Imagens de lapso de tempo de CWR22 Vr1 células tratadas com ADI. Seqüência de imagens mostrando CWR22 RV1 células tratadas com ADI para 80 min. (Imagens 2D obtidas das pilhas de imagens originais de projeção máxima Z). A WHite A linha a tracejado representa o contorno da célula, e a região de luz sombreada representa a posição do núcleo.

Figura 2. Análise Estatística. Tendências estatísticos foram extraídos por meio da análise da sequência da imagem mostrada na Figura 1 como uma demonstração de que os dados da imagem é quantificável. clique aqui para ver figura maior .
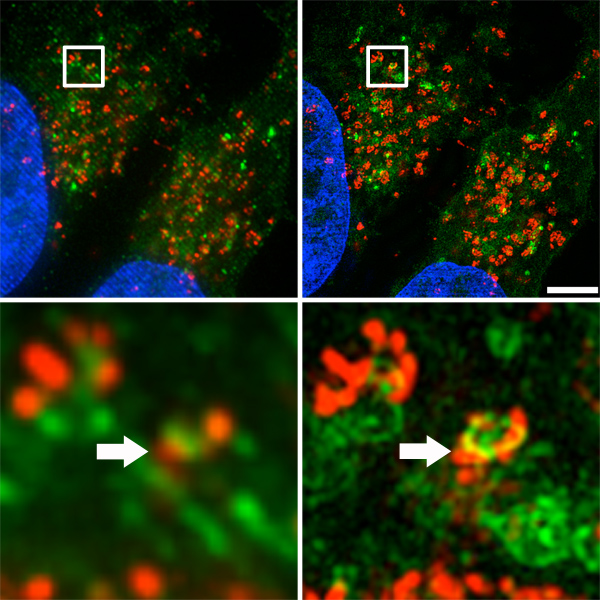
Figura 3. Distribuição autophagosome lisossoma e na célula. A. Top) Side-by-side comparação de imagens adquiridas e reconstruídas no modo DV (simulado widefield deconvolution, à esquerda) e modo de iluminação estruturada usando o microscópio OMX (direita). Barra de escala representa 5 mm. B. Inferior) co-localização em pequena escala de autophagosomes e lisossomos (indicado por setas).
Filme 1 e 2 do filme. Reconstrução 3D de CWR22 Vr1 celular Autophagy passando. Este filme ilustra uma indução de autofagia normal após o tratamento ADI. Imagens Z-Stack foram reconstruídas em um modelo 3-dimensional. Este filme foi gerado pela rotação da célula 360 ° na horizontal e 360 ° na vertical. Sinal verde representa LC3, o sinal vermelho representa lisossomo, o sinal branco representa a E-caderina, e sinal azul representa o núcleo. Clique aqui para ver um filme ou= "_blank"> Clique aqui para ver Movie 2.
Discussão
Enquanto a observação direta de células marcadas com sondas fluorescentes contra LC3 é amplamente aceito como um método padrão para confirmar resposta autofágica 6, imagens 3D quantitativa do mesmo sistema (como temos feito) fornece informações e detalhes sem precedentes sobre o complexo processo de autofagia celular . Em particular, observa-se que centenas (se não milhares) de autophagosomes em células vivas são formadas dentro de 80 min de indução de autofagia. Da mesma forma, observa-se alterações morfológicas muito interessantes na distribuição dos compartimentos lisossomais durante a indução de autofagia. Dentro de uma dada célula, a diminuição do número autophagosome e o correspondente aumento do seu tamanho médio ao longo do tempo, sugerem que autofagossomas crescem por fusão com o outro, antes de combinar com os lisossomas para autolysosomes formulário. Alta resolução de imagem de fluorescência de células vivas 3D nos permite investigar se autophagosomes deve alcançar um tamanho crítico antes da suae fusão com os lisossomos, ou se a autofagia pode proceder simplesmente a um tamanho físico menor. Com efeito, tendo em conta o número de alterações muito pequenas autofagossomas que aparecem após a activação precoce apenas no limite de resolução do microscópio desconvolução, e muito mais claramente resolvido no OMX-iluminação estruturada microscópio 13, pode ser o caso de que a maior parte da actividade autophagic efectivamente ocorre a este nível.
A capacidade de monitorar as células vivas durante o curso da autofagia é extremamente importante, uma vez que as alterações no número e tamanho das autofagossomas numa dada célula pode ser pequeno, em relação às variações dos referidos parâmetros a partir de uma célula para outra. Estudando uma única célula, de estar ao longo do tempo - pode ser mais do que certa de que as alterações morfológicas que observamos são, devido às condições experimentais, ao invés de variância estatística.
Finalmente, com marcadores moleculares específicos, podemos aplicar essas técnicas de imagem para estudar aformação inicial de autophagosomes, para rastrear a origem para as membranas autophagosomal, e identificar possíveis organelas e / ou componentes intracelulares que foram especificamente envolto em autophagosomes durante a indução de autofagia. Esperamos também que esta abordagem irá permitir-nos investigar o papel da autofagia em células de sobrevivência e morte celular, e melhor caracterizar os efeitos de potenciais drogas e inibidores de autofagia.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Apoio Grant: NIH CA165263, CA150197 NIH, NIH CA150197S1 (HJ Kung), NIH CA150197S1 (CA Changou), NSF PHY-0120999 Centro de Biofotônica da Ciência e Tecnologia (DL Wolfson, FYS Chuang), DOD PC073420 (RJ Negrito), a pesquisa Conselho da Noruega, Leiv Eiriksson viagem Grant 209286/F11 (BS Ahluwalia). HJ Kung também agradece o apoio do Fundo Endowment Cancer Comunidade Auburn. RJ Negrito também reconhece o apoio da J. McDonald investidura.
Agradecemos ao Dr. Jenny Wei-Jen Kung e Dr. Bor-wen Wu em DesigneRx para generosa oferta de ADI.
Materiais
| Name | Company | Catalog Number | Comments |
| Arginine Deiminase (ADI) | DesigneRx | ||
| HEPES | Sigma | H4034 | |
| Casein | Sigma | C5890 | |
| Paraformaldehyde | Fisher | 4042 | |
| Saponin | Sigma | S4521 | |
| Alexa anti-mouse 555 | Invitrogen | A21422 | |
| Alexa anti-rabbit 647 | Invitrogen | A21244 | |
| LysoTracker Red DND-99 | Invitrogen | L7528 | |
| anti-Lamp1 | DSHB | H4A3 | |
| anti-Cadherin | Cell Signaling | #3195 | |
| SlowFade Gold | Invitrogen | S36936 | |
| 35 mm poly-d-lysine coated glass bottom plate | MatTek | P35GC-1.5-1.4-C | |
| No.1, 22 mm coverslip | Corning | #2865-22 | |
| Microscope slides | Globe Scientific | 1324G |
Referências
- Kim, R. H., Coates, J. M., Bowles, T. L., McNerney, G. P., Sutcliffe, J., Jung, J. U., Gandour-Edwards, R., Chuang, F. Y., Bold, R. J., Kung, H. J. Arginine deiminase as a novel therapy for prostate cancer induces autophagy and caspase-independent apoptosis. Cancer Res. 69 (2), 700-708 (2009).
- Miyazaki, K., Takaku, H., Umeda, M., et al. Potent growth inhibition of human tumor cells in culture by arginine deiminase purified from a culture medium of a Mycoplasma-infected cell line. Cancer Res. 50, 4522-4527 (1990).
- Takaku, H., Matsumoto, M., Misawa, S., Miyazaki, K. Anti-tumor activity of arginine deiminase from Mycoplasma argini and its growth-inhibitory mechanism. Jpn. J. Cancer Res. 86, 840-846 (1995).
- Levine, B., Klionsky, D. J. Development by self-digestion: molecular mechanisms and biological functions of autophagy. Dev. Cell. 6, 463-477 (2004).
- Mizushima, N., Levine, B., Cuervo, A. M., Klionsky, D. J. Autophagy fights disease through cellular self-digestion. Nature. 451, 1069-1075 (2008).
- Kabeya, Y., Mizushima, N., Ueno, T. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. EMBO J. 19, 5720-5728 (2000).
- Pattingre, S., Espert, L., Biard-Piechaczyk, M., Codogno, P. Regulation of macroautophagy by mTOR and Beclin 1 complexes. Biochimie. 90, 313-323 (2008).
- Maiuri, M. C., Criollo, A., Tasdemir, E., et al. BH3-only proteins and BH3mimetics induce autophagy by competitively disrupting the interaction between Beclin 1 and Bcl-2/Bcl-X(L). Autophagy. 3, 374-376 (2007).
- Crighton, D., Wilkinson, S., O'Prey, J., et al. DRAM, a p53-induced modulator of autophagy, is critical for apoptosis. Cell. 126, 121-134 (2008).
- Dillon, B. J., Prieto, V. G., Curley, S. A., et al. Incidence and distribution of argininosuccinate synthetase deficiency in human cancers: a method for identifying cancers sensitive to arginine deprivation. Cancer. 100, 826-833 (2004).
- Dale, B. M., McNerney, G. P., Thompson, D. L., et al. Cell-to-cell transfer of HIV-1 via virological synapses leads to endosomal virion maturation that activates viral membrane fusion. Cell Host & Microbe. 10 (6), 551-562 (2011).
- Cogger, V. C., McNerney, G. P., Nyunt, T., et al. Three-dimensional structured illumination microscopy of liver sinusoidal endothelial cell fenestrations. J. Struct. Biol. 171 (3), 382-388 (2010).
- York, A. G., Parekh, S. H., Nogare, D. D., et al. Resolution doubling in live, multicellular organisms via multifocal structured illumination microscopy. Nat. Methods. 9 (7), 749-754 (2012).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by factor of two using structured illumination microscopy. Journal of Microscopy. 198, 82-87 (2000).
- Shao, L., Kner, P., Rego, E. H., Gustaffson, M. G. Super-resolution 3D microscopy of live whole cells using structured illumination. Nat. Method. 8 (12), 1044-1046 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados