Method Article
采用先进的3D荧光显微镜定量分析自噬
摘要
细胞自噬是一个无处不在的进程,使细胞蛋白质和细胞器的降解和回收利用。我们采用先进的荧光显微镜可视化和量化小,但必要的,与诱导自噬相关的物理变化,包括自噬体和溶酶体的形成和分布,以及他们融合成autolysosomes。
摘要
手术虽然带有明显的阳痿和尿失禁的风险,前列腺癌是美国男性恶性肿瘤之间的主要形式,传统的化疗方法已基本成功。激素治疗是有效的,在早期阶段,但往往失败,并最终发展为激素难治性肿瘤。我们一直有兴趣在开发针对特定的肿瘤细胞代谢缺陷的疗法。我们最近发现,前列腺肿瘤细胞的特异性缺乏一种酶(精氨琥珀酸合成酶,或ASS)的合成中所涉及的氨基酸精氨酸1。这种情况会导致肿瘤细胞变得依赖于外生精氨酸,他们经过代谢应激游离精氨酸耗尽时由精氨酸脱亚胺酶(ADI)1,10。事实上,我们已经表明,人类前列腺癌细胞CWR22 RV1有效地杀死ADI依赖caspase的细胞凋亡和积极autophaGY(或macroautophagy)1,2,3。细胞自噬是一种进化上保守的过程,使细胞代谢多余的蛋白质在营养饥饿4,5溶酶体破裂。虽然这一途径的重要组成部分,良好的特点6,7,8,9,很多方面的分子机制仍不清楚-特别是,什么是自噬作用的前列腺癌细胞死亡响应ADI治疗后?为了解决这个问题,我们需要的实验方法来衡量的水平和程度在细胞中的自噬反应 - 因为没有已知的分子标记物,可以准确地跟踪这个过程中,我们选择了开发基于成像的方法,使用定量三维荧光显微镜11,12的 。
使用专门CWR22Rv1细胞自噬体和溶酶体的荧光探针标记,我们证明了收购3D图像栈或者无线defield卷积显微镜(以及后来的超高分辨率,结构照明显微镜),可以清晰地捕捉到自噬诱导的早期阶段。市售数字图像分析的应用程序,我们可以很容易地获得统计信息自噬体和溶酶体的数量,规模,分布和程度从任何成像细胞共存。这个信息让我们能够精确地跟踪在活细胞自噬的进展,使我们继续调查自噬作用在癌症化疗。
研究方案
1。第1部分:细胞培养,免疫荧光标记
- CWR22 RV1人前列腺肿瘤细胞生长在盖玻片(#1.5或170微米厚)置于6孔培养板中,含有10%胎牛血清(FBS)和1%青霉素/链霉素/谷氨酰胺的RPMI(敏达,VA)。
- 引起细胞自噬与精氨酸脱亚胺酶(ADI,0.3微克/毫升)的磷酸盐缓冲盐水(PBS)处理选定的样品。
- 固定细胞,用4%多聚甲醛(PFA,Fisher Scientific公司,NH)在PBS中稀释,将100μl每盖玻片,在RT下10分钟。
- 用1毫升HBS / BSA洗涤细胞3次。
- 通透细胞用0.05%皂甙(Sigma公司),HEPES(HBS)/ BSA,加入100μl每盖玻片,在RT下的15分钟。
- 免疫标记,块样品与2.5%的酪蛋白(Sigma公司),每盖玻片100微升,HBS / BSA 1小时RT之前。
- 孵育固定细胞主要抗体稀释在2.5%酪蛋白/ HBS / BSA,每coversl 100微升IP,O / N在4°C。
- 用1毫升HBS / BSA洗涤细胞3次。
- 固定细胞孵育二次抗体稀释在2.5%酪蛋白/ HBS / BSA,每100微升盖玻片,在室温下1小时。
- 用1毫升HBS / BSA洗涤细胞3次。
- SlowFade金制备的盖玻片,在显微镜玻片安装或延长Gold抗淬灭试剂(Invitrogen,CA),含有4',6 - 二脒-2 - 苯基吲哚(DAPI)。
- 指甲油密封盖玻片边缘。为了获得最佳的成像效果,让时间(理想的O / N)为防褪色媒体的彻底渗透细胞。
- 清洁盖玻片的表面与甲醇和/或氯仿。
- 浸油(折射率1.520成像时,在37°C或1.516成像时,在RT)60X 1.42 NA物镜(奥林巴斯,日本)。
- 滑动感兴趣的细胞在显微镜舞台上,并建立重点。
2。第2部分:准备细胞实时成像
- 长大无缝线路22 RV1细胞表达绿色荧光蛋白偶联型轻链3(LC3-GFP)的RPMI培养基中,在35 mm的聚-D-赖氨酸涂覆的玻璃底培养皿(MatTek,MA)中含有10%FBS和1%抗生素。应接种于细胞足够的密度,方便快速扩散,但没有那么多细胞覆盖,成群成像时。
- 治疗选择与ADI(0.3微克/毫升)的PBS的细胞样本。
- 约1小时,在成像之前,稀释红色LysoTracker DND-99(Invitrogen公司,CA),用20毫升的RPMI 1.5微升。含有10%FBS和1%抗生素。使用的解决方案,包含ADI选定样本。预热到37°C前加入培养皿所有媒体。
- 与红的RPMI含有LysoTracker的DND-99 15-45分钟,在37°C孵育
- 约30分钟,在成像之前,打开的WeatherStation环境围封,并允许平衡到37℃和5%CO 2的(humidif与IED的空气)。
- 用PBS洗涤细胞,并与标准的RPMI只含有10%FBS和1%抗生素更换介质。 ADI表示样品。
- 安装35毫米盖玻片底培养皿定制适配器(应用精密公司,WA),并在显微镜舞台上的位置。 60X 1.42 NA物镜用浸油(折射率1.520),并在舞台上的位置安装的培养皿。
3。第3部分:反卷积显微镜和分析
该协议在本节中假定DeltaVision的个人DV卷积显微镜和相关的应用程序套件(应用精密公司,WA)SoftWorX使用。
- 打开显微镜系统,包括采集工作站和仪器控制器。打开Resolve3D应用初始化显微镜阶段,并开启氙灯光源。允许光源的热身和达到稳定的条件下为10分钟。
- 添加滴浸油(折射率1.520活样品在37℃或固定的样品在RT 1.516)60X 1.42 NA物镜,在物镜的中心滑动试样(盖玻片正面朝下)。
- 无论是使用明视场或外部照明和粗调调节旋钮,慢慢地提高胎圈浸油物镜,直到与倒置的玻璃盖接触。 (从这点来说,应该成为焦点的细胞层内几圈微调对焦调节旋钮)。当使用固定的样品在幻灯片上,它被建议,以避免细胞内或附近有气泡的任何领域。当观看直播样品上的玻璃底菜,避免移动物镜玻璃盖玻片的边界之外。
- 自噬体可以识别eGFP的信号(绿色)。溶酶体确定由LysoTracker红(现场采样)或反灯泡1荧光抗体(固定样本)。细胞的细胞核用DAPI圣发现癌宁(固定样本)。
- 选择理想的视野。理想的情况下,细胞应具有良好的荧光信号在所有适当的渠道,并充分地附着盖玻片表面上摊开,使细胞内的内容是很容易想象的。侧的x,y和z聚焦在小的调整,可以控制由计算机使用Acquire3D接口。设置分级1X1或2x2的,这取决于所需的视场(更多的细胞中可以看出分级设置为2x2的领域)。
- 对于一个给定的图像通过一个电池的中部,设置最大像素强度不超过3000个计数的曝光参数( 例如 %的发送功率和曝光时间)。一般情况下,%透光率应尽可能低的设置,相应的曝光时间在1秒内没有提高。重复此过程,每个荧光色进行成像,并为每个单独的视野,以获得最高质量的图像。
- 设置在u稍稍调整焦点分别在顶部和底部的细胞样品,Z堆栈PPER和下限。 Z堆栈中的图像的总数,从而来确定这些限制和层与层之间的间距(默认值:0.2微米)。
- 为了最大限度地减少运动伪影,图像采集过程中,我们建议设置图像“波长,那么Z-Stack的”用于固定试样,“Z-Stack的波长”现场样品采集模式。
- 荧光图像反褶积使用SoftWorX(应用精密,WA)以后分析使用VoloCITY(即兴,现在上Perkin Elmer公司,MA)。
4。第4部分:超高分辨率,结构照明显微镜(OMX)
本节中的协议适用于使用结构照明显微镜OMX(应用精密,WA)。
- 接通主电源和所需的激光器(410纳米,488纳米,和/或532纳米)。等待20英里n为激光热稳定。
- 浸油(用于固定试样在室温下的折射率1.516)60X 1.42 NA物镜。如果气泡出现在油滴,清洁的目的,并再次添加机油。
- 如将样品幻灯片的舞台上,并在必要时,允许10分钟的细胞在盖玻片上定居。
- 初始化收购方案。使用荧光照明,直到锋利的细胞轮廓可以看出,调整的重点。 (在任何时候都应该小心,以尽量减少暴露的细胞荧光激发,直到实际图像采集)。
- 螺旋拼接扫描可以获取的样品预览较大的地区,选择单元格或地区的利益。建议使用短的曝光时间(1-10毫秒),低激磁功率(0.1-1%传输),同时扫描样品或寻找目标。
- GFP-LC3荧光识别自噬。识别溶酶体的Alexa Fluor 555抗LAMP(溶酶体相关膜蛋白)和细胞核用DAPI(固定的样品)。
- 选择的实验条件,包括激发波长,图像区域( 例如,512×512),堆栈的厚度,曝光时间,激光透射百分率。对于超分辨图像,确保结构照明选项启用。在OMX卷积显微镜,选择传统的照明选项。
- 为了获得最佳的重建结果中,选择的实验条件下,例如,在采集过程中在10,000和15,000之间的最大强度计数。漂白如果是一个问题,也可以使用在一个较低的曝光(5000和10000之间的数)的成像。理想情况下,不应该减少计数超过整个采集过程中的两个因素,应避免超过四个因素的减少。如果有必要,减少组的厚度,以确保持续的强度计数。相机饱和,所以在64,000计数最大张力不应该超过此值。为了获得良好的样本,我们发现1%的激光传输的实验条件下,10〜50毫秒的曝光时间。可以反复成像明亮的和耐光样品的首选。需要一个稳定的污渍时间推移,超分辨图像。
- 为了减少噪音,95MHz读出速度(中等速度选项)和采集时间为2毫秒或以上推荐。增加文物得到重建成像,当样品在收购移动。由于这个影响,短期风险,建议非贴壁或快速移动的细胞。
- 多通道成像可以同时或依次进行。顺序模式产量减少串扰和建议。
- 为了减少光漂白,一般建议图像较长的激发波长(532纳米,488纳米和410纳米)。然而,对于在图3所示的实验结果,该顺序是反向。( 即 48纳米,532纳米和410纳米)由于这个特定样品的光稳定性。
- 设置上限/下限的Z堆栈的顶部/底部的细胞移动显微镜阶段,直到稍微失焦。在0.125微米的超分辨率成像所需的每幅图像之间的距离应保持不变,重建软件借此为默认值。
- 图像重建栈使用SoftWorX(应用精密,WA)和分析使用的6.0 VoloCITY(Perkin Elmer公司,MA)。
结果
图1中所示的图象序列显示在第一个80分钟的自噬诱导的在CWR22细胞发生的物理变化。在这方面和其他的研究(图中未示出)中,我们持续观察到:(1)核内的位移相差的小区中心;(2)减少了粘着点,以及(3)朝向中心的自噬体和溶酶体一般易位细胞。此外,我们还观察到自噬体和溶酶体(绿色)(红色)之间的共存小幅增加(黄色),在稍后的时间点。
基于图像流式细胞仪作为示范,我们使用VoloCITY的数字成像应用程序套件(版本6.0,即兴/ Perkin-Elmer公司),以识别,计数,收集统计数据标记的自噬体和溶酶体在CWR22细胞的图像图1。在图2中的图表所示,吨的数量和大小的他自噬体(后,他们最初的形成和外观)不随时间变化的:自噬感应80分钟后,有一个渐进的,但在自噬体的净减少( 图2A)的自噬体的平均规模,并相应增加( 图2B)。此外,有一个可测量的增加,自噬体和溶酶体中的共定位,皮尔逊相关系数分析( 图2C)的基础上。总之,这些研究结果表明,自噬刺激后,无数的小自噬体融合形成较大的自噬体随着时间的推移( 图2D)。虽然图1和图2反映了只有一个单一的成像研究的结果,更严格的定量分析,将需要作出肯定的结论,但它并说明定量荧光显微镜的特征和优点。我们正在积极使用这种技术,在欧细胞自噬的分子机制及过程ŗ继续调查。
图3A显示了图像获取和使用OMX显微镜(右)在DV模式(模拟怀德菲尔德卷积,左)与结构化照明模式重建并排侧比较。横向分辨率提高到高达120纳米,传统的有限衍射显微镜14,15两倍的分辨率。小规模的自噬体和溶酶体( 图3B,用箭头表示)共存显然超高分辨率显微镜,而他们几乎没有明显与传统的荧光成像。
使用解卷积显微镜的优点之一是重建所有的图像转换为3D模型,将揭示不同分子间的更精确的空间信息。 电影1示出了整体画面的CWR22 RV1的细胞undergoi伍自噬在稍后的时间点,其中显着的量之间的共存自噬体和溶酶体开始发生。 E-cadherin的染色(如以白色为主色调)显示靶细胞的轮廓。在电影中 ,三维重建模型自噬体和溶酶体(黄色表示)之间的融合提供了更多的空间细节。我们可以清楚地看到绿色LC3信号和红色溶酶体信号,两个信号的合并可产生黄色信号之间的相互作用。

图1。时间推移图像,CWR22 RV1细胞治疗与ADI CWR22 RV1细胞治疗与ADI公司的80分钟的图像序列。 (2D图像从原始图像栈获得最大的Z轴投影)。 WH伊特虚线表示的轮廓的光的阴影区域代表的细胞,核的位置。

图2。通过分析图像序列如图1所示,作为示范的图像数据量化统计分析。提取统计趋势。 点击这里查看大图 。
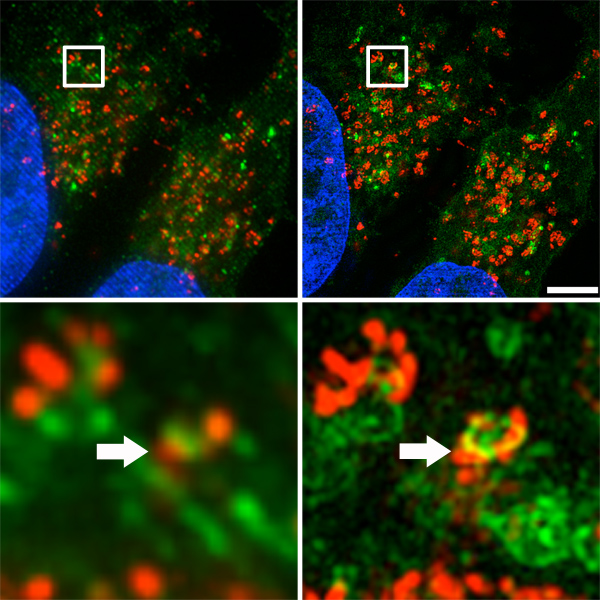
图3。自噬体和溶酶体细胞分布。首页)侧端比较获得的图像和使用OMX显微镜(右)在DV模式(模拟怀德菲尔德卷积,左)和结构化照明模式重建。标尺为5微米。底部)小规模的自噬体和溶酶体(用箭头表示)共存。
电影1,电影2。 CWR22 RV1细胞发生自噬的三维重建。这部电影说明ADI治疗正常后诱导自噬。 Z-Stack的图像重建成3维模型。这部电影所产生的细胞360°水平和360°垂直旋转。绿色信号代表LC3,红色信号表示溶酶体,白色信号代表E-cadherin的,蓝色信号代表细胞核。 点击这里查看或电影1=“_blank”>点击此处查看电影2。
讨论
虽然被广泛接受为一个标准的方法来确认自噬反应6相同的系统(因为我们已经做了),定量三维成像提供了前所未有的信息和细节的复杂过程,细胞自噬对LC3荧光探针标记细胞直接观察。特别是,我们观察到自噬诱导后80分钟内,数百(如果不是数千)在活细胞中的自噬体形成。同样,我们观察到在自噬诱导溶酶体舱室的分布非常有趣的形态学变化。在一个给定的单元格,在自噬体的数量减少,并随着时间的推移他们的平均大小相应增加,表明自噬体的增长大融合与对方相结合的与溶酶体形成autolysosomes的之前。高分辨率三维荧光活细胞成像,让我们去调查,自噬是否必须达到一个临界尺寸江前ê与溶酶体融合,还是自噬可进行简单的在一个较小的物理尺寸。事实上,由于一些非常小的自噬体出现早期活化后,只是在反褶积显微镜的分辨率极限,更清楚在OMX结构照明显微镜13的解决,它可能的情况下,实际上大量的自噬活性发生在这个水平。
能够监测活细胞的自噬过程中是极其重要的,作为自噬体在给定的小区的数量和大小的变化,可以是小的,这些参数的变化相对于从细胞到细胞。通过研究一个单一的,随着时间的推移活细胞 - 我们可以肯定的是,我们观察到的形态变化是由于实验条件,而不是统计方差。
最后,具有特定的分子标记,我们可以应用这些成像技术研究早期形成自噬体,追本溯源的autophagosomal膜的,并找出可能的细胞器和/或细胞内的组件,已专门在自噬体吞噬在细胞自噬诱导。我们也希望,这种方法将使我们研究自噬作用在细胞生存和细胞死亡,并更好地表征潜在的药物和自噬抑制剂的影响。
披露声明
没有利益冲突的声明。
致谢
格兰特支持,国家科学基金会,美国国立卫生研究院NIH CA165263 CA150197:NIH CA150197S1(HJ功夫),:NIH CA150197S1(CA Changou)PHY-0120999 中心生物光子学科学与技术 (DL欧胜,庄花园街),国防部PC073420(RJ大胆),研究挪威理事会,:儿子里夫埃埃里克森旅游格兰特209286/F11(BS阿鲁瓦莉)。 HJ功夫也承认奥本社区癌症基金的支持。 RJ大胆也承认J.麦当劳养老的支持。
我们感谢珍妮博士伟仁西贡博士和吴柏文DesigneRx慷慨供给ADI。
材料
| Name | Company | Catalog Number | Comments |
| Arginine Deiminase (ADI) | DesigneRx | ||
| HEPES | Sigma | H4034 | |
| Casein | Sigma | C5890 | |
| Paraformaldehyde | Fisher | 4042 | |
| Saponin | Sigma | S4521 | |
| Alexa anti-mouse 555 | Invitrogen | A21422 | |
| Alexa anti-rabbit 647 | Invitrogen | A21244 | |
| LysoTracker Red DND-99 | Invitrogen | L7528 | |
| anti-Lamp1 | DSHB | H4A3 | |
| anti-Cadherin | Cell Signaling | #3195 | |
| SlowFade Gold | Invitrogen | S36936 | |
| 35 mm poly-d-lysine coated glass bottom plate | MatTek | P35GC-1.5-1.4-C | |
| No.1, 22 mm coverslip | Corning | #2865-22 | |
| Microscope slides | Globe Scientific | 1324G |
参考文献
- Kim, R. H., Coates, J. M., Bowles, T. L., McNerney, G. P., Sutcliffe, J., Jung, J. U., Gandour-Edwards, R., Chuang, F. Y., Bold, R. J., Kung, H. J. Arginine deiminase as a novel therapy for prostate cancer induces autophagy and caspase-independent apoptosis. Cancer Res. 69 (2), 700-708 (2009).
- Miyazaki, K., Takaku, H., Umeda, M., et al. Potent growth inhibition of human tumor cells in culture by arginine deiminase purified from a culture medium of a Mycoplasma-infected cell line. Cancer Res. 50, 4522-4527 (1990).
- Takaku, H., Matsumoto, M., Misawa, S., Miyazaki, K. Anti-tumor activity of arginine deiminase from Mycoplasma argini and its growth-inhibitory mechanism. Jpn. J. Cancer Res. 86, 840-846 (1995).
- Levine, B., Klionsky, D. J. Development by self-digestion: molecular mechanisms and biological functions of autophagy. Dev. Cell. 6, 463-477 (2004).
- Mizushima, N., Levine, B., Cuervo, A. M., Klionsky, D. J. Autophagy fights disease through cellular self-digestion. Nature. 451, 1069-1075 (2008).
- Kabeya, Y., Mizushima, N., Ueno, T. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. EMBO J. 19, 5720-5728 (2000).
- Pattingre, S., Espert, L., Biard-Piechaczyk, M., Codogno, P. Regulation of macroautophagy by mTOR and Beclin 1 complexes. Biochimie. 90, 313-323 (2008).
- Maiuri, M. C., Criollo, A., Tasdemir, E., et al. BH3-only proteins and BH3mimetics induce autophagy by competitively disrupting the interaction between Beclin 1 and Bcl-2/Bcl-X(L). Autophagy. 3, 374-376 (2007).
- Crighton, D., Wilkinson, S., O'Prey, J., et al. DRAM, a p53-induced modulator of autophagy, is critical for apoptosis. Cell. 126, 121-134 (2008).
- Dillon, B. J., Prieto, V. G., Curley, S. A., et al. Incidence and distribution of argininosuccinate synthetase deficiency in human cancers: a method for identifying cancers sensitive to arginine deprivation. Cancer. 100, 826-833 (2004).
- Dale, B. M., McNerney, G. P., Thompson, D. L., et al. Cell-to-cell transfer of HIV-1 via virological synapses leads to endosomal virion maturation that activates viral membrane fusion. Cell Host & Microbe. 10 (6), 551-562 (2011).
- Cogger, V. C., McNerney, G. P., Nyunt, T., et al. Three-dimensional structured illumination microscopy of liver sinusoidal endothelial cell fenestrations. J. Struct. Biol. 171 (3), 382-388 (2010).
- York, A. G., Parekh, S. H., Nogare, D. D., et al. Resolution doubling in live, multicellular organisms via multifocal structured illumination microscopy. Nat. Methods. 9 (7), 749-754 (2012).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by factor of two using structured illumination microscopy. Journal of Microscopy. 198, 82-87 (2000).
- Shao, L., Kner, P., Rego, E. H., Gustaffson, M. G. Super-resolution 3D microscopy of live whole cells using structured illumination. Nat. Method. 8 (12), 1044-1046 (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。