Method Article
Analisi quantitativa di autofagia usando 3D avanzato microscopia a fluorescenza
In questo articolo
Riepilogo
L'autofagia è un processo onnipresente che permette alle cellule di degradare e riciclare le proteine e organelli. Applichiamo la microscopia a fluorescenza avanzata per visualizzare e quantificare il piccolo, ma, cambiamenti fisici essenziali connessi con l'induzione di autofagia, tra cui la formazione e la distribuzione di autophagosomes e lisosomi, e la loro fusione in autolysosomes.
Abstract
Il cancro della prostata è la forma principale di tumori maligni tra gli uomini negli Stati Uniti Mentre la chirurgia comporta un rischio significativo di impotenza e incontinenza, gli approcci tradizionali chemioterapici sono stati in gran parte senza successo. La terapia ormonale è efficace in fase iniziale, ma spesso non riesce con l'eventuale sviluppo di tumori ormone-refrattario. Siamo stati interessati a sviluppo di terapie mirate specifica carenza metabolica delle cellule tumorali. Recentemente abbiamo dimostrato che le cellule tumorali prostatiche specificamente privi di un enzima (sintasi argininosuccinate, o ASS) coinvolti nella sintesi dell'amminoacido arginina 1. Questa condizione fa sì che le cellule tumorali di diventare dipendente da arginina esogena, e subiscono lo stress metabolico quando arginina libera è esaurito da arginina deiminase (ADI) 1,10. Infatti, abbiamo dimostrato che le cellule del cancro alla prostata umano CWR22 RV1 sono effettivamente uccisi da ADI con apoptosi caspasi-indipendente e autopha aggressivogy (o macroautofagia) 1,2,3. L'autofagia è un processo evolutivamente conservato che permette alle cellule di metabolizzare le proteine indesiderate ripartizione lisosomiale durante il digiuno alimentare 4,5. Anche se i componenti essenziali di questo percorso sono ben caratterizzati 6,7,8,9, molti aspetti del meccanismo molecolare sono ancora poco chiari - in particolare, qual è il ruolo dell'autofagia nella morte-risposta delle cellule tumorali della prostata dopo il trattamento ADI ? Per far fronte a questa domanda, abbiamo richiesto un metodo sperimentale per misurare il livello e la portata della risposta autophagic nelle cellule - e poiché non ci sono marcatori molecolari noti che possono tracciare accuratamente questo processo, abbiamo scelto di sviluppare un approccio di imaging-based, utilizzando quantitativa 3D microscopia a fluorescenza 11,12.
Utilizzando CWR22Rv1 cellule specificamente marcati con sonde fluorescenti per autophagosomes e lisosomi, dimostriamo che gli stack di immagini 3D acquisiti sia con widefield deconvoluzione microscopia (e più tardi, con la super-risoluzione, microscopia strutturato-illuminazione) in grado di catturare in modo chiaro le prime fasi di induzione di autofagia. Con le applicazioni di analisi delle immagini digitali disponibili in commercio, possiamo facilmente ricavare informazioni statistiche sul numero autofagosoma e lisosomi, le dimensioni, la distribuzione, e il grado di colocalizzazione da qualsiasi cellula creata l'immagine. Questa informazione ci permette di tracciare con precisione i progressi di autofagia nelle cellule viventi e consente di continuare la nostra indagine sul ruolo dell'autofagia nella chemioterapia del cancro.
Protocollo
1. Parte 1: coltura cellulare ed Etichettatura Immuno-fluorescenza
- Crescere CWR22 RV1 cellule tumorali della prostata umana su vetro coprioggetto (# 1.5 o 170 micron di spessore) posti in piastre da 6 pozzetti, con RPMI (Mediatech, VA) contenente il 10% di siero fetale bovino (FBS) e 1% di penicillina / streptomicina / glutammina.
- Indurre l'autofagia trattando campioni selezionati con arginina deiminasi (ADI, 0.3 mg / ml) in tampone fosfato salino (PBS).
- Fissare le cellule con paraformaldeide al 4% (PFA, Fisher Scientific, NH) diluita in PBS; 100 microlitri per coprioggetto, per 10 min a RT.
- Lavare le cellule 3 volte con 1 ml HBS / BSA.
- Permeabilize cellule con 0,05% saponina (Sigma, MO) in HEPES (HBS) / BSA, 100 microlitri per coprioggetto, per 15 minuti a RT.
- Prima di immuno-etichettatura, i campioni di blocco con il 2,5% di caseina (Sigma, MO) in HBS / BSA, 100 microlitri per coprioggetto, per 1 ora a temperatura ambiente.
- Incubare le cellule fissate con anticorpi primari diluiti in 2,5% caseina / HBS / BSA, 100 microlitri per coverslip, O / N a 4 ° C.
- Lavare le cellule 3 volte con 1 ml HBS / BSA.
- Incubare le cellule fissate con anticorpi secondari diluiti in 2,5% caseina / HBS / BSA, 100 microlitri per coprioggetto, per 1 ora a temperatura ambiente.
- Lavare le cellule 3 volte con 1 ml HBS / BSA.
- Montare vetrini preparati su regolari vetrini da microscopio in vetro con oro SlowFade o Prolungare reagente antifade Gold (Invitrogen, CA) contenente 4 ',6-diamidino-2-fenilindolo (DAPI).
- Sigillare i bordi coprioggetto con smalto. Per ottenere i migliori risultati di imaging, dare il tempo (idealmente O / N) per i supporti anti-sbiadimento di permeare completamente le cellule.
- Superficie coprioggetto pulito con metanolo e / o cloroformio.
- Aggiungere l'olio di immersione (indice di rifrazione 1.520 quando l'imaging a 37 ° C, o 1.516 se l'imaging a RT) al 1,42 NA lente dell'obiettivo 60X (Olympus, Giappone).
- Posizione della slitta sul palco microscopio e stabilire fuoco con le cellule di interesse.
2. Parte 2: Preparazione di cellule per Live Imaging
- Crescere CWR22 RV1 cellule esprimenti green fluorescent protein-coupled Catena luce 3 (LC3-GFP) in terreni di coltura RPMI contenente 10% FBS e 1% antibiotici da 35 mm rivestite piastre di coltura con fondo poli-D-lisina (MatTek, MA). Le cellule devono essere piastrate a densità sufficiente di facilitare una rapida proliferazione, ma non così tanto che le cellule sono ricoperte e clumped al tempo di imaging.
- Trattare campioni di cellule selezionate con ADI (0,3 mcg / ml) in PBS.
- Circa 1 ora prima di imaging, diluire 1,5 ml di LysoTracker Red ND-99 (Invitrogen, CA) con 20 ml di RPMI. contenente 10% FBS e 1% antibiotici. Utilizzare soluzioni contenenti ADI per i campioni selezionati. Riscaldare tutti i media a 37 ° C prima di aggiungere nel piatto cultura.
- Incubare le cellule con RPMI contenente LysoTracker Red ND-99 per 15-45 minuti a 37 ° C.
- Circa 30 minuti prima di imaging, accendere il contenitore ambientale WeatherStation e lasciare equilibrare a 37 ° C e 5% di CO 2 (con Humidifaria ied).
- Lavare le cellule con PBS e sostituire il supporto con lo standard RPMI contenente solo il 10% FBS e 1% di antibiotici. Aggiungi ADI a campioni come indicato.
- Monte 35 millimetri coverglass-bottom piatti della cultura in adattatore personalizzato (Applied Precision, Inc., WA), e la posizione sul palco microscopio. Usare olio di immersione (indice di rifrazione 1.520) sul 60X 1.42 NA lente dell'obiettivo, e posizionare il piatto cultura montato sul palco.
3. Parte 3: Deconvoluzione Microscopia e analisi
Il protocollo in questa sezione presuppone l'uso del Personal DeltaVision DV deconvoluzione microscopio e associati Softworx Application Suite (Applied Precision, Inc., WA).
- Accendere il sistema di microscopio, compreso stazione di acquisizione e di controllo dello strumento. Aprire l'applicazione Resolve3D per inizializzare il palco microscopio e accendere la sorgente di luce allo xeno. Lasciare 10 minuti per la sorgente luminosa per riscaldarsi e raggiungere condizioni stabili.
- Aggiungere ungoccia di olio di immersione (indice di rifrazione 1.520 per campioni scena a 37 ° C o 1.516 per campioni fissati a RT) sul 60X 1,42 NA lente obiettivo, e campione su vetrino centro (copertura antiscivolo faccia in giù) sopra la lente obiettivo.
- Utilizzando illuminazione in campo chiaro o esterno ed il fuoco grossolana manopola regolare, sollevare lentamente la lente obiettivo finché il tallone di olio di immersione a contatto con il vetro coperchio rovesciato. (Da questo punto, lo strato di cellule dovrebbe entrare in centro in pochi giri di messa a fuoco fine regolare la manopola). Quando si lavora con campioni fissati sui vetrini, si raccomanda di evitare di cellule all'interno o in prossimità di aree con le bollicine. Quando si visualizzano con i campioni dal vivo sul piatto fondo di vetro, evitare di spostare la lente dell'obiettivo di fuori del confine del coprioggetto di vetro.
- Autophagosomes possono essere identificati tramite segnale eGFP (verde). Lisosomi sono identificati da LysoTracker Red (campione dal vivo) o anticorpi fluorescenti anti-Lampada1 (campione fisso). Nuclei delle cellule sono identificate da DAPI staining (campione fisso).
- Selezionare il campo di vista desiderato. Idealmente, le cellule dovrebbero mostrare buon segnale fluorescente in tutti i canali appropriati, e sufficientemente attaccati e sparsi sulla superficie del vetrino in modo che i contenuti intracellulari sono facili da visualizzare. Piccoli aggiustamenti in x laterali, Y e Z-messa a fuoco possono essere controllati dal computer utilizzando l'interfaccia Acquire3D. Impostare binning di 1x1 o 2x2, a seconda campo di vista desiderato (più celle possono essere visti in campo quando binning è impostato su 2x2).
- Per una data immagine attraverso il tronco di una cella, impostare i parametri di esposizione (ad esempio, la potenza di trasmissione% e tempo di esposizione) in modo che l'intensità massima dei pixel non supera i 3.000 conteggi. Generalmente, la trasmissione% dovrebbe essere impostato più basso possibile, senza aumentare il corrispondente tempo di esposizione più di 1 sec. Ripetere questa procedura per ogni colore di fluorescenza per essere ripreso e per ogni campo separato di vista di ottenere immagini di altissima qualità.
- Impostare l'upper limiti inferiore e dello Z-stack regolando il focus solo leggermente sopra la parte superiore e inferiore del campione cellulare, rispettivamente. Il numero totale di immagini in un determinato stack Z sarà pertanto determinato da questi limiti e dalla spaziatura tra strati (valore di default: 0,2 micron).
- Per ridurre al minimo gli artefatti di movimento durante l'acquisizione delle immagini, si consiglia di impostare la modalità di acquisizione delle immagini a "lunghezza d'onda allora z-stack" per campioni fissati, e "z-stack quindi lunghezza d'onda" per i campioni dal vivo.
- Immagini di fluorescenza sono deconvolved utilizzando Softworx (Applied Precision, WA) e successivamente analizzati utilizzando Volocity (Improvision, ora Perkin Elmer, MA).
4. Parte 4: Super-risoluzione, Strutturato illuminazione (OMX) Microscopia
Il protocollo in questa sezione si applica all'uso della OMX Structured Illumination Microscope (Applied Precision, WA).
- Accendere l'alimentazione principale e laser desiderati (410 nm, 488 nm e / o 532 nm). Attendere 20 min per i laser da stabilizzare termicamente.
- Aggiungere l'olio di immersione (indice di rifrazione 1,516 per campioni fissati a RT) sul 60X 1.42 NA lente dell'obiettivo. Se le bolle sono visti nella goccia d'olio, pulire l'obiettivo e aggiungere ancora olio.
- Collocare il campione vetrino sul tavolo e, se necessario, lasciare 10 minuti per le cellule di stabilirsi su vetrino.
- Inizializzare il programma di acquisizione. Utilizzare illuminazione a fluorescenza per regolare la messa a fuoco fino un netto contorno della cella può essere visto. (Si deve prestare attenzione a tutte le ore per ridurre al minimo l'esposizione delle cellule ad eccitazione di fluorescenza, fino a quando l'acquisizione effettiva dell'immagine).
- Una scansione mosaico spirale può essere acquisita per vedere in anteprima le aree più grandi del campione per selezionare le celle o regioni di interesse. Si raccomanda di utilizzare tempi di esposizione brevi (1-10 msec) e bassa potenza di eccitazione (0,1-1% di trasmissione) durante la scansione del campione o la ricerca di bersagli.
- Identificare autophagosomes da GFP-LC3 fluorescenza. Identificare lisosomi da Alexa Fluor 555 anti-LAMP(Proteina di membrana lisosomiale associata) e nuclei di cellule utilizzando DAPI (campioni fissi).
- Scegli condizioni sperimentali tra cui lunghezza d'onda di eccitazione, area dell'immagine (ad esempio 512x512), spessore pila, tempo di esposizione, e la percentuale di trasmissione laser. Per le immagini eccellenti risolte, assicurarsi che l'opzione di illuminazione strutturata è abilitata. Per la microscopia a deconvoluzione sul OMX, selezionare l'opzione di illuminazione convenzionale.
- Per ottenere i migliori risultati di ricostruzione, selezionare condizioni sperimentali tali che il numero massimo di intensità è compresa tra 10.000 e 15.000 durante l'acquisizione. Se fotodecolorazione è un problema, l'imaging ad una esposizione inferiore (conta tra 5.000 e 10.000) può essere utilizzato. Idealmente, i conti non dovrebbero diminuire più di un fattore di due durante l'intera acquisizione, e diminuisce di più di un fattore di quattro dovrebbero essere evitati. Se necessario, ridurre lo spessore pila di garantire un conteggio intensità continua. La fotocamera satura a 64.000 conteggi, quindi massimo inintensità non deve mai superare questo. Per i buoni campioni, abbiamo trovato le condizioni sperimentali da 10 ~ 50 msec di tempo di esposizione con la trasmissione del laser 1%. Campioni luminosi e fotostabili che può essere ripreso più volte sono preferite. Una stabile macchia è richiesto per time-lapse, immagini eccellenti risolti.
- Per ridurre il rumore, una velocità di lettura 95MHz (opzione Media velocità) e tempo di acquisizione di 2 msec o più è raccomandato. Aumento dei manufatti sono ottenuti nel immaginata ricostruito, quando campione si muove durante l'acquisizione. A causa di questo effetto, brevi esposizioni sono raccomandati per le cellule non aderenti o in rapido movimento.
- Imaging multicanale può essere eseguita simultaneamente o in sequenza. Rendimenti modalità sequenziale meno cross-talk ed è quindi raccomandato.
- Per ridurre photobleaching, si raccomanda generalmente di immagine con lunghezze d'onda più lunghe di eccitazione prima (532 nm, 488 nm, e poi 410 Nm). Tuttavia, per i risultati sperimentali mostrati in figura 3, tale ordine era inversod (cioè 48 nm, 532 nm e 410 nm) a causa della fotostabilità di questo particolare campione.
- Impostare il limite superiore / inferiore della Z-stack spostando il tavolino del microscopio fino a quando la parte superiore / inferiore delle celle è leggermente fuori fuoco. La distanza desiderata tra ogni immagine deve essere tenuto costante a 0.125 micron per super risoluzione di imaging, come software di ricostruzione prendere questo valore come impostazione predefinita.
- Ricostruire stack di immagini utilizzando Softworx (Applied Precision, WA) e analizzare con 6,0 Volocity (Perkin Elmer, MA).
Risultati
La sequenza di immagini mostrato in Figura 1 mostra i cambiamenti fisici che si verificano in CWR22 cellule durante il primo 80 min dell'induzione autofagia. In questo ed altri studi (non mostrato) abbiamo costantemente osservato: (1) lo spostamento del nucleo di distanza dal centro della cella; (2) riduzione dei punti di adesione focale, e (3) traslocazione generale autophagosomes e lisosomi verso il centro del cella. Inoltre, abbiamo anche osservato un piccolo aumento della co-localizzazione (indicato in giallo) tra autophagosomes (verde) e lisosomi (rosso), in momenti successivi.
Come dimostrazione di citometria basata sull'immagine, abbiamo usato il Volocity Digital Imaging Application Suite (versione 6.0, Improvision / Perkin-Elmer) per identificare, contare, e raccogliere dati statistici sul autophagosomes e lisosomi etichettati nelle immagini dei CWR22 celle mostrate in figura 1. Come mostrato dai grafici in figura 2, il numero e le dimensioni dei tegli autofagosoma (dopo la loro formazione iniziale e l'aspetto) variano con il tempo: dopo 80 min di induzione dell'autofagia, c'è stata una diminuzione graduale ma netto del numero di autophagosomes (Figura 2A) con un corrispondente aumento della dimensione media delle autophagosomes ( Figura 2B). Inoltre, c'è stato un aumento misurabile della colocalizzazione di autophagosomes e lisosomi, sulla base di analisi di coefficiente di correlazione di Pearson (Figura 2C). Insieme, questi risultati hanno suggerito che dopo stimolazione della autofagia, numerosi piccoli autophagosomes fondono per formare grandi autophagosomes nel tempo (Figura 2D). Mentre figure 1 e 2 riflettono i risultati di uno studio di imaging solo singolo, e saranno necessari analisi quantitativa più rigorosa trarre una conclusione, si illustrano le caratteristiche ei vantaggi di microscopia a fluorescenza quantitativa. Stiamo attivamente utilizzando questa tecnica in our continua investigazione dei meccanismi molecolari e del processo di autofagia cellulare.
Figura 3A mostra un confronto side-by-side di immagini acquisite e ricostruite in modalità DV (simulato widefield deconvoluzione, sinistra) vs modalità strutturata-illuminazione utilizzando il microscopio OMX (a destra). Risoluzione laterale è stata migliorata per fino a 120 nm, il doppio della risoluzione di microscopia convenzionale diffrazione limitata 14,15. Colocalizzazione in piccola scala di autophagosomes e lisosomi (Figura 3B, indicato dalle frecce) erano chiaramente evidente con microscopia super-risoluzione, mentre erano a malapena evidente con imaging di fluorescenza convenzionale.
Uno dei vantaggi di usare Deconvoluzione Microscopia è di ricostruire tutte le immagini in un modello 3D che rivelerà le informazioni spaziali più accurata tra molecole diverse. Movie 1 mostra un quadro complessivo di una CWR22 RV1 cella undergoing autofagia in seguito punto di tempo, in cui la quantità significativa di colocalizzazione tra autofagosoma e lisosomi cominciano a verificarsi. La colorazione E-caderina (come indicato in colore bianco) rivela il contorno della cellula bersaglio. In Movie 2, il modello ricostruito 3D fornisce dettagli più spaziali della fusione tra autophagosomes e lisosomi (come indicato in colore giallo). Siamo in grado di vedere chiaramente l'interazione tra verdi LC3 segnali e segnali lisosomi rossi, e l'unione dei due segnali per produrre segnali gialli.

Figura 1. Time-lapse immagini di CWR22 RV1 cellule trattate con ADI. Sequenza di immagini che mostra CWR22 RV1 cellule trattate con ADI per 80 min. (Immagini 2D ottenute da pile di immagini originali di massima proiezione Z). Il whlinea tratteggiata ite rappresenta il contorno della cella, e la regione di luce ombreggiata rappresenta la posizione del nucleo.

Figura 2. Analisi statistica. Trend statistici sono stati estratti analizzando la sequenza di immagini mostrato in Figura 1 a dimostrazione che i dati immagine è quantificabile. Clicca qui per ingrandire la figura .
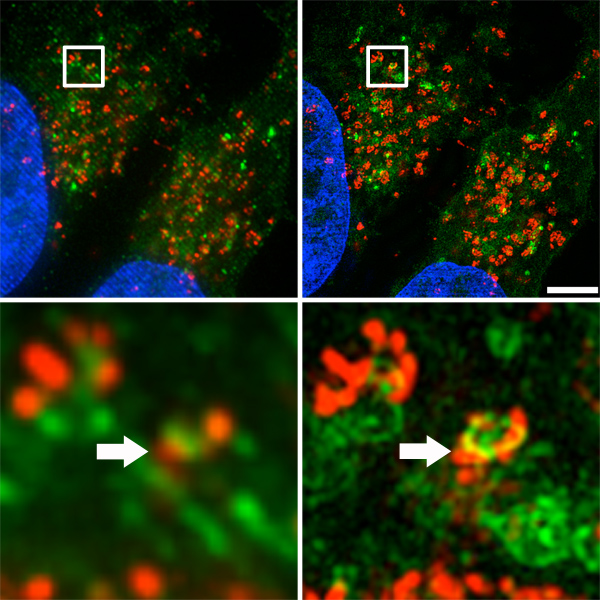
Figura 3. Distribuzione autofagosoma e lisosomi nella cellula. A. Top) Side-by-side confronto delle immagini acquisite e ricostruite in modalità DV (simulato widefield deconvoluzione, a sinistra) e la modalità strutturata-illuminazione utilizzando il microscopio OMX (a destra). Barra di scala rappresenta 5 micron. B. In basso) colocalizzazione in piccola scala di autophagosomes e lisosomi (indicato dalle frecce).
Movie 1 e Movie 2. Ricostruzione 3D di CWR22 RV1 cellulare Sottoposti autofagia. Questo film mostra una normale induzione di autofagia dopo il trattamento ADI. Immagini Z-Stack sono state ricostruite in un modello 3-dimensionale. Questo film è stata generata ruotando la cella 360 ° in orizzontale e 360 ° in verticale. Segnale verde rappresenta LC3, segnale rosso rappresenta lisosomi, segnale bianco rappresenta E-caderina, e il segnale blu rappresenta il nucleo. Clicca qui per vedere film 1 o= "_blank"> Clicca qui per vedere film 2.
Discussione
Mentre l'osservazione diretta di cellule marcate con sonde fluorescenti contro LC3 è ampiamente accettata come un metodo standard per confermare la risposta autophagic 6, l'imaging 3D quantitativa dello stesso sistema (come abbiamo fatto) fornisce informazioni senza precedenti e di dettaglio circa il complesso processo di autofagia cellulare . In particolare, si osserva che centinaia (se non migliaia) di autophagosomes in cellule vive sono formate all'interno di 80 min di induzione di autofagia. Allo stesso modo, si osserva molto interessanti cambiamenti morfologici nella distribuzione dei compartimenti lisosomiali durante induzione dell'autofagia. All'interno di una data cella, la diminuzione del numero autofagosoma e il corrispondente aumento della loro dimensione media nel tempo, suggeriscono che autophagosomes sviluppano più grandi di fusione con l'altro, prima di combinare con i lisosomi per formare autolysosomes. Ad alta risoluzione 3D imaging di fluorescenza delle cellule vive ci permette di indagare se autophagosomes devono raggiungere una dimensione critica before fusione con i lisosomi, o se l'autofagia può procedere semplicemente ad una dimensione fisica più piccola. Infatti, dato il numero di piccolissime autophagosomes che appaiono su attivazione precoce proprio al limite di risoluzione del microscopio deconvoluzione, e molto più chiaramente risolta sulla OMX strutturato-illuminazione microscopio 13, può essere il caso che la maggior parte delle attività effettivamente autofagica avviene a questo livello.
La capacità di monitorare cellule vive durante il corso della autofagia è di fondamentale importanza, in quanto le variazioni del numero e delle dimensioni di autophagosomes in una data cella possono essere piccole, rispetto alle variazioni di questi parametri da cellula a cellula. Attraverso lo studio di un singolo, cellula vivente nel tempo - possiamo essere più certi che i cambiamenti morfologici che osserviamo sono dovuti a condizioni sperimentali, piuttosto che varianza statistica.
Infine, con marcatori molecolari specifici, siamo in grado di applicare queste tecniche di imaging per studiare ilformazione precoce di autophagosomes, per rintracciare l'origine per le membrane autophagosomal, e di individuare possibili organelli e / o componenti intracellulari che sono stati specificamente inghiottito in autophagosomes durante l'induzione dell'autofagia. Speriamo anche che questo approccio ci permetterà di indagare il ruolo dell'autofagia in cellule sopravvivenza e morte cellulare, e meglio caratterizzare gli effetti di potenziali farmaci e inibitori di autofagia.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Concessione di aiuti: NIH CA165263, CA150197 NIH, NIH CA150197S1 (HJ Kung), NIH CA150197S1 (CA Changou), NSF PHY-0.120.999 Centro per Biofotonica Science & Technology (DL Wolfson, FYS Chuang), DOD PC073420 (RJ grassetto), la ricerca Consiglio di Norvegia, Leiv Eiriksson viaggio di Grant 209286/F11 (BS Ahluwalia). HJ Kung riconosce anche il sostegno della Auburn Comunità Cancer Fondo di dotazione. RJ Grassetto riconosce anche il sostegno della J. McDonald dotazione.
Ringraziamo il Dr. Jenny Wei-Jen Kung e il dottor Bor-Wen Wu a DesigneRx per la generosa offerta di ADI.
Materiali
| Name | Company | Catalog Number | Comments |
| Arginine Deiminase (ADI) | DesigneRx | ||
| HEPES | Sigma | H4034 | |
| Casein | Sigma | C5890 | |
| Paraformaldehyde | Fisher | 4042 | |
| Saponin | Sigma | S4521 | |
| Alexa anti-mouse 555 | Invitrogen | A21422 | |
| Alexa anti-rabbit 647 | Invitrogen | A21244 | |
| LysoTracker Red DND-99 | Invitrogen | L7528 | |
| anti-Lamp1 | DSHB | H4A3 | |
| anti-Cadherin | Cell Signaling | #3195 | |
| SlowFade Gold | Invitrogen | S36936 | |
| 35 mm poly-d-lysine coated glass bottom plate | MatTek | P35GC-1.5-1.4-C | |
| No.1, 22 mm coverslip | Corning | #2865-22 | |
| Microscope slides | Globe Scientific | 1324G |
Riferimenti
- Kim, R. H., Coates, J. M., Bowles, T. L., McNerney, G. P., Sutcliffe, J., Jung, J. U., Gandour-Edwards, R., Chuang, F. Y., Bold, R. J., Kung, H. J. Arginine deiminase as a novel therapy for prostate cancer induces autophagy and caspase-independent apoptosis. Cancer Res. 69 (2), 700-708 (2009).
- Miyazaki, K., Takaku, H., Umeda, M., et al. Potent growth inhibition of human tumor cells in culture by arginine deiminase purified from a culture medium of a Mycoplasma-infected cell line. Cancer Res. 50, 4522-4527 (1990).
- Takaku, H., Matsumoto, M., Misawa, S., Miyazaki, K. Anti-tumor activity of arginine deiminase from Mycoplasma argini and its growth-inhibitory mechanism. Jpn. J. Cancer Res. 86, 840-846 (1995).
- Levine, B., Klionsky, D. J. Development by self-digestion: molecular mechanisms and biological functions of autophagy. Dev. Cell. 6, 463-477 (2004).
- Mizushima, N., Levine, B., Cuervo, A. M., Klionsky, D. J. Autophagy fights disease through cellular self-digestion. Nature. 451, 1069-1075 (2008).
- Kabeya, Y., Mizushima, N., Ueno, T. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. EMBO J. 19, 5720-5728 (2000).
- Pattingre, S., Espert, L., Biard-Piechaczyk, M., Codogno, P. Regulation of macroautophagy by mTOR and Beclin 1 complexes. Biochimie. 90, 313-323 (2008).
- Maiuri, M. C., Criollo, A., Tasdemir, E., et al. BH3-only proteins and BH3mimetics induce autophagy by competitively disrupting the interaction between Beclin 1 and Bcl-2/Bcl-X(L). Autophagy. 3, 374-376 (2007).
- Crighton, D., Wilkinson, S., O'Prey, J., et al. DRAM, a p53-induced modulator of autophagy, is critical for apoptosis. Cell. 126, 121-134 (2008).
- Dillon, B. J., Prieto, V. G., Curley, S. A., et al. Incidence and distribution of argininosuccinate synthetase deficiency in human cancers: a method for identifying cancers sensitive to arginine deprivation. Cancer. 100, 826-833 (2004).
- Dale, B. M., McNerney, G. P., Thompson, D. L., et al. Cell-to-cell transfer of HIV-1 via virological synapses leads to endosomal virion maturation that activates viral membrane fusion. Cell Host & Microbe. 10 (6), 551-562 (2011).
- Cogger, V. C., McNerney, G. P., Nyunt, T., et al. Three-dimensional structured illumination microscopy of liver sinusoidal endothelial cell fenestrations. J. Struct. Biol. 171 (3), 382-388 (2010).
- York, A. G., Parekh, S. H., Nogare, D. D., et al. Resolution doubling in live, multicellular organisms via multifocal structured illumination microscopy. Nat. Methods. 9 (7), 749-754 (2012).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by factor of two using structured illumination microscopy. Journal of Microscopy. 198, 82-87 (2000).
- Shao, L., Kner, P., Rego, E. H., Gustaffson, M. G. Super-resolution 3D microscopy of live whole cells using structured illumination. Nat. Method. 8 (12), 1044-1046 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon