Method Article
Nano-Fem: localização de proteínas usando a foto-ativada Microscopia Localização e Microscopia Eletrônica
Neste Artigo
Resumo
Descreve-se um método para localizar proteínas marcadas por fluorescência em micrografias electrónicas. A fluorescência é inicialmente localizado utilizando foto-activado microscopia localização em cortes ultrafinos. Estas imagens são então alinhadas com micrografias electrónicas da mesma secção.
Resumo
Mapear a distribuição de proteínas é essencial para a compreensão da função de proteínas numa célula. A microscopia de fluorescência é usado extensivamente para localização de proteínas, mas contexto subcelular é frequentemente ausente em imagens de fluorescência. Imuno-microscopia electrónica, por outro lado, pode localizar as proteínas, mas a técnica é limitada pela falta de anticorpos compatíveis, a preservação da morfologia pobre e porque a maioria dos antigénios não estão expostos à superfície da amostra. Abordagens correlativas pode obter a imagem de fluorescência a partir de uma célula inteira em primeiro lugar, quer a partir de imuno-fluorescência ou proteínas geneticamente marcadas. A amostra é, em seguida, fixadas e incluídas para a microscopia electrónica, e as imagens são correlacionados 1-3. No entanto, a imagem de baixa resolução da fluorescência e da falta de marcadores fiduciais exclui a localização precisa de proteínas.
Alternativamente, imagens de fluorescência pode ser feito após a preservar o espécime em plástico. EmNesta abordagem, o bloco é seccionado, e as imagens de fluorescência e as micrografias electrónicas de a mesma secção são correlacionados 4-7. No entanto, o limite de difracção de luz na imagem correlacionada obscurece as localizações das moléculas individuais, e a fluorescência frequentemente se estende para além do limite da célula.
Nano-resolução de fluorescência microscopia eletrônica (nano-FEM) é projetado para localizar proteínas em escala nanométrica por imagens usando as mesmas seções de foto-ativado microscopia localização (PALM) e microscopia eletrônica. PALM ultrapassa o limite de difracção por imagem individuais proteínas fluorescentes e, subsequentemente, o mapeamento do centroide de cada mancha fluorescente 8-10.
Nós delineamos a técnica de nano-FEM em cinco etapas. Em primeiro lugar, a amostra é fixa e incorporado utilizando condições que preservam a fluorescência de proteínas marcadas. Em segundo lugar, os blocos de resina são cortados em segmentos ultrafinas (70-80 nm), que são montadossobre uma tampa de vidro. Em terceiro lugar, é criada uma imagem de fluorescência nestas secções, utilizando o microscópio Zeiss PALM. Quarta, estruturas densas de elétrons são gravadas nestas mesmas seções usando um microscópio eletrônico de varredura. Em quinto lugar, as micrografias de fluorescência e de electrões são alinhadas usando partículas de ouro como marcadores fiduciais. Em resumo, a localização subcelular de proteínas marcadas com fluorescência pode ser determinada com alta nanometros em cerca de uma semana.
Protocolo
1. Alta pressão de congelação
- Prepare o crio-protector por adição de 0,2 g de BSA em 1 ml de um meio apropriado para o seu modelo (por exemplo, meio de cultura para cultura de células, M9 para C. elegans). Incubar o tubo num banho de água a 37 ° C até que toda a BSA é dissolvido.
- Antes da congelação, encher o dispositivo de substituição de congelamento-automatizado (Leica AFS) com azoto líquido
- Definir o programa AFS: -90 ° C por 5-30 horas *, 5 ° C / h até -60 ° C, -60 ° C por 2 horas (ou selecione "pausa" opção se você estiver usando Leica AFS 2), 5 ° C / hora a -30 ° C e -30 ° C durante 72 h. Se a máquina de AFS é capaz de ter mais do que cinco passos, a -30 ° C passo deve ser substituída por uma "pausa" opção ajustado para 0 h, e dois passos adicionais devem ser adicionados: 10 ° C / h até -20 ° C e 24 horas à temperatura de -20 ° C. Se a máquina está limitado a cinco passos, criar um outro programa na memória de uma ranhura diferente como se segue: 10 ° C / h a -20 ° C e 24 hr-se a -20 ° C.
* O tempo aqui pode ser ajustada de modo que a -60 ° C passo começa no início da manhã. - Preparar uma solução-mãe de ósmio% tetróxido misturando 0,1 g de cristais de tetróxido de ósmio (EMS, RT19134) com 10 ml de acetona anidra (EMS, # RT10016). Preparar os fixadores de um tubo cónico de 50 ml como se segue. Em primeiro lugar, adicionar 1 ml de água milliQ e dissolver nele 0,02 g de permanganato de potássio (EMS, RT20200). Adicionar 19 ml de acetona e misture bem. Por fim, adicionar 20 ul de uma solução a 1% de ósmio estoque para dentro do tubo. A acetona é um sequestrante de radicais livres 11 e, assim, evita a polimerização de plástico, mas o uso de acetona como um meio de substituição de congelação é necessário para preservar a morfologia devido à sua interacção com os lípidos. Acetona será substituído com etanol antes da infiltração de plástico. Ml 1 para cada alíquota criotubo e manter os fixadores congelados, armazenando os tubos em azoto líquido.
- Encher um transportador de amostra com 20% de BSA ou bactérias.Ambos BSA a 20% e bactérias servir como crio-protectores. A escolha do tipo de espécime do transportador depende da amostra. Para C. elegans, usar um 100 um poço.
- Colocar a amostra no suporte e congelá-lo utilizando um congelador de alta pressão.
- Após a congelação e em atmosfera de azoto líquido, a transferência da amostra para dentro do criotubo contendo fixadores. Certifique-se a amostra permanece sob o líquido em todas as vezes.
- Repita 1.5) - 1.7) até que todas as amostras são congeladas.
- Transferir todos os criotubos na unidade AFS, e retomar o programa.
2. Congelamento de substituição de
- Quando a temperatura atinge -60 ° C, os frascos lugar com 20 ml de acetona a 95% para a AFS.
- Quando a temperatura atinge -50 ° C, substituir os fixadores com acetona a 95% seis vezes ao longo do período de 2 horas.
- Coloque um frasco contendo 20 ml de acetato de uranilo a 0,1% (Polysciences, # 21447-25) em acetona a 95% para a câmara epré-arrefecer a -50 ° C.
- No final da etapa de lavagem, adicionar 1 ml de ~ uma solução de acetato de uranilo a cada frasco. Retomar o programa, se necessário.
- Quando a temperatura atinge -30 ° C, substituir o acetato de uranilo com etanol a 95%, seis vezes ao longo do período de 2 horas.
- Enquanto isso, tornar-se resina de metacrilato de 97% de glicol (GMA, Supplies SPI / Structure Probe, Inc., # 02630-AA) por mistura de 22,3 ml de GMA, 10 ml de metacrilato de n-butilo, 1 ml de água milliQ e 0,2 g de peróxido de benzoílo (catalisador). Armazená-la em frascos de vidro (EMS, # 72632) e preparar-GMA 30%, misturando-o com etanol a 95%. Não armazenar mídia GMA em tubos de plástico, porque a qualidade da polimerização degrada com o armazenamento a longo prazo, em polietileno com base em plástico. Pré-arrefecer a solução GMA a -30 ° C.
Resinas epóxi tradicionais, tais como Epon Araldite e não pode ser utilizada no protocolo, uma vez que são anidro e ácido, que mata as proteínas fluorescentes. Resinas acrílicas, tais como LR White, cuma tolerar uma pequena quantidade de água e pode conservar a proteína fluorescente, mas o seu pH ácido tende a extinguir o fluoróforo 12. GMA tolera, de facto requer, uma pequena quantidade de água e é alcalino (pH 8) 12. GMA é, infelizmente, um pouco frágil. Além disso, a GMA não cross-link com tecidos como resinas epóxi tradicionais. Essas características da GMA inconsistência em causa o corte de qualidade, especialmente quando seccionado mais fino do que 80 nm.
Embora a vida de prateleira do GMA é de cerca de um ano, GMA deve ser usado dentro de 3 meses da compra, para garantir a sua qualidade.
3. Infiltração e Polimerização
3,1-3,3 passos são efectuados nos mesmos utilizados para criotubos freeze-substituição. Infiltração e polimerização são realizados à temperatura de -30 ° C no AFS para preservar a proteína fluorescente.
- Preparar meios de infiltração por mistura da solução de estoque GMA com etanol a 95%. Incubar espécimesem 30% de GMA por 3-5 horas.
- Incubar as amostras em 70% GMA por 4-6 horas.
- Incubar as amostras em 97% GMA durante a noite.
- No dia seguinte, faz-se fresco GMA 97%.
- Transferir as amostras para um molde de encastre (EBSciences, # TC). Preparar um disco de película de ACLAR (EMS, # 50425-10), usando um 3/8 "DISC Punch (Ted Pella, Inc.) e colocar o disco na parte inferior da cápsula BEEM.
- A troca de 97% GMA três vezes ao longo de 6 horas a -30 ° C.
- Após a terceira troca, adicionar o iniciador de N, N-dimetil-p-toluidina (Sigma-Aldrich, # D9912) de GMA, a uma concentração de 1,5 ul / 1 ml GMA e aplicar esta solução para cada um dos moldes de amostra. Imediatamente posicionar a amostra no molde incorporação no AFS.
Note-se que o peróxido de benzoílo é o catalisador e está presente em todas as fases de infiltração de modo a agitação não é necessária. Peróxido de benzoílo não se polimerizar o plástico até que o mesmo fique exposto ao iniciador químico N, N,-dimetil-p-toluidina. O iniciador de pol ativaymerization imediatamente e polimerizar a resina, mesmo em tecidos dentro de 1 hora. No entanto, o abandono de tecidos pode resultar em tecidos profundos devido à polimerização incompleta. Além disso, a GMA não crosslink a amostra para o bloco bem, então separar a amostra da crioprotetor tocando na matriz de bactérias.
Se a polimerização é realizada fora de um AFS, o molde de encaixe deve ser coberto com um disco de Aclar para bloquear a exposição ao oxigénio. GMA não polimerizar totalmente, quando expostos ao oxigénio. - Permitir que o plástico a curar durante a noite, mesmo que polimeriza dentro de 1 hora.
- Armazenar a amostra em um saco de vácuo cheio de gás azoto (Ziploc) no congelador (-20 ° C) até posterior processamento, de modo que as proteínas fluorescentes não são expostos ao oxigénio.
4. Seccionamento
- Seccionamento do GMA-espécimes embebidos pode ser realizada de uma maneira semelhante à do EPON-espécimes embebidos. O cuidado extra deve ser tomadan não molhar a superfície de corte, porque GMA é muito hidrófila e as fitas irão ser empurrados para o banho de água, se eles são humedecidas em ambos os lados.
- Colete fitas de seções (50-80 nm) sobre uma lamela de vidro se um microscópio TIRF está sendo usado para a localização. De outro modo, utilizar uma grelha feita por microscopia electrónica de transmissão. A velocidade de corte deve ser ajustada at1.6 mm / s ou superior. Caso contrário, a fita pode não se formar.
- Seções armazenar a -20 ° C, se não trabalhada imediatamente. Proteger os fluoróforos de luz UV cobrindo detentores secção com folha de alumínio.
5. PALM imagem
- Configure o microscópio PALM de acordo com as recomendações do fabricante. A temperatura de uma câmara EMCCD deve ser ajustado para -70 ° C ou abaixo.
- Aplicar partículas de ouro (# 790122-010 - 2x concentrado, microesferas-nanospheres.com) em solução (cerca de 50 uL) com a amostra a ser trabalhada. Deixar a solução repousar durante 30 segundos sobre as tampaslábio enquanto sob um caso capa preta.
- Remover a solução de ouro fundindo-o para a extremidade da lamela e absorvendo-a com um Kimwipe.
- Insira o lamela em um suporte lamela circular. Se necessário, aplicar vácuo graxa para o aro para manter a lamela no lugar.
- Aplicar o óleo de imersão para a parte inferior da lamela, directamente por baixo das amostras.
- Inserir o porta-amostras para a ranhura no estágio do microscópio. Tome especial cuidado para não tocar os objectivos.
- Ajuste o suporte, de modo que é apertado e centraliza as seções acima do objectivo.
- Localize as seções usando uma lente objetiva de 10x.
- Mude para a lente de 100x objetivo.
- Concentre-se na amostra.
- Localizar uma região de interesse através da observação da intensidade dos sinais de fluorescência. Usar o laser 488 nm e aumentar a intensidade do menu Canais para 10%. Concentre-se em áreas de mais brilhante fluorescência. Note-se que este passo não é necessário se o o regiãof interesse podem ser identificados no campo luminoso por olho.
- Ligar o laser a 561 nm e aumentar a intensidade de 100% de lixívia a autofluorescência de fundo. Lixívia a amostra para ~ 2 min.
- Se o foco muda durante o branqueamento, permitir 5 min a decorrer antes de ajustar o foco e captura de imagens. Esta pausa permite que a temperatura estabilize. Se o alcance de palma está equipado com uma câmara de incubação, ajustar a temperatura a 20 ° C e esperar por ~ 2 horas para estabilizar a temperatura na câmara.
- Quando o branqueamento é completa, ativar o laser de 405 nm e configurá-lo para o menor intensidade.
- Começar a recolher as imagens em 20 quadros por segundo. Nós normalmente recolher 5.000-6.000 quadros por experiência, mas o número de quadros deve ser ajustada, dependendo do objetivo do ensaio. Por exemplo, se todas as proteínas de uma região de interesse deve ser trabalhada, o número de quadro deve ser aumentado.
- Se os sinais são escassos ou desmaio, lentamente ause a intensidade do laser 405 nm.
- Durante o processo de aquisição, não se esqueça de manter as amostras em foco, ajustando cuidadosamente o botão, se necessário.
- Quando as imagens são recolhidos, a análise deve ser realizada PALM. Para tdEos, nós filtrar quaisquer sinais que apresentam fluorescência maior do que 500 ms, porque os sinais são provavelmente devido a autofluorescência.
6. SEM imagem
- Antes de imagem MEV, mancha das secções, utilizando acetato de uranilo a 2,5% (em água) durante 4 min. Lavar o acetato de uranilo cuidadosamente com água milliQ filtrada.
- Após os cortes são secos, o carbono-coat lamela do carbono usando um sputter até a lamela se torna bastante escuro. Aplicar uma extremidade da fita de carbono condutor na extremidade da lamela e a outra extremidade no topo do metal de modo que os electrões que se acumulam sobre a superfície da lamela são fundamentados.
- Monte o espécime para a câmara de SEM (FEI Nova Nano).
- Insira o detector VCD.
- Fechar a câmara e bombeá-lo usando a configuração de alto vácuo.
- Uma vez evacuada, abrir a válvula de coluna de modo que o feixe de electrões é aplicado a uma amostra.
- Realizar um alinhamento de rotina de feixe de elétrons.
- Localize o espécime.
- Uma vez que o foco é ajustado, vincular o estágio exemplar e trazer ao palco para 5 mm abaixo do pólo.
- Tomar uma imagem de baixa ampliação da amostra (~ 5000 x).
- Zoom para a região de interesse e a obtenção de ampliação elevada (50.000 x) imagens.
- Mover para a próxima seção, e repita o passo 6.10) e 6.11) até que todas as seções são gravadas.
7. Alinhando Imagens de palma e EM
- Abra o Photoshop e as imagens obtidas de SEM.
- Criar uma nova janela com dimensões de 5.000 x 5.000 pixels e 300 pixels de resolução / polegada.
- Copie o baixo ampliação da imagem SEM na nova janela (Figura 1A).
- Dimensionar a imagem para que ela se enchetoda a janela, usando a manipulação de transformação (Command + T para um Mac).
- Copie o maior ampliação SEM imagens e escalá-los, se necessário.
- Inverter a imagem TIRF soma horizontalmente, selecionando rotação de imagem na barra de menu drop-down imagem.
- Copie e cole a soma imagem TIRF (de PALM) em uma nova camada.
- A escala da imagem utilizando a transformada de manipulação e, em seguida, se necessário girar para coincidir com a fluorescência das partículas de ouro (setas brancas na Figura 1A), com as correspondentes estruturas visualizadas em SEM (Figura 1B).
- Copie a imagem PALM em uma nova camada e aplicar a mesma transformação (Figura 1C). Uma imagem com maior ampliação pode ser extraído a partir desta imagem (Figura 2).
- Para a apresentação, copie a imagem PALM transformado em uma nova camada. Selecione os sinais de palma, mas não do fundo usando "gama de cores" no "select" no menu drop-down. Certifique-se de select um pixel de fundo como um pixel de referência e ligue o "inverter" a opção.
- Corte os sinais PALM desejados e colá-los para uma nova camada.
- Aplicar transparência para a camada de fundo, e configurá-lo para 10%. Isto permite a visualização dos sinais PALM, fazendo o fundo transparente, sem comprometer a sua intensidade (Figura 2D).
8. Resultados representativos
Histona marcada com tdEos podem ser expressos de forma estável no nemátodo C. elegans, e os animais transgénicos foram processados utilizando o protocolo descrito acima. As micrografias PALM e eletrônica foram adquiridos da mesma seção (Figura 1). Para alinhar as imagens, a soma de imagem TIRF, que resume a fluorescência ao longo do tempo inteiro, é sobreposto sobre a micrografia electrónica. As nanopartículas de ouro aparecem em ambas as micrografias de fluorescência e de electrões e pode ser utilizado para alinhar as duas imagens usando a transf 'função orm 'em Photoshop (Figura 1 A e B). Em seguida, o mesmo valor "transformada" foi aplicado à imagem PALM (Figura 1C). Nesta ampliação, detalhe estrutural não pode ser distinguida, de modo que com o zoom em uma região próxima da extremidade de topo da micrografia (Figura 2). Na imagem de alta ampliação, detalhes subcelulares, como um núcleo, um nucléolo, poros nucleares, retículo endoplasmático e pôde ser observado. Além disso, as moléculas marcadas histona são exclusivamente localizada no núcleo, mas não para o nucléolo, como esperado. A microscopia eletrônica de PALM e correlativa, assim, permite a localização de proteínas com a maior resolução.
Cinco problemas podem comprometer a qualidade das imagens. Em primeiro lugar, os danos de cristais de gelo pode distorcer ultraestrutura (Figura 3A e B). Colocar as amostras a uma bactéria crio-protectores, tais como, o que reduz a propagação de cristais de gelo, pode reduzir este dano. No entanto,deve-se ainda filtrar amostras por microscopia eletrônica e descartar aqueles com artefatos de congelamento. Segundo, GMA não cruzar-link para tecidos como resinas epóxi, e, assim, muitas vezes exemplares romper com o plástico ao redor e esticar, encolher ou até mesmo cair fora da seção (Figura 3C e D). A dissecção da amostra fora das bactérias ou outros crio-protector antes prevê a incorporação de uma maior aderência do plástico para a amostra (Figura 3C). De igual modo, as estruturas, tais como gotículas de gordura no intestino dissociar frequentemente a partir de tecidos, devido à ausência de ligação cruzada (Figura 3E). Terceiro, a polimerização incompleta das causas de plástico que se estendem ou dobragem de tecidos (Figura 3F), a presença de oxigénio na amostra, também impede a polimerização de GMA. Em quarto lugar, a fraca qualidade do corte GMA muitas vezes resulta em uma morfologia inconsistente (Figura 3G e H). Seções GMA deve ser cortado em70 nm ou mais espesso e com uma velocidade de cerca de 1,6 mm / s para minimizar os artefactos de seccionamento. Quinto, autofluorescência de fundo de poeira sobre a lamela ou seção é inevitável. Autofluorescência do pó podem ser minimizados através da utilização pré-limpas lamelas e evitando a contaminação por pó de kimwipes e papel de filtro, tal como descrito no protocolo. O programa de análise de palma pode editar os sinais a partir da amostra ou de plástico que apresentam fluorescência mais do que os sinais típicos de proteínas fluorescentes (Figura 2 A e B). A imagem final será, portanto, livre de tais artefatos.

Figura 1. Alinhando micrografias de fluorescência e eletrônica, utilizando nanopartículas de ouro. (A) Uma micrografia de electrões baixa ampliação de uma secção transversal de C. elegans que expressam as tdEos marcadas histona :: HIS-11. Branco umrrows indicar 100 nm elétron-denso nanopartículas de ouro aplicado antes da palma de imagens que servem como pontos de referência. (B) As contas de ouro fluorescência por exposição à luz ~ nm 580 e criar pontos de referência na imagem de fluorescência. A soma TIRF imagem é alinhada sobre uma micrografia electrónica com base na localização dos pontos de referência. A soma imagem TIRF representa todos os fotões detectados pela câmera durante o tempo experimental. Note-se que os pontos brilhantes no canto superior esquerdo (seta branca) surgir a partir dos aglomerados de partículas de ouro. (C) Uma imagem PALM é então adicionado à micrografia electrónica e girada e traduzida a partir dos valores determinados a partir do alinhamento da imagem TIRF soma em (B). para ver figura maior .

Figura 2. Correlativo nano-FEM utilizando proteínas de fusão histona. (A) Soma imagem TIRF de tdEos :: HIS-11 adquiridos de uma seção fina (70nm). (B) imagem PALM Correspondente da tdEos :: HIS-11. Autofluorescência (seta branca) com duração de mais de 500 ms foi filtrado pelo programa PALM. (C) Electron Micrograph de um núcleo adquiridos a partir da mesma secção. (D) a imagem PALM correlativa e micrografia eletrônica de tdEos :: HIS-11. A fluorescência é fortemente localizada na cromatina no núcleo.
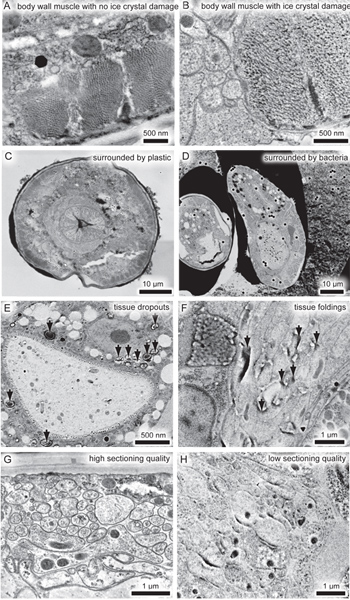
Figura 3. Problemas associados com nano fem. (A) Micrografia eletrônica de um C. elegans músculo do corpo sem danos de cristais de gelo. (B) micrografia de electrões de um músculo do corpo com os danos de cristais de gelo. Em vez de cortes transversais distintas, filamentos de actina e miosina são recolhidos em agregados devido à formação de cristais de gelo. (C, D) baixa ampliação micrografia eletrônicas, que mostra a dissociação de vermes dos meios circundantes. A secção é mais distorcida numa amostra que está rodeado pela bactéria crio-protectores (D) do que quando a amostra encontra-se rodeado de plástico (C). A crio-protector bacteriana no Gallette deve ser dissecado a partir da amostra antes da incorporação de plástico fixa. Note-se que o animal à direita (D) foi seccionado obliquamente, e, assim, a forma não é devido à distorção dos tecidos. (E) micrografia de electrões de intestino, mostrando encaixes dos tecidos (setas pretas). (F) micrografia de electrões de anel nervo, mostrando dobramento das secções devido à polimerização incompleta do plástico (setas pretas). (G, H) Eletromicrografias de neurônios a partir da mesma amostra, seccionado, em datas diferentes. A conservação de tecidos é excelente em um dia (G), mas a morfologia tal é obscurecida pela qualidade inconsistente seccionamento (H). clique aqui para vfigura maior iew.
Discussão
Aqui descrevemos como preservar proteínas fluorescentes em plástico, localizar as proteínas fluorescentes nas seções, ea imagem do ultra-estrutura por microscopia eletrônica. Proteínas foram localizadas abaixo do limite de difração usando microscopia PALM para resolução de nanômetros. Para adaptar este protocolo para espécimes específicos, os seguintes parâmetros devem ser considerados: fluoróforo, quantificação e alinhamento.
A escolha de uma proteína fluorescente ou fluoróforo orgânico depende da aplicação e o sistema de modelo. Temos testado numa variedade de proteínas fluorescentes, incluindo GFP, YFP, citrino, mEosFP, mEos2, tdEos, Morange, PA-mCherry e Dendra 12. A preservação da fluorescência de cada fluoróforo foram semelhantes, sugerindo que todas as proteínas fluorescentes podem ser preservados utilizando o método descrito. Escolhemos tdEos porque expressa bem em C. elegans, as proteínas se funcional, quando fundido a tdEos, e porque a suafoto-ativação características foram ideal para microscopia PALM. No entanto, a agregação ou não expressão de tdEos foi ocasionalmente observado 12.
Dependendo da aplicação, um fluoróforo diferente, pode ser mais adequado. Em muitos casos, não é necessária a utilização de uma proteína de foto-activado fluorescente. Simples correlativo microscopia electrónica de fluorescência não requer foto-activado de proteína fluorescente. GFP ou corantes orgânicos podem ser utilizados para imagem de fluorescência de proteínas marcadas em secções acima do limite de difracção. Por exemplo, pode-se axon uma imagem num neuropil usando microscopia de fluorescência e correlacionar o sinal de fluorescência com um axónio particular em uma micrografia electrónica por imagiologia de fluorescência num microscópio de fluorescência. Outras técnicas de super-resolução, tais como microscopia de emissão estimulada esgotamento (STED) 12, chão de microscopia de depleção estado seguido de retorno molécula individual (GSDIM) 13, emicroscopia de iluminação estruturada (SIM) 14, não precisam de foto-activados proteínas fluorescentes. Além disso, as técnicas de super-resolução de imagem que utilizam corantes orgânicos 9,15,16 ou a propriedade intrínseca de sondas fluorescentes 17 são facilmente aplicáveis.
Em PALM, o número de moléculas pode ser quantificada por fluorescência de cada molécula está separado espacialmente e temporalmente. No entanto, a quantificação pode ser enganosa por quatro motivos: oxidação, subestimação, overcounting e superexpressão. Em primeiro lugar, uma fracção das proteínas fluorescentes pode ser desnaturado ou oxidados durante o processamento da amostra 5,12. Embora ~ 90% do sinal de fluorescência foi preservada por meio da fixação e incorporação no nosso protocolo, a oxidação da proteína fluorescente pode ocorrer depois de o espécime foi seccionado e a superfície exposta ao oxigénio. Em segundo lugar, a activação do foto-activáveis proteínas é estocástico, e, assim, as moléculas podem ser múltiplasativado em um ponto de difração dada limitada 8. Fluorescência das moléculas múltiplas mesma aparece como uma mancha, e, assim, o número total de proteínas será contado regressivamente. Em terceiro lugar, um problema semelhante pode levar a overcounting. Na palma, cada proteína fluorescente é localizado e então "apagados" pelo branqueamento. No entanto, as proteínas fluorescentes pode retornar do estado escuro sem estar permanentemente branqueada 18. Tais moléculas, então, ser contado várias vezes. Em quarto lugar, as proteínas marcadas são expressos como transgenes e são muitas vezes presentes em múltiplas cópias, o que pode conduzir a sobre-expressão. Assim, a quantificação de palma podem ser usados para estimar com precisão, mas não determinar o número de moléculas de uma dada localização.
O alinhamento de uma imagem PALM com uma micrografia de electrões pode também ser um desafio devido à diferença de resolução de microscopia óptica e electrónica e distorção provocada pelo feixe de electrões. Partículas de ouro servir bem locdesmineralizada marcadores fiduciais em microscopia eletrônica. No entanto, fluoresecence de partículas de ouro não é foto-ativado, e aparece como uma mancha de difração limitada de grande porte. Assim, a colocação de uma imagem de fluorescência ao longo de uma fotografia de microscopia electrónica é uma estimativa. Distorções também pode surgir a partir de interações dos elétrons com a secção de plástico. Resinas acrílicas tais como GMA são menos estáveis sob o feixe de electrões, e as dimensões do que o plástico pode ser alterada. Sob estas circunstâncias, o alinhamento da fluorescência com ultraestrutura pode requerer a transformação não linear dos marcadores fiduciais.
Divulgações
Produção e livre acesso a este artigo é patrocinado pela Carl Zeiss, Inc.
Agradecimentos
Agradecemos Harald Hess e Eric Betzig para o acesso ao microscópio PALM para a prova de princípio experimentos, Richard Fetter para a partilha de protocolos de fixação, reagentes e encorajamento. Agradecemos Michael Davidson, Geraldine Seydoux, Stefan Eimer, Rudolf Leube, Keith Nehrke, Christian Frøkjr-Jensen, Aude Ada-Nguema e Marc Hammarlund para construções de DNA. Agradecemos também a Carl Zeiss Inc. para fornecer acesso à Zeiss PAL-M, uma versão beta do P.1 Elyra microscópio Zeiss PALM.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente ou do equipamento | Companhia | Número de catálogo | Comentários |
| Alta pressão congelador | ABRA | HPM 010 | EMPact e HPM 100 da Leica ou hpf-01 a partir de Wohlwend também podem ser usados. |
| Unidade de substituição automática congelamento | Leica | AFS 2 | AFS 1 também pode ser usado. |
| Zeiss PALM | Zeiss | ELYRA P.1 | Nikon e Vutara também vender microscópios PALM comerciais. |
| Microscópio eletrônico de varredura | FEI | Nova nano | Outros microscópios de alta resolução de SEM pode ser usado. |
| Acetona | EMS | RT10016 | |
| Etanol | Sigma-aldrich | 459844-1L | |
| Ósmio tetroxide | EMS | RT19134 | |
| O permanganato de potássio | EMS | RT20200 | |
| Albumina de soro bovino | Sigma-aldrich | A3059-50G | |
| Acetato de uranilo | Polysciences | 21447-25 | pH de acetato de uranilo a partir desta empresa é ligeiramente maior. |
| Metacrilato de glicol (GMA) | SPI | 02630-AA | Ácido baixo, grau TEM. |
| N, N-dimetil-p-toluidina | Sigma-aldrich | D9912 | |
| Cryo frascos | Nalgene | 5000-0020 | |
| Frascos de vidro | EMS | 72632 | |
| Filme Aclar | EMS | 50425-10 | |
| BEEM cápsula | EBSciences | TC | Polipropileno |
| 3/8 "DISCO socos | Ted Pella | 54741 | |
| As nanopartículas de ouro | microesferas-nanospheres.com | 790122-010 | Pedir solução concentrada 2x |
Referências
- Polishchuk, R. S., et al. Correlative light-electron microscopy reveals the tubular-saccular ultrastructure of carriers operating between Golgi apparatus and plasma membrane. J. Cell Biol. 148, 45-58 (2000).
- Oberti, D., Kirschmann, M. A., Hahnloser, R. H. R. Correlative microscopy of densely labeled projection neurons using neural tracers. Front. Neuroanat. 4, 24(2010).
- Bishop, D., et al. Near-infrared branding efficiently correlates light and electron microscopy. Nat. Meth. 8, 568-570 (2011).
- Sims, P. A., Hardin, J. D. Fluorescence-integrated transmission electron microscopy images: integrating fluorescence microscopy with transmission electron microscopy. Methods Mol. Biol. 369, 291-308 (2007).
- Micheva, K., Smith, S. Array tomography: a new tool for imaging the molecular architecture and ultrastructure of neural circuits. Neuron. 55, 25-36 (2007).
- Nixon, S. J., et al. A single method for cryofixation and correlative light, electron microscopy and tomography of zebrafish embryos. Traffic. 10, 131-136 (2009).
- Kukulski, W., et al. Correlated fluorescence and 3D electron microscopy with high sensitivity and spatial precision. J. Cell Biol. 192, 111-119 (2011).
- Betzig, E., et al. Imaging Intracellular Fluorescent Proteins at Nanometer Resolution. Science. 313, 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat. Methods. 3, 793-795 (2006).
- Hess, S. T., Girirajan, T. P. K., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophys. J. 91, 4258-4272 (2006).
- Weibull, C., Christiansson, A. Extraction of proteins and membrane lipids during low temperature embedding of biological material for electron microscopy. J. Microsc. 142, 79-86 (1986).
- Watanabe, S., et al. Protein localization in electron micrographs using fluorescence nanoscopy. Nat. Methods. 8, 80-84 (2011).
- F lling, J., et al. Fluorescence nanoscopy by ground-state depletion and single-molecule return. Nat. Methods. 5, 943-945 (2008).
- Gustafsson, M. G. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy. J. Microsc. 198, 82-87 (2000).
- Wombacher, R., et al. Live-cell super-resolution imaging with trimethoprim conjugates. Nature Methods. 7, 717-719 (2010).
- Dertinger, T., Colyer, R., Iyer, G., Weiss, S., Enderlein, J. Fast, background-free, 3D super-resolution optical fluctuation imaging (SOFI). PNAS. 106, 22287-22292 (2009).
- Burnette, D. T., Sengupta, P., Dai, Y., Lippincott-Schwartz, J., Kachar, B. Bleaching/blinking assisted localization microscopy for superresolution imaging using standard fluorescent molecules. PNAS. , (2011).
- Annibale, P., Vanni, S., Scarselli, M., Rothlisberger, U., Radenovic, A. Identification of clustering artifacts in photoactivated localization microscopy. Nat. Meth. 8, 527-528 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados