Method Article
Nano-FEM: la localisation des protéines en utilisant la microscopie Localisation Photo-actif et microscopie électronique
Dans cet article
Résumé
Nous décrivons une méthode pour localiser les protéines marquées par fluorescence en microscopie électronique. Fluorescence est d'abord localisée en utilisant la microscopie localisation photo-activé sur coupes ultrafines. Ces images sont ensuite alignées à micrographies électroniques de la même section.
Résumé
Cartographie de la répartition des protéines est essentielle pour comprendre la fonction des protéines dans une cellule. La microscopie à fluorescence est largement utilisé pour la localisation des protéines, mais le contexte subcellulaire est souvent absent dans les images de fluorescence. Immuno-microscopie électronique, d'autre part, peuvent localiser les protéines, mais la technique est limitée par un manque d'anticorps compatibles, la préservation de la morphologie des pauvres et parce que la plupart des antigènes sont pas exposés à la surface de l'échantillon. Approches corrélatives peuvent acquérir l'image de fluorescence d'une cellule toute la première, soit à partir de l'immuno-fluorescence ou de protéines génétiquement marqués. L'échantillon est ensuite fixé et intégré pour la microscopie électronique, et les images sont corrélées 1-3. Cependant, l'image basse résolution fluorescence et le manque de marqueurs fiduciaires obstacle à la localisation précise des protéines.
Alternativement, l'imagerie de fluorescence peut être fait après préservant l'échantillon dans du plastique. Encette approche, le bloc est sectionné, et les images de fluorescence et micrographies électroniques de la même section sont corrélées 4-7. Cependant, la limite de diffraction de la lumière dans l'image corrélé obscurcit les emplacements des molécules individuelles, et la fluorescence s'étend souvent au-delà de la limite de la cellule.
Nano-résolution de fluorescence par microscopie électronique (nano-FEM) est conçu pour localiser les protéines à l'échelle nanométrique par imagerie les mêmes sections en utilisant la microscopie localisation photo-activé (PALM) et la microscopie électronique. PALM surmonte la limite de diffraction individuels d'imagerie par fluorescence des protéines et ensuite la cartographie du centroïde de chaque tache fluorescente 8-10.
Nous décrivons la technique de nano-FEM en cinq étapes. Tout d'abord, l'échantillon est fixe et intégrée en utilisant des conditions qui préservent la fluorescence des protéines marquées. D'autre part, les blocs de résine sont coupés en segments ultraminces (70-80 nm) qui sont montéssur un verre de recouvrement. Troisièmement, la fluorescence est représenté dans ces sections en utilisant le microscope Zeiss PALM. Quatrièmement, électrons structures denses sont imagés dans ces mêmes articles à l'aide d'un microscope électronique à balayage. Cinquièmement, les micrographies de fluorescence et électronique sont alignées en utilisant des particules d'or comme marqueurs fiduciaires. En résumé, la localisation subcellulaire des protéines marquées par fluorescence peut être déterminée à une résolution nanométrique dans environ une semaine.
Protocole
1. Congélation à haute pression
- Préparer la cryo-protecteur en ajoutant 0,2 g de BSA dans 1 ml d'un milieu approprié pour votre spécimen (par exemple, un milieu de culture pour les cultures cellulaires, M9 pour C. elegans). Incuber le tube dans un bain à 37 ° C l'eau jusqu'à ce que tout est dissous BSA.
- Avant la congélation, remplissez le gel automatisé de substitution appareil (Leica AFS) avec de l'azote liquide
- Réglez le programme AFS: -90 ° C pour hr * 5-30, 5 ° C / h à -60 ° C, -60 ° C pendant 2 heures (ou sélectionnez "pause" option si vous utilisez Leica AFS 2), 5 ° C / h à -30 ° C et -30 ° C pendant 72 h. Si la machine AFS est capable d'avoir plus de cinq étapes, l'étape de -30 ° C doit être remplacé par une "pause" option est réglée à 0 h, et deux nouvelles mesures devraient être ajoutés: 10 ° C / h à -20 ° C et 24 heures à -20 ° C. Si la machine est limitée à cinq étapes, mettre en place un autre programme dans un emplacement mémoire différent comme suit: 10 ° C / h à -20 ° C et 24 hr à -20 ° C.
* Le temps ici peut être ajustée de sorte que l'-60 ° C étape commence tôt le matin. - Préparez 1% solution de tétroxyde d'osmium en mélangeant 0,1 g de cristaux tétroxyde d'osmium (EMS, RT19134) avec 10 ml d'acétone anhydre (EMS, # RT10016). Préparer les fixateurs dans un tube conique de 50 ml comme suit. D'abord, ajouter 1 ml d'eau milliQ, puis se dissoudre dans l'0,02 g de permanganate de potassium (EMS, RT20200). Ajouter 19 ml d'acétone et mélangez bien. Enfin, ajouter 20 ul d'une solution mère à 1% d'osmium dans le tube. L'acétone est un capteur de radicaux libres 11 et empêche ainsi la polymérisation du plastique, mais l'utilisation de l'acétone comme un moyen gel de substitution est nécessaire pour préserver la morphologie en raison de son interaction avec les lipides. Acétone sera remplacé par de l'éthanol avant l'infiltration en plastique. Aliquote de 1 ml dans chaque tube cryogénique et de garder les fixateurs gelés en stockant les tubes dans l'azote liquide.
- Remplissez un porte-échantillon avec de la BSA 20% ou de bactéries.Les deux BSA 20% et les bactéries servir de cryo-protecteurs. Le choix du type de support du spécimen dépend de l'échantillon. Pour C. elegans, utilisez de 100 um bien.
- Placer l'échantillon dans le support et le congeler en utilisant un congélateur à haute pression.
- Après congélation et sous azote liquide, transférer l'échantillon dans le cryotube contenant fixateurs. Assurez-vous que l'échantillon reste dans le liquide à tout moment.
- Répétez 1,5) - 1,7) jusqu'à ce que tous les échantillons sont congelés.
- Transférez tous les cryotubes dans l'unité AFS, et reprendre le programme.
2. Gel de substitution
- Lorsque la température atteint -60 ° C, les flacons contenant placer 20 ml d'acétone à 95% dans l'AFS.
- Lorsque la température atteint -50 ° C, remplacer les fixateurs à l'acétone 95% six fois au cours de la période de 2 heures.
- Placez un flacon contenant 20 ml d'acétate d'uranyle 0,1% (Polysciences, # 21447-25) dans l'acétone à 95% dans la chambre etpré-refroidir le à -50 ° C.
- À la fin de l'étape de lavage, d'ajouter ~ 1 ml de solution d'acétate d'uranyle dans chaque flacon. Annulez la pause le programme si nécessaire.
- Lorsque la température atteint -30 ° C, remplacer l'acétate d'uranyle avec de l'éthanol 95% six fois au cours de la période de 2 heures.
- Dans le même temps, représentent 97% de résine méthacrylate (GMA, Fournitures SPI / Structure Probe, Inc # 02630-AA) en mélangeant 22,3 ml GMA, 10ml de n-butyle, le méthacrylate, 1 ml d'eau MilliQ, et 0,2 g de peroxyde de benzoyle (catalyseur). Conservez-le dans des flacons en verre (EMS, # 72632) et préparer 30% GMA en le mélangeant avec de l'éthanol à 95%. Ne pas stocker les supports GMA dans des tubes en plastique, car la qualité de la polymérisation se dégrade avec le stockage à long terme à base de polyéthylène plastiques. Pré-refroidir la solution GMA à -30 ° C.
Résines époxydes traditionnels tels que Araldite et Epon ne peut pas être utilisé dans le protocole car ils sont anhydres et acides qui désaltère protéines fluorescentes. Les résines acryliques, tels que LR blanc, cune tolérer une petite quantité d'eau et peut préserver la protéine fluorescente, mais son pH acide tend à éteindre le fluorophore 12. GMA tolère, nécessite en effet, une petite quantité d'eau et est alcaline (pH 8) 12. GMA est malheureusement un peu fragile. En outre, GMA ne réticulation avec des tissus comme les résines époxy traditionnels. Ces caractéristiques de l'incohérence à cause de GMA sectionnement en particulier lorsque la qualité de coupe plus mince que 80 nm.
Bien que la durée de vie de GMA est d'environ un an, GMA devrait être utilisé dans les 3 mois suivant l'achat pour s'assurer de sa qualité.
3. Infiltration et polymérisation
Étapes 3.1 à 3.3 sont effectuées dans les mêmes utilisés pour cryotubes gel de substitution. Imprégnation et la polymérisation sont effectuées à -30 ° C dans l'AFS pour préserver la protéine fluorescente.
- Préparez le support d'infiltration en mélangeant la solution de GMA avec de l'éthanol à 95%. Incuber les échantillonsà 30% pour les 3-5 h GMA.
- Incuber les échantillons dans 70% GMA pour 4-6 heures.
- Incuber les échantillons dans 97% GMA nuit.
- Le lendemain, maquillage fraîche 97% GMA.
- Transférer les échantillons à un moule d'enrobage (EBSciences, # TC). Préparez un disque de film ACLAR (EMS, # 50425-10) en utilisant un 3/8 "DISC Punch (Ted Pella Inc) et placez le disque dans le fond de la capsule BEEM.
- Bourse 97% de la GMA trois fois plus de 6 heures à -30 ° C.
- Après le troisième échange, ajouter l'initiateur de N, N-diméthyl-p-toluidine (Sigma-Aldrich, # D9912) à GMA à une concentration de 1,5 ul / ml et 1 GMA appliquer cette solution à chaque échantillon de moule. Immédiatement positionner l'échantillon dans le moule d'enrobage en AFS.
Noter que le peroxyde de benzoyle est le catalyseur et est présent au cours de toutes les étapes d'infiltration afin d'agitation n'est pas nécessaire. Peroxyde de benzoyle ne pas polymériser la matière plastique jusqu'à ce qu'elle soit exposée à l'amorceur chimique N, N-diméthyl-p-toluidine. L'initiateur active polymerization immédiatement et polymériser la résine, même dans les tissus à l'intérieur de 1 h. Néanmoins, les décrocheurs tissus peut entraîner des tissus profonds en raison de la polymérisation incomplète. En outre, GMA ne réticule pas à l'échantillon et le bloc, afin de séparer l'échantillon de l'agent cryoprotecteur en appuyant sur la matrice de bactéries.
Si la polymérisation est effectuée en dehors d'un AFS, le moule d'enrobage doit être recouvert d'un disque Aclar de bloquer l'exposition à l'oxygène. GMA ne se polymérise pas complètement quand il est exposé à l'oxygène. - Laissez le plastique pour guérir du jour au lendemain, même si elle se polymérise en 1 h.
- Stocker l'échantillon dans un sac sous vide rempli de gaz d'azote (Ziploc) dans le congélateur (-20 ° C) jusqu'à ce que le traitement ultérieur de sorte que les protéines fluorescentes ne sont pas exposés à l'oxygène.
4. Sectionnement
- Sectionnement de GMA-embarqués échantillons peut être effectuée d'une manière similaire à celle-EPON embarqués spécimens. Redoublez de prudence devrait être de prendren ne pas mouiller la surface de sectionnement, car GMA est très hydrophile et les rubans sont enfoncés dans le bain d'eau si elles sont mouillées sur les deux faces.
- Recueillir des rubans de sections (50-80 nm) sur une lamelle de verre d'un microscope TIRF si est utilisé pour la localisation. Sinon, utilisez une grille de microscopie électronique à transmission. La vitesse de coupe doit être réglé at1.6 mm / s ou plus. Dans le cas contraire, un ruban peut pas se former.
- Sections conserver à -20 ° C si non imagée immédiatement. Protéger les fluorophores de la lumière UV en couvrant les détenteurs section de papier d'aluminium.
5. PALM Imaging
- Mettre en place le microscope PALM selon les recommandations du fabricant. La température d'un appareil photo EMCCD doit être réglé à -70 ° C ou moins.
- Appliquer des particules d'or (# 790122-010 - 2x concentré, microsphères-nanospheres.com) dans la solution (environ 50 ul) de l'échantillon à imager. Laisser la solution reposer pendant 30 secondes sur les couverturestandis que sous la lèvre d'un cas de protection noire.
- Retirer la solution de l'or en soufflant sur le bord de la lamelle et l'absorbant avec un Kimwipe.
- Insérez la lamelle dans un porte-lamelle circulaire. Si nécessaire, appliquer de la graisse à vide à la jante de garder la lamelle en place.
- Appliquer de l'huile d'immersion de la face inférieure de la lamelle couvre-objet, directement au-dessous des échantillons.
- Insérez le porte-échantillon dans la fente sur la platine du microscope. Faites attention à ne pas toucher les objectifs.
- Réglez le support de sorte qu'elle soit bien serrée et centre les sections ci-dessus de l'objectif.
- Localisez les sections à l'aide d'un objectif 10x.
- Modification de la lentille objectif 100x.
- Concentrez-vous sur l'échantillon.
- Localiser une zone d'intérêt en observant la puissance des signaux de fluorescence. Utiliser le laser 488 nm et augmenter l'intensité dans le menu Chaînes à 10%. Mettre l'accent sur les domaines de la brillante fluorescence. Notez que cette étape n'est pas nécessaire si la région of intérêts peuvent être identifiées dans le domaine lumineux à l'œil nu.
- Mettez le laser à 561 nm et augmenter l'intensité à 100% à l'eau de Javel sur l'autofluorescence de fond. Bleach l'échantillon pour ~ 2 min.
- Si le focus est modifié pendant le blanchiment, permettent 5 min à s'écouler avant de régler la mise au point et la capture d'images. Cette pause permet à la température de se stabiliser. Si le champ d'application PALM est équipé d'une chambre d'incubation, régler la température à 20 ° C et attendre environ 2 heures pour stabiliser les températures dans la chambre.
- Lorsque blanchiment est terminée, activer le laser 405 nm et réglez-le sur la plus faible intensité.
- Commencer à recueillir des images à 20 images par seconde. En général, nous recueillons 5.000-6.000 images par expérience, mais le nombre d'images doit être ajustée en fonction de l'objectif de l'expérience. Par exemple, si toutes les protéines dans une région d'intérêt doivent être imagé, le numéro de châssis doit être augmenté.
- Si les signaux sont rares, voire faible, lentement augSE l'intensité laser 405 nm.
- Au cours du processus d'acquisition, assurez-vous de conserver les échantillons au point en ajustant avec soin le bouton si nécessaire.
- Lorsque les images sont collectées, l'analyse PALM doit être effectuée. Pour tdEos, nous filtrer les signaux qui sont fluorescents plus de 500 msec parce que ces signaux sont probablement dus à l'autofluorescence.
6. SEM Imaging
- Avant d'imagerie SEM, tacher les sections à l'aide d'acétate d'uranyle 2,5% (dans l'eau) pendant 4 min. Laver l'acétate d'uranyle à fond avec de l'eau milliQ filtré.
- Une fois les sections sont séchées, le carbone-coat la lamelle en utilisant un carbone par pulvérisation jusqu'à ce que la lamelle devient assez sombre. Appliquer une extrémité de ruban de carbone conducteur au niveau du bord de la lamelle couvre-objet et l'autre extrémité sur l'embout métallique de sorte que les électrons qui s'accumulent sur la surface de la lamelle couvre-objet sont mis à la terre.
- Placer le spécimen dans la chambre de SEM (FEI Nouvelle Nano).
- Insérez le détecteur de VCD.
- Fermer la chambre et le pomper en utilisant le réglage vide poussé.
- Une fois vide, ouvrir la soupape de colonne de sorte que le faisceau d'électrons est appliqué à un échantillon.
- Effectuez un alignement systématique de faisceau d'électrons.
- Localisez le spécimen.
- Une fois la mise au point est réglé, relier la platine et mettre en place la scène à 5 mm en dessous de la pièce polaire.
- Prendre une image à faible grossissement de l'échantillon (~ 5.000 x).
- Zoom dans la région de l'intérêt et d'obtenir un fort grossissement (50.000 x) des images.
- Passez à la section suivante et répétez l'étape 6.10) et 6.11) jusqu'à ce que toutes les sections sont imagés.
7. Aligner les images PALM et EM
- Ouvrez Photoshop et les images acquises au MEB.
- Créer une nouvelle fenêtre avec les dimensions de 5.000 x 5.000 pixels et 300 pixels / pouce résolution.
- Copiez le faible grossissement image MEB dans la nouvelle fenêtre (figure 1A).
- Redimensionner l'image afin qu'elle remplisse jusqu'àtoute la fenêtre en utilisant la manipulation de transformation (Ctrl + T pour un Mac).
- Copiez le plus fort grossissement images MEB et d'intensifier autant que nécessaire.
- Retourner l'image horizontalement FRBR somme en sélectionnant rotation de l'image dans la barre de menu de l'image déroulante.
- Copiez et collez l'image somme TIRF (à partir de PALM) dans un nouveau calque.
- Redimensionnez l'image en utilisant la transformée de manipulation et tournez si nécessaire pour correspondre à la fluorescence des particules d'or (flèches blanches dans la figure 1A) avec les structures correspondantes visualisées en SEM (figure 1B).
- Copiez l'image PALM dans un nouveau calque et appliquer la même transformation (figure 1C). A plus fort grossissement, l'image peut être extraite de cette image (figure 2).
- Pour la présentation, copiez l'image PALM transformé en un nouveau calque. Sélectionnez les signaux de palme, mais pas d'arrière-plan à l'aide de "gamme de couleurs" dans le "select" dans le menu déroulant. Assurez-vous de selØter un pixel d'arrière-plan comme un pixel de référence et activer le "inverser" option.
- Couper les signaux PALM souhaités et les coller dans un nouveau calque.
- Appliquer la transparence pour la couche de fond, et le mettre à 10%. Ceci permet la visualisation des signaux PALM en faisant l'arrière-plan transparent, sans compromettre leur intensité (figure 2D).
8. Les résultats représentatifs
Histone marqués avec tdEos peut être exprimée de façon stable dans le nématode C. elegans, et les animaux transgéniques ont été traités selon le protocole décrit ci-dessus. Les micrographies PALM et électronique ont été acquis auprès de la même section (Figure 1). Pour aligner les images, l'image somme FRBR, qui résume la fluorescence au cours du temps ensemble, se superpose sur la micrographie électronique. Les nanoparticules d'or apparaissent dans les micrographies de fluorescence et électronique et peuvent être utilisés pour aligner les deux images en utilisant la transf 'orm «fonction dans Photoshop (figure 1A et B). Ensuite, la valeur de la même «transformation» a été appliqué à l'image PALM (figure 1C). A ce grossissement, détail structurel ne peut être distinguée, nous zoomé dans une région proche de l'extrémité supérieure de la micrographie (Figure 2). Dans l'image à fort grossissement, les détails subcellulaires comme un noyau, un nucléole, les pores nucléaires, et le réticulum endoplasmique a pu être observée. De plus, les molécules marquées sont exclusivement localisés histones du noyau, mais non à l'nucléole, comme prévu. La microscopie corrélative PALM et électronique permet ainsi la localisation des protéines à la plus haute résolution.
Cinq problèmes peuvent compromettre la qualité des images. Tout d'abord, les dommages cristaux de glace peuvent fausser ultrastructure (figure 3A et B). Placer les spécimens dans une bactérie cryo-protecteurs tels que, ce qui réduit la propagation des cristaux de glace, peut réduire ces dommages. Néanmoins,encore faut-il filtrer des échantillons par microscopie électronique et de jeter ceux avec des objets de congélation. Deuxièmement, GMA ne pas réticuler les tissus comme les résines époxy, et donc spécimens souvent se détacher de la matière plastique environnante et d'étirement, de réduire ou même tomber de la section (figure 3C et D). Dissection de l'échantillon à l'écart des bactéries ou d'autres cryo-protecteur avant enrobage assure une plus grande adhérence de la matière plastique de l'échantillon (figure 3C). De même, des structures telles que des gouttelettes lipidiques dans l'intestin souvent dissocier les tissus en raison de l'absence de réticulation (figure 3E). En troisième lieu, la polymérisation incomplète des causes d'étirage plastique, le pliage de tissus (figure 3F), la présence d'oxygène dans l'échantillon empêche également la polymérisation de l'EMM. En quatrième lieu, la mauvaise qualité de sectionnement de GMA se traduit souvent par une morphologie irrégulière (Figure 3G et H). GMA sections doivent être coupés à70 nm ou plus d'épaisseur et à une vitesse de l'ordre de 1,6 mm / s afin de minimiser les artefacts de sectionnement. Cinquièmement, l'autofluorescence fond de la poussière sur la lamelle ou de l'article est inévitable. Autofluorescence de la poussière peut être minimisé en utilisant prénettoyés lamelles et en évitant la contamination de la poussière de papier filtre Kimwipes et tel que décrit dans le protocole. Le programme d'analyse peut PALM modifier des signaux provenant de l'échantillon ou en matière plastique qui émettent une fluorescence plus de signaux typiques des protéines fluorescentes (Figure 2 A et B). L'image finale sera donc libre de ces artefacts.

Figure 1. Alignement micrographies de fluorescence et électronique en utilisant des nanoparticules d'or. (A) Une micrographie électronique à faible grossissement d'une coupe transversale de C. elegans exprimant les tdEos taggés histones :: SA-11. Blanc unrrows indiquent 100 nm dense aux électrons des nanoparticules d'or appliquée avant l'imagerie PALM qui servent de points repères. (B) Les perles d'or fluorescence lors de l'exposition à la lumière ~ nm 580 et créer des points repères dans l'image de fluorescence. L'image TIRF somme est aligné sur une micrographie électronique sur la base de l'emplacement des points repères. L'image TIRF somme représente tous les photons détectés par l'appareil au cours du temps expérimental. Notez que les points lumineux sur le coin supérieur gauche (flèche blanche) proviennent des grappes de particules d'or. (C) Une image PALM est ensuite ajouté à la micrographie électronique et rotation et convertis sur la base des valeurs déterminées à partir de l'alignement de l'image TIRF somme en (B). Cliquez ici pour agrandir la figure .

Figure 2. Corrélative nano-FEM en utilisant des protéines de fusion des histones. (Un) Somme FRBR image de tdEos :: SA-11 acquis auprès d'une section mince (70 nm). (B) image PALM correspondant de tdEos :: SA-11. Autofluorescence (flèche blanche) d'une durée supérieure à 500 ms a été filtré par le programme PALM. (C) micrographie électronique d'un noyau acquis à partir de la même section. (D) image PALM corrélative et micrographie électronique de tdEos :: SA-11. La fluorescence est bien localisée dans la chromatine dans le noyau.
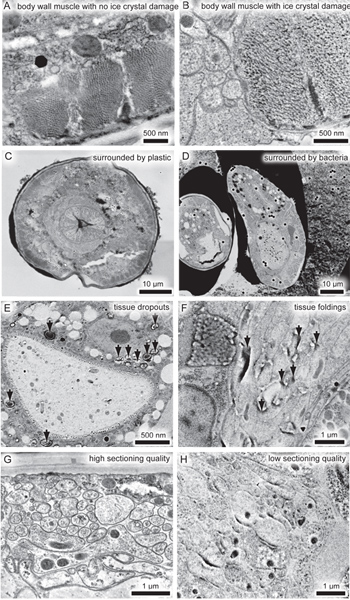
Figure 3. Problèmes associés à nano FEM. (A) d'un microscope électronique C. muscles du corps elegans sans dommage de cristaux de glace. (B) micrographie électronique d'un muscle du corps avec des dommages de cristaux de glace. Au lieu de sections distinctes, l'actine et la myosine sont regroupées en agrégats dus à la formation de cristaux de glace. (C, D) faible grossissement en microscopie électroniques, montrant la dissociation des vers du milieu environnant. La section est plus déformé dans un échantillon qui est entouré par les bactéries cryo-protecteurs (D) que lorsque l'échantillon est entouré par le plastique (C). La cryo-protecteur bactérienne dans la galette doit être disséquée à partir de l'échantillon fixe avant enrobage en matière plastique. Notez que l'animal sur la droite dans (D) a été sectionnée obliquement, et donc la forme n'est pas due à la déformation des tissus. (E) Micrographie électronique de l'intestin, montrant les abandons de tissus (flèches noires). (F) Micrographie électronique d'anneau nerveux, montrant pliage de sections en raison de la polymérisation incomplète de plastique (flèches noires). (G, H) micrographies électroniques de neurones à partir du même échantillon, sectionnés à des dates différentes. La préservation des tissus est superbe sur une journée (G), mais la morphologie telle est obscurcie par la qualité de sectionnement incompatibles (H). Cliquez ici pour vchiffre plus grand ue.
Discussion
Nous décrivons ici comment préserver protéines fluorescentes en plastique, de localiser les protéines fluorescentes dans les sections, et l'image de l'ultrastructure en microscopie électronique. Les protéines ont été localisées en dessous de la limite de diffraction en microscopie PALM à résolution nanométrique. Pour adapter ce protocole aux spécimens particuliers, les paramètres suivants doivent être considérés: fluorophore, la quantification et l'alignement.
Le choix d'une protéine fluorescente ou fluorophore organique dépend de l'application et du système modèle. Nous avons testé une variété de protéines fluorescentes, y compris EGFP, YFP, Citrine, mEosFP, mEos2, tdEos, Morange, PA-mCherry et Dendra 12. La préservation de la fluorescence de chaque fluorophore était similaire, ce qui suggère que toutes les protéines fluorescentes peuvent être conservés à l'aide de la méthode décrite. Nous avons choisi tdEos car il a bien exprimé dans C. elegans, les protéines demeurées fonctionnelles lorsqu'il est fusionné à tdEos, et parce que sonphoto-activation caractéristiques sont optimales pour la microscopie PALM. Cependant, l'expression d'agrégation ou pas de tdEos a parfois été observée 12.
Selon l'application, un fluorophore différent peut-être mieux. Dans de nombreux cas, il n'est pas nécessaire d'utiliser une protéine fluorescente photo-activée. Simple microscopie électronique corrélative de fluorescence ne nécessite pas de photo-activé protéine fluorescente. GFP ou des colorants organiques peuvent être utilisés à fluorescence image à partir de protéines marquées dans les sections ci-dessus de la limite de diffraction. Par exemple, une boîte d'image d'un axone dans un neuropile en utilisant la microscopie par fluorescence et de corréler le signal de fluorescence d'un axone particulier dans une micrographie électronique d'imagerie par fluorescence sur l'un microscope à fluorescence. D'autres techniques de super-résolution, telles que la microscopie d'émission stimulée épuisement (STED) 12, la microscopie appauvrissement de l'état fondamental suivi par le retour molécule individuelle (GSDIM) 13, etmicroscopie illumination structurée (SIM) 14, n'ont pas besoin de photo-activés protéines fluorescentes. En outre, de super-résolution des techniques d'imagerie utilisant des colorants organiques 9,15,16 ou la propriété intrinsèque des sondes fluorescentes 17 sont facilement applicables.
Dans PALM, le nombre de molécules peuvent être quantifiés en raison de fluorescence de chaque molécule est séparé spatialement et temporellement. Cependant, la quantification peut être trompeuse pour quatre raisons: oxydation, sous-estimation, surdénombrement, et la surexpression. Tout d'abord, une fraction des protéines fluorescentes peuvent être dénaturé ou oxydée pendant le traitement d'échantillons 5,12. Bien que ~ 90% du signal de fluorescence a été préservée grâce à la fixation et l'inclusion dans notre protocole, l'oxydation de la protéine fluorescente peut se produire après que l'échantillon a été sectionné et la surface exposée à l'oxygène. D'autre part, l'activation des protéines photo-activables est stochastique, et ainsi de multiples molécules peuvent êtreactivés dans une tache de diffraction donnée limitée 8. Fluorescence des molécules multiples apparaîtra comme une tache, et donc le nombre total de protéines vont être sous-estimés. Troisièmement, un problème similaire peut conduire à surdénombrement. Dans PALM, chaque protéine fluorescente est localisée puis «effacées» par le blanchiment. Toutefois, les protéines fluorescentes peut revenir de l'état sombre sans être définitivement blanchi 18. Ces molécules seront ensuite comptée plusieurs fois. Quatrièmement, les protéines marquées sont exprimés en transgènes et sont souvent présents en plusieurs exemplaires, ce qui peut conduire à une surexpression. Par conséquent, la quantification de PALM peut être utilisée pour estimer, mais pas de déterminer avec précision le nombre de molécules dans un lieu donné.
L'alignement d'une image PALM avec une micrographie électronique peut aussi être difficile à cause de la différence de résolution en microscopie optique et électronique et la distorsion provoquée par le faisceau d'électrons. Les particules d'or servent bien loclisés marqueurs repères dans micrographies électroniques. Cependant, fluoresecence de particules d'or n'est pas photo-activé, et apparaît comme une grande tache limitée par la diffraction. Ainsi, la mise en place d'une image de fluorescence sur une micrographie électronique est une estimation. Distorsions peuvent également survenir des interactions des électrons avec la partie plastique. Résines acryliques tels que les GMA sont moins stables sous le faisceau d'électrons, et les dimensions de la matière plastique peut être modifié. Dans ces circonstances, l'alignement de la fluorescence avec ultrastructure peut exiger une transformation non linéaire des marqueurs fiduciaires.
Déclarations de divulgation
Production et le libre accès à cet article est sponsorisé par Carl Zeiss, Inc
Remerciements
Nous tenons à remercier Harald Hess et Eric Betzig pour l'accès au microscope PALM pour preuve de principe expériences, Richard Fetter pour les protocoles de partage de fixation, des réactifs et des encouragements. Nous remercions Michael Davidson, Geraldine Seydoux, Stefan Eimer, Rudolf Leube, Keith Nehrke, Christian Frøkjr-Jensen, Aude Ada-Nguema et Marc Hammarlund pour des constructions d'ADN. Nous remercions également Carl Zeiss Inc pour fournir un accès à l'Zeiss PAL-M, une version bêta du microscope Zeiss Elyra P.1 PALM.
matériels
| Name | Company | Catalog Number | Comments |
| Le nom du réactif ou de l'équipement | Entreprise | Numéro de catalogue | Commentaires |
| Haute pression congélateur | ABRA | HPM 010 | EMPACT et HPM 100 de Leica ou hpf-01 de Wohlwend peut également être utilisé. |
| Automatisé unité cryosubstitution | Leica | AFS 2 | AFS 1 peut également être utilisé. |
| Zeiss PALM | Zeiss | ELYRA P.1 | Nikon et Vutara vendent aussi des microscopes PALM commerciales. |
| Microscope électronique à balayage | FEI | Nouvelle nano | D'autres haute résolution des microscopes MEB peuvent être utilisées. |
| Acétone | EMS | RT10016 | |
| L'éthanol | Sigma-Aldrich | 459844-1L | |
| Osmium tetroxide | EMS | RT19134 | |
| Le permanganate de potassium | EMS | RT20200 | |
| L'albumine de sérum bovin | Sigma-Aldrich | A3059-50G | |
| L'acétate d'uranyle | Polysciences | 21447-25 | pH de l'acétate d'uranyle de cette compagnie est légèrement plus élevé. |
| Méthacrylate (GMA) | SPI | 02630-AA | Acide faible, la qualité du TEM. |
| N, N-diméthyl-p-toluidine | Sigma-Aldrich | D9912 | |
| Tubes Cryo | Nalgene | 5000-0020 | |
| Flacons en verre | EMS | 72632 | |
| Film de Aclar | EMS | 50425-10 | |
| BEEM capsule | EBSciences | TC | Polypropylène |
| 3/8 "DISC poinçons | Ted Pella | 54741 | |
| Les nanoparticules d'or | microsphères-nanospheres.com | 790122-010 | Demander 2x solution concentrée |
Références
- Polishchuk, R. S., et al. Correlative light-electron microscopy reveals the tubular-saccular ultrastructure of carriers operating between Golgi apparatus and plasma membrane. J. Cell Biol. 148, 45-58 (2000).
- Oberti, D., Kirschmann, M. A., Hahnloser, R. H. R. Correlative microscopy of densely labeled projection neurons using neural tracers. Front. Neuroanat. 4, 24(2010).
- Bishop, D., et al. Near-infrared branding efficiently correlates light and electron microscopy. Nat. Meth. 8, 568-570 (2011).
- Sims, P. A., Hardin, J. D. Fluorescence-integrated transmission electron microscopy images: integrating fluorescence microscopy with transmission electron microscopy. Methods Mol. Biol. 369, 291-308 (2007).
- Micheva, K., Smith, S. Array tomography: a new tool for imaging the molecular architecture and ultrastructure of neural circuits. Neuron. 55, 25-36 (2007).
- Nixon, S. J., et al. A single method for cryofixation and correlative light, electron microscopy and tomography of zebrafish embryos. Traffic. 10, 131-136 (2009).
- Kukulski, W., et al. Correlated fluorescence and 3D electron microscopy with high sensitivity and spatial precision. J. Cell Biol. 192, 111-119 (2011).
- Betzig, E., et al. Imaging Intracellular Fluorescent Proteins at Nanometer Resolution. Science. 313, 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat. Methods. 3, 793-795 (2006).
- Hess, S. T., Girirajan, T. P. K., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophys. J. 91, 4258-4272 (2006).
- Weibull, C., Christiansson, A. Extraction of proteins and membrane lipids during low temperature embedding of biological material for electron microscopy. J. Microsc. 142, 79-86 (1986).
- Watanabe, S., et al. Protein localization in electron micrographs using fluorescence nanoscopy. Nat. Methods. 8, 80-84 (2011).
- F lling, J., et al. Fluorescence nanoscopy by ground-state depletion and single-molecule return. Nat. Methods. 5, 943-945 (2008).
- Gustafsson, M. G. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy. J. Microsc. 198, 82-87 (2000).
- Wombacher, R., et al. Live-cell super-resolution imaging with trimethoprim conjugates. Nature Methods. 7, 717-719 (2010).
- Dertinger, T., Colyer, R., Iyer, G., Weiss, S., Enderlein, J. Fast, background-free, 3D super-resolution optical fluctuation imaging (SOFI). PNAS. 106, 22287-22292 (2009).
- Burnette, D. T., Sengupta, P., Dai, Y., Lippincott-Schwartz, J., Kachar, B. Bleaching/blinking assisted localization microscopy for superresolution imaging using standard fluorescent molecules. PNAS. , (2011).
- Annibale, P., Vanni, S., Scarselli, M., Rothlisberger, U., Radenovic, A. Identification of clustering artifacts in photoactivated localization microscopy. Nat. Meth. 8, 527-528 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon