Method Article
Saccharomyces cerevisiae에서 반수체의 짝짓기 효율 측정
요약
이 연구에서는 효모 Saccharomyces cerevisiae 의 짝짓기 효율을 정량화하는 강력한 방법이 설명되어 있습니다. 이 방법은 종 분화 연구에서 접합 전 장벽의 정량화에 특히 유용합니다.
초록
사카로미세스 세레비시아(Saccharomyces cerevisiae)는 유전학, 진화 및 분자 생물학에서 널리 사용되는 모델 유기체입니다. 최근 몇 년 동안, 그것은 또한 종 분화와 관련된 문제를 연구하는 인기있는 모델 유기체가되었습니다. 효모의 수명주기는 무성 생식 단계와 유성 생식 단계를 모두 포함합니다. 진화 실험을 수행하기 쉽고 유기체의 짧은 생성 시간은 생식 장벽의 진화에 대한 연구를 가능하게합니다. 두 가지 짝짓기 유형(a 및 α)이 짝을 이루어 a/α 이배체를 형성하는 효율을 짝짓기 효율이라고 합니다. 반수체 사이의 짝짓기 효율의 감소는 접합 전 장벽을 나타냅니다. 따라서, 두 반수체 사이의 생식 격리 정도를 정량화하기 위해서는 짝짓기 효율을 정량화하는 강력한 방법이 필요합니다. 이를 위해 간단하고 재현성이 높은 프로토콜이 여기에 제시됩니다. 이 프로토콜에는 YPD 플레이트에 반수체를 패치하고, 동일한 수의 반수체를 혼합하고, 단일 콜로니에 대해 희석 및 도금하고, 마지막으로 드롭아웃 플레이트의 콜로니 수를 기반으로 효율성을 계산하는 등 4가지 주요 단계가 포함됩니다. Auxotrophic 마커는 반수체와 이배체를 명확하게 구별하기 위해 사용됩니다.
서문
일반적으로 신진 효모라고 불리는 Saccharomyces cerevisiae는 단세포 진핵생물입니다. a와 α의 두 가지 짝짓기 유형이 있으며 무성 생식 주기와 유성 생식 주기를 모두 나타냅니다. a 및 α 짝짓기 유형은 반수체이며 주변 환경에 다른 짝짓기 유형이 없을 때 유사분열할 수 있으며, 이는 효모의 무성 주기를 나타냅니다. 두 짝짓기 유형이 가까이 있으면 유사 분열을 멈추고 융합하여 이배체 세포를 형성합니다. 이배체 효모는 영양소가 존재할 때 유사분열을 일으키거나 아세테이트1과 같이 발효되지 않는 열악한 탄소원이 있는 상태에서 질소 결핍 조건에서 감수분열을 겪을 수 있습니다. 그 결과 포자가 형성되어 유리한 성장 조건이 될 때까지 휴면 상태로 유지됩니다. 이 포자가 발아하고 두 가지 반수체 유형이 반수체 풀 2,3으로 다시 방출되면 수명주기가 완료됩니다 (그림 1).
효모 세포의 교배는 응집, 교미 돌기 또는 "shmoo"의 형성, 세포 및 핵융합 4,5와 같은 여러 단계를 포함합니다. 두 가지 결합 유형 a 와 α는 각각 a-인자와 α-인자를 생성하여 결합을 시작합니다. 이러한 인자는 반대 짝짓기 유형5의 세포 표면에 존재하는 수용체(Ste2 및 Ste3)에 결합하는 폴리펩티드 페로몬입니다. 페로몬과 수용체의 결합은 페로몬 반응 경로, 즉 미토겐 활성화 단백질 키나아제(MAPK) 신호 전달 경로 6,7,8을 개시한다. 그 결과 G1 단계에서 세포주기가 정지되어 대사 활성 정지 단계9로 이어집니다. 그런 다음 세포는 유사 분열을 멈추고 짝짓기에 필요한 단백질이 합성됩니다. 반수체 세포가 서로를 향해 움직일 수 없기 때문에 짝짓기 돌출부 또는 "shmoo"가 짝짓기 파트너를 향합니다. 세포가 접촉하게 되면, 세포벽이 분해되고, 세포질 내용물이 융합되어, 짝짓기를 하여 이배체 세포를 형성한다(10,11). 반수체 사이의 짝짓기 효율은 실험실에서 진화한 균주와 현존하는 종 사이의 종분화의 척도로 사용되어 왔다12.
단순한 진핵 생물이기 때문에 효모는 복잡한 진핵 생물과 관련된 많은 연구 질문에 대한 선택 모델입니다. 그러한 질문 중 하나는 종 분화와 생식 장벽의 진화와 관련이 있습니다13,14. 유성 생식 유기체의 경우, 종은 Ernst Mayr15가 제안한 생물학적 종 개념 (BSC)에 의해 정의됩니다. 이 개념에 따르면, 개체군의 두 개체는 교배할 수 없고 생식적으로 고립된 경우 두 개의 다른 종에 속한다고 합니다. 유성 생식주기의 붕괴 (배우자가 접합체를 형성하고, 접합체가 자손으로 발달하고, 자손의 성적 성숙에 도달하는 것을 포함)는 생식 격리로 이어진다. 그림 1에서 볼 수 있듯이 S. cerevisiae의 수명주기는 유성생식주기와 비슷합니다 : a) 두 가지 짝짓기 유형 a와 α의 융합은 유성 생식 유기체에서 배우자의 융합과 유사합니다. b) 유사분열을 겪는 이배체의 능력은 자손으로 발달하는 접합체와 동일하다. c) 포자 형성을 겪고 있는 이배체는 배우자 형성 과정과 비슷하다(14).
접합 전 분리는 구색 짝짓기가 관찰 될 때 발생합니다. 유전 적으로 다른 두 가지 유형과 짝짓기를 할 수 있는 동등한 기회가 주어진다면, α 유형은 다른 유형보다 우선적으로 짝짓기를 하거나 그 반대의 경우도 마찬가지이다14. 반수체가 상이한 환경에서 진화 된 진화 실험의 경우, 짝짓기 분석을 수행하여 사전 짝짓기 장벽의 존재를 결정할 수 있습니다. 조상과 비교할 때 짝짓기 효율의 감소는 짝짓기 전 장벽의 진화를 나타냅니다. 접합체 후 분리(post-zygotic isolation)는 이배체가 효과적인 유사분열 및/또는 포자형성을 통해 반수체 포자를 형성할 수 없기 때문에 발생할 수 있다14. 이들은 이배체의 성장 속도를 측정하고 포자 형성 효율을 각각 계산하여 정량화할 수 있습니다. 따라서 생식 장벽의 진화를 연구하기 위해서는 (a) 짝짓기 효율, (b) 이배체의 유사분열 성장 및 (c) 이배체의 포자 형성 효율을 정량화하기 위한 강력한 방법이 필요합니다. 이 연구에서, 효모 균주의 교배 효율을 정량화하는 강력한 방법이 보고되었다.
실험실 실험에서 짝짓기 발생을 감지할 수 있는 방법 중 하나는 영양 요구 사항을 보완하는 영양요구성 마커를 사용하는 것입니다. 두 가지 짝짓기 유형이 두 개의 다른 아미노산에 대해 영양요구성 인 경우, 두 가지 짝짓기 유형의 융합에 의해 형성된 이배체 세포 만이 두 아미노산이 모두 결핍 된 배지에서 성장할 수 있습니다. 따라서, 영양요구성 마커는 정성적 및 정량적으로 짝짓기를 검출하는데 유용하다. 정성적 검사는 감수 분열 후 균주의 짝짓기 유형을 확인하기에 충분할 것이다16. 정량적 검사는 짝짓기 경로17,18에 관여하는 유전자를 연구하는 동안 짝짓기 감소를 확인하는 데 관심이 있을 때 필수적입니다. 또한, 효모가 종 분화 연구에 점점 더 많이 사용됨에 따라, 짝짓기 효율의 정량화는 접합 전 장벽의 척도이기 때문에 편리하고 재현 가능한 짝짓기 분석이 필요합니다.
두 효모 교배 유형 사이의 교배 효율은 이전에 정량화되었습니다16,19,20. 이전에 사용된 대부분의 방법은 16,21,22,23,24,25와 같은 몇 가지 변형을 제외하고는 디자인이 유사합니다. 그들 중 일부는 초기 로그 단계 배양을 사용하는 반면, 다른 일부는 반수체 균주의 중간 로그 단계 배양을 사용합니다. 두 가지 결합 유형이 혼합되는 비율에는 차이가 있습니다. 거의 모든 프로토콜은 니트로셀룰로오스 막을 사용합니다. 이전에 성장한 배양물로부터 채취한 두 가지 짝짓기 유형의 현탁액을 혼합하고 YPD 플레이트 상에 놓인 니트로셀룰로스 막 상에 여과한다. 프로토콜의 변형들 중 하나에서, 반수체 현탁액은 YPD 플레이트(21) 상에 직접 패치된다. 두 가지 짝짓기 유형의 페로몬 생산에 관여하는 유전자를 다루는 실험에서, 페로몬은 두 가지 짝짓기 유형의 현탁액을 만드는 동안 외부적으로 첨가된다24.
반수체를 혼합한 후 몇 시간(일반적으로 약 5시간) 동안 배양한 후 세포를 멤브레인에서 씻어내고 희석한 다음 선택적 배지에 도말합니다. 1973년에 보고된 초기 방법 중 하나에서, 접합체 형성 또는 짝짓기의 효율은 혈구계를 사용하여 현미경으로 발아 세포, 발아 세포 및 짝짓기 쌍의 수를 계산하여 계산되었습니다26. 그러나 나중에 보고된 대부분의 방법은 반수체와 이배체를 구별하기 위해 영양요구성 마커를 사용합니다. 짝짓기 효율은 세포 풀내의 이배체 및 반수체 세포의 수에 대한 이배체 세포의 백분율로서 계산된다 16,21,23.
그러나 종분화를 연구하기 위해 효모를 모델 유기체로 사용하는 많은 보고에도 불구하고 지금까지 짝짓기 효율을 계산하기 위한 표준화된 프로토콜이 문헌에 보고되지 않았습니다. 로그 단계의 셀은 짝짓기 효율의 정량화에 이상적이지 않을 수 있습니다. 짝짓기 동안, 두 반수체의 세포주기가 정지되고, 따라서 짝짓기 중 세포는 분열하지 않는다9. 세포 주기가 또한 정지 위상27에서 세포에서 유사하게 정지되는 것으로 알려져 있기 때문에, 이러한 세포를 사용하여 프로토콜을 보다 재현가능하게 할 수 있다. 고정상 세포는 짝짓기를 위해 YPD 플레이트(즉, 영양이 풍부한 환경)에 혼합되고 배치될 수 있습니다. 또한 기존 절차에서는 니트로셀룰로오스 멤브레인을 사용하고 세포를 세척해야 하므로 프로세스가 번거롭고 처리 오류가 발생하기 쉽습니다. 또한, 현재까지 사용된 프로토콜은 하나의 반수체 측면에서 짝짓기 효율을 정량화합니다. 그러나, 생식 격리를 측정할 때, 짝짓기 효율은 단일 반수체가 아닌 반수체의 특정 조합에 대해 정량화된다.
이러한 문제를 해결하기 위해 여기에서 우리는 재현성이 높고 사용하기 쉬운 효모의 짝짓기 효율을 정량화하는 강력한 방법을 보고합니다. 또한, 이 방법과 여기에 사용된 효모 균주는 짝짓기 장벽의 진화에 대한 유전자 흐름의 영향을 조사하는 연구에도 사용할 수 있습니다.
이 연구에서는 S. cerevisiae의 두 가지 다른 균주가 사용되었습니다. 균주 중 하나는 SK1 배경에서 파생됩니다. 이것은 MAT 유전자좌 근처에 영양요구성 마커를 추가하여 실험실에서 수정되었습니다. 반수체의 생성된 유전자형은 표 128,29,30에 제공된다. SK1 균주에서, 반수체는 MAT 유전자좌 근처에 삽입된 TRP1 유전자를 가졌고, α 반수체는 MAT 유전자좌 근처에 삽입된 LEU2 유전자를 가졌다. ScAM 균주에서, TRP1 및 URA3 유전자를 각각 a 및 α 반수체에 삽입하였다. 삽입 위치는 염색체 III의 ARS 영역이었습니다(Chr III: 197378..197609). 여기에 보고된 프로토콜의 경우 게놈의 어느 곳에서나 영양요구성 마커로 충분합니다. 그러나 MAT 유전자좌 근처에 영양요구성 마커가 있다는 것은 이러한 균주가 종분화에 대한 유전자 흐름의 영향을 조사하는 연구에도 사용될 수 있음을 의미합니다31,32. 마커는 재조합으로 인한 마커의 재편성을 방지하기 위해 MAT 유전자좌 가까이에 추가되었습니다. 따라서 이 프로토콜은 종분화와 관련된 연구에서 짝짓기 효율을 정량화하고 짝짓기 경로에 관여하는 단백질을 연구할 때 짝짓기 효율의 변화를 확인하는 데 사용할 수 있습니다.
프로토콜
참고: 프로토콜은 광범위하게 다음 단계를 포함합니다: (1) YPD 플레이트의 짝짓기 효율 그리드에 있는 반수체를 패치하고, (2) 24시간 배양 후 동일한 수의 반수체를 혼합하고 혼합된 반수체를 몇 시간 동안 짝짓기(이 연구에서 7시간), (3) 30°C에서 7시간 후 단일 콜로니를 분리하기 위해 혼합 세포를 YPD에 플레이팅하고, 마지막으로, (4) 영양요구성 마커를 사용하여 형성된 이배체의 수를 결정합니다. 이러한 단계는 아래에 자세히 설명되어 있습니다( 그림 2 참조).
1. 짝짓기 효율 그리드에서 반수체 패치
- YPD 한천 플레이트(2% 한천, 2% 덱스트로스, 1% 펩톤, 0.5% 효모 추출물)에 줄무늬를 넣어 냉동고 스톡에서 반수체 a 및 α 을 되살리고 30°C에서 48시간 동안 성장시켜 분리된 단일 콜로니를 얻습니다.
- YPD 플레이트의 단일 콜로니를 5mL의 YPD 배지(2% 덱스트로스, 1% 펩톤, 0.5% 효모 추출물)에 접종하고 30°C에서 250rpm 진탕으로 48시간 동안 배양합니다. 세포는이 잠복기 후에 성장의 정지 단계에 있습니다.
- 새 YPD 플레이트에 결합 효율 그리드를 그립니다. 그림 2A와 같이 각각 1cm x 0.5cm의 치수를 갖도록 세 개의 상자로 분할된 1cm x 1.5cm 직사각형으로 그리드를 그립니다.
- 가장 왼쪽 및 가장 오른쪽 직사각형에 두 가지 짝짓기 유형의 YPD 배양물 5μL를 패치합니다(그림 2B). 이 부피는 각 섹션에 배치된 대략 5 x 105 개의 반수체 세포에 해당합니다. 플레이트를 30°C에서 24시간 동안 인큐베이션한다.
참고: 그리드의 목적은 실험 측정값(예: 실험의 셀 수)을 정확하게 만드는 것입니다. 그리드 크기는 실험적으로 다루기 쉬울 정도로 작지만 쉽게 조작할 수 있고(예: 그리드에서 셀을 들어 올리는 것처럼) 드리프트 또는 우연한 이벤트에 취약하지 않을 만큼 충분히 큽니다.
2. 반수체의 혼합과 짝짓기
참고: 24시간 후(그림 2C), 두 반수체 유형의 동일한 수의 셀이 두 그리드에서 긁어내어 혼합된 다음 중앙 직사각형에 배치됩니다(그림 2D).
- 동일한 수의 세포를 혼합하기 위해 멸균 이쑤시개를 사용하여 외부 상자에 놓인 패치의 약 1/3을 제거하고 각 반수체에 대해 멸균 1.5mL 바이알에 20μL의 물에 다시 현탁합니다.
- 이 현탁액 5μL를 물 2mL에 희석합니다. 이 희석된 현탁액의 OD를 600nm에서 분광광도계를 사용하여 측정합니다. 특정 균주에 대한 OD 값과 1 OD의 세포 수/mL를 기준으로 두 균주에서 동일한 수의 세포를 혼합합니다. 혼합에 필요한 부피를 계산하고 개별 반수체 현탁액의 나머지 15μL에서 흡인합니다.
참고: 중앙 사각형에 패치된 셀의 수는 셀이 단층을 형성하는 정도입니다. 효모 세포가 반경 2.58μm33의 구체인 것을 고려하면, 1 cm x 0.5 cm의 직사각형 상자는 단분자층을 형성하기 위해 대략 1.7 x 106 세포를 필요로 할 것이다. 짝짓기를 위해 패치된 세포와 두 개의 반수체 세포 그리드 사이에 물리적 접촉이 없도록 주의해야 합니다. 각 유형의 반수체 세포가 동일한 수로 혼합되어야 하기 때문에 각 균주에서 8.5 x 105개의 세포가 추가됩니다. 셀 수는 OD 측정에 기초하여 계산되며, 600 nm에서 1 OD가 대략 1 x 107 셀34와 동일하다고 생각한다. 예를 들어, 반수체 현탁액의 OD600이 0.17이고 α 반수체의OD600이 0.11인 경우, 각 반수체 현탁액의 5 μL에 있는 세포의 수를 계산할 수 있다. 각 반수체 유형의 8.5 x 105 세포를 보장하기 위해, 반수체 1.25 μL와 α 반수 체 현탁액 1.93 μL를 혼합한다. - 신선하고 멸균된 1.5mL 바이알에 필요한 양의 반수체를 넣고 피펫을 사용하여 잘 섞습니다. 이 현탁액의 최종 부피는 일반적으로 약 6-8 μL입니다. 이 현탁액을 중앙 그리드에 패치합니다. 플레이트를 30°C에서 7시간 동안 인큐베이션하여 반수체가 짝짓기를 하기에 충분한 시간을 허용합니다(그림 2E).
3. YPD 한천에 혼합 세포 도금
- 7 시간의 배양 기간 후, 이쑤시개 또는 피펫 팁을 사용하여 중앙 사각형에서 세포를 긁어 내고 2mL의 멸균 수로 희석합니다. 이어서, 세포 현탁액을 YPD 한천에 퍼뜨려 단일 콜로니를 얻었다. 단일 콜로니를 얻는 데 필요한 희석 계수를 결정하려면 긁힌 세포가 첨가 된 첫 번째 튜브의 OD를 측정하십시오. 사용되는 각 세포 유형/균주에 대해 특정 희석 계수를 결정해야 합니다.
알림: 예를 들어, 600의 OD0.15는 3mL 현탁액의 106 x 2 세포에 해당합니다(1 OD = 1 x 107 cells/mL 고려). YPD 플레이트에서 수백 개의 콜로니를 얻기 위해 세포 현탁액을 1:20에서 두 번 연속 희석한 다음 최종 희석액 100μL를 사용하여 퍼뜨립니다. - 도금 후, 단일 콜로니가 될 때까지 30 ° C에서 36-48 시간 동안 YPD 플레이트를 배양합니다. 데이터에서 통계적 유의성을 검출할 수 있도록 스크리닝을 위해 각 짝짓기 실험에서 수백 개의 개별 콜로니를 얻었는지 확인합니다(그림 2F).
4. 영양요구성 마커를 이용한 이배체 스크리닝
- 얻은 콜로니의 어떤 부분이 이배체인지 결정하십시오. 플레이트에서 이배체 콜로니를 식별하려면 그림 2G와 같이 균주가 영양요영양성인 아미노산이 결여된 이중 드롭아웃 플레이트(2% 포도당, 0.66% 질소 염기, 0.05% 이중 드롭아웃 아미노산 혼합물 및 2% 한천)에 개별적으로 스트리킹하여 단일 콜로니를 옮깁니다. 플레이트를 30°C에서 48시간 동안 배양합니다.
참고: 콜로니는 복제 도금을 사용하여 이중 드롭아웃 플레이트로 옮길 수도 있습니다. 본 연구에서는 SK1AM 균주의 교배 효율을 정량화할 때 트립토판과 류신(trp-leu-) drop-out 배지를 사용하였고, ScAM 균주의 교배효율을 정량할 때는 트립토판과 우라실(trp-ura-) drop-out medium을 사용하였다. 이배체 콜로니만 이중 탈락 플레이트에서 성장하는데, 이는 SK1AM 균주의 TRP1 및 LEU2 유전자와 ScAM 균주의 TRP1 및 URA3 유전자와 같은 영양요구성 마커를 모두 가지고 있기 때문입니다. - 또한, 단일 드롭아웃 배지(trp- 또는 leu- 또는 ura-)에 콜로니를 줄무늬 또는 복제하여 개체군에서 두 종류의 반수체 각각의 빈도를 정량화합니다.
- 다음과 같이 결합 효율 η 계산하십시오.
 에큐 (1)
에큐 (1)
여기서 짝짓기된 반수체의 수는 이중 탈락판에서 확인된 이배체 수의 두 배와 같습니다(각 이배체는 두 반수체의 짝짓기의 결과이기 때문에). 반수체의 총 수는 줄무늬가 있는 반수체 수에 줄무늬가 있는 이배체 수의 두 배를 더한 합과 같습니다.
참고: 예를 들어, 이중 드롭아웃 배지에서 100개의 콜로니를 스트리킹/복제 도금한 후 60개의 콜로니만 성장하는 경우 짝짓기 효율은 75%로 정량화할 수 있습니다(60 x 2 반수체가 짝을 이루어 60개의 이배체를 형성하고 40개의 반수체가 짝짓기를 하지 않았기 때문에).
결과
두 가지 결합 유형의 결합 효율 정량화
여기에 설명된 프로토콜은 SK1AM a와 SK1AMα 사이 및 ScAMa와 ScAMα 사이의 두 효모 균주 사이의 짝짓기 효율을 정량화하는 데 사용되었습니다(그림 3A). 이 실험에서, 두 반수체 사이의 교배를 적어도 12회 반복하였다. 실험의 각 반복에서, 적어도 100 콜로니가 이중 드롭 아웃 배지에 줄무늬가 있었다. 프로토콜의 견고성으로 인해 SK1AM과 ScAM의 두 균주 간의 결합 효율을 쉽게 구별할 수 있었습니다. SK1 균주는 매우 높은 효율로 교배된 반면, ScAM 균주는 상대적으로 낮은 효율로 교배되었습니다(그림 3A). ScAM 균주는 S. cerevisiae와 S. carlsbergensis 균주35 사이의 잡종 교배에서 유래했기 때문에 이것은 놀라운 일이 아닙니다.
균주 간의 짝짓기 효율을 구별하는 것 외에도 이 방법은 서로 다른 환경에서 균주의 짝짓기 효율 차이를 정량화하는 데에도 사용할 수 있습니다. 도 3B에 나타낸 바와 같이, ScAM 균주의 교배 효율은 1차 탄소원으로서 글루코스를 함유하지 않은 환경에서 유의하게 떨어졌다. 짝짓기는 에너지 비용이 많이 드는 과정이며, 대체 탄소원의 성장은 짝짓기의 효율성을 질적으로 감소시킵니다.
짝짓기 효율성의 역학
이 방법의 높은 반복성으로 인해 결합 효율의 역학을 추적할 수도 있습니다. 상이한 기간 동안 짝짓기를 허용했을 때(그림 3C), 처음 4-5시간 동안 짝짓기가 관찰되지 않았습니다. 첫 번째 이배체는이 기간 후에 만 나타났습니다. 아마도 이것은 주어진 환경에서 짝짓기 과정이 완료되는 데 필요한 시간일 것입니다. 그 후, 짝짓기 효율이 급격히 증가하였다. 그러나 7 h를 초과하면 짝짓기 효율 계산은 유사 분열 성장이 플레이트에서 일어나기 시작한다는 사실에 영향을받습니다. 이러한 방식으로 초기 또는 후기 이배체의 식별 및 선택은 반수체 간의 빠른 또는 지연된 짝짓기를 진화시키는 것을 목표로 하는 실험 설계를 허용할 수 있습니다. 결합 효율이 최고조에 달하는 정확한 시간은 각 변형률에 따라 다릅니다. 예를 들어, 성장 동역학을 사용하여 ScAM 균주가 다른 S. cerevisiae 균주(예: SK1)보다 낮은 성장률을 나타내며 이는 아마도 짝짓기 역학에도 영향을 미칠 수 있음을 실험적으로 확인했습니다.

그림 1 : Saccharomyces cerevisiae의 수명주기. S. cerevisiae에는 a와 α의 두 가지 짝짓기 유형이 있으며 무성 생식 단계와 유성 생식 단계를 모두 나타냅니다. 두 짝짓기 유형은 다른 유형이 없을 때 유사 분열로 나뉩니다. 그러나 그들이 서로 근처에 존재할 때, 그들은 유사 분열을 멈추고 세포와 핵 내용물이 융합하여 이배체를 형성합니다. 이배체 세포는 유사 분열 적으로 더 분열 할 수 있습니다. 그러나 불리한 조건 (기아)이있는 경우 감수 분열을 겪어 4 개의 반수체를 포함하는 포자를 생성합니다. 이 포자는 유리한 조건에서 발아하여 각 종류의 두 반수체를 방출하여 수명주기를 완료합니다. 두 개의 반수체 a 및 α 짝짓기의 효능을 짝짓기 효율이라고 합니다. 이 짝짓기 효율의 감소는 짝짓기에 대한 접합 전 장벽을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
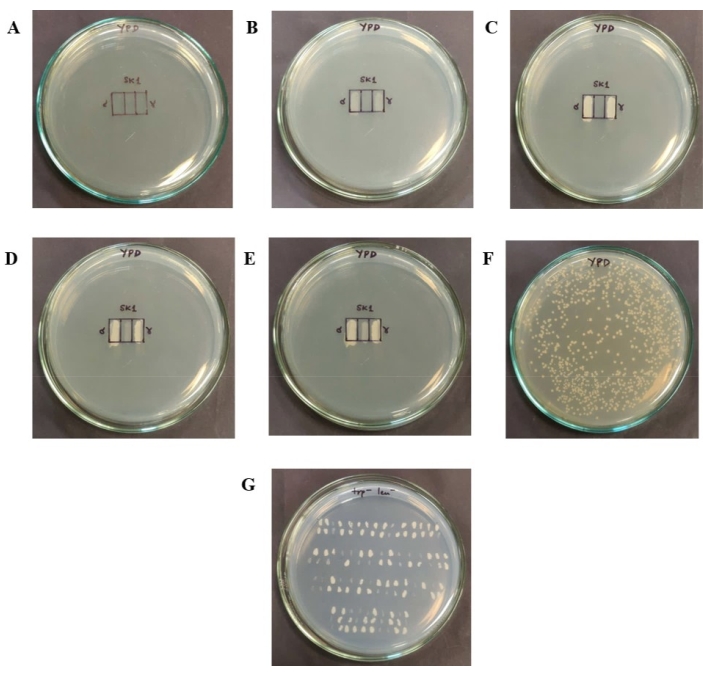
그림 2: 결합 효율 분석과 관련된 단계. (A) 각각 1cm x 0.5cm의 상자로 더 분할되는 1cm x 1.5cm의 결합 효율 그리드가 YPD 플레이트에 그려집니다. (B) 반수체 세포는 극단 상자에 패치됩니다. (c) 30°C에서 24시간 후의 반수체 세포의 성장. (D) 반수체 패치의 1/3을 이쑤시개를 사용하여 제거하고 동일한 세포 번호(OD600 기준)로 혼합한 다음 중앙 그리드에 패치합니다. (e) 세포의 성장을 30°C에서 7시간 후에 중앙 그리드에 패치한다. 여기에는 형성된 새로운 이배체와 짝짓기되지 않은 반수체가 포함됩니다. (f) 세포의 희석 및 도금 후 YPD 플레이트 상에서 수득된 단일콜로니를 중앙 그리드로부터 긁어낸다. 이 플레이트를 30°C에서 36-48시간 동안 인큐베이션하였다. (g) YPD 플레이트 상에 존재하는 이배체의 수를 확인하기 위해, 콜로니는 트립토판 및 류신이 결여된 이중 드롭아웃 플레이트로 옮겨진다. 이 판에서는 이배체 만 자랄 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
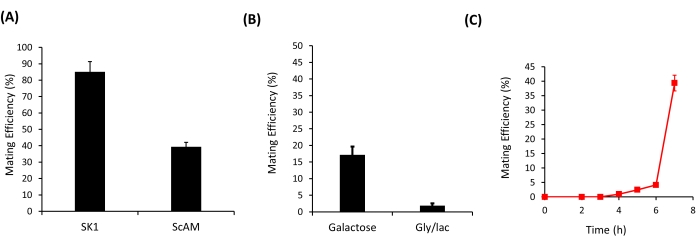
그림 3: 효모 균주의 짝짓기 효율. (A) YPD 플레이트에서 SK1 및 ScAM 균주의 두 반수체 a 및 α의 짝짓기 효율. (B) 2% 갈락토오스를 함유하는 YP 플레이트 및 글리세롤/락테이트 플레이트에서 ScAM 균주의 두 반수체 a 및 α의 교배 효율. (씨) 포도당 환경에서 ScAM 균주에서 짝짓기의 역학. 결과는 12개의 독립적인 반복에서 평균 ± SD로 나타내었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 변종 | 유전자 형 | 주석 | 참조 |
| SK1AMa | ars314::TRP1, MATa, ho::LYS2, lys2, ura3, leu2::hisG, his3::hisG, trp1::hisG | 이 균주는 TRP1 유전자를 가지고 있으므로 배지에 트립토판이 없을 때 자랄 수 있습니다. | |
| SK1AMα | ars314::LEU2, 매트알파, ho::LYS2, lys2, ura3, leu2::hisG, his3::hisG, trp1::hisG | 이 균주는 LEU2 유전자를 가지고 있으므로 배지에 류신이 없을 때 자랄 수 있습니다. | |
| ScAMa | ars314::TRP1, MATa, ho, MEL1, ade1, ile, trp1-HIII ura3-52 | 이 균주는 TRP1 유전자를 가지고 있으므로 배지에 트립토판이 없을 때 자랄 수 있습니다. | 참고문헌 28의 ScPJB644a에서 파생됨 |
| ScAMα | ars314::URA3, 매트알파, 호, MEL1, ade1, ile, trp1-HIII, ura3‐52 | 이 균주는 URA3 유전자를 가지고 있으므로 배지에 우라실이 없을 때 자랄 수 있습니다. | 참고문헌 28의 ScPJB644α로부터 유래 |
표 1: 본 연구에 사용된 S. cerevisiae 균주의 유전자형.
토론
S. cerevisiae의 짝짓기 효율 정량화는 짝짓기 경로에 관여하는 유전자와 관련된 연구를 수행하거나 짝짓기 행동에 대한 외부 환경의 영향을 연구하는 데 필수적입니다. 지난 20년 동안 S. cerevisiae는 종분화 14,36,37,38과 관련된 질문을 해결하는 인기 있는 모델이 되었습니다. 두 가지 짝짓기 유형의 존재와 실험실 환경에서의 유전자 조작 및 유지 관리의 용이성으로 인해 생식 장벽의 진화를 실시간으로 연구하기에 적합한 유기체가 되었습니다. 짝짓기 효율의 정량화는 접합 전 생식 장벽의 척도를 제공하기 때문에 필수적입니다. 따라서 재현성이 높은 편리한 프로토콜이 필요합니다.
상기 프로토콜을 사용하여, 완전히 상이한 교배 거동을 나타내는 2개의 상이한 균주의 교배 효율을 정량화하였다. 따라서, 대부분의 실험실 효모 균주는 선택에 사용될 수 있는 영양요구성 균주를 가지고 있기 때문에 이 프로토콜은 모든 균주에 적용될 수 있다39. 그러나 이 프로토콜을 사용하는 동안 몇 가지 중요한 고려 사항이 있습니다. 초기 YPD 배양이 고정상에 도달하는 데 필요한 기간은 균주에 따라 다릅니다. 느리게 성장하는 균주는 30°C에서 48시간 이상의 배양이 필요할 수 있습니다. 혼합되는 반수체의 부피는 분광 광도계의 OD 측정을 기반으로 계산됩니다. 문헌에 보고된 값은 1 OD34에 대해 107 cells/mL 정도입니다. 그러나 세포 수 대비 OD의 플롯을 사용하여 특정 균주에 대해 이 값을 특성화하는 것이 좋습니다. 동일한 수의 세포를 혼합하는 데 필요한 부피는 균주의 특성화에서 얻은 OD 값과 1 OD의 세포 수/mL를 기반으로 계산할 수 있습니다. 반수체를 혼합한 후 중앙 그리드에 패치되는 세포가 대략적으로 매우 두껍지 않은 단층을 형성하는지 확인해야 합니다. 또한 혼합되는 부피가 약 6-8 μL인지 확인해야합니다. 볼륨이 높을수록 중앙 그리드의 양쪽에 있는 반수체 패치와 겹칠 수 있습니다.
이 방법은 또한 효모의 생태학적 분리주 사이의 접합 전 생식 장벽을 연구하는 데 사용할 수 있습니다. 그러나, 그러한 경우에는, HO 엔도뉴클레아제 유전자는 먼저 유기체의 게놈으로부터 제거되어야 하는데, 이는 HO 엔도뉴클레아제를 운반하는 균주가 그의 짝짓기형 스위칭 활성(40)으로 인해 자동적으로 이배체를 형성하기 때문이다. 포자화된 이배체로부터 분리된 반수체는 앞서 언급한 특이적 프라이머를 사용하여 MAT 유전자좌에 존재하는 서열을 확인함으로써 또는 α로 확인될 수 있다(41). HO 마커는 2개의 반수체 짝짓기 유형을 구별하기 위한 마커로서 2개의 상이한 항생제 내성 유전자로 대체될 수 있다. 이 세부 사항은 특정 연구에 사용된 변형률 구성에 따라 다릅니다.
이 방법의 한계 중 하나는 영양요구성 마커를 사용하려면 콜로니를 YPD 플레이트에서 드롭아웃 플레이트로 옮겨야 한다는 것입니다. 반수체 세포와 이배체 세포를 구별하기 위해 형광 마커를 사용하면 실험 시간을 줄이는 데 도움이 될 수 있습니다. 그러나, 형광 마커의 사용은 형광 단백질을 생산하는데 관여하는 추가 비용이 있기 때문에 세포의 성장 동력에 영향을 미칠 수 있으며, 따라서 세포의 대사 및 생리학적 상태를 변화시킬 수 있다(42). 대안적으로, 2개의 반수체를 식별하기 위한 영양요구성 마커는 항생제 마커로 대체될 수 있다.
짝짓기 효율의 특성화에 사용된 이전 방법 중 일부는 10:1 16,21,24의 비율로 짝짓기 유형 중 하나를 과도하게 사용하는 것을 보고합니다. 경험에 비추어 볼 때, 두 반수체의 편향된 비율을 사용하면 반수체의 유사 분열 성장이 과도하게 발생합니다. 따라서 현재 방법은 두 반수체의 1:1 혼합을 사용합니다.
본 연구에서 사용된 균주의 영양요구성 마커는 MAT 유전자좌 가까이에 삽입되기 때문에 감수분열 중 재조합은 영양요구성 마커와 짝짓기 유형 사이의 연관성을 깨뜨리지 않습니다. 결과적으로, 특정 반수체의 영양요구성 및 짝짓기 유형은 균주가 감수 분열을 겪도록 허용되더라도 변하지 않습니다. 이를 통해 적응 및 종분화에 대한 변수로서 유전자 흐름의 영향과 관련된 질문에 답할 수 있습니다43. 따라서 전반적으로 이 프로토콜은 서로 다른 효모 균주 간의 짝짓기 효율을 정량화하는 간단하고 강력한 방법을 제시합니다.
공개
저자는이 작품에 경쟁 이익이 없다고 선언합니다. 저자는 모든 비영리 용도로 SK1 유래 균주를 공유하게 되어 기쁩니다.
감사의 말
이 연구는 DBT/Wellcome Trust(India Alliance) 보조금(IA/S/19/2/504632)으로 자금을 지원받았습니다. SPN은 DBT/Wellcome Trust(India Alliance) 보조금(IA/S/19/2/504632)의 지원을 받는 연구원입니다. A.M.은 선임 연구원 (09 / 087 (0873) / 2017-EMR-I)로서 인도 정부의 과학 및 산업 연구위원회 (CSIR)의 지원을 받고 있습니다. 저자는 토론에 대해 Paike Jayadeva Bhat에게 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| Adenine | Sigma Life Science | A8626 | |
| Agar Powder regular grade for bacteriology | SRL | 19661 (0140186) | |
| Ammonium Sulphate, Hi-AR | HiMedia | GRM1273 | |
| D-(+)-glucose | Sigma Life Science | G8270 | |
| Glass Petri plates | HiMedia | PW008 | 90 mm x 15 mm dimension |
| L-Arginine | Sigma Life Science | A8094 | |
| L-Aspartic acid | Sigma Life Science | A7219 | |
| L-Histidine monochloride monohydrate | Sigma Life Science | H5659 | |
| L-Isoleucine | Sigma Aldrich | I2752 | |
| L-Leucine | Sigma Life Science | L8912 | |
| L-Lysine | Aldrich | 62840 | |
| L-Methionine | Sigma Life Science | M5308 | |
| L-Phenylalanine | Sigma Life Science | P5482 | |
| L-Threonine | Sigma Aldrich | T8625 | |
| L-Tyrosine | Sigma Life Science | T8566 | |
| L-Valine | Sigma Life Science | V0513 | |
| Mating efficiency grid | 1 cm x 1.5 cm rectangular grid drawn on the Petri plate | ||
| Microcentrifuge tubes | Tarsons | 500010 | |
| Peptone | HiMedia | RM001 | |
| Uracil | Sigma Life Science | U0750 | |
| Yeast Extract Powder | HiMedia | RM027 | |
| Yeast Nitrogen Base w/o Amino acids and Ammonium Sulphate | BD Difco | 233520 |
참고문헌
- Neiman, A. M. Sporulation in the budding yeast Saccharomyces cerevisiae. Genetics. 189 (3), 737-765 (2011).
- Duina, A. A., Miller, M. E., Keeney, J. B. Budding yeast for budding geneticists: A primer on the Saccharomyces cerevisiae model system. Genetics. 197 (1), 33-48 (2014).
- Herskowitz, I. Life cycle of the budding yeast Saccharomyces cerevisiae. Microbiological Reviews. 52 (4), 536-553 (1988).
- Erdman, S., Lin, L., Malczynski, M., Snyder, M. Pheromone-regulated genes required for yeast mating differentiation. Journal of Cell Biology. 140 (3), 461-483 (1998).
- Merlini, L., Dudin, O., Martin, S. G. Mate and fuse: How yeast cells do it. Open Biology. 3 (3), 130008 (2013).
- Gustin, M. C., Albertyn, J., Alexander, M., Davenport, K. MAP kinase pathways in the yeast Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 62 (4), 1264-1300 (1998).
- Bardwell, L. A walk-through of the yeast mating pheromone response pathway. Peptides. 26 (2), 339-350 (2005).
- Reid, B. J., Hartwell, L. H. Regulation of mating in the cell cycle of Saccharomyces cerevisiae. Journal of Cell Biology. 75, 355-365 (1977).
- Williams, T. C., Peng, B., Vickers, C. E., Nielsen, L. K. The Saccharomyces cerevisiae pheromone-response is a metabolically active stationary phase for bio-production. Metabolic Engineering Communications. 3, 142-152 (2016).
- Bagnat, M., Simons, K. Cell surface polarization during yeast mating. Proceedings of the National Academy of Sciences of the United States of America. 99 (22), 14183-14188 (2002).
- Trueheart, J., Boeke, J. D., Fink, G. R. Two genes required for cell fusion during yeast conjugation: Evidence for a pheromone-induced surface protein. Molecular and Cellular Biology. 7 (7), 2316-2328 (1987).
- Sniegowski, P. D., Dombrowski, P. G., Fingerman, E. Saccharomyces cerevisiae and Saccharomyces paradoxus coexist in a natural woodland site in North America and display different levels of reproductive isolation from European conspecifics. FEMS Yeast Research. 1 (4), 299-306 (2002).
- Replansky, T., Koufopanou, V., Greig, D., Bell, G. Saccharomyces sensu stricto as a model system for evolution and ecology. Trends in Ecology and Evolution. 23 (9), 494-501 (2008).
- Greig, D. Reproductive isolation in Saccharomyces. Heredity. 102 (1), 39-44 (2009).
- Mayr, E. . Systematics and the Origin of Species, from the Viewpoint of a Zoologist. , (1999).
- Sprague, G. F. Assay of yeast mating reaction. Methods in Enzymology. 194, 77-93 (1991).
- McCaffrey, G., Clay, F. J., Kelsay, K., Sprague, G. F. Identification and regulation of a gene required for cell fusion during mating of the yeast Saccharomyces cerevisiae. Molecular and Cellular Biology. 7 (8), 2680-2690 (1987).
- Valtz, N., Peter, M., Herskowitz, I. FAR1 is required for oriented polarization of yeast cells in response to mating pheromones. Journal of Cell Biology. 131 (4), 863-873 (1995).
- Maclean, C. J., Greig, D. Prezygotic reproductive isolation between Saccharomyces cerevisiae and Saccharomyces paradoxus. BMC Evolutionary Biology. 8, 1 (2008).
- Murphy, H. A., Kuehne, H. A., Francis, C. A., Sniegowski, P. D. Mate choice assays and mating propensity differences in natural yeast populations. Biology Letters. 2 (4), 553-556 (2006).
- Leu, J. Y., Murray, A. W. Experimental evolution of mating discrimination in budding yeast. Current Biology. 16 (3), 280-286 (2006).
- Kim, J., Hirsch, J. P. A nucleolar protein that affects mating efficiency in Saccharomyces cerevisiae by altering the morphological response to pheromone. Genetics. 149 (2), 795-805 (1998).
- Jin, M., et al. Yeast dynamically modify their environment to achieve better mating efficiency. Science Signaling. 4 (186), (2011).
- Rogers, D. W., Denton, J. A., McConnell, E., Greig, D. Experimental evolution of species recognition. Current Biology. 25 (13), 1753-1758 (2015).
- McClure, A. W., Jacobs, K. C., Zyla, T. R., Lew, D. J. Mating in wild yeast: Delayed interest in sex after spore germination. Molecular Biology of the Cell. 29 (26), 3119-3127 (2018).
- Sena, E. P., Radin, D. N., Fogel, S. Synchronous mating in yeast. Proceedings of the National Academy of Sciences of the United States of America. 70 (5), 1373-1377 (1973).
- Werner-Washburne, M., Braun, E., Johnston, G. C., Singer, R. A. Stationary phase in the yeast Saccharomyces cerevisiae. Microbiology Reviews. 57 (2), 383-401 (1993).
- Johnston, S. A., Hopper, J. E. Isolation of the yeast regulatory gene GAL4 and analysis of its dosage effects on the galactose/melibiose regulon. Proceedings of the National Academy of Sciences of the United States of America. 79 (22), 6971-6975 (1982).
- Mahilkar, A. . Study of metabolic specialization leading to speciation, using yeast as a model system. , (2021).
- Blank, T. E., Woods, M. P., Lebo, C. M., Xin, P., Hopper, J. E. Novel Gal3 proteins showing altered Gal80p binding cause constitutive transcription of Gal4p-activated genes in Saccharomyces cerevisiae. Molecular and Cellular Biology. 17 (5), 2566-2575 (1997).
- Rice, W. R., Hostert, E. E. Laboratory experiments on speciation: What have we learned in 40 years. Evolution. 47 (6), 1637-1653 (1993).
- White, N. J., Snook, R. R., Eyres, I. The past and future of experimental speciation. Trends in Ecology and Evolution. 35 (1), 10-21 (2020).
- Milo, R., Jorgensen, P., Moran, U., Weber, G., Springer, M. BioNumbers--The database of key numbers in molecular and cell biology. Nucleic Acids Research. 38, 750-753 (2010).
- Domitrovic, T., et al. Structural and functional study of YER067W, a new protein involved in yeast metabolism control and drug resistance. PLoS One. 5 (6), 11163 (2010).
- Lindegren, C. C., Spiegelman, S., Lindegren, G. Mendelian inheritance of adaptive enzymes. Proceedings of the National Academy of Sciences of the United States of America. 30 (11), 346-352 (1944).
- Dettman, J. R., Sirjusingh, C., Kohn, L. M., Anderson, J. B. Incipient speciation by divergent adaptation and antagonistic epistasis in yeast. Nature. 447 (7144), 585-588 (2007).
- Jhuang, H. Y., Lee, H. Y., Leu, J. Y. Mitochondrial-nuclear co-evolution leads to hybrid incompatibility through pentatricopeptide repeat proteins. EMBO Reports. 18 (1), 87-101 (2017).
- Lee, H. Y., et al. Incompatibility of nuclear and mitochondrial genomes causes hybrid sterility between two yeast species. Cell. 135 (6), 1065-1073 (2008).
- Pronk, J. T. Auxotrophic yeast strains in fundamental and applied research. Applied and Environmental Microbiology. 68 (5), 2095-2100 (2002).
- Madhani, H. D. . From a to α: Yeast as a Model for Cellular Differentiation. , (2007).
- Huxley, C., Green, E. D., Dunham, I. Rapid assessment of S. cerevisiae mating type by PCR. Trends in Genetics. 6 (8), 236 (1990).
- Rang, C., Galen, J. E., Kaper, J. B., Chao, L. Fitness cost of the green fluorescent protein in gastrointestinal bacteria. Canadian Journal of Microbiology. 49 (9), 531-537 (2003).
- Tusso, S., Nieuwenhuis, B. P. S., Weissensteiner, B., Immler, S., Wolf, J. B. W. Experimental evolution of adaptive divergence under varying degrees of gene flow. Nature Ecology and Evolution. 5 (3), 338-349 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유