Method Article
지역 사회 기반의 설정에서 획득 샘플의 신속한 분별 및 전체 혈액 성분의 분리
요약
We outline a methodology for the processing of whole blood to obtain a variety of components for further analysis. We have optimized a streamlined protocol that enables rapid, high-throughput simultaneous processing of whole blood samples in a non-clinical setting.
초록
비 임상 설정에서 수집 및 전체 혈액 샘플의 처리와 기존 조건없이 모두 지역 사회에 거주하는 개인을 평가하는 독특한 기회를 제공합니다. 이들 샘플의 신속한 처리가 주요 세포 성분의 열화를 방지하는 것이 필수적이다. 동시에 말초 혈액 단핵 세포 (PBMC), DNA에 대한 방법이 여기에 포함된다, RNA 및 처리와 대도시 지역에 걸쳐 참가자를 동의의 가정에서 수행 한 혈액 추첨에서 혈청 분리, 컬렉션의 2 시간 이내에 시작했다. 우리는 1,600 혈액 표본이어서 성공적인 DNA 메틸화, 유전자형, 유전자 발현에 이용하고 유동 세포 계측법 분석 한 항복 일관된 고품질의 재료를 처리하는 이러한 기술을 이용했다. 사용되는 방법 중 일부는 표준; 설명 된 방식으로 조합 그러나, 그들은 인구 문제 및 / 또는 참가자를 커뮤니티의 샘플의 효율적인 처리를 가능하게일반적으로 임상에서 평가되지 않는다 기반 연구. 따라서,이 프로토콜은 일반적인 인구의 대표적인 (후속 데이터) 샘플들을 획득 할 수있는 잠재력을 갖는다.
서문
여러 연구와 정신 (또는 다른) 1-4 질병없이 개인간 혈액 유전자 발현, DNA 메틸화 및 세포 서브셋의 차이를 특징으로하고있다. 이러한 연구는, 그러나, 질병 - 관련 차이 때문에 환자를 치료하고자하는 질병에 대해 일반적으로 더 심한 특성을 확대 할 수있는 임상에서 얻어졌다. 때문에 "오 믹스"접근 방법의 발전으로, 지난 십 질병 유병률의 인구 기반의 추정치를 제공하기 위해, 지역 사회 및 / 또는 역학적 설정 5-7에서 생물학적 샘플을 얻는에 대한 관심의 폭발과의 폭 넓은 사진을 보았다 이러한 정신적 / 육체적 질병의 환경 적 결정.
이에 대한 주요 과제는 수집 된 표본의 신속한 처리에 대한 요구이다. 단핵 세포, frequentl 아르 키 면역 시스템 구성 요소들의 열화Y는 개인의 건강, 수집 8-10의 2 시간 후 복구에 상당한 감소와 혈액 무승부를 즉시 시작 평가하는 데 사용됩니다. 이 문제를 해결하기 위해, 우리는 인간의 전체 혈액의 여러 구성 요소가 동시에 대도시 지역에 사는 주제의 가정에서 얻어진 샘플로부터 고립되는 최적화 된 프로토콜을 제시한다. 프로토콜은 우리의 컴파일 및 미래의 기술이 더 분리를 허용 이벤트에있는 모든 "추가"분수의 저장을 포함하여 현재 기술의 변경에 기반 / 분석한다. 다른 방법 또는 키트가 여기에 설명 된 각각의 방법, 그 윤곽의 장소에서 이용 될 수 있지만 높은 처리량 방법으로 샘플을 처리하기위한 안정적이고 효율적인 방법으로 증명되었다. 양질의 분획 (PBMC를는, DNA는 혈청 및 RNA) 신선한 혈액의 2 일 (도 1 내에서 이용 가능하다 (2)의 수집 시간과 모든 분석 준비 시험편 내에 제조 할 수있다).
이 프로토콜은 지역 사회 주거, 디트로이트 환경 건강 연구에서 테스트 디트로이트시의 성인 거주자에서 수집 된 샘플의 효율적인 처리를 가능하게하기 위해 개발되었다 (DNHS을, DA022720, RC1MH088283, DA022720-05-S1)을, 인구 기반 외상 후 스트레스 장애 (PTSD) 및 기타 정신 질환의 사회적, 생물학적 결정 요인 연구. 디트로이트에서 외상 후 스트레스 장애의 유병률은 두 번 전국 평균 11, 12 이상입니다. 이 집단에서 PTSD의 생물학적 결정을 확인하는 것은이 도시의 인구 및 기타 위험이 높은 집단 (예를 들어, 군 참전 용사를 반환) 모두에서 장애 그 고통을 돕기 위해 적절한 약물 및 / 또는인지 행동 개입을 개발하는 데 도움이 될 수 있습니다. 이전에 디트로이트, 미시간 웨인 주립 대학에있는 우리의 실험은, 다양한에서 파생 된 신선한 조직 샘플을 처리하는 우리의 전문 지식을 기반으로 처리를 위해 선정되었다소스, 필요성이 컬렉션의 2 시간 이내에 샘플을 처리하기 시작하고, 수집 사이트에 우리의 근접. 손이 독특한 기회, 우리의 목표는 각 시료로부터 DNA, RNA, 혈청 및 말초 혈액 단핵 세포 (PBMC)의 최대 수율 (시편 컬렉션 5 파도 위에 N = 1,639 샘플의 합계)에 대한 처리를 최적화하는 것이다. 여기에 설명 된 절차에 따라서, 마이크로 어레이, 성적인, 실시간 RT-PCR을 포함하여 하류 다양한 응용을위한 재료 (평균 수율은 표 1 참조)을 출발 제조, 비 임상 환경에서 동시에 수행하고, 분석 유세포 수있다.
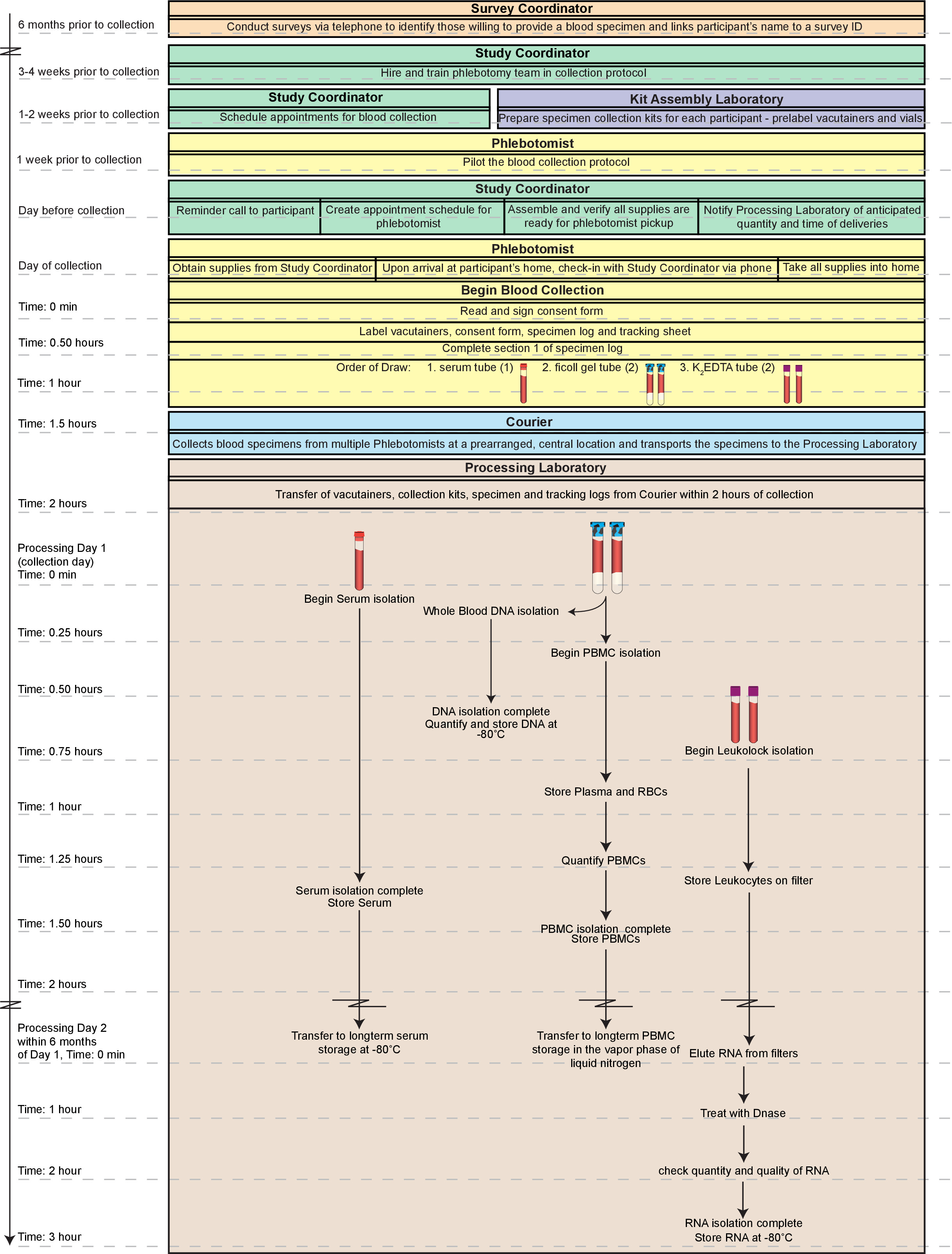
그림 1. 전체 작업 흐름. 여기에 도시 된 전체적인 공정은 동의 partic 식별로부터 혈액 표본을 얻는 물류를 포함혈액에 ipants 자체를 그립니다. 고품질, 분획 (말초 혈액 단핵 세포; PBMC를, DNA, 혈청, 및 RNA) 신선한 전혈의 컬렉션의 2 시간 내에 제조 될 수 있고, 모든 분석 시료 준비 2 일 내에 사용할 수있다. 시료는 즉시 시험 될 수없는 경우 또한,이 방법에 의해 제조 된 분획은 장기간 저장에 적합하다. 여기에 설명 된 전체 타임 라인은 하루 (~ 5 시간의 합계)에 완료 될 수 있습니다. 그러나, 이와 같은 일이 특히 상당한 경험과 기술을 가진 단일 기술자 매우 노동 집약적 일 것이다. 따라서, 우리는 적어도 두 기술자 사이에 1 일의 절차를 나누고 일 2. RNA 처리를 완료 추천 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
디트로이트 환경 건강 연구 검토와 미시간의 임상 시험 심사위원회의 대학에 의해 승인되었다. 모든 참가자는 이전 연구에 참여에 동의 정보를 제공했다.
1. 개요
- 제대로 엔드 포인트 데이터의 분석을 통해 모집에서 모든 단계를 문서화합니다. 단계 전환과 시간이 허락으로 중복, 동시에 1 일에 프로토콜을 수행합니다. 단계의 시퀀스는 성능 효율을 최적화하기 위해 기록된다. (1) 전체의 처리의 개요를도.
참고 : 용어 "참가자는"동의 개인으로부터 그린 드 확인 된 혈액 샘플을 나타 내기 위해 전반에 걸쳐 사용된다. 괄호 안의 시간은 아래의 실습 시간을 나타냅니다. 추적 시트 시험편 로그의 예는 각각 부가 테이블 S1과 S2에서 볼 수있다. 샘플은 가정에서 phlebotomists에 의해 수집(더 전처리 자세한 내용은 보충 정보 (SI 참조) 1) 택배로 연구 코디네이터에 의해 참가자를 동의 및 실험실로 운반로 식별 개인의.
2. 1 일 설치
- 1X 인산염 완충 식염수 (PBS)를 준비합니다.
- 56 ºC에 열 블록을 예열.
- 그날의 배달 (참가자 당 2)에 대한 마이크로 원심 튜브의 적절한 수에 20 μl를 분취하여 단백질 분해 효소 (SI 2 참조) 준비합니다.
- 필요한 경우, 라벨에 표시된 바와 같이 100 % 에탄올을 첨가, 완충제 및 AW1 AW2 (DNA의 순도를 증가 DNA 분리 키트에 세척 버퍼)은 사용할 준비가되도록.
- 4 ℃로 냉장 원심 분리기를 냉각.
- 전송 SPI의 상단에있는 스크류 캡을 유지, 제공된 고무 격막 캡 1X PBS의 병 뚜껑을 교체하고 전송 스파이크와 고무 격막을 관통KE 증발을 방지한다. RNA 안정화 용액 병 반복한다.
- 완충제, 원심 분리기와 열 블록의 제조에 이어, 샘플 추적 시트 vacutainer 전달의 시간을 기록 (SI 표 참조).
3. 1 일 : DNA, PBMC를하고 백혈구 RNA에 대한 처리 항목의 혈액 샘플 (2 시간)
- 개요
- 채혈 및 처리 시작 시간 사이의 지연을 최소화하기 위해 충분한 시간을 할애. 적혈구 세포 오염의 증가 및 단핵 세포 회수의 감소를 방지하기 위해 채혈 2 시간 내에 함유 피콜 vacutainers의 처리를 시작한다.
- 피콜 젤 그라데이션을 포함하는 2 vacutainers, 2 K 2 에틸렌 디아민 테트라 아세트산 (EDTA) vacutainers, 성인 참가자 당 1 혈청 vacutainer의 수집을 가정합니다. 실험실에 vacutainers 전달하면, 기술자가 샘플의 추적 시트 의미 수용에 서명. 빌다표본 로그에 vacutainer별로 기록 시작 시간 즉시 처리한다.
- 혈청 분리 1 단계 (1 분)
- 표본 로그 시작 시간을 기록 (SI 표 2 참조). 원심 분리기 에어로졸 모자와 스윙 버킷 회 사용 (참가자 당 1) 7.0 ㎖의 혈청 (빨간색) vacutainer (브레이크와 가속 OFF 포함) (바이오 안전성 레벨 2를, BSL2가 인증) 20 분, 1,300 XG, 4 ºC합니다.
- DNA 격리 1 단계 (15 분)
참고 :이 격리 절차에 대한 자세한 내용은 제조업체의 프로토콜 13을 참조하십시오.- 시작 시간을 문서화하십시오. BSL2 세포 배양 후드에서 다음 프로테아제의 두 20 μL 씩 (참가자 당 400 ㎕의 총)의 각각에 vacutainers 중 하나의 상부로부터 전혈 200 μL를 추가 피콜 겔 5 배를 함유 vacutainers 반전. 후드에 프로테아제 + 혈액의 microcentrifuge 튜브를 떠나, 센트리 계속단계 3.4.1에서 vacutainers를 포함하는 피콜의 fugation.
참고 : 마음의 단계 3.4.1에 vacutainers 회전 할 때 (즉,이 두 vacutainers에서 각각 200 μl를 제거해야 할 수도 있습니다) 원심 분리기의 균형의 필요성을 유지, 가장 큰 컬렉션 볼륨과 vacutainer을 사용합니다. 또한, 청색 vacutainers 처리하는 동안 적절한 분리를 보장하기 위해, vacutainer에 잔류 혈액 수준은 피콜 층 위 2.5 인치 이하이어야한다.
- 시작 시간을 문서화하십시오. BSL2 세포 배양 후드에서 다음 프로테아제의 두 20 μL 씩 (참가자 당 400 ㎕의 총)의 각각에 vacutainers 중 하나의 상부로부터 전혈 200 μL를 추가 피콜 겔 5 배를 함유 vacutainers 반전. 후드에 프로테아제 + 혈액의 microcentrifuge 튜브를 떠나, 센트리 계속단계 3.4.1에서 vacutainers를 포함하는 피콜의 fugation.
- PBMC 격리 1 단계 (1 분)
- 시작 시간을 기록 vacutainers 8-10 번 함유 피콜 .Invert (적혈구 세포 오염의 증가 및 단핵 세포의 회수의 감소를 방지하기 위해 채혈 2 시간 내에 있어야한다). (브레이크와 가속 OFF)와 원심 분리기 30 분, 1,600 XG, 22 ° C에 대한 에어로졸 모자 (BSL2 인증)와 스윙 버킷 로터를 사용하여 vacutainers (참가자 당 2).
- 의 DNA 분리 2 단계 (20 분)
- 후드로 돌아가서 프로테아제 + 혈액의 microcentrifuge 튜브 (단계 3.3.1)에 각각 200 μL 버퍼 AL를 추가합니다. 모자, 후드에서 소용돌이 15 초 플래시 스핀을 제거합니다.
- 열 블록에서 56 ºC, 10 분을 품어.
- 열 블록에서 튜브를 제거합니다. 플래시 스핀. BSL2 후드로 돌아갑니다. 200 μL 100 % 에탄올을 추가합니다. 모자, 후드에서 소용돌이 15 초 플래시 스핀을 제거합니다. 나머지 단계는 후드 외부에서 완료 될 수있다.
- 해물을 적용 (2 ㎖의 수집 관) 레이블 스핀 컬럼 (단계 3.5.3의 30 분 이내). 에어로졸을 통해 교차 오염을 방지하기 위해 뚜껑을 닫습니다. 원심 분리기 6000 XG, 1 분.
- 여과 액을 포함 포집 관을 버리고 새로운 2 ㎖의 수집 관에 스핀 열을 배치합니다.
- 림 가습없이 열을 500 μL 버퍼 AW1 추가 뚜껑을 닫고, 원심 분리기 6000 XG, 1 분.
- FILTRA를 포함하는 수집 관을 폐기TE는 새로운 2 ㎖의 수집 관에 스핀 열을 배치하고.
- 림 가습없이 열을 500 μL 버퍼 AW2 추가 뚜껑을 닫고, 원심 분리기 20,000 XG, 3 분.
- 여과 액을 포함 포집 관을 버리고 새로운 1.5 ml의 포집 관 (안 키트에 포함)에 스핀 열을 배치하고, 원심 분리기 20,000 XG, 1 분.
- 여과 액을 포함 포집 관을 버리고 새로운 1.5 ml의 포집 관에서 스핀 컬럼을 배치 (키트에 포함되지 않음).
- 각 칼럼에 200 μL 버퍼 AE 또는 물을 추가하고 5 분 동안 실온에서 품어. 원심 분리기 6000 XG, 1 분.
- 동일한 수집 관에 용출 단계를 반복 3.5.11.
- 참가자 당 2 열에서 용출 된 DNA를 풀. 참가자 당 총 수율 ~ 800 μL.
- 시간이 허락 될 때, 분광 광도계를 사용하여 샘플을 계량.
- specime에 각각의 농도를 문서화, 2 ML의 크리오 바이알 (cryovial)에 원하는대로 나누어지는 DNA를N 로그. 전사는 장기 저장을 위해 -80 ℃에서 cryobox 제자리에 크리오 바이알 (cryovial). 냉장고의 시작 시간을 문서화하십시오.
- 백혈구 RNA 격리 1 단계 (30 분)
참고 : 바이오 안전성 레벨 2 인증을 세포 배양 후드에서 수행합니다. 이 격리에 대한 자세한 내용은 제조업체의 지침 (15)을 참조하십시오.- 전송 스파이크와 K 2 EDTA의 vacutainer의 고무 격막을 관통. 칼집을 유지하고 단계 3.6.11에 사용하기 위해 캡 나사. BSL2 표준 관행에 따라, 혈액 유래 병원균에 노출되지 않도록주의하십시오.
- 전송 스파이크의 상단에 흰색 슬립 커넥터를 연결합니다.
- 흰색 슬립 커넥터에 필터의 입구 (플레어 단부)을 연결 한 다음 대응하는 참가자 ID와 필터 레이블.
- 필터의 출구 (테이퍼 끝)에 끼 우고 25⅝ G 바늘을 연결합니다. 인도 전에 필터 조립체가 상당히 따라서 처리를 신속하게 제조필터, 후 피복 바늘을 추가하고 사용할 때까지 배양 튜브 랙에 어셈블리를 설정, 전송 스파이크에 흰색 슬립 커넥터를 부착하는 것이 좋습니다.
- K 2 EDTA 튜브 시스템의 어셈블리에 따라, 안전 (금속 주걱의 끝을 사용) 바늘을 칼집에서 뽑다. 빈 10 ml의 진공 혈액 수집 관 (혈청 수신기 튜브)에 바늘을 찔러 K 2 EDTA의 vacutainer / 필터 / 수신기 튜브 어셈블리를 반전. BSL2 표준 관행에 따라, 혈액 유래 병원균에 노출되지 않도록주의하십시오.
- 필터의 쐐기 모양의 섹션이 피를 해제 할 때까지 혈액을 통해 필터링 할 수 있습니다. 여과는 약 2 분 동안 취해 여과 테스트 튜브 랙에 배치 될 수있다.
- 어셈블리에서 필터를 제거합니다. 여과 액이 들어있는 튜브에 바늘을두고 날카로운 물건 용기에 전체 어셈블리를 폐기합니다.
- 에, 1X PBS의 병에 전송 스파이크로 5 ML의 주사기를 연결합니다병을 수직 및 3 ㎖를 철회.
- (3-5 초당 방울) 필터의 입구에 PBS로 주사기를 연결하고 필터를 세척합니다. 흐름을 통해 생물학적 폐기물 용기에 수거 할 것. 플런저를 철회하지 않고 필터에서 주사기를 분리합니다.
- 여과하고 PBS 세척 후에, 새로운 5 ㎖ 주사기 단계 3.6.8에 기술 된 방법을 사용하여 RNA 안정 화제 3 ㎖를 철회. 단계 3.6.9에서와 같이 필터를 세척합니다. RNA 안정 화제는 필터에 남아 있어야한다. 플런저를 철회하지 않고 필터에서 주사기를 분리합니다.
- RNA 안정화 제로 포화 된 필터를 떠나기 이전부터 스파이크 유지 외피와 스크류 캡이 필터 입구와 출구를 밀봉. 필터는이 시점에서 저장 될 수있다. 5.4.7 5.3 단계를 완료하는 데 시간이 허락 할 때까지 -80 ºC (~ 2 시간)에서 필터를 저장합니다.
참고 : 수집 한 후 1 년까지 -80 ºC에 저장 필터 decrea없이 처리 된RNA 품질 SE.
- PBMC 분리 2 단계 (30 분)
- 원심 분리기 (단계 3.4.1)에서 피콜 포함 vacutainers를 제거하고 BSL2 후드에 계속. Vacutainers 그림 2에서와 같이 분리를 표시해야합니다. 표 2 참조하지 않는 경우.
- vacutainer가 BSL2 후드에 반환되면, 마개를 제거하고 단핵 (일반 / 흰색) 층에 가까운받지 않고 혈청 학적 피펫을 사용하여 위, 황색, 플라즈마 층 (그림 2)의 1.5 ml의 철회. (- 1 참가자 2 vacutainers에서 풀 -) cryovial 5 ml의 플라즈마를 전송합니다. 수집 된 볼륨을 로그인합니다. 저장에 관한 지시 사항 단계 3.7.6을 참조하십시오.
참고 : 최고 분리 및 큰 PBMC 수익률은 이전에 혈액 수집에 적어도 12 시간을 금식 한 참가자에서 왔습니다. - U - 나머지 플라즈마 및 희끄무레, 단핵 층 (그림 2 겔 층 위의 모든 것을) 이동하나의 원뿔형 튜브에 참가자 당 vacutainers 함유 두 피콜 각각으로부터 단핵 층을 풀링, 15ml의 원뿔형 튜브에, 혈청 피펫 노래.
- 15 ml의 원뿔 튜브의 전체 볼륨을 가지고 1X PBS를 추가합니다. 캡 튜브와 반전의 5 배. 15 분, 300 XG, 22 ° C (브레이크와 가속 OFF)와 원심 분리기.
- 후드 피콜 함유 vacutainers로 돌아가서 주위 소용돌이 피콜 겔 층의 외부를 풀고 가능한 경우이를 제거 5¾ "파스퇴르 피펫을 사용하여 적혈구 (적혈구)를 수집한다. 수집 cryovial 5 ml의 적혈구 (~ 4.5 ml)에 전송하는 혈청 학적 피펫을 사용합니다. 수집 된 볼륨을 로그인합니다.
- 제어 속도 냉동 컨테이너 (단계 3.7.5 플라즈마 단계 3.7.2 및 적혈구) 모두 5 ml의 크리오 바이알 (cryovial)를 전송하고 그들이 cryobox로 전송하고 반환 할 수있는 시간 후 최소 24 시간 동안 -80 ºC에 넣어 장기 기억 -80 ºC.
- 공동 반환단계 3.7.4에서 원심 분리가 완료하고, 펠렛을 방해하지 않고 PBS 모든하지만 ~ 500 μl를 흡인 후드로 nical 튜브. PBS의 ~ 200 μL이 단계에서 펠렛 위에두면 PBMC 수율은 크다.
- 10 ml의 볼륨을 가지고 신선한 1X PBS를 추가합니다. 부드럽게 펠렛을 재현 탁. 캡 튜브와 반전의 5 배. 10 분, 300 XG, 22 ° C (브레이크와 가속 OFF)와 원심 분리기.
- 혈청 분리 2 단계 (10 분)
참고 : BSL2 후드에서 수행합니다.- 원하는 2ml의 크리오 바이알 (cryovial)로 원심 분리한다 (단계 3.2.1) 다음, 혈청 vacutainer에서 혈청 상부 층을 나누어지는. 일반적인 수율은 2.5 ㎖ (표 1)입니다. 볼륨을 로그인합니다. 예를 들어, cryovial 1로 200 μl를 분취 4 크리오 바이알 (cryovial), cryovial 2에 1,000 μl를 사용하고 크리오 바이알 (cryovial) 3과 4에 나머지를 나눕니다.
- 장기 보관을 위해 -80 ºC에서 cryobox 장소 크리오 바이알 (cryovial)로 옮긴다. 냉장고의 시작을 문서화시간.
- PBMC 분리 3 단계 (15 분)
- 펠렛을 방해하지 않고 가능한 한 많이 뜨는 / PBS로 원심 분리 (단계 3.7.8)를 흡인 한 후, 후드로 돌아갑니다. 보통 1 냉동 2.5 ml의 PBMC에서 피펫으로 재현 탁 펠릿 (SI 2 참조).
- 단계 3.9.1의 셀 / 매체 솔루션에 중간 2 (SI 2 참조) 냉동 2.5 ml의 PBMC를 추가합니다. 소용돌이 부드럽게.
- 0.65 mL의 분취 량의 microcentrifuge 튜브에 세포 용액 10 μL (상기 희석이 필요할 수있다). 0.65 ㎖의 microcentrifuge 관에 0.4 % 트리 판 블루 염색의 10 μl를 추가하고 여러 번 피펫 팅하여 혼합한다. 자세한 내용은 제조업체의 사용 설명서 (16)를 참조하십시오.
- 피펫 혼합의 3 분 이내에 셀 카운터에 챔버 슬라이드 및 장소 슬라이드를 계산 셀에 혼합물의 10 μL. 확대하고 세포를 집중한다. PBMC 카운트 얻기 위해 "카운트 세포"를 누르십시오.
- 나누어지는, 가능한 경우크리오 바이알 (cryovial)에 원하는 및 3.9.9 단계로 계속 PBMC 번호, 밀리리터 당 300 만 셀 (MC / ㎖) 이상입니다. 적어도 3 MC / ㎖ 각각의 농도에서 최대 5 크리오 바이알 (cryovial)에 보관 된 PBMC.
- 가능한 PBMC 수가 3 미만이면 MC / ㎖, 5 ㎖에 의해 실행 가능한 MC / ㎖를 승산함으로써 세포의 총 수를 계산한다. 3 MC / ㎖와 적어도 하나의 관이 될 수 있도록 4, 3, 2, 1 ml로 그 번호를 나눕니다.
- 원심 분리기 (300) XG (제동 및 가속 OFF)에서 5 분 동안 세포 / 동결 배지 용액을 함유 원추형 튜브. 원심 분리 후, (4, 3, 2, 1 mL)을 상기 계산 된 부피를 떠나 냉동 배지 (상징액)의 적절한 부피를 대기음.
- cryovial / 1 ml의에서 크리오 바이알 (cryovial) (1-4)의 해당 번호에 남아있는 상층 액에서 펠렛과 나누어지는 적어도 3 MC / ㎖을 재현 탁. 최종 동결 배지는 10 % 디메틸 설폭 사이드 (DMSO) / 20 % 소 태아 혈청 (FBS) / 70 % 로스웰 파크 메모리얼 연구소 (RPMI) 1640.
- Documcryovial 당 세포 수를 ENT. 제어 속도 냉동 컨테이너에 크리오 바이알 (cryovial)를 전송하고 크리오 바이알 (cryovial)가 cryobox로 전송 및 장기 저장을위한 액체 질소 탱크 (기상)에 배치 될 수있는 시간 후 최소 24 시간 동안 -80 ºC에 넣어. 문서 냉동 시간을 시작합니다.
4 일 2 설치
- 열 블록에 80 ° C에 뉴 클레아없는 튜브에 뉴 클레아 무료로 0.1 mM의 EDTA의 나누어지는 (필터 당 220 μL)를 가열한다.
- 세척 용액 1 및 2 (SI 2 참조)를 제조 하였다.
5. 주 2 : 장기 샘플 보관 및 백혈구 필터 처리 (3 시간)
- 개요.
- 필터에 백혈구의 적용 6 개월 이내에이 절차를 수행합니다.
주 : 전체적으로는 2 일째 처리는 약 3 시간 소요가 표시된 상태에서 "제 2 일,"핵심 요구 사항은 필터 처리 부가 일 때 수행되어야한다는 것이다실험실에서 발생에는 1 일 처리가 없습니다.
- 필터에 백혈구의 적용 6 개월 이내에이 절차를 수행합니다.
- 장기 샘플 저장소 (15 분)
- 이전에 1 일 처리를 위해 실험실에 새로운 vacutainers의 전달에, 매일이 절차를 수행합니다. 적절하게 레이블이 cryoboxes에 1 일에 제어 속도 냉동 용기에 O / N를 저장 한 크리오 바이알 (cryovial)를 전송하고 장기간 보관시 -80 ºC 또는 액체 질소 (기상)로 돌아갑니다. 종점 분석법 추적 샘플 위치를 신속하게 샘플 유형 (예, DNA, PBMC, 적혈구 등)에 기초 크리오 바이알 (cryovial)를 구성.
- 백혈구 RNA 필터 처리 (45 분).
참고 :이 절차에 대한 자세한 내용은 제조업체의 지침 (17)를 참조하십시오.- RT에 필터를 가지고 (약 5 분 동안 해동).
- 덮개를 제거하고 필터에서 캡 나사. 플런저 우울, 5 ㎖ 주사기의 플런저를 후퇴 필터의 입구 (플레어 단부)에 연결RNA 안정화 에이전트를 추방 생물학적 폐기물 용기에 포트로부터 필터.
- 표지 된 15ml의 원뿔형 튜브에 해물을 수집, 필터를 통해 용액을 세척하기 위해 플런저 우울, RNA 분리 용 페놀 및 구아니딘 이소 티오 시아 네이트 용액을 4 ㎖의 새로운 5 ㎖ 주사기를로드하고 필터의 입구에 부착 (참가자 당 2).
- 플런저를 후퇴, 필터로부터 주사기를 분리 필터에 다시 부착하고, 동일한 15ml의 원뿔형 튜브에 필터 디스크에 갇혀 잔류 샘플을 배출하도록 플런저 우울. 필터 주사기를 폐기하십시오.
- , 원뿔 튜브에 800 μL BCP를 추가 단단히 튜브를 닫고 적극적으로 30 초 동안 준비를 흔들.
- 5 분 동안 실온에서 품어. 2,000 X g에서 10 분 동안 원심 분리기.
- 갓 표시 15 ML 원뿔 튜브 (~ 2.5 ㎖)로 수성 (위) 상을 전송합니다.
- 뉴 클레아 제 무 수분의 성상의 0.5 배 부피를 추가잘 섞는다 거라고. 이어서 100 % 에탄올 수용액 1.25 부피 추가하고 다시 혼합한다.
참고 : 2.5 ml의 수성 볼륨의 경우, 뉴 클레아 무료 물 1.25를 가하여 혼합 한 후 4.7 ml의 100 % 에탄올 (2.5 ml의 X 0.5 = THEN 2.5 ml의 + 1.25 ml의 = 3.75 ml의 새로운 수성 볼륨 1.25 ml의 뉴 클레아 무료 물을 추가 그때 3.75 ml의 X 1.25 = 4.7 ml의 100 % 에탄올). 이 단계는 작은 RNA 분획을 포함하는 총 RNA의 분리 가능하다. 격리에서 작은 RNA를 생략하는 방법이 수동 17에서 찾을 수있다. - 5 ML의 주사기에서 플런저를 제거하고 그 자리에 스핀 카트리지를 삽입합니다. 진공 매니 폴드에 카트리지 / 주사기 어셈블리를 연결합니다. 바닥과 1.5 ML의 microcentrifuge 관이 차단 내부의 진공 매니 폴드에보다 안전한 연결의 경우, 카트리지 / 주사기 어셈블리를 배치합니다.
- 이 카트리지를 당겨주의 깊게 더 많은 샘플을 추가,에 진공으로 천천히 스핀 카트리지 단계 5.3.8에서 샘플을 적용합니다.
주 : 진공이없는 경우선물, http://www.ambion.com/techlib/misc/leuko_iso.pdf에서 원심 분리 방법을 참조하십시오. - 1.5 ml의 microcentrifuge 관에 스핀 카트리지를 전송하고 세척 1. 원심 분리기의 스핀 카트리지 / 튜브 어셈블리 (5) 750 μl를 추가 - 12,000 X g에서 10 초.
- 튜브에서 여과 액을 취소하고 같은 microcentrifuge 관에 스핀 카트리지를 반환합니다.
- 12,000 X g에서 10 초 - 5 워시 2 원심 분리기의 스핀 카트리지 / 튜브를 750 μl를 추가합니다. 단계 5.3.12에서와 같이 여과 액을 버린다. 같은 microcentrifuge 관에 스핀 카트리지를 돌려줍니다.
- 단계 5.3.13에서와 같이 세척 2, 원심 분리의 또 다른 750 μL로 반복합니다. 취소 여과 액. 같은 microcentrifuge 관에 스핀 카트리지를 돌려줍니다. 필터를 건조 1 분 동안 최대 속도로 스핀 카트리지 / 튜브를 원심 분리기.
- 신선한 스핀 카트리지를 이동, 1.5 μL microcentrifuge 관을 표시.
- 뉴 클레아없는 0.1 mM의 EDTA의 200 μl를 추가 (80 & #로 예열스핀 카트리지 필터 참가자 당 (2)의 중심 C) 176. 1 분 동안 실온에서 품어.
- RNA를 용출 12,000 XG에서 1 분 동안 원심 분리기. 여과 액을 보관하십시오. 스핀 카트리지를 폐기하십시오.
- 신선한 1.5 ml의 마이크로 원심 튜브, 라벨에 두, 100 μL 분취에 각 200 μL 여과 액을 분할하고 얼음에 보관하십시오. 이 시점에서, 참가자 당 2 필터로 시작하는 참가자 당 RNA의 네 100 μL 씩을 얻을 것입니다.
- DNase의 처리 (1 시간)
참고 :이 치료에 대한 자세한 내용은 제조업체의 프로토콜 18을 참조하십시오.- (오류를 피펫 팅을 설명하기 위해) 내가 나누어 + 1 당의 DNase I 버퍼의 10 μL 2 μL의 rDNase을 결합하여 마스터 믹스를 만듭니다.
참고 : 4 개의 샘플의 경우, 100 μL 씩 50 μL의 DNase I 버퍼 + 10 μL의 rDNase (I)가 사용 - 나누어지는 12 단계 5.3.18에서 RNA 씩의 각 단계에 5.4.1의 마스터 믹스 μL 가볍게 섞는다. 37 품어열 블록에 30 분 동안 C를 °.
- DNase의 불 활성화 시약 와동 각 나누어지는 11.2 μl를 추가, 잘 섞는다. 배양하는 동안 2 ~ 3 번 텍싱 2 분 동안 실온에서 품어.
- 1.5 분 동안 10,000 XG에 원심 분리기.
- 신선한 1.5 ML 튜브에 뜨는 (RNA)를 전송합니다. 같은 참가자의 분취 량은 여기에 풀링 할 수있다.
- 농도를 얻을 수있는 분광 광도계에 각각의 샘플을 실행합니다. RNA의 품질 분석은 Bioanalyzer을 (단계 5.5 참조)를 사용하는 것이 좋습니다.
- 원하는대로 크리오 바이알 (cryovial)로 RNA를 나누어지는. Bioanalyzer 분석 이후 분석을 위해 마이크로 원심 튜브로 즉시 분취 샘플 1.5 μL를 수행하지 않을 경우. 농도 및 냉동고 시작 시간을 문서화. -80 ° C에서 보관 샘플.
- (오류를 피펫 팅을 설명하기 위해) 내가 나누어 + 1 당의 DNase I 버퍼의 10 μL 2 μL의 rDNase을 결합하여 마스터 믹스를 만듭니다.
- Bioanalyzer 분석 (1 시간)
- 제조업체의 프로토콜 19을 따르십시오. 실행이 완료되면, 데이터를 자동으로 저장되지만 w 다시 저장되고작고, 더 인식 파일 이름 번째. 예상 결과 (그림 3) : 사다리는 샘플 3 피크 (2 리보솜 피크 44 초, 50 초, 각각 25 초에서 1 초 마커 절정을)해야한다, 6 피크를해야한다.
결과
이것은 전체적인 절차 유전자 마이크로 어레이와 RT-PCR 분석을 통해 발현 후성 변형을 검출하고, 셀 집합의 변화를 포함 하류 다수의 애플리케이션에서 분석을위한 높은 품질의 재료를 생성하는 것이 필수적이다. 표 1은 물질의 평균 수율 및 품질을 나타내는 각 프로세스에서. 3 백혈구 RNA 분리 및 필터 처리 방법의 품질의 출력의 예를 그림. 도 3의 좌측 이미지는 모세관 전기 영동에 의한 겔 사진이다. 각 레인은 저하를 나타냅니다 최소한의 그림자와 함께 두 가지 밴드를 생산한다. 아래 겔 크로마토 그램의 피크의 위치와 크기에 기초하여 결정될 수있다 열화의 레벨 및 유형에 추가적인 모습을 제공한다. (; 저하 낮음)에 RNA 무결성 번호 (린) 1 범위 다른 품질 측정10 (높은 순수한, 좋은 품질의 RNA).
이러한 높은 처리량 방법은 가끔 오류로 빌려 준다,하지만 품질 관리를 보장하기 위해 처리의 여러 단계에 걸쳐 몇 가지 체크 포인트가있다. 피콜 포함 vacutainer의 원심 분리 다음이 표시 적절한 분리를 그림. 표 2에 나타낸 바와 같이 vacutainer 원심 속도, 낮은 컬렉션 볼륨, 또는 공복 참가자의 부족에 오류를 포함하여이 거리를 표시 할 수있는 몇 가지 이유가있다.
이 절차를 사용하여 격리 된 표본의 하위 집합 (N = 100) 11 pyrosequencing에 의해 그 결과의 검증을 포함 HumanMethylation27 (HM27) DNA 분석 BeadChips에 의해 성공을 분석 하였다. 유전자형은 중아 pyrosequencing 타겟 및 실시간 RT-PCR이 성공적 먼과 Uddin 20,21,22,23 실험실에서 수행되었다. 세럼, 파에 고립Beadchip와 유전자형 분석을위한 DNA를 모두 분리하기 병렬로 성공적으로 IL-6 및 C 반응성 단백질의 활성 (24)에 대해 분석 하였다. 단리 된 PBMC에서 T 세포의 서브 세트가 성공적으로 25-28 유세포 분석 하였다. 또한, 변성 백혈구 절차에서 RNA 와이드 유전자 발현 프로파일 링 (29)을 게놈을 실시하고있다.
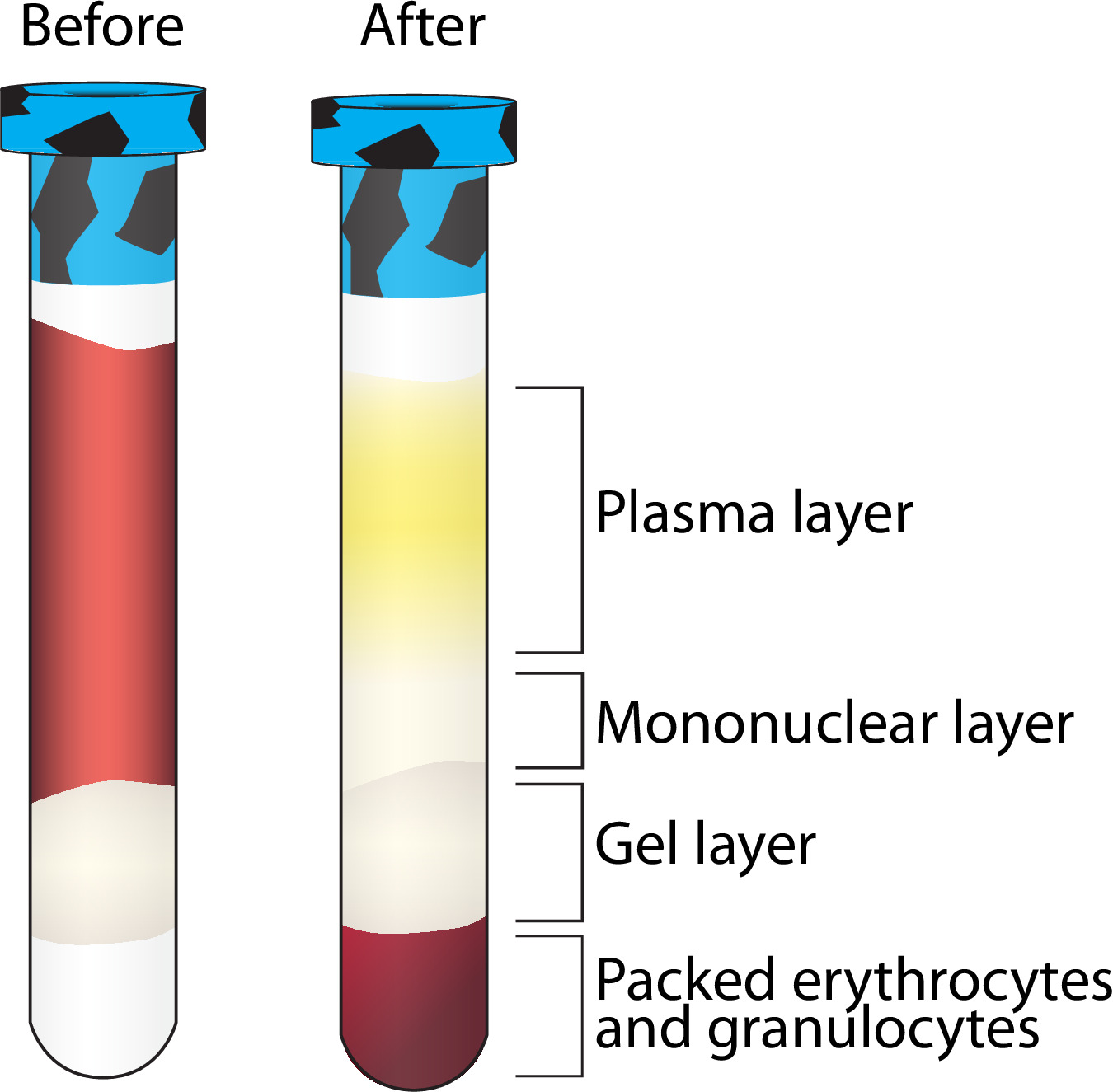
피콜 포함 vacutainer의 원심 분리 다음 그림 2 층 분리. 원심 분리 다음과 같은 여러 혈액 성분의 분리의 시각화. 다른 층은 ° C ~ -80에 저장하는 동안 단핵 층 더 정제입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
: FO "jove_content" 
설명 백혈구 RNA 분리 및 필터 처리 방법 (프로토콜 3.6 및 5.3 참조)와 격리 RNA 무결성 번호 (RINs) 8 위와 18S와 28S 리보솜 RNA를 나타내는 두 가지 대역을 표시하는 그림 3. 예상 백혈구 RNA 처리 결과. Bioanalyzer 결과. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 총 평균 수익률 (N≈500) | 평균 품질 | |
| 혈청 | 2.30 ㎖의 | N / A |
| 전체 혈액에서 DNA | 39.97 μg의 | 280 분의 260 = 1.74에서 흡광도 |
| PBMC | 22250000 가능한 세포 | 전체적인 분리의 적어도 95 %의 생존 |
| 백혈구에서 RNA | 44.09 μg의 | 280 분의 260 = 2.01에서 흡광도 RNA 무결성 번호 (RIN)는 6.48를 = |
표 1. 예상 수익률. 평균 수량과 샘플의 품질은 기술 된 방법을 사용하여 처리.
| 문제 | 잠재적 인 솔루션 |
| 피콜 포함 vacutainer 원심 분리 후 없음 분리 없음 | Vacutainer은 용량에 작성되지 - 적절한 처리를 위해 최소 수집 볼륨 6 ml의입니다. |
| , 원심 분리기 설정을 확인 속도가 G-힘에 있는지 확인합니다. 잘못된 경우, G-힘과 리 스핀으로 설정. | |
| 낮은 PBMC 공동UNT | 혈액의 볼륨이 너무 낮은립니다. |
| 단계 3.7.8의 펠렛에 너무 가까이 흡입 | |
| 비 금식 참가자. | |
| 혈청 vacutainer 원심 분리 후 없음 분리 없음 | , 원심 분리기 설정을 확인 속도가 G-힘에 있는지 확인합니다. 잘못된 경우, G-힘과 리 스핀으로 설정. |
| Nanodrop 1000을 사용하여 예기치 않은 농도 | 측정이 적절한 샘플 유형 (예를 들면, DNA 또는 RNA)에 대한 있는지 확인합니다. |
표 2. 문제 해결. 기술 방법과 잠재적 인 솔루션으로 발생하는 일반적인 문제.

보충 표 1. 추적 시트. 이리저리 vacutainers 추적의 예실험실 배달 M 혈액 수집. 이 테이블을 보려면 여기를 클릭하십시오.
보충 표 2. 시료 로그. 실험실로 전달에 vacutainers의 처리를 문서화하는 데 사용되는 문서의 예. 이 표를 보려면 여기를 클릭하십시오.

보충 표 3. Cryovial 번호 시스템. 각 참가자에 사용되는 번호 체계의 예. 이 표를 보려면 여기를 클릭하십시오.
보충 1. 전처리 세부 정보. 연구 코디네이터, 택배 및 Phlebotomist의 의무에 대해 자세히 설명합니다. 추가 정보를 보려면 여기를 클릭하십시오.

보충 2. 조리법. 필요한 시약을위한 조리법의 목록입니다. 추가 정보 2를 보려면 여기를 클릭하십시오.
토론
우리는 성공적으로 디트로이트 환경 건강 연구에 1,600 개 이상의 전체 혈액 샘플을 처리하기 위해 적용된 간소화 된 프로토콜을 설명했다. 이들 기술의 대부분은 기존 문헌에서 이용할 수 있지만, 각 단계 사이에 정확하게 타이밍 변화를 포함하여 단계별 편집, 성공적 하류 광범위한 응용으로 생물학적 시료의 종류를 생산 최적화 효율적인 프로토콜이 반영 DNA 메틸화, mRNA 발현과 면역 학적 분석을 포함. 이 시편은 이미 국가 회의 11,20,21,23,24,29에서 동료 검토 문헌에 출판 및 / 또는 제출 된 결과를있는 실험의 다양한 테스트되었습니다. 이 프로토콜은 따라서 DNHS 유사한 인구 기반 연구에서 생물 표본을 수집하고자하는 다른 연구자들의 관심을 끌 수있을 것입니다.
이러한 높은 throughp의 샘플을 처리 할 때UT 방식에서는, 모든 단계에서 정확한 기록을 유지하기 위해 매우 중요하다. 우리는 초기에 cryovial 모든 정보를 저장하는 데이터베이스를 개발 권장. 이 데이터베이스는 (상자 안에 보관 상자 번호와 위치) 양, 농도, 품질, 바코드 및 저장 위치를 포함하여 각 cryovial 내 샘플의 모든 측면을 포함해야한다. 우리는 미리 라벨링 크리오 바이알 (cryovial)과 바코드를 포함하는 스토리지 박스를 준비하는 것이 좋습니다. 또한, 우리는 편리한 각 참가자 (표 S3)에 각각 특정 튜브에 대한 바코드를 포함하는 "마스터"문서를 발견했다. 이 샘플에 불필요한 냉동 / 해동 사이클을 도입 샘플 과도한 처리없이 데이터베이스에 cryovial 데이터의 신속한 입력이 가능하고 수동으로 바코드에 입력 실수의 가능성을 제거한다. -150에 액체 질소 -178의 그것은 레이블이 극단적으로 준수하는 것도 필수적이다 (예를 들어, 기상° C) 온도. 문서는 많은 시간이 소요 될 수 있으며, 가진 두 개 이상의 기술자는 프로세스가 가장 큰 효율을 생산하고 나누는 것을 인도시 효율적인 처리의 필요성과 함께, 우리는 발견했다.
이러한 방법의 한계는 수집 사이트 실험실 근방이다. 특히 PBMC의 분리를 위해, 샘플은 적혈구 오염에 상당한 증가를 피하기 위해 수집 2 시간 내에서 처리되어야하고 단핵 세포에서 감소한다. 따라서, 실험실은 발생할 수있는 모든 트래픽 관련 문제를 설명하기 위해 수집 사이트에서 더 이상 30 분 이상이어야한다. 또한, 제안하는 어떤 연구소는 최적의 샘플 처리를 보장하기 위해 손에 두 기술자가 필요합니다 여기에 설명 된 프로토콜을 수행합니다. 또한, 각 기관은 위에서 설명한 각 단계에 필요한 장비에 대한 액세스 권한이 있어야합니다. 따라서, 적은 수의 직원 및 / 또는 제한된 장비 W와 실험실가능성이 프로토콜을 수행 할 수 없습니다 울드.
이 프로토콜의 장점은 동의 개인 가정에 직접 시료를 수집 할 수있는 능력이다. 이것은 일반적으로 아마 때문에 보험이나 교통의 부족, 의료 도움을 요청하지 않을 정신 또는 다른 건강 문제를 가진 개인에 도달 할 수있는 연구를 할 수 있습니다. 또한 비슷한 트리거를 경험하지만, 자신의 정신 건강 증상의 관점에서 차이가있다 같은 지역 사회에 거주 영향 및 영향을받지 않는 개인의 비교를 가능하게한다. 이러한 방식으로 표본을 얻기 분야에서 phlebotomists의 정확한 타이밍과 조정이 필요합니다. 저희 연구실은 우리가 공부 한 사회 내에 위치한 되었기 때문에, phlebotomist는 일반적으로 두 세 가정에서 샘플을 수집하고 첫 컬렉션의 2 시간 창 내 실험실에 샘플을 제공 할 수 있습니다. 효율성은 우리의 샘플 처리의 핵심이며, 같은, 우리는 여러 phlebotomists했다필드는, 정밀한 조정에 대한 필요성을 증가시킨다. 실험실 떨어져 2 시간 간격 배달 당 최대 8 개의 참가자들과 여러 혈액 배달을 받았다. phlebotomists는 지정된 장소에서 만나 하나의 phlebotomist는 하나의 배치로 실험실 표본을 제공하는 자신의 컬렉션을 결합했다. 여기에 기술 된 방법은 쉽게 많은 다른 표현형 검정 및 하류의 다수에 사용될 수 수집 생물학적 표본을 연구하기에 적합 할 수있다.
공개
저자는 그들이 더 경쟁 이익이 없다고 선언합니다.
감사의 말
We would like to thank Henriette Mair-Meijers for invaluable attention to detail and hours devoted to processing the blood collections. We are grateful for the graphic design expertise of Natalie Jameson Kiesling. We also appreciate the approval of the manufacturers (Qiagen (Valencia, CA), BD Biosciences (San Jose, CA), Life Technologies (Grand Island, NY)) mentioned herein to publish the use of their products as described. Funding for this work was generously provided by the National Institutes of Health award numbers DA022720, RC1MH088283, and DA022720-05-S1.
자료
| Name | Company | Catalog Number | Comments |
| QIAamp DNA Blood Mini Kit | Qiagen | 51104 | Day 1: DNA isolation |
| Phosphate-buffered saline (PBS) | Sigma | P5493-1L | Day 1: PBMC isolation |
| 5 ¾” Pasteur pipets | Fisher | 13-678-6A | Day 1: PBMC isolation |
| Fetal Bovine Serum (FBS), heat inactivated | Life Technologies | 10082147 | Day 1: PBMC isolation |
| Dimethyl Sulfoxide (DMSO) | Sigma | D8418-500ml | Day 1: PBMC isolation |
| RPMI Medium 1640, liquid | Invitrogen | 11875119 | Day 1: PBMC isolation |
| 0.4% trypan blue stain | Invitrogen | T10282 | Day 1: PBMC isolation |
| Countess Cell Counting Chamber | Invitrogen | C10283 | Day 1: PBMC isolation |
| Countess Automated Cell Counter or cell counting device such as a microscope and hemocytometer | Invitrogen | C10281 | Day 1: PBMC isolation |
| LeukoLOCK Fractionation & Stabilization Kit | Ambion | 1933 | Day 1: Leukocyte RNA isolation |
| 25 G x 5/8 in. needles | Becton Dickinson | 305122 | Day 1: Leukocyte RNA isolation |
| Syringes (5 ml) | Becton Dickinson | 309646 | Days 1 and 2: Leukocyte RNA isolation |
| Denaturing Lysis Solution | Ambion | 8540G | Day 2: Leukocyte RNA isolation |
| 5 M NaCl | Life Technologies | 24740011 | Day 2: Leukocyte RNA isolation |
| TRI Reagent | Ambion | 9738 | Day 2: Leukocyte RNA isolation |
| Bromo-3-chloro-propane (BCP) | Sigma | B-9673 | Day 2: Leukocyte RNA isolation |
| spin cartridges | Ambion | 10051G | Day 2: Leukocyte RNA isolation |
| 0.1 mM EDTA | Ambion | 9912 | Day 2: Leukocyte RNA isolation |
| DNA-free Kit | Ambion | AM1960 | Day 2: DNase treament |
| RNA 6000 Ladder | Agilent | 5067-1529 | Day 2: Bioanalyzer analysis |
| RNA 6000 Nano Series II Kit | Agilent | 5067-1511 | Day 2: Bioanalyzer analysis |
| RNaseZAP | Ambion | AM8782 | Day 2: Bioanalyzer analysis |
| Ethanol >99% | Sigma | E7023-500ml | |
| Isopropanol >99% | Sigma | I9516-500ml | |
| Nuclease-free ultra pure water | Invitrogen | 9938 | |
| Pipette tips (nuclease-free) | Eppendorf | 22491253 | |
| Pipetter (serological) | Eppendorf | 2223020-4 | |
| Pipetters (for volumes under 1 ml) | Eppendorf | 3120000-054 | |
| Pipettes (serological) | Fisher | 13-678-27E | |
| Controlled rate freezing containers | Nalgene | 5100-0001 | |
| Cryoboxes (to hold 2 ml and 5 ml cryovials and 1.5 ml microcentrifuge tubes) | Fisher | 03-395-464 | |
| Test tube rack | Thermo Scientific | 14-804-134 | |
| 15 ml polypropylene tubes | Fisher | 14-959-49D | |
| 1.5 ml and 0.65 microcentrifuge tubes | Fisher | 07-200-534 and 07-200-185 | |
| 2 ml and 5 ml cryovials | Fisher | 10-500-26 and 10-269-88F | |
| 8 ml CPT vacutainer | BD Biosciences | 362761 | 2 tubes |
| 6 ml K2 EDTA vacutainer | BD Biosciences | 367863 | 2 tubes |
| 8.5 ml SST vacutainer | BD Biosciences | 367988 | 1 tube |
| Vortexer | Fisher | 2215365 | |
| Dry bath incubator with heating block for microcentrifuge tubes | Fisher | 11-715-1250 | |
| Filtration/vacuum system for use within the cell culture hood | Fisher | 01-257-87 | |
| Fixed-angle rotor for microcentrifuge tubes with aerosol-tight lid | Eppendorf | 22637002 | |
| Refrigerated centrifuge with a swing-bucket rotor and aerosol-tight caps for 16 x 125 mm vacutainers and 15 ml polypropylene tubes | Eppendorf | 22628157 | 2, one does not need to be refrigerated |
| Nanodrop 2000 (recommended for accuracy of small volumes) or other spectrophotometric device | Fisher | 13-400-411 | |
| Agilent Bioanalyzer | Agilent Technologies | G2940CA | |
| Liquid nitrogen tank | Thermo Scientific | 11-676-56 | |
| Sharps container | Fisher | 22-037-970 | |
| Biological waste container | Thermo Scientific | 1223P52 | |
| Biosafety Level 2 certified cell culture hood | Thermo Scientific | 13-261-315 |
참고문헌
- Hernandez, M. E., Martinez-Fong, D., Perez-Tapia, M., Estrada-Garcia, I., Estrada-Parra, S., Pavon, L. Evaluation of the effect of selective serotonin-reuptake inhibitors on lymphocyte subsets in patients with a major depressive disorder. Eur Neuropsychopharmacol. 20, 88-95 (2010).
- Weigelt, K., et al. TREM-1 and DAP12 expression in monocytes of patients with severe psychiatric disorders EGR3, ATF3 and PU.1 as important transcription factors. Brain Behav Immun. 25, 1162-1169 (2011).
- Robertson, M. J., et al. Lymphocyte subset differences in patients with chronic fatigue syndrome, multiple sclerosis and major depression. Clin Exp Immunol. 141, 326-332 (2005).
- Rotter, A., Asemann, R., Decker, A., Kornhuber, J., Biermann, T. Orexin expression and promoter-methylation in peripheral blood of patients suffering from major depressive disorder. J Affect Disord. 131, 186-192 (2011).
- Klengel, T., et al. Allele-specific FKBP5 DNA demethylation mediates gene-childhood trauma interactions. Nat Neurosci. 16, 33-41 (2013).
- Segman, R. H., Shefi, N., Goltser-Dubner, T., Friedman, N., Kaminski, N., Shalev, A. Y. Peripheral blood mononuclear cell gene expression profiles identify emergent post-traumatic stress disorder among trauma survivors. Mol Psychiatry. 10, 500-513 (2005).
- Smith, B. H., et al. Cohort Profile: Generation Scotland: Scottish Family Health Study (GS:SFHS). The study, its participants and their potential for genetic research on health and illness. Int J Epidemiol. 42, 689-700 (2013).
- Mallone, R., et al. Isolation and preservation of peripheral blood mononuclear cells for analysis of islet antigen-reactive T cell responses: position statement of the T-Cell Workshop Committee of the Immunology of Diabetes Society. Clin Exp Immunol. 163, 33-49 (2011).
- Duvigneau, J. C., Hartl, R. T., Teinfalt, M., Gemeiner, M. Delay in processing porcine whole blood affects cytokine expression. J Immunol Methods. 272, 11-21 (2003).
- Debey, S., et al. Comparison of different isolation techniques prior gene expression profiling of blood derived cells: impact on physiological responses, on overall expression and the role of different cell types. Pharmacogenomics J. 4, 193-207 (2004).
- Uddin, M., et al. Epigenetic and immune function profiles associated with posttraumatic stress disorder. Proc Natl Acad Sci U S A. 107, 9470-9475 (2010).
- Kessler, R. C., Wang, P. S. The descriptive epidemiology of commonly occurring mental disorders in the United States. Annu Rev Public Health. 29, 115-129 (2008).
- . QIAamp® DNA Mini and Blood Mini Handbook Available from: https://www.qiagen.com/us/resources/resourcedetail?id=67893a91-946f-49b5-8033-394fa5d752ea (2010)
- Koenen, K. C., et al. SLC6A4 methylation modifies the effect of the number of traumatic events on risk for posttraumatic stress disorder. Depress Anxiety. 28, 639-647 (2011).
- Walsh, K., Uddin, M., Soliven, R., Wildman, D. E., Bradley, B. Associations between the SS variant of 5-HTTLPR and PTSD among adults with histories of childhood emotional abuse: Results from two African American independent samples. J Affect Disord. 161, 91-96 (2014).
- Bustamante, A. C., et al. Childhood maltreatment is associated with epigenetic differences in hypothalamic-pituitary-adrenal (HPA) axis genes in the Detroit Neighborhood Health Study. , (2013).
- Sipahi, L., et al. Longitudinal epigenetic variation of DNA methyltransferase genes is associated with vulnerability to post-traumatic stress disorder. Psychol Med. 44, 3165-3179 (2014).
- Uddin, M., Koenen, K. C., Aiello, A. E., Wildman, D., de los Santos, R., Galea, S. Epigenetic and inflammatory marker profiles associated with depression in a community-based epidemiologic sample. Psychol Med. 41, 997-1007 (2011).
- Uddin, M., et al. Post-traumatic stress disorder is associated with immunosenescent T cell phenotypes in the Detroit Neighborhood Health Study. , (2011).
- Uddin, M., et al. Post-traumatic stress disorder is associated with immunosenescent T cell phenotypes in the Detroit Neighborhood Health Study. , (2011).
- Uddin, M. Biological signatures of post-traumatic stress disorder in the Detroit Neighborhood Health Study. , (2010).
- Aiello, A. E. Cytomegalovirus antibodies as a marker of immunosenescence in the Detroit Neighborhood Health Study. , (2010).
- Bustamante, A. C., et al. . Distinct gene expression profiles characterize lifetime PTSD and childhood maltreatment in the Detroit Neighborhood Health Study. , (2013).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유