Method Article
Fraccionamiento rápida y aislamiento de componentes de sangre total en las muestras obtenidas a partir de una configuración basada en la comunidad
En este artículo
Resumen
We outline a methodology for the processing of whole blood to obtain a variety of components for further analysis. We have optimized a streamlined protocol that enables rapid, high-throughput simultaneous processing of whole blood samples in a non-clinical setting.
Resumen
Recogida y tratamiento de muestras de sangre entera en un entorno no-clínica ofrece una oportunidad única para evaluar los individuos residentes en la comunidad con y sin condiciones preexistentes. Procesamiento rápido de estas muestras es esencial para evitar la degradación de los componentes celulares clave. Aquí se incluyen métodos para periférica de células mononucleares de sangre simultánea (CMSP), el ADN, el ARN y el aislamiento suero de una sola extracción de sangre se realiza en los hogares de los participantes consienten a través de un área metropolitana, con el procesamiento de iniciarse dentro de 2 horas de la recolección. Hemos usado estas técnicas para procesar más de 1.600 muestras de sangre rendimiento, material de alta calidad consistente, que posteriormente ha sido utilizado con éxito en la metilación del ADN, genotipificación, expresión génica y análisis de citometría de flujo. Algunos de los métodos empleados son estándar; sin embargo, cuando se combinan en la forma descrita, permiten un procesamiento eficiente de las muestras de los participantes de la población y el / o comunidadestudios basados en que normalmente no se evaluaron en un entorno clínico. Por lo tanto, este protocolo tiene el potencial para obtener muestras (y posteriormente de datos) que son más representativos de la población general.
Introducción
Múltiples estudios han caracterizado las diferencias en la expresión génica, la metilación del ADN y subconjunto de células en la sangre entre individuos con y sin Mental (u otro) enfermedades a 1-4. Estos estudios, sin embargo, se han obtenido a partir de situaciones clínicas en las que las diferencias asociadas a la enfermedad pueden ser magnificados debido a la naturaleza en general más grave de las enfermedades para las que los pacientes están buscando tratamiento. Debido a los avances en "ómicas" enfoques, la última década ha sido testigo de una explosión de interés en la obtención de muestras biológicas de ajustes epidemiológicos 5-7 comunidad y / o, con el fin de proporcionar estimaciones poblacionales de prevalencia de la enfermedad y una imagen más amplia de la determinantes ambientales de estas enfermedades mentales y / o físicos.
Un desafío clave en este sentido es el requisito para el procesamiento rápido de las muestras recogidas. La degradación de las células mononucleares, componentes del sistema inmune clave que son frequently utilizado para evaluar la salud de un individuo, comienza inmediatamente después de la extracción de sangre con una disminución significativa en la recuperación después de 2 hr de la recogida de 8-10. Para hacer frente a este desafío, presentamos un protocolo optimizado en el que varios componentes de la sangre entera humana están simultáneamente aisladas de muestras obtenidas en los hogares de los sujetos que viven en una gran área metropolitana. El protocolo se basa en nuestra recopilación y modificación de las técnicas actuales, incluido el almacenamiento de todas las fracciones "extra" en el caso de las técnicas de futuros permiten un mayor aislamiento / análisis. Mientras que los métodos o kits alternativos pueden ser empleados en lugar de los métodos individuales descritos aquí, aquellos descritos han demostrado ser un medio fiable y eficaz para el procesamiento de muestras de una manera de alto rendimiento. Fracciones de alta calidad (PBMCs, ADN, suero y ARN) de la sangre fresca se pueden producir dentro de 2 horas de recogida y todos los especímenes de ensayo listos pueden estar disponibles dentro de 2 días (Figura 1).
Este protocolo fue desarrollado para permitir el procesamiento eficiente de las muestras obtenidas de la comunidad-vivienda, los residentes adultos de la ciudad de Detroit para la prueba en el Estudio de Salud Comunitario de Detroit (DNHS; DA022720, RC1MH088283, DA022720-05-S1) una base de población, estudio de los factores sociales y biológicos del trastorno de estrés postraumático (TEPT) y otras enfermedades mentales. La prevalencia del trastorno de estrés postraumático en Detroit es más del doble del promedio de 11,12 nacional. La identificación de los determinantes biológicos del trastorno de estrés postraumático en esta población puede ayudar a desarrollar farmacológico apropiado y / o intervenciones cognitivo-conductuales para ayudar a aquellos que sufren de la enfermedad, tanto en esta población urbana, y en otras poblaciones de alto riesgo (por ejemplo, los veteranos que regresan militares). Nuestro laboratorio, anteriormente ubicada en la Universidad Estatal de Wayne en Detroit, Michigan, fue seleccionado para el procesamiento basado en nuestra experiencia en el manejo de muestras de tejidos frescos derivados de una variedadde las fuentes, la necesidad de comenzar el procesamiento de las muestras dentro de las 2 horas de la recolección, y nuestra proximidad a los lugares de recolección. Con esta oportunidad única que nos ocupa, nuestro objetivo fue optimizar el procesamiento para mayor rendimiento de ADN, ARN, suero y células mononucleares de sangre periférica (CMSP) de cada muestra (un total de N = 1.639 muestras de más de 5 oleadas de recogida de muestras). Los procedimientos descritos aquí se pueden realizar de forma simultánea en un entorno no clínico, lo que produce el material (véase la Tabla 1 para los rendimientos medios) a partir de una multitud de aplicaciones posteriores incluyendo microarrays, epigenética, en tiempo real de RT-PCR, y citometría de flujo análisis.
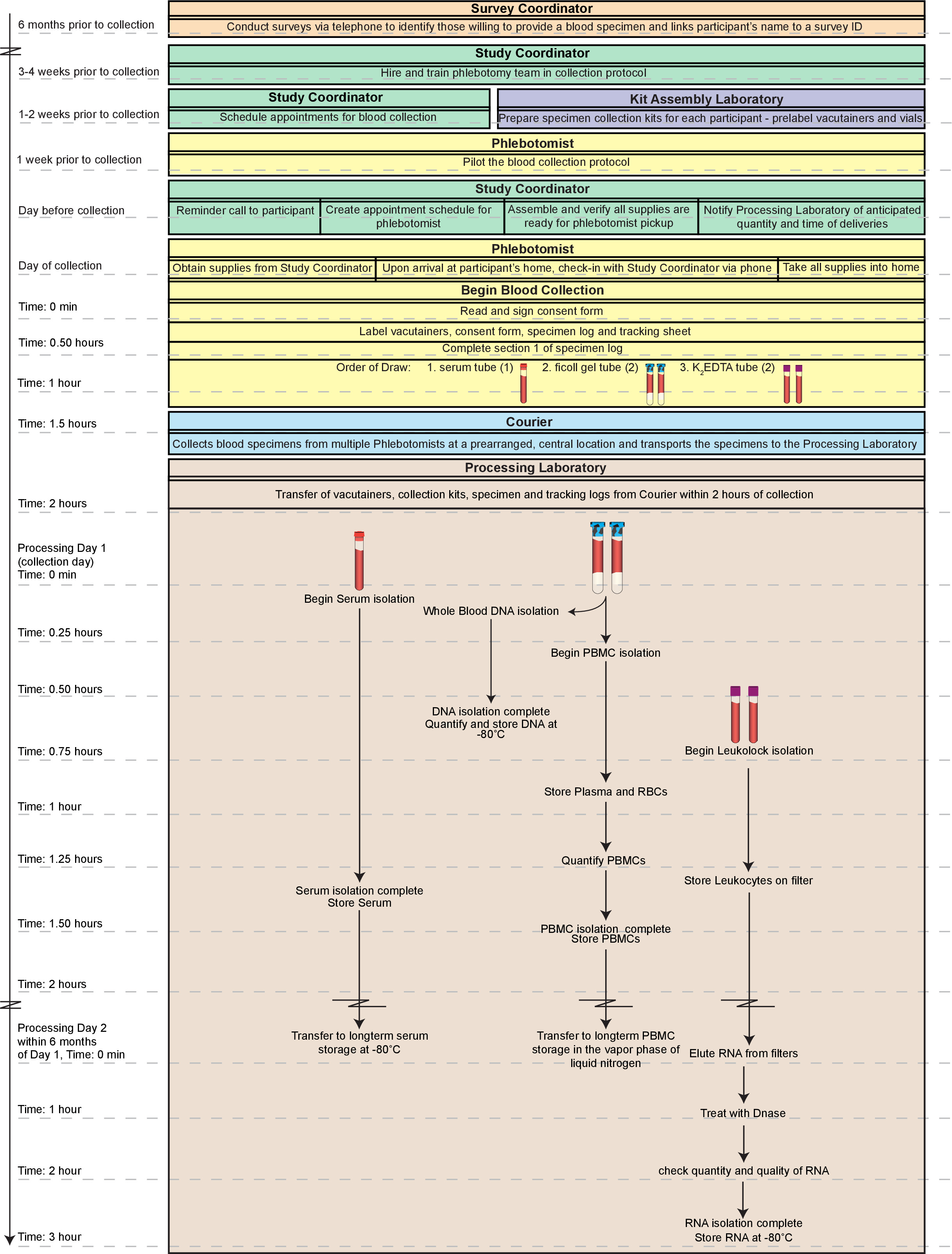
Figura 1. Flujo general de trabajo. El proceso global se muestra aquí incluye la logística de la obtención de las muestras de sangre de la identificación partic consintiendoipants a la sangre dibujan a sí mismo. De alta calidad, fracciones (células mononucleares de sangre periférica; CMSP, ADN, suero y ARN) de sangre entera fresca se puede producir dentro de 2 horas de recogida y todos los especímenes de ensayo preparada puede estar disponible dentro de 2 días. Por otra parte, las fracciones preparadas a través de este método son adecuados para el almacenamiento a largo plazo si las muestras no son para ser probado inmediatamente. Toda la línea de tiempo esbozado aquí se pudo completar en un solo día (~ 5 horas en total). Sin embargo, ese día sería muy laborioso especialmente para un solo técnico con amplia experiencia con las técnicas. Por lo tanto, se recomienda dividir los procedimientos en el Día 1 entre al menos dos técnicos y completar el procesamiento del ARN en el Día 2. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
El Estudio de Salud de Detroit Barrio fue revisado y aprobado por la Universidad de la Junta de Revisión Institucional de Michigan. Todos los participantes proporcionaron su consentimiento informado antes de su participación en el estudio.
1. Información general
- Correctamente documentar todas las etapas de la contratación a través del análisis de los datos de punto final. Lleve a cabo los protocolos en el Día 1 al mismo tiempo, el cambio de fases y solapando el tiempo lo permite. La secuencia de etapas está escrito para optimizar la eficiencia del rendimiento. La Figura 1 proporciona una visión general de todo el proceso.
Nota: El término "participante" se utiliza en todo para significar la muestra de sangre de-identificado extraída de un individuo que consienten. Los tiempos entre paréntesis a continuación indican práctica en el tiempo. Ejemplos de la hoja de seguimiento y registro de la muestra se pueden encontrar en las Tablas suplementarios S1 y S2, respectivamente. Las muestras se recogen por flebotomistas en los hogaresde los individuos identificados como participantes consienten por el coordinador del estudio y transportado al laboratorio por un mensajero (ver Información Suplementaria (SI) 1 para más detalles pre-procesamiento).
2. Día 1 Configuración
- Preparar una solución salina tamponada con fosfato 1x (PBS).
- Precaliente un bloque de calor a 56 ºC.
- Preparar la proteasa (ver SI 2) por alícuotas de 20 l en el número apropiado de tubos de microcentrífuga para las entregas del día (2 por participante).
- Asegúrese de tampones AW1 y AW2 (tampones de lavado provistos en el kit de aislamiento de ADN que aumentan la pureza del ADN) están listos para su uso, la adición de etanol al 100%, como se indica en la etiqueta, si es necesario.
- Enfriar centrífuga refrigerada a 4 ° C.
- Vuelva a colocar el tapón en la botella de 1x PBS con el tapón tapón de goma provisto y perforar el tapón de goma con una punta de transferencia, conservando el tapón de rosca en la parte superior del spi transferenciake para evitar la evaporación. Repita con la botella de solución de estabilización de ARN.
- Después de la preparación del bloque de tampones, centrífuga y el calor, documentar el momento de la entrega vacutainer en la hoja de seguimiento de la muestra (véase la Tabla SI).
3. Día 1: El procesamiento de la muestra de sangre entera por ADN, CMSP y leucocitos ARN (2 h)
- Información general
- Asignar tiempo suficiente para reducir al mínimo el tiempo transcurrido entre la extracción de sangre y la hora de inicio de procesamiento. Comenzar el procesamiento de los que contienen vacutainers de Ficoll dentro de 2 h de extracción de sangre para evitar un aumento en la contaminación de glóbulos rojos y una disminución en la recuperación de células mononucleares.
- Supongamos colección de 2 vacutainers que contienen gel de gradiente de Ficoll, 2 K 2 de ácido etilendiaminotetraacético (EDTA), y 1 vacutainers vacutainer suero por participante adulto. A la entrega de las vacutainers al laboratorio, tiene un técnico de firmar la hoja de seguimiento que significa la aceptación de las muestras. Mendigaren el procesamiento inmediatamente con la hora de inicio documentado en una base por vacutainer en el registro de espécimen.
- Serum etapa de aislamiento 1 (1 min)
- Documentar la hora de inicio en el registro de la muestra (ver Tabla 2 SI). Centrifugar (con freno y aceleración OFF) el vacutainer 7,0 ml de suero (rojo) (1 por participante) usando un rotor de cubeta oscilante con tapas de aerosol (nivel de bioseguridad 2; certificado BSL2) durante 20 min, 1.300 xg, 4 ºC.
- Aislamiento de ADN de la etapa 1 (15 min)
Nota: Para obtener más detalles sobre este procedimiento de aislamiento, ver el protocolo del fabricante 13.- Documentar la hora de inicio. En una campana de cultivo de células BSL2, invertir los vacutainers que contienen el gel de Ficoll 5 veces a continuación, añadir 200 l de sangre entera desde la parte superior de uno de los vacutainers en cada una de dos partes alícuotas de 20 l de proteasa (400 l totales por participante). Dejando a los tubos de la proteasa + microcentrífuga sangre en el capó, continúe con el centrifugation del Ficoll que contiene vacutainers en el paso 3.4.1.
Nota: Utilice el vacutainer con el volumen de recogida más grande, teniendo en cuenta la necesidad de equilibrar la centrífuga (es decir, puede ser necesario para eliminar 200 l de cada uno de los dos vacutainers) al girar los vacutainers en el paso 3.4.1. Además, para asegurar la separación adecuada durante el procesamiento de los vacutainers azules, el nivel de la sangre que queda en el vacutainer no debe ser menor de 2,5 pulgadas por encima de la capa de Ficoll.
- Documentar la hora de inicio. En una campana de cultivo de células BSL2, invertir los vacutainers que contienen el gel de Ficoll 5 veces a continuación, añadir 200 l de sangre entera desde la parte superior de uno de los vacutainers en cada una de dos partes alícuotas de 20 l de proteasa (400 l totales por participante). Dejando a los tubos de la proteasa + microcentrífuga sangre en el capó, continúe con el centrifugation del Ficoll que contiene vacutainers en el paso 3.4.1.
- Aislamiento de PBMC Etapa 1 (1 min)
- Documentar el tiempo de inicio (debe estar dentro de 2 h de extracción de sangre para evitar un aumento en la contaminación de glóbulos rojos y una disminución en la recuperación de células mononucleares) .Invert el Ficoll vacutainers que contiene 8-10 veces. Centrífuga (con freno y aceleración OFF) los vacutainers (2 por participante) que utilizan un rotor basculante con tapas de aerosol (BSL2 certificado) durante 30 min, 1600 xg, 22 ° C.
- etapa de aislamiento de ADN 2 (20 min)
- Volver a la campana y añadir 200 l de tampón AL a cada uno de los tubos de la proteasa + sangre microcentrífuga (paso 3.3.1). Cap, retirar del capó, vórtice 15 seg y flash giro.
- Incubar 56 ºC en un bloque de calor, 10 min.
- Retire los tubos del bloque de calor. Giro de Flash. Volver a la campana BSL2. Añadir 200 l de etanol 100%. Cap, retirar del capó, vórtice 15 seg y flash giro. Los pasos restantes se pueden completar exterior de la campana.
- Aplicar lisado (a menos de 30 minutos del paso 3.5.3) a una columna numérica llamada (en un tubo de recogida de 2 ml). Cierre la tapa para evitar la contaminación cruzada a través de aerosoles. Centrífuga 6000 xg, 1 min.
- Deseche el tubo de recogida que contiene el filtrado y coloque la columna de centrifugación en un nuevo tubo de recogida de 2 ml.
- Añadir 500 l Buffer AW1 a la columna sin humedecer el borde, cerrar la tapa y centrifugar 6000 xg, 1 min.
- Deseche el tubo de recogida que contiene la filtracite y coloque la columna de centrifugación en un nuevo tubo de recogida de 2 ml.
- Añadir 500 l Buffer AW2 a la columna sin humedecer el borde, cerrar la tapa y centrifugar 20.000 xg, 3 min.
- Desechar el tubo de recogida que contiene el filtrado y coloque la columna de centrifugación en un nuevo tubo de recogida de 1,5 ml (no incluido en el kit), y centrifugar 20.000 xg, 1 min.
- Deseche el tubo de recogida que contiene el filtrado y coloque la columna de centrifugación en un nuevo tubo de recogida de 1,5 ml (no incluido en el kit).
- Añadir 200 l de tampón AE o agua a cada columna y se incuba a temperatura ambiente durante 5 min. Centrífuga 6000 xg, 1 min.
- Repita el paso 3.5.11 liberadores en el mismo tubo de recogida.
- En común el ADN eluido a partir de las 2 columnas por participante. Rendimiento total ~ 800 l por participante.
- Cuantificar las muestras usando un espectrofotómetro, cuando el tiempo lo permite.
- ADN Alícuota como se desee en 2 ml crioviales, la documentación de la concentración de cada uno en el specimelog n. Transferencia crioviales a un cryobox y lugar a -80 ° C para almacenamiento a largo plazo. Documentar la hora de inicio del congelador.
- Aislamiento de ARN de leucocitos Etapa 1 (30 min)
Nota: Realice en una campana de cultivo celular certificada de Bioseguridad Nivel 2. Para más detalles sobre este aislamiento consulte las instrucciones del fabricante 15.- Perforar el tapón de goma del vacutainer K 2 EDTA con un pico de transferencia. Conserve la funda y tapón de rosca para su uso en el paso 3.6.11. Siguiendo las prácticas estándar BSL2, tenga cuidado para evitar la exposición a patógenos transmitidos por la sangre.
- Conecte el conector de engobe blanco a la parte superior de la punta de transferencia.
- Etiquetar el filtro con el ID de participante correspondiente a continuación, conecte la entrada (extremo acampanado) del filtro al conector engobe blanco.
- Adjuntar un enfundados 25⅝ G aguja a la salida (extremo cónico) del filtro. Preparación del conjunto de filtro antes de la entrega acelera considerablemente el proceso y por lo tanto,Fijación del conector de deslizamiento blanco a la punta de transferencia, añadiendo el filtro, a continuación, la aguja enfundada y estableciendo el conjunto en un bastidor de tubo de cultivo hasta su uso, se recomienda.
- Después del montaje del sistema de tubos K 2 EDTA, desenvainar de forma segura la aguja (utilizar el extremo de una espátula de metal). Apuñalar a la aguja en un 10 ml tubo de recogida de sangre evacuada vacío (tubo receptor sérico) e invertir el conjunto del tubo K 2 EDTA vacutainer / filtro / receptor. Siguiendo las prácticas estándar BSL2, tenga cuidado para evitar la exposición a patógenos transmitidos por la sangre.
- Deje que la sangre se filtre a través hasta que las secciones en forma de cuña del filtro han limpiado de sangre. Filtración tarda aproximadamente 2 min y se puede colocar en un bastidor de tubo de ensayo durante la filtración.
- Retire el filtro de la asamblea. Deje la aguja en el tubo que contiene el filtrado y deseche todo el conjunto en un contenedor de objetos punzantes.
- Conecte una jeringa de 5 ml a la punta de transferencia en la botella de 1x PBS, envert la botella, y retirar 3 ml.
- Coloque la jeringa con PBS a la entrada del filtro y lavar el filtro (3-5 gotas por segundo). Recoger el flujo a través de un contenedor de residuos biológico. Desconecte la jeringa del filtro sin retraer el émbolo.
- Después de la filtración y un lavado de PBS, 3 ml de retirar el agente de estabilización de ARN utilizando una nueva jeringa de 5 ml y el método descrito en el paso 3.6.8. Limpie el filtro como en el paso 3.6.9. El agente de estabilización de ARN debe permanecer en el filtro. Desconecte la jeringa del filtro sin retraer el émbolo.
- Selle la entrada del filtro y la salida con la tapa de la funda y tornillo retenido desde la punta de transferencia dejando el filtro saturado con el agente de estabilización de ARN. El filtro puede ser almacenado en este punto. Guarde el filtro a -80 ºC hasta que el tiempo lo permite (~ 2 horas) para completar los pasos 5.3 a 5.4.7.
Nota: Los Filtros almacenadas a -80 ºC hasta por un año después de la recolección se han procesado sin decrease en la calidad del ARN.
- Aislamiento de PBMC Etapa 2 (30 min)
- Retire los que contienen vacutainers Ficoll de la centrífuga (paso 3.4.1) y continuar en una campana BSL2. Vacutainers deben mostrar la separación como en la figura 2. Si no es así, consulte la Tabla 2.
- Una vez que el vacutainer se devuelve a la campana BSL2, retire el tapón y retirar 1,5 ml de la parte superior, de color amarillento, capa de plasma (Figura 2) utilizando una pipeta serológica sin llegar cerca de la capa mononuclear (claro / blanco). Transferir el plasma a un 5 ml criovial - (piscina de 2 vacutainers - 1 participante). Registrar el volumen recogido. Vea el paso 3.7.6 para obtener instrucciones de almacenamiento.
Nota: La mejor separación y mayores rendimientos PBMC provienen de participantes que han ayunado por lo menos 12 horas antes de la recogida de sangre. - Transferir el plasma restante y la capa de color blanquecino, mononuclear (todo por encima de la capa de gel - la figura 2) ucantar una pipeta serológica, a un tubo cónico de 15 ml, puesta en común de la capa mononuclear de cada uno de los dos Ficoll que contiene vacutainers por participante en un tubo cónico.
- Añadir 1x PBS para llevar el volumen total en el tubo cónico de 15 ml. Tubo de tapa e invierta 5 veces. Centrífuga (con freno y aceleración OFF) 15 min, 300 xg, 22 ° C.
- Vuelta a la que contienen vacutainers de Ficoll en el capó y recoger las células rojas de la sangre (glóbulos rojos) mediante el uso de un "pipeta Pasteur 5¾ a girar alrededor y aflojar el exterior de la capa de gel de Ficoll y quitarlo si es posible. Usar una pipeta serológica para recoger y transferir los RBC (~ 4,5 ml) a una 5 ml criovial. Registrar el volumen recogido.
- Transferencia ambos 5 ml crioviales (plasma en el paso 3.7.2 y glóbulos rojos en el paso 3.7.5) a un recipiente de congelación velocidad controlada y poner a -80 ºC durante al menos 24 horas después de lo cual se pueden transferir a un cryobox y volvieron a -80 ºC para el almacenamiento a largo plazo.
- Devuelva la cotubo técnica a la campana, cuando la centrifugación en el paso 3.7.4 es completa, y aspirar todo pero ~ 500 l de la PBS sin perturbar el sedimento. PBMC rendimiento es mayor si ~ 200 l de PBS se deja encima de la pastilla en esta etapa.
- Añadir 1x PBS fresco para llevar el volumen a 10 ml. Resuspender el precipitado suavemente. Tubo de tapa e invierta 5 veces. Centrífuga (con freno y aceleración OFF) 10 min, 300 xg, 22 ° C.
- Serum etapa de aislamiento 2 (10 min)
Nota: Realice en una campana BSL2.- Alícuota la capa de suero superior de la vacutainer suero, después de la centrifugación (paso 3.2.1), en 2 ml crioviales lo deseas. Rendimiento típico es de 2,5 ml (Tabla 1). Entrar el volumen. Por ejemplo, usar 4 crioviales alícuotas de 200 l en criovial 1, 1.000 l en criovial 2 y luego dividir el restante en crioviales 3 y 4.
- Transfiera los crioviales a un cryobox y lugar a -80 ºC para el almacenamiento a largo plazo. Documentar el inicio congeladorhora.
- Aislamiento de PBMC Etapa 3 (15 min)
- Volver a la campana, después de la centrifugación (paso 3.7.8), y aspirar tanto sobrenadante / PBS como sea posible y sin perturbar el sedimento. Resuspender pellet con la pipeta de 2.5 ml PBMC Congelación Medio 1 (ver SI 2).
- Añadir 2,5 ml de PBMC Freezing Medium 2 (ver SI 2) para la solución de células / medio en el paso 3.9.1. Vortex suavemente.
- Alícuota de 10 l de la solución celular en un tubo de microcentrífuga de 0,65 ml (dilución adicional puede ser necesaria). Añadir 10 l de 0,4% mancha azul tripán en el tubo de microcentrífuga de 0,65 ml y mezclar con la pipeta varias veces. Para más detalles, véase el manual del fabricante 16.
- Pipetear 10 l de la mezcla en una célula contando diapositiva cámara y el lugar diapositiva en el contador de células dentro de 3 min de la mezcla. Acercar y enfocar las células. Pulse el botón "contar las células" para obtener el recuento PBMC.
- Alícuota, si la viabilidadNúmero PBMC está por encima de 3 millones de células por mililitro (mc / ml), según se desee en crioviales y continúe con el paso 3.9.9. PBMCs Almacenar en un máximo de 5 crioviales a una concentración de al menos 3 mc / ml cada una.
- Si el número de PBMC viable está por debajo de 3 mc / ml, calcular el número total de células multiplicando el viable mc / ml por 5 ml. Divida ese número por 4, 3, 2 o 1 ml por lo que habrá al menos un tubo con 3 mc / ml.
- Centrifugar el tubo cónico que contiene las células / solución del medio de congelación durante 5 min a 300 xg (OFF de freno y aceleración). Después de la centrifugación, aspirar el volumen apropiado de medio de congelación (sobrenadante), dejando el volumen calculado anteriormente (4, 3, 2 o 1 ml).
- Resuspender el precipitado en el sobrenadante restante y parte alícuota de al menos 3 mc / ml en el número apropiado de crioviales (1-4) a 1 ml / criovial. Medio de congelación final es de 10% sulfóxido de dimetilo (DMSO) / 20% suero bovino fetal (FBS) / 70% Roswell Park Memorial Institute (RPMI) 1.640.
- Document el recuento de células por criovial. La transferencia de los crioviales a un recipiente de congelación velocidad controlada y poner a -80 ºC durante al menos 24 horas después de cuyo tiempo los crioviales pueden ser transferidos a un cryobox y poner en un tanque de nitrógeno líquido (fase de vapor) para almacenamiento a largo plazo. Congelador Documento hora de inicio.
4. Día 2 Configuración
- Calentar una alícuota (220 l por filtro) de nucleasa libre de EDTA 0,1 mM en un tubo libre de nucleasa a 80 ° C en un bloque de calor.
- Preparar soluciones de lavado 1 y 2 (ver SI 2).
5. Día 2: a largo plazo de almacenamiento de muestras y procesamiento de filtro de leucocitos (3 horas)
- Información general.
- Realice este procedimiento dentro de los 6 meses de la aplicación de los leucocitos al filtro.
Nota: En general, el día 2 de proceso toma alrededor de 3 horas y si bien tiene la etiqueta "Día 2", el requisito clave es que la parte de procesamiento de filtro se debe realizar en un día en queno hay día 1 de procesamiento se produce en el laboratorio.
- Realice este procedimiento dentro de los 6 meses de la aplicación de los leucocitos al filtro.
- Almacenamiento de muestras a largo plazo (15 min)
- Realice este procedimiento todos los días, antes de la entrega de nuevos vacutainers al laboratorio para el día 1 de procesamiento. Transfiera los crioviales que se almacenaron O / N en recipientes de congelación velocidad controlada en el Día 1 en criocajas debidamente etiquetados y devolverlos a -80 ºC o nitrógeno líquido (fase de vapor) para almacenamiento a largo plazo. Organizar los crioviales en función del tipo de muestra (por ejemplo, ADN, CMSP, glóbulos rojos, etc.) para acelerar la ubicación de la muestra de seguimiento para los ensayos de punto final.
- Leucocitos de ARN filtros de procesamiento (45 min).
Nota: Para obtener más información sobre este procedimiento consulte las instrucciones del fabricante 17.- Llevar el filtro a RT (descongelar sobre 5 min).
- Retire la funda y tapón de rosca del filtro. Retraer el émbolo de una jeringa de 5 ml y conectarlo a la entrada (extremo abocinado) del filtro, presione el émbolopara expulsar el agente de estabilización de ARN desde los puertos de filtro en un contenedor de residuos biológico.
- Cargar una nueva jeringa de 5 ml con 4 ml de una solución de isotiocianato de fenol y guanidina para el aislamiento de ARN y adjuntarlo a la entrada del filtro, presione el émbolo para vaciar la solución a través del filtro, recogiendo el lisado en un tubo de 15 ml cónico marcado (2 por participante).
- Desconecte la jeringa del filtro, retraer el émbolo, vuelva a adjuntarlo al filtro, y presione el émbolo para expulsar la muestra residual atrapado en el disco de filtro en los mismos 15 ml tubo cónico. Deseche el filtro y la jeringa.
- Añadir 800 l BCP al tubo cónico, cerrar el tubo herméticamente y agitar vigorosamente la preparación durante 30 segundos.
- Incubar a temperatura ambiente durante 5 min. Centrifugar durante 10 min a 2000 x g.
- Transferir la fase acuosa (superior) a un tubo recién marcado 15 ml cónico (~ 2,5 ml).
- Añadir 0,5 veces el volumen de la fase acuosa de un agua libre de nucleasad mezclar bien. A continuación, agregue 1.25x el volumen acuosa de etanol al 100% y mezclar de nuevo.
Nota: Para un volumen acuoso 2,5 ml, añadir 1,25 ml de agua libre de nucleasa, mezclar, a continuación, añadir 4,7 ml de etanol al 100% (2,5 ml x 0,5 = 1,25 ml nucleasa agua libre ENTONCES 2,5 ml + 1,25 ml = 3,75 ml nuevo volumen acuoso ENTONCES 3,75 ml x 1,25 = 4,7 ml 100% de etanol). Este paso permite el aislamiento de ARN total que incluye la fracción de ARN pequeño. Un método para omitir los pequeños RNAs del aislamiento se puede encontrar en el manual 17. - Retire el émbolo de una jeringa de 5 ml y se inserte un cartucho de giro en su lugar. Coloque el conjunto del cartucho / jeringa para un colector de vacío. Para una conexión más segura al colector de vacío, colocar el conjunto de cartucho / jeringa dentro de un tubo de microcentrífuga de 1,5 ml con la parte inferior cortada.
- Aplicar la muestra de la etapa 5.3.8 al cartucho giro lentamente con el vacío, añadiendo cuidadosamente más muestra a medida que se tira a través del cartucho.
Nota: Si no hay vacío esActualmente, ver método de centrifugación a http://www.ambion.com/techlib/misc/leuko_iso.pdf. - Transferir el cartucho de giro a un tubo de microcentrífuga de 1,5 ml y añadir 750 l de Lavado 1. Centrifugar el cartucho de giro / conjunto de tubo de 5 a 10 segundos a 12.000 x g.
- Desechar filtrado del tubo y devolver el cartucho de giro a la misma tubo de microcentrífuga.
- Añadir 750 l de Wash 2 y centrifugar el cartucho de giro / tubo de 5-10 segundos a 12.000 x g. Deseche filtrado como en el paso 5.3.12. Devuelva el cartucho de vuelta para el mismo tubo de microcentrífuga.
- Repita con otros 750 l de Wash 2 y centrifugación como en el paso 5.3.13. Filtrado Descartar. Devuelva el cartucho de vuelta para el mismo tubo de microcentrífuga. Centrifugar el cartucho de giro / tubo a velocidad máxima durante 1 min para secar el filtro.
- Transferir el cartucho de giro a un nuevo máximo, marcado 1,5 l tubo de microcentrífuga.
- Añadir 200 l de EDTA mM libre de nucleasa 0,1 (precalentado a 80 & #176; C) hasta el centro del filtro de cartucho de giro (2 por participante). Incubar a temperatura ambiente durante 1 min.
- Centrifugar durante 1 min a 12.000 xg para eluir el ARN. Conservar el filtrado. Deseche el cartucho de centrifugado.
- Dividir cada filtrado 200 l en dos, 100 alícuotas en tubos de 1,5 ml de microcentrífuga frescas, etiqueta y mantener en hielo. En este punto, a partir de 2 filtros por participante producirá cuatro alícuotas de 100 l de RNA por participante.
- Tratamiento DNasa (1 hora)
Nota: Para más detalles sobre este tratamiento ver el protocolo del fabricante 18.- Crear una mezcla maestra mediante la combinación de 10 l de DNasa I Buffer y 2 l rDNase yo por alícuota + 1 (a la cuenta para el error pipeteo).
Nota: Para cuatro muestras, 100 alícuotas utilizan 50 l DNasa I Buffer + 10 l rDNase I. - Alícuota de 12 l de la mezcla maestra en el paso 5.4.1 en cada una de las alícuotas de ARN a partir del paso 3.5.18 y mezclar suavemente. Incubar a 37° C durante 30 min en un bloque de calor.
- Vortex la DNasa inactivación reactivo y añadir 11,2 l de cada alícuota, mezcle bien. Incubar a temperatura ambiente durante 2 min vórtex 2-3 veces durante la incubación.
- Se centrifuga a 10.000 xg durante 1,5 min.
- Transferir el sobrenadante (ARN) a un tubo de 1,5 ml frescos. Las alícuotas del mismo participante pueden agruparse aquí.
- Ejecutar cada muestra en un espectrofotómetro para obtener la concentración. Se recomienda un análisis de la calidad del ARN usando un Bioanalyzer (véase el paso 5.5).
- Alícuota de la ARN en crioviales lo deseas. Si un análisis Bioanalyzer no se realizará inmediatamente, alícuota de 1,5 l de muestra en un tubo de microcentrífuga para su posterior análisis. Documentar las concentraciones y la hora de inicio del congelador. Almacene las muestras a -80 ° C.
- Crear una mezcla maestra mediante la combinación de 10 l de DNasa I Buffer y 2 l rDNase yo por alícuota + 1 (a la cuenta para el error pipeteo).
- Análisis Bioanalyzer (1 hora)
- Siga el protocolo del fabricante 19. Cuando la carrera se ha completado, los datos se almacenan de forma automática, pero guardarlo nuevamente won una, nombre de archivo más reconocibles menor. Resultados esperados (Figura 3): la escalera deben tener 6 picos, las muestras deben tener 3 picos (2 picos ribosomal en 44 segundos y 50 segundos, respectivamente, y 1 pico marcador temprano a 25 seg).
Resultados
Es esencial que el procedimiento global producir material de alta calidad para el análisis en una multitud de aplicaciones posteriores, incluyendo la expresión de genes a través de microarray y análisis RT-PCR, la detección de modificaciones epigenéticas, y las variaciones subconjunto de células. La Tabla 1 indica el rendimiento medio y la calidad de los materiales de cada uno de los procesos. La Figura 3 proporciona un ejemplo de la salida de calidad de los métodos de procesamiento de aislamiento de ARN de leucocitos y el filtro. La imagen en la parte superior izquierda de la figura 3 es la imagen de gel resultante de la electroforesis capilar. Cada carril debe producir dos bandas distintas con sombreado mínimo lo que indicaría la degradación. Los cromatogramas por debajo del gel proporcionan un aspecto adicional en el nivel y tipo de degradación que puede ser determinado en base a la localización y el tamaño de los picos. El Número de la integridad del ARN (RIN) es otra medida de la calidad que va desde 1 (bajo; degradado) a10 (alto, puro, bueno ARN de calidad).
Dicha metodología de alto rendimiento se presta a un error de vez en cuando, pero hay varios puestos de control a lo largo de las diferentes etapas de procesamiento para garantizar el control de calidad. Figura 2 muestra la separación adecuada después de la centrifugación de la que contiene vacutainer Ficoll. Hay varias razones un vacutainer no puede mostrar esta separación que incluye un error en la velocidad de centrifugado, bajo volumen de recogida, o la falta de un participante en ayunas como se indica en la Tabla 2.
Subconjuntos (n = 100) de los ejemplares aislados mediante este procedimiento se han analizado con éxito por HumanMethylation27 (HM27) BeadChips análisis de ADN, incluyendo la validación de los resultados por pirosecuenciación 11. Genotipado, dirigido pirosecuenciación bisulfito, y en tiempo real de RT-PCR se ha realizado con éxito en el Wildman y Uddin laboratorios 20,21,22,23. Suero, aislado en el parallel con el aislamiento de ADN, tanto para el BeadChip y genotipado análisis, se analizó con éxito para la IL-6 y C-reactiva actividad de la proteína 24. Subconjuntos de células T a partir de las PBMC aisladas se han analizado con éxito por citometría de flujo 25-28 de. Además, el ARN a partir del procedimiento de leucocitos modificado ha sido sometido a todo el genoma perfiles de expresión génica 29.
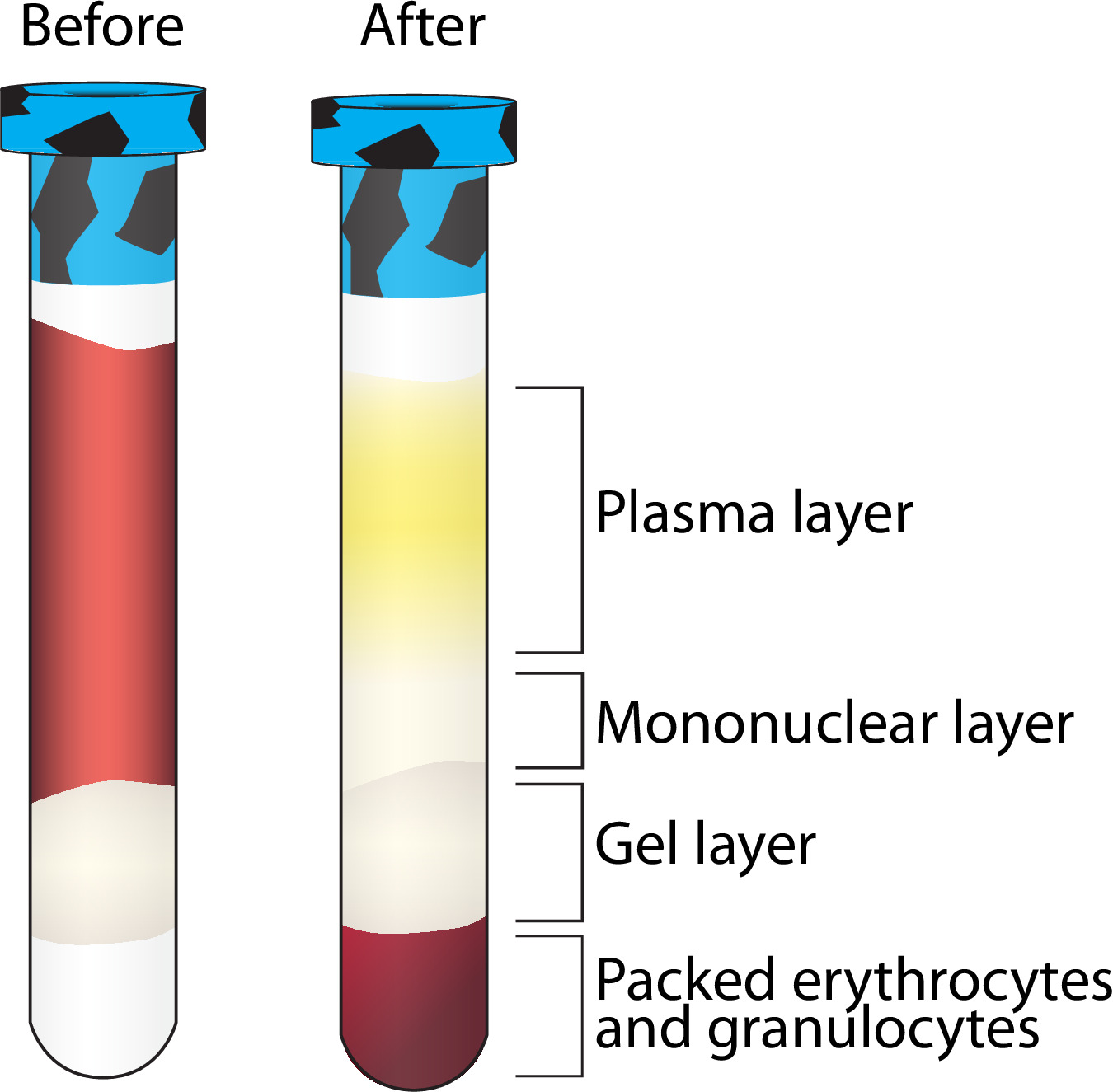
Figura 2. separación de la capa después de la centrifugación de Ficoll que contiene vacutainer. Una visualización de la separación de múltiples componentes de la sangre después de la centrifugación. La capa de mononucleares se purifica mientras que las otras capas se almacenan a -80 ° C. Por favor haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Resultados esperados de procesamiento del ARN de leucocitos. Bioanalyzer resultados que muestran dos bandas distintas que representan 18S y 28S ARN ribosomal con los números de integridad del ARN (RINs) por encima de 8 aislados con los métodos de leucocitos se describe el aislamiento de ARN y el filtro de procesamiento (véase el protocolo 3.6 y 5.3). Haga clic aquí para ver una versión más grande de esta figura.
| Total Rentabilidad Media (N≈500) | Calidad media | |
| Suero | 2,30 ml | N / A |
| ADN a partir de sangre entera | 39.97 g | Absorbancia a 260/280 = 1,74 |
| PBMC | 22.25 millones de células viables | Al menos 95% de viabilidad de aislamiento general |
| RNA a partir de leucocitos | 44.09 g | La absorbancia a 260/280 = 2,01 Número integridad del ARN (RIN) = 6,48 |
Tabla 1. esperados rendimientos. Cantidad promedio y la calidad de las muestras procesadas usando los métodos descritos.
| Problema | Posible solución |
| Sin separación después de Ficoll contiene centrifugación vacutainer | Vacutainer no está lleno a capacidad - el volumen mínimo de recogida para el procesamiento adecuado es de 6 ml. |
| Compruebe la configuración de centrifugadoras, asegúrese de que la velocidad es en fuerza g. Si no es correcta, ajuste de fuerza-g y volver a girar. | |
| Co bajo CMSPunt | El volumen de la sangre dibuja demasiado bajo. |
| Aspirado demasiado cerca del pellet en el paso 3.7.8 | |
| Participante no ayuno. | |
| Sin separación después de la centrifugación vacutainer suero | Compruebe la configuración de centrifugadoras, asegúrese de que la velocidad es en fuerza g. Si no es correcta, ajuste de fuerza-g y volver a girar. |
| Concentración inesperado utilizando el Nanodrop 1000 | Asegúrese de que la medida es para el tipo apropiado de la muestra (por ejemplo, ADN o ARN). |
Tabla 2. Solución de problemas. Los problemas más comunes encontrados con los métodos descritos y las posibles soluciones.

Hoja de seguimiento de la Tabla 1. Complementario. Un ejemplo de seguimiento de los vacutainers from extracción de sangre para la entrega de laboratorio. Por favor haga clic aquí para ver esta tabla.
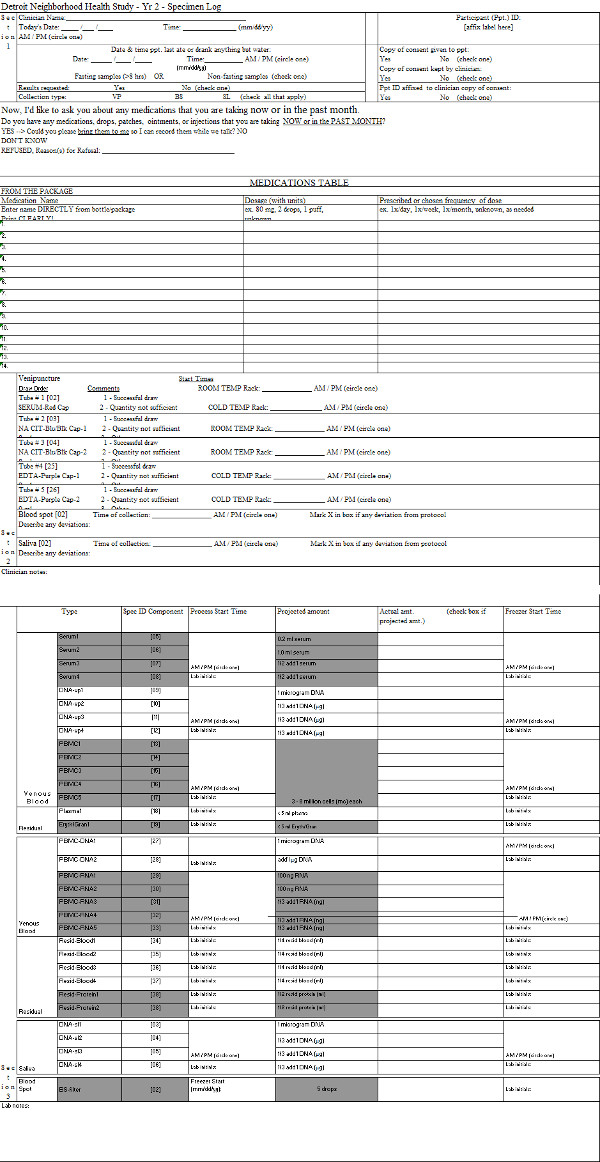
Que complementa el cuadro 2. registro de muestras. Un ejemplo del documento utilizado para documentar el proceso de los vacutainers momento de la entrega al laboratorio. Haga clic aquí para ver esta tabla.

Suplementaria Cuadro 3. Sistema de Numeración criovial. Un ejemplo del sistema de numeración utilizado para cada participante. Por favor haga clic aquí para ver esta tabla.
Información complementaria 1. Datos de preprocesamiento. Detalla las funciones del Coordinador de Estudio, Courier y Flebotomista. Por favor haga clic aquí para ver información complementaria 1.

Información complementaria 2. Recetas. Una lista de recetas para los reactivos necesarios. Haga clic aquí para ver información complementaria 2.
Discusión
Hemos descrito un protocolo simplificado que se ha aplicado con éxito para procesar más de 1.600 muestras de sangre entera en el Estudio de Salud de Detroit Barrio. Aunque muchas de estas técnicas están disponibles en la literatura existente, nuestra compilación paso a paso, incluidas las modificaciones precisamente cronometrados entre cada paso, refleja una optimizado, protocolo eficiente que produce con éxito una variedad de muestras biológicas con una amplia gama de aplicaciones posteriores, incluyendo la metilación del ADN, la expresión de ARNm y análisis inmunológico. Estas muestras ya han sido probados en una variedad de experimentos, cuyos resultados han sido publicados en la literatura revisada por pares y / o presentados en reuniones nacionales 11,20,21,23,24,29. Así, este protocolo debe ser de interés para otros investigadores que tratan de recoger muestras biológicas en estudios poblacionales similares a la DNHS.
Al procesar las muestras en un alto throughp talesforma ut, es de suma importancia para mantener registros precisos en todas las etapas. Se recomienda el desarrollo de una base de datos para almacenar toda la información criovial al principio. Esta base de datos debe incluir todos los aspectos de la muestra dentro de cada criovial incluyendo el volumen, la concentración, la calidad, código de barras, y la ubicación de almacenamiento (número de caja de almacenamiento y la ubicación dentro de la caja). Se recomienda preparar crioviales pre-etiquetados y cajas de almacenamiento que contienen un código de barras. Además, hemos encontrado que es conveniente tener un documento "maestro" que contiene los códigos de barras para cada tubo, específicos para cada participante (Tabla S3). Esto permite la entrada rápida de los datos criovial en la base de datos sin una manipulación excesiva de las muestras, la introducción de los ciclos de congelación / descongelación innecesarias a las muestras y elimina la posibilidad de error humano en entrar en los códigos de barras manualmente. También es esencial que las etiquetas se adhieran a extremo (por ejemplo, fase de vapor de nitrógeno líquido a -178 -150ºC) temperaturas. La documentación puede llevar mucho tiempo, y con la necesidad de un procesamiento eficiente en la entrega, hemos encontrado que tienen al menos dos técnicos dividen los procesos produce la mayor eficiencia.
Una limitación de este método es la proximidad del laboratorio al sitio de recolección. Para el aislamiento de PBMC, en particular, las muestras deben ser procesadas dentro de 2 horas de la recolección para evitar un aumento significativo en la contaminación de glóbulos rojos y disminuye en las células mononucleares viables. Como tal, el laboratorio debe ser no más de 30 minutos de un punto de recogida para dar cuenta de los problemas de tráfico relacionados que puedan surgir. Además, cualquier laboratorio que propone para llevar a cabo el protocolo descrito aquí requerirá dos técnicos en la mano para asegurar un procesamiento óptimo de la muestra. Además, cada laboratorio debe tener acceso a los equipos necesarios para cada paso descrito anteriormente. Por lo tanto, los laboratorios con menos personal y / o equipo limitado wOuld probable que no pueda llevar a cabo este protocolo.
Una ventaja de este protocolo es la capacidad de recoger muestras directamente en las casas de las personas que consienten. Esto permite que el estudio para llegar a las personas con problemas de salud mental o de otro tipo que no se suelen buscar ayuda médica, tal vez debido a la falta de seguro o el transporte. También la permite la comparación de los individuos afectados y no afectados que viven en la misma comunidad que han experimentado los disparadores similares, pero difieren en cuanto a sus síntomas de salud mental. La obtención de especímenes de esta manera requiere una sincronización precisa y la coordinación de flebotomistas en el campo. Debido a que nuestro laboratorio se encuentra dentro de la comunidad que estábamos estudiando, un flebotomista normalmente podría recoger muestras de dos de tres casas y entregar las muestras al laboratorio dentro de la ventana de 2 horas de la primera colección. Eficiencia es clave para nuestro procesamiento de la muestra, y como tal, hemos tenido múltiples flebotomistas en elcampo, el aumento de la necesidad de una coordinación precisa. El laboratorio recibió múltiples entregas de sangre, con un máximo de ocho participantes por la entrega espaciados 2 horas de diferencia. Los flebotomistas se reunieron en un lugar designado y se combinan sus colecciones con sólo un flebotomista entregar las muestras al laboratorio como un solo lote. Los métodos descritos aquí se pueden adaptar fácilmente para estudiar muchos otros fenotipos y los especímenes biológicos recogidos se pueden utilizar en una multitud de ensayos de aguas abajo.
Divulgaciones
Los autores declaran que no tienen intereses en conflicto.
Agradecimientos
We would like to thank Henriette Mair-Meijers for invaluable attention to detail and hours devoted to processing the blood collections. We are grateful for the graphic design expertise of Natalie Jameson Kiesling. We also appreciate the approval of the manufacturers (Qiagen (Valencia, CA), BD Biosciences (San Jose, CA), Life Technologies (Grand Island, NY)) mentioned herein to publish the use of their products as described. Funding for this work was generously provided by the National Institutes of Health award numbers DA022720, RC1MH088283, and DA022720-05-S1.
Materiales
| Name | Company | Catalog Number | Comments |
| QIAamp DNA Blood Mini Kit | Qiagen | 51104 | Day 1: DNA isolation |
| Phosphate-buffered saline (PBS) | Sigma | P5493-1L | Day 1: PBMC isolation |
| 5 ¾” Pasteur pipets | Fisher | 13-678-6A | Day 1: PBMC isolation |
| Fetal Bovine Serum (FBS), heat inactivated | Life Technologies | 10082147 | Day 1: PBMC isolation |
| Dimethyl Sulfoxide (DMSO) | Sigma | D8418-500ml | Day 1: PBMC isolation |
| RPMI Medium 1640, liquid | Invitrogen | 11875119 | Day 1: PBMC isolation |
| 0.4% trypan blue stain | Invitrogen | T10282 | Day 1: PBMC isolation |
| Countess Cell Counting Chamber | Invitrogen | C10283 | Day 1: PBMC isolation |
| Countess Automated Cell Counter or cell counting device such as a microscope and hemocytometer | Invitrogen | C10281 | Day 1: PBMC isolation |
| LeukoLOCK Fractionation & Stabilization Kit | Ambion | 1933 | Day 1: Leukocyte RNA isolation |
| 25 G x 5/8 in. needles | Becton Dickinson | 305122 | Day 1: Leukocyte RNA isolation |
| Syringes (5 ml) | Becton Dickinson | 309646 | Days 1 and 2: Leukocyte RNA isolation |
| Denaturing Lysis Solution | Ambion | 8540G | Day 2: Leukocyte RNA isolation |
| 5 M NaCl | Life Technologies | 24740011 | Day 2: Leukocyte RNA isolation |
| TRI Reagent | Ambion | 9738 | Day 2: Leukocyte RNA isolation |
| Bromo-3-chloro-propane (BCP) | Sigma | B-9673 | Day 2: Leukocyte RNA isolation |
| spin cartridges | Ambion | 10051G | Day 2: Leukocyte RNA isolation |
| 0.1 mM EDTA | Ambion | 9912 | Day 2: Leukocyte RNA isolation |
| DNA-free Kit | Ambion | AM1960 | Day 2: DNase treament |
| RNA 6000 Ladder | Agilent | 5067-1529 | Day 2: Bioanalyzer analysis |
| RNA 6000 Nano Series II Kit | Agilent | 5067-1511 | Day 2: Bioanalyzer analysis |
| RNaseZAP | Ambion | AM8782 | Day 2: Bioanalyzer analysis |
| Ethanol >99% | Sigma | E7023-500ml | |
| Isopropanol >99% | Sigma | I9516-500ml | |
| Nuclease-free ultra pure water | Invitrogen | 9938 | |
| Pipette tips (nuclease-free) | Eppendorf | 22491253 | |
| Pipetter (serological) | Eppendorf | 2223020-4 | |
| Pipetters (for volumes under 1 ml) | Eppendorf | 3120000-054 | |
| Pipettes (serological) | Fisher | 13-678-27E | |
| Controlled rate freezing containers | Nalgene | 5100-0001 | |
| Cryoboxes (to hold 2 ml and 5 ml cryovials and 1.5 ml microcentrifuge tubes) | Fisher | 03-395-464 | |
| Test tube rack | Thermo Scientific | 14-804-134 | |
| 15 ml polypropylene tubes | Fisher | 14-959-49D | |
| 1.5 ml and 0.65 microcentrifuge tubes | Fisher | 07-200-534 and 07-200-185 | |
| 2 ml and 5 ml cryovials | Fisher | 10-500-26 and 10-269-88F | |
| 8 ml CPT vacutainer | BD Biosciences | 362761 | 2 tubes |
| 6 ml K2 EDTA vacutainer | BD Biosciences | 367863 | 2 tubes |
| 8.5 ml SST vacutainer | BD Biosciences | 367988 | 1 tube |
| Vortexer | Fisher | 2215365 | |
| Dry bath incubator with heating block for microcentrifuge tubes | Fisher | 11-715-1250 | |
| Filtration/vacuum system for use within the cell culture hood | Fisher | 01-257-87 | |
| Fixed-angle rotor for microcentrifuge tubes with aerosol-tight lid | Eppendorf | 22637002 | |
| Refrigerated centrifuge with a swing-bucket rotor and aerosol-tight caps for 16 x 125 mm vacutainers and 15 ml polypropylene tubes | Eppendorf | 22628157 | 2, one does not need to be refrigerated |
| Nanodrop 2000 (recommended for accuracy of small volumes) or other spectrophotometric device | Fisher | 13-400-411 | |
| Agilent Bioanalyzer | Agilent Technologies | G2940CA | |
| Liquid nitrogen tank | Thermo Scientific | 11-676-56 | |
| Sharps container | Fisher | 22-037-970 | |
| Biological waste container | Thermo Scientific | 1223P52 | |
| Biosafety Level 2 certified cell culture hood | Thermo Scientific | 13-261-315 |
Referencias
- Hernandez, M. E., Martinez-Fong, D., Perez-Tapia, M., Estrada-Garcia, I., Estrada-Parra, S., Pavon, L. Evaluation of the effect of selective serotonin-reuptake inhibitors on lymphocyte subsets in patients with a major depressive disorder. Eur Neuropsychopharmacol. 20, 88-95 (2010).
- Weigelt, K., et al. TREM-1 and DAP12 expression in monocytes of patients with severe psychiatric disorders EGR3, ATF3 and PU.1 as important transcription factors. Brain Behav Immun. 25, 1162-1169 (2011).
- Robertson, M. J., et al. Lymphocyte subset differences in patients with chronic fatigue syndrome, multiple sclerosis and major depression. Clin Exp Immunol. 141, 326-332 (2005).
- Rotter, A., Asemann, R., Decker, A., Kornhuber, J., Biermann, T. Orexin expression and promoter-methylation in peripheral blood of patients suffering from major depressive disorder. J Affect Disord. 131, 186-192 (2011).
- Klengel, T., et al. Allele-specific FKBP5 DNA demethylation mediates gene-childhood trauma interactions. Nat Neurosci. 16, 33-41 (2013).
- Segman, R. H., Shefi, N., Goltser-Dubner, T., Friedman, N., Kaminski, N., Shalev, A. Y. Peripheral blood mononuclear cell gene expression profiles identify emergent post-traumatic stress disorder among trauma survivors. Mol Psychiatry. 10, 500-513 (2005).
- Smith, B. H., et al. Cohort Profile: Generation Scotland: Scottish Family Health Study (GS:SFHS). The study, its participants and their potential for genetic research on health and illness. Int J Epidemiol. 42, 689-700 (2013).
- Mallone, R., et al. Isolation and preservation of peripheral blood mononuclear cells for analysis of islet antigen-reactive T cell responses: position statement of the T-Cell Workshop Committee of the Immunology of Diabetes Society. Clin Exp Immunol. 163, 33-49 (2011).
- Duvigneau, J. C., Hartl, R. T., Teinfalt, M., Gemeiner, M. Delay in processing porcine whole blood affects cytokine expression. J Immunol Methods. 272, 11-21 (2003).
- Debey, S., et al. Comparison of different isolation techniques prior gene expression profiling of blood derived cells: impact on physiological responses, on overall expression and the role of different cell types. Pharmacogenomics J. 4, 193-207 (2004).
- Uddin, M., et al. Epigenetic and immune function profiles associated with posttraumatic stress disorder. Proc Natl Acad Sci U S A. 107, 9470-9475 (2010).
- Kessler, R. C., Wang, P. S. The descriptive epidemiology of commonly occurring mental disorders in the United States. Annu Rev Public Health. 29, 115-129 (2008).
- . QIAamp® DNA Mini and Blood Mini Handbook Available from: https://www.qiagen.com/us/resources/resourcedetail?id=67893a91-946f-49b5-8033-394fa5d752ea (2010)
- Koenen, K. C., et al. SLC6A4 methylation modifies the effect of the number of traumatic events on risk for posttraumatic stress disorder. Depress Anxiety. 28, 639-647 (2011).
- Walsh, K., Uddin, M., Soliven, R., Wildman, D. E., Bradley, B. Associations between the SS variant of 5-HTTLPR and PTSD among adults with histories of childhood emotional abuse: Results from two African American independent samples. J Affect Disord. 161, 91-96 (2014).
- Bustamante, A. C., et al. Childhood maltreatment is associated with epigenetic differences in hypothalamic-pituitary-adrenal (HPA) axis genes in the Detroit Neighborhood Health Study. , (2013).
- Sipahi, L., et al. Longitudinal epigenetic variation of DNA methyltransferase genes is associated with vulnerability to post-traumatic stress disorder. Psychol Med. 44, 3165-3179 (2014).
- Uddin, M., Koenen, K. C., Aiello, A. E., Wildman, D., de los Santos, R., Galea, S. Epigenetic and inflammatory marker profiles associated with depression in a community-based epidemiologic sample. Psychol Med. 41, 997-1007 (2011).
- Uddin, M., et al. Post-traumatic stress disorder is associated with immunosenescent T cell phenotypes in the Detroit Neighborhood Health Study. , (2011).
- Uddin, M., et al. Post-traumatic stress disorder is associated with immunosenescent T cell phenotypes in the Detroit Neighborhood Health Study. , (2011).
- Uddin, M. Biological signatures of post-traumatic stress disorder in the Detroit Neighborhood Health Study. , (2010).
- Aiello, A. E. Cytomegalovirus antibodies as a marker of immunosenescence in the Detroit Neighborhood Health Study. , (2010).
- Bustamante, A. C., et al. . Distinct gene expression profiles characterize lifetime PTSD and childhood maltreatment in the Detroit Neighborhood Health Study. , (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados