Method Article
단백질 - 단백질 상호 작용 및 알로 스테 릭 리간드 효과의 속도론을 측정하는 바이오 층 간섭
요약
여기에 프로토콜은 바이오 층 간섭과 단백질 - 단백질 상호 작용의 운동 분석을 설명합니다. 셀룰러 에너지 대사에 관여 F 형 ATP 합성 효소는, 세균에서의 ε 서브 유닛에 의해 억제 될 수있다. 우리는 ε의 억제 C-말단 도메인과 촉매 복합체의 상호 작용을 연구하는 바이오 층 간섭을 채택했다.
초록
우리는 대장균 ATP 합성 효소의 촉매 복합체 소단위 ε의 억제 작용을 연구하는 바이오 층 간섭계를 사용하는 방법을 설명한다. 세균성 F 형 ATP 신타 약물 내성 결핵에 대처하기 위해 새로운, FDA 승인 항생제의 대상입니다. 서브 유닛 ε의 C-말단 도메인에 의해 ATP 합성 효소의 이해 박테리아 별 자동 억제 항균 약물의 발견을위한 효소를 표적으로하는 새로운 방법을 제공 할 수있다. ε의 C-말단 도메인은 활성 및 비활성 상태 및 촉매 사이트 리간드 간의 전이 효소가 우세한 ε의 배좌의 영향을 미칠 수있는 극적인 형태 적 변화를 겪는다. 간접적으로 ε의 바인딩 / 촉매 복합과 분리하고, MEA의 분석 측정 반응 속도슈어 분명히 nondissociable 억제 형태 및에서 효소 결합 ε의 변화. 바이오 - 레이어 간섭 신호는 용액 조성물에 지나치게 민감하지이므로도 ε의 형태 적 변화에 촉매 사이트 리간드의 알로 스테 릭 효과를 모니터하는데 사용될 수있다.
서문
단백질 - 단백질 상호 작용은 많은 생물 학적 과정에 중요하며, 표면 플라스 몬 공명 (SPR) 같은 레이블의 광학 방법은 1 바인딩과 해리의 속도를 연구하기 위해 체외에서 사용되어왔다. 대부분의 라벨없는 방법은 센서면에 하나의 생체 분자를 고정화하고 고정화 한 생체 분자와 연관로서 용액으로부터 바인딩 파트너를 검출하는 광 신호를 사용한다. SPR은 매우 민감한 방법이지만,이 때문에 센서 (2)를 통해 흐르는 용액의 굴절률 변화에 간섭하는 경향이있다. SPR 바이오 층 간섭계 (BLI)만큼 민감하지가 적은 샘플 구성 1,3의 변화에 의해 영향을받지 않는다. BLI는 끝 독점 생체 적합성 코팅이 광섬유 바이오 센서를 사용합니다. (진수 RED96) 여기에 사용 된 시스템에 8 개의 분광 광도계가 포함되어 있습니다. 흰색 빛이 로봇 팔에 이동 프로브의 행으로 파이프됩니다. 광섬유 센서는 아칸소전자는 프로브에 의해 포착 및 샘플을 포함하는 96 - 웰 플레이트로 옮겼다. 표적 분자 중 하나는 바이오 센서 표면에 고정된다. 그런 다음 센서 솔루션의 결합 파트너를 포함하는 우물로 이동합니다. BLI는 고정 된 분자와 결합 파트너의 연결을 모니터링하고 바인딩 파트너없이 솔루션에 센서를 이동 한 후 해리를 모니터링합니다. 바이오 센서의 표면에 분자의 결합은 내부 표면 및 센서 및 솔루션 사이의 외부 인터페이스에서 다시 분광 광도계에 반영하는 빛의 파장과 광 간섭의 변화로 연결됩니다. 간섭의 이러한 변화는 정량화 및도 1의 애니메이션에 요약 바인딩 및 해리의 운동 속도를 결정하는데 사용될 수있다.
우리는 촉매 세균 ATP 합성 효소의 복잡한 효소를 자동으로 억제 할 수는 ε의 서브 유닛 사이의 상호 작용을 측정하는 BLI를 적용했습니다. ATP 싸이nthase 합성 및 ATP 4의 가수 분해를 촉매 막 - 내장 된 회전을 nanomotor입니다. 촉매 복합체 (F 1) ATP 아제로 작동하는 수용성 형태로 분리 할 수있다. 소단위 ε는 두 도메인있다 : N-말단 영역 (NTD) 적절한 조립 및 효소의 기능적인 결합을 위해 필요하지만, 촉매 서브 유닛과 직접적으로 상호 작용하지 않고, C-말단 도메인 (CTD)에 상호 작용하여 효소를 억제 할 여러 촉매 서브 유닛 5,6. 이 ε - 중재 규정은 세균 ATP 합성 효소에 고유하며 미토콘드리아 상동에서 관찰되지 않습니다. 약물 내성 결핵 7을 치료하는 bedaquiline의 최근 FDA의 승인에 의해 도시로 ATP 합성 효소는, 항균 약물의 대상으로 떠오르고있다. 따라서, 약물 발견을위한 ε의 억제 역할을 대상으로하는 미토콘드리아 ATP 합성 효소를 억제하지 않는 항균제를 얻을 수 있습니다. 절연 촉매 착체 (F 1), ε와 함께해리 서브 유닛이됩니다. 그러나, F (1)에 바인딩 ε와 εCTD 부분적 효소의 중앙 공동 내로 삽입하고 직접 6,8 해리 가능성은 억제 상태를 형성 극적인 형태 적 변화를 겪을 수있다. 우리는 촉매 사이트는 ε의 형태에 대한 리간드의 알로 스테 릭 효과를 조사하기 위해, 간접적으로 F 1 / ε 결합과 해리의 반응 속도를 측정하는 BLI를 사용합니다.
우리의 시스템에서, ε는 (SPR 등) BLI 신호 이후 센서 표면에 고정화를 위해 선택 하였다는 표면에 결합 분자의 질량에 민감하다. ε 서브 유닛은 메인 F 1 단지 (~ 347 kDa의) 작은 (~ 15 kDa의)를 기준으로합니다. 따라서, 더 큰 BLI 신호는 고정 ε에 F 1의 결합에서 발생합니다. 매우 느릴 수 있습니다 F 1 해리를 모니터링하기 위해, ε 강하게 고정해야합니다. 따라서 우리는 biotinylate하기로 결정했습니다; 및 스트렙 타비 딘 - 코팅 된 바이오 센서를 이용하여 그것을 고정시킨다. 단백질 표면 리신 (9)의 (I)의 임의의 변형, 비오틴 - 말레이 미드 시약 (10) 또는 (ⅲ) 유전자 효소 중에 비오틴되는 특정 비오틴-수용체 펩티드 첨가와 고유 네이티브 또는 엔지니어링 시스테인 (II)의 반응에 의해 비오틴 수 태그 단백질 (11)의 생체 내 발현. 우리의 시스템에서, ε는 방법 (III) 8 사용 비오틴된다. 비오틴 태그 ε는 스트렙 타비 딘 센서에 고정되면, BLI는 바인딩과 서브 유닛 ε (F 1 (-ε))의 소진 된 F 1의 분리를 측정 할 수 있습니다. 여기 기재된 실험을 위해, 예비 분석은 센서에 고정화 바이오틴 단백질의 적당한 양을 결정하기 위해 수행되었다. 이 단백질의 분자량 및 그 결합 파트너에 따라 달라질 수 있지만, 목표는 고정화 단백질 t의 최소한의 양을 결정하는 것이다모자를 제공합니다 (I) 허용 신호 대 잡음 (K D 이하) 바인딩 파트너의 낮은 농도와 결합 파트너의 거의 포화 농도와 반응 속도를 결합 (II) 최소한의 왜곡과 반응 속도를 바인딩. 또한, 바이오 티 닐화의 화학 양론은 일관된 BLI 신호가 스트렙 타비 딘 - 코팅에 고정하는 동안 달성 될 수 있음을 확인하기 위해 변경 (만> 1 몰 비오틴 / 몰 단백질을 방지), 그래서 몇 가지 초기 분석은 바이오틴 단백질의 새 많이 필요 할 수도 있습니다 센서.
프로토콜
1. BLI 분석 실험을위한 장비를 프로그래밍
램프가 예열 할 수 있도록 사전에 기기에 적어도 한 시간에 켜고,이 소음을 최소화하고 실험 기간 동안 광 신호에서 표류 할 필요가있다. 샘플 플레이트 홀더를 prewarm 악기 탭을 통해 원하는 온도를 설정합니다. 그런 다음 데이터 수집 소프트웨어의 실험적인 디자인을 설정합니다. 실험 마법사 탭에서 "새 속도론 실험"을 선택합니다. 이 정의해야하는 모든 단계에 탭 메뉴를 제공합니다.
1.1. 플레이트의 정의
96 - 웰 샘플 플레이트에 사용되는 열을 정의. 버퍼, 고정 된 단백질 또는 바인딩 파트너를 포함하는 열을 지정합니다. 각 웰 협회, 사용될 파트너 결합 농도를 입력한다. 참고 : 그림 2의 플레이트 정의가 서로 다른 분석을위한 옵션이 있습니다.
1.2. 분석의 정의
ontent "> 분석에 필요한 모든 단계를 정의합니다. 다음은 (내가)베이스 라인 (수), (II)로드, (ⅲ) 협회와 파트너를 바인딩 (IV) 해리 (가) 있습니다. 그런 다음, 아래에 설명 된대로, 개별 분석 단계를 링크 각각의 열 분석 단계를 두 번 클릭을 선택하여 샘플 접시에 우물의 열.이 프로그램은 센서는 분석하는 동안 다음에 우물 하나의 컬럼에서 이동합니다.- 기준
96 - 웰 플레이트에 초기 BLI 신호를 설정하기 위해, 분석 버퍼 센서 (그림 2, 열 1)과, 간단한 초기 단계 (≥ 60 초)로 시작합니다. - (센서의 바이오틴 단백질을 고정)로드
바이오틴 단백질 (그림 2, 2 열)를 포함한다 우물에 센서를 할당합니다. 바인딩의 일정 수준 (소개를 참조하십시오) 달성하기 위해 임계 값 기능을 사용합니다. 모든 센서가 이동 될 수 있도록 임계 값 옵션을 설정센서들 중 하나가 임계 값에 도달 웰 다음 열로 D. - 기준
센서에서 멀리 nonimmobilized 바이오틴 단백질을 씻고 새로운 안정 기준 신호를 설정, 버퍼의 또 다른 기본 단계 (일반적으로> 5 분 그림 2, 3 열)를 포함합니다. 만 (그림 3)을 기본 결합 / 분리 분석법의 경우, 협회의 단계 전에 버퍼의 새로운 우물에 센서와 별도의 기본 단계 (60 초) (그림 2, 4 열) 등이 있습니다. - (고정 된 단백질에 결합 파트너의) 협회
(그림 3)을 기본 결합 / 분리 분석을 위해, 다른 센서 / 우물에 사용되는 바인딩 파트너의 농도 범위 (그림 2, 5 열)을 선택합니다. 바인딩 파트너의 적어도 포화 농도가 신호를 결합 평형에 접근해야되도록 단계 시간을 조정한다. 에 대한(그림 5에서와 같이) 단백질 - 단백질 복합체의 해리에 작은 리간드의 알로 스테 릭 효과를 테스트하는 분석은, 모든 협회 우물에 사용 (10 배 추정 K D 이상 ~) 바인딩 파트너의 하나의 높은 농도를 선택합니다. - (바인딩 파트너의) 분리
만 (그림 3)을 기본 결합 / 분리 분석의 경우, 4 열 (그림 2), 협회 전에 별도의 기준에 사용 된 것과 동일한 버퍼 우물로 돌아 센서를 지정합니다. 작은 리간드의 알로 스테 릭 효과를 테스트 할 수있는 분석의 경우, 대신에 열 6 각 잘 버퍼 플러스 다른 알로 스테 릭 리간드를 포함 할 수 있습니다 (그림 2)으로 이동 센서를 지정합니다.
1.3. 센서 할당
분석을 위해 prewetted 센서를 포함 할 센서 트레이에 위치를 나타냅니다. 실험 FAI 때문에 빈 위치를 확인L에는 센서가 포착되지 않은 경우.
1.4. 검토 실험
실수를 확인하고이를 수정로 돌아가 모든 계획 단계를 시각화.
1.5. 실험을 실행
데이터 파일의 위치를 포함하여 필요한 세부 사항을 입력합니다. 실험에 원하는 온도를 입력합니다. 센서는 여전히 prewetting이 필요한 경우, 실험을 시작 지연하는 옵션을 선택합니다. 마지막으로, 한 번 모든 샘플 준비 (2 단계) 완료하고 샘플 플레이트와 센서 트레이 모두가 악기에로드, 분석을 실행하는 바로 가기 버튼을 클릭합니다.
2. 샘플 준비
- 적절한 분석 버퍼를 준비합니다. (8.0에 첨가하여 pH를 조정할) - ((N-모르 폴리 노) 프로판 술폰산 3), 트리스 (트리스 (히드 록시 메틸) 아미노 메탄), 50 mM의 KCl을 20mM의 MOPS : 버퍼는도 3 및도 5에 도시 된 실험에 사용. BSA (소 혈청 알부민, 자유 지방산, 0.5 마련되어모든 분석 단계와 닐화 단백질 또는 센서 단백질의 비특이적 결합을 최소화하기 위해 결합 파트너의 모든 희석에 대한 버퍼) 최종 ML ㎎ /.
- (소개 참조) 적절한 농도로 분석 버퍼에 바이오틴 단백질 (ε)를 희석.
- 분석 버퍼에 바인딩 파트너 (F 1 (-ε)) (사용하는 농도의 범위에 대한 설명을 참조)의 희석을 준비합니다.
- 그들의 보호 자당 코팅을 제거하기 위해 적어도 10 분 동안 스트렙 타비 딘 - 코팅 센서 Prewet. 센서 트레이에서 센서를 포함하는 랙을 제거하고 트레이에 배향 노치에 판의 한 구석 슬라이딩 트레이의 바닥에 96 - 웰 플레이트를 삽입합니다. 센서의 열을 사용하는 경우, 96 - 웰 플레이트의 열을 잘 당 분석 완충액 200 μl를 추가합니다. (슬롯 탭)를 올바른 방향으로 트레이 센서의 랙을 반환합니다.
- 1 단계에서 프로그래밍하는 동안 할당 된, O 우물을 채우기그림 2에서와 같이 분석 버퍼 또는 적합한 단백질 희석와 F 샘플 플레이트. 그들은 광 신호의 노이즈를 일으킬 수, 거품을 도입하지 마십시오. (I) 비오틴 단백질 또는 (ⅱ) 결합 파트너 중 하나를 생략 하나 이상의 참조 우물을 포함합니다.
- 악기의 문을 열고 무대의 슬롯에 삽입 된 트레이의 탭으로, 단계 (왼쪽)에 센서 트레이를 삽입합니다. 판 홀더 (오른쪽)에 샘플 플레이트를 삽입 한 판 홀더에 표시된대로 판은, 평평하고 올바른 방향으로 장착되어 있는지 확인합니다. 문을 닫고 데이터 수집 프로그램 (단계 1.5)에서 분석을 시작합니다.
3. 데이터 처리
- 분석이 실행 된 후, 데이터 분석 소프트웨어를 열고 데이터를 포함하는 폴더를로드. 각 센서 (왼쪽) 단계별 처리 메뉴와 원시 운동 데이터를 확인하려면 "취소"탭을 클릭 (AH) 다른 색상을 할당 (참조그림 3).
- "데이터 선택"에서 "센서 선택"버튼을 클릭합니다. "샘플 플레이트지도"에서 참조 우물로 1 또는 2 제어 센서 (G, 그림 3의 분석에서 H)에 대한 우물을 지정합니다. 처리 메뉴에서 "빼기"확인란을 선택하고 다른 모든 센서의 신호로부터 기준 신호 (단일 또는 평균)을 뺄 "참조 웰스"를 선택합니다.
- "정렬 Y 축"단계를 사용하여 = 0 Y에 모든 흔적을 맞 춥니 다. 정렬 단계 (이것은 협회의 단계 전에 마지막 기준 인) 등의 "기준"을 선택합니다. "시간 범위"에 대한, 그 기준 (그림 3에서와 같이 60 초 기준에 대한 즉, 50 ~ 60 초)의 마지막 10 초를 입력합니다.
- 협회와 해리 단계 사이의 신호 변화를 최소화하기 위해 "간 단계 보정"확인란을 선택합니다. 베이스 라인 또는 해리에 정렬을 선택합니다.
- 대부분의 경우 Savitzky - 골 레이 필터링 기능을 선택하고 "프로세스 데이터를!" 진행합니다. 육안 검사최종 처리 된 데이터 (오른쪽 패널). 신호 트레이스는 협회와 해리 단계 사이에 상당한 변화를 표시하는 경우 (그림 4에서와 같이) 글로벌 데이터 분석을위한 분석과 함께, "간 단계의 보정"의 해리 또는 기준의 선택을 변경하고 데이터를 진행하기 전에 데이터를 다시 처리 분석.
4. 데이터 분석
시작하는 데이터 분석에서 "분석"탭을 클릭합니다. 참고 : 예를 들어 여러 바인딩 / 해리 곡선의 글로벌 분석 (그림 3에서와 같이)입니다 아래의 단계를 반복합니다.
- "분석하는 단계"의 경우, 협회와 해리를 선택합니다. "모델"에 대해 1:1를 선택합니다. 참고 : 제한된 옵션을 사용할 수 있습니다.
- "피팅"의 경우, (전체) 글로벌을 선택합니다. "별 그룹"에 대한 (그래프 등) 색상을 선택합니다. (R 최대의 독립적 인 피팅을 할 수 있도록 "센서가 연결되지 않은 R 최대"를 선택 파의 결합 포화에 따라 최대한의 신호 응답) 고정 된 단백질에 tner. 참고 : 센서는 고정 된 단백질의 양이 다소 다를로 우리는 (그림 3,로드 참조), 대부분의 분석에 대해이 작업을 수행합니다.
- 표시된 테이블에서 분석 할 모든 센서 흔적 같은 색깔이있다, 그래서 그들은 글로벌 세트로 분석 될 것이라는 점을 확인한다. 필요한 경우, 하나 이상의 센서가 글로벌 피팅에서 생략 추적 선택 : "포함"열에서 원하는 센서 위치를 마우스 오른쪽 버튼으로 클릭하고 "웰스 제외"를 선택합니다.
- "적당한 곡선을!"클릭 비선형 회귀 분석을 시작합니다. (그림 4)를 센서 데이터 추적으로 회귀 곡선 (I) 오버레이, 피팅 잔차 (II) 플롯, 그리고 결정된 매개 변수 값 (속도 상수, R 최대로 (III) 테이블을 포함 피팅 결과를 검토, K D) 및 통계 매개 변수 (표준 오차, 카이 제곱, R 2).
- "데이터 내보내기"에서 "내보내기 테이블에. CSV 파일"을 클릭하여 피팅 결과를 저장합니다. GRAP에 대한힝 또는 다른 소프트웨어와 데이터 / 장착 곡선의 추가 분석은 텍스트 파일의 각 센서의 데이터 및 장착 곡선을 저장하려면 "수출 피팅 결과"를 클릭합니다.
결과
실시간 바인딩 및 해리 BLI의 반응 속도는 그림 3에 나와 있습니다. 이 실험은 협회와 해리의 분석 완충액으로 이루어졌다. 센서가 잠깐 동안 만 prewet했다 이후이 실험은 10 분 기준 단계를 시작했다. 다음으로, 비오틴 ε는 센서에로드했습니다. ε의 검출 가능한 해리은 바인딩 파트너가 추가되지 있었다 기준 곡선 (G)에서 본 나머지 모든 단계에 걸쳐 발생했습니다. 제 2 기준 센서 (H)가 고정화 단백질의 결여이고 결합 파트너의 낮은 비특이적 결합을 나타내었다. F 1 (-ε)의 다양한 농도는 전 세계적으로 결과를 맞게하고 케이, 케이 D 및 K D에 대한 최고의 가치를 얻기 위해 센서 AF 협회 단계에서 사용되었다. 결과는 그림 4의 데이터 트레이스 (파란색)를 산출 프로토콜로 처리했다. 이 경우, 기준 센서 추적 H는 샘플 TR로부터 감산 하였다바인딩 파트너 및 시스템 신호 드리프트의 비특이적 결합을 보정하는 에이스. 데이터 분석 단계에서, 모든 곡선은 1:1 모델 (단일 케이, 하나의 케이 D)와 함께 세계적으로 적합했다; "글로벌 (전체)"맞는 바인딩 파트너의 완전한 분리를 가정 (신호는 무한에서 0으로 돌아갑니다 시간) 빨강 (그림 4). 것을 유의 한 F (-ε)의 두 가장 높은 농도에서, 해외 1:1 모델로부터 데이터 작지만 상당한 편차가있다. 고정화 리간드 (비오틴 "ε"서브 유닛 내)의 낮은 수준이 사용되었고, 적당한은 모델 물질 전달 계수 (도시되지 않음)를 포함함으로써 개선되지 않았다. 그러나, F 1 (- "ε")는 큰 착체 (~ 350 kDa의)이고, (도시되지 않은) 다른 실험은 이러한 편차가 고정화 된 리간드의 분획의 감소 바인딩 접근성 때문이라고 제안, 즉 F (1)에 바인딩 한 "ε"는 입체적으로 다른 바이오틴에 대한 액세스를 방해 할 수같은 스트렙 타비 딘 테트라에 바인딩 "ε".
그림 5는 효소가 1 ㎜ ATP / EDTA (에틸렌 다이아 민 테트라 초산)의 존재에 ε 센서 고정화에 바인딩하는 다른 실험을 보여줍니다, 이것은 대부분의 F 1 / "ε"단지 쉽게 8 해리 noninhibitory 형태를 가정에 걸리기. 센서는 다음 리간드와 분석 버퍼를 포함하는 우물을 분리하기 위해 이동했다. 이 ε의 형태에 다른 리간드의 효과를 보여줍니다. 예를 들어, MgADP / 파이에 대한 노출이 크게 ε가 긴밀하게 결합 된, 억제 상태로 형태를 이동을 나타내는 순 해리 둔화. 리간드는 F 1의 비율에서 다른 변화를 야기하기 때문에 복잡한 해리 반응 속도가 관찰되었다. 활성 (해리) 또는 억제 (nondissociable) 입체 형태의 ε와 ε 단지를. 또한 가능하다 바인딩 단백질의 상당한 구조적 변화. BLI 신호의 변화에 직접적으로 기여하지만, 우리의 연구에서,이 (샤 등 8, 그림 5D를 참조 큰 F 1 복합체의 결합 / 분리에 대한 신호에 비해 무시할 수있는 것으로 나타납니다, 곡선 3, 6은 전환 매우 유사한 행동, 자신의 조건이 결합 된 F의 독특한 입체 형태를 선호하지만, 1). 데이터 분석 소프트웨어는 복잡한 분해 반응 속도를 위해 설계되지 않았습니다. 따라서, 우리는 다른 소프트웨어와의 비선형 회귀 분석을 위해 텍스트 파일로 데이터를 내보낼.
그림 1. 바이오 층 간섭의 원리. 애니메이션 BLI에 의해 생체 분자 상호 작용을 검출하는 기본 물리학을 요약 한 것입니다. 프로브를 통과 한 빛은 센서의 내부면으로부터 및 (II)의 외부로부터 위로 분광 광도계 (I)로 반사된다검액으로 센서의 터 페이스. 이것은 간섭 패턴 초래한다. 센서 표면에 분자의 결합은 간섭 패턴을 변화시킨다. 이 파장 대 상대 강도의 나노 미터의 변화로 그릴 수 있습니다. 시간이 지남에 따라이 나노 미터의 변화를 모니터링 바인딩 및 해리 반응 속도를 제공한다. 그래픽은 FortéBio 12의 허가를 맞게된다.

도 2. 96 - 웰 샘플 플레이트의 샘플 개략 구성. 센서는 열에서 열 평행 이동되며 좌우 특정 분석 프로토콜에 정의 된 하나를 이동시킬 수있다. 빨강, 파랑, 녹색 파트너 리가 결합, 고정 된 단백질을 나타내는 반면 회색 웰스는 분석 버퍼를 나타냅니다각각 NDS,.
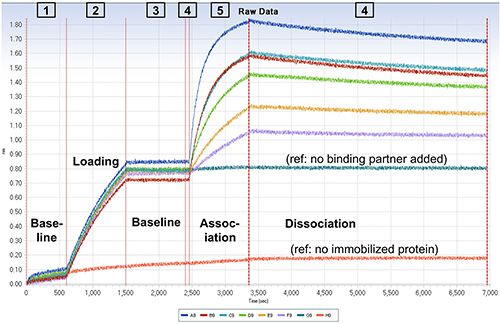
BLI 분석에서 원시 데이터를 보여주는 그림 3. 화면 캡처. 빨간색 세로 줄이 프로토콜의 분석 단계 사이에 센서의 움직임을 나타냅니다. 단계 유형은 화상에 표시된다. 버퍼와 샘플의 위치는 같은 그림 2에 나타내었다. 그래프의 상단에 숫자로 언급 한 바와 같이, 스트렙 타비 딘 - 코팅 된 센서는 이동되었다 123,454 : 다음과 같은 순서로 샘플 플레이트의 상이한 열 사이. 그래프 아래의 범례는 각 센서의 데이터 트레이스의 색을 나타냅니다. 로드 단계에서 H를 제외한 각 센서는 50 nM의 비오틴 "ε"서브 유닛으로 배양 하였다. 30 (: 협회 단계에서, 센서는 "ε"고갈 F 1 nM의 농도로 배양 하였다A, H), 20 (B), 15 (C), 10 (D), 5 (E), 2.5 (F), 0 nm의 (G). 더 큰 이미지를 보려면 여기를 클릭하십시오.
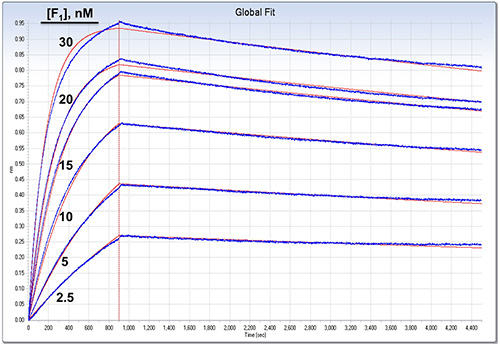
도 3의 BLI 분석에서 분석 된 데이터의도 4. 화면 캡처. 데이터 처리 및 텍스트에서 설명한 장착 하였다. 단지 협회와 해리 단계는 빨간색 세로 선으로 나누어 표시됩니다. 처리 된 데이터 곡선은 파란색, 비선형 회귀는 빨간색으로 표시됩니다 1시 1분 글로벌 분석에 적합하다. 글로벌 피팅 결과 8 : K = 2 × 5m -1의 1 (± 0.1 %), 케이 D = 4.8 × 10 -5의 -1 K D = 0.24 nm의 항복 ( "±"0.1 %). 적합도 : R= 0.999054 2. 최대 매개 변수 바인딩 (R 최대)의 평균 값 = 0.813 nm의 ( "±"0.0803)로, 각각의 센서에 대해 개별적으로 적합했다. 더 큰 이미지를 보려면 여기를 클릭하십시오.
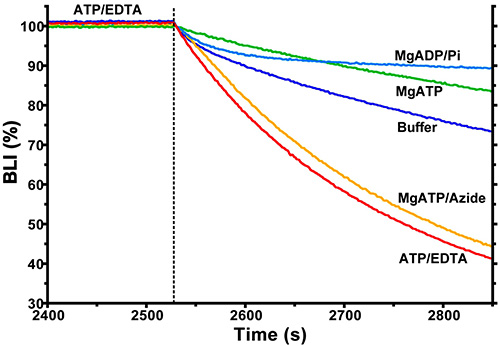
그림 5. ε의 형태에 대한 리간드의 알로 스테 릭 효과를 보여주는 BLI 분석의 결과. F 1 (-ε)은 1 mM의 ATP / EDTA (에틸렌 다이아 민 테트라 초산)의 존재에 ε 센서 고정화에 바인딩했다. 연관 상과 해리 상 부분의 단부가 도시된다. 해리 단계에서, (각 추적 나열) F 1 촉매 사이트에 대한 별개의 리간드는 F 1의 분리에 자신의 알로 스테 릭 효과를 관찰 하였다 서브.
토론
BLI에 대한 현재 사용 가능한 장비는 생체 분자의 상호 작용에 대한 분석에 상당한 처리량과 유연성을 허용합니다. 각종 용액 시료 블랙 마이크로 타이 터 플레이트의 웰에 분배하고, 병렬 BLI 센서 세트는 플레이트에 웰의 열 사이에서 앞뒤로 이동하도록 프로그램되어있다. 시료는 분석을 통해 궤도 흔들어 교반. 여기에 사용 된 시스템은 8 센서를 가지고 있으며, 96 웰 샘플 플레이트를 사용하지만, 다른 시스템 (16) 센서와 384도 샘플 플레이트를 사용합니다. 따라서, 센서가 고정 된 생체 분자와 솔루션의 결합 파트너 사이의 상호 작용은 병렬로 여러 개의 센서와 다른 조건에서 테스트 할 수 있습니다. 예를 들어, 고정화 억제 ε 서브 유닛으로 여기에 제시된 제 분석에서, 상호 작용은 평행 효소 (결합 파트너)의 여섯 가지 농도의 측정 (도 3 및 4) 하였다. 이것은 도전 분석 determi 허용NE에 대한 속도 상수의 단일 쌍 (A, K d을 (를) K) 순 결합 및 분리, 따라서 평형 해리 상수 (K D는 = K D / K), 이는 억제 상수와 밀접하게 동의 (K I)에서 결정 ε (8)에 의해 효소 억제의 솔루션 분석. 실험의 두 번째 유형 (그림 5) BLI는 또한 두 단백질 사이의 상호 작용에 작은 리간드의 알로 스테 릭 효과를 모니터링 할 수 있음을 보여, 다른 리간드의 효과를 병렬로 비교 하였다. BLI 신호는 결합 분자의 질량에 의존하며, 직접적으로 최적화 된 조건 하에서 13 작은 화합물의 바인딩을 검출 할 수 있기 때문에 친화력 리간드의 효과를 검출하는 한계가있다. 그러나,도 5의 분석에서, 결합 파트너, F (1)는, 대규모 단백질 복합체 (~ 347 kDa의), 그래서 그것의 결합 / 분리를위한 BLI 신호는 크게 광고에 의해 영향을받지 않았다추가적인는 F 1 복잡한 같은 ATP (500 ~ 다)와 같은 작은 리간드의 결합. 이는 이러한 리간드 억제 CTD 8 결여 ε의 절두 형태에 바인딩 F 1 BLI 신호에 영향을 미치지 않았다되는 분석법으로 확인 하였다. 따라서, 알로 스테 릭 효과의 분석은 상호 작용하는 단백질의 질량에 알로 스테 릭 리간드 상대의 질량을 고려해야합니다, 바람직하게는 리간드의 결합에 직접 BLI의 응답을 테스트하기 위해 컨트롤을 포함합니다.
실험 프로토콜의 특정 단계 및 변형이 더 나은 결과를보다 정확한 분석을 달성하기위한 중요 할 수 있습니다. 예를 들어, 여기에 설명 된 프로토콜로, 결합 및 해리 속도 상수의 글로벌 분석을위한 분석은 새로운 열에서 별도의 기준 단계 (단계 1.2.3 참조)이 포함되어야합니다 (1.2.3, 1.2.5 단계를 참조) 협회 단계, 및 센서 연구해야한다 버퍼 우물 (그림 2, 4 열) 전에해리 단계 버퍼 우물 같은 열 (단계 1.2.5 참조) eturned. 협회 공정 전후에 (동일한 광학 특성을 가진) 같은 잘 각 센서를 갖는 것은 협회 / 해리 단계는 인터 - 단계 보정 (단계 3.4)을 개선하고, 이는와 같이 (다중 데이터 트레이스 해외 운동 피팅을 용이하게 할 그림 4). 또한, 여기에 표시된 것보다 더 많은 예비 분석을 위해, 프로그램은 (그것을 활성 단계 동안) 단축하거나 분석하는 동안 모든 단계의 시간을 연장 할 수있는 옵션이 있습니다. 더 많은 시간이 해리 속도에 적합한를 달성하기에 충분한 해리를 허용하는 경우, 필요에 따라서 최적의 로딩 신호가 이미 알려져 있지 않거나 경우에 유용하다.
센서 표면에 대한 결합 파트너의 결합 특이성은 SPR 및 BLI 같은 라벨없는 방법에 대해 문제가 될 수있다. 우리의 분석에서,이 효과적으로 분석 완충액에 BSA를 포함함으로써 최소화 하였다. 면역 블롯에서와 같이딸랑 딸랑 소리 BSA 이외의 단백질이 사용할 수있는 프로토콜 및 세제의 낮은 농도도 최소화 할 수 비특이적 (트윈 20은 공급 업체의 반응 속도 버퍼에 사용되는) 바인딩. 결합 잔류 비특이적의 뺄셈 (및 부패 동안 심지어 최소한의 비특이적 결합으로, 대부분의 분석은 고정 된 단백질하지 않고 있지만, 협회 단계에서 결합 파트너의 높은 농도 (그림 3, 센서 H 참조) 적어도 하나의 제어 센서를 포함한다 해리 단계는) 크게 운동은 특정 결합 / 분리가 연구되고에 대한 분석을 향상시킬 수 있습니다. 한편, 일단 초기 분석이 바이오틴 단백질 (도 3, 센서 G 참조) 안정적으로 적재 단계 후 바운드 남아 있는지 확인하고, 그 제어는 생략 될 수 있고, 그 센서는 샘플 조건에 사용될 수있다.
BLI를 사용하여 연결 및 분리 속도 상수, B의 여러 농도의 강력한 결정을위한inding 파트너가 사용되어야하고, 가능하면 모든 데이터의 흔적, 글로벌 분석에 의해 적합해야한다 (그림 3과 같이 4). 이상적으로, 농도의 범위는 ~ 10 배량 위와 잡은 K D의 아래 스팬한다. 우리의 경우, 높은 친 화성 상호 작용 (K D = 0.25 NM)와, 신호 대 잡음은 서브 K D의 농도 속도론 바인딩 불량 하였다. 따라서, K D 위에 파트너를 바인딩의 농도는 (그림 3 참조) 관찰 요금과 결합 수준에 큰 변화가 얻어 지도록 하였다. 또한, 긴 해리 단계는 느린 해리 속도 피트의 신뢰도를 향상시키기 위해 사용되었다. 선호도가 높은 상호 작용, 그것은 BLI의 분석이 아니라 SPR의 경우와 센서를 통해 지속적인 흐름,보다 교반 샘플에서 수행되기 때문에 속도가 느린 해리가 해리 단계에서 리 바인딩으로 과장하는 것도 가능하다. 이 잠재적 인 이슈는 테스트 할 수 있습니다해리 버퍼가 해리 바인딩 파트너를 결합하여 센서 고정화 단백질 (14)에 다시 바인딩 것을 방지 과잉 경쟁 단백질의 '싱크'를 포함하는 말한다. 우리의 시스템이 잘 분리에 함께하고 nonbiotinylated (K D 이상> 10 배)하지 않고 결과를 비교하여 수행하고, 우리는 그 리 바인딩이 중요한 문제 8 아니었다 확인되었다. 운동의 결과 세트가 글로벌 분석 (사용 가능한 1시 1분 2:1 모델) 잘 맞지 않을 경우 마지막으로, 문제 해결을위한 다른 분석 옵션이 있습니다. 예를 들어, 대신 (단계 4.2) "(전체) 글로벌"선택의 "지역"은 독립적으로 각 센서의 추적에 맞게 선택할 수 있습니다. 바인딩은 2 차, 고유 바인딩 속도와 동일 기울기와 파트너가 선형 의존도를 표시해야합니다 바인딩의 농도 대 관찰 된 결합 요금 (케이 OBS)를 세우고 간단한 1시 1분 모델을 다음합니다. 소프트웨어의 분석 탭은 "XY가이 관계를 신속하게 시각화 할 수 있습니다 그래프 "창.
우리가 사용하는 악기의 한 가지 제한이 가능한 온도 범위입니다. 40 º C에 주위 온도보다 2 ° C의 범위로, 그 범위에서 안정 분자만을 사용할 수 있습니다. 또한, 열역학적 분석은 좁은 온도 범위에 의해 제한된다. 또 다른 일반적인 제한은 최대 시험 시간 ~ 4 시간 것입니다,이 후, 유물은 오픈 마이크로 플레이트에서 샘플의 증발에 의한 개발.
BLI는 SPR에 비해 비교적 간단한 구성을 포함한다. 센서는 오히려 센서를 통해 시료의 일정한 흐름보다, 고정 볼륨과 우물의 열 사이에 이동합니다. SPR만큼 민감하지 않지만, BLI 덜 인해 샘플 조성의 변화에 굴절율의 변화에 의해 영향을 받는다. 전반적으로, 분석 조건 사이의 이동에 사용 기기의 유연성이 상대적으로 쉽게, BLI는 제공SA 생체 분자 상호 작용의 체외 분석을위한 다양한 도구.
공개
저자는 관심 없음 충돌이 없다는 것을 선언합니다.
감사의 말
우리는 그림 1에 사용 된 그래픽을 제공 FortéBio 감사합니다. 이 작품은 TMD에 NIH 교부금 GM083088에 의해 지원되었다
자료
| Name | Company | Catalog Number | Comments |
| Octet-RED96 | Pall/FortéBio | 30-5048 | |
| Bovine Serum Albumin | Sigma | A6003-10G | Fatty Acid free |
| Biosensor/Streptavidin | Pall/FortéBio | 18-5019 | Tray of 96 sensors |
| Microtiter plate | Greiner Bio-one | 655209 | Black, Polypropylene |
| Data Acquisition software | Pall/FortéBio | Version 6.4 | Newer versions available |
| Data Analysis software | Pall/FortéBio | Version 6.4 | Newer versions available |
참고문헌
- Nirschl, M., Reuter, F., Voros, J. Review of Transducer Principles for Label-Free Biomolecular Interaction Analysis. Biosensors. 1, 70-92 (2011).
- Homola, J. Present and future of surface plasmon resonance biosensors. Anal. Bioanal. Chem. 377, 528-539 (2003).
- Piehler, J., Brecht, A., Gauglitz, G. Affinity Detection of Low Molecular Weight Analytes. Anal. Chem. 68, 139-143 (1996).
- Duncan, T. M., Hackney, D. D., Tamanoi, F. The Enzymes. Energy Coupling and Molecular Motors Vol. XXIII, 203-275 (2004).
- Feniouk, B. A., Suzuki, T., Yoshida, M. The role of subunit ε in the catalysis and regulation of FOF1-ATP synthase. Biochim. Biophys. Acta. 1757, 326-338 (2006).
- Cingolani, G., Duncan, T. M. Structure of the ATP synthase catalytic complex (F1) from Escherichia coli in an autoinhibited conformation. Nat. Struc. Mol. Biol. 18, 701-707 (2011).
- Mirsaeidi, M. After 40 years, new medicine for combating TB. Int. J. Mycobacteriol. 2, 1-2 (2013).
- Shah, N. B., Hutcheon, M. L., Haarer, B. K., Duncan, T. M. F1-ATPase of Escherichia coli: The ϵ-inhibited state forms after ATP hydrolysis, is distinct from the ADP-inhibited state, and responds dynamically to catalytic site ligands. J. Biol. Chem. 288, 9383-9395 (2013).
- Burgess, T. L., Ross, S. L., Qian, Y. -. x., Brankow, D., Hu, S. Biosynthetic Processing of neu Differentiation Factor: glycosylation, trafficking, and regulated cleavage from the cell surface. J. Biol. Chem. 270, 19188-19196 (1995).
- Loo, T. W., Clarke, D. M. Membrane Topology of a Cysteine-less Mutant of Human P-glycoprotein. J. Biol. Chem. 270, 843-848 (1995).
- Tsao, K. -. L., DeBarbieri, B., Michel, H., Waugh, D. S. A versatile plasmid expression vector for the production of biotinylated proteins by site-specific, enzymatic modification in Escherichia coli. Gene. 169, 59-64 (1996).
- Dayne, D. BioLayer Interferometry (BLI) - How Does it Work?. FortéBio newsletter In Interactions. 5, 8-9 (2012).
- Wartchow, C., et al. Biosensor-based small molecule fragment screening with biolayer interferometry. J. Comput. Aided Mol. Des. 25, 669-676 (2011).
- Abdiche, Y., Malashock, D., Pinkerton, A., Pons, J. Determining kinetics and affinities of protein interactions using a parallel real-time label-free biosensor, the Octet. Anal. Biochem. 377, 209-217 (2008).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유