Method Article
Bio-couche interférométrie pour Cinétique des interactions protéine-protéine et allostériques Effets ligand mesure
Dans cet article
Résumé
Les protocoles décrivent ici les essais cinétiques des interactions protéine-protéine avec Bio-couche interférométrie. Type F ATP synthase, qui est impliquée dans le métabolisme énergétique cellulaire, peut être inhibée par sa sous-unité ε dans des bactéries. Nous avons adapté Bio-couche interférométrie pour étudier les interactions du complexe catalytique avec le domaine C-terminal inhibiteur de ε.
Résumé
Nous décrivons l'utilisation de Bio-couche interférométrie pour étudier les interactions inhibitrices de la sous-unité ε avec le complexe catalytique d'Escherichia coli ATP synthase. Bactérienne de type F ATP synthase est la cible d'une nouvelle antibiotique approuvé par la FDA pour combattre la tuberculose résistante aux médicaments. Comprendre spécifiques-bactéries auto-inhibition de l'ATP synthase par le domaine C-terminal de la sous-unité ε pourrait fournir un nouveau moyen de cibler l'enzyme pour la découverte de médicaments antibactériens. Le domaine C-terminal de ε subit un changement conformationnel dramatique lorsque les transitions de l'enzyme entre les états actifs et inactifs, et des ligands au site catalytique, qui peuvent influencer des conformations de ε est prédominante. Les mesures d'analyse cinétique de liaison / dissociation avec le complexe catalytique, et indirectement de mea εSures le décalage ε de l'enzyme lié à et à partir de la conformation d'inhibition apparemment nondissociable. Le signal d'interférométrie Bio-couche n'est pas excessivement sensible à la composition de la solution, de sorte qu'il peut également être utilisé pour surveiller les effets allostériques de ligands au site catalytique sur les changements conformationnels de ε.
Introduction
Interactions protéine-protéine sont importantes pour de nombreux processus biologiques, et les méthodes optiques sans étiquette comme résonance plasmonique de surface (SPR) ont été utilisés in vitro pour étudier la cinétique de liaison et de dissociation 1. La plupart des méthodes sans étiquette immobiliser une biomolécule sur une surface du capteur et d'utiliser un signal optique pour détecter un partenaire de liaison à partir de la solution telle qu'elle s'associe à la biomolécule immobilisée 1. Bien que SPR est une méthode très sensible, elle est sujette à des interférences dues à des changements dans l'indice de réfraction de la solution s'écoulant sur le capteur 2. Bien que pas aussi sensible que SPR, Bio-couche interférométrie (BLI) est moins affectée par des changements dans la composition de l'échantillon 1,3. BLI utilise des biocapteurs à fibres optiques qui ont un revêtement biocompatible propriétaire à la pointe. Le système utilisé ici (Octet-RED96) contient huit spectrophotomètres. La lumière blanche est canalisée vers une rangée de sondes qui se déplacent sur un bras robotisé. Capteurs à fibre optique are captés par les sondes et déplacé à une plaque de 96 puits contenant des échantillons. L'une des molécules cibles est immobilisé sur la surface du biocapteur. Puis capteurs sont déplacés aux puits contenant le partenaire de liaison en solution. BLI surveille association du partenaire de liaison avec la molécule immobilisée, puis surveille dissociation après le déplacement des capteurs de solution sans le partenaire de liaison. La liaison de molécules à la surface du biocapteur conduit à des changements de l'interférence optique entre les ondes lumineuses qui reflètent vers les spectrophotomètres à partir d'une surface interne et externe à partir de l'interface entre le capteur et la solution. Ces changements dans les interférences peuvent être quantifiés et utilisés pour déterminer les taux cinétiques de liaison et de dissociation, résumées dans l'animation de la figure 1.
Nous avons appliqué BLI pour mesurer les interactions entre le complexe catalytique de l'ATP synthase bactérienne et sa sous-unité ε, qui peut auto-inhiber l'enzyme. ATP synthase est un nanomoteur rotatif enrobé dans une membrane qui catalyse la synthèse et l'hydrolyse de l'ATP 4. Le complexe catalytique (F 1) peut être isolé sous une forme soluble qui fonctionne comme une ATPase. Sous-unité ε a deux domaines: le domaine N-terminal (NTD) est nécessaire pour un montage correct et couplage fonctionnel de l'enzyme mais n'interagit pas directement avec les sous-unités catalytiques; du domaine C-terminal (CTD) peut inhiber l'enzyme en interagissant avec de multiples sous-unités catalytiques 5,6. Ce règlement de ε-médiation est spécifique à ATP synthases bactériennes et n'est pas observée dans l'homologue mitochondrial. ATP synthase a apparu comme une cible pour les médicaments antibactériens, comme le montre la récente approbation de la FDA de bedaquiline pour traiter la tuberculose résistante aux médicaments 7. Ainsi, le ciblage rôle inhibiteur de ε pour la découverte de médicaments pourrait donner antibactériens qui ne inhibent l'ATP synthase mitochondriale. Avec le complexe catalytique isolé (F 1), εdevient une sous-unité indissociable. Cependant, avec ε lié à F 1, la εCTD peut subir un changement de conformation dramatique, insertion partielle dans la cavité centrale de l'enzyme et la formation d'un état d'inhibition qui est peu probable de dissocier directement 6,8. Nous utilisons BLI pour mesurer la cinétique de F 1 / ε liaison et de dissociation, et indirectement, pour examiner les effets allostériques du site catalytique ligands sur la conformation de ε.
Dans notre système, ε a été choisie pour l'immobilisation sur la surface du capteur étant donné que le signal BLI (comme SPR) est sensible à la masse des molécules de liaison à la surface. La sous-unité ε est petit (~ 15 kDa) par rapport à la principale F 1 complexe (~ 347 kDa). Ainsi, un signal de plus grande BLI va résulter de la liaison de F 1 à ε immobilisé. Afin de surveiller F 1 dissociation, qui peut être très lent, ε doit être fermement immobilisée. Ainsi nous avons choisi de biotinylerEt l'immobiliser sur biocapteurs revêtues de streptavidine. Les protéines peuvent être biotinylés par (i) la modification aléatoire des lysines de surface 9, (ii) réaction d'une cystéine natif ou composite unique avec un réactif de biotine-maléimide 10 ou (iii) l'addition génétiquement un peptide biotine-accepteur spécifique qui est enzymatiquement biotinylé pendant l'expression in vivo de la protéine marquée 11. Dans notre système, ε est biotinylé en utilisant la méthode (iii) 8. Une fois ε-biotine marqué est immobilisé sur des capteurs de streptavidine, BLI peut mesurer la liaison et la dissociation de F 1 qui a été épuisée de la sous-unité ε (F 1 (-ε)). Pour les expériences décrites ici, des essais préliminaires ont été faites pour déterminer des quantités raisonnables de la protéine biotinylé à immobiliser sur les capteurs. Cela peut varier, en fonction du poids moléculaire de la protéine et son partenaire de liaison, mais le but est de déterminer une quantité minimale de protéine immobilisée tchapeau fournit (i) acceptable signal sur bruit pour la cinétique de liaison avec une faible concentration du partenaire de liaison (K D ci-dessous) et (ii) un minimum de distorsion de la cinétique de liaison de concentration quasi-saturation du partenaire de liaison. En outre, la stoechiométrie de biotinylation peut varier (mais évitez> 1 mole de biotine / protéine mol), de sorte que certains dosage initial peut être nécessaire pour chaque nouveau lot de protéines biotinylé pour confirmer qu'un signal BLI convergentes peut être réalisé au cours de l'immobilisation sur la streptavidine capteurs.
Protocole
Une. Programmation de l'instrument de BLI Assay
Allumez l'instrument au moins une heure à l'avance pour permettre à la lampe de se réchauffer, ce qui est nécessaire pour réduire le bruit et la dérive du signal optique pendant l'expérience. Réglez la température souhaitée via l'onglet instrument Préchauffez le support de plaque échantillon. Ensuite, mettre en place le plan expérimental dans le logiciel d'acquisition de données. Sélectionnez "Nouvelle Expérience Kinetics" dans l'onglet de l'assistant de l'expérience. Cela présente un menu à onglets avec toutes les mesures qui doivent être définis.
1.1. Plaque Définition
Définir les colonnes à utiliser sur la plaque d'échantillons à 96 puits. Assigner les colonnes pour contenir tampon, protéine immobilisée ou partenaire de liaison. Pour chaque association ainsi, entrer la concentration de partenaire de liaison à utiliser. Remarque: la définition de la plaque représentée sur la figure 2 a des options pour des dosages distincts.
1.2. Essai Définition
ontenu "> Définir toutes les mesures nécessaires pour le dosage. s'agit notamment (i) de base (plusieurs), (ii) en cours de chargement, (iii) Association et (iv) La dissociation de partenaire de liaison. Ensuite, comme décrit ci-dessous, un lien étapes de test avec colonnes de puits sur la plaque de l'échantillon en sélectionnant l'étape de test, puis double-cliquant sur la colonne correspondante. Cette programmes les capteurs à être déplacés d'une colonne de puits à l'autre pendant le test.- Baseline
Commencer avec un pas de ligne de base courte (≥ 60 sec), avec des capteurs dans le tampon d'essai (figure 2, colonne 1), pour établir des signaux de BLI initiales dans la plaque à 96 puits. - Chargement (immobilisation de la protéine biotinylée sur les capteurs)
Assigner des détecteurs à puits qui contiennent la protéine biotinylé (Figure 2, colonne 2). Utilisez la fonction de seuil pour atteindre un niveau prédéterminé de liaison (voir Introduction). Réglez l'option de seuil de sorte que tous les capteurs seront mouvementd à l'autre colonne de puits lorsque l'un quelconque des capteurs atteigne le seuil. - Baseline
Inclure une autre étape de base dans un tampon (figure 2, colonne 3; généralement> 5 min) pour laver protéines biotinylées non immobilisés par les capteurs et établir de nouveaux signaux de référence, stable. Pour de base des essais de liaison / de dissociation que (comme dans la figure 3), inclure une étape supplémentaire de base (60 sec) avec des capteurs dans de nouveaux puits de tampon (figure 2, colonne 4) avant l'étape d'association. - Association (du partenaire de liaison à la protéine immobilisée)
Pour une analyse de base de liaison / de dissociation (comme dans la figure 3), sélectionnez une plage de concentrations de partenaire de liaison à utiliser pour différents capteurs / puits (figure 2, colonne 5). Ajuster le temps de l'étape de telle sorte qu'au moins une concentration saturante du partenaire de liaison devrait approcher le signal liaison à l'équilibre. Pour unessai pour tester les effets allostériques de petits ligands sur la dissociation du complexe protéine-protéine (comme dans la Figure 5), sélectionnez une seule concentration élevée de partenaire de liaison (~ 10 fois supérieure à la estimée K D) pour une utilisation dans tous les puits de l'association. - Dissociation (du partenaire de liaison)
Pour un dosage de liaison de dissociation de base / seulement (comme sur la figure 3), désigner les capteurs pour revenir à la colonne 4 (Figure 2), les mêmes puits de tampon que celui utilisé pour la ligne de base supplémentaire avant d'association. Pour un essai pour tester les effets allostériques de petits ligands, au lieu de désigner des capteurs de passer à la colonne 6 (figure 2), où chaque puits peut contenir un tampon en plus différents ligands allostériques.
1.3. Capteur Affectation
Indiquer les endroits dans le plateau de capteur qui va contenir les capteurs préalablement mouillée pour le dosage. Identifier toutes les positions vides puisque l'expérience sera fail si aucun capteur sont pris en charge.
1.4. Expérience avis
Visualisez toutes les mesures prévues à vérifier pour les erreurs et aller de nouveau à les corriger.
1.5. Exécutez l'expérience
Entrez les détails nécessaires, y compris l'emplacement des fichiers de données. Entrez la température désirée pour l'expérience. Si les capteurs nécessitent encore prémouillage, sélectionnez l'option de retarder le démarrage de l'expérience. Enfin, une fois toute la préparation de l'échantillon est terminée (étape 2) et à la fois la plaque de l'échantillon et le plateau de capteur sont chargés dans l'instrument, cliquez sur le bouton OK pour lancer l'analyse.
2. Préparation de l'échantillon
- Préparer un tampon de dosage approprié. Tampon utilisé pour les expériences représentées sur les figures 3 et 5: 20 mM de MOPS (3 - (N-morpholino) propanesulfonique), le Tris (Tris (hydroxyméthyl) amino-méthane) (ajouté pour ajuster le pH à 8,0), 50 mM de KCl. Inclure BSA (albumine de sérum bovin, acides gras libres, de 0,5mg / ml final) dans le tampon pour toutes les étapes de l'analyse et pour toutes les dilutions de la protéine partenaire de liaison biotinylé ou de réduire au minimum la liaison non spécifique de protéines à des capteurs.
- Diluer la protéine biotinylée (ε) dans du tampon analytique à une concentration appropriée (voir l'introduction).
- Préparer des dilutions du partenaire de liaison (F 1 (-ε)) dans un tampon de dosage (voir discussion pour la gamme de concentrations à utiliser).
- Pré-imbibé les capteurs revêtues de streptavidine pendant au moins 10 minutes pour éliminer de leur revêtement protecteur de saccharose. Retirer le porte-capteur contenant le capteur et le plateau à insérer une plaque à 96 puits dans le fond du plateau, en glissant l'un des coins de la plaque dans l'encoche d'orientation sur le plateau. Pour la colonne de capteurs à utiliser, ajouter 200 ul de tampon d'essai par puits dans la colonne de la plaque de 96 puits. Remettre le panier de capteurs sur le plateau dans le bon sens (avec onglets dans les fentes).
- Comme attribué lors de la programmation à l'étape 1, remplir les puits of la plaque d'échantillons avec un tampon de dosage ou des dilutions appropriées de la protéine, comme dans la figure 2. Éviter d'introduire des bulles, car ils peuvent provoquer du bruit dans le signal optique. Inclure un ou plusieurs puits de référence qui omettent soit (i) la protéine biotinylé ou (ii) un partenaire de liaison.
- Ouvrez la porte de l'appareil et insérez le plateau de capteur sur la scène (à gauche), avec les onglets du plateau insérés dans les fentes de la scène. Insérez la plaque de l'échantillon dans le support de plaque (à droite), assurez-vous que la plaque est assis à plat et dans le bon sens, comme indiqué sur le support de plaque. Fermez la porte et commencer le test du programme d'acquisition de données (étape 1.5).
3. Traitement des données
- Après le test a été exécuté, ouvrez le logiciel d'analyse de données et de charger le dossier contenant les données. Cliquez sur l'onglet "Traitement" pour voir un menu étape par étape de traitement (à gauche) et les données cinétiques premières, à chaque capteur (AH) attribué une couleur différente (voirLa figure 3).
- Sous la rubrique «Sélection de données", cliquez sur le bouton "Sélection capteur". Sur la "plaque d'échantillons de carte", désigner les puits pour 1 ou 2 capteurs de contrôle (G, H dans le test de la figure 3) que les puits de référence. Dans le menu de traitement, cochez la case «Soustraction» et sélectionnez «Référence Wells" pour soustraire le signal de référence (simple ou moyenne) à partir du signal de chaque autre capteur.
- Aligner toutes les traces de Y = 0 en utilisant l'étape "Aligner l'axe Y". Sélectionnez "de base" comme l'étape d'alignement (ce qui est la dernière base avant l'étape d'association). Pour "Plage de temps", entrez les 10 dernières secondes de cette ligne de base (c.-à 50-60 secondes pour une base de référence de 60 sec, comme dans la figure 3).
- Cochez la case "Inter-étape de correction» pour minimiser les changements de signaux entre les étapes d'association et de dissociation. Sélectionnez aligner à base ou de dissociation.
- Sélectionner la fonction de filtrage Savitzky-Golay dans la plupart des cas et cliquez sur "Process Data!" de procéder. Inspecter visuellementles données finales transformés (panneau en bas à droite). Avec des essais destinés à l'analyse globale des données (comme dans la figure 4), si des traces de signaux montrent un changement important entre l'Association et les étapes de dissociation, changer la sélection de dissociation ou de référence pour le "Correction Inter-étape» et retraiter les données avant de procéder à des données Analyse.
4. Analyse des données
Cliquez sur l'onglet "Analyse" dans l'analyse des données pour commencer. Remarque: l'exemple ci-dessous sont les étapes de l'analyse globale de plusieurs courbes de liaison / dissociation (comme sur la figure 3).
- Pour "l'étape pour l'analyse", choisissez Association et dissociation. Pour "modèle", sélectionnez 1:1. Remarque: d'autres options disponibles sont limitées.
- Pour "assemblage", choisissez mondial (complet). Pour "Groupe par" sélectionnez Couleur (comme sur le graphique). Sélectionnez «R max UNLINKED par capteur" d'autoriser le montage indépendant de R max (réponse maximale du signal sur la liaison de la saturation parTENAIRE à la protéine immobilisée). Note: nous le faisons pour la plupart des tests, comme des capteurs varient légèrement dans la quantité de protéine immobilisée (voir la figure 3, Chargement).
- Sur le tableau ci, assurez-vous que toutes les traces de capteurs à analyser ont la même couleur, afin qu'ils soient analysés comme un ensemble global. Si nécessaire, sélectionnez un ou plusieurs capteur traces d'omettre de montage mondiale: dans la colonne "Inclure", cliquez-droit sur la position du capteur souhaité et sélectionnez "Exclure Wells".
- Cliquez sur "ajustement des courbes!" commencer l'analyse de régression non linéaire. Examiner les résultats de montage, qui comprennent (i) superposition des courbes de régression avec des traces de données de capteurs (comme sur la figure 4), (ii) des graphiques de résidus de montage, et (iii) un tableau avec des valeurs de paramètre déterminées (constantes de taux, R max, K D) et statistiques (erreurs types de paramètres, Chi-squared, R 2).
- Sous "Exporter des données", enregistrer les résultats de montage en cliquant sur "Exporter le tableau. Fichier csv". Pour graphing ou une analyse plus approfondie des données / courbes ajustées avec un autre logiciel, cliquez sur "Exporter les résultats de montage" pour enregistrer les données de chaque capteur et courbe ajustée dans un fichier texte.
Résultats
Liaison en temps réel et la cinétique de dissociation de BLI sont le montre la figure 3. Cette expérience a été effectuée avec un tampon de dosage en association et de dissociation. Cette expérience a été commencé avec un pas de ligne de base de 10 min depuis les capteurs ont été brièvement pré-imbibé. Ensuite, ε biotinylé a été chargé sur les capteurs. Pas de dissociation détectable de ε s'est produite dans toutes les étapes restantes comme on le voit à partir de la courbe de référence (G) qui n'avait pas de partenaire de liaison ajouté. Un deuxième capteur de référence (H) est dépourvue de protéine immobilisée et a montré faible liaison non spécifique du partenaire de liaison. Différentes concentrations de F 1 (-ε) ont été utilisés dans l'étape d'association pour les capteurs AF pour s'adapter à des résultats à l'échelle mondiale et d'obtenir les meilleures valeurs pour k a, k d et K D. Les résultats ont été traités comme dans le protocole, ce qui donne les traces de données (en bleu) dans la figure 4. Dans ce cas, le capteur de référence trace H a été soustraite de l'échantillon tras pour corriger la liaison non spécifique du partenaire et le signal de système de liaison dérive. Dans l'étape d'analyse de données, toutes les courbes sont globalement en forme avec un modèle 1:1 (seule k un, unique k d), le «Global (Full)" ajustement suppose dissociation complète du partenaire de liaison (signal de retour à zéro à l'infini temps) (Figure 4, rouge). Notez que, pour les deux plus fortes concentrations de F 1 (-ε), il ya des petits mais importants écarts entre les données du modèle global 1:1. Les faibles niveaux de ligand immobilisé (biotinylé "ε" de la sous-unité) ont été utilisés, et l'ajustement n'a pas été améliorées par l'introduction d'un facteur de transport de masse dans le modèle (non représenté). Cependant, F 1 (- «ε») est un grand complexe (~ 350 kDa), et d'autres expériences (non représentés) suggèrent que ces écarts sont dus à l'accessibilité de liaison réduite d'une fraction de ligand immobilisé, c'est à dire une F 1 lié à un "ε" peut encombrement entraver l'accès à un autre biotinylé"Ε" lié à la même tétramère streptavidine.
La figure 5 montre une autre expérience dans laquelle l'enzyme est lié à la sonde immobilisée ε en présence d'ATP 1 mM / EDTA, ce qui prédispose la plupart des F 1 / complexes de "ε" pour assumer la conformation non inhibitrice, qui se dissocie facilement 8. Capteurs ont ensuite été déplacés à la dissociation des puits contenant un tampon d'essai avec différents ligands. Ceci démontre les effets de différents ligands sur la conformation de ε. Par exemple, l'exposition à MgADP / Pi considérablement ralenti dissociation nette, indiquant que ε décalé conformation à l', état d'inhibition étroitement lié. Cinétique de dissociation du complexe ont été observés depuis différents ligands causés différents quarts de travail dans la fraction de F 1. Complexes ε avec ε dans les (dissociables) ou inhibiteurs conformations actives (nondissociable). Il est également possible que des changements de conformation des protéines liées substantiellescontribuent directement à des changements dans le signal BLI mais, dans nos études, ce qui semble être négligeable par rapport au signal pour la liaison / dissociation du grand complexe F 1 (voir Shah et al 8, figure 5D;. transition pour les courbes 3 et 6 montrent comportement très similaire, bien que leurs conditions favorisent conformations distinctes de F lié 1). Le logiciel d'analyse des données n'a pas été conçu pour ces cinétique de dissociation complexes. Ainsi, nous avons exporté les données dans des fichiers texte pour l'analyse de régression non linéaire avec un autre logiciel.
Figure 1. principes de la bio-couche interférométrie. Animations résume la physique sous-jacents pour détecter une interaction biomoléculaire par BLI. La lumière passant à travers les sondes est réfléchie vers les spectrophotomètres (i) à partir de la surface interne de la sonde et (ii) de l'extérieur dansinterface du capteur avec la solution d'échantillon. Il en résulte un motif d'interférence. La liaison de molécules à la surface du capteur modifie la figure d'interférence. Cela peut être tracée comme un changement de nanomètres de longueur d'onde intensité relative vs. Suivre ce nanomètre décalage dans le temps fournit la cinétique de liaison et de dissociation. Les graphiques sont adaptés avec la permission de FortéBio 12.

Figure 2. D'agencement schématique des échantillons dans la plaque d'échantillons à 96 puits. Ces capteurs seront déplacés en parallèle de colonne en colonne, et peuvent être déplacés vers la gauche ou droite tel que défini dans un protocole d'essai particulier. Wells avec la couleur gris représentent tampon d'essai alors que le rouge, le bleu et le vert représentent protéine immobilisée, partenaire et liga liaisonnds, respectivement.
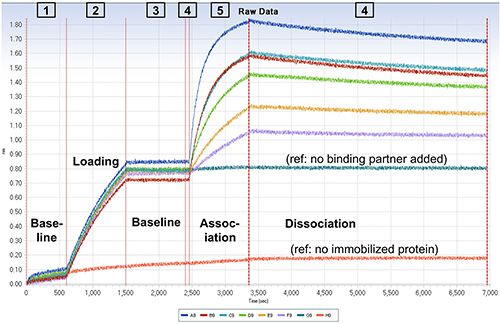
Figure 3. Capture d'écran montrant les données brutes d'un essai de BLI. Lignes rouges verticales indiquent le mouvement des capteurs entre les étapes d'analyse du protocole. types d'étape sont indiqués sur l'image. Lieux de mémoire tampon et les échantillons sont conformes à la figure 2. Comme il est indiqué par des nombres le long de la partie supérieure du graphique, des capteurs revêtues de streptavidine ont été déplacées entre les différentes colonnes de la plaque de l'échantillon dans l'ordre suivant: 123454. La légende sous le graphique indique la couleur des données de trace de chaque capteur. Dans l'étape de chargement, chaque capteur exception de H a été incubé avec 50 nM biotinylé "ε" sous-unités. Dans l'étape d'association, des capteurs ont été incubées avec des concentrations nM de "ε" appauvri F 1: 30 (A, H), 20 (B), 15 (C), 10 (D), 5 (E), 2.5 (F), 0 nM (G). Cliquez ici pour agrandir l'image.
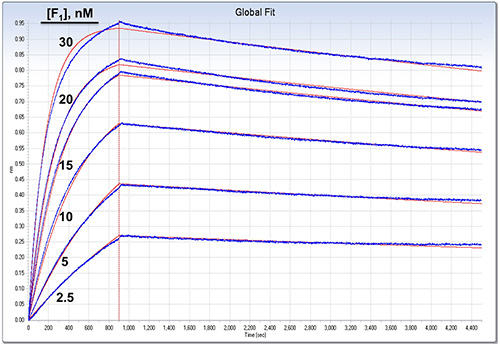
Figure 4. Capture d'écran des données analysées à partir de l'analyse BLI de la figure 3. L'données ont été traitées et équipé comme décrit dans le texte. Seuls association et de dissociation étapes sont représentées, divisé par une ligne verticale rouge. Les courbes de données traitées sont en bleu; la régression non linéaire s'inscrit de 1:1 analyse globale sont indiqués en rouge. Résultats de l'ajustement global 8: k a = 2 x 10 5 m -1 s -1 (± 0,1%), k d = 4,8 x 10 -5 s -1 («±» 0,1%), ce qui donne K D = 0,24 nM. Qualité de l'ajustement: R2 = 0,999054. Le paramètre de liaison maximale (R max) était apte séparément pour chaque capteur, avec une valeur moyenne = 0,813 nm («±» 0,0803). Cliquez ici pour agrandir l'image.
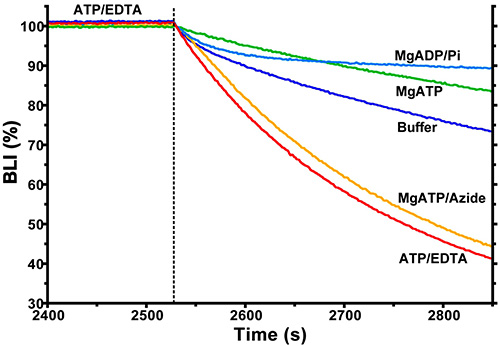
Figure 5. Résultats d'un essai de BLI montrant des effets allostériques des ligands sur la conformation de ε. F 1 (-ε) ε est lié à capteur immobilisé en présence d'ATP 1 mM / EDTA. La fin de la phase de liaison et une partie de la phase de dissociation sont présentées. Au cours de l'étape de dissociation des ligands distincts pour 1 F (voir la liste des sites catalytiques pour chaque trace) ont été inclus pour observer leurs effets allostériques sur la dissociation de F 1 de ε de capteur lié.
Discussion
Instruments actuellement disponibles pour BLI permettent un débit important et la flexibilité dans des essais pour les interactions biomoléculaires. Différents échantillons de solution sont distribués dans des puits d'une plaque de microtitration noire, et un ensemble de capteurs BLI parallèles sont programmés pour se déplacer d'avant en arrière entre les colonnes de puits de la plaque. Les échantillons sont agités par agitation orbitale tout au long de l'essai. Le système utilisé ici a huit capteurs et utilise une plaque de 96 puits à échantillon, mais un autre système utilise des capteurs 16 et une plaque d'échantillon de 384 puits. Ainsi, l'interaction entre une biomolécule sonde immobilisée et son partenaire de liaison en solution peut être testé dans des conditions différentes avec plusieurs capteurs en parallèle. Par exemple, dans le premier essai présenté ici avec la sous-unité ε inhibiteur immobilisé, les interactions ont été mesurées avec six concentrations différentes de l'enzyme (partenaire de liaison) en parallèle (figures 3 et 4). Cela a permis une analyse globale de déterminationnir une seule paire de constantes de vitesse (k a, k d) de filet de liaison et de dissociation, et donc une constante d'équilibre de dissociation (KD = k d / k a), ce qui concorde étroitement avec la constante d'inhibition (K I) déterminée à partir de des dosages d'inhibition de la solution enzymatique par ε 8. Un deuxième type d'expérience (Figure 5) a démontré que BLI peut également surveiller les effets allostériques de petits ligands sur les interactions entre deux protéines, les effets de différents ligands ont été comparés en parallèle. Il existe des limites pour les effets de ligands allostériques de détection, puisque le signal BLI dépend de la masse de la molécule de liaison, et peut directement détecter la liaison de petits composés 13 dans des conditions optimisées. Cependant, dans l'essai de la figure 5, le partenaire de liaison, F 1, est un grand complexe de protéines (~ 347 kDa), de sorte que le signal de BLI pour sa liaison / dissociation n'a pas été significativement affectée par adliaison conditionnelle de petits ligands tels que l'ATP (~ 500 Da) au complexe F 1. Cela a été confirmé par des tests dans lesquels ces ligands n'ont pas d'incidence sur le signal de BLI pour F 1 lié à une forme tronquée de ε manque la inhibitrice CTD 8. Ainsi, les tests d'effets allostériques doivent tenir compte de la masse du ligand allostérique par rapport à la masse des protéines qui interagissent, et comprennent de préférence des contrôles pour tester la réponse BLI direct à la liaison du ligand.
Notez que certaines étapes et des modifications dans le protocole expérimental peut être critique pour obtenir de meilleurs résultats et une analyse plus précise. Par exemple, comme dans le protocole décrit ici (voir les étapes 1.2.3, 1.2.5), un essai destiné à l'analyse globale des constantes de vitesse de dissociation de liaison et devrait inclure une étape de base supplémentaire (voir l'étape 1.2.3) dans une nouvelle colonne des puits tampons (figure 2, colonne 4) avant l'étape d'association, et les capteurs doivent être returned à la même colonne de puits de tampon pour l'étape de dissociation (voir l'étape 1.2.5). Ayant chacun des capteurs dans le même puits (avec les mêmes propriétés optiques) avant et après l'étape d'association peut améliorer la correction inter-étape (étape 3.4) pour les étapes d'association / de dissociation, ce qui facilite le montage cinétique globale de multiples traces de données (comme dans l' la figure 4). En outre, pour les essais qui sont plus avant que montré ici, le programme a une option pour raccourcir ou allonger le temps de n'importe quelle étape de l'essai (alors qu'il est l'étape active). Ceci est utile, par exemple, si le signal de chargement optimal n'est pas déjà connu, ou si plus de temps est nécessaire pour permettre la dissociation assez pour obtenir un bon ajustement pour le taux de dissociation.
La liaison non spécifique du partenaire de liaison à la surface du capteur peut être un problème pour les méthodes sans marquage tels que SPR et BLI. Dans nos essais, cela a été efficacement réduit au minimum en incluant de BSA dans le tampon d'essai. Comme dans immunoblotTing, des protocoles autres que les protéines BSA pourraient être utilisés, et de faibles concentrations de détergent peuvent également réduire la liaison non spécifique (Tween 20 est utilisé dans un tampon cinétique du fournisseur). Même avec un minimum de liaison non spécifique, la plupart des essais devraient inclure au moins un capteur de contrôle sans protéine immobilisée, mais avec la plus forte concentration de partenaire de liaison à l'étape d'association (voir la figure 3, le capteur H); soustraction de la liaison non spécifique résiduelle (et son déclin au cours l'étape de dissociation) peut améliorer considérablement l'analyse cinétique de la liaison / dissociation spécifique à l'étude. D'autre part, une fois que les dosages initiaux confirment que la protéine biotinylé reste lié de manière stable après l'étape de chargement (voir la figure 3, le capteur G), que le contrôle peut être omise et que le capteur peut être utilisé pour une condition de l'échantillon.
Pour la détermination robuste des constantes de vitesse d'association et de dissociation à l'aide de BLI, plusieurs concentrations de brouver partenaire doit être utilisé et toutes les traces de données doit être adapté par une analyse globale, si possible (comme dans les figures 3 et 4). Idéalement, la plage de concentrations doit s'étendre ~ 10 fois au-dessus et au-dessous de la valeur de KD estimée. Dans notre cas, avec une interaction de haute affinité (K D = 0,25 nM), le rapport signal sur bruit, était médiocre pour la cinétique de liaison avec des concentrations sous-K D. Ainsi, les concentrations de partenaire de liaison au-dessus du K D ont été utilisés tels que des changements significatifs dans les taux et niveaux de consolidation observés ont été obtenus (voir figure 3). En outre, une étape de dissociation de long a été utilisée pour améliorer la confiance dans l'ajustement du taux de dissociation lente. Avec des interactions de haute affinité, il est également possible que la dissociation lente est exagérée par reconsolidation pendant l'étape de dissociation, puisque des tests de BLI sont effectuées dans un échantillon sous agitation plutôt que flux continu sur le capteur, comme par SPR. Cet artefact potentiel peut être testé avec commedans laquelle le dit tampon de dissociation contient un «puits» de l'excès de protéine qui se lie compétitif dissocié partenaire de liaison et l'empêche de reconsolidation de la protéine de la sonde immobilisée 14. Dans notre système, ce qui fut fait en comparant les résultats avec et sans (> 10 fois supérieure à K D) non biotinylé dans la dissociation, et nous a confirmé que la reconsolidation n'était pas un problème important 8. Enfin, si des ensembles de résultats cinétiques ne sont pas adaptés et par l'analyse globale (1h01 ou 2h01 modèles disponibles), il existe d'autres options d'analyse pour le dépannage. Par exemple, au lieu de choisir "Global (Full)" (dans l'étape 4.2), "Local" peut être choisi pour s'adapter à chaque trace de capteur indépendamment. Si la liaison suit un modèle 1:01 simple, traçant les taux observés de liaison (k obs) vs les concentrations de partenaire de liaison doit montrer une dépendance linéaire, avec la pente égale à la deuxième ordre, le taux de liaison intrinsèque. L'onglet Analyse du logiciel a une "XYgraphique "fenêtre qui permet la visualisation rapide de cette relation.
Une limitation de l'instrument que nous avons utilisé est la plage de température disponible. Avec une plage de 2 ° C au-dessus de la température ambiante à 40 ° C, seules les molécules qui sont stables dans cette plage peuvent être utilisées. Par ailleurs, l'analyse thermodynamique est limitée par la plage de température étroite. Une autre limitation générale est que le temps de dosage maximal est de ~ 4 heures, après cela, les artefacts se développent en raison de l'évaporation d'échantillons à partir de la plaque de microtitration ouverte.
BLI implique un arrangement relativement simple par rapport à SPR. Les capteurs sont déplacés entre des colonnes de puits avec des volumes fixes, plutôt que d'un débit constant de l'échantillon sur le capteur. Bien que pas aussi sensible que SPR, BLI est moins affectée par les variations de l'indice de réfraction en raison de changements dans la composition de l'échantillon. Dans l'ensemble, avec cette relative facilité d'utilisation et la flexibilité de l'instrument en se déplaçant entre les conditions de dosage, BLI fournirsa outil polyvalent pour les tests in vitro des interactions biomoléculaires.
Déclarations de divulgation
Les auteurs déclarent qu'ils n'ont pas de conflits d'intérêts.
Remerciements
Nous remercions FortéBio pour fournir graphiques utilisés dans la figure 1. Ce travail a été soutenu par NIH GM083088 de TMD
matériels
| Name | Company | Catalog Number | Comments |
| Octet-RED96 | Pall/FortéBio | 30-5048 | |
| Bovine Serum Albumin | Sigma | A6003-10G | Fatty Acid free |
| Biosensor/Streptavidin | Pall/FortéBio | 18-5019 | Tray of 96 sensors |
| Microtiter plate | Greiner Bio-one | 655209 | Black, Polypropylene |
| Data Acquisition software | Pall/FortéBio | Version 6.4 | Newer versions available |
| Data Analysis software | Pall/FortéBio | Version 6.4 | Newer versions available |
Références
- Nirschl, M., Reuter, F., Voros, J. Review of Transducer Principles for Label-Free Biomolecular Interaction Analysis. Biosensors. 1, 70-92 (2011).
- Homola, J. Present and future of surface plasmon resonance biosensors. Anal. Bioanal. Chem. 377, 528-539 (2003).
- Piehler, J., Brecht, A., Gauglitz, G. Affinity Detection of Low Molecular Weight Analytes. Anal. Chem. 68, 139-143 (1996).
- Duncan, T. M. The Enzymes. Energy Coupling and Molecular Motors Vol. Hackney, D. D., Tamanoi, F. XXIII, Elsevier Academic Press. 203-275 (2004).
- Feniouk, B. A., Suzuki, T., Yoshida, M. The role of subunit ε in the catalysis and regulation of FOF1-ATP synthase. Biochim. Biophys. Acta. 1757, 326-338 (2006).
- Cingolani, G., Duncan, T. M. Structure of the ATP synthase catalytic complex (F1) from Escherichia coli in an autoinhibited conformation. Nat. Struc. Mol. Biol. 18, 701-707 (2011).
- Mirsaeidi, M. After 40 years, new medicine for combating TB. Int. J. Mycobacteriol. 2, 1-2 (2013).
- Shah, N. B., Hutcheon, M. L., Haarer, B. K., Duncan, T. M. F1-ATPase of Escherichia coli: The ϵ-inhibited state forms after ATP hydrolysis, is distinct from the ADP-inhibited state, and responds dynamically to catalytic site ligands. J. Biol. Chem. 288, 9383-9395 (2013).
- Burgess, T. L., Ross, S. L., Qian, Y. -x, Brankow, D., Hu, S. Biosynthetic Processing of neu Differentiation Factor: glycosylation, trafficking, and regulated cleavage from the cell surface. J. Biol. Chem. 270, 19188-19196 (1995).
- Loo, T. W., Clarke, D. M. Membrane Topology of a Cysteine-less Mutant of Human P-glycoprotein. J. Biol. Chem. 270, 843-848 (1995).
- Tsao, K. -L., DeBarbieri, B., Michel, H., Waugh, D. S. A versatile plasmid expression vector for the production of biotinylated proteins by site-specific, enzymatic modification in Escherichia coli. Gene. 169, 59-64 (1996).
- Dayne, D. BioLayer Interferometry (BLI) - How Does it Work?. FortéBio newsletter In Interactions. 5, 8-9 (2012).
- Wartchow, C., et al. Biosensor-based small molecule fragment screening with biolayer interferometry. J. Comput. Aided Mol. Des. 25, 669-676 (2011).
- Abdiche, Y., Malashock, D., Pinkerton, A., Pons, J. Determining kinetics and affinities of protein interactions using a parallel real-time label-free biosensor, the Octet. Anal. Biochem. 377, 209-217 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon