Method Article
膨潤組織の振動イメージングと解析によるラベルフリー超解像イメージング
要約
このプロトコールでは、サンプル膨張ハイドロゲル化学とラベルフリーの化学特異的刺激ラマン散乱顕微鏡を組み合わせることで、生体サンプルでラベルフリーの超解像体積イメージングを実現する方法を説明しています。追加の機械学習画像セグメンテーションアルゴリズムにより、抗体標識のない組織でタンパク質特異的な多成分画像が得られました。
要約
蛍光顕微鏡、特に超解像顕微鏡の普遍的な利用は、現代生物学に関する知識を大きく進歩させました。逆に、蛍光法における蛍光色素の標識要件は、蛍光プローブの光退色や不均一な標識、サンプル処理の長期化など、大きな課題をもたらします。このプロトコルでは、腫れた組織の振動イメージングと分析(VISTA)の詳細な作業手順が提示されます。VISTAは、蛍光色素に関連する障害を回避し、78nmまでの空間分解能で生体サンプルのラベルフリー超解像体積イメージングを実現します。この手順は、細胞や組織をハイドロゲルに埋め込み、ハイドロゲルサンプルハイブリッドを等方的に拡大し、刺激ラマン散乱顕微鏡による振動イメージングにより内因性タンパク質分布を可視化することで確立されます。この方法は、細胞とマウスの脳組織の両方で実証されています。相関性の高いVISTA画像と免疫蛍光画像が観察され、イメージング特異性のタンパク質起源が検証されました。このような相関関係を利用して、機械学習ベースの画像セグメンテーションアルゴリズムを訓練し、ラベルフリーのマウス脳画像から核、血管、神経細胞、樹状突起の多成分予測を実現しました。この手順は、細胞内の病理学的ポリグルタミン(polyQ)凝集体および脳組織中のアミロイドベータ(Aβ)プラークを高スループットで調査するためにさらに適応され、大規模な臨床サンプルの可能性を正当化しました。
概要
光学イメージング法の開発は、細胞内タンパク質から臓器全体まで、さまざまなスケールの標的について前例のない空間的および時間的情報を提供するため、現代生物学の理解に革命をもたらしました1。その中でも、蛍光顕微鏡法は最も確立されており、高い吸光係数と量子収率2を持つ有機色素の大規模なパレット2、使いやすい遺伝学的にコードされた蛍光タンパク質3、ナノメートルスケールの構造をイメージングするためのSTED、PALM、STORMなどの超解像法4,5があります.さらに、膨潤性ポリマーハイドロゲル6,7,8に埋め込まれた標本を拡大するサンプルエンジニアリングと保存化学の最近の進歩により、従来の蛍光顕微鏡での回折限界以下の分解能が可能になります。例えば、一般的な膨張顕微鏡(ExM)は、4倍の等方性サンプル膨張7により、画像の解像度を4倍に効果的に向上させます。
その利点にもかかわらず、超解像蛍光顕微鏡は、蛍光色素の標識に由来する制限を共有しています。まず、蛍光色素の光退色と不活性化は、反復的および定量的な蛍光評価の能力を損ないます。光退色は、光が電子を電子的に励起した状態に送り続けるときに避けられないイベントです9。次に、蛍光色素を目的のターゲットに標識することは、必ずしも簡単な作業ではありません。例えば、免疫染色は長くて手間のかかるサンプル調製プロセスを必要とし、イメージングのスループットを妨げます10。また、不均一な抗体標識、特に組織の深部11によるアーチファクトを導入する可能性もあります。さらに、目的のタンパク質の蛍光色素を標的とする適切な標識戦略は未発達である可能性があります。例えば、Aβプラーク12に対する有効な抗体を見つけるためには、広範なスクリーニングが必要でした。コンゴレッドのような小さな有機色素は、特異性が限られていることが多く、Aβプラークの芯のみを染色します。したがって、蛍光色素標識の欠点を回避し、細胞から組織、さらには大規模なヒトサンプルまで補完的な高解像度イメージングを提供するラベルフリーの超解像モダリティを開発することが非常に望ましいです。
ラマン顕微鏡は、化学特異的な構造に対してラベルフリーのコントラストを提供し、励起された振動遷移13を調べることにより、他の方法では見えない化学結合の分布をマッピングします。特に、ラベルフリーまたは微小標識サンプル上の誘導ラマン散乱(SRS)イメージングは、蛍光顕微鏡法と同様の速度と分解能を有することが実証されています14,15。例えば、健康な脳領域は、ヒトおよびマウス組織において腫瘍浸潤領域から容易に区別されている16,17。Aβプラークは、標識なしで新鮮凍結脳切片上のタンパク質CH3振動(2940cm-1)およびアミドI(1660cm-1)を標的とすることによりも明確に画像化されました18。したがって、ラマン散乱は、蛍光色素の限界を克服する堅牢なラベルフリーコントラストを提供します。そこで問題となったのは、ラマン散乱を使用して超解像能力をどのように達成できるかということであり、これにより、生体試料のナノスケールの構造詳細と機能的意味を明らかにすることができました。
エレガントな光学機器を用いたラマン顕微鏡の超解像を達成するために広範な努力が払われてきたが、生物学的サンプルの分解能向上はかなり限られてきた19,20,21。ここでは、最近の研究22,23に基づいて、サンプル拡張戦略と誘導ラマン散乱を組み合わせた超解像ラベルフリー振動イメージングのためのプロトコル、Vibrational Imaging of Swelled Tissues and Analysis(VISTA)を紹介します。まず、細胞および組織を、最適化されたタンパク質-ヒドロゲルハイブリダイゼーションプロトコルを通じてハイドロゲルマトリックスに包埋した。次に、ハイドロゲル組織ハイブリッドを界面活性剤が豊富な溶液でインキュベートして脱脂し、続いて水中で膨張させました。次に、拡大したサンプルを、保持された内因性タンパク質からのCH3振動を標的とすることにより、通常のSRS顕微鏡でイメージングしました。VISTAは、ラベルフリーのイメージング機能により、蛍光色素の標識から生じる光退色や不均一な標識を回避し、サンプル処理のスループットが大幅に向上します。これは、報告された最初のサブ100 nm(78 nmまで)のラベルフリーイメージングでもあります。典型的なSRSセットアップ22,24以外の追加の光学計装は必要なく、容易に適用できます。相関VISTA画像と免疫蛍光画像を用いて、確立された機械学習画像セグメンテーションアルゴリズムを学習させ、シングルチャネル画像からタンパク質特異的なマルチプレックス画像を生成した25,26。さらに、マウス脳組織におけるAβプラークの探索にも応用し、細胞核と血管に囲まれたプラークコアと末梢フィラメントの微細な観察から、サブフェノタイピングに適した全体像を得ることができました。
プロトコル
この研究で実施されたすべての動物手順は、カリフォルニア工科大学の施設用動物管理および使用委員会(IACUC)によって承認され、プロトコル手順は関連するすべての倫理規則に準拠していました。
1. 固定およびサンプルの増殖のための原液の調製
- まず、12 gのアクリルアミド(30% w/v)固体を26 mLのヌクレアーゼフリー水に溶解して、40 mLの固定溶液を調製します。次に、10 mLの16%PFA原液を混合物に加えます。最後に、4 mLの10倍リン酸緩衝生理食塩水(PBS、pH 7.4)を加えます。調製した溶液は、4°Cで最大2週間保存できます。
注:アクリルアミドは危険であるため、アクリルアミド固体を水に溶解するステップはヒュームフードで行う必要があります。 - 70 mgのアクリル酸ナトリウム(7% w/v)、200 mgのアクリルアミド(20% w/v)、および50 μLのN、N'-メチレンビスアクリルアミド(0.1% w/v)を420 μLの超純水に溶解して、ゲル化溶液(ストックX)を調製します。滅菌ろ過した10x PBS(pH 7.4)を57 μL加え、この溶液を-20°Cで最大1週間保存します。
注:塊を形成するアクリル酸ナトリウム固体は避けてください。使用するアクリル酸ナトリウムが分散粉末の形であることを確認してください。作られたストックXは無色の液体になります。液体が淡黄色に見える場合は、新しいアクリル酸ナトリウムを入手してください。 - 1 gの過硫酸アンモニウム(APS、10%w / w)を9 mLのヌクレアーゼフリー水に溶解して、重合開始剤溶液を調製します。同様に、1 gのテトラメチルエチレンジアミン(TEMED、10%w / w)を9 mLのヌクレアーゼフリー水に溶解して重合促進剤溶液を調製します。得られた溶液を分注し、-20°Cで保存します。
- 2.88 gのドデシル硫酸ナトリウム(SDS、200 mM)、0.584 gの塩化ナトリウム(200 mM)、および2.5 mLの1M Tris-HCl緩衝液(50 mM、pH 8)をヌクレアーゼフリー水に溶解して、50 mLの変性緩衝液を調製します。
注:SDSの濃度は飽和状態に近いです。バッファー内の結晶化は正常です。37°Cまでわずかに温めると、溶液が透明になり、すぐに使用できます。
2. 哺乳動物細胞サンプルの調製
- 3.5 x 104 HeLa細胞を24ウェルプレートの12 mmホウケイ酸カバースリップ (#1.5) に播種し、400 μLのDulbecco's Modified Eagle Medium (DMEM) で10%ウシ胎児血清および1%ペニシリン-ストレプトマイシン抗生物質 (complete DMEM) を添加し、5% CO2 、37°Cの加湿雰囲気下で培養します。
- 異なる有糸分裂期の細胞を得るには、細胞を完全なDMEMで60%のコンフルエンシーになるまでインキュベートします。次に、ウシ胎児血清を含まないDMEMで細胞を20時間処理し、細胞のステージを同期させます。同期後、細胞の培地を切り替えてDMEMを完了し、さらに2〜6時間インキュベートして、有糸分裂のさまざまな段階を標的にします。
- カバースリップ上のHeLa細胞を滅菌PBSで洗浄し、固定溶液(PBS中の4%PFA、30%アクリルアミド[AA])と37°Cで7〜8時間インキュベートしてから、さらに処理します。
注:高濃度のアクリルアミドと一緒に高い固定温度は、固定プロセス中にタンパク質間の分子間架橋を消すのに役立ち、タンパク質の詳細な超微細構造を保持することが報告されています8。 - polyQ凝集体を有する細胞の場合、細胞が70%〜90%のコンフルエンシーに達するまで、完全なDMEMで細胞を増殖させます。新しい完全DMEMに変更した後、トランスフェクション剤を使用して、mHtt97Q-GFPをコードするプラスミド1 μgを細胞にトランスフェクションします(詳細は 材料表を参照)。トランスフェクションは、メーカーのプロトコールに従って行ってください。
- トランスフェクションの24〜48時間後、培地を取り出し、800μLの固定溶液(4%PFA、30%AA)を細胞のカバースリップに加えます。カバーガラスを37°Cで6〜8時間インキュベートします。カバーガラスをPBS 3xで洗浄すると、得られた細胞サンプルをさらに処理する準備が整います。
3. マウス脳サンプルの調製
- C57BL/6Jマウス(6週齢から8週齢、雌雄、マウス6匹)およびアルツハイマーマウス(生後9ヶ月、雌、マウス3匹)は、市販の情報源から購入してください(詳細は 資料表を参照)。標準プロトコル27 に従って、二酸化炭素ナルコーシスによる安楽死を行います。簡単に言えば、マウスをチャンバーに入れ、チャンバーの体積/分の30%〜70%のオーダーで100%CO2 の流れでチャンバーを満たし、臨床的死亡後少なくとも1分間の流れを維持します。
- 人道的な安楽死を確認した後、ハサミを使用してマウスをすばやく斬首します。正中線の下の皮膚を通して大きな切開で頭蓋骨を露出させます。皮膚を動物の鼻に向かって引っ張ると、頭蓋骨が完全に露出します。
- 頭蓋骨を細いハサミで正中線に沿って切り開き、頭蓋骨の半分を横に剥がします。ピンセットを使用して、PBSで氷の上のシャーレに脳をすくい取ります。新鮮なマウスの脳を氷冷PBSで洗浄し、脳を10 mLの固定緩衝液(4%PFA、30%AA)に移します。
- マウスの脳を4°Cの固定緩衝液で24〜48時間インキュベートし、その後、37°Cに一晩移します。滅菌PBSで洗浄した後、ビブラトームを使用してマウスの脳を150μmの切片に切断し、4°CでPBSに保存します。
4. 細胞および組織サンプルのハイドロゲル包埋、変性、および増殖
- 上記のように、サンプルが固定バッファー内で必要な時間インキュベートされていることを確認してください。ストックX、フリーラジカル開始剤、および加速器ストック溶液を解凍し、プロセス全体を通して氷上に保ちます。
注:インキュベーション時間が不十分な場合、ハイドロゲルハイブリッド上に保持されるタンパク質の数が減少し、シグナルが低下する可能性があります。 - セルの入ったカバースリップを手に取り、ピンセットを使用してスライドガラスの上に置きます。脳のスライス(厚さ150μm)を選び、柔らかいウールの絵筆を使用して別のスライドガラスに置き、平らであることを確認します。サンプルに付属する余分な液体を取り除きます。
- スライドガラス上のサンプルの両側に、それらの間に水滴を追加して、2つのカバースリップ(#1.5)を積み重ねてゲル化チャンバーを作ります。スライドガラスを上に向けてサンプルを氷冷ヒートブロックに置き、4°Cで1分間冷却します。
- ストックXに10%(w/w)TEMEDを添加し、次に10%APS溶液を添加します。素早くボルテックスし、混合後1分以内に、得られた溶液をゆっくりとチャンバー内に滴下し、サンプルの表面を覆い、気泡を避けます。溶液とサンプルを氷上(または氷冷ヒートブロック)に保管して、サンプルに添加する前にゲル形成(高度な重合)を避けてください。
- サンプルが溶液に完全に浸されたら、チャンバーの蓋として、サンプルの上に平らで透明なフィルムで覆われたカバースリップを置きます。チャンバーを氷上または氷冷ヒートブロックの上にさらに1分間置いてから、37°Cの加湿インキュベーターで30分間インキュベートします。
- スライドガラスのゲル化チャンバーを37°Cインキュベーターから取り出します。蓋を取り外した後、不透明なゲルが観察されるはずです。カミソリの刃で切断してゲルを回収するか、またはスライドガラスを室温で15分間変性緩衝液に入れます。ゲルはスライドガラスから分離します。
- 単離したゲルを熱条件下で変性緩衝液中でインキュベートし、さらなる変性および脱脂を実現します。細胞サンプルの場合は、95°Cで1時間変性させます。厚さ150μmの脳スライスの場合、70°Cで3時間変性し、続いて95°Cで1時間変性します。
注:ゲルは、室温で変性緩衝液と最初にインキュベートすると、巻き毛になることがあります。これは正常であり、高温変性後は平らになります。より厚い組織サンプルを扱う場合、より長い変性時間が必要になります。 - 熱処理後、変性したサンプルをPBSで3回10分間洗浄します。この時点で、ゲルは元のサイズの約1.5倍に膨張しているはずです。洗浄したゲルを大型容器(ゲルの容量の20倍以上)で超純水とインキュベートし、より高い膨張率を達成します。1時間ごとに水を3回交換し、サンプルを暗闇に一晩放置します。得られたゲルは、イメージングする準備ができています。
注:膨張率は、元のサンプルの機械的特性に大きく依存します。細胞サンプルで4.2倍、脳組織サンプルで3.4倍の拡大が得られました。
5. 拡大した細胞および組織サンプル中の内因性タンパク質分布のラベルフリーイメージング
- イメージングプロセス中は、膨張したゲルサンプルを水中に保持してください。ゲルは膨張後壊れやすいため、取り扱いには注意が必要です。膨張したサンプル含有ゲルを、適切な開口部のサイズと深さの顕微鏡スペーサーを水で満たした顕微鏡スライド(厚さ1 mm)の上に置きます。スペーサーをカバースリップ(#1.5)で覆い、サンプルが移動しないように適切に密封されていることを確認します。
- 拡張した細胞または組織のいずれかを入れたサンプルを、カバーガラスを対物レンズに向けて電動ステージに置きます。ソフトウェアの [接眼 ]をクリックして、光路を明視野に変更し、手動ノブを使用してz位置を調整します。
- サンプルの上部に液浸油を追加し、対物レンズから25倍の倍率で明視野下を観察しながら、コンデンサーをケーラー照明の正しい位置に調整します。レーザーパネルのOPO波長ウィンドウに791.3nmを入力して、2940cm-1のタンパク質CH3振動を標的とします。
- LSMボタンをクリックして顕微鏡の光を明視野(接眼レンズ)からレーザースキャンモードに切り替え、レーザーコントロールパネルのシャッターボタンを押してSRSレーザーシャッターを開きます。顕微鏡ソフトウェアのLIVEボタンを押すと、リアルタイムの画像取得が開始されます。
- 短いピクセル滞留時間 (12.5 μs/ピクセル) と低い画像解像度 (256 ピクセル x 256 ピクセル) で SRS 画像を見ながら z を変更して、適切なピントを見つけます。理想的なz位置が見つかったら、顕微鏡ソフトウェアでそれぞれのバーを引いて、スキャンサイズとピクセルドウェル時間を変更します。
- スーパーサンプリング条件(1024ピクセル×1024ピクセル)を使用し、ロックインアンプの時定数と一致する長いピクセル滞留時間(80μs/ピクセル)で LSM ボタンを押して画像を取得します。z方向に1μmのステップサイズのzスタックを収集して、体積画像を取得します。ImageJを使用して、ソフトウェアから保存されたOIRファイルを処理および分析します。
注:詳細なSRSセットアップは、最近Mutlu et al.24で報告されています。ここでは、ポンプ(770-990 nm)とストークス(1031.2 nm)の80 MHzの繰り返し周波数と2 psの帯域幅を持つ調整可能なピコ秒レーザーをレーザー走査型共焦点顕微鏡にルーティングしました(詳細については 、材料の表を参照)。純粋な D2O の信号の 2 つのビームの時間的および空間的なオーバーラップを最適化します。屈折率はゲル全体で非常に均一であるため、明視野下で膨張したサンプルに適したzを見つけるには、ある程度の努力が必要です。 - 25倍対物レンズと60倍対物レンズの両方を使用して、ポリスチレンビーズ(100 nm)のSRS画像を撮影することにより、VISTAの解像度を決定します。ポンプレーザーを784.5nmに設定し、これは3050 cm-1のラマンシフトに対応し、芳香族C-Hはポリスチレンの振動を伸ばします。
注:ここで使用されるポンプレーザーの波長は、VISTAでタンパク質に使用されるものと似ています。 - 60倍対物レンズでは、ビード像の実験的な全波半値(FWHM、振幅の半分の線形状の幅)は276.17nmでした。ビード オブジェクトの機能を半円としてモデル化します。
注:PSFガウス関数がc(σ)= 269 nm / 2.35の場合、畳み込みビーズ画像のFWHMは276.17nmで測定されます。その結果、SRSシステムの分解能は269nm×レイリー基準では1.22 = 328nmです。VISTAは細胞サンプルの平均4.2倍に増殖するため、有効分解能は328 nm/4.2 = 78 nmに低下します。
6. 免疫標識および拡大組織サンプルの相関VISTAおよび蛍光イメージング
- 変性後、ハイドロゲル包埋サンプル(例:150 μm 脳冠状切片)を 1% (v/v) Triton X-100 (PBST) で 15 分間プレインキュベートします。インキュベーションバッファーを、1:100希釈の一次抗体を含むPBSTに切り替えます。マルチプレックスイメージングに複数のタンパク質ターゲットが必要な場合は、異なるターゲットに対するそれぞれの一次抗体を同じカクテルで適切な濃度に希釈し、サンプルと同時にインキュベートします。
- 希釈した一次抗体とゲルを37°Cでインキュベートし、80 rpmで16〜18時間穏やかに振とうした後、PBSTで3回3回、37°Cで1〜2時間広範囲に洗浄します。 インキュベーション中にゲルを裏返して、ここおよびその後のすべてのインキュベーションで不均一な抗体標識を防ぎます。
- サンプルを、対応する分子種ターゲットの二次抗体と1:100希釈で、PBSTを37°Cで12〜16時間インキュベートし、光から保護します。標識したヒドロゲルサンプルをPBSTで3回、37°C、1〜2時間、穏やかに振とうしながら洗浄します。
- DAPIをPBSの最終濃度3μMに希釈します。サンプルハイドロゲルに十分な量のDAPI溶液を加えて、ゲルが溶液に浸るようにします。80 rpmで穏やかに振とうしながら、室温で1〜2時間インキュベートします。サンプルをPBSで3回洗浄します。
- 免疫標識ゲルサンプルを、大量の二重脱イオンH2Oとインキュベートして増殖させ、1時間ごとに水を3回交換し、サンプルをH2Oで一晩インキュベートし、光から保護します。標識中、ゲルはPBSバッファーと比較して1.5倍に膨張する必要があります。
- 手順5.1の説明に従ってイメージングサンプルを準備します。蛍光イメージング用の25倍、1.05 NAの水浸対物レンズを備えたレーザー走査型共焦点顕微鏡にイメージングサンプルを置きます。
- 顕微鏡ソフトウェアのドロップダウンメニューで、それぞれの励起レーザー(405 nm、488 nm、561 nm、640 nm)とPMTペアで、標的抗体に従って適切なチャンネルを選択します。LIVEボタンを押すと、リアルタイムの画像取得が可能です。
- リアルタイムの蛍光信号に基づく手動ノブでフォーカス位置を調整します。顕微鏡ソフトウェアのレーザー出力、ピクセル滞留時間、PMTゲインをリアルタイムの蛍光信号に応じて最適化し、信号の薄暗さや過飽和を回避します。
注:報告されたさまざまな抗体の構造に基づいて外観と特徴を評価し、潜在的な抗体の交差反応性を排除し、異なる蛍光チャネル間のクロストークを回避します。 - LSMボタンを押すと、相関SRS画像と蛍光画像の取得が開始されます。まず、免疫標識サンプル上の体積蛍光画像を収集します。蛍光イメージングが終了したら、顕微鏡の光路を赤外(IR)透明状態に切り替えます。
- 顕微鏡ソフトウェアで検出チャネルを蛍光PMTからSRSに変更します。SRSレーザーのシャッターを開き、同じz範囲の同じサンプルの視野でSRSボリュームイメージングを実行します。正常範囲は100〜200μmで、最大700μmまで上がることができます。
注:励起レーザーの波長差により、蛍光画像とSRS画像の間でz位置がずれるわずかな色収差があります。SRSと蛍光zスタック画像を手動で並べて比較する必要があります。SRSと蛍光チャネルの両方から、z位置に正確に一致する特徴を探します。
7. U-Netアーキテクチャの構築・訓練・検証
注: Linux でのインストールをお勧めします。>10GBのRAMを搭載したグラフィックカードが必要です。
- 環境のセットアップ
- Anaconda または Miniconda を 3-5.3.0-linux-x86_64 にインストールします。次のファイルをクローンまたはダウンロードします:https://github.com/Li-En-Good/VISTA。次のコマンド・ラインを使用して、プラットフォームのConda環境を作成します:
conda env create -f environment.yml
- Anaconda または Miniconda を 3-5.3.0-linux-x86_64 にインストールします。次のファイルをクローンまたはダウンロードします:https://github.com/Li-En-Good/VISTA。次のコマンド・ラインを使用して、プラットフォームのConda環境を作成します:
- 予測モデルのトレーニング
- 対応する SRS 画像のディレクトリを csv ファイル内のグラウンド トゥルース画像とペアリングします。SRS 画像のディレクトリを path_signal の下に配置して、グラウンド トゥルース画像を path_target 列の下に配置します。
- csvファイルをdata/csvsフォルダに入れます。必要に応じて、scripts/s train_model_2d.sh の設定を変更します。コマンドラインを使用して環境をアクティブ化します。
conda activate fnet - コマンドラインを使用してモデルトレーニングを開始します。
./scripts/train_model_2d.sh 0
その後、トレーニングが開始されます。各反復の損失はコマンドラインに表示され、csv file> のフォルダ save_models/
- コマンド ラインを使用して、トレーニング セットとテスト セット内の画像を検証します。
./scripts/train_model_2d.sh 0
予測結果は、csvファイルの名前>のresults/
8. VISTAとU-Netの併用によるラベルフリー画像におけるタンパク質特異的多重性
- data/csvs//test.csv で csv ファイルを変更します。path_signal と path_target の両方のディレクトリを、新しい SRS イメージのディレクトリに置き換えます。
- トレーニングから予測結果 (csv file> の results/
./scripts/train_model_2d.sh 0
予測結果は、csv file>/testのresults/<ファイル名のフォルダに保存されます。
結果
イメージングおよび分析法の動作原理を確立した後、膨張率を評価し、サンプル処理中の等方性膨張を確保するために、画像レジストレーションが行われました(図1A、B)。未処理のサンプルとVISTAサンプルの両方を、内因性タンパク質のCH3に由来する2940 cm-1の結合振動を標的にしてイメージングしました。未処理のサンプルでは、核のようなタンパク質に富む構造は、周囲の組織からの圧倒的な脂質含有量のために暗かった22(図1A)。脱脂処理を含むサンプル処理後、得られた画像は、コントラストが逆になった同じ特徴を示しました(図1B)。核と血管の形状と相対的な位置はまったく変更されておらず(図1A、B、番号付き構造)、処理が等方性プロセスであることが確認されました。対応する核のサイズを比較することにより、この方法は、未処理のサンプルと比較して脳組織サンプルの3.4倍の拡大を達成すると結論付けました22,23。
脳組織の拡大率を知ることで、VISTAは、以前は解決できなかったラベルフリーのSRS画像の新機能を解決できるようになりました。アクチンおよびチューブリン構造は超解像デモンストレーションのゴールドスタンダードでしたが、アクチンおよびチューブリン構造の分解能向上は、同様のハイブリダイゼーションケミストリーを使用した蛍光ベースのサンプル膨張戦略によって十分に特徴付けられています28。さらに、この手法では、シグナルが内因性タンパク質の全アンサンブルから来ているため、特定のアクチン/チューブリン構造のイメージングは、チューブリンのような細胞骨格構造を明確に区別するのに十分なコントラスト(シグナル対バックグラウンド比)を持たないため、実現性が低くなります。そこで、他のナノスケール構造のイメージングを追求することにしました。マウスの皮質から150 nmまで特徴を捕捉できることを示しました(図1C、D)。ニューロン樹状突起の周りの分散パターンに基づいて、観察された小さな構造は、146nmのサイズを持つ樹状突起脊椎頭部7である可能性が高い(図1D)。さらに、この方法は、厚さが約100 nm29,30であると考えられているAβプラークの線維構造を画像化するために使用されました。実際、この方法を用いて、代表的な拡散性Aβプラークにおいて~130 nmの線維構造を分解できることが実証されました(図1E、F)。
VISTAは効果的なタンパク質保持とタンパク質イメージングを可能にするため22、核内のタンパク質リッチ核小体と培養HeLa細胞のサイトゾル内のリボン状細胞骨格構造を明確に区別できます(図2A、矢印)。この方法は、哺乳類細胞で一過性に発現するポリグルタミン(polyQ)凝集体の研究にさらに適用されました(図2B、C)。その結果、凝集体は、予想通り高密度に充填された構造として、複数の反復サンプル23にわたって膨張前後の同じ凝集構造を比較することにより、等方的に膨張したことが確認された。この方法により、通常解像度のSRS画像にはない/ぼやけている高解像度の構造が得られました。VISTA凝集体画像では、polyQ凝集体の周辺に線維様の突起があり、中央に中空構造があることが明らかになりました(図2B、矢印)。突起が細胞質の内容物にシームレスに付着するという観察は、凝集体が細胞質の機能タンパク質と関与していることを示唆している可能性があります。後から考えると、固定試薬のホルムアルデヒドとヒドロゲルモノマーのアクリルアミドおよびアクリル酸ナトリウムはすべて、タンパク質凝集体に出入りする小さな分子であるため、高密度の凝集体を膨張させる能力も妥当になります。凝集体がモノマーと共重合してヒドロゲルになると、膨張プロセスは通常どおり進行するはずです。
その後、この手法をマウスの脳組織に適用し、さらにその範囲を広げました。組織サンプルには、透過性の減少、厚さの増加、不均一な機械的強度などの課題がありますが、この方法を使用してマウスの脳サンプルをうまくイメージングすることができました(図2D)。細胞サンプルと同様に、細胞核、血管、ニューロンプロセスなどのタンパク質に富む構造が観察されました(図2D、矢印)。脳組織の限界は、3.4倍の拡大しか達成されなかったため、脳サンプルの有効分解能は99nm22になることです。SRSシグナルの構造起源は、相関色素と抗体染色により検証し、DAPIは核を染色し、レクチンは血管を染色しました(図2E、F)。ニューロンの細胞体とプロセスは、NeuNおよびMAP2からの免疫蛍光法によっても描写されました22。相関蛍光画像をグラウンドトゥルースとして利用する訓練された畳み込みニューラルネットワーク(CNN)アルゴリズムにより、シングルチャネル画像は、マルチプレックス画像22のために特定のタンパク質構造チャネルに分割された。
最後に、アルツハイマー病の動物モデルとしてよく知られている5xFADマウスの脳内の病理学的Aβプラークを調べることを目指しました31。手順に従った後、脳組織に沈着したアミロイドプラークの3次元SRS画像を取得しました(図2G)。高タンパク質濃度の点が観察され(図2G、オレンジ色の矢印)、Aβプラークのコアを表しています。このような画像は、Aβコアのみを標的とする従来のコンゴレッド染色では見過ごされがちな周辺Aβプラーク(図2G、マゼンタの矢印)も明らかにしました。訓練されたセグメンテーションアルゴリズムと組み合わせると、ラベルフリー画像を標的特異的なマルチプレックス画像に変換でき(図2F)、免疫蛍光法23 と共同で実施して、プラーク-アストロサイトおよびプラーク-ミクログリア微小環境相互作用32 を包括的かつハイスループットな方法で研究することができる。
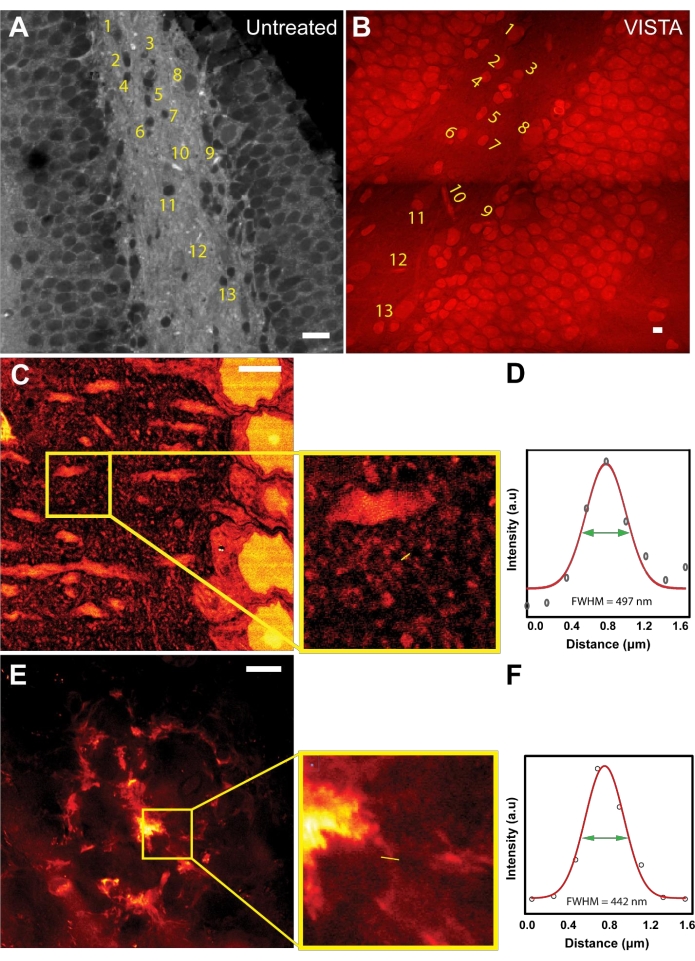
図1:サンプル拡大戦略により、マウス脳組織における超解像ラベルフリーイメージングが可能に (A) マウス海馬のCH3 周波数でのSRS画像。(B)マウス海馬の同じ視野内のVISTA画像。ラベル付き領域は、治療前と治療後に対応する特徴を示しています。(C)より細かい特徴を示す正常なマウス皮質のVISTA画像。挿入図は、関心領域を示します。(D)膨張したサンプルで観察された微細構造の分解能定量化。497nmのFWHMは、3.4倍の膨張で146nmの有効分解能に相当します。(E)マウス脳組織におけるアミロイドベータ(Aβ)プラークのVISTA画像。挿入図は、拡大された関心領域を示しています。(F)膨張したアミロイドベータプラークの押出繊維構造の分解能定量化。442nmのFWHMは、3.4倍の膨張で130nmの有効分解能に相当します。スケールバー = 20 μm. この図の拡大版を表示するには、ここをクリックしてください。
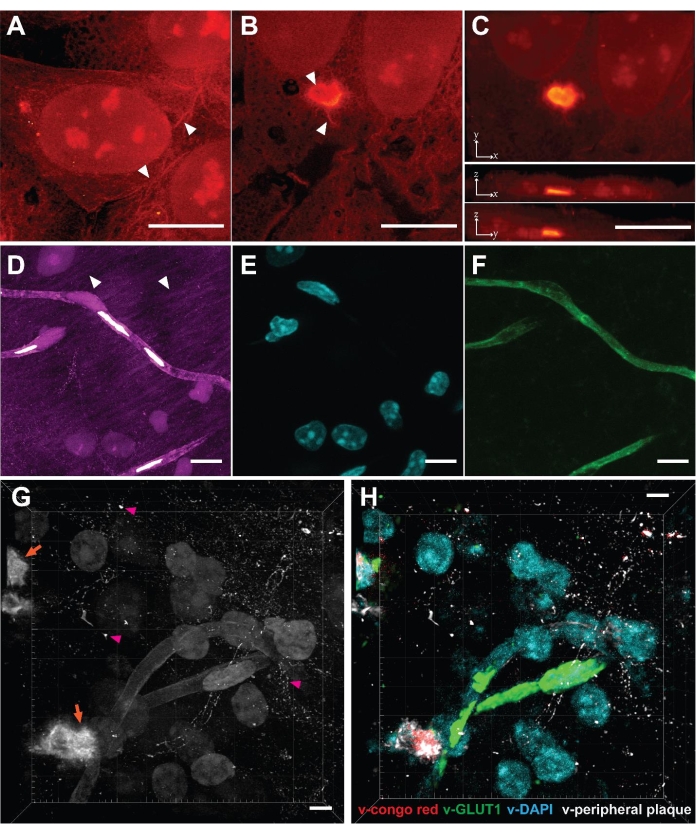
図2:VISTAによって可能になった細胞および組織におけるラベルフリーの超解像体積イメージング(A)正常なHeLa細胞の体積画像。アローヘッド:細胞骨格様構造。(B)HeLa細胞で発現したpolyQ凝集体の単一のzスライス画像。矢印:中空構造とフィブリル押出成形。(C)x-y、x-z、およびy-z方向の最大強度投影は、polyQ凝集体含有セルの体積ビューを示しています。(D)マウス脳の冠状切片の体積画像。矢印:ニューロンのプロセス。(E)同じサンプル領域での核の蛍光画像(DAPI染色)は、VISTA画像の核と1対1の相関を示しています。(F)同じ試料領域の血管(抗レクチン)の蛍光画像は、VISTA画像の血管構造と1対1の相関を示しています。(G)脳組織を含むAβ(オレンジ色の矢印)の体積画像。ピンクの矢印:周辺のAβプラーク。(H)訓練された画像セグメンテーションアルゴリズムによって予測された(G)からの多重画像。v-congo redはAβプレートのコアを表しています。v-GLUT1は血管を表します。v-DAPIは原子核を表します。v末梢プラークは、コンゴレッド色素で染色されていないAβプラークを表しています。スケールバー = 10 μm。長さのスケールは、拡張前の距離で表されます(さまざまな拡張比率に合わせて調整されます)。この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
要約すると、細胞や組織のタンパク質に富む細胞および細胞内構造を画像化するためのラベルフリーモダリティであるVISTAのプロトコールを紹介します。この方法は、ハイドロゲルに包埋された細胞や組織のタンパク質から内因性CH3を標的とすることにより、生体サンプル中の78 nmまでの効果的なイメージング分解能を達成し、Aβプラーク中のハンチンチン凝集体およびフィブリルのわずかな押し出しを解決します。この技術は、ラベルフリーイメージングモダリティ22で100nm未満の解像度を報告した最初の例です。既存の拡張方法6,7,8,28,33,34と比較して、この技術はラベルフリーSRSイメージングのメリットを継承しているため、レーザー照明による光退色、不活性化、または消光がありません。さらに、ラベルフリーの方法として、DISCO 12,35 や ExM33,34 などの方法に常に関与する、要求が厳しく、非効率的で、アーティファクトを引き起こす可能性のある抗体標識を回避し、ハイスループットなサンプル調製と組織全体の均一なイメージングを提供します。ラベルフリーアプローチにおける多重性の欠如に対処するために、CNNベースの画像セグメンテーションアルゴリズム25で実装されたVISTAは、脳組織22に任意の標識なしでタンパク質特異的な多成分画像を提供する。この方法はさらに5xFADマウス脳に適用され、凝集体、コアおよび末梢線維、核、および血管の全体像の体積ビューを可能にした23。私たちは、VISTAが霊長類やヒトの脳スライスなどのより大きなサンプルに対しても十分にスケールアップし、最終的には臨床研究に役立つと想定しています。
この方法を成功させるためには、3つの重要なステップがあります。まず、ハイドロゲルサンプルハイブリッドにおける最大タンパク質保持が重要であり、必要である22。この目標を達成するために、固定条件を高濃度のアクリルアミド28を含有するように変更し、タンパク質消化手順を高濃度の界面活性剤脱脂処理に置き換えることで、タンパク質消化によるタンパク質損失を大幅に防ぎました。AAの添加は、タンパク質の分子間架橋を消光し、タンパク質消化を伴わない等方性拡大を可能にする28。以前の研究では、アクリルアミドハイドロゲル中の脂肪族CH結合が一定のバックグラウンドのみを生じさせることを証明するために、重水素化モノマーが使用されました22。次に、SRSと免疫標識との間の適切な相関関係、および異なるタンパク質標的間の区別を確立する必要があります。この手法は、モノクロSRS画像に多重性を追加するために画像セグメンテーションアルゴリズムに依存しているため、免疫蛍光法における異なるタンパク質ターゲット間のクロストークは、画像の品質を大幅に損なうことになります。SRS画像で明らかなタンパク質に富む構造を細心の注意を払って選択し、それらに対応する免疫蛍光特性を検証しました。第 3 に、モデルを使用して新しい SRS データ セットから蛍光パターンを予測する前に、トレーニング済みの機械学習モデルの有効性と信頼性を証明する必要があります。トレーニング セットに含まれていない個別の特徴は、予測に問題を引き起こす可能性があります。予測結果が満足のいくものでない場合、ユーザーはトレーニングにより多くのデータを含めるようにし、トレーニング セットに含まれていないパターンの予測を避ける必要があります。Pearsonのテストセットと検証セットの相関も、予測22,23の品質を確保するために監視する必要があります。トレーニングには、少なくとも 100 個の対応する画像セットを用意することをお勧めします。
この方法は生物学的研究に計り知れない可能性を秘めていますが、創造的な解決策を待っている一定の制限があります。何よりもまず、感度をさらに向上させる必要があります。ラベルフリーの模擬ラマン散乱の検出限界は低ミリモル範囲であり、サンプルの3次元での等方性膨張は化学結合を大幅に希釈し、シグナルを弱めます。したがって、私たちは内因性タンパク質の全アンサンブルをイメージングすることに限定されており、特異性と多重性に欠けています。VISTAと超高感度SRSを組み合わせると、これを低存在量のタンパク質を画像化し、直交する化学結合を標的とすることにより超解像レベルで凝集構造と組成を研究するために拡張できる可能性があります37。第二に、現在の脳組織の3.4倍の拡張比では、解像度は中程度の改善にとどまります。以前は区別できなかったAβプラークの小さな押し出しをすでに解決していますが、より高い分解能が常に望まれます。この場合、タンパク質アンカーとハイドロゲル化学の革新が大きな恩恵を受けるでしょう。例えば、異なるゲル配合により、さらに高い画像解像度38,39,40に対してより大きな膨張比を可能にすることができる。サンプル処理の新しい手順により、広く利用可能なFFPE組織学サンプル38,41に適用できるようになり、大規模な臨床研究にさらに適しています。
開示事項
著者は、競合する利益を宣言しません。
謝辞
私たちは、ソフトウェアサポートについてCaltech Biological Imaging Facilityに感謝します。L.W.は、国立衛生研究所(NIH Director's New Innovator Award、DP2 GM140919-01)、アムジェン社(Amgen Early Innovation Award)、およびカリフォルニア工科大学からのスタートアップ資金の支援に感謝しています。
資料
| Name | Company | Catalog Number | Comments |
| 1.0 M Tris pH 8 | Sigma-Aldrich | 648314 | |
| 16% Paraformaldehyde | Electron microscopy science | 15710 | diluted to 4% in PBS |
| 25x water immersion objective | Olympus | XLPLN25XWMP2 | NA 1.05 |
| 5XFAD Mice | Mutant Mouse Resource and Research Centers and the Jackson Laboratory | B6SJL-Tg (APPSwFlLon, PSEN1*M146L*L286 V) 6799Vas/Mmjax | Alzheimer brain |
| 60x water immersion objective | Olympus | UPLSAPO60XWIR | NA 1.2 |
| Acrylamide | Sigma-Aldrich | A9099 | |
| ammonium persulfate | Sigma-Aldrich | A3678 | |
| anti-MAP2 | Cell Signaling Technology | 8707 | |
| anti-NeuN | Cell Signaling Technology | 24307 | |
| borosilicate coverslip #1.5 | Fisher Scientific | 1254581 | |
| C57BL/6J Mice | Jackson Laboratory (JAX) | 664 | Normal mice |
| D2O | Sigma-Aldrich | 151882 | for SRS calibration |
| DAPI | Thermo Fisher | D1306 | |
| DMEM | GIBCO | 10566-016 | |
| FBS | GIBCO | A4766 | |
| glass slide 3" x 1" x 1 mm | VWR | 16004-430 | |
| goat anti-chicken IgY, Alexa Fluor 647 | Invitrogen | A-21449 | |
| goat anti-mouse IgG, Alexa Fluor 647 | Invitrogen | A-21236 | |
| goat anti-rabbit IgG, Alexa Fluor 488 | Invitrogen | A-11034 | |
| goat anti-rat IgG, Alexa Fluor 568 | Invitrogen | A-11077 | |
| Grace Bio-Labs Press-To-Seal silicone isolators | Sigma-Aldrich | GBL664108 | microscope spacer |
| Htt-97Q-GFP Plasmid | Gift from Prof. R. Kopito and Prof. F.-U.Hartl. | ||
| Laser scanning microscope | Olympus | FV3000 | laser scanning confocal microscope |
| lipofectamine 3000 | Thermo Fisher | L3000001 | transfection agent |
| Lycopersicon Esculentum Lectin DyLight®594 (lectin) | Vector Laboratories | DL-1177-1 | |
| Microscope spacer | Grace Bio-Labs | 621502 | |
| N,N′-methylenebisacrylamide (BIS) | Sigma-Aldrich | M1533 | bought as 2% solution in water |
| Nuclease free water | Thermo Fisher | 10977-015 | |
| Penicillin-Streptomycin | GIBCO | 15140-122 | |
| poly-strene beads | Sigma-Aldrich | 43302 | for resolution characterization |
| Sodium Acrylate | Sigma-Aldrich | 408220 | |
| sodium dodecyl sulfate | Sigma-Aldrich | 71725 | |
| soft-wool paint brush #3 | TANIS | 000333 | |
| SRS Laser | A.P.E | picoEmerald | 2ps pulse width |
| tetramethylethylenediamine | Sigma-Aldrich | T9281 | |
| Tissue culture flask 25 cm2 | Corning | 430639 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween-20 | Sigma-Aldrich | P9416 | |
| tweezer | Fine Science Tool | 11295-51 | |
| Vibrotome | Leica | VT1200S | the vibratome |
参考文献
- Ntziachristos, V. Going deeper than microscopy: The optical imaging frontier in biology. Nature Methods. 7 (8), 603-614 (2010).
- Lavis, L. D., Bright Raines, R. T. ideas for chemical biology. ACS Chemical Biology. 3 (3), 142-155 (2008).
- Tsien, R. Y. Constructing and exploiting the fluorescent protein paintbox (Nobel Lecture). Angewandte Chemie (International Edition in English). 48 (31), 5612-5626 (2009).
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annual Review of Biochemistry. 78, 993-1016 (2009).
- Sahl, S. J., Hell, S. W., Jakobs, S. Fluorescence nanoscopy in cell biology. Nature Reviews Molecular Cell Biology. 18 (11), 685-701 (2017).
- Wassie, A. T., Zhao, Y., Boyden, E. S. Expansion microscopy: Principles and uses in biological research. Nature Methods. 16 (1), 33-41 (2019).
- Chen, F., Tillberg, P. W., Boyden, E. S. Expansion microscopy. Science. 347 (6221), 543-548 (2015).
- Gambarotto, D., et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nature Methods. 16 (1), 71-74 (2019).
- Demchenko, A. P. Photobleaching of organic fluorophores: Quantitative characterization, mechanisms, protection. Methods and Applications in Fluorescence. 8 (2), 022001(2020).
- Murray, E., et al. scalable proteomic imaging for high-dimensional profiling of intact systems. Cell. 163 (6), 1500-1514 (2015).
- Kim, S. -Y., et al. Stochastic electrotransport selectively enhances the transport of highly electromobile molecules. Proceedings of the National Academy of Sciences. 112 (46), 6274-6283 (2015).
- Liebmann, T., et al. Three-dimensional study of Alzheimer's disease hallmarks using the IDISCO clearing method. Cell Report. 16 (4), 1138-1152 (2016).
- Min, W., Freudiger, C. W., Lu, S., Xie, X. S. Coherent nonlinear optical imaging: Beyond fluorescence microscopy. Annual Review of Physical Chemistry. 62 (1), 507-530 (2011).
- Saar, B. G., et al. Video-rate molecular imaging in vivo with stimulated Raman scattering. Science. 330 (6009), 1368-1370 (2010).
- Cheng, J. -X., Xie, X. S. Vibrational spectroscopic imaging of living systems: An emerging platform for biology and medicine. Science. 350 (6264), 1054-1063 (2015).
- Ji, M., et al. Rapid, label-free detection of brain tumors with stimulated Raman scattering microscopy. Science Translational Medicine. 5 (201), (2013).
- Wei, M., et al. Volumetric chemical imaging by clearing-enhanced stimulated Raman scattering microscopy. Proceedings of the National Academy of Sciences. 116 (14), 6608-6617 (2019).
- Ji, M., et al. Label-free imaging of amyloid plaques in Alzheimer's disease with stimulated Raman scattering microscopy. Science Advances. 4 (11), 39(2018).
- Silva, W. R., Graefe, C. T., Frontiera, R. R. Toward label-free super-resolution microscopy. ACS Photonics. 3 (1), 79-86 (2016).
- Gong, L., Zheng, W., Ma, Y., Huang, Z. Higher-order coherent anti-stokes Raman scattering microscopy realizes label-free super-resolution vibrational imaging. Nature Photonics. 14 (2), 115-122 (2020).
- Watanabe, K., et al. Structured line illumination Raman microscopy. Nature Communication. 6 (1), 10095(2015).
- Qian, C., et al. Super-resolution label-free volumetric vibrational imaging. Nature Communications. 12 (1), 3648(2021).
- Lin, L. -E., Miao, K., Qian, C., Wei, L. High spatial-resolution imaging of label-free in vivo protein aggregates by VISTA. Analyst. 146 (13), 4135-4145 (2021).
- Mutlu, A. S., Chen, T., Deng, D., Wang, M. C. Label-free imaging of lipid storage dynamics in Caenorhabditis elegans using stimulated Raman scattering microscopy. Journal of Visualized Experiments. (171), e61870(2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nature Methods. 15 (11), 917-920 (2018).
- Falk, T., et al. U-Net: Deep learning for cell counting, detection, and morphometry. Nature Methods. 16 (1), 67-70 (2019).
- Yang, B., et al. Single-cell phenotyping within transparent intact tissue through whole-body clearing. Cell. 158 (4), 945-958 (2014).
- Ku, T., et al. Multiplexed and scalable super-resolution imaging of three-dimensional protein localization in size-adjustable tissues. Nature Biotechnology. 34 (9), 973-981 (2016).
- Mlodzianoski, M. J., et al. Active PSF shaping and adaptive optics enable volumetric localization microscopy through brain sections. Nature Methods. 15 (8), 583-586 (2018).
- Querol-Vilaseca, M., et al. Nanoscale structure of amyloid-β plaques in Alzheimer's disease. Scientific Reports. 9 (1), 5181(2019).
- Oakley, H., et al. Intraneuronal beta-amyloid aggregates, neurodegeneration, and neuron loss in transgenic mice with five familial Alzheimer's disease mutations: Potential factors in amyloid plaque formation. Journal of Neuroscience. 26 (40), 10129-10140 (2006).
- Bartels, T., De Schepper, S., Hong, S. Microglia modulate neurodegeneration in Alzheimer's and Parkinson's diseases. Science. 370, 66-69 (2020).
- Chen, F., et al. Nanoscale imaging of RNA with expansion microscopy. Nature Methods. 13 (8), 679-684 (2016).
- Tillberg, P. W., et al. Protein-retention expansion microscopy of cells and tissues labeled using standard fluorescent proteins and antibodies. Nature Biotechnology. 34 (9), 987-992 (2016).
- Renier, N., et al. IDISCO: A simple, rapid method to immunolabel large tissue samples for volume imaging. Cell. 159 (4), 896-910 (2014).
- Zhuge, M., et al. Ultrasensitive vibrational imaging of retinoids by visible preresonance stimulated Raman scattering microscopy. Advanced Science. 8 (9), 2003136(2021).
- Miao, K., Wei, L. Live-cell imaging and quantification of PolyQ aggregates by stimulated Raman scattering of selective deuterium labeling. ACS Central Science. 6 (4), 478-486 (2020).
- Klimas, A., et al. Nanoscale imaging of biomolecules using molecule anchorable gel-enabled nanoscale in-situ fluorescence microscopy. Research Square. , (2021).
- Shi, L., et al. Super-resolution vibrational imaging using expansion stimulated Raman scattering microscopy. Advanced Science. , (2022).
- M'Saad, O., et al. All-optical visualization of specific molecules in the ultrastructural context of brain tissue. bioRxiv. , (2022).
- Zhao, Y., et al. Nanoscale imaging of clinical specimens using pathology-optimized expansion microscopy. Nature Biotechnology. 35 (8), 757-764 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved