Method Article
通过肿胀组织的振动成像和分析实现无标记超分辨率成像
摘要
通过将样品扩增水凝胶化学与无标记化学特异性刺激拉曼散射显微镜相结合,该方案描述了如何在生物样品中实现无标记超分辨率体积成像。使用额外的机器学习图像分割算法,可以在没有抗体标记的组织中获得蛋白质特异性多组分图像。
摘要
荧光显微镜,尤其是超分辨率显微镜的普遍应用,极大地推动了现代生物学的知识。相反,荧光技术中荧光团标记的要求带来了重大挑战,例如荧光探针的光漂白和不均匀标记以及样品处理时间延长。在该协议中,介绍了肿胀组织振动成像和分析 (VISTA) 的详细工作程序。VISTA 绕过了与荧光基团相关的障碍,并在生物样品中实现了空间分辨率低至 78 nm 的无标记超分辨率体积成像。该程序是通过将细胞和组织包埋在水凝胶中,各向同性扩增水凝胶样品杂交体,并通过受激拉曼散射显微镜的振动成像可视化内源性蛋白质分布来建立的。该方法在细胞和小鼠脑组织上均得到验证。观察到高度相关的 VISTA 和免疫荧光图像,验证了成像特异性的蛋白质来源。利用这种相关性,训练了一种基于机器学习的图像分割算法,以实现对无标记小鼠大脑图像中的细胞核、血管、神经元细胞和树突的多组分预测。该程序进一步适用于研究细胞中的病理性聚谷氨酰胺 (polyQ) 聚集体和脑组织中的淀粉样蛋白 β (Aβ) 斑块,具有高通量,证明了其用于大规模临床样本的潜力。
引言
光学成像方法的发展彻底改变了对现代生物学的理解,因为它们提供了从亚细胞蛋白到整个器官的不同尺度上靶标的前所未有的空间和时间信息1。其中,荧光显微镜是最成熟的,拥有大量具有高消光系数和量子产率的有机染料2、易于使用的遗传编码荧光蛋白3,以及用于纳米级结构成像的超分辨率方法,如 STED、PALM 和 STORM 4,5.此外,样品工程和保存化学的最新进展扩大了包埋在可溶胀聚合物水凝胶中的样品 6,7,8,使传统荧光显微镜的亚衍射极限分辨率成为可能。例如,典型的扩增显微镜 (ExM) 通过四倍各向同性样品扩增7 有效地将图像分辨率提高了四倍。
尽管具有优势,但超分辨率荧光显微镜存在荧光团标记带来的局限性。首先,荧光团的光漂白和失活损害了重复和定量荧光评估的能力。当光不断将电子泵入电子激发态时,光漂白是不可避免的事件9。其次,将荧光基团标记到所需的靶标并不总是一件容易的事。例如,免疫染色需要漫长而费力的样品制备过程,并阻碍成像通量10。由于抗体标记不均匀,它还可能引入伪影,尤其是组织深处11。此外,靶向所需蛋白质荧光团的适当标记策略可能尚未开发。例如,需要进行广泛的筛查才能找到针对 Aβ 斑块的有效抗体12。较小的有机染料,如刚果红,通常特异性有限,只能对 Aβ 噬菌斑的核心进行染色。因此,非常需要开发一种无标记的超分辨率模式,以规避荧光团标记的缺点,并提供从细胞到组织,甚至到大规模人类样品的互补高分辨率成像。
拉曼显微镜为化学特异性结构提供无标记的对比度,并通过观察激发的振动跃迁来绘制原本不可见的化学键的分布图13。特别是,无标记或微小标记样品上的受激拉曼散射 (SRS) 成像已被证明具有与荧光显微镜相似的速度和分辨率14,15。例如,在人类和小鼠组织中,健康的大脑区域很容易与肿瘤浸润区域区分开来 16,17。通过在新鲜冷冻的脑切片上靶向蛋白 CH3 振动 (2940 cm-1) 和酰胺 I (1660 cm-1),在没有任何标记的情况下,Aβ 斑块也清晰成像18。因此,拉曼散射提供了强大的无标记对比度,克服了荧光团的局限性。然后,问题就变成了如何使用拉曼散射来实现超分辨率能力,拉曼散射可以揭示生物样品中的纳米级结构细节和功能意义。
尽管人们已经做出了广泛的努力来使用优雅的光学仪器实现拉曼显微镜的超分辨率,但生物样品的分辨率增强相当有限 19,20,21。在这里,基于最近的工作22,23,我们提出了一种协议,该协议将样品扩展策略与受激拉曼散射相结合,用于超分辨率无标记振动成像,称为肿胀组织的振动成像和分析 (VISTA)。首先,通过优化的蛋白质-水凝胶杂交方案将细胞和组织包埋在水凝胶基质中。然后将水凝胶组织杂交体在富含去污剂的溶液中孵育以进行脱脂,然后在水中膨胀。然后,通过常规 SRS 显微镜靶向保留内源性蛋白质的 CH3 振动,对扩增的样品进行成像。VISTA 由于其无标记成像功能,绕过了荧光团标记引起的光漂白和不均匀标记,具有更高的样品处理通量。这也是首次报道的亚 100 nm(低至 78 nm)无标记成像。除了典型的 SRS 设置22,24 之外,不需要额外的光学仪器,使其易于应用。使用相关 VISTA 和免疫荧光图像,训练了一种已建立的机器学习图像分割算法25,26 从单通道图像生成蛋白质特异性多重图像。该方法进一步应用于研究小鼠脑组织中的 Aβ 斑块,根据细胞核和血管包围的斑块核心和外周细丝的精细视图提供适合亚表型的整体图像。
研究方案
本研究中执行的所有动物程序均已获得加州理工学院机构动物护理和使用委员会 (IACUC) 的批准,并且协议程序符合所有相关的伦理法规。
1. 制备用于固定和样品扩增的储备液
- 首先将 12 g 丙烯酰胺 (30% w/v) 固体溶解在 26 mL 无核酸酶水中,制备 40 mL 固定溶液。然后,向混合物中加入 10 mL 16% PFA 储备液。最后,加入 4 mL 10x 磷酸盐缓冲盐水 (PBS,pH 7.4)。制备的溶液可在 4 °C 下储存长达 2 周。
注意:丙烯酰胺是危险的,因此将丙烯酰胺固体溶解在水中的步骤应在通风橱中完成。 - 通过将 70 mg 丙烯酸钠 (7% w/v)、200 mg 丙烯酸酰胺 (20% w/v) 和 50 μL N,N′-亚甲基双丙烯酰胺 (0.1% w/v) 溶解在 420 μL 超纯水中来制备凝胶溶液(原液 X)。最后加入 57 μL 无菌过滤的 10x PBS (pH 7.4),并将该溶液在 -20 °C 下储存长达 1 周。
注:避免使用会形成团块的丙烯酸钠固体;确保使用的丙烯酸钠为分散粉末。制成的原液 X 将是无色液体;如果液体呈浅黄色,则获取新的丙烯酸钠。 - 将 1 g 过硫酸铵(APS,10% w/w)溶解在 9 mL 无核酸酶水中,制备聚合引发剂溶液。同样,通过将 1 g 四甲基乙二胺(TEMED,10% w/w)溶解在 9 mL 无核酸酶的水中来制备聚合促进剂溶液。分装所得溶液并储存在 -20 °C。
- 将 2.88 g 十二烷基硫酸钠(SDS,200 mM)、0.584 g 氯化钠 (200 mM) 和 2.5 mL 1M Tris-HCl 缓冲液(50 mM,pH 8)溶于无核酸酶的水中,制备 50 mL 变性缓冲液。
注:SDS 的浓度接近其饱和条件。缓冲液中的一些结晶是正常的。将其略微加热至 37 °C 可使溶液澄清并立即使用。
2. 哺乳动物细胞样品的制备
- 将 3.5 x 104 个 HeLa 细胞接种到 24 孔板中的 12 mm 硼硅酸盐盖玻片 (#1.5) 上,然后在 400 μL 含有 10% 胎牛血清和 1% 青霉素-链霉素抗生素(完全 DMEM)的 Dulbecco 改良 Eagle 培养基 (DMEM) 中培养细胞,在 5% CO2 的潮湿气氛中,37 °C。
- 为了获得处于不同有丝分裂阶段的细胞,将细胞在完全 DMEM 中孵育至 60% 汇合。然后,用不含胎牛血清的 DMEM 处理细胞 20 小时以同步细胞阶段。同步后,切换细胞培养基以完成 DMEM 并再孵育 2-6 小时以靶向有丝分裂的各个阶段。
- 用无菌PBS洗涤盖玻片上的HeLa细胞,并在37°C下与固定液(4%PFA,30%丙烯酰胺[AA]的PBS溶液)在37°C下孵育7-8小时,然后进一步处理。
注:较高的固定温度与高浓度的丙烯酰胺一起有助于在固定过程中淬灭蛋白质之间的分子间交联,据报道可以保留蛋白质的详细超结构8。 - 对于具有 polyQ 聚集体的细胞,在完整的 DMEM 中培养细胞,直到它们达到 70%-90% 汇合。更换为新的完全 DMEM 后,使用转染试剂将 1 μg 编码 mHtt97Q-GFP 的质粒转染到细胞中(详情见 材料表)。根据制造商的方案进行转染。
- 转染 24-48 小时后,去除培养基,向带有细胞的盖玻片中加入 800 μL 固定液(4% PFA、30% AA)。将盖玻片在 37 °C 下孵育 6-8 小时。用 PBS 3x 清洗盖玻片,所得细胞样品即可用于进一步处理。
3. 小鼠脑样品的制备
- 从商业来源购买 C57BL/6J 小鼠(6 至 8 周龄,雄性和雌性,6 只小鼠)和阿尔茨海默病小鼠(9 个月大,雌性,3 只小鼠)(详情见 材料表)。根据标准方案27 通过二氧化碳麻醉进行安乐死。简而言之,将小鼠置于腔室中,并以腔室体积/分钟的 30%-70% 的量级用 100% CO2 的流量填充腔室,并在临床死亡后保持流量至少 1 分钟。
- 确认人道安乐死后,用剪刀迅速将老鼠斩首。从中线向下穿过皮肤,露出一个大切口的颅骨。将皮肤拉向动物的鼻子,以完全露出头骨。
- 用细剪刀沿中线切开头骨,然后将头骨的两半剥到一边。用镊子将大脑舀入装有 PBS 的冰培养皿中。用冰冷的 PBS 洗涤新鲜的小鼠大脑,并将大脑转移到 10 mL 固定缓冲液(4% PFA、30% AA)中。
- 将小鼠大脑在4°C的固定缓冲液中孵育24-48小时,然后将其转移至37°C过夜。用无菌 PBS 洗涤后,使用振动切片机将小鼠大脑切成 150 μm 切片,并在 4 °C 的 PBS 中储存。
4. 细胞和组织样品的水凝胶包埋、变性和扩增
- 确保样品在固定缓冲液中孵育所需的时间,如上所述。解冻原液 X、自由基引发剂和促进剂原液,并在整个过程中将它们保存在冰上。
注:孵育时间不足可能导致水凝胶杂交体上保留的蛋白质数量减少,从而导致信号减弱。 - 拿起带有细胞的盖玻片,用镊子将其放在载玻片上。挑选脑切片(150 μm 厚)并使用软羊毛画笔将它们放在另一个载玻片上,确保它们是平坦的。去除样品中多余的液体。
- 将两个盖玻片 (#1.5),通过在它们之间添加水滴,在载玻片上样品的两侧堆叠,以形成凝胶室。将载玻片样品朝上放在冰冷的加热块上,并将其冷却至 4 °C 1 分钟。
- 将 10% (w/w) TEMED 添加到原液 X 中,然后加入 10% APS 溶液。快速涡旋,并在混合后 1 分钟内,将所得溶液缓慢滴入腔室中,以覆盖样品表面,避免气泡。在添加到样品中之前,将溶液和样品保存在冰上(或冰冷的加热块)上,以避免形成凝胶(高度聚合)。
- 样品完全浸入溶液中后,将一个平坦、透明、覆盖有薄膜的盖玻片放在样品顶部作为腔室的盖子。将腔室放在冰上或冰冷的加热块上再放置 1 分钟,然后在 37 °C 的加湿培养箱中孵育 30 分钟。
- 从37°C培养箱中取出载玻片上的凝胶室。取下盖子后应观察不透明的凝胶。通过用剃须刀片切割来取回凝胶,或者,将载玻片放入室温下的变性缓冲液中 15 分钟。凝胶会从载玻片上分离出来。
- 在加热条件下将分离的凝胶在变性缓冲液中孵育,以实现进一步的变性和脱脂。对于细胞样品,在 95 °C 下变性 1 小时。对于 150 μm 厚的脑切片,在 70 °C 下变性 3 小时,然后在 95 °C 下变性 1 小时。
注:首次在室温下与变性缓冲液一起孵育时,凝胶可能会变成卷曲。属正常现象,高温变性后会变平。当处理较厚的组织样品时,需要更长的变性时间。 - 热处理后,用 PBS 洗涤变性样品 3 次,每次 10 分钟。此时,凝胶应该已经膨胀到原始大小的 1.5 倍左右。将洗涤后的凝胶与超纯水在大容器(至少是凝胶体积的 20 倍)中孵育,以获得更高的膨胀率。每 1 小时 3 次换水,并将样品在黑暗中放置过夜。所得凝胶可直接进行成像。
注:膨胀率在很大程度上取决于原始样品的机械性能。细胞样品的扩增率为 4.2 倍,脑组织样品的扩增率为 3.4 倍。
5. 扩增细胞和组织样品中内源性蛋白质分布的无标记成像
- 在成像过程中,将膨胀的凝胶样品保存在水中。凝胶在膨胀后很脆弱,因此应谨慎处理。将扩增的含样品凝胶放在显微镜载玻片(1 mm 厚)上,并带有具有适当开口尺寸和深度的显微镜垫片,并装满水。用盖玻片 (#1.5) 盖住垫片,并确保其正确密封以避免样品移动。
- 将带有扩增细胞或组织的样品放在电动载物台上,盖玻片朝向物镜。单击软件上的 Ocular 将光路更改为明场并使用手动旋钮调整 z 位置。
- 在样品顶部添加浸油,并将聚光镜调整到正确的位置以进行科勒照明,同时在物镜的 25 倍放大倍率下在明场下观察。在激光面板上的 OPO 波长窗口中输入 791.3 nm,以靶向 2940 cm-1 处的蛋白质 CH3 振动。
- 单击 LSM 按钮将显微镜的光线从明场(目镜)切换到激光扫描模式,然后通过点击激光控制面板上的 快门 按钮打开 SRS 激光快门。按下显微镜软件中的 LIVE 按钮开始实时图像采集。
- 在较短的像素停留时间 (12.5 μs/像素) 和低图像分辨率 (256 像素 x 256 像素) 下,通过更改 z 值来查看 SRS 图像,从而找到合适的焦点。找到理想的 z 位置后,通过拉动显微镜软件中的相应条来更改扫描大小和像素停留时间。
- 使用超级采样条件(1024 像素 x 1024 像素)获取图像,像素停留时间较长(80 μs/像素),与锁相放大器的时间常数相匹配。 通过在 z 方向上收集步长为 1 μm 的 z 堆栈来获取体积图像。使用 ImageJ 处理和分析来自软件保存的 OIR 文件。
注意:最近的 Mutlu 等人24 报道了详细的 SRS 设置。在这里,将具有 80 MHz 重复频率和 2 ps 带宽的可调谐皮秒激光器(泵浦 (770-990 nm) 和斯托克斯 (1031.2 nm) 路由到激光扫描共聚焦显微镜中(详见 材料表)。优化纯 D2O 信号的两束的时间和空间重叠。在明场下为膨胀的样品找到合适的 z 值需要付出一些努力,因为整个凝胶的折射率非常均匀。 - 通过使用 25 倍物镜和 60 倍物镜拍摄聚苯乙烯珠 (100 nm) 的 SRS 图像来确定 VISTA 的分辨率。将泵浦激光器设置为 784.5 nm,对应于 3050 cm-1 的拉曼位移,这是聚苯乙烯芳香族 C-H 拉伸振动的特征。
注:此处使用的泵浦激光波长与 VISTA 中用于蛋白质的波长相似。 - 使用 60 倍物镜时,磁珠图像的实验全波半最大值(FWHM,即振幅一半处线形的宽度)为 276.17 nm。将拉延筋对象的功能建模为半圆。
注意:当 PSF 高斯函数的 c (σ) = 269 nm/2.35 时,卷积珠子图像的测量 FWHM 为 276.17 nm。因此,根据瑞利准则,SRS 系统的分辨率为 269 nm × 1.22 = 328 nm。由于 VISTA 的平均扩增是细胞样品的 4.2 倍,因此有效分辨率低至 328 nm/4.2 = 78 nm。
6. 免疫标记和扩增组织样品的 VISTA 和荧光成像
- 变性后,将水凝胶包埋样品(例如,150 μm 脑冠状切片)在 1% (v/v) Triton X-100 (PBST) 中预孵育 15 分钟。将孵育缓冲液以 1:100 稀释度的一抗切换到 PBST。如果多重成像需要多个蛋白质靶标,请在同一混合物中将不同靶标的相应一抗稀释至适当的浓度,并同时与样品一起孵育。
- 将凝胶与稀释的一抗在 37 °C 下孵育,以 80 rpm 轻轻摇动 16-18 小时,然后在 37 °C 下用 PBST 充分洗涤 3 次 1-2 小时。 在孵育过程中翻转凝胶,以防止此处和所有后续孵育中出现不均匀的抗体标记。
- 将样品与相应物种靶标的二抗以 1:100 稀释度与 PBST 在 37 °C 下孵育 12-16 小时,避光。用 PBST 在 37 °C 下洗涤标记的水凝胶样品 3 次,轻轻摇动 1-2 小时。
- 在 PBS 中将 DAPI 稀释至 3 μM 终浓度。向样品水凝胶中加入足够体积的 DAPI 溶液,使凝胶浸没在溶液中。在室温下孵育 1-2 小时,以 80 rpm 轻轻摇动。用 PBS 洗涤样品 3 次。
- 通过与大量双去离子 H2O 孵育来扩增免疫标记的凝胶样品。每 1 小时 3 次换水,并将样品在 H2O 中孵育过夜,避光。在标记过程中,凝胶应比 PBS 缓冲液膨胀 1.5 倍。
- 按照步骤 5.1 中的说明准备成像样品。将成像样品放在带有 25 倍、1.05 NA 水浸物镜的激光扫描共聚焦显微镜上,以进行荧光成像。
- 根据显微镜软件下拉菜单中的靶向抗体,使用相应的激发激光(405 nm、488 nm、561 nm 和 640 nm)和 PMT 对选择合适的通道。按下 LIVE 按钮进行实时图像采集。
- 通过手动旋钮根据实时荧光信号调整焦点位置。根据实时荧光信号,在显微镜软件中优化激光功率、像素驻留时间和 PMT 增益,以避免信号变暗或过饱和。
注:根据报告的不同抗体的结构评估外观和特征,以消除潜在的抗体交叉反应性并避免不同荧光通道之间的串扰。 - 点击 LSM 按钮开始采集相关 SRS 和荧光图像。首先,在免疫标记的样品上收集体积荧光图像。荧光成像完成后,将显微镜光路切换到红外 (IR) 透明状态。
- 在显微镜软件上将检测通道从荧光 PMT 更改为 SRS。打开 SRS 激光器的快门,并在具有相同 z 范围的同一样品的视场上执行 SRS 体积成像。正常范围在 100-200 μm 之间,最高可达 700 μm。
注意:由于激发激光器的波长差异,存在轻微的色差,导致荧光图像和 SRS 图像之间的 z 位置偏移。需要在 SRS 和荧光 z 堆栈图像之间进行手动并排比较。从 SRS 和荧光通道中寻找精确匹配的特征,以精确匹配 z 位置。
7. U-Net 架构的构建、培训和验证
注意:建议在 Linux 上安装。需要具有 >10 GB RAM 的显卡。
- 设置环境
- 在 3-5.3.0-linux-x86_64上安装 Anaconda 或 Miniconda。克隆或下载以下文件:https://github.com/Li-En-Good/VISTA。使用以下命令行为平台创建 Conda 环境:
conda env create -f environment.yml
- 在 3-5.3.0-linux-x86_64上安装 Anaconda 或 Miniconda。克隆或下载以下文件:https://github.com/Li-En-Good/VISTA。使用以下命令行为平台创建 Conda 环境:
- 训练预测模型
- 将相应 SRS 图像的目录与 csv 文件中的真实图像配对。将 SRS 图像的目录放在 path_signal 下,将 ground truth 图像放在 path_target 列下。
- 将 csv 文件放在文件夹 data/csvs 中。如果需要,请在 scripts/train_model_2d.sh 中修改配置。使用命令行激活环境:
Conda 激活 FNET - 使用命令行启动模型训练:
./scripts/train_model_2d.sh 0
然后,训练将开始。每次迭代的损失将显示在命令行中,并与模型一起保存在 csv 文件>的文件夹 save_models/
- 使用命令行验证训练集和测试集中的图像:
./scripts/train_model_2d.sh 0
预测结果将保存在 csv 文件的 results/<文件名 > 文件夹中。
8. VISTA 结合 U-Net 预测无标记图像中蛋白质特异性多重性
- 在 data/csvs/< csv 文件的文件名>/test.csv 中修改 csv 文件。将 path_signal 和 path_target 的目录替换为新的 SRS 映像目录。
- 从训练中删除预测结果,即 csv 文件的文件夹 results/。使用命令行运行预测:
./scripts/train_model_2d.sh 0
预测结果将保存在 csv 文件的 results//test 文件夹中。
结果
在确定了成像和分析方法的工作原理后,进行了图像配准以评估膨胀率并确保样品处理过程中的各向同性膨胀(图 1A、B)。未处理样品和 VISTA 样品均在靶向 2940 cm-1 处的键振动时进行成像,该振动源自内源性蛋白质的 CH3。在未经处理的样品中,由于周围组织的脂质含量过多,细胞核等富含蛋白质的结构是深色的 22(图 1A)。在包括脱脂处理的样品处理后,所得图像显示出相同的特征,但具有反向对比度(图 1B)。细胞核和血管的形状和相对位置完全没有改变(图 1A、B;编号结构),证实了该处理是一个各向同性过程。通过比较相应细胞核的大小,得出结论,与未处理的样品相比,该方法在脑组织样品中的扩增增加了 3.4倍 22,23。
了解脑组织中的扩增率后,VISTA 现在可以分辨无标记 SRS 图像中以前无法分辨的新特征。尽管肌动蛋白和微管蛋白结构一直是超分辨率演示的金标准,但肌动蛋白和微管蛋白结构的分辨率改进已经通过使用类似的杂交化学的基于荧光的样品扩增策略得到了很好的表征28。此外,使用这种技术对特异性肌动蛋白/微管蛋白结构进行成像不太可行,因为信号来自内源性蛋白质的总集合,其中像微管蛋白这样的细胞骨架结构没有足够的对比度(信噪比)来清楚地区分。因此,我们决定对其他纳米级结构进行成像。我们表明可以从低至 150 nm 的小鼠皮层中捕获特征(图 1C、D)。根据神经元树突周围的分散模式,观察到的小结构可能是树突棘头7,其大小为 146 nm(图 1D)。此外,该方法还用于对 Aβ 斑块中的纤维结构进行成像,据信其厚度约为 100 nm29,30。事实上,已经证明使用这种方法可以在代表性的扩散性 Aβ 斑块中解析 ~130 nm 的纤维结构(图 1E,F)。
由于 VISTA 能够有效地保留蛋白质和蛋白质成像22,因此可以清楚地区分细胞核中富含蛋白质的核仁和培养的 HeLa 细胞胞质溶胶中的带状细胞骨架结构(图 2A,箭头)。该方法进一步应用于研究在哺乳动物细胞中瞬时表达的聚谷氨酰胺 (polyQ) 聚集体(图 2B、C)。结果证实,通过比较多个重复样品中膨胀前后的相同聚集体结构,聚集体作为预期的密集堆积结构进行了各向同性膨胀23。使用这种方法获得了正常分辨率 SRS 图像中不存在/模糊的高分辨率结构。VISTA 聚集体图像显示 polyQ 聚集体外围有原纤维状突起,中心有一个空心结构(图 2B,箭头)。突起无缝附着在胞质内容物上的观察结果可能表明聚集体与胞质溶胶中的功能性蛋白结合。事后看来,膨胀致密聚集体的能力也变得合理,因为固定试剂甲醛和水凝胶单体丙烯酸酰胺和丙烯酸钠都是可以在蛋白质聚集体中扩散的小分子。一旦聚集体与单体共聚成水凝胶,膨胀过程应照常进行。
然后,我们将该方法应用于小鼠脑组织,以进一步扩展其范围。尽管组织样本存在渗透性降低、厚度增加和机械强度不均等挑战,但使用这种方法成功地对小鼠脑样本进行了成像(图 2D)。与细胞样品类似,观察到富含蛋白质的结构,包括细胞核、血管和神经元过程(图 2D,箭头)。脑组织的局限性在于仅实现了 3.4 倍的扩增,这使得脑样本中的有效分辨率为 99 nm22。我们通过相关染料和抗体染色验证了 SRS 信号的结构起源,其中细胞核的 DAPI 染色和血管的凝集素染色(图 2E、F)。还通过来自 NeuN 和 MAP2 的免疫荧光描绘神经元细胞体和过程22。使用经过训练的卷积神经网络 (CNN) 算法,利用相关荧光图像作为真实数据,然后将单通道图像分割成特定的蛋白质结构通道以进行多路复用图像22。
最后,我们旨在询问 5xFAD 小鼠大脑中的病理性 Aβ 斑块,这是一种著名的阿尔茨海默病动物模型31。遵循这些程序后,获得了淀粉样蛋白斑块在脑组织中沉积的 3 维 SRS 图像(图 2G)。观察到高蛋白质浓度的 Puncta (图 2G,橙色箭头),代表 Aβ 斑块的核心。该图像还揭示了外周 Aβ 斑块(图 2G,洋红色箭头),这些斑块通常被仅针对 Aβ 核心的传统刚果红染色所忽略。当与经过训练的分割算法相结合时,无标记图像可以转化为靶标特异性多重图像(图 2F),并且可以与免疫荧光23 联合进行,以全面和高通量的方式研究斑块-星形胶质细胞和斑块-小胶质细胞微环境相互作用32 。
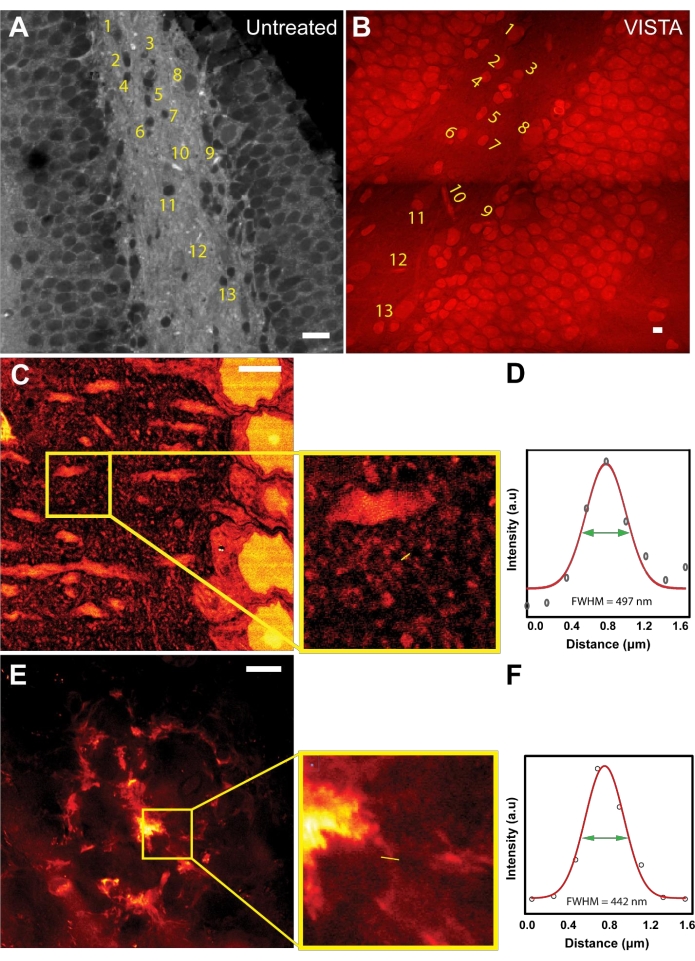
图 1:样品扩展策略可在小鼠脑组织中实现超分辨率无标记成像。 (A) 小鼠海马体 CH3 频率的 SRS 图像。(B) 小鼠海马体同一视野中的 VISTA 图像。标记区域显示处理前后的相应特征。(C) 显示更精细特征的正常小鼠皮层的 VISTA 图像。Inset 显示感兴趣的区域。(D) 在扩增样品中观察到的精细结构的分离度定量。497 nm 的 FWHM 对应于 146 nm 的有效分辨率,扩展率为 3.4 倍。(E) 小鼠脑组织中 β 淀粉样蛋白 (Aβ) 斑块的 VISTA 图像。插图显示扩大的感兴趣区域。(F) 扩增的淀粉样蛋白-β 斑块的挤出纤维结构的分辨率定量。442 nm 的 FWHM 对应于 130 nm 的有效分辨率,扩展为 3.4 倍。比例尺 = 20 μm。 请点击此处查看此图的较大版本。
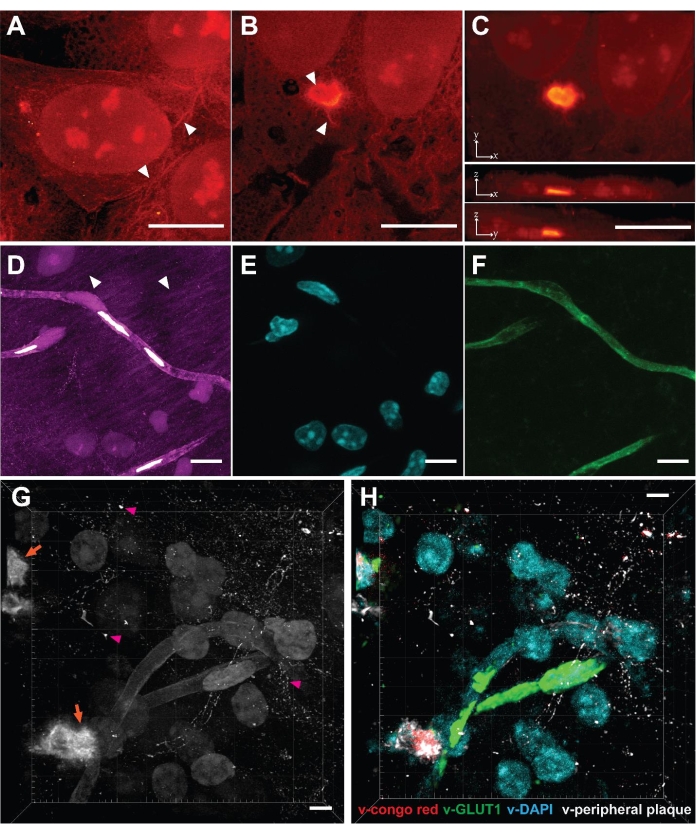
图 2:VISTA 实现的细胞和组织中无标记超分辨率体积成像。 (A) 正常 HeLa 细胞的体积图像。箭头:细胞骨架样结构。(B) 在 HeLa 细胞中表达的 polyQ 聚集体的单个 z 切片图像。箭头:中空结构和原纤维挤压。(C) x-y、x-z 和 y-z 方向的最大强度投影显示了包含 polyQ 聚集体的像元的体积视图。(D) 小鼠大脑冠状切片的体积图像。箭头:神经元过程。(E) 同一样品区域的细胞核荧光图像(DAPI 染色)与 VISTA 图像中的细胞核呈 1 比 1 的相关性。(F) 同一样品区域的血管(抗凝集素)的荧光图像与 VISTA 图像中的血管结构呈 1 比 1 的相关性。(G) 含有脑组织的 Aβ(橙色箭头)的体积图像。粉红色箭头:外周 Aβ 斑块。(H) 来自 (G) 的多路复用图像,由经过训练的图像分割算法预测。v-刚果红代表 Aβ 斑块的核心;v-GLUT1 代表血管;v-DAPI 代表原子核;v 外周斑块表示未被刚果红染料染色的 Aβ 斑块。比例尺 = 10 μm。长度刻度是根据膨胀前的距离(根据不同的膨胀率进行调整)。 请单击此处查看此图的较大版本。
讨论
总之,我们提出了 VISTA 的方案,这是一种无标记模式,用于对细胞和组织的富含蛋白质的细胞和亚细胞结构进行成像。通过靶向水凝胶包埋的细胞和组织中蛋白质的内源性 CH3,该方法在生物样品中实现了低至 78 nm 的有效成像分辨率,并解决了 Aβ 噬菌斑中亨廷顿聚集体和原纤维的轻微挤出。该技术是第一个报告无标记成像模式的分辨率低于 100 nm 的实例22。与现有的扩增方法 6,7,8,28,33,34 相比,该技术继承了无标记 SRS 成像的优点,因此不会因激光照射而发生光漂白、灭活或淬灭。此外,作为一种无标记方法,它避免了 DISCO 12,35 和 ExM33,34 等方法中始终涉及的要求高、效率低下且可能引起伪影的抗体标记,因此,提供了高通量样品制备和整个组织的均匀成像。为了解决无标记方法中缺乏多路复用的问题,使用基于 CNN 的图像分割算法25 实现的 VISTA 提供了蛋白质特异性多组分图像,在脑组织中没有任何标记22。该方法进一步应用于 5xFAD 小鼠大脑,并实现了聚集体核心和外周原纤维、细胞核和血管的整体体积视图23。我们设想 VISTA 将很好地扩大到更大的样本,例如灵长类动物或人脑切片,并最终可用于临床研究。
有三个基本步骤可以确保成功实施此方法。首先,水凝胶样品杂交体中的最大蛋白质保留率至关重要,并且是必需的22.为了实现这一目标,将固定条件修改为含有高浓度的丙烯酰胺28,并用高浓度去污剂脱脂代替蛋白质消化程序,从而避免蛋白质消化造成的大量蛋白质损失。添加 AA 可淬灭蛋白质的分子间交联,并在不消化蛋白质的情况下实现各向同性扩增28。在之前的一项研究中,氘代单体用于证明丙烯酰胺水凝胶中的脂肪族 CH 键仅产生恒定的背景22。其次,需要建立 SRS 和免疫标记之间的适当相关性以及不同蛋白质靶标之间的区别。由于该方法依赖于图像分割算法为单色 SRS 图像增加多路性,因此免疫荧光中不同蛋白质靶标之间的串扰将显着影响图像质量。我们精心挑选了在 SRS 图像中明显的富含蛋白质的结构,并验证了它们相应的免疫荧光特征。第三,在使用模型预测新 SRS 数据集的荧光模式之前,应验证经过训练的机器学习模型的有效性和可靠性。训练集中未包含的不同特征可能会导致预测问题。如果预测结果不令人满意,用户应尝试包含更多数据进行训练,并避免预测训练集中未包含的模式。还应监控测试集和验证集的 Pearson 相关性,以确保预测的质量22,23。建议至少有 100 个对应的 image set 用于训练。
虽然该方法在生物学研究方面具有巨大潜力,但仍存在一些限制,等待创造性的解决方案。首先,灵敏度需要进一步提高。无标记模拟拉曼散射的检出限在低毫摩尔范围内,样品在三维空间中的各向同性膨胀显著稀释了化学键并减弱了信号。因此,我们仅限于对缺乏特异性和多重性的内源性蛋白质的整个集合进行成像。将 VISTA 与超灵敏 SRS36 相结合,可能会将其扩展到对低丰度蛋白质进行成像,并通过靶向正交化学键37 在超分辨率水平上研究聚集体结构和组成。其次,目前脑组织中 3.4 倍的扩展比只能提供适度的分辨率提升。尽管我们已经解决了以前无法区分的 Aβ 斑块中的微小挤压,但更高的分辨率始终是可取的。在这种情况下,蛋白质锚定和水凝胶化学的创新将受益匪浅。例如,不同的凝胶配方可以实现更大的膨胀率,以获得更高的图像分辨率 38,39,40。样本处理的新程序将使其能够应用于广泛可用的 FFPE 组织学样本38,41,使其更适合大规模临床研究。
披露声明
作者声明没有利益冲突。
致谢
我们感谢加州理工学院生物成像设施提供的软件支持。L.W. 感谢美国国立卫生研究院(NIH 主任新创新者奖,DP2 GM140919-01)、安进(安进早期创新奖)和加州理工学院启动资金的支持。
材料
| Name | Company | Catalog Number | Comments |
| 1.0 M Tris pH 8 | Sigma-Aldrich | 648314 | |
| 16% Paraformaldehyde | Electron microscopy science | 15710 | diluted to 4% in PBS |
| 25x water immersion objective | Olympus | XLPLN25XWMP2 | NA 1.05 |
| 5XFAD Mice | Mutant Mouse Resource and Research Centers and the Jackson Laboratory | B6SJL-Tg (APPSwFlLon, PSEN1*M146L*L286 V) 6799Vas/Mmjax | Alzheimer brain |
| 60x water immersion objective | Olympus | UPLSAPO60XWIR | NA 1.2 |
| Acrylamide | Sigma-Aldrich | A9099 | |
| ammonium persulfate | Sigma-Aldrich | A3678 | |
| anti-MAP2 | Cell Signaling Technology | 8707 | |
| anti-NeuN | Cell Signaling Technology | 24307 | |
| borosilicate coverslip #1.5 | Fisher Scientific | 1254581 | |
| C57BL/6J Mice | Jackson Laboratory (JAX) | 664 | Normal mice |
| D2O | Sigma-Aldrich | 151882 | for SRS calibration |
| DAPI | Thermo Fisher | D1306 | |
| DMEM | GIBCO | 10566-016 | |
| FBS | GIBCO | A4766 | |
| glass slide 3" x 1" x 1 mm | VWR | 16004-430 | |
| goat anti-chicken IgY, Alexa Fluor 647 | Invitrogen | A-21449 | |
| goat anti-mouse IgG, Alexa Fluor 647 | Invitrogen | A-21236 | |
| goat anti-rabbit IgG, Alexa Fluor 488 | Invitrogen | A-11034 | |
| goat anti-rat IgG, Alexa Fluor 568 | Invitrogen | A-11077 | |
| Grace Bio-Labs Press-To-Seal silicone isolators | Sigma-Aldrich | GBL664108 | microscope spacer |
| Htt-97Q-GFP Plasmid | Gift from Prof. R. Kopito and Prof. F.-U.Hartl. | ||
| Laser scanning microscope | Olympus | FV3000 | laser scanning confocal microscope |
| lipofectamine 3000 | Thermo Fisher | L3000001 | transfection agent |
| Lycopersicon Esculentum Lectin DyLight®594 (lectin) | Vector Laboratories | DL-1177-1 | |
| Microscope spacer | Grace Bio-Labs | 621502 | |
| N,N′-methylenebisacrylamide (BIS) | Sigma-Aldrich | M1533 | bought as 2% solution in water |
| Nuclease free water | Thermo Fisher | 10977-015 | |
| Penicillin-Streptomycin | GIBCO | 15140-122 | |
| poly-strene beads | Sigma-Aldrich | 43302 | for resolution characterization |
| Sodium Acrylate | Sigma-Aldrich | 408220 | |
| sodium dodecyl sulfate | Sigma-Aldrich | 71725 | |
| soft-wool paint brush #3 | TANIS | 000333 | |
| SRS Laser | A.P.E | picoEmerald | 2ps pulse width |
| tetramethylethylenediamine | Sigma-Aldrich | T9281 | |
| Tissue culture flask 25 cm2 | Corning | 430639 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween-20 | Sigma-Aldrich | P9416 | |
| tweezer | Fine Science Tool | 11295-51 | |
| Vibrotome | Leica | VT1200S | the vibratome |
参考文献
- Ntziachristos, V. Going deeper than microscopy: The optical imaging frontier in biology. Nature Methods. 7 (8), 603-614 (2010).
- Lavis, L. D., Bright Raines, R. T. ideas for chemical biology. ACS Chemical Biology. 3 (3), 142-155 (2008).
- Tsien, R. Y. Constructing and exploiting the fluorescent protein paintbox (Nobel Lecture). Angewandte Chemie (International Edition in English). 48 (31), 5612-5626 (2009).
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annual Review of Biochemistry. 78, 993-1016 (2009).
- Sahl, S. J., Hell, S. W., Jakobs, S. Fluorescence nanoscopy in cell biology. Nature Reviews Molecular Cell Biology. 18 (11), 685-701 (2017).
- Wassie, A. T., Zhao, Y., Boyden, E. S. Expansion microscopy: Principles and uses in biological research. Nature Methods. 16 (1), 33-41 (2019).
- Chen, F., Tillberg, P. W., Boyden, E. S. Expansion microscopy. Science. 347 (6221), 543-548 (2015).
- Gambarotto, D., et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nature Methods. 16 (1), 71-74 (2019).
- Demchenko, A. P. Photobleaching of organic fluorophores: Quantitative characterization, mechanisms, protection. Methods and Applications in Fluorescence. 8 (2), 022001 (2020).
- Murray, E., et al. scalable proteomic imaging for high-dimensional profiling of intact systems. Cell. 163 (6), 1500-1514 (2015).
- Kim, S. -. Y., et al. Stochastic electrotransport selectively enhances the transport of highly electromobile molecules. Proceedings of the National Academy of Sciences. 112 (46), 6274-6283 (2015).
- Liebmann, T., et al. Three-dimensional study of Alzheimer's disease hallmarks using the IDISCO clearing method. Cell Report. 16 (4), 1138-1152 (2016).
- Min, W., Freudiger, C. W., Lu, S., Xie, X. S. Coherent nonlinear optical imaging: Beyond fluorescence microscopy. Annual Review of Physical Chemistry. 62 (1), 507-530 (2011).
- Saar, B. G., et al. Video-rate molecular imaging in vivo with stimulated Raman scattering. Science. 330 (6009), 1368-1370 (2010).
- Cheng, J. -. X., Xie, X. S. Vibrational spectroscopic imaging of living systems: An emerging platform for biology and medicine. Science. 350 (6264), 1054-1063 (2015).
- Ji, M., et al. Rapid, label-free detection of brain tumors with stimulated Raman scattering microscopy. Science Translational Medicine. 5 (201), (2013).
- Wei, M., et al. Volumetric chemical imaging by clearing-enhanced stimulated Raman scattering microscopy. Proceedings of the National Academy of Sciences. 116 (14), 6608-6617 (2019).
- Ji, M., et al. Label-free imaging of amyloid plaques in Alzheimer's disease with stimulated Raman scattering microscopy. Science Advances. 4 (11), 39 (2018).
- Silva, W. R., Graefe, C. T., Frontiera, R. R. Toward label-free super-resolution microscopy. ACS Photonics. 3 (1), 79-86 (2016).
- Gong, L., Zheng, W., Ma, Y., Huang, Z. Higher-order coherent anti-stokes Raman scattering microscopy realizes label-free super-resolution vibrational imaging. Nature Photonics. 14 (2), 115-122 (2020).
- Watanabe, K., et al. Structured line illumination Raman microscopy. Nature Communication. 6 (1), 10095 (2015).
- Qian, C., et al. Super-resolution label-free volumetric vibrational imaging. Nature Communications. 12 (1), 3648 (2021).
- Lin, L. -. E., Miao, K., Qian, C., Wei, L. High spatial-resolution imaging of label-free in vivo protein aggregates by VISTA. Analyst. 146 (13), 4135-4145 (2021).
- Mutlu, A. S., Chen, T., Deng, D., Wang, M. C. Label-free imaging of lipid storage dynamics in Caenorhabditis elegans using stimulated Raman scattering microscopy. Journal of Visualized Experiments. (171), e61870 (2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nature Methods. 15 (11), 917-920 (2018).
- Falk, T., et al. U-Net: Deep learning for cell counting, detection, and morphometry. Nature Methods. 16 (1), 67-70 (2019).
- Yang, B., et al. Single-cell phenotyping within transparent intact tissue through whole-body clearing. Cell. 158 (4), 945-958 (2014).
- Ku, T., et al. Multiplexed and scalable super-resolution imaging of three-dimensional protein localization in size-adjustable tissues. Nature Biotechnology. 34 (9), 973-981 (2016).
- Mlodzianoski, M. J., et al. Active PSF shaping and adaptive optics enable volumetric localization microscopy through brain sections. Nature Methods. 15 (8), 583-586 (2018).
- Querol-Vilaseca, M., et al. Nanoscale structure of amyloid-β plaques in Alzheimer's disease. Scientific Reports. 9 (1), 5181 (2019).
- Oakley, H., et al. Intraneuronal beta-amyloid aggregates, neurodegeneration, and neuron loss in transgenic mice with five familial Alzheimer's disease mutations: Potential factors in amyloid plaque formation. Journal of Neuroscience. 26 (40), 10129-10140 (2006).
- Bartels, T., De Schepper, S., Hong, S. Microglia modulate neurodegeneration in Alzheimer's and Parkinson's diseases. Science. 370, 66-69 (2020).
- Chen, F., et al. Nanoscale imaging of RNA with expansion microscopy. Nature Methods. 13 (8), 679-684 (2016).
- Tillberg, P. W., et al. Protein-retention expansion microscopy of cells and tissues labeled using standard fluorescent proteins and antibodies. Nature Biotechnology. 34 (9), 987-992 (2016).
- Renier, N., et al. IDISCO: A simple, rapid method to immunolabel large tissue samples for volume imaging. Cell. 159 (4), 896-910 (2014).
- Zhuge, M., et al. Ultrasensitive vibrational imaging of retinoids by visible preresonance stimulated Raman scattering microscopy. Advanced Science. 8 (9), 2003136 (2021).
- Miao, K., Wei, L. Live-cell imaging and quantification of PolyQ aggregates by stimulated Raman scattering of selective deuterium labeling. ACS Central Science. 6 (4), 478-486 (2020).
- Klimas, A., et al. Nanoscale imaging of biomolecules using molecule anchorable gel-enabled nanoscale in-situ fluorescence microscopy. Research Square. , (2021).
- Shi, L., et al. Super-resolution vibrational imaging using expansion stimulated Raman scattering microscopy. Advanced Science. , (2022).
- M'Saad, O., et al. All-optical visualization of specific molecules in the ultrastructural context of brain tissue. bioRxiv. , (2022).
- Zhao, Y., et al. Nanoscale imaging of clinical specimens using pathology-optimized expansion microscopy. Nature Biotechnology. 35 (8), 757-764 (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。
我们使用 cookie 来增强您在我们网站上的体验。
继续使用我们的网站或单击“继续”,即表示您同意接受我们的 cookie。
