Method Article
合成のバクテリオファージの建設は合わせた多様性と fab 抗体ライブラリーを表示
* これらの著者は同等に貢献しました
要約
このプロトコルでは、合わせた多様性を持つ合成抗体のバクテリオファージの表示ライブラリの構築の詳細な手順について説明します。合成抗体は、病気の診断、治療に基礎研究から広範なアプリケーションを持っています。
要約
基礎研究と医学におけるモノクローナル抗体 (Mab) の需要は毎年増加しています。ハイブリドーマ技術は、1975 年にその最初のレポート以来のモノクローナル抗体開発の支配的な方法をされています。代替技術としてモノクローナル抗体開発のバクテリオファージの表示方法は、ヒュミラ、最初のファージ由来の抗体と 1 つのベストセラーの Mab が 2002 年に関節リウマチの臨床治療のため承認されたのでますます魅力的です。非動物ベースのモノクローナル抗体開発技術、バクテリオファージの表示は抗原免疫原性、人間性、および伝統的なハイブリドーマ技術による抗体の開発から必要とされる動物のメンテナンスをバイパスします。このプロトコルでは 109-1010単一電気穿孔法を用いて得られる多様性を持つ合成工場バクテリオファージ表示のライブラリの構築手法について述べる.このプロトコルで構成されています: 1) 高効率電子有能なセルの準備;2) ウラシルを含む単一座礁させた DNA (デュ ssDNA); の抽出3) クンケル法オリゴヌクレオチド指示された突然変異誘発;4) エレクトロポレーションとライブラリのサイズの計算5) 蛋白質 A/L ベース酵素免疫測定法 (ELISA 折りたたみ) と機能の多様性評価;・ 6) 多様性の DNA シーケンス解析。
概要
Mab 基礎研究から疾患の診断、治療に至るまで幅広い用途があります。2016 年時点以上 60 Mab は、自己免疫疾患、がん、感染症1,2の臨床治療のためアメリカ合衆国の食品と医薬品管理 (USFDA) によって承認されています。
1975 年ケーラーとミルスタイン、'ハイブリドーマ'、このテクニックと呼ばれる細胞ソースから単一クローンの特異性の抗体の連続的な生成のための技術、薬および企業3 の礎となってその後の報告で ,4。世代この法によるモノクローナル抗体の抗原の生産、マウス免疫、B リンパ球の抽出、不滅のハイブリドーマ細胞、クローンの選択を形成する骨髄腫細胞と治療への応用のための B 細胞の融合など様々 なステップが必要です。人間性がひと抗マウス抗体 (浜)4,5を避けるために必要です。しかし、この技術の毒素や病原体、非常に節約された蛋白質を含む抗原は mAb 生産5生体内で免疫反応をトリガーに比較的有効であります。
1978 年にハチソンらは単一座礁させたファージ ウイルス6残基の直接変異するオリゴヌクレオチドの使用を報告しました。1985 年、スミスは外国の遺伝子断片が III バクテリオファージのコート蛋白質を符号化する遺伝子とフレームに融合することができ、その感染7を損なうことがなくファージ表面に表示することができますはこうしてを報告しました。免疫、世間知らずで抗体のバクテリオファージの表示ライブラリの後続の建設の基礎を築いた先駆これらと合成形作る単一鎖可変フラグメント (scFv) と抗原結合フラグメント (Fab) の形式の治療モノクローナル抗体開発8,9。技術的な観点からバクテリオファージ表示ベースの抗体開発を提供していますいくつかの抗原をもたらすことができます制限を回避することができますハイブリドーマによるモノクローナル抗体開発に相補的なアプローチと、人間化のプロセスハイブリドーマ培養由来の抗体は、5を必要があります。2016 年現在 6 バクテリオファージ表示由来モノクローナル抗体はヒュミラ、リウマチ性関節炎の治療に使用される最も成功したモノクローナル抗体のいずれかを含む市場に承認されているし、多くのバクテリオファージ表示由来抗体の候補者は現在臨床のさまざまな段階で調査10。
免疫と素朴なバクテリオファージの抗体ライブラリ、多様性の相補性決定の地域 (Cdr) 軽鎖重鎖は自然免疫レパートリー (すなわち、B 細胞から) に由来します。対照的に、合成のバクテリオファージの抗体ライブラリの Cdr の多様性は、完全に人工的です。合成ライブラリ構築への配列の多様性のデザインを正確に制御を提供して抗体の構造の解明のための機会を提供して機能の11,12。さらに、大規模な工業開発11,12下流に、容易にするために図書館の建設前に合成ライブラリのためのフレームワークを最適化できます。
1985 年にクンケルは M13 バクテリオファージにサイト指示された突然変異を効率的に導入する一本鎖 DNA 変異のテンプレート ベース アプローチを報告した13。このアプローチは、phage 表示ライブラリの構築のため広く使用された後。化学的に合成した DNA のオリゴヌクレオチド Fab Cdr に多様性を導入するように設計は、抗体のバックボーン テンプレート phagemid に組み込まれます。このプロセスで phagemid はウラシルを含む ssDNA (デュ ssDNA) として表現され、オリゴヌクレオチドは、Cdr に焼鈍、T7 DNA ポリメラーゼと T4 DNA リガーゼの存在下で二本鎖 DNA (dsDNA) を合成する拡張します。最後に、生成された ds DNA は、電気穿孔法による大腸菌に導入することができます。
高い多様性、phage 表示ライブラリ構築、高電圧電気穿孔法と電子有能なセルの二成分混合物の閉じた円形 dsDNA (CCC dsDNA) を慎重に準備されるべき。シドゥらは、従来から大幅に強化されたライブラリ多様性14DNA と電子有能なセルの準備を変更しました。
このプロトコルでは 109-1010単一電気穿孔法を用いて得られる多様性を持つ合成工場バクテリオファージ表示のライブラリの構築手法について述べる.図 1ライブラリ構築などの概要を示します: 1) 高効率電子有能なセルの準備;2) デュ一本鎖 Dna の抽出3) クンケル法オリゴヌクレオチド指示された突然変異誘発;4) エレクトロポレーションとライブラリのサイズの計算5) A/L ベース ELISA のための蛋白質折りたたみと機能の多様性評価;・ 6) 多様性の DNA シーケンス解析。すべての試薬、緊張および機器は、材料のテーブルに表示されます。表 1は、試薬のセットアップを示しています。
プロトコル
注: フィルター滅菌ヒント使わなければならない全体バクテリオファージを扱うときピペット郡とその周辺地域への汚染を避けるために。細菌およびバクテリオファージの処理実験するとき、無菌エリアやフードを使用する必要があります。バクテリオファージ実験エリアは、バクテリオファージ汚染を避けるために 2% ドデシル硫酸ナトリウム (SDS) 続いて 70% エタノールを使用してクリーンアップする必要があります。このプロトコルのシリアル希薄を作るための新しいヒントを使用して、各希釈用ください。
1大腸菌SS320 電子有能なセルの準備。
- あらかじめ暖かい LB/テト寒天プレート (準備とに格納されて 4 ° C 1 週間の古いよりも小さいため) 1 h. の 37 ° C の定温器でジグザグ方向滅菌接種ループまたは予め温めておいたテト/LB 寒天プレート大腸菌SS320 細胞のグリセロール ストックを連勝する滅菌チップで使用します。プレートの表面に沿って優しくグローバリゼ-ション。約 12-15 時間のために夜通し 37 ° C の定温器でプレートを孵化させなさい。
- 次の日は、ジグザグの線に沿って十分に分離単一コロニーをピックアップし、媒体にループを浸漬し、簡単に攪拌によって 50 mL 丸底チューブで 2YT/テト培地 10 mL に接種する滅菌接種ループまたは滅菌チップを使用します。
- 37 ° C で約 0.4 0.8 (ログ相成長) で外径600に達するまで約 3-4 時間 200 rpm で振とうしながらインキュベートします。2 h でモニター外径600 。この期間中に前 5 LB/テト寒天プレート (準備しに格納されて 4 ° C 以下の 1 週間の古い) 少なくとも 1 h の 37 ° C の定温器を温めます。
- ラボ株式価約 1 × 10 から 20 μ L M13KO7 助手のバクテリオファージを追加13コロニー形成単位 (cfu)/mL 180 μ L 滅菌 1 の 10 を準備するオートクレーブ 1.5 mL チューブ X PBS 10 倍のシリアル希薄。
- 5 X 14 mL に分注 4 mL オートクレーブ、液体 2YT 上の寒天は、ラウンド下部チューブそれぞれ、10 倍希釈の第 9 第五から 1 希釈でそれぞれのチューブにラベルを付けるし、液体の状態で 2YT 上の寒天を維持するために 65 ° C のインキュベーターで孵化させなさい。
- M13KO7 希釈チューブに細胞のログ相大腸菌SS320 ミックス 500 μ L (選択 5 9 に 10 倍希釈 10 倍希釈)、5-10 分の 37 ° C のインキュベーターで孵化させなさい。
- ステップ 1.6 の孵化の間に 1.5 のステップから部屋の温度 (RT) 約 42 ° c. に約 5 分間クールダウンの 2YT 上の寒天を転送する.内側の手首を使用して、2YT 上の寒天; の温度をテストするのにはそれは残るべきである液体として。各対応する 2YT 上の寒天チューブに 1.5 のステップから各希釈混合物を転送します。各チューブの蓋を締め、ミックス優しく数回を逆さま回すことによって、簡単に気泡の発生を避けるためにします。
- 慎重に (手順 1.3) からあらかじめ暖かい LB/テト寒天プレートの端に沿ってそれぞれの混合物を注ぎ、わずかに完全かつ均等に泡の導入を回避しながらの混合物とのプレートを埋める板を傾斜します。常温約 5-10 分内各プレート上の寒天を固めるし、約 15-18 時間一晩 37 ° C で 2YT 上の寒天プレートをインキュベートするプレートを保ちます。
- 1.8 ステップ周りから一晩成長後希釈プレートを選択プラーク接種 100 200 平均サイズ、シングル、良く分離斑。光源、および垂直方向に上の寒天に刺す長い滅菌ピペット チップを搭載ピペット銃を保持するために他の手に対して寒天プレートを保持する 1 つの手を使用し、シングルと十分に分離プラークを収集します。
- 少し上の寒天を区切りますプラークにピペット チップを傾けます。2YT/菅/テト培地 1 mL でプリロード済 14 mL 丸底培養管にプラークと寒天を取り除くために数回上下ピペット (表 1参照)。
- 合計で 3-5 プラークをピックアップするこの手順を繰り返します。いくつかプラクを狩りの目的は、プラークは細菌コロニーと比較して比較的小さいすることができます成功した接種を確保するためです。200 rpm で振とうしながら 37 ° C で 8 h のプラクを成長します。
- 2YT/菅/テト中 250 mL 困惑してフラスコ 50 mL に文化を成長のチューブを転送します。37 ° C で約 12 時間のために夜通し 200 rpm で振とうしながら成長します。
- Superbroth/テト/菅媒体ごとの一晩の文化の 5 mL と 900 mL を含む 3 つの 2 L 困惑してフラスコを接種します。37 ° C で約 0.6 0.8 外径600 6-7 h の 200 rpm で振とうしながらインキュベートします。
- 穏やかな一定旋回の手で 5 分間氷浴で細菌培養の 3 個のフラスコを冷やします。次の手順は、prechilled ソリューションと機器の氷の上の冷蔵室でされるべきであります。
- 各フラスコ; の外側のガラスを乾燥するのに吸光度組織タオルを使用します。ベンチと各遠心ボトルに各フラスコからそっと中を注ぐ一方 1 L オートクレーブ遠心ボトルを斜めに 1 つの手を使用します。
- スピン 5,000 × g とペレット細菌.に 10 分の 4 ° C
- 以下の遠心分離、5 L のオートクレーブ廃棄物ビーカーに上清をデカント優しく。
- 100 ml の滅菌フィルター 1.0 mM HEPES、pH 7.4 の各ボトルを記入し、200 rpm でペレットを再懸濁で支援するために、各ボトルにオートクレーブの電磁攪拌バーを追加します。旋回、電磁攪拌中に数分おきボトル壁から全体のペレットを外します。ペレットが解散した後は、追加 400 ml 滅菌フィルター 1.0 mM HEPES、pH 7.4 の各ボトルを入力します。
- 遠心 5,000 × g、4 ° C で 10 分間デカント清、瓶の中の攪拌棒を保持します。
- 1.16 に 1.17 の手順をもう一度繰り返します。
- 攪拌棒の助けを借りて無菌ろ過、10% の超高純度グリセリン 500 mL にペレットを再懸濁します。旋回、電磁攪拌中に数分おきボトル壁から全体のペレットを外します。
- 遠心分離機し、1.17 の手順とデカントします。1.19 の手順をもう一度繰り返します。攪拌棒を削除するのに長い腕オートクレーブ鉗子を使用します。
- 5,000 × g、15 分上清をデカントの 4 ° C で遠心し、各遠心ボトルからピペットで上澄みの任意の残りのトレースを削除します。
- 1 つのボトルに 10% の超高純度グリセリン 3.0 mL を追加し、軽くピペッティングでペレットを再懸濁します。次のボトルに懸濁液を転送し、ペレットのすべて再停止、ボトル 1 本の組み合わせまで繰り返します。高濃度細胞の約 6 mL、11 cfu/mL 約 3 x 10 の力価が得られます。
- 事前チル 1.5 mL オートクレーブ マイクロ遠心チューブ用、この手順の前に少なくとも 1 時間-80 ° c の仕切りなし 1 つの 96 ウェル チューブ収納ボックス。細胞ペレット懸濁系の因数をピペッティングする前に冷蔵室に氷の上あらかじめ冷やしたマイクロ遠心チューブ用を転送します。各 1.5 mL 容マイクロ チューブに細胞懸濁液の 350 μ L 分注にピペットを使用します。
- 発泡ボックス コンテナー フラッシュ凍結液体窒素で満たされた (3-5 分) に試料を転送します。
- -80 ° C のフリーザーに液体窒素で発泡ボックスを輸送 (1.23 の手順を参照してください)。
- -80 ° C のフリーザーから 1.5 mL 容マイクロ チューブ収納ボックスを削除する (手順 1.23 参照) と氷に置きます。
- すぐに液体窒素から因数をふるい、チューブ収納ボックス内に配置する金属製のメッシュを使用します。-80 ° C でストア
注意: 液体窒素がやけどを引き起こすことができるし、安全保護のため注意が必要があります。Fab バックボーン phagemid を使用して、(Fab バックボーン phagemid ストックが 400 ng/μ L の純水 (MilliQ) はず) 準備のエレクトロ有能なセルの効率を確認します。1 μ g の 10 μ L の純水で Fab バックボーン phagemid そしてエレクトロ有能な SS320 氷の上の 350 μ 因数を解凍し、氷の上 0.2 cm ギャップ エレクトロポレーション キュベットを prechill します。
- 解凍後 10 μ L の DNA に 350 μ L エレクトロ有能な SS320 セル、氷の上の混合物を保ちながら数回をピペッティングで混ぜます。気泡を導入することは避けてください。
- エレクトロポレーションの前に少なくとも 30 分の 37 ° C の水浴内の 50 mL チューブに SOC 培地 15 mL は事前に暖かい。キュヴェットへ 360 μ L 混合物を転送し、製造元の指示に従ってエレクトロポレーションを行います。
- エレクトロポレーション後ピペッティングを (ステップ 1.27) から予め温めておいた SOC 培地 1 mL を追加してセルをすぐに救出します。125 mL のフラスコに予め温めておいた SOC の 5 mL でプリロード済にキュベットからメディアを転送します。
- キュベットを 2 回すすぎ、たびに予め温めておいたの SOC 培地 1 ml と 125 mL のフラスコにメディアを転送 (1.27 の手順を参照してください)。125 mL フラスコに 10 mL の最終巻に予め温めておいた SOC 培地を追加します。
- 10 mL 培養 200 rpm で振とうしながら 37 ° C で 30 分間インキュベートします。
- トランスフェクション効率と M13KO7 前感染率を決定するためのシリアル希薄を作る。
- 96 マイクロウェル プレートの 1 つの列の各ウェルに 2YT メディアの 180 μ L を追加するのにには、マルチ チャンネル ピペットを使用します。
- 8 倍シリアルを作る希釈: ピペッティングによるステップ 1.29 ミックス プレートの最初の井戸から文化の 20 μ L を転送し、よく混合物の 20 μ L を次に転送します。シリアル希薄化の最後にこの手順を繰り返します。
- 各シリアル希薄を重複して LB/炭水化物、LB/菅、LB/テト プレート上の 10 μ L をプレートします。
- 37 ° C で一晩インキュベートします。
- 効率計算式: M 希釈倍 10Nから数えて平均コロニー数であると仮定 (N は 1 ~ 8)。LB/炭水化物プレートから Fab バックボーン phagemid のエシェリヒア属大腸菌SS320 エレクトロ有能なセル トランスフェクション効率はN + 3 M X 10 cfu/μ g と同じです。M13KO7 前の感染率は、LB/菅と LB/テトの植民地の比から推定しました。Fab バックボーン phagemid をトランスフェクトした大腸菌SS320 有能なセルの割合は、LB/炭水化物および LB/菅の植民地の比から推定されます。
2. Phagemid テンプレートからウラシルを含む ssDNA (デュ ssDNA) を準備します。
注: A 以前 Fab バックボーン phagemid はデュ ssDNA 準備15のテンプレートとして使用された報告。Fab バックボーン phagemid のアーキテクチャを図 2に示します。プラスミド スピン キット (QIAprep スピン M13) は、わずかな修正とデュ ssDNA の抽出のために使用されます。
- 中古滅菌ループまたはヒントを予め温めておいた LB/cmp プレート大腸菌 CJ236 (または別の dut−/ung− 株) のグリセロール ストックを 1 h. 連勝の 37 ° C のインキュベーターで LB/cmp プレート (準備しに格納されて 4 ° C 以下の 1 週間の古い)、暖かい。約 12-15 時間一晩 37 ° C でプレートを孵化させなさい。
- 滅菌チップ シングル コロニーをピックアップし、14 mL ポリプロピレン製丸底チューブで 2YT/cmp 媒体の 2 mL に接種します。
- 外径600が約 0.4 0.8 (ログ相成長) に達するまで 3-4 時間 200 rpm で振とうしながら 37 ° C で媒体を孵化させなさい。
- 文化に Fab バックボーン テンプレートの 5-10 μ L ファージを追加し、バクテリオファージ感染できるように 30 分間、200 rpm で振とうしながら, 37 ° C で。
- インキュベーション後、37 ° C で予め温めておいた LB/炭水化物プレート上に文化の 10 μ L を連勝する滅菌チップを使用します。約 12-15 時間一晩 37 ° C で孵化させなさい。
- 3 mL 2YT/炭水化物/cmp 14 mL ポリプロピレン製丸底チューブ内のスターター培養に接種し、37 ° C で約 12 時間のために夜通し 200 rpm で振とうしながら成長するエシェリヒア属大腸菌CJ236 含む Fab バックボーン phagemid 単一コロニーを拾います。
- 30 mL 2YT/炭水化物/cmp 250 mL 困惑して瓶の中に 0.3 mL スターター文化を接種します。
- 外径600が約 0.4 0.8 (ログ相成長) に達するまで 3-4 時間 200 rpm で振とうしながら 37 ° C で培養を孵化させなさい。
- 追加、M13KO7 (ラボ株式価約 1 × 1013 cfu/mL) から文化にステップ約 10 の感染 (MOI) の多様性と 2.8 と M13KO7 の最終的な力価は約 1 × 1010 cfu/mL。
- 200 rpm と 37 ° C 1 時間孵化させなさい。
- 5,000 × g および 25 ° C、20 分で 50 mL 丸底チューブの遠心分離によって文化をペレットします。
- 上清をデカントし、30 ml の新鮮な 2YT/炭水化物/菅/ウリジンのペレットを再懸濁します。新しい 250 mL 困惑してボトルに再懸濁を転送します。
- 200 rpm および 25 ° C、22-24 時間で孵化させなさい。
- 50 mL 丸底チューブ ステップ 2.13 から文化を転送し、細菌の細胞ペレットからファージ培養上清を分離する遠心分離機の 12,000 × g 20 分での文化。新しい 50 mL 丸底チューブに上清のバクテリオファージを転送し、1/5 追加をバクテリオファージを沈殿させたペグ/食塩の最終巻。よく混合し、バクテリオファージの粒子を沈殿させる 30 分間氷の上を孵化させなさい。
- 遠心 12,000 × g、4 ° C 30 分上清をデカントし、4,000 × g と 4 ° C で 2 分間遠心は、残りの上清を吸引します。
- 無菌ろ過 1 × PBS と 1.5 mL 遠心チューブに転送の 2 mL でバクテリオファージのペレットを再懸濁します。任意の残りの細菌の残骸を取除く、ファージ培養上清を新しい 1.5 mL 遠心チューブに転送し、4 ° C で保存するベンチトップ遠心機で 5 分間 12,000 × g で遠心分離します。
- 大腸菌SS320 と並べて比較することにより大腸菌CJ236 ウラシル混入効率を確認してください。
- 2YT メディアの 180 μ L を各ウェルの 96 ウェル プレートの 1 つの行に追加します。
- 10 倍シリアル 10 を作る希釈: ファージ培養上清中の 20 μ L をプレートの最初の井戸に転送、ピペッティングで混ぜるし、よく混合物の 20 μ L を次に転送します。シリアル希薄化の最後にこの手順を繰り返します。
- ログの段階で 90 μ CJ236大腸菌と大腸菌SS320 に感染する最後の 8 のシリアル希薄からファージの 10 μ L を追加 (外径600 = 0.4 0.8)。37 ° c 穏やかな揺れで 30 分間インキュベートします。
- 各感染症大腸菌CJ236 重複の 2YT/炭水化物プレート大腸菌SS320 のシリアル希薄化から 10 μ L をプレートします。
- 37 ° C で一晩インキュベートします。
- 力価計算式: M 希釈倍 10Nから数えて平均コロニー数であると仮定 (N は 1 から 10 から)。CJ236大腸菌または大腸菌SS320 価はN + 2 M X 10 cfu/mL と同じです。CJ236大腸菌と大腸菌SS320 の力価比からウラシル設立の効率を推定します。
- 1.5 mL 遠心チューブに上清のバクテリオファージ バクテリオファージ沈殿バッファー MP の 1/100 ボリュームを追加し、数回のミックスに軽く上下します。RT でファージ粒子が沈殿培養液から、少なくとも 2 分間インキュベートし、したがって、曇りソリューション必要がありますこの時点で表示されます。
- 1.5 mL 遠心チューブにステップ 2.18 からサンプルを (例えばQIAprep) のプラスミド スピン列に適用します。SsDNA の 1 つの回転カラムのバインディングの容量まで少なくとも 10 μ g です。 遠心分離機 6,000 × g と 30 の 25 ° C でベンチトップ遠心機で s。1.5 mL 遠心チューブにあるフロースルーを破棄します。バクテリオファージの粒子は、この段階で列のマトリックスにバインドされたまま。
- 列に UT-MLB バクテリオファージ溶解と結合バッファー (ディスカッションを参照) の 0.7 mL を追加し、RT で少なくとも 1 分間インキュベートします。 遠心分離機の 6,000 x g、25 ° C、30 s と流れを破棄。
- 別の 0.7 mL の UT-MLB バッファーを追加し、RT で少なくとも 1 分間インキュベートします。
- 流れを 30 s. 破棄の 6,000 × g で遠心分離機します。この段階でバクテリオファージのコート蛋白質はそのまま列行列にバインドするデュ ssDNA から分離されます。
- 製造元の指示に従って洗浄バッファー PE 含むエタノールの 0.7 mL を追加します。30 s の流れを通して破棄 6,000 × g で遠心分離機します。
- 2.23、手順を繰り返しますし、もう一度 30 6,000 × g で遠心分離機 s PE 残留バッファーを削除します。
- 列を新しい 1.5 mL 遠心チューブに転送し、溶出バッファー EB (10 mM Tris· の 100 μ L を追加CL、pH 8.5) 列膜の中心に。
- 10 分と 1 分の溶出がデュ ssDNA の 6,000 × g で遠心する室温で孵化させなさい。およそ、1.5-2.5 μ g デュ-ssDNA/mL 文化が得られます。
- TAE ゲル 1% アガロースゲルの 1 μ L electrophoresing によって溶離された DNA を分析します。DNA はないスミアと主に単一バンドとして表示されます。
- 260 nanodrop 分光光度計で吸光度を測定することによって DNA 濃度を決定する nm (260 ssDNA の 33 ng/μ L の 1.0 を =)。典型的なデュ ssDNA 濃度は 200-500 ng/μ L 以内です。
3. クンケル法オリゴヌクレオチド特異的変異導入
ノート: 変異原性オリゴヌクレオチドと反応成分の品質を確保する本格的反応の前に小規模な反応を実施することをお勧めです。クンケルの手法の漫画オリゴヌクレオチド指示された突然変異誘発は図 3に示します。様々 なアミノ酸の多様性は、命名法16 (表 2) を番号 IMGT と CDRH1、CDRH2、CDRH3、および CDRL3 の地域に導入されます。表 2に各 CDR の理論的なアミノ酸の多様性、総理論的なアミノ酸の多様性、およびオリゴヌクレオチドのシーケンスのとおりです。
- T4 ポリヌクレオチド キナーゼをオリゴヌクレオチドを用いたリン酸化
- 0.6 μ g を 1 μ L 100 mM DTT や 2 μ L 10 mM ATP、バッファー TM X 2 μ 10 の単一の CDR を変異させる設計されている変異のオリゴヌクレオチドを結合します。超純水 H2O を 1.5 mL チューブに 18 μ L の総ボリュームに追加します。ライブラリ構築に 4 別々 と並列のリン酸化反応はそれぞれ CDRH1、CDRH2、CDRH3、CDRL3 に対応して設定されます。
- 20 U を追加 (2 uL) それぞれに T4 ポリヌクレオチド キナーゼのチューブし、37 ° C (表 3に反応 1) で 1 時間インキュベートします。アニーリングのためすぐに使用します。
- テンプレートにオリゴヌクレオチドの焼鈍
- 20 μ g デュ ssDNA テンプレートに 0.5 mL チューブで 250 μ L の最終巻に 10 X TM バッファーの 25 μ L、各のリン酸化オリゴヌクレオチド ソリューションおよび超純水 H2O の 20 μ L を追加します。よく混ぜるし、表 3の反応 2 として 0.20 mL PCR チューブ (50 μ) に移転します。オリゴヌクレオチドとテンプレートの長さの比が 1: 100 と仮定すると、これらの DNA 量はオリゴヌクレオチドとテンプレート、3:1 のモル比を提供します。
- 90 ° C で 3 分、3 分、50 ° C のための PCR マシンで反応をインキュベートし、20 ° C、5 分。
- CCC dsDNA の酵素的合成
- オリゴヌクレオチド/テンプレートの焼鈍 250 μ L 混合物に表 3の反応 3 として 10 mM ATP の 10 μ L、25 mM dNTP ミックスの 10 μ L、100 mM DTT の 15 μ L、30 のヴァイスシュヴァルツ ユニット T4 DNA リガーゼ、30 U T7 DNA ポリメラーゼを追加します。
- 一晩 20 ° C で 1.5 mL チューブに反応を孵化させなさい。
- 洗って室温 30 kDa の孔サイズの膜を 0.5 mL 遠心ろ過デバイス合成 CCC dsDNA を集中
- フィルター デバイスに一晩反応混合物を転送し、超純水 H2O を 400 μ L 最終巻に追加します。14,000 × g 10 分; でスピンします。未満 50 μ L です。
- 流れを破棄、フィルターに超純水 H2O の 400 μ L を追加し、14,000 × g 10 分で回転します。
- 3.3.3.2 の手順をもう一度繰り返します。
- 逆さま CCC dsDNA を回復するきれいな遠心管にフィルターを配置します。2 分; 1,000 × g でスピンします。回収量は一般におよそ 20-40 μ L。回復された CCC dsDNA はエシェリヒア属大腸菌のエレクトロポレーション用すぐにまたは後で使用するための-20 ° C で凍結できます。通常 20-40 μ g CCC DNA が得られます。
- 反応の結果を視覚化するデュ ssDNA テンプレートと一緒に溶出反応生成物の 1.0 μ L を electrophorese します。
4. エレクトロポレーションとライブラリのサイズの計算
- 精製された CCC dsDNA の寒さ (50 μ L の最大音量で 20 μ g) 1.5 mL 遠心チューブに 0.2 cm ギャップ エレクトロポレーション キュベット氷の上。
- 少なくとも 30 分の 37 ° C の水浴中 50 mL ポリプロピレン製円錐形遠心管に SOC のメディアの事前暖かい 20 mL。
- エレクトロ有能なエシェリヒア属大腸菌の 350 μ 因数を融解氷の上 SS320。数回ピペッティングにより徹底的に DNA とミックスにセルを追加します。気泡を導入することは避けてください。
- キュベットに混合物を転送し、製造元の指示に従ってエレクトロポレーションを実行します。たとえば、BTX ECM 630 エレクトロポレーション システムを使用すると、関連の設定が 2.5 kV 電界強度、125 Ω 抵抗と静電容量 μ F 50。
- すぐに予め温めておいた SOC 培地 1 mL を追加し、SOC 培地 125 mL のフラスコの中の 17 mL に転送することによって electroporated 細胞を救出します。同じフラスコに SOC 培地および転送の 1 つの mL で 2 回キュベットをリンス (最終巻は 20 mL)。
- 200 rpm で振とうしながら 37 ° C で 30 分間インキュベートします。
- エレクトロポレーションの効率を決定します。
- 96 マイクロウェル プレートの 1 つの列の各ウェルに 2YT メディアの 180 μ L を追加します。
- 8 の 10 倍シリアルを作る希釈: 20 mL 文化の 20 μ L をプレートの最初の井戸に転送、ピペッティングで混ぜるし、よく混合物の 20 μ L を次に転送します。シリアル希薄化の最後にこの手順を繰り返します。
- 重複の 1 つの LB/炭水化物プレートにシリアルの希薄のそれぞれの 10 μ L をプレートします。各コロニー カウント クロス チェックのため別の LB/炭水化物プレートにシリアルの希薄から残り 100 μ L をプレートします。これらのプレートは、elisa 法とシーケンス解析 (セクション 5 を参照) の単一クローンをまた提供します。
- 37 ° C で一晩インキュベートします。
- LB/炭水化物プレートに 10 μ L の重複からコロニーをカウントします。M が平均コロニー数のカウント 10N倍ポンド/炭水化物プレート (N は 1 ~ 8) であると仮定します。合計ライブラリのサイズは、10N + 3コロニー X 2 M と同じです。
- 因数 2 2 L 困惑してフラスコ、各含む 500 mL に均等にバクテリオファージ ライブラリ生成のための 2YT/炭水化物/菅媒体の 4.6 ステップから文化。
- 37 ° C で 16 時間前後のために夜通し 200 rpm で振とうしながらインキュベートします。
- 2 つの 1 L オートクレーブ遠心ボトル、4 ° C で 12,000 × g で 30 分間遠心する文化を転送します。
- 2 つの新しい 1 L オートクレーブ遠心ボトルに上澄みを転送し、1/5 追加をバクテリオファージを沈殿させたペグ/食塩の最終巻。氷上で 30 分間インキュベートします。
- 4 ° C、12,000 × g で 30 分間遠心慎重に上清をデカント、バクテリオファージの餌を妨害しません。4,000 x g で 1 分のスピンし、ピペットと残りの上清を除去します。
- 20 ml 1 × PBS バッファーの無菌ろ過、新しい 50 mL チューブへの転送のバクテリオファージ ペレットを再懸濁します。
- 4 ° C、12,000 × g で 5 分間遠心分離によって不溶性物質をペレットします。新しい 50 mL チューブに上清を転送します。
- 分光光度計によるファージ濃度の測定 (外径268 = 5 X 1012バクテリオファージ/mL の溶液 1.0)。
- 5 X 1012バクテリオファージ/ml 1x PBS で 10% の超高純度グリセリンにバクテリオファージの濃度を調整します。
- 1.5 mL 遠心チューブごとの解決をバクテリオファージの分注 1 mL。すぐにパンのライブラリを使用してまたは-80 ° C で保存
5. 品質評価蛋白質 A/L ダイレクト バインディング ELISA の試金とシーケンス
- 2YT/炭水化物各ウェル内の 800 μ L を含む 96 深いよく培養プレートへのステップ 4.7.3 から LB/炭水化物プレートに 96 のシングル コロニーをランダムに選択します。外径600 1,000 rpm で振とうしながら 37 ° C で 3-4 時間インキュベート 0.4 0.8 を =。
- M13KO7 の 100 μ L を追加 (1 X 1011 cfu/mL) 96-深いの各ウェルにも文化、マルチ チャンネル ピペットのプレート。37 ° C で 1 時間 1,000 rpm で振とうしながらインキュベートします。
- マルチ チャンネル ピペットの各ウェルにカナマイシン (500 μ g/mL) の 10 倍の濃度を含む 2YT の 100 μ L を追加します。1,000 rpm で振とうしながら 37 ° C で一晩インキュベートします。
- 1 μ g/mL の 1x PBS で L タンパク質を溶解します。L 蛋白質の解決の 30 μ L/ウェルの 384 ウェル高蛋白結合ポリスチレン板で井戸のコート 3/4。4 ° C で一晩インキュベートします。
- ステップ 5.4 から一晩タンパク質 L コーティング溶液を破棄します。M PBST の 50 μ L を追加 (ブロッキング液) 384 ウェル高マルチ チャンネル ピペットと蛋白結合ポリスチレン板で井戸のすべてに。室温 1 時間マイクロ プレート シェーカーでプレートを孵化させなさい
- 4 ° C で 10 分間 3,000 x g で深い 96 ウェル培養プレートにステップ 5.3 から一晩かけて培養スピンダウンします。バクテリオファージは上清です。
- ステップ 5.5 からブロッキング液を破棄します。千九百六十八とマルチ チャンネル ピペットを使用して 1 非コーティングも負の値として制御タンパク質コーティング L 井戸の 3 M PBST の 15 μ L と上清 (ステップ 5.6) 各バクテリオファージの 15 μ L を追加します。200 rpm で振とうしながら 1 時間室温で孵化させなさい。
- バクテリオファージ ソリューションを破棄します。自動プレート洗濯機 PBST の 80 μ L で 6 回洗浄します。
- マルチ チャンネル ピペットの各ウェルに M pbst; 15 μ、15 μ L のタンパク質 A HRP (1:1, 500 1x PBS で希釈) を追加し、, RT で 1 h 約 200 rpm で振とうしながら。
- タンパク質 A-HRP/M-PBST ソリューションを破棄します。PBST の 80 μ L で 6 回洗浄します。
- マルチ チャンネル ピペットで TMB 基板 (30 μ L/ウェル) を追加し、色を開発するまで 2-3 分間インキュベートします。1.0 M H3PO4 (30 μ L/ウェル) との反応を停止します。
- 450 でオンカラム プレートを読む nm。肯定的なクローンは、外径450 M PBST 3.0 よりも大きい蛋白質 L 井戸吸光度の平均比率を示すものとして定義されます。
- 96 深いさて、室温 30 分間 (ステップ 5.6) ELISA に使用される同じバクテリオファージの 5 μ L とログの段階で 2YT/テトで SS320 の 50 μ L を感染します。
- 5.13 のステップからも 96 深く 2YT/炭水化物の 950 μ L を追加し、1,000 rpm で振とうしながら 37 ° C で一晩インキュベートします。
- 小型準備 DNA 抽出キットで phagemid DNA を抽出します。VL の上流のプライマーを使用して (5'-TCGCTTTGTTTTTATTTTTTAATGTA-3') と VH (5'-GACTACTAATAACATAAAGTCTACGCCG-3') ライブラリ配列の多様性を推定するためのシーケンス分析のため。
結果
Fab 抗体ライブラリーの構築のフローチャートを次 (図 1参照)、我々 は M13KO7 ヘルパーに事前に感染するファージ大腸菌SS320 エレクトロ有能なセル準備します。図書館建設のため工場 phagemid バックボーンを使用した場合、これらの電子有能なセルの効率が 2 X 109 cfu/μ g として推定される (図 4)。
ウラシル混入効率比較では大腸菌CJ236 と大腸菌SS320 細胞に抗体価のチェックされました。CJ236大腸菌と大腸菌SS320 細胞は、デュ ssDNA をかくまっているバクテリオファージによって感染しました。大腸菌SS320 は酵素 (dUTPase ・ ウラシル グリコシラーゼ) ウラシル含む DNA、大腸菌CJ236 これらの酵素を欠いているし、ウラシルを含む DNA を低下させることはできませんが低下させることができます。許容できるウラシル混入効率を達成する、大腸菌CJ236 から抗体は大腸菌SS320 からのそれらより高い、少なくとも 10 の4倍になる必要があります。そうでなければ非効率的なウラシル定款により構築された抗体ライブラリ野生型人口が増加します。図 5一本鎖 Dna ファージに効率的なウラシル定款を示す大腸菌CJ236 から価が約 3 X 10 の5倍大腸菌SS320 からのそれよりも高いことが分かった。
次に、我々 は準備し、デュに一本鎖 Dna を抽出します。デュ ssDNA 純度は agarose のゲルの電気泳動 (図 6) によってチェックされます。オリゴヌクレオチド特異的変異導入を行ったし、CCC DNA へデュ ssDNA の変換の効率を評価した (図 6)。デュ ssDNA より低い運動の 3 つの製品は、最速実行バンド (CCC dsDNA)、中間の弱いバンド (ニック バンド)、遅い実行バンド (ストランド避難 DNA) などのゲルに視覚化できます。
一晩培養プレート大腸菌SS320 に穿孔後ライブラリのサイズを推定した (4.7.5 の手順を参照してください)。平均ライブラリのサイズは 5 × 109 LB/炭水化物プレート (図 7) に重複シリアル希薄だったただし、この段階での推定サイズは、フレーム シフトの存在のために工場を表示しないまたは終止コドン、または誤って折りたたまれた Fab を表示するファージを含めることができます。シーケンスおよび elisa 法は、構築されたライブラリの機能の多様性を推定する使用されました。96 のランダムに選択された単一のクローンは、シーケンス解析のために送られました。表 4に示します (少なくとも 1 つの CDR 変異 53 クローン) と野生型のシーケンスを持つ 17 のクローンは時期尚早停止コドンなし 70 クローンと 20 クローンとを含んだ、96 のランダムに選択された単一クローンのうち 90 が正常に塩基配列をさまざまな地域で時期尚早停止コドン。70 クローン内 CDRH1、CDRH2、CDRH3、および CDRL3 の突然変異率は、50%、57%、53%、56%、それぞれ、少なくとも 1 つの CDR と突然変異率は 76%。時期尚早停止コドン (90%) 20 クローン、時期尚早停止コドンは主に 45% (CDRH1)、10% (CDRH2)、15% (CDRH3)、20% (CDRL3) などのオリゴヌクレオチドの突然変異誘発のプライマーのフレーム シフトから派生しました。
適切に折り畳まれた工場、タンパク質 A のディスプレイを検出/L ベース ELISA タンパク質 A とタンパク質 L VH フレームワークと VL フレームワーク、それぞれ17,18の折り畳み式の適切な認識することができます知られている採用されました。シーケンス解析に一致して 3 通 (図 8) ELISA アッセイを示した陽性率であった場合、野生型シーケンスを持つ 17 のクローンはすべて陽性時期尚早停止コドンと 20 のクローンがすべて陰性だった経験的 3.0 に設定します。10 クローンが負の場合、少なくとも 1 つの CDR 変異 53 複製 43 クローンされた ELISA で陽性これはクローンのほとんどが 10 クローンから Cdr は、折り畳み式の Fab に有害な影響を持つことができますしながらも折り畳まれていることを示します。合計では、90 のクローン (48%) から 43 のクローンも折り畳まれた, し、変異体 CDR が少なくとも 1 つ含まれています。タンパク質 A に基づいて構築されたライブラリの機能性アミノ酸の多様性のため、L ELISA とシーケンス解析推定された 2.4 × 10/9 (すなわち5 X 10 の9の 48%)。
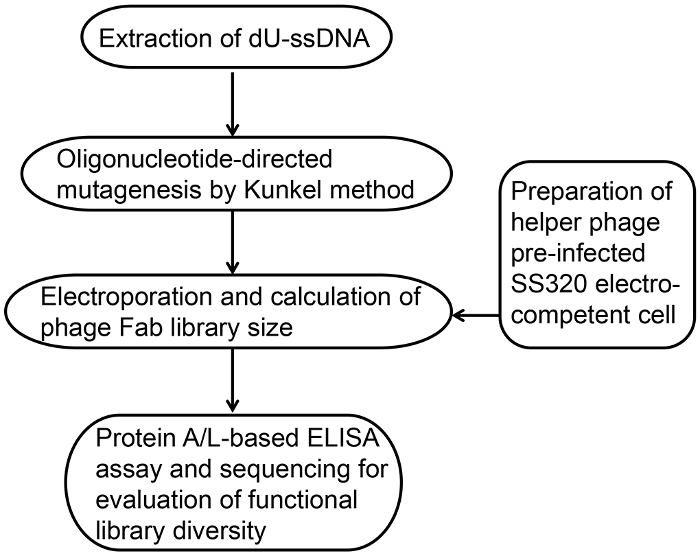
図 1: バクテリオファージ表示 fab 抗体ライブラリーの構築の概要。Fab 抗体ライブラリーのバクテリオファージ表示工事では、基本的な一連の手順を従います。それを含む高効率電気有能な細菌のセルの準備、デュ ssDNA、クンケルの法オリゴヌクレオチド特異的変異導入、エレクトロポレーション、ファージによる機能性評価、fab 抗体ライブラリー サイズの計算の抽出タンパク質 A L elisa 法と DNA シーケンス解析/。この図の拡大版を表示するのにはここをクリックしてください。
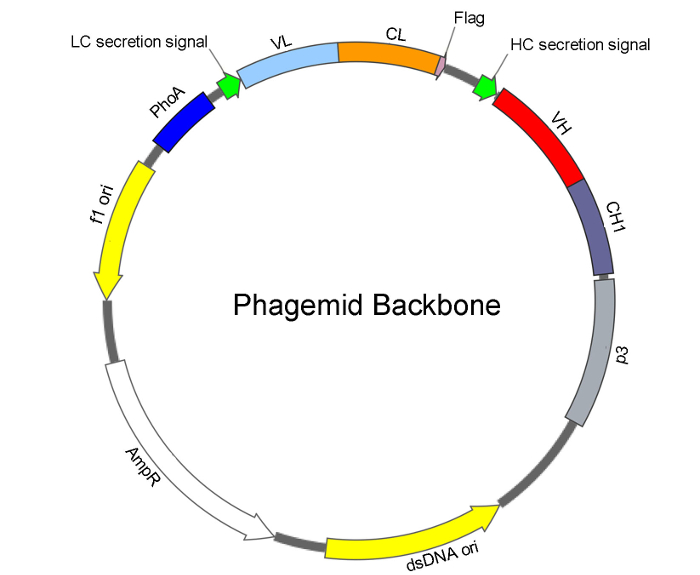
図 2: fab 抗体ライブラリーの構築 Phagemid アーキテクチャ。Phagemid バックボーンの基本的な機能から成っている単一座礁させた (f1 ori) と DNA 二重鎖 (dsDNA ori) の起源レプリケーション、およびアンピシリン/carbenicillin 抵抗性遺伝子 (AmpR)。アルカリホスファターゼ プロモーター (PhoA) の制御の下での Fab 表示には phagemid にはドライブ式との分泌する bicistronic カセットが含まれています: 光の分泌シグナル、VL (軽鎖の可変領域) CL (定数で構成されるチェーン (LC)軽鎖の領域)、および C ターミナル フラグ タグ;重鎖 (HC) 分泌シグナル、VH (重鎖の可変領域) と CH1 から成る (重鎖の一定領域 1) p3 バクテリオファージ マイナーな外被蛋白質と融合。大腸菌ペリプラズム内工場に軽鎖と重鎖のアセンブリは、ファージ表面で Fab の表示を指示します。この図の拡大版を表示するのにはここをクリックしてください。

図 3: クンケルの法の模式図によるオリゴヌクレオチド特異的変異導入します。このプロトコルでは dU ssDNA テンプレートを準備するのにクンケルのメソッドを使用しました。デザインの多様性と CDRH1、CDRH2、CDRH3、および CDRL3 のオリゴヌクレオチドがリン酸化、テンプレートに焼鈍、CCC dsDNA ss DNA に変換するために使用します。野生型と変異フォーム エレクトロポレーション大腸菌SS320 エレクトロ有能なセルに、次のヘテロ二本鎖 DNA が修復されます。野生型ストランドのウラシルの存在のために修復プロセスを支持する突然変異体のフォームと変異フォームがライブラリを支配するため。この図の拡大版を表示するのにはここをクリックしてください。
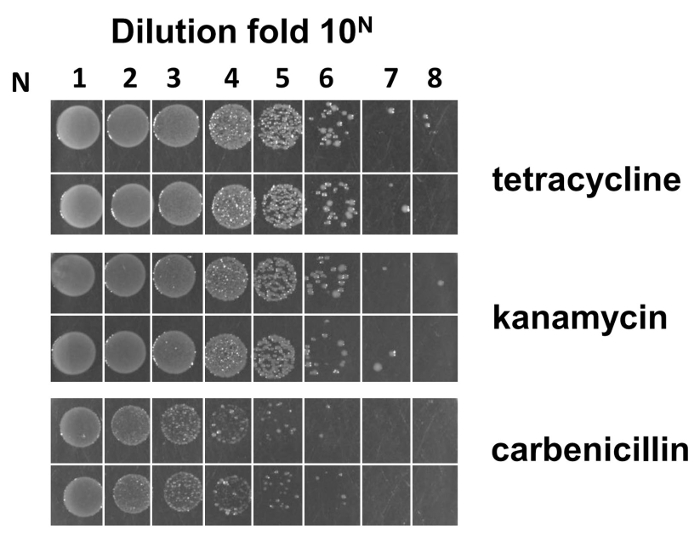
図 4: M13KO7 の推定に事前大腸菌SS320 エレクトロ有能なセル効率に感染します。Phagemid バックボーン ベクトルは、有能な細胞のエレクトロポレーション効率を確認していました。効率を計算する数式は次のように: M が最も希釈倍 10Nから数えて平均コロニー数であると仮定 (N は 1 ~ 8) 重複して。エシェリヒア属大腸菌SS320 LB/炭水化物板から効率はN + 3 M X 10 cfu/μ g と同じです。電子有能なセルの効率は約 2 × 109 cfu/μ g.この図の拡大版を表示するのにはここをクリックしてください。

図 5: ウラシル混入 CJ236エシェリヒア属大腸菌および大腸菌SS320 細胞のファージ感染によって ssDNA の評価。クンケルのメソッドに基づいて、ウラシルの取り込み効率はエシェリヒア属大腸菌CJ236 および大腸菌SS320 細胞におけるファージ感染力価の比較によってチェックします。力価の計算式は次のように: M が最も希釈倍 10Nから数えて平均コロニー数であると仮定 (N は 1 から 10 から)、エシェリヒア属大腸菌CJ236 または大腸菌SS320 価はN + 2 M X 10 cfu/mL に等しいことと。ウラシル定款の効率はエシェリヒア属大腸菌CJ236、大腸菌SS320 の力価比から推定できます。大腸菌CJ236 の力価は 9 X 1012 cfu/mL大腸菌SS320 の力価は 3 X 107 cfu/mL。CJ236大腸菌と大腸菌SS320 の力価比は 3 X 10 の5だったこの図の拡大版を表示するのにはここをクリックしてください。

図 6: デュ ssDNA のオリゴヌクレオチド特異的変異導入による CCC DNA への変換。オリゴヌクレオチド特異的変異導入、次の CCC dna デュ ssDNA 変換効率を評価しました。デュ ssDNA は、dsDNA に完全に変換されました。支配的なバンドは CCC dsDNA ニック dsDNA や DNA のストランド避難のマイナーな部分があります。この図の拡大版を表示するのにはここをクリックしてください。
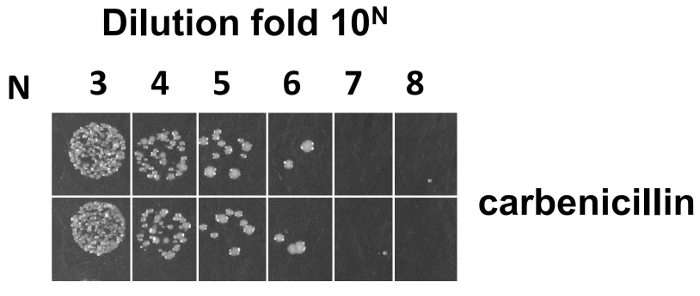
図 7: ライブラリのサイズの計算のためのバクテリオファージの滴定します。大腸菌SS320 に穿孔後ライブラリのサイズは、LB/炭水化物プレートのシリアル希薄から推定しました。サイズの計算式は次のように: M、最も希釈倍 10N (N は 1 ~ 8) 2YT/炭水化物プレートから数えて平均コロニー数をサイズが 2 M × 10N + 3想定します。この図の拡大版を表示するのにはここをクリックしてください。

図 8: 蛋白質 A/L ダイレクト バインディング バクテリオファージ ELISA 。タンパク質 L も折り畳まれた kappa の軽鎖 VL のフレームワークとタンパク質ことができます認識も折り畳まれた重鎖 VH のフレームワークを認識できます。L ・ A 蛋白 Fab の結合は、重鎖と軽鎖の両方の適切な折りたたみを示します。簡単に言えば、3 通と負の制御 M pbst; L 蛋白質がプレートにコーティングされた、異なるクローンから Fab バクテリオファージ清培養タンパク質 L と M PBST、洗浄後、タンパク質 A HRP がバインドされた Fab バクテリオファージをキャプチャする使用されました。バクテリオファージの ELISA の測定値は、成功したシーケンスの読み出しと 90 のランダムに選択されたクローンを示した。肯定的なクローンを表すしきい値を表す線だった経験的どこの比率タンパク質 L から OD450吸光度値 (誤差範囲と 3 通の平均) と負の制御は 3.0 以上を定義します。青 (17 クローン) と緑 (20 クローン) で停止コドン変異なし赤 (53 クローン)、野生型 (WT) で終止コドンの変異に対応するシーケンス解析に基づく 3 つのグループが示されました。この図の拡大版を表示するのにはここをクリックしてください。
| 試薬セットアップ | コンポーネント | 量 | コメント/説明 |
| 2YT 培地 | 酵母エキス | 10 g | 7.0 に pH を調整したり、1.0 L にボリュームを構成する超純水を追加オートクレーブ。 |
| トリプトン | 16 g | ||
| 塩化ナトリウム | 5 g | ||
| 2YT 上の寒天 | 酵母エキス | 10 g | 1.0 L にボリュームを構成して、熱分解、オートクレーブを 7.0 に pH を調整する超純水を追加します。 |
| トリプトン | 16 g | ||
| 塩化ナトリウム | 5 g | ||
| グラニュー寒天 | 7.5 g | ||
| 2YT/炭水化物/cmp 媒体 | Carbenicillin | 100 μ g/mL | |
| クロラムフェニ コール | 10 μ g/mL | ||
| 2YT/炭水化物/菅/ウリジン媒体 | Carbenicillin | 100 μ g/mL | |
| カナマイシン | 50 μ g/mL | ||
| ウリジン | 0.25 μ g/mL | ||
| 2YT/炭水化物/テト中 | Carbenicillin | 100 μ g/mL | |
| テトラサイクリン | 10 μ g/mL | ||
| 2YT/炭水化物中 | Carbenicilin | 100 μ g/mL | |
| 2YT/菅媒体 | カナマイシン | 50 μ g/mL | |
| 2YT/菅/テト中 | カナマイシン | 50 μ g/mL | |
| テトラサイクリン | 10 μ g/mL | ||
| 2YT/テト中 | テトラサイクリン | 10 μ g/mL | |
| 2YT/cmp 媒体 | クロラムフェニ コール | 10 μ g/mL | |
| LB 培地寒天培地 | 酵母エキス | 5 g | 7.0 に pH を調整したり、1.0 L にボリュームを構成する超純水を追加オートクレーブ。LB 寒天、グラニュー寒天 20 g を追加オートクレーブ。 |
| トリプトン | 10 g | ||
| 塩化ナトリウム | 10 g | ||
| LB/炭水化物プレート | LB 寒天培地 | 1 L | |
| Carbenicillin | 100 μ g/mL | ||
| LB/テト プレート | LB 寒天培地 | 1 L | |
| テトラサイクリン | 10 μ g/mL | ||
| LB/cmp プレート | クロラムフェニ コール | 10 μ g/mL | |
| LB/菅プレート | カナマイシン | 50 μ g/mL | |
| SOC 培地 | 酵母エキス | 5 g | 1.0 L にボリュームをアップし、7.0 に pH を調整する超純水を追加オートクレーブ。 |
| トリプトン | 20 g | ||
| 塩化ナトリウム | 0.5 g | ||
| KCl | 0.2 g | ||
| 2.0 M MgCl2 | 5.0 mL | ||
| 1.0 ブドウ糖 M | 20 mL | ||
| Superbroth 培地 | トリプトン | 12 g | 900 ml の純水を追加オートクレーブ、オートクレーブ 0.17 M KH2PO4, 0.72 M K2HPO4100 mL を加えます。 |
| 酵母エキス | 24 g | ||
| グリセロール | 5 mL | ||
| Superbroth 菅/テト中 | カナマイシン | 50 μ g/mL | |
| テトラサイクリン | 10 μ g/mL | ||
| 1X PBS | 塩化ナトリウム | 137 mM | 7.2、pH を調整するオートクレーブ。 |
| KCl | 3 mM | ||
| Na2HPO4 | 8 mM | ||
| KH2PO4 | 1.5 mM | ||
| 泰/agarose のゲル | TAE バッファー | ||
| アガロース | 1% (w/v) | ||
| GelRed | 1:10000 (v/v) | ||
| TMB 基板 | TMB | 50% (v/v) | |
| H2O2ペルオキシダーゼ基質 | 50% (v/v) | ||
| M PBST バッファー | 1X PBS | 100 ml | |
| トゥイーン 20 | 0.05% (v/v) | ||
| 脱脂粉乳 | 5% (v/v) | ||
| 5 X ペグ/食塩 | ペグ-8000 | 20% (w/v) | 1 L とオートクレーブをボリューム アップする超純水を追加します。 |
| 塩化ナトリウム | 2.5 M | ||
| PBST バッファー | 1X PBS | 1 L | 0.22 μ m のフィルター滅菌します。 |
| トゥイーン 20 | 0.05% (v/v) | ||
| TM バッファー x 10 | MgCl2 | 0.1 M | 7.5 pH を調整します。 |
| トリス | 0.5 M | ||
| 1.0 mM HEPES、pH 7.4 | 1.0 M HEPES | 4.0 mL | 0.22 μ m のフィルター滅菌します。 |
| 超純水 | 4.0 L | ||
| 10% (v/v) 超純水グリセロール | 超高純度グリセリン | 100 ml | 0.22 μ m のフィルター滅菌します。 |
| 超純水 | 900 mL | ||
| 超純水 | H20 | 無料の Dnase、Rnase フリー、パイロジェン フリーします。 |
表 1: 試薬のセットアップ。

表 2: CDR 多様性および変異原性プライマー 。赤; で変異する CDR 領域の DNA シーケンスが表示されます。シーケンスは、IUPAC ヌクレオチド コードを使用して書式設定されます。"X"は異なるアミノ酸のセットを格納するために設計された混合物からトライ ヌクレオチドを示します"n"は、X. 5 プライマー X の異なる数を持つの別の番号が CDRL3 または CDRH3 をそれぞれ多様化する、可変長の CDRL3 と CDRH3 を生成するために使用されたことを示します。残基番号が IMGT 命名法によって定義されます。この表をダウンロードするにはここをクリックしてください。
| クンケルの手法の突然変異誘発 | ||
| 反応 1.T4 ポリヌクレオチド キナーゼをオリゴヌクレオチドを用いたリン酸化 | ||
| コンポーネント | 量 | 最終的な |
| 変異原性オリゴヌクレオチド | 0.6 μ g | |
| TM バッファー x 10 | 2 Μ L | 1 X |
| 10 mM ATP | 2 Μ L | 1 mM |
| 100 mM DTT | 1 Μ L | 5 mM |
| T4 ポリヌクレオチド キナーゼ (10 U/μ L) | 2 Μ L | 20 U |
| 超純水の H20 | 20 μ L まで | |
| 反応の設定 | ||
| ステップ 1。 | 1 h の 37 ° C | |
| 反応 2.テンプレートにオリゴヌクレオチドの焼鈍 | ||
| コンポーネント | 量 | 最終的な |
| デュ ssDNA テンプレート | 20 μ g | 20 μ g |
| TM バッファー x 10 | 25 Μ L | 1 X |
| リン酸化 CDRH1 オリゴヌクレオチド | 20 Μ L | 0.6 μ g |
| リン酸化 CDRH2 オリゴヌクレオチド | 20 Μ L | 0.6 μ g |
| リン酸化 CDRH3 オリゴヌクレオチド | 20 Μ L | 0.6 μ g |
| リン酸化 CDRL3 オリゴヌクレオチド | 20 Μ L | 0.6 μ g |
| 超純水の H20 | 250 μ L まで | |
| 反応の設定 | ||
| ステップ 1。 | 90 ° C、3 分 | |
| ステップ 2。 | 50 ° C、5 分 | |
| ステップ 3。 | 20 ° C、5 分 | |
| 反応 3.CCC dsDNA の酵素的合成 | ||
| コンポーネント | 量 | 最終的な |
| 混合焼なましオリゴヌクレオチド/テンプレート | 250 Μ L | |
| 10 mM ATP | 10 Μ L | 各ヌクレオチドの 346 μ M |
| dNTP ミックス (各ヌクレオチドの 25 mM) | 10 Μ L | 各ヌクレオチドの 865 μ M |
| 100 mM DTT | 15 Μ L | 5 mM |
| T4 DNA リガーゼ | 1 Μ L | ヴァイスシュヴァルツに 30 単位 |
| T7 DNA ポリメラーゼ | 3 Μ L | 30 U |
| 反応の設定 | ||
| ステップ 1。 | 一晩のための 20 の ° C | |
表 3: プロシージャおよびクンケルのメソッドのコンポーネント ベースの反応。
| グループ | クローン番号 | 地域 | 割合 | ||
| ないのは時期尚早停止コドン | 70 | CDRH1 変異 | 50% (35/70) | ||
| CDRH2 変異 | 57% (40/70) | ||||
| CDRH3 変異 | 53% (37/70) | ||||
| CDRL3 変異 | 56% (39/70) | ||||
| 少なくとも 1 つの CDR 変異 | 76% (53/70) | ||||
| 時期尚早停止コドン | 20 | CDRH1 欠陥 | 45% (9/20) | ||
| CDRH2 欠陥 | 10% (2/20) | ||||
| CDRH3 欠陥 | 15% (3/20) | ||||
| CDRL3 欠陥 | 20% (4/20) | ||||
| 他の欠陥 | 10% (2/20) | ||||
表 4: CDRH1、CDRH2、CDRH3、および CDRL3 合成の fab 抗体ライブラリーからの解析。
ディスカッション
電子有能なセル、デュ ssDNA テンプレート、効率性の能力を含む建設プロセスのさまざまな段階を監視する必要がチェック ポイント高い多様性、Fab ライブラリのバクテリオファージ表示、品質管理を構築するにはCCC dsDNA 合成、エレクトロポレーション、Fab 折りたたみと Fab phage クローンの解析から Cdr のアミノ酸の多様性後価。
高収量とデュ ssDNA の純度高い突然変異率のために不可欠です。我々 の経験で一晩 25 ° C でバクテリオファージの誘導は一晩 37 ° C でバクテリオファージの誘導からそれよりもっとデュ ssDNA をもたらすことができます。これは以前のレポート19のと一致しています。SsDNA 抽出に関するスピン キット (QIAprep) の初期のプラスミドはバクテリオファージ溶解とバインディングの MLB を含まれています。その後、MLB は未知の理由で中止、PB に置き換え。デュ ssDNA の収量は MLB の治療からと PB 治療から低くわかった.このプロトコルでは MLB を置き換える、デュ ssDNA の収量は類似している初期のスピン キットから UT-MLB20という試薬を使用しました。
CDRH3 と CDRL3、抗原認識21、最も多様な地域特定のアミノ酸の組み合わせや比率のセットに合わせた多様性を紹介する冗長性バイアスを取り外して、終止コドンの縮退のコドンによって導入されるようNNK (N、A/C/G/t のモルK、G/T の等モル)、三量体コドン ホスホロアミダイト ベース オリゴヌクレオチド22 CDRH3 と CDRL3 で設計された特定のアミノ酸に対応する 1 つの三量体コドン。さらに、CDRH3 と CDRL3 のオリゴヌクレオチドの変数の長さを用いて多様性をさらに高めます。
後に CCC dsDNA の酵素的合成は、一般的に 3 つのバンドは agarose のゲルの電気泳動によって観察され、バンドが、にじみのないクリアする必要があります。その中で、最速実行バンドは CCC dsDNA エレクトロポレーション23後高い突然変異率 (約 80%) をもたらすことができます。遅い実行しているバンドは、T7 DNA ポリメラーゼの固有にともなう鎖置換活性から発生している低い突然変異率 (約 20%)23ストランド避難 DNA です。中間の弱いバンドは不十分な T4 DNA リガーゼ活性や不十分なオリゴヌクレオチドのリン酸化のための延長の後 DNA を傷です。
小さなシーケンス サンプル プールが正確ではない24ライブラリ多様性を推定する使用されました。本当の多様性を正確に推定するには、次世代シークエンシング (NGS)25構築されたライブラリの多様性の深さをマイニングの良いオプションがあります。実際には、NGS の現在の課題のため技術を含む読む長さ、精度、コスト、および高スループット、約 950 の長さでこのプロトコルで使われる Fab バクテリオファージライブラリのシーケンス CDRH1、CDRH2、CDRH3、および CDRL3 にまたがる bp はないです。達成されます。しかし、それは、scFv を推定することが可能 (約 700 bp) 何百万の範囲内でライブラリ多様性24,25。構築されたライブラリの多様性を評価する別のキー標準ライブラリを使用してさまざまな種類の抗原に対してパンおよびキャプチャ ライブラリ多様性直接抗原の成功率と相関しているので肯定的なクローンを計算することです。26をパン。この目的のため、ハイスループット選択プラットフォームが適しており、読者は Mierschらによって報告された詳細なプロトコルを参照することができます。27
理論上は、任意の抗原をターゲットしてこのように広範なアプリケーションに合わせた多様性を持つ合成抗体のバクテリオファージの表示ライブラリを使用できます。現在、ケンブリッジ抗体技術 (猫)、MedImmune、ジェネンテック、Dyax、Bioinvent、ファイザー、MorphoSys などの企業は、治療用抗体開発28バクテリオファージ表示プラットフォームに重く頼る。また、多くのバクテリオファージ表示コア技術特許の29 を切れた。間違いなく、これが抗体のバクテリオファージ表示技術のポテンシャルを最大限を発揮します。
開示事項
著者が明らかに何もありません。
謝辞
著者は、シンドゥ ラボ クンケルの法合成工場バクテリオファージ図書館建設に批判的なコメントから博士フレデリック Fellouse を感謝しています。著者夫人 Alevtina Pavlenco と高効率電気主務の準備の貴重な助けのシドゥ ラボから他のメンバーに感謝エシェリヒア属大腸菌のセルと高品質デュ ssDNA。この作品は中国の国家自然科学基金によって支えられた (グラント号: 81572698、31771006) DW と ShanghaiTech 大学 (グラント号: F-0301-13-005) 抗体工学研究室に。
資料
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1.0 M H3PO4 | Fisher | AC29570 | |
| 1.0 M Tris, pH 8.0 | Invitrogen | 15568-025 | |
| 10 mM ATP | Invitrogen | 18330-019 | |
| 100 mM dithiothreitol | Fisher | BP172 | |
| 100 mM dNTP mix | GE Healthcare | 28-4065-60 | solution containing 25 mM each of dATP, dCTP, dGTP and dTTP. |
| 3,3’,5,5’-tetramethylbenzidine (TMB) | Kirkegaard & Perry Laboratories Inc | 50-76-02 | |
| 50X TAE | Invitrogen | 24710030 | |
| Agarose | Fisher | BP160 | |
| Carbenicillin, carb | Sigma | C1389 | 100 mg/mL in water, 0.22 μm filter-sterilize, work concentration: 100 μg/mL. |
| Chloramphenicol, cmp | Sigma | C0378 | 100 mg/mL in ethanol, 0.22 μm filter-sterilize, work concentration: 10 μg/mL. |
| EDTA 0.5 M, pH 8.0 | Invitrogen | AM9620G | |
| Granulated agar | VWR | J637-500G | |
| H2O2 peroxidase substrate | Kirkegaard & Perry Laboratories Inc | 50-65-02 | |
| K2HPO4 | Sigma | 795488 | |
| Kanamycin, kan | Fisher | AC61129 | 50 mg/mL in water, 0.22 μm filter-sterilize, work concentration: 50 μg/mL. |
| KH2PO4 | Sigma | P2222 | |
| Na2HPO4 | Sigma | 94046 | |
| NaCl | Alfa Aesar | U19C015 | |
| Nanodrop | Fisher | ND2000C | |
| NaOH | Fisher | SS256 | ! CAUTION NaOH causes burns. |
| NON-Fat Powdered Milk | Sangon Biotech | A600669 | |
| PEG-8000 | Fisher | BP233 | |
| Protein A-HRP conjugate | Invitrogen | 101123 | |
| QIAprep Spin M13 Kit | Qiagen | 22704 | |
| QIAquick Gel Extraction Kit | Qiagen | 28706 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| Recombinant Protein L | Fisher | 77679 | |
| T4 DNA polymerase | New England Biolabs | M0203S | |
| T4 polynucleotide kinase | New England Biolabs | M0201S | |
| T7 DNA polymerase | New England Biolabs | M0274S | |
| Tetracycline, tet | Sigma | T7660 | 50 mg/mL in water, 0. 22 μm filter-sterilize, work concentration: 10 μg/mL. |
| Tryptone | Fisher | 0123-07-5 | |
| Tween-20 | Sigma | P2287 | |
| Ultrapure glycerol | Invitrogen | 15514-011 | |
| Uridine | Sigma | U3750 | 25 mg/mL in ethanol, work concentration: 0.25 μg/mL. |
| Yeast extract | VWR | DF0127-08 | |
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E.coli CJ236 | New England Biolabs | E4141 | Genotype: dut- ung- thi-1 relA1 spoT1 mcrA/pCJ105(F' camr). Used for preparation of dU-ssDNA. |
| E.coli SS320 | Lucigen | 60512 | Genotype: [F'proAB+lacIq lacZΔM15 Tn10 (tetr)] hsdR mcrB araD139 Δ(araABC-leu)7679 ΔlacX74 galUgalK rpsL thi. Optimized for high-efficiency electroporation and filamentous bacteriophage production. |
| M13KO7 | New England Biolabs | N0315S | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| 0.2-cm gap electroporation cuvette | BTX | ||
| 96-well 2mL Deep-well plates | Fisher | 278743 | |
| 96-well Maxisorp immunoplates | Nunc | 151759 | |
| Baffled flasks | Corning | ||
| Benchtop centrifuge | Eppendorf | 5811000096 | |
| Centrifuge bottles | Nalgene | ||
| ECM-630 electroporator | BTX | ||
| Magnetic stir bars | Nalgene | ||
| Thermo Fisher centrifuge | Fisher | ||
| High speed shaker | TAITEK | MBR-034P | |
| Microplate shaker | QILINBEIER | QB-9002 | |
| Liquid handler for 96 and 384 wells | RAININ | ||
| Mutil-channel pipette | RAININ | E4XLS | |
| Amicon concentrator | Merck | UFC803096 |
参考文献
- Singh, S., et al. Monoclonal Antibodies: A Review. Curr Clin Pharmacol. , (2017).
- Reichert, J. M. Antibodies to watch in 2017. MAbs. 9 (2), 167-181 (2017).
- Kohler, G., Milstein, C. Continuous cultures of fused cells secreting antibody of predefined specificity. Nature. 256 (5517), 495-497 (1975).
- Milstein, C. The hybridoma revolution: an offshoot of basic research. Bioessays. 21 (11), 966-973 (1999).
- Gray, A. C., Sidhu, S. S., Chandrasekera, P. C., Hendriksen, C. F., Borrebaeck, C. A. Animal-Friendly Affinity Reagents: Replacing the Needless in the Haystack. Trends Biotechnol. 34 (12), 960-969 (2016).
- Hutchison, C. A., et al. Mutagenesis at a specific position in a DNA sequence. J Biol Chem. 253 (18), 6551-6560 (1978).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Sidhu, S. S. . Phage Display in Biotechnology and Drug Discovery. , (2005).
- Weiss, G. A., Watanabe, C. K., Zhong, A., Goddard, A., Sidhu, S. S. Rapid mapping of protein functional epitopes by combinatorial alanine scanning. Proc Natl Acad Sci U S A. 97 (16), 8950-8954 (2000).
- Frenzel, A., Schirrmann, T., Hust, M. Phage display-derived human antibodies in clinical development and therapy. MAbs. 8 (7), 1177-1194 (2016).
- Sidhu, S. S., Fellouse, F. A. Synthetic therapeutic antibodies. Nat Chem Biol. 2 (12), 682-688 (2006).
- Adams, J. J., Sidhu, S. S. Synthetic antibody technologies. Curr Opin Struct Biol. 24, 1-9 (2014).
- Kunkel, T. A. Rapid and efficient site-specific mutagenesis without phenotypic selection. Proc Natl Acad Sci U S A. 82 (2), 488-492 (1985).
- Sidhu, S. S., Lowman, H. B., Cunningham, B. C., Wells, J. A. Phage display for selection of novel binding peptides. Methods Enzymol. 328, 333-363 (2000).
- Persson, H., et al. CDR-H3 diversity is not required for antigen recognition by synthetic antibodies. J Mol Biol. 425 (4), 803-811 (2013).
- Lefranc, M. P., et al. IMGT unique numbering for immunoglobulin and T cell receptor variable domains and Ig superfamily V-like domains. Dev Comp Immunol. 27 (1), 55-77 (2003).
- Graille, M., et al. Complex between Peptostreptococcus magnus protein L and a human antibody reveals structural convergence in the interaction modes of Fab binding proteins. Structure. 9 (8), 679-687 (2001).
- Graille, M., et al. Crystal structure of a Staphylococcus aureus protein A domain complexed with the Fab fragment of a human IgM antibody: structural basis for recognition of B-cell receptors and superantigen activity. Proc Natl Acad Sci U S A. 97 (10), 5399-5404 (2000).
- Huang, R., Fang, P., Kay, B. K. Improvements to the Kunkel mutagenesis protocol for constructing primary and secondary phage-display libraries. Methods. 58 (1), 10-17 (2012).
- Chen, G., Sidhu, S. S. Design and generation of synthetic antibody libraries for phage display. Methods Mol Biol. 1131, 113-131 (2014).
- Padlan, E. A. Anatomy of the antibody molecule. Mol Immunol. 31 (3), 169-217 (1994).
- Virnekas, B., et al. Trinucleotide phosphoramidites: ideal reagents for the synthesis of mixed oligonucleotides for random mutagenesis. Nucleic Acids Res. 22 (25), 5600-5607 (1994).
- Fellouse, F. A., Sidhu, S. S. . Making and Using Antibodies: A Practical Handbook. , 157-180 (2006).
- Fantini, M., et al. Assessment of antibody library diversity through next generation sequencing and technical error compensation. PLoS One. 12 (5), e0177574 (2017).
- Glanville, J., et al. Deep sequencing in library selection projects: what insight does it bring?. Curr Opin Struct Biol. 33, 146-160 (2015).
- Perelson, A. S., Oster, G. F. Theoretical studies of clonal selection: minimal antibody repertoire size and reliability of self-non-self discrimination. J Theor Biol. 81 (4), 645-670 (1979).
- Miersch, S., et al. Scalable high throughput selection from phage-displayed synthetic antibody libraries. J Vis Exp. (95), e51492 (2015).
- Ponsel, D., Neugebauer, J., Ladetzki-Baehs, K., Tissot, K. High affinity, developability and functional size: the holy grail of combinatorial antibody library generation. Molecules. 16 (5), 3675-3700 (2011).
- Petering, J., McManamny, P., Honeyman, J. Antibody therapeutics - the evolving patent landscape. N Biotechnol. 28 (5), 538-544 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved