Method Article
Construction du bactériophage synthétique affiche Fab bibliothèque avec diversité sur mesure
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole décrit une procédure détaillée pour la construction d’une bibliothèque d’anticorps synthétiques phage-affiché avec diversité sur mesure. Anticorps synthétiques ont des applications larges depuis la recherche fondamentale pour le diagnostic de la maladie et la thérapeutique.
Résumé
Demande d’anticorps monoclonaux (ACM) dans la recherche fondamentale et de la médecine est en augmentation chaque année. Hybridome a été la méthode dominante pour mAb développement depuis son premier rapport en 1975. Comme une technologie alternative, méthodes d’affichage bactériophage mAb développement sont plus en plus attrayantes car Humira, du premier anticorps dérivés de phage et l’un du mAbs Best-seller, a été approuvé pour le traitement clinique de la polyarthrite rhumatoïde en 2002. Comme un animal non fondée mAb développement de la technologie, affichage de phage contourne immunogénicité des antigènes, humanisation et maintenance animale qui sont exigés d’hybridome traditionnel technicisées anticorps développement. Dans ce protocole, nous décrivons une méthode de construction des synthétiques bibliothèques Fab phage-affichée avec les diversités de de9-10 1010 obtenue avec une seule électroporation. Ce protocole se compose de : 1) préparation de haut rendement cellule electro-compétente ; 2) extraction de l’uracile-contenant de l’ADN simple brin (dU-ssDNA) ; 3) méthode de Kunkel base mutagénèse oligonucléotide-dirigée ; 4) électroporation et calcul de la taille de la bibliothèque ; 5) protéine A/L-base-immuno enzymatique (ELISA) pour le pliage et l’évaluation de la diversité fonctionnelle ; et 6) analyse des séquences ADN de diversité.
Introduction
mAbs ont des applications larges, allant de la recherche fondamentale à la thérapeutique et le diagnostic de la maladie. À partir de 2016, plus de 60 mAbs ont été approuvés par les United States Food Drug Administration (FDA) pour le traitement clinique des maladies auto-immunes, le cancer et les maladies infectieuses1,2.
En 1975, Kohler et Milstein a signalé une technique pour la production continue d’anticorps d’une spécificité unique clonale provenant d’une source cellulaire dénommée « hybridomes » et cette technique est par la suite devenu la pierre angulaire de la médecine et l’industrie3 ,4. Génération du mAbs par cette méthode nécessite différentes étapes, y compris la production de l’antigène, immunisation de souris, extraction des lymphocytes B, fusion de cellules de B avec cellules myélomateuses pour former des hybridomes immortel, sélection de clone et pour des applications thérapeutiques, humanisation est nécessaire pour éviter les humains d’anticorps anti-souris (HAMA)4,5. Toutefois, pour cette technologie, y compris les toxines, agents pathogènes et des protéines hautement conservées des antigènes sont relativement inefficaces pour déclencher une réponse immunitaire in vivo pour mAb production5.
En 1978, Hutchison et coll. ont signalé l’utilisation d’un oligonucléotide de mutagenèse directe d’un résidu dans un simple brin bactériophage virus6. En 1985, Smith a indiqué que les fragments de gènes étrangers peuvent être fusionnés dans le cadre avec le gène codant pour la protéine de capside phage III et donc peuvent être affichées sur l’aire de phage sans compromettre son infectiosité7. Ces travaux de pionniers jeté les bases pour la construction ultérieure des bibliothèques phage-affiche anticorps immunitaire, naïf et synthétique forme avec les formats de chaîne unique variable fragment (scFv) et fragment de liaison de l’antigène (Fab) pour thérapeutique mAb development8,9. Du point de vue technique, développement axée sur l’affichage des anticorps phage propose une approche complémentaire au développement axée sur les hybridomes mAb qui peut aider à contourner les limitations que peuvent poser certains antigènes et l’humanisation du processus qui anticorps hybridome nécessitent souvent5. À partir de 2016, 6 phage affichage dérivé mAbs ont été approuvés dans le marché, y compris Humira, l’un des plus réussis mAbs utilisé pour le traitement de la polyarthrite rhumatoïde, et de nombreux candidats d’anticorps dérivés affichage phage sont actuellement à différents stades de la clinique enquête10.
Immunitaire et naïve bibliothèques anticorps phage, la diversité des déterminant la complémentarité régions (CDRs) de chaîne légère et lourde est dérivé du répertoire immunitaire naturel (c'est-à-diredes cellules B). En revanche, la diversité des CDRs dans les bibliothèques d’anticorps bactériophage synthétique est entièrement artificielle. Des approches synthétiques pour la construction de la bibliothèque offrent un contrôle précis sur la conception de la diversité des séquences et offrent des possibilités d’études mécanistes de la structure des anticorps et fonctionnent11,12. En outre, le cadre pour les bibliothèques synthétiques peut être optimisé avant la construction de la bibliothèque pour faciliter en aval, le développement industriel à grande échelle11,12.
En 1985, Kunkel a signalé une approche de mutagenèse basés sur un modèle simple brin ADN (ADN simple brin) à introduire des mutations dirigée dans bactériophage M13 efficacement13. Cette approche a été par la suite largement utilisée pour la construction de bibliothèques phage-affiché. Chimiquement synthétisés oligonucléotides d’ADN visant à introduire la diversité dans les CDRs Fab sont incorporés dans un phagemid avec un modèle de colonne vertébrale d’anticorps. Dans ce processus, le phagemid s’exprime comme un ADN simple brin contenant de l’uracile (dU-ADN simple brin) et les oligonucléotides sont recuites sur les CDRs et étendus pour synthétiser l’ADN à double brin (dsDNA) en présence de T7 ADN polymérase et T4 DNA ligase. Enfin, ds-ADN généré peut être introduit dans Escherichia coli par électroporation.
Pour forte diversité, construction de la bibliothèque de phage-affiché, électroporation haute tension d’un mélange de deux composants de cellules capables d’electro et de façon covalente dsDNA circulaire fermé (CCC-ADNdb) doit être préparé soigneusement. Sidhu et coll. modifié la préparation de cellules capables d’electro et d’ADN de méthodes traditionnelles et grandement amélioré bibliothèque diversité14.
Dans ce protocole, nous décrivons une méthode de construction des synthétiques bibliothèques Fab phage-affichée avec les diversités de de9-10 1010 obtenue avec une seule électroporation. La figure 1 représente une vue générale de construction de la bibliothèque, y compris : 1) préparation de haut rendement cellule electro-compétente ; 2) extraction dU-ADN simple brin ; 3) méthode de Kunkel base mutagénèse oligonucléotide-dirigée ; 4) électroporation et calcul de la taille de la bibliothèque ; 5) protéine A/L-base ELISA pour le pliage et l’évaluation de la diversité fonctionnelle ; et 6) analyse des séquences ADN de diversité. Tous les réactifs, les souches et les équipements sont répertoriées dans le tableau du matériau. Le tableau 1 illustre la configuration du réactif.
Protocole
NOTE : Pointes à filtre stériles doivent servir tout au long de lorsqu’ils traitent de phage pour éviter la contamination de pistolet de pipette et environs. Aire aseptique ou la hotte doit être utilisé lorsque les expériences de manipulation avec les bactéries et les bactériophages. Phage expérience aire doit être nettoyée utilisant 2 % dodécylsulfate de sodium (SDS) suivie de l’éthanol à 70 % pour éviter la contamination du phage. Pour faire des dilutions en série dans le présent protocole, les nouvelles astuces devraient servir pour chaque dilution.
1. préparation de cellules Electro-compétente pour le SS320 e. Coli
- Préchauffez gélose LB/tet (préparés et stockés à 4 ° C pour moins de 1 - semaine vieux) à l’incubateur de 37 ° C pendant 1 h. utiliser une anse à inoculation stérile ou un embout stérile à ensemencer sur un stock de glycérol d’e. coli SS320 cellules sur la gélose LB/tet au pré chauffée dans une direc zig-zag tion doucement le long de la surface de la plaque. Incuber la plaque à l’incubateur de 37 ° C pendant la nuit pour environ 12-15 h.
- Le lendemain, utiliser une anse à inoculation stérile ou un embout stérile pour prélever une colonie unique bien séparée le long de la ligne de zig-zag et ensemencer dans 10 mL de milieu de 2YT/tet dans un tube de 50mL fond rond en plongeant la boucle dans le milieu et en agitant brièvement.
- Incuber à 37 ° C sous agitation à 200 tr/min pendant environ 3-4 h, jusqu'à ce que OD600 est atteint à autour de 0,4 à 0,8 (log phase de croissance). Moniteur OD600 à 2 h. Durant cette période, réchauffez 5 plaques de gélose LB/tet (préparés et stockés à 4 ° C pour moins de 1 - vieux semaine) dans un incubateur à 37 ° C pendant au moins 1 h.
- Ajouter phage de helper 20 μL M13KO7 du stock de laboratoire avec titre environ 1 x 1013 colonies formant unité (UFC) par millilitre dans 180 μL de stérile 1 X PBS dans des tubes de 1,5 mL stérilisés à l’autoclave de préparer 10 dix fois des dilutions successives.
- Aliquote 4 mL stérilisés à l’autoclave et liquide 2YT albums agar dans 5 X 14 mL bas tubes ronds, étiqueter chaque tube avec une dilution de la cinquième à la neuvième de la dilution dix fois, respectivement et incuber dans un incubateur à 65 ° C pour maintenir la gélose haut de 2YT à l’état liquide.
- Mix 500 μL d’e. coli SS320 de la phase logarithmique de cellules dans le tube de dilution M13KO7 (choisissez le cinquième dilution dix fois à la neuvième dix fois dilution) et incuber dans un incubateur à 37 ° C pendant 5-10 min.
- Au cours de l’incubation de l’étape 1.6, transfert 2YT agar haut de la page de l’étape 1.5 à température ambiante (RT) se refroidir pendant environ 5 min à environ 42 ° C. Poignet intérieur permet de tester la température de 2YT dessus agar ; il doit rester liquide. Transférer chaque mélange de dilution de l’étape 1.5 dans chaque tube de gélose albums 2YT correspondante. Serrer le couvercle de chaque tube et mélanger en tournant à l’envers plusieurs fois doucement et brièvement pour éviter la génération de bulle.
- Soigneusement versez chaque mélange le long d’une plaque de gélose LB/tet Préchauffez (de l’étape 1.3) et incliner la plaque légèrement pour pleinement et régulièrement remplir la plaque avec le mélange tout en évitant l’introduction de bulles. Maintenir les plaques à RT pendant environ 5-10 min pour solidifier l’agar albums au sein de chaque plaque et incuber les géloses haut 2YT à 37 ° C durant la nuit pendant environ 15-18 h.
- Choisissez la plaque de dilution après la croissance durant la nuit à l’étape 1.8 avec autour de 100-200 plaques de taille moyenne, simples, bien séparées pour l’inoculation de la plaque. Utilisez une main pour tenir la gélose contre une source de lumière et l’autre main pour tenir un pipette pistolet chargé avec un embout de la pipette longtemps stérile de poignarder verticalement sur l’agar de haut et de recueillir une plaque unique et bien séparée.
- Incliner l’embout de la pipette légèrement pour séparer les albums agar avec plaque. Pipette de monter et descendre plusieurs fois pour déloger l’agar avec plaque dans un tube à culture fond rond 14 mL pré-chargé avec 1 mL de milieu 2YT/kan/tet (voir tableau 1).
- Répétez cette procédure pour récupérer des plaques de 3-5 au total. La cueillette de plusieurs plaques vise à assurer l’inoculation réussie, comme une plaque peut être faible et relativement faible en comparaison avec une colonie de bactéries. Pousser les plaques pendant 8 h à 37 ° C sous agitation à 200 tr/min.
- Transfert d’un tube de culture de plus en plus dans 50 mL de milieu de 2YT/kan/tet dans un ballon jaugé de 250 mL dérouté. Croissance à 37 ° C sous agitation à 200 tr/min pendant la nuit pendant environ 12 h.
- Inoculer les trois flacons déroutés 2-L, contenant 900 mL de superbroth/tet/kan milieu chaque avec 5 mL de la culture au jour le jour. Incuber à 37 ° C sous agitation à 200 tr/min pour 6-7 h à OD600 autour de 0,6 à 0,8.
- Faites refroidir les trois flacons de culture bactérienne dans un bain de glace pendant 5 min avec tourbillonnant douce constante à la main. Les étapes suivantes devraient être faites dans une chambre froide, sur la glace, avec des solutions prérefroidies et équipement.
- Utiliser un chiffon tissu absorbance pour sécher la vitre extérieure de chaque fiole ; Utilisez une main pour incliner la bouteille de 1 L autoclavés centrifugeuse sur le banc et l’autre à verser le milieu doucement de chaque fiole dans chaque bouteille de centrifugeuse.
- Tourner à 5 000 × g et 4 ° C pendant 10 min granuler les bactéries.
- Après centrifugation, délicatement éliminer le surnageant dans un bécher de déchets autoclavé 5 L.
- Remplir chaque flacon de 100 mL stérile filtré 1,0 mm HEPES, pH 7,4 et ajouter une barre d’autoclavés agitation magnétique à chaque bouteille pour aider à la remise en suspension de pellet à 200 tr/min. Agiter et déloger le culot d’ensemble de la paroi de la bouteille chaque plusieurs minutes au cours de l’agitateur magnétique. Une fois que le plomb se dissout, remplir chaque bouteille avec une autre 400 mL stérile filtré 1,0 mm HEPES, pH 7,4.
- Centrifuger à 5 000 × g et 4 ° C pendant 10 min. décanter le liquide surnageant, conservant la barre de l’émoi dans la bouteille.
- Répétez les étapes 1.16 à 1,17 fois.
- Resuspendre chaque culot dans 500 mL de stérile filtré, 10 % glycérol ultrapure à l’aide des barres de remuer. Agiter et déloger le culot d’ensemble de la paroi de la bouteille chaque plusieurs minutes au cours de l’agitateur magnétique.
- Centrifuger et décanter comme à l’étape 1.17. Répétez l’étape 1,19 fois. Personam pinces stérilisés à l’autoclave permet d’enlever la barre de remuer.
- Centrifuger à 5 000 × g et 4 ° C pendant 15 min. décanter le liquide surnageant et supprimer toutes les traces restantes de surnageant de chaque bouteille de centrifugeuse avec une pipette.
- Ajouter 3,0 mL de 10 % de glycérol ultrapure à une bouteille et doucement Resuspendre le culot de pipetage. Transférer la suspension dans la bouteille suivante et répéter jusqu'à ce que toutes les granules sont remis en suspension et combinées dans une bouteille. Environ 6 mL de cellules hautement concentrés sont obtenus avec un titre d’environ 3 x 1011 cfu/mL.
- Avant chill 1,5 mL stérilisés à l’autoclave microtubes à centrifuger et boîte de rangement d’un tube de 96 puits sans séparateurs à-80 ° C pendant au moins 1 h avant cette étape. Transférer les tubes de microcentrifuge préalablement réfrigérées à la chambre froide sur glace avant de pipetage aliquotes de la suspension du culot cellulaire. Utiliser une pipette pour Aliquoter 350 μL de la suspension cellulaire dans chaque tube de microtubes de 1,5 mL.
- Transférer les aliquotes dans un mousse boîte contenant rempli d’azote liquide pour congélation flash (3-5 min).
- Transport de la boîte de mousse à l’azote liquide pour un congélateur à-80 ° C (Voir l’étape 1,23).
- Supprimer la boîte de rangement de tube microtubes de 1,5 mL du congélateur à-80 ° C (Voir l’étape 1,23) et mettre sur la glace.
- Rapidement, utiliser un treillis métallique à Tamiser les aliquotes de l’azote liquide et les placer dans la boîte de rangement de tube. Conserver à-80 ° C.
ATTENTION : L’azote liquide peut causer des brûlures et le soin doit être pris pour des mesures de protection. Utilisez une colonne vertébrale Fab phagemid pour vérifier l’efficacité des cellules capables d’electro préparées (le stock de phagemid Fab épine dorsale devrait être dans l’eau ultrapure (MilliQ) à 400 ng/µL). Décongeler 1 μg de la phagemid colonne vertébrale Fab à 10 µL d’eau ultrapure et une portion de 350 μL de SS320 electro-compétente sur la glace et utiliser son préalable d’une cuvette d’électroporation de 0,2 cm écart sur la glace.
- Ajouter les 350 μL electro-SS320 les cellules compétentes à l’ADN de 10 µL après décongélation et mélanger en pipettant également plusieurs fois tout en gardant les mélanges sur la glace. Éviter d’introduire des bulles.
- Réchauffez 15 mL de milieu SOC dans un tube de 50mL dans un bain d’eau de 37 ° C pendant au moins 30 min avant l’électroporation. Transvaser le mélange de 360 μL dans la cuvette et effectuer l’électroporation suivant les instructions du fabricant.
- Immédiatement, sauver les cellules après électroporation en ajoutant 1 mL de milieu SOC préchauffé (de l’étape 1.27) et de pipetage. Transférer le milieu de la cuve dans une fiole de 125 mL pré-chargé avec 5 mL de SOC préchauffé.
- Rincer la cuvette deux fois, chaque fois avec 1 mL de milieu SOC préchauffé et transférer le support dans le flacon de 125mL (voir étape 1.27). Ajouter préchauffé moyen SOC pour un volume final de 10 mL pour le flacon de 125mL.
- Incuber la culture cellulaire de 10 mL pour 30 min à 37 ° C sous agitation à 200 tr/min.
- Faire des dilutions successives afin de déterminer l’efficacité de transfection et le taux d’infection avant de M13KO7.
- Une pipette multicanaux permet d’ajouter 180 μL de médias 2YT dans chaque puits d’une seule colonne d’une plaque de 96 puits.
- Faire 8 série dix fois les dilutions : 20 μL de la culture de l’étape 1,29 pour le premier puits de la plaque, mix en pipettant également et du transfert 20 μl du mélange à l’autre bien. Répétez cette étape jusqu'à la fin de la dilution en série.
- Plaque de 10 μl de chaque dilution en série sur les plaques LB/carb, LB/kan et LB/tet en double exemplaire.
- Incuber une nuit à 37 ° C.
- Formule de calcul de l’efficacité : Supposons que M est le nombre moyen de colonie compté à partir du pli dilué 10N (N étant de 1 à 8). L’efficacité de transfection de cellule electro-compétente de SS320 e. coli de la phagemid colonne vertébrale Fab de plaque LB/carb est égale à M X 10N + 3 UFC/µg. Le taux d’infection avant de M13KO7 est estimé à partir du ratio des colonies en LB/kan et LB/tet. Le pourcentage des e. coli SS320 compétentes cellules transfectées avec le phagemid Fab épine dorsale est estimé par le ratio des colonies en LB/carb et LB/kan.
2. préparation contenant de l’uracile ADNsb (dU-ssDNA) du modèle Phagemid
NOTE : A signalé auparavant épine dorsale Fab phagemid a été utilisé comme modèle pour dU-ssDNA préparation15. L’architecture de la colonne vertébrale Fab phagemid est illustré à la Figure 2. Un kit de spin du plasmide (QIAprep Spin M13) est utilisé pour l’extraction dU-ssDNA avec de légères modifications.
- Réchauffez une plaque LB/cmp (préparés et stockés à 4 ° C pour moins de 1 - vieux semaine) dans un incubateur à 37 ° C pendant 1 h. Streak sur un stock de glycérol de cellules CJ236 d’Escherichia coli (ou une autre souche de dut−/ung−) avec boucles stériles ou des conseils sur la plaque de LB/cmp préchauffée. Incuber les plaques à 37 ° C durant la nuit pendant environ 12-15 h.
- Prélever une colonie unique avec une pointe stérile et ensemencer dans 2 mL de milieu de 2YT/cmp dans un tube en polypropylène à fond rond 14 mL.
- Incuber le milieu à 37 ° C sous agitation à 200 tr/min pendant 3-4 h, jusqu'à ce que OD600 est atteint à autour de 0,4 à 0,8 (log phase de croissance).
- Ajouter 5-10 phages μL de modèle de colonne vertébrale Fab dans la culture et incuber à 37 ° C sous agitation à 200 tr/min pendant 30 min permettre l’infection par le phage.
- Après incubation, utiliser un embout stérile à strie sur 10 μL de culture sur une plaque LB/carb pré chauffée à 37 ° C. Incuber à 37 ° C durant la nuit pendant environ 12-15 h.
- Choisir une seule colonie de la CJ236 d’e. coli contenant épine dorsale Fab phagemid pour ensemencer un démarreur de 2YT/carb/cmp 3 mL dans un tube en polypropylène à fond rond 14 mL et se développer à 37 ° C sous agitation à 200 tr/min pendant la nuit pendant environ 12 h.
- Ensemencer la culture starter 0,3 mL 30 mL 2YT/carb/cmp dans une bouteille de 250 mL déroutée.
- Incuber les cultures cellulaires à 37 ° C sous agitation à 200 tr/min pendant 3-4 h, jusqu'à ce que OD600 est atteint à autour de 0,4 à 0,8 (log phase de croissance).
- Ajouter le M13KO7 (titre stock, laboratoire d’environ 1 x 1013 UFC/mL) à la culture de l’étape 2.8 avec une multiplicité d’infection (MOI) d’environ 10 et le titre final du M13KO7 est d’environ 1 x 1010 UFC/mL.
- Incuber à 200 tr/min et 37 ° C pendant 1 h.
- La culture de granule par centrifugation dans un tube à fond rond 50 mL à 5 000 × g et 25 ° C pendant 20 min.
- Décanter le liquide surnageant et Resuspendre le culot avec 30 mL de frais 2YT/carb/kan/uridine. Transvaser la remise en suspension dans une nouvelle bouteille déroutée de 250 mL.
- Incuber à 200 tr/min et 25 ° C pendant 22 à 24 heures.
- Transfert de la culture de l’étape 2.13 dans un tube à fond rond 50 mL et centrifuger la culture à 12 000 x g pendant 20 min pour séparer le phage surnageant du culot cellulaire bactérienne. Transvaser le phage surnageant dans un nouveau tube fond rond de 50 mL et ajouter 1/5 le volume final de PEG/NaCl solution pour précipiter le phage. Bien mélanger et laisser incuber sur glace pendant 30 min à précipiter les particules phagiques.
- Centrifuger à 12 000 × g et 4 ° C pendant 30 min. décanter le liquide surnageant et centrifuger à 4 000 × g et 4 ° C pendant 2 min. aspirer le surnageant restant.
- Resuspendre le culot de phage dans 2 mL de stérile filtrée 1 X PBS et transfert pour microtubes de 1,5 mL à. Centrifuger à 12 000 x g pendant 5 min dans une micro-centrifugeuse benchtop enlever tous les débris bactériens restants et de transférer le phage surnageant dans de nouveaux tubes de microcentrifuge de 1,5 mL et de stocker à 4 ° C.
- Vérifier l’efficacité d’incorporation d’uracile dans e. coli CJ236 par comparaison côte à côte avec e. coli SS320.
- Ajouter 180 μL de médias 2YT dans chaque puits d’une seule ligne d’une plaque de 96 puits.
- Faire dix 10 fois série dilutions : transférer 20 μL du phage surnageant dans le premier puits de la plaque, mélanger en pipettant également et transférer 20 μl du mélange à l’autre bien. Répétez cette étape jusqu'à la fin de la dilution en série.
- Ajouter 10 μL de phage de huit derniers des dilutions successives d’infecter 90 μL d’e. coli CJ236 et SS320 d’e. coli en phase logarithmique (OD600 = 0,4 à 0,8). Incuber à 37 ° C pendant 30 min avec agitant doucement.
- Plaque de 10 μl de la dilution en série de chaque infection à e. coli CJ236 et SS320 d’e. coli sur les plaques 2YT/Carb en double exemplaire.
- Incuber une nuit à 37 ° C.
- Formule de calcul de titre : Supposons que M est le nombre moyen de colonie compté à partir du pli dilué 10N (N étant de 1 à 10). Le titre de CJ236 d’e. coli ou e. coli SS320 est égal à M X 10N + 2 UFC/mL. Évaluer l’efficacité de l’incorporation d’uracile le rapport entre le titre d’e. coli CJ236 et e. coli SS320.
- Ajouter du volume 1/100 de la mémoire tampon de précipitations de phage MP dans le phage surnageant dans des microtubes de 1,5 mL à et faire tourner à l’envers doucement pour mélanger plusieurs fois. Incuber à RT pendant au moins 2 min. Phage particules sont précipités en milieu de culture, et ainsi, une solution trouble doit être visible à ce stade.
- Déposer l’échantillon de l’étape 2.18 à une colonne de spin de plasmide (p. ex., QIAprep) dans un tube de microtubes de 1,5 mL. La capacité de liaison d’une colonne d’ADNsb peut atteindre au moins 10 µg. Centrifuger à 6 000 × g et 25 ° C pendant 30 s dans une micro-centrifugeuse benchtop. Jeter le cheminement qui est dans le tube de microtubes de 1,5 mL. Particules phagiques restent liés à la matrice colonne à ce stade.
- Ajouter 0,7 mL de l’UT-MLB phage lyse et le tampon de liaison (voir Discussion) à la colonne et incuber à RT pendant au moins 1 minute. Centrifuger à 6 000 x g et 25 ° C pendant 30 s et jeter le cheminement.
- Ajouter un autre 0,7 mL de tampon UT-MLB et incuber à RT pendant au moins 1 minute.
- Centrifuger à 6 000 × g pendant 30 s. jeter le cheminement. À ce stade, la protéine de capside du phage est séparée dU-ADN simple brin, qui reste lié à la matrice colonne.
- Ajouter 0,7 mL d’éthanol contenant de lavage tampon PE suivant les instructions du fabricant. Centrifuger à 6 000 × g pendant 30 s et jeter le cheminement.
- Répétez l’étape 2.23, puis centrifuger une fois de plus à 6 000 × g pendant 30 s pour retirer le tampon résiduel de PE.
- Transférer la colonne dans un nouveau tube de microtubes de 1,5 mL et ajouter 100 μL de tampon d’élution EB (10 mM Tris· CL, pH 8,5) au centre de la membrane de la colonne.
- Incuber à RT pendant 10 min et centrifuger à 6 000 × g pendant 1 min éluer dU-ADN simple brin. Approximativement, la culture de 1,5 à 2,5 μg dU ADNsb/mL peut être obtenue.
- Analyser l’ADN éluée par electrophoresing 1 μL sur un TAE gel agarose à 1 %. L’ADN doit apparaître comme une bande unique principalement avec sans bavures.
- Déterminer la concentration d’ADN en mesurant l’absorbance sur un spectrophotomètre nanodrop à 260 nm (un260 = 1,0 pour 33 ng/μL d’ADN simple brin). Les concentrations typiques dU-ssDNA relèvent de 200 à 500 ng/μl.
3. méthode de Kunkel basée mutagénèse oligonucléotide-dirigée
Notes : Il est conseillé d’effectuer des réactions à petite échelle avant de réactions de la pleine échelle pour assurer la qualité des composants mutagènes oligonucléotide et réaction. Un dessin animé de méthode de Kunkel basée oligonucléotide-dirigée mutagénèse est illustré à la Figure 3. Différents acides aminés diversités sont introduites dans les régions CDRH1, CDRH2, CDRH3 et CDRL3 avec IMGT numérotation nomenclature16 (tableau 2). La diversité théorique d’acide aminé de chaque CDR, diversité théorique totale en acides aminés et les séquences oligonucléotidiques sont répertoriés dans le tableau 2.
- Phosphorylation des oligonucléotides avec T4 polynucléotide kinase
- Combiner les 0,6 μg d’oligonucléotides mutagènes, conçu pour muter un CDR unique, avec 2 μl 10 X TM tampon, 2 μL ATP 10 mM et 1 μL 100 mM TNT. Ajouter ultrapure H2O pour un volume total de 18 μL dans un tube de 1,5 mL. Pour la construction de la bibliothèque, 4 réactions de phosphorylation séparées et parallèles sont mises en place correspondant à CDRH1, CDRH2, CDRH3 et CDRL3, respectivement.
- Ajouter 20 U (uL 2) T4 polynucléotide kinase à chaque tube et incuber pendant 1 heure à 37 ° C (réaction 1 dans le tableau 3). Utilisation immédiatement de recuit.
- Recuit des oligonucléotides au modèle
- À 20 μg de modèle dU-ADN simple brin, ajouter 25 μL de tampon de TM X 10, 20 μL de chaque solution oligonucléotide phosphorylée et ultrapure H2O pour un volume final de 250 μL dans un tube de 0,5 mL. Bien mélanger et transférer dans des tubes PCR de 0,20 mL (50 μl) en tant que réaction 2 dans le tableau 3. Ces quantités d’ADN fournissent un rapport molaire de 3:1 entre les oligonucléotides et modèle, en supposant que le ratio de la longueur des oligonucléotides et modèle est au 1/100.
- Incubez la réaction dans une machine PCR à 90 ° C pendant 3 min, 50 ° C pendant 3 min et 20 ° C pendant 5 min.
- Synthèse enzymatique du CCC-ADN double brin
- Pour le mélange d’oligonucléotide/modèle 250 μL recuit, ajouter 10 μL d’ATP 10 mM, 10 μL de mélange dNTP 25 mM, 15 μL de 100 mM DTT, Weiss 30 unités T4 DNA ligase et 30 U T7 DNA polymérase comme réaction 3 dans le tableau 3.
- Incubez la réaction dans le tube de 1,5 mL à 20 ° C durant la nuit.
- Laver et concentrer la CCC-ADN double brin synthétisé dans un dispositif de filtration centrifuge de 0,5 mL d’une membrane de taille de pore de 30 kDa à température ambiante.
- Transférer le mélange réactionnel au jour le jour dans le dispositif de filtration et ajoutez ultrapure H2O au volume final de 400 µL. Tournant à 14 000 x g pendant 10 min ; le volume est inférieur à 50 μL.
- Jeter le débit à travers, ajouter 400 µL d’ultrapure H2O dans le filtre et essorer à 14 000 × g pendant 10 min.
- Répétez l’étape 3.3.3.2 une fois de plus.
- Placer le filtre à l’envers dans un tube de microcentrifuge propres pour récupérer le CCC-dsDNA. Tourner à 1 000 × g pendant 2 min ; le volume récupéré est généralement autour de 20-40 μL. La CCC-dsDNA récupéré peut être utilisé immédiatement pour l’électroporation d’e. coli ou congelé à-20 ° C pour une utilisation ultérieure. Normalement de 20 à 40 μg CCC-DNA peut être obtenue.
- ELECTROPHORESE 1,0 µL du produit éluées réaction aux côtés du modèle dU-ssDNA pour visualiser le résultat de la réaction.
4. électroporation et calcul de la taille de la bibliothèque
- Refroidissez la CCC-ADN purifié (20 μg dans un volume maximum de 50 μL) dans un tube de microtubes de 1,5 mL et une cuvette d’électroporation de 0,2 cm écart sur la glace.
- Préchauffer 20 mL de médias SOC dans un tube à centrifuger coniques en polypropylène 50mL au bain-marie à 37 ° C pendant au moins 30 min.
- Décongeler une aliquote de 350 μL d' electro-compétent e. coli SS320 sur la glace. Ajouter des cellules à l’ADN et mélangez soigneusement en pipettant également plusieurs fois. Éviter d’introduire des bulles.
- Transvaser le mélange dans la cuve et effectuer l’électroporation suivant les instructions du fabricant. Par exemple, lorsque le système d’électroporation BTX ECM-630 est utilisé, les paramètres pertinents sont l’intensité de champ 2,5 kV, 125 Ω résistance et capacité µF 50.
- Sauvetage immédiatement l’électroporation des cellules en ajoutant 1 mL de milieu SOC préchauffé et en transférant dans 17 mL de milieu SOC dans un flacon de 125mL. Rincer la cuvette deux fois avec 1 mL de milieu SOC et transfert dans le même ballon (volume final est de 20 mL).
- Incuber 30 min à 37 ° C sous agitation à 200 tr/min.
- Déterminer l’efficacité d’électroporation.
- Ajouter 180 μL de médias 2YT dans chaque puits d’une seule colonne d’une plaque de 96 puits.
- Faire 8 série dix fois les dilutions : transférer 20 μL de la culture de 20 mL dans le premier puits de la plaque, se mêlent pipetage et transférer 20 μl du mélange à l’autre bien. Répétez cette étape jusqu'à la fin de la dilution en série.
- Plaque de 10 μl de chacune des dilutions séries sur une plaque LB/carb en double exemplaire. Plaque de 100 µL restant de chacune des dilutions serial sur plaques LB/carb distinctes pour la colonie comte contre-vérification. Ces plaques fournira également des clones unique pour l’analyse de l’ELISA et de séquence (voir section 5).
- Incuber une nuit à 37 ° C.
- Compter les colonies de 10 doublons μL sur la plaque LB/carb. Supposons que M est la colonie moyenne nombre compté sur 10N plier LB/carb support (N est de 1 à 8). La taille de la bibliothèque totale est égale à 2 M X 10 colonies deN + 3 .
- Aliquote la culture de l’étape 4.6 également dans les deux 2-L déroutés fioles, chacune contenant 500 mL de milieu 2YT/carb/kan pour la génération de bibliothèque phage.
- Incuber à 37 ° C sous agitation à 200 tr/min pendant la nuit pendant environ 16 h.
- Transfert de la culture à deux bouteilles de 1 L centrifugeuse autoclavé et centrifuger pendant 30 min à 12 000 × g à 4 ° C.
- Transvaser le surnageant à deux nouvelles bouteilles 1 L centrifugeuse autoclavé et ajouter 1/5 le volume final de PEG/NaCl solution pour précipiter le phage. Incuber sur glace pendant 30 min.
- Centrifuger pendant 30 min à 12 000 × g et 4 ° C. Soigneusement, éliminer le surnageant et éviter de déranger de l’extrait concentré de phage. Faites tourner pendant 1 min à 4 000 x g et enlever le surnageant restant avec une pipette.
- Resuspendre le culot de phages avec 20 mL de stérile filtré la solution tampon PBS de X 1 et le transfert dans un nouveau tube de 50 mL.
- Granulés de la matière insoluble par centrifugation pendant 5 min à 12 000 × g et 4 ° C. Transférer le surnageant dans un nouveau tube de 50 mL.
- Mesurer la concentration de phages par spectrophotomètre (OD268 = 1,0 pour une solution de 5 X 1012 phage/mL).
- Ajuster la concentration du phage à 5 X 1012 phage/mL dans du PBS de X 1 avec 10 % de glycérol ultrapure.
- Aliquote 1 mL de solution de phages par tube microtubes de 1,5 mL. Utiliser les bibliothèques immédiatement pour panoramique ou stocker à-80 ° C.
5. évaluation de la protéine A / L Direct analyse ELISA obligatoire et le séquençage
- Au hasard choisir 96 colonies individuelles sur plaque LB/carb de l’étape 4.7.3 dans une plaque de culture de profondeur 96 puits contenant 800 μL de 2YT/carb dans chaque puits. Incuber pendant 3-4 h à 37 ° C sous agitation à 1 000 tr/min à OD600 = 0,4 à 0,8.
- Ajouter 100 μL de M13KO7 (1 X 1011 UFC/mL) pour chaque puits d’une profondeur 96 bien la culture plaque avec une pipette multicanaux. Incuber à 37 ° C sous agitation à 1 000 tr/min pendant 1 h.
- Ajouter 100 μL de 2YT contenant 10 X concentration kanamycine (500 μg/mL) dans chaque puits avec une pipette multicanaux. Incuber une nuit à 37 ° C sous agitation à 1 000 tr/min.
- Dissoudre les protéines L à 1 µg/mL en solution 1 PBS X. Manteau 3/4 des puits dans une plaque en polystyrène de haute 384 puits-la liaison aux protéines avec 30 µL/puits de la solution de protéine L. Incuber une nuit à 4 ° C.
- Jeter la solution de revêtement protéine L nuit d’étape 5.4. Ajouter 50 µL de M-PBST (la solution de saturation) à tous les puits de la plaque en polystyrène de haute 384 puits-la liaison aux protéines avec une pipette multicanaux. Incuber les plaques sur un agitateur de microplaque pendant 1 h à température ambiante.
- Tournez en bas la culture au jour le jour de l’étape 5.3 dans une plaque de 96 puits profond culture à 3 000 x g pendant 10 min à 4 ° C. Le phage se trouve dans le surnageant.
- Jeter la solution de saturation de l’étape 5.5. Ajouter 15 ml de M-PBST et 15 μL de chaque phage surnageante (point 5.6) à 3 des puits L enduit protéique en triple exemplaire et 1 contrôle bien que négatif non couché à l’aide d’une pipette multicanaux. Incuber à RT pendant 1 h avec agitation à 200 tr/min.
- Jeter la solution phage. Laver la plaque 6 fois avec 80 μL de PBST par un laveur de plaques automatisé.
- Ajouter 15 μL de PBST-M et 15 μl de protéine A-HRP (1:1, 500 dilué dans une solution 1 PBS X) dans chaque puits avec une pipette multicanaux et incuber à RT pendant 1 h avec agitation à environ 200 tr/min.
- Jeter la solution de protéine A-HRP/M-PBST. Laver la plaque 6 fois avec 80 μL de PBST.
- Ajouter le substrat TMB (30 μL/puits) avec une pipette multicanaux et incuber pendant 2-3 min jusqu'à ce que la couleur se développe. Arrêter la réaction avec 1,0 M H3PO4 (30 μL/puits).
- Lire les plaques par spectrophotométrie à 450 nm. Les clones positifs sont définis comme ceux qui présentent un ratio moyen de l’absorbance450 OD de puits de protéine L pour le M-PBST bien supérieure à 3.0.
- Dans un profond-96, infecter 50 μL de SS320 dans 2YT/tet à phase logarithmique avec 5 μL du phage même utilisé pour les tests ELISA (point 5.6) pendant 30 min à température ambiante.
- Ajouter 950 μL de 2YT/carb en 96-profondeur bien de l’étape 5.13 et incuber une nuit à 37 ° C sous agitation à 1 000 tr/min.
- Extraire l’ADN de phagemid par kit d’extraction d’ADN mini-préparent. Utiliser les amorces en amont de VL (5'-TCGCTTTGTTTTTATTTTTTAATGTA-3') et VH (5'-GACTACTAATAACATAAAGTCTACGCCG-3') pour l’analyse de la séquence estimer la diversité des séquences de la bibliothèque.
Résultats
Suivant l’organigramme de la construction de la bibliothèque Fab (voir Figure 1), nous avons établi M13KO7 helper phage préalablement infectées par e. coli SS320 cellules capables d’electro. L’efficacité de ces cellules capables d’electro est estimée à 2 X 109 UFC/µg lorsqu’il est l’épine dorsale de Fab phagemid pour la construction de la bibliothèque a été utilisé (Figure 4).
L’efficacité de l’incorporation d’uracile en comparaison du titre dans les cellules des e. coli CJ236 et SS320 d’e. coli a été vérifiée. Les cellules d’Escherichia coli CJ236 et SS320 d’e. coli étaient infectés par le phage nourrissait dU-ADN simple brin. E. coli SS320 a enzymes (glycosylase dUTPase et uracile) qui peuvent dégrader l’uracil-contenant de l’ADN, tandis qu’e. coli CJ236 ne dispose pas de ces enzymes et ne peut pas dégrader l’ADN contenant de l’uracile. Pour atteindre une efficacité d’incorporation uracile acceptable, les titres d’e. coli CJ236 doivent être au moins 104 fois plus élevées que celles d’e. coli SS320. Dans le cas contraire, la population sauvage augmentera dans la bibliothèque d’anticorps construit en raison de l’incorporation de l’uracile inefficace. Figure 5 montre que le titre de CJ236 d’e. coli est environ 3 X 10,5 fois supérieurs à celui d’e. coli SS320, indiquant une incorporation de l’uracile efficace dans phage ADN simple brin.
Ensuite, nous avons préparé et extraite dU-ADN simple brin. La pureté dU-ssDNA est vérifiée par électrophorèse sur gel d’agarose (Figure 6). Puis la mutagénèse oligonucléotide-dirigée a été menée et a évalué l’efficacité de la conversion dU-ADN simple brin d’ADN CCC (Figure 6). Trois produits à plus faible motilité que dU-ssDNA peuvent être visualisées sur le gel, y compris la bande plus rapide d’exécution (CCC-ADNdb), la moyenne faible bande (ébréchée) et la bande de durée plus lente (DNA strand déplacées).
Après électroporation dans SS320 d’e. coli , la bibliothèque a été estimée de la plaque de l’incubation durant la nuit (voir étape 4.7.5). La taille de la bibliothèque moyenne était de 5 X 109 de dilutions en série en double sur LB/carb plaques (Figure 7). Toutefois, la taille estimée à cette étape peut contenir des bactériophages qui ne pas afficher les Fabs en raison de la présence d’un changement de phase ou arrêtez le codon ou exposer Fabs mal repliées. Séquençage et ELISA ont été utilisés pour estimer la diversité fonctionnelle de bibliothèque construite. 96 clones unique choisis au hasard ont été envoyés pour analyse de la séquence. Le tableau 4 montre que 90 sur 96 clones unique choisis au hasard ont été séquencées avec succès, qui contient 70 clones sans un codon stop prématuré (53 clones au moins un CDR mutant) et 17 clones avec la séquence de type sauvage et 20 clones avec une codon d’arrêt prématuré dans différentes régions. Dans les 70 clones, mutants taux de CDRH1, CDRH2, CDRH3 et CDRL3 sont 50 %, 57 %, 53 % et 56 %, respectivement, tandis que le taux de mutant avec au moins un CDR est de 76 %. Dans les 20 clones avec un codon stop prématuré (90 %), le codon stop prématuré provenait principalement du déphasage des amorces de mutagenèse, dont 45 % (CDRH1), 10 % (CDRH2), 15 % (CDRH3) et 20 % (CDRL3).
Pour détecter l’affichage des Fabs correctement pliées, une protéine A / L base ELISA travaillait comme il est connu que la protéine A et la protéine L peuvent reconnaître un repliement adéquat du VH cadre et cadre de VL, respectivement de17,18. En accord avec l’analyse de la séquence, le test ELISA en triple exemplaire (Figure 8) a montré que les 20 clones avec un codon stop prématuré étaient tous négatifs, tandis que les 17 clones avec une séquence de type sauvage sont tous positifs lorsque le ratio positif était empiriquement, fixé à 3.0. Pour les 53 clones au moins un CDR mutant, 43 clones ont été positifs en ELISA tandis que 10 clones étaient négatifs ; Cela indique que la plupart des clones ont été bien pliée, tandis que les CDRs de 10 clones peuvent avoir des effets préjudiciables sur pliage Fab. Au total, 43 clones de 90 clones (48 %) ont été bien pliés et contenaient au moins un CDR mutant. Ainsi, la diversité fonctionnelle des acides aminés de la bibliothèque construite basée sur la protéine A / L ELISA et séquence d’analyse a été estimée à 2,4 X 109 (soit48 % de 5 X 10-9).
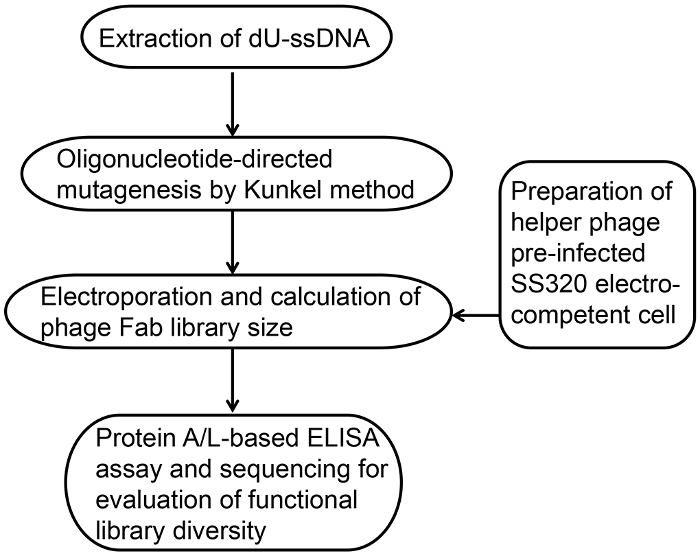
Figure 1 : vue d’ensemble de la construction de la bibliothèque de Fab phage-affiché. Construction de la bibliothèque de Fab phage-affiche suit une série de base des étapes. Il implique la préparation de cellules bactériennes capables d’electro haute efficacité, extraction dU-ADN simple brin, mutagénèse oligonucléotide-dirigée du méthode basée de Kunkel, électroporation et calcul du phage taille bibliothèque Fab, évaluation fonctionnelle par protéine A / analyse de séquence L ELISA et l’ADN. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
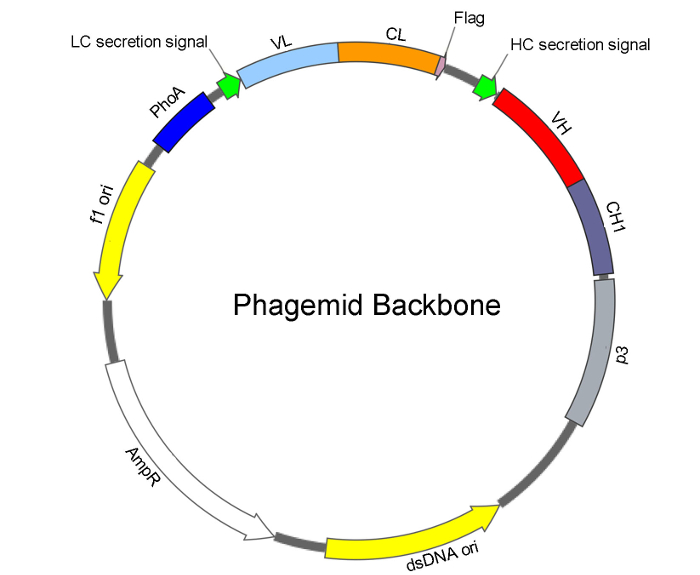
Figure 2 : architecture de Phagemid pour la construction de la bibliothèque Fab. Les fonctionnalités de base de l’épine dorsale de phagemid se composent d’origines du simple brin (f1 ori) et ADN à double brin (dsDNA ori) réplication et un gène de résistance de l’ampicilline/carbénicilline (AmpR). Pour l’affichage Fab, sous le contrôle du promoteur phosphatase alcaline (PhoA), le phagemid contient une cassette bicistronique à disque expression et la sécrétion de : chaîne (LC), consistant en un signal de sécrétion, VL (région variable de la chaîne légère), CL (constante légère région de chaîne légère) et étiquette C-terminal d’indicateur ; et la chaîne lourde (HC), consistant en un signal de sécrétion et VH (région variable de la chaîne lourde) CH1 (1 région constante des chaînes lourdes) fusionné avec une protéine de capside mineures de phage p3. Assemblage de la chaîne légère et la chaîne lourde en Fab dans le périplasme d’e. coli ordonne l’affichage de Fab à la surface du phage. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 3 : représentation schématique du méthode de Kunkel basé mutagénèse dirigée oligonucléotide. Dans ce protocole, nous avons utilisé des méthode de Kunkel pour préparer dU-ssDNA modèle. Oligonucléotides pour CDRH1, CDRH2, CDRH3 et CDRL3 avec la diversité conçue sont phosphorylées, recuits au modèle et utilisés pour convertir les ss-ADN CCC-dsDNA. Suite d’électroporation dans e. coli SS320 cellules capables d’electro, l’ADN hétéroduplex est réparé pour le type sauvage ou la forme mutante ; En raison de la présence de l’uracile dans le brin de type sauvage, le processus de réparation favorise la forme mutante, et ainsi, la forme mutante domine la bibliothèque. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
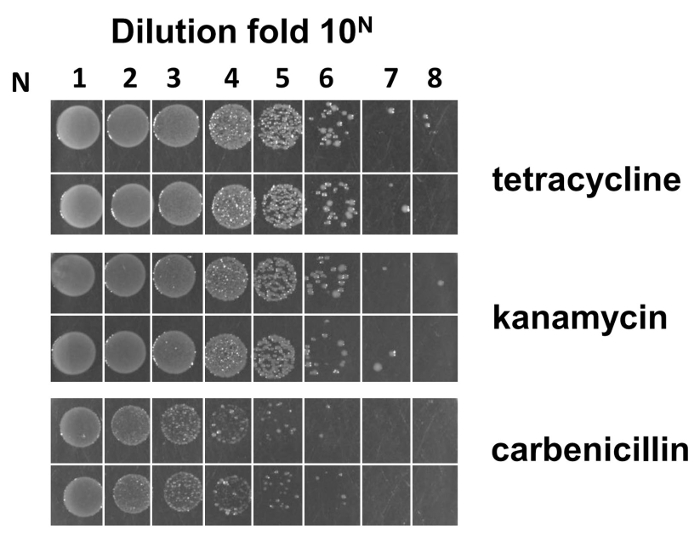
Figure 4 : Estimation de le M13KO7 infectés avant l’efficacité e. coli SS320 cellule electro-compétente. Un vecteur de colonne vertébrale de phagemid a été utilisé pour vérifier l’efficacité de l’électroporation des cellules compétentes. Formule pour calculer l’efficacité est la suivante : Supposons que M est le nombre moyen de colonie compté à partir du pli plus dilué 10N (N étant de 1 à 8) en double exemplaire. E. coli SS320 efficacité de plaque LB/carb est égale à M X 10N + 3 UFC/µg. L’efficacité des cellules capables d’electro est environ 2 X 109 UFC / µg. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 5 : Évaluation de l’incorporation d’uracile dans ADNsb par infection par le phage de cellules d’Escherichia coli CJ236 et e. coli SS320. Basé sur la méthode de Kunkel, l’efficacité de l’incorporation d’uracile est vérifiée par comparaison de titre infection bactériophage dans les cellules des e. coli CJ236 et e. coli SS320. La formule de calcul du titre est la suivante : Supposons que M est le nombre moyen de colonie compté à partir du pli plus dilué 10N (N est de 1 à 10), et que le titre de CJ236 d’e. coli ou e. coli SS320 est égal à M X 10N + 2 UFC/mL. Le rapport de titre d’e. coli CJ236 et SS320 d’e. coli , on peut estimer l’efficacité de l’incorporation de l’uracile. Le titre en CJ236 d’e. coli a été 9 X 1012 UFC/mL alors que le titre en SS320 d’e. coli a été 3 X 107 UFC/mL. Le ratio de titre d’e. coli CJ236 et SS320 d’e. coli a été 3 X 10,5. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 6 : Conversion dU-ADN simple brin en ADN CCC par mutagénèse oligonucléotide-dirigée. Après mutagénèse oligonucléotide-dirigée, a évalué l’efficacité de conversion dU-ADN simple brin d’ADN CCC. dU-ssDNA est complètement transformé en ADN double brin. Le groupe dominant est CCC-dsDNA alors qu’il y a une petite partie de dsDNA ébréchée et déplacées brin ADN. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
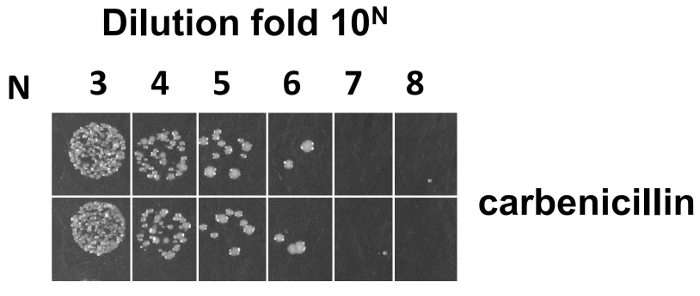
Figure 7 : titrage de phages pour le calcul de la taille de la bibliothèque. Après électroporation dans SS320 d’e. coli , la bibliothèque a été estimée de dilutions en série sur des plaques LB/carb. La formule de calcul de taille est la suivante : Supposons M est le nombre moyen de colonie compté à partir du pli plus dilué 10N d’une plaque de 2YT/Carb (N est de 1 à 8), la taille est égale à 2 M X 10N + 3. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 8 : la protéine A / L direct phage liaison ELISA. Protéine L peut reconnaître que le cadre de la chaîne légère kappa bien plié VL et la protéine un peut reconnaissent le cadre de la chaîne lourde bien plié VH. Liaison de Fab avec protéine L et A indique un repliement adéquat de chaîne lourde et de chaîne légère. En bref, protéine L en triple exemplaire et le contrôle négatif PBST-M ont été revêtus de la plaque, les surnageants phage Fab provenant de différents clones ont été incubées avec protéine L et M-PBST, puis après le lavage, protéine Qu'a-HRP a été utilisé pour capturer les phages Fab lié. Lectures de phage ELISA a montré 90 clones choisis au hasard avec une lecture réussie de séquençage. Une ligne de seuil représentant le clone comme positif a été empiriquement définies où le ratio de la valeur d’absorbance de450 OD de protéine L (moyenne de trois exemplaires avec barre d’erreur) versus contrôle négatif était de plus de 3,0. Trois groupes basés sur l’analyse de la séquence apparaissaient, correspondant à un mutant sans codon stop en rouge (53 clones), type sauvage (WT) en bleu (17 clones) et mutant avec le codon d’arrêt en vert (20 clones). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Installation de réactif | Composant | Montant | Commentaires/description |
| 2YT moyen | Extrait de levure | 10 g | Ajouter de l’eau ultrapure à porter le volume à 1,0 L, ajuster le pH à 7.0, autoclave. |
| Tryptone | 16 g | ||
| NaCl | 5 g | ||
| Albums agar 2YT | Extrait de levure | 10 g | Ajouter de l’eau ultrapure pour porter le volume à 1,0 L et ajuster le pH à 7.0, chaleur de dissolution, autoclave. |
| Tryptone | 16 g | ||
| NaCl | 5 g | ||
| Gélose granulé | 7,5 g | ||
| 2YT/carb/CPM moyen | Carbénicilline | 100 μg/mL | |
| Chloramphénicol | 10 μg/mL | ||
| 2YT/carb/kan/uridine moyen | Carbénicilline | 100 μg/mL | |
| Kanamycine | 50 μg/mL | ||
| Uridine | 0,25 μg/mL | ||
| 2YT/carb/tet moyen | Carbénicilline | 100 μg/mL | |
| Tétracycline | 10 μg/mL | ||
| Moyenne de 2YT/carb | Carbenicilin | 100 μg/mL | |
| Moyenne de 2YT/kan | Kanamycine | 50 μg/mL | |
| 2YT/kan/tet moyen | Kanamycine | 50 μg/mL | |
| Tétracycline | 10 μg/mL | ||
| Moyenne de 2YT/tet | Tétracycline | 10 μg/mL | |
| 2YT/CPM moyen | Chloramphénicol | 10 μg/mL | |
| Milieu gélosé LB | Extrait de levure | 5 g | Ajouter de l’eau ultrapure à porter le volume à 1,0 L, ajuster le pH à 7.0, autoclave. Pour LB agar, ajouter 20 g de granulé agar, autoclave. |
| Tryptone | 10 g | ||
| NaCl | 10 g | ||
| Plaques LB/carb | LB agar | 1L | |
| Carbénicilline | 100 μg/mL | ||
| Plaques LB/tet | LB agar | 1 L | |
| Tétracycline | 10 μg/mL | ||
| Plaques LB/cmp | Chloramphénicol | 10 μg/mL | |
| Plaques LB/kan | Kanamycine | 50 μg/mL | |
| Moyen SOC | Extrait de levure | 5 g | Ajouter de l’eau ultrapure pour porter le volume à 1,0 L et ajuster le pH à 7.0, autoclave. |
| Tryptone | 20 g | ||
| NaCl | 0,5 g | ||
| KCl | 0,2 g | ||
| 2.0 M MgCl2 | 5,0 mL | ||
| 1,0 M de glucose | 20 mL | ||
| Moyen superbroth | Tryptone | 12 g | Ajouter l’eau ultrapure à 900 mL, autoclave, ajouter 100 mL d’autoclave 0,17 M KH2PO4, 0,72 M K2HPO4. |
| Extrait de levure | 24 g | ||
| Glycérol | 5 mL | ||
| Moyen de kan/tet superbroth | Kanamycine | 50 μg/mL | |
| Tétracycline | 10 μg/mL | ||
| 1 X PBS | NaCl | 137 mM | Ajuster le pH à 7,2, autoclave. |
| KCl | 3 mM | ||
| Na2HPO4 | 8 mM | ||
| KH2PO4 | 1,5 mM | ||
| Gel TAE / d’agarose | Tampon TAE | ||
| Gel d’agarose | 1 % (p/v) | ||
| GelRed | 1 : 10000 (v/v) | ||
| Substrat TMB | TMB | 50 % (v/v) | |
| Substrat peroxydase de H2O2 | 50 % (v/v) | ||
| Mémoire tampon PBST-M | 1 X PBS | 100 ml | |
| Tween-20 | 0,05 % (v/v) | ||
| Lait en poudre écrémé | 5 % (v/v) | ||
| 5 X PEG/NaCl | PEG-8000 | 20 % (p/v) | Ajouter de l’eau ultrapure à porter le volume à 1L et stériliser. |
| NaCl | 5 2. M | ||
| Mémoire tampon PBST | 1 X PBS | 1 L | Filtre de 0,22 μm-stériliser. |
| Tween-20 | 0,05 % (v/v) | ||
| Mémoire tampon TM 10 x | MgCl2 | 0,1 M | Ajuster le pH à 7,5. |
| Tris | 0. 5 M | ||
| 1,0 mM HEPES, pH 7,4 | 1.0 HEPES M | 4,0 mL | Filtre de 0,22 μm-stériliser. |
| Eau ultrapure | 4.0 L | ||
| 10 % (v/v) de glycérol ultrapure | Glycérol ultrapure | 100 ml | Filtre de 0,22 μm-stériliser. |
| Eau ultrapure | 900 mL | ||
| Eau ultrapure | H20 | Exempt de DNase, Rnase-libre, exempt de substances pyrogènes. |
Tableau 1 : Configuration du réactif.

Tableau 2 : CDR des diversités et des amorces de mutagénèse. Les séquences d’ADN des régions CDR être muté figurent en rouge ; séquences sont formatés en utilisant le code de nucléotides IUPAC. « X » indique tri-nucléotide à partir d’un mélange conçu pour contenir des acides aminés différents ensembles ; « n » indique un nombre différent de X. cinq amorces avec un nombre différent de X ont été utilisé pour diversifier les CDRL3 ou CDRH3, respectivement, pour générer une longueur variable de CDRL3 et CDRH3. Le nombre de résidus est défini par la nomenclature IMGT. S’il vous plaît cliquez ici pour télécharger ce tableau.
| Mutagenèse méthode basée de Kunkel | ||
| Réaction 1. Phosphorylation des oligonucléotides avec T4 polynucléotide kinase | ||
| Composant | Montant | Finale |
| oligonucléotides mutagènes | 0,6 μg | |
| Mémoire tampon TM 10 x | 2 ΜL | 1 X |
| ATP 10 mM | 2 ΜL | 1 mM |
| 100 mM TNT | 1 ΜL | 5 mM |
| T4 polynucléotide kinase (10 U/μl) | 2 ΜL | 20 U |
| Ultrapure H20 | Jusqu'à 20 μL | |
| Paramètre de réaction | ||
| Étape 1. | 37 ° C pendant 1 h | |
| Réaction 2. Recuit des oligonucléotides au modèle | ||
| Composant | Montant | Finale |
| modèle dU-ADN simple brin | 20 μg | 20 μg |
| Mémoire tampon TM 10 x | 25 ΜL | 1 X |
| oligonucléotides CDRH1 phosphorylés | 20 ΜL | 0,6 μg |
| oligonucléotides CDRH2 phosphorylés | 20 ΜL | 0,6 μg |
| oligonucléotides CDRH3 phosphorylés | 20 ΜL | 0,6 μg |
| oligonucléotides CDRL3 phosphorylés | 20 ΜL | 0,6 μg |
| Ultrapure H20 | Jusqu'à 250 μL | |
| Paramètre de réaction | ||
| Étape 1. | 90 ° C pendant 3 min | |
| Étape 2. | 50 ° C pendant 5 min | |
| Étape 3. | 20 ° C pendant 5 min | |
| 3 la réaction. Synthèse enzymatique du CCC-ADN double brin | ||
| Composant | Montant | Finale |
| mélanges de recuit oligonucléotides/modèle | 250 ΜL | |
| ATP 10 mM | 10 ΜL | 346 µM de chaque nucléotide |
| Mélange dNTP (25 mM de chaque nucléotide) | 10 ΜL | 865 µM de chaque nucléotide |
| 100 mM TNT | 15 ΜL | 5 mM |
| T4 DNA ligase | 1 ΜL | 30 unités de Weiss |
| T7 ADN polymérase | 3 ΜL | 30 U |
| Paramètre de réaction | ||
| Étape 1. | 20 ° C pour la nuit | |
Tableau 3 : Procédures et éléments de méthode de Kunkel base réaction.
| Groupe | Nombre de clone | Région | Pourcentage | ||
| Aucun codon stop prématuré | 70 | CDRH1 mutation | 50 % (35/70) | ||
| CDRH2 mutation | 57 % (40/70) | ||||
| CDRH3 mutation | 53 % (37/70) | ||||
| CDRL3 mutation | 56 % (39/70) | ||||
| Au moins une mutation de la CDR | 76 % (53/70) | ||||
| Codon stop prématuré | 20 | CDRH1 défaut | 45 % (9/20) | ||
| CDRH2 défaut | 10 % (2/20) | ||||
| CDRH3 défaut | 15 % (3/20) | ||||
| CDRL3 défaut | 20 % (4/20) | ||||
| Autre défaut | 10 % (2/20) | ||||
Tableau 4 : Analyse de la séquence de CDRH1, CDRH2, CDRH3 et CDRL3 de la bibliothèque de Fab synthétique.
Discussion
Pour construire la grande diversité, phage-affiche les bibliothèques Fab, contrôle qualité check-points sont nécessaires pour surveiller les différentes étapes de la construction, y compris la compétence des cellules capables d’electro, de qualité du modèle dU-ADN simple brin, efficacité de CCC-dsDNA synthèse, titre après électroporation, pliage Fab et la diversité des acides aminés des CDRs par séquençage de clones de Fab-phage.
Rendement élevé et la pureté dU-ssDNA est essentiel pour taux élevé de mutagénèse. Dans notre expérience, induction phage à 25 ° C pendant la nuit peut donner dU-ssDNA plus que celle d’induction de phage à 37 ° C pendant la nuit. C’est en accord avec un précédent rapport19. Au sujet de l’extraction de l’ADN simple brin, le plasmide initial Spin kit (QIAprep) contenait MLB pour la liaison et la lyse de phages. Plus tard, MLB a été abandonnée avec raison inconnue et remplacé par PB. Nous avons constaté que le rendement dU-ssDNA est beaucoup plus faible de traitement PB par rapport à celui du traitement de la MLB. Dans ce protocole, nous avons utilisé un réactif appelé UT-MLB20 pour remplacer la MLB et constaté que le rendement dU-ssDNA est semblable à celle de la trousse initiale de Spin.
Comme CDRH3 et CDRL3 sont les régions les plus diversifiées sur la reconnaissance de l’antigène21, d’introduire une diversité sur mesure avec un ensemble de combinaisons d’acides aminés et des rapports spécifiques et pour éliminer les biais de redondance et arrêt-codons introduits par les codons dégénérés tels que NNK (N, équimolaire d’a/c/G/T ; K, équimolaire de G/T), trimère codon phosphoramidite axée sur les oligonucléotides22 avec exactement un codon trimère correspondant à l’un acides aminés spécifiques ont été conçus pour CDRH3 et CDRL3. En outre, des longueurs variables des oligonucléotides CDRH3 et CDRL3 ont été utilisés pour accroître encore les diversités.
Après la synthèse enzymatique du CCC-ADN double brin, généralement trois bandes sont observées par électrophorèse sur gel d’agarose et les bandes devraient être clairs sans frottis. Parmi eux, le groupe le plus rapide d’exécution est le CCC-ADN double brin qui peut produire un taux de mutation élevé (environ 80 %) après électroporation23. La bande en cours d’exécution plus lente est l’ADN brin-déplacés qui découle de l’activité inhérente strand-déplacement de T7 ADN polymérase et a une mutation faible taux (20 %)23. La bande faible moyenne est entaillée ADN après prolongation en raison d’une activité insuffisante T4 DNA ligase ou phosphorylation oligonucléotide insuffisante.
Un pool d’échantillon petit séquençage a été utilisé pour estimer la diversité de la bibliothèque mais pas précis24. Pour estimer avec précision la diversité réelle, prochaine génération de séquençage (NGS) peut être une bonne option dans l’exploitation minière de la profondeur de la diversité de la bibliothèque construite à25. Dans la pratique, en raison des défis actuels de la NGS technology y compris lire longueur, précision, coût et haut débit, le séquençage de la bibliothèque de Fab phages utilisé dans le présent protocole avec la longueur d’environ 950 bp s’étendant sur CDRH1, CDRH2, CDRH3 et CDRL3 n’est pas réalisables ; Toutefois, il est possible d’estimer la scFv (environ 700 bp) diversité de bibliothèque au sein de la gamme de millions24,25. Une autre norme clée pour évaluer la diversité de la bibliothèque construite est d’utiliser la bibliothèque de pan contre différents types d’antigènes et de calculer les clones positifs capturées en raison de la diversité de bibliothèque est directement corrélée avec le taux de succès de l’antigène panoramique de26. Plateforme de sélection de haut débit est bien adapté à cet effet et lecteurs peuvent se référer à un protocole détaillé rapporté par Mersch et al. 27
Théoriquement, bibliothèques d’anticorps synthétiques phage-affiché avec diversité sur mesure peuvent être utilisés pour cibler tout antigène et ainsi avoir des applications larges. Actuellement, sociétés dont Cambridge Antibody Technology (CAT), MedImmune, Genentech, Dyax, Bioinvent, Pfizer et MorphoSys dépendent beaucoup de plates-formes affichage phage d’anticorps thérapeutiques développement28. En outre, les nombreux phage display core technologie brevets ont expiré29. Sans aucun doute, cela va déclencher le potentiel maximum de la technologie des anticorps phage-affiché.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs apprécient Dr Frederic Fellouse du labo pour des commentaires critiques sur la construction de bibliothèque de phage Fab synthétique de Kunkel méthode basée Sidhu. Les auteurs apprécient Mme Alevtina Pavlenco et autres membres du laboratoire pour l’aide précieuse de préparation haute efficacité electro-compétente Sidhu cellules d’Escherichia coli et haute qualité dU-ADN simple brin. Ce travail a été soutenu par la Fondation nationale des sciences naturelles de Chine (Grant No. : 81572698, 31771006) à DW et par l’Université de ShanghaiTech (Grant No. : F-0301-13-005) au laboratoire de génie d’anticorps.
matériels
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1.0 M H3PO4 | Fisher | AC29570 | |
| 1.0 M Tris, pH 8.0 | Invitrogen | 15568-025 | |
| 10 mM ATP | Invitrogen | 18330-019 | |
| 100 mM dithiothreitol | Fisher | BP172 | |
| 100 mM dNTP mix | GE Healthcare | 28-4065-60 | solution containing 25 mM each of dATP, dCTP, dGTP and dTTP. |
| 3,3’,5,5’-tetramethylbenzidine (TMB) | Kirkegaard & Perry Laboratories Inc | 50-76-02 | |
| 50X TAE | Invitrogen | 24710030 | |
| Agarose | Fisher | BP160 | |
| Carbenicillin, carb | Sigma | C1389 | 100 mg/mL in water, 0.22 μm filter-sterilize, work concentration: 100 μg/mL. |
| Chloramphenicol, cmp | Sigma | C0378 | 100 mg/mL in ethanol, 0.22 μm filter-sterilize, work concentration: 10 μg/mL. |
| EDTA 0.5 M, pH 8.0 | Invitrogen | AM9620G | |
| Granulated agar | VWR | J637-500G | |
| H2O2 peroxidase substrate | Kirkegaard & Perry Laboratories Inc | 50-65-02 | |
| K2HPO4 | Sigma | 795488 | |
| Kanamycin, kan | Fisher | AC61129 | 50 mg/mL in water, 0.22 μm filter-sterilize, work concentration: 50 μg/mL. |
| KH2PO4 | Sigma | P2222 | |
| Na2HPO4 | Sigma | 94046 | |
| NaCl | Alfa Aesar | U19C015 | |
| Nanodrop | Fisher | ND2000C | |
| NaOH | Fisher | SS256 | ! CAUTION NaOH causes burns. |
| NON-Fat Powdered Milk | Sangon Biotech | A600669 | |
| PEG-8000 | Fisher | BP233 | |
| Protein A-HRP conjugate | Invitrogen | 101123 | |
| QIAprep Spin M13 Kit | Qiagen | 22704 | |
| QIAquick Gel Extraction Kit | Qiagen | 28706 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| Recombinant Protein L | Fisher | 77679 | |
| T4 DNA polymerase | New England Biolabs | M0203S | |
| T4 polynucleotide kinase | New England Biolabs | M0201S | |
| T7 DNA polymerase | New England Biolabs | M0274S | |
| Tetracycline, tet | Sigma | T7660 | 50 mg/mL in water, 0. 22 μm filter-sterilize, work concentration: 10 μg/mL. |
| Tryptone | Fisher | 0123-07-5 | |
| Tween-20 | Sigma | P2287 | |
| Ultrapure glycerol | Invitrogen | 15514-011 | |
| Uridine | Sigma | U3750 | 25 mg/mL in ethanol, work concentration: 0.25 μg/mL. |
| Yeast extract | VWR | DF0127-08 | |
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E.coli CJ236 | New England Biolabs | E4141 | Genotype: dut- ung- thi-1 relA1 spoT1 mcrA/pCJ105(F' camr). Used for preparation of dU-ssDNA. |
| E.coli SS320 | Lucigen | 60512 | Genotype: [F'proAB+lacIq lacZΔM15 Tn10 (tetr)] hsdR mcrB araD139 Δ(araABC-leu)7679 ΔlacX74 galUgalK rpsL thi. Optimized for high-efficiency electroporation and filamentous bacteriophage production. |
| M13KO7 | New England Biolabs | N0315S | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| 0.2-cm gap electroporation cuvette | BTX | ||
| 96-well 2mL Deep-well plates | Fisher | 278743 | |
| 96-well Maxisorp immunoplates | Nunc | 151759 | |
| Baffled flasks | Corning | ||
| Benchtop centrifuge | Eppendorf | 5811000096 | |
| Centrifuge bottles | Nalgene | ||
| ECM-630 electroporator | BTX | ||
| Magnetic stir bars | Nalgene | ||
| Thermo Fisher centrifuge | Fisher | ||
| High speed shaker | TAITEK | MBR-034P | |
| Microplate shaker | QILINBEIER | QB-9002 | |
| Liquid handler for 96 and 384 wells | RAININ | ||
| Mutil-channel pipette | RAININ | E4XLS | |
| Amicon concentrator | Merck | UFC803096 |
Références
- Singh, S., et al. Monoclonal Antibodies: A Review. Curr Clin Pharmacol. , (2017).
- Reichert, J. M. Antibodies to watch in 2017. MAbs. 9 (2), 167-181 (2017).
- Kohler, G., Milstein, C. Continuous cultures of fused cells secreting antibody of predefined specificity. Nature. 256 (5517), 495-497 (1975).
- Milstein, C. The hybridoma revolution: an offshoot of basic research. Bioessays. 21 (11), 966-973 (1999).
- Gray, A. C., Sidhu, S. S., Chandrasekera, P. C., Hendriksen, C. F., Borrebaeck, C. A. Animal-Friendly Affinity Reagents: Replacing the Needless in the Haystack. Trends Biotechnol. 34 (12), 960-969 (2016).
- Hutchison, C. A., et al. Mutagenesis at a specific position in a DNA sequence. J Biol Chem. 253 (18), 6551-6560 (1978).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Sidhu, S. S. . Phage Display in Biotechnology and Drug Discovery. , (2005).
- Weiss, G. A., Watanabe, C. K., Zhong, A., Goddard, A., Sidhu, S. S. Rapid mapping of protein functional epitopes by combinatorial alanine scanning. Proc Natl Acad Sci U S A. 97 (16), 8950-8954 (2000).
- Frenzel, A., Schirrmann, T., Hust, M. Phage display-derived human antibodies in clinical development and therapy. MAbs. 8 (7), 1177-1194 (2016).
- Sidhu, S. S., Fellouse, F. A. Synthetic therapeutic antibodies. Nat Chem Biol. 2 (12), 682-688 (2006).
- Adams, J. J., Sidhu, S. S. Synthetic antibody technologies. Curr Opin Struct Biol. 24, 1-9 (2014).
- Kunkel, T. A. Rapid and efficient site-specific mutagenesis without phenotypic selection. Proc Natl Acad Sci U S A. 82 (2), 488-492 (1985).
- Sidhu, S. S., Lowman, H. B., Cunningham, B. C., Wells, J. A. Phage display for selection of novel binding peptides. Methods Enzymol. 328, 333-363 (2000).
- Persson, H., et al. CDR-H3 diversity is not required for antigen recognition by synthetic antibodies. J Mol Biol. 425 (4), 803-811 (2013).
- Lefranc, M. P., et al. IMGT unique numbering for immunoglobulin and T cell receptor variable domains and Ig superfamily V-like domains. Dev Comp Immunol. 27 (1), 55-77 (2003).
- Graille, M., et al. Complex between Peptostreptococcus magnus protein L and a human antibody reveals structural convergence in the interaction modes of Fab binding proteins. Structure. 9 (8), 679-687 (2001).
- Graille, M., et al. Crystal structure of a Staphylococcus aureus protein A domain complexed with the Fab fragment of a human IgM antibody: structural basis for recognition of B-cell receptors and superantigen activity. Proc Natl Acad Sci U S A. 97 (10), 5399-5404 (2000).
- Huang, R., Fang, P., Kay, B. K. Improvements to the Kunkel mutagenesis protocol for constructing primary and secondary phage-display libraries. Methods. 58 (1), 10-17 (2012).
- Chen, G., Sidhu, S. S. Design and generation of synthetic antibody libraries for phage display. Methods Mol Biol. 1131, 113-131 (2014).
- Padlan, E. A. Anatomy of the antibody molecule. Mol Immunol. 31 (3), 169-217 (1994).
- Virnekas, B., et al. Trinucleotide phosphoramidites: ideal reagents for the synthesis of mixed oligonucleotides for random mutagenesis. Nucleic Acids Res. 22 (25), 5600-5607 (1994).
- Fellouse, F. A., Sidhu, S. S. . Making and Using Antibodies: A Practical Handbook. , 157-180 (2006).
- Fantini, M., et al. Assessment of antibody library diversity through next generation sequencing and technical error compensation. PLoS One. 12 (5), e0177574 (2017).
- Glanville, J., et al. Deep sequencing in library selection projects: what insight does it bring?. Curr Opin Struct Biol. 33, 146-160 (2015).
- Perelson, A. S., Oster, G. F. Theoretical studies of clonal selection: minimal antibody repertoire size and reliability of self-non-self discrimination. J Theor Biol. 81 (4), 645-670 (1979).
- Miersch, S., et al. Scalable high throughput selection from phage-displayed synthetic antibody libraries. J Vis Exp. (95), e51492 (2015).
- Ponsel, D., Neugebauer, J., Ladetzki-Baehs, K., Tissot, K. High affinity, developability and functional size: the holy grail of combinatorial antibody library generation. Molecules. 16 (5), 3675-3700 (2011).
- Petering, J., McManamny, P., Honeyman, J. Antibody therapeutics - the evolving patent landscape. N Biotechnol. 28 (5), 538-544 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon