Method Article
Costruzione del fago sintetico visualizzato favolosa biblioteca con diversità su misura
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo descrive una procedura dettagliata per la costruzione di una libreria dei fagi-visualizzato anticorpo sintetico con diversità su misura. Gli anticorpi sintetici hanno vaste applicazioni dalla ricerca di base alla malattia diagnostica e terapeutica.
Abstract
Domanda per gli anticorpi monoclonali (mAbs) nella ricerca di base e la medicina è in aumento ogni anno. Tecnologia dell'ibridoma è stato il metodo dominante per lo sviluppo di mAb dal suo primo rapporto nel 1975. Come una tecnologia alternativa, metodi di visualizzazione dei fagi per sviluppo di mAb sono sempre più attraente poiché Humira, il primo anticorpo dei fagi-derivato e uno dei Best-Seller mAbs, è stato approvato per il trattamento clinico dell'artrite reumatoide nel 2002. Come un non-animale basato su tecnologia di sviluppo di mAb, esposizione dei fagi bypassa l'immunogenicità dell'antigene, umanizzazione e manutenzione degli animali che sono richiesti da ibridomi tradizionale tecnologia basato lo sviluppo di anticorpi. In questo protocollo, descriviamo un metodo per la costruzione di librerie Fab dei fagi-visualizzato sintetiche con le diversità del9-10 1010 ottenibili con un singolo elettroporazione. Questo protocollo si compone di: 1) preparazione delle cellule electro-competente ad alta efficienza; 2) estrazione di uracile-contenenti DNA single-stranded (dU-ssDNA); 3) metodo di Kunkel base mutagenesi oligonucleotide-diretta; 4) elettroporazione e calcolo della dimensione di biblioteca; 5) analisi della proteina A/L-based enzima-collegata dell'immunosorbente (ELISA) per la piegatura e la valutazione della diversità funzionale; e 6) analisi di sequenza del DNA della diversità.
Introduzione
anticorpi monoclonali hanno vaste applicazioni che vanno dalla ricerca di base alla malattia diagnostica e terapeutica. A partire dal 2016, mAbs più di 60 sono stati approvati dagli Stati Uniti Food and Drug Administration (USFDA) per il trattamento clinico delle malattie autoimmuni, il cancro e malattie infettive1,2.
Nel 1975, Kohler e Milstein segnalato una tecnica per la generazione continua di anticorpi di una singola specificità clonale da una fonte cellulare indicata come 'ibridomi' e questa tecnica è successivamente diventato una pietra miliare nella medicina e nell'industria3 ,4. Generazione di anticorpi monoclonali da questo metodo richiede vari passaggi tra cui la produzione dell'antigene, l'immunizzazione del mouse, estrazione dei linfociti B, fusione di cellule B con cellule di mieloma per formare cellule di ibridoma immortale, selezione clonale e per le applicazioni terapeutiche, umanizzazione è necessaria per evitare di anticorpo umano anti-topo (HAMA)4,5. Tuttavia, per questa tecnologia, gli antigeni incluse proteine altamente conservati tossine e agenti patogeni sono relativamente inefficaci nell'innescare la risposta immunitaria in vivo per mAb produzione5.
Nel 1978, Hutchison et al ha segnalato l'uso di un oligonucleotide di mutagenesi diretta di un residuo in un singolo filamento batteriofago virus6. Nel 1985, Smith ha riferito che frammenti di gene estraneo possono essere fuso nel telaio con del gene che codifica la proteina di cappotto dei fagi III e pertanto possono essere visualizzati sulla superficie dei fagi senza compromettere la sua infettività7. Questi lavori pionieristici gettato le basi per la successiva costruzione di librerie anticorpali dei fagi-visualizzati nel sistema immunitario, ingenuo, e sintetico forme con i formati del frammento variabile singola catena (scFv) e antigene-legante frammento (Fab) per terapeutico mAb sviluppo8,9. Dal punto di vista tecnico, lo sviluppo dei fagi dell'anticorpo basato su display offre un approccio complementare allo sviluppo basato su ibridoma mAb che possa aiutare ad per aggirare le limitazioni che possono rappresentare alcuni antigeni e processo di umanizzazione che anticorpi derivati ibridoma spesso richiedono5. A partire dal 2016, 6 phage display-derivato mAbs sono state approvate nel mercato compreso Humira, uno dei più riusciti mAbs utilizzato per il trattamento dell'artrite reumatoide, e molti dei fagi display-derivato dell'anticorpo candidati sono attualmente in varie fasi della clinica indagine10.
Per librerie anticorpali fagiche immunitario e ingenuo, la diversità di dideterminazione regioni (CDR) nella catena leggera e pesante è derivato dal repertorio immune naturale (vale a dire, dalle cellule di B). Al contrario, la diversità di CDRs in librerie anticorpali sintetico dei fagi è interamente artificiale. Approcci sintetici per la costruzione della libreria forniscono il controllo preciso sopra il disegno della diversità di sequenza e offrono opportunità per studi meccanicistici di struttura dell'anticorpo e funzionano11,12. Inoltre, il framework per le librerie sintetiche può essere ottimizzato prima costruzione della libreria per facilitare a valle, lo sviluppo industriale su larga scala11,12.
Nel 1985, Kunkel segnalati un approccio di mutagenesi basate su modelli single-stranded DNA (ssDNA) per introdurre le mutazioni sito-diretta in batteriofago M13 efficientemente13. Questo approccio è stato successivamente utilizzato ampiamente per costruzione delle librerie dei fagi-visualizzato. Oligonucleotidi di DNA sintetizzati chimicamente progettati per introdurre diversità nella Fab CDRs sono incorporati in un phagemid con un modello di spina dorsale dell'anticorpo. In questo processo, il phagemid è espressa come un uracile-contenente ssDNA (dU-ssDNA) e gli oligonucleotidi sono ricotto sul CDRs ed estesa per sintetizzare DNA a doppia elica (dsDNA) in presenza di DNA di T7 polimerasi e T4 DNA ligasi. Infine, generato ds-DNA può essere introdotto in Escherichia coli mediante elettroporazione.
Per elevata diversità, costruzione della libreria dei fagi-visualizzati, elettroporazione ad alta tensione di una miscela di due componenti delle cellule electro-competenti e covalenza dsDNA circolare chiusa (CCC-dsDNA) deve essere preparato con cura. Sidhu et per volta la preparazione di cellule competenti di electro e DNA da metodi tradizionali e notevolmente migliorato biblioteca diversità14.
In questo protocollo, descriviamo un metodo per la costruzione di librerie Fab dei fagi-visualizzato sintetiche con le diversità del9-10 1010 ottenibili con un singolo elettroporazione. La figura 1 Mostra una panoramica della libreria costruzione tra cui: 1) preparazione delle cellule electro-competente ad alta efficienza; 2) estrazione di dU-ssDNA; 3) metodo di Kunkel base mutagenesi oligonucleotide-diretta; 4) elettroporazione e calcolo della dimensione di biblioteca; 5) proteina A/L-based ELISA per la piegatura e la valutazione della diversità funzionale; e 6) analisi di sequenza del DNA della diversità. Tutti i reagenti, ceppi e attrezzature sono elencati nella tabella del materiale. La tabella 1 Mostra la configurazione di reagente.
Protocollo
Nota: Puntali sterili con filtro devono essere utilizzati in tutto quando si tratta dei fagi per evitare la contaminazione per pistola pipetta e la zona circostante. Zona asettica o cappa deve essere utilizzato quando esperimenti di manipolazione con batteri e fagi. Zona di esperimento dei fagi dovrà essere ripulita utilizzando 2% sodio dodecil solfato (SDS) seguito da etanolo al 70% per evitare la contaminazione dei fagi. Per effettuare diluizioni seriali nel presente protocollo, le nuove punte devono essere utilizzati per ogni diluizione.
1. preparazione di Electro-competenti delle cellule di e. Coli 320
- Pre-riscaldare piastra di agar LB/tet (preparati e conservati a 4 ° C per meno di 1 - settimana-vecchio) presso Incubatore 37 ° C per 1 h. utilizzare un ciclo di inoculazione sterile o la punta sterile per striscia fuori uno stock di glicerolo di cellule di e. coli 320 sulla piastra di agar LB/tet pre-riscaldata in una direzione a zig-zag zione delicatamente lungo la superficie della piastra. Incubare la piastra a incubatore 37 ° C durante la notte per circa 12-15 h.
- Il giorno seguente, utilizzare un ciclo di inoculazione sterile o la punta sterile per prendere una singola Colonia ben separata lungo la linea a zig-zag e inoculare in 10 mL di terreno di 2YT/tet in un tubo da 50mL fondo tondo immergendo il ciclo in mezzo e mescolare brevemente.
- Incubare a 37 ° C con agitazione a 200 giri/min per circa 3-4 h fino a OD600 viene raggiunta a circa 0.4-0.8 (crescita del log di fase). Monitor OD600 a 2 h. Durante questo periodo, pre-riscaldare le piastre di agar 5 LB/tet (preparati e conservati a 4 ° C per meno di 1 - settimana-vecchio) in un incubatore a 37 ° C per almeno 1 h.
- Aggiungere dei fagi helper di 20 μL M13KO7 da Stock in laboratorio con titolo circa 1 x 1013 colony forming unit (cfu) /mL in 180 μL di sterile 1 X PBS in provette da 1,5-mL sterilizzato nell'autoclave per preparare 10 diluizioni seriali dieci volte.
- Aliquota 4 mL sterilizzato nell'autoclave e liquido 2YT agar superiore in 5 X 14 mL fondo tubi tondi, etichettare ogni provetta con una diluizione dalla quinta alla nona della diluizione dieci volte, rispettivamente e incubare in un'incubatrice di 65 ° C per mantenere l'agar superiore 2YT allo stato liquido.
- Mix 500 μL di fase di registro 320 Escherichia coli cellule nella provetta di diluizione M13KO7 (scegliere il quinto diluizione dieci volte alla nona diluizione dieci volte) ed incubare in un incubatore a 37 ° C per 5-10 min.
- Durante l'incubazione del passaggio 1.6, trasferimento 2YT agar superiore da passo 1.5 a temperatura ambiente (TA) raffreddare per circa 5 min a 42 ° C. Utilizzare il polso interno per verificare la temperatura di 2YT agar superiore; dovrebbe rimanere come liquido. Trasferire ciascuna miscela di diluizione da passo 1.5 a ciascuna provetta di agar superiore 2YT corrispondente. Stringere il coperchio di ogni provetta e miscelare capovolgendo più volte delicatamente e brevemente per evitare la generazione di bolle.
- Attentamente ogni versate lungo il bordo di una piastra di agar LB/tet pre-calda (dal punto 1.3) e inclinano la piastra leggermente per completamente e uniformemente riempire il piatto con la miscela, evitando l'introduzione di bolle. Tenere le piastre a temperatura ambiente per circa 5-10 min per solidificare l'agar superiore all'interno di ogni piatto e incubare le piastre di agar superiore 2YT a 37 ° C durante la notte per circa 15-18 h.
- Scegliere la piastra di diluizione dopo una notte crescita dal passaggio 1.8 con intorno 100-200 targhe medie, singoli, ben separati per inoculazione di placca. Usare una mano per tenere la piastra di agar contro una sorgente di luce e l'altra mano per tenere una pistola di pipetta caricata con una pipetta sterile lungo verticalmente pugnalare l'agar superiore e raccogliere una piastra singola e ben separata.
- Inclinano il puntale della pipetta leggermente per separare top agar con placca. Pipetta su e giù più volte per sloggiare l'agar con placca in un tubo di cultura di fondo tondo 14-mL pre-caricato con 1 mL di terreno di 2YT/kan/tet (Vedi tabella 1).
- Ripetere questa procedura per prendere 3-5 placche in totale. Lo scopo di raccogliere diverse placche è per garantire successo inoculazione, come una targa può essere debole e relativamente piccolo se confrontato con una colonia di batteri. Crescere le placche per 8 h a 37 ° C con agitazione a 200 giri/min.
- Trasferire un filmato di crescere cultura in 50 mL di terreno di 2YT/kan/tet in un pallone di sconcertato da 250 mL. Crescere a 37 ° C con agitazione a 200 giri/min durante la notte per circa 12 h.
- Inoculare 2-L tre boccette sconcertati che contiene 900 mL di superbroth/tet/kan medio ogni 5 ml della coltura durante la notte. Incubare a 37 ° C con agitazione a 200 giri/min per 6-7 h a OD600 intorno a 0.6-0.8.
- Raffreddare i tre fiaschi di coltura batterica in un bagno di ghiaccio per 5 min con costante agitando delicatamente a mano. La procedura seguente dovrebbe essere fatto in una stanza fredda, sul ghiaccio, con attrezzature e soluzioni prechilled.
- Utilizzare assorbanza tessuto asciugamano per asciugare il vetro esterno di ciascuna beuta; usare una mano per inclinare la bottiglia 1-L autoclavato centrifuga sulla panca e l'altra mano per versare il mezzo delicatamente da ciascuna beuta in ogni bottiglia di centrifuga.
- Spin a 5.000 × g e 4 ° C per 10 min a pellet i batteri.
- Dopo centrifugazione, delicatamente decantare il supernatante in un becher di rifiuti autoclavato 5-L.
- Riempire ogni flacone con 100 mL di filtrato sterile 1,0 mM HEPES, pH 7.4 e aggiungere un'ancoretta magnetica in autoclave ad ogni bottiglia per aiutare nella risospensione del pellet a 200 giri/min. Il turbinio e sloggiare il pellet intero da pareti della bottiglia ogni alcuni minuti durante l'agitazione magnetica. Una volta che la pallina è dissolto, riempire ogni bottiglia con un ulteriore 400 mL di filtrato sterile 1,0 mM HEPES, pH 7.4.
- Centrifugare a 5.000 × g e 4 ° C per 10 min. decantare il supernatante, mantenendo l'ancoretta in bottiglia.
- Ripetere i passaggi da 1,16-1,17 una volta.
- Ogni risospendere in 500 mL di filtrato sterile, 10% glicerolo ultrapura con l'ausilio delle barre mescolare. Il turbinio e sloggiare il pellet intero da pareti della bottiglia ogni alcuni minuti durante l'agitazione magnetica.
- Centrifugare e decantare come descritto al punto 1.17. Ripetere il passaggio 1.19 una volta. Utilizzare forcipe autoclavato lungo-braccio per rimuovere la barra per l'agitazione.
- Centrifugare a 5.000 × g e 4 ° C per 15 min. decantare il supernatante e rimuovere qualsiasi residuo di surnatante da ogni flacone di centrifuga con una pipetta.
- Aggiungere 3,0 mL di glicerolo al 10% ultrapura a una bottiglia e Risospendere delicatamente la pipettando. Trasferire la sospensione nel flacone successivo e ripetere fino a quando tutti i pellet sono risospese e combinati in una bottiglia. Circa 6 mL di cellule altamente concentrate sono ottenuti con un titolo di circa 3 x 1011 cfu/mL.
- Pre-chill per microcentrifuga da 1,5 mL sterilizzato nell'autoclave e scatola di immagazzinaggio di un tubo di 96 pozzetti senza divisori a-80 ° C per almeno 1 h prima di questo passaggio. Trasferire la microcentrifuga pre-refrigerati a camera fredda su ghiaccio prima di pipettaggio aliquote della sospensione pellet cellulare. Utilizzare una pipetta per aliquota 350 µ l di sospensione cellulare in ciascuna provetta per microcentrifuga da 1,5 mL.
- Trasferire le aliquote in un contenitore di scatola schiuma riempito con azoto liquido per congelamento flash (3-5 min).
- Trasportare la scatola della gomma piuma con azoto liquido per un congelatore a-80 ° C (vedi passo 1,23).
- Rimuovere la scatola di immagazzinaggio del tubo per microcentrifuga da 1,5 mL dal congelatore-80 ° C (vedi passo 1,23) e mettere il ghiaccio.
- Utilizzare rapidamente una rete metallica per setacciare le aliquote da azoto liquido e metterli in scatola per la conservazione di tubo. Conservare a-80 ° C.
Attenzione: Azoto liquido può causare ustioni e cura deve essere presa per la protezione di sicurezza. Utilizzare un phagemid Fab spina dorsale per verificare l'efficienza delle celle electro-competente preparate (lo stock di phagemid Fab spina dorsale dovrebbe essere in acqua ultrapura (MilliQ) a 400 ng / µ l). Scongelare 1 μg della spina dorsale Fab phagemid in acqua ultrapura di 10 µ l e un'aliquota di 350 μL di 320 electro-competente sul ghiaccio e prechill una cuvetta di elettroporazione di 0,2 centimetri gap sul ghiaccio.
- Aggiungere il 350 μL electro-competente 320 celle al 10 µ l di DNA dopo lo scongelamento e Miscelare pipettando diverse volte, mantenendo le miscele sul ghiaccio. Evitare l'introduzione di bolle.
- Pre-riscaldare 15 mL di terreno SOC in un tubo da 50mL in bagnomaria a 37 ° C per almeno 30 min prima di elettroporazione. Trasferire il composto di 360 μL in cuvetta ed eseguire elettroporazione seguendo le istruzioni del produttore.
- Salvato immediatamente le cellule dopo elettroporazione aggiungendo 1 mL di terreno SOC pre-riscaldato (da passo 1,27) e pipettaggio. Trasferire il mezzo dalla cuvette in un pallone di 125 mL pre-caricato con 5 mL di pre-riscaldato Soc.
- Sciacquare la cuvetta due volte, ogni volta con 1 mL di mezzo SOC pre-riscaldato e trasferire il mezzo al pallone da 125mL (vedi passo 1,27). Aggiungere il pre-riscaldato SOC terreno in un volume finale di 10 mL al pallone da 125mL.
- Incubare la coltura delle cellule di 10 mL per 30 min a 37 ° C con agitazione a 200 giri/min.
- Effettuare diluizioni seriali per determinare l'efficienza di trasfezione e il tasso di pre-infezione M13KO7.
- Utilizzare una pipetta multicanale per aggiungere 180 μL di 2YT media in ciascun pozzetto di una singola colonna di una piastra microtiter di 96.
- Fare dieci volte seriale 8 diluizioni: trasferire 20 μL di cultura dal passaggio 1,29 per il primo pozzetto della piastra, mix pipettando e trasferire 20 μL di miscela al successivo bene. Ripetere questo passaggio fino alla fine della diluizione seriale.
- Piastra 10 μL di ogni diluizione seriale sulle piastre LB/carb, LB/kan e LB/tet in duplice copia.
- Incubare per una notte a 37 ° C.
- Formula di calcolo di efficienza: si supponga che M è il numero medio di Colonia contato dalla piega diluita 10N (N è da 1-8). L'efficienza di trasfezione Escherichia coli 320 electro-competente delle cellule del phagemid Fab spina dorsale dalla piastra LB/carb è uguale a M X 10N + 3 cfu / µ g. Il tasso di pre-infezione M13KO7 è stimato dal rapporto delle colonie in LB/kan e LB/tet. La percentuale di cellule competenti di Escherichia coli 320 transfettate con la spina dorsale Fab phagemid è stimata dal rapporto delle colonie in LB/carb e LB/kan.
2. preparazione contenente uracile ssDNA (dU-ssDNA) dal modello Phagemid
Nota: A precedentemente segnalato phagemid Fab spina dorsale è stato utilizzato come modello per dU-ssDNA preparazione15. L'architettura della spina dorsale Fab phagemid è illustrata nella Figura 2. Un kit di spin di plasmide (QIAprep Spin M13) viene utilizzato per l'estrazione di dU-ssDNA con lievi modifiche.
- Pre-riscaldare una piastra LB/cmp (preparati e conservati a 4 ° C per meno di 1 - settimana-vecchio) in un incubatore a 37 ° C per 1 h. striscia fuori uno stock di glicerolo di cellule di Escherichia coli CJ236 (o un altro ceppo di dut−/ung−) con Anse sterili o suggerimenti sulla piastra LB/cmp pre-riscaldata. Incubare la piastra a 37 ° C durante la notte per circa 12-15 h.
- Scegli una singola Colonia con una punta sterile e inoculare in 2 mL di terreno di 2YT/cmp in una provetta di turno-fondo 14 mL in polipropilene.
- Incubare il mezzo a 37 ° C con agitazione a 200 giri/min per 3-4 h fino a OD600 viene raggiunta a circa 0.4-0.8 (crescita del log di fase).
- Aggiungere dei fagi di 5-10 μL di modello di Fab spina dorsale nella cultura e incubare a 37 ° C con agitazione a 200 rpm per 30 min consentire l'infezione fagica.
- Dopo l'incubazione, utilizzare una punta sterile per striscia fuori 10 μL di coltura su una piastra di LB/carb pre-riscaldata a 37 ° C. Incubare a 37 ° C durante la notte per circa 12-15 h.
- Selezionare una singola Colonia del CJ236 di e. coli contenenti spina dorsale Fab phagemid a inoculare una coltura starter di 3ml 2YT/carb/cmp in una provetta di turno-fondo 14 mL in polipropilene, crescere a 37 ° C con agitazione a 200 giri/min durante la notte per circa 12 h.
- Inoculare la coltura starter 0,3 mL in 30 mL 2YT/carb/cmp in un flacone di 250 mL sconcertato.
- Incubare la coltura cellulare a 37 ° C con agitazione a 200 giri/min per 3-4 h fino a OD600 viene raggiunta a circa 0.4-0.8 (crescita del log di fase).
- Aggiungere il M13KO7 (titolo stock, laboratorio di circa 1 x 1013 cfu/mL) alla cultura da passo 2.8 con una molteplicità di infezione (MOI) di circa 10 e il titolo finale degli M13KO7 è circa 1 x 1010 cfu/mL.
- Incubare a 200 giri/min e 37 ° C per 1 h.
- Pallina la cultura mediante centrifugazione in un tubo di turno-fondo 50 mL a 5.000 × g e 25 ° C per 20 min.
- Decantare il supernatante e risospendere il sedimento con 30 mL di fresco 2YT/carb/kan/uridina. Trasferire la risospensione in un nuovo flacone di 250 mL sconcertato.
- Incubare a 200 giri/min e 25 ° C per 22-24 h.
- Trasferire la cultura dal punto 2.13 in una provetta di turno-fondo 50 mL e centrifugare la coltura a 12.000 × g per 20 minuti per separare il fago supernatante dal pellet cellulare batterica. Trasferire il surnatante fago in un nuovo tubo tondo-fondo 50 mL e aggiungere 1/5 il volume finale della soluzione PEG/NaCl per precipitare il fago. Mescolare bene e incubare in ghiaccio per 30 min per far precipitare le particelle dei fagi.
- Centrifugare a 12.000 × g e 4 ° C per 30 min. decantare il supernatante e centrifugare a 4.000 × g e 4 ° C per 2 min. aspirare il supernatante rimanente.
- Risospendere il pellet di fago in 2 mL di filtrato sterile 1X PBS e trasferimento per provette per microcentrifuga da 1,5 mL. Centrifugare a 12.000 × g per 5 min in una microcentrifuga da banco per rimuovere eventuali detriti batterici residui e trasferire il surnatante fago in nuove provette microcentrifuga da 1,5 mL e conservare a 4 ° C.
- Verificare l'efficienza di incorporazione di uracile in CJ236 di Escherichia coli mediante confronto fianco a fianco con 320 di e. coli .
- Aggiungere 180 μL di 2YT media in ciascun pozzetto di una singola riga di una piastra a 96 pozzetti.
- Fare dieci 10 volte seriale diluizioni: trasferire 20 μL del surnatante fago al primo pozzetto della piastra, Miscelare pipettando e trasferire ben 20 μL di miscela a quella successiva. Ripetere questo passaggio fino alla fine della diluizione seriale.
- Aggiungere 10 μL di fago dalle ultime otto diluizioni seriali di infettare 90 μL di e. coli CJ236 e 320 di e. coli in fase di log (OD600 = 0.4-0.8). Incubare a 37 ° C per 30 min con agitazione delicata.
- Piastra 10 μL dalla diluizione seriale di ogni infezione in e. coli CJ236 e 320 di e. coli sulle piastre 2YT/Carb in duplice copia.
- Incubare per una notte a 37 ° C.
- Formula di calcolo del titolo: si supponga che M è il numero medio di Colonia contato dalla piega diluita 10N (N è da 1 a 10). Il titolo da e. coli CJ236 o e. coli 320 è uguale a M X 10N + 2 cfu/mL. Stimare l'efficienza dell'incorporazione di uracile dal rapporto titolo di e. coli CJ236 e 320 di e. coli .
- Aggiungere volume 1/100 di buffer precipitazione dei fagi MP nel fago surnatante in provette per microcentrifuga da 1,5 mL e girare sottosopra delicatamente per mescolare per diverse volte. Incubare a temperatura ambiente per almeno 2 min dei fagi particelle sono precipitate da terreno di coltura, e quindi, una soluzione nuvolosa dovrebbe essere visibile a questo punto.
- Applicare il campione dal punto 2.18 a una colonna di spin del plasmide (ad esempio, QIAprep) in una provetta da microcentrifuga da 1,5 mL. La capacità legante della colonna uno spin per ssDNA può raggiungere almeno 10 µ g. centrifuga a 6.000 × g e 25 ° C per 30 s in una microcentrifuga da banco. Scartare il flusso-attraverso la quale è nel tubo per microcentrifuga da 1,5 mL. Particelle dei fagi rimangono legate alla matrice colonna in questa fase.
- Aggiungere 0,7 mL della lisi dei fagi UT-MLB e buffer obbligatorio (Vedi discussione) alla colonna e incubare a temperatura ambiente per almeno 1 min. Centrifugare a 6.000 x g e a 25 ° C per 30 s e scartare il flusso continuo.
- Aggiungere un altro 0,7 mL di tampone UT-MLB e incubare a temperatura ambiente per almeno 1 min.
- Centrifugare a 6.000 × g per 30 s. scartare il flusso continuo. In questa fase, la proteina del cappotto dei fagi è separata dal dU-ssDNA, che rimane legato alla matrice colonna.
- Aggiungere 0,7 mL di etanolo contenente di wash buffer PE seguendo le istruzioni del produttore. Centrifugare a 6.000 × g per 30 s e scartare il flusso continuo.
- Ripetere il passaggio 2.23, poi centrifugare ancora una volta a 6.000 × g per 30 s per rimuovere residui buffer PE.
- Trasferire la colonna ad un nuovo tubo per microcentrifuga da 1,5 mL e aggiungere 100 μL di tampone di eluizione EB (10 mM Tris· CL, pH 8.5) al centro della membrana colonna.
- Incubare a temperatura ambiente per 10 min e centrifugare a 6.000 × g per 1 min eluire il dU-ssDNA. Circa, 1,5-2,5 μg dU-ssDNA/mL coltura può essere ottenuta.
- Analizzare il DNA eluito per electrophoresing 1 μL su una TAE del gel dell'agarosi di 1%. Il DNA deve essere visualizzato come una banda principalmente singola con senza sbavature.
- Determinare la concentrazione di DNA di misurare l'assorbanza su uno spettrofotometro nanodrop a 260 nm (un260 = 1.0 per 33 ng/μL di ssDNA). Le concentrazioni tipiche dU-ssDNA sono nel raggio di 200-500 ng/μL.
3. metodo di Kunkel basato mutagenesi Oligonucleotide-diretta
Note: Si consiglia di condurre reazioni su piccola scala prima di reazioni di fondo scala per garantire la qualità dei componenti del oligonucleotide e reazione mutagene. Un fumetto di metodo di Kunkel basato del oligonucleotide-regia mutagenesi è illustrata nella Figura 3. Varie dell'amminoacido diversità vengono introdotti nelle regioni di CDRH1, CDRH2, CDRH3 e CDRL3 con IMGT numerazione nomenclatura16 (tabella 2). Il teorico dell'amminoacido diversità di ogni CDR, aminoacido teorica totale diversità e sequenze di oligonucleotidi sono elencati nella tabella 2.
- Fosforilazione del oligonucleotide con chinasi di polinucleotide T4
- Combinare 0,6 μg di oligonucleotidi mutageni, progettato per mutare un CDR singolo, con 2 μL 10 X buffer TM, 2 μL 10 mM ATP e 1 μL 100 mM DTT. Aggiungere ultrapura H2O a un volume totale di 18 μL in una provetta da 1,5 mL. Per la costruzione della libreria, 4 reazioni di fosforilazione separate e parallele sono impostate corrisponde a CDRH1, CDRH2, CDRH3 e CDRL3, rispettivamente.
- Aggiungere 20 U (2 uL) della chinasi di polinucleotide T4 a ciascun tubo e incubare per 1h a 37 ° C (reazione 1, tabella 3). Utilizzare immediatamente per ricottura.
- Ricottura di oligonucleotidi per il modello
- A 20 μg di dU-ssDNA modello, aggiungere 25 μL di tampone di TM X 10, 20 μL di ogni soluzione del oligonucleotide fosforilato e ultrapura H2O ad un volume finale di 250 μL in una provetta 0,5 mL. Mescolare bene e trasferire in provette per PCR 0,20 mL (50 µ l) come reazione 2 nella tabella 3. Queste quantità di DNA forniscono un rapporto molare di 3:1 tra oligonucleotide e modello, supponendo che il rapporto di lunghezza di oligonucleotidi e modello è 1: 100.
- Incubare la reazione in una macchina PCR a 90 ° C per 3 min, 50 ° C per 3 min e 20 ° C per 5 min.
- Enzimatico di sintesi della CCC-dsDNA
- Alla miscela del oligonucleotide/modello 250 μL ricotto, aggiungere 10 μL di 10 mM ATP, 10 μL di miscela del dNTP 25mm, 15 µ l di 100 mM DTT, Weiss 30 unità T4 DNA ligasi e 30 U T7 polimerasi come reazione 3 nella tabella 3.
- Incubare la reazione nel tubo da 1,5 mL a 20 ° C durante la notte.
- Lavare e concentrare il CCC-dsDNA sintetizzato in un dispositivo di filtro centrifugo 0.5 mL con una membrana di dimensione del poro di 30 kDa a TA.
- Trasferire la miscela di reazione durante la notte per il dispositivo di filtro e aggiungere ultrapura H2O 400 µ l di volume finale. Spin a 14.000 × g per 10 min; il volume è inferiore a 50 μL.
- Scartare il flusso attraverso, aggiungere 400 µ l di ultrapura H2O nel filtro e girano a 14.000 × g per 10 min.
- Ripetere il punto 3.3.3.2 ancora una volta.
- Capovolgere il filtro in una microcentrifuga pulito per recuperare il CCC-dsDNA. Spin a 1.000 × g per 2 minuti; il volume recuperato è generalmente circa 20-40 μL. Il CCC-dsDNA recuperato possa essere utilizzato immediatamente per elettroporazione di e. coli o congelato a-20 ° C per un uso successivo. Normalmente 20-40 μg CCC-DNA può essere ottenuta.
- Electrophorese 1,0 µ l del prodotto di reazione eluiti a fianco il modello dU-ssDNA per visualizzare il risultato della reazione.
4. elettroporazione e calcolo delle dimensioni della libreria
- Raffreddare il CCC-dsDNA purificato (20 μg in un volume massimo di 50 μL) in una provetta da microcentrifuga da 1,5 mL e una cuvetta di elettroporazione di 0,2 centimetri gap sul ghiaccio.
- Pre-riscaldare 20ml di media SOC in una provetta conica per centrifuga in polipropilene da 50mL in bagnomaria a 37 ° C per almeno 30 min.
- Scongelare un'aliquota di 350 μL di electro-competente e. coli 320 sul ghiaccio. Aggiungere le cellule del DNA e mescolare accuratamente pipettando diverse volte. Evitare l'introduzione di bolle.
- Trasferire il composto in cuvetta ed eseguire elettroporazione seguendo le istruzioni del produttore. Ad esempio, quando viene utilizzato il sistema di elettroporazione di BTX ECM-630, le impostazioni rilevanti sono 2,5 kV intensità di campo, 125 Ω resistenza e capacità di 50 µF.
- Salvare immediatamente le cellule elettroporate aggiungendo 1 mL di terreno SOC pre-riscaldato e trasferimento in 17 mL di terreno SOC in un matraccio da 125mL. Sciacquare la cuvetta due volte con 1 mL di medium SOC e trasferimento al pallone stesso (volume finale è di 20 mL).
- Incubare per 30 min a 37 ° C con agitazione a 200 giri/min.
- Determinare l'efficienza di elettroporazione.
- Aggiungere 180 μL di 2YT media in ciascun pozzetto di una singola colonna di una piastra microtiter di 96.
- Fare dieci volte seriale 8 diluizioni: trasferire 20 μL della cultura 20 mL nel primo pozzetto della piastra, mescolare con il pipettaggio e trasferire 20 μL di miscela bene a altro. Ripetere questo passaggio fino alla fine della diluizione seriale.
- Piastra 10 μL di ciascuna delle diluizioni seriali su una piastra LB/carb in duplice copia. Piastra di 100 µ l restanti da ciascuna delle diluizioni seriali su piastre LB/carb separate per croce-verifica del conteggio di Colonia. Queste piastre fornirà anche singoli cloni per analisi di sequenza ed ELISA (Vedi sezione 5).
- Incubare per una notte a 37 ° C.
- Contare le colonie dai duplicati μL 10 sulla piastra LB/carb. Si supponga che M sia la Colonia Media numero contato su 10N piega piastra LB/carb (N è da 1-8). La dimensione di raccolta totale è uguale a 2 M X 10N + 3 colonie.
- Aliquotare la cultura dal punto 4.6 equamente in due 2-L sconcertati boccette, ciascuno contenente 500 mL di mezzo di 2YT/carb/kan per la generazione della libreria dei fagi.
- Incubare a 37 ° C con agitazione a 200 giri/min durante la notte per circa 16 h.
- Trasferire la cultura a due bottiglie 1-L autoclavato centrifuga e centrifugare per 30 min a 12.000 × g a 4 ° C.
- Trasferire il surnatante in due nuove bottiglie di centrifuga in autoclave 1-L e aggiungere 1/5 il volume finale della soluzione PEG/NaCl per precipitare il fago. Incubare in ghiaccio per 30 min.
- Centrifuga per 30 min a 12.000 × g e 4 ° C. Attentamente decantare il supernatante ed evitare di disturbare il precipitato dei fagi. Centrifugare per 1 min a 4.000 x g e rimuovere il surnatante con una pipetta rimanente.
- Risospendere il pellet di fago con 20 mL di filtrato sterile 1 X PBS buffer e trasferirlo in una nuova provetta 50 mL.
- A pellet la materia insolubile mediante centrifugazione per 5 min a 12.000 × g e 4 ° C. Trasferire il surnatante in una provetta da 50 mL nuova.
- Misurare la concentrazione dei fagi mediante lo spettrofotometro (OD268 = 1.0 per una soluzione di 5 X 1012 fago/mL).
- Regolare la concentrazione dei fagi a 5 X 1012 fago/mL in 1X PBS con 10% glicerolo ultrapura.
- Aliquotare 1 mL di soluzione dei fagi per microcentrifuga da 1,5 mL. Utilizzare le librerie immediatamente per il panning o conservare a-80 ° C.
5. qualità valutazione di proteina A / L diretta analisi ELISA obbligatoria e sequenziamento
- Scegliere casualmente 96 singole colonie sulla piastra LB/carb dal passaggio 4.7.3 in una piastra di coltura ben 96-profondo contenente 800 μL di 2YT/carb in ciascun pozzetto. Incubare per 3-4 h a 37 ° C con agitazione a 1.000 giri/min a OD600 = 0.4-0.8.
- Aggiungere 100 μL di M13KO7 (1 X 1011 cfu/mL) in ciascun pozzetto di una 96-profonda ben cultura piastra con una pipetta multicanale. Incubare a 37 ° C con agitazione a 1.000 giri/min per 1 h.
- Aggiungere 100 μL di 2YT contenente 10 X concentrazione di kanamicina (500 μg/mL) ad ogni pozzetto con una pipetta multicanale. Incubare per una notte a 37 ° C con agitazione a 1.000 giri/min.
- Sciogliere la proteina L a 1 µ g/mL in 1X PBS. Cappotto 3/4 dei pozzi in un piatto di polistirolo di legame alle proteine 384 pozzetti elevata con 30 µ l/pozzetto di soluzione della proteina L. Incubare per una notte a 4 ° C.
- Scartare la soluzione di rivestimento di proteina L durante la notte dal punto 5.4. Aggiungere 50 µ l di M-PBST (la soluzione bloccante) a tutti i pozzetti della piastra di polistirolo legame 384 pozzetti alta con una pipetta multicanale. Incubare le piastre su un agitatore per micropiastre per 1 h a TA.
- Rotazione verso il basso la cultura durante la notte dal punto 5.3 in una piastra a 96 pozzetti profondi cultura a 3.000 x g per 10 min a 4 ° C. Il fago è nel surnatante.
- Scartare la soluzione bloccante dal punto 5.5. Aggiungere 15 μL di M-PBST e 15 μL di ogni fago surnatante (punto 5.6) 3 dei pozzi della proteina L rivestito come triplice copia e 1 controllo bene come negativo non rivestiti, usando una pipetta multicanale. Incubare per 1h a RT con agitazione a 200 giri/min.
- Scartare la soluzione dei fagi. Lavare la piastra 6 volte con 80 μL di PBST da una rondella di piatto automatizzato.
- Aggiungere 15 μL di M-PBST e 15 µ l di proteina A-HRP (1:1, 500 diluito in 1X PBS) ad ogni pozzetto con una pipetta multicanale e incubare per 1h a RT con agitazione a circa 200 giri/min.
- Scartare la soluzione di proteina A-HRP/M-PBST. Lavare la piastra 6 volte con 80 μL di PBST.
- Aggiungere substrato TMB (30 μL/pozzetto) con una pipetta multicanale e incubare per 2-3 minuti fino a quando il colore si sviluppa. Fermare la reazione con 1,0 M H3PO4 (30 μL/pozzetto).
- Leggere le piastre spettrofotometro a 450 nm. Cloni positivi sono definiti come coloro che presentano un rapporto medio di assorbanza450 OD di pozzi di proteina L per il bene superiore a 3.0 M-PBST.
- In un 96-profondo bene, infettare 50 μL di 320 in 2YT/tet in fase di log con 5 μL del fago stesso utilizzato per l'ELISA (punto 5.6) per 30 minuti a TA.
- Aggiungere 950 μL di 2YT/carb il 96-largo passaggio 5.13 bene e incubare per una notte a 37 ° C con agitazione a 1.000 giri/min.
- Estrarre il DNA phagemid da kit di estrazione del DNA di mini-preparazione. Utilizzare il primer a Monte di VL (5'-TCGCTTTGTTTTTATTTTTTAATGTA-3') e VH (5'-GACTACTAATAACATAAAGTCTACGCCG-3') per l'analisi di sequenza valutare la diversità di sequenza di biblioteca.
Risultati
Seguendo il diagramma di flusso della costruzione favolosa biblioteca (Vedi Figura 1), abbiamo preparato M13KO7 helper dei fagi pre-infettato 320 Escherichia coli cellule electro-competenti. L'efficienza di queste cellule electro-competente è stimato come 2 X 109 cfu / µ g quando la spina dorsale phagemid Fab per la costruzione della libreria è stata utilizzata (Figura 4).
L'efficienza di incorporazione di uracile in confronto del titolo in cellule CJ236 di e. coli e di e. coli 320 stato controllato. Le cellule di e. coli CJ236 e 320 di Escherichia coli sono state infettate da fagi che harboring dU-ssDNA. E. coli 320 ha enzimi (dUTPase e uracile glicosilasi) che possono degradare l'uracile-contenenti DNA, mentre Escherichia coli CJ236 manca di questi enzimi e non può degradare l'uracile-contenenti DNA. Per ottenere un'efficienza di incorporazione di uracile accettabile, i titoli da e. coli CJ236 devono essere almeno 104 volte superiori a quelli da 320 e. coli . In caso contrario la popolazione di selvaggio-tipo aumenterà nella libreria dell'anticorpo costruito grazie alla incorporazione di uracile inefficiente. Figura 5 ha mostrato che il titolo da e. coli CJ236 è di circa 3 X 105 volte superiore a quella da 320 e. coli , che indica un'incorporazione di uracile efficiente dei fagi ssDNA.
Successivamente, abbiamo preparato ed estratte dU-ssDNA. La purezza di dU-ssDNA è controllata mediante elettroforesi su gel di agarosio (Figura 6). Poi è stata condotta la mutagenesi oligonucleotide-diretta e l'efficienza della conversione dU-ssDNA al CCC-DNA è stato valutato (Figura 6). Tre prodotti con bassa motilità di dU-ssDNA possono essere fruiti sul gel tra cui la band più veloce esecuzione (CCC-dsDNA), la banda centrale debole (intaccato band) e la band di più lenta esecuzione (DNA strand-spostato).
Dopo elettroporazione in e. coli 320, la dimensione della biblioteca è stata stimata dalla piastra di incubazione overnight (Vedi punto 4.7.5). La dimensione media biblioteca era 5 X 109 da diluizioni seriali duplicati su piastre LB/carb (Figura 7). Tuttavia, la dimensione stimata in questa fase può contenere dei fagi che non visualizzare Fabs dovuta alla presenza di un frameshift o codone di arresto oppure misfolded Fabs. Sequenziamento ed ELISA sono stati usati per stimare la diversità funzionale della biblioteca costruita. 96 a caso selezionati singoli cloni sono stati inviati per l'analisi di sequenziamento. La tabella 4 Mostra che 90 su 96 cloni singoli casualmente scelto con successo sono stati sequenziati, che contiene 70 cloni senza un codone di stop prematuro (53 cloni con almeno un CDR mutante) e 17 cloni con la sequenza wild-type e 20 cloni con un codone di stop prematuro alle diverse regioni. Entro i 70 cloni, mutanti tassi di CDRH1, CDRH2, CDRH3 e CDRL3 sono 50%, 57%, 53% e 56%, rispettivamente, mentre il tasso di mutanti con almeno un CDR è del 76%. In the 20 cloni con un codone di stop prematuro (90%), il codone di stop prematuro era principalmente derivato da frameshift degli iniettori di mutagenesi oligonucleotide, tra cui 45% (CDRH1), 10% (CDRH2), 15% (CDRH3) e 20% (CDRL3).
Per rilevare la visualizzazione dei Fabs correttamente piegato, una proteina A / L base ELISA è stato impiegato come è noto che la proteina A e proteina L può riconoscere corretto ripiegamento il quadro VH e VL quadro, rispettivamente17,18. In accordo con l'analisi di sequenziamento, il test ELISA in triplice copia (Figura 8) ha mostrato che i 20 cloni con un codone di stop prematuro erano tutti negativi, mentre i 17 cloni con una sequenza wild-type sono state tutte positivi, quando il rapporto positivo era impostare empiricamente a 3.0. Per i 53 cloni con almeno un CDR mutante, 43 cloni erano positivi in ELISA mentre 10 cloni erano negativi; Questo indica che la maggior parte dei cloni sono stata piegata bene mentre il CDRs da 10 cloni possa avere effetti negativi sul pieghevole Fab. In totale, 43 cloni fuori i 90 cloni (48%) sono stati ben piegati e conteneva almeno un CDR mutante. Così, la diversità funzionale dell'amminoacido della biblioteca costruita basato su proteina A / analisi di sequenza e L ELISA è stata stimata per essere 2.4 X 109 (cioè, 48% di 5 X 109).
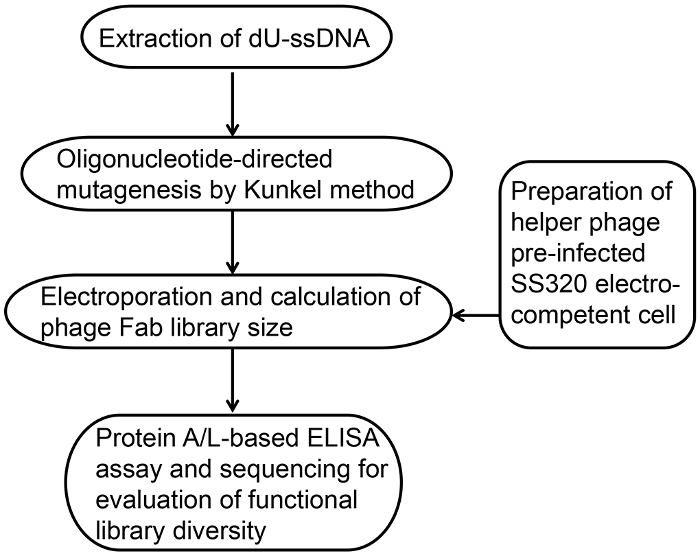
Figura 1: Panoramica della costruzione dei fagi-visualizzato favolosa biblioteca. Costruzione dei fagi-visualizzato favolosa biblioteca segue una serie di passaggi di base. Si tratta di preparazione delle cellule batteriche electro-competente ad alta efficienza, l'estrazione di dU-ssDNA, mutagenesi oligonucleotide-diretta metodo basato di Kunkel, elettroporazione e calcolo del fago dimensione favolosa biblioteca, valutazione funzionale di proteina A / L ELISA e DNA sequence analysis. Clicca qui per visualizzare una versione più grande di questa figura.
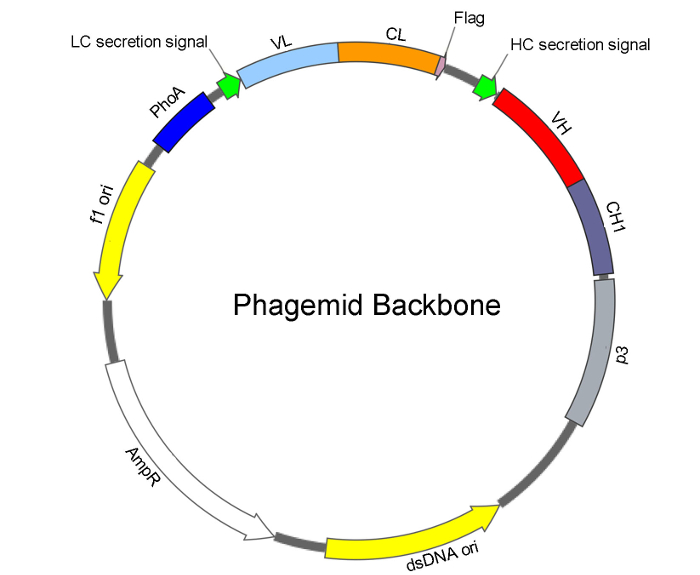
Figura 2: Phagemid architettura per la costruzione della libreria Fab. Le caratteristiche di base della spina dorsale phagemid consistono delle origini del singolo filamento (f1 ori) e DNA a doppia elica (dsDNA ori) replica e un gene di resistenza all'ampicillina/carbenicillina (AmpR). Per la visualizzazione di Fab, sotto il controllo del promotore della fosfatasi alcalina (PhoA), il phagemid contiene una cassetta di sialophosphoprotein di auto espressione e la secrezione di: catena (LC), costituita da un segnale di secrezione, VL (regione variabile della catena leggera), CL (costante di luce regione della catena leggera) e C-terminale bandiera tag; e catena pesante (HC) costituito da un segnale di secrezione, VH (regione variabile della catena pesante) e CH1 (1 regione costante della catena pesante) fuso con una proteina di cappotto minori dei fagi p3. Montaggio della catena leggera e pesante catena in Fab entro il Periplasma Escherichia coli dirige il display di Fab sulla superficie dei fagi. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: schema del metodo di Kunkel basato mutagenesi oligonucleotide-. In questo protocollo, abbiamo usato il metodo di Kunkel per preparare dU-ssDNA modello. Oligonucleotidi per CDRH1, CDRH2, CDRH3 e CDRL3 con diversità progettato sono fosforilati, ricotto al modello e utilizzati per convertire ss-DNA in CCC-dsDNA. Elettroporazione in cellule di Escherichia coli 320 electro-competente, a seguito dell'eteroduplex DNA è riparato a wild type o la forma mutata; A causa della presenza dell'uracile nel filo del tipo selvatico, il processo di riparazione favorisce la forma mutata, e così, la forma mutante domina la libreria. Clicca qui per visualizzare una versione più grande di questa figura.
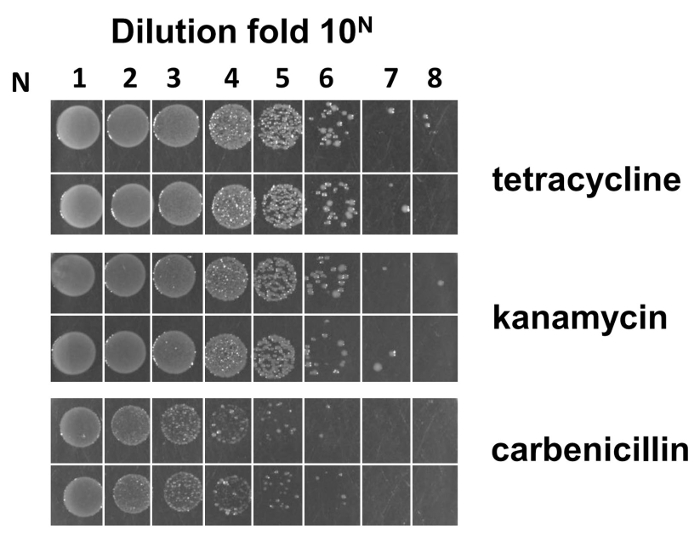
Figura 4: Stima della M13KO7 pre-infettati efficienza elettro-competente delle cellule di Escherichia coli 320. Un vettore di spina dorsale phagemid è stato utilizzato per verificare l'efficienza di elettroporazione delle cellule competenti. Formula per calcolare l'efficienza è la seguente: si supponga che M è il numero medio di Colonia contato dalla piega più diluita 10N (N è da 1-8) in duplice copia. E. coli 320 efficienza dalla piastra LB/carb è uguale a M X 10N + 3 cfu / µ g. L'efficienza delle celle electro-competente è di circa 2 X 109 cfu / µ g. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Valutazione dell'incorporazione di uracile nel ssDNA da infezione dei fagi delle cellule e. coli CJ236 e 320 di e. coli . Basato sul metodo di Kunkel, l'efficienza di incorporazione di uracile è controllato tramite il confronto del titolo di infezione dei fagi in cellule CJ236 di e. coli e di e. coli 320. La formula di calcolo del titolo è come segue: si supponga che M è il numero medio di Colonia contato dalla piega più diluita 10N (N è da 1 a 10), e che il titolo da e. coli CJ236 o e. coli 320 è uguale a M X 10N + 2 cfu/mL. L'efficienza dell'incorporazione di uracile può essere stimato dal rapporto titolo di e. coli CJ236 e 320 di e. coli . Il titolo in CJ236 di e. coli era 9 X 1012 cfu/mL mentre il titolo a 320 di e. coli era 3 X 107 cfu/mL. Il rapporto di titolo di CJ236 di e. coli ed e. coli 320 era 3 X 105. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: conversione di dU-ssDNA in CCC DNA mediante mutagenesi oligonucleotide-. A seguito di mutagenesi oligonucleotide-diretta, è stata valutata l'efficienza di conversione di dU-ssDNA al CCC-DNA. dU-ssDNA completamente è stato convertito a dsDNA. Il gruppo dominante è CCC-dsDNA mentre c'è una parte minore del dsDNA intaccato e DNA strand-spostato. Clicca qui per visualizzare una versione più grande di questa figura.
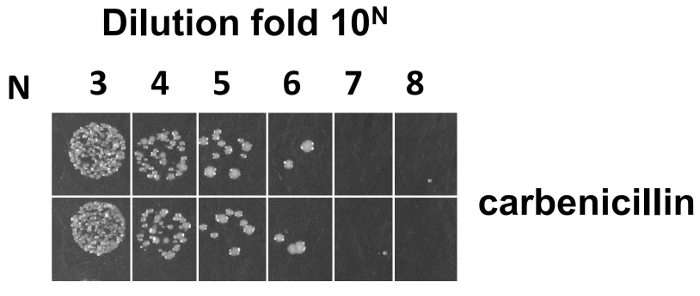
Figura 7: titolazione dei fagi per il calcolo della dimensione di biblioteca. Dopo elettroporazione in e. coli 320, la dimensione della biblioteca è stata stimata da diluizioni seriali su piastre LB/carb. La formula di calcolo di dimensioni è come segue: si supponga di M è il numero medio di Colonia contato dalla piega più diluita 10N da una piastra di 2YT/Carb (N è da 1-8), è pari a 2 M X 10N + 3. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 8: proteina A / L diretto dei fagi associazione ELISA. Proteina L può riconoscere che il quadro della catena chiara del kappa ben piegato VL e la proteina un può riconoscere il quadro della catena pesante ben piegato VH. Associazione di Fab con proteina L e A indica il corretto ripiegamento della catena pesante sia catena leggera. In breve, proteina L in triplice copia ed il controllo negativo M-PBST sono stati rivestiti alla piastra, surnatanti di Fab fago da diversi cloni sono stati incubati con proteina L e M-PBST, quindi dopo il lavaggio, proteina A-HRP è stata utilizzata per catturare dei fagi Fab associato. Letture di ELISA fago ha mostrate 90 casualmente selezionati cloni con lettura di sequenziamento di successo. Una linea di soglia che rappresenta il clone come positivo è stato empiricamente definito dove il rapporto tra valore di assorbanza OD450 da proteina L (triplice copia con barre di errore in media) rispetto al controllo negativo è stato più di 3.0. Sono stati mostrati tre gruppi basati sull'analisi di sequenziamento, corrispondente a un mutante senza codone di stop in rosso (53 cloni), wild type (WT) in blu (17 cloni) e mutante con codone di stop in verde (20 cloni). Clicca qui per visualizzare una versione più grande di questa figura.
| Installazione di reagente | Componente | Importo | Commenti/Descrizione |
| 2YT medio | Estratto di lievito | 10 g | Aggiungere acqua ultrapura per portare il volume di 1.0 L, regolare il pH a 7.0, autoclave. |
| TRYPTONE | 16 g | ||
| NaCl | 5 g | ||
| 2YT top agar | Estratto di lievito | 10 g | Aggiungere acqua ultrapura per portare il volume di 1.0 L e regolare il pH a 7.0, calore per sciogliere, autoclave. |
| TRYPTONE | 16 g | ||
| NaCl | 5 g | ||
| Agar granulato | 7,5 g | ||
| 2YT/carb/cmp media | Carbenicillina | 100 μg/mL | |
| Cloramfenicolo | 10 μg/mL | ||
| 2YT/carb/kan/uridina medio | Carbenicillina | 100 μg/mL | |
| Kanamicina | 50 μg/mL | ||
| Uridina | 0,25 μg/mL | ||
| 2YT/carb/tet medio | Carbenicillina | 100 μg/mL | |
| Tetraciclina | 10 μg/mL | ||
| 2YT/carb medio | Carbenicilin | 100 μg/mL | |
| Medio 2YT/kan | Kanamicina | 50 μg/mL | |
| 2YT/kan/tet medio | Kanamicina | 50 μg/mL | |
| Tetraciclina | 10 μg/mL | ||
| Medio 2YT/tet | Tetraciclina | 10 μg/mL | |
| 2YT/cmp media | Cloramfenicolo | 10 μg/mL | |
| Agar medio LB | Estratto di lievito | 5 g | Aggiungere acqua ultrapura per portare il volume di 1.0 L, regolare il pH a 7.0, autoclave. Per LB agar, aggiungere 20 g di granulato agar, autoclave. |
| TRYPTONE | 10 g | ||
| NaCl | 10 g | ||
| Piastre di LB/carb | LB agar | 1L | |
| Carbenicillina | 100 μg/mL | ||
| Piastre di LB/tet | LB agar | 1 L | |
| Tetraciclina | 10 μg/mL | ||
| Piastre di LB/cmp | Cloramfenicolo | 10 μg/mL | |
| Piastre di LB/kan | Kanamicina | 50 μg/mL | |
| Mezzo SOC | Estratto di lievito | 5 g | Aggiungere acqua ultrapura per portare il volume a 1,0 L e regolare il pH a 7.0, autoclave. |
| TRYPTONE | 20 g | ||
| NaCl | 0,5 g | ||
| KCl | 0,2 g | ||
| 2.0 M MgCl2 | 5,0 mL | ||
| 1.0 glucosio di M | 20 mL | ||
| Medio superbroth | TRYPTONE | 12 g | Aggiungere acqua ultrapura a 900 mL, autoclave, aggiungere 100 mL di autoclavato 0.17 M KH2PO4, 0,72 M K2HPO4. |
| Estratto di lievito | 24 g | ||
| Glicerolo | 5 mL | ||
| Medie di superbroth kan/tet | Kanamicina | 50 μg/mL | |
| Tetraciclina | 10 μg/mL | ||
| 1X PBS | NaCl | 137 mM | Aggiustare il pH a 7,2, autoclave. |
| KCl | 3 mM | ||
| Na2HPO4 | 8 mM | ||
| KH2PO4 | 1,5 mM | ||
| Gel di agarosio/TAE | Tampone TAE | ||
| Agarosio | 1% (p/v) | ||
| GelRed | 1: 10000 (v/v) | ||
| Substrato TMB | TMB | 50% (v/v) | |
| Substrato perossidasi di H2O2 | 50% (v/v) | ||
| Buffer di M-PBST | 1X PBS | 100 ml | |
| Tween-20 | 0,05% (v/v) | ||
| Latte in polvere senza grassi | 5% (v/v) | ||
| 5 X PEG/NaCl | PEG-8000 | 20% (p/v) | Aggiungere acqua ultrapura per portare il volume di 1L e autoclave. |
| NaCl | 2.5 M | ||
| Buffer di PBST | 1X PBS | 1 L | Filtro di 0,22 μm-sterilizzare. |
| Tween-20 | 0,05% (v/v) | ||
| TM tampone 10x | MgCl2 | 0.1 M | Regolare il pH a 7,5. |
| Tris | 0,5 M | ||
| 1,0 mM HEPES, pH 7.4 | 1.0 HEPES M | 4,0 mL | Filtro di 0,22 μm-sterilizzare. |
| Acqua ultrapura | 4.0 L | ||
| 10% (v/v) glicerolo ultrapura | Glicerolo ultrapura | 100 ml | Filtro di 0,22 μm-sterilizzare. |
| Acqua ultrapura | 900 mL | ||
| Acqua ultrapura | H20 | Privo di DNasi, RNasi-free, apirogeno. |
Tabella 1: Installazione di reagente.

Tabella 2: diversità di CDR e mutagenesi primer. Le sequenze di DNA delle regioni CDR per essere subito una mutazione sono mostrate in rosso; sequenze vengono formattate utilizzando il codice del nucleotide IUPAC. "X" indica tri-nucleotide da una miscela studiata per contenere insiemi differenti dell'amminoacido; "n" indica il diverso numero di X. cinque primer con un diverso numero di X sono stato usato per diversificare CDRL3 o CDRH3, rispettivamente, per generare la lunghezza variabile di CDRL3 e CDRH3. I numeri di residui sono definiti dalla nomenclatura IMGT. Per favore clicca qui per scaricare questa tabella.
| Mutagenesi metodo basato di Kunkel | ||
| Reazione 1. Fosforilazione del oligonucleotide con chinasi di polinucleotide T4 | ||
| Componente | Importo | Finale |
| oligonucleotidi mutageni | 0,6 μg | |
| TM tampone 10x | 2 ΜL | 1 X |
| 10 mM ATP | 2 ΜL | 1 mM |
| 100 mM DTT | 1 ΜL | 5 mM |
| Chinasi di polinucleotide T4 (10 U/μL) | 2 ΜL | 20 U |
| Ultrapura H20 | Fino a 20 μL | |
| Impostazione di reazione | ||
| Passaggio 1. | 37 ° C per 1 h | |
| Reazione 2. Ricottura di oligonucleotidi per il modello | ||
| Componente | Importo | Finale |
| modello di dU-ssDNA | 20 μg | 20 μg |
| TM tampone 10x | 25 ΜL | 1 X |
| fosforilato CDRH1 oligonucleotidi | 20 ΜL | 0,6 μg |
| fosforilato CDRH2 oligonucleotidi | 20 ΜL | 0,6 μg |
| fosforilato CDRH3 oligonucleotidi | 20 ΜL | 0,6 μg |
| fosforilato CDRL3 oligonucleotidi | 20 ΜL | 0,6 μg |
| Ultrapura H20 | Fino a 250 μL | |
| Impostazione di reazione | ||
| Passaggio 1. | 90 ° C per 3 min | |
| Passaggio 2. | 50 ° C per 5 min | |
| Passaggio 3. | 20 ° C per 5 min | |
| Reazione 3. Enzimatico di sintesi della CCC-dsDNA | ||
| Componente | Importo | Finale |
| miscele di oligonucleotidi ricotto/modello | 250 ΜL | |
| 10 mM ATP | 10 ΜL | 346 µM di ogni nucleotide |
| miscela del dNTP (25 mM di ogni nucleotide) | 10 ΜL | 865 µM di ogni nucleotide |
| 100 mM DTT | 15 ΜL | 5 mM |
| Ligasi del DNA T4 | 1 ΜL | 30 unità di Weiss |
| T7 polimerasi | 3 ΜL | 30 U |
| Impostazione di reazione | ||
| Passaggio 1. | 20 ° C per una notte | |
Tabella 3: Componenti del metodo di Kunkel e procedure basano di reazione.
| Gruppo | Clonare il numero | Regione | Percentuale | ||
| Nessun codone di stop prematuro | 70 | Mutazione CDRH1 | 50% (35/70) | ||
| Mutazione CDRH2 | 57% (40/70) | ||||
| Mutazione CDRH3 | 53% (37/70) | ||||
| Mutazione CDRL3 | 56% (39/70) | ||||
| Almeno una mutazione di CDR | 76% (53/70) | ||||
| Codone di stop prematuro | 20 | CDRH1 difetto | 45% (9/20) | ||
| CDRH2 difetto | 10% (2/20) | ||||
| Difetto CDRH3 | 15% (3/20) | ||||
| CDRL3 difetto | 20% (4/20) | ||||
| Altro difetto | 10% (2/20) | ||||
Tabella 4: Analisi di sequenza di CDRH1, CDRH2, CDRH3 e CDRL3 dalla libreria Fab sintetica.
Discussione
Per costruire alta diversità, dei fagi-visualizzato Fab biblioteche, controllo di qualità sono necessari punti di controllo per monitorare le varie fasi del processo di costruzione, tra cui la competenza delle celle electro-competenti, qualità del modello dU-ssDNA, efficienza di CCC-dsDNA sintesi, titolo dopo elettroporazione, Fab pieghevole e diversità dell'amminoacido di CDRs mediante analisi di sequenza di cloni di Fab-fago.
Alta resa e purezza di dU-ssDNA è essenziale per tasso di alta mutagenesi. Nella nostra esperienza, induzione dei fagi a 25 ° C durante la notte può produrre più dU-ssDNA di quello da induzione dei fagi a 37 ° C durante la notte. Questo è in accordo con un precedente report19. Per quanto riguarda estrazione ssDNA, il plasmide iniziale Spin kit (QIAprep) conteneva MLB per l'associazione e lisi dei fagi. Più tardi, MLB fu interrotto con motivo sconosciuto e sostituito da PB. Abbiamo trovato che la resa di dU-ssDNA è molto più bassa dal trattamento PB rispetto a quello dal trattamento di MLB. In questo protocollo, abbiamo usato un reagente chiamato UT-MLB20 per sostituire MLB e trovato che la resa di dU-ssDNA è simile a quello del kit iniziale di Spin.
Come CDRH3 e CDRL3 sono le regioni più diverse per antigene riconoscimento21, per introdurre una diversità su misura con un insieme specifico di rapporti e combinazioni di aminoacidi e rimuovere ridondanza bias e introdotti da codoni degenerati come codoni di stop NNK (N, equimolare di a/c/G/T; K, equimolare di G/T), trimero codone basati su phosphoramidite oligonucleotidi22 con esattamente un codone trimero corrispondente a uno specifico amminoacido sono stati progettati per CDRH3 e CDRL3. Inoltre, lunghezze variabili di oligonucleotidi CDRH3 e CDRL3 sono state utilizzate per aumentare ulteriormente la diversità.
Dopo la sintesi enzimatica di CCC-dsDNA, generalmente tre bande sono osservate mediante elettroforesi su gel di agarosio e le bande dovrebbero essere chiare senza sbavatura. Tra questi, la band più veloce esecuzione è il CCC-dsDNA che può produrre un alto tasso di mutazione (80% circa) dopo elettroporazione23. La band di esecuzione più lento è il DNA strand-sfollati che nasce dall'attività inerente strand-spostamento della polimerasi T7 e ha una mutazione basso tasso (20% circa)23. La banda centrale debole è scalfita DNA dopo l'estensione a causa di insufficiente attività della ligasi del DNA T4 o fosforilazione del oligonucleotide insufficiente.
Una piscina di sequenziamento piccolo campione fu usata per valutare la diversità di libreria anche se non accurato24. Per stimare con precisione la reale diversità, prossima generazione sequenziamento (NGS) può essere una buona opzione nella profondità di diversità della biblioteca costruita25di data mining. In pratica, a causa delle attuali sfide di NGS tecnologia tra cui leggere lunghezza, precisione, costo ed elevata velocità effettiva, il sequenziamento della libreria dei fagi Fab utilizzata in questo protocollo con la lunghezza di circa 950 bp che attraversa CDRH1, CDRH2, CDRH3 e CDRL3 non è realizzabile; Tuttavia, è possibile stimare il scFv (circa 700 bp) diversità di biblioteca all'interno della gamma di milioni24,25. Un'altra chiave standard per valutare la diversità della biblioteca costruita è utilizzare la libreria di pan contro diversi tipi di antigeni e calcolare i cloni positivi acquisiti dal diversità biblioteca è direttamente correlata con il tasso di successo dell'antigene panning26. Piattaforma di selezione di una velocità effettiva elevata è adatto per questo scopo e i lettori possono riferirsi a un protocollo dettagliato riportato da Miersch et al. 27
Teoricamente, librerie dei fagi-visualizzato anticorpo sintetico con diversità su misura possono essere utilizzate per indirizzare qualsiasi antigene e così avere applicazioni ampie. Attualmente, le aziende tra cui Cambridge anticorpo Technology (CAT), MedImmune, Genentech, Dyax, Bioinvent, Pfizer e MorphoSys si basano molto su piattaforme di visualizzazione dei fagi per anticorpo terapeutico sviluppo28. Inoltre, molti dei fagi display core, tecnologia, brevetti sono scaduti il29. Senza dubbio, Ciò libererà il massimo potenziale della tecnologia degli anticorpi dei fagi-visualizzato.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori apprezzano Dr. Frederic Fellouse dal laboratorio Sidhu per osservazioni critiche sulla costruzione della libreria di Kunkel metodo basata sintetica dei fagi Fab. Gli autori apprezzano la signora Alevtina Pavlenco e gli altri membri dal laboratorio Sidhu per il prezioso aiuto di preparazione ad alta efficienza elettro-competenti le cellule e. coli e alta qualità dU-ssDNA. Questo lavoro è stato supportato dalla Fondazione nazionale di scienze naturali della Cina (Grant No.: 81572698, 31771006) a DW e dall'Università di ShanghaiTech (Grant No.: F-0301-13-005) al laboratorio di ingegneria dell'anticorpo.
Materiali
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1.0 M H3PO4 | Fisher | AC29570 | |
| 1.0 M Tris, pH 8.0 | Invitrogen | 15568-025 | |
| 10 mM ATP | Invitrogen | 18330-019 | |
| 100 mM dithiothreitol | Fisher | BP172 | |
| 100 mM dNTP mix | GE Healthcare | 28-4065-60 | solution containing 25 mM each of dATP, dCTP, dGTP and dTTP. |
| 3,3’,5,5’-tetramethylbenzidine (TMB) | Kirkegaard & Perry Laboratories Inc | 50-76-02 | |
| 50X TAE | Invitrogen | 24710030 | |
| Agarose | Fisher | BP160 | |
| Carbenicillin, carb | Sigma | C1389 | 100 mg/mL in water, 0.22 μm filter-sterilize, work concentration: 100 μg/mL. |
| Chloramphenicol, cmp | Sigma | C0378 | 100 mg/mL in ethanol, 0.22 μm filter-sterilize, work concentration: 10 μg/mL. |
| EDTA 0.5 M, pH 8.0 | Invitrogen | AM9620G | |
| Granulated agar | VWR | J637-500G | |
| H2O2 peroxidase substrate | Kirkegaard & Perry Laboratories Inc | 50-65-02 | |
| K2HPO4 | Sigma | 795488 | |
| Kanamycin, kan | Fisher | AC61129 | 50 mg/mL in water, 0.22 μm filter-sterilize, work concentration: 50 μg/mL. |
| KH2PO4 | Sigma | P2222 | |
| Na2HPO4 | Sigma | 94046 | |
| NaCl | Alfa Aesar | U19C015 | |
| Nanodrop | Fisher | ND2000C | |
| NaOH | Fisher | SS256 | ! CAUTION NaOH causes burns. |
| NON-Fat Powdered Milk | Sangon Biotech | A600669 | |
| PEG-8000 | Fisher | BP233 | |
| Protein A-HRP conjugate | Invitrogen | 101123 | |
| QIAprep Spin M13 Kit | Qiagen | 22704 | |
| QIAquick Gel Extraction Kit | Qiagen | 28706 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| Recombinant Protein L | Fisher | 77679 | |
| T4 DNA polymerase | New England Biolabs | M0203S | |
| T4 polynucleotide kinase | New England Biolabs | M0201S | |
| T7 DNA polymerase | New England Biolabs | M0274S | |
| Tetracycline, tet | Sigma | T7660 | 50 mg/mL in water, 0. 22 μm filter-sterilize, work concentration: 10 μg/mL. |
| Tryptone | Fisher | 0123-07-5 | |
| Tween-20 | Sigma | P2287 | |
| Ultrapure glycerol | Invitrogen | 15514-011 | |
| Uridine | Sigma | U3750 | 25 mg/mL in ethanol, work concentration: 0.25 μg/mL. |
| Yeast extract | VWR | DF0127-08 | |
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E.coli CJ236 | New England Biolabs | E4141 | Genotype: dut- ung- thi-1 relA1 spoT1 mcrA/pCJ105(F' camr). Used for preparation of dU-ssDNA. |
| E.coli SS320 | Lucigen | 60512 | Genotype: [F'proAB+lacIq lacZΔM15 Tn10 (tetr)] hsdR mcrB araD139 Δ(araABC-leu)7679 ΔlacX74 galUgalK rpsL thi. Optimized for high-efficiency electroporation and filamentous bacteriophage production. |
| M13KO7 | New England Biolabs | N0315S | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| 0.2-cm gap electroporation cuvette | BTX | ||
| 96-well 2mL Deep-well plates | Fisher | 278743 | |
| 96-well Maxisorp immunoplates | Nunc | 151759 | |
| Baffled flasks | Corning | ||
| Benchtop centrifuge | Eppendorf | 5811000096 | |
| Centrifuge bottles | Nalgene | ||
| ECM-630 electroporator | BTX | ||
| Magnetic stir bars | Nalgene | ||
| Thermo Fisher centrifuge | Fisher | ||
| High speed shaker | TAITEK | MBR-034P | |
| Microplate shaker | QILINBEIER | QB-9002 | |
| Liquid handler for 96 and 384 wells | RAININ | ||
| Mutil-channel pipette | RAININ | E4XLS | |
| Amicon concentrator | Merck | UFC803096 |
Riferimenti
- Singh, S., et al. Monoclonal Antibodies: A Review. Curr Clin Pharmacol. , (2017).
- Reichert, J. M. Antibodies to watch in 2017. MAbs. 9 (2), 167-181 (2017).
- Kohler, G., Milstein, C. Continuous cultures of fused cells secreting antibody of predefined specificity. Nature. 256 (5517), 495-497 (1975).
- Milstein, C. The hybridoma revolution: an offshoot of basic research. Bioessays. 21 (11), 966-973 (1999).
- Gray, A. C., Sidhu, S. S., Chandrasekera, P. C., Hendriksen, C. F., Borrebaeck, C. A. Animal-Friendly Affinity Reagents: Replacing the Needless in the Haystack. Trends Biotechnol. 34 (12), 960-969 (2016).
- Hutchison, C. A., et al. Mutagenesis at a specific position in a DNA sequence. J Biol Chem. 253 (18), 6551-6560 (1978).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Sidhu, S. S. . Phage Display in Biotechnology and Drug Discovery. , (2005).
- Weiss, G. A., Watanabe, C. K., Zhong, A., Goddard, A., Sidhu, S. S. Rapid mapping of protein functional epitopes by combinatorial alanine scanning. Proc Natl Acad Sci U S A. 97 (16), 8950-8954 (2000).
- Frenzel, A., Schirrmann, T., Hust, M. Phage display-derived human antibodies in clinical development and therapy. MAbs. 8 (7), 1177-1194 (2016).
- Sidhu, S. S., Fellouse, F. A. Synthetic therapeutic antibodies. Nat Chem Biol. 2 (12), 682-688 (2006).
- Adams, J. J., Sidhu, S. S. Synthetic antibody technologies. Curr Opin Struct Biol. 24, 1-9 (2014).
- Kunkel, T. A. Rapid and efficient site-specific mutagenesis without phenotypic selection. Proc Natl Acad Sci U S A. 82 (2), 488-492 (1985).
- Sidhu, S. S., Lowman, H. B., Cunningham, B. C., Wells, J. A. Phage display for selection of novel binding peptides. Methods Enzymol. 328, 333-363 (2000).
- Persson, H., et al. CDR-H3 diversity is not required for antigen recognition by synthetic antibodies. J Mol Biol. 425 (4), 803-811 (2013).
- Lefranc, M. P., et al. IMGT unique numbering for immunoglobulin and T cell receptor variable domains and Ig superfamily V-like domains. Dev Comp Immunol. 27 (1), 55-77 (2003).
- Graille, M., et al. Complex between Peptostreptococcus magnus protein L and a human antibody reveals structural convergence in the interaction modes of Fab binding proteins. Structure. 9 (8), 679-687 (2001).
- Graille, M., et al. Crystal structure of a Staphylococcus aureus protein A domain complexed with the Fab fragment of a human IgM antibody: structural basis for recognition of B-cell receptors and superantigen activity. Proc Natl Acad Sci U S A. 97 (10), 5399-5404 (2000).
- Huang, R., Fang, P., Kay, B. K. Improvements to the Kunkel mutagenesis protocol for constructing primary and secondary phage-display libraries. Methods. 58 (1), 10-17 (2012).
- Chen, G., Sidhu, S. S. Design and generation of synthetic antibody libraries for phage display. Methods Mol Biol. 1131, 113-131 (2014).
- Padlan, E. A. Anatomy of the antibody molecule. Mol Immunol. 31 (3), 169-217 (1994).
- Virnekas, B., et al. Trinucleotide phosphoramidites: ideal reagents for the synthesis of mixed oligonucleotides for random mutagenesis. Nucleic Acids Res. 22 (25), 5600-5607 (1994).
- Fellouse, F. A., Sidhu, S. S. . Making and Using Antibodies: A Practical Handbook. , 157-180 (2006).
- Fantini, M., et al. Assessment of antibody library diversity through next generation sequencing and technical error compensation. PLoS One. 12 (5), e0177574 (2017).
- Glanville, J., et al. Deep sequencing in library selection projects: what insight does it bring?. Curr Opin Struct Biol. 33, 146-160 (2015).
- Perelson, A. S., Oster, G. F. Theoretical studies of clonal selection: minimal antibody repertoire size and reliability of self-non-self discrimination. J Theor Biol. 81 (4), 645-670 (1979).
- Miersch, S., et al. Scalable high throughput selection from phage-displayed synthetic antibody libraries. J Vis Exp. (95), e51492 (2015).
- Ponsel, D., Neugebauer, J., Ladetzki-Baehs, K., Tissot, K. High affinity, developability and functional size: the holy grail of combinatorial antibody library generation. Molecules. 16 (5), 3675-3700 (2011).
- Petering, J., McManamny, P., Honeyman, J. Antibody therapeutics - the evolving patent landscape. N Biotechnol. 28 (5), 538-544 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon