Method Article
Un protocollo efficiente per valutare la modulazione dell'attività di ERK nei primi modelli di sindrome di Noonan del pesce zebra tramite microscopia FRET dal vivo e immunofluorescenza
In questo articolo
Riepilogo
Le RASopatie sono sindromi genetiche multisistemiche causate dall'iperattivazione della via RAS-MAPK. Varianti potenzialmente patogene in attesa di convalida emergono continuamente, mentre le scarse evidenze precliniche limitano la terapia. Qui, descriviamo il nostro protocollo in vivo per testare e convalidare in modo incrociato i livelli di attivazione ERK associati alla RASopathy e la sua modulazione farmacologica durante l'embriogenesi mediante imaging FRET dal vivo in zebrafish teen-reporter.
Abstract
Le RASopatie sono sindromi genetiche causate dall'iperattivazione di ERK e che provocano malattie multisistemiche che possono anche portare alla predisposizione al cancro. Nonostante un'ampia eterogeneità genetica, la maggior parte dei casi è alla base di mutazioni germinali con guadagno di funzione nei regolatori chiave della via RAS-MAPK e, grazie a tecniche avanzate di sequenziamento, continuano ad essere identificate varianti potenzialmente patogene che influenzano la via RAS-MAPK. La validazione funzionale della patogenicità di queste varianti, essenziale per una diagnosi accurata, richiede protocolli rapidi e affidabili, preferibilmente in vivo. Data la scarsità di trattamenti efficaci nella prima infanzia, tali protocolli, soprattutto se scalabili in modelli animali economicamente vantaggiosi, possono essere strumentali nell'offrire un terreno preclinico per il riposizionamento/riposizionamento dei farmaci.
Qui descriviamo passo dopo passo il protocollo per la generazione rapida di modelli transitori di RASopathy in embrioni di zebrafish e l'ispezione diretta dei cambiamenti di attività ERK associati alla malattia che si verificano già durante la gastrulazione attraverso l'imaging FRET (Förster resonance energy transfer) multispettrale in tempo reale. Il protocollo utilizza un reporter ERK transgenico di recente costituzione e integrato con l'hardware dei microscopi commerciali. Forniamo un esempio di applicazione per i modelli di zebrafish della sindrome di Noonan (NS) ottenuti dall'espressione di Shp2D61G. Descriviamo un metodo semplice che consente la registrazione del cambiamento del segnale ERK nel modello di pesce NS prima e dopo la modulazione farmacologica del segnale da parte degli inibitori MEK a basso dosaggio disponibili. Descriviamo in dettaglio come generare, recuperare e valutare i segnali FRET raziometrici da acquisizioni multispettrali prima e dopo il trattamento e come convalidare in modo incrociato i risultati tramite l'immunofluorescenza classica su embrioni interi nelle fasi iniziali. Descriviamo quindi come, attraverso l'esame di parametri morfometrici standard, interrogare i cambiamenti tardivi nella forma dell'embrione, indicativi di una conseguente compromissione della gastrulazione, negli stessi embrioni la cui attività ERK è valutata mediante FRET vivo a 6 ore dopo la fecondazione.
Introduzione
Le RASopatie sono sindromi genetiche che compromettono il normale sviluppo e colpiscono vari organi e tessuti. Queste condizioni sono spesso causate da mutazioni germinali con guadagno di funzione (GoF) nei geni chiave e negli attori coinvolti nella segnalazione RAS/MAK, con conseguente iperattivazione (aumento della fosforilazione) della chinasi regolata dal segnale extracellulare (ERK). ERK regola alcuni processi fondamentali importanti durante lo sviluppo - crescita dei tessuti - traslocando nel nucleo 1,2. Le mutazioni somatiche nei geni coinvolti nella via RAS-MAPK sono gli eventi più comuni che portano al cancro3. Pertanto, non sorprende che la predisposizione al cancro si osservi anche nelle RASopatie. La sindrome di Noonan (NS), caratterizzata da ritardo dello sviluppo, bassa statura, deficit cognitivi di gravità variabile e cardiomiopatia, è la forma più comune diRASopathy 2. Nella maggior parte dei casi, la malattia è causata da mutazioni di GoF in PTPN11, il primo gene RASopathy ad essere scoperto all'inizio del 20004 che codifica per la proteina tirosina fosfatasi SHP2, che agisce come regolatore positivo della via.
Da allora, grazie all'uso esponenziale di approcci di sequenziamento dell'esoma in pazienti non diagnosticati, varianti potenzialmente patogene che influenzano i fattori coinvolti nella RAS-MAPK, e probabilmente legate a varie forme di RASopatie, continuano ad essere scoperte e attendono una caratterizzazione funzionale per un'efficiente stratificazione dei pazienti2. Per raggiungere questo obiettivo, sono necessari protocolli sperimentali che garantiscano una rapida e informativa validazione funzionale a livello di organismo. L'impiego di modelli di mammiferi classici e standardizzati per testare varianti con significato sconosciuto sarebbe costoso, estremamente dispendioso in termini di tempo e richiederebbe metodi invasivi in animali di grandi dimensioni non trasparenti. Tale strategia non è chiaramente compatibile con l'obbligo di test rapidi, dato l'onere sociale rappresentato dai pazienti con RASopathy poveri o non diagnosticati, attualmente privi di gestione o trattamento. I protocolli per la valutazione quantitativa dei tratti fenotipici chiave e dei correlati molecolari in interi organismi servirebbero anche ad accelerare la possibile traduzione clinica di farmaci possibilmente disponibili per i pazienti con RASopathy mediante riposizionamento/riposizionamento.
Il pesce zebra è un modello di vertebrato ideale per studiare le malattie che influenzano lo sviluppo precoce. Per cominciare, il pesce zebra condivide un alto livello di omologia genetica con gli esseri umani. L'elevata fecondità dei pesci adulti si traduce in una grande produzione di embrioni che sono piccoli e si sviluppano rapidamente. Gli embrioni sono trasparenti nelle fasi iniziali, in modo tale che i principali processi di sviluppo - epibolia, gastrulazione, assi e formazione del piano corporeo - possono essere visualizzati senza sforzo utilizzando la microscopia standard. Inoltre, la disponibilità di linee transgeniche che possono essere utilizzate per tracciare il comportamento cellulare specifico e gli eventi molecolari dinamici nello spazio e nel tempo durante lo sviluppo, in combinazione con tecniche avanzate per generare modelli genetici, è imbattibile. Inoltre, le letture fenotipiche possono essere valutate a più livelli nel pesce zebra (dai difetti dell'organismo a quelli cellulari) e sono già stati stabiliti test dedicati per diverse malattie, tra cui le RASopatie5. Inoltre, metodi relativamente semplici di immersione in bagno per la somministrazione di farmaci durante le fasi iniziali, almeno per i composti idrosolubili, consentono lo screening di farmaci ad alto rendimento in vivo in un formato a 96 pozzetti.
Da un punto di vista molecolare, gli studi che utilizzano approcci standard, come l'immunoistochimica e l'immunoblot, dimostrano in modo robusto la correlazione tra l'attivazione di ERK e i difetti di sviluppo associati alla RASopathy negli embrioni di pesce 6,7. Il biosensore FRET di tipo EKAR, sviluppato di recente nel pesce zebra (Tg[ef1a:ERK biosensor-nes], Teen) fornisce uno strumento affidabile in vivo per registrare l'attivazione di ERK durante l'embriogenesi in modo spazio-temporalmente risolto. Pertanto, potrebbe essere utile per una migliore valutazione delle alterazioni dinamiche di ERK e delle modulazioni farmacologiche nei modelli di pesce RASopathy.
Nel sensore Teen , uno specifico substrato di ERK nel reporter viene fosforilato all'attivazione di ERK, innescando un cambiamento conformazionale che porta nelle immediate vicinanze il donatore fluorescente di CFP (D) e l'accettore fluorescente Ypet (YFP migliorato) (A). Se lo spettro di emissione D si sovrappone considerevolmente allo spettro di assorbimento dell'A, può verificarsi FRET (assorbimento di energia da D ad A). Questo è proporzionale alla distanza tra D e A e, quindi, in Teen, allo stato di attivazione dell'ERK. È possibile impostare diversi protocolli di imaging utilizzando moduli di imaging standard e avanzati di microscopi standard o confocali sia in campioni vivi che fissi. Dopo l'eccitazione D, l'acquisizione di scansioni multispettrali lungo uno spettro definito di emissione (λ) da CFP a YFP seguita da algoritmi spettrali di "unmixing" è tra i metodi più affidabili per registrare e quantificare i dati FRET8. Può essere applicato anche a esemplari vivi di zebrafish per registrare in vivo la dinamica dei tessuti.
Seguendo i precedenti rapporti 6,9 e la nostra recente applicazione7, qui, descriviamo in dettaglio il flusso di lavoro passo-passo utilizzando Teen fish per valutare l'attivazione di ERK nelle cellule al margine del polo animale dei modelli NS all'inizio della gastrulazione e correlarla con i difetti caratteristici degli assi corporei visibili solo più tardi nello sviluppo. Mostriamo come ottenere ed esaminare i dati quantitativi di FRET da gastrule NS vive prima e dopo il trattamento con un MEKi disponibile e come convalidare i risultati tramite immunoistochimica standard contro ERK fosforilato (attivo) o eseguire analisi morfometriche correlate dei difetti di allungamento dell'embrione.
Il flusso di lavoro potrebbe essere applicato per potenziare il test funzionale delle varianti emergenti e dei geni malattia putativamente associati alle RASopatie e per ottenere informazioni sulla correlazione delle dinamiche di attivazione delle ERK spazialmente e temporalmente durante lo sviluppo dei vertebrati e sui difetti morfologici negli embrioni. Dimostriamo che questo protocollo può essere utilizzato anche per testare l'efficacia di farmaci candidati che agiscono per modulare l'attivazione di ERK.
Protocollo
Tutte le procedure sperimentali che hanno coinvolto la stabulazione e l'allevamento degli animali sono state condotte secondo le linee guida ARRIVE per l'utilizzo del pesce zebra nella ricerca animale e autorizzate dalla Direzione Generale della Sanità Animale e dei Farmaci veterinari - DGSAF. Tutte le reazioni DNA/RNA e le sessioni di imaging possono essere aumentate o ridotte a piacere, a seconda del materiale finale richiesto o del numero di geni e varianti testate.
1. Generazione e trattamento farmacologico di modelli transitori di RASopathy di zebrafish
NOTA: Per monitorare l'espressione di varianti associate alla RASopathy, possono essere utilizzati costrutti specifici che ospitano la sequenza codificante desiderata (cds) della proteina di interesse in frame con i cds di piccoli tag non fluorescenti (come myc o simili). In questo modo, i livelli di espressione della proteina mutante possono essere valutati mediante western blot standard rispetto al tag. Se sono disponibili anticorpi contro la proteina specifica di interesse, i tag possono essere evitati. L'immunofluorescenza può essere utilizzata anche per valutare l'espressione proteica all'interno del tessuto embrionale seguendo protocolli standard. Questo tipo di esperimento di controllo può essere utile per correlare l'espressione di proteine mutanti con i livelli di attivazione indotti di ERK. L'uso di tag fluorescenti non è consigliabile in combinazione con l'imaging FRET, data la possibile interazione tra le emissioni di fluorescenza durante la microscopia.
- Linearizzazione plasmidi (giorni 1-2) (Figura 1A)
NOTA: Quando si genera un modello di malattia transitoria nel pesce zebra (RAsopathy qui) causata da una mutazione GoF che colpisce un gene specifico, un approccio rapido consiste nel preparare l'mRNA che codifica per il wild-type (WT) e la proteina mutante di interesse, che dovrebbe poi essere iniettato in embrioni di zebrafish seguendo i passaggi seguenti. Il plasmide utilizzato come stampo per trascrivere l'mRNA dovrebbe contenere il CDS (sequenza codificante) a lunghezza intera desiderato per il gene di interesse clonato a valle di un promotore della polimerasi T7, Sp6 o T3 e a monte del segnale di poliadenilazione (polyA). Se non disponibile, il primo passo consiste nel produrlo clonando il pesce zebra o il CDS desiderato dall'uomo in un vettore adatto10 e convalidarlo mediante sequenziamento SANGER. Per la clonazione, considerare un tempo extra (in media 1 settimana compresa la clonazione, lo screening delle colonie e l'isolamento e l'espansione dei cloni corretti).- Linearizzare il plasmide con opportuni enzimi di restrizione tagliando una sola volta a valle il segnale della poliA (qui KpnI). Per ottenere un'efficiente linearizzazione plasmidica, mescolare 3 γ (μg/μL) di DNA plasmidico, 2 μL di enzima di restrizione (20.000 Unità/mL) e 10 μL di tampone di reazione 10x in un volume finale di 100 μL in acqua priva di nucleasi. Miscelare accuratamente i componenti pipettandoli alcune volte e incubare la miscela di reazione a 37 °C per 2-4 ore.
- Verificare l'esito della linearizzazione mediante elettroforesi su un gel di agarosio all'1,5%.
- Diluire 10 volte la soluzione madre di TBE (48,5 g di Tris, 11,4 mL di acido acetico glaciale, 20 mL di EDTA 0,5 M [pH 8,0]) con acqua priva di nucleasi.
- Preparare il gel di agarosio sciogliendo nel microonde 1,5 g di agarosio in un volume finale di 100 ml di soluzione 1x TBE. Quindi, aggiungere 3,5 μL di colorante in gel.
- Colare, il gel versando la soluzione di agarosio in camere di dimensioni adeguate, a seconda del numero di condizioni/plasmidi. Fissare i pettini e attendere circa 1 ora fino a quando il gel non si sarà solidificato; Quindi, rimuovere i pettini e posizionare il gel polimerizzato nell'apposito vassoio per gel per l'elettroforesi.
- Mescolare alcuni μL del plasmide digerito ("taglio") e 1,5 μL di tampone di caricamento 6x (contenente un colorante per monitorare l'elettroforesi) in un volume finale di 10 μL di acqua priva di nucleasi. Preparare allo stesso modo anche un campione di controllo separato con il plasmide non digerito ("non tagliato"). Caricare i campioni nelle corsie dell'agarosio e, nella prima corsia, caricare una scala del DNA da 1 Kb. Eseguire l'elettroforesi del DNA a 100 V per 30 minuti.
- Visualizzare e documentare le bande di DNA risultanti dalla corsa su un transilluminatore UV standard. Verificare l'efficienza della linearizzazione dei plasmidi ispezionando il modello delle bande di DNA confrontando "taglio" e "non tagliato".
NOTA: I plasmidi sufficientemente linearizzati dovrebbero funzionare più velocemente come una banda affilata. Assicurarsi che la scissione del DNA sia completa perché l'inizio della reazione di trascrizione è un passaggio limitante chiave che può essere ostacolato dalla presenza di forme plasmidiche circolarizzate, con conseguente scarsa resa dell'mRNA.
- Procedere alla purificazione del plasmide linearizzato utilizzando i kit di purificazione del DNA basati su colonna di rotazione disponibili in commercio e conservare la preparazione di DNA purificato a -20 °C fino al momento del bisogno.
NOTA: Invece della linearizzazione, è possibile utilizzare un prodotto della PCR come stampo per la trascrizione dell'mRNA, a condizione che includa la sequenza del promotore della polimerasi a monte e la sequenza del segnale della poliA a valle del codone di stop. I prodotti PCR devono anche essere purificati e ispezionati su un gel di agarosio prima di procedere.
- In vitro Trascrizione, purificazione e controllo di qualità dell'mRNA (Giorni 2-3) (Figura 1A)
NOTA: pulire accuratamente l'area di lavoro e le pipette con soluzioni di decontaminazione a base di RNAsi. Utilizzare materiali e reagenti privi di nucleasi; Indossare i guanti. Ciò ridurrà al minimo la contaminazione da RNasi e consentirà una migliore resa di mRNA a lunghezza intera.
Trascrivi mRNA capped e poliadenilati che codificano per WT e proteine mutanti da plasmidi linearizzati utilizzando kit standard per la trascrizione di mRNA in vitro e seguendo le istruzioni del produttore.- Centrifugare il plasmide linearizzato purificato per alcuni secondi a 17.949 × g per assicurarsi che i detriti vengano raccolti sul fondo della provetta e non trasferiti nella reazione di trascrizione.
- Scongelare tutti i componenti a temperatura ambiente (RT) ad eccezione della miscela di analoghi NTP e CAP, che vengono mantenuti in ghiaccio. Mantenere la polimerasi a -20 °C fino al momento dell'uso.
- Centrifugare brevemente tutti i reagenti a 17.949 × g prima dell'uso per evitare la perdita di reagenti o la contaminazione accidentale e agitare brevemente il tampone di reazione 10x e la miscela analogica NTP/CAP pronta all'uso fino a quando non sono completamente in soluzione. Mantieni il tampone di reazione 10x su RT.
- Preparare la reazione di trascrizione a RT aggiungendo 600-800 ng del plasmide linearizzato e purificato a una soluzione contenente 10 μL di miscela analogica 2x NTP/CAP, 2 μL di tampone di reazione 10x, 2 μL di enzima SP6, T7 o T3 (a seconda del promotore specifico a monte del CDS nel plasmide) in un volume finale di 20 μL costituito con acqua priva di nucleasi. Miscelare accuratamente pipettando su e giù alcune volte.
- Incubare la reazione di trascrizione a 37 °C in un termociclatore. Assicurati di impostare anche la temperatura del coperchio a 37 °C ed esegui la reazione per 2 ore.
- Per rimuovere il plasmide residuo che non è stato trascritto durante la reazione, aggiungere 1 μL di soluzione standard di DNAsi (2 Unità/μL). Miscelare accuratamente la reazione pipettando alcune volte e incubare a 37 °C per altri 30 minuti.
- Prima di procedere, verificare la qualità e l'integrità dell'mRNA sintetizzato in vitro mediante elettroforesi di agarosio TBE/formammide. Sciogliere 1,0 g di agarosio in 50 mL di soluzione 1x TBE, aggiungere 5,5 mL di formammide al 37% e 3,5 μL di colorante in gel, mescolare e fondere il gel come spiegato sopra.
ATTENZIONE: Quando si maneggia la formammide, indossare dispositivi di protezione individuale (DPI) e utilizzare un cappuccio chimico. - Miscelare 1 μL di mRNA trascritto con 2,5 μL di tampone di caricamento colorante 2x Formamide in acqua priva di nucleasi in un volume totale di 5 μL. Miscelare accuratamente i componenti pipettandoli alcune volte e caricare i campioni nelle corsie di gel. Far funzionare il gel a 100 mV per 10-15 minuti in un tampone preraffreddato 1x TBE. Eseguire anche una scala di DNA e/o RNA da 1 kb.
- Opzionale: Denaturare la scala e l'mRNA a 70 °C per 10 minuti.
- Visualizza e documenta le bande di mRNA risultanti su un transilluminatore UV standard.
NOTA: Lunghe tirature e alte temperature aumentano la probabilità di degradazione dell'RNA. - Se l'integrità e le dimensioni dell'RNA incappucciato sintetizzato sono ottimali, procedere con la poliadenilazione del C-terminale aggiungendo alla reazione: 36 μL di acqua priva di nucleasi, 20 μL di tampone di carico denaturante 5x, 10 μL di 25 mM MnCl2, 10 μL di 10 mM ATP e 4 μL di enzima di poliadenilazione (come la Poli(A) Polimerasi purificata di E. coli ). Miscelare accuratamente la reazione pipettandola alcune volte e incubare i 100 μl della reazione finale a 37 °C per un'ulteriore 1 ora.
NOTA: Non è consigliabile controllare la qualità dell'RNA tramite elettroforesi su gel dopo la reazione polyA perché l'RNA apparirà macchiato a causa delle code polyA. - Precipitare e recuperare l'mRNA sintetizzato, incappucciato e poliadenilato utilizzando sali appropriati come LiCl. In breve, mescolare volumi uguali di acqua priva di nucleasi e una soluzione standard di LiCl 2,5 M (30 μL) e incubare per >30 minuti a -20 °C.
NOTA: Una precipitazione efficiente può essere ottenuta dopo 2 ore, ma l'incubazione notturna in genere aumenta la resa finale dell'mRNA. - Per pellettare l'mRNA purificato, centrifugare la reazione per 30 minuti a 4 °C a 17.949 × g e rimuovere il surnatante. Ora, lavare il pellet con ~1 mL di etanolo al 70% (EtOH) e centrifugare per 15 minuti a 4 °C a 17.949 × g per rimuovere i contaminanti e i nucleotidi residui dalla reazione.
- Asciugare il pellet all'aria e poi rimetterlo in sospensione in 20 μL di acqua priva di nucleasi. Sciogliere bene l'RNA pipettandolo delicatamente e ripetutamente a RT e incubare a 10 minuti.
NOTA: Controllare il pellet ogni 5 minuti per garantire l'evaporazione dell'EtOH. Durante la risospensione, data la viscosità dell'RNA, pipettarlo fino a quando non è adeguatamente sciolto in acqua. - Valutare la concentrazione e la purezza dell'mRNA preparato misurando l'assorbanza a 260 nm e 280 nm (rapporto 260/280) in uno spettrofotometro. Misurare diverse diluizioni scalari della preparazione di RNA per garantire una corretta stima della concentrazione.
NOTA: Normalmente, la concentrazione dell'RNA sintetizzato varia da 1 a 2 μg/mL, ma la quantità finale può variare a seconda della lunghezza del CDS e della quantità di materiale di partenza. - Durante la valutazione della qualità, mantenere lo stock di mRNA in ghiaccio, quindi aliquotarlo in aliquote da 5 μL e conservarlo a -80 °C fino al momento dell'iniezione.
- Preparazione di soluzioni e materiali standard per l'iniezione di embrioni e il trattamento farmacologico (giorni 2-3, Figura 1B)
- Aghi per microiniezione per embrioni di zebrafish utilizzando capillari sterili (dimensioni standard dei capillari: 1,0 OD x 0,58 ID x 100 L mm). Utilizzare un estrattore disponibile in commercio e applicare le impostazioni desiderate, in termini di tensione di uscita, modalità di trazione (uno o due passaggi) e forza di trazione per ottenere lunghezza, diametro e forma dell'ago della punta variabili. Per le caratteristiche dell'ago comunemente usate per il pesce zebra, fare riferimento ad Abdelrahman e colleghi11.
NOTA: Le impostazioni ottimali possono variare da un laboratorio all'altro perché le condizioni ambientali (come l'umidità o la temperatura ambiente) possono influenzare i risultati dell'estrazione dell'ago. Le impostazioni devono essere regolarmente ritestate e calibrate. Gli aghi estratti possono essere conservati presso RT in contenitori puliti per diversi mesi. - Preparare la soluzione madre 30x "Danieau" a pH 7,6 sciogliendo 101,7 g di NaCl, 1,56 g di KCl, 2,96 g di MgSO4 x 7H2O, 4,25 g di Ca(NO3)2 e 35,75 g di HEPES in 1 L di acqua a doppia distillazione (DD) per preparare la soluzione di microiniezione. Preparare anche una soluzione madre di rosso fenolo al 5% in acqua DD da utilizzare come tracciante visibile della miscela iniettabile.
- Preparare la soluzione di terreno E3 per la crescita embrionale diluendo 4 mL di NaCl (5 M), 680 μL di KCl (1 M), 1,32 mL di CaCl2 x 2H2O (1 M) e 1,32 mL di MgSO4 (1 M) in un volume finale di 4 L di acqua ad osmosi inversa.
- Preparare le soluzioni madre dei farmaci desiderati (in questo esempio: l'inibitore MEK [MEKi] PD032590) come soluzioni madre secondo la scheda tecnica del prodotto e la solubilità (10 mM del MEKi di scelta sciogliendo 5 mg in 1,036 mL di DMSO). Mescolare accuratamente e generare piccole aliquote da conservare a -80 °C fino al momento dell'uso.
NOTA: Il DMSO è un solvente comune utilizzato per dissolvere composti sia polari che non polari e mostra solubilità in un'ampia gamma di solventi organici e in acqua. - Preparare una sospensione di artemia salina viva lasciando schiudere le cisti di Artemia salina in soluzione di schiusa (2 g/L di cisti di Artemia salina in 30 g/L di sale oceanico precedentemente disciolto in acqua ad osmosi inversa) a 28-30 °C per almeno 18 ore, garantendo un'aerazione costante.
NOTA: Le condizioni di coltura e il tempo di schiusa potrebbero essere influenzati dalle condizioni ambientali dell'impianto e dai lotti di cisti e dovrebbero essere riesaminati regolarmente e reimpostati se necessario.
- Aghi per microiniezione per embrioni di zebrafish utilizzando capillari sterili (dimensioni standard dei capillari: 1,0 OD x 0,58 ID x 100 L mm). Utilizzare un estrattore disponibile in commercio e applicare le impostazioni desiderate, in termini di tensione di uscita, modalità di trazione (uno o due passaggi) e forza di trazione per ottenere lunghezza, diametro e forma dell'ago della punta variabili. Per le caratteristiche dell'ago comunemente usate per il pesce zebra, fare riferimento ad Abdelrahman e colleghi11.
- Preparazione delle coppie riproduttive per i pesci Tg[ef1α:ERK biosensor-nes] (Teen) (Giorno 3)
- Il giorno dell'accoppiamento, nutrire regolarmente le coppie adulte secondo il protocollo approvato della struttura animale.
- Pulire la coltura contenente artemia salina schizzata filtrando le cisti non schiuse o vuote con due filtri di diverse dimensioni (fase di filtro I: 180 μm, fase di filtro II: 112 μm).
- Raccogliere e lavare i gamberetti in salamoia filtrati in 200 ml di acqua ad osmosi inversa per nutrire 10-15 pesci adulti con circa 5-10 ml di soluzione di artemia salina.
NOTA: Controlla la qualità della soluzione di artemia salina schiusa osservando la loro vitalità, motilità, dimensioni e colore e assicurati di rimuovere le cisti non schiuse o vuote, che sono indigeribili per i pesci e possono causare danni al tratto gastrointestinale dei pesci. Per le coppie riproduttive, è preferibile fornire l'ultimo pasto della giornata almeno 3 ore prima di isolare le coppie nella vasca di riproduzione per consentire la digestione del cibo e mantenere alti i livelli di qualità dell'acqua.
- Seleziona coppie di pesci che non sono state accoppiate per l'accoppiamento (almeno) nelle 2 settimane precedenti, per ridurre al minimo l'angoscia e consentire lo sviluppo dei gameti. Acclimatare le coppie di pesci adolescenti adulti selezionate (normalmente con questa composizione: 1 ♂ : 2 ♀ ) in vasche di riproduzione appropriate. Separa le femmine dai maschi usando un divisorio fino al mattino successivo. Garantire un ciclo di luce/buio standard di 14/10 h.
NOTA: È possibile organizzare anche croci di gruppo. Tuttavia, in questo caso, la deposizione delle uova può avvenire in momenti diversi e la selezione degli embrioni nella stessa fase da covate miste diventa più difficile. Il passaggio dal buio alla luce è fondamentale per il successo dell'accoppiamento.
- Il giorno dell'accoppiamento, nutrire regolarmente le coppie adulte secondo il protocollo approvato della struttura animale.
- Raccolta di embrioni e microiniezione di mRNA WT e mutanti (Giorno 4)
- Non appena si accende la luce all'interno dell'impianto, rimuovere il divisorio della vasca che separa i maschi e le femmine e, se necessario, sostituire circa il 20% dell'acqua della vasca con acqua dolce proveniente dal sistema di acquacoltura a ricircolo per mantenere alta la qualità dell'acqua senza diluire eccessivamente gli ormoni rilasciati dai pesci.
- Lasciare il pesce indisturbato e controllare regolarmente la deposizione delle uova (di solito nei primi 30 minuti a 1 ora di luce diurna). Le uova (2-3 mm) vengono deposte e cadono sul fondo della vasca separate da una griglia tale che gli adulti non possono mangiarle. Isolare gli adulti che si sono riprodotti con successo e filtrare l'acqua della vasca contenente le uova attraverso un colino standard (come un colino da tè) per trattenere le uova.
NOTA: È possibile rimettere lo stesso pesce nella vasca di riproduzione per un altro giro di accoppiamento o riportare il pesce nelle vasche di stabulazione originali, registrando la coppia di accoppiamento, la data e le prestazioni. - Lavare gli embrioni raccolti con terreno E3 fresco e rimuovere le uova non fecondate e degenerate, le feci e altri tipi di detriti dalle coppie utilizzando una pipetta Pasteur.
NOTA: Raccogli ~n = 100 uova di adolescenti fecondate in una capsula di Petri di 90 mm di diametro per evitare la sovrappopolazione di embrioni. - Disporre e allineare gli ovuli di adolescenti fecondati in una piastra di microiniezione personalizzata ottenuta modellando agarosio al 2% in terreno E3 per generare corsie appropriate che devono contenere e limitare gli ovuli durante la microiniezione.
NOTA: A questo scopo possono essere utilizzati stampi su misura o commerciali. - Preparare una miscela di microiniezione fresca per generare embrioni mutanti come segue: 30-60 pg di mRNA incappucciato e poliadenilato (qui shp2) disciolto in una soluzione di Danieau 0,3x (diluita dalla soluzione madre) per un volume finale di 20 μL. Aggiungere 0,2 μL (0,05%) di soluzione madre di Rosso Fenolo come tracciante per microiniezione.
- Caricare l'ago con 2 μL di materiale per iniezione utilizzando una pipetta microloader. Iniettare la soluzione nello stadio monocellulare di embrioni di pesce zebra adolescente utilizzando un dispositivo di microiniezione a pressione disponibile in commercio, regolando manualmente le impostazioni di pressione e tempo per calibrare ogni iniezione in base alla qualità dell'ago e dell'embrione. Fare riferimento ai protocolli standard di microiniezione di zebrafish disponibili in letteratura11,12.
- Allevare embrioni adolescenti microiniettati in condizioni di allevamento controllate (temperatura: 28 °C, umidità: 70%, ciclo luce/buio: 14/10 h) per uno sviluppo ottimale e ripulire le uova che appaiono torbide o degenerate nelle successive 3 ore dopo la deposizione.
- A ~4 ore dopo la fecondazione (hpf), esaminare gli embrioni per la fluorescenza (espressione reporter) utilizzando uno stereomicroscopio a fluorescenza standard con lampade e filtri appropriati (465-500 nm). Utilizzare pesci adolescenti positivi per l'imaging FRET e fratelli negativi per le valutazioni immunoistochimiche (IHC) (vedi sotto).
NOTA: Data la nota variabilità nell'espressione dei transgeni13 basati su Tol2, gli embrioni possono mostrare livelli variabili di fluorescenza Teen . Si raccomanda di ispezionare i fratelli non iniettati per controllare la variabilità interindividuale della fluorescenza all'interno di un lotto e, considerando la bassa gamma dinamica dell'imaging FRET, scartare gli embrioni con livelli estremamente bassi di fluorescenza basale.
- Trattamento di embrioni di zebrafish con l'inibitore MEK (MEKi) (Giorno 4)
- Preparare una soluzione intermedia di MEKi PD0325901 (1 mM) diluendo 1 μl di soluzione madre da 10 mM con 9 μl con terreno E3. Utilizzare la soluzione intermedia per ottenere soluzioni finali a basso dosaggio. Preparare una dose bassa (0,25 μM) del PD0325901 MEKi in un volume totale di 3 mL mescolando 0,75 μL della soluzione intermedia 1 mM con 2,25 μL di terreno E3. Diluire 0,75 μl di DMSO al 10% con 2,25 ml di terreno E3 per ottenere la soluzione di controllo (Ctrl).
- Posizionare pool di embrioni di numero uguale in pozzetti diversi di una piastra a 6 pozzetti e iniziare il trattamento mediante immersione nel bagno: per ogni pozzetto, sostituire il terreno E3 con un terreno E3 contenente il veicolo di controllo (0,0025% DMSO) o farmaci diluiti alla concentrazione desiderata (0,25 μM, 0,0025% DMSO) come menzionato in precedenza. Utilizzare 3 ml di soluzione farmacologica per pozzetto.
NOTA: Per eliminare gli effetti causati dalla variazione delle concentrazioni del vettore (DMSO), è importante assicurarsi che sia la soluzione di controllo che quella di trattamento abbiano la stessa concentrazione finale di DMSO. - Conservare gli embrioni trattati a 28 °C prima di raccoglierli fino allo stadio di sviluppo desiderato per i saggi di follow-up (qui: analisi morfometrica e convalida IHC).
2. Imaging FRET multispettrale dal vivo di modelli di zebrafish RASopathy allo stadio di gastrula e analisi dei dati
- Montaggio di gastrule per l'imaging dal vivo (Giorno 4) (Figura 2A)
- Preparare il terreno di montaggio per embrioni vivi sciogliendo 1,5 g di agarosio a basso punto di fusione (LMA) in 1x PBS per ottenere l'1,5% di LMA/E3.
NOTA: Scegliere la concentrazione di agarosio da 0,8 a 1,5% a seconda dello stadio di sviluppo e della necessità specifica di trattenere il pesce. Distribuire la soluzione di LMA appena prodotta in aliquote del formato desiderato e conservare l'LMA allo stato polimerizzato a RT (stabile per alcuni mesi). In questo modo si riduce al minimo la contaminazione batterica e fungina, ma si controlla regolarmente la qualità dell'aliquota LMA prima dell'uso. - Prima di montare il campione, fondere l'aliquota LMA all'1,5% in un termomiscelatore o in un bagno d'acqua sterile a 50 °C. Una volta sciolto, diminuire la temperatura del termomiscelatore a circa 30 °C.
- Posizionare un singolo embrione Teen+ iniettato (in questo caso una gastrula che esprime ShpD61G) al centro di un piatto di vetro con fondo di 35 mm di diametro per l'imaging dal vivo e orientarlo utilizzando un capello sottile. Rimuovere il terreno E3 in eccesso e immobilizzare l'embrione mettendo sopra una goccia di LMA e lasciandolo polimerizzare a RT.
NOTA: All'inizio del processo di polimerizzazione, il pesce può essere orientato nella posizione desiderata. Si consiglia vivamente di utilizzare l'LMA a una temperatura non superiore a 35 °C per evitare danni ai tessuti.
- Preparare il terreno di montaggio per embrioni vivi sciogliendo 1,5 g di agarosio a basso punto di fusione (LMA) in 1x PBS per ottenere l'1,5% di LMA/E3.
- Impostazione dei parametri di microscopia ed esecuzione dell'imaging FRET multispettrale delle gastrule (Giorno 4) (Figura 2B)
NOTA: Il protocollo si applica all'uso di qualsiasi piattaforma di microscopia confocale dotata di tutto l'hardware e il software necessari per eseguire l'imaging FRET. Qui abbiamo utilizzato un microscopio confocale dotato di un laser a ioni argon con le linee di lunghezza d'onda di 458-476-488-496-514 nm, un divisore di fascio acusto-ottico programmabile (AOBS) che separa la luce di eccitazione e di emissione, due rivelatori ibridi spettrali (HyD) e un incubatore da palco per mantenere stabili le condizioni di temperatura (a 28 °C) e umidità durante l'imaging dal vivo dei campioni.
Il protocollo di imaging FRET multispettrale dal vivo, come descritto, può essere applicato a embrioni in diversi stadi di sviluppo oltre alle gastrule precoci, come dimostrato da Fasano et al.7. Uno z-stack più grande dovrebbe essere preso in considerazione per le fasi di sviluppo successive (ad esempio, 24 hpf) così come le impostazioni appropriate degli intervalli z e t per consentire fasi di rilevamento multispettrale durante l'acquisizione di immagini FRET spettrali.- Accendere il controller dell'incubatrice almeno 1 ora prima di iniziare l'acquisizione e impostare la temperatura a 28 °C per mantenere gli embrioni di zebrafish in uno stato sano. Una volta che la temperatura dell'incubatore si è stabilizzata, posizionare la piastra di imaging con l'embrione sul supporto del campione e utilizzare un obiettivo 10x/dry (apertura numerica di 0,4) per visualizzare rapidamente il campione.
- Nella configurazione laser del software di riferimento, accendere il laser agli ioni di argon e utilizzare il cursore per regolare la potenza del laser al 50%. In Impostazioni hardware, scegli la risoluzione di profondità a 8 bit con cui verranno acquisite le immagini.
- Selezionare la modalità di scansione XYλZ di rilevamento spettrale nel pannello di acquisizione e impostare i seguenti parametri di acquisizione: formato immagine: 512 x 512 px; velocità di scansione di 400 Hz; zoom ottico di 0,75.
- Attivare la linea laser a 458 nm del laser agli ioni di argon e impostarne il valore di intensità, normalmente <10% (qui 8,5%).
- Selezionare un rivelatore HyD e regolare la sensibilità del rivelatore (guadagno) inserendo il valore desiderato (qui 500).
NOTA: Per campioni vivi, debolmente fluorescenti e per accumulare meno rumore di fondo, è consigliabile utilizzare rivelatori ibridi piuttosto che tubi fotomoltiplicatori (PMT).
Nel software di riferimento, la visualizzazione dello spettro di emissione di un colorante richiede l'attivazione di un rivelatore. - Per iniziare, attivare il primo rivelatore (HyD) e selezionare la proteina di fluorescenza ciano (CFP) per visualizzare la sua curva di emissione. Aprire il menu a tendina della barra del rivelatore del software per aprire l'elenco di selezione per la curva di emissione CFP. Ora, attivare un secondo rivelatore per visualizzare la curva di emissione della seconda proteina di fluorescenza gialla colorante (YFP). Per visualizzare anche la curva di emissione Ypet , attivare un secondo rivelatore, quindi selezionare la curva di emissione YFP che apparirà nell'immagine dello spettro, dopo aver spento il secondo rivelatore.
NOTA: Una volta completato questo passaggio, il secondo rivelatore deve essere disattivato, poiché in modalità di acquisizione spettrale viene utilizzato un solo rivelatore. - Per avviare le acquisizioni in tempo reale, posizionare il cursore di rilevamento nell'intervallo di emissione del segnale più intenso (in questo caso, quello di YFP) per visualizzare il campione, decidere e impostare le posizioni di inizio e fine dello spessore del campione nella finestra z-stack LAS X.
- Per le proprietà dell'intervallo di scansione λ, impostare i seguenti parametri: inizio (460 nm) e fine (570 nm) dell'intervallo di rilevamento; Larghezza della banda di rilevamento: 5 nm; λ-scan Dimensione del passo: 5 nm. Avvia l'acquisizione z-stack.
NOTA: I parametri indicati consentono agli utenti di ottenere un'immagine relativamente veloce con una risoluzione accettabile, ma è possibile utilizzare impostazioni diverse. Nelle impostazioni del disco del fluorifier, il filtro NOTCH inserito automaticamente può essere deselezionato per evitare la perdita di intensità singola. - Per valutare i cambiamenti del segnale in tempo reale dopo l'esposizione a MEKi, montare un singolo embrione mutante (qui Shp2D61G) in un piatto di vetro e creare un'immagine prima e dopo il trattamento farmacologico. Per questi esperimenti dal vivo, eseguire direttamente il trattamento farmacologico mediante immersione in bagno durante la sessione di imaging con un massimo di 2 ml della soluzione contenente il farmaco disciolto.
- Post-elaborazione delle immagini e separazione multispettrale dei coloranti per ottenere immagini FRET raziometriche (Giorno 5) (Figura 2C)
- Per separare l'emissione del donatore (CFP) e dell'accettore (Ypet), assicurarsi di eliminare il contributo dell'emissione di fluorescenza di entrambe le molecole nel canale FRET, chiamato bleedthrough spettrale (SBT).
- Fare riferimento ai database di coloranti/proteine fluorescenti disponibili al pubblico per visualizzare e scaricare il file .cvs relativo a uno spettro di emissione CFP standard (spettri di eccitazione ed emissione).
- Nel pannello Dati del file .cvs, scegli l'opzione Testo in colonne e suddividi i dati in colonne utilizzando una virgola come delimitatore. Il file risultante includerà informazioni aggiuntive (ad esempio, eccitazione CFP, CFP 2P) che dovrebbero essere rimosse. Conservare solo i dati della colonna relativi alla lunghezza d'onda e all'emissione.
- Nella colonna delle emissioni CFP, rimuovere tutti i dati sull'intensità a partire da 501 nm.
- Salvare il file dello spettro di emissione CFP adattato da 460 a 500 nm in formato .xls (denominato "eCFP modificato" nella Figura 2C).
- Ripetere la stessa procedura per scaricare ed elaborare lo spettro di emissione della proteina YFP e conservare solo i dati della colonna relativi alla lunghezza d'onda e all'emissione. Rimuovere tutti i valori di emissione fino a 524 nm. Salvare il file dello spettro di emissione YFP adattato da 525 a 650 nm in.xls formato (chiamato "Ypet da 525 nm" nella Figura 2C).
- Per consentire l'esclusione del bleed-through spettrale (SBT) nel database dei coloranti disponibile nella finestra di configurazione del software, inserire e salvare due spettri di emissione di riferimento adattati per CFP e YFP.
- Selezionare il file di immagine spettrale risultante dalla sessione di imaging, aprire la finestra Processo e selezionare Separazione colorante spettrale nello strumento Separazione colorante. Configurare le impostazioni per la separazione dei coloranti come segue: negli elenchi a discesa sul lato sinistro della finestra di dialogo, selezionare il nuovo spettro di emissione CFP (eCFP modificato) in prima posizione e il nuovo spettro di emissione YFP (Ypet da 525 nm) dal database dello spettro.
- In Riscala, selezionare Per canale per scalare i canali singolarmente quindi, nella scansione λ delle immagini, scegliere quello con la maggiore intensità del segnale (corrispondente al passo 14 della scansione spettrale a livello del picco di emissione del canale FRET, indicato dalla freccia bianca nel pannello che mostra i passi di rilevamento a destra della Figura 2B). Quindi, spostarsi lungo la scansione Z del campione e scegliere la sezione ottica che evidenzia l'area di interesse sulla zona del margine.
- Utilizzare la modalità di selezione del ROI sulla zona di margine del palo dell'animale per definire l'area con lo spettro migliore. Cliccare su ROICrosshair indicato in alto nella finestra di visualizzazione per richiamare il mirino e regolare la dimensione del ROI di riferimento inserendo il valore di 40 voxel nell'Area Misure. Definisci con precisione l'area di interesse dal ROICrosshair. Nel diagramma immagine, scegliere la normalizzazione dei dati, quindi fare clic su Applica per eseguire la separazione dei coloranti con le impostazioni configurate.
- Apri questo file appena generato ottenuto con i due canali separati e produci un'immagine di proiezione bidimensionale dalla serie di immagini tridimensionali (proiezione di massima intensità) per la visualizzazione dei dati.
- Nella finestra Processo , selezionare Ritaglia per separare i canali in due file separati, i canali CH1 e CH2 (o FRET).
- Nella finestra Processo , seleziona Combina immagini, seleziona il file CH2 e inseriscilo nella prima opzione, quindi seleziona il file CH1 e inseriscilo nella seconda opzione. Impostare una riscala con fattore 5 e scegliere l'operazione Rapporto. Quindi, fare clic su Applica per generare il nuovo file contenente l'immagine raziometrica (YFP/CFP). Salvare il file per l'analisi dei dati di follow-up.
- Per separare l'emissione del donatore (CFP) e dell'accettore (Ypet), assicurarsi di eliminare il contributo dell'emissione di fluorescenza di entrambe le molecole nel canale FRET, chiamato bleedthrough spettrale (SBT).
- Analisi quantitativa dei segnali FRET e resa delle immagini (Giorni 5-6) (Figura 2D)
- Per misure quantitative dei segnali FRET da immagini raziometriche FRET/CFP, eseguire l'esperimento su N>2 embrioni (repliche), sulla base di una valutazione statistica a priori basata sull'effetto atteso. Utilizza un pacchetto di elaborazione delle immagini come il software open source Fiji. Importa i file di immagine ottenuti dall'imaging spettrale e dalla separazione dei coloranti in un pacchetto di analisi delle immagini come l'open source Fiji.
- Prima di iniziare con la selezione della ROI sulle immagini delle gastrule, impostare i parametri come letture delle misure utilizzando la funzione Analizza | Imposta le misure | selezionare i parametri di interesse, ad esempio Area, Densità integrata e Valore medio di grigio.
- Selezionare la regione di interesse (in questo studio specifico: il margine della gastrula) utilizzando lo strumento di selezione dei poligoni dalla barra degli strumenti. Salva le specifiche ROI x,y facendo clic su Analizza | Strumenti | Responsabile del ROI.
- Per eseguire misurazioni in un ROI selezionato, fare clic su Analizza | Misurare. Ripeti questi passaggi per ogni ROI e immagine/condizione.
NOTA: Se necessario, salvare l'immagine come . Formato TIFF. È possibile salvare tutte le misure di ROI derivanti da più immagini in un unico file ROI. Rinomina ogni misura nell'elenco di gestione ROI con il nome corretto relativo a ciascun campione/immagine. - Per ogni condizione (mutante e mutante + trattamento qui), estrarre tutti i valori del rapporto FRET/CFP per ogni ROI selezionato ottenuto e organizzarli in un foglio di lavoro organizzando, ad esempio, i gruppi sperimentali in colonne e ogni valore grezzo in righe.
- Utilizzare qualsiasi software adatto per valutare le differenze statistiche dei segnali FRET tra diverse condizioni sperimentali e per la generazione di grafici e la visualizzazione dei dati.
NOTA: Sono disponibili software open source e con licenza per l'analisi dei dati e le soluzioni grafiche. - Organizzare un progetto specifico per la valutazione statistica considerando tutte le misure grezze e il numero di repliche per ogni condizione sperimentale. Per l'analisi statistica di una singola replica biologica, creare una tabella di colonne con una variabile di raggruppamento, con ogni gruppo definito da una colonna. Per l'analisi statistica di più di una replica biologica, creare tabelle raggruppate con almeno due variabili di raggruppamento, una definita da colonne (ad esempio, condizioni sperimentali) e l'altra definita da righe (ad esempio, valori medi delle repliche).
- Dopo aver organizzato in modo appropriato i gruppi sperimentali nel foglio di lavoro, valutare la distribuzione dei dati (test di normalità, ad esempio, D'Agostino-Pearson, Anderson-Darling) e, in base al risultato, scegliere il test statistico più appropriato.
- Eseguire un'ANOVA unidirezionale per i dati parametrici (seguendo la distribuzione gaussiana) e il test di Kruskal-Wallis per i dati non parametrici. Se sono presenti più fattori (concentrazioni genetiche e di farmaci), prendere in considerazione un'ANOVA a due vie. Confrontare il valore FRET medio per ciascuna condizione l'una con l'altra (confronti multipli) e correggere i confronti multipli utilizzando un test post hoc (ad esempio, il test di Tukey e quello di Dunn per dati parametrici e non parametrici, rispettivamente).
3. Validazione IHC dei risultati FRET e analisi morfometrica correlativa dei difetti di gastrulazione
- Preparazione delle soluzioni e fissazione e montaggio dell'embrione per IHC contro tERK e pERK (Giorno 7) (Figura 3A)
- Preparare le soluzioni funzionanti necessarie per IHC.
- Preparare una soluzione madre di 10x PBS sciogliendo 80 g di NaCl, 2 g di KCl, 14,4 g di Na2HPO4 e 2,4 g di KH2PO4 in 1 L di acqua DD. Autoclavare la soluzione in autoclave per mantenerla sterile.
- Preparare una soluzione di PBS-Triton allo 0,8% (PBSTr) sciogliendo 8 mL di Triton X-100 al 10% in un volume finale di 100 mL di 1x PBS.
- Preparare soluzioni di glicerolo al 20%, 50% e 80% diluendo rispettivamente 3 ml, 7,5 ml e 12 ml di glicerolo al 100% in un volume finale di 15 ml di 1x PBS.
- Preparare paraformaldeide al 4% (PFA)/PBSTr allo 0,25% (soluzione fissativa) mescolando 2,5 mL di PFA al 16% in 250 μL di Triton X-100 al 10% precedentemente disciolto in un volume finale di 10 mL di 1x PBS. Utilizzare questa soluzione per fissare gli embrioni a 6 hpf durante la notte a 4 °C. Utilizzare provette da 2 ml e un massimo di cinque embrioni per provetta per consentire una fissazione e un lavaggio efficienti.
NOTA: Si consiglia di preparare un nuovo fissativo per ogni giro di fissaggio. ATTENZIONE: Quando si maneggia la formaldeide al 16%, indossare dispositivi di protezione individuale (DPI) e sotto un cappuccio chimico. - Lavare gli embrioni nelle provette da 2 mL riempiendole con 1,5 mL di PBSTr allo 0,8% per alcune volte. Trasferire gli embrioni in nuove provette da 2 ml e conservare gli embrioni in 1x PBS fino al momento dell'uso.
NOTA: Se non si ha familiarità con la manipolazione degli embrioni precoci di zebrafish, lavorare al microscopio stereoscopico per evitare la perdita di embrioni durante il pipettaggio. Utilizzare materiale plastico "a basso legante" per ridurre la possibile appiccicosità degli embrioni alla parete del tubo.
- Preparare le soluzioni funzionanti necessarie per IHC.
- Permeabilizzazione tissutale e IHC contro pERK e tERK (giorni 7-9)
NOTA: I risultati dell'IHC dipendono da più variabili che possono differire tra i laboratori e potrebbe essere necessaria un'ottimizzazione specifica dei protocolli.- Lavare gli embrioni di zebrafish fissati in 1 mL di PBSTr allo 0,8% per 3 x 10 minuti a RT.
- Permeabilizzare i campioni con 2 μl di proteinasi K diluita in PBSTr allo 0,8% (1:1.000) per 2 minuti a RT.
- Arrestare la permeabilizzazione del tessuto lavando i campioni in 1 mL di PBSTr allo 0,8% per 3 x 10 minuti e postfissare i campioni in 500 μL di PFA al 4%/1x PBS per 20 minuti a RT. Quindi, lavare i campioni in 1 mL di PBSTr allo 0,8% per 3 x 10 minuti.
NOTA: Il metodo e il tempo utilizzato per la permeabilizzazione dei tessuti sono passaggi critici che possono essere influenzati dal tipo di antigene, dalla qualità della conservazione dei tessuti e dai fattori ambientali. Ottimizza il protocollo prima di applicarlo su campioni preziosi. - Incubare i campioni con 1 tampone bloccante (BB) contenente il 5% di siero di capra normale (NGS), l'1% di albumina sierica bovina (BSA), l'1% di DMSO, preparati come segue: 75 μL di 100% NGS, 150 μL di 10% BSA e 15 μL di DMSO al 100% in un volume finale di 1,5 mL di 0,8% PBSTr. Utilizzare 200 μL di soluzione per provetta e incubare gli embrioni per 20-30 minuti a RT nella soluzione.
- Rimuovere 1x BB dal passaggio precedente e incubare i campioni in una soluzione contenente una miscela degli anticorpi primari desiderati (qui: diluendo 1 μL di anticorpo monoclonale primario di topo p44/42 MAPK, ERK totale, tERK, + 1 μL di fosfo-p44/42 MAPK policlonale di coniglio, ERK fosforilato, pERK) in 250 μL di soluzione 1x BB appena preparata. Utilizzare 200 μl di soluzione per provetta e incubare gli embrioni nella soluzione per una notte a 20 °C.
- Per eliminare il legame aspecifico, lavare i campioni con 1 mL di PBSTr allo 0,8% più volte (prima lavaggio ogni 10 minuti e poi ogni 30 minuti).
- Incubare i campioni in 1 BB appena preparato per 20 minuti a RT. Utilizzare 200 μl per provetta.
- Rimuovere 1x BB dal passaggio precedente e incubare i campioni con una miscela di anticorpi secondari (1 μL di anti-topo di capra coniugato fluorescente 488, + 1 μL di anti-coniglio di capra 633 coniugato fluorescente in 600 μL di soluzione di BB 1x). Lasciare gli embrioni agitati delicatamente per una notte a 20 °C.
NOTA: Abbiamo eseguito l'IHC su pesci adolescenti negativi (non transgenici) dello stesso lotto utilizzato per l'imaging FRET. In alternativa, è possibile utilizzare anche il pesce Teen+ , ma in questo caso dovrebbero essere impiegati anticorpi secondari coniugati a fluorofori che non si sovrappongono agli spettri di emissione di CFP o YFP. - Eliminare l'associazione non specifica come descritto in precedenza.
- Lavare i campioni in un gradiente di soluzioni di glicerolo/1x PBS (20%, 50%, 80%) per 15 minuti per ciascuna soluzione a RT. Conservare i campioni in glicerolo al 90%/1x PBS a 4 °C.
NOTA: Per ridurre al minimo il possibile decadimento della fluorescenza, conservare i campioni al buio.
- Microscopia confocale di tessuto immunocolorato e analisi quantitativa (Giorni 10-11) (Figura 3B-E)
- Monta gli embrioni come descritto in precedenza.
- Impostare i parametri di acquisizione al microscopio confocale come segue: laser bianco all'80%, intervallo di emissione della fluorescenza a 507 - 551 nm per anti-topo di capra coniugato fluorescentmente (in questo caso con lo spettro di emissione di 488 nm, utilizzato per tERK), 644 - 740 nm per capra coniugata fluorescentmente anti-coniglio (in questo caso con lo spettro di emissione di 633 nm, utilizzato per pERK). Seleziona la modalità di acquisizione sequenziale, un formato di scansione di 512 x 512 px e una velocità di 400 Hz. Definisci l'inizio e la fine della scansione con uno spessore della dimensione z-step di 4 - 5 μm e uno zoom digitale standard di 1. Utilizzare l'obiettivo 25x ad immersione in acqua (con apertura numerica di 0,05) per acquisire l'intera gastrula a 6 hpf
ATTENZIONE: Questo protocollo include l'applicazione di laser che possono essere dannosi; Pertanto, richiede la formazione del personale secondo i requisiti nazionali specifici. - Dopo l'acquisizione dell'immagine confocale, ispezionare il file di immagine grezzo ottenuto utilizzando un pacchetto di elaborazione delle immagini come il software open source Fiji.
- Nelle Figi, utilizzare il comando Dividi canali dal menu Immagine e dal sottomenu Colore per ottenere un'immagine di un singolo canale (totale ERK: canale = 0, C = 0; pERK: canale = 1, C = 1) e generare un'immagine di proiezione z di massima intensità per entrambi i canali facendo clic su Immagine | Pile | Progetto Z.
- Ripetere la procedura per le misurazioni dell'intensità del ROI come descritto in precedenza ed estrarre i dati in un foglio di lavoro.
- Per dedurre le variazioni di intensità del segnale p-ERK nel ROI desiderato tra i gruppi sperimentali, calcolare il rapporto p-ERK/t-ERK dividendo i valori grezzi di densità integrata della colorazione p-ERK per la densità grezza integrata del segnale t-ERK. Procedere alla valutazione della significatività statistica come al punto 2.4.
NOTA: Le impostazioni dei parametri di imaging possono essere modificate in base alle esigenze specifiche e considerando la durata di ciascun campione, la qualità delle immagini desiderata e il numero di piani da acquisire.
- Misure degli assi in 11 embrioni hpf (Giorno 4 per la fissazione dell'embrione, Giorno 12 per l'acquisizione e l'analisi dei dati) (Figura 4A-D)
- Al termine della gastrulazione (11 hpf), fissare gli embrioni con PFA al 4% in 1x PBS per 20 minuti a RT, lavare più volte in 1x PBS e conservare a 4 °C fino all'acquisizione dell'immagine.
- Orientare gli embrioni lateralmente utilizzando una pipetta Pasteur in un singolo pozzetto di una piastra a 12 pozzetti contenente 1x PBS fresco.
- Per valutare la presenza di una forma ovale (rapporto assi embrionali Y/X > 1), un fenotipo caratteristico della rasopatia nel pesce zebra e il rapporto di salvataggio morfologico degli assi embrionali come risultato dell'efficacia del trattamento, acquisire immagini di embrioni interi utilizzando uno stereomicroscopio con un obiettivo di ingrandimento 3,4x (obiettivo di scansione 0,63x x fattore di zoom 8,60) semplicemente catturando immagini in modalità campo chiaro.
- Importa il file immagine in un pacchetto di analisi delle immagini come l'open source Fiji.
- Misura la lunghezza degli assi degli embrioni (x, asse minore; y, asse maggiore) selezionando lo strumento dritto dalla barra degli strumenti e facendo clic su Analizza | Misurare. Dopo aver eseguito le misurazioni selezionate, aggiungerle all'elenco del ROI manager e salvare il file ROI come spiegato sopra per l'analisi dell'imaging FRET.
NOTA: Ripetere questi passaggi per ogni immagine dell'embrione.
- Misura la lunghezza degli assi degli embrioni (x, asse minore; y, asse maggiore) selezionando lo strumento dritto dalla barra degli strumenti e facendo clic su Analizza | Misurare. Dopo aver eseguito le misurazioni selezionate, aggiungerle all'elenco del ROI manager e salvare il file ROI come spiegato sopra per l'analisi dell'imaging FRET.
- Procedi con l'esportazione dei dati in un foglio di lavoro e calcola il rapporto degli assi dividendo la lunghezza dell'asse maggiore (y) per la lunghezza dell'asse minore (x). Calcola la media, la deviazione standard della media o l'errore standard della media di diverse repliche.
- Procedere all'analisi statistica dei dati come al punto 2.4.
Risultati
Questo protocollo mostra un flusso di lavoro semplice per generare rapidamente modelli di RASopathy transitori in embrioni di zebrafish e valutare le fluttuazioni ERK nei mutanti precoci con un metodo di imaging FRET live standard applicato a un sensore ERK zebrafish di recente costituzione 6,9. Come recentemente dimostrato 6,7 all'interno dello stesso flusso di lavoro sperimentale, i risultati FRET possono essere convalidati in modo incrociato con IHC standard con ERK fosforilato e totale su embrioni interi. La compromissione della formazione degli assi corporei correlata all'aumento dell'attivazione di ERK può essere seguita in fasi successive, anche negli stessi embrioni utilizzati per FRET7. Qui vengono mostrate le tipiche fluttuazioni di ERK nelle gastrule precoci registrate da FRET, che sono legate all'espressione di Shp2D61G che causa NS. Sono stati acquisiti anche dati da mutanti in cui il segnale è stato modulato negativamente dal trattamento a basse dosi con un MEKi, inibendo la cascata di segnalazione RAS/MAPK.
Per generare modelli NS, abbiamo prima prodotto mRNA poliadenilato e poliadenilato di alta qualità che codifica Shp2 (forma wild-type e mutante) mediante trascrizione in vitro da un opportuno plasmide linearizzato che ospita shp2 full-length CDS. Come mostrato nella Figura 1A, quando la maggior parte del plasmide viene linearizzata con successo ("tagliata"), si può osservare una banda netta che scorre in un gel di agarosio standard all'1-1,5% (qui nell'esempio della dimensione di 7.500 bp) mentre il DNA "non tagliato", usato qui come controllo del plasmide non digerito, mostra diverse possibili conformazioni, tipiche del DNA circolare non digerito. L'mRNA incappucciato e poliadenilato può quindi essere prodotto in vitro. Sul gel di agarosio contenente formammide, l'RNA intatto dovrebbe apparire come una banda cospicua intorno alle dimensioni previste (Figura 1A, a destra) senza strisci evidenti (indicativi di degradazione dell'mRNA) o presenza di frammenti più piccoli. Si consiglia di non procedere se si ottiene RNA frammentato o degradato. La preparazione deve essere ripetuta durante la risoluzione dei problemi. Come mostrato negli schemi della Figura 1B, l'mRNA poliadenilato e poliadenilato di buona qualità che codifica per l'allele RASopathy desiderato e un controllo WT (nel nostro caso Shp2D61G e Shp2WT come controllo) possono quindi essere iniettati in embrioni di zebrafish a stadio unicellulare che esprimono il Teen reporter. Gli embrioni vengono quindi allevati fino allo stadio desiderato (in questo caso le prime fasi durante la gastrulazione).
La Figura 2 illustra gli schemi e i risultati rappresentativi del flusso di lavoro utilizzato per registrare i segnali FRET nel nostro modello di RASopathy in vivo. Le gastrule teen+ che esprimono l'allele NS mutante Shp2D61G sollevate e raccolte allo stadio desiderato (4 hpf) sono state montate in agar a basso punto di fusione (LMA) all'1,5% in terreno E3 (Figura 2A). Un esempio delle impostazioni generali del microscopio e delle impostazioni di acquisizione dello stack lambda (λ) che abbiamo utilizzato per ottenere l'imaging FRET multispettrale dall'eccitazione del donatore (405 nm) negli embrioni adolescenti è mostrato nella Figura 2B. È importante sottolineare che, per garantire una raccolta sufficiente di spettri, abbiamo impostato la larghezza di banda λ a 5 nm. Abbiamo utilizzato un passo relativamente alto (qui 8 nm) per ottenere scansioni x, y, λ, z in un tempo ragionevole, considerando la modalità di acquisizione in tempo reale (acquisizioni con intervallo di tempo di circa 13 minuti).
Dopo l'acquisizione dell'immagine, l'intensità del segnale registrato è stata ispezionata lungo l'intero spettro λ per assegnare le migliori finestre di emissione e procedere all'unmixing degli spettri CFP (D) e Ypet (A) (Figura 2C, a sinistra). Qui, ispezionando la regione del margine del polo animale dell'embrione con un approccio basato sul ROI integrato nel software Spectral dye separation wizard, ci siamo concentrati sull'assegnazione delle migliori finestre spettrali di emissione per i due fluorofori, evitando qualsiasi sovrapposizione spettrale tra le molecole D e A. I singoli canali grezzi rappresentativi (CFP o Ypet, verde e rosso, rispettivamente) ottenuti con questo approccio e l'immagine raziometrica FRET/CFP (grigio) risultante dall'applicazione della funzione Rapporto sono mostrati nella Figura 2C, pannello in alto a destra. Il pannello inferiore mostra un risultato rappresentativo non ottimale da embrioni reporter adolescenti , che occasionalmente mostrano un'intensità del segnale insufficiente, come spesso osservato all'interno di una popolazione di pesci transgenici. Questi embrioni devono essere scartati in anticipo e si deve prestare attenzione a valutare gli embrioni con livelli simili di espressione di Teen nell'analisi.
Dopo l'ispezione del segnale al margine del palo animale, dove l'attività di ERK è concentrata durante i movimenti delle cellule di epibolia e gastrulazione, il rendering delle immagini è stato eseguito utilizzando la pseudocolorazione LUT "intelligente" nelle Fiji. Ciò consente una migliore visualizzazione delle differenze di intensità del segnale spaziale sull'immagine raziometrica risultante. Le immagini FRET raziometriche risultanti nella Figura 2D mostrano già una chiara tendenza alla riduzione dell'intensità del segnale in Shp2D61G nella regione del margine dopo circa 13 minuti di trattamento con il MEKi selezionato, PD0325901 (qui PD) a basse dosi (0,25 μM)7. I dati dimostrano l'idoneità del sensore Teen e del protocollo di imaging FRET multispettrale utilizzato qui per rilevare le variazioni dinamiche di ERK in tempo reale in un modello di RASopathy durante le prime fasi di sviluppo, nonché la modulazione fine del segnale associato alla malattia dopo il trattamento con MEKi a basso dosaggio.
Parallelamente, abbiamo convalidato i risultati del nostro approccio FRET applicato a modelli di pesci RASopathy mediante IHC a fluorescenza rispetto a ERK totale (t-ERK) e alla sua forma fosforilata (p-ERK). Normalizzando il segnale ottenuto (p-ERK/t-ERK) all'interno di un ROI, emergono cellule con alti livelli di ERK. La Figura 3 mostra un esempio di convalida dell'attività di ERK tramite IHC per Shp2WT, Shp2D61G e Shp2D61G trattati con PD a basso dosaggio da 4 hpf a 6 hpf (trattamento più lungo rispetto alla finestra di trattamento di 13 minuti mostrata durante l'imaging FRET dal vivo). L'immunocolorazione è stata eseguita utilizzando anticorpi secondari fluorescenti con spettri di emissione non sovrapposti per t-ERK e p-ERK (488 e 633 nm, rispettivamente).
I 6 embrioni hpf sono stati montati in LMA all'1,5% disciolto in PBS e sono state impostate impostazioni confocali standard per ottenere scansioni x, y, z dell'intero volume dell'embrione (Figura 3A, B). Le impostazioni confocali standard con risoluzione di 512 x 512 px a 400 Hz ci consentono di ispezionare la localizzazione del segnale, importante per determinare il buon esito dell'immunofluorescenza prima della quantificazione del segnale. Come previsto, e come mostrato nel risultato rappresentativo, per t-ERK (verde), abbiamo osservato una colorazione quasi ubiquitaria all'interno delle cellule del margine, mentre il segnale p-ERK (rosso) era per lo più confinato ai nuclei (Figura 3C). Le immagini grezze, come immagini a canale singolo e unite, sono state elaborate inizialmente eseguendo una proiezione z-stack per ottenere immagini dell'intero embrione. La pseudocolorazione di p-ERK (ciliegia) è stata applicata per risolvere meglio il contrasto con t-ERK (verde) (Figura 3D). Rispetto a Shp2WT, in Shp2D61G si è potuto osservare un aumento del numero di nuclei p-ERK+ all'interno della regione del margine, che è stato recuperato negli embrioni Shp2D61G trattati da 4 hpf a 6 hpf con 0,25 μM PD. I dati confermano i risultati ottenuti dall'imaging FRET e dimostrano l'efficace modulazione molecolare ottenuta dal MEKi a basso dosaggio di scelta. Analogamente alle immagini FRET, il segnale proveniente dalle acquisizioni z-stack a canale singolo è stato quindi ispezionato nella regione del margine del polo animale utilizzando l'analisi basata sul ROI utilizzando le Fiji, e i valori p-ERK e t-ERK sono stati misurati separatamente ed estratti per la normalizzazione del follow-up (p-ERK/t-ERK) e l'analisi statistica (Figura 3E).
Infine, la Figura 4 fornisce dati morfometrici rappresentativi che possono essere ottenuti e misurati, idealmente sugli stessi campioni provenienti dall'imaging FRET dal vivo e successivamente aumentati da 6 a 11/12 hpf o sui loro fratelli. Misure alterate degli assi minore e maggiore (rapporto assi maggiore/minore) in questa fase embrionale possono infatti evidenziare difetti dei movimenti di gastrulazione associati alla RASopathy. I dati possono essere utilizzati per esaminare l'esito fenotipico di nuove varianti scoperte in pazienti con patogenicità incerta e per correlare la forza del fenotipo con quella dell'alterazione molecolare (attivazione di ERK) misurata con FRET. Nell'esempio qui, i fratelli degli embrioni adolescenti (gruppo di controllo 1: Shp2WT e gruppi 2 e 3: Shp2D61G e Shp2D61G trattati con PD a basso dosaggio tra 4 e 6 hpf) sono stati allevati separatamente fino a 11 hpf e poi fissati in PFA al 4% (Figura 4A) per evitare un ulteriore sviluppo embrionale che potrebbe interferire con il confronto dei dati tra gli individui. Le immagini degli embrioni laterali sono state acquisite utilizzando una semplice modalità di campo chiaro disponibile per le impostazioni standard. L'allungamento dell'embrione è stato valutato in termini di misurazioni degli assi maggiori e minori (Figura 4B, C). Le misure dei risultati (Figura 4D) riflettono e corroborano i dati ottenuti con l'imaging FRET e dimostrano una correlazione tra l'attività dinamica di ERK al margine del polo animale e gli esiti della gastrulazione. Nel complesso, il set di dati rappresentativo mostrato qui per l'allele NS-associato shp2D61G con o senza trattamento MEKi PD0325901 mostra l'utilità dell'imaging FRET multispettrale nei pesci reporter Tg[ef1α:ERK biosensor-nes] (Teen) per prevedere la patogenicità delle varianti per NS nell'embrione precoce.

Figura 1: Analisi dei preparati di plasmidi e mRNA necessari per generare modelli transitori di NS zebrafish. (A) Il pannello mostra i risultati schematici e rappresentativi dell'esito dell'elettroforesi del DNA visualizzato da un transilluminatore UV, utilizzato per valutare l'efficiente linearizzazione dei plasmidi e la trascrizione di buona qualità dell'mRNA. I plasmidi linearizzati ("cut") sono ottenuti per digestione con un enzima di restrizione (in questo caso KpnI) e confrontati con il plasmide di controllo non digerito ("uncut"). Una singola banda che corre più veloce di quella "non tagliata" dovrebbe essere idealmente osservata dopo la linearizzazione plasmidica. Dimensioni del plasmide: ~ 7.500 bps: (plasmide dorsale + shp2 CDS). Si noti come il plasmide circolare "non tagliato" mostri diverse forme di DNA di molecole rilassate e superavvolte. Una scala da 1 kb (corsia 1), i cui frammenti di peso molecolare variavano da 0,5 a 10 kilobasi (kb), è stata caricata sullo stesso gel per l'ispezione delle dimensioni dei frammenti di DNA. A destra, è mostrata una serie rappresentativa di mRNA appena trascritti (in questo esempio, mRNA trascritti che codificano per SPRED2 umano associato alla rasopathy). Due scale che funzionano in modo leggermente diverso (scala del DNA, corsia 1 e scala dell'RNA, corsia 2) con intervallo molecolare simile da 0,5 a 10 kb vengono caricate per l'ispezione delle dimensioni dei frammenti di RNA. (B) Panoramica schematica che illustra l'uso degli mRNA trascritti che codificano varianti associate alla RASopathy per generare modelli di malattia transitoria mediante iniezione in embrioni di zebrafish in uno stadio cellulare. L'mRNA che codifica per la forma wild-type della proteina di interesse viene iniettato come controllo. La miscela di mRNA viene preparata nella soluzione di Deneau con il colorante vitale Phenol Red. Gli embrioni vengono allevati e valutati al microscopio a 6 hpf (per i test FRET e IHC) o a 11/12 hpf (per gli assi corporei). Abbreviazioni: NS = sindrome di Noonan; CDS = sequenza di codifica; FRET = Trasferimento di energia per risonanza di fluorescenza; IHC = immunoistochimica ; HPF = ore dopo la fecondazione . Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Flusso di lavoro e risultati rappresentativi dell'imaging FRET multispettrale dal vivo in embrioni Teen Shp2D61G precoci prima e dopo il trattamento con MEKi. (A) Panoramica delle fasi necessarie per l'imaging FRET spettrale in tempo reale in campioni di zebrafish. Gli embrioni allevati e stadiati in terreno E3 sono stati montati in una capsula con fondo di vetro in LMA all'1,5% per l'acquisizione di FRET spettrale dal vivo. L'imaging FRET viene eseguito prima e dopo la somministrazione di PD0325901 a basso dosaggio. Viene mostrato uno schema del sistema Teen reporter sviluppato da Sari et al.9. Le immagini raziometriche (FRET/CFP) sono ottenute in post-elaborazione. (B) Dettagli delle impostazioni di acquisizione del microscopio e dell'imaging multispettrale nel software LAS X per entrambi i fluorofori (CFP, D; Ypet, A). (C) Comandi e metodo per ottenere la separazione spettrale del colorante e immagini raziometriche applicate a una gastrula Shp2D61GTeen . A sinistra, un segnale forte dal margine del polo animale è stato selezionato per la separazione spettrale con uno strumento di selezione del ROI. In alto a destra, vengono mostrate le impostazioni e i risultati rappresentativi delle immagini risultanti per entrambi i canali D e A come proiezioni z di massima intensità da un singolo pesce, nonché le impostazioni per ottenere l'immagine raziometrica e i risultati rappresentativi. Viene indicata la regione di margine al polo dell'embrione animale con segnale FRET alto (linea gialla tratteggiata). (D) Pannello di sinistra: schemi della selezione del ROI sul margine (linea gialla tratteggiata) dall'immagine raziometrica grezza per l'analisi dei dati nelle Fiji. Immagini FRET/CFP rappresentative generate tramite il plug-in LUT "intelligente" delle Fiji mostrano di visualizzare i cambiamenti nell'attivazione di ERK nell'embrione che esprime Shp2D61G rispetto a Shp2WT (pannello centrale) o nello stesso embrione Shp2D61G prima e dopo il trattamento PD (pannello di destra). Per questo esperimento, vengono mostrati i valori grezzi di densità di intensità del ROI selezionato. Viene visualizzata la barra della scala. Abbreviazioni: FRET = Trasferimento di energia di risonanza a fluorescenza; MEKi = Inibitore degli enzimi proteina chinasi attivata da mitogeno; LMA = agarosio a basso punto di fusione; CFP = proteina di fluorescenza ciano ; Ypet = proteina gialla a fluorescenza; D = donatore; A = accettore; ROI = regione di interesse; ERK = chinasi regolata dal segnale extracellulare. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Flusso di lavoro e risultati rappresentativi dell'IHC contro t-ERK e p-ERK in embrioni precoci Shp2D61G con o senza trattamento con MEKi. (A) Schemi che illustrano la raccolta di campioni di zebrafish immunocolorati contro t-ERK (488) e p-ERK (633) e montati in LMA/PBS all'1,5% per acquisizioni confocali z-stack. (B) Impostazioni del microscopio confocale per due linee laser (488, 633 nm), utilizzate per catturare in sequenza i segnali fluorescenti derivanti dagli anticorpi secondari coniugati fluorescente. Il riquadro illustra i parametri Z-stack per l'acquisizione di immagini raw multistack con risoluzione accettabile. (C) Schemi (pannello di sinistra) e immagini confocali grezze rappresentative (pannello di destra) di gastrule di pesce zebra correttamente colorate a 6 hpf che mostrano la localizzazione ubiquitaria e per lo più nucleare di t-ERK (verde) e p-ERK (rosso), rispettivamente, al margine del polo animale. (D) Proiezioni z rappresentative di massima intensità da pile confocali di embrioni da 6 hpf che esprimono Shp2WT (controllo), Shp2D61G e Shp2D61G dopo trattamento con 0,25 μM di PD0325901 tra 4 e 6 hpf. Le immagini sono mostrate come canale singolo a 633 nm (qui p-ERK, pseudocolorato in ciliegia) e combinate (p-ERK in ciliegia e t-ERK in verde). (E) Nei piccoli schemi di riquadro della selezione del ROI sulla regione del margine (linea gialla tratteggiata) dalle immagini z-stack grezze per l'analisi dei dati nelle Fiji . Sono inclusi gli zoom delle immagini z-stacks che mostrano la regione del margine, così come le misure grezze di densità di intensità recuperate e valutate per dedurre l'attivazione di ERK nel ROI da fratelli che esprimono Shp2WT o Shp2D61G con o senza trattamento PD0325901 0,25 μM. La barra della scala è indicata nei pannelli delle immagini. Abbreviazioni: IHC = immunoistochimica; ERK = chinasi regolata dal segnale extracellulare; t-ERK = chinasi totale regolata dal segnale extracellulare; p-ERK = chinasi fosforilata regolata dal segnale extracellulare; FRET = trasferimento di energia per risonanza di fluorescenza; MEKi = Inibitore degli enzimi proteina chinasi attivata da mitogeno; LMA = agarosio a basso punto di fusione; PBS = tampone fosfatasi soluzione salina; HPF = ore dopo la fecondazione; ROI = regione di interesse. Clicca qui per visualizzare una versione più grande di questa figura.
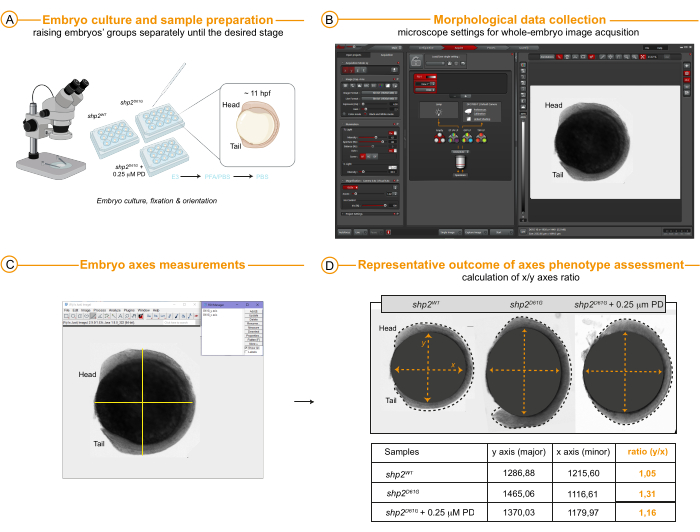
Figura 4: Analisi morfologica a 11 hpf. (A) Gli embrioni sono stati allevati in terreno E3 fino allo stadio di sviluppo desiderato, qui 11 hpf, quindi fissati in PFA al 4%, conservati in 1x PBS e disposti lateralmente in pozzetti per l'acquisizione di immagini al microscopio e la valutazione degli assi. (B) Impostazioni di acquisizione dell'immagine in uno stereomicroscopio standard e un esempio dell'immagine risultante che mostra un intero embrione (qui Shp2WT) valutato con la modalità a campo chiaro. (C, D) Risultati rappresentativi e risultati delle misurazioni degli assi in Shp2WT (controllo), Shp2D61G e Shp2D61G dopo trattamento con 0,25 μM PD0325901 tra 4 e 6 hpf. Il contorno dell'embrione è indicato da una linea nera tratteggiata. La barra della scala è indicata nei pannelli delle immagini. Abbreviazioni: hpf = ore dopo la fecondazione; PFA = paraformaldeide; PBS = tampone fosfatasi soluzione salina. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Nonostante decenni di ricerca e miriadi di mutazioni che portano a forme altamente eterogenee di RASopatie ora mappate, varianti genetiche con significato sconosciuto continuano ad emergere dagli sforzi di sequenziamento su pazienti non diagnosticati. In effetti, in molti casi, la diagnosi basata esclusivamente sulle caratteristiche cliniche può essere impegnativa e gli approcci genomici funzionali per convalidare i risultati del sequenziamento rimangono fondamentali. Inoltre, nonostante alcune molecole antitumorali disponibili (ad esempio, gli inibitori MEK) siano state proposte per il trattamento di un sottogruppo di RASopatie e alcune storie di successo inizino ad emergere, esiste un consenso limitato. Ciò è causato da scarse evidenze precliniche per la maggior parte dei farmaci disponibili, anche per quanto riguarda le dosi efficaci di potenti farmaci antitumorali e le finestre terapeutiche per i pazienti pediatrici.
L'analisi funzionale nei modelli animali rappresenta un passo importante per la sottoclassificazione della malattia, la stratificazione dei pazienti e la valutazione iniziale dei farmaci. Il pesce zebra può essere un modello in vivo efficiente per la validazione funzionale di varianti potenzialmente patogene che causano malattie pediatriche, tra cui la RASopatie14. La valutazione fenotipica all'avanguardia dell'impatto delle varianti associate alla RASopathy esiste per i modelli di RASopathy di zebrafish e si basa principalmente su letture morfologiche: il "test ovale" dell'embrione, in cui gli assi del corpo vengono misurati alla fine della gastrulazione. Tuttavia, la correlazione con l'attivazione di ERK è comunemente eseguita principalmente con metodi standard di post-fissazione - immunoblots/IHC.
La pipeline che abbiamo messo a punto aveva lo scopo di valutare la fluttuazione di ERK in tempo reale in grado di testare rapidamente la potenziale efficacia dei farmaci disponibili all'inizio della gastrulazione, anticipando l'analisi delle alterazioni morfometriche risultanti. Il protocollo qui presentato inizia con la generazione del modello transitorio di RASopathy di zebrafish desiderato mediante sovraespressione dell'allele GoF di interesse come mRNA nello stadio a una cellula del reporter in vivo ERK (Tg[ef1a:ERK biosensor-nes] Teen)6. In questo sensore FRET di tipo EKAR, l'imaging FRET multispettrale è un metodo non invasivo e non distruttivo che può essere utilizzato per rilevare con precisione la fluttuazione del segnale ERK spazialmente e temporalmente in embrioni vivi che sono fisiologici durante lo sviluppo, aberranti nei modelli di RASopathy e corretti dalla modulazione farmacologicadel segnale 6,7.
Diverse fasi critiche della pipeline presentata dovrebbero essere prese in considerazione per ottenere risultati informativi dai campioni di embrioni. Data la nota bassa sensibilità dei sensori FRET15 e i possibili effetti variabili delle mutazioni che influenzano la segnalazione di ERK (soprattutto per mutazioni non precedentemente convalidate con metodi standard), prima di impostare l'esperimento, è consigliabile decidere il tipo di disegno sperimentale (cioè n. di gruppi e condizioni) e quindi fare una stima del numero richiesto di embrioni (dimensione del campione, n) per la dimensione dell'effetto atteso e i valori standard dati per gli errori di tipo I e II. È anche importante condurre esperimenti pilota per stimare l'effettivo minimo n richiesto per osservare differenze rilevanti tra WT e proteina mutante. È inoltre essenziale la convalida incrociata dei risultati FRET utilizzando approcci alternativi e complementari, come l'IF o i saggi morfologici (come indicato qui).
In generale, una dimensione standard del campione di n = 30 può essere appropriata per rilevare grandi effetti di valori normalmente distribuiti16. In dettaglio, per l'analisi a priori della dimensione del campione, si possono utilizzare programmi liberamente disponibili come "G-power"17 , impostando questi parametri standard: livello di significatività (α): tipicamente impostato a 0,05; livello di confidenza (1 - β): tipicamente impostato al 95%; potenza di un test di ipotesi: tipicamente impostata su 80% (1 - β, dove β è la probabilità di commettere un errore di Tipo II); dimensione dell'effetto d (l'entità attesa delle differenze tra i gruppi sperimentali) che può avere valori diversi a seconda che sia grande, media o piccola, secondo Cohen18; variazione statistica (dispersione in una distribuzione di frequenza).
Nell'esempio specifico fornito, un noto effetto di grandi dimensioni del mutante Shp2D61G è stato precedentemente convalidato anche nel pesce zebra utilizzando letture morfologiche ed esperimenti pilota di FRET dal vivo, inclusi saggi complementari come FRET su campioni fissati come IF e letture morfologiche sono stati utilizzati per convalidare l'effetto osservato (vedi in Fasano et al.7).
Inoltre, l'mRNA di alta qualità è un fattore di partenza chiave per generare modelli di malattia transitori appropriati. Infatti, mentre sarebbe ideale impiegare modelli geneticamente modificati per ricapitolare l'eterozigosi dei pazienti, la generazione di questi modelli non è compatibile con la necessità di test rapidi delle varianti emergenti in quanto richiederebbe un aumento della linea. I modelli transitori che vengono generati e valutati solo durante l'embriogenesi sono da preferire per lo screening di grandi dimensioni.
Tuttavia, per garantire l'informatività e la riproducibilità dei risultati, è necessario prestare particolare attenzione alla valutazione della qualità dell'RNA. Oltre ai classici gel di formammide, l'RNA può essere controllato anche utilizzando strumenti come il Bioanalyzer che fornisce la quantificazione diretta e gli elettroferogrammi visivi per l'ispezione della qualità. I preparati di RNA di alta qualità sembrano un singolo picco acuto della dimensione desiderata. Si consiglia di non procedere con la microiniezione se l'RNA è vistosamente degradato. È importante sottolineare che, quando si tratta di microiniezione, per lotti di mRNA di nuova sintesi, è necessario un esperimento iniziale per calibrare la dose di iniezione corretta. A questa dose ideale, l'espressione della forma WT dell'mRNA di interesse nei fratelli dello stesso lotto non dovrebbe causare un fenotipo visibile.
Come raccomandazione generale, quando si preparano gli embrioni per l'iniezione, sarebbe meglio ottenere, raccogliere e valutare gli embrioni da coppie singole di accoppiamento (1 femmina + 1 maschio) per essere sicuri di ridurre al minimo la variabilità del lotto. Tuttavia, per aumentare la resa embrionale, la deposizione delle uova dei pesci può essere impostata da incroci di gruppo con più maschi e femmine. In questo caso, gli embrioni raccolti derivano probabilmente da più eventi di fecondazione che si verificano con un leggero ritardo all'interno della vasca ed è, quindi, importante selezionare accuratamente gli individui allo stesso stadio di sviluppo (gastrulazione precoce) ispezionando gli embrioni più volte nelle prime 2 ore dopo la raccolta. Per consentire una buona crescita dell'embrione nella delicata fase iniziale, è necessario prestare particolare attenzione anche all'utilizzo di tutte le soluzioni appena preparate. Inoltre, data la possibile presenza di materiale fecale e detriti dell'evento di accoppiamento, può verificarsi la contaminazione dei lotti di embrioni da funghi e batteri se gli embrioni nel terreno E3 non vengono lavati e puliti a partire immediatamente dopo la raccolta. La vitalità del lotto di embrioni deve essere monitorata prima, durante e dopo l'esperimento. Se il tasso di mortalità è superiore al tasso standard (normalmente 20-30%), l'esperimento deve essere interrotto.
Per quanto riguarda i trattamenti farmacologici, almeno per la classe di MEKi qui testata, è meglio evitare trattamenti prima di 4 hpf, poiché questi potrebbero influenzare irreversibilmente lo sviluppo embrionale (tossicità), come descritto in precedenza19. Un altro passo fondamentale per l'affidabilità dell'esperimento è quello di utilizzare la stessa concentrazione di veicolo farmacologico (in questo caso DMSO) per tutte le condizioni sperimentali ed evitare il sovraffollamento di embrioni per pozzetto durante il trattamento per mantenere buoni livelli di ossigenazione (un massimo di 20 embrioni dovrebbero essere conservati in una piastra a 6 pozzetti).
Anche l'ottimizzazione dei parametri di imaging è estremamente importante per il successo della pipeline che include in particolare l'imaging FRET multispettrale ma anche l'imaging confocale di campioni IHC. In primo luogo, data la variabilità nell'espressione transgenica, un aspetto decisivo è quello di selezionare accuratamente gli embrioni di Teen e scartare quelli con un'espressione molto bassa, che, data la bassa gamma dinamica dei sensori FRET, potrebbero impedire una sufficiente rilevazione e quantificazione del segnale (vedi esempio Figura 2). L'espressione del transgene può essere visibile sotto uno stereomicroscopio standard con lampade fluorescenti e ruote portafiltri appropriate ~2 ore dopo l'iniezione.
Nella nostra esperienza per l'acquisizione di dati multispettrali di pesci sensore Teen , le impostazioni del laser e i parametri della modalità di acquisizione della definizione del colorante spettrale (x, y, λ, z) sono passaggi iniziali critici del microscopio e delle configurazioni hardware e devono essere ottimizzati in base alle esigenze specifiche. In linea di principio, i parametri dovrebbero essere impostati in modo tale da raggiungere un buon compromesso tra la raccolta del segnale e la velocità di acquisizione. Ciò è particolarmente importante per le registrazioni in tempo reale su embrioni quasi interi, come mostrato qui. La velocità di acquisizione di 400-600 Hz con una dimensione del passo compresa tra 8 μm e 10 μm e l'impostazione di una singola scansione per piano z potrebbero non fornire una risoluzione cellulare, ma potrebbero essere sufficienti per catturare i cambiamenti dinamici dell'attività ERK nei tessuti degli embrioni. In linea di principio, è possibile ottenere immagini con risoluzione quasi cellulare con impostazioni del microscopio ad hoc , ma a scapito della velocità e del campionamento 3D. Questo potrebbe essere interessante se solo una certa popolazione cellulare viene studiata all'interno degli embrioni anche nelle fasi successive6.
La separazione spettrale dei coloranti può essere eseguita con algoritmi diversi da quello qui indicato. Indipendentemente da ciò, la selezione della regione di riferimento necessaria per assegnare i migliori spettri di emissione sia per la CFP che per l'YFP è fondamentale. A tal fine, è consigliabile verificare la coerenza dei risultati ottenuti selezionando e facendo la media di diverse ROI, dove il segnale tissutale/cellulare è chiaramente visibile. Al termine dell'acquisizione spettrale e una volta ottenuta la separazione spettrale del colorante, è possibile ottenere immagini raziometriche anche a partire da scansioni CFP e YFP grezze e funzioni di rapporto liberamente disponibili nelle Fiji. Tuttavia, per visualizzare ed evidenziare le differenze nei livelli di segnale FRET spazialmente, potrebbe essere consigliabile ridimensionare le immagini. Nelle nostre mani, la pseudocolorazione utilizzando le tabelle di ricerca "intelligenti" (LUT) codificate all'interno del software gratuito Fiji funziona bene per mostrare meglio i livelli di attivazione ERK. Altre LUT e altre scale possono essere scelte in base alle preferenze dell'utente.
Va sottolineato che la pipeline sperimentale qui presentata è limitata ai sensori Teen che ospitano CFP come donatore (D) e Ypet (simile a YFP) come accettore (A). In generale, quando si decide il reporter FRET, si dovrebbe considerare la gamma dinamica dei sensori (la capacità di rilevare piccoli ma significativi cambiamenti) e l'efficienza FRET (E), influenzata dalla distanza D-A e dall'orientamento all'interno del sensore, nonché la sovrapposizione spettrale tra l'emissione D e l'assorbimento A. Teen è un sensore EKAREV che, attraverso un design ottimizzato della costruzione, offre una gamma dinamica e una sensibilità (E) migliorate rispetto alle versioni precedenti. Tuttavia, la gamma dinamica relativamente bassa, normalmente peggiore in vivo, può essere considerata il principale limite a questo approccio. Tuttavia, Sari et al. e Wong et al. hanno dimostrato che i cambiamenti dinamici dell'ERK che si verificano fisiologicamente durante lo sviluppo sono comunque visibili e misurabili negli embrioni adolescenti e osserviamo fluttuazioni del segnale in un modello di pesce NS trattato farmacologicamente 6,9. Tuttavia, prevediamo che le fluttuazioni del segnale ERK estremamente basse potrebbero non essere facili da valutare utilizzando il metodo mostrato qui. Sarà importante testare le prestazioni della nostra pipeline con biosensori ERK migliorati che saranno probabilmente disponibili in futuro.
Per la convalida incrociata dei risultati con IHC, un passaggio critico è la qualità della fissazione tissutale. Il tessuto embrionale a 6 hpf è estremamente delicato e sottile e, quindi, sensibile a una scarsa fissazione e sovrafissazione. Data la qualità da lotto a lotto e la variabilità delle prestazioni per le soluzioni fissative, nonché per gli anticorpi, il tempo di fissazione e il successo dell'intero protocollo IHC dovrebbero essere valutati in un esperimento pilota poco prima di valutare i campioni sperimentali. È sempre consigliabile un fissativo sterile e preparato al momento.
In sintesi, il protocollo sperimentale mostrato qui nel modello di zebrafish rappresenta una pipeline robusta e relativamente veloce per valutare l'impatto di alleli NS selezionati sull'attivazione di ERK nelle cellule gastrulanti di embrioni precoci. Il metodo, che prevede l'imaging FRET multispettrale nel pesce sensore Teen di nuova concezione per rilevare le fluttuazioni molecolari in tempo reale, supera l'IHC classico. Tuttavia, l'IHC può essere utilizzato come approccio complementare per convalidare i risultati ottenuti. L'approccio anticipa le classiche letture fenotipiche, vale a dire il rapporto assi maggiori/minori (test dell'"embrione ovale"), un gold standard per i modelli di pesci RASopathy, ma misurabile solo alla fine della gastrulazione. Il protocollo può anche essere applicato per testare la capacità del MEKi proposto di correggere le alterazioni del segnale e degli assi negli embrioni.
Considerando che abbiamo testato solo l'allele shp2D61G associato a NS e un farmaco, l'ulteriore sviluppo del test dovrebbe includere la valutazione delle prestazioni rispetto ad altre mutazioni associate a RASopathy con impatti diversi sull'attivazione di ERK e ulteriori farmaci, dosi e finestre di trattamento proposti. Sarà interessante valutare la sensibilità di questo approccio multilivello per altre varianti emergenti che influenzano molecole a diversi livelli della cascata di segnali RAS-MAPK. Infine, per potenziare i test delle varianti, sarebbe necessaria la futura standardizzazione di un metodo dedicato ad alto rendimento per l'imaging raziometrico FRET e la quantificazione in sistemi di analisi dedicati ad alto contenuto.
Riconoscimenti
Ringraziamo il Dr. Jeroen den Hertog (Hubrecht Institute, Utrecht, Paesi Bassi) per aver gentilmente fornito pCS2+_eGFP-2a-Shp2a da cui è stato estratto il CDS completo shp2 per generare il modello plasmidico che abbiamo utilizzato7. Ringraziamo l'Istituto di Scienza e Tecnologia di Nara (Takaaki Matsui), l'Istituto Nazionale di Genetica (NIG/ROIS) (Koichi Kawakami), per aver fornito la linea di reporter per adolescenti transgenici. Questo lavoro è stato sostenuto dal Ministero della Salute italiano - Fondi di ricerca in corso 2021 e Fondi per la ricerca in corso 2024 e Ricerca finalizzata Giovani Ricercatori GR-2019-12368907 ad AL; Fondi di ricerca in corso 2019, PNRRMR1-2022-12376811, 5x1000 2019, AIRC (IG-21614 e IG-28768) e LazioInnova (A0375-2020-36719) a MT.
Materiali
| Name | Company | Catalog Number | Comments |
| Plasticwares | |||
| 1.7 L Breeding Tank - Beach style Design | Tecniplast | 1.7L SLOPED | Breeding tank |
| Capillaries GC100F-10 | Harvard apparatus | 30-0019 | One-cell stage embryo microinjection |
| Cell and Tissue Culture Plates - 12 well | BIOFIL | TCP011012 | Embryo collection and treatment |
| Cell and Tissue Culture Plates - 6 well | BIOFIL | TCP011006 | Embryo collection and treatment |
| Cell Culture Dish | SPL Life Sciences | 20100 | Embryo collection |
| Nunc Glass Dishes 12mm | Thermo Fisher | 150680 | Embryo FRET spectral imaging |
| Pipette Pasteur | Corning | 357524 | Embryo transfer |
| Protein Lobind Tubes 2ml | Eppendorf | 30108450 | IHC assay |
| Reagents and others | |||
| Caviar 500-800 µm | Rettenmaier Italia | BE2269 (500-800) | Dry fish food |
| Great Salt Lake Blue Artemia Cysts | Sanders | 00004727 | Live fish food |
| Instant Ocean salt | Tecniplast | XPSIO25R | Dehydrated sea salt for live food preparation |
| Tg(EF1a:ERK Biosensor-nes) (Teen) | Contacts for ordering*: National BioResource Project Zebrafish, Support Unit for Animal Resources Development, RRD, RIKEN Center for Brain Science, Japan. https://shigen.nig.ac.jp/zebra/index_en.html *upon MTA signature. | - | Supplier of ERK Reporter zebrafish line. Fish embryos can be obtained upon MTA signature from National BioResource Project of Japan for Zebrafish (RIKEN, Japan). The zebrafish line is deposited by Nara Institute of Science and Technology (Takaaki Matsui) and the National Institute of Genetics (NIG/ROIS) (Koichi Kawakami, patent for Tol2 system) (Wong et al., 2018, Urasaki et al., 2006, Okamoto and Ishioka, 2010). |
| 6x loading dye | Cell Signaling | B7024S | Gel Elecrophoresis |
| 100 bp DNA ladder | NEB | N3231S | Gel Elecrophoresis |
| Agarose | Sigma-Aldrich | 1,01,236 | Gel Elecrophoresis |
| Agarose, low gelling temperature | Sigma-Aldrich | A9414-10G | Embryo mounting for FRET spectral imaging and IHC assay |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A8022 | IHC assay |
| Calcium chloride | Sigma-Aldrich | 223506 | E3 medium component |
| Calcium nitrate | Sigma-Aldrich | 237124 | Danieau stock solution component |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418-100ML | IHC assay |
| EDTA | Sigma-Aldrich | E9884 | TBE buffer component for gel preparation |
| Ethanol 99%+ | Fisher Scientific | 10048291 | In vitro RNA purification |
| Formaldeide 16% | Thermo Fisher | 28908 | Embryo fixation |
| Formamide | Sigma-Aldrich | F9037 | Gel Elecrophoresis |
| Gel Loading Buffer II (Denaturing PAGE) | Thermo Fisher | AM8546G | In vitro RNA transcription |
| Glacial Acetic Acid | Sigma-Aldrich | 695092 | TBE buffer component for gel preparation |
| Glycerol | Sigma-Aldrich | G6279-1L | IHC assay |
| Goat anti-mouse Alexa Fluor 488 | Thermo Fisher | A11001 | IHC assay |
| Goat anti-rabbit Alexa Fluor 633 | Thermo Fisher | A21070 | IHC assay |
| HEPES | Sigma-Aldrich | H3375 | Danieau stock solution component |
| KpnI - HF (Enzyme + rCutSmart Buffer) | NEB | R3142 | Plasmid linearization |
| Magnesium sulfate | Sigma-Aldrich | 230391 | E3 medium component/Danieau stock solution component |
| Millennium RNA Markers | Thermo Fisher | AM7150 | Gel Elecrophoresis |
| Monarch Genomic DNA purification Kit | NEB | T3010L | Plasmid linearization |
| Mouse monoclonal p44/42 MAPK | Cell Signaling | 4696S | IHC assay |
| mMACHINE SP6 Transcription Kit | Thermo Fisher | AM1340 | In vitro RNA transcription |
| Normal Goat serum (NGS) | Sigma-Aldrich | G9023 | IHC assay |
| Nuclease-free water Ambion | Thermo Fisher | AM9937 | In vitro RNA transcription |
| PD0325901 | Sigma-Aldrich | PZ0162 | MEK inhibitor |
| Phenol Red solution | Sigma-Aldrich | P0290 | Microinjection mix component |
| Poly A Tailing Kit | Thermo Fisher | AM1350 | In vitro RNA transcription |
| Potassium chloride bioxtra | Sigma-Aldrich | P9333 | E3 medium component/Danieau stock solution component/PBS stock solution component |
| Potassium dihydrogen phosphate | Sigma-Aldrich | P0662 | PBS stock solution component |
| Proteinase K | Sigma-Aldrich | P2308 | IHC assay |
| Rabbit polyclonal phospho-p44/42 MAPK | Cell Signaling | 4695S | IHC assay |
| SYBR safe DNA gel staining | Thermo Fisher | S33102 | Gel Elecrophoresis |
| Sodium Chloride | Sigma-Aldrich | 31434-M | E3 medium component/Danieau stock solution component/PBS stock solution component |
| Sodium phosphate dibasic | Sigma-Aldrich | 71643 | PBS stock solution component |
| Trizma base | Sigma-Aldrich | T1503 | TBE buffer component for gel preparation |
| Triton X-100 | Sigma-Aldrich | T8787 | PBSTr buffer component |
| Equipment | |||
| Alliance Mini HD9 | Uvitec | - | Imaging system |
| Centrifuge 5430 R | Eppendorf | 5428000205 | Microcentrifuge |
| Eppendorf ThermoMixer C | Eppendorf | - | Embryo mounting |
| FemtoJet 4x | Eppendorf | - | Microinjection system |
| Infinite M Plex | Tecan | - | Multimode plate reader |
| Leica M205FA | Leica Microsystems | - | Fluorescence stereo microscope |
| Leica TCS-SP8X equipped with incubator (OkoLab) | Leica Microsystems | - | Confocal microscope |
| Mini-sub Cell GT Horiziontal Electrophoresis System | Bio-Rad | 1704406 | Gel Elecrophoresis |
| PC-100 Vertical puller | Narishige | - | Needle puller |
| PowerPac Universal Power Supply | Bio-Rad | 1645070 | Gel Elecrophoresis |
| Stellaris 5 | Leica Microsystems | - | Confocal microscope |
| Vortex MiniStar silverline | VWR | - | Plasmid preparation |
| Softwares | |||
| Biorender | Biorender | CC-BY 4.0 license | Cartoon elaboration for Figures |
| Excel | Microsoft Office Professional Plus 2019 | - | Data analyses |
| Fiji software | ImageJ | 15.3t | Imaging rendering and quantitative analyses (FRET signals measurements, ERK fluorescence intensity in IHC assay, embryo axes lenght) |
| GraphPad Prism | GraphPad Software LLC | v. 9 | Statistical data analyses |
| iControl spectrophotometer software | Tecan | v. 2.0 | RNA quantification |
| Illustrator | Adobe | 26.0.3 (64-bit) | Figure assembling |
| LASX software | Leica Microsystems | v. 4.5 (Stellaris 5), v. 3.0 (M205FA), v. 3.5 (TCS-SP8X) | Imaging acquisition for spectral FRET experiments and embryo imaging for axes lenght measurements |
| Q9 Mini 18.02-SN software | Uvitec | - | Gel image acquisition |
Riferimenti
- Iroegbu, J. D., Ijomone, O. K., Femi-Akinlosotu, O. M., Ijomone, O. M. ERK/MAPK signalling in the developing brain: Perturbations and consequences. Neurosci Biobehav Rev. 131, 792-805 (2021).
- Tartaglia, M., Aoki, Y., Gelb, B. D. The molecular genetics of RASopathies: An update on novel disease genes and new disorders. Am J Med Genet C Semin Med Genet. 190 (4), 425-439 (2022).
- Tate, J. G., et al. COSMIC: the catalogue of somatic mutations in cancer. Nucleic Acids Res. 47 (D1), D941-D947 (2019).
- Tartaglia, M., et al. Mutations in PTPN11, encoding the protein tyrosine phosphatase SHP-2, cause Noonan syndrome. Nat Genet. 29 (4), 465-468 (2001).
- Jopling, C., van Geemen, D., den Hertog, J. Shp2 knockdown and Noonan/LEOPARD mutant Shp2-induced gastrulation defects. PLoS Genet. 3 (12), e225 (2007).
- Wong, K. -. L., Akiyama, R., Bessho, Y., Matsui, T. ERK activity dynamics during zebrafish embryonic development. Int J Mol Sci. 20 (1), 109 (2019).
- Fasano, G., et al. Assessment of the FRET-based Teen sensor to monitor ERK activation changes preceding morphological defects in a RASopathy zebrafish model and phenotypic rescue by MEK inhibitor. Mol Med. 30 (1), 47 (2024).
- Zimmermann, T., Rietdorf, J., Girod, A., Georget, V., Pepperkok, R. Spectral imaging and linear un-mixing enables improved FRET efficiency with a novel GFP2-YFP FRET pair. FEBS Lett. 531 (2), 245-249 (2002).
- Sari, D. W. K., et al. Time-lapse observation of stepwise regression of Erk activity in zebrafish presomitic mesoderm. Sci Rep. 8 (1), 4335 (2018).
- Wang, H., Li, J., Xue, T., Cheng, N., Du, H. Construction of a series of pCS2+ backbone-based Gateway vectors for overexpressing various tagged proteins in vertebrates. Acta Biochim Biophys Sin (Shanghai). 48 (12), 1128-1134 (2016).
- Abdelrahman, D., Hasan, W., Da'as, S. Microinjection quality control in zebrafish model for genetic manipulations. MethodsX. 8, 101418 (2021).
- Yuan, S., Sun, Z. Microinjection of mRNA and morpholino antisense oligonucleotides in zebrafish embryos. J Vis Exp. (27), e1113 (2009).
- Rossant, J., Nutter, L. M. J., Gertsenstein, M. Engineering the embryo. Proc Natl Acad Sci USA. 108 (19), 7659-7660 (2011).
- Jindal, G. A., et al. In vivo severity ranking of Ras pathway mutations associated with developmental disorders. Proc Natl Acad Sci USA. 114 (3), 510-515 (2017).
- Leavesley, S. J., Rich, T. C. Overcoming lmitations of FRET measurements. Cytometry A. 89 (4), 325-327 (2016).
- Mascha, E., Vetter, T. Significance, errors, power, and sample size: Theblocking and tackling of statistics. Anesth Analg. 126 (2), 691-698 (2018).
- Faul, F., Erdfelder, E., Lang, A. -. G., Buchner, A. G*Power 3: a flexible statistical power analysis program for the social, behavioral, and biomedical sciences. Behav Res Methods. 39 (2), 175-191 (2007).
- Cohen, J. . Statistical power analysis for the behavioral sciences. , (1988).
- Anastasaki, C., Rauen, K. A., Patton, E. E. Continual low-level MEK inhibition ameliorates cardio-facio-cutaneous phenotypes in zebrafish. Dis Model Mech. 5 (4), 546-552 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon