Method Article
Ein effizientes Protokoll zur Beurteilung der Modulation der ERK-Aktivität in frühen Modellen des Zebrafisch-Noonan-Syndroms mittels Live-FRET-Mikroskopie und Immunfluoreszenz
In diesem Artikel
Zusammenfassung
RASopathien sind multisystemische genetische Syndrome, die durch eine Hyperaktivierung des RAS-MAPK-Signalwegs verursacht werden. Potenziell pathogene Varianten, die auf ihre Validierung warten, treten kontinuierlich auf, während schlechte präklinische Evidenz die Therapie einschränkt. Hier beschreiben wir unser in vivo-Protokoll zum Testen und Kreuzvalidieren von RASopathie-assoziierten ERK-Aktivierungsniveaus und ihrer pharmakologischen Modulation während der Embryogenese durch Live-FRET-Bildgebung in Teen-Reporter-Zebrafischen.
Zusammenfassung
RASopathien sind genetische Syndrome, die durch eine ERK-Hyperaktivierung verursacht werden und zu multisystemischen Erkrankungen führen, die auch zu einer Krebsveranlagung führen können. Trotz einer breiten genetischen Heterogenität liegen der Mehrzahl der Fälle Keimbahn-Gain-of-Function-Mutationen in Schlüsselregulatoren des RAS-MAPK-Signalwegs zugrunde, und dank fortschrittlicher Sequenzierungstechniken werden weiterhin potenziell pathogene Varianten identifiziert, die den RAS-MAPK-Signalweg beeinflussen. Die funktionelle Validierung der Pathogenität dieser Varianten, die für eine genaue Diagnose unerlässlich ist, erfordert schnelle und zuverlässige Protokolle, vorzugsweise in vivo. Angesichts des Mangels an wirksamen Behandlungen in der frühen Kindheit können solche Protokolle, insbesondere wenn sie in kostengünstigen Tiermodellen skalierbar sind, dazu beitragen, eine präklinische Grundlage für die Neupositionierung/Umwidmung von Arzneimitteln zu bieten.
Hier beschreiben wir Schritt für Schritt das Protokoll für die schnelle Generierung von transienten RASopathie-Modellen in Zebrafischembryonen und die direkte Inspektion von lebenden krankheitsassoziierten ERK-Aktivitätsänderungen, die bereits während der Gastrulation auftreten, durch multispektrale Echtzeit-Bildgebung des Förster-Resonanzenergietransfers (FRET). Das Protokoll verwendet einen transgenen ERK-Reporter, der kürzlich etabliert und in die Hardware kommerzieller Mikroskope integriert wurde. Wir stellen eine beispielhafte Anwendung für Zebrafischmodelle des Noonan-Syndroms (NS) vor, die durch Expression des Shp2D61G erhalten wurden. Wir beschreiben eine einfache Methode, die es ermöglicht, die ERK-Signaländerung im NS-Fischmodell vor und nach der pharmakologischen Signalmodulation durch verfügbare niedrig dosierte MEK-Inhibitoren zu registrieren. Wir beschreiben, wie ratiometrische FRET-Signale aus multispektralen Aufnahmen vor und nach der Behandlung generiert, abgerufen und bewertet werden und wie die Ergebnisse über klassische Immunfluoreszenz an ganzen Embryonen in frühen Stadien validiert werden können. Anschließend beschreiben wir, wie durch die Untersuchung morphometrischer Standardparameter späte Veränderungen der Embryonenform, die auf eine daraus resultierende Beeinträchtigung der Gastrulation hinweisen, bei denselben Embryonen abgefragt werden können, deren ERK-Aktivität 6 h nach der Befruchtung durch lebende FRET beurteilt wird.
Einleitung
RASopathien sind genetische Syndrome, die die normale Entwicklung beeinträchtigen und verschiedene Organe und Gewebe betreffen. Diese Zustände werden häufig durch Keimbahn-Gain-of-Function-Mutationen (GoF) in den Schlüsselgenen und Akteuren verursacht, die an der RAS/MAK-Signalübertragung beteiligt sind, was zu einer Hyperaktivierung (erhöhte Phosphorylierung) der extrazellulären signalregulierten Kinase (ERK) führt. ERK reguliert einige grundlegende Prozesse, die während der Entwicklung wichtig sind - das Gewebewachstum -, indem es in den Zellkern transloziert 1,2. Somatische Mutationen in Genen, die am RAS-MAPK-Signalweg beteiligt sind, sind die häufigsten Ereignisse, die zu Krebs führen3. So ist es nicht verwunderlich, dass eine Krebsveranlagung auch bei RASopathien beobachtet wird. Das Noonan-Syndrom (NS), gekennzeichnet durch Entwicklungsverzögerung, Kleinwuchs, kognitive Defizite mit unterschiedlichem Schweregrad und Kardiomyopathie, ist die häufigste Form der RASopathie2. In den meisten Fällen wird die Krankheit durch GoF-Mutationen in PTPN11 verursacht, dem ersten RASopathie-Gen, das Anfang 2000 entdeckt wurde4 und für das Protein Tyrosinphosphatase SHP2 kodiert, das als positiver Regulator des Signalwegs fungiert.
Seitdem werden dank des exponentiellen Einsatzes von Exom-Sequenzierungsansätzen bei nicht diagnostizierten Patienten weiterhin potenziell pathogene Varianten entdeckt, die Faktoren beeinflussen, die an der RAS-MAK beteiligt sind und wahrscheinlich mit verschiedenen Formen von RASopathien in Verbindung stehen, und warten auf eine funktionelle Charakterisierung für eine effiziente Patientenstratifizierung2. Um dieses Ziel zu erreichen, sind experimentelle Protokolle erforderlich, die eine schnelle und aussagekräftige funktionelle Validierung auf organismischer Ebene gewährleisten. Der Einsatz klassischer und standardisierter Säugetiermodelle zur Untersuchung von Varianten mit unbekannter Signifikanz wäre kostspielig, extrem zeitaufwändig und würde invasive Methoden bei intransparenten Großtieren erfordern. Eine solche Strategie ist angesichts der gesellschaftlichen Belastung durch arme oder nicht diagnostizierte RASopathie-Patienten, die derzeit ohne Behandlung oder Behandlung sind, eindeutig nicht mit dem Erfordernis schneller Tests vereinbar. Protokolle zur quantitativen Bewertung wichtiger phänotypischer Merkmale und molekularer Korrelate in ganzen Organismen würden auch dazu dienen, die mögliche klinische Translation von Medikamenten, die möglicherweise RASopathie-Patienten zur Verfügung stehen, durch Umwidmung/Neupositionierung zu beschleunigen.
Der Zebrafisch ist ein ideales Wirbeltiermodell, um Krankheiten zu untersuchen, die die frühe Entwicklung beeinflussen. Zunächst einmal teilen Zebrafische ein hohes Maß an genetischer Homologie mit dem Menschen. Die hohe Fruchtbarkeit erwachsener Fische führt zu einer großen Produktion von Embryonen, die klein sind und sich schnell entwickeln. Embryonen sind in frühen Stadien transparent, so dass wichtige Entwicklungsprozesse - Epibolie, Gastrulation, Achsen und Körperbaubildung - mit der Standardmikroskopie mühelos sichtbar gemacht werden können. Darüber hinaus ist die Verfügbarkeit transgener Linien, die zur Verfolgung des spezifischen zellulären Verhaltens und dynamischer molekularer Ereignisse in Raum und Zeit während der Entwicklung in Verbindung mit fortschrittlichen Techniken zur Generierung genetischer Modelle verwendet werden können, unschlagbar. Darüber hinaus können phänotypische Messwerte bei Zebrafischen auf mehreren Ebenen (von organismischen bis hin zu zellulären Defekten) bewertet werden, und spezielle Assays sind bereits für mehrere Krankheiten, einschließlich RASopathien5, etabliert. Darüber hinaus ermöglichen relativ einfache Bad-Immersionsmethoden für die Verabreichung von Arzneimitteln in den frühen Stadien, zumindest für wasserlösliche Verbindungen, ein Hochdurchsatz-Wirkstoffscreening in vivo in einem 96-Well-Format.
Aus molekularer Sicht zeigen Studien mit Standardansätzen wie Immunhistochemie und Immunoblot die Korrelation zwischen ERK-Aktivierung und RASopathie-assoziierten Entwicklungsdefekten in Fischembryonen 6,7. Der kürzlich entwickelte FRET-Biosensor vom EKAR-Typ im Zebrafisch (Tg[ef1a:ERK biosensor-nes], Teen) bietet ein zuverlässiges in vivo Werkzeug, um die ERK-Aktivierung während der Embryogenese räumlich und zeitaufgelöst zu registrieren. Daher könnte es für eine bessere Beurteilung dynamischer ERK-Veränderungen und pharmakologischer Modulationen in RASopathie-Fischmodellen wertvoll sein.
Im Teen-Sensor wird ein spezifisches ERK-Substrat im Reporter bei ERK-Aktivierung phosphoryliert, wodurch eine Konformationsänderung ausgelöst wird, die den fluoreszierenden CFP-Donor (D) und den fluoreszierenden Ypet-Akzeptor (A) in die Nähe bringt. Überlappt sich das D-Emissionsspektrum stark mit dem Absorptionsspektrum des A, kann es zu FRET kommen (Energieabsorption von D nach A). Dies ist proportional zum Abstand zwischen D und A und damit in Teen zum ERK-Aktivierungsstatus. Unterschiedliche Bildgebungsprotokolle können sowohl mit Standard- als auch mit fortschrittlichen Bildgebungsmodulen von Standard- oder Konfokalmikroskopen sowohl in lebenden als auch in fixierten Proben eingerichtet werden. Nach D-Anregung gehört die Erfassung von multispektralen Scans entlang eines definierten Emissionsspektrums (λ) von CFP bis YFP, gefolgt von spektralen "Entmischungs"-Algorithmen, zu den zuverlässigsten Methoden zur Registrierung und Quantifizierung von FRET-Daten8. Es kann auch auf lebende Zebrafischproben angewendet werden, um die in vivo Gewebedynamik zu erfassen.
In Anlehnung an frühere Berichte 6,9 und unsere jüngste Anwendung7 beschreiben wir hier den Schritt-für-Schritt-Workflow mit Teen Fish zur Beurteilung der ERK-Aktivierung in Zellen am Rand des tierischen Pols von NS-Modellen zu Beginn der Gastrulation und korrelieren sie mit charakteristischen Defekten der Körperachsen, die erst später in der Entwicklung sichtbar werden. Wir zeigen, wie quantitative FRET-Daten aus lebenden NS-Gastruten vor und nach der Behandlung mit einem verfügbaren MEKi gewonnen und untersucht werden können und wie die Ergebnisse mittels Standardimmunhistochemie gegen phosphoryliertes (aktives) ERK validiert oder korrelative morphometrische Analysen von embryonalen Elongationsdefekten durchgeführt werden können.
Der Workflow könnte angewendet werden, um den Funktionstest von neu auftretenden Varianten und Krankheitsgenen, die mutmaßlich mit RASopathien assoziiert sind, zu verbessern und Einblicke in die Korrelation der ERK-Aktivierungsdynamik räumlich und zeitlich während der Wirbeltierentwicklung und der morphologischen Defekte in Embryonen zu erhalten. Wir zeigen, dass dieses Protokoll auch verwendet werden kann, um die Wirksamkeit von Medikamentenkandidaten zu testen, die die ERK-Aktivierung modulieren.
Protokoll
Alle Versuchsverfahren zur Haltung und Zucht von Tieren wurden gemäß den ANARRIVE-Richtlinien für die Verwendung von Zebrafischen in Tierversuchen durchgeführt und vom italienischen Gesundheitsministerium (Direzione Generale della Sanità Animale e dei Farmaci veterinari - DGSAF) genehmigt. Alle DNA/RNA-Reaktionen und Bildgebungssitzungen können je nach gewünschtem Material oder der Anzahl der getesteten Gene und Varianten nach oben oder unten skaliert werden.
1. Generierung und medikamentöse Behandlung von transienten Zebrafisch-RASopathie-Modellen
HINWEIS: Um die Expression von RASopathie-assoziierten Varianten zu überwachen, können spezifische Konstrukte verwendet werden, die die gewünschte kodierende Sequenz (cds) des interessierenden Proteins im Rahmen mit den cds kleiner nicht fluoreszierender Tags (wie myc oder ähnliches) enthalten. Auf diese Weise können die Expressionsniveaus des mutierten Proteins mit einem Standard-Western-Blot gegen den Tag bestimmt werden. Wenn Antikörper gegen das spezifische Protein von Interesse verfügbar sind, können Tags vermieden werden. Immunfluoreszenz kann auch verwendet werden, um die Proteinexpression im Embryogewebe nach Standardprotokollen zu beurteilen. Diese Art von Kontrollexperiment kann nützlich sein, um die Expression mutierter Proteine mit den induzierten ERK-Aktivierungsniveaus zu korrelieren. Die Verwendung von Fluoreszenzmarkierungen in Kombination mit FRET-Bildgebung ist aufgrund der möglichen Fluoreszenzemissionsübergriffe während der Mikroskopie nicht ratsam.
- Plasmid-Linearisierung (Tage 1-2) (Abbildung 1A)
HINWEIS: Bei der Generierung eines vorübergehenden Krankheitsmodells im Zebrafisch (hier RAsopathie), das durch eine GoF-Mutation verursacht wird, die ein bestimmtes Gen betrifft, besteht ein schneller Ansatz darin, die mRNA vorzubereiten, die für den Wildtyp (WT) und das mutierte Protein von Interesse kodiert, die dann in Zebrafischembryonen injiziert werden sollten, indem die folgenden Schritte ausgeführt werden. Das Plasmid, das als Template zur Transkription der mRNA verwendet wird, sollte die gewünschte CDS (kodierende Sequenz) in voller Länge für das interessierende Gen enthalten, das stromabwärts eines T7-, Sp6- oder T3-Polymerase-Promotors kloniert und dem Polyadenylierungssignal (polyA) vorgeschaltet ist. Falls nicht verfügbar, besteht der erste Schritt darin, es durch Klonierung des von Zebrafischen oder Menschen gewünschten CDS in einem geeigneten Vektor10 herzustellen und durch SANGER-Sequenzierung zu validieren. Planen Sie für das Klonen zusätzliche Zeit ein (durchschnittlich 1 Woche einschließlich Klonen, Screening von Kolonien und Isolierung und Erweiterung der richtigen Klone).- Linearisieren Sie das Plasmid mit geeigneten Restriktionsenzymen, indem Sie das polyA-Signal (hier KpnI) nur einmal stromabwärts schneiden. Um eine effiziente Plasmidlinearisierung zu erreichen, mischen Sie 3 γ (μg/μl) Plasmid-DNA, 2 μl Restriktionsenzym (20.000 Einheiten/ml) und 10 μl 10x Reaktionspuffer in einem Endvolumen von 100 μl in nukleasefreiem Wasser. Mischen Sie die Komponenten gründlich, indem Sie sie einige Male pipettieren und inkubieren Sie das Reaktionsgemisch bei 37 °C für 2-4 h.
- Überprüfen Sie das Ergebnis der Linearisierung durch Elektrophorese an einem 1,5%igen Agarosegel.
- Verdünnen Sie eine 10-fache Stammlösung FSME (48,5 g Tris, 11,4 mL Eisessig, 20 mL 0,5 M EDTA [pH 8,0]) mit nukleasefreiem Wasser.
- Bereiten Sie das Agarose-Gel vor, indem Sie 1,5 g Agarose in der Mikrowelle in einem Endvolumen von 100 mL 1x FSME-Lösung auflösen. Fügen Sie dann 3,5 μl Farbstoff-Gel-Beize hinzu.
- Gießen Sie das Gel, indem Sie die Agaroselösung in Kammern geeigneter Größe gießen, abhängig von der Anzahl der Bedingungen/Plasmide. Setzen Sie die Waben fest und warten Sie ca. 1 Stunde, bis das Gel verfestigt ist; Entfernen Sie dann die Waben und positionieren Sie das polymerisierte Gel in der entsprechenden Gelschale für die Elektrophorese.
- Mischen Sie einige μL des verdauten Plasmids ("Cut") und 1,5 μL 6x Ladepuffer (enthält einen Farbstoff zur Überwachung der Elektrophorese) in einem Endvolumen von 10 μL nukleasefreiem Wasser. In gleicher Weise wird auch eine separate Kontrollprobe mit dem unverdauten Plasmid ("ungeschnitten") hergestellt. Laden Sie die Proben in die Agarose-Lanes und in die erste Lane eine 1-KB-DNA-Leiter. Führen Sie die DNA-Elektrophorese bei 100 V für 30 min durch.
- Visualisieren und dokumentieren Sie die DNA-Banden, die aus dem Lauf auf einem Standard-UV-Transilluminator resultieren. Überprüfen Sie die Effizienz der Plasmidlinearisierung, indem Sie das Muster der DNA-Banden untersuchen, indem Sie "geschnitten" und "ungeschnitten" vergleichen.
HINWEIS: Ausreichend linearisierte Plasmide sollten schneller als ein scharfes Band laufen. Stellen Sie sicher, dass die DNA-Spaltung abgeschlossen ist, da die Initiierung der Transkriptionsreaktion ein wichtiger limitierender Schritt ist, der durch das Vorhandensein von zirkularisierten Plasmidformen behindert werden kann, was zu einer schlechten mRNA-Ausbeute führt.
- Fahren Sie mit der Aufreinigung des linearisierten Plasmids mit kommerziell erhältlichen DNA-Aufreinigungskits auf Basis von Spin-Säulen fort und lagern Sie das gereinigte DNA-Präparat bis zur Verwendung bei -20 °C.
HINWEIS: Anstelle der Linearisierung ist es möglich, ein PCR-Produkt als Template für die mRNA-Transkription zu verwenden, vorausgesetzt, es enthält die Polymerase-Promotorsequenz stromaufwärts und die polyA-Signalsequenz stromabwärts des Stoppcodons. PCR-Produkte sollten ebenfalls gereinigt und auf einem Agarosegel inspiziert werden, bevor sie fortfahren.
- In vitro mRNA-Transkription, -Reinigung und -Qualitätskontrolle (Tage 2-3) (Abbildung 1A)
HINWEIS: Reinigen Sie den Arbeitsbereich und die Pipetten sorgfältig mit RNAse-Dekontaminationslösungen. Verwendung von nukleasefreien Materialien und Reagenzien; Tragen Sie Handschuhe. Dies minimiert die RNase-Kontamination und ermöglicht eine bessere Ausbeute an mRNA in voller Länge.
Transkribieren Sie gekappte und polyadenylierte mRNAs, die für WT- und mutierte Proteine kodieren, aus linearisierten Plasmiden unter Verwendung von Standardkits für die In-vitro-mRNA-Transkription und gemäß den Anweisungen des Herstellers.- Zentrifugieren Sie das gereinigte linearisierte Plasmid einige Sekunden lang bei 17.949 × g , um sicherzustellen, dass sich die Ablagerungen am Boden des Röhrchens sammeln und nicht in die Transkriptionsreaktion überführt werden.
- Tauen Sie alle Komponenten bei Raumtemperatur (RT) auf, mit Ausnahme der Mischung aus NTP- und CAP-Analoga, die auf Eis aufbewahrt werden. Halten Sie die Polymerase bis zur Verwendung bei -20 °C.
- Alle Reagenzien vor der Verwendung bei 17.949 × g kurz zentrifugieren, um Reagenzienverlust oder versehentliche Kontamination zu vermeiden, und den 10x Reaktionspuffer und das gebrauchsfertige NTP/CAP-Analoggemisch kurz vortexen, bis sie vollständig in Lösung sind. Halten Sie den 10x Reaktionspuffer auf RT.
- Bereiten Sie die Transkriptionsreaktion bei RT vor, indem Sie 600-800 ng des linearisierten und gereinigten Plasmids zu einer Lösung hinzufügen, die 10 μl 2x NTP/CAP-Analoggemisch, 2 μl 10x Reaktionspuffer, 2 μl SP6-, T7- oder T3-Enzym (abhängig vom spezifischen Promotor stromaufwärts des CDS im Plasmid) in einem Endvolumen von 20 μl enthält, das aus nukleasefreiem Wasser besteht. Gründlich mischen, indem Sie ein paar Mal auf und ab pipettieren.
- Die Transkriptionsreaktion wird bei 37 °C in einem Thermocycler inkubiert. Stellen Sie sicher, dass Sie auch die Temperatur des Deckels auf 37 °C einstellen und die Reaktion 2 h lang laufen lassen.
- Um das restliche Plasmid zu entfernen, das während der Reaktion nicht transkribiert wurde, fügen Sie 1 μl Standard-DNAse-Lösung (2 Einheiten/μl) hinzu. Die Reaktion wird durch mehrmaliges Pipettieren gründlich gemischt und weitere 30 Minuten bei 37 °C inkubiert.
- Bevor Sie fortfahren, überprüfen Sie die Qualität und Integrität der in vitro synthetisierten mRNA durch FSME/Formamid-Agarose-Elektrophorese. 1,0 g Agarose in 50 mL 1x FSME-Lösung auflösen, 5,5 mL 37% Formamid und 3,5 μl Farbstoffgel hinzufügen, mischen und gießen Sie das Gel wie oben beschrieben.
ACHTUNG: Tragen Sie beim Umgang mit Formamid persönliche Schutzausrüstung (PSA) und verwenden Sie eine chemische Haube. - Mischen Sie 1 μL der transkribierten mRNA mit 2,5 μL 2x Formamide Dye Loading Buffer in nukleasefreiem Wasser in einem Gesamtvolumen von 5 μL. Mischen Sie die Komponenten durch mehrmaliges Pipettieren gründlich und laden Sie die Proben in die Gelbahnen. Lassen Sie das Gel bei 100 mV für 10-15 min in vorgekühltem 1x TBE-Puffer laufen. Führen Sie auch eine 1 kb DNA- und/oder RNA-Leiter durch.
- Optional: Denaturieren Sie die Leiter und die mRNA bei 70 °C für 10 min.
- Visualisieren und dokumentieren Sie die resultierenden mRNA-Banden auf einem Standard-UV-Transilluminator.
HINWEIS: Lange Läufe und hohe Temperaturen erhöhen die Wahrscheinlichkeit eines RNA-Abbaus. - Wenn die Integrität und Größe der synthetisierten gedeckelten RNA optimal sind, fahren Sie mit der Polyadenylierung des C-Terminus fort, indem Sie der Reaktion Folgendes hinzufügen: 36 μl nukleasefreies Wasser, 20 μl 5x denaturierender Ladepuffer, 10 μl 25 mM MnCl2, 10 μl 10 mM ATP und 4 μl Polyadenylierungsenzym (z. B. gereinigte E. coli Poly(A)-Polymerase). Mischen Sie die Reaktion gründlich, indem Sie sie einige Male pipettieren und inkubieren Sie die 100 μl der endgültigen Reaktion bei 37 °C für weitere 1 h.
HINWEIS: Es ist nicht ratsam, die RNA-Qualität nach der polyA-Reaktion per Gelelektrophorese zu überprüfen, da die RNA aufgrund der polyA-Schwänze verschmiert erscheint. - Die synthetisierte gedeckelte und polyadenylierte mRNA wird mit geeigneten Salzen wie LiCl ausgefällt und zurückgewonnen. Kurz gesagt, gleiche Volumina nukleasefreies Wasser und 2,5 M LiCl-Standardlösung (30 μl) mischen und >30 min bei -20 °C inkubieren.
HINWEIS: Eine effiziente Fällung kann nach 2 Stunden erzielt werden, aber eine Inkubation über Nacht erhöht in der Regel die endgültige Ausbeute an mRNA. - Um die gereinigte mRNA zu pelletieren, zentrifugieren Sie die Reaktion 30 Minuten lang bei 4 °C bei 17.949 × g und entfernen Sie den Überstand. Waschen Sie nun das Pellet mit ~1 mL 70 % Ethanol (EtOH) und zentrifugieren Sie es 15 Minuten lang bei 4 °C bei 17.949 × g , um Verunreinigungen und Nukleotidreste aus der Reaktion zu entfernen.
- Trocknen Sie das Pellet an der Luft und suspendieren Sie es dann wieder in 20 μl nukleasefreiem Wasser. Die RNA durch vorsichtiges und wiederholtes Pipettieren bei RT gut auflösen und bei 10 min inkubieren.
HINWEIS: Überprüfen Sie das Pellet alle 5 Minuten, um sicherzustellen, dass EtOH verdampft. Wenn Sie resuspendieren, pipettieren Sie die RNA angesichts der Klebrigkeit, bis sie richtig in Wasser aufgelöst ist. - Beurteilen Sie die Konzentration und Reinheit der präparierten mRNA durch Messung der Extinktion bei 260 nm und 280 nm (Verhältnis 260/280) in einem Spektralphotometer. Messen Sie verschiedene skalare Verdünnungen des RNA-Präparats, um eine korrekte Konzentrationsschätzung zu gewährleisten.
HINWEIS: Normalerweise liegt die Konzentration der synthetisierten RNA zwischen 1 und 2 μg/ml, aber die endgültige Menge kann je nach CDS-Länge und Menge des Ausgangsmaterials variieren. - Bewahren Sie den mRNA-Bestand bei der Beurteilung der Qualität in Eis auf, aliquotieren Sie ihn dann in 5-μl-Aliquote und lagern Sie ihn bei -80 °C, bis er für die Injektion benötigt wird.
- Vorbereitung von Standardlösungen und -materialien für die Embryoneninjektion und medikamentöse Behandlung (Tage 2-3, Abbildung 1B)
- Ziehen Sie Mikroinjektionsnadeln für Zebrafischembryonen mit sterilen Kapillaren (Standardkapillarabmessungen: 1,0 OD x 0,58 ID x 100 L mm). Verwenden Sie eine handelsübliche Abziehvorrichtung und wenden Sie die gewünschten Einstellungen in Bezug auf die Ausgangsspannung, den Zugmodus (ein oder zwei Schritte) und die Zugkraft an, um eine variable Spitzenlänge, einen Durchmesser und eine Nadelform zu erhalten. Für häufig verwendete Nadelmerkmale für Zebrafische siehe Abdelrahman und Kollegen11.
HINWEIS: Die optimalen Einstellungen können von Labor zu Labor variieren, da Umgebungsbedingungen (z. B. Luftfeuchtigkeit oder Raumtemperatur) die Ergebnisse des Nadelziehens beeinflussen können. Die Einstellungen sollten regelmäßig neu getestet und kalibriert werden. Gezogene Nadeln können bei RT in sauberen Behältern mehrere Monate gelagert werden. - Die 30-fache "Danieau"-Stammlösung bei einem pH-Wert von 7,6 wird durch Auflösen von 101,7 g NaCl, 1,56 g KCl, 2,96 g MgSO4 x 7H2O, 4,25 g Ca(NO3)2 und 35,75 g HEPES in 1 l doppelt destilliertem (DD) Wasser zur Herstellung der Mikroinjektionslösung hergestellt. Bereiten Sie auch eine Stammlösung von Phenolrot mit 5 % in DD-Wasser vor, die als sichtbarer Tracer der Injektionsmischung verwendet werden kann.
- Bereiten Sie die E3-Mediumlösung für das Embryowachstum vor, indem Sie 4 ml NaCl (5 M), 680 μl KCl (1 M), 1,32 ml CaCl2 x 2H2O (1 M) und 1,32 ml MgSO4 (1 M) in einem Endvolumen von 4 l Umkehrosmosewasser verdünnen.
- Herstellung von Stammlösungen der gewünschten Arzneimittel (in diesem Beispiel: der MEK-Inhibitor [MEKi] PD032590) als Stammlösungen gemäß Produktdatenblatt und Löslichkeit (10 mM des MEKi der Wahl durch Auflösen von 5 mg in 1.036 mL DMSO). Gründlich mischen und kleine Aliquots erzeugen, die bis zur Verwendung bei -80 °C gelagert werden.
HINWEIS: DMSO ist ein gängiges Lösungsmittel, das zum Auflösen von polaren und unpolaren Verbindungen verwendet wird und in einer Vielzahl von organischen Lösungsmitteln sowie in Wasser löslich ist. - Bereiten Sie eine lebende Salinengarnelensuspension vor, indem Sie Artemia salina-Zysten in Schlupflösung (2 g/l Artemia salina-Zysten in 30 g/l Meersalz, das zuvor in Umkehrosmosewasser gelöst wurde) bei 28-30 °C für mindestens 18 h schlüpfen lassen und dabei für eine konstante Belüftung sorgen.
HINWEIS: Die Kultivierungsbedingungen und die Schlupfzeit können durch die Umgebungsbedingungen der Einrichtung und die Zystenmengen beeinflusst werden und sollten regelmäßig erneut getestet und bei Bedarf neu eingestellt werden.
- Ziehen Sie Mikroinjektionsnadeln für Zebrafischembryonen mit sterilen Kapillaren (Standardkapillarabmessungen: 1,0 OD x 0,58 ID x 100 L mm). Verwenden Sie eine handelsübliche Abziehvorrichtung und wenden Sie die gewünschten Einstellungen in Bezug auf die Ausgangsspannung, den Zugmodus (ein oder zwei Schritte) und die Zugkraft an, um eine variable Spitzenlänge, einen Durchmesser und eine Nadelform zu erhalten. Für häufig verwendete Nadelmerkmale für Zebrafische siehe Abdelrahman und Kollegen11.
- Vorbereitung von Zuchtpaaren für Tg[ef1α:ERK biosensor-nes] (Teen) Fische (Tag 3)
- Füttern Sie die erwachsenen Paare am Tag der Paarung regelmäßig gemäß dem genehmigten Protokoll der Tiereinrichtung.
- Reinigen Sie die Kultur mit geschlüpften Salinengarnelen, indem Sie die nicht geschlüpften oder leeren Zysten mit zwei Sieben unterschiedlicher Filtergröße herausfiltern (Filterstufe I: 180 μm, Filterstufe II: 112 μm).
- Sammeln und waschen Sie die gefilterten Salinengarnelen in 200 ml Umkehrosmosewasser, um 10-15 erwachsene Fische mit etwa 5-10 ml der Salinengarnelenlösung zu füttern.
HINWEIS: Überprüfen Sie die Qualität der geschlüpften Salinengarnelenlösung anhand ihrer Vitalität, Beweglichkeit, Größe und Farbe und achten Sie darauf, nicht geschlüpfte oder leere Zysten zu entfernen, die für Fische unverdaulich sind und den Magen-Darm-Trakt der Fische schädigen können. Für Brutpaare ist es vorzuziehen, die letzte Mahlzeit des Tages mindestens 3 Stunden vor der Isolierung der Paare im Brutbecken zu verabreichen, um die Verdauung der Nahrung zu ermöglichen und eine hohe Wasserqualität zu erhalten.
- Wählen Sie Fischpaare aus, die in den letzten 2 Wochen nicht zur Paarung gepaart wurden, um den Stress zu minimieren und die Entwicklung der Gameten zu ermöglichen. Akklimatisieren Sie die ausgewählten erwachsenen Teen-Fischpaare (normalerweise mit dieser Zusammensetzung: 1 ♂ : 2 ♀ ) in entsprechenden Zuchtbecken. Trennen Sie Weibchen von Männchen mit einer Tanktrennwand bis zum nächsten Morgen. Stellen Sie einen standardmäßigen Hell-Dunkel-Zyklus von 14/10 h sicher.
HINWEIS: Es können auch Gruppenkreuze angeordnet werden. In diesem Fall kann das Laichen jedoch zu unterschiedlichen Zeiten erfolgen und die Auswahl von Embryonen im gleichen Stadium aus gemischten Gelegen wird schwieriger. Der Übergang von dunkel zu hell ist entscheidend für eine erfolgreiche Paarung.
- Füttern Sie die erwachsenen Paare am Tag der Paarung regelmäßig gemäß dem genehmigten Protokoll der Tiereinrichtung.
- Embryonenentnahme und Mikroinjektion von WT und mutierten mRNAs (Tag 4)
- Sobald das Licht in der Anlage eingeschaltet ist, entfernen Sie die Tanktrennwand, die die Männchen und Weibchen trennt, und wechseln Sie gegebenenfalls ca. 20 % des Aquarienwassers durch frisches Wasser aus dem Aquakulturkreislaufsystem, um die Wasserqualität hoch zu halten, ohne die von den Fischen freigesetzten Hormone übermäßig zu verdünnen.
- Lassen Sie den Fisch ungestört und kontrollieren Sie regelmäßig die Eiablage (in der Regel in den ersten 30 min bis 1 h Tageslicht). Die Eier (2-3 mm) werden abgelegt und fallen auf den Boden des Beckens, der durch ein Gitter getrennt ist, so dass die Erwachsenen sie nicht essen können. Isolieren Sie die adulten Tiere, die erfolgreich gebrütet haben, und seihen Sie das Aquarienwasser mit den Eiern durch ein Standardsieb (z. B. ein Teesieb), um die Eier aufzubewahren.
HINWEIS: Es ist möglich, denselben Fisch für eine weitere Paarungsrunde wieder in das Zuchtbecken zu setzen oder den Fisch in die ursprünglichen Haltungstanks zurückzubringen, wobei das Paar, das Datum und die Leistung aufgezeichnet werden. - Waschen Sie die entnommenen Embryonen mit frischem E3-Medium und entfernen Sie unbefruchtete, degenerierte Eizellen, Kot und andere Arten von Ablagerungen von den Paaren mit einer Pasteurpipette.
HINWEIS: Sammeln Sie ~n = 100 befruchtete Teen-Eier in einer Petrischale mit einem Durchmesser von 90 mm, um eine Überpopulation der Embryonen zu vermeiden. - Ordnen Sie befruchtete Teenager-Eier in einer benutzerdefinierten Mikroinjektionsplatte an, die durch Formen von 2 % Agarose in E3-Medium erhalten wird, um geeignete Lanes zu erzeugen, die die Eier während der Mikroinjektion enthalten und einschränken müssen.
HINWEIS: Zu diesem Zweck können kundenspezifische oder kommerzielle Formen verwendet werden. - Bereiten Sie eine frische Mikroinjektionsmischung vor, um mutierte Embryonen wie folgt zu erzeugen: 30-60 μg verschlossene und polyadenylierte mRNA (hier shp2), gelöst in 0,3x Danieau-Lösung (verdünnt aus Stammlösung) für ein Endvolumen von 20 μl. Fügen Sie 0,2 μl (0,05%) Phenolrot-Stammlösung als Mikroinjektionstracer hinzu.
- Laden Sie die Nadel mit 2 μl Injektionsmaterial mit einer Microloader-Pipette zurück. Injizieren Sie die Lösung mit einem handelsüblichen Druck-Mikroinjektionsgerät in das Einzelzellstadium von Teenager-Zebrafischembryonen und passen Sie die Druck- und Zeiteinstellungen manuell an, um jede Injektion basierend auf der Nadel- und Embryoqualität zu kalibrieren. Beziehen Sie sich auf die in der Literatur verfügbaren Standard-Mikroinjektionsprotokolle für Zebrafische11,12.
- Züchten Sie mikroinjizierte Teen-Embryonen unter kontrollierten Haltungsbedingungen (Temperatur: 28 °C, Luftfeuchtigkeit: 70%, Hell-Dunkel-Zyklus: 14/10 h) für eine optimale Entwicklung und reinigen Sie alle Eizellen, die trüb oder degeneriert aussehen, in den nächsten 3 Stunden nach der Ablagerung.
- Untersuchen Sie die Embryonen ~4 h nach der Befruchtung (hpf) auf Fluoreszenz (Reporterexpression) mit einem Standard-Fluoreszenz-Stereomikroskop mit geeigneten Lampen- und Filtereinstellungen (465-500 nm). Verwenden Sie Teen-positive Fische für die FRET-Bildgebung und negative Geschwister für immunhistochemische (IHC) Untersuchungen (siehe unten).
HINWEIS: Angesichts der bekannten Variabilität in der Expression von Tol2-basierten Transgenen13 können Embryonen unterschiedliche Fluoreszenzniveaus von Teen aufweisen. Es wird empfohlen, nicht injizierte Geschwister zu untersuchen, um die interindividuelle Variabilität der Fluoreszenz innerhalb einer Charge zu kontrollieren, und unter Berücksichtigung des geringen Dynamikbereichs der FRET-Bildgebung Embryonen mit extrem niedrigen basalen Fluoreszenzwerten zu verwerfen.
- Behandlung von Zebrafischembryonen mit dem MEK-Inhibitor (MEKi) (Tag 4)
- Bereiten Sie eine Zwischenlösung von MEKi PD0325901 (1 mM) vor, indem Sie 1 μl 10 mM Stammlösung mit 9 μl mit E3-Medium verdünnen. Verwenden Sie die Zwischenlösung, um endgültige Lösungen mit niedriger Dosis zu erhalten. Bereiten Sie eine niedrige Dosis (0,25 μM) des MEKi PD0325901 in einem Gesamtvolumen von 3 mL vor, indem Sie 0,75 μl der 1 mM Zwischenlösung mit 2,25 μl E3-Medium mischen. 0,75 μl 10 % DMSO mit 2,25 ml E3-Medium verdünnen, um die Kontrolllösung (Strg) zu erhalten.
- Platzieren Sie gleich viele Embryopools in verschiedenen Vertiefungen einer 6-Well-Platte und beginnen Sie die Behandlung durch Eintauchen in das Bad: Tauschen Sie für jede Vertiefung das E3-Medium gegen ein E3-Medium aus, das die Vehikelkontrolle (0,0025 % DMSO) oder verdünnte Arzneimittel in der gewünschten Konzentration (0,25 μM, 0,0025 % DMSO) enthält, wie zuvor erwähnt. Verwenden Sie 3 ml Arzneimittellösung pro Vertiefung.
HINWEIS: Um Auswirkungen zu vermeiden, die durch unterschiedliche Konzentrationen des Trägers (DMSO) verursacht werden, ist es wichtig sicherzustellen, dass sowohl die Kontroll- als auch die Behandlungslösung die gleiche DMSO-Endkonzentration aufweisen. - Halten Sie die behandelten Embryonen bei 28 °C, bevor Sie sie bis zum gewünschten Entwicklungsstadium für Folgeuntersuchungen (hier: morphometrische Analyse und IHC-Validierung) entnehmen.
2. Multispektrale Live-FRET-Bildgebung von RASopathy-Zebrafischmodellen im Gastrula-Stadium und Datenanalyse
- Einbetten von Gastrulen für die Live-Bildgebung (Tag 4) (Abbildung 2A)
- Bereiten Sie Eindeckmedium für lebende Embryonen vor, indem Sie 1,5 g niedrigschmelzende Agarose (LMA) in 1x PBS auflösen, um 1,5% LMA/E3 herzustellen.
HINWEIS: Wählen Sie die Agarosekonzentration zwischen 0,8 und 1,5 %, abhängig vom Entwicklungsstadium und der spezifischen Notwendigkeit, den Fisch zu bändigen. Verteilen Sie die frisch hergestellte LMA-Lösung in Aliquots des gewünschten Formats und lagern Sie LMA in polymerisiertem Zustand bei RT (einige Monate haltbar). Dadurch wird die Kontamination mit Bakterien und Pilzen minimiert, aber überprüfen Sie die Qualität des LMA-Aliquots regelmäßig vor der Anwendung. - Vor der Probenaufnahme das 1,5%ige LMA-Aliquot in einem Thermomischer oder einem sterilen Wasserbad bei 50 °C schmelzen. Nach dem Auflösen die Temperatur des Thermomischers auf ca. 30 °C senken.
- Positionieren Sie einen einzelnen injizierten Teen+ Embryo (hier eine Gastrula, die ShpD61G exprimiert) in der Mitte einer Glasbodenschale mit einem Durchmesser von 35 mm für die Live-Bildgebung und richten Sie ihn mit einem dünnen Haar aus. Entfernen Sie das überschüssige E3-Medium und immobilisieren Sie den Embryo, indem Sie einen Tropfen LMA darauf geben und ihn bei RT polymerisieren lassen.
HINWEIS: Zu Beginn des Polymerisationsprozesses kann der Fisch in die gewünschte Position gebracht werden. Es wird strengstens empfohlen, das LMA bei einer Temperatur von nicht mehr als 35 °C zu verwenden, um Gewebeschäden zu vermeiden.
- Bereiten Sie Eindeckmedium für lebende Embryonen vor, indem Sie 1,5 g niedrigschmelzende Agarose (LMA) in 1x PBS auflösen, um 1,5% LMA/E3 herzustellen.
- Einstellen von Mikroskopieparametern und Durchführen einer multispektralen FRET-Bildgebung von Gastrulae (Tag 4) (Abbildung 2B)
HINWEIS: Das Protokoll gilt für die Verwendung von konfokalen Mikroskopieplattformen, die mit der gesamten erforderlichen Hard- und Software für die Durchführung der FRET-Bildgebung ausgestattet sind. Hier verwendeten wir ein konfokales Mikroskop, das mit einem Argon-Ionen-Laser mit den Wellenlängenlinien von 458-476-488-496-514 nm, einem programmierbaren akusto-optischen Strahlteiler (AOBS), der Anregungs- und Emissionslicht trennt, zwei spektralen Hybriddetektoren (HyD) und einem Tischinkubator ausgestattet ist, um stabile Temperatur- (bei 28 °C) und Luftfeuchtigkeit während der Live-Bildgebung von Proben aufrechtzuerhalten.
Das beschriebene multispektrale FRET-Live-Bildgebungsprotokoll kann auf Embryonen in verschiedenen Entwicklungsstadien jenseits der frühen Gastrulae angewendet werden, wie Fasano et al. gezeigt haben.7. Für spätere Entwicklungsphasen sollte ein größerer Z-Stack (z. B. 24 hpf) sowie geeignete Einstellungen für Z- und T-Intervalle in Betracht gezogen werden, um multispektrale Detektionsschritte während der spektralen FRET-Bildaufnahme zu ermöglichen.- Schalten Sie den Inkubatorregler mindestens 1 Stunde vor Beginn der Aufnahme ein und stellen Sie die Temperatur auf 28 °C ein, um die Zebrafischembryonen in einem gesunden Zustand zu halten. Sobald sich die Temperatur des Inkubators stabilisiert hat, platzieren Sie die Bildgebungsschale mit dem Embryo auf dem Probenhalter und verwenden Sie ein 10x/Dry-Objektiv (numerische Apertur von 0,4), um die Probe schnell zu visualisieren.
- Schalten Sie in der Laserkonfiguration der referenzierten Software den Argon-Ionen-Laser ein und stellen Sie mit dem Schieberegler die Laserleistung auf 50% ein. Wählen Sie in den Hardwareeinstellungen die Tiefenauflösung von 8 Bit , mit der die Bilder aufgenommen werden.
- Wählen Sie im Erfassungsfenster den Scanmodus für die spektrale Detektion XYλZ und stellen Sie die folgenden Erfassungsparameter ein: Bildformat: 512 x 512 px; Scangeschwindigkeit von 400 Hz; optischer Zoom von 0,75.
- Aktivieren Sie die 458 nm Laserlinie des Argon-Ionen-Lasers und stellen Sie deren Intensitätswert ein, normalerweise <10% (hier 8,5%).
- Wählen Sie einen HyD-Detektor aus und stellen Sie die Empfindlichkeit des Detektors (Gain) ein, indem Sie den gewünschten Wert (hier 500) eingeben.
HINWEIS: Für lebende, schwach fluoreszierende Proben und um weniger Hintergrundrauschen zu erzeugen, ist es ratsam, Hybriddetektoren anstelle von Photomultiplier-Röhren (PMTs) zu verwenden.
In der referenzierten Software muss für die Darstellung des Emissionsspektrums eines Farbstoffs ein Detektor aktiviert werden. - Aktivieren Sie zunächst den ersten Detektor (HyD) und wählen Sie das Cyan-Fluoreszenzprotein (CFP) aus, um seine Emissionskurve anzuzeigen. Öffnen Sie das Dropdown-Menü der Detektorleiste der Software, um die Auswahlliste für die CFP-Emissionskurve zu öffnen. Aktivieren Sie nun einen zweiten Detektor, um die Emissionskurve des zweiten Farbstoffs, des gelben Fluoreszenzproteins (YFP), anzuzeigen. Um auch die Ypet-Emissionskurve anzuzeigen, aktivieren Sie einen zweiten Detektor und wählen Sie dann die YFP-Emissionskurve aus, die nach dem Ausschalten des zweiten Detektors im Spektrumbild angezeigt wird.
HINWEIS: Sobald dieser Schritt abgeschlossen ist, sollte der zweite Detektor deaktiviert werden, da nur ein Detektor im spektralen Erfassungsmodus verwendet wird. - Um Live-Aufnahmen zu starten, positionieren Sie den Detektionscursor im Bereich der intensivsten Signalemission (in diesem Fall die von YFP), um die Probe zu visualisieren, und legen Sie die Start- und Endpositionen der Probendicke im z-stack LAS X-Fenster fest.
- Legen Sie für die Eigenschaften des λ-Scan-Bereichs die folgenden Parameter fest: Anfang (460 nm) und Ende (570 nm) des Erfassungsbereichs; Detektionsbandbreite: 5 nm; λ-Scan Schrittweite: 5 nm. Starten Sie die Z-Stack-Erfassung.
HINWEIS: Die angegebenen Parameter ermöglichen es dem Benutzer, eine relativ schnelle Abbildung mit akzeptabler Auflösung zu erhalten, es können jedoch auch andere Einstellungen verwendet werden. In den Einstellungen der Fluorifierscheibe kann der automatisch eingefügte NOTCH-Filter abgewählt werden, um einen einzelnen Intensitätsverlust zu vermeiden. - Um Echtzeit-Signaländerungen nach MEKi-Exposition zu beurteilen, montieren Sie einen einzelnen mutierten Embryo (hier Shp2D61G) in eine Glasschale und fotografieren Sie ihn vor und nach der medikamentösen Behandlung. Für diese Live-Experimente führen Sie während der Bildgebungssitzung direkt eine Arzneimittelbehandlung durch Eintauchen in ein Bad mit maximal 2 ml der Lösung durch, die das gelöste Arzneimittel enthält.
- Bildnachbearbeitung und multispektrale Farbstofftrennung zur Gewinnung ratiometrischer FRET-Bilder (Tag 5) (Abbildung 2C)
- Um die Donor- (CFP) und Akzeptor- (Ypet) Emission zu trennen, stellen Sie sicher, dass der Beitrag der Fluoreszenzemission beider Moleküle im FRET-Kanal, der als spektraler Durchlass (SBT) bezeichnet wird, eliminiert wird.
- In öffentlich zugänglichen Datenbanken für Farbstoff-/Fluoreszenzproteine können Sie die .cvs-Datei relativ zu einem Standard-CFP-Emissionsspektrum (Anregungs- und Emissionsspektren) anzeigen und herunterladen.
- Wählen Sie im Bereich "Daten" der CVS-Datei die Option "Text in Spalten " aus und teilen Sie die Daten mit einem Komma als Trennzeichen in Spalten auf. Die resultierende Datei enthält zusätzliche Informationen (z. B. CFP-Anregung, CFP 2P), die entfernt werden sollten. Behalten Sie nur die Spaltendaten relativ zu Wellenlänge und Emission bei.
- Entfernen Sie in der Spalte CFP-Emission alle Intensitätsdaten ab 501 nm.
- Speichern Sie die angepasste CFP-Emissionsspektrumdatei von 460 bis 500 nm in .xls Format (in Abbildung 2C als "eCFP modified" bezeichnet).
- Wiederholen Sie den gleichen Vorgang, um das YFP-Proteinemissionsspektrum herunterzuladen und zu verarbeiten, und behalten Sie nur die Spaltendaten relativ zu Wellenlänge und Emission bei. Entfernen Sie alle Emissionswerte bis zu 524 nm. Speichern Sie die angepasste YFP-Emissionsspektrumdatei von 525 bis 650 nm in.xls Format (in Abbildung 2C als "Ypet ab 525 nm" bezeichnet).
- Um den Ausschluss von spektralem Durchscheinen (SBT) in der Farbstoffdatenbank zu ermöglichen, die im Konfigurationsfenster der Software verfügbar ist, fügen Sie zwei angepasste Referenzemissionsspektren für CFP und YFP ein und speichern Sie sie.
- Wählen Sie die resultierende Spektralbilddatei aus der Imaging-Sitzung aus, öffnen Sie das Prozessfenster und wählen Sie im Farbseparationswerkzeug die Option "Spektrale Farbstofftrennung". Konfigurieren Sie die Einstellungen für die Farbstofftrennung wie folgt: Wählen Sie in den Dropdown-Listen auf der linken Seite des Dialogs an erster Stelle das neue CFP-Emissionsspektrum (eCFP modifiziert) und das neue YFP-Emissionsspektrum (Ypet ab 525 nm) aus der Spektrumdatenbank aus.
- Wählen Sie unter Rescale die Option Pro Kanal aus, um die Kanäle einzeln zu skalieren, und wählen Sie dann im λ-Scan der Bilder denjenigen mit der größten Signalintensität aus (entsprechend Schritt 14 des Spektralscans auf dem Niveau der FRET-Kanal-Emissionsspitze, gekennzeichnet durch einen weißen Pfeil in der Tafel mit den Detektionsschritten rechts in Abbildung 2B). Bewegen Sie sich dann entlang des Z-Scans der Probe und wählen Sie den optischen Abschnitt aus, der den Interessenbereich im Margin-Bereich hervorhebt.
- Verwenden Sie den ROI-Auswahlmodus in der Randzone des Tiermastes, um den Bereich mit dem besten Spektrum zu definieren. Klicken Sie auf das oben im Anzeigefenster angezeigte ROICrosshair, um das Fadenkreuz aufzurufen und die Größe des Referenz-ROI anzupassen, indem Sie im Messbereich den Wert von 40 Voxeln eingeben. Definieren Sie genau den Interessenbereich durch den ROICrosshair. Wählen Sie im Bilddiagramm die Option Datennormalisierung und klicken Sie dann auf Anwenden, um die Farbstofftrennung mit den konfigurierten Einstellungen durchzuführen.
- Öffnen Sie diese neu generierte Datei, die mit den beiden Kanälen getrennt erhalten wurde, und erzeugen Sie ein zweidimensionales Projektionsbild aus der dreidimensionalen Bildserie (Projektion mit maximaler Intensität) zur Datenvisualisierung.
- Wählen Sie im Prozessfenster die Option "Zuschneiden ", um die Kanäle in zwei separate Dateien zu trennen, den Kanal CH1 und den Kanal CH2 (oder FRET).
- Wählen Sie im Prozessfenster die Option Bilder kombinieren aus, wählen Sie die CH2-Datei aus, und fügen Sie sie in die erste Option ein, und wählen Sie dann die CH1-Datei aus, und fügen Sie sie in die zweite Option ein. Legen Sie eine Neuskalierung mit Faktor 5 fest, und wählen Sie die Operation Verhältnis. Klicken Sie dann auf Übernehmen , um die neue Datei mit dem ratiometrischen Bild (YFP/CFP) zu generieren. Speichern Sie die Datei für die anschließende Datenanalyse.
- Um die Donor- (CFP) und Akzeptor- (Ypet) Emission zu trennen, stellen Sie sicher, dass der Beitrag der Fluoreszenzemission beider Moleküle im FRET-Kanal, der als spektraler Durchlass (SBT) bezeichnet wird, eliminiert wird.
- Quantitative Analyse von FRET-Signalen und Bildwiedergabe (Tage 5-6) (Abbildung 2D)
- Für quantitative Messungen von FRET-Signalen aus ratiometrischen FRET/CFP-Bildern führen Sie das Experiment an N>2-Embryonen (Replikaten) durch, abhängig von einer a priori statistischen Bewertung auf der Grundlage des erwarteten Effekts. Verwenden Sie ein Bildverarbeitungspaket wie die Open-Source-Software Fiji. Importieren Sie die Bilddateien, die aus der spektralen Bildgebung und der Farbstofftrennung gewonnen wurden, in ein Bildanalysepaket, wie z. B. das Open-Source-Projekt Fiji.
- Bevor Sie mit der ROI-Auswahl der Gastrulae-Bilder beginnen, stellen Sie die Parameter mit der Funktion Analysieren | Maße einstellen | Wählen Sie die relevanten Parameter aus, z. B. Fläche, Integrierte Dichte und Mittlerer Grauwert.
- Wählen Sie den interessierenden Bereich (in dieser speziellen Studie: den Rand der Gastrula) mit dem Polygonauswahlwerkzeug aus der Symbolleiste aus. Speichern Sie die ROI x,y-Spezifikationen, indem Sie auf Analysieren | Werkzeuge | ROI-Manager.
- Um Messungen in einem ausgewählten ROI durchzuführen, klicken Sie auf Analysieren | Messen. Wiederholen Sie diese Schritte für jeden ROI und jedes Bild/jede Bedingung.
HINWEIS: Speichern Sie das Bild bei Bedarf unter . TIFF-Format. Es ist möglich, alle ROI-Messungen, die von mehreren Bildern abgeleitet wurden, in einer ROI-Datei zu speichern. Benennen Sie jede Kennzahl in der ROI-Manager-Liste mit dem korrekten Namen relativ zu jedem Beispiel/Bild um. - Extrahieren Sie für jede Bedingung (hier Mutante und Mutante + Behandlung) alle Werte des FRET/CFP-Verhältnisses für jeden ausgewählten ROI und organisieren Sie sie in einem Arbeitsblatt, indem Sie z. B. experimentelle Gruppen in Spalten und jeden Rohwert in Zeilen organisieren.
- Verwenden Sie eine geeignete Software, um statistische Unterschiede von FRET-Signalen zwischen verschiedenen experimentellen Bedingungen zu bewerten und für die Erstellung von Diagrammen und die Datenvisualisierung.
HINWEIS: Open-Source- und lizenzierte Software sind für Datenanalyse- und Grafiklösungen verfügbar. - Organisieren Sie ein spezifisches Projekt für die statistische Bewertung unter Berücksichtigung aller Rohmessungen und der Anzahl der Wiederholungen für jede Versuchsbedingung. Für die statistische Analyse eines einzelnen biologischen Replikats erstellen Sie eine Spaltentabelle mit einer Gruppierungsvariablen, wobei jede Gruppe durch eine Spalte definiert wird. Erstellen Sie für die statistische Analyse von mehr als einem biologischen Replikat gruppierte Tabellen mit mindestens zwei Gruppierungsvariablen, von denen eine durch Spalten (z. B. Versuchsbedingungen) und die andere durch Zeilen (z. B. Mittelwerte von Replikaten) definiert ist.
- Nachdem Sie die Versuchsgruppen im Arbeitsblatt entsprechend angeordnet haben, beurteilen Sie die Datenverteilung (Normalitätstest, z. B. D'Agostino-Pearson, Anderson-Darling) und wählen Sie auf der Grundlage des Ergebnisses den am besten geeigneten statistischen Test aus.
- Führen Sie eine unidirektionale ANOVA für parametrische Daten (nach Gaußscher Verteilung) und einen Kruskal-Wallis-Test für nicht-parametrische Daten durch. Wenn mehr Faktoren vorhanden sind (genetische und Arzneimittelkonzentrationen), sollten Sie eine Zwei-Wege-ANOVA in Betracht ziehen. Vergleichen Sie den mittleren FRET-Wert für jede Bedingung miteinander (Mehrfachvergleiche) und korrigieren Sie Mehrfachvergleiche mit einem Post-hoc-Test (z. B. Tukey-Test und Dunn-Test für parametrische bzw. nicht-parametrische Daten).
3. IHC-Validierung der FRET-Ergebnisse und korrelative morphometrische Analyse von Gastrulationsdefekten
- Vorbereitung der Lösungen und Embryofixierung und -einbettung für IHC gegen tERK und pERK (Tag 7) (Abbildung 3A)
- Bereiten Sie die für die IHC erforderlichen Arbeitslösungen vor.
- Bereiten Sie eine Stammlösung von 10x PBS her, indem Sie 80 g NaCl, 2 g KCl, 14,4 g Na2HPO4 und 2,4 g KH2PO4 in 1 l DD Wasser auflösen. Autoklavieren Sie die Lösung, um sie steril zu halten.
- Bereiten Sie eine 0,8%ige PBS-Triton (PBSTr)-Lösung her, indem Sie 8 mL 10% Triton X-100 in einem Endvolumen von 100 mL 1x PBS auflösen.
- Bereiten Sie 20 %, 50 % und 80 % ige Glycerinlösungen vor, indem Sie 3 ml, 7,5 ml bzw. 12 ml 100 % Glycerin in einem Endvolumen von 15 ml 1x PBS verdünnen.
- Bereiten Sie 4 % Paraformaldehyd (PFA)/0,25 % PBSTr (Fixierlösung) vor, indem Sie 2,5 ml 16 % PFA in 250 μl 10 % Triton X-100 mischen, das zuvor in einem Endvolumen von 10 ml 1x PBS gelöst wurde. Verwenden Sie diese Lösung, um Embryonen bei 6 hpf über Nacht bei 4 °C zu fixieren. Verwenden Sie 2 ml-Röhrchen und maximal fünf Embryonen pro Röhrchen, um eine effiziente Fixierung und Reinigung zu ermöglichen.
HINWEIS: Es wird empfohlen, für jede Fixierrunde frisches Fixiermittel zuzubereiten. ACHTUNG: Tragen Sie beim Umgang mit Formaldehyd mit einem Anteil von 16 % persönliche Schutzausrüstung (PSA) und unter einer chemischen Haube. - Waschen Sie die Embryonen in den 2-ml-Röhrchen, indem Sie die Röhrchen einige Male mit 1,5 ml 0,8 % PBSTr füllen. Übertragen Sie die Embryonen in neue 2-ml-Röhrchen und lagern Sie die Embryonen bis zur Verwendung in 1x PBS.
HINWEIS: Wenn Sie mit dem Umgang mit frühen Zebrafischembryonen nicht vertraut sind, arbeiten Sie unter einem Stereomikroskop, um Embryoverluste während des Pipettierens zu vermeiden. Verwenden Sie Kunststoffe mit geringer Bindung, um eine mögliche Klebrigkeit der Embryonen an der Eileiterwand zu verringern.
- Bereiten Sie die für die IHC erforderlichen Arbeitslösungen vor.
- Gewebepermeabilisierung und IHC gegen pERK und tERK (Tage 7-9)
HINWEIS: Die IHC-Ergebnisse hängen von mehreren Variablen ab, die sich von Labor zu Labor unterscheiden können, und eine spezifische Optimierung der Protokolle kann erforderlich sein.- Waschen Sie die fixierten Zebrafischembryonen in 1 mL 0,8 % PBSTr für 3 x 10 min bei RT.
- Permeabilisieren Sie die Proben mit 2 μl Proteinase K, verdünnt in 0,8 % PBSTr (1:1.000) für 2 min bei RT.
- Stoppen Sie die Permeabilisierung des Gewebes, indem Sie die Proben 3 x 10 Minuten lang in 1 ml 0,8 % PBSTr waschen und die Proben 20 Minuten lang in 500 μl 4 % PFA/1x PBS bei RT postfixieren. Waschen Sie dann die Proben in 1 mL 0,8 % PBSTr für 3 x 10 Minuten.
HINWEIS: Die Methode und die Zeit, die für die Gewebepermeabilisierung benötigt wird, sind kritische Schritte, die durch den Antigentyp, die Qualität der Gewebekonservierung und Umweltfaktoren beeinflusst werden können. Optimieren Sie das Protokoll, bevor Sie es auf wertvolle Proben anwenden. - Die Proben werden mit 1x Blockierungspuffer (BB) inkubiert, der 5 % normales Ziegenserum (NGS), 1 % Rinderserumalbumin (BSA) und 1 % DMSO enthält, hergestellt wie folgt: 75 μl 100 % NGS, 150 μl 10 % BSA und 15 μl 100 % DMSO in einem Endvolumen von 1,5 ml 0,8 % PBSTr. Verwenden Sie 200 μl der Lösung pro Röhrchen und inkubieren Sie die Embryonen 20-30 Minuten lang bei RT in der Lösung.
- Entfernen Sie das 1x BB aus dem vorherigen Schritt und inkubieren Sie die Proben in einer Lösung, die eine Mischung der gewünschten Primärantikörper enthält (hier: Verdünnung von 1 μL des primären monoklonalen Maus-Antikörpers p44/42 MAPK, Gesamt-ERK, tERK, + 1 μL polyklonaler Kaninchenphospho-p44/42 MAPK, phosphoryliertes ERK, pERK) in 250 μL frisch zubereiteter 1x BB-Lösung. Verwenden Sie 200 μl der Lösung pro Röhrchen und inkubieren Sie die Embryonen über Nacht bei 20 °C in der Lösung.
- Um unspezifische Bindungen zu vermeiden, waschen Sie die Proben mehrmals mit 1 mL 0,8 % PBSTr (zuerst alle 10 min und dann alle 30 min).
- Inkubieren Sie die Proben in frisch zubereitetem 1x BB für 20 min bei RT. Verwenden Sie 200 μL pro Röhrchen.
- Entfernen Sie das 1x BB aus dem vorherigen Schritt und inkubieren Sie die Proben mit einer Mischung der Sekundärantikörper (1 μl fluoreszierend 488 konjugiertes Ziegen-Anti-Maus, + 1 μl fluoreszierend konjugiertes 633 Ziegen-Anti-Kaninchen in 600 μl 1x BB-Lösung). Lassen Sie die Embryonen über Nacht bei 20 °C leicht schütteln.
HINWEIS: Wir führten die IHC an negativen (nicht transgenen) Teen-Fischen aus der gleichen Charge durch, die auch für die FRET-Bildgebung verwendet wurde. Alternativ kann auch Teen+ Fisch verwendet werden, aber in diesem Fall sollten stattdessen Sekundärantikörper verwendet werden, die an Fluorophore konjugiert sind und sich nicht mit den Emissionsspektren von CFP oder YFP überlappen. - Beseitigen Sie unspezifische Bindungen, wie oben beschrieben.
- Waschen Sie die Proben in einem Gradienten aus Glycerin/1x PBS-Lösungen (20 %, 50 %, 80 %) für 15 Minuten pro Lösung bei RT. Lagern Sie die Proben in 90 % Glycerin/1x PBS bei 4 °C.
HINWEIS: Um einen möglichen Fluoreszenzzerfall zu minimieren, lagern Sie die Proben im Dunkeln.
- Konfokale Mikroskopie von immungefärbtem Gewebe und quantitative Analyse (Tage 10-11) (Abbildung 3B-E)
- Montieren Sie die Embryonen wie zuvor beschrieben.
- Stellen Sie die Erfassungsparameter für die konfokale Mikroskopie wie folgt ein: weißer Laser auf 80 %, Fluoreszenzemissionsbereich auf 507 - 551 nm für fluoreszierend konjugierte Ziegen-Anti-Maus (in diesem Fall mit dem Emissionsspektrum von 488 nm, verwendet für tERK), 644 - 740 nm für fluoreszierend konjugierte Ziegen-Anti-Kaninchen (in diesem Fall mit dem Emissionsspektrum von 633 nm, verwendet für pERK). Wählen Sie den sequenziellen Aufnahmemodus , ein Scanformat von 512 x 512 px und eine Geschwindigkeit von 400 Hz. Definieren Sie Start und Ende des Scans mit einer Z-Schritt-Größe von 4 - 5 μm und einem Standard-Digitalzoom von 1. Verwenden Sie ein 25-faches Wasserimmersionsobjektiv (mit einer numerischen Apertur von 0,05), um die gesamte Gastrula bei 6 hpf zu erfassen
ACHTUNG: Dieses Protokoll beinhaltet die Anwendung von Lasern, die schädlich sein können; Daher ist eine Schulung des Personals gemäß den spezifischen nationalen Anforderungen erforderlich. - Überprüfen Sie nach der konfokalen Bildaufnahme die Rohbilddatei, die mit einem Bildverarbeitungspaket wie der Open-Source-Software Fiji erhalten wurde.
- In Fidschi verwenden Sie den Befehl Kanäle teilen aus dem Menü Bild und dem Untermenü Farbe , um ein Einzelkanalbild zu erhalten (Gesamt-ERK: Kanal = 0, C = 0; pERK: Kanal = 1, C = 1) und erzeugen Sie ein Z-Projektionsbild mit maximaler Intensität für beide Kanäle, indem Sie auf Bild | Stapel | Z-Projekt.
- Wiederholen Sie das Verfahren für ROI-Intensitätsmessungen wie zuvor beschrieben, und extrahieren Sie die Daten in ein Arbeitsblatt.
- Um auf Änderungen der p-ERK-Signalintensität des gewünschten ROI zwischen Versuchsgruppen zu schließen, berechnen Sie das p-ERK/t-ERK-Verhältnis, indem Sie die Rohwerte der integrierten Dichte der p-ERK-Färbung durch die integrierte Rohdichte des t-ERK-Signals dividieren. Fahren Sie mit der Bewertung der statistischen Signifikanz wie in Schritt 2.4 fort.
HINWEIS: Die Einstellungen der Bildgebungsparameter können je nach den spezifischen Anforderungen und unter Berücksichtigung der Zeitdauer für jede Probe, der gewünschten Bildqualität und der Anzahl der zu erfassenden Ebenen geändert werden.
- Achsenmessungen bei 11 hpf-Embryonen (Tag 4 für die Embryofixierung, Tag 12 für die Datenerfassung und -analyse) (Abbildung 4A-D)
- Am Ende der Gastrulation (11 hpf) fixieren Sie die Embryonen mit 4% PFA in 1x PBS für 20 min bei RT, waschen Sie sie mehrmals in 1x PBS und lagern Sie sie bei 4 °C bis zur Bildaufnahme.
- Richten Sie die Embryonen mit einer Pasteur-Pipette seitlich in einer Einzelvertiefung einer 12-Well-Platte aus, die frisches 1x PBS enthält.
- Um das Vorhandensein einer ovalen Form (Y/X-Embryoachsenverhältnis > 1), eines charakteristischen Rasopathie-Phänotyps beim Zebrafisch und des morphologischen Verhältnisses der Embryoachsen als Ergebnis der Wirksamkeit der Behandlung zu beurteilen, nehmen Sie Bilder ganzer Embryonen mit einem Stereomikroskop mit einem Objektiv mit 3,4-facher Vergrößerung (0,63-faches Scanning-Objektiv x 8,60 Zoomfaktor) auf, indem Sie einfach Bilder im Hellfeldmodus aufnehmen.
- Importieren Sie die Bilddatei in ein Bildanalysepaket, z. B. das Open-Source-Programm Fiji.
- Messen Sie die Achsenlänge der Embryonen (x, Nebenachse; y, Hauptachse), indem Sie das gerade Werkzeug aus der Symbolleiste auswählen und auf Analysieren | Messen. Nachdem Sie ausgewählte Messungen durchgeführt haben, fügen Sie sie der ROI-Manager-Liste hinzu und speichern Sie die ROI-Datei, wie oben für die FRET-Bildgebungsanalyse beschrieben.
HINWEIS: Wiederholen Sie diese Schritte für jedes Embryobild.
- Messen Sie die Achsenlänge der Embryonen (x, Nebenachse; y, Hauptachse), indem Sie das gerade Werkzeug aus der Symbolleiste auswählen und auf Analysieren | Messen. Nachdem Sie ausgewählte Messungen durchgeführt haben, fügen Sie sie der ROI-Manager-Liste hinzu und speichern Sie die ROI-Datei, wie oben für die FRET-Bildgebungsanalyse beschrieben.
- Fahren Sie mit dem Exportieren der Daten in einem Arbeitsblatt fort und berechnen Sie das Achsenverhältnis , indem Sie die Länge der Hauptachse (y) durch die Länge der Nebenachse (x) dividieren. Berechnen Sie den Mittelwert, die Standardabweichung des Mittelwerts oder den Standardfehler des Mittelwerts verschiedener Replikate.
- Fahren Sie mit der statistischen Datenanalyse wie in Schritt 2.4 fort.
Ergebnisse
Dieses Protokoll zeigt einen einfachen Arbeitsablauf zur schnellen Generierung transienter RASopathie-Modelle in Zebrafischembryonen und zur Beurteilung von ERK-Fluktuationen in frühen Mutanten mit einer standardmäßigen lebenden FRET-Bildgebungsmethode, die auf einen kürzlich etablierten ERK-Zebrafischsensor angewendet wird 6,9. Wie kürzlich gezeigtwurde, können FRET-Ergebnisse innerhalb desselben experimentellen Arbeitsablaufs durch Standard-IHC gegen phosphorylierte und totale ERK an ganzen Embryonen kreuzvalidiert werden. Eine Beeinträchtigung der Körperachsenbildung, die mit einer erhöhten ERK-Aktivierung korreliert, kann in späteren Stadien verfolgt werden, selbst bei denselben Embryonen, die für FRET7 verwendet wurden. Hier werden typische ERK-Fluktuationen in frühen Gastrula, die von FRET registriert werden, gezeigt, die mit der Expression von NS-verursachendem Shp2D61G verbunden sind. Es wurden auch Daten von Mutanten gewonnen, bei denen das Signal durch eine niedrig dosierte Behandlung mit einem MEKi negativ moduliert wurde, wodurch die RAS/MAPK-Signalkaskade gehemmt wurde.
Um NS-Modelle zu generieren, haben wir zunächst hochwertige gekapselte und polyadenylierte mRNA hergestellt, die für Shp2 (Wildtyp- und Mutantenform) kodiert, indem wir in vitro Transkription von einem geeigneten linearisierten Plasmid mit shp2-CDS in voller Länge durchgeführt haben. Wie in Abbildung 1A gezeigt, kann bei erfolgreicher Linearisierung ("Schnitt") des größten Teils des Plasmids eine scharfe Bande in einem Standard-1-1,5%igen Agarosegel beobachtet werden (hier im Beispiel der Größe von 7.500 bp), während die "ungeschnittene" DNA, die hier als Kontrolle für unverdautes Plasmid verwendet wird, verschiedene mögliche Konformationen aufweist, die für unverdaute zirkuläre DNA typisch sind. Verdeckelte und polyadenylierte mRNA kann dann in vitro hergestellt werden.Auf dem Agarose-Gel, das Formamid enthält, sollte die intakte RNA wie eine auffällige Bande um die erwartete Größe aussehen (Abbildung 1A, rechts), ohne erkennbaren Abstrich (was auf einen mRNA-Abbau hinweist) oder das Vorhandensein kleinerer Fragmente. Es ist ratsam, nicht fortzufahren, wenn fragmentierte oder abgebaute RNA erhalten wird. Die Vorbereitung sollte bei der Fehlerbehebung wiederholt werden. Wie in den Schemata in Abbildung 1B gezeigt, kann dann gekappte und polyadenylierte mRNA guter Qualität, die für das gewünschte RASopathie-Allel kodiert, und eine WT-Kontrolle (in unserem Fall Shp2D61G und Shp2WT als Kontrolle) in Zebrafischembryonen im Einzelzellstadium injiziert werden, die den Teen-Reporter exprimieren. Die Embryonen werden dann bis zum gewünschten Stadium (hier frühe Stadien während der Gastrulation) aufgezogen.
Abbildung 2 zeigt schematische Schemata und repräsentative Ergebnisse des Arbeitsablaufs, der zur Registrierung von FRET-Signalen in unserem RASopathy-Modell in vivo verwendet wird. Teen+ -Gastrula, die das mutierte NS-Allel Shp2D61G exprimieren, aufgezogen und im gewünschten Stadium (4 hpf) gesammelt, wurden in 1,5 % niedrigschmelzendem Agar (LMA) im E3-Medium montiert (Abbildung 2A). Ein Beispiel für die allgemeinen Mikroskopeinstellungen und die Lambda (λ)-Stack-Aufnahmeeinstellungen, die wir verwendet haben, um eine multispektrale FRET-Bildgebung aus der Spenderanregung (405 nm) in Teen-Embryonen zu erhalten, ist in Abbildung 2B dargestellt. Um eine ausreichende Spektrenerfassung zu gewährleisten, haben wir die λ-Bandbreite auf 5 nm eingestellt. Wir haben eine relativ hohe Schrittweite (hier 8 nm) verwendet, um einen x-, y-, λ-, z-Scan in angemessener Zeit zu erhalten, wenn man den Live-Erfassungsmodus (Aufnahmen mit einem Zeitintervall von ca. 13 Minuten) berücksichtigt.
Nach der Bildaufnahme wurde die aufgezeichnete Signalintensität entlang des gesamten λ-Spektrums überprüft, um die besten Emissionsfenster zuzuweisen und mit der Entmischung der Spektren CFP (D) und Ypet (A) fortzufahren (Abbildung 2C, links). Bei der Untersuchung des Randbereichs des tierischen Pols des Embryos mit einem ROI-basierten Ansatz, der in die Software Spectral dye separation wizard integriert ist, konzentrierten wir uns darauf, den beiden Fluorophoren die besten Emissionsspektralfenster zuzuweisen und dabei jegliche spektrale Überlappung zwischen D- und A-Molekülen zu vermeiden. Repräsentative rohe Einzelkanäle (CFP oder Ypet, grün bzw. rot), die mit diesem Ansatz erhalten wurden, und das ratiometrische FRET/CFP-Bild (grau), das sich aus der Anwendung der Verhältnisfunktion ergibt, sind in Abbildung 2C (rechtes oberes Bild) dargestellt. Das untere Feld zeigt ein repräsentatives suboptimales Ergebnis von Teen Reporter-Embryonen, die gelegentlich eine unzureichende Signalintensität aufweisen, wie sie häufig in einer Population transgener Fische beobachtet wird. Diese Embryonen sollten vorher verworfen werden, und es sollte vorsichtig vorgegangen werden, um Embryonen mit ähnlichem Ausblutungsgrad von Teenagern in der Analyse zu beurteilen.
Nach der Signalinspektion am Rand des Tierpols, wo sich die ERK-Aktivität während der Zellbewegungen der Epibolie und Gastrulation konzentriert, wurde die Bildwiedergabe mit "intelligenter" LUT-Pseudofärbung in Fidschi durchgeführt. Dies ermöglicht eine bessere Visualisierung von räumlichen Signalintensitätsunterschieden auf dem resultierenden ratiometrischen Bild. Die resultierenden ratiometrischen FRET-Bilder in Abbildung 2D zeigen bereits einen deutlichen Trend zur Reduktion der Signalintensität in Shp2D61G im Randbereich nach etwa 13 min Behandlung mit dem ausgewählten MEKi, PD0325901 (hier PD) bei niedrigen Dosen (0,25 μM)7. Die Daten zeigen die Eignung des Teen-Sensors und des hier verwendeten multispektralen FRET-Bildgebungsprotokolls zur Erkennung dynamischer ERK-Veränderungen in einem RASopathie-Modell in frühen Entwicklungsstadien sowie zur Feinmodulation des krankheitsassoziierten Signals nach niedrig dosierter MEKi-Behandlung.
Parallel dazu validierten wir die Ergebnisse unseres FRET-Ansatzes, der auf RASopathie-Fischmodelle angewendet wurde, durch Fluoreszenz-IHC gegen Gesamt-ERK (t-ERK) und seine phosphorylierte (p-ERK) Form. Durch die Normalisierung des erhaltenen Signals (p-ERK/t-ERK) innerhalb eines ROI entstehen Zellen mit hohen ERK-Werten. Abbildung 3 zeigt ein Beispiel für die Validierung der ERK-Aktivität mittels IHC für Shp2WT, Shp2D61G und Shp2D61G , die mit niedrig dosierter PD von 4 hpf bis 6 hpf behandelt wurden (längere Behandlung im Vergleich zu dem 13-minütigen Behandlungsfenster, das während der Live-FRET-Bildgebung gezeigt wurde). Die Immunfärbung wurde unter Verwendung von fluoreszierenden Sekundärantikörpern mit nicht überlappenden Emissionsspektren für t-ERK und p-ERK (488 bzw. 633 nm) durchgeführt.
Die 6 hpf-Embryonen wurden in 1,5 % LMA eingelegt, das in PBS gelöst war, und es wurden konfokale Standardeinstellungen eingestellt, um X,Y,Z-Scans des gesamten Embryovolumens zu erhalten (Abbildung 3A,B). Konfokale Standardeinstellungen mit einer Auflösung von 512 x 512 px bei 400 Hz ermöglichen es uns, die Signallokalisation zu untersuchen, was wichtig ist, um das gute Ergebnis der Immunfluoreszenz vor der Signalquantifizierung zu bestimmen. Wie erwartet und wie im repräsentativen Ergebnis gezeigt, beobachteten wir für t-ERK (grün) eine nahezu ubiquitäre Färbung in den Zellen des Randes, während das p-ERK-Signal (rot) hauptsächlich auf die Zellkerne beschränkt war (Abbildung 3C). Rohbilder, als Einkanal- und zusammengeführte Bilder, wurden zunächst durch eine Z-Stapel-Projektion verarbeitet, um Bilder des gesamten Embryos zu erhalten. Die Pseudofärbung von p-ERK (Kirsche) wurde angewendet, um den Kontrast mit t-ERK (grün) besser aufzulösen (Abbildung 3D). Im Vergleich zu Shp2WT konnte in Shp2D61G eine Zunahme der Anzahl von p-ERK+ Kernen im Randbereich beobachtet werden, was bei Shp2D61G Embryonen, die von 4 hpf auf 6 hpf mit 0,25 μM PD behandelt wurden, gerettet wurde. Die Daten bestätigen die Ergebnisse der FRET-Bildgebung und zeigen die effektive molekulare Modulation, die durch das niedrig dosierte MEKi der Wahl erzielt wird. Ähnlich wie bei FRET-Bildern wurde dann das Signal von Einkanal-Z-Stack-Aufnahmen im Bereich des Polrandes von Tieren mittels ROI-basierter Analyse mit Fidschi untersucht, und die p-ERK- und t-ERK-Werte wurden separat gemessen und für die anschließende Normalisierung (p-ERK/t-ERK) und statistische Analyse extrahiert (Abbildung 3E).
Schließlich liefert Abbildung 4 repräsentative morphometrische Daten, die erhalten und gemessen werden können, idealerweise an denselben Proben aus der Live-FRET-Bildgebung und anschließend von 6 auf 11/12 hpf oder an ihren Geschwistern erhöht. Veränderte Messungen der kleinen und großen Achse (Verhältnis Haupt- und Nebenachse) in diesem embryonalen Stadium können in der Tat Defekte der Gastrulationsbewegungen aufzeigen, die mit der RASopathie verbunden sind. Die Daten können verwendet werden, um das phänotypische Ergebnis neuer Varianten zu untersuchen, die bei Patienten mit unsicherer Pathogenität entdeckt wurden, und um die Stärke des Phänotyps mit der der molekularen Veränderung (ERK-Aktivierung) zu korrelieren, die mit FRET gemessen wurde. Im vorliegenden Beispiel wurden Geschwister der Teen-Embryonen (Kontrollgruppe 1: Shp2WT sowie Gruppen 2 und 3: Shp2D61G und Shp2D61G, behandelt mit niedrig dosiertem PD zwischen 4 und 6 hpf) getrennt bis 11 hpf aufgezogen und dann in 4% PFA fixiert (Abbildung 4A), um eine weitere Embryonalentwicklung zu vermeiden, die den Datenvergleich zwischen Individuen beeinträchtigen könnte. Bilder von lateralen Embryonen wurden mit einer einfachen Hellfeldmodalität aufgenommen, die für Standardeinstellungen verfügbar ist. Die Embryonenverlängerung wurde anhand von Messungen der Haupt- und Nebenachsen beurteilt (Abbildung 4B,C). Die Messergebnisse (Abbildung 4D) spiegeln und bestätigen die mit der FRET-Bildgebung erhaltenen Daten und zeigen eine Korrelation zwischen der dynamischen ERK-Aktivität am Rand des Tierpols und den Gastrulationsergebnissen. Insgesamt zeigt der hier gezeigte repräsentative Datensatz für das NS-assoziierte Allel shp2D61G mit oder ohne Behandlung MEKi PD0325901 den Nutzen der multispektralen FRET-Bildgebung in Tg[ef1α:ERK biosensor-nes] (Teen) Reporterfischen zur Vorhersage der Pathogenität von Varianten für NS im frühen Embryo.

Abbildung 1: Analyse von Plasmid- und mRNA-Präparaten, die zur Generierung transienter NS-Zebrafischmodelle erforderlich sind. (A) Das Panel zeigt ein schematisches und repräsentatives Ergebnis des DNA-Elektrophorese-Ergebnisses, das an einem UV-Transilluminator visualisiert wurde, der zur Bewertung einer effizienten Plasmidlinearisierung und einer qualitativ hochwertigen mRNA-Transkription verwendet wird. Linearisierte Plasmide ("Cut") werden durch Verdau mit einem Restriktionsenzym (in diesem Fall KpnI) erhalten und mit dem unverdauten Kontrollplasmid ("uncut") verglichen. Ein einzelnes Band, das schneller als das "ungeschnittene" Band läuft, sollte idealerweise bei der Plasmidlinearisierung beobachtet werden. Plasmidgröße: ~ 7.500 bps: (Rückgratplasmid + shp2 CDS). Beachten Sie, dass das "ungeschliffene" zirkuläre Plasmid verschiedene DNA-Formen von entspannten und supergewickelten Molekülen aufweist. Eine 1-kb-Leiter (Spur 1), deren Molekulargewichtsfragmente zwischen 0,5 und 10 Kilobasen (kb) lagen, wurde für die Größenkontrolle der DNA-Fragmente auf dasselbe Gel geladen. Auf der rechten Seite ist ein repräsentativer Gellauf von frisch transkribierten mRNAs zu sehen (in diesem Beispiel transkribierte mRNAs, die für Rasopathie-assoziiertes humanes SPRED2 kodieren). Zwei leicht unterschiedlich verlaufende Leitern (DNA-Leiter, Bahn 1 und RNA-Leiter, Bahn 2) mit ähnlichem Molekularbereich von 0,5 bis 10 kb werden für die Größenkontrolle der RNA-Fragmente geladen. (B) Schematische Übersicht, die die Verwendung der transkribierten mRNAs, die für RASopathie-assoziierte Varianten kodieren, zur Generierung transienter Krankheitsmodelle durch Injektion in Zebrafischembryonen im Einzelzellstadium darstellt. mRNA, die für die Wildtyp-Form des interessierenden Proteins kodiert, wird als Kontrolle injiziert. Das mRNA-Gemisch wird in der Lösung von Deneau mit dem lebenswichtigen Farbstoff Phenolrot hergestellt. Die Embryonen werden bei 6 hpf (für FRET- und IHC-Assays) oder bei 11/12 hpf (für Körperachsen) aufgezogen und mikroskopisch beurteilt. Abkürzungen: NS = Noonan-Syndrom; CDS = Codierungssequenz; FRET = Fluoreszenz-Resonanz-Energieübertragung ; IHC = Immunhistochemie ; HPF = Stunden nach der Befruchtung . Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Arbeitsablauf und repräsentative Ergebnisse der multispektralen FRET-Live-Bildgebung in frühen Teen Shp2D61G-Embryonen vor und nach der MEKi-Behandlung. (A) Überblick über die Schritte, die für die Live-spektrale FRET-Bildgebung in Zebrafischproben erforderlich sind. Embryonen, die in E3-Medium aufgezogen und inszeniert wurden, wurden in einer Glasbodenschale mit 1,5 % LMA für die lebende spektrale FRET-Erfassung eingesetzt. Die FRET-Bildgebung wird vor und nach der Verabreichung von niedrig dosierten PD0325901 durchgeführt. Ein Schema des von Sari et al. entwickelten Teen-Reportersystems ist dargestellt9. Ratiometrische Bilder (FRET/CFP) werden in der Nachbearbeitung erstellt. (B) Einzelheiten zu den Einstellungen für die Mikroskop- und multispektrale Bildaufnahme in der LAS X-Software für beide Fluorophore (CFP, D; Ypet, A). (C) Befehle und Verfahren zur Erlangung einer spektralen Farbstofftrennung und ratiometrischer Bilder, die auf ein Shp2D61GTeen-Gastrulae angewendet werden. Auf der linken Seite wurde ein starkes Signal vom Polrand des Tieres für die spektrale Trennung mit einem ROI-Auswahlwerkzeug ausgewählt. Oben rechts werden die Einstellungen und repräsentativen Ergebnisse der resultierenden Bilder für den D- und A-Kanal als maximale Intensität der Z-Projektionen eines einzelnen Fisches sowie die Einstellungen zum Erreichen des ratiometrischen Bildes und der repräsentativen Ergebnisse angezeigt. Angedeutet ist die Randregion am Pol des embryonalen Tieres mit hohem FRET-Signal (gestrichelte gelbe Linie). (D) Linkes Bild: Schematische Darstellung der ROI-Auswahl am Rand (gestrichelte gelbe Linie) aus dem ratiometrischen Rohbild für die Datenanalyse in Fidschi. Repräsentative FRET/CFP-Bilder, die über das "intelligente" LUT-Plugin von Fidschi erzeugt wurden, zeigen Veränderungen in der ERK-Aktivierung bei Embryonen, die Shp2D61G exprimieren, im Vergleich zu Shp2WT (mittleres Bild) oder im selben Shp2D61G-Embryo vor und nach der Parkinson-Behandlung (rechtes Bild). Für dieses Experiment werden die Rohintensitätsdichtewerte des ausgewählten ROI angezeigt. Die Maßstabsleiste wird angezeigt. Abkürzungen: FRET = Fluoreszenz-Resonanz-Energietransfer; MEKi = Mitogen-aktivierter Protein-Kinase-Enzyme-Inhibitor; LMA = niedrigschmelzende Agarose; CFP = Cyan-Fluoreszenz-Protein ; Ypet = gelbes Fluoreszenzprotein; D = Spender; A = Akzeptor; ROI = Region von Interesse; ERK = Extrazelluläre signalregulierte Kinase. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Arbeitsablauf und repräsentative Ergebnisse von IHC gegen t-ERK und p-ERK bei Shp2D61G frühen Embryonen mit oder ohne MEKi-Behandlung. (A) Schematische Darstellung der Entnahme von Zebrafischproben, die gegen t-ERK (488) und p-ERK (633) immungefärbt und in 1,5 % LMA/PBS für konfokale Z-Stack-Aufnahmen montiert wurden. (B) Konfokale Mikroskopeinstellungen für zwei Laserlinien (488, 633 nm), die zur sequentiellen Erfassung der Fluoreszenzsignale verwendet werden, die von den fluoreszierend konjugierten Sekundärantikörpern stammen. Der Einschub veranschaulicht die Z-Stack-Parameter für die Aufnahme von Multistack-Rohbildern mit akzeptabler Auflösung. (C) Schematische Darstellungen (linkes Bild) und repräsentative konfokale Rohbilder (rechtes Bild) von korrekt gefärbten Zebrafisch-Gastrutellen bei 6 hpf, die eine ubiquitäre und überwiegend nukleare Lokalisation von t-ERK (grün) bzw. p-ERK (rot) am Rand des Tierpols zeigen. (D) Repräsentative maximale Intensität von z-Projektionen aus konfokalen Stacks von 6 hpf-Embryonen, die Shp2WT (Kontrolle), Shp2D61G sowie Shp2D61G exprimieren, nach Behandlung mit 0,25 μM PD0325901 zwischen 4 und 6 hpf. Die Bilder werden als einzelner 633-nm-Kanal (hier p-ERK, pseudogefärbt in Kirsche) und kombiniert (p-ERK in Kirsche und t-ERK in grün) dargestellt. (E) In den kleinen Einschubschemata der ROI-Auswahl im Randbereich (gestrichelte gelbe Linie) aus den rohen Z-Stack-Bildern für die Datenanalyse in Fidschi ist zu sehen. Zooms der Z-Stack-Bilder, die den Randbereich zeigen, sind ebenso enthalten wie die rohen Intensitätsdichtemessungen, die abgerufen und bewertet wurden, um die ERK-Aktivierung im ROI von Geschwistern abzuleiten, die Shp2WT exprimieren, oder das Shp2D61G mit oder ohne 0,25 μM PD0325901 Behandlung. Die Maßstabsleiste wird in den Bildfenstern angezeigt. Abkürzungen: IHC = Immunhistochemie; ERK = Extrazelluläre signalregulierte Kinase; t-ERK = gesamte extrazelluläre signalregulierte Kinase; p-ERK = phosphorylierte extrazelluläre signalregulierte Kinase; FRET = Übertragung der Fluoreszenz-Resonanz-Energie; MEKi = Mitogen-aktivierter Protein-Kinase-Enzyme-Inhibitor; LMA = niedrigschmelzende Agarose; PBS = Phosphatase-Puffer-Kochsalzlösung; hpf = Stunden nach der Befruchtung; ROI = Region von Interesse. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
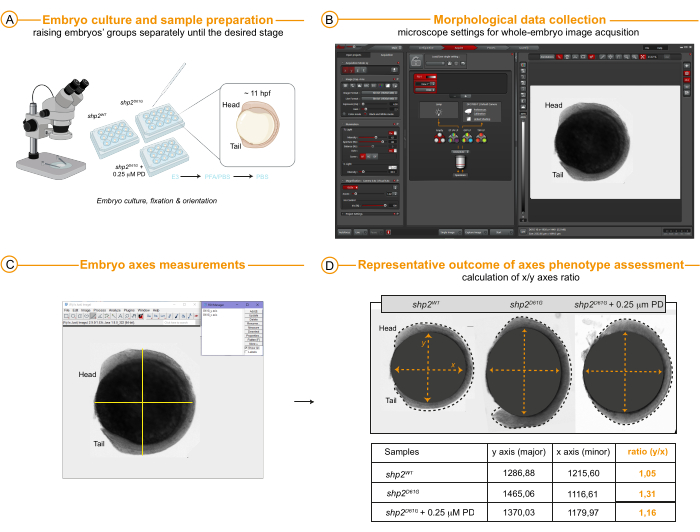
Abbildung 4: Morphologische Analyse bei 11 hpf. (A) Die Embryonen wurden bis zum gewünschten Entwicklungsstadium in E3-Medium aufgezogen, hier 11 hpf, dann in 4% PFA fixiert, in 1x PBS aufbewahrt und seitlich in Vertiefungen für die Aufnahme von Mikroskopbildern und die Beurteilung der Achsen angeordnet. (B) Bildaufnahmeeinstellungen in einem Standard-Stereomikroskop und ein Beispiel für das resultierende Bild, das einen ganzen Embryo (hier Shp2WT) zeigt, der mit dem Hellfeldmodus beurteilt wurde. (C,D) Repräsentative Ergebnisse und Achsenmessungen bei Shp2WT (Kontrolle), Shp2D61G und Shp2D61G nach Behandlung mit 0,25 μM PD0325901 zwischen 4 und 6 hpf. Der Umriss des Embryos ist durch eine gestrichelte schwarze Linie gekennzeichnet. Die Maßstabsleiste wird in den Bildfenstern angezeigt. Abkürzungen: hpf = Stunden nach der Befruchtung; PFA = Paraformaldehyd; PBS = Phosphatase-Puffer-Kochsalzlösung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Trotz jahrzehntelanger Forschung und Myriaden von Mutationen, die zu sehr heterogenen Formen von RASopathien führen, die jetzt kartiert sind, tauchen weiterhin genetische Varianten mit unbekannter Bedeutung aus Sequenzierungsbemühungen an nicht diagnostizierten Patienten auf. In der Tat kann eine Diagnose, die ausschließlich auf klinischen Merkmalen basiert, in vielen Fällen eine Herausforderung darstellen, und funktionelle genomische Ansätze zur Validierung der Sequenzierungsergebnisse sind nach wie vor von entscheidender Bedeutung. Darüber hinaus besteht trotz einiger verfügbarer Antikrebsmoleküle (d. h. MEK-Inhibitoren), die zur Behandlung einer Untergruppe von RASopathien vorgeschlagen werden, und einiger Erfolgsgeschichten, die sich abzeichnen, ein begrenzter Konsens. Dies ist auf die schlechte präklinische Evidenz für die meisten verfügbaren Medikamente zurückzuführen, auch wenn es um die wirksamen Dosen wirksamer Krebsmedikamente und therapeutische Fenster für pädiatrische Patienten geht.
Die Funktionsanalyse im Tiermodell stellt einen wichtigen Schritt für die Subklassifizierung von Krankheiten, die Stratifizierung der Patienten und die erste Arzneimittelbewertung dar. Der Zebrafisch kann ein effizientes In-vivo-Modell für die funktionelle Validierung potenziell pathogener Varianten sein, die pädiatrische Erkrankungen verursachen, einschließlich RASopathien14. Für Zebrafisch-RASopathie-Modelle gibt es eine hochmoderne phänotypische Bewertung der Auswirkungen von RASopathie-assoziierten Varianten, die hauptsächlich auf morphologischen Messwerten basieren - dem "ovalen Test" des Embryos, bei dem die Körperachsen am Ende der Gastrulation gemessen werden. Die Korrelation mit der ERK-Aktivierung wird jedoch häufig hauptsächlich durch Standardmethoden nach der Fixierung durchgeführt -- Immunblots / IHC.
Die von uns eingerichtete Pipeline zielte darauf ab, die ERK-Fluktuation live zu bewerten, um die potenzielle Wirksamkeit verfügbarer Medikamente zu Beginn der Gastrulation schnell zu testen und die daraus resultierenden morphometrischen Veränderungen zu analysieren. Das hier vorgestellte Protokoll beginnt mit der Generierung des gewünschten transienten Zebrafisch-RASopathie-Modells durch Überexpression des interessierenden GoF-Allels als mRNA in das Einzelzellstadium des ERK in vivo Reporters (Tg[ef1a:ERK biosensor-nes] Teen)6. Bei diesem FRET-Sensor vom EKAR-Typ ist die multispektrale FRET-Bildgebung eine nicht-invasive, unterbrechungsfreie Methode, die verwendet werden kann, um ERK-Signalschwankungen räumlich und zeitlich in lebenden Embryonen genau zu detektieren, die während der Entwicklung physiologisch sind, in RASopathie-Modellen aberrant sind und durch pharmakologische Signalmodulation korrigiert werden 6,7.
Verschiedene kritische Schritte der vorgestellten Pipeline sollten berücksichtigt werden, um aussagekräftige Ergebnisse aus den Embryonenproben zu erhalten. Angesichts der bekannten geringen Empfindlichkeit von FRET-Sensoren15 und möglicher variabler Effekte der Mutationen, die die ERK-Signalübertragung beeinflussen (insbesondere bei Mutationen, die zuvor nicht mit Standardmethoden validiert wurden), ist es ratsam, vor Beginn des Experiments die Art des Versuchsdesigns (d. h. Anzahl der Gruppen und Bedingungen) festzulegen und damit eine Schätzung der erforderlichen Anzahl von Embryonen vorzunehmen (Stichprobengröße, n) für die erwartete Effektstärke und gegebene Standardwerte für Fehler vom Typ I und Typ II. Es ist auch wichtig, Pilotversuche durchzuführen, um das tatsächliche Minimum an n abzuschätzen, das erforderlich ist, um relevante Unterschiede zwischen WT und mutiertem Protein zu beobachten. Eine Kreuzvalidierung der FRET-Ergebnisse mit alternativen und komplementären Ansätzen, wie z. B. IF- oder morphologischen Assays (wie hier angegeben), ist ebenfalls unerlässlich.
Im Allgemeinen kann ein Standardstichprobenumfang von n = 30 angemessen sein, um große Effekte normalverteilter Werte16 zu erkennen. Im Detail können für die A-priori-Analyse der Stichprobengröße frei verfügbare Programme wie "G-Power"17 verwendet werden, die diese Standardparameter einstellen: Signifikanzniveau (α): typischerweise auf 0,05 gesetzt; Konfidenzniveau (1 - β): in der Regel auf 95 % festgelegt; Trennschärfe eines Hypothesentests: in der Regel auf 80 % eingestellt (1 - β, wobei β die Wahrscheinlichkeit ist, einen Fehler vom Typ II zu begehen); Effektstärke d (das erwartete Ausmaß der Unterschiede zwischen experimentellen Gruppen), die unterschiedliche Werte haben kann, je nachdem, ob sie groß, mittel oder klein ist, laut Cohen18; Statistische Variation (Streuung in einer Häufigkeitsverteilung).
Im konkreten Beispiel wurde zuvor auch ein bekannter großer Effekt der Shp2D61G-Mutante im Zebrafisch mit Hilfe morphologischer Auslesungen und Pilot-Live-FRET-Experimente validiert, einschließlich komplementärer Assays wie FRET an fixierten Proben wie IF und morphologischen Messungen, die zur Validierung des beobachteten Effekts verwendet wurden (siehe Fasano et al.7).
Darüber hinaus ist mRNA von hoher Qualität ein wichtiger Ausgangspunkt, um geeignete transiente Krankheitsmodelle zu generieren. In der Tat wäre es zwar ideal, genetisch veränderte Modelle zu verwenden, um die Heterozygotie der Patienten zu rekapitulieren, aber die Generierung dieser Modelle ist nicht mit der Forderung nach einem schnellen Test neu auftretender Varianten vereinbar, da dies eine Anhebung der Grenze erfordern würde. Transiente Modelle, die erst während der Embryogenese erzeugt und bewertet werden, sind für große Screenings zu bevorzugen.
Um jedoch die Aussagekraft und Reproduzierbarkeit der Ergebnisse zu gewährleisten, sollte bei der Beurteilung der Qualität der RNA besondere Sorgfalt walten. Neben den klassischen Formamid-Gelen kann RNA auch mit Instrumenten wie dem Bioanalyzer überprüft werden, der eine direkte Quantifizierung und visuelle Elektropherogramme für die Qualitätskontrolle bietet. Hochwertige RNA-Präparate sehen aus wie ein einziger scharfer Peak in der gewünschten Größe. Es ist ratsam, nicht mit der Mikroinjektion fortzufahren, wenn die RNA auffällig abgebaut ist. Wichtig ist, dass bei der Mikroinjektion für neu synthetisierte mRNA-Chargen ein erstes Experiment zur Kalibrierung der richtigen Injektionsdosis ein Muss ist. Bei dieser idealen Dosis sollte die Expression der WT-Form der interessierenden mRNA bei Geschwistern aus derselben Charge keinen sichtbaren Phänotyp verursachen.
Als allgemeine Empfehlung gilt, dass es bei der Vorbereitung von Embryonen für die Injektion am besten wäre, Embryonen von paarungslosen Einzelpaaren (1 Weibchen + 1 Männchen) zu gewinnen, zu sammeln und zu beurteilen, um sicherzustellen, dass die Chargenvariabilität minimiert wird. Um die Embryonenausbeute zu erhöhen, kann das Laichen der Fische jedoch aus Gruppenkreuzungen mit mehreren Männchen und Weibchen eingerichtet werden. In diesem Fall stammen die entnommenen Embryonen wahrscheinlich aus mehr Befruchtungsereignissen, die mit leichter Verzögerung im Aquarium ablaufen, und es ist daher wichtig, Individuen im gleichen Entwicklungsstadium (frühe Gastrulation) genau auszuwählen, indem die Embryonen in den ersten 2 Stunden nach der Entnahme mehrmals untersucht werden. Um ein gutes Embryowachstum in der zarten Anfangsphase zu ermöglichen, sollte besonders darauf geachtet werden, auch alle frisch zubereiteten Lösungen zu verwenden. Darüber hinaus kann es angesichts des möglichen Vorhandenseins von Fäkalien und Ablagerungen aus dem Paarungsereignis zu einer Kontamination der Embryochargen durch Pilze und Bakterien kommen, wenn Embryonen im E3-Medium nicht unmittelbar nach der Entnahme gewaschen und gereinigt werden. Die Lebensfähigkeit der Embryonencharge sollte vor, während und nach dem Experiment überwacht werden. Wenn die Mortalitätsrate über der Standardrate (normalerweise 20-30%) liegt, sollte der Versuch abgebrochen werden.
In Bezug auf medikamentöse Behandlungen, zumindest für die hier getestete Klasse von MEKi, ist es am besten, Behandlungen vor 4 hpf zu vermeiden, da diese die Embryonalentwicklung (Toxizität) irreversibel beeinträchtigen können, wie zuvor beschrieben19. Ein weiterer kritischer Schritt für die Zuverlässigkeit des Experiments besteht darin, die gleiche Konzentration des Wirkstoffvehikels (in diesem Fall DMSO) für alle Versuchsbedingungen zu verwenden und eine Überfüllung der Embryonen pro Vertiefung während der Behandlung zu vermeiden, um eine gute Sauerstoffversorgung zu gewährleisten (maximal 20 Embryonen sollten in einer 6-Well-Platte aufbewahrt werden).
Die Optimierung der Bildgebungsparameter ist ebenfalls äußerst wichtig für den Erfolg der Pipeline, die insbesondere die multispektrale FRET-Bildgebung, aber auch die konfokale Bildgebung von IHC-Proben umfasst. Erstens besteht angesichts der Variabilität der Transgenexpression ein entscheidender Aspekt darin, Teen-Embryonen genau auszuwählen und diejenigen mit sehr geringer Expression zu verwerfen, die aufgrund des geringen Dynamikbereichs von FRET-Sensoren eine ausreichende Signaldetektion und -quantifizierung behindern könnten (siehe Beispiel Abbildung 2). Die Transgenexpression kann unter einem Standard-Stereomikroskop mit Leuchtstofflampen und geeigneten Filterrädern ~2 h nach der Injektion sichtbar sein.
Nach unserer Erfahrung mit der multispektralen Datenerfassung von Teen-Sensorfischen sind die Lasereinstellungen und die Parameter für den Spektralfarbstoffdefinitionserfassungsmodus (x, y, λ, z) wichtige erste Schritte der Mikroskop- und Hardwarekonfigurationen und sollten auf der Grundlage der spezifischen Anforderungen optimiert werden. Prinzipiell sollten die Parameter so eingestellt werden, dass ein guter Kompromiss zwischen Signalerfassung und Geschwindigkeit der Erfassung erreicht wird. Dies ist besonders wichtig für Echtzeitregistrierungen an nahezu vollständigen Embryonen, wie hier gezeigt. Eine Aufnahmegeschwindigkeit von 400 bis 600 Hz mit einer Schrittweite zwischen 8 μm und 10 μm und die Einstellung eines einzelnen Scans pro z-Ebene bieten möglicherweise keine zelluläre Auflösung, können aber ausreichen, um dynamische ERK-Aktivitätsänderungen im gesamten Gewebe von Embryonen zu erfassen. Es ist prinzipiell möglich, Bilder mit nahezu zellulärer Auflösung mit Ad-hoc-Mikroskopeinstellungen zu erhalten, jedoch auf Kosten der Geschwindigkeit und der 3D-Abtastung. Dies könnte interessant sein, wenn auch in späteren Stadien nur eine bestimmte Zellpopulation innerhalb der Embryonen untersucht wird6.
Die spektrale Farbstofftrennung kann mit anderen als den hier angegebenen Algorithmen durchgeführt werden. Unabhängig davon ist die Auswahl des Referenzbereichs, der erforderlich ist, um die besten Emissionsspektren sowohl für GFP als auch für YFP zuzuweisen, von entscheidender Bedeutung. Zu diesem Zweck ist es ratsam, die Konsistenz der erzielten Ergebnisse zu überprüfen, indem verschiedene ROIs ausgewählt und gemittelt werden, bei denen das Gewebe-/Zellsignal deutlich sichtbar ist. Am Ende der spektralen Erfassung und sobald die Farbstoffspektraltrennung erreicht ist, können ratiometrische Bilder auch ausgehend von CFP- und YFP-Rohscans und frei verfügbaren Ratio-Funktionen in Fidschi erhalten werden. Um Unterschiede in den FRET-Signalpegeln räumlich sichtbar zu machen und hervorzuheben, kann jedoch eine Neuskalierung der Bilder ratsam sein. In unseren Händen funktioniert die Pseudofärbung mit "intelligenten" Nachschlagetabellen (LUT), die in der kostenlosen Fiji-Software kodiert sind, gut, um die ERK-Aktivierungsstufen besser anzuzeigen. Andere LUTs und andere Skalen können je nach Vorliebe des Benutzers ausgewählt werden.
Es sollte betont werden, dass die hier vorgestellte experimentelle Pipeline auf Teen-Sensoren beschränkt ist, die CFP als Donor (D) und Ypet (ähnlich wie YFP) als Akzeptor (A) enthalten. Im Allgemeinen sollte man bei der Entscheidung für den FRET-Reporter den Dynamikbereich der Sensoren (die Fähigkeit, kleine, aber signifikante Änderungen zu erkennen) und die FRET-Effizienz (E) berücksichtigen, die durch den D-A-Abstand und die Orientierung innerhalb des Sensors beeinflusst wird, sowie die spektrale Überlappung zwischen D-Emission und A-Absorption. Teen ist ein EKAREV-Sensor, der durch optimiertes Konstruktdesign im Vergleich zu früheren Versionen einen verbesserten Dynamikbereich und eine verbesserte Empfindlichkeit (E) bietet. Der relativ geringe Dynamikbereich, der in vivo normalerweise schlechter ist, kann jedoch als die größte Einschränkung dieses Ansatzes angesehen werden. Nichtsdestotrotz zeigten Sari et al. und Wong et al., dass dynamische ERK-Veränderungen, die physiologisch während der Entwicklung auftreten, in Teen-Embryonen dennoch sichtbar und messbar sind, und wir beobachten Signalschwankungen in einem pharmakologisch behandelten NS-Fischmodell 6,9. Wir gehen jedoch davon aus, dass extrem geringe ERK-Signalschwankungen mit der hier gezeigten Methode nicht einfach zu beurteilen sind. Es wird wichtig sein, die Leistungsfähigkeit unserer Pipeline mit verbesserten ERK-Biosensoren zu testen, die wahrscheinlich in Zukunft verfügbar sein werden.
Für die Kreuzvalidierung der Ergebnisse mit IHC ist die Qualität der Gewebefixierung ein entscheidender Schritt. Embryonalgewebe bei 6 hpf ist extrem empfindlich und dünn und daher empfindlich gegenüber schlechter Fixierung und Überfixierung. Angesichts der Qualitäts- und Leistungsvariabilität von Charge zu Charge sowohl für Fixierlösungen als auch für Antikörper sollten die Fixierungszeit und der Erfolg des gesamten IHC-Protokolls in einem Pilotversuch kurz vor der Bewertung von Versuchsproben evaluiert werden. Frisch zubereitetes und steriles Fixiermittel ist immer ratsam.
Zusammenfassend lässt sich sagen, dass das hier gezeigte experimentelle Protokoll im praktischen Zebrafischmodell eine robuste und relativ schnelle Pipeline darstellt, um den Einfluss ausgewählter NS-Allele auf die ERK-Aktivierung in gastrulierenden Zellen früher Embryonen zu bewerten. Die Methode, bei der die multispektrale FRET-Bildgebung im neu entwickelten Teen-Sensorfisch zum Nachweis lebender molekularer Fluktuationen zum Einsatz kommt, übertrifft die klassische IHC. Nichtsdestotrotz kann IHC als ergänzender Ansatz zur Validierung der erzielten Ergebnisse verwendet werden. Der Ansatz nimmt klassische phänotypische Messwerte vorweg, nämlich das Verhältnis von Haupt- und Nebenachsen ("ovaler Embryo"-Test), ein Goldstandard für RASopathie-Fischmodelle, der jedoch nur am Ende der Gastrulation messbar ist. Das Protokoll kann auch angewendet werden, um die Fähigkeit des vorgeschlagenen MEKi zu testen, Signal- und Achsenveränderungen in Embryonen zu korrigieren.
In Anbetracht der Tatsache, dass wir nur das NS-assoziierte shp2D61G-Allel und ein Medikament getestet haben, sollte die weitere Entwicklung des Assays die Bewertung der Leistung in Bezug auf andere RASopathie-assoziierte Mutationen mit unterschiedlichen Auswirkungen auf die ERK-Aktivierung und zusätzliche vorgeschlagene Medikamente, Dosen und Behandlungsfenster umfassen. Es wird interessant sein, die Sensitivität dieses mehrstufigen Ansatzes für andere neu auftretende Varianten zu bewerten, die Moleküle auf verschiedenen Ebenen der RAS-MAPK-Signalkaskade betreffen. Zu guter Letzt wäre eine zukünftige Standardisierung einer speziellen Hochdurchsatzmethode für die ratiometrische FRET-Bildgebung und -Quantifizierung in speziellen High-Content-Analysesystemen erforderlich, um die Variantenprüfung zu fördern.
Danksagungen
Wir danken Dr. Jeroen den Hertog (Hubrecht Institute, Utrecht, Niederlande) für die freundliche Bereitstellung von pCS2+_eGFP-2a-Shp2a, aus dem das shp2-CDS in voller Länge extrahiert wurde, um das von uns verwendete Plasmid-Templatezu generieren 7. Wir danken dem Nara Institute of Science and Technology (Takaaki Matsui) und dem National Institute of Genetics (NIG/ROIS) (Koichi Kawakami) für die Bereitstellung der transgenen Teen-Reporterlinie . Diese Arbeit wurde unterstützt durch das italienische Gesundheitsministerium - Laufende Forschungsfonds 2021 und Laufende Forschungsfonds 2024 und Ricerca Finalizzata Giovani Ricercatori GR-2019-12368907 an AL; Aktuelle Forschungsfonds 2019, PNRRMR1-2022-12376811, 5x1000 2019, AIRC (IG-21614 und IG-28768) und LazioInnova (A0375-2020-36719) bis MT.
Materialien
| Name | Company | Catalog Number | Comments |
| Plasticwares | |||
| 1.7 L Breeding Tank - Beach style Design | Tecniplast | 1.7L SLOPED | Breeding tank |
| Capillaries GC100F-10 | Harvard apparatus | 30-0019 | One-cell stage embryo microinjection |
| Cell and Tissue Culture Plates - 12 well | BIOFIL | TCP011012 | Embryo collection and treatment |
| Cell and Tissue Culture Plates - 6 well | BIOFIL | TCP011006 | Embryo collection and treatment |
| Cell Culture Dish | SPL Life Sciences | 20100 | Embryo collection |
| Nunc Glass Dishes 12mm | Thermo Fisher | 150680 | Embryo FRET spectral imaging |
| Pipette Pasteur | Corning | 357524 | Embryo transfer |
| Protein Lobind Tubes 2ml | Eppendorf | 30108450 | IHC assay |
| Reagents and others | |||
| Caviar 500-800 µm | Rettenmaier Italia | BE2269 (500-800) | Dry fish food |
| Great Salt Lake Blue Artemia Cysts | Sanders | 00004727 | Live fish food |
| Instant Ocean salt | Tecniplast | XPSIO25R | Dehydrated sea salt for live food preparation |
| Tg(EF1a:ERK Biosensor-nes) (Teen) | Contacts for ordering*: National BioResource Project Zebrafish, Support Unit for Animal Resources Development, RRD, RIKEN Center for Brain Science, Japan. https://shigen.nig.ac.jp/zebra/index_en.html *upon MTA signature. | - | Supplier of ERK Reporter zebrafish line. Fish embryos can be obtained upon MTA signature from National BioResource Project of Japan for Zebrafish (RIKEN, Japan). The zebrafish line is deposited by Nara Institute of Science and Technology (Takaaki Matsui) and the National Institute of Genetics (NIG/ROIS) (Koichi Kawakami, patent for Tol2 system) (Wong et al., 2018, Urasaki et al., 2006, Okamoto and Ishioka, 2010). |
| 6x loading dye | Cell Signaling | B7024S | Gel Elecrophoresis |
| 100 bp DNA ladder | NEB | N3231S | Gel Elecrophoresis |
| Agarose | Sigma-Aldrich | 1,01,236 | Gel Elecrophoresis |
| Agarose, low gelling temperature | Sigma-Aldrich | A9414-10G | Embryo mounting for FRET spectral imaging and IHC assay |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A8022 | IHC assay |
| Calcium chloride | Sigma-Aldrich | 223506 | E3 medium component |
| Calcium nitrate | Sigma-Aldrich | 237124 | Danieau stock solution component |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418-100ML | IHC assay |
| EDTA | Sigma-Aldrich | E9884 | TBE buffer component for gel preparation |
| Ethanol 99%+ | Fisher Scientific | 10048291 | In vitro RNA purification |
| Formaldeide 16% | Thermo Fisher | 28908 | Embryo fixation |
| Formamide | Sigma-Aldrich | F9037 | Gel Elecrophoresis |
| Gel Loading Buffer II (Denaturing PAGE) | Thermo Fisher | AM8546G | In vitro RNA transcription |
| Glacial Acetic Acid | Sigma-Aldrich | 695092 | TBE buffer component for gel preparation |
| Glycerol | Sigma-Aldrich | G6279-1L | IHC assay |
| Goat anti-mouse Alexa Fluor 488 | Thermo Fisher | A11001 | IHC assay |
| Goat anti-rabbit Alexa Fluor 633 | Thermo Fisher | A21070 | IHC assay |
| HEPES | Sigma-Aldrich | H3375 | Danieau stock solution component |
| KpnI - HF (Enzyme + rCutSmart Buffer) | NEB | R3142 | Plasmid linearization |
| Magnesium sulfate | Sigma-Aldrich | 230391 | E3 medium component/Danieau stock solution component |
| Millennium RNA Markers | Thermo Fisher | AM7150 | Gel Elecrophoresis |
| Monarch Genomic DNA purification Kit | NEB | T3010L | Plasmid linearization |
| Mouse monoclonal p44/42 MAPK | Cell Signaling | 4696S | IHC assay |
| mMACHINE SP6 Transcription Kit | Thermo Fisher | AM1340 | In vitro RNA transcription |
| Normal Goat serum (NGS) | Sigma-Aldrich | G9023 | IHC assay |
| Nuclease-free water Ambion | Thermo Fisher | AM9937 | In vitro RNA transcription |
| PD0325901 | Sigma-Aldrich | PZ0162 | MEK inhibitor |
| Phenol Red solution | Sigma-Aldrich | P0290 | Microinjection mix component |
| Poly A Tailing Kit | Thermo Fisher | AM1350 | In vitro RNA transcription |
| Potassium chloride bioxtra | Sigma-Aldrich | P9333 | E3 medium component/Danieau stock solution component/PBS stock solution component |
| Potassium dihydrogen phosphate | Sigma-Aldrich | P0662 | PBS stock solution component |
| Proteinase K | Sigma-Aldrich | P2308 | IHC assay |
| Rabbit polyclonal phospho-p44/42 MAPK | Cell Signaling | 4695S | IHC assay |
| SYBR safe DNA gel staining | Thermo Fisher | S33102 | Gel Elecrophoresis |
| Sodium Chloride | Sigma-Aldrich | 31434-M | E3 medium component/Danieau stock solution component/PBS stock solution component |
| Sodium phosphate dibasic | Sigma-Aldrich | 71643 | PBS stock solution component |
| Trizma base | Sigma-Aldrich | T1503 | TBE buffer component for gel preparation |
| Triton X-100 | Sigma-Aldrich | T8787 | PBSTr buffer component |
| Equipment | |||
| Alliance Mini HD9 | Uvitec | - | Imaging system |
| Centrifuge 5430 R | Eppendorf | 5428000205 | Microcentrifuge |
| Eppendorf ThermoMixer C | Eppendorf | - | Embryo mounting |
| FemtoJet 4x | Eppendorf | - | Microinjection system |
| Infinite M Plex | Tecan | - | Multimode plate reader |
| Leica M205FA | Leica Microsystems | - | Fluorescence stereo microscope |
| Leica TCS-SP8X equipped with incubator (OkoLab) | Leica Microsystems | - | Confocal microscope |
| Mini-sub Cell GT Horiziontal Electrophoresis System | Bio-Rad | 1704406 | Gel Elecrophoresis |
| PC-100 Vertical puller | Narishige | - | Needle puller |
| PowerPac Universal Power Supply | Bio-Rad | 1645070 | Gel Elecrophoresis |
| Stellaris 5 | Leica Microsystems | - | Confocal microscope |
| Vortex MiniStar silverline | VWR | - | Plasmid preparation |
| Softwares | |||
| Biorender | Biorender | CC-BY 4.0 license | Cartoon elaboration for Figures |
| Excel | Microsoft Office Professional Plus 2019 | - | Data analyses |
| Fiji software | ImageJ | 15.3t | Imaging rendering and quantitative analyses (FRET signals measurements, ERK fluorescence intensity in IHC assay, embryo axes lenght) |
| GraphPad Prism | GraphPad Software LLC | v. 9 | Statistical data analyses |
| iControl spectrophotometer software | Tecan | v. 2.0 | RNA quantification |
| Illustrator | Adobe | 26.0.3 (64-bit) | Figure assembling |
| LASX software | Leica Microsystems | v. 4.5 (Stellaris 5), v. 3.0 (M205FA), v. 3.5 (TCS-SP8X) | Imaging acquisition for spectral FRET experiments and embryo imaging for axes lenght measurements |
| Q9 Mini 18.02-SN software | Uvitec | - | Gel image acquisition |
Referenzen
- Iroegbu, J. D., Ijomone, O. K., Femi-Akinlosotu, O. M., Ijomone, O. M. ERK/MAPK signalling in the developing brain: Perturbations and consequences. Neurosci Biobehav Rev. 131, 792-805 (2021).
- Tartaglia, M., Aoki, Y., Gelb, B. D. The molecular genetics of RASopathies: An update on novel disease genes and new disorders. Am J Med Genet C Semin Med Genet. 190 (4), 425-439 (2022).
- Tate, J. G., et al. COSMIC: the catalogue of somatic mutations in cancer. Nucleic Acids Res. 47 (D1), D941-D947 (2019).
- Tartaglia, M., et al. Mutations in PTPN11, encoding the protein tyrosine phosphatase SHP-2, cause Noonan syndrome. Nat Genet. 29 (4), 465-468 (2001).
- Jopling, C., van Geemen, D., den Hertog, J. Shp2 knockdown and Noonan/LEOPARD mutant Shp2-induced gastrulation defects. PLoS Genet. 3 (12), e225 (2007).
- Wong, K. -. L., Akiyama, R., Bessho, Y., Matsui, T. ERK activity dynamics during zebrafish embryonic development. Int J Mol Sci. 20 (1), 109 (2019).
- Fasano, G., et al. Assessment of the FRET-based Teen sensor to monitor ERK activation changes preceding morphological defects in a RASopathy zebrafish model and phenotypic rescue by MEK inhibitor. Mol Med. 30 (1), 47 (2024).
- Zimmermann, T., Rietdorf, J., Girod, A., Georget, V., Pepperkok, R. Spectral imaging and linear un-mixing enables improved FRET efficiency with a novel GFP2-YFP FRET pair. FEBS Lett. 531 (2), 245-249 (2002).
- Sari, D. W. K., et al. Time-lapse observation of stepwise regression of Erk activity in zebrafish presomitic mesoderm. Sci Rep. 8 (1), 4335 (2018).
- Wang, H., Li, J., Xue, T., Cheng, N., Du, H. Construction of a series of pCS2+ backbone-based Gateway vectors for overexpressing various tagged proteins in vertebrates. Acta Biochim Biophys Sin (Shanghai). 48 (12), 1128-1134 (2016).
- Abdelrahman, D., Hasan, W., Da'as, S. Microinjection quality control in zebrafish model for genetic manipulations. MethodsX. 8, 101418 (2021).
- Yuan, S., Sun, Z. Microinjection of mRNA and morpholino antisense oligonucleotides in zebrafish embryos. J Vis Exp. (27), e1113 (2009).
- Rossant, J., Nutter, L. M. J., Gertsenstein, M. Engineering the embryo. Proc Natl Acad Sci USA. 108 (19), 7659-7660 (2011).
- Jindal, G. A., et al. In vivo severity ranking of Ras pathway mutations associated with developmental disorders. Proc Natl Acad Sci USA. 114 (3), 510-515 (2017).
- Leavesley, S. J., Rich, T. C. Overcoming lmitations of FRET measurements. Cytometry A. 89 (4), 325-327 (2016).
- Mascha, E., Vetter, T. Significance, errors, power, and sample size: Theblocking and tackling of statistics. Anesth Analg. 126 (2), 691-698 (2018).
- Faul, F., Erdfelder, E., Lang, A. -. G., Buchner, A. G*Power 3: a flexible statistical power analysis program for the social, behavioral, and biomedical sciences. Behav Res Methods. 39 (2), 175-191 (2007).
- Cohen, J. . Statistical power analysis for the behavioral sciences. , (1988).
- Anastasaki, C., Rauen, K. A., Patton, E. E. Continual low-level MEK inhibition ameliorates cardio-facio-cutaneous phenotypes in zebrafish. Dis Model Mech. 5 (4), 546-552 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten