Method Article
Un protocolo eficiente para evaluar la modulación de la actividad de ERK en modelos tempranos de síndrome de Noonan de pez cebra mediante microscopía FRET en vivo e inmunofluorescencia
En este artículo
Resumen
Las rasopatías son síndromes genéticos multisistémicos causados por la hiperactivación de la vía RAS-MAPK. Las variantes potencialmente patógenas que están a la espera de validación surgen continuamente, mientras que la escasa evidencia preclínica limita el tratamiento. Aquí, describimos nuestro protocolo in vivo para probar y validar los niveles de activación de ERK asociados a RASopatía y su modulación farmacológica durante la embriogénesis mediante imágenes FRET en vivo en pez cebra reportero.
Resumen
Las rasopatías son síndromes genéticos causados por la hiperactivación de ERK y que dan lugar a enfermedades multisistémicas que también pueden conducir a la predisposición al cáncer. A pesar de la amplia heterogeneidad genética, en la mayoría de los casos subyacen mutaciones de ganancia de función de la línea germinal en reguladores clave de la vía RAS-MAPK y, gracias a técnicas de secuenciación avanzadas, se siguen identificando variantes potencialmente patógenas que afectan a la vía RAS-MAPK. La validación funcional de la patogenicidad de estas variantes, esencial para un diagnóstico preciso, requiere protocolos rápidos y fiables, preferiblemente in vivo. Dada la escasez de tratamientos eficaces en la primera infancia, estos protocolos, especialmente si son escalables en modelos animales rentables, pueden ser fundamentales para ofrecer una base preclínica para el reposicionamiento/readaptación de fármacos.
Aquí describimos paso a paso el protocolo para la generación rápida de modelos transitorios de RASopatía en embriones de pez cebra y la inspección directa de los cambios vivos en la actividad de ERK asociados a enfermedades que ocurren ya durante la gastrulación a través de imágenes multiespectrales de transferencia de energía por resonancia de Förster (FRET) en tiempo real. El protocolo utiliza un reportero ERK transgénico recientemente establecido e integrado con el hardware de los microscopios comerciales. Proporcionamos un ejemplo de aplicación para modelos de pez cebra con síndrome de Noonan (SN) obtenidos por expresión del Shp2D61G. Describimos un método sencillo que permite el registro del cambio de señal de ERK en el modelo de peces NS antes y después de la modulación de la señal farmacológica por los inhibidores de MEK de dosis baja disponibles. Detallamos cómo generar, recuperar y evaluar señales FRET ratiométricas a partir de adquisiciones multiespectrales antes y después del tratamiento y cómo validar los resultados mediante inmunofluorescencia clásica en embriones enteros en etapas tempranas. A continuación, describimos cómo, mediante el examen de los parámetros morfométricos estándar, se pueden consultar los cambios tardíos en la forma del embrión, indicativos de un deterioro resultante de la gastrulación, en los mismos embriones cuya actividad de ERK se evalúa mediante FRET vivo a las 6 h después de la fertilización.
Introducción
Las rastopías son síndromes genéticos que alteran el desarrollo normal y afectan a diversos órganos y tejidos. Estas afecciones a menudo son causadas por mutaciones de ganancia de función (GoF) de la línea germinal en los genes clave y los actores involucrados en la señalización de RAS/MAK, lo que resulta en una hiperactivación (aumento de la fosforilación) de la quinasa regulada por señales extracelulares (ERK). ERK regula algunos procesos fundamentales importantes durante el desarrollo, como el crecimiento de los tejidos, mediante la translocación al núcleo 1,2. Las mutaciones somáticas en genes implicados en la vía RAS-MAPK son los eventos más comunes que conducen al cáncer3. Así, no es sorprendente que la predisposición al cáncer también se observe en las rasopatías. El síndrome de Noonan (SN), caracterizado por retraso en el desarrollo, baja estatura, déficits cognitivos de gravedad variable y miocardiopatía, es la forma más común de RASopatía2. En la mayoría de los casos, la enfermedad está causada por mutaciones GoF en PTPN11, el primer gen de RASopathy que se descubrió a principios de 20004 que codifica para la proteína tirosina fosfatasa SHP2, que actúa como regulador positivo de la vía.
Desde entonces, gracias al uso exponencial de los enfoques de secuenciación del exoma en pacientes no diagnosticados, se siguen descubriendo variantes potencialmente patógenas que afectan a factores implicados en el RAS-MAPK, y probablemente relacionadas con diversas formas de RASopatías, a la espera de una caracterización funcional para una estratificación eficiente de los pacientes2. Para lograr este objetivo, se requieren protocolos experimentales que garanticen una validación funcional rápida e informativa a nivel de organismo. El empleo de modelos clásicos y estandarizados de mamíferos para probar variantes con significado desconocido sería costoso, llevaría mucho tiempo y requeriría métodos invasivos en animales grandes no transparentes. Es evidente que esta estrategia no es compatible con el requisito de realizar pruebas rápidas, dada la carga social que representan los pacientes pobres o no diagnosticados con RASopatía, que actualmente carecen de tratamiento ni tratamiento. Los protocolos para la evaluación cuantitativa de los rasgos fenotípicos clave y los correlatos moleculares en organismos completos también servirían para acelerar la posible traslación clínica de los fármacos posiblemente disponibles para los pacientes con RASopatía mediante la reutilización/reposicionamiento.
El pez cebra es un modelo de vertebrado ideal para estudiar enfermedades que afectan al desarrollo temprano. Para empezar, el pez cebra comparte un alto nivel de homología genética con los humanos. La alta fecundidad de los peces adultos da como resultado una gran producción de embriones que son pequeños y se desarrollan rápidamente. Los embriones son transparentes en las primeras etapas, de modo que los principales procesos de desarrollo (epibolia, gastrulación, ejes y formación del plan corporal) se pueden visualizar sin esfuerzo utilizando la microscopía estándar. Además, la disponibilidad de líneas transgénicas que se pueden utilizar para rastrear el comportamiento celular específico y los eventos moleculares dinámicos en el espacio y el tiempo durante el desarrollo, junto con técnicas avanzadas para generar modelos genéticos, es inmejorable. Además, las lecturas fenotípicas se pueden evaluar a múltiples niveles en el pez cebra (desde defectos del organismo hasta defectos celulares), y ya se han establecido ensayos específicos para varias enfermedades, incluidas las RASopatías5. Además, los métodos relativamente sencillos de baño de inmersión para la administración de fármacos durante las primeras etapas, al menos para los compuestos solubles en agua, permiten el cribado de fármacos de alto rendimiento in vivo en un formato de 96 pocillos.
Desde el punto de vista molecular, los estudios que utilizan enfoques estándar, como la inmunohistoquímica y el inmunoblot, demuestran de manera robusta la correlación entre la activación de ERK y los defectos de desarrollo asociados a RASopatía en embriones de peces 6,7. El biosensor FRET de tipo EKAR recientemente desarrollado en pez cebra (Tg[ef1a:ERK biosensor-nes], Teen) proporciona una herramienta in vivo fiable para registrar la activación de ERK durante la embriogénesis de una manera resuelta espacio-temporalmente. Por lo tanto, podría ser valioso para una mejor evaluación de las alteraciones dinámicas de ERK y las modulaciones farmacológicas en modelos de peces RASopathy.
En el sensor Teen , un sustrato específico de ERK en el reportero se fosforila tras la activación de ERK, lo que desencadena un cambio conformacional que acerca al donante fluorescente de CFP (D) y al aceptor fluorescente Ypet (YFP mejorado) (A). Si el espectro de emisión D se superpone considerablemente con el espectro de absorción del A, puede producirse FRET (absorción de energía de D a A). Esto es proporcional a la distancia entre D y A y, por lo tanto, en Teen, al estado de activación de ERK. Se pueden configurar diferentes protocolos de imagen utilizando módulos de imagen estándar y avanzados de microscopios estándar o confocales en muestras vivas y fijas. Tras la excitación D, la adquisición de exploraciones multiespectrales a lo largo de un espectro definido de emisión (λ) de CFP a YFP seguidas de algoritmos de "desmezcla" espectral es uno de los métodos más fiables para registrar y cuantificar los datos FRET8. También se puede aplicar a especímenes vivos de pez cebra para registrar la dinámica de los tejidos in vivo .
Siguiendo los informes anteriores 6,9 y nuestra reciente aplicación7, aquí, detallamos el flujo de trabajo paso a paso utilizando peces adolescentes para evaluar la activación de ERK en células en el margen del polo animal de modelos NS al comienzo de la gastrulación y correlacionarlo con defectos característicos de los ejes corporales visibles solo más tarde en el desarrollo. Mostramos cómo obtener y examinar datos cuantitativos de FRET de NS gasstrulae vivas antes y después del tratamiento con un MEKi disponible y cómo validar los resultados a través de la inmunohistoquímica estándar contra ERK fosforilada (activa) o realizar un análisis morfométrico correlativo de defectos de elongación embrionaria.
El flujo de trabajo podría aplicarse para impulsar la prueba funcional de variantes emergentes y genes de enfermedades supuestamente asociados con RASopatías y para obtener información sobre la correlación de la dinámica de activación de ERK espacial y temporalmente durante el desarrollo de vertebrados y los defectos morfológicos en los embriones. Demostramos que este protocolo también se puede utilizar para probar la eficacia de los fármacos candidatos que actúan para modular la activación de ERK.
Protocolo
Todos los procedimientos experimentales relacionados con el alojamiento y la cría de animales se llevaron a cabo de acuerdo con las directrices de ARRIVE para el uso de pez cebra en la investigación animal y autorizadas por el Ministerio de Salud italiano (Direzione Generale della Sanità Animale e dei Farmaci veterinari - DGSAF). Todas las reacciones de ADN/ARN y las sesiones de imágenes pueden ampliarse o reducirse según se desee, en función del material final necesario o del número de genes y variantes analizados.
1. Generación y tratamiento farmacológico de modelos transitorios de RASopatía de pez cebra
NOTA: Para monitorizar la expresión de las variantes asociadas a la RASopathy, se pueden utilizar construcciones específicas que albergan la secuencia codificante deseada (cds) de la proteína de interés en el marco con las cds de pequeñas etiquetas no fluorescentes (como myc o similar). De esta manera, los niveles de expresión de la proteína mutante se pueden evaluar mediante Western blot estándar frente a la etiqueta. Si se dispone de anticuerpos contra la proteína específica de interés, se pueden evitar las marcas. La inmunofluorescencia también se puede utilizar para evaluar la expresión de proteínas dentro del tejido embrionario siguiendo protocolos estándar. Este tipo de experimento de control puede ser útil para correlacionar la expresión de proteínas mutantes con los niveles de activación inducida de ERK. No se aconseja el uso de marcadores fluorescentes en combinación con imágenes FRET, dada la posible diafonía de emisiones de fluorescencia durante la microscopía.
- Linealización de plásmidos (días 1-2) (Figura 1A)
NOTA: Al generar un modelo de enfermedad transitoria en pez cebra (RAsopatía aquí) causada por una mutación GoF que afecta a un gen específico, un enfoque rápido es preparar el ARNm que codifica el tipo salvaje (WT) y la proteína mutante de interés, que luego debe inyectarse en embriones de pez cebra siguiendo los pasos a continuación. El plásmido utilizado como molde para transcribir el ARNm debe contener la CDS (secuencia codificante) de longitud completa deseada para el gen de interés clonado aguas abajo de un promotor de la polimerasa T7, Sp6 o T3 y aguas arriba de la señal de poliadenilación (poliA). Si no se dispone de él, el primer paso es producirlo clonando el pez cebra o el CDS deseado por el ser humano en un vector10 adecuado y validarlo mediante secuenciación SANGER. Para la clonación, considere tiempo adicional (en promedio 1 semana que incluye la clonación, la selección de colonias y el aislamiento y expansión de los clones correctos).- Linealizar el plásmido con enzimas de restricción adecuadas, cortando solo una vez aguas abajo la señal de poliA (aquí KpnI). Para lograr una linealización eficiente del plásmido, mezcle 3 γ (μg/μL) de ADN plasmídico, 2 μL de enzima de restricción (20.000 Unit/mL) y 10 μL de tampón de reacción 10x en un volumen final de 100 μL en agua libre de nucleasas. Mezcle bien los componentes pipeteando varias veces e incube la mezcla de reacción a 37 °C durante 2-4 h.
- Comprobar el resultado de la linealización por electroforesis en un gel de agarosa al 1,5%.
- Diluir 10 veces la solución madre de TBE (48,5 g de Tris, 11,4 mL de ácido acético glacial, 20 mL de EDTA 0,5 M [pH 8,0]) con agua libre de nucleasas.
- Preparar el gel de agarosa disolviendo en el microondas 1,5 g de agarosa en un volumen final de 100 mL de 1x solución de TBE. A continuación, añade 3,5 μL de tinte en gel de tinte.
- Lanzar el gel vertiendo la solución de agarosa en cámaras de tamaño adecuado, dependiendo del número de condiciones/plásmidos. Coloque los peines y espere aproximadamente 1 h hasta que el gel se solidifique; A continuación, retire los peines y coloque el gel polimerizado en la bandeja de gel adecuada para la electroforesis.
- Mezclar unos pocos μL del plásmido digerido ("corte") y 1,5 μL de tampón de carga 6x (que contiene un colorante para controlar la electroforesis) en un volumen final de 10 μL de agua libre de nucleasas. Prepare de la misma manera también una muestra de control separada con el plásmido sin digerir ("sin cortar"). Cargue las muestras en los carriles de agarosa y en el primer carril, cargue una escalera de ADN de 1 Kb. Realizar la electroforesis de ADN a 100 V durante 30 min.
- Visualice y documente las bandas de ADN resultantes de la ejecución en un transiluminador UV estándar. Compruebe la eficiencia de la linealización de plásmidos inspeccionando el patrón de las bandas de ADN comparando "cortado" y "sin cortar".
NOTA: Los plásmidos suficientemente linealizados deberían correr más rápido como una banda aguda. Asegúrese de que la escisión del ADN esté completa porque el inicio de la reacción de transcripción es un paso limitante clave que puede verse obstaculizado por la presencia de formas de plásmidos circularizadas, lo que resulta en un rendimiento deficiente del ARNm.
- Proceda a purificar el plásmido linealizado utilizando kits de purificación de ADN basados en columna de centrifugación disponibles en el mercado y almacene la preparación de ADN purificado a -20 °C hasta que sea necesario.
NOTA: En lugar de linealización, es posible utilizar un producto de PCR como plantilla para la transcripción del ARNm, siempre que incluya la secuencia del promotor de la polimerasa aguas arriba y la secuencia de señal de poliA aguas abajo del codón de parada. Los productos de PCR también deben purificarse e inspeccionarse en un gel de agarosa antes de continuar.
- In vitro Transcripción, purificación y control de calidad del ARNm (días 2-3) (Figura 1A)
NOTA: limpie cuidadosamente el área de trabajo y las pipetas con soluciones de descontaminación de ARNasa. Utilice materiales y reactivos libres de nucleasas; Usa guantes. Esto minimizará la contaminación por ARNasa y permitirá un mejor rendimiento de ARNm de longitud completa.
Transcriba ARNm tapados y poliadenilados que codifican WT y proteínas mutantes a partir de plásmidos linealizados utilizando kits estándar para la transcripción de ARNm in vitro y siguiendo las instrucciones del fabricante.- Centrifugar el plásmido linealizado purificado durante unos segundos a 17.949 × g para asegurarse de que los residuos se recogen en el fondo del tubo y no se transfieren a la reacción de transcripción.
- Descongele todos los componentes a temperatura ambiente (RT) excepto la mezcla de análogos NTP y CAP, que se mantienen en hielo. Mantenga la polimerasa a -20 °C hasta su uso.
- Centrifugar brevemente todos los reactivos a 17.949 × g antes de usarlos para evitar la pérdida de reactivos o la contaminación accidental y vórtelos brevemente el tampón de reacción 10x y la mezcla análoga NTP/CAP lista para usar hasta que estén completamente en solución. Mantenga el tampón de reacción 10x en RT.
- Prepare la reacción de transcripción en RT añadiendo 600-800 ng del plásmido linealizado y purificado a una solución que contenga 10 μL de mezcla análoga NTP/CAP 2x, 2 μL de tampón de reacción 10x, 2 μL de enzima SP6, T7 o T3 (dependiendo del promotor específico aguas arriba del CDS en el plásmido) en un volumen final de 20 μL compuesto por agua libre de nucleasas. Mezcle bien pipeteando hacia arriba y hacia abajo varias veces.
- Incubar la reacción de transcripción a 37 °C en un termociclador. Asegúrese también de ajustar la temperatura de la tapa a 37 °C y ejecute la reacción durante 2 h.
- Para eliminar el plásmido residual que no se transcribió durante la reacción, agregue 1 μL de solución de ADNasa estándar (2 unidades/μL). Mezcle bien la reacción pipeteando varias veces e incube a 37 °C durante 30 minutos más.
- Antes de continuar, compruebe la calidad e integridad del ARNm sintetizado in vitro mediante electroforesis de TBE/formamida y agarosa. Disuelva 1,0 g de agarosa en 50 mL de solución 1x TBE, agregue 5,5 mL de formamida al 37% y 3,5 μL de tinte en gel de tinte, mezcle y mezcle el gel como se explicó anteriormente.
PRECAUCIÓN: Cuando manipule formamida, use equipo de protección personal (EPP) y use una capucha química. - Mezcle 1 μL del ARNm transcrito con 2,5 μL de tampón de carga de colorante de formamida 2x en agua libre de nucleasas en un volumen total de 5 μL. Mezcle bien los componentes pipeteando varias veces y cargue las muestras en los carriles de gel. Haga funcionar el gel a 100 mV durante 10-15 minutos en un tampón 1x TBE preenfriado. Ejecute también una escalera de ADN y/o ARN de 1 kb.
- Opcional: Desnaturalizar la escalera y el ARNm a 70 °C durante 10 min.
- Visualice y documente las bandas de ARNm resultantes en un transiluminador UV estándar.
NOTA: Las tiradas largas y las altas temperaturas aumentan la probabilidad de degradación del ARN. - Si la integridad y el tamaño del ARN encapsulado sintetizado son óptimos, se procede a la poliadenilación del C-terminal añadiendo a la reacción: 36 μL de agua libre de nucleasas, 20 μL de tampón de carga desnaturalizante 5x, 10 μL de 25 mM MnCl2, 10 μL de 10 mM de ATP y 4 μL de enzima de poliadenilación (como la poli(A) polimerasa purificada de E. coli ). Mezcle bien la reacción pipeteando varias veces e incube los 100 μL de la reacción final a 37 °C durante 1 h más.
NOTA: No es aconsejable comprobar la calidad del ARN mediante electroforesis en gel después de la reacción de poliA porque el ARN aparecerá manchado debido a las colas de poliA. - Precipitar y recuperar el ARNm sintetizado, tapado y poliadenilado utilizando sales apropiadas como el LiCl. Brevemente, mezcle volúmenes iguales de agua libre de nucleasas y una solución estándar de LiCl de 2,5 M (30 μL) e incube durante >30 min a -20 °C.
NOTA: Se puede obtener una precipitación eficiente después de 2 h, pero la incubación nocturna generalmente aumenta el rendimiento final del ARNm. - Para granular el ARNm purificado, centrifugar la reacción durante 30 min a 4 °C a 17.949 × g y eliminar el sobrenadante. Ahora, lave el pellet con ~ 1 mL de etanol al 70% (EtOH) y centrifugue durante 15 minutos a 4 °C a 17,949 × g para eliminar los contaminantes y los nucleótidos residuales de la reacción.
- Seque el pellet al aire y vuelva a suspenderlo en 20 μL de agua sin nucleasas. Disuelva bien el ARN pipeteando suave y repetidamente en RT e incube a los 10 min.
NOTA: Revise el pellet cada 5 minutos para asegurar la evaporación del EtOH. Al resuspender, dada la pegajosidad del ARN, pipetearlo hasta que se disuelva correctamente en agua. - Evalúe la concentración y la pureza del ARNm preparado midiendo la absorbancia a 260 nm y 280 nm (relación 260/280) en un espectrofotómetro. Mida diferentes diluciones escalares de la preparación de ARN para garantizar una estimación correcta de la concentración.
NOTA: Normalmente, la concentración del ARN sintetizado oscila entre 1 y 2 μg/mL, pero la cantidad final puede variar en función de la longitud del CDS y de la cantidad de material de partida. - Al evaluar la calidad, mantenga el stock de ARNm en hielo, luego alícueste en alícuotas de 5 μL y almacene a -80 °C hasta que sea necesario para la inyección.
- Preparación de soluciones y materiales estándar para la inyección de embriones y el tratamiento farmacológico (Días 2-3, Figura 1B)
- Tire de las agujas de microinyección para embriones de pez cebra utilizando capilares estériles (dimensiones capilares estándar: 1,0 OD x 0,58 ID x 100 L mm). Utilice un aparato extractor disponible en el mercado y aplique los ajustes deseados, en términos de voltaje de salida, modo de tracción (un paso o dos pasos) y fuerza de tracción para obtener una longitud de punta, diámetro y forma de aguja variables. Para conocer las características de las agujas de uso común para el pez cebra, consulte Abdelrahman y colegas11.
NOTA: Los ajustes óptimos pueden variar de un laboratorio a otro, ya que las condiciones ambientales (como la humedad o la temperatura ambiente) pueden influir en los resultados de la extracción de la aguja. Los ajustes deben volver a probarse y calibrarse periódicamente. Las agujas extraídas se pueden almacenar en RT en recipientes limpios durante varios meses. - Prepare la solución madre 30x "Danieau" a pH 7,6 disolviendo 101,7 g de NaCl, 1,56 g de KCl, 2,96 g de MgSO4 x 7H2O, 4,25 g de Ca(NO3)2 y 35,75 g de HEPES en 1 L de agua bidestilada (DD) para preparar la solución de microinyección. Prepare también una solución madre de rojo de fenol al 5% en agua DD para su uso como trazador visible de la mezcla de inyección.
- Prepare la solución de medio E3 para el crecimiento embrionario diluyendo 4 mL de NaCl (5 M), 680 μL de KCl (1 M), 1,32 mL de CaCl2 x 2H2O (1 M) y 1,32 mL de MgSO4 (1 M) en un volumen final de 4 L de agua de ósmosis inversa.
- Prepare soluciones madre de los medicamentos deseados (en este ejemplo: el inhibidor de MEK [MEKi] PD032590) como soluciones madre de acuerdo con la hoja de datos del producto y la solubilidad (10 mM del MEKi de elección disolviendo 5 mg en 1.036 mL de DMSO). Mezclar bien y generar pequeñas alícuotas para almacenar a -80 °C hasta su uso.
NOTA: El DMSO es un disolvente común que se utiliza para disolver compuestos polares y no polares y muestra solubilidad en una amplia gama de disolventes orgánicos, así como en agua. - Prepare una suspensión de camarones en salmuera vivos permitiendo que los quistes de Artemia salina eclosionen en solución de incubación (2 g/L de quistes de Artemia salina en 30 g/L de sal oceánica previamente disuelta en agua de ósmosis inversa) a 28-30 °C durante al menos 18 h, proporcionando una aireación constante.
NOTA: Las condiciones de cultivo y el tiempo de eclosión pueden verse influenciados por las condiciones ambientales de la instalación y los lotes de quistes, y deben volver a probarse regularmente y reajustarse si es necesario.
- Tire de las agujas de microinyección para embriones de pez cebra utilizando capilares estériles (dimensiones capilares estándar: 1,0 OD x 0,58 ID x 100 L mm). Utilice un aparato extractor disponible en el mercado y aplique los ajustes deseados, en términos de voltaje de salida, modo de tracción (un paso o dos pasos) y fuerza de tracción para obtener una longitud de punta, diámetro y forma de aguja variables. Para conocer las características de las agujas de uso común para el pez cebra, consulte Abdelrahman y colegas11.
- Preparación de parejas reproductoras para peces Tg[ef1α:ERK biosensor-nes] (Adolescentes) (Día 3)
- El día del apareamiento, alimente a las parejas adultas con regularidad de acuerdo con el protocolo aprobado de la instalación de animales.
- Limpie el cultivo que contiene camarones en salmuera eclosionados filtrando los quistes no eclosionados o vacíos con dos coladores de diferentes tamaños de filtro (paso de filtro I: 180 μm, paso de filtro II: 112 μm).
- Recoja y lave los camarones en salmuera filtrados en 200 ml de agua de ósmosis inversa para alimentar a 10-15 peces adultos con aproximadamente 5-10 ml de la solución de camarones en salmuera.
NOTA: Verifique la calidad de la solución de camarones en salmuera eclosionados observando su vitalidad, motilidad, tamaño y color y asegúrese de eliminar los quistes vacíos o sin eclosionar, que son indigeribles para los peces y pueden causar daño al tracto gastrointestinal de los peces. Para las parejas reproductoras, es preferible proporcionar la última comida del día al menos 3 h antes de aislar las parejas en el tanque de cría para permitir la digestión de los alimentos y mantener altos niveles de calidad del agua.
- Seleccione parejas de peces que no hayan sido emparejados para el apareamiento en (al menos) las 2 semanas anteriores, para minimizar la angustia y permitir el desarrollo de los gametos. Aclimatar a las parejas de peces adolescentes adultos seleccionados (normalmente con esta composición: 1 ♂ : 2 ♀ ) en tanques de cría adecuados. Separe a las hembras de los machos usando un divisor de tanque hasta la mañana siguiente. Asegure un ciclo de luz/oscuridad estándar de 14/10 h.
NOTA: También se pueden organizar cruces grupales. Sin embargo, en este caso, el desove puede ocurrir en diferentes momentos y la selección de embriones en la misma etapa de nidadas mixtas se vuelve más difícil. La transición de la oscuridad a la luz es fundamental para el éxito del apareamiento.
- El día del apareamiento, alimente a las parejas adultas con regularidad de acuerdo con el protocolo aprobado de la instalación de animales.
- Recolección de embriones y microinyección de WT y ARNm mutantes (Día 4)
- Tan pronto como se encienda la luz en la instalación, retire el divisor del tanque que separa a los machos y las hembras y, si es necesario, cambie aproximadamente el 20% del agua del tanque con agua dulce del sistema de acuicultura de recirculación para mantener la calidad del agua alta sin diluir excesivamente las hormonas liberadas por los peces.
- Deje a los peces intactos y verifique regularmente la puesta de huevos (generalmente en los primeros 30 minutos a 1 hora de luz diurna). Los huevos (2-3 mm) se ponen y caen al fondo del tanque separados por una rejilla tal que los adultos no pueden comerlos. Aísle a los adultos que se han reproducido con éxito y cuele el agua del tanque que contiene los huevos a través de un colador estándar (como un colador de té) para retener los huevos.
NOTA: Es posible volver a poner el mismo pez en el tanque de cría para otra ronda de apareamiento o devolver los peces a los tanques de alojamiento originales, registrando la pareja de apareamiento, la fecha y el rendimiento. - Lave los embriones recolectados con medio E3 fresco y elimine los huevos, heces y otros tipos de desechos no fertilizados y degenerados de las parejas con una pipeta Pasteur.
NOTA: Recoja ~n = 100 óvulos de adolescentes fertilizados en una placa de Petri de 90 mm de diámetro para evitar la sobrepoblación de embriones. - Organice y alinee los óvulos fertilizados de Teen en una placa de microinyección personalizada obtenida mediante moldeo de agarosa al 2% en medio E3 para generar carriles apropiados que deben contener y restringir los óvulos durante la microinyección.
NOTA: Para ello, se pueden utilizar moldes a medida o comerciales. - Prepare la mezcla fresca de microinyección para generar embriones mutantes de la siguiente manera: 30-60 pg de ARNm tapado y poliadenilado (aquí shp2) disuelto en una solución Danieau 0,3x (diluida a partir de una solución madre) para un volumen final de 20 μL. Añadir 0,2 μL (0,05%) de solución madre de rojo de fenol como trazador de microinyección.
- Retrocargue la aguja con 2 μL de material de inyección utilizando una pipeta con microcargador. Inyecte la solución en la etapa de una célula de embriones adolescentes de pez cebra utilizando un dispositivo de microinyección de presión disponible en el mercado, ajustando manualmente la configuración de presión y tiempo para calibrar cada inyección en función de la calidad de la aguja y el embrión. Consulte los protocolos estándar de microinyección de pez cebra disponibles en la literatura 11,12.
- Cría embriones adolescentes microinyectados en condiciones de cría controladas (temperatura: 28 °C, humedad: 70%, ciclo de luz/oscuridad: 14/10 h) para un desarrollo óptimo y limpia los huevos que parezcan turbios o degenerados en las próximas 3 horas después de la deposición.
- A ~4 h después de la fertilización (hpf), cribar los embriones para determinar la fluorescencia (expresión del reportero) utilizando un microscopio estereoscópico de fluorescencia estándar con los ajustes adecuados de lámpara y filtro (465-500 nm). Utilice los peces adolescentes positivos para las imágenes FRET y los hermanos negativos para las evaluaciones inmunohistoquímicas (IHC) (ver más abajo).
NOTA: Dada la variabilidad conocida en la expresión de los transgenes basados en Tol213, los embriones pueden mostrar niveles variables de fluorescencia adolescente . Se recomienda inspeccionar los hermanos no inyectados para controlar la variabilidad interindividual en la fluorescencia dentro de un lote y, teniendo en cuenta el bajo rango dinámico de las imágenes FRET, descartar los embriones con niveles extremadamente bajos de fluorescencia basal.
- Tratamiento de embriones de pez cebra con el inhibidor de MEK (MEKi) (Día 4)
- Prepare una solución intermedia de MEKi PD0325901 (1 mM) diluyendo 1 μL de solución madre de 10 mM con 9 μL con medio E3. Utilice la solución intermedia para obtener soluciones finales de dosis bajas. Prepare una dosis baja (0,25 μM) del PD0325901 MEKi en un volumen total de 3 mL mezclando 0,75 μL de la solución intermedia de 1 mM con 2,25 μL de medio E3. Diluir 0,75 μL de DMSO al 10% con 2,25 mL de medio E3 para obtener la solución de control (Ctrl).
- Coloque piscinas de embriones de igual número en diferentes pocillos de una placa de 6 pocillos e inicie el tratamiento por inmersión en baño: para cada pocillo, intercambie el medio E3 por un medio E3 que contenga un vehículo de control (0,0025% DMSO) o fármacos diluidos a la concentración deseada (0,25 μM, 0,0025% DMSO) como se mencionó anteriormente. Use 3 mL de solución de medicamento por pocillo.
NOTA: Para eliminar cualquier efecto causado por las diferentes concentraciones del portador (DMSO), es importante asegurarse de que tanto las soluciones de control como las de tratamiento tengan la misma concentración final de DMSO. - Mantener los embriones tratados a 28 °C antes de recolectarlos hasta la etapa de desarrollo deseada para los ensayos de seguimiento (aquí: análisis morfométrico y validación de IHQ).
2. Imágenes FRET multiespectrales en vivo de modelos de pez cebra RASopathy en la etapa de gástrula y análisis de datos
- Montaje de gástrulas para imágenes en vivo (Día 4) (Figura 2A)
- Prepare el medio de montaje para embriones vivos disolviendo 1,5 g de agarosa (LMA) de bajo punto de fusión en 1x PBS para producir 1,5% de LMA/E3.
NOTA: Elija la concentración de agarosa de 0.8 a 1.5% dependiendo de la etapa de desarrollo y la necesidad específica de sujetar a los peces. Distribuya la solución de LMA recién hecha en alícuotas del formato deseado y almacene LMA en un estado polimerizado en RT (estable durante unos meses). Esto minimiza la contaminación bacteriana y fúngica, pero verifique la calidad de la alícuota LMA regularmente antes de usarla. - Antes de montar la muestra, fundir la alícuota de LMA al 1,5% en un termomezclador o en un baño de agua estéril a 50 °C. Una vez disuelto, disminuya la temperatura de la termobatidora a aproximadamente 30 °C.
- Coloque un solo embrión Teen+ inyectado (en este caso, una gástrula que expresa ShpD61G) en el centro de una placa con fondo de vidrio de 35 mm de diámetro para obtener imágenes en vivo y oriéntelo con un cabello fino. Retirar el exceso de medio E3 e inmovilizar el embrión poniendo una gota de LMA encima y dejándolo polimerizar a RT.
NOTA: Al comienzo del proceso de polimerización, el pez se puede orientar a la posición deseada. Se recomienda estrictamente utilizar el LMA a una temperatura no superior a 35 °C para evitar daños en los tejidos.
- Prepare el medio de montaje para embriones vivos disolviendo 1,5 g de agarosa (LMA) de bajo punto de fusión en 1x PBS para producir 1,5% de LMA/E3.
- Ajuste de los parámetros de microscopía y realización de imágenes FRET multiespectrales de gástrulas (Día 4) (Figura 2B)
NOTA: El protocolo se aplica al uso de cualquier plataforma de microscopía confocal equipada con todo el hardware y software necesarios para realizar imágenes FRET. Aquí se utilizó un microscopio confocal equipado con un láser de iones de argón con líneas de longitud de onda de 458-476-488-496-514 nm, un divisor de haz acústico-óptico programable (AOBS) que separa la luz de excitación y emisión, dos detectores híbridos espectrales (HyD) y una incubadora de etapa para mantener condiciones estables de temperatura (a 28 °C) y humedad durante la obtención de imágenes en vivo de las muestras.
El protocolo de imagen FRET multiespectral en vivo, tal como se describe, se puede aplicar a embriones en diferentes etapas de desarrollo más allá de las gástrulas tempranas, como lo demuestran Fasano et al.7. Se debe considerar una pila z más grande para las etapas de desarrollo posteriores (por ejemplo, 24 hpf), así como la configuración adecuada de los intervalos z y t para permitir pasos de detección multiespectrales durante la adquisición de imágenes FRET espectrales.- Encienda el controlador de la incubadora al menos 1 h antes de iniciar la adquisición y ajuste la temperatura a 28 °C para mantener los embriones de pez cebra en un estado saludable. Una vez que la temperatura de la incubadora se haya estabilizado, coloque la placa de imágenes con el embrión en el soporte de muestras y use un objetivo 10x/seco (apertura numérica de 0,4) para visualizar rápidamente la muestra.
- En la configuración del láser del software al que se hace referencia, encienda el láser de iones de argón y utilice el control deslizante para ajustar la potencia del láser al 50%. En Configuración de hardware, elija la resolución de profundidad de 8 bits a la que se adquirirán las imágenes.
- Seleccione el modo de escaneo XYλZ de detección espectral en el panel de adquisición y configure los siguientes parámetros de adquisición: formato de imagen: 512 x 512 px; velocidad de escaneo de 400 Hz; Zoom óptico de 0,75.
- Active la línea láser de 458 nm del láser de iones de argón y ajuste su valor de intensidad, normalmente <10% (aquí 8,5%).
- Seleccione un detector HyD y ajuste la sensibilidad del detector (ganancia) introduciendo el valor deseado (aquí 500).
NOTA: Para muestras vivas débilmente fluorescentes y para acumular menos ruido de fondo, es aconsejable utilizar detectores híbridos en lugar de tubos fotomultiplicadores (PMT).
En el software al que se hace referencia, la visualización del espectro de emisión de un tinte requiere la activación de un detector. - Para comenzar, active el primer detector (HyD) y seleccione la proteína de fluorescencia cian (CFP) para mostrar su curva de emisión. Abra el menú desplegable de la barra de detectores del software para abrir la lista de selección de la curva de emisión CFP. Ahora, active un segundo detector para mostrar la curva de emisión de la segunda proteína de fluorescencia amarilla (YFP) del colorante. Para mostrar también la curva de emisión Ypet , active un segundo detector, luego seleccione la curva de emisión YFP que aparecerá en la imagen del espectro, después de apagar el segundo detector.
NOTA: Una vez completado este paso, se debe desactivar el segundo detector, ya que solo se utiliza un detector en el modo de adquisición espectral. - Para iniciar las adquisiciones en vivo, coloque el cursor de detección en el rango de emisión de señal más intensa (en este caso, el de YFP) para visualizar la muestra, decida y establezca las posiciones inicial y final del grosor de la muestra en la ventana z-stack LAS X.
- Para las propiedades del rango de escaneo λ, establezca los siguientes parámetros: inicio (460 nm) y fin (570 nm) del rango de detección; Ancho de la banda de detección: 5 nm; λ-scan Tamaño del paso: 5 nm. Inicie la adquisición de z-stack.
NOTA: Los parámetros indicados permiten a los usuarios obtener una imagen relativamente rápida con una resolución aceptable, pero se pueden utilizar diferentes configuraciones. En la configuración del disco fluorador, el filtro NOTCH insertado automáticamente se puede deseleccionar para evitar la pérdida de intensidad única. - Para evaluar los cambios en la señal en tiempo real tras la exposición a MEKi, monte un único embrión mutante (en este caso, Shp2D61G) en un plato de vidrio y tome una imagen antes y después del tratamiento con fármaco. Para estos experimentos en vivo, realice directamente el tratamiento farmacológico mediante inmersión en baño durante la sesión de imagen con un máximo de 2 mL de la solución que contiene el fármaco disuelto.
- Postprocesamiento de imágenes y separación de colorantes multiespectrales para obtener imágenes FRET radiométricas (Día 5) (Figura 2C)
- Para separar la emisión del donante (CFP) y del aceptor (Ypet), asegúrese de eliminar la contribución de la emisión de fluorescencia de ambas moléculas en el canal FRET, lo que se denomina sangrado espectral (SBT).
- Consulte las bases de datos de colorantes/proteínas fluorescentes disponibles públicamente para mostrar y descargar el archivo .cvs relativo a un espectro de emisión CFP estándar (espectros de excitación y emisión).
- En el panel Datos del archivo .cvs, elija la opción Texto a columnas y divida los datos en columnas utilizando una coma como delimitador. El archivo resultante incluirá información adicional (por ejemplo, excitación CFP, CFP 2P) que debe eliminarse. Conserve solo los datos de la columna relativos a la longitud de onda y la emisión.
- En la columna de emisiones de CFP, elimine todos los datos de intensidad a partir de 501 nm.
- Guarde el archivo de espectro de emisión CFP adaptado de 460 a 500 nm en formato .xls (denominado "eCFP modificado" en la Figura 2C).
- Repita el mismo procedimiento para descargar y procesar el espectro de emisión de proteínas YFP y conserve solo los datos de la columna relativos a la longitud de onda y la emisión. Elimine todos los valores de emisión de hasta 524 nm. Guarde el archivo de espectro de emisión YFP adaptado de 525 a 650 nm in.xls formato (llamado "Ypet de 525 nm" en la Figura 2C).
- Para permitir la exclusión del sangrado espectral (SBT) en la base de datos de colorantes disponible en la ventana Configuración del software, inserte y guarde dos espectros de emisión de referencia adaptados para CFP e YFP.
- Seleccione el archivo de imagen espectral resultante de la sesión de imágenes, abra la ventana Proceso y seleccione Separación de colorante espectral en la herramienta Separación de color. Configure los ajustes para la separación de colorantes de la siguiente manera: en las listas desplegables en el lado izquierdo del cuadro de diálogo, seleccione el nuevo espectro de emisión CFP (eCFP modificado) en la primera posición y el nuevo espectro de emisión YFP (Ypet a partir de 525 nm) de la base de datos de espectro.
- En Reescalar, seleccione Por canal para escalar los canales individualmente y, a continuación, en el escaneo λ de las imágenes, elija el que tenga la mayor intensidad de señal (correspondiente al paso 14 del escaneo espectral al nivel del pico de emisión del canal FRET, indicado por una flecha blanca en el panel que muestra los pasos de detección a la derecha de la Figura 2B). A continuación, desplácese a lo largo del escaneo Z de la muestra y elija la sección óptica que resalte el área de interés en la zona de margen.
- Utilice el modo de selección de ROI en la zona de margen del poste del animal para definir el área con el mejor espectro. Haga clic en ROICrosshair indicado en la parte superior de la ventana de visualización para acceder a la cruz y ajustar el tamaño del ROI de referencia introduciendo el valor de 40 vóxeles en el Área de mediciones. Defina con precisión el área de interés por el ROICrosshair. En el diagrama de la imagen, elija Normalización de datos y, a continuación, haga clic en Aplicar para realizar la separación del tinte con los ajustes configurados.
- Abra este archivo recién generado obtenido con los dos canales separados y produzca una imagen de proyección bidimensional de la serie de imágenes tridimensionales (proyección de máxima intensidad) para la visualización de datos.
- En la ventana Proceso , seleccione Recortar para separar los canales en dos archivos separados, los canales CH1 y CH2 (o FRET).
- En la ventana Proceso , seleccione Combinar imágenes, seleccione el archivo CH2 e insértelo en la primera opción y, a continuación, seleccione el archivo CH1 e insértelo en la segunda opción. Establezca un cambio de escala con el factor 5 y elija la operación Ratio. A continuación, haga clic en Aplicar para generar el nuevo archivo que contiene la imagen radiométrica (YFP/CFP). Guarde el archivo para el análisis de datos de seguimiento.
- Para separar la emisión del donante (CFP) y del aceptor (Ypet), asegúrese de eliminar la contribución de la emisión de fluorescencia de ambas moléculas en el canal FRET, lo que se denomina sangrado espectral (SBT).
- Análisis cuantitativo de señales FRET y representación de imágenes (Días 5-6) (Figura 2D)
- Para las mediciones cuantitativas de las señales FRET a partir de imágenes radiométricas FRET/CFP, se realiza el experimento en N>2 embriones (réplicas), basándose en una evaluación estadística a priori basada en el efecto esperado. Emplee un paquete de procesamiento de imágenes como el software de código abierto Fiji. Importe los archivos de imagen obtenidos a partir de imágenes espectrales y separación de colorantes en un paquete de análisis de imágenes como el Fiji de código abierto .
- Antes de comenzar con la selección del ROI en las imágenes de gástrulae, establezca los parámetros como mediciones de lectura mediante la función Analizar | Establecer medidas | seleccione los parámetros de interés, como Área, Densidad integrada y Valor medio de gris.
- Seleccione la región de interés (en este estudio específico: el margen de gástrula) utilizando la herramienta de selección de polígonos de la barra de herramientas. Guarde las especificaciones del ROI x,y haciendo clic en Analizar | Herramientas | Gerente de ROI.
- Para realizar mediciones en un ROI seleccionado, haga clic en Analizar | Medir. Repita estos pasos para cada ROI e imagen/condición.
NOTA: Si es necesario, guarde la imagen como . Formato TIFF. Es posible guardar todas las mediciones de ROI derivadas de varias imágenes como un archivo de ROI. Cambie el nombre de cada medida en la lista del administrador de ROI con el nombre correcto en relación con cada muestra o imagen. - Para cada condición (mutante y mutante + tratamiento aquí), extraiga todos los valores de la relación FRET/CFP para cada ROI seleccionado obtenido y organícelos en una hoja de trabajo organizando, por ejemplo, los grupos experimentales en columnas y cada valor bruto en filas.
- Utilice cualquier software adecuado para evaluar las diferencias estadísticas de las señales FRET entre diferentes condiciones experimentales y para la generación de gráficos y la visualización de datos.
NOTA: El software de código abierto y con licencia está disponible para soluciones de análisis de datos y gráficos. - Organizar un proyecto específico para la evaluación estadística considerando todas las mediciones brutas y el número de réplicas para cada condición experimental. Para el análisis estadístico de una sola réplica biológica, cree una tabla de columnas que tenga una variable de agrupación, con cada grupo definido por una columna. Para el análisis estadístico de más de una réplica biológica, cree tablas agrupadas que tengan al menos dos variables de agrupación, una definida por columnas (por ejemplo, condiciones experimentales) y la otra definida por filas (por ejemplo, valores medios de réplicas).
- Después de organizar adecuadamente los grupos experimentales en la hoja de trabajo, evalúe la distribución de los datos (prueba de normalidad, por ejemplo, D'Agostino-Pearson, Anderson-Darling) y, en función del resultado, elija la prueba estadística más adecuada.
- Realice un ANOVA de un factor para datos paramétricos (siguiendo la distribución gaussiana) y una prueba de Kruskal-Wallis para datos no paramétricos. Si hay más factores presentes (genética y concentraciones de fármacos), considere un ANOVA de dos vías. Compare el valor medio de FRET para cada condición entre sí (comparaciones múltiples) y corrija las comparaciones múltiples utilizando una prueba post hoc (por ejemplo, la prueba de Tukey y la de Dunn para datos paramétricos y no paramétricos, respectivamente).
3. IHQ: validación de los resultados de FRET y análisis morfométrico correlativo de los defectos de gastrulación
- Preparación de soluciones y fijación y montaje de embriones para IHQ frente a tERK y pERK (Día 7) (Figura 3A)
- Preparar las soluciones de trabajo necesarias para IHC.
- Prepare una solución madre de 10x PBS disolviendo 80 g de NaCl, 2 g de KCl, 14,4 g deNa2HPO4 y 2,4 g de KH2PO4 en 1 L de agua DD. Esterilizar la solución en autoclave para mantenerla estéril.
- Prepare una solución de PBS-Triton (PBSTr) al 0,8 % disolviendo 8 ml de Triton X-100 al 10 % en un volumen final de 100 ml de 1x PBS.
- Prepare soluciones de glicerol al 20%, 50% y 80% diluyendo 3 mL, 7,5 mL y 12 mL de glicerol al 100%, respectivamente, en un volumen final de 15 mL de 1x PBS.
- Prepare paraformaldehído (PFA) al 4%/PBSTr al 0,25% (solución fijadora) mezclando 2,5 mL de PFA al 16% en 250 μL de Triton X-100 al 10% previamente disuelto en un volumen final de 10 mL de 1x PBS. Utilice esta solución para fijar embriones a 6 hpf durante la noche a 4 °C. Utilice tubos de 2 ml y un máximo de cinco embriones por tubo para permitir una fijación y lavados eficientes.
NOTA: Se recomienda preparar un fijador nuevo para cada ronda de fijación. PRECAUCIÓN: Cuando manipule formaldehído al 16%, use equipo de protección personal (EPP) y debajo de una capucha química. - Lave los embriones en los tubos de 2 mL llenando los tubos con 1,5 mL de PBSTr al 0,8% durante varias veces. Transfiera los embriones a nuevos tubos de 2 ml y almacene los embriones en 1x PBS hasta su uso.
NOTA: Si no está familiarizado con el manejo de embriones tempranos de pez cebra, trabaje bajo un microscopio estereoscópico para evitar la pérdida de embriones durante el pipeteo. Utilice material de plástico de "baja unión" para reducir la posible pegajosidad de los embriones a la pared del tubo.
- Preparar las soluciones de trabajo necesarias para IHC.
- Permeabilización tisular e IHQ frente a pERK y tERK (Días 7-9)
NOTA: Los resultados de IHQ dependen de múltiples variables que pueden diferir entre los laboratorios y puede ser necesaria una optimización específica de los protocolos.- Lave los embriones fijos de pez cebra en 1 mL de PBSTr al 0,8% durante 3 x 10 min a RT.
- Permeabilizar las muestras con 2 μL de proteinasa K diluida en PBSTr al 0,8% (1:1.000) durante 2 min en RT.
- Detenga la permeabilización del tejido lavando las muestras en 1 mL de PBSTr al 0,8% durante 3 x 10 min, y fije las muestras en 500 μL de PFA al 4%/1x PBS durante 20 min a RT. A continuación, lavar las muestras en 1 mL de PBSTr al 0,8% durante 3 x 10 min.
NOTA: El método y el tiempo utilizados para la permeabilización de los tejidos son pasos críticos que pueden verse influenciados por el tipo de antígeno, la calidad de la conservación del tejido y los factores ambientales. Optimice el protocolo antes de aplicarlo en muestras valiosas. - Incubar las muestras con 1x tampón de bloqueo (BB) que contenga 5% de suero de cabra normal (NGS), 1% de albúmina sérica bovina (BSA), 1% de DMSO, preparado de la siguiente manera: 75 μL de NGS al 100%, 150 μL de BSA al 10% y 15 μL de DMSO al 100% en un volumen final de 1,5 mL de PBSTr al 0,8%. Utilice 200 μL de la solución por tubo e incube los embriones durante 20-30 min a RT en la solución.
- Retire el 1x BB del paso anterior e incube las muestras en una solución que contenga una mezcla de los anticuerpos primarios deseados (en este caso: diluyendo 1 μL de anticuerpo monoclonal primario de ratón p44/42 MAPK, ERK total, tERK, + 1 μL de fosfo-p44/42 MAPK policlonal de conejo, ERK fosforilado, pERK) en 250 μL de solución 1x BB recién preparada. Utilice 200 μL de la solución por tubo e incube los embriones en la solución durante la noche a 20 °C.
- Para eliminar la unión inespecífica, lavar las muestras con 1 mL de PBSTr al 0,8% varias veces (primero lavar cada 10 min y luego cada 30 min).
- Incubar las muestras en 1x BB recién preparado durante 20 min a RT. Utilice 200 μL por tubo.
- Retire el 1x BB del paso anterior e incube las muestras con una mezcla de los anticuerpos secundarios (1 μL de anti-ratón de cabra conjugado fluorescente 488, + 1 μL de anti-conejo de cabra 633 conjugado con fluorescencia en 600 μL de solución de 1x BB). Dejar los embriones agitando suavemente durante la noche a 20 °C.
NOTA: Realizamos IHQ en peces adolescentes negativos (no transgénicos) del mismo lote utilizado para la obtención de imágenes FRET. Alternativamente, también se pueden usar peces Teen+ , pero en este caso, se deben emplear anticuerpos secundarios conjugados con fluoróforos que no se superponen con los espectros de emisión de CFP o YFP. - Elimine el enlace no específico como se ha descrito anteriormente.
- Lave las muestras en un gradiente de soluciones de glicerol/1x PBS (20%, 50%, 80%) durante 15 min por cada solución en RT. Almacene las muestras en glicerol al 90%/1x PBS a 4 °C.
NOTA: Para minimizar el posible decaimiento de la fluorescencia, almacene las muestras en la oscuridad.
- Microscopía confocal de tejido inmunoteñido y análisis cuantitativo (Días 10-11) (Figura 3B-E)
- Monte los embriones como se describió anteriormente.
- Ajuste los parámetros de adquisición de microscopía confocal de la siguiente manera: láser blanco al 80%, rango de emisión de fluorescencia a 507 - 551 nm para el anti-ratón de cabra conjugado con fluorescencia (en este caso con el espectro de emisión de 488 nm, utilizado para tERK), 644 - 740 nm para el anti-conejo de cabra conjugado con fluorescencia (en este caso con el espectro de emisión de 633 nm, utilizado para pERK). Seleccione el modo secuencial de adquisición, un formato de escaneo de 512 x 512 px y una velocidad de 400 Hz. Defina el inicio y el final del escaneo con un grosor de tamaño de paso z de 4 a 5 μm y un zoom digital estándar de 1. Utilice el objetivo de inmersión en agua 25x (con una apertura numérica de 0,05) para adquirir toda la gástrula a 6 hpf
ATENCIÓN: Este protocolo incluye la aplicación de láseres que pueden ser dañinos; Por lo tanto, requiere capacitación del personal de acuerdo con los requisitos nacionales específicos. - Después de la adquisición de la imagen confocal, inspeccione el archivo de imagen sin procesar obtenido mediante un paquete de procesamiento de imágenes como el software de código abierto Fiji.
- En Fiji, utilice el comando Dividir canales del menú Imagen y submenú Color para obtener una imagen de un solo canal (ERK total: canal = 0, C = 0; pERK: canal = 1, C = 1) y genere una imagen de proyección z de intensidad máxima para ambos canales haciendo clic en Imagen | Pilas | Proyecto Z.
- Repita el procedimiento para las mediciones de la intensidad del ROI como se describió anteriormente y extraiga los datos en una hoja de cálculo.
- Para inferir los cambios en la intensidad de la señal p-ERK en el ROI deseado entre los grupos experimentales, calcule la relación p-ERK/t-ERK dividiendo los valores de densidad integrada bruta de la tinción p-ERK por la densidad integrada bruta de la señal t-ERK. Proceda a la evaluación de la significación estadística como en el paso 2.4.
NOTA: La configuración de los parámetros de imagen se puede modificar en función de las necesidades específicas y teniendo en cuenta la duración de cada muestra, la calidad deseada de las imágenes y el número de planos a adquirir.
- Mediciones de ejes en 11 embriones hpf (Día 4 para la fijación embrionaria, Día 12 para la adquisición y análisis de datos) (Figura 4A-D)
- Al final de la gastrulación (11 hpf), fijar los embriones con PFA al 4% en 1x PBS durante 20 min en RT, lavar varias veces en 1x PBS y almacenar a 4 °C hasta la adquisición de la imagen.
- Oriente los embriones lateralmente con una pipeta Pasteur en un solo pocillo de una placa de 12 pocillos que contenga 1x PBS fresco.
- Para evaluar la presencia de una forma ovalada (relación de ejes embrionarios Y/X > 1), un fenotipo característico de rasopatía en el pez cebra y la relación de rescate morfológico de los ejes embrionarios como resultado de la eficacia del tratamiento, adquiera imágenes de embriones enteros utilizando un microscopio estereoscópico con un objetivo de aumento de 3,4x (objetivo de escaneo de 0,63x x factor de zoom de 8,60) simplemente capturando imágenes en un modo de campo claro.
- Importe el archivo de imagen en un paquete de análisis de imágenes, como el Fiji de código abierto.
- Mida la longitud de los ejes de los embriones (x, eje menor; y, eje mayor) seleccionando la herramienta recta de la barra de herramientas y haciendo clic en Analizar | Medir. Después de realizar las mediciones seleccionadas, agréguelas a la lista del administrador de ROI y guarde el archivo de ROI como se explicó anteriormente para el análisis de imágenes FRET.
NOTA: Repita estos pasos para cada imagen de embrión.
- Mida la longitud de los ejes de los embriones (x, eje menor; y, eje mayor) seleccionando la herramienta recta de la barra de herramientas y haciendo clic en Analizar | Medir. Después de realizar las mediciones seleccionadas, agréguelas a la lista del administrador de ROI y guarde el archivo de ROI como se explicó anteriormente para el análisis de imágenes FRET.
- Proceda a exportar los datos en una hoja de trabajo y calcule la relación de ejes dividiendo la longitud del eje mayor (y) por la longitud del eje menor (x). Calcule la media, la desviación estándar de la media o el error estándar de la media de diferentes réplicas.
- Proceda al análisis estadístico de datos como en el paso 2.4.
Resultados
Este protocolo muestra un flujo de trabajo simple para generar rápidamente modelos transitorios de RASopatía en embriones de pez cebra y evaluar las fluctuaciones de ERK en mutantes tempranos con un método estándar de imágenes FRET en vivo aplicado a un sensor de pez cebra ERK recientemente establecido 6,9. Como se ha demostrado recientemente 6,7 dentro del mismo flujo de trabajo experimental, los resultados de FRET pueden ser validados de forma cruzada por IHQ estándar contra ERK fosforilada y total en embriones enteros. El deterioro de la formación de los ejes corporales que se correlaciona con una mayor activación de ERK puede seguirse en etapas posteriores, incluso en los mismos embriones utilizados para FRET7. Aquí, se muestran las fluctuaciones típicas de ERK en las gástrulas tempranas registradas por FRET, que están relacionadas con la expresión de Shp2D61G causante de NS. También se adquirieron datos de mutantes en los que la señal fue modulada negativamente por el tratamiento con dosis bajas con un MEKi, inhibiendo la cascada de señalización RAS/MAPK.
Para generar modelos NS, primero producimos ARNm tapado y poliadenilado de alta calidad que codifica Shp2 (forma salvaje y mutante) mediante transcripción in vitro a partir de un plásmido linealizado adecuado que alberga CDS de longitud completa shp2 . Como se muestra en la Figura 1A, cuando la mayor parte del plásmido se linealiza con éxito ("corte"), se puede observar una banda afilada que corre en un gel de agarosa estándar de 1-1,5% (aquí en el ejemplo del tamaño de 7.500 pb), mientras que el ADN "sin cortar", utilizado aquí como control de plásmido no digerido, muestra diferentes conformaciones posibles, típicas del ADN circular no digerido. A continuación, se puede producir ARNm tapado y poliadenilado in vitro. En el gel de agarosa que contiene formamida, el ARN intacto debe verse como una banda conspicua alrededor del tamaño esperado (Figura 1A, derecha) sin frotis notables (indicativo de degradación del ARNm) o presencia de fragmentos más pequeños. Es aconsejable no proceder si se obtiene ARN fragmentado o degradado. La preparación debe repetirse al solucionar el problema. Como se muestra en los esquemas de la Figura 1B, se puede inyectar ARNm de buena calidad con tapa y poliadenilado que codifica el alelo RASopatía deseado y un control WT (en nuestro caso Shp2D61G y Shp2WT como control) en embriones de pez cebra en etapa unicelular que expresan el reportero adolescente . A continuación, los embriones se crían hasta la etapa deseada (en este caso, las primeras etapas durante la gastrulación).
La Figura 2 ilustra los esquemas y los resultados representativos del flujo de trabajo utilizado para registrar las señales FRET en nuestro modelo RASopathy in vivo. Las gástrulas teen+ que expresan el alelo NS mutante Shp2D61G se elevaron y recolectaron en la etapa deseada (4 hpf) en agar (LMA) de bajo punto de fusión al 1,5% en medio E3 (Figura 2A). En la Figura 2B se muestra un ejemplo de los ajustes generales del microscopio y los ajustes de adquisición de la pila lambda (λ) que utilizamos para obtener imágenes FRET multiespectrales a partir de la excitación del donante (405 nm) en embriones adolescentes. Es importante destacar que, para garantizar una recopilación suficiente de espectros, establecemos el ancho de banda λ en 5 nm. Utilizamos un tamaño de paso relativamente alto (aquí 8 nm) para obtener escaneos x, y, λ, z en un tiempo razonable, considerando el modo de adquisición en vivo (adquisiciones con un intervalo de tiempo de aproximadamente 13 minutos).
Después de la adquisición de la imagen, se inspeccionó la intensidad de la señal registrada a lo largo de todo el espectro λ para asignar las mejores ventanas de emisión y proceder a la desmezcla de los espectros CFP (D) e Ypet (A) (Figura 2C, izquierda). Aquí, al inspeccionar la región del margen del polo animal del embrión mediante un enfoque basado en el ROI incorporado en el asistente de separación de colorantes espectrales del software, nos enfocamos en asignar las mejores ventanas espectrales de emisión para los dos fluoróforos, evitando cualquier superposición espectral entre las moléculas D y A. En la Figura 2C, panel superior derecho, se muestran los canales individuales sin procesar representativos (CFP o Ypet, verde y rojo, respectivamente) obtenidos por este enfoque y la imagen radiométrica FRET/CFP (gris) resultante de la aplicación de la función Ratio. El panel inferior muestra un resultado subóptimo representativo de embriones reporteros adolescentes, que ocasionalmente muestran una intensidad de señal insuficiente, como se observa a menudo dentro de una población de peces transgénicos. Estos embriones deben descartarse de antemano y se debe tener cuidado para evaluar los embriones con niveles similares de expresión adolescente en el análisis.
Después de la inspección de la señal en el margen del polo animal, donde se concentra la actividad de ERK durante los movimientos de las células de epibolía y gastrulación, se realizó la representación de la imagen utilizando pseudocoloración LUT "inteligente" en Fiji. Esto permite una mejor visualización de las diferencias de intensidad de la señal espacial en la imagen radiométrica resultante. Las imágenes FRET radiométricas resultantes en la Figura 2D ya muestran una clara tendencia de reducción de la intensidad de la señal en Shp2D61G en la región del margen después de aproximadamente 13 min de tratamiento con el MEKi, PD0325901 seleccionado (aquí PD) a dosis bajas (0,25 μM)7. Los datos demuestran la idoneidad del sensor Teen y el protocolo de imagen FRET multiespectral utilizado aquí para detectar cambios dinámicos de ERK en vivo en un modelo RASopathy durante las primeras etapas de desarrollo, así como la modulación fina de la señal asociada a la enfermedad tras el tratamiento con MEKi a dosis bajas.
Paralelamente, validamos los resultados de nuestro enfoque FRET aplicado a modelos de peces RASopathy mediante fluorescencia IHC frente a ERK total (t-ERK) y su forma fosforilada (p-ERK). Al normalizar la señal obtenida (p-ERK/t-ERK) dentro de un ROI, emergen células con altos niveles de ERK. La Figura 3 muestra un ejemplo de validación de la actividad de ERK a través de IHQ para Shp2WT, Shp2D61G y Shp2D61G tratados con PD en dosis bajas de 4 hpf a 6 hpf (tratamiento más largo en comparación con la ventana de tratamiento de 13 minutos que se muestra durante las imágenes FRET en vivo). La inmunotinción se realizó utilizando anticuerpos secundarios fluorescentes con espectros de emisión no superpuestos para t-ERK y p-ERK (488 y 633 nm, respectivamente).
Los 6 embriones hpf se montaron en LMA al 1,5% disueltos en PBS y se establecieron configuraciones confocales estándar para obtener escaneos x, y, z de todo el volumen embrionario (Figura 3A, B). La configuración confocal estándar con una resolución de 512 x 512 px a 400 Hz nos permite inspeccionar la localización de la señal, lo que es importante para determinar el buen resultado de la inmunofluorescencia antes de la cuantificación de la señal. Como era de esperar, y como se muestra en el resultado representativo, para t-ERK (verde), observamos una tinción casi ubicua dentro de las células del margen, mientras que la señal de p-ERK (rojo) se limitó principalmente a los núcleos (Figura 3C). Las imágenes en bruto, como imágenes monocanal y fusionadas, se procesaron inicialmente mediante la realización de una proyección en z para obtener imágenes de todo el embrión. Se aplicó pseudocoloración de p-ERK (cereza) para resolver mejor el contraste con t-ERK (verde) (Figura 3D). En comparación con Shp2WT, en Shp2D61G se pudo observar un aumento en el número de núcleos de p-ERK+ dentro de la región del margen, que se rescató en los embriones Shp2D61G tratados de 4 hpf a 6 hpf con 0,25 μM de DP. Los datos confirman los resultados obtenidos por la imagen FRET y demuestran la modulación molecular efectiva obtenida por la dosis baja de MEKi de elección. De manera similar a las imágenes FRET, la señal de las adquisiciones de pila z de un solo canal se inspeccionó en la región del margen del polo animal utilizando un análisis basado en ROI utilizando Fiji, y los valores de p-ERK y t-ERK se midieron por separado y se extrajeron para la normalización de seguimiento (p-ERK/t-ERK) y el análisis estadístico (Figura 3E).
Por último, la Figura 4 proporciona datos morfométricos representativos que se pueden obtener y medir, idealmente en las mismas muestras de imágenes FRET en vivo y subsecuentemente aumentadas de 6 a 11/12 hpf o en sus hermanos. Las mediciones alteradas de los ejes menor y mayor (relación ejes mayor/menor) en esta etapa embrionaria pueden resaltar los defectos de los movimientos de gastrulación asociados con la RASopapatía. Los datos se pueden utilizar para examinar el resultado fenotípico de las nuevas variantes descubiertas en pacientes con patogenicidad incierta y para correlacionar la fuerza del fenotipo con la de la alteración molecular (activación de ERK) medida con FRET. En el ejemplo que nos caracteriza, los hermanos de los embriones adolescentes (grupo control 1: Shp2WT, así como grupos 2 y 3: Shp2D61G y Shp2D61G tratados con dosis bajas de PD entre 4 y 6 hpf) se criaron por separado hasta 11 hpf y luego se fijaron en PFA al 4% (Figura 4A) para evitar un mayor desarrollo embrionario que pudiera interferir con la comparación de datos entre individuos. Las imágenes de los embriones laterales se adquirieron utilizando una modalidad simple de campo claro disponible para configuraciones estándar. La elongación embrionaria se evaluó en términos de medidas de ejes mayores y menores (Figura 4B,C). Las medidas de los resultados (Figura 4D) reflejan y corroboran los datos obtenidos con las imágenes FRET y demuestran una correlación entre la actividad dinámica de ERK en el margen del polo animal y los resultados de gastrulación. En conjunto, el conjunto de datos representativo que se muestra aquí para el alelo shp2D61G asociado a NS con o sin tratamiento MEKi PD0325901 muestra la utilidad de las imágenes FRET multiespectrales en peces reporteros Tg[ef1α:ERK biosensor-nes] (Teen) para predecir la patogenicidad de las variantes para NS en embriones tempranos.

Figura 1: Análisis de las preparaciones de plásmidos y ARNm necesarias para generar modelos transitorios de pez cebra NS. (A) El panel representa un esquema y resultados representativos del resultado de la electroforesis de ADN visualizado en un transiluminador UV, utilizado para evaluar la linealización eficiente de plásmidos y la transcripción de ARNm de buena calidad. Los plásmidos linealizados ("cortados") se obtienen por digestión con una enzima de restricción (en este caso KpnI) y se comparan con el plásmido de control no digerido ("sin cortar"). Lo ideal es observar una sola banda que corra más rápido que la "sin cortar" en la linealización del plásmido. Tamaño del plásmido: ~ 7.500 bps: (plásmido de columna + shp2 CDS). Observe cómo el plásmido circular "sin cortar" muestra diferentes formas de ADN de moléculas relajadas y superenrolladas. Una escalera de 1 kb (carril 1), cuyos fragmentos de peso molecular oscilaban entre 0,5 y 10 kilobases (kb), se cargó en el mismo gel para la inspección del tamaño de los fragmentos de ADN. A la derecha, se muestra una serie de gel representativos de ARNm recién transcritos (en este ejemplo, ARNm transcritos que codifican SPRED2 humano asociado a Rasopatía). Dos escaleras que corren de manera ligeramente diferente (escalera de ADN, carril 1 y escalera de ARN, carril 2) con un rango molecular similar de 0,5 a 10 kb se cargan para la inspección del tamaño de los fragmentos de ARN. (B) Descripción general del esquema que describe el uso de los ARNm transcritos que codifican variantes asociadas a RASopathy, para generar modelos de enfermedades transitorias mediante inyección en embriones de pez cebra en etapa de una célula. El ARNm que codifica la forma de tipo salvaje de la proteína de interés se inyecta como control. La mezcla de ARNm se prepara en la solución de Deneau con el colorante vital Phenol Red. Los embriones se crían y evalúan mediante microscopía a 6 hpf (para ensayos FRET e IHC) o a 11/12 hpf (para ejes corporales). Abreviaturas: NS = Síndrome de Noonan; CDS = secuencia de codificación; FRET = Transferencia de energía de resonancia de fluorescencia; IHQ = inmunohistoquímica ; HPF = horas después de la fecundación. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Flujo de trabajo y resultados representativos de imágenes FRET multiespectrales en vivo en embriones Teen Shp2D61G tempranos antes y después del tratamiento con MEKi. (A) Descripción general de los pasos necesarios para la obtención de imágenes FRET espectrales en vivo en muestras de pez cebra. Los embriones criados y estadizados en medio E3 se montaron en un plato con fondo de vidrio en LMA al 1,5% para la adquisición de FRET espectral en vivo. Las imágenes FRET se realizan antes y después de la administración de PD0325901 de dosis bajas. Se muestra un esquema del sistema Teen reporter desarrollado por Sari et al.9. Las imágenes radiométricas (FRET/CFP) se obtienen en el postprocesamiento. (B) Detalles de la configuración del microscopio y la adquisición de imágenes multiespectrales en el software LAS X para ambos fluoróforos (CFP, D; Ypet, A). (C) Comandos y método para obtener separación de colorantes espectrales e imágenes radiométricas aplicadas a una gástrula Shp2D61GTeen . A la izquierda, se seleccionó una señal fuerte del margen del polo animal para la separación espectral con una herramienta de selección de ROI. En la parte superior derecha, se muestran los ajustes y los resultados representativos de las imágenes resultantes para los canales D y A como proyecciones z de intensidad máxima de un solo pez, así como los ajustes para obtener la imagen radiométrica y los resultados representativos. Se indica la región del margen en el polo del animal embrionario con alta señal de FRET (línea amarilla punteada). (D) Panel izquierdo: esquemas de la selección del ROI en el margen (línea amarilla punteada) de la imagen radiométrica en bruto para el análisis de datos en Fiji. Se muestran imágenes representativas de FRET/CFP generadas a través del plugin LUT "inteligente" de Fiji para visualizar los cambios en la activación de ERK en embriones que expresan Shp2D61G en comparación con Shp2WT (panel central) o en el mismo embrión Shp2D61G antes y después del tratamiento con DP (panel derecho). Para este experimento, se muestran los valores de densidad de intensidad bruta del ROI seleccionado. Se indica la barra de escala. Abreviaturas: FRET = Transferencia de energía de resonancia de fluorescencia; MEKi = Proteína quinasa inhibidor de las enzimas activadas por mitógenos; LMA = agarosa de bajo punto de fusión; CFP = proteína de fluorescencia cian ; Ypet = proteína de fluorescencia amarilla; D = donante; A = aceptante; ROI = región de interés; ERK = Quinasa regulada por señal extracelular. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Flujo de trabajo y resultados representativos de IHQ frente a t-ERK y p-ERK en embriones tempranos Shp2D61G con o sin tratamiento con MEKi. (A) Esquemas que ilustran la colección de muestras de pez cebra inmunoteñidas contra t-ERK (488) y p-ERK (633) y montadas en LMA/PBS al 1,5% para adquisiciones confocales de z-stack. (B) Configuraciones de microscopio confocal para dos líneas láser (488, 633 nm), utilizadas para capturar secuencialmente las señales fluorescentes derivadas de los anticuerpos secundarios conjugados con fluorescencia. El recuadro ilustra los parámetros de la pila Z para la adquisición de imágenes RAW multipila con una resolución aceptable. (C) Esquemas (panel izquierdo) e imágenes confocales representativas en bruto (panel derecho) de gastrulae de pez cebra correctamente teñidas a 6 hpf que muestran la localización ubicua y principalmente nuclear de t-ERK (verde) y p-ERK (rojo), respectivamente, en el margen del polo del animal. (D) Proyecciones z representativas de intensidad máxima de pilas confocales de embriones de 6 hpf que expresan Shp2WT (control), Shp2D61G , así como Shp2D61G después del tratamiento con 0,25 μM de PD0325901 entre 4 y 6 hpf. Las imágenes se muestran como un solo canal de 633 nm (aquí p-ERK, pseudocoloreado en cereza) y combinadas (p-ERK en cereza y t-ERK en verde). (E) En el pequeño recuadro se muestra la selección del ROI en la región del margen (línea amarilla punteada) de las imágenes sin procesar de la pila z para el análisis de datos en Fiji . Se incluyen los zooms de las imágenes de las pilas z que muestran la región del margen, así como las medidas de densidad de intensidad bruta recuperadas y evaluadas para inferir la activación de ERK en el ROI de hermanos que expresan Shp2WT o Shp2D61G con o sin tratamiento PD0325901 de 0,25 μM. La barra de escala se indica en los paneles de la imagen. Abreviaturas: IHQ = inmunohistoquímica; ERK = Quinasa regulada por señal extracelular; t-ERK = quinasa regulada por señal extracelular total; p-ERK = quinasa regulada por señal extracelular fosforilada; FRET = Transferencia de energía de resonancia de fluorescencia; MEKi = Proteína quinasa inhibidor de las enzimas activadas por mitógenos; LMA = agarosa de bajo punto de fusión; PBS = solución salina tampón fosfatasa; HPF = horas después de la fertilización; ROI = región de interés. Haga clic aquí para ver una versión más grande de esta figura.
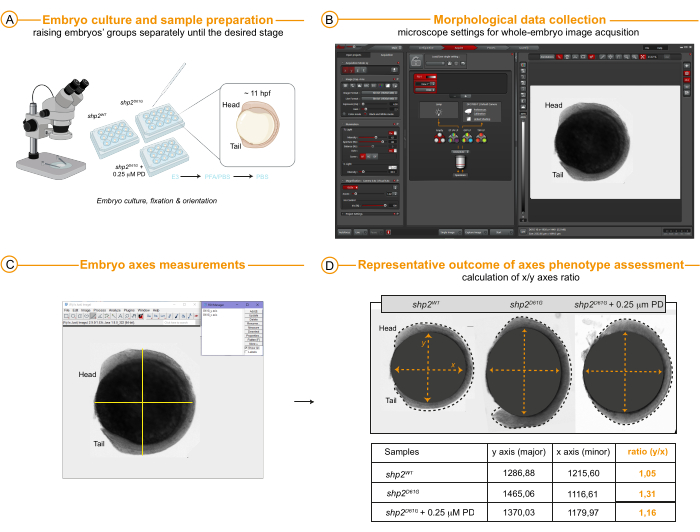
Figura 4: Análisis morfológico a 11 hpf. (A) Los embriones se criaron en medio E3 hasta la etapa de desarrollo deseada, aquí 11 hpf, luego se fijaron en PFA al 4%, se mantuvieron en 1x PBS y se dispusieron lateralmente en pocillos para la adquisición de imágenes al microscopio y la evaluación de los ejes. (B) Ajustes de adquisición de imágenes en un microscopio estereoscópico estándar y un ejemplo de la imagen resultante que muestra un embrión completo (en este caso Shp2WT) evaluado con modo de campo claro. (C,D) Resultados representativos y mediciones de ejes en Shp2WT (control), Shp2D61G y Shp2D61G después del tratamiento con 0,25 μM PD0325901 entre 4 y 6 hpf. El contorno del embrión se indica con una línea negra punteada. La barra de escala se indica en los paneles de la imagen. Abreviaturas: hpf = horas después de la fertilización; PFA = paraformaldehído; PBS = solución salina tampón fosfatasa. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
A pesar de décadas de investigación y de una miríada de mutaciones que conducen a formas muy heterogéneas de rasopatías ahora mapeadas, siguen surgiendo variantes genéticas de significado desconocido a partir de los esfuerzos de secuenciación en pacientes no diagnosticados. De hecho, en muchos casos, el diagnóstico basado únicamente en las características clínicas puede ser un desafío y los enfoques genómicos funcionales para validar los resultados de la secuenciación siguen siendo cruciales. Además, a pesar de que se han propuesto algunas moléculas anticancerosas disponibles (es decir, inhibidores de MEK) para tratar un subconjunto de rasopatías y de que están empezando a surgir algunas historias de éxito, existe un consenso limitado. Esto se debe a la escasa evidencia preclínica de la mayoría de los fármacos disponibles, también en lo que respecta a las dosis efectivas de potentes fármacos contra el cáncer y las ventanas terapéuticas para los pacientes pediátricos.
El análisis funcional en modelos animales representa un paso importante para la subclasificación de enfermedades, la estratificación de los pacientes y la evaluación inicial del fármaco. El pez cebra puede ser un modelo eficiente in vivo para validaciones funcionales de variantes potencialmente patógenas que causan enfermedades pediátricas, incluidas las rasopatías14. Existe una evaluación fenotípica de última generación del impacto de las variantes asociadas a la RASopatía para los modelos de RASopatía del pez cebra y se basan principalmente en lecturas morfológicas: la "prueba ovalada" del embrión, en la que se miden los ejes corporales al final de la gastrulación. Sin embargo, la correlación con la activación de ERK se realiza comúnmente principalmente mediante métodos estándar de posfijación: inmunoblots/IHQ.
El pipeline que establecimos tenía como objetivo evaluar la fluctuación de ERK en vivo que pueda probar rápidamente la eficacia potencial de los fármacos disponibles al comienzo de la gastrulación, anticipando el análisis de las alteraciones morfométricas resultantes. El protocolo presentado aquí comienza con la generación del modelo RASopathy transitorio deseado del pez cebra por sobreexpresión del alelo GoF de interés como ARNm en la etapa de una célula del reportero in vivo ERK (Tg[ef1a:ERK biosensor-nes] Teen)6. En este sensor FRET de tipo EKAR, la imagen FRET multiespectral es un método no invasivo y no disruptivo que se puede utilizar para detectar con precisión la fluctuación de la señal ERK espacial y temporalmente en embriones vivos que son fisiológicos durante el desarrollo, aberrantes en modelos de RASopatía y corregidos por modulación de señal farmacológica 6,7.
Se deben considerar los diferentes pasos críticos de la tubería presentada para obtener resultados informativos de las muestras de embriones. Dada la conocida baja sensibilidad de los sensores FRET15 y los posibles efectos variables de las mutaciones que afectan a la señalización de ERK (especialmente para mutaciones no validadas previamente por métodos estándar), antes de establecer el experimento, es aconsejable decidir el tipo de diseño experimental (es decir, n. de grupos y condiciones) y así hacer una estimación del número requerido de embriones (tamaño de la muestra, n) para el tamaño del efecto esperado y los valores estándar dados para los errores de tipo I y tipo II. También es importante realizar experimentos piloto para estimar el n mínimo real requerido para observar las diferencias relevantes entre WT y la proteína mutante. También es esencial la validación cruzada de los resultados de la FRET utilizando enfoques alternativos y complementarios, como el FI o los ensayos morfológicos (como se indica aquí).
En general, un tamaño de muestra estándar de n = 30 puede ser apropiado para detectar grandes efectos de valores distribuidos normalmente16. En detalle, para el análisis a priori del tamaño de la muestra, se pueden utilizar programas disponibles gratuitamente como "G-power"17 , estableciendo estos parámetros estándar: nivel de significación (α): normalmente establecido en 0,05; Nivel de confianza (1 - β): normalmente se establece en 95%; potencia de una prueba de hipótesis: normalmente se establece en 80% (1 - β, donde β es la probabilidad de cometer un error de tipo II); tamaño del efecto d (la magnitud esperada de las diferencias entre los grupos experimentales) que puede tener diferentes valores dependiendo de si es grande, mediano o pequeño, según Cohen18; variación estadística (dispersión en una distribución de frecuencias).
En el ejemplo específico proporcionado, también se validó previamente un gran efecto bien conocido del mutante Shp2D61G en pez cebra utilizando lecturas morfológicas y experimentos piloto de FRET en vivo, incluidos ensayos complementarios como FRET en muestras fijas como IF y lecturas morfológicas para validar el efecto observado (ver en Fasano et al.7).
Además, el ARNm de alta calidad es un factor de partida clave para generar modelos de enfermedades transitorias adecuados. De hecho, si bien sería ideal emplear modelos modificados genéticamente para recapitular la heterocigosis de los pacientes, la generación de estos modelos no es compatible con el requisito de pruebas rápidas de variantes emergentes, ya que requeriría aumentar la línea. Los modelos transitorios que se generan y evalúan solo durante la embriogénesis son preferibles para el cribado a gran escala.
Sin embargo, para garantizar la informatividad y la reproducibilidad de los resultados, se debe tener especial cuidado en la evaluación de la calidad del ARN. Además de los geles de formamida clásicos, el ARN también se puede comprobar utilizando instrumentos como el Bioanalyzer, que proporciona cuantificación directa y electroferogramas visuales para la inspección de calidad. Las preparaciones de ARN de alta calidad parecen un único pico afilado del tamaño deseado. Es aconsejable no proceder con la microinyección si el ARN se degrada notablemente. Es importante destacar que, cuando se trata de microinyección, para lotes de ARNm recién sintetizados, es imprescindible un experimento inicial para calibrar la dosis de inyección correcta. A esta dosis ideal, la expresión de la forma WT del ARNm de interés en hermanos del mismo lote no debería causar un fenotipo visible.
Como recomendación general, al preparar embriones para inyectar, sería mejor obtener, recolectar y evaluar embriones de parejas individuales (1 hembra + 1 macho) para asegurarse de minimizar la variabilidad del lote. Sin embargo, para aumentar el rendimiento embrionario, el desove de los peces se puede establecer a partir de cruces grupales con múltiples machos y hembras. En este caso, es probable que los embriones recolectados se deriven de más eventos de fertilización que ocurren con un ligero retraso dentro del tanque y, por lo tanto, es importante seleccionar con precisión a los individuos en la misma etapa de desarrollo (gastrulación temprana) inspeccionando los embriones varias veces en las primeras 2 horas después de la recolección. Para permitir un buen crecimiento embrionario en la delicada fase inicial, también se debe prestar especial atención al uso de todas las soluciones recién preparadas. Además, dada la posible presencia de material fecal y desechos del evento de apareamiento, la contaminación de los lotes de embriones por hongos y bacterias puede ocurrir si los embriones en medio E3 no se lavan y limpian inmediatamente después de la recolección. La viabilidad del lote de embriones debe controlarse antes, durante y después del experimento. Si la tasa de mortalidad está por encima de la tasa estándar (normalmente 20-30%), el experimento debe suspenderse.
En cuanto a los tratamientos farmacológicos, al menos para la clase de MEKi analizada aquí, es mejor evitar los tratamientos antes de 4 hpf, ya que estos podrían afectar irreversiblemente el desarrollo embrionario (toxicidad), como se describió anteriormente19. Otro paso crítico para la fiabilidad del experimento es utilizar la misma concentración de fármaco (en este caso DMSO) para todas las condiciones experimentales y evitar el hacinamiento de embriones por pocillo durante el tratamiento para mantener buenos niveles de oxigenación (se debe mantener un máximo de 20 embriones en una placa de 6 pocillos).
La optimización de los parámetros de adquisición de imágenes también es extremadamente importante para el éxito de la línea de producción, que incluye especialmente imágenes FRET multiespectrales, pero también imágenes confocales de muestras IHC. En primer lugar, dada la variabilidad en la expresión de transgenes, un aspecto decisivo es seleccionar con precisión los embriones adolescentes y descartar aquellos con muy baja expresión, que, proporcionado el bajo rango dinámico de los sensores FRET, podrían impedir una detección y cuantificación de señales suficientes (véase el ejemplo de la Figura 2). La expresión del transgén puede ser visible bajo un microscopio estereoscópico estándar con lámparas fluorescentes y ruedas de filtro apropiadas ~ 2 h después de la inyección.
En nuestra experiencia para la adquisición de datos multiespectrales de peces sensor adolescentes, la configuración del láser y los parámetros del modo de adquisición de definición de colorante espectral (x, y, λ, z) son pasos iniciales críticos de las configuraciones del microscopio y del hardware y deben optimizarse en función de la necesidad específica. En principio, los parámetros deben establecerse de manera que se alcance un buen compromiso entre la recogida de la señal y la velocidad de adquisición. Esto es particularmente importante para los registros en tiempo real en embriones casi completos, como se muestra aquí. La velocidad de adquisición de 400-600 Hz con un tamaño de paso entre 8 μm y 10 μm y el ajuste de un solo escaneo por plano z podrían no proporcionar resolución celular, pero podrían ser suficientes para capturar los cambios dinámicos en la actividad de ERK en los tejidos de los embriones. En principio, es posible obtener imágenes con una resolución casi celular con ajustes de microscopio ad hoc , pero a expensas de la velocidad y el muestreo 3D. Esto podría ser interesante si solo se investiga una determinada población celular dentro de los embriones, incluso en etapas posteriores6.
La separación espectral del colorante se puede realizar con algoritmos diferentes al aquí indicado. En cualquier caso, la selección de la región de referencia necesaria para asignar los mejores espectros de emisión tanto para CFP como para YFP es crucial. Para ello, es aconsejable comprobar la consistencia de los resultados obtenidos seleccionando y promediando diferentes ROIs, donde la señal tisular/celular es claramente visible. Al final de la adquisición espectral y una vez que se obtiene la separación espectral del colorante, también se pueden obtener imágenes radiométricas a partir de escaneos CFP e YFP sin procesar y funciones de relación disponibles gratuitamente en Fiji. Sin embargo, para visualizar y resaltar espacialmente las diferencias en los niveles de señal FRET, puede ser aconsejable reescalar las imágenes. En nuestras manos, el pseudocoloreado utilizando tablas de búsqueda (LUT) "inteligentes" codificadas dentro del software gratuito de Fiji funciona bien para mostrar mejor los niveles de activación de ERK. Se pueden elegir otras LUT y otras escalas en función de las preferencias del usuario.
Cabe destacar que el pipeline experimental presentado aquí se limita a los sensores Teen que albergan CFP como donante (D) e Ypet (similar a YFP) como aceptor (A). En general, a la hora de decidir el indicador FRET, se debe tener en cuenta el rango dinámico de los sensores (la capacidad de detectar cambios pequeños pero significativos) y la eficiencia FRET (E), influenciada por la distancia y la orientación D-A dentro del senor, así como la superposición espectral entre la emisión D y la absorción A. Teen es un sensor EKAREV que, a través de un diseño de construcción optimizado, ofrece un rango dinámico y una sensibilidad (E) mejorados en comparación con las versiones anteriores. Sin embargo, el rango dinámico relativamente bajo, normalmente peor in vivo, puede considerarse la principal limitación de este enfoque. Sin embargo, Sari et al. y Wong et al. demostraron que los cambios dinámicos de ERK que ocurren fisiológicamente durante el desarrollo son, sin embargo, visibles y medibles en embriones adolescentes y observamos fluctuaciones de señal en un modelo de pez NS tratado farmacológicamente 6,9. Sin embargo, esperamos que las fluctuaciones extremadamente bajas de la señal ERK no sean fáciles de evaluar utilizando el método que se muestra aquí. Será importante probar el rendimiento de nuestra cartera de productos con biosensores ERK mejorados que probablemente estarán disponibles en el futuro.
Para la validación cruzada de los resultados con IHQ, un paso crítico es la calidad de la fijación tisular. El tejido embrionario a 6 hpf es extremadamente delicado y delgado y, por lo tanto, sensible a una mala fijación y sobrefijación. Dada la variabilidad de la calidad y el rendimiento de un lote a otro para las soluciones fijadoras, así como para los anticuerpos, el tiempo de fijación y el éxito de todo el protocolo de IHQ deben evaluarse en un experimento piloto poco antes de evaluar las muestras experimentales. Siempre es aconsejable un fijador estéril y recién preparado.
En resumen, el protocolo experimental que se muestra aquí en el modelo conveniente de pez cebra representa una tubería robusta y relativamente rápida para evaluar el impacto de los alelos NS seleccionados en la activación de ERK en células gastrulantes de embriones tempranos. El método, que involucra imágenes FRET multiespectrales en el pez sensor Teen recientemente desarrollado para detectar fluctuaciones moleculares vivas, supera a la IHC clásica. Sin embargo, la IHQ puede utilizarse como un enfoque complementario para validar los resultados obtenidos. El enfoque anticipa las lecturas fenotípicas clásicas, a saber, la relación de ejes mayor / menor (prueba de "embrión ovalado"), un estándar de oro para los modelos de peces RASopathy, pero medible solo al final de la gastrulación. El protocolo también se puede aplicar para probar la capacidad del MEKi propuesto para corregir alteraciones de señales y ejes en embriones.
Teniendo en cuenta que solo probamos el alelo shp2D61G asociado a NS y un fármaco, el desarrollo posterior del ensayo debe incluir la evaluación del rendimiento con respecto a otras mutaciones asociadas a RASopathy, con diferentes impactos en la activación de ERK y los fármacos, dosis y ventanas de tratamiento adicionales propuestos. Será interesante evaluar la sensibilidad de este enfoque multinivel para otras variantes emergentes que afectan a moléculas en diferentes niveles de la cascada de señales RAS-MAPK. Por último, sería necesaria la futura estandarización de un método específico de alto rendimiento para la obtención de imágenes y cuantificación radiométricas FRET en sistemas de análisis de alto contenido dedicados para impulsar las pruebas de variantes.
Agradecimientos
Agradecemos al Dr. Jeroen den Hertog (Instituto Hubrecht, Utrecht, Países Bajos) por proporcionar amablemente pCS2+_eGFP-2a-Shp2a de la que se extrajo el CDS completo de shp2 para generar la plantilla de plásmido que utilizamos7. Agradecemos al Instituto de Ciencia y Tecnología de Nara (Takaaki Matsui), al Instituto Nacional de Genética (NIG/ROIS) (Koichi Kawakami), por proporcionar la línea de reporteros adolescentes transgénicos. Este trabajo fue apoyado por el Ministerio de Salud italiano - Fondos de Investigación Corriente 2021 y Fondos de Investigación Corriente 2024 y Ricerca Finalizzata Giovani Ricercatori GR-2019-12368907 a AL; Fondos de Investigación Actuales 2019, PNRRMR1-2022-12376811, 5x1000 2019, AIRC (IG-21614 e IG-28768) y LazioInnova (A0375-2020-36719) a MT.
Materiales
| Name | Company | Catalog Number | Comments |
| Plasticwares | |||
| 1.7 L Breeding Tank - Beach style Design | Tecniplast | 1.7L SLOPED | Breeding tank |
| Capillaries GC100F-10 | Harvard apparatus | 30-0019 | One-cell stage embryo microinjection |
| Cell and Tissue Culture Plates - 12 well | BIOFIL | TCP011012 | Embryo collection and treatment |
| Cell and Tissue Culture Plates - 6 well | BIOFIL | TCP011006 | Embryo collection and treatment |
| Cell Culture Dish | SPL Life Sciences | 20100 | Embryo collection |
| Nunc Glass Dishes 12mm | Thermo Fisher | 150680 | Embryo FRET spectral imaging |
| Pipette Pasteur | Corning | 357524 | Embryo transfer |
| Protein Lobind Tubes 2ml | Eppendorf | 30108450 | IHC assay |
| Reagents and others | |||
| Caviar 500-800 µm | Rettenmaier Italia | BE2269 (500-800) | Dry fish food |
| Great Salt Lake Blue Artemia Cysts | Sanders | 00004727 | Live fish food |
| Instant Ocean salt | Tecniplast | XPSIO25R | Dehydrated sea salt for live food preparation |
| Tg(EF1a:ERK Biosensor-nes) (Teen) | Contacts for ordering*: National BioResource Project Zebrafish, Support Unit for Animal Resources Development, RRD, RIKEN Center for Brain Science, Japan. https://shigen.nig.ac.jp/zebra/index_en.html *upon MTA signature. | - | Supplier of ERK Reporter zebrafish line. Fish embryos can be obtained upon MTA signature from National BioResource Project of Japan for Zebrafish (RIKEN, Japan). The zebrafish line is deposited by Nara Institute of Science and Technology (Takaaki Matsui) and the National Institute of Genetics (NIG/ROIS) (Koichi Kawakami, patent for Tol2 system) (Wong et al., 2018, Urasaki et al., 2006, Okamoto and Ishioka, 2010). |
| 6x loading dye | Cell Signaling | B7024S | Gel Elecrophoresis |
| 100 bp DNA ladder | NEB | N3231S | Gel Elecrophoresis |
| Agarose | Sigma-Aldrich | 1,01,236 | Gel Elecrophoresis |
| Agarose, low gelling temperature | Sigma-Aldrich | A9414-10G | Embryo mounting for FRET spectral imaging and IHC assay |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A8022 | IHC assay |
| Calcium chloride | Sigma-Aldrich | 223506 | E3 medium component |
| Calcium nitrate | Sigma-Aldrich | 237124 | Danieau stock solution component |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418-100ML | IHC assay |
| EDTA | Sigma-Aldrich | E9884 | TBE buffer component for gel preparation |
| Ethanol 99%+ | Fisher Scientific | 10048291 | In vitro RNA purification |
| Formaldeide 16% | Thermo Fisher | 28908 | Embryo fixation |
| Formamide | Sigma-Aldrich | F9037 | Gel Elecrophoresis |
| Gel Loading Buffer II (Denaturing PAGE) | Thermo Fisher | AM8546G | In vitro RNA transcription |
| Glacial Acetic Acid | Sigma-Aldrich | 695092 | TBE buffer component for gel preparation |
| Glycerol | Sigma-Aldrich | G6279-1L | IHC assay |
| Goat anti-mouse Alexa Fluor 488 | Thermo Fisher | A11001 | IHC assay |
| Goat anti-rabbit Alexa Fluor 633 | Thermo Fisher | A21070 | IHC assay |
| HEPES | Sigma-Aldrich | H3375 | Danieau stock solution component |
| KpnI - HF (Enzyme + rCutSmart Buffer) | NEB | R3142 | Plasmid linearization |
| Magnesium sulfate | Sigma-Aldrich | 230391 | E3 medium component/Danieau stock solution component |
| Millennium RNA Markers | Thermo Fisher | AM7150 | Gel Elecrophoresis |
| Monarch Genomic DNA purification Kit | NEB | T3010L | Plasmid linearization |
| Mouse monoclonal p44/42 MAPK | Cell Signaling | 4696S | IHC assay |
| mMACHINE SP6 Transcription Kit | Thermo Fisher | AM1340 | In vitro RNA transcription |
| Normal Goat serum (NGS) | Sigma-Aldrich | G9023 | IHC assay |
| Nuclease-free water Ambion | Thermo Fisher | AM9937 | In vitro RNA transcription |
| PD0325901 | Sigma-Aldrich | PZ0162 | MEK inhibitor |
| Phenol Red solution | Sigma-Aldrich | P0290 | Microinjection mix component |
| Poly A Tailing Kit | Thermo Fisher | AM1350 | In vitro RNA transcription |
| Potassium chloride bioxtra | Sigma-Aldrich | P9333 | E3 medium component/Danieau stock solution component/PBS stock solution component |
| Potassium dihydrogen phosphate | Sigma-Aldrich | P0662 | PBS stock solution component |
| Proteinase K | Sigma-Aldrich | P2308 | IHC assay |
| Rabbit polyclonal phospho-p44/42 MAPK | Cell Signaling | 4695S | IHC assay |
| SYBR safe DNA gel staining | Thermo Fisher | S33102 | Gel Elecrophoresis |
| Sodium Chloride | Sigma-Aldrich | 31434-M | E3 medium component/Danieau stock solution component/PBS stock solution component |
| Sodium phosphate dibasic | Sigma-Aldrich | 71643 | PBS stock solution component |
| Trizma base | Sigma-Aldrich | T1503 | TBE buffer component for gel preparation |
| Triton X-100 | Sigma-Aldrich | T8787 | PBSTr buffer component |
| Equipment | |||
| Alliance Mini HD9 | Uvitec | - | Imaging system |
| Centrifuge 5430 R | Eppendorf | 5428000205 | Microcentrifuge |
| Eppendorf ThermoMixer C | Eppendorf | - | Embryo mounting |
| FemtoJet 4x | Eppendorf | - | Microinjection system |
| Infinite M Plex | Tecan | - | Multimode plate reader |
| Leica M205FA | Leica Microsystems | - | Fluorescence stereo microscope |
| Leica TCS-SP8X equipped with incubator (OkoLab) | Leica Microsystems | - | Confocal microscope |
| Mini-sub Cell GT Horiziontal Electrophoresis System | Bio-Rad | 1704406 | Gel Elecrophoresis |
| PC-100 Vertical puller | Narishige | - | Needle puller |
| PowerPac Universal Power Supply | Bio-Rad | 1645070 | Gel Elecrophoresis |
| Stellaris 5 | Leica Microsystems | - | Confocal microscope |
| Vortex MiniStar silverline | VWR | - | Plasmid preparation |
| Softwares | |||
| Biorender | Biorender | CC-BY 4.0 license | Cartoon elaboration for Figures |
| Excel | Microsoft Office Professional Plus 2019 | - | Data analyses |
| Fiji software | ImageJ | 15.3t | Imaging rendering and quantitative analyses (FRET signals measurements, ERK fluorescence intensity in IHC assay, embryo axes lenght) |
| GraphPad Prism | GraphPad Software LLC | v. 9 | Statistical data analyses |
| iControl spectrophotometer software | Tecan | v. 2.0 | RNA quantification |
| Illustrator | Adobe | 26.0.3 (64-bit) | Figure assembling |
| LASX software | Leica Microsystems | v. 4.5 (Stellaris 5), v. 3.0 (M205FA), v. 3.5 (TCS-SP8X) | Imaging acquisition for spectral FRET experiments and embryo imaging for axes lenght measurements |
| Q9 Mini 18.02-SN software | Uvitec | - | Gel image acquisition |
Referencias
- Iroegbu, J. D., Ijomone, O. K., Femi-Akinlosotu, O. M., Ijomone, O. M. ERK/MAPK signalling in the developing brain: Perturbations and consequences. Neurosci Biobehav Rev. 131, 792-805 (2021).
- Tartaglia, M., Aoki, Y., Gelb, B. D. The molecular genetics of RASopathies: An update on novel disease genes and new disorders. Am J Med Genet C Semin Med Genet. 190 (4), 425-439 (2022).
- Tate, J. G., et al. COSMIC: the catalogue of somatic mutations in cancer. Nucleic Acids Res. 47 (D1), D941-D947 (2019).
- Tartaglia, M., et al. Mutations in PTPN11, encoding the protein tyrosine phosphatase SHP-2, cause Noonan syndrome. Nat Genet. 29 (4), 465-468 (2001).
- Jopling, C., van Geemen, D., den Hertog, J. Shp2 knockdown and Noonan/LEOPARD mutant Shp2-induced gastrulation defects. PLoS Genet. 3 (12), e225 (2007).
- Wong, K. -. L., Akiyama, R., Bessho, Y., Matsui, T. ERK activity dynamics during zebrafish embryonic development. Int J Mol Sci. 20 (1), 109 (2019).
- Fasano, G., et al. Assessment of the FRET-based Teen sensor to monitor ERK activation changes preceding morphological defects in a RASopathy zebrafish model and phenotypic rescue by MEK inhibitor. Mol Med. 30 (1), 47 (2024).
- Zimmermann, T., Rietdorf, J., Girod, A., Georget, V., Pepperkok, R. Spectral imaging and linear un-mixing enables improved FRET efficiency with a novel GFP2-YFP FRET pair. FEBS Lett. 531 (2), 245-249 (2002).
- Sari, D. W. K., et al. Time-lapse observation of stepwise regression of Erk activity in zebrafish presomitic mesoderm. Sci Rep. 8 (1), 4335 (2018).
- Wang, H., Li, J., Xue, T., Cheng, N., Du, H. Construction of a series of pCS2+ backbone-based Gateway vectors for overexpressing various tagged proteins in vertebrates. Acta Biochim Biophys Sin (Shanghai). 48 (12), 1128-1134 (2016).
- Abdelrahman, D., Hasan, W., Da'as, S. Microinjection quality control in zebrafish model for genetic manipulations. MethodsX. 8, 101418 (2021).
- Yuan, S., Sun, Z. Microinjection of mRNA and morpholino antisense oligonucleotides in zebrafish embryos. J Vis Exp. (27), e1113 (2009).
- Rossant, J., Nutter, L. M. J., Gertsenstein, M. Engineering the embryo. Proc Natl Acad Sci USA. 108 (19), 7659-7660 (2011).
- Jindal, G. A., et al. In vivo severity ranking of Ras pathway mutations associated with developmental disorders. Proc Natl Acad Sci USA. 114 (3), 510-515 (2017).
- Leavesley, S. J., Rich, T. C. Overcoming lmitations of FRET measurements. Cytometry A. 89 (4), 325-327 (2016).
- Mascha, E., Vetter, T. Significance, errors, power, and sample size: Theblocking and tackling of statistics. Anesth Analg. 126 (2), 691-698 (2018).
- Faul, F., Erdfelder, E., Lang, A. -. G., Buchner, A. G*Power 3: a flexible statistical power analysis program for the social, behavioral, and biomedical sciences. Behav Res Methods. 39 (2), 175-191 (2007).
- Cohen, J. . Statistical power analysis for the behavioral sciences. , (1988).
- Anastasaki, C., Rauen, K. A., Patton, E. E. Continual low-level MEK inhibition ameliorates cardio-facio-cutaneous phenotypes in zebrafish. Dis Model Mech. 5 (4), 546-552 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados