Method Article
Pipeline per lo studio anatomico tridimensionale multiscala del cuore umano
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo presenta una pipeline completa per analizzare campioni ottenuti da cuori umani che abbracciano le scale microscopiche e macroscopiche.
Abstract
Lo studio dettagliato dei cuori umani non falliti rigettati per il trapianto offre un'opportunità unica per eseguire analisi strutturali su scala microscopica e macroscopica. Queste tecniche includono la pulizia dei tessuti (imaging tridimensionale (3D) di organi puliti con solvente abilitati per l'immunomarcatura modificata) e la colorazione immunoistochimica. Le procedure di esame mesoscopico comprendono la dissezione stereoscopica e la tomografia microcomputerizzata (TC). Le procedure di esame macroscopico includono la dissezione macroscopica, la fotografia (compresi gli anaglifi e la fotogrammetria), la TC e la stampa 3D del cuore sezionato fisicamente o virtualmente o dell'intero cuore. Prima dell'esame macroscopico, può essere eseguita la fissazione pressione-perfusione per mantenere l'architettura 3D e la morfologia fisiologicamente rilevante del cuore. L'applicazione di queste tecniche in combinazione per studiare il cuore umano è unica e cruciale per comprendere la relazione tra caratteristiche anatomiche distinte come la vascolarizzazione coronarica e l'innervazione miocardica nel contesto dell'architettura 3D del cuore. Questo protocollo descrive le metodologie in dettaglio e include risultati rappresentativi per illustrare i progressi nella ricerca sull'anatomia cardiaca umana.
Introduzione
Poiché la funzione segue la forma, la comprensione dell'architettura del cuore è fondamentale per apprezzarne la fisiologia. Sebbene numerose indagini abbiano rivelato l'anatomia cardiaca dalla micro alla macroscala 1,2,3, molte questioni rimangono irrisolte, in particolare quelle relative all'anatomia cardiaca umana. Ciò è in parte dovuto al fatto che gli studi di base incentrati sull'anatomia funzionale hanno generalmente utilizzato cuori di animali 4,5,6, che sono spesso distinti dai cuori umani 1,7,8. Inoltre, ogni singolo studio, anche quelli che utilizzano campioni di cuore umano, tende a concentrarsi su strutture molto specifiche, il che rende difficile applicare i risultati nel contesto dell'intero cuore. Questo è ancora più vero se le strutture focalizzate si trovano su micro o mesoscale, come il perinexus9 e i plessi gangliari10.
In questo contesto, lo studio strutturale sistemico del cuore umano rigettato per il trapianto offre un'opportunità unica e rara per ottenere un atlante completo delle strutture cardiache a fuoco su scale microscopiche e macroscopiche11. I protocolli di esame microscopico includono la pulizia dei tessuti (imaging tridimensionale (3D) di organi puliti con solvente abilitati per l'immunomarcatura modificata, iDISCO+)12,13 e la colorazione immunoistochimica. I protocolli di esame mesoscopico includono la dissezione stereoscopica, la macrofotografia e la scansione tomografica microcomputerizzata (TC). I protocolli di esame macroscopico includono la dissezione macroscopica14, la fotografia (inclusi anaglifi e fotogrammetria)15,16,17, la TC, la dissezione virtuale 18 e la stampa 3D del cuore sezionato fisicamente o virtualmente o dell'intero cuore17. In preparazione all'esame macroscopico, viene eseguita la fissazione pressione-perfusione per mantenere l'architettura 3D e la morfologia fisiologicamente rilevante del cuore 14,19,20,21. L'applicazione combinata di queste tecniche è unica e cruciale per correlare caratteristiche anatomiche distinte nel contesto dell'architettura 3D del cuore umano.
Poiché l'opportunità di ottenere un campione di cuore umano non patologico è estremamente limitata, un approccio multiscala qui descritto massimizza l'uso del campione. Applicando le varie procedure descritte di seguito, i risultati rappresentativi illustreranno al lettore come i risultati possano essere utilizzati per molteplici scopi, tra cui la scoperta nella ricerca scientifica11 (analisi complete dell'innervazione cardiaca, distribuzione dei plessi ganglionati), il miglioramento delle procedure cliniche (simulazione per approcci chirurgici e interventistici) e l'educazione anatomica (vera dimostrazione 3D dell'anatomia cardiaca).
Protocollo
Questo studio ha utilizzato campioni di tessuto anonimizzati raccolti da cuori umani di donatori non falliti ed è stato approvato dall'Institutional Review Board dell'Università della California, Los Angeles (UCLA). I campioni sono stati ottenuti da cuori non falliti che sono stati rifiutati per il trapianto. I cuori sono stati perfusi a pressione, fissati in paraformaldeide al 4% (PFA) e sottoposti a imaging prima dell'elaborazione dei tessuti con i seguenti metodi. La Figura 1 riassume il diagramma di flusso dell'ordine dello studio. I dettagli dei reagenti e delle attrezzature utilizzate nello studio sono elencati nella tabella dei materiali.
1. Esame su microscala
- Pulizia dei tessuti utilizzando il protocollo iDISCO+ (Modified Immunolabeling-enabled 3D imaging of solvent-cleared organis).
- Sezionare il tessuto fissato al 4% con PFA con un bisturi per adattarlo alla camera da 3 mm × 16 mm × 25 mm per la microscopia confocale. Per visualizzare i tessuti più spessi, è possibile impilare camere e/o distanziatori aggiuntivi sul vetrino.
- Disidratare i campioni utilizzando la serie di metanolo graduato (MeOH) (20%, 40%, 60%, 80% e 100% MeOH in H2O deionizzato [vol/vol]) per 1 ora ciascuno a temperatura ambiente (RT) con agitazione.
- Lavare con 100% MeOH per 1 ora a RT e immergere in 66% diclorometano/33% MeOH a RT agitando per una notte.
- Il giorno successivo, lavare due volte in MeOH (100%) per 1 ora a RT, raffreddare a 4 °C e trattare con 5% di H2O2 in MeOH (vol/vol) per una notte a 4 °C.
- Reidratare con la serie di MeOH graduata (80%, 60%, 40% e 20% MeOH) e lavare in 0,01 mol/L PBS per 1 ora ciascuno a RT con agitazione.
- Lavare i fazzoletti due volte a 0,01 mol/L PBS con Triton X-100 allo 0,2% per 1 ora a RT.
- Prepararsi per l'immunomarcatura mediante permeabilizzazione in 0,01 mol/L di PBS, 20% dimetil solfossido (DMSO), 0,2% Triton X-100 e 0,3 mol/L di glicina per 2 giorni a 37 °C con agitazione.
- Bloccare in 0,01 mol/L di PBS con il 10% di DMSO, lo 0,2% di Triton X-100 e il 5% di siero d'asino normale per altri 2 giorni a 37 °C con agitazione.
- Etichettare con un anticorpo primario compatibile con MeOH coniugato a fluorofori diluito in PBS 0,01 mol/L con 10 mg/mL di eparina (PTwH), 0,2% Tween-20, 5% DMSO e 3% siero d'asino normale per 3-4 giorni a 37 °C con agitazione.
- Reintegrare la soluzione anticorpale e incubare per altri 3-4 giorni a 37 °C con agitazione.
- Dopo 1 settimana di incubazione in soluzione di anticorpi primari, lavare da 4 a 5 volte in PTwH durante la notte a RT.
- Incubare con anticorpo secondario coniugato a fluorofori diluiti in PTwH, siero d'asino normale al 3% per 3 giorni a 37 °C con agitazione.
- Reintegrare la soluzione di anticorpi secondari, incubare per altri 3 giorni a 37 °C con agitazione.
- Dopo 6 giorni di incubazione in soluzione di anticorpi secondari, lavare in PTwH 4-5 volte durante la notte a RT.
- Disidratare con una serie di MeOH graduata (20%, 40%, 60%, 80%, 100% e 100% MeOH). Il campione può essere conservato per una notte a RT.
- Incubare in 66% diclorometano/33% MeOH per 3 ore a RT con agitazione.
- Lavare due volte in diclorometano al 100% per 15 minuti a RT con agitazione.
- Incubare e conservare i campioni in etere benzilico. Riempire la provetta per ridurre al minimo l'ossidazione del campione da parte dell'aria.
- Imaging di campioni puliti dal tessuto
- Fissare una camera contenente adesivo su un vetrino e applicare lo smalto per unghie attorno al perimetro della camera. Per tessuti più spessi, è possibile impilare camere e/o distanziatori aggiuntivi sul vetrino.
- Posizionare il tessuto pulito nella camera, riempirlo con etere benzilico e applicare un vetrino coprioggetti.
- Applicare lo smalto intorno al vetrino coprioggetti per creare un sigillo.
- Ottenete immagini tilecan e Z stack utilizzando un microscopio confocale a scansione laser verticale con una lente 5x o 10x per ottenere immagini a una profondità fino alla distanza di lavoro della lente.
- Immagine con una risoluzione di 1024 x 1024 utilizzando linee laser appropriate per gli spettri di emissione dei fluorofori utilizzati. L'autofluorescenza muscolare è visibile utilizzando la linea laser a 488 nm.
- Assicurarsi che la dimensione del passo dell'asse z sia commisurata al campionamento di Nyquist in base all'apertura numerica dell'obiettivo11 specificato.
- Unisci le immagini e usa il software per la visualizzazione 3D.
- Crea figure utilizzando immagini MIP (Maximum Intensity Projection) di stack Z per canali singoli e uniti (Figura 2).
- Immunoistochimica
NOTA: Dopo che il tessuto è stato incorporato in paraffina22, la seguente procedura viene utilizzata per creare vetrini per lo studio immunoistochimico.- Preparazione della soluzione di corrispondenza dell'indice di rifrazione (RIMS).
- Preparare 0,1 mol/L di tampone fosfato aggiungendo 10,9 g di Na2HPO4 (anidro) e 3,1 g di NaH2PO4 (monoidrato) all'H2O deionizzato per un volume totale di 1 L (pH 7,4). Sterilizzare con filtro la soluzione e conservarla in RT.
- Diluire il tampone fosfato a 0,02 mol/L.
- Sciogliere Histodenz in 30 mL di tampone fosfato 0,02 mol/L agitando la soluzione per 10 minuti con un'ancoretta magnetica nel flacone di conservazione finale che può essere sigillato per ridurre al minimo l'evaporazione e la contaminazione.
- Aggiungere sodio azide a una concentrazione totale dello 0,01% (p/v) e regolare il pH a 7,5 con NaOH.
- Regola l'indice ricursale variando la concentrazione finale di Histodenz.
- Conserva i RIMS presso RT per mesi. Scartare se si nota una contaminazione microbica.
NOTA: NON sterilizzare in autoclave soluzioni contenenti sodio azide.
- Preparazione di vetrini per lo studio immunoistochimico
- Crea sezioni di 5 μm di spessore con il microtomo. Applicare la sezione di tessuto ai vetrini carichi.
- Rimuovere la paraffina incubando i vetrini in xilene al >75% per 10 minuti. Spostare i vetrini in un secondo contenitore con xilene per altri 10 minuti.
- Rimuovere lo xilene immergendo i vetrini in EtOH al 100% per 10 minuti, poi in EtOH al 95% per 5 minuti e EtOH al 70% per 5 minuti.
- Sciacquare i vetrini con H2O deionizzato per 5 min.
- Immergere i vetrini nel tampone di recupero dell'antigene per 25 minuti a 90-95°C.
- Lasciare raffreddare il contenitore a RT per 1 ora agitando.
- Immergere i vetrini nel tampone di ammollo (0,01 mol/L PBS + 0,4% Triton X-100) per 30 minuti a 4°C.
- Circondare il fazzoletto con una penna PAP. Aggiungere PBS a ciascun vetrino e posizionarlo in una camera umidificata per evitare l'essiccazione.
- Lavare i vetrini con PBS a RT agitando per 5 min.
- Blocco con tampone bloccante (0,01 mol/L PBS + 10% siero d'asino + 0,1% TX-100) per 1 ora con agitazione.
- Incubare con un anticorpo primario diluito in tampone bloccante per una notte a 4°C.
- Il giorno successivo, lasciare che i vetrini si ritirino a RT per 15 minuti.
- Lavare i vetrini 3 volte con 0,01 mol/L PBS + 0,2% TritonX-100 per 5 minuti.
- Incubare con un anticorpo secondario diluito in tampone bloccante per 1 ora a RT con agitazione.
- Lavare i vetrini 3 volte con 0,01 mol/L PBS + 0,2% TritonX-100 per 5 minuti.
- Lavare i vetrini 3 volte con 0,01 mol/L di PBS per 5 min.
- Posiziona 1 goccia di RIMS con un contagocce e applica un vetrino coprioggetti.
- Applicare lo smalto per unghie intorno al vetrino coprioggetti per creare il sigillo.
- Come controllo negativo, eseguire un campione senza l'anticorpo primario per dimostrare l'assenza di colorazione specifica.
- Vetrini immunocolorati per imaging
- Visualizzare i vetrini con un microscopio confocale a scansione laser con obiettivi 10x, 20x e 40x.
- Immagine con una risoluzione di 1024 x 1024 utilizzando linee laser appropriate per gli spettri di emissione degli anticorpi secondari utilizzati.
- Crea figure utilizzando immagini MIP (Maximum Intensity Projection) di stack Z per canali singoli e uniti (Figura 3).
- Preparazione della soluzione di corrispondenza dell'indice di rifrazione (RIMS).
2. Esame in mesoscala
- Dissezione stereoscopica
- Esegui dissezioni delicate concentrandosi su strutture minuscole o sottili, come il nodo atrioventricolare, l'arteria del nodo atrioventricolare e il plesso nervoso cardiaco (scala da submillimetrica a millimetri) con una lampada da tavolo ingranditrice con morsetto, telescopi chirurgici o stereomicroscopio.
- Scansione micro-CT
NOTA: L'imaging TC viene eseguito dopo la perfusione e la fissazione a pressione e in qualsiasi fase della dissezione utilizzando una tomografia a emissione di micropositroni (PET)/scanner CT (Figura 4).- Riscaldare la sorgente di raggi X CT per 25 minuti prima dell'imaging del campione.
- Posizionare il campione di cuore sul piano dello scanner.
- Spostare il piano dello scanner in una posizione orizzontale di 544 mm e in una posizione verticale di 14 mm per centrare il cuore nel campo visivo (FOV) della TC.
- Acquisizione di immagini TC a 80 kVp, 150 μA, con 720 proiezioni durante un tempo di scansione di 1 minuto a una risoluzione spaziale di 200 μm.
- Ricostruisci i dati CT con un campo visivo di 12 cm x 12 cm x 10 cm e una matrice di 600 x 600 x 500 voxel e salvali come file DICOM.
3. Esame su macroscala
- Perfusione e fissazione a pressione
NOTA: Gli autori modificano le tecniche di perfusione e fissazione pressoria precedentemente descritte e le applicano ai cuori umani non falliti respinti per il trapianto 14,19,20,21.- Utilizzare pompe ad alto flusso per la fissazione della perfusione. Utilizzare il 100% di etanolo14, il 4% di PFA o il 10% di formalina per il fissativo.
NOTA: Il cuore viene recuperato con l'aorta ascendente, il tronco polmonare e entrambe le vene cave e polmonari resecate il più distalmente possibile e consegnate nella soluzione23 dell'Università del Wisconsin. - Utilizzare due cannule chirurgiche da 20-24 Fr per la perfusione del cuore destro e sinistro. Per la perfusione del cuore destro, incannulare la vena cava superiore e posizionare uno sfiato sul tronco polmonare o sull'arteria polmonare con siringhe di plastica da 12-30 ml tagliate a metà con punte Luer-Lock attaccate a rubinetti a tre vie.
- Occludere la vena cava inferiore e l'altra arteria polmonare con lo spago dopo aver inserito una siringa di plastica semitagliata bloccata di dimensioni adeguate o una provetta da centrifuga da 1,5-5,0 ml.
- Per la perfusione anterograda del cuore sinistro, incannulare una delle vene polmonari e posizionare uno sfiato all'estremità tagliata distale dell'aorta con le siringhe di plastica da 12-30 ml tagliate a metà con punte Luer-Lock attaccate a rubinetti a tre vie.
- Per la perfusione retrograda del cuore sinistro, incannulare uno dei rami dell'arco aortico e posizionare uno sfiato in un altro ramo dell'arco aortico. Posizionare le punte delle cannule in entrambi i ventricoli.
- Occludere gli altri orifizi dei vasi con lo spago dopo aver inserito una siringa di plastica semitagliata bloccata di dimensioni adeguate o una provetta per microcentrifuga da 1,5-5,0 mL. Utilizzare una garza sottile per coprire la parte di inserimento delle siringhe/tubi/cannule per evitare perdite e scivolamenti. Ripara le perdite di grandi dimensioni usando sutura, fasciatura o agglomerazione. Sono consentite piccole perdite.
- Sospendi il cuore in un contenitore di plastica.
- Collegare un tubo di plastica morbida da 22-24 Fr a ciascuna cannula e inserire l'altra estremità del tubo nel contenitore riempito di fissativo.
- Far circolare il fissativo attraverso i circuiti del cuore destro e sinistro utilizzando una pompa ad alto flusso impostata a circa 100-300 mL/min per il cuore destro e 200-400 mL/min per il cuore sinistro per raggiungere rispettivamente circa 20 mmHg nel ventricolo destro e 80 mmHg nel ventricolo sinistro.
- Mantenere la perfusione a 4 °C per 24 ore.
- Lavare il cuore con 0,01 mol/L di PBS per 30 minuti agitando quattro volte.
- Conservare il cuore in 0,01 mol/L di PBS/0,02% di sodio azide a 4 °C.
NOTA: La fissazione pressione-perfusione è efficace solo per un cuore fresco, non per un cuore recuperato da un cadavere imbalsamato.
- Utilizzare pompe ad alto flusso per la fissazione della perfusione. Utilizzare il 100% di etanolo14, il 4% di PFA o il 10% di formalina per il fissativo.
- Dissezione grossolana
- Eseguire la dissezione progressiva con registrazioni fotografiche in ogni fase della dissezione.
- Per mantenere la rilevanza clinica, prestare particolare attenzione a evitare di distorcere/deformare qualsiasi struttura per mantenere la morfologia fisiologica del cuore.
- Immagine delle strutture target utilizzando l'orientamento clinicamente rilevante, come l'orientamento obliquo anteriore destro.
- Fotografia
- Posizionare il cuore fisso e a pressione su un treppiede con una piattaforma montata con più rebbi e la possibilità di ruotare di 360o.
- Fotografare il cuore utilizzando una fotocamera reflex digitale a obiettivo singolo (Figura 5)24 utilizzando più pannelli luminosi a diodi emettitori di luce posizionati sui cavalletti C e un ampio panno di sfondo nero per piumino.
- Fotografare utilizzando l'obiettivo con una lunghezza focale lunga (200 mm) per una distanza di lavoro di 4-6 piedi per ridurre al minimo la distorsione del soggetto14.
- Anaglifi
- Per visualizzare immagini anaglifiche, ricostruire una coppia di fotografie o immagini renderizzate in volume da set di dati CT con una differenza di 10° nell'angolo di rotazione sul piano orizzontale.
- Converti una serie di queste immagini bidimensionali (2D), denominate stereogrammi, in anaglifi utilizzando il freeware16.
- Per visualizzare un anaglifo, utilizzare occhiali rossi/ciano.
- Fotogrammetria
NOTA: La fotogrammetria è la scienza applicata alla generazione di una ricostruzione tridimensionale della superficie da più fotografie bidimensionali scattate a varie angolazioni17.- Sospendi il campione su C-Stand o posizionalo sulla tavola rotante per ottenere centinaia di fotografie multidirezionali con uno smartphone.
- Genera il modello 3D in formato FBX utilizzando software disponibili in commercio.
- Scansione TC
NOTA: La TAC può essere eseguita dopo la perfusione a pressione e la fissazione e in qualsiasi fase della dissezione.- Sospendi il campione di cuore da una barra posta sulla parte superiore del contenitore. Per evitare che il cuore oscilli durante la scansione, sostenere la base del cuore con rebbi di plastica fissati sul fondo del contenitore. Pertanto, l'aria fungerà da contrasto negativo.
- Eseguire la scansione TC utilizzando uno scanner CT multi-rivelatore disponibile in commercio con i seguenti parametri: tensione del tubo di 120 kV, corrente del tubo di 800-900 mA e rotazione del gantry di 280 ms. La lunghezza della dose del prodotto è generalmente di 500-1200 mGy.cm.
- Ricostruire i dati dell'immagine assiale utilizzando i seguenti parametri: uno spessore di sezione, 0,6 mm; un intervallo incrementale, 0,3 mm; un campo visivo, il più piccolo possibile (generalmente 100-200 mm); e una matrice, 512 × 512.
- Dissezione virtuale
- Analizza le immagini della scansione TC utilizzando il software disponibile in commercio per generare immagini di dissezione virtuale.
NOTA: La dissezione virtuale è una modifica del processo di rendering del volume in cui l'attenzione viene spostata sulle pareti delle camere cardiache e dei vasi18. In questo processo, la soglia manuale rimuove virtualmente la camera migliorata dai set di dati originali. - Visualizza le pareti, i setti e le valvole non migliorate con la dissezione virtuale per produrre immagini simili alla dissezione grossolana. A differenza della dissezione grossolana dei campioni di cuore, i piani di taglio durante la dissezione virtuale sono praticamente illimitati. Quasi tutte le viste possono essere ricreate per visualizzare le strutture di interesse in base alle esigenze.
- Analizza le immagini della scansione TC utilizzando il software disponibile in commercio per generare immagini di dissezione virtuale.
- Stampa 3D
- Apri il file compatibile del campione di cuore nel software della stampante 3D.
- Utilizzare il profilo di 0,10 mm Fast DETAIL per le impostazioni di stampa nella stampante 3D e ridurre la velocità di stampa a 20 mm/s. Abilitare Genera materiale di supporto.
- Utilizzare il profilo del filamento TPU per "Impostazioni filamento" nella stampante 3D.
- Utilizzare il profilo dell'ugello Original Prusa MK4 Input Shaper 0.4 per "Impostazioni stampante" nella stampante.
- Al termine dello slicing, salvare il file BGCODE in un'unità flash USB per la stampa 3D.
- Usa il filamento TPU da 1,75 mm per stampare in 3D il campione di cuore umano. Prima della stampa 3D, asciugare il filamento TPU per 6 ore utilizzando un essiccatore di filamenti.
- Per ridurre la tensione del filamento durante la stampa 3D, posiziona la bobina di filamento su un supporto per bobina con un cuscinetto incorporato per facilitare la rotazione della bobina di filamento. Esegui la stampa 3D utilizzando una stampante 3D disponibile in commercio con una lamiera di acciaio verniciata a polvere testurizzata.
- Al termine della stampa 3D, rimuovere con cautela i materiali di supporto.
Risultati
Esami su microscala
L'applicazione della pulizia dei tessuti consente l'imaging di volumi maggiori di tessuto in 3D utilizzando la microscopia confocale. Nel cuore è possibile visualizzare i gangli contenenti i neuroni cardiaci e il pattern neurale dell'innervazione miocardica (Figura 2). La Figura 3 mostra un'immagine confocale del miocardio del ventricolo sinistro umano immunocolorato per i nervi e le cellule muscolari lisce. Si nota che i vasi sanguigni attraversano il miocardio e vengono identificate numerose fibre nervose, sia in associazione che indipendentemente dai vasi sanguigni.
Esami su meso e macroscala
Quando si utilizza l'etanolo assoluto per la perfusione e la fissazione a pressione di 24 ore, il colore naturale del campione viene sbiancato, il tessuto viene disidratato25 e l'elasticità è significativamente ridotta. D'altra parte, dopo la fissazione con PFA e formalina, il colore naturale e l'elasticità vengono notevolmente mantenuti. Per questi motivi, il PFA o la formalina sono principalmente utilizzati come fissativo preferito.
La Figura 6 mostra immagini rappresentative della dissezione grossolana, della dissezione virtuale, del modello poligonale STL e della stampa tridimensionale. La Figura 7 mostra immagini rappresentative degli anaglifi creati da immagini di dissezione sia grossolane che virtuali. La percezione della profondità può essere ottenuta con vetri anaglifici. Il singolo modello fotogrammetrico acquisito può essere osservato da quasi tutte le direzioni utilizzando il software disponibile in commercio e dimostra caratteristiche anatomiche complesse rilevanti per le procedure cardiache transcatetere di routine. Applicando queste tecniche al cuore preparate con la perfusione e la fissazione della pressione, le informazioni tridimensionali sul cuore possono essere conservate quasi eternamente sia digitalmente che fisicamente e condivise senza confini. La Figura 8 mostra le stampe 3D in scala del 50% replicate dai cuori sezionati rifiutati per il trapianto.

Figura 1: Diagramma di flusso del protocollo. Fare clic qui per visualizzare una versione più grande di questa figura.
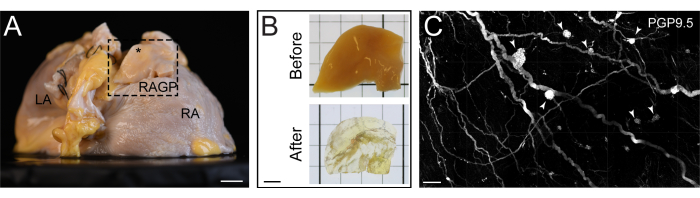
Figura 2: Sezione dell'atrio destro umano ripulita con tessuto pulito. (A) Vista obliqua posteriore destra di un atrio destro con il plesso ganglionato atriale destro (RAGP) sezionato per la pulizia dei tessuti. (B) Campione RAGP prima e dopo la pulizia dei tessuti. (C) Proiezione della massima intensità di una porzione di RAGP umano ripulita da iDISCO+ che dimostra gangli (punte di freccia) immunocolorati con prodotto genico 9.5 (PGP9.5) del marcatore panneuronale. Le barre della scala sono di 1 cm (A), 5 mm (B) e 500 μm (C). Questa figura è adattata da Hanna et al.11. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Vetrini immunocolorati del ventricolo sinistro umano. Immagine confocale della fetta di miocardio del ventricolo sinistro umano immunocolorata con il prodotto genico 9.5 (PGP9.5) del marcatore delle cellule muscolari lisce e con il marcatore delle cellule muscolari lisce α actina muscolare liscia (αSMA). L'autofluorescenza muscolare è visibile utilizzando la linea laser a 488 nm (verde). La barra della scala è di 50 μm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Imaging tomografico microcomputerizzato di campioni cardiaci. (A) Configurazione della tomografia microcomputerizzata per l'imaging di campioni cardiaci. (B) Interfaccia utente per l'imaging con tomografia microcomputerizzata. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Impostazione dello studio fotografico presso l'UCLA Cardiac Arrhythmia Center. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: Dissezione grossolana (in alto a sinistra), dissezione virtuale (in alto a destra), modello poligonale STL (in basso a sinistra) e stampa tridimensionale (in basso a destra) immagini del complesso valvolare aortico e mitralico. Clicca qui per visualizzare una versione più grande di questa figura.
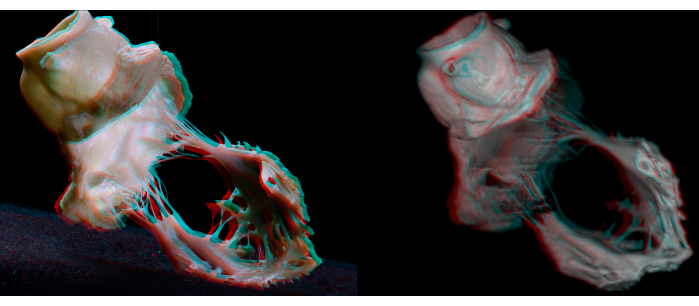
Figura 7: Anaglifi di una dissezione grossolana (a sinistra) e di una dissezione virtuale (a destra) del complesso valvolare aortico e mitralico. I vetri anaglifici (rosso/ciano) sono necessari per ottenere la percezione della profondità. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 8: Stampa tridimensionale di campioni di cuore. (A) Configurazione della stampante tridimensionale (3D) per la stampa 3D di campioni di cuore con un filamento TPU. (B) Stampe 3D a cuore rappresentative prodotte utilizzando i metodi descritti in questo studio. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Il presente studio dimostra la pipeline completa per analizzare campioni ottenuti da cuori umani interi. I risultati rappresentativi mostrano esami anatomici su micro-macroscala eseguiti di routine per un singolo cuore. Poiché un campione di cuore umano è estremamente prezioso, un approccio multiscala è ideale ed efficace in modo da non sprecare alcuna parte del campione applicando più protocolli per vari scopi, tra cui la scoperta nella ricerca scientifica, il miglioramento delle procedure cliniche e l'educazione anatomica con il mantenimento della correlazione anatomica nel contesto dell'intero cuore.
Per quanto riguarda l'esame su microscala, l'immunocolorazione e la microscopia possono essere applicate per comprendere la citoarchitettura di campioni cardiaci umani. Qui viene dimostrata l'applicazione della chiarificazione tissutale e dell'immunoistochimica per studiare la neuroanatomia cardiaca su scala cellulare. L'uso di queste tecniche è utile nella caratterizzazione del sistema nervoso cardiaco e dei pattern di innervazione miocardica in relazione alle strutture di interesse, come il sistema di conduzione cardiaca e le camere cardiache. Sebbene si ottenga un'eccellente risoluzione spaziale, l'uso della microscopia confocale, in particolare per i campioni puliti dai tessuti, richiede molto tempo. La microscopia Lightsheet può essere utilizzata per ridurre il tempo di acquisizione delle immagini a scapito della risoluzione spaziale.
Per quanto riguarda l'esame su meso-macroscala, la risoluzione spaziale degli scanner micro-CT nell'istituto degli autori varia da 10 a 200 μm. La dimensione del campione è limitata a 20 mm per una scansione di 10 μm e a 120 mm per una scansione di 100-200 μm. Gli scanner Micro-CT nell'istituto degli autori non possono ospitare tutto il cuore. Pertanto, presso l'istituto degli autori, una scansione dell'intero cuore richiede l'uso di uno scanner CT clinico con risoluzione spaziale di 600 μm, sebbene i progressi abbiano consentito lo sviluppo di scanner micro-CT in grado di visualizzare l'intero cuore2. Lo sviluppo tecnologico, come la CT a conteggio di fotoni, amplierà sicuramente ulteriori possibilità. Il miglioramento della risoluzione spaziale del file STL dovrebbe essere il primo passo per migliorare ulteriormente la qualità della stampa 3D. Il costo più elevato della stampa 3D limita l'applicazione della tecnica alla pratica clinica di routine. Le immagini fotogrammetriche generate da qualsiasi applicazione per smartphone sono facili da sviluppare e di qualità accettabile, ma richiedono ulteriori sistemi sofisticati ma costosi e dispendiosi in termini di tempo per migliorare la risoluzione26. Per visualizzare in 3D, la realtà estesa con copricapo dedicato27,28 e monitor olografici29 sono strumenti aggiuntivi ma sono anche limitati da costi più elevati.
In sintesi, attraverso analisi strutturali complete su scale microscopiche e macroscopiche, l'anatomia su microscala di ciascuna struttura e il suo contributo funzionale possono essere compresi nel contesto dell'intero cuore. Insieme allo sviluppo dell'imaging ad alta risoluzione, la distanza tra l'anatomia su micro e macroscala si sta espandendo notevolmente. Gli esperti nell'analisi al microscopio elettronico dei cardiomiociti potrebbero non avere familiarità con il numero di lecci mitralici e viceversa. Per facilitare la comprensione completa della morfologia cardiaca, gli scienziati devono continuare a esplorare ulteriori dettagli di ogni albero, mantenendo la vista a volo d'uccello dell'intera foresta.
Divulgazioni
Nessuno.
Riconoscimenti
Ringraziamo le persone che hanno donato i loro corpi per il progresso dell'istruzione e della ricerca. Siamo grati alla OneLegacy Foundation, che ha costituito la base per ottenere i cuori dei donatori per la ricerca. Siamo anche grati ad Anthony A. Smithson e Arvin Roque-Verdeflor dell'UCLA Translational Research Imaging Center (Dipartimento di Radiologia) per il loro supporto nell'acquisizione dei dati TC. Questo progetto è stato sostenuto dall'UCLA Amara Yad Project. Siamo grati ai dottori Kalyanam Shivkumar e Olujimi A. Ajijola per aver stabilito e mantenuto una pipeline di cuore umano per la ricerca. Apprezziamo il nostro Research Operations Manager, Amiksha S. Gandhi, per la sua dedizione nel sostenere i nostri progetti. Questo lavoro è stato reso possibile dal sostegno delle sovvenzioni NIH OT2OD023848 e P01 HL164311 e della sovvenzione Leducq 23CVD04 a Kalyanam Shivkumar, del premio per lo sviluppo della carriera dell'American Heart Association 23CDA1039446 a PH e del progetto UCLA Amara-Yad (https://www.uclahealth.org/medical-services/heart/arrhythmia/about-us/amara-yad-project). Lo scanner microPET/CT GNEXT utilizzato in questo studio è stato finanziato da una sovvenzione NIH Shared Instrumentation for Animal Research (1 S10 OD026917-01A1).
Materiali
| Name | Company | Catalog Number | Comments |
| 1x Phosphate buffered saline | Sigma-Aldrich | P3813 | |
| 3D Viewer | Microsoft | ||
| 647 AffiniPure Donkey Anti-Rabbit IgG | Jackson ImmunoResearch Laboratories | 711-605-152 | |
| 647 AffiniPure Donkey Anti-Sheep IgG | Jackson ImmunoResearch Laboratories | 713-605-147 | |
| AF Micro-NIKKOR 200 mm f/4D IF-ED lens | Nikon | ||
| Anti-Actin, α-Smooth Muscle - Cy3 antibody | Sigma-Aldrich | C6198 | |
| Antigen Retrieval Buffer (100x EDTA Buffer, pH 8.0) | Abcam | ab93680 | |
| Anti-PGP9.5 (protein gene product 9.5) | Abcam | ab108986 | |
| Anti-TH (tyrosine hydrox ylase) | Abcam | ab1542 | |
| Anti-VAChT (vesicular acetylcholine transporter) | Synaptic Systems | 139 103 | |
| Benzyl ether | Sigma-Aldrich | 108014 | |
| Bovine serum albumin | Sigma-Aldrich | A4503-10G | |
| Cheetah 3D printer filament (95A), 1.75 mm | NinjaTek | ||
| Coverslip, 22 mm x 30mm, No. 1.5 | VWR | 48393 151 | |
| Cy3 AffiniPure Donkey Anti-Rabbit IgG | Jackson ImmunoResearch Laboratories | 711-165-152 | |
| Dichloromethane | Sigma-Aldrich | 270997-100ML | |
| Dimethyl sulfoxide | Sigma-Aldrich | D8418-500ML | |
| Ethanol, 100% | Decon laboratories | 2701 | |
| Glycine | Sigma-Aldrich | G7126-500G | |
| GNEXT PET/CT | SOFIE Biosciences | ||
| Heparin sodium salt from porcine intestinal mucosa | Sigma-Aldrich | H3149-50KU | |
| Histodenz | Sigma-Aldrich | D2158-100G | |
| Hydrogen peroxide solution | Sigma-Aldrich | H1009-500ML | |
| Imaging software | Zeiss | ZEN (black edition) | |
| Imaging software | Oxford Instruments | Imaris 10 | |
| iSpacer | Sunjin Labs | iSpacer 3mm | |
| KIRI Engine | KIRI Innovation | ||
| Laser scanning confocal microscope | Zeiss | LSM 880 | |
| LEAD-2 - Vertical & Multi-channels Peristaltic Pump | LONGER | ||
| Lightview XL | Brightech | ||
| Methanol (Certified ACS) | Fischer Scientific | A412-4 | |
| Nikon D850 | Nikon | ||
| NinjaTek NinjaFlex TPU @MK4 | NinjaTek | ||
| Normal donkey serum | Jackson ImmunoResearch Laboratories | 017-000-121 | |
| Original Prusa MK4 3D printer | Prusa Research | ||
| PAP pen | Abcam | ab2601 | |
| Paraformaldehyde, 32% | Electron Microscopy Sciences | 15714-S | |
| Polycam | Polycam | ||
| Primary antibody | |||
| PrusaSlicer 2.7.1 | Prusa Research | ||
| SARA-Engine | pita4 mobile LLC | ||
| Scaniverse | Niantic | ||
| Secondary antibody | |||
| SlowFade Gold Antiface Mountant | Invitrogen | S36936 | |
| Sodium azide, 5% (w/v) | Ricca Chemical Company | 7144.8-32 | |
| SOMATOM Definition AS | Siemens Healthcare | ||
| Standard Field Surgi-Spec Telescopes, | Designs for Vision | ||
| Stereomicroscope System SZ61 | OLYMPUS | ||
| StereoPhoto Maker | Free ware developed by Masuji Suto | ||
| Superfrost Plus Microscope Slides, Precleaned | Fisher Scientific | 12-550-15 | |
| Triton X-100 | Sigma-Aldrich | T8787-50ML | |
| Tween-20 | Sigma-Aldrich | P9416-100ML | |
| Xylene | Sigma-Aldrich | 534056-4L | |
| Ziostation2 | Ziosoft, AMIN |
Riferimenti
- Tawara, S. Das reizleitungssystem des säugetierherzens. Eine anatomisch-histologische studie über das atrioventrikularbündel und die purkinjeschen fäden. Jena: Gustav fischer. , https://wellcomecollection.org/works/gnxjxg9e (1906).
- Stephenson, R. S., et al. High-resolution 3-dimensional imaging of the human cardiac conduction system from microanatomy to mathematical modeling. Sci Rep. 7 (1), 7188(2017).
- Kawashima, T., Sato, F. First in situ 3D visualization of the human cardiac conduction system and its transformation associated with heart contour and inclination. Sci Rep. 11 (1), 8636(2021).
- Bojsen-Moller, F., Tranum-Jensen, J. Whole-mount demonstration of cholinesterase-containing nerves in the right atrial wall, nodal tissue, and atrioventricular bundle of the pig heart. J Anat. 108, Pt 3 375-386 (1971).
- Zhao, Y., et al. Ganglionated plexi and ligament of marshall ablation reduces atrial vulnerability and causes stellate ganglion remodeling in ambulatory dogs. Heart Rhythm. 13 (10), 2083-2090 (2016).
- Chung, W. H., et al. Ischemia-induced ventricular proarrhythmia and cardiovascular autonomic dysreflexia after cardioneuroablation. Heart Rhythm. 20 (11), 1534-1545 (2023).
- Crick, S. J., Sheppard, M. N., Ho, S. Y., Gebstein, L., Anderson, R. H. Anatomy of the pig heart: Comparisons with normal human cardiac structure. J Anat. 193, Pt 1 105-119 (1998).
- Saburkina, I., Pauziene, N., Solomon, O. I., Rysevaite-Kyguoliene, K., Pauza, D. H. Comparative gross anatomy of epicardiac ganglionated nerve plexi on the human and sheep cardiac ventricles. Anat Rec (Hoboken). 306 (9), 2302-2312 (2023).
- Hoagland, D. T., Santos, W., Poelzing, S., Gourdie, R. G. The role of the gap junction perinexus in cardiac conduction: Potential as a novel anti-arrhythmic drug target. Prog Biophys Mol Biol. 144, 41-50 (2019).
- Aksu, T., Gopinathannair, R., Gupta, D., Pauza, D. H. Intrinsic cardiac autonomic nervous system: What do clinical electrophysiologists need to know about the "heart brain". J Cardiovasc Electrophysiol. 32 (6), 1737-1747 (2021).
- Hanna, P., et al. Innervation and neuronal control of the mammalian sinoatrial node a comprehensive atlas. Circ Res. 128 (9), 1279-1296 (2021).
- Rajendran, P. S., et al. Identification of peripheral neural circuits that regulate heart rate using optogenetic and viral vector strategies. Nature Communications. 10, 1944(2019).
- Renier, N., et al. Mapping of brain activity by automated volume analysis of immediate early genes. Cell. 165 (7), 1789-1802 (2016).
- Mcalpine, W. Heart and coronary arteries: An anatomical atlas for clinical diagnosis, radiological investigation, and surgical treatment. Springer-verlag. , ISBN 13: 9783540069850 (1975).
- Mori, S., Shivkumar, K. Stereoscopic three-dimensional anatomy of the heart: Another legacy of dr. Wallace a. Mcalpine. Anat Sci Int. 96 (3), 485-488 (2021).
- Izawa, Y., Nishii, T., Mori, S. Stereogram of the living heart, lung, and adjacent structures. Tomography. 8 (2), 824-841 (2022).
- Sato, T., Hanna, P., Ajijola, O. A., Shivkumar, K., Mori, S. Photogrammetry of perfusion-fixed heart: Innovative approach to study 3-dimensional cardiac anatomy. JACC Case Rep. 21, 101937(2023).
- Tretter, J. T., Gupta, S. K., Izawa, Y., Nishii, T., Mori, S. Virtual dissection: Emerging as the gold standard of analyzing living heart anatomy. J Cardiovasc Dev Dis. 7 (3), 30(2020).
- Thomas, A. C., Davies, M. J. The demonstration of cardiac pathology using perfusion-fixation. Histopathology. 9 (1), 5-19 (1985).
- Glagov, S., Eckner, F. A., Lev, M. Controlled pressure fixation apparatus for hearts. Arch Pathol. 76, 640-646 (1963).
- Iaizzo, P. A. The visible heart(r) project and free-access website 'atlas of human cardiac anatomy. Europace. 18, suppl 4 163-172 (2016).
- Yang, Y., Huang, H., Li, L., Yang, Y. Multiplex immunohistochemistry staining for paraffin-embedded lung cancer tissue. J Vis Exp. (201), e65850(2023).
- Tripathy, S., Das, S. K. Strategies for organ preservation: Current prospective and challenges. Cell Biol Int. 47 (3), 520-538 (2023).
- Mori, S., Shivkumar, K. Atlas of cardiac anatomy (anatomical basis of cardiac interventions. Vol. 1). Cardiotext. , (2022).
- Crosado, B., et al. Phenoxyethanol-based embalming for anatomy teaching: An 18 years' experience with crosado embalming at the university of otago in new zealand. Anat Sci Educ. 13 (6), 778-793 (2020).
- Titmus, M., et al. A workflow for the creation of photorealistic 3d cadaveric models using photogrammetry. J Anat. 243 (2), 319-333 (2023).
- Silva, J. N. A., Southworth, M., Raptis, C., Silva, J. Emerging applications of virtual reality in cardiovascular medicine. JACC Basic Transl Sci. 3 (3), 420-430 (2018).
- Maresky, H. S., et al. Virtual reality and cardiac anatomy: Exploring immersive three-dimensional cardiac imaging, a pilot study in undergraduate medical anatomy education. Clin Anat. 32 (2), 238-243 (2019).
- Mori, S., Shivkumar, K. Real three-dimensional cardiac imaging using leading-edge holographic display. Clin Anat. 34 (6), 966-968 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon