Method Article
Pipeline pour l’étude anatomique tridimensionnelle multi-échelle du cœur humain
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole présente un pipeline complet pour analyser des échantillons obtenus à partir de cœurs humains qui couvrent les échelles microscopique et macroscopique.
Résumé
L’étude détaillée des cœurs humains non défaillants rejetés pour la transplantation offre une occasion unique d’effectuer des analyses structurelles à des échelles microscopiques et macroscopiques. Ces techniques comprennent l’élimination des tissus (imagerie tridimensionnelle (3D) modifiée par immunomarquage des organes éliminés par solvant) et la coloration immunohistochimique. Les procédures d’examen mésoscopique comprennent la dissection stéréoscopique et la tomodensitométrie (TDM). Les procédures d’examen macroscopique comprennent la dissection macroscopique, la photographie (y compris les anaglyphes et la photogrammétrie), la tomodensitométrie et l’impression 3D du cœur disséqué physiquement ou virtuellement ou entier. Avant l’examen macroscopique, une fixation par perfusion de pression peut être effectuée pour maintenir l’architecture 3D et la morphologie physiologiquement pertinente du cœur. L’application combinée de ces techniques à l’étude du cœur humain est unique et cruciale pour comprendre la relation entre des caractéristiques anatomiques distinctes telles que la coronaroplastie et l’innervation myocardique dans le contexte de l’architecture 3D du cœur. Ce protocole décrit les méthodologies en détail et comprend des résultats représentatifs pour illustrer les progrès de la recherche sur l’anatomie cardiaque humaine.
Introduction
Comme la fonction suit la forme, la compréhension de l’architecture du cœur est fondamentale pour apprécier sa physiologie. Bien que de nombreuses recherches aient révélé l’anatomie cardiaque à l’échelle microscopique 1,2,3, de nombreuses questions restent en suspens, en particulier celles liées à l’anatomie cardiaque humaine. Cela s’explique en partie par le fait que les études fondamentales axées sur l’anatomie fonctionnelle ont généralement utilisé des cœurs d’animaux 4,5,6, qui sont souvent distincts des cœurs humains 1,7,8. De plus, chaque étude individuelle, même celles utilisant des échantillons de cœur humain, a tendance à se concentrer sur des structures très spécifiques, ce qui rend difficile l’application des résultats dans le contexte de l’ensemble du cœur. C’est encore plus vrai si les structures focalisées se trouvent à des échelles micro ou mésoscopiques, comme le perinexus9 et les plexus ganglionnaires10.
Dans ce contexte, l’étude structurale systémique du cœur humain rejeté pour la transplantation offre une occasion unique et rare d’obtenir un atlas complet des structures cardiaques en mettant l’accent sur les échelles microscopiques et macroscopiques11. Les protocoles d’examen microscopique comprennent l’élimination des tissus (imagerie tridimensionnelle (3D) modifiée des organes éliminés par immunomarquage, iDISCO+)12,13 et la coloration immunohistochimique. Les protocoles d’examen mésoscopique comprennent la dissection stéréoscopique, la macrophotographie et la tomodensitométrie (TDM). Les protocoles d’examen macroscopique comprennent la dissection macroscopique14, la photographie (y compris les anaglyphes et la photogrammétrie)15,16,17, la tomodensitométrie, la dissection virtuelle 18 et l’impression 3D du cœur disséqué physiquement ou virtuellement ou du cœur entier17. En préparation à l’examen macroscopique, une fixation par perfusion de pression est effectuée pour maintenir l’architecture 3D et la morphologie physiologiquement pertinente du cœur 14,19,20,21. L’application combinée de ces techniques est unique et cruciale pour corréler des caractéristiques anatomiques distinctes dans le contexte de l’architecture 3D du cœur humain.
Comme la possibilité d’obtenir un échantillon de cœur humain non pathologique est extrêmement limitée, une approche multi-échelle décrite dans le présent document maximise l’utilisation de l’échantillon. En appliquant les différentes procédures décrites ci-dessous, des résultats représentatifs illustreront au lecteur comment les résultats peuvent être utilisés à des fins multiples, y compris la découverte dans la recherche scientifique11 (analyses complètes de l’innervation cardiaque, de la distribution des plexus ganglionnaires), l’amélioration des procédures cliniques (simulation pour les approches chirurgicales et interventionnelles) et l’éducation anatomique (démonstration 3D réelle de l’anatomie cardiaque).
Protocole
Cette étude a utilisé des échantillons de tissus anonymisés prélevés sur des cœurs humains de donneurs non défaillants et a été approuvée par le conseil d’examen institutionnel de l’Université de Californie à Los Angeles (UCLA). Des échantillons ont été prélevés à partir de cœurs non défaillants qui ont été rejetés pour la transplantation. Les cœurs ont été perfusés sous pression, fixés dans du paraformaldéhyde (PFA) à 4 % et imagés avant le traitement des tissus selon les méthodes suivantes. La figure 1 résume l’organigramme de l’ordre de l’étude. Les détails des réactifs et de l’équipement utilisés dans l’étude sont énumérés dans la table des matériaux.
1. Examen à l’échelle microscopique
- Nettoyage tissulaire à l’aide de l’imagerie 3D modifiée d’organes clairsemés par immunomarquage (protocole iDISCO+).
- Disséquez du tissu fixé à 4 % de PFA à l’aide d’un scalpel pour l’adapter à la chambre de 3 mm × 16 mm × 25 mm pour la microscopie confocale. Pour imager des tissus plus épais, des chambres et/ou des espaceurs supplémentaires peuvent être empilés sur la lame.
- Déshydrater les échantillons à l’aide de la série de méthanol gradué (MeOH) (20 %, 40 %, 60 %, 80 % et 100 % MeOH dans du H2O désionisé [vol/vol]) pendant 1 h chacun à température ambiante (RT) avec agitation.
- Laver avec 100% de MeOH pendant 1 h à RT et immerger dans 66% de dichlorométhane/33% de MeOH à RT avec agitation pendant la nuit.
- Le lendemain, laver deux fois au MeOH (100%) pendant 1 h à RT, réfrigérer à 4 °C et traiter à 5% H2O2 in MeOH (vol/vol) pendant la nuit à 4 °C.
- Réhydratez-vous avec les séries MeOH graduées (80 %, 60 %, 40 % et 20 % MeOH) et lavez à 0,01 mol/L PBS pendant 1 h chacun à RT avec agitation.
- Laver les mouchoirs deux fois dans du PBS 0,01 mol/L avec 0,2 % de Triton X-100 pendant 1 h à RT.
- Préparez-vous à l’immunomarquage par perméabilisation dans 0,01 mol/L de PBS, 20 % de diméthylsulfoxyde (DMSO), 0,2 % de Triton X-100 et 0,3 mol/L de glycine pendant 2 jours à 37 °C avec agitation.
- Bloquer dans 0,01 mol/L de PBS avec 10 % de DMSO, 0,2 % de Triton X-100 et 5 % de sérum d’ânesse normal pendant encore 2 jours à 37 °C avec agitation.
- Marquage avec un anticorps primaire compatible avec le MeOH conjugué à des fluorophores dilués dans 0,01 mol/L de PBS avec 10 mg/mL d’héparine (PTwH), 0,2 % de Tween-20, 5 % de DMSO et 3 % de sérum d’âne normal pendant 3 à 4 jours à 37 °C avec agitation.
- Réapprovisionnez la solution d’anticorps et incubez pendant encore 3 à 4 jours à 37 °C en agitant.
- Après 1 semaine d’incubation dans une solution d’anticorps primaires, laver 4 à 5 fois dans PTwH pendant la nuit à RT.
- Incuber avec des anticorps secondaires conjugués à des fluorophores dilués dans du PTwH, 3% de sérum d’âne normal pendant 3 jours à 37 °C avec agitation.
- Réapprovisionnez la solution d’anticorps secondaires, incubez pendant encore 3 jours à 37 °C en agitant.
- Après une incubation de 6 jours dans une solution secondaire d’anticorps, laver dans PTwH 4 à 5 fois pendant la nuit à RT.
- Déshydrater avec une série MeOH graduée (20 %, 40 %, 60 %, 80 %, 100 % et 100 % MeOH). L’échantillon peut être stocké pendant la nuit à RT.
- Incuber dans 66% de dichlorométhane/33% de MeOH pendant 3 h à la RT avec agitation.
- Laver deux fois au 100% de dichlorométhane pendant 15 min à RT avec agitation.
- Incuber et stocker les échantillons dans de l’éther benzylique. Remplissez le tube pour minimiser l’oxydation de l’échantillon par l’air.
- Imagerie d’un échantillon nettoyé des tissus
- Fixez une chambre contenant de l’adhésif sur une lame et appliquez du vernis à ongles sur le pourtour de la chambre. Pour les tissus plus épais, des chambres et/ou des entretoises supplémentaires peuvent être empilées sur la lame.
- Placez le mouchoir transparent dans la chambre, remplissez-le d’éther benzylique et appliquez une lamelle.
- Appliquez du vernis à ongles autour de la lamelle pour créer une étanchéité.
- Obtenez des images tilescan et Z stack à l’aide d’un microscope confocal à balayage laser vertical avec un objectif 5x ou 10x pour imager à une profondeur allant jusqu’à la distance de travail de l’objectif.
- Image à une résolution de 1024 x 1024 en utilisant des raies laser appropriées pour les spectres d’émission des fluorophores utilisés. L’autofluorescence musculaire est visible à l’aide de la ligne laser de 488 nm.
- Assurez-vous que la taille du pas de l’axe z est proportionnelle à l’échantillonnage de Nyquist basé sur l’ouverture numérique de l’objectif11 spécifié.
- Assemblez des images et utilisez un logiciel de visualisation 3D.
- Créez des figures à l’aide d’images de projection d’intensité maximale (MIP) d’empilements Z pour des canaux individuels et fusionnés (Figure 2).
- Immunohistochimie
REMARQUE : Une fois que le tissu est inclus dans la paraffine22, la procédure suivante est utilisée pour créer des lames pour l’étude immunohistochimique.- Préparation de la solution d’adaptation de l’indice de réfraction (RIMS).
- Préparez un tampon phosphate de 0,1 mol/L en ajoutant 10,9 g de Na2HPO4 (anhydre) et 3,1 g de NaH2PO4 (monohydraté) à du H2O désionisé pour obtenir un volume total de 1 L (pH 7,4). Filtrez-stérilisez la solution et conservez-la à RT.
- Diluer le tampon phosphate à 0,02 mol/L.
- Dissoudre Histodenz dans 30 mL de tampon phosphate à 0,02 mol/L en agitant la solution pendant 10 minutes avec une barre d’agitation magnétique dans le dernier flacon de stockage qui peut être scellé pour minimiser l’évaporation et la contamination.
- Ajoutez de l’azoture de sodium à une concentration totale de 0,01 % (p/v) et ajustez le pH à 7,5 avec du NaOH.
- Ajustez l’IR en faisant varier la concentration finale d’Histodenz.
- Stockez le RIMS chez RT pendant des mois. Jeter si une contamination microbienne est constatée.
REMARQUE : Ne pas autoclaver des solutions contenant de l’azoture de sodium.
- Préparation de lames pour l’étude immunohistochimique
- Créez des sections de 5 μm d’épaisseur avec un microtome. Appliquez une section de tissu sur les lames chargées.
- Retirer la paraffine en incubant les lames dans du xylène à >75 % pendant 10 min. Déplacer les lames dans un deuxième contenant contenant du xylène pendant 10 minutes supplémentaires.
- Retirez le xylène en immergeant les lames dans 100% d’EtOH pendant 10 min, puis dans 95% d’EtOH pendant 5 min et 70% d’EtOH pendant 5 min.
- Rincez les lames avec du H2O déminéralisé pendant 5 min.
- Immerger les lames dans le tampon de récupération d’antigène pendant 25 min à 90-95°C.
- Laisser refroidir le récipient à RT pendant 1 h en agitant.
- Immergez les lames dans un tampon de trempage (0,01 mol/L PBS + 0,4 % Triton X-100) pendant 30 min à 4°C.
- Encerclez le tissu avec un stylo PAP. Ajoutez du PBS à chaque lame et placez-la dans une chambre humidifiée pour éviter la dessiccation.
- Laver les lames avec PBS à RT avec agitation pendant 5 min.
- Bloc avec tampon bloquant (0,01 mol/L PBS + 10% sérum d’ânesse + 0,1% TX-100) pendant 1 h avec agitation.
- Incuber avec un anticorps primaire dilué dans un tampon de blocage pendant la nuit à 4°C.
- Le lendemain, laissez les diapositives se réchauffer à RT pendant 15 min.
- Lavez les lames 3 fois avec 0,01 mol/L de PBS + 0,2 % de TritonX-100 pendant 5 min.
- Incuber avec un anticorps secondaire dilué dans un tampon de blocage pendant 1 h à RT avec agitation.
- Lavez les lames 3 fois avec 0,01 mol/L de PBS + 0,2 % de TritonX-100 pendant 5 min.
- Lavez les lames 3 fois avec 0,01 mol/L de PBS pendant 5 min.
- Placez 1 goutte de RIMS à l’aide d’un compte-gouttes et appliquez une lamelle.
- Appliquez du vernis à ongles autour de la lamelle pour créer l’étanchéité.
- En tant que témoin négatif, prélevez un échantillon sans l’anticorps primaire pour démontrer l’absence de coloration spécifique.
- Imagerie de lames immunomarquées
- Imagez les lames à l’aide d’un microscope confocal à balayage laser avec des objectifs 10x, 20x et 40x.
- Image à une résolution de 1024 x 1024 à l’aide de raies laser appropriées pour les spectres d’émission des anticorps secondaires utilisés.
- Créez des figures à l’aide d’images de projection d’intensité maximale (MIP) d’empilements Z pour des canaux individuels et fusionnés (Figure 3).
- Préparation de la solution d’adaptation de l’indice de réfraction (RIMS).
2. Examen à méso-échelle
- Dissection stéréoscopique
- Effectuez des dissections délicates en se concentrant sur des structures minuscules ou minces, telles que le nœud auriculo-ventriculaire, l’artère du ganglion auriculo-ventriculaire et le plexus nerveux cardiaque (échelle submillimétrique à millimétrique) avec une lampe de bureau loupe avec pince, des télescopes chirurgicaux ou un stéréomicroscope.
- Balayage Micro-CT
REMARQUE : L’imagerie par tomodensitométrie est réalisée après la perfusion sous pression et la fixation et à tous les stades de la dissection à l’aide d’un scanner par tomographie par émission de micropositons (TEP)/TDM (Figure 4).- Réchauffez la source de rayons X CT pendant 25 minutes avant l’imagerie de l’échantillon.
- Placez l’échantillon de cœur sur le lit du scanner.
- Déplacez le lit du scanner en position horizontale de 544 mm et en position verticale de 14 mm pour centrer le cœur dans le champ de vision de la tomodensitométrie.
- Acquisition d’images CT à 80 kVp, 150 μA, avec 720 projections pendant 1 min de balayage à une résolution spatiale de 200 μm.
- Reconstruisez les données CT avec un champ de vision de 12 cm x 12 cm x 10 cm et une matrice de 600 x 600 x 500 voxels, et enregistrez-les en tant que fichier DICOM.
3. Examen à grande échelle
- Perfusion et fixation sous pression
REMARQUE : Les auteurs modifient les techniques de perfusion et de fixation par pression précédemment décrites et les appliquent aux cœurs humains non défaillants rejetés pour la transplantation 14,19,20,21.- Utilisez des pompes à haut débit pour la fixation de la perfusion. Utilisez soit de l’éthanolà 100 %, du PFA à 4 % ou du formol à 10 % pour le fixateur.
REMARQUE : Le cœur est récupéré avec l’aorte ascendante, le tronc pulmonaire et la veine cave et les veines pulmonaires réséqués aussi distalement que possible et délivrés dans la solution23 de l’Université du Wisconsin. - Utilisez deux canules chirurgicales de 20-24 Fr pour la perfusion cardiaque droite et gauche. Pour la perfusion du cœur droit, canulez la veine cave supérieure et placez un évent au niveau du tronc pulmonaire ou de l’artère pulmonaire avec des seringues en plastique demi-coupées de 12 à 30 ml avec des embouts Luer-Lock fixés à des robinets d’arrêt à trois voies.
- Occlure la veine cave inférieure et l’autre artère pulmonaire avec de la ficelle après avoir mis une seringue en plastique demi-coupée verrouillée de taille appropriée ou un tube à centrifuger de 1,5 à 5,0 ml.
- Pour la perfusion antérograde du cœur gauche, canuler l’une des veines pulmonaires et placer un évent à l’extrémité distale coupée de l’aorte avec les seringues en plastique demi-coupées de 12 à 30 ml avec des embouts Luer-Lock fixés à des robinets d’arrêt à trois voies.
- Pour une perfusion rétrograde du cœur gauche, canulez l’une des branches de l’arc aortique et placez un évent sur une autre branche de l’arc aortique. Placez les extrémités des canules dans les deux ventricules.
- Obstruez les autres orifices vasculaires à l’aide d’une ficelle après avoir inséré une seringue en plastique demi-coupée verrouillée de taille appropriée ou un tube de microcentrifugation de 1,5 à 5,0 ml. Utilisez une gaze fine pour couvrir la partie d’insertion des seringues/tubes/canules afin d’éviter les fuites et les glissades. Réparez les fuites importantes à l’aide de sutures, de bandes ou d’agglomérations. Les petites fuites sont autorisées.
- Suspendez le cœur dans un récipient en plastique.
- Connectez un tube en plastique souple 22-24 Fr à chaque canule et insérez l’autre extrémité du tube dans le récipient rempli de fixateur.
- Faire circuler le fixateur dans les circuits du cœur droit et du cœur gauche à l’aide d’une pompe à haut débit réglée à environ 100-300 ml/min pour le cœur droit et 200-400 mL/min pour le cœur gauche afin d’atteindre environ 20 mmHg dans le ventricule droit et 80 mmHg dans le ventricule gauche, respectivement.
- Maintenir la perfusion à 4 °C pendant 24 h.
- Lavez le cœur avec 0,01 mol/L de PBS pendant 30 minutes en agitant quatre fois.
- Stocker le cœur dans 0,01 mol/L de PBS/0,02 % d’azoture de sodium à 4 °C.
REMARQUE : La fixation par perfusion de pression n’est efficace que pour un cœur frais, pas pour un cœur récupéré d’un cadavre embaumé.
- Utilisez des pompes à haut débit pour la fixation de la perfusion. Utilisez soit de l’éthanolà 100 %, du PFA à 4 % ou du formol à 10 % pour le fixateur.
- Dissection macroscopique
- Effectuer une dissection progressive à l’aide d’enregistrements photographiques à chaque étape de la dissection.
- Pour maintenir la pertinence clinique, faites particulièrement attention à ne pas déformer/déformer les structures afin de maintenir la morphologie physiologique du cœur.
- Imagez les structures cibles en utilisant une orientation cliniquement pertinente, telle que l’orientation oblique antérieure droite.
- Photographie
- Placez le cœur perfusé et fixe sur un trépied avec une plate-forme montée avec plusieurs broches et la possibilité de pivoter à 360°.
- Photographiez le cœur à l’aide d’un appareil photo reflex numérique à objectif unique (Figure 5)24 tout en utilisant plusieurs panneaux lumineux à diodes électroluminescentes placés sur les supports en C et un large tissu d’arrière-plan noir duvetyn.
- Photographiez à l’aide de l’objectif avec une longue distance focale (200 mm) pour une distance de travail de 4 à 6 pieds afin de minimiser la distorsion du sujet14.
- Anaglyphes
- Pour afficher des images anaglyphiques, reconstruisez une paire de photographies ou d’images rendues en volume à partir d’ensembles de données CT avec une différence de 10° dans l’angle de rotation sur le plan horizontal.
- Convertissez un ensemble de ces images bidimensionnelles (2D), appelé stéréogramme, en anaglyphes à l’aide du logiciel gratuit16.
- Pour afficher un anaglyphe, utilisez des lunettes rouges/cyan.
- Photogrammétrie
REMARQUE : La photogrammétrie est la science appliquée qui consiste à générer une reconstruction tridimensionnelle de surface à partir de plusieurs photographies bidimensionnelles prises sous différents angles17.- Suspendez l’échantillon sur des C-Stands ou placez-le sur la table rotative pour obtenir des centaines de photographies multidirectionnelles avec un smartphone.
- Générez le modèle 3D au format FBX à l’aide d’un logiciel disponible dans le commerce.
- Tomodensitométrie
REMARQUE : La tomodensitométrie peut être effectuée après la perfusion sous pression et la fixation et à n’importe quel stade de la dissection.- Suspendez l’échantillon de cœur à une barre placée sur le dessus du récipient. Pour éviter que le cœur ne se balance pendant l’examen, soutenez la base du cœur à l’aide de griffes en plastique fixées au fond du récipient. Ainsi, l’air servira de contraste négatif.
- Effectuez la tomodensitométrie à l’aide d’un tomodensitomètre multi-détecteurs disponible dans le commerce avec les paramètres suivants : tension du tube de 120 kV, courant du tube de 800-900 mA et rotation du portique de 280 ms. La durée de la dose est généralement de 500 à 1200 mGy.cm.
- Reconstruisez les données de l’image axiale à l’aide des paramètres suivants : une épaisseur de section, 0,6 mm ; un intervalle incrémentiel, 0,3 mm ; un champ de vision, le plus petit possible (généralement 100-200 mm) ; et une matrice, 512 × 512.
- Dissection virtuelle
- Analysez les images de tomodensitométrie à l’aide d’un logiciel disponible dans le commerce pour générer des images de dissection virtuelles.
REMARQUE : La dissection virtuelle est une modification du processus de rendu de volume dans laquelle l’accent est déplacé vers les parois des cavités et des vaisseaux cardiaques18. Dans ce processus, le seuillage manuel supprime pratiquement la chambre améliorée des ensembles de données d’origine. - Visualisez les parois, les septa et les vannes non améliorées avec une dissection virtuelle pour produire des images similaires à la dissection grossière. Contrairement à la dissection macroscopique des échantillons cardiaques, les plans de coupe lors de la dissection virtuelle sont pratiquement illimités. Presque n’importe quelle vue peut être recréée pour visualiser les structures d’intérêt selon les besoins.
- Analysez les images de tomodensitométrie à l’aide d’un logiciel disponible dans le commerce pour générer des images de dissection virtuelles.
- Impression 3D
- Ouvrez le fichier compatible de l’échantillon de cœur dans le logiciel d’impression 3D.
- Utilisez le profil de 0,10 mm Fast DETAIL pour les paramètres d’impression dans l’imprimante 3D et réduisez la vitesse d’impression à 20 mm/s. Activez l’option Générer du matériel de support.
- Utilisez le profil du filament TPU pour les « Paramètres du filament » dans l’imprimante 3D.
- Utilisez le profil de la buse Original Prusa MK4 Input Shaper 0.4 pour les « Paramètres de l’imprimante » dans l’imprimante.
- Une fois le découpage terminé, enregistrez le fichier BGCODE sur une clé USB pour l’impression 3D.
- Utilisez le filament TPU de 1,75 mm pour imprimer en 3D l’échantillon de cœur humain. Avant l’impression 3D, faites sécher le filament TPU pendant 6 h à l’aide d’un séchoir à filament.
- Pour réduire la tension du filament lors de l’impression 3D, placez la bobine de filament sur un porte-bobine avec un roulement intégré pour faciliter la rotation de la bobine de filament. Effectuez l’impression 3D à l’aide d’une imprimante 3D disponible dans le commerce avec une tôle d’acier texturée enduite de poudre.
- Retirez soigneusement les matériaux de support lorsque l’impression 3D est terminée.
Résultats
Examens à l’échelle microscopique
L’application d’un nettoyage tissulaire permet d’imager de plus grands volumes de tissus en 3D à l’aide de la microscopie confocale. Dans le cœur, les ganglions contenant les neurones cardiaques et la structure neuronale de l’innervation myocardique peuvent être visualisés (Figure 2). La figure 3 montre une image confocale du myocarde du ventricule gauche humain immunomarqué pour les nerfs et les cellules musculaires lisses. On note que les vaisseaux sanguins traversent le myocarde et que de nombreuses fibres nerveuses sont identifiées, à la fois en association avec et indépendamment des vaisseaux sanguins.
Examens à l’échelle méso et macroscopique
Lors de l’utilisation d’éthanol absolu pour la perfusion et la fixation sous pression de 24 heures, la couleur naturelle de l’échantillon est blanchie, le tissu est déshydratéet l’élasticité est considérablement réduite. D’autre part, lors de la fixation avec du PFA et du formol, la couleur naturelle et l’élasticité sont remarquablement maintenues. Pour ces raisons, le PFA ou le formol est principalement utilisé comme fixateur préféré.
La figure 6 montre des images représentatives de la dissection grossière, de la dissection virtuelle, du modèle polygonal STL et de l’impression tridimensionnelle. La figure 7 montre des images représentatives des anaglyphes créés à partir d’images de dissection grossière et virtuelle. La perception de la profondeur peut être obtenue avec des lunettes anaglyphiques. Le modèle photogrammétrique unique capturé peut être observé de presque toutes les directions à l’aide d’un logiciel disponible dans le commerce et démontre des caractéristiques anatomiques complexes pertinentes pour les procédures cardiaques transcathéter de routine. En appliquant ces techniques au cœur préparé avec perfusion de pression et fixation, les informations tridimensionnelles sur le cœur peuvent être presque éternellement conservées, numériquement ou physiquement, et partagées sans frontières. La figure 8 montre les impressions 3D à l’échelle 50 % reproduites à partir des cœurs disséqués rejetés pour la transplantation.

Figure 1 : Organigramme du protocole. Veuillez cliquer ici pour voir une version agrandie de cette figure.
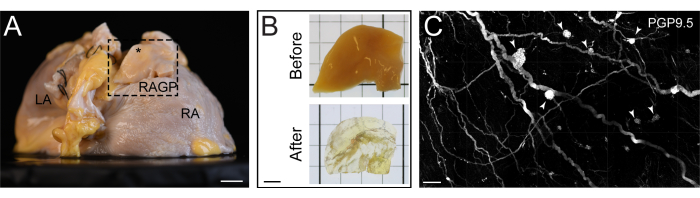
Figure 2 : Coupe de l’oreillette droite nettoyée des tissus. (A) Vue oblique postérieure droite d’un oreillette droite avec le plexus ganglionnaire de l’oreillette droite (RAGP) disséqué pour l’élimination des tissus. (B) Échantillon RAGP avant et après l’élimination des tissus. (C) Projection de l’intensité maximale d’une partie de RAGP humaine éliminée par iDISCO+ démontrant que les ganglions (pointes de flèches) sont immunomarqués avec le produit génique protéique marqueur pan-neuronal 9.5 (PGP9.5). Les barres d’échelle mesurent 1 cm (A), 5 mm (B) et 500 μm (C). Cette figure est adaptée de Hanna et al.11. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Lames immunomarquées du ventricule gauche humain. Image confocale d’une tranche de myocarde du ventricule gauche humain immunomarquée avec le gène marqueur pan-neuronal produit génique 9.5 (PGP9.5) et le marqueur des cellules musculaires lisses α-actine musculaire lisse (αSMA). L’autofluorescence musculaire est visible à l’aide de la ligne laser de 488 nm (verte). La barre d’échelle est de 50 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 4 : Imagerie par micro-tomodensitométrie d’échantillons cardiaques. (A) Installation de micro-tomodensitométrie pour l’imagerie d’échantillons cardiaques. (B) Interface utilisateur pour l’imagerie par micro-tomodensitométrie. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Décor d’un studio photo au Centre d’arythmie cardiaque de l’UCLA. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 6 : Dissection macroscopique (en haut à gauche), dissection virtuelle (en haut à droite), modèle de polygone STL (en bas à gauche) et images d’impression tridimensionnelle (en bas à droite) du complexe valvulaire aortique et mitral. Veuillez cliquer ici pour voir une version agrandie de cette figure.
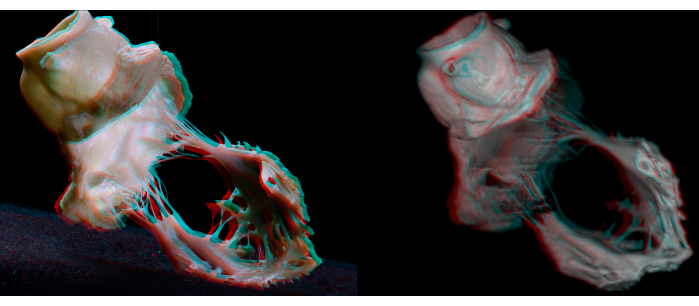
Figure 7 : Anaglyphes d’une dissection macroscopique (à gauche) et d’une dissection virtuelle (à droite) du complexe valvulaire aortique et mitral. Des lunettes anaglyphiques (rouge/cyan) sont nécessaires pour obtenir une perception de la profondeur. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 8 : Impression tridimensionnelle d’échantillons cardiaques. (A) Configuration d’une imprimante tridimensionnelle (3D) pour l’impression 3D d’échantillons cardiaques avec un filament TPU. (B) Impressions 3D cardiaques représentatives produites à l’aide des méthodes décrites dans cette étude. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
La présente étude démontre le pipeline complet d’analyse d’échantillons obtenus à partir de cœurs humains entiers. Les résultats représentatifs montrent des examens anatomiques à l’échelle microscopique et macroscopique effectués en routine pour un seul cœur. Comme un échantillon de cœur humain est extrêmement précieux, une approche multi-échelle est idéale et efficace afin de ne gaspiller aucune partie de l’échantillon en appliquant plusieurs protocoles à diverses fins, y compris la découverte dans la recherche scientifique, l’amélioration des procédures cliniques et l’éducation anatomique avec le maintien de la corrélation anatomique dans le contexte de l’ensemble du cœur.
En ce qui concerne l’examen à l’échelle microscopique, l’immunocoloration et la microscopie peuvent être appliquées pour comprendre la cytoarchitecture des échantillons cardiaques humains. Ici, l’application du nettoyage tissulaire et de l’immunohistochimie pour étudier la neuroanatomie cardiaque à l’échelle cellulaire est démontrée. L’utilisation de ces techniques est utile dans la caractérisation du système nerveux cardiaque et des modèles d’innervation myocardique en relation avec des structures d’intérêt, telles que le système de conduction cardiaque et les cavités cardiaques. Bien qu’une excellente résolution spatiale soit obtenue, l’utilisation de la microscopie confocale, en particulier pour les échantillons dont les tissus ont été éliminés, prend beaucoup de temps. La microscopie à feuille de lumière peut être utilisée pour réduire le temps d’acquisition d’images au détriment de la résolution spatiale.
En ce qui concerne l’examen à l’échelle méso à macroscopique, la résolution spatiale des scanners micro-CT dans l’établissement des auteurs varie de 10 à 200 μm. La taille de l’échantillon est limitée à 20 mm pour un balayage de 10 μm et à 120 mm pour un balayage de 100-200 μm. Les scanners Micro-CT de l’institution des auteurs ne peuvent pas accueillir tout le cœur. Ainsi, dans l’établissement des auteurs, un scanner du cœur entier nécessite l’utilisation d’un scanner clinique avec une résolution spatiale de 600 μm, bien que les progrès aient permis le développement de scanners micro-CT capables d’imager l’ensemble du cœur2. Le développement technologique, comme la tomodensitométrie par comptage de photons, élargira certainement d’autres possibilités. L’amélioration de la résolution spatiale du fichier STL devrait être la première étape pour améliorer encore la qualité de l’impression 3D. Le coût plus élevé de l’impression 3D limite l’application de la technique à la pratique clinique de routine. Les images photogrammétriques générées à partir de n’importe quelle application pour smartphone sont faciles à développer et de qualité acceptable, mais nécessiteront d’autres systèmes sophistiqués mais coûteux et chronophages pour améliorer la résolution26. Pour visualiser en 3D, la réalité étendue avec un casque dédié27,28 et des moniteurs holographiques29 sont des outils supplémentaires mais sont également limités par un coût plus élevé.
En résumé, grâce à des analyses structurelles complètes à travers les échelles microscopiques et macroscopiques, l’anatomie à l’échelle microscopique de chaque structure et sa contribution fonctionnelle peuvent être comprises dans le contexte de l’ensemble du cœur. Parallèlement au développement de l’imagerie à haute résolution, la distance entre l’anatomie à l’échelle micro et macro s’étend considérablement. Les experts en analyse au microscope électronique des cardiomyocytes ne connaissent peut-être pas le nombre de feuillets mitrals et vice versa. Pour faciliter la compréhension complète de la morphologie cardiaque, les scientifiques doivent continuer à explorer davantage de détails de chaque arbre, tout en conservant une vue d’ensemble de l’ensemble de la forêt.
Déclarations de divulgation
Aucun.
Remerciements
Nous remercions les personnes qui ont fait don de leur corps pour l’avancement de l’éducation et de la recherche. Nous sommes reconnaissants envers la Fondation OneLegacy, qui a servi de base à l’obtention de cœurs de donneurs pour la recherche. Nous sommes également reconnaissants à Anthony A. Smithson et Arvin Roque-Verdeflor du Translational Research Imaging Center de l’UCLA (Département de radiologie) pour leur soutien dans l’acquisition de données CT. Ce projet a été soutenu par le projet Amara Yad de l’UCLA. Nous sommes reconnaissants aux Drs Kalyanam Shivkumar et Olujimi A. Ajijola d’avoir établi et maintenu un pipeline de recherche sur le cœur humain. Nous apprécions notre directrice des opérations de recherche, Amiksha S. Gandhi, pour son dévouement à soutenir nos projets. Ce travail a été rendu possible grâce au soutien des subventions NIH OT2OD023848 & P01 HL164311 et de la subvention Leducq 23CVD04 à Kalyanam Shivkumar, du prix de développement de carrière 23CDA1039446 de l’American Heart Association à PH et du projet Amara-Yad de l’UCLA (https://www.uclahealth.org/medical-services/heart/arrhythmia/about-us/amara-yad-project). Le scanner microPET/CT GNEXT utilisé dans cette étude a été financé par une subvention NIH Shared Instrumentation for Animal Research Grant (1 S10 OD026917-01A1).
matériels
| Name | Company | Catalog Number | Comments |
| 1x Phosphate buffered saline | Sigma-Aldrich | P3813 | |
| 3D Viewer | Microsoft | ||
| 647 AffiniPure Donkey Anti-Rabbit IgG | Jackson ImmunoResearch Laboratories | 711-605-152 | |
| 647 AffiniPure Donkey Anti-Sheep IgG | Jackson ImmunoResearch Laboratories | 713-605-147 | |
| AF Micro-NIKKOR 200 mm f/4D IF-ED lens | Nikon | ||
| Anti-Actin, α-Smooth Muscle - Cy3 antibody | Sigma-Aldrich | C6198 | |
| Antigen Retrieval Buffer (100x EDTA Buffer, pH 8.0) | Abcam | ab93680 | |
| Anti-PGP9.5 (protein gene product 9.5) | Abcam | ab108986 | |
| Anti-TH (tyrosine hydrox ylase) | Abcam | ab1542 | |
| Anti-VAChT (vesicular acetylcholine transporter) | Synaptic Systems | 139 103 | |
| Benzyl ether | Sigma-Aldrich | 108014 | |
| Bovine serum albumin | Sigma-Aldrich | A4503-10G | |
| Cheetah 3D printer filament (95A), 1.75 mm | NinjaTek | ||
| Coverslip, 22 mm x 30mm, No. 1.5 | VWR | 48393 151 | |
| Cy3 AffiniPure Donkey Anti-Rabbit IgG | Jackson ImmunoResearch Laboratories | 711-165-152 | |
| Dichloromethane | Sigma-Aldrich | 270997-100ML | |
| Dimethyl sulfoxide | Sigma-Aldrich | D8418-500ML | |
| Ethanol, 100% | Decon laboratories | 2701 | |
| Glycine | Sigma-Aldrich | G7126-500G | |
| GNEXT PET/CT | SOFIE Biosciences | ||
| Heparin sodium salt from porcine intestinal mucosa | Sigma-Aldrich | H3149-50KU | |
| Histodenz | Sigma-Aldrich | D2158-100G | |
| Hydrogen peroxide solution | Sigma-Aldrich | H1009-500ML | |
| Imaging software | Zeiss | ZEN (black edition) | |
| Imaging software | Oxford Instruments | Imaris 10 | |
| iSpacer | Sunjin Labs | iSpacer 3mm | |
| KIRI Engine | KIRI Innovation | ||
| Laser scanning confocal microscope | Zeiss | LSM 880 | |
| LEAD-2 - Vertical & Multi-channels Peristaltic Pump | LONGER | ||
| Lightview XL | Brightech | ||
| Methanol (Certified ACS) | Fischer Scientific | A412-4 | |
| Nikon D850 | Nikon | ||
| NinjaTek NinjaFlex TPU @MK4 | NinjaTek | ||
| Normal donkey serum | Jackson ImmunoResearch Laboratories | 017-000-121 | |
| Original Prusa MK4 3D printer | Prusa Research | ||
| PAP pen | Abcam | ab2601 | |
| Paraformaldehyde, 32% | Electron Microscopy Sciences | 15714-S | |
| Polycam | Polycam | ||
| Primary antibody | |||
| PrusaSlicer 2.7.1 | Prusa Research | ||
| SARA-Engine | pita4 mobile LLC | ||
| Scaniverse | Niantic | ||
| Secondary antibody | |||
| SlowFade Gold Antiface Mountant | Invitrogen | S36936 | |
| Sodium azide, 5% (w/v) | Ricca Chemical Company | 7144.8-32 | |
| SOMATOM Definition AS | Siemens Healthcare | ||
| Standard Field Surgi-Spec Telescopes, | Designs for Vision | ||
| Stereomicroscope System SZ61 | OLYMPUS | ||
| StereoPhoto Maker | Free ware developed by Masuji Suto | ||
| Superfrost Plus Microscope Slides, Precleaned | Fisher Scientific | 12-550-15 | |
| Triton X-100 | Sigma-Aldrich | T8787-50ML | |
| Tween-20 | Sigma-Aldrich | P9416-100ML | |
| Xylene | Sigma-Aldrich | 534056-4L | |
| Ziostation2 | Ziosoft, AMIN |
Références
- Tawara, S. Das reizleitungssystem des säugetierherzens. Eine anatomisch-histologische studie über das atrioventrikularbündel und die purkinjeschen fäden. Jena: Gustav fischer. , https://wellcomecollection.org/works/gnxjxg9e (1906).
- Stephenson, R. S., et al. High-resolution 3-dimensional imaging of the human cardiac conduction system from microanatomy to mathematical modeling. Sci Rep. 7 (1), 7188(2017).
- Kawashima, T., Sato, F. First in situ 3D visualization of the human cardiac conduction system and its transformation associated with heart contour and inclination. Sci Rep. 11 (1), 8636(2021).
- Bojsen-Moller, F., Tranum-Jensen, J. Whole-mount demonstration of cholinesterase-containing nerves in the right atrial wall, nodal tissue, and atrioventricular bundle of the pig heart. J Anat. 108, Pt 3 375-386 (1971).
- Zhao, Y., et al. Ganglionated plexi and ligament of marshall ablation reduces atrial vulnerability and causes stellate ganglion remodeling in ambulatory dogs. Heart Rhythm. 13 (10), 2083-2090 (2016).
- Chung, W. H., et al. Ischemia-induced ventricular proarrhythmia and cardiovascular autonomic dysreflexia after cardioneuroablation. Heart Rhythm. 20 (11), 1534-1545 (2023).
- Crick, S. J., Sheppard, M. N., Ho, S. Y., Gebstein, L., Anderson, R. H. Anatomy of the pig heart: Comparisons with normal human cardiac structure. J Anat. 193, Pt 1 105-119 (1998).
- Saburkina, I., Pauziene, N., Solomon, O. I., Rysevaite-Kyguoliene, K., Pauza, D. H. Comparative gross anatomy of epicardiac ganglionated nerve plexi on the human and sheep cardiac ventricles. Anat Rec (Hoboken). 306 (9), 2302-2312 (2023).
- Hoagland, D. T., Santos, W., Poelzing, S., Gourdie, R. G. The role of the gap junction perinexus in cardiac conduction: Potential as a novel anti-arrhythmic drug target. Prog Biophys Mol Biol. 144, 41-50 (2019).
- Aksu, T., Gopinathannair, R., Gupta, D., Pauza, D. H. Intrinsic cardiac autonomic nervous system: What do clinical electrophysiologists need to know about the "heart brain". J Cardiovasc Electrophysiol. 32 (6), 1737-1747 (2021).
- Hanna, P., et al. Innervation and neuronal control of the mammalian sinoatrial node a comprehensive atlas. Circ Res. 128 (9), 1279-1296 (2021).
- Rajendran, P. S., et al. Identification of peripheral neural circuits that regulate heart rate using optogenetic and viral vector strategies. Nature Communications. 10, 1944(2019).
- Renier, N., et al. Mapping of brain activity by automated volume analysis of immediate early genes. Cell. 165 (7), 1789-1802 (2016).
- Mcalpine, W. Heart and coronary arteries: An anatomical atlas for clinical diagnosis, radiological investigation, and surgical treatment. Springer-verlag. , ISBN 13: 9783540069850 (1975).
- Mori, S., Shivkumar, K. Stereoscopic three-dimensional anatomy of the heart: Another legacy of dr. Wallace a. Mcalpine. Anat Sci Int. 96 (3), 485-488 (2021).
- Izawa, Y., Nishii, T., Mori, S. Stereogram of the living heart, lung, and adjacent structures. Tomography. 8 (2), 824-841 (2022).
- Sato, T., Hanna, P., Ajijola, O. A., Shivkumar, K., Mori, S. Photogrammetry of perfusion-fixed heart: Innovative approach to study 3-dimensional cardiac anatomy. JACC Case Rep. 21, 101937(2023).
- Tretter, J. T., Gupta, S. K., Izawa, Y., Nishii, T., Mori, S. Virtual dissection: Emerging as the gold standard of analyzing living heart anatomy. J Cardiovasc Dev Dis. 7 (3), 30(2020).
- Thomas, A. C., Davies, M. J. The demonstration of cardiac pathology using perfusion-fixation. Histopathology. 9 (1), 5-19 (1985).
- Glagov, S., Eckner, F. A., Lev, M. Controlled pressure fixation apparatus for hearts. Arch Pathol. 76, 640-646 (1963).
- Iaizzo, P. A. The visible heart(r) project and free-access website 'atlas of human cardiac anatomy. Europace. 18, suppl 4 163-172 (2016).
- Yang, Y., Huang, H., Li, L., Yang, Y. Multiplex immunohistochemistry staining for paraffin-embedded lung cancer tissue. J Vis Exp. (201), e65850(2023).
- Tripathy, S., Das, S. K. Strategies for organ preservation: Current prospective and challenges. Cell Biol Int. 47 (3), 520-538 (2023).
- Mori, S., Shivkumar, K. Atlas of cardiac anatomy (anatomical basis of cardiac interventions. Vol. 1). Cardiotext. , (2022).
- Crosado, B., et al. Phenoxyethanol-based embalming for anatomy teaching: An 18 years' experience with crosado embalming at the university of otago in new zealand. Anat Sci Educ. 13 (6), 778-793 (2020).
- Titmus, M., et al. A workflow for the creation of photorealistic 3d cadaveric models using photogrammetry. J Anat. 243 (2), 319-333 (2023).
- Silva, J. N. A., Southworth, M., Raptis, C., Silva, J. Emerging applications of virtual reality in cardiovascular medicine. JACC Basic Transl Sci. 3 (3), 420-430 (2018).
- Maresky, H. S., et al. Virtual reality and cardiac anatomy: Exploring immersive three-dimensional cardiac imaging, a pilot study in undergraduate medical anatomy education. Clin Anat. 32 (2), 238-243 (2019).
- Mori, S., Shivkumar, K. Real three-dimensional cardiac imaging using leading-edge holographic display. Clin Anat. 34 (6), 966-968 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon