Method Article
Riprogrammazione dell'adenocarcinoma duttale pancreatico alla pluripotenza
In questo articolo
Riepilogo
Il presente protocollo descrive la riprogrammazione dell'adenocarcinoma duttale pancreatico (PDAC) e delle cellule epiteliali duttali pancreatiche normali in cellule staminali pluripotenti indotte (iPSC). Forniamo una procedura ottimizzata e dettagliata, passo dopo passo, dalla preparazione dei lentivirus alla creazione di linee iPSC stabili.
Abstract
La generazione di cellule staminali pluripotenti indotte (iPSC) utilizzando fattori di trascrizione è stata ottenuta da quasi tutti i tipi di cellule differenziate e si è dimostrata molto preziosa per la ricerca e le applicazioni cliniche. È interessante notare che la riprogrammazione iPSC delle cellule tumorali, come l'adenocarcinoma duttale pancreatico (PDAC), ha dimostrato di invertire il fenotipo PDAC invasivo e di ignorare l'epigenoma del cancro. La differenziazione delle iPSC derivate da PDAC può ricapitolare la progressione di PDAC dal suo precursore precoce della neoplasia intraepiteliale pancreatica (PanIN), rivelando i cambiamenti molecolari e cellulari che si verificano precocemente durante la progressione di PDAC. Pertanto, le iPSC derivate da PDAC possono essere utilizzate per modellare le prime fasi di PDAC per la scoperta di marcatori diagnostici di diagnosi precoce. Ciò è particolarmente importante per i pazienti con PDAC, che in genere vengono diagnosticati nelle fasi metastatiche tardive a causa della mancanza di biomarcatori affidabili per le fasi iniziali di PanIN. Tuttavia, la riprogrammazione delle linee cellulari tumorali, incluso il PDAC, in pluripotenza rimane impegnativa, laboriosa e altamente variabile tra le diverse linee. Qui, descriviamo un protocollo più coerente per la generazione di iPSC da varie linee cellulari PDAC umane utilizzando vettori lentivirali bicistronici. Le linee di iPSC risultanti sono stabili e non mostrano alcuna dipendenza dall'espressione esogena di fattori di riprogrammazione o farmaci inducibili. Nel complesso, questo protocollo facilita la generazione di un'ampia gamma di iPSC derivate da PDAC, che è essenziale per scoprire biomarcatori precoci che sono più specifici e rappresentativi dei casi di PDAC.
Introduzione
L'adenocarcinoma duttale pancreatico (PDAC) è una delle neoplasie maligne più fatali e la diagnosi precoce rimane difficile a causa della natura asintomatica della malattia. La maggior parte dei pazienti affetti da PDAC viene diagnosticata in fase metastatica avanzata, quando sono disponibili opzioni terapeutiche molto limitate 1,2. Ciò è dovuto principalmente alla mancanza di biomarcatori affidabili per le fasi iniziali, come quelli che potrebbero essere comodamente rilevati come proteine rilasciate nel flusso sanguigno.
Il PDAC può diffondersi molto presto durante la sua progressione e una prognosi migliore è stata collegata alla diagnosi precoce del cancro quando il PDAC è localizzato nel pancreas3. Tuttavia, a meno di un decimo dei pazienti con PDAC viene diagnosticata una prognosi favorevole, consentendo la resezione chirurgica. Tuttavia, i pochi con tumori resecabili sono anche inclini alla recidiva del tumore entro 12 mesi4.
Negli ultimi cinquant'anni sono stati apportati notevoli miglioramenti alle tecniche chirurgiche, alla cura del paziente e alle modalità di trattamento 5,6. Tuttavia, il tasso di sopravvivenza a 5 anni nei pazienti PDAC resecati chirurgicamente è salito a malapena al 17%. Ciononostante, questo dato è ancora migliore di quello dei pazienti non resecati, che è rimasto pressoché invariato (0,9%)4,7. La chemioterapia è l'unico altro trattamento alternativo per la PDAC. Tuttavia, questa opzione è molto limitata in quanto la grande maggioranza dei pazienti con PDAC mostra una forte resistenza ai farmaci chemioterapici come la gemcitabina 7,8. Altri farmaci, come l'erlotinib, sono disponibili solo per un piccolo gruppo di pazienti affetti da PDAC con mutazioni specifiche, la maggior parte dei quali mostra resistenza all'erlotinib9. Gli effetti collaterali avversi associati alla chemioterapia nella maggior parte dei pazienti con PDAC sono un altro svantaggio di questo trattamento10. Recentemente, strategie promettenti hanno dimostrato che gli inibitori del checkpoint immunitario (ICI) e gli inibitori delle chinasi a piccole molecole (SMKI) possono essere efficaci nel trattamento del PDAC, ma le risposte durature a queste terapie mirate rimangono limitate a una minoranza di pazienti11,12. Nel complesso, la scoperta di biomarcatori precoci specifici per PDAC può aprire nuove strade per la diagnosi e il trattamento precoci.
La PDAC si sviluppa da lesioni precursori delle neoplasie intraepiteliali pancreatiche (PanIN) che derivano da proliferazioni epiteliali del dotto pancreatico non invasive13,14. Mentre la formazione di PanIN è iniziata da mutazioni oncogeniche come KRAS, sono necessarie ulteriori alterazioni genetiche ed epigenetiche per la progressione verso PDAC. È stato previsto che la progressione di PanIN attraverso i diversi stadi in PDAC invasivo richieda circa 10 anni 13,15,16,17. Questo lasso di tempo offre una grande opportunità per trarre vantaggio dalla diagnosi precoce del PDAC. Pertanto, sono state condotte ricerche approfondite per stabilire modelli animali di xenotrapianto tumorale e colture di organoidi per studiare la progressionedel PDAC 18,19,20,21. Questi modelli sono stati molto utili per studiare gli stadi invasivi del PDAC, anche se non la transizione dalle prime fasi di PanIN. È quindi importante sviluppare modelli sperimentali in grado di ricapitolare la progressione precoce degli stadi di PanIN per consentire la scoperta di biomarcatori di diagnosi precoce.
La riprogrammazione delle cellule somatiche in cellule staminali pluripotenti indotte (iPSC) utilizzando i quattro fattori di trascrizione OCT4, SOX2, KLF4 e c-MYC (OSKM) ha illustrato l'estensione della plasticità cellulare22. La plasticità delle cellule tumorali è stata ben documentata e la riprogrammazione delle cellule tumorali umane in iPSC è stata utilizzata con successo per riportare le cellule al loro stato cellulare originale, rimuovendo molti degli insulti epigenetici che si sono accumulati durante la progressione del cancro 23,24,25,26,27,28,29. La possibilità di utilizzare questa strategia di riprogrammazione per manipolare l'identità delle cellule tumorali ha, quindi, presentato una grande promessa nel trattamento del cancro30,31. Infatti, abbiamo precedentemente dimostrato che la differenziazione delle iPSC derivate dai PDAC può ricapitolare la progressione del PDAC attraverso i primi stadi PanIN32. Identificando i geni e i percorsi specifici per gli stadi preco-intermedi del PDAC, sono stati identificati biomarcatori candidati che possono essere utilizzati clinicamente per la diagnosi precoce del PDAC32,33. Tuttavia, i biomarcatori scoperti utilizzando una singola linea di iPSC hanno mostrato una copertura limitata nella maggior parte dei pazienti con PDAC32. Le sfide legate alla generazione di linee di iPSC da altri pazienti affetti da PDAC hanno interrotto la capacità di scoprire biomarcatori più affidabili. Ciò è dovuto a molti fattori tecnici, tra cui l'eterogeneità della somministrazione di OSKM, poiché solo una piccola porzione di cellule PDAC primarie umane conteneva tutti e quattro i fattori e rispondeva con successo alla riprogrammazione. Qui, viene presentato un protocollo dettagliato per la riprogrammazione delle cellule PDAC primarie utilizzando una doppia somministrazione lentivirale più efficiente e coerente di OSKM.
Protocollo
Tutti i protocolli sperimentali sono stati approvati dall'Institutional Review Board dell'OHSU. Tutti i metodi sono stati eseguiti in conformità con le linee guida e i regolamenti pertinenti. Tutti i lavori sugli animali per i tumori PDX sono stati eseguiti con l'approvazione del Comitato istituzionale per l'uso e la cura degli animali (IACUC) dell'OHSU. Questo protocollo è stato testato in cellule PDAC primarie da xenotrapianto derivato da paziente (PDX), linea cellulare BxPc3 che presenta morfologia epiteliale isolata dal tessuto pancreatico di una paziente di 61 anni con adenocarcinoma, la linea cellulare epiteliale immortalizzata H6C7 derivata da un normale epitelio del dotto pancreatico umano e fibroblasti umani primari derivati da biopsia cutanea di individui sani. I campioni umani di PDAC sono stati ottenuti nell'ambito dello studio Oregon Pancreas Tissue Registry (IRB00003609). Il consenso informato è stato ottenuto da tutti i soggetti. I fibroblasti primari umani sono stati derivati in RBiomedical, Edimburgo, Regno Unito, da campioni di pelle di donatori anonimi sottoposti a chirurgia di routine presso l'Edinburgh Royal Infirmary, Little France, Regno Unito, con il loro consenso e approvazione etica (09/MRE00/91). Tutto il lavoro sui lentivirus è stato svolto nell'ambito dell'attività di ricerca di Classe 2 (GM207/16.6) e approvato dal Dipartimento di Salute e Sicurezza dell'Università di Edimburgo e notificato all'autorità competente HSE del governo scozzese. Tutti gli esperimenti di riprogrammazione utilizzando cellule pancreatiche umane sono stati condotti sotto l'approvazione etica del comitato etico della School of Biological Sciences dell'Università di Edimburgo (riferimento # asoufi-0002).
1. Preparazione dei lentivirus
- Per la preparazione dei lentivirus, preparare plasmidi di confezionamento ed espressione di alta qualità (privi di endotossine) con concentrazioni comprese tra 1-2 μg/μL tra cui psPAX2, pMDG34 e due vettori bicistronici contenenti RES; la codifica di pSIN4-EF1a-O2S per l'espressione di OCT4 e SOX2 guidata dal promotore EF-1α, e pSIN4-CMV-K2M per l'espressione di KLF4 e c-MYC sotto l'enhancer/promoter CMV35 (vedi Tabella dei Materiali). Inoltre, preparare il plasmide pWPT-GFP da utilizzare come controllo della trasfezione.
- Scongelare la linea cellulare di rene embrionale umano (293T) e la coltura nel terreno minimo essenziale di Glasgow (GMEM), integrata con il 10% di siero fetale di vitello (FCS), 1x aminoacidi non essenziali, 1 mM di piruvato di sodio e 1 mM di glutammina a (vedi tabella dei materiali) 37 °C e 5% di CO2 .
NOTA: Si consiglia di utilizzare celle 293T entro quattro passaggi dopo lo scongelamento. - Seme 293T ad una densità di 3 milioni di cellule per piatto da 15 cm, 24 ore prima della trasfezione. Sono necessari un totale di tre piatti. È preferibile seminare le cellule nel tardo pomeriggio ~ 16:00.
- Il giorno successivo (~16:00), quando le cellule raggiungono ~40%-50% di confluenza, preparare quanto segue per tre reazioni di trasfezione:
NOTA: Tenere sempre conto dell'errore di pipettaggio aggiungendo il 10% del volume in più.- Etichettare tre provette di plastica da 15 mL con il nome del lentivirus appropriato (pSIN4-EF1a-O2S, pSIN4-CMV-K2M e controllo pwPT-GFP). Aggiungere 1,710 mL di terreno sierico ridotto (vedere la tabella dei materiali) in ciascuna provetta.
- Diluire il reagente di trasfezione 90 μL (vedere Tabella dei materiali) in terreno di coltura ridotto da 1,710 mL, mescolare per vertice, per 2 s e incubare a temperatura ambiente per 5 min.
- Mescolare i vettori di imballaggio; 5,1 μg di psPAX2 e 2,4 μg di pMDG (7,5 μg totali).
- Aggiungere la miscela vettoriale di imballaggio al mezzo di trasfezione (dal punto 1.4.2) e vorticare per 2 s.
- Aggiungere 7,5 μg di ciascun vettore di riprogrammazione: pSIN4-EF1a-O2S e pSIN4-CMV-K2M e il controllo pwPT-GFP alla miscela di trasfezione dal passo (1.4.4) e al vortice per 2 s.
NOTA: Utilizzare l'espressione vector: vettore virale con un rapporto 1:1.
- Incubare le provette di trasfezione per 15 minuti a temperatura ambiente.
- Trasfettare le cellule 293T con ciascun lentivirus aggiungendo direttamente la miscela di trasfezione-DNA del passaggio (1.4.5) al terreno in modo goccia a goccia. Agitare il piatto per garantire una distribuzione uniforme su tutta la superficie.
- Incubare le cellule trasfettate in un incubatore a 37 °C, 5% di CO2 per una notte.
NOTA: Utilizzare rifiuti specifici per virus, un secchio metallico per lo smaltimento, un doppio sacchetto con sacchetti per autoclave e prendere un contenitore per rifiuti liquidi per virus e aggiungere una compressa disinfettante. Smaltire tutte le pipette, i puntali e le provette in un secchio di metallo. Smaltire i vecchi supporti in un contenitore per rifiuti liquido virale. Evitare l'uso di pipette e vetreria in vetro per eliminare la contaminazione accidentale da virus. - Dopo 14-16 ore dalla trasfezione, sostituire il terreno con 30 mL di terreno fresco 293T.
- Incubare le cellule trasfettate a 37 °C, 5% di CO2 per 60-72 ore dopo il cambio del terreno. Osservare quotidianamente le cellule e controllare l'efficienza di trasfezione mediante fluorescenza GFP.
NOTA: Idealmente, l'efficienza di trasfezione dovrebbe essere >90% da GFP. Per gli altri virus, si dovrebbero osservare chiari cambiamenti morfologici delle cellule 293T, poiché tendono a diventare più rotonde quando producono particelle virali. - Raccogliere i terreni da ciascuna coltura di trasfezione virale in provette da 50 mL e centrifugarli per eliminare i detriti cellulari a 1932 x g per 10 minuti a 4 °C.
- Filtrare ciascun surnatante lentivirus attraverso un filtro a siringa da 0,45 μM per rimuovere i detriti più piccoli e raccoglierli in nuove provette da 50 mL.
- Dividere ogni surnatante lentivirus in aliquote da 6 mL e congelare a scatto ciascuno in azoto liquido.
- Conservare le aliquote di lentivirus a -80 °C fino al momento dell'uso.
2. Riprogrammazione della trasduzione dei lentivirus
- Scongelare le cellule PDAC primarie e la coltura in terreno libero da siero di cheratinociti (KSFM) completamente definito, integrato con estratto ipofisario bovino (BPE), fattore di crescita epidermico (EGF) ricombinante umano a 5 ng/mL e tossina del colera a 50 ng/mL in un'incubatrice a 37 °C, 5% CO2 e 5% O2 (ipossia) (vedere Tabella dei materiali).
- Scongelare BxPc3, una linea cellulare di adenocarcinoma duttale pancreatico squamoso, e coltura in terreno RPMI 1640 integrato con il 10% di siero fetale di vitello (FCS) a 37 °C e il 5% di CO2 .
- Scongelare le cellule H6C7, le cellule epiteliali duttali pancreatiche e la coltura in KSFM integrata con BPE ed EGF a 5 ng/mL, a 37 °C e 5% di CO2 .
- Scongelare il fibroblasto umano (hFib) e la coltura nel terreno minimo essenziale di Glasgow (GMEM), integrato con il 10% di FCS, 1x aminoacidi non essenziali, 1 mM di piruvato di sodio e 1 mM di glutammina e 0,05 mM (finale) di beta-mercaptoetanolo a 37 °C e 5% di CO2 .
- Un giorno prima della trasduzione del lentivirus (preferibilmente nel tardo pomeriggio), preparare due pozzetti di una piastra a 6 pozzetti contenente 100.000 cellule per pozzetto di ciascuna delle cellule PDAC, BxPc3, H6C7 e hFib. Infettare un pozzetto con i lentivirus OSKM e utilizzare il secondo come controllo non infetto.
NOTA: Utilizzare una piastra separata per ciascun tipo di cella. - Il giorno seguente, nel pomeriggio (circa 24 ore dopo), assicurarsi che la confluenza cellulare raggiunga almeno il 70% prima di procedere alla successiva fase di infezione da lentivirus.
3. Trasduzione lentivirale del PDAC
- Scongelare 5 mL di ciascun surnatante lentivirus in un incubatore virale a 37 °C.
- Preparare due provette da 15 mL, ciascuna contenente 2 mL di terreno di coltura PDAC preriscaldato.
- Alla prima provetta, aggiungere 6 mL di ciascun lentivirus di riprogrammazione (pSIN4-CMV-K2M e pSIN4-EF1a-O2S) e 12 μL di poliferene 4,5 mg/mL (4,5 μg/mL finali, vedere la tabella dei materiali) e mescolare.
- Alla seconda provetta, aggiungere 2 μL di polibrone 4,5 mg/mL (4,5 μg/mL finali).
- Scartare il terreno da ogni pozzetto e lavare una volta con PBS a temperatura ambiente.
- Aggiungere il terreno di infezione di riprogrammazione (provetta 1) al primo pozzetto.
- Aggiungere la miscela della provetta 2 nel secondo pozzetto. Questo sarà il controllo non infetto.
- Incubare le cellule PDAC in un incubatore a 37 °C, 5% CO2 e 5% O2 (ipossia) per una notte.
- Il giorno successivo, nel pomeriggio, scartare il terreno da entrambi i pozzetti e sostituirlo con terreno di coltura PDAC fresco.
- Incubare le cellule a 37 °C, 5% di CO2 e 5% di O2 (ipossia) per 48 ore.
4. Trasduzione lentivirale delle cellule BxPc3
- Scongelare 3 mL di ciascun surnatante lentivirus in un incubatore virale a 37 °C.
- Preparare due provette da 15 mL, ciascuna contenente 2 mL di terreno di coltura BxPc3.
- Alla prima provetta, aggiungere 3 mL di ciascun lentivirus di riprogrammazione (pSIN4-CMV-K2M e pSIN4-EF1a-O2S) e 6 μL di porlorene 4,5 mg/mL (4,5 μg/mL finali) e mescolare.
- Alla seconda provetta, aggiungere 2 μL di polibrone 4,5 mg/mL (4,5 μg/mL finali).
- Scartare il terreno da ogni pozzetto e lavare una volta con PBS.
- Aggiungere il terreno di infezione di riprogrammazione (provetta 1) al primo pozzetto.
- Aggiungere la miscela della provetta 2 nel secondo pozzetto. Questo sarà il controllo non infetto.
- Incubare le cellule BxPc3 infette a 37 °C per il virus e al 5% di CO2 durante la notte.
- Il giorno successivo, nel pomeriggio, scartare il terreno da entrambi i pozzetti e sostituirlo con terreno di coltura BxPc3 fresco.
- Incubare le cellule a 37 °C e 5% di CO2 per 48 ore.
5. Trasduzione lentivirale dell'infezione da cellule H6c7
- Scongelare 4 mL di ciascun surnatante lentivirale in un incubatore virale a 37 °C.
- Preparare due provette da 15 mL, ciascuna contenente 2 mL di terreno di coltura H6c7.
- Alla prima provetta, aggiungere 4 mL di ciascun lentivirus di riprogrammazione (pSIN4-CMV-K2M e pSIN4-EF1a-O2S) e 8 μL di polibrone 4,5 mg/mL (4,5 μg/mL finali) e mescolare.
- Alla seconda provetta, aggiungere 2 μL di polibrone 4,5 mg/mL (4,5 μg/mL finali).
- Scartare il terreno da entrambi i pozzetti e lavare una volta con PBS.
- Aggiungere il terreno di infezione di riprogrammazione (provetta 1) al primo pozzetto.
- Aggiungere la miscela della provetta 2 nel secondo pozzetto. Questo sarà il controllo non infetto.
- Incubare le cellule H6C7 infette a 37 °C per il virus e al 5% di CO2 durante la notte.
- Il giorno successivo, nel pomeriggio, scartare il terreno da entrambi i pozzetti e sostituirlo con un terreno di coltura H6c7 fresco.
- Incubare le cellule a 37 °C e 5% di CO2 per 48 ore.
6. Trasduzione lentivirale delle cellule hFib
- Scongelare 2 mL di ciascun surnatante lentivirus in un incubatore virale a 37 °C.
- Preparare due provette da 15 mL, ciascuna contenente 2 mL di terreno di coltura hFib.
- Alla prima provetta, aggiungere 2 mL di ciascun virus di riprogrammazione (pSIN4-CMV-K2M e pSIN4-EF1a-O2S) e aggiungere 4 μL di porlorene 4,5 mg/mL (4,5 μg/mL finali) e mescolare.
- Alla seconda provetta, aggiungere 2 μL di polibrone 4,5 mg/mL (4,5 μg/mL finali) e mescolare.
- Scartare il terreno da ogni pozzetto e lavare una volta con PBS.
- Aggiungere il terreno di infezione di riprogrammazione (provetta 1) al primo pozzetto.
- Aggiungere la miscela della provetta 2 nel secondo pozzetto. Questo sarà il controllo non infetto.
- Incubare le cellule hFib infette a 37 °C e 5% di CO2 per una notte.
- Il giorno successivo, nel pomeriggio, scartare il terreno da entrambi i pozzi e sostituirlo con terreno di coltura hFib fresco.
- Incubare le cellule a 37 °C e 5% di CO2 per altre 48 ore.
NOTA: Per un'efficiente trasduzione dei lentivirus, titolare attentamente la quantità di lentivirus, poiché varia notevolmente tra i diversi tipi di cellule. Per alcuni tipi di cellule possono essere necessarie trasduzioni multiple di lentivirus. Le linee cellulari PDAC richiedono fino a tre dosi, mentre una dose è stata sufficiente per riprogrammare le linee BxPc3, H6C7 e hFib.
7. Preparazione delle celle di alimentazione iMEF
- Preparare 40 mL di soluzione di gelatina allo 0,2% diluendo la soluzione madre di gelatina all'1% con PBS.
- Rivestire quattro piastre a 6 pozzetti coprendo ogni pozzetto con 2 mL di soluzione di gelatina allo 0,2%.
- Incubare le piastre ricoperte di gelatina a 37 °C e 5% di CO2 per almeno 30 min.
- Aspirare la soluzione di gelatina in eccesso e lasciare asciugare le piastre sotto la cappa di flusso.
- Scongelare due fiale (~ 4 milioni di cellule per flaconcino) di fibroblasti embrionali di topo irradiati (iMEF) agitandoli in un bagno d'acqua a 37 °C.
NOTA: Per ridurre la possibilità di contaminazione, fare attenzione a evitare che l'acqua schizzi vicino all'apertura del tappo. Asciugare il flaconcino con un tovagliolo di carta e spruzzarlo con etanolo al 70%. - Nella cappa di coltura tissutale, pipettare il contenuto di ciascun flaconcino di iMEFs scongelato in una provetta da 15 mL contenente 10 mL di terreno di coltura hFib preriscaldato goccia per goccia e mescolare bene capovolgendo delicatamente la provetta.
- Centrifugare la sospensione cellulare a 309 x g per 3 minuti a temperatura ambiente e rimuovere il surnatante da ciascuna provetta. Risospendere ogni pellet cellulare in 12 mL di terreno hFib.
- Piastra 2 mL di sospensione cellulare per pozzetto delle piastre a 6 pozzetti rivestite di gelatina. Distribuire uniformemente le cellule scuotendo delicatamente le piastre in tutte le direzioni.
- Incubare le piastre a 37 °C e 5% di CO2 per una notte.
NOTA: Preparare nuove piastre iMEF 24 ore prima di utilizzarle per l'esperimento di riprogrammazione.
8. Trasferimento delle cellule infette sullo strato di alimentazione iMEF
- Scartare i terreni dalle cellule PDAC, BxPc3, H6c7 e hFib infette e non infette. Lavare ogni pozzetto due volte con PBS a temperatura ambiente.
- Dissociare le cellule dalla piastra aggiungendo 0,5 mL di tripsina a ciascun pozzetto. Incubare le cellule PDAC in un incubatore a 37 °C, 5% CO2 e 5% O2 (ipossia) per 15 minuti.
- Incubare le cellule BxPc3 a 37 °C e 5% di CO2 per 5 min.
- Incubare le cellule H6c7 e hFib a 37 °C e 5% di CO2 per 4 minuti.
- Trasferire la sospensione cellulare dissociata da ciascun pozzetto in provette da 15 mL etichettate di conseguenza come infette o non infette.
- Prelevare le cellule mediante centrifugazione come segue: per PDAC, far girare la sospensione cellulare a 300 x g per 5 minuti; per BxPc3, far girare la sospensione cellulare a 120 x g per 5 min; per H6C7, far girare la sospensione cellulare a 112 x g per 4 minuti; per hFib, far girare la sospensione cellulare a 161 x g per 3 min. Eseguire tutte le centrifugazioni a temperatura ambiente.
- Risospendere ogni tipo di pellet cellulare in 1 mL del terreno di coltura appropriato.
- Lavare due volte le piastre iMEF preparate nella fase 7 con ciascun terreno di coltura per tipo di cellula. cioè, lavare una lastra con un mezzo PDAC, una con un mezzo BxPc3, una con un mezzo H6C7 e l'altra con un mezzo hFib.
- Piastra 50.000 cellule infette per pozzetto in cinque pozzetti di piastra iMEF a 6 pozzetti. Piastra 50.000 cellule di controllo non infette nel restante pozzetto della piastra a 6 pozzetti iMEF.
NOTA: Ottimizzare il numero di cellule infette da placcare da 1.000 a 50.000 cellule per pozzetto. - Incubare le cellule PDAC a 37 °C, 5% di CO2 e 5% di O2 (ipossia) durante la notte.
- Incubare le cellule BxPc3, H6C7 e hFib a 37 °C e 5% di CO2 per una notte.
- Il giorno successivo, preparare il terreno di riprogrammazione come segue: aggiungere 100 mL di siero sostitutivo knockout (25% finale), 5 mL di glutammina 200 mM (1 mM finale), 5 mL di 100 amminoacidi non essenziali (100 μM finali), 1,5 mL di Beta-mercaptoetanolo (0,1 mM finale) a 400 mL di Modified Eagle Medium (DMEM).
- Conservare i terreni di riprogrammazione in aliquote da 100 mL a 4 °C per un massimo di 4 settimane o a -20 °C più a lungo.
- Al pronto per l'uso, aggiungere 100 μL di 10 mg/mL di fattore di crescita basico dei fibroblasti (bFGF) (10 ng/mL finale) a un'aliquota di terreno di riprogrammazione da 100 mL.
- Riscaldare il mezzo di riprogrammazione a bagnomaria a 37 °C.
- Lavare due volte le cellule che crescono sui feeder iMEFs con terreni di riprogrammazione preriscaldati.
Aggiungere 2 mL di terreno di riprogrammazione a ciascun pozzetto. - Trasferire le piastre di riprogrammazione in un incubatore a 37 °C, 5% CO2e 3% O2 (ipossia).
NOTA: Il giorno in cui viene aggiunto il supporto di riprogrammazione è considerato il giorno 1 della riprogrammazione. - Nutrire le cellule ogni giorno con terreni di riprogrammazione freschi fino a quando non iniziano a comparire le colonie di iPSC.
NOTA: Identificare le colonie di iPSC in base alla loro morfologia simile alle ESC (colonie compatte con bordi ben definiti e composte da cellule con un elevato rapporto nucleo/citoplasma). - Monitorare quotidianamente le colonie di iPSC e, dopo averne formato un numero adeguato, farle passare come un pool.
NOTA: Assicurarsi che le colonie iPSC non si tocchino, poiché ciò comporterebbe una differenziazione e influirebbe sulla loro stabilità a lungo termine. - Per stabilire le linee clonali, far passare il pool di iPSC circa 5 volte, quindi scegliere colonie robuste che mantengano la loro morfologia simile a quella delle ESC.
- Per identificare le colonie di iPSC completamente riprogrammate, eseguire la colorazione viva con TRA-160 (vedere la tabella dei materiali) e raccogliere l'RNA dalle colonie di iPSC per la caratterizzazione dell'espressione genica.
9. Passaggio di colonie di iPSC
- Preparare una quantità sufficiente di piastre di alimentazione iMEF 24 ore prima di passare le colonie di iPSC come descritto al punto 7.
- Lavare due volte le piastre di alimentazione iMEF con un mezzo di riprogrammazione preriscaldato.
- Aggiungere 2 mL di terreno di riprogrammazione integrato con l'inibitore ROCK (Y2) (10 μM) a ciascun pozzetto della piastra iMEF.
- Incubare le piastre iMEF in un incubatore a 37 °C, 5% CO2 e 3% O2 fino al momento dell'uso.
- Preparare una soluzione salina tamponata con acido etilendiamminotetraacetico/fosfato (EDTA/PBS) diluendo 0,5 M EDTA 1:1000 in PBS (0,5 mM finale).
- Sostituire il terreno di coltura di riprogrammazione con 0,5 mL di EDTA/PBS in ciascun pozzetto.
- Incubare in un incubatore a 37 °C, 5% CO2 e 3% O2 o a temperatura ambiente, a seconda del tipo di cellula. Le iPSC BxPc3 e PDAC richiedono un'incubazione di 15 minuti a 37 °C. Le iPSC H6C7 e hFib devono essere incubate a temperatura ambiente per 5 minuti.
- Controllare le cellule delle iPSCs al microscopio fino a quando non iniziano a dissociarsi uniformemente dallo strato di alimentazione in tutta la colonia.
- Raccogliere la sospensione di cellule iPSC dissociate in una provetta da centrifuga da 15 mL. Centrifugare a 300 x g per 5 minuti a temperatura ambiente.
- Risospendere il pellet cellulare in 1 mL di terreno di riprogrammazione integrato con Y2 (10 μM).
- Placcare le cellule sullo strato di alimentazione iMEF trasferendo ogni 1 mL di sospensione cellulare iPSC in tre pozzetti della piastra iMEF.
NOTA: Il rapporto di divisione può variare a seconda del numero di colonie iPSC raccolte. - Incubare in un incubatore a 37 °C, 5% CO2 e 3% O2 .
10. Colorazione viva di colonie di iPSC con TRA-1-60
- Preparare 0,5 mL per pozzetto di anticorpo TRA-1-60 da 4 μg/mL in terreni di riprogrammazione.
- Scartare il terreno di riprogrammazione e sostituirlo con una miscela di anticorpi da 0,5 mL in ciascun pozzetto. Incubare in un incubatore a 37 °C, 5% CO2 e 3% O2 per 30 min.
- Lavare le celle due volte con il terreno di riprogrammazione.
- Acquisizione di immagini a fluorescenza con un sistema di imaging cellulare utilizzando il filtro GFP.
- Controllare la qualità dell'immagine considerando il canale di controllo negativo.
Risultati
Nella Figura 1 sono mostrate immagini rappresentative che mostrano la morfologia delle colonie di iPSC derivate da cellule PDAC, BXPc3, H6C7 e hFib. Le colonie PDAC-iPSC hanno iniziato a formarsi il giorno 25 della riprogrammazione. Colonie di iPSC robuste con una morfologia più consolidata simile a quella delle ESC sono state identificate al 40° giorno di riprogrammazione (Figura 1). Allo stesso modo, la formazione di BxPc3-iPSC è iniziata il giorno 23 ed è diventata più consolidata il giorno 35. La formazione di H6C7-iPSC era simile a quella delle PDAC-iPSC e ha iniziato a stabilirsi il giorno 45. Le colonie di hFib-iPSC hanno iniziato a formarsi il giorno 15 della riprogrammazione.
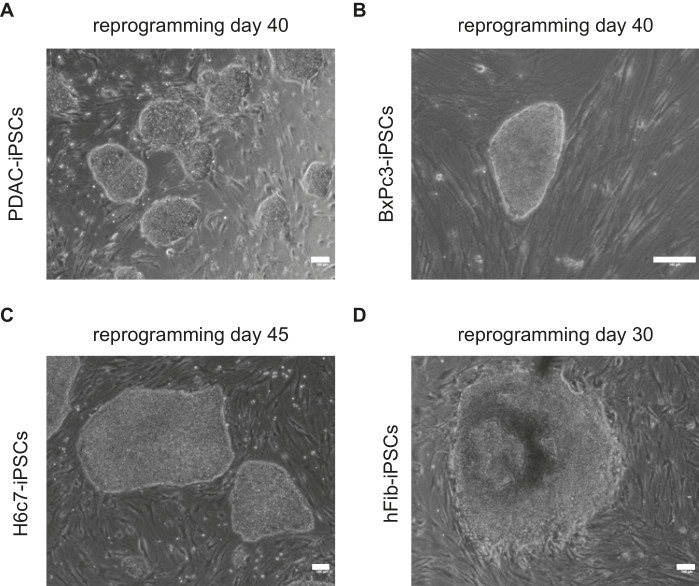
Figura 1: Immagini rappresentative delle colonie di iPSC. Le immagini mostrano una morfologia simile a hESC derivata da (A) PDAC, (B) BxPc3, (C) H6c7 e (D) hFib. I giorni di riprogrammazione sono indicati sopra ogni immagine. Barre di scala = 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Le linee clonali di iPSC sono state stabilite da ogni riprogrammazione di cellule PDAC, BxPc3, H6C7 e hFib. Tutte le linee di iPSC consolidate sono risultate positive per TRA-1-60, un marcatore indifferenziato della superficie cellulare hESC, confermando la loro riprogrammazione alla pluripotenza (Figura 2).

Figura 2: Immagini rappresentative di linee clonali di iPSC. Le immagini sono derivate da (A) PDAC, (B) BxPc3, (C) H6c7 e (D) hFib che mostrano una morfologia simile a ESC (pannelli superiori) e una colorazione positiva per TRA-1-60 (pannello inferiore). Barre di scala = 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Per facilitare l'uso della riprogrammazione delle iPSC per lo studio della progressione del cancro, è stato stabilito un robusto protocollo per la riprogrammazione delle cellule tumorali del pancreas. La riprogrammazione delle cellule tumorali in pluripotenza si è dimostrata finora molto impegnativa, poiché solo pochi studi hanno generato con successo iPSC da cellule tumorali 32,36,37,38,39,40,41,42,43,44,45,46. La maggior parte di questi studi ha utilizzato linee cellulari tumorali immortalizzate per generare linee iPSC, non cellule primarie derivate da pazienti 36,37,38,40,42,43,44,46. Ad esempio, è stata tentata la riprogrammazione di quattro diverse linee cellulari di cancro al fegato utilizzando l'introduzione retrovirale di fattori OSKM, ma solo una singola linea cellulare è stata riprogrammata con successo41. Tuttavia, la linea iPSC generata per il cancro del fegato ha mostrato una perdita di staminalità dopo pochi passaggi, evidenziando l'elevata resistenza delle cellule tumorali alla riprogrammazione OSKM41. In precedenza, sono stati fatti tentativi di riprogrammare le cellule PDAC utilizzando la somministrazione lentivirale di OSKM individualmente; tuttavia, è stata generata una sola linea iPSC, dipendente dall'espressione esogena di OSKM e quindi non completamente riprogrammata37. Un altro studio ha utilizzato vettori episomiali per fornire fattori OSKM senza integrazione genomica, ma è riuscito a generare solo un singolo clone di iPSC da PDAC39. Il limitato successo nella riprogrammazione delle cellule tumorali si aggiunge alle molte sfide che ostacolano l'uso della tecnologia iPSC nella ricerca sul cancro.
Qui, viene spiegata la generazione di iPSC da campioni primari di PDAC derivati da due diversi pazienti e da una linea cellulare PDAC consolidata (BxPc3). Inoltre, le iPSC sono state generate anche da H6c7, una linea cellulare epiteliale duttale pancreatica. Per quanto ne sappiamo, questo è il primo rapporto di linee iPSC stabili derivate con successo da BxPc3 e H6c7. Seguendo questo protocollo, le iPSC sono state generate con successo anche da fibroblasti umani primari derivati da individui sani, ampliando l'applicabilità del metodo oltre la ricerca sul cancro.
Uno degli elementi chiave alla base del successo del protocollo è l'uso di vettori lentivirali bicistronici per co-esprimere i fattori OS e KM. Questi vettori contengono il sito di ingresso del ribosoma interno 2 (IRES2) per esprimere OCT4 e SOX2 in un vettore e KLF4 e cMYC nell'altro. Diversi studi che utilizzano vettori monocistronici in cui ogni fattore di riprogrammazione è stato fornito individualmente hanno dimostrato che l'assorbimento di ciascun vettore è diverso, influenzando la stechiometria OSKM e l'efficienza di riprogrammazione47. L'uso di vettori bicistronici può aiutare a mitigare questo problema. Inoltre, l'utilizzo di IRES in vettori lentivirali ha mostrato un significativo aumento dell'efficienza di riprogrammazione48. In questo sistema lentivirale, l'espressione di OS è guidata dal promotore EF1a, mentre l'espressione di KM è sotto il controllo di un promotore di CMV. È noto che il promotore del CMV può essere sottoposto a silenziamento altamente efficiente mediante metilazione del DNA e deacetilazione degli istoni, a differenza di EF1a49,50. Pertanto, il silenziamento precoce di KM prima di OS durante la riprogrammazione può essere un fattore chiave per il successo del protocollo. Ciò è coerente con studi precedenti che mostrano l'importanza delle dinamiche di espressione di OSKM durante la riprogrammazione 51,52,53,54,55. Pertanto, il vettore bicistronico è anche più vantaggioso del vettore policistronico, in cui tutti i fattori OSKM sono espressi da un singolo promotore56. Altri fattori che contribuiscono al successo del protocollo includono le dosi di infezione da lentivirus e il numero di cellule infette utilizzate per la riprogrammazione, che sono state personalizzate per ogni tipo di cellula.
In sintesi, viene presentato un metodo ottimizzato per la riprogrammazione delle cellule PDAC primarie insieme ad altre linee cellulari e cellule normali. Questo metodo aiuterà ad espandere l'uso della riprogrammazione delle iPSC per modellare la progressione del cancro e, in questo caso, scoprire i primi biomarcatori PDAC.
Divulgazioni
Gli autori dichiarano di non avere alcun conflitto di interessi.
Riconoscimenti
A.S e J.K desiderano ringraziare Cancer Research UK e OHSU per il finanziamento (CRUK-OHSU Project Award C65925/A26986). A.S è supportato da un premio per lo sviluppo della carriera MRC (MR/N024028/1). A.A è finanziato da una borsa di dottorato di ricerca (Scholarship ref. 1078107040) della King Abdulaziz City for Science and Technology. J.K è finanziato da MRF New Investigator Grant (GCNCR1042A) e Knight CEDAR grant (68182-933-000, 68182-939-000). Ringraziamo il Prof. Keisuke Kaji per averci gentilmente fornito il vettore di riprogrammazione pSIN4-EF1a-O2S e pSIN4-CMV-K2M. Ai fini dell'accesso aperto, l'autore ha applicato una licenza Creative Commons Attribution (CC BY) a qualsiasi versione del manoscritto accettato dall'autore derivante da questa presentazione.
Materiali
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol (50 mM) | Thermo Fisher | 31350010 | |
| Alexa Fluor 488 anti- human TRA-1-60-R | BioLegend | 330613 | |
| Bovine Pituitary Extract (BPE) | Thermo Fisher | 13028014 | |
| BxPc3 | ATCC | CRL-1687 | |
| Cholera Toxin from Vibrio cholerae | Merck | C8052-1MG | |
| Collagen, Type I solution from rat tail | Merck | C3867 | |
| Completed Defined K-SFM | Thermo Fisher | 10744-019 | |
| Corning Costar TC-Treated Multiple Well Plates | Merck | CLS3516 | |
| Corning syringe filters | Merck | CLS431231 | |
| Corning tissue-culture treated culture dishes | Merck | CLS430599 | |
| Day Impex Virkon Disinfectant Virucidal Tablets | Thermo Fisher | 12328667 | |
| Dulbecco′s Phosphate Buffered Saline (PBS) | Merck | D8537 | |
| Fetal Calf Serum (FCS) | Thermo Fisher | 10270-106 | |
| Fugene HD Transfection Reagent | Promega | E2312 | |
| Gelatin solution, Type B, 2% in H2O | Merck | G1393-100ML | |
| Glasgow Minimum Essential Media (GMEM) | Merck | G5154 | |
| Human EGF Recombinant Protein | Thermo Fisher | PHG0311 | |
| Human FGF-basic (FGF-2/bFGF) (154 aa) Recombinant Protein, PeproTech | Thermo Fisher | 100-18B | |
| Human Pancreatic Duct Epithelial Cell Line (H6c7) | Kerafast | ECA001-FP | |
| iMEF feeder cells | iXcells Biotechnologies | 10MU-001-1V | |
| Keratinocyte Serum Free Media (KSFM) | Thermo Fisher | 17005-042 | |
| KnockOut DMEM | Thermo Fisher | 10829018 | |
| KnockOut serum Replacement | Thermo Fisher | 10828028 | |
| L-Glutamine (200 mM) | Thermo Fisher | 25030-024 | |
| MEM Non-Essential Amino Acids Solution (100x) | Thermo Fisher | 11140050 | |
| Millex-HP 0.45 μM syringe Filter Unit (Sterile) | Merck | SLHP033RS | |
| Opti-MEM Reduced Serum Medium | Thermo Fisher | 31985062 | |
| pMDG | AddGene | 187440 | |
| Polybrene (Hexadimethrine bromide) | Merck | H9268-5G | |
| pSIN4-CMV-K2M | AddGene | 21164 | |
| pSIN4-EF2-O2S | AddGene | 21162 | |
| psPAX2 | AddGene | 12260 | |
| pWPT-GFP | AddGene | 12255 | |
| RPMI 1640 Medium (ATCC modification) | Thermo Fisher | A1049101 | |
| Sodym Pyruvate | Thermo Fisher | 11360-039 | |
| Sterile Syringes for Single Use (60 mL) | Thermo Fisher | 15899152 | |
| TrypLE Express Enzyme (1x), phenol red | Thermo Fisher | 12605036 | |
| UltraPure 0.5M EDTA, pH 8.0 | Thermo Fisher | 15575020 | |
| Y-27632 (Dihydrochloride) | STEMCELL Technologies | 72304 |
Riferimenti
- Rahib, L., et al. Projecting cancer incidence and deaths to 2030: the unexpected burden of thyroid, liver, and pancreas cancers in the United States. Cancer Research. 74 (11), 2913-2921 (2014).
- Hu, J. X., et al. Pancreatic cancer: A review of epidemiology, trend, and risk factors. World Journal of Gastroenterology. 27 (27), 4298 (2021).
- Howlader, N., et al. SEER cancer statistics review, 1975-2013. National Cancer Institute. 19, (2016).
- Bengtsson, A., Andersson, R., Ansari, D. The actual 5-year survivors of pancreatic ductal adenocarcinoma based on real-world data. Scientific Reports. 10 (1), 16425 (2020).
- He, J., et al. 2564 resected periampullary adenocarcinomas at a single institution: trends over three decades. HPB. 16 (1), 83-90 (2014).
- Dusch, N., et al. Factors predicting long-term survival following pancreatic resection for ductal adenocarcinoma of the pancreas: 40 years of experience. Journal of Gastrointestinal Surgery. 18 (4), 674-681 (2014).
- Principe, D. R., et al. The current treatment paradigm for pancreatic ductal adenocarcinoma and barriers to therapeutic efficacy. Frontiers in Oncology. 11, 688377 (2021).
- Papademetrio, D. L., et al. Interplay between autophagy and apoptosis in pancreatic tumors in response to gemcitabine. Targeted Oncology. 9 (2), 123-134 (2014).
- Ng, S. S., Tsao, M. S., Nicklee, T., Hedley, D. W. Effects of the epidermal growth factor receptor inhibitor OSI-774, Tarceva, on downstream signaling pathways and apoptosis in human pancreatic adenocarcinoma 1 supported by the National Cancer Institute of Canada and the Pat Myhal Fund for Pancreatic Cancer Research. Molecular Cancer Therapeutics. 1 (10), 777-783 (2002).
- Sultana, A., et al. Meta-analyses of chemotherapy for locally advanced and metastatic pancreatic cancer. Journal of Clinical Oncology. 25 (18), 2607-2615 (2007).
- Sun, J., Russell, C. C., Scarlett, C. J., McCluskey, A. Small molecule inhibitors in pancreatic cancer. RSC Medicinal Chemistry. 11 (2), 164-183 (2020).
- Jiang, H., et al. Targeting focal adhesion kinase renders pancreatic cancers responsive to checkpoint immunotherapy. Nature Medicine. 22 (8), 851-860 (2016).
- Hruban, R. H., et al. Pancreatic intraepithelial neoplasia: a new nomenclature and classification system for pancreatic duct lesions. The American Journal of Surgical Pathology. 25 (5), 579-586 (2001).
- Maitra, A., Hruban, R. H. Pancreatic cancer. Annual Review of Pathology: Mechanisms of Disease. 3, 157-188 (2008).
- Vincent, A., Herman, J., Schulick, R., Hruban, R. H., Goggins, M. Pancreatic cancer. The Lancet. 378 (9791), 607-620 (2011).
- Yachida, S., et al. Distant metastasis occurs late during the genetic evolution of pancreatic cancer. Nature. 467 (7319), 1114-1117 (2010).
- Lennon, A. M., et al. The early detection of pancreatic cancer: what will it take to diagnose and treat curable pancreatic neoplasia. Cancer Research. 74 (13), 3381-3389 (2014).
- Rubio-Viqueira, B., et al. An in vivo platform for translational drug development in pancreatic cancer. Clinical Cancer Research. 12 (15), 4652-4661 (2006).
- Li, C., et al. Identification of pancreatic cancer stem cells. Cancer Research. 67 (3), 1030-1037 (2007).
- Hermann, P. C., et al. Distinct populations of cancer stem cells determine tumor growth and metastatic activity in human pancreatic cancer. Cell Stem Cell. 1 (3), 313-323 (2007).
- Boj, S. F., et al. Organoid models of human and mouse ductal pancreatic cancer. Cell. 160 (1-2), 324-338 (2015).
- Takahashi, K., Yamanaka, S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 126 (4), 663-676 (2006).
- Suvà, M. L., et al. Reconstructing and reprogramming the tumor-propagating potential of glioblastoma stem-like cells. Cell. 157, 580-594 (2014).
- Kotini, A. G., et al. Stage-specific human induced pluripotent stem cells map the progression of myeloid transformation to transplantable leukemia. Cell Stem Cell. 20 (3), 315-328 (2017).
- Stricker, S. H., et al. Widespread resetting of DNA methylation in glioblastoma-initiating cells suppresses malignant cellular behavior in a lineage-dependent manner. Genes Development. 27 (6), 654-669 (2013).
- Chao, M. P., et al. Human AML-iPSCs reacquire leukemic properties after differentiation and model clonal variation of disease. Cell Stem Cell. 20 (3), 329-344 (2017).
- Aparicio, L. A., et al. Clinical implications of epithelial cell plasticity in cancer progression. Cancer letters. 366 (1), 1-10 (2015).
- Grimont, A., Leach, S. D., Chandwani, R. Uncertain beginnings: acinar and ductal cell plasticity in the development of pancreatic cancer. Cellular and Molecular Gastroenterology and Hepatology. 13 (2), 369-382 (2022).
- Greenspan, L. J., Weinstein, B. M. To be or not to be: endothelial cell plasticity in development, repair, and disease. Angiogenesis. 24 (2), 251-269 (2021).
- Papapetrou, E. P. Patient-derived induced pluripotent stem cells in cancer research and precision oncology. Nature Medicine. 22 (12), 1392-1401 (2016).
- Zhang, X., Cruz, F. D., Terry, M., Remotti, F., Matushansky, I. Terminal differentiation and loss of tumorigenicity of human cancers via pluripotency-based reprogramming. Oncogene. 32 (18), 2249-2260 (2013).
- Kim, J., et al. An iPSC line from human pancreatic ductal adenocarcinoma undergoes early to invasive stages of pancreatic cancer progression. Cell Reports. 3 (6), 2088-2099 (2013).
- Kim, J., et al. Detection of early pancreatic ductal adenocarcinoma with thrombospondin-2 and CA19-9 blood markers. Science Translational Medicine. 9 (398), (2017).
- Susac, L., et al. Structure of a fully assembled tumor-specific T cell receptor ligated by pMHC. Cell. 185 (17), 3201-3213 (2022).
- Yu, J., et al. Human induced pluripotent stem cells free of vector and transgene sequences. Science. 324, 797-801 (2009).
- Carette, J. E., et al. Generation of iPSCs from cultured human malignant cells. Blood, The Journal of the American Society of Hematology. 115 (20), 4039-4042 (2010).
- Choi, S. M., et al. Reprogramming of EBV-immortalized B-lymphocyte cell lines into induced pluripotent stem cells. Blood, The Journal of the American Society of Hematology. 118 (7), 1801-1805 (2011).
- Hochedlinger, K., et al. Reprogramming of a melanoma genome by nuclear transplantation. Genes & Development. 18 (15), 1875-1885 (2004).
- Hu, K., et al. Efficient generation of transgene-free induced pluripotent stem cells from normal and neoplastic bone marrow and cord blood mononuclear cells. Blood, The Journal of the American Society of Hematology. 117 (14), e109-e119 (2011).
- Iskender, B., Izgi, K., Canatan, H. Reprogramming bladder cancer cells for studying cancer initiation and progression. Tumor Biology. 37, 13237-13245 (2016).
- Khoshchehreh, R., et al. Epigenetic reprogramming of primary pancreatic cancer cells counteracts their in vivo tumourigenicity. Oncogene. 38 (34), 6226-6239 (2019).
- Kim, H. J., et al. Establishment of hepatocellular cancer induced pluripotent stem cells using a reprogramming technique. Gut and Liver. 11 (2), 261 (2017).
- Lin, S. L., et al. Mir-302 reprograms human skin cancer cells into a pluripotent ES-cell-like state. RNA. 14 (10), 2115-2124 (2008).
- Miyoshi, N., et al. Defined factors induce reprogramming of gastrointestinal cancer cells. Proceedings of the National Academy of Sciences. 107 (1), 40-45 (2010).
- Singovski, G., et al. In vivo epigenetic reprogramming of primary human colon cancer cells enhances metastases. Journal of Molecular Cell Biology. 8 (2), 157-173 (2016).
- Zhao, H., et al. A highly optimized protocol for reprogramming cancer cells to pluripotency using nonviral plasmid vectors. Cellular Reprogramming (Formerly" Cloning and Stem Cells". 17 (1), 7-18 (2015).
- Lo, C. A., et al. Quantification of protein levels in single living cells). Cell Reports. 13 (11), 2634-2644 (2015).
- Yu, J., et al. Human induced pluripotent stem cells free of vector and transgene sequences. Science. 324 (5928), 797-801 (2009).
- Prösch, S., et al. Inactivation of the very strong HCMV immediate early promoter by DNA CpG methylation in vitro. Biological Chemistry Hoppe-Seyler. 377 (3), 195-201 (1996).
- Mehta, A. K., Majumdar, S. S., Alam, P., Gulati, N., Brahmachari, V. Epigenetic regulation of cytomegalovirus major immediate-early promoter activity in transgenic mice. Gene. 428 (1-2), 20-24 (2009).
- Chen, X., et al. Integration of external signaling pathways with the core transcriptional network in embryonic stem cells. Cell. 133 (6), 1106-1117 (2008).
- Kim, J., Chu, J., Shen, X., Wang, J., Orkin, S. H. An extended transcriptional network for pluripotency of embryonic stem cells. Cell. 132 (6), 1049-1061 (2008).
- Chronis, C., et al. Cooperative binding of transcription factors orchestrates reprogramming. Cell. 168 (3), 442-459 (2017).
- Li, D., et al. Chromatin accessibility dynamics during iPSC reprogramming. Cell Stem Cell. 21 (6), 819-833 (2017).
- Soufi, A., Donahue, G., Zaret, K. S. Facilitators and impediments of the pluripotency reprogramming factors' initial engagement with the genome. Cell. 151 (5), 994-1004 (2012).
- Carey, B. W., et al. Reprogramming of murine and human somatic cells using a single polycistronic vector. Proceedings of the National Academy of Sciences of the United States of America. 106, 157-162 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon