Method Article
Reprogrammation de l’adénocarcinome canalaire pancréatique à la pluripotence
Dans cet article
Résumé
Le présent protocole décrit la reprogrammation de l’adénocarcinome canalaire pancréatique (PDAC) et des cellules épithéliales canalaires pancréatiques normales en cellules souches pluripotentes induites (CSPi). Nous fournissons une procédure optimisée et détaillée, étape par étape, de la préparation du lentivirus à l’établissement de lignées iPSC stables.
Résumé
La génération de cellules souches pluripotentes induites (CSPi) à l’aide de facteurs de transcription a été réalisée à partir de presque tous les types de cellules différenciées et s’est avérée très précieuse pour la recherche et les applications cliniques. Il est intéressant de noter que la reprogrammation des cellules cancéreuses par iPSC, comme l’adénocarcinome canalaire pancréatique (PDAC), inverse le phénotype invasif du PDAC et remplace l’épigénome du cancer. La différenciation des iPSC dérivées du PDAC peut récapituler la progression du PDAC à partir de son précurseur précoce de la néoplasie intra-épithéliale pancréatique (PanIN), révélant les changements moléculaires et cellulaires qui se produisent tôt au cours de la progression du PDAC. Par conséquent, les iPSC dérivées du PDAC peuvent être utilisées pour modéliser les premiers stades du PDAC pour la découverte de marqueurs diagnostiques de détection précoce. Ceci est particulièrement important pour les patients atteints de PDAC, qui sont généralement diagnostiqués aux stades métastatiques tardifs en raison d’un manque de biomarqueurs fiables pour les stades antérieurs de PanIN. Cependant, la reprogrammation des lignées cellulaires cancéreuses, y compris le PDAC, en pluripotence reste difficile, laborieuse et très variable entre les différentes lignées. Ici, nous décrivons un protocole plus cohérent pour générer des iPSC à partir de diverses lignées cellulaires PDAC humaines à l’aide de vecteurs lentiviraux bicistroniques. Les lignées iPSC résultantes sont stables, ne montrant aucune dépendance à l’expression exogène de facteurs de reprogrammation ou de médicaments inductibles. Dans l’ensemble, ce protocole facilite la génération d’un large éventail d’iPSC dérivées du PDAC, ce qui est essentiel pour découvrir des biomarqueurs précoces plus spécifiques et représentatifs des cas de PDAC.
Introduction
L’adénocarcinome canalaire pancréatique (PDAC) est l’une des tumeurs malignes les plus mortelles, et le diagnostic précoce reste difficile en raison de la nature asymptomatique de la maladie. La majorité des patients atteints de PDAC sont diagnostiqués au stade métastatique avancé lorsque les options de traitement disponibles sont très limitées 1,2. Cela est principalement dû au manque de biomarqueurs fiables pour les stades précoces, tels que ceux qui pourraient être facilement détectés comme des protéines libérées dans la circulation sanguine.
Le PDAC peut se disséminer très tôt au cours de sa progression, et un meilleur pronostic a été associé à la détection précoce du cancer lorsque le PDAC est localisé dans le pancréas3. Cependant, moins d’un dixième des patients atteints de PDAC reçoivent un diagnostic de pronostic favorable, ce qui permet une résection chirurgicale. Néanmoins, les quelques personnes atteintes de tumeurs résécables sont également sujettes à une récidive tumorale dans les 12 mois4.
Au cours des cinq dernières décennies, des améliorations remarquables ont été apportées aux techniques chirurgicales, aux soins aux patients et aux modalités de traitement 5,6. Cependant, le taux de survie à 5 ans chez les patients PDAC réséqués chirurgicalement a à peine atteint 17 %. Néanmoins, cela reste meilleur que celui des patients non réséqués, qui est resté pratiquement inchangé (0,9 %)4,7. La chimiothérapie est le seul autre traitement alternatif du PDAC. Cependant, cette option est très limitée car la grande majorité des patients atteints de PDAC présentent une forte résistance aux médicaments de chimiothérapie tels que la gemcitabine 7,8. D’autres médicaments, comme l’erlotinib, ne sont disponibles que pour un petit groupe de patients atteints de PDAC présentant des mutations spécifiques, dont la plupart présentent une résistance à l’erlotinib9. Les effets secondaires indésirables associés à la chimiothérapie chez la plupart des patients PDAC sont un autre inconvénient de ce traitement10. Récemment, des stratégies prometteuses ont montré que les inhibiteurs de point de contrôle immunitaire (ICI) et les inhibiteurs de kinases à petites molécules (SMKI) peuvent être efficaces dans le traitement du PDAC, mais les réponses durables à ces thérapies ciblées restent limitées à une minorité de patients11,12. Dans l’ensemble, la découverte de biomarqueurs précoces spécifiques au PDAC peut ouvrir de nouvelles voies pour un diagnostic et un traitement précoces.
Le PDAC se développe à partir de lésions précurseurs de néoplasmes intra-épithéliales pancréatiques (PanIN) qui résultent de proliférations épithéliales non invasives du canal pancréatique13,14. Bien que la formation de PanIN soit initiée par des mutations oncogènes telles que KRAS, des altérations génétiques et épigénétiques supplémentaires sont nécessaires pour la progression vers le PDAC. Il a été prévu que la progression du PanIN à travers les différentes étapes vers le PDAC invasif prend environ 10 ans 13,15,16,17. Ce délai offre une excellente occasion de bénéficier d’un diagnostic précoce de l’ACPE. Par conséquent, des recherches approfondies ont été menées pour établir des modèles animaux de xénogreffes tumorales et des cultures d’organoïdes afin d’étudier la progression du PDAC 18,19,20,21. Ces modèles ont été très utiles pour étudier les stades invasifs du PDAC, mais pas la transition des premières phases de PanIN. Il est donc important de développer des modèles expérimentaux capables de récapituler la progression précoce des stades PanIN pour permettre la découverte de biomarqueurs de détection précoce.
La reprogrammation des cellules somatiques en cellules souches pluripotentes induites (iPSC) à l’aide des quatre facteurs de transcription OCT4, SOX2, KLF4 et c-MYC (OSKM) a illustré l’étendue de la plasticité cellulaire22. La plasticité des cellules cancéreuses a été bien documentée, et la reprogrammation des cellules cancéreuses humaines en iPSC a été utilisée avec succès pour réinitialiser les cellules à leur état cellulaire d’origine, éliminant ainsi de nombreuses agressions épigénétiques qui se sont accumulées au cours de la progression du cancer 23,24,25,26,27,28,29. La possibilité d’utiliser cette stratégie de reprogrammation pour manipuler l’identité des cellules cancéreuses s’est donc révélée très prometteuse dans le traitement du cancer30,31. En effet, nous avons précédemment montré que la différenciation des iPSC dérivées des PDAC peut récapituler la progression du PDAC à travers les premiers stades PanIN32. En identifiant les gènes et les voies spécifiques aux stades précoces et intermédiaires du PDAC, des biomarqueurs candidats ont été identifiés qui peuvent être utilisés cliniquement pour le diagnostic précoce du PDAC32,33. Cependant, les biomarqueurs découverts à l’aide d’une seule lignée d’iPSC ont montré une couverture limitée chez la majorité des patients atteints de PDAC32. Les défis liés à la génération de lignées iPSC à partir d’autres patients PDAC ont mis fin à la capacité de découvrir des biomarqueurs plus fiables. Cela est dû à de nombreux facteurs techniques, notamment l’hétérogénéité de l’administration d’OSKM, car seule une petite partie des cellules primaires humaines du PDAC contenait les quatre facteurs et répondait avec succès à la reprogrammation. Ici, un protocole détaillé est présenté pour reprogrammer les cellules primaires PDAC en utilisant une double administration lentivirale plus efficace et cohérente d’OSKM.
Protocole
Tous les protocoles expérimentaux ont été approuvés par le comité d’examen institutionnel de l’OHSU. Toutes les méthodes ont été mises en œuvre conformément aux directives et réglementations en vigueur. Tous les travaux sur les animaux pour les tumeurs PDX ont été effectués avec l’approbation du comité institutionnel d’utilisation et de soins des animaux (IACUC) de l’OHSU. Ce protocole a été testé sur des cellules PDAC primaires issues d’une xénogreffe dérivée d’un patient (PDX), une lignée cellulaire BxPc3 présentant une morphologie épithéliale isolée du tissu pancréatique d’une patiente de 61 ans atteinte d’adénocarcinome, la lignée cellulaire épithéliale immortalisée H6C7 dérivée de l’épithélium normal du canal pancréatique humain et des fibroblastes humains primaires dérivés d’une biopsie cutanée d’individus sains. Des échantillons de PDAC humains ont été obtenus dans le cadre de l’étude Oregon Pancreas Tissue Registry (IRB00003609). Le consentement éclairé a été obtenu de tous les sujets. Des fibroblastes primaires humains ont été obtenus à RBiomedical, Édimbourg, Royaume-Uni, à partir d’échantillons de peau provenant de donneurs anonymes subissant une intervention chirurgicale de routine à l’Edinburgh Royal Infirmary, Little France, Royaume-Uni, avec leur consentement et leur approbation éthique (09/MRE00/91). Tous les travaux sur les lentivirus ont été effectués dans le cadre d’une activité de recherche de classe 2 (GM207/16.6) et approuvés par le département de la santé et de la sécurité de l’Université d’Édimbourg et notifiés à l’autorité compétente du gouvernement écossais en matière de HSE. Toutes les expériences de reprogrammation utilisant des cellules pancréatiques humaines ont été réalisées sous l’approbation éthique du comité d’éthique de l’École des sciences biologiques de l’Université d’Édimbourg (référence # asoufi-0002).
1. Préparation des lentivirus
- Pour la préparation de lentivirus, préparer des plasmides d’emballage et d’expression de haute qualité (sans endotoxines) avec des concentrations comprises entre 1 et 2 μg/μL, y compris psPAX2, pMDG34 et deux vecteurs bicistroniques contenant des SRE ; le codage pSIN4-EF1a-O2S pour l’expression d’OCT4 et SOX2 pilotée par le promoteur EF-1α, et pSIN4-CMV-K2M pour l’expression de KLF4 et c-MYC sous l’amplificateur/promoteur CMV35 (voir Tableau des matériaux). Préparez également le plasmide pWPT-GFP à utiliser comme contrôle de transfection.
- Décongélation d’une lignée cellulaire de rein embryonnaire humain (293T) et culture dans le milieu essentiel minimum (GMEM) de Glasgow, complétée par 10 % de sérum de veau fœtal (FCS), 1 x acides aminés non essentiels, 1 mM de pyruvate de sodium et 1 mM de glutamine à (voir tableau des matières) à 37 °C et 5 % de CO2.
REMARQUE : Il est recommandé d’utiliser des cellules 293T dans quatre passages après la décongélation. - Semez le 293T à une densité de 3 millions de cellules par boîte de 15 cm, 24 h avant la transfection. Un total de trois plats sont nécessaires. Il est préférable d’ensemencer les cellules plus tard dans l’après-midi ~16h00.
- Le lendemain (~16h00), lorsque les cellules atteignent ~40%-50% de confluence, préparez les éléments suivants pour trois réactions de transfection :
REMARQUE : Tenez toujours compte de l’erreur de pipetage en ajoutant 10 % de volume supplémentaire.- Étiqueter trois tubes en plastique de 15 mL avec le nom de lentivirus approprié (pSIN4-EF1a-O2S, pSIN4-CMV-K2M et témoin pwPT-GFP). Ajouter 1,710 mL de milieu sérique réduit (voir le tableau des matériaux) dans chaque tube.
- Diluer 90 μL de réactif de transfection (voir Tableau des matériaux) dans un milieu sérique réduit de 1,710 mL, mélanger par vertexation pendant 2 s et incuber à température ambiante pendant 5 min.
- Mélanger les vecteurs d’emballage ; 5,1 μg de psPAX2 et 2,4 μg de pMDG (7,5 μg au total).
- Ajouter le mélange de vecteurs d’emballage au milieu de transfection (à partir de l’étape 1.4.2) et agiter pendant 2 s.
- Ajouter 7,5 μg de chaque vecteur de reprogrammation : pSIN4-EF1a-O2S et pSIN4-CMV-K2M et le témoin pwPT-GFP au mélange de transfection de l’étape (1.4.4) et du vortex pendant 2 s.
REMARQUE : Utilisez le vecteur d’expression : vecteur viral dans un rapport de 1:1.
- Incuber les tubes de transfection pendant 15 min à température ambiante.
- Transfectez les cellules 293T avec chaque lentivirus en ajoutant directement le mélange transfection-ADN de l’étape (1.4.5) au milieu de manière goutte à goutte. Faites tourner le plat pour assurer une répartition uniforme sur toute la surface.
- Incuber les cellules transfectées dans un incubateur à 37 °C et 5 % de CO2 pendant la nuit.
REMARQUE : Utilisez des déchets spécifiques au virus, un seau d’élimination en métal, un sac double avec des sacs d’autoclave, et prenez un récipient à déchets liquides pour les virus et ajoutez un comprimé désinfectant. Jetez toutes les pipettes, embouts et tubes dans un seau en métal. Jetez les anciens supports dans un conteneur à déchets liquides à virus. Évitez d’utiliser des pipettes en verre et de la verrerie pour éliminer la contamination accidentelle par des virus. - Après 14 à 16 h après la transfection, remplacer le milieu par 30 mL de milieu frais 293T.
- Incuber les cellules transfectées à 37 °C, 5 % de CO2 pendant 60 à 72 heures après le changement de milieu. Observez les cellules quotidiennement et vérifiez l’efficacité de la transfection par fluorescence GFP.
REMARQUE : Idéalement, l’efficacité de la transfection devrait être de >90 % par GFP. Pour les autres virus, il faut observer des changements morphologiques clairs des cellules 293T, car elles ont tendance à devenir plus rondes lors de la production de particules virales. - Prélever le milieu de chaque culture de transfection virale dans des tubes de 50 mL et tourner vers le bas pour éliminer les débris cellulaires à 1932 x g pendant 10 min à 4 °C.
- Filtrer chaque surnageant lentiviral à l’aide d’un filtre à seringue de 0,45 μM pour éliminer les petits débris et les recueillir dans de nouveaux tubes de 50 mL.
- Diviser chaque surnageant de lentivirus en aliquotes de 6 mL et congeler chacune d’elles dans de l’azote liquide.
- Conservez les aliquotes de lentivirus à -80 °C jusqu’au moment de les utiliser.
2. Reprogrammation de la transduction des lentivirus
- Décongeler les cellules primaires de PDAC et les cultiver dans un milieu libre sérique de kératinocytes (KSFM) complètement défini, complété par de l’extrait hypophysaire bovin (BPE), du facteur de croissance épidermique recombinant humain (EGF) à 5 ng/mL et de la toxine cholérique à 50 ng/mL dans un incubateur à 37 °C, 5 % de CO2 et 5 % d’O2 (hypoxie) (voir le tableau des matériaux).
- Décongélation de BxPc3, une lignée cellulaire d’adénocarcinome canalaire pancréatique squameux, et culture dans un milieu RPMI 1640 complété par 10 % de sérum de veau fœtal (FCS) à 37 °C et 5 % de CO2.
- Décongeler les cellules H6C7, les cellules épithéliales canalaires pancréatiques et les mettre en culture dans des KSFM supplémentées en BPE et EGF à 5 ng/mL, à 37 °C et 5 % de CO2.
- Décongeler des fibroblastes humains (hFib) et culture dans le milieu essentiel minimum (GMEM) de Glasgow, complété par 10 % de FCS, 1 x acides aminés non essentiels, 1 mM de pyruvate de sodium et 1 mM de glutamine, et 0,05 mM (final) de bêta-mercaptoéthanol à 37 °C et 5 % de CO2.
- Un jour avant la transduction du lentivirus (de préférence en fin d’après-midi), préparer deux puits d’une plaque de 6 puits contenant 100 000 cellules par puits de chacune des cellules PDAC, BxPc3, H6C7 et fibrillation fibique. Infectez un puits avec des lentivirus OSKM et utilisez le second comme témoin non infecté.
REMARQUE : Utilisez une plaque séparée pour chaque type de cellule. - Le lendemain, dans l’après-midi (environ 24 h plus tard), assurez-vous que la confluence cellulaire atteint au moins 70 % avant de passer à l’étape suivante de l’infection par le lentivirus.
3. Transduction du PDAC par les lentivirus
- Décongeler 5 mL de chaque surnageant de lentivirus dans un incubateur à virus à 37 °C.
- Préparez deux tubes de 15 mL, contenant chacun 2 mL de milieu de culture PDAC préchauffé.
- Dans le premier tube, ajouter 6 mL de chaque lentivirus reprogrammé (pSIN4-CMV-K2M et pSIN4-EF1a-O2S) et 12 μL de polybrène à 4,5 mg/mL (4,5 μg/mL final, voir le tableau des matières) et mélanger.
- Dans le deuxième tube, ajouter 2 μL de polybrène à 4,5 mg/mL (4,5 μg/mL final).
- Jeter le milieu de chaque puits et laver une fois avec du PBS à température ambiante.
- Ajouter le milieu de reprogrammation (tube 1) dans le premier puits.
- Ajouter le mélange du tube 2 au deuxième puits. Ce sera le contrôle non infecté.
- Incuber les cellules PDAC dans un incubateur à 37 °C, 5 % de CO2 et 5 % d’O2 (hypoxie) pendant la nuit.
- Le lendemain, dans l’après-midi, jeter le milieu des deux puits et le remplacer par un milieu de culture PDAC frais.
- Incuber les cellules à 37 °C, 5 % de CO2 et 5 % d’O2 (hypoxie) pendant 48 h.
4. Transduction lentivirale des cellules BxPc3
- Décongeler 3 mL de chaque surnageant de lentivirus dans un incubateur à virus à 37 °C.
- Préparez deux tubes de 15 mL, contenant chacun 2 mL de milieu de culture BxPc3.
- Dans le premier tube, ajouter 3 mL de chaque lentivirus en reprogrammation (pSIN4-CMV-K2M et pSIN4-EF1a-O2S) et 6 μL de polybrène à 4,5 mg/mL (4,5 μg/mL final) et mélanger.
- Dans le deuxième tube, ajouter 2 μL de polybrène à 4,5 mg/mL (4,5 μg/mL final).
- Jeter le milieu de chaque puits et laver une fois avec du PBS.
- Ajouter le milieu de reprogrammation (tube 1) dans le premier puits.
- Ajouter le mélange du tube 2 au deuxième puits. Ce sera le contrôle non infecté.
- Incuber les cellules BxPc3 infectées à 37 °C de virus et 5 % de CO2 pendant la nuit.
- Le lendemain, dans l’après-midi, jetez le milieu des deux puits et remplacez-le par un milieu de culture BxPc3 frais.
- Incuber les cellules à 37 °C et 5 % de CO2 pendant 48 h.
5. Transduction lentivirale des cellules H6c7 infection
- Décongeler 4 mL de chaque surnageant de lentivirus dans un incubateur à virus à 37 °C.
- Préparez deux tubes de 15 mL contenant chacun 2 mL de milieu de culture H6c7.
- Dans le premier tube, ajouter 4 mL de chaque lentivirus reprogrammé (pSIN4-CMV-K2M et pSIN4-EF1a-O2S) et 8 μL de polybrène à 4,5 mg/mL (4,5 μg/mL final) et mélanger.
- Dans le deuxième tube, ajouter 2 μL de polybrène à 4,5 mg/mL (4,5 μg/mL final).
- Jeter le milieu des deux puits et laver une fois avec du PBS.
- Ajouter le milieu de reprogrammation (tube 1) dans le premier puits.
- Ajouter le mélange du tube 2 au deuxième puits. Ce sera le contrôle non infecté.
- Incuber les cellules H6C7 infectées à 37 °C de virus et 5 % de CO2 pendant la nuit.
- Le lendemain, dans l’après-midi, jeter le milieu des deux puits et le remplacer par un milieu de culture H6c7 frais.
- Incuber les cellules à 37 °C et 5 % de CO2 pendant 48 h.
6. Transduction des cellules hFib par lentivirus
- Décongeler 2 mL de chaque surnageant de lentivirus dans un incubateur à virus à 37 °C.
- Préparez deux tubes de 15 mL, contenant chacun 2 mL de milieu de culture de Fibrillation h.
- Dans le premier tube, ajouter 2 mL de chaque virus reprogrammé (pSIN4-CMV-K2M et pSIN4-EF1a-O2S) et ajouter 4 μL de polybrène à 4,5 mg/mL (4,5 μg/mL final) et mélanger.
- Dans le deuxième tube, ajouter 2 μL de polybrène à 4,5 mg/mL (4,5 μg/mL final) et mélanger.
- Jeter le milieu de chaque puits et laver une fois avec du PBS.
- Ajouter le milieu de reprogrammation (tube 1) dans le premier puits.
- Ajouter le mélange du tube 2 au deuxième puits. Ce sera le contrôle non infecté.
- Incuber les cellules de Fibrillation auriculaire infectées à 37 °C et 5 % de CO2 pendant la nuit.
- Le lendemain, dans l’après-midi, jetez le milieu des deux puits et remplacez-le par un milieu de culture de fibrillation hépatique fraîche.
- Incuber les cellules à 37 °C et 5 % de CO2 pendant 48 heures supplémentaires.
REMARQUE : Pour une transduction efficace des lentivirus, titrez soigneusement la quantité de lentivirus, car elle varie considérablement entre les différents types de cellules. Des transductions multiples de lentivirus peuvent être nécessaires pour certains types de cellules. Les lignées cellulaires PDAC nécessitent jusqu’à trois doses, alors qu’une dose suffisait pour reprogrammer les lignées BxPc3, H6C7 et hFib.
7. Préparation des cellules d’alimentation iMEF
- Préparer 40 mL de solution de gélatine à 0,2 % en diluant la solution mère de gélatine à 1 % avec du PBS.
- Enduire quatre plaques de 6 puits en recouvrant chaque puits de 2 ml de solution de gélatine à 0,2 %.
- Incuber les plaques revêtues de gélatine à 37 °C et 5 % de CO2 pendant au moins 30 min.
- Aspirer l’excès de solution de gélatine et laisser sécher les plaques sous la hotte.
- Décongeler deux flacons (~ 4 millions de cellules par flacon) de fibroblastes embryonnaires de souris irradiés (iMEF) en les faisant tourner dans un bain-marie à 37 °C.
REMARQUE : Pour réduire le risque de contamination, veillez à empêcher l’eau d’éclabousser près de l’ouverture du bouchon. Séchez le flacon avec une serviette en papier et vaporisez-le avec de l’éthanol à 70 %. - Dans le capot de culture tissulaire, pipeter le contenu de chaque fiole de FIMi décongelée dans un tube de 15 mL contenant 10 mL de milieu de culture de Fibrillation hébraïque préchauffé en goutte à goutte et bien mélanger en inversant doucement le tube.
- Faire tourner la suspension cellulaire à 309 x g pendant 3 min à température ambiante et retirer le surnageant de chaque tube. Remettre en suspension chaque pastille de cellule dans un milieu de Fibrillation prolongée de 12 mL.
- Plaque de 2 mL de suspension cellulaire par puits des plaques à 6 puits recouvertes de gélatine. Répartissez uniformément les cellules en secouant doucement les plaques dans toutes les directions.
- Incuber les plaques à 37 °C et 5 % de CO2 pendant la nuit.
REMARQUE : Préparez des plaques iMEF fraîches 24 h avant de les utiliser pour l’expérience de reprogrammation.
8. Transfert des cellules infectées sur la couche nourricière iMEF
- Jetez les milieux des cellules PDAC, BxPc3, H6c7 et hFib infectées et non infectées. Lavez chaque puits deux fois avec du PBS à température ambiante.
- Dissociez les cellules de la plaque en ajoutant 0,5 mL de trypsine dans chaque puits. Incuber les cellules PDAC dans un incubateur à 37 °C, 5 % de CO2 et 5 % d’O2 (hypoxie) pendant 15 min.
- Incuber les cellules BxPc3 à 37 °C et 5 % de CO2 pendant 5 min.
- Incuber les cellules H6c7 et hFib à 37 °C et 5 % de CO2 pendant 4 min.
- Transférez la suspension cellulaire dissociée de chaque puits dans des tubes de 15 mL étiquetés comme infectés ou non infectés.
- Récolter les cellules par centrifugation comme suit : pour le PDAC, essorer la suspension cellulaire à 300 x g pendant 5 min ; pour BxPc3, essorer la suspension cellulaire à 120 x g pendant 5 min ; pour H6C7, essorer la suspension cellulaire à 112 x g pendant 4 minutes ; pour la fibrillation hépatique, faire tourner la suspension cellulaire à 161 x g pendant 3 min. Effectuez toutes les centrifugations à température ambiante.
- Remettre en suspension chaque pastille de type cellulaire dans 1 mL du milieu de culture approprié.
- Laver deux fois les plaques iMEF préparées à l’étape 7 avec chaque milieu de culture de type cellulaire. c’est-à-dire laver une plaque avec un milieu PDAC, une avec un milieu BxPc3, une avec un milieu H6C7 et l’autre avec un milieu hFib.
- Plaque 50 000 cellules infectées par puits dans cinq puits de plaque iMEF à 6 puits. Plaque 50 000 cellules témoins non infectées dans le puits restant de la plaque iMEF à 6 puits.
REMARQUE : Optimisez le nombre de cellules infectées à mettre en plaque de 1 000 à 50 000 cellules par puits. - Incuber les cellules PDAC à 37 °C, 5 % de CO2 et 5 % d’O2 (hypoxie) pendant la nuit.
- Incuber les cellules BxPc3, H6C7 et hFib à 37 °C et 5 % de CO2 pendant la nuit.
- Le lendemain, préparer le milieu de reprogrammation comme suit : ajouter 100 mL de sérum de remplacement (25 % final), 5 mL de glutamine 200 mM (1 mM final), 5 mL de 100 acides aminés non essentiels (100 μM final), 1,5 mL de bêta-mercaptoéthanol (0,1 mM final) à 400 mL de milieu Eagle modifié (DMEM).
- Conserver le milieu de reprogrammation dans des aliquotes de 100 mL à 4 °C jusqu’à 4 semaines ou à -20 °C plus longtemps.
- Au moment de l’utiliser, ajouter 100 μL de facteur de croissance des fibroblastes basiques (FGFb) à 10 mg/mL (10 ng/mL final) à une aliquote de milieu de reprogrammation de 100 mL.
- Réchauffez le support de reprogrammation dans un bain-marie à 37 °C.
- Lavez deux fois les cellules qui se développent sur les mangeoires iMEF avec un milieu de reprogrammation préchauffé.
Ajouter 2 mL de milieu de reprogrammation dans chaque puits. - Transvaser les plaques de reprogrammation dans un incubateur à 37 °C, 5 % de CO2et 3 % d’O2 (hypoxie).
REMARQUE : Le jour où le support de reprogrammation est ajouté est considéré comme le jour 1 de la reprogrammation. - Nourrissez les cellules quotidiennement avec des milieux de reprogrammation frais jusqu’à ce que les colonies d’iPSC commencent à apparaître.
REMARQUE : Identifier les colonies d’iPSC par leur morphologie de type ESC (colonies compactes avec des bords bien définis et composées de cellules avec un rapport noyau/cytoplasme élevé). - Surveillez quotidiennement les colonies de CSPi et, une fois qu’un nombre suffisant a été formé, faites-les passer en piscine.
REMARQUE : S’assurer que les colonies de CSPi ne se touchent pas, car cela entraînerait une différenciation et affecterait leur stabilité à long terme. - Pour établir des lignées clonales, passez le pool d’iPSC environ 5 fois, puis choisissez des colonies robustes qui conservent leur morphologie semblable à celle de l’ESC.
- Pour identifier les colonies d’iPSC entièrement reprogrammées, effectuer une coloration vivante avec TRA-160 (voir Tableau des matériaux) et récolter l’ARN des colonies d’iPSC pour la caractérisation de l’expression génique.
9. Passage des colonies de CSPi
- Préparez suffisamment de plaques d’alimentation iMEF 24 h avant de passer les colonies iPSC comme décrit à l’étape 7.
- Lavez deux fois les plaques d’alimentation iMEF avec un support de reprogrammation préchauffé.
- Ajouter 2 mL de milieu de reprogrammation complété par un inhibiteur de ROCK (Y2) (10 μM) dans chaque puits de la plaque iMEF.
- Incuber les plaques iMEF dans un incubateur à 37 °C, 5 % de CO2 et 3 % d’O2 jusqu’au moment de l’utiliser.
- Préparer une solution saline tamponnée d’acide éthylènediaminetétraacétique et de phosphate (EDTA/PBS) en diluant 0,5 M d’EDTA 1:1000 dans du PBS (0,5 mM final).
- Remplacer le milieu de culture de reprogrammation par 0,5 mL d’EDTA/PBS dans chaque puits.
- Incuber dans un incubateur à 37 °C, 5 % de CO2 et 3 % d’O2 ou à température ambiante, selon le type de cellule. Les iPSC BxPc3 et PDAC nécessitent une incubation de 15 minutes à 37 °C. Les CSPi H6C7 et hFib doivent être incubées à température ambiante pendant 5 min.
- Vérifiez les cellules iPSC au microscope jusqu’à ce qu’elles commencent à se dissocier uniformément de la couche nourricière dans toute la colonie.
- Prélever la suspension de cellules iPSC dissociées dans un tube à centrifuger de 15 mL. Essorer à 300 x g pendant 5 min à température ambiante.
- Remettre la pastille en suspension dans 1 mL de milieu de reprogrammation complété par Y2 (10 μM).
- Plaquez les cellules sur la couche d’alimentation iMEF en transférant chaque 1 mL de suspension de cellules iPSC dans trois puits de la plaque iMEF.
REMARQUE : Le rapport de répartition peut varier en fonction du nombre de colonies de CSPi récoltées. - Incuber dans un incubateur à 37 °C, 5 % de CO2 et 3 % d’O2 .
10. Coloration vivante des colonies iPSC avec TRA-1-60
- Préparer 0,5 mL par puits d’anticorps TRA-1-60 à 4 μg/mL dans un milieu de reprogrammation.
- Jeter le milieu de reprogrammation et le remplacer par un mélange d’anticorps de 0,5 mL dans chaque puits. Incuber dans un incubateur à 37 °C, 5 % de CO2 et 3 % d’O2 pendant 30 min.
- Lavez les cellules deux fois avec le support de reprogrammation.
- Capturez des images de fluorescence avec un système d’imagerie cellulaire à l’aide du filtre GFP.
- Vérifiez la qualité de l’image en tenant compte du canal de contrôle négatif.
Résultats
Des images représentatives montrant la morphologie des colonies d’iPSC dérivées de cellules PDAC, BXPc3, H6C7 et hFib sont présentées à la figure 1. Les colonies PDAC-iPSC ont commencé à se former au jour 25 de la reprogrammation. Des colonies de CSPi robustes avec une morphologie plus établie de type ESC ont été identifiées au jour 40 de la reprogrammation (Figure 1). De même, la formation des CSPi BxPc3 a commencé le 23e jour et s’est établie au 35e jour. La formation des CSPi H6C7 était similaire à celle des CSPi du CADPE et a commencé à s’établir au jour 45. Les colonies de hFib-iPSC ont commencé à se former au jour 15 de la reprogrammation.
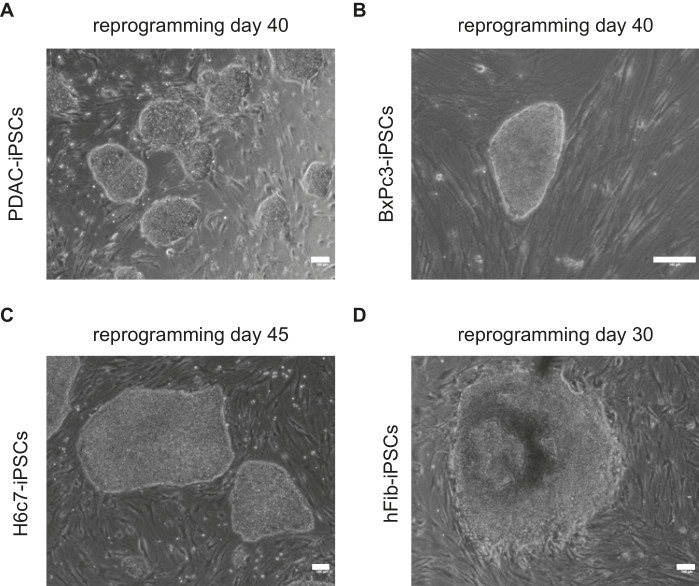
Figure 1 : Images représentatives des colonies d’iPSC. Les images montrent une morphologie établie de type CSEh dérivée de (A) PDAC, (B) BxPc3, (C) H6c7 et (D) hFib. Les jours de reprogrammation sont indiqués au-dessus de chaque image. Barres d’échelle = 100 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Des lignées clonales d’iPSC ont été établies à partir de chaque reprogrammation de cellules PDAC, BxPc3, H6C7 et hFib. Toutes les lignées iPSC établies ont été colorées positives pour TRA-1-60, un marqueur de surface cellulaire hESC indifférencié, confirmant leur reprogrammation en pluripotence (Figure 2).

Figure 2 : Images représentatives des lignées iPSC clonales. Les images sont dérivées de (A) PDAC, (B) BxPc3, (C) H6c7 et (D) hFib montrant une morphologie de type ESC (panneaux supérieurs) et une coloration positive pour TRA-1-60 (panneau inférieur). Barres d’échelle = 100 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Pour faciliter l’utilisation de la reprogrammation des iPSC pour étudier la progression du cancer, un protocole robuste a été établi pour la reprogrammation des cellules cancéreuses du pancréas. La reprogrammation des cellules cancéreuses en pluripotence s’est avérée très difficile jusqu’à présent, car seules quelques études ont réussi à générer des iPSC à partir de cellules cancéreuses 32,36,37,38,39,40,41,42,43,44,45,46. La plupart de ces études ont utilisé des lignées cellulaires cancéreuses immortalisées pour générer des lignées iPSC, et non des cellules primaires dérivées de patients 36,37,38,40,42,43,44,46. Par exemple, la reprogrammation de quatre lignées cellulaires différentes de cancer du foie a été tentée en utilisant l’introduction rétrovirale de facteurs OSKM, mais une seule lignée cellulaire a été reprogrammée avec succès41. Cependant, la lignée iPSC du cancer du foie générée a montré une perte de souche après quelques passages, mettant en évidence la forte résistance des cellules cancéreuses à la reprogrammation d’OSKM41. Auparavant, des tentatives ont été faites pour reprogrammer les cellules PDAC en utilisant l’administration lentivirale d’OSKM individuellement ; cependant, une seule lignée iPSC a été générée, dépendante de l’expression exogène d’OSKM et donc pas entièrement reprogrammée37. Une autre étude a utilisé des vecteurs épisomiques pour délivrer des facteurs OSKM sans intégration génomique, mais n’a réussi à générer qu’un seul clone iPSC à partir du PDAC39. Le succès limité de la reprogrammation des cellules cancéreuses s’ajoute aux nombreux défis qui entravent l’utilisation de la technologie iPSC dans la recherche sur le cancer.
Ici, la génération d’iPSC à partir d’échantillons primaires de PDAC dérivés de deux patients différents et d’une lignée cellulaire PDAC établie (BxPc3) est expliquée. De plus, des iPSC ont également été générées à partir de H6c7, une lignée de cellules épithéliales canalaires pancréatiques. À notre connaissance, il s’agit du premier rapport de lignées iPSC stables dérivées avec succès de BxPc3 et H6c7. À la suite de ce protocole, des iPSC ont également été générées avec succès à partir de fibroblastes humains primaires dérivés d’individus sains, élargissant ainsi l’applicabilité de la méthode au-delà de la recherche sur le cancer.
L’un des éléments clés du succès du protocole est l’utilisation de vecteurs lentiviraux bicistroniques pour co-exprimer les facteurs OS et KM. Ces vecteurs contiennent le site d’entrée interne du ribosome 2 (IRES2) pour exprimer OCT4 et SOX2 dans un vecteur et KLF4 et cMYC dans l’autre. De multiples études utilisant des vecteurs monocistroniques où chaque facteur de reprogrammation a été délivré individuellement ont montré que l’absorption de chaque vecteur est différente, affectant la stœchiométrie OSKM et l’efficacité de la reprogrammation47. L’utilisation de vecteurs bicistroniques peut aider à atténuer ce problème. De plus, l’utilisation d’IRES dans les vecteurs lentiviraux a montré une augmentation significative de l’efficacité de la reprogrammation48. Dans ce système lentivirus, l’expression de la SG est pilotée par le promoteur EF1a, tandis que l’expression de KM est sous le contrôle d’un promoteur CMV. On sait que le promoteur du CMV peut être soumis à un silençage très efficace par méthylation de l’ADN et désacétylation des histones, contrairement à EF1a49,50. Par conséquent, le silence précoce de KM avant la SG pendant la reprogrammation peut être un facteur clé pour le succès du protocole. Ceci est cohérent avec des études antérieures montrant l’importance de la dynamique d’expression d’OSKM lors de la reprogrammation 51,52,53,54,55. Ainsi, le vecteur bicistronique est également plus avantageux que le vecteur polycistronique, où tous les facteurs OSKM sont exprimés à partir d’un seul promoteur56. D’autres facteurs contribuent au succès du protocole, notamment les doses d’infection par les lentivirus et le nombre de cellules infectées utilisées pour la reprogrammation, qui ont été personnalisées pour chaque type de cellule.
En résumé, une méthode optimisée de reprogrammation des cellules primaires PDAC avec d’autres lignées cellulaires et cellules normales est présentée. Cette méthode permettra d’étendre l’utilisation de la reprogrammation des iPSC pour modéliser la progression du cancer et, dans ce cas, découvrir des biomarqueurs précoces du PDAC.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
A.S et J.K tiennent à remercier Cancer Research UK et OHSU pour leur financement (CRUK-OHSU Project Award C65925/A26986). A.S est soutenu par une bourse de développement de carrière du CMR (MR/N024028/1). Les AA sont financés par une bourse de doctorat (bourse réf. 1078107040) de la Cité du roi Abdulaziz pour la science et la technologie. J.K est financé par la subvention de nouveau chercheur MRF (GCNCR1042A) et la subvention Knight CEDAR (68182-933-000, 68182-939-000). Nous remercions le professeur Keisuke Kaji d’avoir aimablement fourni les vecteurs de reprogrammation pSIN4-EF1a-O2S et pSIN4-CMV-K2M. Aux fins du libre accès, l’auteur a appliqué une licence Creative Commons Attribution (CC BY) à toute version du manuscrit accepté par l’auteur découlant de cette soumission.
matériels
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol (50 mM) | Thermo Fisher | 31350010 | |
| Alexa Fluor 488 anti- human TRA-1-60-R | BioLegend | 330613 | |
| Bovine Pituitary Extract (BPE) | Thermo Fisher | 13028014 | |
| BxPc3 | ATCC | CRL-1687 | |
| Cholera Toxin from Vibrio cholerae | Merck | C8052-1MG | |
| Collagen, Type I solution from rat tail | Merck | C3867 | |
| Completed Defined K-SFM | Thermo Fisher | 10744-019 | |
| Corning Costar TC-Treated Multiple Well Plates | Merck | CLS3516 | |
| Corning syringe filters | Merck | CLS431231 | |
| Corning tissue-culture treated culture dishes | Merck | CLS430599 | |
| Day Impex Virkon Disinfectant Virucidal Tablets | Thermo Fisher | 12328667 | |
| Dulbecco′s Phosphate Buffered Saline (PBS) | Merck | D8537 | |
| Fetal Calf Serum (FCS) | Thermo Fisher | 10270-106 | |
| Fugene HD Transfection Reagent | Promega | E2312 | |
| Gelatin solution, Type B, 2% in H2O | Merck | G1393-100ML | |
| Glasgow Minimum Essential Media (GMEM) | Merck | G5154 | |
| Human EGF Recombinant Protein | Thermo Fisher | PHG0311 | |
| Human FGF-basic (FGF-2/bFGF) (154 aa) Recombinant Protein, PeproTech | Thermo Fisher | 100-18B | |
| Human Pancreatic Duct Epithelial Cell Line (H6c7) | Kerafast | ECA001-FP | |
| iMEF feeder cells | iXcells Biotechnologies | 10MU-001-1V | |
| Keratinocyte Serum Free Media (KSFM) | Thermo Fisher | 17005-042 | |
| KnockOut DMEM | Thermo Fisher | 10829018 | |
| KnockOut serum Replacement | Thermo Fisher | 10828028 | |
| L-Glutamine (200 mM) | Thermo Fisher | 25030-024 | |
| MEM Non-Essential Amino Acids Solution (100x) | Thermo Fisher | 11140050 | |
| Millex-HP 0.45 μM syringe Filter Unit (Sterile) | Merck | SLHP033RS | |
| Opti-MEM Reduced Serum Medium | Thermo Fisher | 31985062 | |
| pMDG | AddGene | 187440 | |
| Polybrene (Hexadimethrine bromide) | Merck | H9268-5G | |
| pSIN4-CMV-K2M | AddGene | 21164 | |
| pSIN4-EF2-O2S | AddGene | 21162 | |
| psPAX2 | AddGene | 12260 | |
| pWPT-GFP | AddGene | 12255 | |
| RPMI 1640 Medium (ATCC modification) | Thermo Fisher | A1049101 | |
| Sodym Pyruvate | Thermo Fisher | 11360-039 | |
| Sterile Syringes for Single Use (60 mL) | Thermo Fisher | 15899152 | |
| TrypLE Express Enzyme (1x), phenol red | Thermo Fisher | 12605036 | |
| UltraPure 0.5M EDTA, pH 8.0 | Thermo Fisher | 15575020 | |
| Y-27632 (Dihydrochloride) | STEMCELL Technologies | 72304 |
Références
- Rahib, L., et al. Projecting cancer incidence and deaths to 2030: the unexpected burden of thyroid, liver, and pancreas cancers in the United States. Cancer Research. 74 (11), 2913-2921 (2014).
- Hu, J. X., et al. Pancreatic cancer: A review of epidemiology, trend, and risk factors. World Journal of Gastroenterology. 27 (27), 4298 (2021).
- Howlader, N., et al. SEER cancer statistics review, 1975-2013. National Cancer Institute. 19, (2016).
- Bengtsson, A., Andersson, R., Ansari, D. The actual 5-year survivors of pancreatic ductal adenocarcinoma based on real-world data. Scientific Reports. 10 (1), 16425 (2020).
- He, J., et al. 2564 resected periampullary adenocarcinomas at a single institution: trends over three decades. HPB. 16 (1), 83-90 (2014).
- Dusch, N., et al. Factors predicting long-term survival following pancreatic resection for ductal adenocarcinoma of the pancreas: 40 years of experience. Journal of Gastrointestinal Surgery. 18 (4), 674-681 (2014).
- Principe, D. R., et al. The current treatment paradigm for pancreatic ductal adenocarcinoma and barriers to therapeutic efficacy. Frontiers in Oncology. 11, 688377 (2021).
- Papademetrio, D. L., et al. Interplay between autophagy and apoptosis in pancreatic tumors in response to gemcitabine. Targeted Oncology. 9 (2), 123-134 (2014).
- Ng, S. S., Tsao, M. S., Nicklee, T., Hedley, D. W. Effects of the epidermal growth factor receptor inhibitor OSI-774, Tarceva, on downstream signaling pathways and apoptosis in human pancreatic adenocarcinoma 1 supported by the National Cancer Institute of Canada and the Pat Myhal Fund for Pancreatic Cancer Research. Molecular Cancer Therapeutics. 1 (10), 777-783 (2002).
- Sultana, A., et al. Meta-analyses of chemotherapy for locally advanced and metastatic pancreatic cancer. Journal of Clinical Oncology. 25 (18), 2607-2615 (2007).
- Sun, J., Russell, C. C., Scarlett, C. J., McCluskey, A. Small molecule inhibitors in pancreatic cancer. RSC Medicinal Chemistry. 11 (2), 164-183 (2020).
- Jiang, H., et al. Targeting focal adhesion kinase renders pancreatic cancers responsive to checkpoint immunotherapy. Nature Medicine. 22 (8), 851-860 (2016).
- Hruban, R. H., et al. Pancreatic intraepithelial neoplasia: a new nomenclature and classification system for pancreatic duct lesions. The American Journal of Surgical Pathology. 25 (5), 579-586 (2001).
- Maitra, A., Hruban, R. H. Pancreatic cancer. Annual Review of Pathology: Mechanisms of Disease. 3, 157-188 (2008).
- Vincent, A., Herman, J., Schulick, R., Hruban, R. H., Goggins, M. Pancreatic cancer. The Lancet. 378 (9791), 607-620 (2011).
- Yachida, S., et al. Distant metastasis occurs late during the genetic evolution of pancreatic cancer. Nature. 467 (7319), 1114-1117 (2010).
- Lennon, A. M., et al. The early detection of pancreatic cancer: what will it take to diagnose and treat curable pancreatic neoplasia. Cancer Research. 74 (13), 3381-3389 (2014).
- Rubio-Viqueira, B., et al. An in vivo platform for translational drug development in pancreatic cancer. Clinical Cancer Research. 12 (15), 4652-4661 (2006).
- Li, C., et al. Identification of pancreatic cancer stem cells. Cancer Research. 67 (3), 1030-1037 (2007).
- Hermann, P. C., et al. Distinct populations of cancer stem cells determine tumor growth and metastatic activity in human pancreatic cancer. Cell Stem Cell. 1 (3), 313-323 (2007).
- Boj, S. F., et al. Organoid models of human and mouse ductal pancreatic cancer. Cell. 160 (1-2), 324-338 (2015).
- Takahashi, K., Yamanaka, S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 126 (4), 663-676 (2006).
- Suvà, M. L., et al. Reconstructing and reprogramming the tumor-propagating potential of glioblastoma stem-like cells. Cell. 157, 580-594 (2014).
- Kotini, A. G., et al. Stage-specific human induced pluripotent stem cells map the progression of myeloid transformation to transplantable leukemia. Cell Stem Cell. 20 (3), 315-328 (2017).
- Stricker, S. H., et al. Widespread resetting of DNA methylation in glioblastoma-initiating cells suppresses malignant cellular behavior in a lineage-dependent manner. Genes Development. 27 (6), 654-669 (2013).
- Chao, M. P., et al. Human AML-iPSCs reacquire leukemic properties after differentiation and model clonal variation of disease. Cell Stem Cell. 20 (3), 329-344 (2017).
- Aparicio, L. A., et al. Clinical implications of epithelial cell plasticity in cancer progression. Cancer letters. 366 (1), 1-10 (2015).
- Grimont, A., Leach, S. D., Chandwani, R. Uncertain beginnings: acinar and ductal cell plasticity in the development of pancreatic cancer. Cellular and Molecular Gastroenterology and Hepatology. 13 (2), 369-382 (2022).
- Greenspan, L. J., Weinstein, B. M. To be or not to be: endothelial cell plasticity in development, repair, and disease. Angiogenesis. 24 (2), 251-269 (2021).
- Papapetrou, E. P. Patient-derived induced pluripotent stem cells in cancer research and precision oncology. Nature Medicine. 22 (12), 1392-1401 (2016).
- Zhang, X., Cruz, F. D., Terry, M., Remotti, F., Matushansky, I. Terminal differentiation and loss of tumorigenicity of human cancers via pluripotency-based reprogramming. Oncogene. 32 (18), 2249-2260 (2013).
- Kim, J., et al. An iPSC line from human pancreatic ductal adenocarcinoma undergoes early to invasive stages of pancreatic cancer progression. Cell Reports. 3 (6), 2088-2099 (2013).
- Kim, J., et al. Detection of early pancreatic ductal adenocarcinoma with thrombospondin-2 and CA19-9 blood markers. Science Translational Medicine. 9 (398), (2017).
- Susac, L., et al. Structure of a fully assembled tumor-specific T cell receptor ligated by pMHC. Cell. 185 (17), 3201-3213 (2022).
- Yu, J., et al. Human induced pluripotent stem cells free of vector and transgene sequences. Science. 324, 797-801 (2009).
- Carette, J. E., et al. Generation of iPSCs from cultured human malignant cells. Blood, The Journal of the American Society of Hematology. 115 (20), 4039-4042 (2010).
- Choi, S. M., et al. Reprogramming of EBV-immortalized B-lymphocyte cell lines into induced pluripotent stem cells. Blood, The Journal of the American Society of Hematology. 118 (7), 1801-1805 (2011).
- Hochedlinger, K., et al. Reprogramming of a melanoma genome by nuclear transplantation. Genes & Development. 18 (15), 1875-1885 (2004).
- Hu, K., et al. Efficient generation of transgene-free induced pluripotent stem cells from normal and neoplastic bone marrow and cord blood mononuclear cells. Blood, The Journal of the American Society of Hematology. 117 (14), e109-e119 (2011).
- Iskender, B., Izgi, K., Canatan, H. Reprogramming bladder cancer cells for studying cancer initiation and progression. Tumor Biology. 37, 13237-13245 (2016).
- Khoshchehreh, R., et al. Epigenetic reprogramming of primary pancreatic cancer cells counteracts their in vivo tumourigenicity. Oncogene. 38 (34), 6226-6239 (2019).
- Kim, H. J., et al. Establishment of hepatocellular cancer induced pluripotent stem cells using a reprogramming technique. Gut and Liver. 11 (2), 261 (2017).
- Lin, S. L., et al. Mir-302 reprograms human skin cancer cells into a pluripotent ES-cell-like state. RNA. 14 (10), 2115-2124 (2008).
- Miyoshi, N., et al. Defined factors induce reprogramming of gastrointestinal cancer cells. Proceedings of the National Academy of Sciences. 107 (1), 40-45 (2010).
- Singovski, G., et al. In vivo epigenetic reprogramming of primary human colon cancer cells enhances metastases. Journal of Molecular Cell Biology. 8 (2), 157-173 (2016).
- Zhao, H., et al. A highly optimized protocol for reprogramming cancer cells to pluripotency using nonviral plasmid vectors. Cellular Reprogramming (Formerly" Cloning and Stem Cells". 17 (1), 7-18 (2015).
- Lo, C. A., et al. Quantification of protein levels in single living cells). Cell Reports. 13 (11), 2634-2644 (2015).
- Yu, J., et al. Human induced pluripotent stem cells free of vector and transgene sequences. Science. 324 (5928), 797-801 (2009).
- Prösch, S., et al. Inactivation of the very strong HCMV immediate early promoter by DNA CpG methylation in vitro. Biological Chemistry Hoppe-Seyler. 377 (3), 195-201 (1996).
- Mehta, A. K., Majumdar, S. S., Alam, P., Gulati, N., Brahmachari, V. Epigenetic regulation of cytomegalovirus major immediate-early promoter activity in transgenic mice. Gene. 428 (1-2), 20-24 (2009).
- Chen, X., et al. Integration of external signaling pathways with the core transcriptional network in embryonic stem cells. Cell. 133 (6), 1106-1117 (2008).
- Kim, J., Chu, J., Shen, X., Wang, J., Orkin, S. H. An extended transcriptional network for pluripotency of embryonic stem cells. Cell. 132 (6), 1049-1061 (2008).
- Chronis, C., et al. Cooperative binding of transcription factors orchestrates reprogramming. Cell. 168 (3), 442-459 (2017).
- Li, D., et al. Chromatin accessibility dynamics during iPSC reprogramming. Cell Stem Cell. 21 (6), 819-833 (2017).
- Soufi, A., Donahue, G., Zaret, K. S. Facilitators and impediments of the pluripotency reprogramming factors' initial engagement with the genome. Cell. 151 (5), 994-1004 (2012).
- Carey, B. W., et al. Reprogramming of murine and human somatic cells using a single polycistronic vector. Proceedings of the National Academy of Sciences of the United States of America. 106, 157-162 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon