Method Article
Uso della bisezione per ridurre il DNA mitocondriale nell'ovocita bovino
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per ridurre significativamente il numero di copie del DNA mitocondriale in un ovocita bovino (P < 0,0001). Questo metodo utilizza la centrifugazione e la bisezione per ridurre sostanzialmente i mitocondri degli ovociti e può consentire una maggiore possibilità di sviluppo negli embrioni di trasferimento nucleare delle cellule somatiche interspecie ricostruiti.
Abstract
Il trasferimento nucleare di cellule somatiche interspecie (iSCNT) può essere utilizzato per salvare specie in via di estinzione, ma esistono due distinte popolazioni di DNA mitocondriale (mtDNA) all'interno dell'embrione ricostruito: una all'interno dell'ooplasma ricevente e una all'interno della cellula somatica donatrice. Questa eteroplasmia mitocondriale può portare a problemi di sviluppo nell'embrione e nel feto. I protocolli di clonazione fatti a mano includono la bisezione degli ovociti, che può essere utilizzata per ridurre il numero di copie del mtDNA, riducendo il grado di eteroplasmia mitocondriale in un embrione ricostruito. La centrifugazione di ovociti bovini maturi denudati ha prodotto una frazione densa di mitocondri visibile a un polo dell'ovocita. Le zonae pellucidae degli ovociti sono state rimosse mediante esposizione a una soluzione di pronasi. La bisezione è stata eseguita utilizzando una microlama per rimuovere la frazione mitocondriale visibile. qPCR è stato utilizzato per quantificare il mtDNA presente in campioni di DNA estratti da ovociti interi e ooplasti tagliati in due, fornendo un confronto dei numeri di copie del mtDNA prima e dopo la bisezione. I numeri di copia sono stati calcolati utilizzando i valori di soglia del ciclo, la formula della linea di regressione di una curva standard e un rapporto che includeva le rispettive dimensioni dei prodotti MTDNA PCR e dei prodotti PCR genomici. Un ovocita bovino aveva un numero medio di copie di mtDNA (deviazione standard ±) di 137.904 ± 94.768 (n = 38). Un ooplasto impoverito di mitocondri aveva un numero medio di copie di mtDNA di 8.442 ± 13.806 (n = 33). Le copie medie di mtDNA presenti in un ooplasto ricco di mitocondri erano 79.390 ± 58.526 copie di mtDNA (n = 28). Le differenze tra queste medie calcolate indicano che la centrifugazione e la successiva bisezione possono ridurre significativamente il numero di copie di mtDNA presenti nell'ooplasto impoverito di mitocondri rispetto all'ovocita originale (P < 0,0001, determinato da ANOVA unidirezionale). La riduzione del mtDNA dovrebbe diminuire il grado di eteroplasmia mitocondriale in un embrione ricostruito, favorendo eventualmente lo sviluppo embrionale e fetale standard. L'integrazione con estratto mitocondriale dalla cellula somatica donatrice può anche essere essenziale per ottenere uno sviluppo embrionale di successo.
Introduzione
Il trasferimento nucleare di cellule somatiche (SCNT) include la fusione di un ovocita enucleato da un animale e una cellula somatica da un animale della stessa specie. Nella maggior parte dei casi, l'ovocita e la cellula somatica provengono dalla stessa specie e i tassi di natalità vivi sono inferiori al 6%1. Alcune ricerche prevedono l'uso di SCNT interspecie (iSCNT), che include la fusione di una cellula somatica e di un ovocita che provengono da due specie diverse. In questi studi, i tassi di natalità vivi sono ancora più bassi rispetto a SCNT, in genere inferiori all'1%1. Tuttavia, iSCNT ha la capacità di essere utilizzato come metodo per salvare le specie in via di estinzione, poiché le cellule somatiche di questi animali sono più accessibili delle loro cellule germinali1. Gli ovociti riceventi utilizzati in iSCNT sono spesso specie domestiche o di laboratorio comuni, come mucche, maiali e topi. Alcuni tentativi fatti finora hanno prodotto con successo giovani vivi, sebbene la prole prodotta sia stata costituita da animali intragenerici (le specie di ovociti riceventi e le specie di cellule donatrici erano membri dello stesso genere)2,3,4. I modelli intergenerici (che utilizzano un ovocita e una cellula somatica di animali di generi diversi) non hanno ancora prodotto animali vivi e la maggior parte degli embrioni ricostruiti arresta allo stadio di 8-16 cellule dello sviluppo in vitro 5,6,7,8. Una possibile spiegazione di questo arresto dello sviluppo embrionale è l'insorgenza di eteroplasmia mitocondriale negli embrioni, la presenza di più di un tipo di DNA mitocondriale (mtDNA) in una singola cellula. L'eteroplasmia può portare a problemi come l'inefficienza dello sviluppo o il fallimento nell'embrione o nell'animale vivo1. La patogenesi può anche verificarsi più tardi nel corso della vita dell'animale9. Sebbene questo problema sia presente anche nella prole SCNT, la componente interspecifica all'interno degli embrioni iSCNT aggrava il problema.
Quando il mtDNA embrionale proviene da due specie diverse, i mitocondri ovocitari riceventi, che rappresentano la maggioranza, non funzionano in modo efficiente o efficace con il nucleo1,10 della cellula donatrice. Maggiori lacune tassonomiche tra le due specie utilizzate in iSCNT probabilmente intensificano questo problema; la prole viva intragenerica prodotta (bos gaurus e bos indicus che utilizza ovociti Bos taurus), così come la prole prodotta tramite SCNT tradizionale (ad esempio la prole Ovis aries che utilizza ovociti Ovis aries) si sono dimostrate chimere (mtDNA di due individui era presente in questi animali 11,12,13). Tuttavia, si sono sviluppati molto più lontano degli embrioni SCNT intergenerici14,15. Lo scambio di informazioni tra i mitocondri ovocitari e il nucleo della cellula donatrice potrebbe avere più successo nell'embrione intragenerico che nell'embrione intergenerico16.
La quantità di mtDNA in un ovocita bovino maturo è circa 100 volte maggiore della quantità trovata in una cellula somatica12. Ridurre questo rapporto potrebbe incoraggiare i mitocondri delle cellule somatiche a proliferare all'interno dell'embrione ricostruito, consentendo a una maggiore popolazione di mitocondri produttivi di essere presente16. Questo potrebbe a sua volta fornire più energia per soddisfare i requisiti dell'embrione in via di sviluppo15. I precedenti tentativi fatti per ridurre il numero di copie del mtDNA dell'ovocita o dell'embrione includono l'applicazione chimica, la micromanipolazione e l'integrazione dell'ovocita o dell'embrione con mitocondri aggiuntivi della specie cellulare donatrice 16,17,18,19,20. Tuttavia, l'applicazione chimica (come la 2',3'-dideossicitidina) non è ideale per lo sviluppo embrionale e ha ridotto il numero di copie di mtDNA degli ovociti di circa la metà18. La precedente riduzione del mtDNA ovocitario mediante micromanipolazione ha rimosso solo una media del 64% del mtDNA17 dell'ovocita. Sebbene l'integrazione di mitocondri di cellule donatrici possa essere un'opzione praticabile, il suo uso non ha ancora prodotto un animale intergenerico vivo all'interno degli studi iSCNT21.
L'uso della bisezione per ridurre il numero di copie di mtDNA degli ovociti non è stato ancora utilizzato negli studi pubblicati. La bisecting degli ovociti con l'intenzione di fondere gli ooplasti con una cellula somatica è la premessa della clonazione fatta a mano (HMC), che in genere utilizza la bisezione come metodo per rimuovere il corpo polare e la piastra di metafase dall'ovocita della metafase II (MII). HMC ha prodotto con successo prole in diverse specie, tra cui capre, bovini, suini, pecore e cavalli 22,23,24,25,26, ma in genere non include una fase di centrifugazione prima della bisezione. L'integrazione della centrifugazione ad alta velocità dell'ovocita consente l'isolamento dei mitocondri (e quindi del mtDNA) in un polo dell'ovocita, che può quindi essere diviso in due utilizzando una microlama per rimuovere quelle frazioni densamente denshe di mitocondri. Due ooplasti impoveriti di mitocondri possono quindi essere fusi con una cellula somatica, come nel caso dell'HMC, per formare un embrione ricostruito che contiene molto meno mtDNA delle specie ovocitarie.
La domanda a cui tentiamo di rispondere con questo protocollo è come ridurre il mtDNA nell'ovocita bovino al fine di produrre un embrione ricostruito vitale che contenga meno mtDNA eteroplasmatico. In questo protocollo, gli ovociti sono stati centrifugati e tagliati in due. I numeri di copie di ooplast e mtDNA di ovociti intatti sono stati calcolati per determinare l'efficacia di questa tecnica nel ridurre il numero di copie di mtDNA dell'ovocita bovino.
Protocollo
Il seguente protocollo segue le linee guida per la cura e l'etica degli animali fornite dalla Utah State University.
1. Preparazione dei media
- Prima della manipolazione degli ovociti, preparare le seguenti soluzioni, come descritto nella Tabella 1: 400 μL di soluzione di ialuronidasi, 500 μL di mezzi T2, 1.020 μL di media T20 e 800 μL di mezzi T10.
- Dividere il supporto T10 in due pozzetti di una piastra a quattro pozzetti, 400 μL per pozzetto. Etichettare un pozzo con "M" e il secondo pozzo con "MR". Mettere in un incubatore a CO2 al 5% fino a dopo la bisezione.
- Preparare 500 μL di Citocalasina B (CB)/Liquido Oviduttale Sintetico con HEPES (HSOF). Aliquota 50 μL della soluzione CB/HSOF in un tubo centrifugo separato da 1,5 mL.
- Preparare 40 μL di soluzione di pronasi. Centrifuga per almeno 30 s a 2.680 x g. Miscelare 20 μL del surnatante con 20 μL di T2 in un nuovo tubo centrifugo; questo forma una soluzione diluita di pronasi da 5 mg/mL.
- Preparare 3 ml di HSOF. Su una piastra di ricerca, depositare separatamente quattro gocce da 400 μL; posizionare questa piastra sotto uno stereomicroscopio.
- Preparare 500 μL di soluzione CB/T20.
2. Maturazione in vitro (IVM) degli ovociti bovini
- IVM: Complessi cumulo-ovociti aspirati di coltura (COC) a 38,5 °C in un incubatore co2 al 5% per 21 ore in un piatto a quattro pozzetti di terreno di maturazione contenente il 10% di FBS, 0,26 UI/mL di FSH e 100 U/mL di penicillina/streptomicina.
- Eseguire i seguenti passaggi per denudere gli ovociti.
- Raccogliere il numero desiderato di COC utilizzando una pipetta da 200 μL e depositarli sul fondo di un tubo centrifugo da 1,5 μL.
- Aggiungere lo stesso volume di 0,6 mg/mL di ialuronidasi alla provetta della centrifuga con gli ovociti (cioè, se il volume degli ovociti e dei mezzi IVM è di 100 μL, aggiungere 100 μL di ialuronidasi).
- Pipettare la soluzione su e giù senza creare bolle, fino a quando tutte le cellule del cumulo sono state rimosse.
- Controllare la maturazione degli ovociti.
- Utilizzare la pipetta per aggiungere 200 μL di HSOF da una delle quattro gocce di HSOF sulla piastra di ricerca alla soluzione di ovociti/ialuronidasi.
- Trasferire gli ovociti dalla goccia di ialuronidasi/HSOF e metterli in 400 μL di goccia di HSOF inutilizzati. Lavarli con due gocce HSOF aggiuntive per aiutare a rimuovere i resti di cellule di ialuronidasi e cumulo.
- Utilizzando una pipetta orale (o una pipetta e una punta da 10 μL) e un elevato ingrandimento microscopico, rotolare e selezionare gli ovociti in base alla presenza del corpo polare.
- Dopo aver selezionato gli ovociti, raccogliere il numero desiderato di ovociti MII da dividere in due e metterli in 400 μL di goccia HSOF.
NOTA: Se gli ovociti sono stati al di fuori dell'incubatrice per più di 30 minuti, gli ovociti possono essere riposizionati in un pozzo di terreno di maturazione e posti in un'incubatrice controllata da CO2 per riposare per almeno 30 minuti. Limitare il numero di ovociti da dividere in due a una quantità che è possibile terminare la biecting in circa 30 minuti per ridurre al minimo il tempo al di fuori dell'incubazione. Se gli ooplasti devono essere fusi con una cellula somatica, attivati e coltivati come embrioni, gli ovociti devono essere enucleati in questo momento.
3. Centrifugazione degli ovociti
NOTA: Se gli ovociti sono stati collocati nell'incubatrice in mezzi di maturazione, spostarli nella goccia HSOF da cui sono stati raccolti più di recente.
- Utilizzando una pipetta orale, raccogliere gli ovociti maturi selezionati dalla goccia HSOF e posizionarli nel tubo da 1,5 ml contenente 50 μL della soluzione HSOF/CB. Centrifugare gli ovociti a 15.000 x g per 12 min.
NOTA: Non è dannoso che le bolle siano presenti nel tubo della centrifuga. - Mentre gli ovociti vengono centrifugati, preparare la piastra di bisezione.
- Realizzare il modello come mostrato nella Figura 1 sul coperchio di una capsula di Petri da 60 mm (20 μL per goccia) e coprire completamente le gocce con olio minerale.
- Utilizzando un pennarello a punta sottile, contrassegnare le linee sotto il piatto, come mostrato nella Figura 2.
- Disegna una casella attorno alla goccia della pronasi e una linea tra le gocce CB / T20 e la fila inferiore di gocce T20.
- Coprire il piatto con un coperchio opaco fino a quando la centrifugazione è completa per evitare cambiamenti di osmolarità delle microgocce.
- Non appena la centrifugazione è completa, utilizzare una pipetta da 200 μL per raccogliere gli ovociti all'interno della soluzione e spostarli in una porzione vuota di una nuova piastra di ricerca con quattro gocce HSOF da 400 μL.
- Raccogliere e lavare gli ovociti attraverso le quattro gocce HSOF.
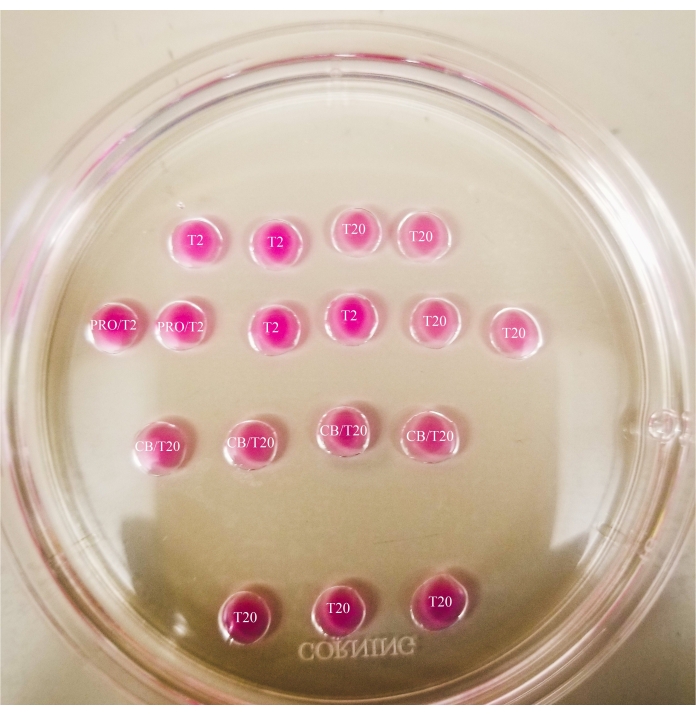
Figura 1: Piastra di bisezione. Tutte le gocce mostrate hanno un volume di 20 μL. La piastra ha un diametro di 60 mm. Le gocce sono state completamente ricoperte di olio minerale. Gli ovociti saranno prima posizionati nella goccia T2 più in alto e più a sinistra (indicata qui con una stella). PRO/T2: 10 μL di pronasi e 10 μL di T2, combinati prima di creare microgocce. CB/T20: 1 μL di citocalasina B per 1 mL di T20, combinati prima di creare microgocce. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Piastra di bisezione marcata. Le linee sono disegnate con un marcatore a punta sottile sul fondo della piastra, per fornire riferimenti di posizione per osservazioni e trasferimenti di ovociti e ooplasti effettuati sotto il microscopio. Gli ovociti saranno prima posizionati nella goccia T2 più in alto e più a sinistra (indicata qui con una stella). Fare clic qui per visualizzare una versione più grande di questa figura.
4. Preparazione dell'ovocita per la bisezione
NOTA: Il seguente processo prevede la preparazione degli ovociti per la bisezione.
- Accendere lo stadio di riscaldamento a 37 °C.
- Utilizzare una pipetta orale per spostare gli ovociti centrifugati dalla goccia HSOF alla goccia T2 in alto a sinistra della piastra di bisezione, quindi lavare gli ovociti attraverso le successive tre gocce sulla riga superiore (T2, T20, T20).
- Deposita gli ovociti in una delle gocce di pronasi, assicurando che ci sia poco o nessun contatto tra di loro.
NOTA: La rimozione della zona pellucida può richiedere quantità di tempo variabili, ma in genere avviene tra 30-120 s con l'uso di uno stadio di riscaldamento. L'assenza di una fase di riscaldamento si estenderà questo tempo. - Osservare gli ovociti fino a quando non vi è una chiara deformazione delle zonae pellucidae (vedere Figura 3).
- Una volta che un singolo ovocita zona pellucida si è deformato, spostare quell'ovocita alla goccia T2 vicina. Ripetere come ulteriori ovociti si deformano, fino a quando tutti gli ovociti sono stati rimossi dalla pronasi e collocati nella goccia T2.
- Osservare gli ovociti fino a quando rimane solo un sottile strato della zona pellucida.
- Lavare gli ovociti attraverso le successive tre gocce all'interno della fila (T2, T20, T20), quindi depositarli in linee verticali all'interno delle gocce CB/T20 (vedere Figura 4).
- Inizia con meno ovociti nelle linee verticali e aumenta il numero di ovociti per goccia man mano che le abilità di bisezione progrediscono.
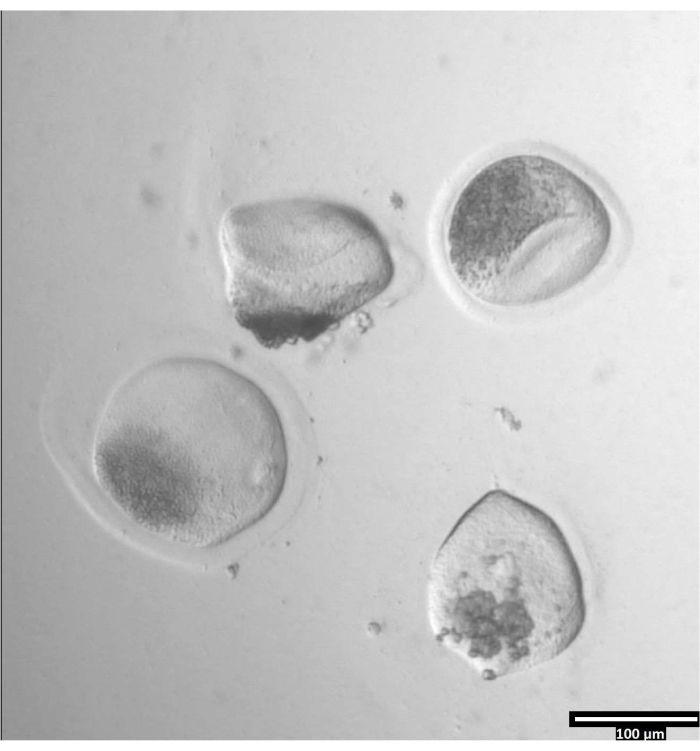
Figura 3: Rimozione della zona pellucida usando la pronasi. (80x) Una zona pellucida dell'ovocita inizierà ad apparire deformata quando la pronasi ha colpito la zona pellucida abbastanza da consentire all'ovocita di essere spostato nella goccia T2 adiacente. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Orientamento degli ovociti nelle cadute di bisezione. (80x) Gli ovociti privi di zona si depositano in un orientamento quasi verticale all'interno di ogni goccia CB/T20 prima della bisezione. Fare clic qui per visualizzare una versione più grande di questa figura.
5. Bisezione degli ovociti
- Concentrati sulla prima goccia di CB / T20 (bisezione) più a sinistra che contiene ovociti e usa una pipetta orale per ruotare gli ovociti, in modo che la porzione densa di mitocondri di ciascun ovocita sia rivolta verso o lontano dal braccio del microscopio.
NOTA: La porzione densa di mitocondri non conterrà lipidi, che appaiono come la porzione più scura dell'ovocita dopo la centrifugazione. - Appoggiare la punta della microlama a sinistra dell'ovocita più in alto, in linea con lo spazio direttamente sopra la porzione densa di mitocondri. Mantenendo la punta della lama nello stesso punto, abbassare con attenzione la lama per tagliare fino in fondo l'ovocita.
- Assicurarsi che l'ooplasto denso di mitocondri e l'ooplasto ridotto ai mitocondri siano di dimensioni simili; bisezione dovrebbe tagliare ogni ovocita a metà per produrre due ooplasti.
NOTA: Le dimensioni comparative degli ooplasti prodotti da un singolo ovocita possono variare in base agli obiettivi della ricerca; se gli ooplasti ridotti con mitocondri devono essere fusi con una cellula somatica, il loro volume deve essere maggiore degli ooplasti densi di mitocondri». - Assicurarsi che la lama stia bisecando l'ovocita sopra la porzione densa di mitocondri e sotto la porzione densa di lipidi, nel segmento più chiaro dell'ovocita centrifugato.
- Assicurarsi che l'ooplasto denso di mitocondri e l'ooplasto ridotto ai mitocondri siano di dimensioni simili; bisezione dovrebbe tagliare ogni ovocita a metà per produrre due ooplasti.
- Quando si solleva la lama, assicurarsi che venga mantenuta la stessa linea e che la punta della lama rimanga nello stesso punto, quindi sollevare delicatamente la punta dalla piastra.
- Ripetere i passaggi di bisezione per tutti gli ovociti entro la prima goccia di bisezione. Se gli ovociti sono presenti in gocce aggiuntive, orientare e dividere in due gli ovociti rimanenti.
- Usando una pipetta orale, raccogliere gli ooplasti ridotti ai mitocondri dalla prima goccia di bisezione. Posizionali nella goccia T20 sinistra situata nella fila inferiore della piastra. Ripetere l'operazione per tutte le gocce di bisezione rimanenti.
NOTA: Gli ooplasti ridotti ai mitocondri conterranno lipidi, che sono di colore più scuro rispetto al resto dell'ovocita. - Usando una pipetta orale, raccogliere ooplasti densi di mitocondri dalla prima goccia di bisezione. Posizionali nella goccia T20 di destra situata nella fila inferiore della piastra. Ripetere l'operazione per tutte le gocce di bisezione rimanenti.
NOTA: le regioni dense di mitocondri avranno un conglomerato visibile di organelli che sarà di colore grigio chiaro in apparenza. - Recupera il piatto a quattro pozzetti T10 dall'incubatrice. Usando una pipetta orale, spostare tutti gli ooplasti ridotti dai mitocondri sul ben etichettato "MR" e spostare gli ooplasti densi di mitocondri sul pozzo etichettato "M".
- Riposizionare la piastra a quattro pozzetti nell'incubatore controllato da CO2. Lasciare riposare gli ooplasti per almeno 30 minuti prima della quantificazione del mtDNA.
6. Quantificazione del mtDNA
- Utilizzare un kit di estrazione del DNA progettato per estrarre materiale da un piccolo campione per estrarre il DNA da singoli campioni (singoli ovociti, singoli ooplasti densi di mitocondri, singoli ooplasti impoveriti di mitocondri).
- Utilizzare un metodo di quantificazione del DNA di scelta per garantire che il DNA sia stato estratto con successo da ciascun campione. Se la reazione a catena quantitativa della polimerasi (qPCR) non sarà completata in quel momento, congelare il DNA estratto in un tubo etichettato in un congelatore a -80 °C.
- Eseguire qPCR
- Fare riferimento alla Tabella 2 per determinare il volume di ciascun reagente da aggiungere a ciascun tubo qPCR, insieme alle sequenze di primer. Questi primer sono progettati per amplificare la regione 12S del mtDNA bovino.
- Impostare il tempo di denaturazione iniziale per 10 minuti, seguito da 35 cicli di: 30 s di denaturazione a 94 °C, 15 s di ricottura a 60 °C e 15 s di estensione a 72 °C. Quando la reazione è completa, registrare i valori della soglia del ciclo (Ct).
NOTA: Per ottenere valori relativi del numero di copie di mtDNA, è necessario produrre una curva standard utilizzando campioni con quantità note di numeri di copie di mtDNA, che aumentano esponenzialmente. I valori di soglia del ciclo devono quindi essere manipolati utilizzando le seguenti formule per determinare i numeri di copia relativi del mtDNA. - Ottenere la concentrazione del DNA usando l'equazione seguente:
Concentrazione di DNA = (10(Ct-intercept/slope)) x volume testato - Ottenere il numero di copia utilizzando l'equazione seguente:
Numero di copia =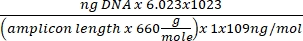
- Ottenere il numero di copia utilizzando l'equazione seguente:
Numero di copia per cella = 2 x

Figura 5: Curva standard del mtDNA delle cellule somatiche. Questa curva standard è stata creata attraverso la quantificazione del mtDNA delle concentrazioni logaritmiche delle cellule somatiche bovine utilizzando i reagenti e il programma qPCR come descritto nella fase di protocollo 6.3. Fare clic qui per visualizzare una versione più grande di questa figura.
Risultati
I risultati quantitativi della PCR (qPCR) vengono utilizzati per determinare le quantità relative di mtDNA presenti in ciascun ooplasto. La reazione descritta è progettata per amplificare la regione 12S del mtDNA bovino.
Se la bisezione ha avuto successo, i campioni di ovociti interi e ooplasti densi di mitocondri avranno valori di Ct simili. I campioni di ooplasti ridotti con mitocondri avranno valori di Ct più elevati rispetto ai campioni degli altri due gruppi. Di seguito è riportato un grafico Ct che mostra i risultati positivi della bisezione. Questi risultati indicano che la bisezione ha ridotto efficacemente il contenuto di mtDNA negli ooplasti ridotti dai mitocondri (Figura 6).
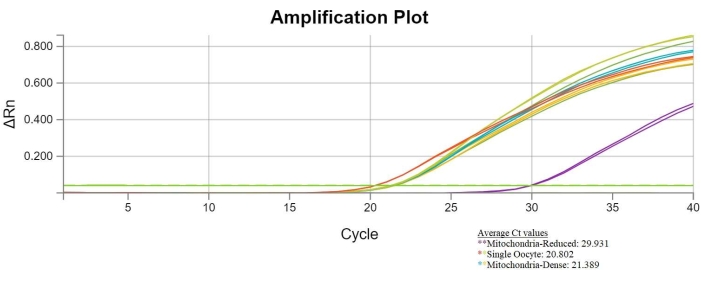
Figura 6: Risultati qPCR bisezione di successo. Il grafico che confronta il numero di ciclo e la fluorescenza mostra i valori di soglia del ciclo di singoli ovociti, ooplasti ridotti ai mitocondri (rappresentati da linee viola) e ooplasti densi di mitocondri. I valori ct degli ooplasti ridotti ai mitocondri hanno una media di 29.931 (linee viola), mentre il valore medio ct dei singoli ovociti è 20.802 (linee rosse e verdi) e il valore medio ct degli ooplasti densi di mitocondri è 21.389 (linee blu e oro). L'aumento dei valori di Ct dei campioni di ooplasto ridotti ai mitocondri indica una diminuzione del numero di copie di mtDNA all'interno di questi campioni. Fare clic qui per visualizzare una versione più grande di questa figura.
Un grafico Ct che mostra i risultati di bisezione non riusciti è mostrato nella Figura 7. Questi risultati indicano che la bisezione non ha ridotto efficacemente il contenuto di mtDNA negli ooplasti ridotti ai mitocondri:

Figura 7: Risultati qPCR di bisezione non riusciti. Il grafico a linee che confronta il numero di ciclo e la fluorescenza mostra i valori di soglia del ciclo di singoli ovociti (linee verde scuro e verde chiaro), ooplasti ridotti ai mitocondri (linee viola) e ooplasti densi di mitocondri (linee blu e oro), che hanno tutti valori ct compresi tra 20.232-20.757. Ciò indica l'assenza di un cambiamento significativo nel numero di copie di mtDNA nei campioni di ooplasto ridotti ai mitocondri. Fare clic qui per visualizzare una versione più grande di questa figura.
A seguito della produzione di una curva standard e dell'uso delle formule di numero di copie del mtDNA fornite, è stata ottenuta una riduzione media del mtDNA degli ovociti del 93,88% utilizzando questo protocollo (Figura 8).

Figura 8: Boxplot che confronta i numeri relativi di copie di DNA mitocondriale ottenute da ovociti interi, ooplasti ridotti con mitocondri e ooplasti densi di mitocondri. Ovociti interi (n = 38), ooplasti ridotti ai mitocondri (n = 34) e ooplasti densi di mitocondri (n = 29). Gli ooplasti ridotti ai mitocondri hanno un numero significativamente inferiore di copie rispetto agli altri due gruppi (P < 0,001). Fare clic qui per visualizzare una versione più grande di questa figura.
| Soluzione | Volume totale | Solvente | Soluto |
| Soluzione di ialuronidasi | 400 μL | 1 mL M-199 | 0,6 mg di ialuronidasi |
| T2 Media | 500 μL | M-199 · | 2% Siero Bovino Fetale (v/v) |
| T20 Media | 1020 μL | M-199 · | 20% Siero Fetale Bovino (v/v) |
| T10 Media | 800 μL | 400 μL T2 | 400 μL T20 |
| CB/HSOF | 500 μL | 499,5 μL di liquido oviduttale sintetico con HEPES (HSOF) | 0,5 μL di 10 mg/mL di citocalasina B (CB) |
| Soluzione Pronase | 40 μL | 1 mL M-199 | 10 mg di pronasi |
| T20/CB | 500 μL | 499,5 μL T20 | 0,5 μL di 10 mg/mL CB |
Tabella 1: Soluzioni richieste. Fornisce volumi di soluti e solventi per ogni soluzione richiesta all'interno del protocollo.
| Reagente | Volume |
| qPCR master mix | 10 μL |
| Primer in avanti | 0,6 μL |
| Primer inverso | 0,6 μL |
| Campione di DNA | 4,4 μL |
| Acqua senza DNasi | 4,4 μL |
| Volume totale | 20 μL |
| Primer in avanti GGGCTACATTCTCTACACCAAG | |
| Primer inverso GTGCTTCATGGCCTAATTCAAC | |
Tabella 2: Composizione quantitativa della PCR. Fornisce sequenze di primer avanti e indietro e volumi di tutti i reagenti necessari per ogni tubo PCR quantitativo.
Discussione
I metodi precedentemente utilizzati per ridurre il numero di copie di mtDNA negli ovociti hanno i loro rispettivi svantaggi. La rimozione basata sulla micromanipolazione dei mitocondri dagli ovociti riduce il numero di copie del mtDNA in media del 64%27. Un metodo unico, precedentemente utilizzato per l'enucleazione, prevede l'uso di pipette Pasteur di piccolo diametro e la scissione di un ovocita privo di zona pellucida al confine tra una microgoccia di media e l'olio minerale circostante. Insieme all'utilizzo della centrifugazione degli ovociti, questo approccio potrebbe produrre ooplasti vitali ridotti ai mitocondri28. Tuttavia, la combinazione di centrifugazione e questo metodo di scissione degli ovociti non è stata ancora segnalata per produrre embrioni. I metodi chimici di riduzione dei mitocondri (come l'esposizione degli ovociti alla 2′,3′-dideossicitidina (ddC), un inibitore della sintesi del mtDNA) hanno ridotto il numero di copie di mtDNA degli ovociti fino al 50% e richiedono che i COC siano esposti alla sostanza chimica per oltre 40 ore durante la maturazione in vitro (IVM)16,27. Sebbene l'incubazione di ovociti suini in un mezzo integrato con ddC per 40 ore non abbia presentato problemi per gli embrioni ricostruiti16, non ci sono ancora dati pubblicati riguardanti l'esposizione degli ovociti bovini a ddC durante il loro IVM di 20-22 ore o qualsiasi successivo sviluppo embrionale. Il metodo descritto in questo articolo riduce il numero relativo di copie di mtDNA di circa il 93,88%, da una media di 45.565 ± 37.169 copie (media ± SD) a 8.396 ± 13.287.
Diversi passaggi all'interno del protocollo sono fondamentali per ottenere risultati soddisfacenti in termini di riduzione efficace del numero di copie del DNA dei mitocondri (mtDNA) di un ovocita. Centrifugando gli ovociti alla velocità ottimizzata (15.000 x g) e al tempo (12 min) si creerà una frazione mitocondriale più concentrata a un polo di ciascun ovocita. Il volume corretto di Cytochalasin B/Synthetic Oviductal Fluid con soluzione di HEPES (CB/HSOF) (50 μL) probabilmente eliminerà la lisi degli ovociti dovuta alla centrifugazione. Dopo la centrifugazione, la zona pellucida (ZP) viene rimossa dall'esposizione a una soluzione di pronasi; la rimozione completa della ZP garantirà una bisezione più precisa e dovrebbe anche separare le cellule del cumulo rimanenti dagli ovociti. L'uso di uno stadio di riscaldamento sul microscopio durante la rimozione dello ZP è facoltativo, ma accelera la rimozione dello ZP di almeno 1 minuto. L'orientamento degli ovociti privi di zona pellucida (ZF) in gocce di bisezione è fondamentale per tagliarli in due in modo accurato. La frazione mitocondriale, che si trova in un polo di ciascun ovocita, deve essere chiaramente visibile e situata nella parte superiore o inferiore dell'ovocita prima della bisezione. Il polo opposto conterrà goccioline lipidiche scure. A seguito della bisezione, la selezione e la corretta classificazione degli ooplasti (come mitocondri densi o impoveriti di mitocondri) è fondamentale per ottenere una valutazione accurata della bisezione. Anche la selezione di quali ovociti della Metafase II (MII) dividere in due è importante. Garantire che gli ovociti utilizzati siano sferici e abbiano un citoplasma omogeneo probabilmente farà la differenza nel numero di copie di mtDNA contenute in ciascun ovocita e ooplasto. Quando si progettano e sintetizzano singole reazioni quantitative a catena della polimerasi (qPCR), è importante assicurarsi che in ogni reazione vengano utilizzati i primer e le concentrazioni di reagenti corretti per ottenere risultati accurati. Se viene prodotta una curva standard, dovrebbe essere creata nel modo più preciso possibile e tutte le reazioni dovrebbero essere eseguite in triplice copia per ottenere una maggiore precisione. I tempi e le temperature di ogni fase qPCR dovrebbero rimanere gli stessi per ogni corsa.
Alcuni problemi possono sorgere durante la sequela del protocollo. Quando si aggiunge olio alla piastra di bisezione, assicurarsi che le gocce siano appena coperte; troppo olio renderà l'uso delle pipette meno efficiente, mentre troppo poco porterà all'evaporazione delle gocce. Questa evaporazione causerebbe un cambiamento nell'osmolarità delle soluzioni, che può essere dannoso per gli ovociti e gli ooplasti. Se gli ovociti lisano dopo la digestione dello ZP, ciò è probabilmente dovuto a uno o entrambi i seguenti, in base all'esperienza personale e all'osservazione: gli ovociti non sono stati centrifugati per il tempo e la velocità prescritti e / o gli ovociti sono stati esposti alla pronasi per troppo tempo. Alcuni ovociti ZF potrebbero non riacquistare una forma completamente sferica all'interno delle gocce di bisezione. Tieni presente che non tutti gli ovociti ZF saranno in grado di essere posizionati in modo ottimale per la bisezione. In questo caso, ci sono tre opzioni: dare agli ovociti ZF più tempo per riacquistare una forma sferica; tenere delicatamente gli ovociti in una posizione più eretta con la microlama prima della bisezione; non tagliano in due gli ovociti che non possono essere posizionati correttamente. Determinare quali ooplasti sono densi di mitocondri o impoveriti di mitocondri dopo la bisezione influenzerà i risultati della quantificazione. Se la classificazione degli ooplasti è difficile, posizionare gli ovociti che verranno tagliati in due in modo che la frazione dei mitocondri sia sempre puntata verso la parte superiore o inferiore della goccia e/o cercare una porzione scura dell'ovocita in uno dei suoi poli (queste goccioline lipidiche sono concentrate al polo opposto rispetto alla frazione mitocondriale). A volte, gli ooplasti possono attaccarsi l'uno all'altro e / o la rientranza fatta dalla microlama nella piastra. A volte, fornire agli ooplasti più tempo per riacquistare una forma sferica aiuterà con la separazione. Altre opzioni includono picchiettare delicatamente la piastra, aspirarli delicatamente dentro e fuori dalla pipetta e / o utilizzare il bordo della pipetta per separare delicatamente gli ooplasti.
Questo metodo di riduzione del numero di copie del mtDNA ha i suoi limiti; uno si basa sulla fonte di ovociti e l'altro è dovuto alla rimozione dello ZP dall'ovocita. La fonte di ovociti (l'animale stesso, l'ovaio specifico e il follicolo specifico) influenzerà notevolmente i numeri di copie del mtDNA all'interno di quell'ovocita specifico. C'è una grande deviazione standard intorno al numero medio di copie di mtDNA di un singolo ovocita, che indica quanto può essere variabile il numero di copie. Molte reazioni di quantificazione di singoli ovociti sono necessarie per avere un robusto numero di copie standard di singolo ovocita che verrà confrontato con ooplasti e altri ovociti. La considerevole deviazione standard rende anche il confronto tra ooplast e ovocita meno assoluto, poiché il numero di copie dell'ooplasto non può essere paragonato al suo ovocita di origine.
La digestione delle zonae pellucidae dell'ovocita rende l'ovocita ZF e gli ooplasti risultanti molto più fragili di quanto non fossero prima della rimozione delle loro zonae pellucidae. Questa fragilità porta a tassi di lisi più elevati degli ooplasti e degli ovociti ZF stessi, in particolare con l'uso di una microlama: è necessario utilizzare una grande quantità di cura e precisione. Se gli ooplasti vengono utilizzati per la fusione, richiedono riposo dopo la bisezione prima che la fusione possa essere tentata. Altrimenti, molti degli ooplasti probabilmente lisceranno. Gli ooplasti impoveriti di mitocondri tendono ad essere più delicati delle loro controparti dense di mitocondri. I microtubuli dell'ovocita tendono a migrare con i mitocondri allo stesso polo dell'ovocita durante la centrifugazione, il che lascia gli ooplasti impoveriti dai mitocondri con meno componenti strutturali29. Sono necessarie ulteriori ricerche e colorazioni per determinare se altri organelli ovocitari sono influenzati negativamente dalla combinazione di centrifugazione e bisezione utilizzata per produrre ooplasti in questo protocollo.
La riduzione dei mitocondri degli ovociti ridurrebbe i mitocondri delle specie ovocitarie in un embrione iSCNT risultante, diminuendo così l'incidenza dell'eteroplasmia dei mitocondri in quell'embrione ricostruito. Le possibilità di sviluppo dell'embrione aumenterebbero comparativamente, ma probabilmente richiederebbero l'integrazione di mitocondri dalle specie di cellule donatrici per soddisfare il fabbisogno energetico durante lo sviluppo. Finora, i tentativi iSCNT che hanno utilizzato croci intergeneriche non hanno prodotto prole viva. La diminuzione dell'eteroplasmia negli embrioni iSCNT può aumentare la probabilità di gravidanze di successo. iSCNT è un metodo che potrebbe aiutare a salvare alcune delle 40.000 specie classificate come in via di estinzione30. Poiché le cellule somatiche di queste specie sono in genere più accessibili delle loro cellule germinali, iSCNT può utilizzare queste cellule somatiche, insieme agli ovociti delle specie domestiche. I tentativi intragenerici hanno avuto successo nella produzione di animali vivi 31,32,33, anche se la prole è tipicamente chimerica per il mtDNA, e trovare una fonte di ovociti intragenerici non è sempre possibile. La bisezione degli ovociti con l'obiettivo di ridurre il numero di copie di mtDNA, seguita dalla fusione di questi ooplasti con una cellula somatica interspecifica, ridurrebbe il chimerismo e l'eteroplasmia negli embrioni iSCNT. A causa di queste riduzioni, l'uso del metodo descritto consente un maggiore potenziale di nascite vive intergeneriche e la continuazione di specie in via di estinzione.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori desiderano ringraziare i loro colleghi della Utah State University, i ricercatori di Scienze Riproduttive dello Zoo di San Diego e la Dott.ssa Rebecca Krisher di Genus PLC.
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL centrifuge tubes | Fisher Scientific | 5408129 | |
| 60 mm dish | Sigma-Aldrich | D8054 | |
| Centrifuge | Eppendorf | 5424 | |
| Cytochalasin B | Sigma-Aldrich | C6762 | |
| Fetal Bovine Serum | Sigma-Aldrich | F2442 | |
| M199 Media | Sigma-Aldrich | M4530 | |
| Mineral Oil | Sigma-Aldrich | M8410 | |
| Mini Centrifuge | SCILOGEX | D1008 | |
| mtDNA Primer: Forward (12S) | GGGCTACATTCTCTACACCAAG | ||
| mtDNA Primer: Reverse (12S) | GTGCTTCATGGCCTAATTCAAC | ||
| NanoDrop Spectrophotometer | Thermo Scientific | ND2000 | |
| Opthalmic Scalpel with Aluminum Handle | PFM Medical | 207300633 | Microblade for bisection |
| Protease/pronase | Sigma-Aldrich | P5147 | |
| QIAamp DNA Micro Kit | Qiagen | 56304 | |
| QuantStudio™ 3 - 96-Well 0.2-mL | ThermoFisher | A28567 | |
| Search plate | Fisher Scientific | FB0875711A | |
| SYBR Green qPCR Master Mix | ThermoFisher | K0221 | qPCR master mix |
| Synthetic Oviductal Fluid with HEPES (HSOF) | |||
| ThermoPlate | Tokai Hit | TPi-SMZSSX | Heating stage |
Riferimenti
- Loi, P., Modlinski, J. A., Ptak, G. Interspecies somatic cell nuclear transfer: A salvage tool seeking first aid. Theriogenology. 76 (2), 217-228 (2011).
- Wani, N. A., Vettical, B. S., Hong, S. B. First cloned Bactrian camel (camelus bactrianus) calf produced by interspecies somatic cell nuclear transfer: A step towards preserving the critically endangered wild Bactrian camels. PLOS ONE. 12 (5), 0177800(2017).
- Oh, H. J., et al. Cloning endangered gray wolves (canis lupus) from somatic cells collected postmortem. Theriogenology. 70 (4), 638-647 (2008).
- Srirattana, K., et al. Full-term development of gaur-bovine interspecies somatic cell nuclear transfer embryos: Effect of trichostatin a treatment. Cellular Reprogramming. 14 (3), 248-257 (2012).
- Kwon, D. K., et al. Blastocysts derived from adult fibroblasts of a rhesus monkey (macaca mulatta) using interspecies somatic cell nuclear transfer. Zygote. 19 (3), 199-204 (2011).
- Lee, E., et al. Production of cloned sei whale (Balaenoptera borealis) embryos by interspecies somatic cell nuclear transfer using enucleated pig oocytes. Journal of Veterinary Science. 10 (4), 285(2009).
- Lorthongpanich, C., Laowtammathron, C., Chan, A. W., Kedutat-Cairns, M., Parnpai, R. Development of interspecies cloned monkey embryos reconstructed with bovine enucleated oocytes. Journal of Reproduction and Development. 54 (5), 306-313 (2008).
- Hong, S. G., et al. Production of transgenic canine embryos using interspecies somatic cell nuclear transfer. Zygote. 20 (1), 67-72 (2011).
- Stewart, J. B., Chinnery, P. F. The dynamics of mitochondrial DNA heteroplasmy: Implications for human health and disease. Nature Reviews Genetics. 16 (9), 530-542 (2015).
- Takeda, K. Mitochondrial DNA transmission and confounding mitochondrial influences in cloned cattle and pigs. Reproductive Medicine and Biology. 12 (2), 47-55 (2013).
- Lanza, R. P., et al. Cloning of an endangered species (Bos Gaurus) using interspecies nuclear transfer. Cloning. 2 (2), 79-90 (2000).
- Evans, M. J., et al. Mitochondrial DNA genotypes in nuclear transfer-derived cloned sheep. Nature Genetics. 23 (1), 90-93 (1999).
- Meirelles, F. V., et al. Complete replacement of the mitochondrial genotype in a Bos indicus calf reconstructed by nuclear transfer to a Bos taurus oocyte. Genetics. 158 (1), 351-356 (2001).
- Beyhan, Z., Iager, A. E., Cibelli, J. B. Interspecies nuclear transfer: Implications for embryonic stem cell biology. Cell Stem Cell. 1 (5), 502-512 (2007).
- Lagutina, I., Fulka, H., Lazzari, G., Galli, C. Interspecies somatic cell nuclear transfer: advancements and problems. Cellular Reprogramming. 15 (5), 374-384 (2013).
- Jiang, Y., et al. Interspecies somatic cell nuclear transfer is dependent on compatible mitochondrial DNA and reprogramming factors. PLoS ONE. 6 (4), 14805(2011).
- Chiaratti, M. R., et al. Embryo mitochondrial DNA depletion is reversed during early embryogenesis in cattle. Biology of Reproduction. 82 (1), 76-85 (2010).
- Spikings, E. C., Alderson, J., John, J. C. Regulated mitochondrial DNA replication during oocyte maturation is essential for successful porcine embryonic development. Biology of Reproduction. 76 (2), 327-335 (2007).
- Cagnone, G. L., et al. Restoration of normal embryogenesis by mitochondrial supplementation in pig oocytes exhibiting mitochondrial DNA deficiency. Scientific Reports. 6 (1), 1-15 (2016).
- Spikings, E. C., Alderson, J., John, J. C. Regulated mitochondrial DNA replication during oocyte maturation is essential for successful porcine embryonic development. Biology of Reproduction. 76 (2), 327-335 (2007).
- Ferreira, A. F., et al. Does supplementation with mitochondria improve oocyte competence? A systematic review. Reproduction. 161 (3), 269-287 (2021).
- Bhat, M. H., et al. Live birth of a pashmina goat kid after transfer of handmade cloned embryos. Journal of Reproduction and Development. , (2019).
- Tecirlioglu, R. T., et al. Birth of a cloned calf derived from a vitrified hand-made cloned embryo. Reproduction, Fertility and Development. 15 (7), 361(2003).
- Zhang, P., et al. Handmade cloned transgenic piglets expressing the nematode fat-1 gene. Cellular Reprogramming. 14 (3), 258-266 (2012).
- Zhang, P., et al. Handmade cloned transgenic sheep rich in omega-3 fatty acids. PLOS ONE. 8 (2), 55941(2013).
- Lagutina, I., et al. Somatic cell nuclear transfer in horses: Effect of oocyte morphology, embryo reconstruction method and donor cell type. Reproduction. 130 (4), 559-567 (2005).
- Chiaratti, M. R., et al. Embryo mitochondrial DNA depletion is reversed during early embryogenesis in cattle. Biology of Reproduction. 82 (1), 76-85 (2010).
- Hosseini, S. M., et al. and efficient method of manual oocyte enucleation using a pulled pasteur pipette. In Vitro Cellular and Developmental Biology - Animal. 49 (8), 569-575 (2013).
- Zampolla, T., Spikings, E., Rawson, D., Zhang, T. Cytoskeleton proteins F-actin and tubulin distribution and interaction with mitochondria in the granulosa cells surrounding stage III zebrafish (danio rerio) oocytes. Theriogenology. 76 (6), 1110-1119 (2011).
- International Union for Conservation of Nature. The IUCN Red List of Threatened Species. International Union for Conservation of Nature. , (2021).
- Berg, D. K., Li, C., Asher, G., Wells, D. N., Oback, B. Red deer cloned from antler stem cells and their differentiated progeny. Biology of Reproduction. 77 (3), 384-394 (2007).
- Gómez, M. C., et al. Birth of African wildcat cloned kittens born from domestic cats. Cloning and Stem Cells. 6 (3), 247-258 (2004).
- Lanza, R. P., et al. Cloning of an endangered species (Bos gaurus) using interspecies nuclear transfer. Cloning. 2 (2), 79-90 (2000).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon