Method Article
Un metodo robusto per la produzione su larga scala di sferoidi per applicazioni di screening e analisi ad alto contenuto
In questo articolo
Riepilogo
Questo protocollo descrive in dettaglio un metodo per la produzione di tre diversi tipi di sferoidi in un modo che li rende adatti per lo screening e l'analisi ad alto contenuto su larga scala. Inoltre, vengono presentati esempi che mostrano come possono essere analizzati a livello sferoidale e di singole cellule.
Abstract
Lo screening ad alto contenuto (HCS) e l'analisi ad alto contenuto (HCA) sono tecnologie che forniscono ai ricercatori la capacità di estrarre misurazioni fenotipiche quantitative su larga scala dalle cellule. Questo approccio si è dimostrato potente per approfondire la nostra comprensione di una vasta gamma di eventi sia fondamentali che applicati nella biologia cellulare. Ad oggi, la maggior parte delle applicazioni per questa tecnologia si è basata sull'uso di cellule cresciute in monostrati, anche se è sempre più consapevole che tali modelli non ricapitolano molte delle interazioni e dei processi che si verificano nei tessuti. Come tale, c'è stato un emergere nello sviluppo e nell'uso di assemblaggi cellulari tridimensionali (3D), come sferoidi e organoidi. Sebbene questi modelli 3D siano particolarmente potenti nel contesto della biologia del cancro e degli studi sulla somministrazione di farmaci, la loro produzione e analisi in modo riproducibile adatto a HCS e HCA presenta una serie di sfide. Il protocollo qui dettagliato descrive un metodo per la generazione di sferoidi tumorali multicellulari (MCTS) e dimostra che può essere applicato a tre diverse linee cellulari in modo compatibile con HCS e HCA. Il metodo facilita la produzione di diverse centinaia di sferoidi per pozzo, fornendo il vantaggio specifico che, se utilizzato in un regime di screening, i dati possono essere ottenuti da diverse centinaia di strutture per pozzo, tutte trattate in modo identico. Vengono inoltre forniti esempi, che descrivono in dettaglio come elaborare gli sferoidi per l'imaging a fluorescenza ad alta risoluzione e come l'HCA può estrarre caratteristiche quantitative sia a livello sferoide che da singole cellule all'interno di ciascun sferoide. Questo protocollo potrebbe essere facilmente applicato per rispondere a una vasta gamma di domande importanti nella biologia cellulare.
Introduzione
Tradizionalmente, i saggi basati su cellule sono stati eseguiti in monostrati che crescono su un substrato solido, che può effettivamente essere considerato come un ambiente bidimensionale (2D). Tuttavia, sta diventando sempre più riconosciuto che i modelli di coltura cellulare 2D mancano di rilevanza fisiologica in alcuni contesti e non possono replicare molte delle complesse interazioni che si verificano tra le cellule1. I metodi di coltura cellulare tridimensionale (3D) stanno rapidamente diventando popolari tra i ricercatori e i modelli cellulari 3D mostrano un alto potenziale per imitare meglio le condizioni fisiologiche incontrate dalle cellule nell'ambiente tissutale2. Esistono diversi tipi di assemblaggi di celle 3D che sono stati impiegati, ma i due tipi più comuni sono sferoidi e organoidi. Gli sferoidi possono essere coltivati da molte linee cellulari diverse e possono adottare varie forme e dimensioni a seconda del tipo di cellula utilizzata e del loro metodo di assemblaggio3. Inoltre, gli sferoidi possono anche essere indicati come sferoidi tumorali multicellulari (MCTS) quando sono cresciuti da linee cellulari tumorali, e questi modelli hanno trovato particolare uso per la somministrazione preclinica in vitro di farmaci e studi di tossicità4,5. Gli organoidi, d'altra parte, mirano a imitare meglio i tessuti e gli organi del nostro corpo e possono adottare disposizioni morfologiche più complesse. La produzione di organoidi comporta l'uso di cellule staminali adulte o cellule staminali pluripotenti, che possono essere riprogrammate nelle cellule appropriate per assomigliare al tessuto o all'organo di interesse. Sono utilizzati principalmente per studiare lo sviluppo di organi e per modellare malattie e interazioni ospite-patogeno6.
Esiste una gamma di metodi diversi utilizzati per generare assiemi di celle 3D. I metodi basati su scaffold forniscono un substrato o un supporto a cui le cellule possono attaccarsi o crescere all'interno. Queste impalcature possono avere varie forme e possono essere realizzate con una varietà di materiali diversi. I più comuni sono i componenti della matrice extracellulare (ECM) e gli idrogel, e sono progettati per assomigliare all'ambiente extracellulare naturale delle cellule e quindi facilitare le interazioni fisiologiche4,7. Il materiale basale ECM è stato estratto dal tumore del sarcoma del topo Engelbreth-Holm-Swarm e ha dimostrato di contenere una ricca miscela di componenti ECM, tra cui laminina, collagene di tipo IV e perlecan8. Tuttavia, nonostante la sua composizione vantaggiosa, ci sono due sfide principali con il suo uso, vale a dire la sua variabilità da lotto a lotto e che ha due diversi stati aggregati al di sotto e al di sopra di 10 °C8,9. Al contrario, gli idrogel hanno il vantaggio di essere flessibili rispetto ai loro componenti e alla rigidità e possono essere personalizzati per adattarsi allo specifico assemblaggio di celle 3D desiderato7,10. I metodi basati su scaffold sono essenziali per la crescita degli organoidi, ma sono anche ampiamente utilizzati per gli sferoidi. I metodi senza impalcature, che funzionano impedendo alle cellule di attaccarsi alla superficie su cui stanno crescendo, sono solitamente compatibili solo con l'assemblaggio sferoide. Gli esempi includono piastre di attacco ultra-basso (ULA), con fondo piatto o fondo a U, che consentono l'aggregazione delle cellule in sferoidi o l'uso di agitazione continua delle cellule in palloni spinner/rotazione10.
L'uso di assemblaggi cellulari 3D per studiare un'ampia varietà di eventi biologici sta rapidamente guadagnando popolarità; tuttavia, è essenziale che il metodo scelto per la loro cultura sia appropriato e compatibile con i piani per la loro analisi a valle. Ad esempio, l'uso di piastre ULA genera sferoidi di elevata consistenza; tuttavia, questo metodo è limitato alla produzione di un singolo sferoide per pozzetto, limitando così la produttività. Particolare considerazione è necessaria quando si pianifica l'imaging a fluorescenza della struttura 3D. Il substrato o la piastra su cui viene coltivato l'assemblaggio deve essere otticamente compatibile e occorre prestare attenzione per ridurre al minimo gli effetti della dispersione della luce causati da eventuali impalcature che potrebbero essere state utilizzate11. Questo particolare problema diventa più acuto all'aumentare dell'apertura numerica delle lenti degli obiettivi del microscopio.
Probabilmente uno dei motivi principali per scegliere di lavorare con un modello di cella 3D è quello di estrarre dati di imaging volumetrico non solo sull'intero assemblaggio ma anche sulle singole celle al suo interno. I modelli MCTS, in particolare, stanno iniziando a dimostrarsi molto potenti per approfondire la nostra comprensione di come le terapie transitano dall'esterno alle cellule centrali (come avrebbero bisogno in un tumore)12, e quindi acquisire conoscenze da singole cellule a diversi strati è essenziale. La tecnologia di imaging che estrae informazioni quantitative dalle singole cellule è definita analisi ad alto contenuto (HCA) ed è un approccio potente nel contesto dello screening13. Ad oggi, l'HCA è stato applicato quasi esclusivamente alle colture monostrato, ma c'è una crescente consapevolezza che questo approccio ha il potere di essere applicato alle colture 3D consentendo di studiare una vasta gamma di funzioni e processi cellulari14. Avrebbe il chiaro vantaggio che un gran numero di assiemi 3D potrebbe essere analizzato, fornendo potenzialmente dati a livello di cella da ogni struttura. Tuttavia, le sfide associate all'imaging di assemblaggi di celle potenzialmente spesse, nonché i grandi set di dati generati, devono essere superati.
In questo articolo viene presentato un robusto metodo basato su scaffold per la produzione su larga scala di MCTS in un formato a 96 pozzetti. Il metodo facilita la produzione di diverse centinaia di assemblaggi di celle 3D in ogni pozzo. Esempi sono mostrati per tre diversi tipi di cellule, che rappresentano modelli di tumore solido del fegato, del polmone e del colon. Gli sferoidi che si formano possono essere di una varietà di dimensioni, e quindi HCA viene utilizzato per selezionare strutture di una particolare dimensione e / o morfologia. Questa caratteristica fornisce l'ulteriore vantaggio che qualsiasi fenotipo osservato può essere confrontato tra sferoidi di diverse dimensioni, ma tutti trattati allo stesso modo nello stesso pozzo. Questo approccio è compatibile con l'imaging ad alta risoluzione, fornendo dati quantitativi sia a livello cellulare che subcellulare dagli stessi assemblaggi cellulari. Questo metodo di produzione di sferoidi ha l'ulteriore vantaggio rispetto ai metodi che generano un singolo sferoide per pozzo, che il gran numero di sferoidi prodotti in ciascun pozzo fornisce potenzialmente biomassa sufficiente per altre analisi a valle, come il trascrittoma e la profilazione del proteoma.
Protocollo
1. Coltura cellulare
- Preparare i supporti
- Preparare specifici terreni di coltura cellulare a seconda del tipo di linea cellulare. Assicurarsi che tutti i mezzi per il mantenimento cellulare contengano il 10% di siero bovino fetale (FBS).
NOTA: linee cellulari diverse utilizzano supporti diversi. Le cellule di carcinoma del colon HT-29 (ATCC HTB-38) sono coltivate in McCoys 5A + 10% FBS. Le cellule di carcinoma epatocellulare HepG2 (ATCC HB-8065) sono coltivate in Minimum Essential Medium + 1% L-glutammina + 10% FBS. Le cellule di carcinoma broncoalveolare H358 (ATCC CRL-5807) sono coltivate in rpmI 1640 medio + 1% L-glutammina + 10% FBS. Tutti i supporti contenenti L-glutammina e FBS sono indicati come supporti completi. Quando si placcano le cellule per crescere come sferoidi, è necessario un mezzo privo di fenolo rosso con il 10% di FBS per evitare la colorazione del materiale basale ECM, che a sua volta può causare artefatti durante il processo di imaging.
- Preparare specifici terreni di coltura cellulare a seconda del tipo di linea cellulare. Assicurarsi che tutti i mezzi per il mantenimento cellulare contengano il 10% di siero bovino fetale (FBS).
- Celle di sottocoltura
- Quando le cellule sono confluenti all'80% circa, lavare brevemente con 2 ml di tripsina-EDTA (0,25% (p / v) di tripsina / 0,53 mM EDTA). Aspirare la tripsina-EDTA, aggiungere 3 ml di tripsina-EDTA fresca e incubare per 3-5 minuti a 37 °C.
- Quando le cellule vengono staccate dal piatto di coltura cellulare, aggiungere 7 ml di terreno fresco completo.
- Pipettare 1 mL di sospensione cellulare in una nuova capsula di coltura cellulare da 10 cm e aggiungere 9 mL di mezzo fresco completo per dare una diluizione 1:10 delle cellule.
- Colturare le cellule a 37 °C in un incubatore umidificato con il 5% di CO2.
- Sottocultura le cellule ogni 3-5 giorni, a seconda della linea cellulare.
2. Generazione di sferoidi
- Rivestire piastre a 96 pozzetti con materiale seminterrato ECM
- Scongelare il materiale del seminterrato ECM sul ghiaccio durante la notte.
- Diluire il materiale basale ECM in mezzo privo di siero privo di fenolo rosso freddo utilizzando punte pre-refrigerate mantenute a -20 °C.
NOTA: La concentrazione finale di materiale basale ECM dipende dalla linea cellulare utilizzata. Per le celle HT-29 e H358, utilizzare 4 mg·mL-1 di materiale basale ECM. Per le cellule HepG2, utilizzare 4 mg·mL-1 di materiale basale ECM a fattore di crescita ridotto. - Pipettare 15 μL del materiale basale ECM/soluzione media in una piastra di imaging a 96 pozzetti.
NOTA: per la produzione di sferoidi su larga scala, questa fase può essere eseguita sia con una pipetta a 8 canali che con una testa di pipettaggio a 96 canali, purché le punte e il serbatoio contenenti il materiale seminterrato ECM siano refrigerati. - Centrifugare la piastra per 20 min a 91 x g a 4 °C.
- Incubare la piastra per non più di 30 minuti a 37 °C.
- Sottocultura e celle di conteggio
- Durante il periodo di incubazione della piastra, incubare le cellule con tripsina-EDTA e risospenderle in un mezzo completo contenente il 10% di FBS.
- Pipettare 10 μL della sospensione cellulare in una camera di conteggio emocitometrica. Contare il numero di celle in 4 quadranti della camera.
- Calcolare il numero di celle all'interno della sospensione cellulare determinando il numero medio di cellule all'interno dei 4 quadranti.
NOTA: le celle possono anche essere contate utilizzando dispositivi automatici di conteggio delle celle. - Centrifugare le celle a 135 x g per 4 minuti a temperatura ambiente (RT).
- Una volta che le cellule sono state pellettate, aspirare il surnatante e risospese le cellule in un mezzo completo privo di fenolo rosso per ottenere una concentrazione di 1 x 106 cellule per mL.
- Cellule di semi in piastre rivestite di materiale seminterrato ECM
- Diluire le cellule in un mezzo completo privo di fenoli rossi.
NOTA: la densità delle celle seminate dipende dalla linea cellulare utilizzata. Per le cellule HT-29 e HepG2, 3 x 104 cellule sono seminate per pozzetto; per le cellule H358, 4 x 104 cellule sono seminate per pozzetto. - Seminare le cellule in un volume totale di 35 μL per pozzetto. Assicurati di aggiungerli a goccia con un movimento circolare.
NOTA: per la produzione di sferoidi su larga scala, questo passaggio può essere eseguito con una pipetta a 8 canali, purché sia possibile ottenere il movimento circolare del pipettaggio. - Incubare le cellule per 1 ora a 37 °C in un incubatore umidificato con il 5% di CO2.
- Preparare una soluzione di materiale basale ECM in mezzo completo privo di fenolo rosso, che si traduce in una concentrazione finale del 2% di materiale basale ECM nel pozzo. Per raggiungere questo obiettivo, aggiungere 1,2 μL di materiale basale ECM a 23,8 μL di mezzo completo privo di fenolo rosso per pozzetto, ottenendo un volume finale di 60 μL per pozzetto.
- Incubare le cellule a 37 °C in un incubatore umidificato con il 5% di CO2.
- Il giorno seguente, sostituire il mezzo con 100 μL di mezzo completo fresco senza fenolo rosso.
- Sostituire il mezzo ogni due giorni con 100 μL di mezzo completo fresco privo di fenolo rosso.
- Diluire le cellule in un mezzo completo privo di fenoli rossi.
3. Colorazione immunofluorescenza degli sferoidi
NOTA: Questo protocollo è adattato da Nürnberg et al., 202015. Questi adattamenti si basano principalmente sul fatto che gli sferoidi descritti in questo protocollo sono più piccoli di quelli utilizzati nel lavoro di Norimberga et al.15 e, come tali, facilitano tempi di incubazione più brevi. Ad esempio, i tempi di blocco sono ridotti da 2 h a 1 h. Inoltre, poiché il protocollo è progettato per la produzione ad alto rendimento di sferoidi, tutte le fasi di lavorazione vengono eseguite nel pozzo in cui vengono coltivati gli sferoidi, il che significa che non è richiesto il trasferimento di singoli sferoidi in tubi.
- Preparare tutte le soluzioni e i tamponi necessari per il fissaggio e la colorazione
- Preparare Phosphate Buffered Saline (PBS) aggiungendo 1 compressa PBS per 200 ml di acqua. Autoclave il PBS.
- Preparare la paraformaldeide (PFA) seguendo i passaggi 3.1.3-3.1.4.
- Utilizzare un agitatore riscaldato per riscaldare 400 mL di PBS a 60-70 °C in una cappa aspirante. Aggiungere 15 g di PFA mescolando. Una volta che il PFA si è sciolto, aggiungere 50 μL di 1 M CaCl2 e 50 μL di 1 M MgCl2.
- Aggiungere 100 mL di PBS per creare un volume finale di 500 ml. Utilizzare NaOH per equilibrare il PFA a pH 7,4. Filtrare il PFA attraverso filtri sottovuoto sterili (dimensione dei pori 0,22 μm), aliquota e conservare a -20 °C.
- Preparare una soluzione madre di glicina 1 M aggiungendo 3,75 g di glicina a 50 ml di PBS. Conservare a 4 °C.
- Preparare 5 mL di tampone di permeabilizzazione aggiungendo 100 μL di Triton X-100, 1 mL di dimetilsolfossido (DMSO) e 1,5 mL di glicina 1 M a 2,4 mL di PBS. Conservare a 4 °C per non più di 2 settimane.
- Preparare 5 mL di tampone bloccante aggiungendo 100 μL di Triton X-100, 0,5 mL di DMSO e 0,05 g di albumina sierica bovina (BSA) a 4,9 mL di PBS. Aliquotare il tampone di bloccaggio in tubi da 1,5 ml e conservare a -20 °C.
- Preparare 5 mL di tampone di incubazione anticorpale aggiungendo 10 μL di polisorbato 20, 0,05 g di BSA e 250 μL di DMSO a 4,74 mL di PBS. Aliquotare il tampone di incubazione degli anticorpi in tubi da 1,5 ml e conservarlo a -20 °C.
NOTA: Nel protocollo qui descritto, gli sferoidi sono assemblati solo nei 60 pozzetti interni di una piastra a 96 pozzetti, sebbene possano essere preparati in tutti i 96 pozzetti della piastra. La ragione per preparare solo gli sferoidi nei 60 pozzetti interni è che alcuni obiettivi ad alta apertura numerica non sono in grado di essere fisicamente in grado di visualizzare l'intero pozzo dei pozzetti esterni a causa della "gonna" su alcuni tipi di piastre. I volumi di cui sopra sono sufficienti per la preparazione di 60 pozzi contenenti sferoidi.
- Fissare e permeabilizzare gli sferoidi
- Rimuovere con attenzione il mezzo dagli sferoidi, assicurando che lo strato di materiale basale ECM non venga disturbato.
- Lavare gli sferoidi due volte con 70 μL di PBS per 3 minuti ogni volta.
- Fissare gli sferoidi con 70 μL di PFA al 3% per 1 ora a 37 °C.
- Lavare gli sferoidi due volte con 70 μL di PBS per 3 minuti ogni volta.
- Spegnere con 70 μL di soluzione di glicina da 0,5 M per 30 minuti a 37 °C.
- Permeabilizzare gli sferoidi utilizzando 70 μL del tampone di permeabilizzazione per 30 min a RT.
- Lavare gli sferoidi due volte con 70 μL di PBS per 3 minuti ogni volta.
- Incubare gli sferoidi in 70 μL di tampone bloccante per 1 ora a 37 °C.
NOTA: una volta fissati gli sferoidi, tutte le successive fasi di lavorazione possono essere eseguite utilizzando una pipetta a 8 canali o una testa di pipettaggio a 96 pozzetti, se disponibile. Ciò aumenta la velocità di preparazione degli sferoidi prima dell'imaging.
- Immunostain sferoidi utilizzando anticorpi primari e secondari
- Diluire l'anticorpo primario ad una diluizione appropriata nel tampone di incubazione degli anticorpi e aggiungere 70 μL a ciascun pozzetto. Incubare gli sferoidi con l'anticorpo primario durante la notte.
NOTA: La diluizione dipende dall'anticorpo specifico utilizzato. Nell'esempio mostrato qui, i lisosomi sono immunocolorati con anticorpi anti-LAMP1 di topo usando una diluizione di 1:500. - Il giorno seguente, lavare gli sferoidi 5 volte con 70 μL di PBS per 3 minuti ogni volta.
- Diluire l'anticorpo secondario a una diluizione di 1:500, falloidina coniugata fluorescentemente a 1:500 e Hoechst 33342 (da uno stock di 1 mg mL-1 ) a 1:5000 nel tampone di incubazione degli anticorpi e aggiungere 70 μL a ciascun pozzetto. Incubare durante la notte.
NOTA: La diluizione anticorpale dipende dall'anticorpo specifico utilizzato. Si raccomandano anticorpi secondari altamente adsorbiti incrociati che mostrano un'elevata fotostabilità e un'elevata luminosità. - Il giorno seguente, lavare gli sferoidi 5 volte con 70 μL di PBS per 3 minuti ogni volta.
NOTA: le piastre possono essere conservate per un massimo di due settimane a RT prima dell'imaging, purché gli sferoidi rimangano coperti con PBS.
- Diluire l'anticorpo primario ad una diluizione appropriata nel tampone di incubazione degli anticorpi e aggiungere 70 μL a ciascun pozzetto. Incubare gli sferoidi con l'anticorpo primario durante la notte.
4. Acquisizione di immagini e analisi di sferoidi
- Acquisizione di immagini
- Inserire la piastra a 96 pozzetti contenente sferoidi in un microscopio di screening confocale ad alto contenuto.
- Selezionare i laser appropriati per eccitare i fluorofori utilizzati.
- Acquisire i canali in sequenza per evitare il cross-talk.
- Immagina gli sferoidi usando obiettivi appropriati.
NOTA: L'uso di obiettivi di immersione in acqua è raccomandato se disponibile. Ad esempio, un obiettivo 20x/1.0 NA può visualizzare un intero pozzo in ~ 77 campi visivi. Un obiettivo 63x/1,15 NA può visualizzare un intero pozzo in circa 826 campi visivi. - Immagine ~ 30-40 fette, a seconda della profondità dello sferoide, con ogni fetta presa ad un intervallo di 1,5 μm da quella precedente.
- Analisi delle immagini
- Per l'analisi morfologica degli sferoidi, seguire i passaggi 4.2.2-4.2.6.
- Segmentare gli sferoidi in base alla colorazione di falloidina coniugata fluorescentemente.
- Segmentare i nuclei in base alla colorazione Hoechst 33342.
- Segmentare il citoplasma di ciascuna cellula dalla colorazione residua di Hoechst 33342 presente nel citoplasma cellulare.
- Calcola diverse proprietà morfologiche degli sferoidi, tra cui volume, superficie, sfericità e numero di nuclei per sferoide.
NOTA: queste informazioni vengono estratte utilizzando vari algoritmi integrati nel software di analisi delle immagini preferito. - Elaborare ulteriormente le immagini per rimuovere sferoidi più piccoli di 75.000 μm3 e più grandi di 900.000 μm3.
Nota : questi valori sono suggerimenti per consentire la rimozione di assiemi di celle molto piccole e assiemi di grandi dimensioni che potrebbero essere sorti a seguito della fusione di più sferoidi. Gli sferoidi possono anche essere classificati in diverse classi di dimensioni in questa fase. - Per analizzare singole cellule all'interno degli sferoidi, seguire i passaggi 4.2.8-4.2.12.
- Segmentare gli sferoidi in base alla colorazione di falloidina coniugata fluorescentemente.
- Segmentare i nuclei in base alla colorazione Hoechst 33342.
- Segmentare il citoplasma di ciascuna cellula dalla colorazione residua di Hoechst 33342 presente nel citoplasma cellulare.
- Segmentare i lisosomi in base alla colorazione secondaria degli anticorpi.
- Calcola diverse proprietà morfologiche delle cellule all'interno di ciascun sferoide, incluso il numero di lisosomi per cellula, il volume cellulare e l'area della superficie cellulare.
NOTA: queste informazioni vengono estratte utilizzando vari algoritmi integrati nel software di analisi delle immagini preferito.
Risultati
In questo protocollo, viene dettagliato un metodo robusto per produrre assemblaggi di colture cellulari 3D sotto forma di sferoidi, utilizzando diversi tipi di cellule per rappresentare vari tessuti tumorali. Questo metodo consente la generazione di centinaia di sferoidi per pozzo, il che consente di eseguire saggi basati su cellule in modo ad alto contenuto (Figura 1). Questo approccio è stato precedentemente utilizzato per studiare l'assorbimento di nanoparticelle negli sferoidi HT-2916 e la tossicità indotta da nanoparticelle negli sferoidi HepG217. Il metodo si basa sulla produzione di una distribuzione sottile e uniforme del materiale dell'impalcatura sul fondo del pozzo di una piastra di qualità ottica. Le cellule vengono seminate in ciascun pozzetto e un ulteriore materiale di impalcatura, a bassa concentrazione, viene aggiunto come sovrapposizione alle cellule. Ciò si traduce in una base che supporta anche la crescita degli sferoidi, il che significa che diverse centinaia di sferoidi possono essere coltivati in ogni pozzetto di una piastra a 96 pozzetti. Un obiettivo di basso ingrandimento viene utilizzato per generare un'immagine panoramica dell'intera popolazione (Figura 2). Le sottoregioni del pozzo possono quindi essere selezionate e visualizzate utilizzando un obiettivo ad alta risoluzione e, in ogni posizione, viene acquisita una pila confocale completa. Questo fornisce le informazioni necessarie per la successiva analisi volumetrica di ogni sferoide.
I singoli sferoidi possono quindi essere analizzati da HCA, fornendo così informazioni sulle proprietà morfologiche degli sferoidi. In genere il primo passo è identificare ogni sferoide come un singolo oggetto. Per fare ciò, viene selezionato un canale di fluorescenza che potrebbe dare una colorazione o una distribuzione coerente in tutto lo sferoide. Nell'esempio mostrato qui, viene utilizzato il segnale nel canale della falloidina coniugato fluorescentemente, poiché l'actina si trova vicino alla membrana plasmatica in tutte le cellule nei diversi tipi di sferoidi (Figura 2 e Figura 3A). Come risultato della generazione di pile confocali complete, tutte le fasi HCA possono essere eseguite in modo volumetrico piuttosto che su base fetta per fetta. Questo è importante in quanto rimuove qualsiasi distorsione associata all'analisi di un piccolo numero di piani selezionati. Questo approccio volumetrico viene quindi applicato agli altri canali dell'immagine. I nuclei sono stati rilevati e segmentati sulla base della colorazione Hoechst 33342, e questo stesso canale può essere utilizzato per rilevare il citoplasma cellulare, in quanto vi è un residuo di Hoechst 33342 presente a bassi livelli (Figura 3B). Se è necessaria l'analisi a valle di singole cellule all'interno dello sferoide, anche gli altri canali (ad esempio, lisosomi immunocolorati con anticorpi anti-LAMP1) possono essere elaborati in modo simile. L'approccio volumetrico consente di visualizzare tutte le strutture etichettate in modo fluorescente da tutte le viste (Figura 3C).
Avendo identificato lo sferoide come un oggetto distinto, è possibile effettuare varie misurazioni morfologiche a livello sferoide. Queste misurazioni includono il calcolo del numero di nuclei per sferoide (Figura 4A), nonché la sfericità (Figura 4B), il volume (Figura 4C) e l'area superficiale (Figura 4D) dello sferoide. A seconda del software HCA utilizzato, è possibile calcolare anche altre caratteristiche morfologiche come l'area della sezione trasversale, il raggio del disco interno e il raggio della sfera interna. Negli esempi mostrati qui, gli sferoidi inferiori a 75.000 μm3 e superiori a 900.000 μm3 sono stati scartati dalle misurazioni, ma questi parametri possono essere impostati dall'utente, a seconda delle proprietà sferoidi desiderate per l'analisi. I fogli di calcolo sono stati generati dal software di analisi delle immagini e importati in RStudio per l'analisi. Sono stati prodotti grafici Boxplot per visualizzare le proprietà morfologiche dei diversi tipi di sferoidi (Figura 4). Questi boxplot rivelano il livello di eterogeneità sferoide nella popolazione, motivo per cui è importante quantificare un gran numero di sferoidi in ogni esperimento. Questo è un vantaggio chiave rispetto ai metodi che generano un singolo sferoide per pozzo. I tre tipi di sferoidi mostrati qui mostrano un alto livello di somiglianza rispetto al loro volume e alla loro superficie, anche se è notevole che la sfericità degli sferoidi HepG2 sia leggermente inferiore a quella degli altri tipi mostrati qui (Figura 4B).
L'imaging di sferoidi con un obiettivo ad alta risoluzione, come l'obiettivo di immersione in acqua 63x utilizzato qui, consente di raggiungere la risoluzione subcellulare delle singole cellule all'interno dello sferoide. I diversi organelli possono essere segmentati a seconda degli anticorpi utilizzati. Nell'esempio mostrato qui, i lisosomi sono stati identificati sulla base dell'immunocolorazione utilizzando anticorpi anti-LAMP1, seguiti dall'aggiunta di anticorpi secondari (Figura 3C). La segmentazione di ciascun organello all'interno di ciascuna cellula consente quindi la quantificazione delle misurazioni a livello cellulare. Vengono mostrati il volume tipico di ciascuna cellula nei tre tipi di sferoidi (Figura 5A), nonché il numero di lisosomi per cellula (Figura 5B). La necessità di tale dettaglio subcellulare è determinata dal test in cui verranno utilizzati gli sferoidi.
L'ottimizzazione della densità iniziale di semina cellulare all'inizio della generazione di sferoidi è un passo fondamentale. L'aggiunta di troppe cellule consente ancora la formazione di sferoidi; tuttavia, ciò può comportare la fusione di sferoidi (Figura 6). Ciò porta alla generazione di assemblaggi di forma irregolare, che a loro volta possono essere altamente problematici per l'identificazione a valle degli sferoidi. Quando gli sferoidi sono erroneamente identificati come strutture distinte, ci possono anche essere problemi con l'analisi delle singole cellule. Pertanto, un'attenta ottimizzazione di ogni linea cellulare utilizzata per la generazione di sferoidi, compresa la densità di semina cellulare e la durata del tempo di crescita, sono parametri critici da stabilire.

Figura 1: Schema che descrive in dettaglio il protocollo utilizzato per generare sferoidi per HCS e HCA. Le piastre a 96 pozzetti sono rivestite con uno strato di materiale basale ECM, seguito dalla semina delle cellule, che quindi avviano la formazione di sferoidi. Medium viene sostituito il giorno seguente e successivamente ogni secondo giorno. In preparazione per l'imaging, gli sferoidi sono fissi, permeabilizzati e immunocolorati con anticorpi specifici. Gli sferoidi vengono quindi ripresi utilizzando un microscopio HCS confocale. Grafica creata con BioRender.com. Fare clic qui per visualizzare una versione più grande di questa figura.
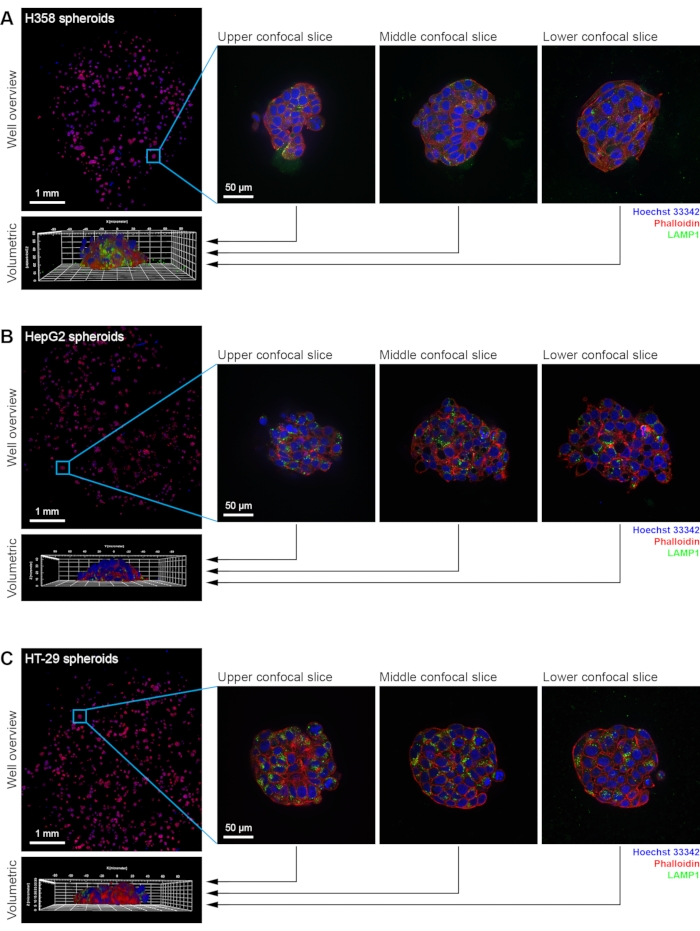
Figura 2: Immagini di esempio di sferoidi H358, HepG2 e HT-29. Imaging confocale HCS completamente automatizzato di (A) H358, (B) HepG2 e (C) HT-29 sferoidi. I pannelli mostrano una singola panoramica del pozzo, oltre a tre fette confocali di esempio e la ricostruzione volumetrica di uno sferoide da ciascuna linea cellulare. I nuclei colorati con Hoechst 33342 sono mostrati in blu, l'actina colorata con falloidina coniugata fluorescente in rosso e i lisosomi immunocolorati per LAMP1 in verde. Le immagini della panoramica del pozzo sono state acquisite con un obiettivo di immersione in acqua 20x (NA 1.0). Le immagini dei singoli sferoidi sono state acquisite con un obiettivo di immersione in acqua 63x (NA 1.15). Fare clic qui per visualizzare una versione più grande di questa figura.
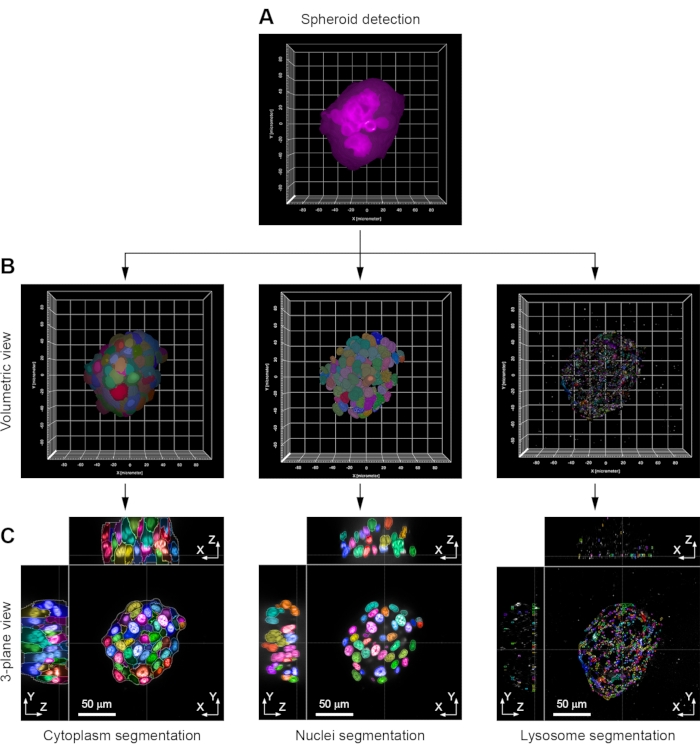
Figura 3: Esempi di passaggi chiave nell'analisi delle immagini. (A) Ogni sferoide viene identificato per la prima volta in modalità volumetrica mediante il rilevamento della colorazione di falloidina coniugata fluorescentemente. (B) Usando queste informazioni come maschera, il citoplasma di ogni cellula è segmentato dalla colorazione residua di Hoechst 33342 che è presente nel citoplasma cellulare. I nuclei sono segmentati dalla macchia Hoechst 33342. I lisosomi sono segmentati utilizzando l'immunocolorazione anticorpale (anti-LAMP1). Vengono visualizzate le viste volumetriche. (C) Viste a 3 piani dello stesso sferoide. L'esempio mostrato è di uno sferoide a cellule H358. Le immagini sono state acquisite con un obiettivo di immersione in acqua 63x (NA 1.15) Fare clic qui per visualizzare una versione più grande di questa figura.
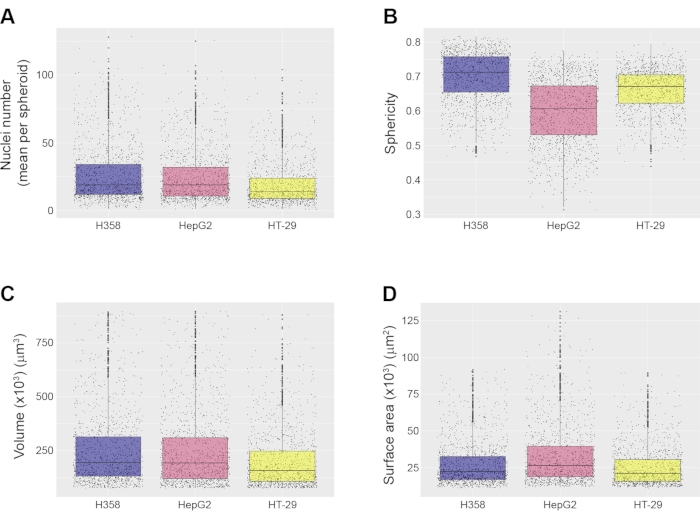
Figura 4: Esempi di misurazioni a livello sferoidale. Tutte le misurazioni sono state effettuate utilizzando una pipeline di analisi delle immagini automatizzata e per tre tipi di sferoidi, vale a dire H358, HepG2 e HT-29, ripresi con un obiettivo 20x. Sono mostrati (A) il numero medio di nuclei per sferoide, (B) la sfericità sferoide, (C) il volume sferoide e (D) l'area della superficie sferoide. Tutti i boxplot mostrano il valore mediano e i quartili per ogni tipo di sferoide. I dati provengono da 3 pozzi replicati; il numero totale di sferoidi analizzati è stato di 1259 (H358), 1522 (HepG2) e 1326 (HT-29). Le immagini sono state acquisite con un obiettivo di immersione in acqua 20x (NA 1.0). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Esempi di misurazioni a livello cellulare da sferoidi. Tutte le misurazioni sono state effettuate utilizzando una pipeline di analisi delle immagini automatizzata e per tre tipi di sferoidi, vale a dire H358, HepG2 e HT-29, ripresi con un obiettivo 63x. Sono mostrati (A) volumi di singole cellule e (B) numeri medi di lisosomi per cellula. Il numero totale di cellule analizzate è stato di 1410 (H358), 1625 (HepG2) e 1401 (HT-29), da 20 sferoidi di ogni linea cellulare. Le immagini sono state acquisite con un obiettivo di immersione in acqua 63x (NA 1.15) Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 6: Immagini di esempio che mostrano gli effetti della sovra-placcatura delle cellule sulla formazione di sferoidi. Vengono mostrate immagini di esempio per gli sferoidi H358, HepG2 e HT-29. Le frecce indicano i luoghi in cui gli sferoidi si sono probabilmente fusi. Le immagini sono state acquisite con un obiettivo di immersione in acqua 20x (NA 1.0). Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
L'approccio qui descritto descrive una piattaforma per generare diverse centinaia di sferoidi per pozzo in un modo adatto per HCS e HCA. Rispetto ad altri metodi popolari, come l'uso di piastre ULA a fondo piatto e a fondo tondo, che consentono la formazione di un solo sferoide per pozzo18,19, questo metodo offre l'opportunità di estrarre informazioni ad alta risoluzione da un gran numero di sferoidi in un formato di screening. In particolare, questo metodo è stato dimostrato in 3 diverse linee cellulari, che rappresentano vari tipi di tumore, evidenziando la sua ampia idoneità a studiare una serie di processi cellulari in diversi modelli. Sebbene gli sferoidi generati utilizzando questo metodo mostrino una certa variabilità in termini di dimensioni e forma, HCA può facilmente classificarli per dimensione (o qualsiasi altro parametro di interesse). Il nostro laboratorio ha utilizzato con successo questo approccio per studiare la tossicità indotta da nanoparticelle in funzione delle dimensioni degli sferoidi. È importante sottolineare che tutte le diverse classi di dimensioni degli sferoidi possono essere studiate nello stesso pozzo, il che significa che tutte sono state esposte a trattamenti identici, fornendo risultati altamente robusti da una grande popolazione di sferoidi17. La metodologia qui descritta potrebbe essere facilmente adattata per l'uso con altri tipi di cellule che rappresentano tessuti diversi. Inoltre, tali sferoidi potrebbero essere utilizzati per valutare diversi processi biologici14. Se gli sferoidi sono generati con cellule che esprimono stabilmente proteine marcate fluorescenti, è anche possibile l'imaging di cellule vive.
Un passo fondamentale in questo protocollo è la produzione dello strato di materiale basale ECM su cui sono placcate le celle. Quando lo strato è troppo spesso, può formare un menisco20 che favorisce la crescita di monostrati al centro del pozzo e sferoidi solo ai bordi esterni del pozzo. Pertanto, è essenziale che venga scelta la corretta concentrazione e volume del materiale basale ECM per consentire una formazione sferoide uniforme in tutto il pozzo; questo è sempre dipendente dalla linea cellulare. Da notare anche che l'eccesso di materiale seminterrato ECM può causare problemi durante il processo di immunocolorazione, in quanto non è facilmente dissolto. Questo può a sua volta portare a complicazioni durante l'acquisizione delle immagini quando si utilizza un sistema di microscopio confocale HCS. Un'altra considerazione è che diverse linee cellulari possono richiedere diverse formulazioni ECM. Ad esempio, gli sferoidi HepG2 richiedono materiale basale ECM con una concentrazione ridotta di fattori di crescita, rispetto a quello utilizzato dagli sferoidi H358 e HT-29. Anche il metodo di placcatura cellulare è importante, in quanto determina come le cellule sono distribuite nel pozzo. Quando troppe cellule vengono seminate o quando sono scarsamente distribuite, questo può portare alla fusione sferoidale (Figura 6). Un'altra sfida dell'utilizzo di scaffold ECM è che devono essere maneggiati a basse temperature (inferiori a 10 ° C), il che è particolarmente problematico quando si utilizza l'automazione per HCS. Tuttavia, questo problema è stato recentemente risolto da Eismann e colleghi (2020), che hanno impiegato la tecnologia microarray per individuare goccioline da 0,2 μL di materiale seminterrato ECM e celle in vetrini camerati. Questo era materiale sufficiente per facilitare la crescita di piccoli sferoidi21. Resta da dimostrare se un tale approccio funzionerebbe anche per la placcatura di volumi maggiori di materiale seminterrato ECM in pozzi di piastre multi-pozzo.
Un problema associato all'analisi di modelli cellulari 3D utilizzando la microscopia è la capacità di estrarre informazioni dai piani centrali di questi assiemi. A questo proposito, la permeabilizzazione e l'accesso agli anticorpi durante il processo di immunocolorazione possono essere problematici. Tuttavia, il protocollo pubblicato da Norimberga e colleghi15 consente un'immunocolorazione altamente coerente dell'intero sferoide. Utilizzando lievi modifiche a questo protocollo, questa tecnica può immunostain anche piccole strutture subcellulari (ad esempio, lisosomi), con un alto grado di consistenza, indipendentemente dal fatto che le cellule siano centrali o periferiche rispetto alla struttura sferoide complessiva. È importante che questo passaggio sia attentamente ottimizzato quando si utilizzano nuovi anticorpi.
Un altro passo critico è il regime di imaging scelto. L'imaging del numero appropriato di z-slice attraverso lo sferoide è importante, in quanto determina il volume di dati che devono essere analizzati e archiviati. Ad esempio, l'imaging di un numero limitato di piani z a un intervallo troppo grande può comportare una sottostima delle dimensioni e del volume complessivi dello sferoide. D'altra parte, l'acquisizione di troppi z-plane può comportare problemi di analisi e archiviazione dei dati. Set di dati molto grandi richiedono anche una potenza di calcolo più intensa per la loro analisi, in particolare in modalità volumetrica. L'intervallo di campionamento ottimale nel piano z è in gran parte dettato dalle caratteristiche della lente dell'obiettivo, ma per gli esempi mostrati qui, sono necessarie circa 40 fette confocali ad un intervallo di 1,5 μm per facilitare l'imaging attraverso l'intera profondità dello sferoide.
Come accennato, al fine di ridurre la distorsione dall'analisi di piani selezionati, l'uso di un approccio di analisi delle immagini basato su volumetrico è fortemente raccomandato. Ciò consente non solo di estrarre informazioni a livello di sferoide, ma anche dati a livello di cella da ogni singolo sferoide. In definitiva, i gasdotti HCA devono essere progettati in modo tale da affrontare la specifica domanda biologica posta. Gli output multiparametrici consentono di descrivere quantitativamente fenotipi complessi. Questo ha dimostrato di funzionare per assemblaggi di celle 3D utilizzando software HCA commerciale16,17, nonché software liberamente accessibile come CellProfiler22,23. I metodi di analisi volumetrica hanno il potenziale per essere applicati ad altri tipi di modelli cellulari 3D, come gli organoidi e gli espianti ex vivo derivati dal paziente (PDE), che replicano ancora meglio le condizioni in vivo. Recentemente, è stata descritta una pipeline ad alto rendimento per la produzione di organoidi cerebrali24. Ciò comporta la generazione di organoidi in una piastra a 96 pozzetti e successivamente l'imaging utilizzando un microscopio di screening ad alto contenuto24. Le PDE sono molto vantaggiose in quanto esibiscono l'istologia originale del tumore, fornendo così un modello cellulare che ricapitola le condizioni in vivo, comprese caratteristiche come il microambiente tumorale25. Questi modelli possono anche, in linea di principio, essere immunocolorati, ripresi e analizzati utilizzando l'approccio qui descritto26,27.
In sintesi, questo protocollo descrive un metodo accessibile e robusto per la produzione su larga scala di sferoidi per applicazioni HCS e HCA. La sua applicabilità è mostrata con tre diverse linee cellulari, producendo sferoidi che possono essere ripresi ad alta risoluzione. Questo protocollo dimostra anche che l'analisi volumetrica può fornire informazioni quantitative sulla popolazione sferoide, nonché a livello di singola cellula e subcellulare, rendendola così utile per un'ampia varietà di saggi.
Divulgazioni
Gli autori dichiarano che non ci sono interessi concorrenti associati a questo lavoro.
Riconoscimenti
Gli autori riconoscono il sostegno di una sovvenzione per la ricerca sulle infrastrutture da parte della Science Foundation Ireland (SFI) (16/RI/3745) a JCS. Il lavoro nel laboratorio di screening delle cellule UCD è supportato dall'UCD College of Science. ASC è finanziato da una borsa di studio post-laurea del governo irlandese del Consiglio della ricerca (IRC) (GOIPG / 2019/68). Gli autori ringraziano anche tutti i membri del laboratorio per il loro contributo e le discussioni utili. La grafica nella Figura 1 è stata generata in BioRender.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA (1x), phenol red | Gibco | 25300054 | |
| Bovine Serum Albumin (BSA) | Sigma Aldrich | A6003 | |
| Calcium chloride | Fisher Scientific | 10050070 | |
| CellCarrier-96 Ultra Microplates, tissue culture treated, black, 96-well with lid | Perkin Elmer | 6055302 | These plates have been renamed as Phenoplates |
| Dimethyl sulfoxide (DMSO) | Sigma Aldrich | D2650 | |
| Foetal Bovine Serum (FBS), qualified, EU approved, South America origin, heat inactivated | Gibco | 10500064 | |
| Glycine | Fisher Scientific | BP381-1 | |
| Goat anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Invitrogen | A-11029 | |
| L-Glutamine solution, 200 mM | Gibco | 25030024 | |
| Hoechst 33342 | Sigma Aldrich | 14533 | |
| Magnesium chloride | Fisher Scientific | 10647032 | |
| Matrigel Basement Membrane Matrix, Phenol Red-free, LDEV-free, 10 mL | Corning | 356237 | This Matrigel formulation can be also found with the same catalogue number at BD Biosciences |
| Matrigel Growth Factor Reduced Matrigel | BD Biosciences | 356231 | This Matrigel formulation can be also found with the same catalogue number at Corning |
| McCoy's 5A medium | Gibco | 26600023 | |
| McCoy's 5A medium with L glutamine and sodium bicarbonate, without phenol red | Hyclone | 10358633 | |
| Minimum Essential Medium (MEM) | Gibco | 21090022 | |
| Minimum Essential Medium (MEM), without glutamine, without phenol red | Gibco | 51200046 | |
| Mouse monoclonal anti-LAMP1 antibody (concentrate) | Developmental Studies Hybridoma Bank | H4A3-a | |
| Neubauer counting chamber | Hirschmann | 8100203 | |
| Nunclon tissue culture dish with lid, polystyrene, 92 mm x 17 mm | ThermoFisher Scientific | 150350 | |
| Opera Phenix HCS System and Harmony HCA software | Perkin Elmer | HCSHH14000000 | |
| Paraformaldehyde (PFA) | Sigma Aldrich | P6148 | |
| Phalloidin Alexa Fluor 568 | Invitrogen | A12380 | |
| Phosphate Buffered Saline (PBS) tablets | Sigma Aldrich | P4417 | |
| Polysorbate 20 | Sigma Aldrich | P5927 | |
| RPMI 1640 Medium, GlutaMAX Supplement | Gibco | 61870010 | |
| RPMI 1640 Medium, without glutamine, without phenol red | Gibco | 11835063 | |
| Triton X-100 | Sigma Aldrich | T9284 | |
| Stericup sterile vacuum filter units | Millipore | SCGVU05RE |
Riferimenti
- Jensen, C., Teng, Y. Is it time to start transitioning from 2D to 3D cell culture. Frontiers in Molecular Biosciences. 7, 33(2020).
- Langhans, S. A. Three-dimensional in vitro cell culture models in drug discovery and drug repositioning. Frontiers in Pharmacology. 9, 6(2018).
- Edmondson, R., Broglie, J. J., Adcock, A. F., Yang, L. Three-dimensional cell culture systems and their applications in drug discovery and cell-based biosensors. Assay and Drug Development Technologies. 12 (4), 207-218 (2014).
- Lv, D., Hu, Z., Lu, L., Lu, H., Xu, X. Three-dimensional cell culture: A powerful tool in tumor research and drug discovery. Oncology Letters. 14 (6), 6999-7010 (2017).
- Zhang, X., Jiang, T., Chen, D., Wang, Q., Zhang, L. W. Three-dimensional liver models: state of the art and their application for hepatotoxicity evaluation. Critical Reviews in Toxicology. 50 (4), 279-309 (2020).
- Dutta, D., Heo, I., Clevers, H. Disease modeling in stem cell-derived 3D organoid systems. Trends in Molecular Medicine. 23 (5), 393-410 (2017).
- Nunes, A. S., Barros, A. S., Costa, E. C., Moreira, A. F., Correia, I. J. 3D tumor spheroids as in vitro models to mimic in vivo human solid tumors resistance to therapeutic drugs. Biotechnology and Bioengineering. 116 (1), 206-226 (2019).
- Kleinman, H. K., Martin, G. R. Matrigel: Basement membrane matrix with biological activity. Seminars in Cancer Biology. 15, 378-386 (2005).
- Caliari, S. R., Burdick, J. A. A practical guide to hydrogels for cell culture. Nature Methods. 13 (5), 405-414 (2016).
- Foglietta, F., Canaparo, R., Muccioli, G., Terreno, E., Serpe, L. Methodological aspects and pharmacological applications of three-dimensional cancer cell cultures and organoids. Life Sciences. 254, 117784(2020).
- Bardsley, K., Deegan, A. J., El Haj, A., Yang, Y. Current state-of-the-art 3D tissue models and their compatibility with live-cell imaging. Advances in Experimental Medicine and Biology. 1035, 3-18 (2017).
- Darrigues, E., et al. Tracking gold nanorods' interaction with large 3D pancreatic-stromal tumor spheroids by multimodal imaging: Fluorescence, photoacoustic, and photothermal microscopies. Scientific Reports. 10 (1), 3362(2020).
- Boutros, M., Heigwer, F., Laufer, C. Microscopy-based high-content screening. Cell. 163 (6), 1314-1325 (2015).
- Mysior, M. M., Simpson, J. C. Cell3: A new vision for study of the endomembrane system in mammalian cells. Bioscience Reports. , (2021).
- Nürnberg, E., et al. Routine optical clearing of 3D-cell cultures: Simplicity forward. Frontiers in Molecular Biosciences. 7 (20), (2020).
- Cutrona, M. B., Simpson, J. C. A High-throughput automated confocal microscopy platform for quantitative phenotyping of nanoparticle uptake and transport in spheroids. Small. 15 (37), 1902033(2019).
- Kelly, S., Byrne, M. H., Quinn, S. J., Simpson, J. C. Multiparametric nanoparticle-induced toxicity readouts with single cell resolution in HepG2 multicellular tumour spheroids. Nanoscale. 13 (41), 17615-17628 (2021).
- Sirenko, O., Mitlo, T., Hesley, J., Luke, S., Owens, W., Cromwell, E. F. High-content assays for characterizing the viability and morphology of 3D cancer spheroid cultures. Assay and Drug Development Technologies. 13 (7), 402-414 (2015).
- Redondo-Castro, E., Cunningham, C. J., Miller, J., Cain, S. A., Allan, S. M., Pinteaux, E. Generation of human mesenchymal stem cell 3D spheroids using low-binding plates. Bio-protocol. 8 (16), (2018).
- Lee, G. Y., Kenny, P. A., Lee, E. H., Bissell, M. J. Three-dimensional culture models of normal and malignant breast epithelial cells. Nature Methods. 4 (4), 359-365 (2007).
- Eismann, B., et al. Automated 3D light-sheet screening with high spatiotemporal resolution reveals mitotic phenotypes. Journal of Cell Science. 133 (11), 245043(2020).
- Alsehli, H., et al. An integrated pipeline for high-throughput screening and profiling of spheroids using simple live image analysis of frame to frame variations. Methods. 190, 33-43 (2021).
- Stirling, D. R., et al. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 1-11 (2021).
- Renner, H., et al. A fully automated high-throughput workflow for 3D-based chemical screening in human midbrain organoids. eLife. 9, 52904(2020).
- Powley, I. R., et al. Patient-derived explants (PDEs) as a powerful preclinical platform for anti-cancer drug and biomarker discovery. British Journal of Cancer. 122 (6), 735-744 (2020).
- Collins, A., Miles, G. J., Wood, J., MacFarlane, M., Pritchard, C., Moss, E. Patient-derived explants, xenografts and organoids: 3-dimensional patient-relevant preclinical models in endometrial cancer. Gynecologic Oncology. 156 (1), 251-259 (2020).
- Miles, G. J., et al. Evaluating and comparing immunostaining and computational methods for spatial profiling of drug response in patient-derived explants. Laboratory Investigation. 101 (3), 396-407 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon