Method Article
Ricostituzione dell'attività estrattiva msp1 con componenti completamente purificati
In questo articolo
Riepilogo
Qui presentiamo un protocollo dettagliato per la ricostituzione dell'attività di estrazione di Msp1 con componenti completamente purificati in proteolipolisi definiti.
Abstract
Come centro per la fosforilazione ossidativa e la regolazione apoptotica, i mitocondri svolgono un ruolo vitale nella salute umana. La corretta funzione mitocondriale dipende da un robusto sistema di controllo della qualità per mantenere l'omeostasi proteica (proteostasi). Il declino della proteostasi mitocondriale è stato collegato al cancro, all'invecchiamento, alla neurodegenerazione e a molte altre malattie. Msp1 è un'ATPasi AAA+ ancorata nella membrana mitocondriale esterna che mantiene la proteostasi rimuovendo le proteine non localizzate ancorate alla coda. Utilizzando componenti purificati ricostituiti in proteoliposomi, abbiamo dimostrato che Msp1 è necessario e sufficiente per estrarre una proteina modello ancorata alla coda da un doppio strato lipidico. Il nostro sistema ricostituito semplificato supera molte delle barriere tecniche che hanno ostacolato lo studio dettagliato dell'estrazione delle proteine di membrana. Qui, forniamo metodi dettagliati per la generazione di liposomi, la ricostituzione delle proteine di membrana e il test di estrazione Msp1.
Introduzione
La corretta funzione cellulare dipende da un processo chiamato proteostasi, che assicura che le proteine funzionali siano alla corretta concentrazione e posizione cellulare1. I fallimenti nella proteostasi portano a compromissione della funzione degli organelli e sono associati a molte malattie neurodegenerative2,3,4. Le proteine di membrana presentano sfide uniche per la rete di proteostasi in quanto devono essere mirate alla membrana corretta evitando l'aggregazione dai domini transmembrana idrofobici (TMD)5. Di conseguenza, macchinari specializzati si sono evoluti per proteggere la TMD idrofobica dal citosol e facilitare il targeting e l'inserimento nella corretta membrana cellulare6,7,8,9,10,11, 12,13,14,15.
I mitocondri sono il fulcro metabolico della cellula e sono coinvolti in numerosi processi cellulari essenziali quali: fosforilazione ossidativa, generazione di cluster ferro-zolfo e regolazione apoptotica16,17. Questi organelli endosimbiotici contengono due membrane, denominate membrana mitocondriale interna (IMM) e membrana mitocondriale esterna (OMM). Oltre il 99% delle 1.500 proteine mitocondriali umane sono codificate nel genoma nucleare e devono essere traslocate attraverso una o due diverse membrane18,19. La corretta funzione mitocondriale dipende quindi da una robusta rete di proteostasi per correggere eventuali errori nel targeting o nella traslocazione delle proteine.
Il nostro laboratorio si concentra su un sottoinsieme di proteine di membrana mitocondriale chiamate proteine ancorate alla coda (TA), che hanno un singolo dominio transmembrana al C-terminus20,21,22,23,24. Le proteine TA sono coinvolte in una serie di processi essenziali, come l'apoptosi, il trasporto delle vescicole e la traslocazione delle proteine25. La topologia unica delle proteine TA richiede l'inserimento post-traduzionale, che avviene nel reticolo endoplasmatico (ER) da parte del Guided Entry of Tail-anchored (GET) o delle vie EMC (EMC) del reticolo endoplasmatico o nell'OMM da una via scarsamente caratterizzata20,26,27,28. Le proprietà biofisiche della TMD sono necessarie e sufficienti per guidare le proteine TA alla corretta membrana29. Il riconoscimento delle caratteristiche biofisiche piuttosto che di un motivo di sequenza definito limita la fedeltà delle vie di targeting5. Pertanto, l'errata localizzazione delle proteine TA è uno stress comune per le reti di proteostasi. Lo stress cellulare, come l'inibizione della via GET, provoca un aumento della mislocalizzazione proteica all'OMM e disfunzione mitocondriale a meno che queste proteine non vengano prontamente rimosse30,31.
Un tema comune nella proteostasi di membrana è l'uso di proteine AAA+(ATPasi Associate con attività cellulari A)per rimuovere proteine vecchie, danneggiate o mal localizzate dal doppio strato lipidico1,32,33,34,35,36,37,38 . Le proteine AAA+ sono motori molecolari che formano anelli esamerici e subiscono movimenti dipendenti dall'ATP per rimodellare un substrato, spesso per traslocazione attraverso uno stretto poro assiale39,40. Sebbene grande sforzo sia stato dedicato allo studio dell'estrazione di proteine di membrana da parte delle ATPasi AAA+, le ricostituzioni sono complesse o coinvolgono una miscela di lipidi e detergente41,42,che limita il potere sperimentale di esaminare il meccanismo di estrazione del substrato dal doppio strato lipidico.
Msp1 è un'ATPasi AAA+ altamente conservata ancorata nell'OMM e nei perossisomi che svolge un ruolo critico nella proteostasi di membrana rimuovendo le proteine TA43, 44,45,46,47,erroneamente non localizzate. Msp1 ha anche recentemente dimostrato di alleviare lo stress da importazione di proteine mitocondriali rimuovendo le proteine di membrana che si bloccano durante la traslocazione attraverso l'OMM48. La perdita di Msp1 o dell'omologo umano ATAD1 provoca frammentazione mitocondriale, fallimenti nella fosforilazione ossidativa, convulsioni, aumento delle lesioni a seguito di ictus e morte precoce31,49,50,51,52,53,54,55,56.
Abbiamo dimostrato che è possibile co-ricostituire le proteine TA con Msp1 e rilevare l'estrazione dal doppio strato lipidico57. Questo sistema semplificato utilizza proteine completamente purificate ricostituite in liposomi definiti che imitano l'OMM (Figura 1)58,59. Questo livello di controllo sperimentale può affrontare dettagliate questioni meccanicistiche di estrazione del substrato che sono sperimentalmente intrattabili con ricostituzioni più complesse che coinvolgono altre proteine AAA +. Qui, forniamo protocolli sperimentali che descrivono in dettaglio i nostri metodi per la preparazione dei liposomi, la ricostituzione delle proteine di membrana e il test di estrazione. La nostra speranza è che questi dettagli sperimentali facilitino ulteriori studi sul processo essenziale ma poco compreso della proteostasi di membrana.
Protocollo
1. Preparazione dei liposomi
- Combinare le scorte di cloroformio dei lipidi in rapporti appropriati per imitare la membrana mitocondriale esterna.
- Preparare 25 mg di miscela lipidica. Utilizziamo una miscela precedentemente stabilita di lipidi che imitano le membrane mitocondriali, costituita da un rapporto molare 48:28:10:10:4 di fosfatidilcolina (PC) uovo di gallina, fosfatidil etanolammina (PE) dell'uovo di gallina, fosfatidil-inositolo (PI) del fegato bovino, 1,2-dioleoil-sn-glicero-3-fosfo-L-serina (DOPS) e sintetico 1',3'-bis[1,2-dioleoil-sn-glicero-3-fosfo]-glicerolo (TOCL)58,59. I calcoli di esempio sono mostrati nella Tabella 1.
- Portare tutte le scorte lipidiche a temperatura ambiente prima dell'apertura in quanto ciò limiterà la condensa. Poiché la maggior parte dei laboratori non ha un modo preciso per misurare la concentrazione dei lipidi, qualsiasi acqua assorbita dallo stock di cloroformio cambierà la concentrazione del materiale lipidico e quindi il rapporto dei lipidi utilizzati nel test.
- Poiché le scorte lipidiche vengono fornite in fiale di vetro, trasferire la quantità necessaria di lipidi in una fiala di vetro utilizzando una siringa da 1 mL. Aggiungere 2 mg di ditiotreitolo (DTT) al flaconcino per prevenire l'ossidazione lipidica. Lavora rapidamente poiché l'evaporazione del cloroformio cambierà la concentrazione del lipide.
- Trasferire qualsiasi lipide rimanente in un flaconcino di vetro separato e adattarlo con un setto PFTE. Aggiungere 2 mg di DTT al flaconcino, avvolgere con parafilm e conservare a -20 °C per evitare l'ossidazione lipidica. Cerca di usare i lipidi entro 3 mesi dal trasferimento alle fiale. Per prevenire la potenziale contaminazione delle scorte di cloroformio da parte del deflusso del marcatore, trasferire gli adesivi dalle fiale originali alle fiale di vetro piuttosto che etichettare con il pennarello.
- Evaporare il cloroformio sotto un flusso molto delicato di azoto mentre si fa girare continuamente la fiala di vetro a mano in una cappa aspirante, agendo essenzialmente come un rotovap manuale. Ruotare il flaconcino a una velocità costante (20-40 giri/min) a mano per mantenere i lipidi in movimento. L'obiettivo è quello di far evaporare tutto il cloroformio e ottenere un rivestimento uniforme di lipidi su tutta la fiala di vetro.
ATTENZIONE: il cloroformio è neurotossico e questo passaggio deve essere eseguito nella cappa aspirante.- Collegare una pipetta Pasteur fresca al tubo di azoto. Non lasci che nessuno dei lipidi schizzi fuori dal flaconcino o sulla pipetta di Pasteur. Puntare la punta sul fondo della fiala in modo che l'aria rimbalzi sul fondo e spinga i lipidi verso il centro della fiala. Ottenere un rivestimento uniforme su tutta la fiala evitando qualsiasi accumulo negli angoli o dal cappuccio. L'intero processo richiede circa 5 minuti.
- Mentre la miscela si addensa in una "perla" di lipidi, guidala al centro della fiala cambiando l'angolo della fiala. Una volta che il tallone inizia a diventare più piccolo, alzare leggermente il flusso di azoto per disperdere il tallone, assicurandosi che nessuno dei lipidi esoffi fuori dalla fiala.
- Rimuovere il cloroformio rimanente sotto vuoto.
- Mettere il flaconcino di vetro su una pompa per vuoto o a membrana per 1 ora per rimuovere la maggior parte del cloroformio residuo. Questi aspirapolvere non sono generalmente abbastanza forti da rimuovere tutto il cloroformio, ma possono tollerare piccole quantità di solvente meglio dei vuoti rotativi.
- Mettere il flaconcino sotto un forte vuoto (<1 mTorr) per 12-16 ore per rimuovere tutto il cloroformio residuo. Assicurarsi di evitare di urtare il flaconcino durante questo processo.
- Sospendere i lipidi in 1,25 mL di tampone liposomico (50 mM HEPES KOH pH 7,5, 15% glicerolo, 1 mM DTT). Come abbiamo iniziato con 25 mg di lipidi, questo si traduce in una concentrazione di 20 mg / ml. Risospendare completamente i lipidi senza pezzi visibili. Se i lipidi si accumulano nell'angolo della fiala, questo può essere un processo lungo.
- Vorticare vigorosamente il flaconcino fino a ottenere un composto liscio come il latte. Scopriamo che se l'evaporazione del cloroformio è stata fatta correttamente la sera prima, questo processo richiede circa 5-10 minuti.
- Per garantire la completa risospensione dei lipidi, ruotare su una ruota a temperatura ambiente per 3 ore a ~ 80 giri / min. Rimuovere il flaconcino dalla ruota una volta ogni ora per 1 minuto di vortice per garantire una miscelazione uniforme.
- Trasferire accuratamente i lipidi in un tubo microcentrifuga pulito da 1,5 ml. Eseguire 5 cicli di congelamento-scongelamento utilizzando azoto liquido per congelare e un blocco di calore a 30 °C per scongelare. Questo passaggio aiuta a convertire le vescicole multilamellari in vescicole unilamellari.
- Estrudere i lipidi.
- Durante i cicli di congelamento del disgelo, preparare il mini-estrusore. Assemblare il mini-estrusore con supporti filtranti da 10 mm e una membrana in policarbonato della dimensione dei pori desiderata (utilizziamo 200 nm). La dimensione del filtro influenzerà la dimensione del liposoma, che influenzerà la concentrazione di proteine necessarie per la ricostituzione (Fase 2.3).
- Posizionare il miniestrusore su una piastra calda e portare la temperatura dell'estrusore fino a 60 °C.
- Aspirare i lipidi in una siringa di vetro a tenuta di gas da 1 mL e posizionarli con cura in un'estremità del miniestrusore. Posizionare la siringa vuota a tenuta di gas nell'altro lato del miniestrusore. Lasciare che i lipidi si equilibrino alla temperatura di supporto dell'estrusore per 5-10 minuti.
- Trasferire i lipidi sulla siringa alternativa premendo delicatamente lo stantuffo della siringa riempita. Spingere la soluzione dalla siringa alternativa nella siringa originale. Ripeti questo processo avanti e indietro 15 volte, in modo che al 15° passaggio i lipidi finiscano nella siringa alternativa. Monitorare il volume in ogni passaggio per assicurarsi che non vi siano perdite.
- Preparare aliquote monouso dei lipidi, congelare in azoto liquido e conservare a -80 °C. I liposomi sono stabili a -80 °C per diversi mesi. Le ricostituzioni richiedono 10 μL di liposomi alla volta (Step 2.3.4), quindi è conveniente preparare aliquote da 10 μL o 20 μL.
2 Ricostituzione di Msp1 e proteina TA modello
- Preparare il tampone di ricostituzione: 50 mM HEPES pH 7,5, 200 mM acetato di potassio, 7 mM acetato di magnesio, 2 mM DTT, 10% saccarosio, 0,01% sodio azide, 0,2-0,8% Deoxy Big Chaps (DBC).
- Ottimizzare le condizioni di ricostituzione per il nuovo lotto di liposomi. La concentrazione di DBC e biosfere necessarie per una ricostituzione ottimale varia a seconda del lotto di liposomi utilizzati. Per limitare la preparazione alla variabilità della preparazione, utilizzare lo stesso lotto di DBC per tutti gli esperimenti. Quando si modificano molti DBC, ripetere il processo di ottimizzazione.
- Impostare una serie di ricostituzioni con diverse concentrazioni di DBC (0,2% - 0,8%) e biosfere (25 mg - 100 mg) ogni volta che viene preparato un nuovo lotto di liposomi. È importante non abbassare il DBC al di sotto della concentrazione critica di micella (CMC) di ~ 0,12%. Una volta ottimizzate le condizioni, si consiglia di raccogliere tutti i dati utilizzando le stesse condizioni di preparazione e ricostituzione dei liposomi.
- Valutare l'efficacia delle varie condizioni di ricostituzione utilizzando il saggio di estrazione descritto nella Fase 3.
- Preparare le biosfere ad una concentrazione finale di 250 mg/ml.
- Pesare 2,7 g di biosfere essiccate e risospesse in un tubo centrifugo da 50 ml di metanolo al 100% (circa 45 ml) per bagnare le perline. Inizialmente bagnare le biosfere in metanolo per evitare che l'aria rimanga intrappolata nei pori delle perline. Una volta in metanolo, mantenere le biosfere bagnate poiché qualsiasi aria intrappolata dalle biosfere altererà la loro capacità di assorbire il detergente.
- Rimuovere il metanolo lavando le perline 8x con circa 45 mL di acqua ultrapura (18,2 mΩ), di seguito denominate ddH2O. Perle di pellet ruotando a 3.200 x g per 1 minuto. Decantare il liquido e risospenderlo in ddH2O.
- Dopo il lavaggio, risospendere in 10 ml di ddH2O con 0,02% di azide di sodio e conservare a 4 °C. Le biosfere possono essere conservate a 4 °C per diversi mesi. Questo stock è di 250 mg / mL in quanto si presume che ~ 0,2 g vengano persi durante le fasi di lavaggio.
- Calcola la dimensione del liposoma e il numero desiderato di molecole di proteina TA e Msp1 per liposoma. Questo determinerà la concentrazione di msp1 e proteina TA necessaria per la ricostituzione.
- Innanzitutto, calcola il numero di molecole lipidiche per liposoma unilamellare (Ntotale) usando l'equazione
 dove d è il diametro del liposoma, h è lo spessore del doppio strato e a è l'area del gruppo di testa lipidico.
dove d è il diametro del liposoma, h è lo spessore del doppio strato e a è l'area del gruppo di testa lipidico.- Misurare il diametro del liposoma in base al DLS. Nel nostro esempio, è stato ottenuto un valore di 70 nm per il diametro del liposoma (d).
- Utilizzare un valore di 5 nm per h e 0,71 nm2 per a, che è la dimensione del gruppo di testa per la fosfatidilcolina. In questa particolare situazione, iltotale N è 37.610.
- Quindi, calcolare la concentrazione molare del lipide M Lipid utilizzando il peso molecolare medio dei lipidi nella miscela. In questo esempio, la concentrazione di lipidi è di 20 mg/mL (Step 1.4) e il peso molecolare medio dei lipidi è di 810 g/mol (Tabella 1). Ciò si traduce in un valore di 0,0247 M per MLipid.
- Successivamente, calcola la concentrazione molare dei liposomi, MLiposoma, usando l'equazione
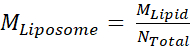 in cui M Lipid è la concentrazione molare di lipidi dal passo 2.3.2e Ntotale è il numero totale di lipidi per liposoma calcolato nel passaggio 2.3.1. In questo esempio, la concentrazione di 20 mg/mL di liposomi è di circa 660 nM.
in cui M Lipid è la concentrazione molare di lipidi dal passo 2.3.2e Ntotale è il numero totale di lipidi per liposoma calcolato nel passaggio 2.3.1. In questo esempio, la concentrazione di 20 mg/mL di liposomi è di circa 660 nM. - Calcolare la quantità di proteine Msp1 e TA necessarie per una reazione di ricostituzione da 100 μL.
NOTA: La concentrazione finale di lipidi nella ricostituzione è di 2 mg/mL, che è una diluizione 10x del ceppo liposomico. Questo dà una concentrazione finale di liposomi di 66 nM. La concentrazione finale di Msp1 è di 792 nM, che dà una media di 12 copie totali (2 esameri funzionali) per liposoma. La concentrazione finale della proteina TA è di 660 nM, che dà una media di 10 copie per liposoma.
- Innanzitutto, calcola il numero di molecole lipidiche per liposoma unilamellare (Ntotale) usando l'equazione
- In un tubo PCR, mescolare insieme Msp1 purificato, proteina TA e liposomi in tampone di ricostituzione. L'ordine di aggiunta è tampone, proteine e liposomi per ultimi. Il volume totale è di 100 μL. Lasciare riposare la miscela sul ghiaccio per 10 minuti. Purificare la proteina Msp1 e TA come descritto in precedenza57.
- Utilizzare la proteina TA modello ben caratterizzata His-Flag-Sumo-Sec22 come substrato di controllo positivo quando si stabilisce per la prima volta il test. Questo costrutto ha un His-tag per una facile purificazione, un tag 3x-Flag per il rilevamento da parte di western blot, un dominio Sumo per una maggiore solubilità e il TMD della proteina ER-TA Sec22 per la ricostituzione e il riconoscimento da parte di Msp1.
- Assicurarsi che le soluzioni madre sia di Msp1 che della proteina TA siano di circa 100 μM per ridurre al minimo l'effetto di N-Dodecyl β-D-maltoside (DDM) dalla purificazione delle proteine sulla ricostituzione. Purificato Msp1 è in 20 mM HEPES pH 7,5, 100 mM cloruro di sodio, 0,1 mM Tris (2-carbossietil) fosfina cloridrato (TCEP), 0,05% DDM mentre la proteina TA purificata è in 50 mM Tris pH 7,4, 150 mM cloruro di sodio, 10 mM cloruro di magnesio, 5 mM β-mercaptoetanolo, 10% glicerolo, 0,1% DDM. Le soluzioni stock di circa 100 μM di proteina TA e monomero Msp1 assicurano che questi componenti costituiranno < 5% del volume di ricostituzione finale, con conseguente diluizione del DDM al di sotto del CMC.
- Aggiungere la quantità desiderata di biosfere al campione per rimuovere il detergente.
- Tagliare la punta di una punta della pipetta p200 a circa1/8 di pollice di diametro in modo che le perline possano adattarsi alla punta. Ruotare accuratamente il tubo delle biosfere per ottenere una miscela uniforme e rimuovere rapidamente il coperchio e la pipetta fino al volume prima che le biosfere si depositino. Trasferire le biosfere in un tubo PCR vuoto.
- Quando la ricostituzione ha terminato la sua incubazione di 10 minuti su ghiaccio, utilizzare una punta di pipetta non tagliata per rimuovere tutto il liquido dalle biosfere. Quindi trasferire la ricostituzione da 100 μL nel tubo con le biosfere. Questo deve essere fatto rapidamente in modo che le biosfere non intrappolano l'aria, il che causerà il galleggiamento delle perline.
- Lasciare ruotare la ricostituzione su una ruota a ~80 giri/min per 16 ore a 4 °C.
- Rimuovere il materiale ricostituito dalle biosfere. Fai un giro rapido in un picofuge per pellettare le biosfere, quindi usa una punta di pipetta non tagliata per trasferire il materiale ricostituito in un tubo PCR pulito. Ripetere questo processo 1-2 volte fino a quando non rimangono biosfere nel campione. Mantenere la ricostituzione sul ghiaccio.
- Pre-cancellare il materiale ricostituito per rimuovere eventuali proteine che non sono riuscite a ricostituirsi nei liposomi.
- Preparare il tampone di estrazione: 50 mM HEPES pH 7,5, 200 mM acetato di potassio, 7 mM acetato di magnesio, 2 mM DTT, 100 nM cloruro di calcio.
- Equilibrare le colonne di spin del glutatione con il buffer di estrazione secondo le indicazioni del produttore. Ciò comporta in genere 3 cicli di lavaggio con 400 μL di tampone e quindi centrifugazione a 700 x g per 2 minuti a temperatura ambiente per rimuovere il tampone.
- Aggiungere 5 μM di ogni chaperone (GST-SGTA e GST-Calmodulin) al materiale ricostituito. Questi chaperoni si legheranno alla TMD di tutte le proteine che non sono riuscite a ricostituirsi nei liposomi. La purificazione degli accompagnatori è stata descritta in precedenza6,57.
NOTA: Questi accompagnatori sono disponibili in commercio, ma preferiamo purificare in casa per controllare costi e qualità. Entrambe le proteine sono in 20 mM Tris pH 7,5, 100 mM di cloruro di sodio e 0,1 mM TCEP e hanno una concentrazione di stock intorno a 160 μM. SGTA riconosce substrati con una TMD altamente idrofobica mentre Calmodulin lega TMD con moderata idrofobicità6,29. Insieme, questo cocktail chaperone può riconoscere una vasta gamma di substrati. - Aggiungere 100 μL di tampone di estrazione al materiale ricostituito, portando il volume fino a 200 μL. Aggiungi questo alle colonne di spin del glutatione equilibrate. Si noti che le colonne di spin del glutatione forniscono il massimo recupero del campione quando il volume di pre-clearing è 200 – 400 μL.
- Collegare le colonne di rotazione e ruotare a ~ 80 giri / min a 4 ° C per 30 minuti per consentire agli accompagnatori di legarsi alla resina.
- Ruotare le colonne a 700 x g per 2 minuti a temperatura ambiente. Il flusso attraverso è materiale pre-eliminato che è impoverito di proteine aggregate. Mantenere il materiale sul ghiaccio e procedere direttamente con il test di estrazione.
3. Saggio di estrazione
- Preparare i tubi per l'analisi SDS PAGE. Ogni reazione avrà 4 tubi: INPUT (I), FLOW THROUGH (FT), WASH (W) ed ELUTE (E).
- Aggiungere 45 μL di campione ddH2O al tubo INPUT, 40 μL di ddH2O al tubo FLOW THROUGH e 0 μL ai tubi WASH ed ELUTE.
NOTA: Il miglior rapporto segnale/rumore in questo test si ottiene quando i campioni WASH ed ELUTE sono concentrati 5x rispetto ai campioni INPUT e FLOW THROUGH. A causa delle diluizioni durante il test, ciò richiede l'assunzione di 5 μL di campione per il campione INPUT, 10 μL di campione per il campione FLOW THROUGH e 50 μL di campione per i campioni WASH ed ELUTE. - Aggiungere 16,6 μL di 4x SDS PAGE Loading Buffer a ciascun tubo. Il volume totale di ciascun campione è di 50 μL prima di SDS PAGE Loading Buffer. Il volume finale è di 66,6 μL (campione da 50 μL + 16,6 μL da 4x SDS PAGE Loading Buffer).
- Aggiungere 45 μL di campione ddH2O al tubo INPUT, 40 μL di ddH2O al tubo FLOW THROUGH e 0 μL ai tubi WASH ed ELUTE.
- Assemblare il test di estrazione.
- Preparare la reazione di estrazione contenente 60 μL di proteolipolisomi pre-eliminati, 5 μM di GST-SGTA, 5 μM di GST-Calmodulina e 2 mM di ATP. Combinare tutti i reagenti tranne ATP, che viene utilizzato per avviare la reazione. Portare ad un volume finale di 200 μL con Extraction Buffer.
NOTA: poiché per ogni test di estrazione vengono utilizzati 60 μL di campione, è possibile utilizzare una ricostituzione per tre diversi saggi di estrazione. Eseguire controlli positivi e negativi (+ATP e -ATP) su materiale proveniente dalla stessa ricostituzione. - Saggio di estrazione preriscaldato in blocco termico a 30 °C per 2 minuti.
- Preparare la reazione di estrazione contenente 60 μL di proteolipolisomi pre-eliminati, 5 μM di GST-SGTA, 5 μM di GST-Calmodulina e 2 mM di ATP. Combinare tutti i reagenti tranne ATP, che viene utilizzato per avviare la reazione. Portare ad un volume finale di 200 μL con Extraction Buffer.
- Avviare il test di estrazione aggiungendo ATP alla concentrazione finale di 2 mM e avviare il timer.
- Dare un giro di 5 secondi in un picofuge per mescolare ATP nella reazione. Reazione di incubazione a 30 °C per 30 minuti.
- Durante l'incubazione, prendere 5 μL della reazione e aggiungere al tubo INPUT. I tempi di questo sono flessibili.
- Durante questo periodo di incubazione, equilibrare una colonna di spin del glutatione per ciascun campione nel test di estrazione.
- Eseguire il pull down sugli accompagnatori per isolare il materiale estratto.
- Una volta terminata l'incubazione di 30 minuti, aggiungere 200 μl di tampone di estrazione al tubo per portare il volume totale a 400 μL. Aggiungere alla resina glutatione equilibrata e lasciare legare su ruota a 4° C per 30 minuti.
- Ruotare le colonne a 700 x g per 2 minuti a temperatura ambiente per raccogliere il flusso attraverso. Prendere 10 μL per il tubo FLOW THROUGH. Questo campione contiene substrati che sono ancora integrati nel doppio strato lipidico.
- Lavare la resina due volte con 400 μL di tampone di estrazione, scartando il flusso attraverso. Al terzo lavaggio, mantenere il flusso e prendere 50 μL per il tubo WASH.
- Preparare 5 mL di Elution Buffer aggiungendo glutatione ridotto ad una concentrazione finale di 5 mM in Extraction Buffer. Preparare questo buffer fresco ogni volta.
- Aggiungere 200 μL di Elution Buffer alla colonna di spin. Incubare a temperatura ambiente per 5 minuti. Girare a 700 x g per 2 minuti a temperatura ambiente per eluire. Mantieni il flusso attraverso. Ripetere il processo una seconda volta in modo che il volume totale di eluizione sia di 400 μL.
- Prelevare 50 μL di campione dal campione Elution e aggiungerlo alla provetta ELUTE.
- Analizza l'attività di estrazione utilizzando SDS-PAGE e western blot.
NOTA: poiché un western blot è una procedura abbastanza standard, viene fornito un protocollo di base che evidenzia alcuni dei dettagli unici di questo test.- Caricare i campioni in un gel di poliacrilammide privo di macchie (4% impilamento, 15% separazione) ed eseguire a 200 V per 50 minuti nel tampone Tris-glicina. Se lo spazio lo consente, utilizzare sia una scala non macchiata che macchiata per consentire la visualizzazione su un imager in gel privo di macchie e il trasferimento alla membrana PVDF rispettivamente. Il gel senza macchie consente la visualizzazione quantitativa delle proteine contenenti triptofano all'attivazione con la luce ultravioletta, pur consentendo al gel di essere utilizzato per un western blot.
- Immagina il gel senza macchie per confermare che c'è un carico uguale su tutti i campioni. Questo è un controllo essenziale che garantisce che eventuali cambiamenti nel segnale rilevati da Western blot non siano il risultato di un carico proteico variabile. Dovrebbero esserci solo bande visibili per gli accompagnatori (GST-calmodulina e GST-SGTA) nei campioni INPUT ed ELUTION. Ricordiamo che il campione ELUTE sarà più concentrato del campione INPUT.
- Assemblare una cassetta western blot utilizzando una membrana PVDF da 45 μm. Trasferimento a corrente costante di 300 mA per 60 minuti.
- Dopo aver bloccato la membrana, legarsi all'anticorpo primario per 16 ore a 4 °C con un leggero scuotimento ~15 rpm. Macchia per substrato con coniglio Anti-FLAG a diluizione 1:1.000.
NOTA: gli anticorpi primari e secondari utilizzati saranno specifici del substrato e potrebbe essere necessario ottimizzare la concentrazione per l'uso. - Lavare la membrana e incubare con anticorpo secondario, capra anti-coniglio a una diluizione 1:10.000, con agitazione delicata per 1 ora a temperatura ambiente.
- Lavare la membrana e l'immagine per l'analisi utilizzando l'agente di rilevamento western blotting.
Risultati
Per interpretare correttamente i risultati, il gel antimacchia e il western blot devono essere visti insieme. Il gel antimacchia garantisce un carico uguale su tutti i campioni. Quando si visualizza il gel antimacchia, gli accompagnatori (GST-calmodulina e GST-SGTA) saranno visibili nelle corsie INPUT (I) ed ELUTE (E). Ricontrollare che l'intensità di queste bande sia uniforme su tutti i campioni INPUT. Allo stesso modo, assicurarsi che l'intensità sia uniforme tra i campioni ELUTE. L'ELUTE è 5 volte più concentrato dell'INPUT e questa differenza di intensità sarà visibile nei gel.
Dopo aver utilizzato il gel antimacchia per confermare il corretto caricamento, esaminare la macchia occidentale per determinare l'attività di estrazione. Misurare l'attività di estrazione confrontando la quantità di substrato nella frazione ELUTE (E) rispetto alla frazione INPUT (I). Il segnale nel Flow Through (FT) mostra una certa variabilità, ma è generalmente simile alla frazione INPUT. Non dovrebbe esserci alcun segnale nella frazione WASH (W). In genere, c'è ~ 10% di efficienza di estrazione per il controllo positivo e 1-2% di efficienza di estrazione nel controllo negativo (Figura 2). Ricordiamo che la frazione ELUTE è 5 volte più intensa della frazione INPUT, quindi questo deve essere preso in considerazione quando si giudica l'efficienza di estrazione. Se le condizioni di ricostituzione non sono ottimizzate, in genere esistono livelli di estrazione comparabili in entrambi i campioni + ATP e - ATP (Figura 3). Questo risultato è attribuito a un fallimento di Msp1 nel ricostituirsi in modo efficiente, con conseguente numerosi proteolipolisomi senza un esamero Msp1 funzionale.
| Lipide | Mole % | MW | MW medio | μmol in 25 mg | mg in 25 mg | Cloro (mg/mL) | μL per 25 mg |
| PC | 48% | 770 | 369.6 | 14.82 | 11.41 | 25 | 456.4 |
| PE | 28% | 746 | 208.88 | 8.64 | 6.45 | 25 | 258.0 |
| PI GRECO | 10% | 902 | 90.2 | 3.09 | 2.78 | 10 | 278.5 |
| DOPS | 10% | 810 | 81 | 3.09 | 2.50 | 10 | 250.1 |
| TOCL · | 4% | 1502 | 60.08 | 1.23 | 1.85 | 25 | 74.2 |
| Corr Media MW | 809.76 | ||||||
| μmol in 25 mg | 30.87 | ||||||
Tabella 1: Calcoli del campione per la preparazione dei liposomi. Gli obiettivi principali di questa tabella sono calcolare la concentrazione corretta del peso molecolare medio della miscela lipidica e il volume di ciascuna azione lipidica necessaria per produrre i liposomi. MW e concentrazione di scorte provengono dalle etichette dei prodotti. I lipidi e la percentuale di talpa sono scelti dall'utente.

Figura 1: Saggio di estrazione del fumetto ed elenco dei passaggi chiave. Fare clic qui per visualizzare una versione più grande di questa figura.
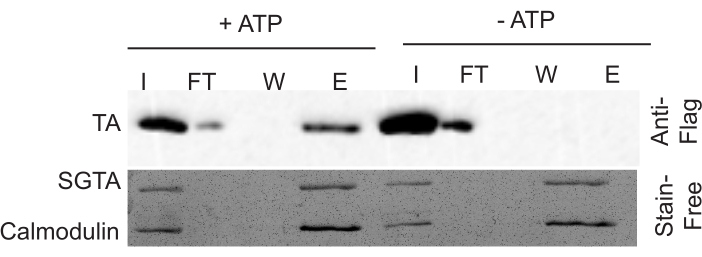
Figura 2: Dati rappresentativi che mostrano un test correttamente funzionante. L'efficienza di estrazione è determinata confrontando la quantità di substrato nella frazione ELUTE con la frazione INPUT. Ricordiamo che il gel ha un carico 5 volte superiore della frazione ELUTE rispetto alla frazione INPUT. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Dati rappresentativi di un test di ricostituzione ed estrazione fallito. Qui, l'attività nel campione + ATP è paragonabile all'attività nel campione - ATP. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
La corretta funzione mitocondriale dipende da un robusto sistema di controllo della qualità delle proteine. A causa dei limiti intrinseci nella fedeltà delle vie di targeting delle proteine TA, le proteine TA mal localizzate sono una fonte costante di stress per i mitocondri. Un componente chiave della rete di proteostasi mitocondriale è Msp1, che è una membrana ancorata AAA + ATPasi che rimuove le proteine TA mal localizzate dall'OMM. Qui, abbiamo descritto come preparare i proteoliposomi, co-ricostituire Msp1 e una proteina TA modello ed eseguire un test di estrazione. In precedenza abbiamo utilizzato questo test per dimostrare che Msp1 riconosce direttamente le proteine TA mal localizzate ed è in grado di estrarre queste proteine da un doppio strato lipidico senza proteine o cofattori accessori57.
Uno svantaggio del test è che c'è una certa variabilità nel processo di ricostituzione. Per controllare la variabilità da prep-to-prep, includiamo sempre un controllo positivo e negativo sullo stesso gel /western blot per garantire che il nostro test funzioni come previsto. Evitiamo di fare confronti tra ricostituzioni che sono state fatte in giorni diversi o di fare confronti tra diversi blots occidentali. Gli unici confronti che facciamo sono per campioni ricostituiti in parallelo ed eseguiti sullo stesso gel/western blot. Siamo anche piuttosto prudenti nella nostra interpretazione dei dati. Sebbene sia possibile quantificare l'efficienza di estrazione utilizzando ImageJ, in genere descriviamo i nostri esperimenti come aventi attività completa, attività intermedia o nessuna attività.
Una fonte di variabilità è la quantità totale di proteine ricostituite. Mentre la maggior parte delle proteine non incorporate vengono rimosse dal processo di pre-clearing, l'efficienza di ricostituzione non perfetta di Msp1 può avere un effetto fuori misura sull'efficienza osservata dell'estrazione del substrato. Questo effetto deriva dal fatto che Msp1 funziona come un omoesamero, ma purifica come un monomero57. I liposomi contenenti qualcosa di diverso da 6x copie di Msp1 saranno inattivi. Ad esempio, un liposoma con solo 5 copie di Msp1 sarà inattivo. L'unico modo per formare esami Msp1 stabili a lunghezza intera è inattivare l'attività dell'ATPasi con analoghi ATP non idrolizzabili (ATPγS) o il mutante inattivante E193Q Walker B, nessuno dei quali è compatibile con un test di attività. Superare questo ostacolo tecnico è un'area di ricerca attiva nel nostro laboratorio.
Un'altra area di ricerca attiva si concentra sul rendere il test di estrazione più quantitativo. Il metodo attuale si basa su pull down e western blotting per il rilevamento del segnale, entrambi solo semi-quantitativi e mostrano variabilità da test a saggio. La modifica covalente dei substrati estratti eliminerebbe la variabilità che deriva dai pull down. Allo stesso modo, l'uso di etichette radioattive o fluorescenti sui substrati eliminerebbe la necessità di macchie occidentali e la variabilità associata.
Uno dei principali punti di forza del test è che il sistema è completamente definito. Le proteine di membrana sono espresse e purificate in modo ricombinante ed è possibile realizzare mutanti definiti sia in Msp1 che nel substrato per studiare aspetti specifici della reazione. Il ruolo dell'ambiente lipidico nella proteostasi è stato in gran parte ignorato a causa delle sfide tecniche di studiarlo in modo dettagliato. Poiché il nostro test utilizza liposomi con una composizione lipidica definita, ciò consente un pieno controllo sperimentale dell'ambiente lipidico. Possiamo facilmente modulare fattori come: fluidità lipidica, spessore del doppio strato, identità del gruppo di testa e dimensione del liposoma. Stiamo lavorando attivamente per utilizzare il nostro test per esaminare il ruolo dell'ambiente lipidico sull'attività di Msp1. La nostra speranza è che il saggio di ricostituzione ed estrazione in vitro qui descritto possa servire come sistema modello semplificato per studiare il processo cellulare comune dell'estrazione mediata da AAA + ATPasi di proteine di membrana da un doppio strato lipidico.
Divulgazioni
Nessuno
Riconoscimenti
MLW ha sviluppato parte di questo protocollo durante i suoi studi post-dottorato con il Dr. Robert Keenan presso l'Università di Chicago.
Questo lavoro è finanziato dalla sovvenzione NIH 1R35GM137904-01 a MLW.
Materiali
| Name | Company | Catalog Number | Comments |
| Biobeads | Bio-Rad | 1523920 | |
| Bovine liver phosphatidyl inositol | Avanti | 840042C | PI |
| Chicken egg phosphatidyl choline | Avanti | 840051C | PC |
| Chicken egg phosphatidyl ethanolamine | Avanti | 840021C | PE |
| ECL Select western blotting detection reagent | GE | RPN2235 | |
| Filter supports | Avanti | 610014 | |
| Glass vial | VWR | 60910L-1 | |
| Glutathione spin column | Thermo Fisher | PI16103 | |
| Goat anti-rabbit | Thermo Fisher | NC1050917 | |
| Mini-Extruder | Avanti | 610020 | |
| Polycarbonate membrane | Avanti | 610006 | 200 nM |
| PVDF membrane | Thermo Fisher | 88518 | 45 µM |
| Rabbit anti-FLAG | Sigma-Aldrich | F7245 | |
| Synthetic 1,2-dioleoyl-sn-glycero-3-phospho-L-serine | Avanti | 840035C | DOPS |
| Synthetic 1',3'-bis[1,2-dioleoyl-sn-glycero-3-phospho]-glycerol | Avanti | 710335C | TOCL |
| Syringe, 1 mL | Norm-Ject | 53548-001 | |
| Syringe, 1 mL, gas-tight | Avanti | 610017 |
Riferimenti
- Song, J., Herrmann, J. M., Becker, T. Quality control of the mitochondrial proteome. Nature Reviews Molecular Cell Biology. 22, 54-70 (2021).
- Phillips, B. P., Miller, E. A. Membrane protein folding and quality control. Current Opinion in Structural Biology. 69, 50-54 (2021).
- Jiang, H. Quality control pathways of tail-anchored proteins. Biochimica et Biophysica Acta - Molecular Cell Research. 1868, 118922(2020).
- McKenna, M. J., et al. The endoplasmic reticulum P5A-ATPase is a transmembrane helix dislocase. Science. 369, New York, N.Y. (2020).
- Hegde, R. S., Zavodszky, E. Recognition and Degradation of Mislocalized Proteins in Health and Disease. Cold Spring Harbor Perspectives in Biology. 11, 033902(2019).
- Shao, S., Hegde, R. S. A calmodulin-dependent translocation pathway for small secretory proteins. Cell. 147, 1576-1588 (2011).
- Samuelson, J. C., et al. YidC mediates membrane protein insertion in bacteria. Nature. 406, 637-641 (2000).
- Anghel, S. A., McGilvray, P. T., Hegde, R. S., Keenan, R. J. Identification of Oxa1 Homologs Operating in the Eukaryotic Endoplasmic Reticulum. Cell Reports. 21, 3708-3716 (2017).
- Aviram, N., et al. The SND proteins constitute an alternative targeting route to the endoplasmic reticulum. Nature. 540, 134-138 (2016).
- Voorhees, R. M., Hegde, R. S. Structure of the Sec61 channel opened by a signal sequence. Science. 351, New York, NY. 88-91 (2016).
- Cichocki, B. A., Krumpe, K., Vitali, D. G., Rapaport, D. Pex19 is involved in importing dually targeted tail-anchored proteins to both mitochondria and peroxisomes. Traffic. 19, Copenhagen, Denmark. 770-785 (2018).
- Mateja, A., et al. Protein targeting. Structure of the Get3 targeting factor in complex with its membrane protein cargo. Science. 347, New York, NY. 1152-1155 (2015).
- Chacinska, A., Koehler, C. M., Milenkovic, D., Lithgow, T., Pfanner, N. Importing mitochondrial proteins: machineries and mechanisms. Cell. 138, 628-644 (2009).
- Chitwood, P. J., Hegde, R. S. An intramembrane chaperone complex facilitates membrane protein biogenesis. Nature. , (2020).
- Chitwood, P. J., Juszkiewicz, S., Guna, A., Shao, S., Hegde, R. S. EMC Is Required to Initiate Accurate Membrane Protein Topogenesis. Cell. 175, 1-30 (2018).
- Bock, F. J., Tait, S. W. G. Mitochondria as multifaceted regulators of cell death. Nature Reviews Molecular Cell Biology. 21, 85-100 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 20, (2019).
- Bykov, Y. S., Rapaport, D., Herrmann, J. M., Schuldiner, M. Cytosolic Events in the Biogenesis of Mitochondrial Proteins. Trends in Biochemical Sciences. 45, 650-667 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 427, 1135(2019).
- Borgese, N., Coy-Vergara, J., Colombo, S. F., Schwappach, B. The Ways of Tails: the GET Pathway and more. The Protein Journal. , 1-17 (2019).
- Mateja, A., Keenan, R. J. A structural perspective on tail-anchored protein biogenesis by the GET pathway. Current Opinion in Structural Biology. 51, 195-202 (2018).
- Chio, U. S., Cho, H., Shan, S. Mechanisms of Tail-Anchored Membrane Protein Targeting and Insertion. Annual review of cell and developmental biology. 33, 417-438 (2017).
- Denic, V. A portrait of the GET pathway as a surprisingly complicated young man. Trends in biochemical sciences. , (2012).
- Hegde, R. S., Keenan, R. J. Tail-anchored membrane protein insertion into the endoplasmic reticulum. Nature Reviews Molecular Cell Biology. 12, 787-798 (2011).
- Kalbfleisch, T., Cambon, A., Wattenberg, B. W. A bioinformatics approach to identifying tail-anchored proteins in the human genome. Traffic. 8, Copenhagen, Denmark. 1687-1694 (2007).
- Doan, K. N., et al. The Mitochondrial Import Complex MIM Functions as Main Translocase for α-Helical Outer Membrane Proteins. Cell Reports. 31, (2020).
- McDowell, M. A., et al. Structural Basis of Tail-Anchored Membrane Protein Biogenesis by the GET Insertase Complex. Molecular Cell. 80, (2020).
- Guna, A., Volkmar, N., Christianson, J. C., Hegde, R. S. The ER membrane protein complex is a transmembrane domain insertase. Science. 591, New York, NY. 3099(2017).
- Rao, M., et al. Multiple selection filters ensure accurate tail-anchored membrane protein targeting. eLife. 5, 21301(2016).
- Schuldiner, M., et al. The GET complex mediates insertion of tail-anchored proteins into the ER membrane. Cell. 134, 634-645 (2008).
- Chen, Y. -C., et al. Msp1/ATAD1 maintains mitochondrial function by facilitating the degradation of mislocalized tail-anchored proteins. The EMBO journal. 33, 1548-1564 (2014).
- Wu, X., Rapoport, T. A. Translocation of Proteins through a Distorted Lipid Bilayer. Trends in Cell Biology. , (2021).
- Phillips, B. P., Gomez-Navarro, N., Miller, E. A. Protein quality control in the endoplasmic reticulum. Current Opinion in Cell Biology. 65, 96-102 (2020).
- van de Weijer, M. L., et al. Quality Control of ER Membrane Proteins by the RNF185/Membralin Ubiquitin Ligase Complex. Molecular Cell. 79, (2020).
- Weir, N. R., Kamber, R. A., Martenson, J. S., Denic, V. The AAA protein Msp1 mediates clearance of excess tail-anchored proteins from the peroxisomal membrane. eLife. 6, 28507(2017).
- Gardner, B. M., et al. The peroxisomal AAA-ATPase Pex1/Pex6 unfolds substrates by processive threading. Nature communications. 9, 135(2018).
- Puchades, C., et al. Unique Structural Features of the Mitochondrial AAA+ Protease AFG3L2 Reveal the Molecular Basis for Activity in Health and Disease. Molecular Cell. , (2019).
- Castanzo, D. T., LaFrance, B., Martin, A. The AAA+ ATPase Msp1 is a processive protein translocase with robust unfoldase activity. Proceedings of the National Academy of Sciences of the United States of America. 117, 14970-14977 (2020).
- Wang, L., Myasnikov, A., Pan, X., Walter, P. Structure of the AAA protein Msp1 reveals mechanism of mislocalized membrane protein extraction. eLife. 9, (2020).
- Puchades, C., Sandate, C. R., Lander, G. C. The molecular principles governing the activity and functional diversity of AAA+ proteins. Nature Reviews Molecular Cell Biology. , 1-16 (2019).
- Yang, Y., et al. Folding-Degradation Relationship of a Membrane Protein Mediated by the Universally Conserved ATP-Dependent Protease FtsH. Journal of the American Chemical Society. , 10(2018).
- Baldridge, R. D., Rapoport, T. A. Autoubiquitination of the Hrd1 Ligase Triggers Protein Retrotranslocation in ERAD. Cell. 166, 394-407 (2016).
- Fresenius, H. L., Wohlever, M. L. Sorting out how Msp1 maintains mitochondrial membrane proteostasis. Mitochondrion. 49, 128-134 (2019).
- Wang, L., Walter, P. Msp1/ATAD1 in Protein Quality Control and Regulation of Synaptic Activities. Annual Review of Cell and Developmental Biology. 36, 1-24 (2020).
- Dederer, V., et al. Cooperation of mitochondrial and ER factors in quality control of tail-anchored proteins. eLife. 8, 1126(2019).
- Matsumoto, S., et al. Msp1 Clears Mistargeted Proteins by Facilitating Their Transfer from Mitochondria to the ER. Molecular Cell. , (2019).
- Li, L., Zheng, J., Wu, X., Jiang, H. Mitochondrial AAA-ATPase Msp1 detects mislocalized tail-anchored proteins through a dual-recognition mechanism. EMBO Reports. 20, (2019).
- Weidberg, H., Amon, A. MitoCPR - a surveillance pathway that protects mitochondria in response to protein import stress. Science. 360, New York, NY. (2018).
- Okreglak, V., Walter, P. The conserved AAA-ATPase Msp1 confers organelle specificity to tail-anchored proteins. Proceedings of the National Academy of Sciences of the United States of America. 111, (2014).
- Piard, J., et al. A homozygous ATAD1 mutation impairs postsynaptic AMPA receptor trafficking and causes a lethal encephalopathy. Brain. , (2018).
- Zhang, J., et al. The AAA+ ATPase Thorase regulates AMPA receptor-dependent synaptic plasticity and behavior. Cell. 145, 284-299 (2011).
- Prendergast, J., et al. Ganglioside regulation of AMPA receptor trafficking. The Journal of Neuroscience. 34, 13246-13258 (2014).
- Umanah, G. K. E., et al. Thorase variants are associated with defects in glutamatergic neurotransmission that can be rescued by Perampanel. Science Translational Medicine. 9, 4985(2017).
- Pignatelli, M., et al. Synaptic Plasticity onto Dopamine Neurons Shapes Fear Learning. Neuron. 93, 425-440 (2017).
- Zhang, J., et al. The AAA Thorase is neuroprotective against ischemic injury. Journal of Cerebral Blood Flow and Metabolism. , 271678(2018).
- Umanah, G. K. E., et al. AMPA Receptor Surface Expression Is Regulated by S-Nitrosylation of Thorase and Transnitrosylation of NSF. Cell Reports. 33, 108329(2020).
- Wohlever, M. L., Mateja, A., McGilvray, P. T., Day, K. J., Keenan, R. J. Msp1 Is a Membrane Protein Dislocase for Tail-Anchored Proteins. Molecular Cell. 67, 194-202 (2017).
- Lovell, J. F., et al. Membrane binding by tBid initiates an ordered series of events culminating in membrane permeabilization by Bax. Cell. 135, 1074-1084 (2008).
- Leshchiner, E. S., Braun, C. R., Bird, G. H., Walensky, L. D. Direct activation of full-length proapoptotic BAK. Proceedings of the National Academy of Sciences of the United States of America. 110, 986-995 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon